Микология и фитопатология, 2021, T. 55, № 5, стр. 353-370
Микобиота черники, произрастающей на Северо-Западе России и в Финляндии
М. М. Гомжина 1, *, Е. Л. Гасич 1, **, Т. Ю. Гагкаева 1, ***, Ф. Б. Ганнибал 1, ****
1 Всероссийский научно-исследовательский институт защиты растений
196608 Санкт-Петербург, Россия
* E-mail: gomzhina91@mail.ru
** E-mail: elena_gasich@mail.ru
*** E-mail: t.gagkaeva@mail.ru
**** E-mail: fgannibal@vizr.spb.ru
Поступила в редакцию 30.03.2021
После доработки 15.05.2021
Принята к публикации 24.05.2021
Аннотация
Черника широко распространена в хвойных, хвойно-широколиственных лесах и произрастает на территории России, Северной Европы, Азии, Северной Америки и Канады. Среди микроорганизмов, вызывающих заболевания черники, наиболее вредоносными являются Diaporthe vaccinii и Colletotrichum acutatum. Эти грибы входят в единый перечень карантинных объектов Евразийского экономического союза и их появление и распространение на территории стран, входящих в этот союз, требует строгого контроля. Корректная идентификация большинства микромицетов до таксонов уровня вида возможна только по молекулярным признакам, к таким таксонам относятся и представители родов Diaporthe и Colletotrichum. Целью данной работы была оценка биоразнообразия грибов, ассоциированных с черникой, произрастающей на территории северо-запада России и в Финляндии, с помощью морфологических и молекулярно-генетических методов. Всего проанализировано 17 образцов дикорастущей черники с некротическими пятнами на листьях и язвами на стеблях, собранных в 2017 г. на территории Санкт-Петербурга и пяти районов Ленинградской обл., а также в республике Карелия и в Финляндии. В результате идентификации выделенных из черники штаммов по морфологическим и молекулярно-генетическим признакам нами идентифицировано 12 видов: Boeremia exigua, Colletotrichum salicis, Diaporthe eres, D. pulla, Fusarium avenaceum, F. incarnatum, F. sporotrichioides, Heterophoma sylvatica, Kalmusia longispora, Microsphaeropsis olivacea, Neocucurbitaria cava, Sporocadus rosigena. Также выявлены грибы из двух секций рода Alternaria: Alternaria и Infectoriae и микромицеты из родов Chaetomium, Cladosporium, Coniothyrium, Curvularia, Epicoccum, Penicillium, Pestalotiopsis, Sordaria, Trichoderma. Впервые на территории России обнаружены виды Colletotrichum salicis, Heterophoma sylvatica, Kalmusia longispora, Microsphaeropsis olivacea и Neocucurbitaria cava. Вид Sporocadus rosigena впервые выявлен в Финляндии. Fusarium avenaceum, F. incarnatum и F. sporotrichioides впервые зарегистрированы в ассоциации с черникой. Входящие в единый перечень карантинных объектов Евразийского экономического союза виды Diaporthe vaccinii и Colletotrichum acutatum нами обнаружены не были.
ВВЕДЕНИЕ
Черника (Vaccinium myrtillus L.) – ягодный кустарничек, широко распространенный в хвойных и хвойно-широколиственных лесах. Это многолетнее растение произрастает на территории Северной Европы, Азии, Северной Америки, Канады. Среди микроорганизмов, вызывающих заболевания черники, фитопатогенные грибы и грибоподобные организмы составляют значительную часть и играют важную роль. Наиболее широко распространенными считаются микромицеты, развивающиеся на стеблях, среди них такие виды как Diaporthe vaccinii Shear, Topospora myrtilli (Feltg.) Boerema, Monilinia vaccinii-corymbosi (Reade.) Honey, Phytophthora cinnamomi Rands. и др. (Szmagara, 2009; Lombard et al., 2014; Hilário et al., 2020).
Самым вредоносным для черники является гриб Diaporthe vaccinii. Известно, что этот микромицет способен вызывать такие заболевания, как гнили, пятнистости листьев, рак стеблей и приводить к существенным потерям урожая (до 65%) ягод черники (Lombard et al., 2014). Вид D. vaccinii включен в единый перечень карантинных объектов Евразийского экономического союза в первую часть (отсутствующие на территории ЕЭС) (Eurasian economic commission, 2016).
Другой опасный и вредоносный для растений рода Vaccinium микромицет – возбудитель антракноза – гриб Colletotrichum acutatum J.H. Simmonds. Поражает этот фитопатоген не только чернику, голубику, клюкву, клубнику, вызывая гнили их ягод (Wharton, Schilder, 2008; Garrido et al., 2009; Polashock et al., 2009), но и плодовые (Lee et al., 2007) и хвойные деревья (Dingley, Gilmour, 1972). Гриб C. acutatum также входит в единый перечень карантинных объектов ЕЭС, но в его вторую часть (ограниченно распространенные на территории ЕЭС) (Eurasian economic commission, 2016).
При проведении идентификации того или иного возбудителя по симптомам на растении или по морфологическим признакам в чистой культуре, неизбежны ошибки, которые приводят к получению недостоверной информации. Известно, что корректная идентификация большинства грибов до таксонов уровня вида возможна только по молекулярным признакам. К таким грибам относятся и представители родов Diaporthe (Gomes et al., 2013; Udayanga et al., 2014; Gao et al., 2017; Santos et al., 2017) и Colletotrichum (Lee et al., 2007).
Например, длительное время считалось, что Diaporthe vaccinii, вызывающий заболевания черники, повсеместно распространен на территории Северной Америки, Канады, Европы, также сообщалось о находках на территории Китая и Чили (Naraouei-Khandan et al., 2017). Все находки были сделаны в результате идентификации этого гриба по морфологическим признакам спороносных структур или по симптомам на чернике. Однако, по морфологическим признакам D. vaccinii сходен с такими видами, как D. conorum (Desm.) Niessl, D. viticola Nitschke, D. columnaris (D. F. Farr et Castl.) Udayanga et Castl. (Farr et al., 2002b). Методы молекулярной филогении позволили заключить, что значительная часть изолятов из Северной Америки, Польши, Латвии, Нидерландов и все из Германии, которые по морфологическим признакам конидий и пикнид и по связи с питающим растением идентифицировали, как D. vaccinii, являются близкородственным видом D. eres Nitschke (Farr et al., 2002a; Vilka, Volkova, 2015; Michalecka et al., 2017). Кроме того, методы молекулярной филогении позволили заключить, что с видами рода Vaccinium, помимо видов D. eres и D. vaccinii, могут быть ассоциированы еще шесть видов этого рода: D. ambigua Nitschke, D. passiflorae Crous et L. Lombard (Elfar et al., 2013), D. asheicola L. Lombard et Crous, D. baccae L. Lombard, G. Polizzi et Crous, D. neotheicola A. J.L. Phillips et J.M. Santos, D. ster-ilis L. Lombard, G. Polizzi et Crous (Lombard et al., 2014) и D. australafricana Crous et Van Niekerk (Latorre et al., 2012; Elfar et al., 2013). Таким образом, данные о распространении этого опасного организма на территории Европы, Северной Америки, Канады и Чили требуют уточнения и ревизии. Так, на настоящий момент, в Европе достоверные находки D. vaccinii были сделаны только в Польше (Michalecka et al., 2017), Нидерландах, Латвии и Литве (Lombard et al., 2014).
Идентификация штаммов Colletotrichum acutatum, осуществляемая по морфологическим признакам, также может приводить к ошибкам в определении видовой принадлежности. Такой признак как форма конидий, имеющих веретеновидную форму и заостренные концы (Simmonds, 1965), который длительное время считался диагностическим, является нестабильным и может быть характерен и для других видов этого рода. Кроме того, конидии C. acutatum могут иметь и другую форму (Damm et al., 2012). Методы молекулярной филогении позволили заключить, что вид C. acutatum является сборным и включает в свой состав 29 видов Colletotrichum (Damm et al., 2012). Из них с кустарничками рода Vaccinium могут быть ассоциированы C. acutatum, C. fioriniae (Marcelino et Gouli) Pennycook, C. rhombiforme Damm, P.F. Cannon et Crous (Wharton, Schilder, 2008; Damm et al., 2012).
Изучение биоразнообразия грибов, вызывающих заболевания черники в России не проводилось. Опубликована информация об отдельных находках видов той или иной группы грибов, но нет результатов комплексного исследования микобиоты, сопровождаемого молекулярным анализом.
Целью данной работы была оценка биоразнообразия грибов, ассоциированных с черникой, произрастающей на территории северо-запада России и Финляндии, с помощью морфологических и молекулярно-генетических методов.
МАТЕРИАЛЫ И МЕТОДЫ
Полевой материал. В период с июня по август 2017 г. на территории Пискаревского лесопарка в г. Санкт-Петербурге, в Ленинградской обл. (Лужский, Приозерский, Ломоносовский, Всеволожский, Гатчинский р-ны), в Республике Карелия (Пудожский р-н), а также на юго-западе Финляндии было собрано 17 образцов дикорастущей черники с некротическими пятнами на листьях и язвами на стеблях (рис. 1).
Рис. 1.
Симптомы листовой и стеблевой пятнистостей черники, наиболее часто наблюдаемые на северо-западе России и в Финляндии.

Изоляты. Из каждого образца брали фрагменты растений с симптомами поражения, которые поверхностно стерилизовали 5%-м р-ром гипохлорита натрия. В дальнейшем фрагменты тканей растений раскладывали на картофельно-сахарозную питательную среду (КСА) (Samson et al., 2000) с добавлением смеси антибиотиков (ампициллин, стрептомицин, пенициллин, HyClone, Austria) и 0.4 мкл/л р-ра Triton X-100 (Panreac, Spain). Чашки Петри инкубировали при 24°C в темноте. Отсев выросших грибов проводили на 7–10-е сутки, из каждой культуры гриба получали моноспоровые или монопикнидиальные изоляты. Все 67 анализированных изолятов хранятся в коллекции лаборатории микологии и фитопатологии ВИЗР.
Морфологический анализ. Идентификация изолятов до уровня секции, рода или семейства (в отдельных случаях до уровня вида) осуществлялась по морфологическим признакам, согласно определителям (Morochkovskiy et al., 1969; Boerema et al., 2004; Simmons, 2007; Gerlach, Nirenberg, 1982).
Выделение и секвенирование ДНК. Среди выделенных изолятов 31 был выбран для изучения с применением молекулярно-генетических методов (табл. 1). Мицелий для экстракции ДНК собрали с поверхности культур грибов, выращенных на КСА. Размол осуществляли с использованием шаровой мельницы MM 400 (Retsch, Germany) и стерильного стеклянного песка (0.3 мм диам.). Экстракцию ДНК проводили стандартным (СТАB/хлороформ) методом (Doyle, Doyle, 1990).
Таблица 1.
Исследуемая коллекция штаммов грибов, выделенных из черники
| № штамма | Вид гриба | Место сбора | Орган | Номера доступа нуклеотидных последовательностей в GenBank | ||||||
|---|---|---|---|---|---|---|---|---|---|---|
| ITS | LSU | ACT | apn2 | TEF | TUB | RPB2 | ||||
| MF-Vm17-035 | Boeremia exigua | Санкт-Петербург, Пискаревский лесопарк | листья | MZ046048 | – | – | – | – | MZ054672 | MZ054693 |
| MF-Vm17-043 | Colletotrichum salicis | ” | листья | MZ046049 | – | MZ054645 | – | MZ054663 | MZ054673 | – |
| MF-Vm17-044 | C. salicis | Ленинградская обл., Лужский р-н | листья | MZ046050 | – | MZ054646 | – | MZ054664 | MZ054674 | – |
| MF-Vm17-001 | Diaporthe eres | Ленинградская обл., Всеволожский р-н | стебель | MZ046051 | – | – | MZ054647 | MZ054665 | MZ054675 | – |
| MF-Vm17-008 | ” | ” | стебель | MZ046052 | – | – | MZ054648 | MZ054666 | MZ054676 | – |
| MF-Vm17-009 | ” | Финляндия | стебель | MZ046053 | – | – | MZ054649 | MZ054667 | MZ054677 | – |
| MF-Vm17-019 | ” | Ленинградская обл., Гатчинский р-н, Меньково | листья | MZ046054 | – | – | MZ054650 | MZ054669 | MZ054678 | – |
| MF-Vm17-030 | ” | Ленинградская обл., Ломоносовский р-н, Горелово | стебель | MZ046055 | – | – | MZ054651 | MZ054670 | MZ054679 | – |
| MF-Vm17-013 | D. pulla | Ленинградская обл., Гатчинский р-н, Меньково | листья | MZ046056 | – | – | MZ054652 | MZ054668 | MZ054680 | – |
| MF-Vm17-029 | ” | Санкт-Петербург, Пискаревский лесопарк | листья | MZ046057 | – | – | MZ054653 | MZ054670 | MZ054681 | – |
| MF-G60101 | Fusarium avenaceum | Карелия, Пудожский р-н | стебель | – | – | – | – | MZ054654 | – | – |
| MF-G60102 | ” | Ленинградская обл., Лужский р-н | листья | – | – | – | – | MZ054655 | – | – |
| MF-G60103 | ” | ” | стебель | – | – | – | – | MZ054656 | – | – |
| MF-G60104 | ” | ” | листья | – | – | – | – | MZ054657 | – | – |
| MF-G60105 | ” | ” | листья | – | – | – | – | MZ054658 | – | – |
| MF-G60106 | ” | Санкт-Петербург, Пискаревский парк | стебель | – | – | – | – | MZ054659 | – | – |
| MF-G60107 | ” | Ленинградская обл., Ломоносовский р-н, Горелово |
стебель | – | – | – | – | MZ054660 | – | – |
| MF-G60108 | ” | Ленинградская обл., Лужский р-н | стебель | – | – | – | – | MZ054661 | – | – |
| MF-G60109 | ” | Финляндия | стебель | – | – | – | – | MZ054662 | – | – |
| MF-Vm17-010 | Heterophoma sylvatica | Ленинградская обл., Ломоносовский р-н | стебель | MZ046058 | – | – | – | – | MZ054682 | – |
| MF-Vm17-012 | ” | ” | стебель | MZ046059 | – | – | – | – | MZ054683 | MZ054694 |
| MF-Vm17-015 | ” | Ленинградская обл., Всеволожский р-н | стебель | MZ046060 | – | – | – | – | MZ054684 | MZ054695 |
| MF-Vm17-016 | ” | ” | стебель | MZ046061 | – | – | – | – | MZ054685 | MZ054696 |
| MF-Vm17-017 | ” | ” | стебель | MZ046062 | – | – | – | – | MZ054686 | MZ054697 |
| MF-Vm17-018 | ” | Ленинградская обл., Ломоносовский р-н | листья | MZ046063 | – | – | – | – | MZ054687 | MZ054698 |
| MF-Vm17-028 | ” | Ленинградская обл., Всеволожский р-н | стебель | MZ046064 | – | – | – | – | MZ054688 | – |
| MF-Vm17-039 | Heterophoma sp. | Ленинградская обл., Лужский р-н | стебель | MZ046065 | – | – | – | – | MZ054689 | MZ054699 |
| MF-Vm17-041 | Kalmusia longispora | Санкт-Петербург, Пискаревский лесопарк | листья | MZ046066 | – | – | – | – | MZ054690 | – |
| MF-Vm17-006 | Microsphaeropsis olivacea | Ленинградская обл., Лужский р-н | стебель | MZ046067 | – | – | – | – | MZ054691 | MZ054700 |
| MF-Vm17-040 | Neocucurbitaria cava | Ленинградская обл., Ломоносовский р-н, Горелово | стебель | MZ046068 | – | – | – | – | MZ054692 | – |
| MF-Vm17-047 | Sporocadus lichenicola |
Финляндия | стебель | MZ046069 | MZ046070 | – | – | – | – | – |
Для реконструкции молекулярной филогении и идентификации изолятов была произведена амплификация и последующее секвенирование таксономически информативных для анализируемой группы грибов локусов ДНК. Для Diaporthe анализировали последовательности областей внутренних транскрибируемых спейсеров рДНК (ITS), участков генов β-тубулина (TUB), фактора элонгации трансляции 1-α (TEF) и фрагмента гена, ответственного за синтез фермента ДНК-лиазы (apn2); Colletotrichum – ITS, TUB, TEF и фрагмента гена актина (ACT); Sporocadus – ITS и область большой субъединицы рибосомы рДНК (LSU); Kalmusia и Neocucurbitaria – ITS, TUB; Fusarium – TEF; изолятов грибов представителей семейства Didymellaceae – ITS, TUB и участка гена второй большой субъединицы фермента РНК-полимеразы II (RPB2).
Амплификацию ITS-области проводили с использованием праймеров ITS1F (Gardes, Bruns, 1993) и ITS4 (White et al., 1990); LSU – LR0R (Rehner, Samuels, 1994) и LR5 (White et al., 1990), ACT – Act-512F и Act-728R (Carbone, Kohn, 1999), apn2 – apn2fw2 и apn2rw2 (Udayanga et al., 2014), RPB2 – fRPB2-5F2 (Sung et al., 2007) и fRPB2-7cR (Liu et al., 1999), TEF – EF1-983F и EF1-1567R (Rehner, Buckley, 2005) или EF1-728F/EF1-986R (Carbone, Kohn, 1999), TUB – βtub2Fw и βtub4Rd (Aveskamp et al., 2009) или Т1 и Т2 (O’Donnell, Cigelnik, 1997; Saleh, Leslie, 2004).
Очищенные по стандартному протоколу (Boyle, Lew, 1995) фрагменты секвенировали методом Сэнгера (1977) на секвенаторе ABI Prism 3500 (Applied Biosystems – Hitachi, Япония) в соответствии с рекомендациями производителя с использованием набора реактивов Big Dye Terminator v3.1 Cycle Sequencing Kit (ABI, США).
Молекулярно-филогенетический анализ. Нуклеотидные последовательности выравнивали с помощью программы ClustalX 1.8 (Thompson et al., 1997), после чего при необходимости выравнивание корректировали вручную. Филогенетические деревья были построены согласно трем алгоритмам. Метод максимального правдоподобия (maximum likelihood – ML) применяли с использованием программного обеспечения RAxML (randomized accelerated maximum likelihood) v. 7.2.8 (Stamatakis et al., 2006). Принцип максимальной экономии (maximum parsimony – MP) применяли с использованием программного обеспечения Molecular Evolutionary Genetics Analysis версии 10 (MEGA X; Kumar et al., 2018). Анализ последовательностей методом Байесовской статистики (BPP) проводили с использованием программы Mr. Bayes v. 3.2.1., интегрированной в платформу Armadillo v. 1.1 (Lord et al., 2012). Надежность топологии дендрограмм, построенных разными методами, оценивали с помощью бутстреп-анализа c 1000 повторностей. В качестве референсных использовали полученные из базы данных GenBank последовательности ITS и LSU локусов рДНК, генов ACT, apn2, TEF, TUB, RPB2 разных видов и штаммов грибов (табл. 2).
Таблица 2.
Нуклеотидные последовательности референсных видов и штаммов грибов, использованные для реконструкции молекулярной филогении
| Вид гриба | № штамма | Номера доступа нуклеотидных последовательностей в GenBank | ||||||
|---|---|---|---|---|---|---|---|---|
| ITS | LSU | ACT | apn2 | TEF | TUB | RPB2 | ||
| Alloconiothyrium aptrootii | CBS 981.95 | JX496122 | – | – | – | – | JX496461 | – |
| Boeremia exigua | CBS 101150 | GU237715 | – | – | – | – | GU237495 | KT389568 |
| ” | MF 010-037 | MF065726 | – | – | – | – | MF065770 | MT293559 |
| ” | MF 010-039 | MF065727 | – | – | – | – | MF065771 | MT293560 |
| ” | MF 010-065 | MF065732 | – | – | – | – | MF065775 | MT293564 |
| ” | MF 010-071 | MF065736 | – | – | – | – | MF065777 | MT293573 |
| ” | MF 010-076 | MF065738 | – | – | – | – | MF065779 | MT293567 |
| ” | CBS 100354 | GU237711 | – | – | – | – | GU237506 | KT389577 |
| B. heteromorpha | CBS 443.94 | GU237866 | – | – | – | – | GU237497 | KT389573 |
| B. linicola | CBS 116.76 | GU237754 | – | – | – | – | GU237500 | KT389574 |
| B. lycopersici | CBS 378.67 | GU237848 | – | – | – | – | GU237512 | KT389580 |
| B. opuli | LC 8118 | KY742046 | – | – | – | – | KY742288 | KY742134 |
| B. populi | CBS 100167 | GU237707 | – | – | – | – | GU237501 | MT018058 |
| B. trachelospermi | CGMCC 3.18222; LC 8105 | KY064028 | – | – | – | – | KY064051 | KY064033 |
| Colletotrichum acerbum | CBS 128530 | JQ948459 | – | JQ949780 | – | – | JQ950110 | – |
| C. australe | CBS 116478 | JQ948455 | – | JQ949776 | – | – | JQ950106 | – |
| C. godetiae | CBS 133.44 | JQ948402 | – | JQ949723 | – | – | JQ950053 | – |
| C. johnstonii | CBS 128532 | JQ948444 | – | JQ949765 | – | – | JQ950095 | – |
| C. kinghornii | CBS 198.35 | JQ948454 | – | JQ949775 | – | – | JQ950105 | – |
| C. phormii | CBS 118194 | JQ948446 | – | JQ949767 | – | – | JQ950097 | – |
| C. pyricola | CBS 128531 | JQ948445 | – | JQ949766 | – | – | JQ950096 | – |
| C. rhombiforme | CBS 129953 | JQ948457 | – | JQ949778 | – | – | JQ950108 | – |
| C. salicis | CBS 607.94 | JQ948460 | – | JQ949781 | – | – | JQ950111 | – |
| ” | CBS 465.83 | JQ948468 | – | JQ949789 | – | – | JQ950119 | – |
| Diaporthe alleghaniensis | CBS 495.72 | – | – | – | KJ380963 | KC343733 | KC343975 | – |
| D. alnea | CBS 146.46 | – | – | – | KJ380969 | KC343734 | KC343976 | – |
| ” | LCM22B.02b | – | – | – | KJ380972 | KJ210558 | KJ420826 | – |
| ” | CBS159.47 | – | – | – | KJ380970 | KC343735 | KC343977 | – |
| ” | LCM22B.02a | – | – | – | KJ380971 | KJ210557 | KJ420825 | – |
| D. bicincta | CBS 121004 | – | – | – | KJ380976 | KC343860 | KC344102 | – |
| D. celastrina | CBS 139.27 | – | – | – | KJ380974 | KC343773 | KC344015 | – |
| D. citri | AR3405 | – | – | – | KJ380981 | KC343778 | KC344020 | – |
| D. eres | AR5193 | – | – | – | KJ380958 | KJ210550 | KJ420799 | – |
| ” | AR5196 | – | – | – | KJ380932 | KJ210554 | KJ420817 | – |
| ” | DP0438 | – | – | – | KJ380935 | KJ210553 | KJ420816 | – |
| ” | LCM11401a | – | – | – | KJ380919 | KJ210545 | KJ420787 | – |
| ” | FAU483 | – | – | – | KJ380933 | JQ807422 | KJ420827 | – |
| ” | DAN001A | – | – | – | KJ380914 | KJ210540 | KJ420781 | – |
| ” | DAN001B | – | – | – | KJ380915 | KJ210541 | KJ420782 | – |
| ” | AR5197 | – | – | – | KJ380931 | KJ210552 | KJ420812 | – |
| ” | AR3519 | – | – | – | KJ380922 | JQ807410 | KJ420789 | – |
| ” | FAU570 | – | – | – | KJ380926 | JQ807354 | KJ420794 | – |
| ” | AR3723 | – | – | – | KJ380941 | JQ807351 | KJ420793 | – |
| ” | AR3560 | – | – | – | KJ380939 | KJ210551 | KJ420795 | – |
| ” | AR5224 | – | – | – | KJ380961 | KJ210555 | KJ420802 | – |
| ” | AR5231 | – | – | – | KJ380936 | KJ210549 | KJ420818 | – |
| ” | AR5223 | – | – | – | KJ380938 | KJ210549 | KJ420830 | – |
| ” | DLR12a | – | – | – | KJ380916 | KJ210543 | KJ420783 | – |
| ” | AR4347 | – | – | – | KJ380929 | JQ807359 | KJ420805 | – |
| ” | AR4355 | – | – | – | KJ380942 | JQ807364 | KJ420797 | – |
| ” | AR4367 | – | – | – | KJ380962 | JQ807355 | KJ420824 | – |
| ” | AR4348 | – | – | – | KJ380952 | JQ807415 | KJ420811 | – |
| ” | AR3669=MAFF 625029 | – | – | – | KJ380930 | JQ807416 | KJ420808 | – |
| ” | DP0591 | – | – | – | KJ380946 | JQ807366 | KJ420821 | – |
| ” | AR4369 | – | – | – | KJ380953 | JQ807384 | KJ420813 | – |
| ” | DP0180 | – | – | – | KJ380928 | JQ807383 | KJ420804 | – |
| ” | DP0179 | – | – | – | KJ380944 | JQ807394 | KJ420803 | – |
| ” | AR4371 | – | – | – | KJ380927 | JQ807408 | KJ420796 | – |
| ” | CBS 113470 | – | – | – | KJ380956 | KC343872 | KC344114 | – |
| ” | AR4374 | – | – | – | KJ380943 | JQ807369 | KJ420785 | – |
| ” | AR4357 | – | – | – | KJ380949 | JQ807360 | KJ420806 | – |
| ” | DNP128 | – | – | – | KJ380960 | KJ210561 | KJ420801 | – |
| ” | FAU532 | – | – | – | KJ380934 | JQ807408 | KJ420815 | – |
| ” | AR5231 | – | – | – | KJ380936 | KJ210555 | KJ420818 | – |
| ” | AR3671=MAFF625033 | – | – | – | KJ380954 | JQ807417 | KJ420814 | – |
| ” | AR3672=MAFF625034 | – | – | – | KJ380937 | JQ807418 | KJ420819 | – |
| ” | AR3670=MAFF625030 | – | – | – | KJ380950 | JQ807416 | KJ420807 | – |
| ” | AR4373 | – | – | – | KJ380957 | JQ807368 | KJ420798 | – |
| ” | AR4346 | – | – | – | KJ380955 | JQ807355 | KJ420823 | – |
| ” | DP0590 | – | – | – | KJ380951 | JQ807394 | KJ420810 | – |
| ” | AR3538=CBS 109767 | – | – | – | KJ380940 | KC343801 | KC344043 | – |
| ” | DNP129 | – | – | – | KJ380959 | KJ210560 | KJ420800 | – |
| ” | CBS587.79 | – | – | – | KJ380975 | KC343879 | KC344121 | – |
| ” | AR4349 | – | – | – | KJ380947 | JQ807358 | KJ420822 | – |
| ” | AR4363 | – | – | – | KJ380948 | JQ807362 | KJ420809 | – |
| D. helicis | AR5211=CBS 138596 | – | – | – | KJ380977 | KJ210559 | KJ420828 | – |
| D. neilliae | CBS 144. 27 | – | – | – | KJ380973 | MH121567 | KC344112 | – |
| D. pulla | CBS 338.89 | – | – | – | KJ380978 | JQ807380 | KC344120 | – |
| D. vaccinii | DF5032=CBS 135436 | – | – | – | KJ380964 | JQ807413 | KC843225 | – |
| ” | FAU468 | – | – | – | KJ380965 | JQ807399 | KC843227 | – |
| ” | FAU446 | – | – | – | KJ380967 | JQ807398 | KC843224 | – |
| ” | CBS160.32 | – | – | – | KJ380968 | KC343954 | KC344196 | – |
| Heterophoma adonidis | CBS 114309; UPSC 2982 | KT389506 | – | – | – | – | KT389803 | MT018057 |
| H. nobilis | CBS 507.91; PD 74/148 | GU237877 | – | – | – | – | GU237603 | KT389638 |
| H. poolensis | CBS 253.38 | MN973475 | – | – | – | – | MT005574 | MT018054 |
| H. sylvatica | CBS 874.97; PD 93/764 | GU237907 | – | – | – | – | GU237662 | MT018052 |
| H. verbascicola | CGMCC 3.18364; LC 8163 | KY742119 | – | – | – | – | KY742361 | KY742187 |
| H. novae-verbascicola | CBS 127.93; PD 92/347 | GU237774 | – | – | – | – | GU237639 | MT018051 |
| Kalmusia longispora | CBS 582.83 | JX496097 | – | – | – | – | JX496436 | – |
| ” | CBS 824.84 | JX496115 | – | – | – | – | JX496454 | – |
| K. variispora | CBS 121517 | JX496030 | – | – | – | – | JX496369 | – |
| K. variispora | CBS 197.82 | JX496053 | – | – | – | – | JX496392 | – |
| Macroventuria anomochaeta | CBS 525.71 | GU237881 | – | – | – | – | GU237544 | GU456346 |
| Microsphaeropsis fusca | CBS 116669 | MN973572 | – | – | – | – | MT005675 | MT018219 |
| M. olivacea | CBS 233.77 | GU237803 | – | – | – | – | GU237549 | MT018217 |
| M. taxicola | CBS 442.83 | GU237865 | – | – | – | – | GU237547 | MT018211 |
| M. viridis | CBS 639.80 | MN973564 | – | – | – | – | MT005667 | MT018208 |
| M. proteae | CBS 111303; STE-U 1423 | JN712495 | – | – | – | – | MT005677 | MT018221 |
| Neocucurbitaria cava | CBS 257.68 | MH859129 | – | – | – | – | KT389844 | – |
| N. cava | CBS 115979 | MH862993 | – | – | – | – | LT623234 | – |
| ” | CBS 143400 | MH107903 | – | – | – | – | MH108046 | – |
| N. citisisicola | CBS 142402 | NR_156357 | – | – | – | – | MF795900 | – |
| N. juglandicola | CBS 142390 | NR_156358 | – | – | – | – | MF795901 | – |
| N. hakeae | CPC28920 | KY173436 | – | – | – | – | KY173613 | – |
| N. populi | CBS 142393 | NR_156359 | – | – | – | – | MF795902 | – |
| Sporocadus cornicola | CBS 143889 | MH554121 | MH554326 | – | – | – | – | – |
| S. cotini | CBS 139966 | MH554003 | MH554222 | – | – | – | – | – |
| S. lichenicola | NBRC 32680 | AB594806 | AB593739 | – | – | – | – | – |
| ” | MFLUCC:14-0052 | KT005515 | KT005514 | – | – | – | – | – |
| ” | CPC 24528 | MH554127 | MH554332 | – | – | – | – | – |
| ” | NBRC 32625 | AB594794 | AB593726 | – | – | – | – | – |
| ” | CBS 354.90 | AB594803 | AB593735 | – | – | – | – | – |
| S. rosigena | CBS 116498 | MH553983 | MH554200 | – | – | – | – | – |
| ” | CBS 129166 | MH553996 | MH554215 | – | – | – | – | – |
| ” | CBS 182.50 | MH554013 | MH554233 | – | – | – | – | – |
| ” | CBS 466.96 | MH554052 | MH554265 | – | – | – | – | – |
| S. sorbi | CBS 160.25 | MH854829 | MH866330 | – | – | – | – | – |
РЕЗУЛЬТАТЫ
Всего из листьев и стеблей черники было выделено 67 изолятов грибов. В результате идентификации по морфологическим признакам определено 16 родов аскомицетов, относящихся к 8 порядкам (Amphisphaerellales, Diaporthales, Capnodiales, Eurotiales, Glomerellales, Hypocreales, Pleosporales, Sordariales) и 13 семействам (Capnodiaceae, Chaetomiaceae, Cladosporiaceae, Coniothyriaceae, Diaporthaceae, Didymellaceae, Glomerellaceae, Hypocreaceae, Nectriaceae, Pestalotiopsidaceae, Pleosporaceae, Sordariaceae, Trichocomaceae).
Из исследуемой коллекции изолятов до уровня вида были определены Fusarium avenaceum (Fr.) Sacc., F. incarnatum (Desm.) Sacc., F. sporotrichioides Sherb. До таксона уровня секции идентифицировали Alternaria sect. Alternaria D.P. Lawr., Gannibal, Peever et B.M. Pryor, Alternaria sect. Infectoriae Woudenb. et Crous. До уровня рода идентифицировали Boeremia Aveskamp, Gruyter et Verkley, Chaetomium Kunze, Cladosporium Link., Colletotrichum Сorda, Coniothyrium Corda, Curvularia Boedijn, Diaporthe Nitschke, Epicoccum Link., Penicillium Link., Pestalotiopsis Steyaert, Sordaria Ces. et De Not, Trichoderma Pers. Кроме того, часть изолятов была идентифицирована только до уровня семейства Didymellaceae.
Также идентифицировали грибы из двух родов базидиомицетов – Rhizoctonia DC. и Sporotrichum Link., относящихся к порядкам Cantharellales и Polyporales и семействам Ceratobasidiaceae и Fomitopsidaceae, соответственно.
Всего 31 изолят из групп, надежное определение которых на настоящий момент рекомендуется осуществлять по молекулярно-генетическим признакам (роды Colletotrichum, Diaporthe, представители Didymellaceae, вид F. avenaceum), был изучен с применением методов молекулярной идентификации. В результате проведенного анализа установлена видовая принадлежность 30 штаммов, идентификация одного осуществлена до рода.
В мультилокусный анализ нуклеотидных последовательностей трех локусов семи наших изолятов Diaporthe были включены последовательности 58 референсных штаммов Diaporthe, составляющих комплекс видов D. eres, в качестве внешней группы взят штамм D. citri F.A. Wolf (AR3405). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 1418 оснований (394 – TUB, 290 – TEF, 734 – apn2), число вариабельных сайтов составляло 265 (18.7%). Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности совпадала с топологией комбинированной филограммы. Участки ITS-локуса были проанализированы, но не включены в филогенетический анализ из-за низкой информативности. Филогенетическое дерево, сконструированное в результате анализа последовательностей трех локусов с применением метода ML, приведено на рисунке 2. Все наши штаммы Diaporthe кластеризовались в пределах комплекса видов D. eres. Два штамма MF-Vm17-013, MF-Vm17-029 формировали одну кладу со штаммом D. pulla Nitschke (CBS 338.89). Остальные пять штаммов (MF-Vm17-001, MF-Vm17-008, MF-Vm17-009, MF-Vm17-019, MF-Vm17-030) формировали субклады в пределах вида D. eres.
Рис. 2.
Комбинированное филогенетическое древо комплекса видов Diaporthe eres, построенное методом ML, основанное на нуклеотидных последовательностях TUB, TEF, apn2. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.

Для видов родов представителей семейства Didymellaceae осуществлено мультилокусное секвенирование нуклеотидных последовательностей трех таксономически информативных фрагментов. В анализ включены последовательности 10 наших изолятов представителей семейства Di-dymellaceae и 25 референсных штаммов, в качестве внешней группы взят штамм Macroventuria anomochaeta AA (CBS 525.71). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 1333 оснований (454 – ITS, 289 – TUB, 590 – RPB2), число вариабельных сайтов составляло 298 (22.5%). Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности совпадала с топологией комбинированной филограммы. Филогенетическое дерево, сконструированное в результате анализа последовательностей трех локусов с применением метода ML, приведено на рис. 3.
Рис. 3.
Комбинированное филогенетическое древо семейства Didymellaceae (роды Boeremia, Heterophoma, Microsphaeropsis), построенное методом ML, основанное на нуклеотидных последовательностях ITS, TUB, RPB2. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.

Восемь штаммов (MF-Vm17-010, MF-Vm17-012, MF-Vm17-015, MF-Vm17-016, MF-Vm17-017, MF-Vm17-018, MF-Vm17-028, MF-Vm17-039) кластеризовались в пределах клады, сформированной видами рода Heterophoma Qian Chen et L. Cai. Внутри этой клады семь штаммов формировали единую субкладу с типовым штаммом Heterophoma sylvatica (Sacc.) Qian Chen et L. Cai (CBS 874.97). Штамм MF-Vm17-039 формировал отдельную субкладу, не включающую референсные штаммы этого рода. Штамм MF-Vm17-006 входил в состав клады Microsphaeropsis Höhn., внутри которой был в одной субкладе с репрезентативным штаммом Microsphaeropsis olivacea (Bonord.) Höhn. (CBS 233.77). Штамм MF-Vm17-035 располагался в кладе с репрезентативными штаммами Boeremia exigua (Desm.) Aveskamp, Gruyter et Verkley.
В мультилокусный анализ видов рода Colletotrichum включены последовательности двух наших изолятов и 10 референсных штаммов, в качестве внешней группы взят штамм Colletotrichum godetiae Neerg. (CBS 133.44). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 1220 оснований (538 – ITS, 461 – TUB, 221 – ACT), число вариабельных сайтов составляло 88 (7.2%). Участки гена TEF были проанализированы, но не включены в филогенетический анализ. Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности и комбинированная совпадали. Филогенетическое дерево, сконструированное в результате анализа последовательностей трех локусов с применением метода ML, приведено на рис. 4. Два исследованных штамма Colletotrichum (MF-Vm17-043, MF-Vm17-044) внутри комплекса видов C. acutatum J.H. Simmonds формировали единую кладу вместе со штаммами C. salicis (Fuckel) Damm, P.F. Cannon et Crous, включая типовой штамм CBS 607.94.
Рис. 4.
Комбинированное филогенетическое древо видов Colletotrichum, построенное методом ML, основанное на нуклеотидных последовательностях ITS, TUB, ACT. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.

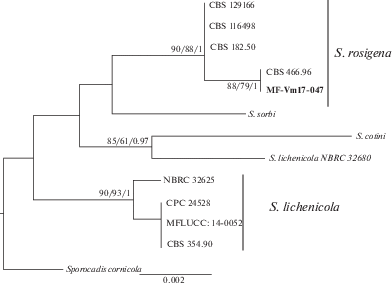
Вид рода Sporocadus идентифицирован в результате мультилокусного секвенирования нуклеотидных последовательностей двух фрагментов рДНК. В анализ включены последовательности одного нашего изолята MF-Vm17-047 и 11 референсных штаммов, в качестве внешней группы взят штамм Sporocadus cornicola (Wijayaw. et Camporesi) F. Liu, L. Cai et Crous (CBS 143889). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 1304 оснований (515 – ITS, 789 – LSU), число вариабельных сайтов составляло 29 (2.2%). Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности и комбинированная совпадали. Филогенетическое дерево, сконструированное в результате анализа последовательностей двух локусов с применением метода ML, приведено на рис. 5. Исследованный штамм MF-Vm17-047 внутри рода Sporocadus формирует единую кладу вместе со штаммами S. rosigena F. Liu, L. Cai et Crous.
Рис. 5.
Комбинированное филогенетическое древо видов Sporocadus, построенное методом ML, основанное на нуклеотидных последовательностях ITS и LSU. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.
В анализ результатов мультилокусного секвенирования видов рода Kalmusia включены последовательности одного нашего изолята MF-Vm17-041 и пяти референсных штаммов. В качестве внешней группы взят штамм Alloconiothyrium aptrootii Verkley, Göker et Stielow (CBS 981.95). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 856 оснований (457 – ITS, 399 – TUB), число вариабельных сайтов составляло 132 (15.4%). Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности и комбинированная, совпадали. Филогенетическое дерево, сконструированное в результате анализа последовательностей двух локусов с применением метода ML, приведено на рис. 6. Исследованный штамм MF-Vm17-041 внутри рода Kalmusia формирует единую кладу вместе со штаммами Kalmusia longispora (Verkley, Göker et Stielow) Ariyaw. et K.D. Hyde, включая типовой штамм CBS582.83.
Рис. 6.
Комбинированное филогенетическое древо видов Kalmusia, построенное методом ML, основанное на нуклеотидных последовательностях ITS и TUB. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.

Мультилокусное секвенирование двух фрагментов ДНК видов рода Neocucurbitaria проведено для одного нашего изолята MF-Vm17-040 и семи референсных штаммов, в качестве внешней группы взят штамм Neocucurbitaria hakeae (Crous) Valenz.-Lopez, Crous, Stchigel, Guarro et J.F. Cano (CPC28920). После проведения выравнивания, матрицы каждого штамма, используемые для реконструкции филогении, имели длину 810 оснований (491 – ITS, 319 – TUB), число вариабельных сайтов составляло 74 (9.1%). Топология филограмм, построенных разными методами и на основании последовательностей каждого локуса в отдельности и комбинированная, совпадали. Филогенетическое дерево, сконструированное в результате анализа последовательностей двух локусов с применением метода ML, приведено на рис. 7. Исследованный штамм MF-Vm17-040 внутри рода Neocucurbitaria формировал единую кладу вместе со штаммами N. cava (Schulzer) Valenz.-Lopez, Crous, Stchigel, Guarro et J.F. Cano, включая типовой штамм CBS 257.68.
Рис. 7.
Комбинированное филогенетическое древо видов Neocucurbitaria, построенное методом ML, основанное на нуклеотидных последовательностях ITS и TUB. Числовые значения бутстреп-поддержки, полученные методами ML, MP и BPP, приведены в узлах ветвей филограммы, соответственно. Номера исследованных штаммов выделены полужирным.

На основании нуклеотидных последовательностей фрагмента гена TEF девяти штаммов Fusarium, предварительно отнесенных к Fusarium avenaceum по морфологическим признакам, показано наибольшее сходство (100%) со штаммом F. avenaceum BRIP:64445 (KU529161), выделенным из пиретрума, собранного в Австралии.
ОБСУЖДЕНИЕ
В результате проведенных исследований коллекции штаммов грибов, выделенных из пораженных листьев и стеблей черники, по морфологическим и молекулярно-генетическим признакам нами идентифицировано 12 видов грибов: Boeremia exigua, Colletotrichum salicis, Diaporthe eres, D. pulla, Fusarium avenaceum, F. incarnatum, F. sporotrichioides, Heterophoma sylvatica, Kalmusia longispora, Micro-sphaeropsis olivacea, Neocucurbitaria cava, Sporocadus rosigena. Были выявлены грибы из двух секций рода Alternaria: Alternaria и Infectoriae. Кроме того, показано, что на растениях черники, растущей на северо-западе Европы, могут развиваться представители относящиеся, как минимум, к 21 роду грибов из двух отделов, 10 порядков, 15 семейств. Помимо вышеперечисленных родов грибов, обнаружены микромицеты из родов Chaetomium, Cladosporium, Coniothyrium, Curvularia, Epicoccum, Penicillium, Pestalotiopsis, Sordaria, Trichoderma.
Все выделенные из черники штаммы Diaporthe идентифицированы как представители комплекса видов Diaporthe eres. Известно, что этот гриб является весьма полиморфным и филогенетически гетерогенным, его представители могут быть ассоциированы с разными. Нуклеотидные последовательности ITS-локуса оказались неинформативными для реконструкции филогении в этой группе грибов, что было показано ранее (Udayanga et al., 2014). Поэтому они были депонированы в базу GenBank, но исключены из филогенетического анализа. Два штамма MF-Vm17-001, MF-Vm17-008 формировали субкладу в кладе, которую ранее трактовали, как вид D. cotoneastri (Punith.) Udayanga, Crous et K.D. Hyde. Штамм MF-Vm17-030 формировал отдельную субкладу, близкородственную кладам, которые ранее понимали, как виды D. cotoneastri и D. nobilis Sacc. et Speg. Штамм MF-Vm17-019 занимал на филогенетическом древе обособленное положение, формируя отдельную субкладу. В кладе, включающей штамм D. eres AR5193, являющийся эпитипом этого вида, оказался штамм MF-Vm17-009. В результате ревизии вида D. eres и видов, составляющих комплекс D. eres, выделение таких видов как D. cotoneastri, D. nobilis и других признано избыточным, и наименования этих таксонов синонимизировали с именем D. eres (Udayanga et al., 2014; Yang et al., 2018). Вероятно, это ожидает и вид D. pulla, который на филогенетических деревьях располагался среди штаммов D. eres и включал в состав формируемой им клады два штамма MF-Vm17-013 и MF-Vm17-029. На настоящий момент вид D. pulla еще является легитимным и эти штаммы, выделенные из черники, собранной в Санкт-Петербурге и Гатчинском р-не Ленинградской обл., согласно актуальным взглядам на филогению комплекса видов D. eres являются видом D. pulla. Входящий в список карантинных объектов Евразийского экономического союза, вид D. vaccinii, который также входит в состав комплекса видов D. eres нами обнаружен не был.
Гриб Colletotrichum acutatum, также входящий в единый перечень карантинных объектов, среди исследованных штаммов нами обнаружен не был. Два штамма Colletotrichum MF-Vm17-043, MF-Vm17-044 идентифицированы, как C. salicis. Этот микромицет входит в состав комплекса видов C. acutatum. Данный гриб не имеет узкой субстратной специализации и может быть ассоциирован с растениями разных семейств, однако, считается, что чаще он развивается на древесных растениях. Помимо разных видов ивы, из которых часто выделяют этот вид гриба, известны его штаммы из клена, араукарии, яблони, тополя, груши, томата и клубники (Cunnington et al., 2007; Pennycook, 1989; Guerber et al., 2003; Gadgil, 2005; Sun et al., 2011; Mulenko et al., 2008; Damm et al., 2012). Распространен C. salicis на территории стран Европы, в США, Австралии и Новой Зеландии, но в России этот гриб выявлен нами впервые.
Корректная и достоверная идентификация карантинных вредоносных видов Diaporthe vaccinii и Colletotrichum acutatum только на основании морфологических признаков невозможна и требует молекулярно-филогенетического анализа. Выявление и идентификацию этих грибов следует проводить в соответствии с современными взглядами на биоразнообразие и таксономию этих микромицетов.
Опубликованной информации о выявлении грибов Fusarium на чернике нами не обнаружено. Однако на культурных посадках голубики (Vaccinium corymbosum) виды Fusarium solani (Mart.) Sacc. (Pérez et al., 2007), F. proliferatum (Matsush.) Nirenberg ex Gerlach et Nirenberg (Pérez et al., 2011), F. oxysporum Schltdl. (Moya-Elizondo et al., 2019) приводили к корневой гнили и увяданию растений, а F. acuminatum Ellis et Everh. вызывал некрозы стеблей (Wright et al., 2014). В проведенных исследованиях идентифицировано три вида: F. avenaceum, F. incarnatum, F. sporotrichioides, среди которых F. avenaceum встречался повсеместно на несущих симптомы растениях. По всей видимости, это первое упоминание видов грибов Fusarium на чернике.
Считается, что вид Heterophoma sylvatica, к которому относятся семь исследованных штаммов, широко распространен в Европе на растениях семейства Scrophulariaceae и встречается также в ассоциации с корнями растений семейства Poaceae (Boerema et al., 2004). Нами этот вид впервые был выделен из черники, собранной во Всеволожском и Ломоносовском р-нах Ленинградской области. Несмотря на то, что для реконструкции молекулярной филогении штаммов Heterophoma в анализ были включены все шесть известных на настоящий момент видов этого рода, штамм MF-Vm17-039, выделенный из стеблей черники, собранной в Лужском районе Ленинградской обл. не входит в состав ни одной из клад, сформированной каждым видом, а формирует отдельную кладу. Таким образом, вероятно, этот штамм Heterophoma sp. является новым для науки видом этого рода.
Микромицет Microsphaeropsis olivacea (штамм MF-Vm17-006) был выявлен на многих растениях разных семейств, как сапротроф или эндофит (Hormazabal et al., 2014), в редких случаях, как фитопатоген (Razaghi, Zafari, 2016) или патоген, вызывающий микозы человека с ослабленным иммунитетом (Guarro et al., 1999). Данный вид впервые обнаружен на территории России – штамм выделен из черники, собранной в Лужском р-не.
Гриб Kalmusia longispora также выявлен на территории России впервые, штамм этого гриба MF-Vm17-041 выделен из черники, собранной на территории Санкт-Петербурга. До настоящего момента этот гриб был выделен из омелы (Arceuthobium pusillum Peck.) с территории Канады и из листьев пшеницы с симптомами пятнистости в Германии (Verkley et al., 2014).
Штамм MF-Vm17-040, который идентифицирован, как гриб Neocucurbitaria cava является первой находкой этого микромицета в России, он выявлен в Ломоносовском районе Ленинградской области. Это гриб известен как почвенный сапроторф или эндофит, ассоциированный преимущественно с древесными растениями или кустарниками (de Gruyter et al., 2010, Jaklitsch et al., 2018).
Sporocadus rosigena описан на территории Италии как сапротроф с мертвых иголок розы (Wanasinghe et al., 2018), впоследствии этот гриб также выявляли на растениях других семейств с территории других стран Европы и в Иране (Liu et al., 2019). Нами штамм этого гриба MF-Vm17-047 был впервые обнаружен на чернике, собранной на территории Финляндии.
Полученные результаты позволяют предварительно оценить биоразнообразие микромицетов на чернике, собранной на территории северо-запада России и в Финляндии. Для более детальной оценки, несомненно, необходим многолетний мониторинг и расширение географии сбора образцов, а также дальнейшие исследования, с включением в молекулярно-генетический анализ бόльшего числа штаммов.
Авторы выражают искреннюю благодарность сотрудникам лаборатории микологии и фитопатологии ВИЗР О.П. Гавриловой и И.А. Казарцеву за помощь со сбором пораженных растений черники. Работа выполнена при финансовой поддержке Российского научного фонда (проект № 19-76-30005).
Список литературы
Aveskamp M.M., Verkley G.J.M., de Gruyter J. et al. DNA phylogeny reveals polyphyly of Phoma section Peyronellaea and multiple taxonomic novelties. Mycologia. 2009. V. 101 (3). P. 363–382. https://doi.org/10.3852/08-199
Boerema G.H., Gruyter J., Noordeloos M.E. et al. Phoma identification Manual. CABI Publishing, 2004.
Boyle J.S., Lew A.M. An inexpensive alternative to glassmilk for DNA purification. Trends in Genetics. 1995. V. 11(1). P. 8. https://doi.org/10.1016/S0168-9525(00)88977-5
Carbone I., Kohn L.M. A method for designing primer sets for speciation studies in filamentous ascomycetes. Mycologia. 1999. V. 91. P. 553–556. https://doi.org/10.2307/3761358
Cunnington J.H., Powney R.A., Adair R.J. et al. Glomerella miyabeana on willows in Australia. Austral. Mycol. 2007. V. 25. P. 69–72.
Damm U., Cannon P.F., Woudenberg J.H.C. et al. The Colletotrichum boninense species complex. Stud. Mycol. 2012. V. 73. P. 1–36. https://doi.org/10.3114/sim0002
de Gruyter J.D., Woudenberg J.H.C., Aveskamp M.M. et al. Systematic reappraisal of species in Phoma section Paraphoma, Pyrenochaeta and Pleurophoma. Mycologia. 2010. V. 102. P. 1066–1081. https://doi.org/10.3852/09-240
Dingley J.M., Gilmour J.W. Colletotrichum acutatum Simmds. f. sp. pineum associated with terminal crook disease of Pinus spp. N. Z. J. For. Sci. 1972. V. 2. P. 192–201.
Doyle J.J., Doyle J.L. Isolation of plant DNA from fresh tissue. Focus. 1990. V. 12. P. 13–15. https://doi.org/10.1007/978-3-642-83962-7_18
Elfar K., Torres R., Díaz G.A. et al. Characterization of Diaporthe australafricana and Diaporthe spp. associated with stem canker of blueberry in Chile. Plant Dis. 2013. V. 97. P. 1042–1050. https://doi.org/10.1094/PDIS-11-12-1030-RE
Eurasian economic commission council decision. On approval of the common list of quarantine pests of the Eurasian Economic Union. 30.11.2016. No. 158. Moscow, 2016.
Farr D.F., Castlebury L.A., Rossman A.Y. Morphological and molecular characterization of Phomopsis vaccinii and additional isolates of Phomopsis from blueberry and cranberry in the eastern United States. Mycologia. 2002a. V. 94. P. 494–504. https://doi.org/10.2307/3761783
Farr D.F., Castlebury L.A., Rossman A.Y. et al. A new species of Phomopsis causing twig dieback of Vaccinium vitis-idaea (lingonberry). Mycol. Res. 2002b. V. 10. P. 745–752. https://doi.org/10.1017/S095375620200583X
Gadgil P.D. Fungi on trees and shrubs in New Zealand. In: Z.Y. Gao, J. Gan (eds). Fungi of New Zealand Volume 4. Fungal Diversity Press, Hong Kong, 2005. P. 437.
Gao Y., Liu F., Duan W. et al. Diaporthe is paraphyletic. IMA fungus. 2017. V. 8 (1). P. 153–187. https://doi.org/10.5598/imafungus.2017.08.01.11
Gardes M., Bruns T.D. ITS primers with enhanced specificity for basidiomycetes – application to the identification of mycorrhizae and rusts. Mol. Ecol. 1993. V. 2. P. 113–118. https://doi.org/10.1111/j.1365-294X.1993.tb00005.x
Garrido C., Carbú M., Fernández-Acero F.J. et al. Phylogenetic relationships and genome organisation of Colletotrichum acutatum causing anthracnose in strawberry. Eur. J. Plant Pathol. 2009. V. 125. P. 397–411. https://doi.org/10.1007/s10658-009-9489-0
Gerlach W., Nirenberg H. The genus Fusarium – a pictorial atlas. Mitt. Biol. Bundesanst. Land- Forstwirtsch. Berlin, Dahlem, 1982. 406 p.
Gomes R.R., Glienke C., Videira S.I.R. et al. Diaporthe: a genus of endophytic, saprobic and plant pathogenic fungi. Persoonia. 2013. V. 31. P. 1–41. https://doi.org/10.3767/003158513X666844
Guarro J., Mayayo E., Tapiol J. et al. Microsphaeropsis olivacea as an etiological agent of human skin infection. Med. Mycol. 1999. V. 37(2). P. 133–137. https://doi.org/10.1080/02681219980000211
Guerber J.C., Liu B., Correll J.C., Johnston P.R. Characterization of diversity in Colletotrichum acutatum sensu lato by sequence analysis of two gene introns, mtDNA and intron RFLPs, and mating compatibility. Mycologia. 2003. V. 95. P. 872–895. https://doi.org/10.2307/3762016
Hilário S., Amaral I.A., Gonçalves M.F.M. et al. Diaporthe species associated with twig blight and dieback of Vaccinium corymbosum in Portugal, with description of four new species. Mycologia. 2020. V. 112 (2). P. 293–308. https://doi.org/10.1080/00275514.2019.1698926
Hormazabal E., Astudillo L., Schmeda-Hirschmann G. et al. Metabolites from Microsphaeropsis olivacea, an endophytic fungus of Pilgerodendron uviferum. Z. Naturforsch. 2014. https://doi.org/10.1515/znc-2005-1-203
Jaklitsch W.M., Checa J., Blanco M.N. et al. A preliminary account of the Cucurbitariaceae. Stud. Mycol. 2018. V. 90. P. 71–118. https://doi.org/doi.org/10.1016/j.simyco.2017.11.002
Kumar S., Stecher G., Li M. et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Mol. Biol. Evol. 2018. V. 35. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Latorre B.A., Elfar K., Espinoza J.G. et al. First report of Diaporthe australafricana associated with stem canker on blueberry in Chile. Plant Dis. 2012. V. 96. P. 768. https://doi.org/10.1094/PDIS-12-11-1025-PDN
Lee D.H., Kim D.H., Jeon Y.A. et al. Molecular and cultural characterization of Colletotrichum spp. causing bitter rot of apples in Korea. Plant Pathol. 2007. V. 23. P. 37–44. https://doi.org/10.5423/PPJ.2007.23.2.037
Liu F., Bonthond G., Groenewald J.Z. et al. Sporocadaceae, a family of coelomycetous fungi with appendagebearing conidia. Stud. Mycol. 2019. V. 92. P. 287–415. https://doi.org/10.1016/j.simyco.2018.11.001
Liu Y.J., Whelen S., Hall B.D. Phylogenetic relationships among ascomycetes: evidence from an RNA polymerase II subunit. Mol. Biol. Evol. 1999. V. 16. P. 1799–1808. https://doi.org/10.1093/oxfordjournals.molbev.a026092
Lombard L., van Leeuwen G., Guarnaccia V. et al. Diaporthe species associated with Vaccinium, with specific refe-rence to Europe. Phytopathol. Mediterr. 2014. V. 53 (2). P. 287–299. https://doi.org/10.14601/Phytopathol_Mediterr-14034
Lord E., Leclercq M., Boc A. et al. Armadillo 1.1: An original workflow platform for designing and conducting phylogenetic analysis and simulations. Plos One. 2012. V. 7(1): e29903. https://doi.org/10.1371/journal.pone.0029903
Michalecka M., Bryk H., Seliga P. Identification and characterization of Diaporthe vaccinii Shear causing upright dieback and viscid rot of cranberry in Poland. Eur. J. Plant Pathol. 2017. V. 148. P. 595–605. https://doi.org/10.1007/s10658-016-1114-4
Morochkovskiy S.F., Zerova M.Ya., Lavitskaya Z.G. et al. Opredelitel gribov Ukrainy tom 2. Naukova dumka, Kiev, 1969 (in Russ.).
Moya-Elizondo E.A., Doussoulin H., San Martin J. et al. First report of Fusarium oxysporum causing Fusarium wilt on blueberry (Vaccinium corymbosum) in Chile. Plant Dis. 2019. V. 103 (10). P. 2669. https://doi.org/10.1094/PDIS-02-19-0275-PDN
Mułenko W., Majewski T., Ruszkiewicz-Michalska M. A preliminary checklist of micromycetes in Poland. W. Szafer Institute of Botany. Polish Academy of Sciences, 2008. P. 1–752.
Naraouei-Khandan H.A., Harmon C.L., Harmon P. et al. Potential global and regional geographic distribution of Phomopsis vaccinii on Vaccinium species projected by two species distribution models. Eur. J. Plant Pathol. 2017. V. 148. P. 919–930. https://doi.org/10.1007/s10658-017-1146-4
O’Donnell K., Cigelnik E. Two divergent intragenomic rDNA ITS2 types within a monophyletic lineage of the fungus Fusarium are non orthologous. Mol. Phylogenetics Evol. 1997. V. 7. P. 103–116. https://doi.org/10.1006/mpev.1996.0376
Pennycook S.R. Plant diseases recorded in New Zealand Vol. 3. Plant Diseases Division, DSIR, New Zealand, Auckland, 1989.
Pérez B.A., Berretta M.F., Carrión E. et al. First report of root rot caused by Fusarium proliferatum on blueberry in Argentina. Plant Dis. 2011. V. 95(11). P. 1478. https://doi.org/10.1094/PDIS-04-11-0307
Pérez B.A., Murillo F., Divo de Sesar M. et al. Occurrence of Fusarium solani on blueberry in Argentina. Plant Dis. 2007. V. 91(8). P. 1053. https://doi.org/10.1094/PDIS-91-8-1053C
Polashock J.J., Oudemans P.V., Caruso F.L. et al. Population structure of the North American cranberry fruit rot complex. Plant Pathol. 2009. V. 58. P. 1116–1127. https://doi.org/10.1111/j.1365-3059.2009.02120.x
Razaghi P., Zafari D. First report of Microsphaeropsis olivacea causing brown spine rot on Alhagi maurorum in Iran. J. Plant Pathol. 2016. V. 98 (3). P. 677–697. https://doi.org/10.4454/JPP.V98I2.044
Rehner S.A., Samuels G.J. Taxonomy and phylogeny of Gliocladium analysed from nuclear large subunit ribosomal DNA sequences. Mycol. Res. 1994. V. 98. P. 625–634. https://doi.org/10.1016/S0953-7562(09)80409-7
Rehner S.A., Buckley E.A. Beauveria phylogeny inferred from nuclear ITS and EF1-a sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia. 2005. V. 97. P. 84–98. https://doi.org/10.1080/15572536.2006.11832842
Saleh A.A., Leslie J.F. Cephalosporium maydis is a distinct species in the Gaeumannomyces–Harpophora species complex. Mycologia. 2004. V. 96 (6). P. 1294–1305. https://doi.org/10.2307/3762146
Samson R.A., Hoekstra E.S., Frisvad J.C. et al. Introduction to food-and airborne fungi. 6th edition. Centraalbureau voor schimmelcultures, Utrecht, 2000.
Sanger F., Nicklen S., Coulson A.R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U S A. 1977. V. 74 (12). P. 5463–5467. https://doi.org/10.1073/pnas.74.12.5463
Santos L., Alves A., Alves R. Evaluating multi-locus phylogenies for species boundaries determination in the genus Diaporthe. PerrJ. 2017. V. 5. P. 1–26. https://doi.org/10.7717/peerj.3120
Simmonds J.H. A study of the species of Colletotrichum causing ripe fruit rots in Queensland. Queensland J. Agric. Anim. Sci. 1965. V. 22. P. 437–459.
Simmons E.G. Alternaria. An identification manual. CBS biodiversity series n 6. CBS fungal biodiversity centre, Utrecht, 2007.
Stamatakis A. RAxML-VI-HPC: Maximum likelihood-based phylogenetic analyses with thousands of taxa and mixed models. Bioinformatics. 2006. V. 22. P. 2688–2690. https://doi.org/10.1093/bioinformatics/btl446
Sun X., Guo L.-D., Hyde K.D. Community composition of endophytic fungi in Acer truncatum and their role in decomposition. Fungal Diversity. 2011. V. 47. P. 85–95. https://doi.org/10.1007/s13225-010-0086-5
Sung G.-H., Sung J.-M., Hywel-Jones N.L. et al. A multi-gene phylogeny of Clavicipitaceae (Ascomycota, Fungi): identification of localized incongruence using a combinational bootstrap approach. Mol. Phylogenetics Evol. 2007. V. 31. P. 1204–1223. https://doi.org/10.1016/j.ympev.2007.03.011
Szamagara M. Biodiversity of fungi inhabiting the highbush blueberry stems. Acta Sci. Pol., Hortorum Cultus. 2009. V. 8 (1). P. 37–50.
Thompson J.D., Gibson T.J., Plewniak F. et al. The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 1997. V. 24. P. 4876–4882. https://doi.org/10.1093/nar/25.24.4876
Udayanga D., Castlebury L.A., Rossman L.A. et al. Insights into the genus Diaporthe: phylogenetic species delimitation in the D. eres species complex. Fungal Diversity. 2014. V. 64. P. 203–229. https://doi.org/10.1007/s13225-014-0297-2
Verkley G.J.M., Dukik K., Renfurm R. et al. Novel genera and species of coniothyrium-like fungi in Montagnulaceae (Ascomycota). Persoonia. 2014. V. 32. P. 25–51. https://doi.org/10.3767/003158514X679191
Vilka L., Volkova J. Morphological diversity of Phomopsis vaccinii isolates from Cranberry (Vaccinium macrocarpon Ait.) in Latvia. Proceedings of the Latvia University of agriculture. 2015. V. 3 (328). P. 8–18. https://doi.org/10.1515/plua-2015-0002
Wanasinghe D.N., Phukhamsakda C., Hyde K.D. et al. Fungal diversity notes 709–839: taxonomic and phylogenetic contributions to fungal taxa with an emphasis on fungi on Rosaceae. Fungal Diversity. 2018. V. 89. P. 1–236. https://doi.org/10.1007/s13225-018-0395-7
Wharton P.S., Schilder A.M.C. Novel infection strategies of Colletotrichum acutatum on ripe blueberry fruit. Plant Pathol. 2008. V. 57. P. 122–134. https://doi.org/10.1111/j.1365-3059.2007.01698.x
White T.J., Bruns T., Lee S., et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols. In: M.A. Innis (eds). A guide to methods and Aapplications. San Diego, Academic Press, 1990. P. 315–322.
Wright E.R., Rivera M.C., Campanella E.R. et al. Fusarium branch blight on highbush blueberry in Argentina. Afr. J. Biotechnol. 2014. V. 13 (51). P. 4628–4634. https://doi.org/10.5897/AJB2014.14156
Yang Q., Fan X.-L., Guarnaccia V. et al. High diversity of Diaporthe species associated with dieback diseases in China, with twelve new species described. MycoKeys. 2018. V. 39. P. 97–149. https://doi.org/10.3897/mycokeys.39.26914
Морочковский С.Ф., Зерова М.Я., Левицкая З.Г. и др. (Morochkovskiy et al.) Определитель грибов Украины. Т. 2. Киев: Наукова думка, 1969. 517 с.
Дополнительные материалы отсутствуют.
Инструменты
Микология и фитопатология


