Молекулярная биология, 2019, T. 53, № 1, стр. 109-119
Паттерн сплайсинга мРНК в клетках эпителия тимуса ограничивает транскриптом, доступный для отрицательной селекции аутореактивных Т-лимфоцитов
Е. С. Шилов a, *, Е. А. Горшкова a, А. Р. Миннегалиева a, Д. М. Поташникова a
a Биологический факультет Московского государственного университета
им. М.В. Ломоносова
119234 Москва, Россия
* E-mail: shilov_evgeny@inbox.ru
Поступила в редакцию 26.08.2018
После доработки 17.09.2018
Принята к публикации 17.09.2018
Аннотация
Для успешной отрицательной селекции аутореактивных Т-лимфоцитов необходимо представление в тимусе максимального количества эпитопов от всех возможных изоформ собственных белков организма. Один из ограничивающих отрицательную селекцию факторов – отсутствие в тимусе части возможных сплайсоформ белка (ограниченный паттерн сплайсинга). Нами показано, что примерно для 25% (87 из 339) кандидатных генов мыши, для которых в литературе описано наличие альтернативного сплайсинга, ограниченность паттерна сплайсинга может приводить к появлению не представленных в тимусе эпитопов. Десять случайно выбранных генов из 87 прошли экспериментальную проверку, для пяти из них (Add2, Dst, Golga7, Lmna, Nasp) выявлена такая неполнота паттерна сплайсинга в тимусе. Таким образом, долю генов, для которых паттерн сплайсинга в тимусе может ограничивать набор доступных отрицательной селекции эпитопов, можно оценить в 10–15%.
ВВЕДЕНИЕ
Стохастические перестройки генов Т-клеточных рецепторов позволяют получать Т-клетки с огромным разнообразием узнаваемых ими пептидных антигенов. Это потенциальное разнообразие включает в себя и рецепторы, специфические к собственным пептидам организма, иммунный ответ на которые может привести к развитию аутоиммунных заболеваний. Ключевым этапом, предотвращающим возникновение аутореактивных клеток в ходе созревания Т-лифоцитов в тимусе, служит отрицательная селекция тимоцитов. Она осуществляется с помощью презентации эпителиальными клетками тимуса аутологичных пептидов на молекулах главного комплекса гистосовместимости (MHC) и последующего апоптоза тимоцитов, проявляющих достаточно высокое сродство к таким аутоантигенам. Для более полного удаления аутореактивных Т-клеток эпителий тимуса млекопитающих экспрессирует бóльшую часть белоккодирующих генов генома. Основную роль в этом процессе играют клетки медуллярного эпителия тимуса (mTEC). Необходимость проведения отрицательной селекции приводит к наиболее широкому спектру экспрессии белоккодирующих генов в клетках mTEC из всех типов клеток организма. В результате секвенирования транскриптома выделенных путем клеточного сортинга mTEC мыши в них достоверно зарегистрирована экспрессия 19 239 белоккодирующих генов, что составляет 89% от общего числа генов в каталоге Ensemble [1]. Только 1788 белоккодирующих генов не экспрессировались в mTEC, при этом бóльшая их часть была представлена генами обонятельных рецепторов (49%) и рецепторов вомероназального органа (15%), а также генами без известной на сегодняшний день функции (22%). Таким образом, большинство потенциальных эпитопов аутореактивных Т-клеток может быть теоретически представлено в тимусе в ходе отрицательной селекции. Взаимосвязь между аутоиммунными процессами и нарушением отрицательной селекции Т-лимфоцитов уже давно выявлена для ряда заболеваний человека. Так, у людей с аутоиммунной полиэндокринопатией–кандидозом–эпидермальной дистрофией (APECED) обнаружены множественные мутации гена AIRE [2]. Традиционно именно ремодулирующий хроматин белок AIRE считался основным фактором, ответственным за экспрессию в mTEC большого числа белоккодирующих генов. Однако при анализе транскриптома нокаутных по гену Aire мышей показано, что из 15 137 работающих в mTEC генов уровень экспрессии снизился, причем неполностью, а только для 3272 [3]. Интересно, что при этом у Aire-независимых генов репертуар сплайсоформ, экспрессирующихся в тимусе, был шире, чем у животных без нокаута.
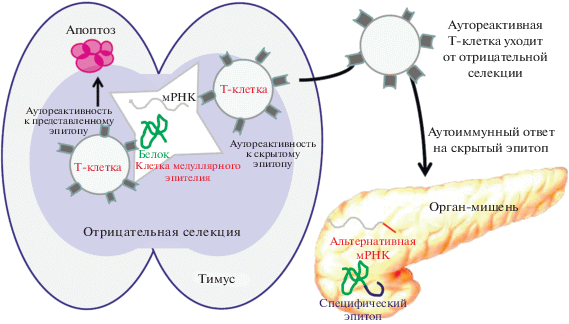
Сложный набор мРНК, возникающих в результате альтернативного сплайсинга и соответствующих разным сплайсоформам белков в разных клетках и тканях для одного и того же гена, может приводить к тому, что, несмотря на экспрессию гена в тимусе, не все его сплайсоформы будут там представлены. Гипотеза о том, что различия в паттернах сплайсинга одних и тех же пре-мРНК в тимусе и органах функциональной экспрессии могут быть причиной возникновения аутоиммунных заболеваний впервые была высказана относительно диабета первого типа и белка IA-2 поджелудочной железы [4]. В одном из первых исследований выборки белков человека, обладающих аутоиммунными эпитопами, показано, что все их гены (45/45, 100%) обладали сложным паттерном сплайсинга, а для белков было характерно наличие нескольких различающихся изоформ (сплайсоформ), в то время как в выборке контрольных генов альтернативный сплайсинг был характерен всего для 42% генов. Однако характер распределения сплайсоформ между тимусом и другими органами авторы не анализировали [5]. Из 339 исследованных тканеспецифических сплайсоформ мРНК в mTEC присутствовало только 72 (21%) [6]. Гибкость программы экспрессии генов в эпителиальных клетках тимуса позволяет презентировать тимоцитам в ходе отрицательной селекции практически любые белки, хотя по причине выраженного тканеспецифического характера сплайсинга не все эпитопы этих белков могут присутствовать в протеоме тимуса. Вследствие этого в системе отрицательной селекции может возникать “уязвимость”, благодаря которой тимус могут покидать аутореактивные Т-клетки, специфичные к тем изоформспецифическим эпитопам, которые не возникали в тимусе, либо были представлены в нем в недостаточном количестве. Эта гипотеза наглядно представлена на рис. 1.
Рис. 1.
Общая схема влияния ограничений альтернативного сплайсинга мРНК в тимусе на уход аутореактивных Т‑клеток от отрицательной селекции. Аутореактивная Т-клетка, распознающая представленный в тимусе эпитоп, в случае достаточной аффинности Т-клеточного рецептора к антигену подвергается апоптозу. Другая аутореактивная Т-клетка, распознающая не представленный в тимусе эпитоп, избегает этого и выходит из тимуса на периферию. В дальнейшем она может вызывать развитие аутоиммунного ответа в органе-мишени, где есть экспрессия не представленного в тимусе эпитопа.
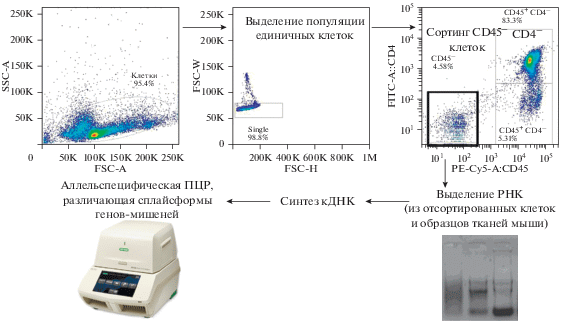
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Биоинформатические методы. Для теоретической части работы был взят перечень из 45 генов человека, имеющих альтернативный сплайсинг и аутоиммунные эпитопы, из публикации Ng и др. [5], а также список из 297 мышиных генов с различающейся сложностью паттерна сплайсинга в клетках эпителия тимуса и контрольных эпителиальных клетках из работы Keane и соавт. [6]. Для генов человека были найдены мышиные ортологи, после чего списки генов объединили, исключив гены, не кодирующие белки. В результате сформировали список из 339 генов мыши с тканеспецифичным альтернативным сплайсингом (список приведен в разделе “Результаты”). Для всех генов проверили их экспрессию в медуллярном и кортикальном эпителии тимуса по материалам работы Sansom и др. [1] (https://www.ncbi.nlm.nih.gov/ pmc/articles/PMC4248310/bin/supp_24_12_1918_ index.html), а также на основе данных об уровне экспрессии в базе ENCODE (https://www.encodeproject.org). Для изучения спектра белковых изоформ, соответствующих альтернативным сплайсоформам мРНК, использовали базы данных Uniprot (http://www.uniprot.org/) и NCBI protein (https://www.ncbi.nlm.nih.gov/protein/). Для предсказания потенциальных Т-клеточных эпитопов использовали базу данных IEDB (Immune Epitope Database, http://tools.immuneepitope. org/main/tcell/), инструменты Peptide binding to MHC class I molecules [7] и Peptide binding to MHC class II molecules [8]. Для анализа использовали гаплотип H-2b, характерный для линии мышей C57BL/6, пептиды проверяли на возможность презентации с помощью MHC-аллелей H-2Db, H-2Kb (MHC I) и H-2IAb (MHC II), пороговое значение процентиля ранга эпитопа составило 5% (Top 5%). При предсказании эпитопов в область поиска включали аминокислотную последовательность, уникальную для анализируемой сплайсоформы, а также по 10 аминокислотных остатков с ее N- и C-концов, общих с другими сплайсоформами, для того, чтобы не пропустить эпитопы, возникающие на границе двух соединяющихся экзонов. Биоинформатический анализ последовательностей проводили с использованием программы CLC Sequence Viewer 7 (“QIAGEN Bioinformatics”).
Экспериментальные методы. В работе использованы здоровые мыши линии C57BL/6, самцы и самки в возрасте 8–16 недель. После эвтаназии и препарирования из мышей выделяли тимус, селезенку, головной мозг, печень, почки, сердце, гонады, легкие и бедренные мышцы. Аликвоты клеток тимуса (1 × 106) окрашивали флуоресцентными антителами к маркерным белкам отдельных субпопуляций CD45 (клон 30-F11; флуорофор PE-Cy5), CD4 (клон GK 1.5; флуорофор FITC) и CD8 (клон 53-6.72; флуорофор APC). Для этих аликвот на проточном цитофлуориметре-сортере FACSAria SORP (“BD BioSciences”, США) провели сортинг эпителиальных клеток (CD45–, CD4–, CD8–). Из цельного тимуса, прочих органов и отсортированных клеток выделили тотальную РНК, которую использовали для синтеза кДНК и последующего определения соотношения различных сплайсоформ мРНК указанных генов-мишеней. Выделение РНК проводили по методу Хомчинского с использованием тризола [9], синтез кДНК – на основе тотальной клеточной РНК, предварительно обработанной ДНКазой I, при помощи обратной транскриптазы SuperScript III (модифицированная MMLV) и олиго(dT)-праймеров (“ThermoFisher Scientific”, США). ПЦР в реальном времени проводили с использованием сплайсоформспецифических праймеров и готовых наборов, содержащих флуоресцентный интеркалятор EVA Green (“Синтол”, Россия). Относительный уровень экспрессии сплайсоформ и их соотношение для 10 выбранных в ходе анализа кандидатных генов определяли с помощью метода ΔΔСt [10], последовательности использованных для этого праймеров приведены в табл. 1. Для амплификации в реальном времени использовали следующую программу: 3 мин при 95°C, далее 50 циклов: 30 с при 95°C, 30 с при температуре отжига (варьировала для разных пар праймеров от 56°C до 62°C, использовали градиент температур на одной плашке), 30 с при 72°C. По завершении амплификации специфичность продуктов оценивали с помощью анализа кривой плавления и агарозного гель-электрофореза. Данные по уровню экспрессии сплайсоформ сравнивали при помощи рангового U-критерия Манна–Уитни.
Таблица 1.
Праймеры для сравнения относительной экспрессии сплайсоформ
| Название | Последовательность | Название | Последовательность |
|---|---|---|---|
| G6cp2F | 5'-GCCCATGTCTTGAGCAGTTTC-3' | LMNAcomF | 5'-AGGAGGAGCTTGACTTCCA-3' |
| G6cp2shortR | 5'-TACTAGCATCCCTGTGCAGAG-3' | LMNAcomR | 5'-CCTTCTTATACTGTTCCACCTG-3' |
| G6cp2longR | 5'-CACTCCACAGAAAGGACCAG-3' | Dclk1NF | 5'-GCTTCTACCGCACCCGCAC-3' |
| GolMBPF | 5'-CTGGAAAGAAGAGAAGCGTG-3' | Dclk1NR | 5'-CGGGTCAAATCAGCCAGCAG-3' |
| GolMBPR | 5'-CCTGTCTTTGAAGGTGTTGTC-3' | Dclk1CF | 5'-GTCCACCGTGACATCAAGCC-3' |
| MBPF | 5'-ACCCATTATGGCTCCCTG-3' | Dclk1CR | 5'-CCAGATGTCCACCTTGAGGC-3' |
| MBPR | 5'-ATGGAGGTGGTGTTCGAG-3' | Dclk1AF | 5'-GCACTGGACCACGGGTTTAC-3' |
| DSTshortF | 5'-CAGGATGACAAACTTGAAAGGG-3' | Dclk1AR | 5'-AAGCAGCATATGGACTGGGTAA-3' |
| DSTshortR | 5'-GCCCTTGAAACTGAAACTCC-3' | Magi2NF | 5'-AGAAAGCGGGACCTATGAAGA-3' |
| DSTlongF | 5'-TCTCTTCCTCTTGTTGCTCC-3' | Magi2NR | 5'-CTTATTTCTTTTCCGCTTCCC-3' |
| DSTlongR | 5'-TGTTCACTCTGATGATGGAC-3' | Magi2CF | 5'-AAGAAGAGTGCGGCTGCTG-3' |
| DSTmidF | 5'-ACAAGAATTACAACCCTCAGGA-3' | Magi2CR | 5'-CAGCGGCAGCACTCCAGG-3' |
| DSTmidR | 5'-TGCTTGGACTGGTACTTCTG-3' | MagimidF | 5'-ACAAGTTTATCTGCTCCGGC-3' |
| NASP1F | 5'-ATGGAGAATGGAGTGTTGGG-3' | MagimidR | 5'-GAAAACTGGTCCTGGGTGG-3' |
| NASP1R | 5'-TTTCTCTGTCCTCTTCCTCTG-3' | Golga71F | 5'-ACACCATCTTCTTATGCATGG-3' |
| NASP2F | 5'-GTTGAGACCAAGGTAGCCC-3' | Golga71R | 5'-TCTGTGGTTTATCTTCCACTGC-3' |
| NASP2R | 5'-TTTCTTCACCCTCTTTCATCTG-3' | Golga72F | 5'-AGGTATGGCTACATTTGTGTTC-3' |
| NASP3F | 5'-AATGGAGAGTCTGGATGTGG-3' | Golga72R | 5'-GCAGACTTGTACATGCTGACC-3' |
| NASP3R | 5'-TCTAAGGCATTTCCCAACACTC-3' | Golga7cF | 5'-GGAAGGTGTTCATTCAGCG-3' |
| UBTF1F | 5'-TGACCTTATCCAGAATGCCA-3' | Golga7cR | 5'-ACAAGCCAAACAGCCTTCAAG-3' |
| UBTF1R | 5'-GTCCTTCACCTCCTTCGT-3' | Add2NF | 5'-GGGCAACAACTCCTCCAAC-3' |
| UBTF2F | 5'-GGTTTCTTCCGGTGAGGT-3' | Add2NR | 5'-GCCAGGTTCAGGGAGTCG-3' |
| UBTF2R | 5'-GCCATTTCCAGGTCTGTG-3' | Add2midF | 5'-GGATGCTGGACAATTTAGGC-3' |
| UBTF3F | 5'-GAAGAACAACCTCCCATCCA-3' | Add2midR | 5'-TTCTGCTGCTTCTGGGCG-3' |
| UBTF3R | 5'-TGAAGAAGCGGAAGTAAGGG-3' | Add2CF | 5'-GGAGAAGGACATAGCCACAGAG-3' |
| LMNA-AF | 5'-CTCTTCTGCCTCCAGTGTC-3' | Add2CR | 5'-CTCTGTGTTGGCTTCGCTGAC-3' |
| LMNA-AR | 5'-GGTCCCAGATTACATGATGC-3' | ActbF | 5'-CAGGGTGTGATGGTGGGAATG-3' |
| LMNAC2F | 5'-CGCAACACCAAGAAGGAG-3' | ActbR | 5'-CCAGAGGCATACAGGGACAGC-3' |
| LMNAC2R | 5'-GAGTTGAGAAGAGCCTCGA-3' | CycsF | 5'- CGTCTGTCTTCGAGTCCG-3' |
| CycsR | 5'-TTCCAGGGATGTACTTTTTG-3' |
Для трех кандидатных генов проанализировали экспрессию полноразмерных минорной и мажорной сплайсоформ мРНК. Выбранные для этого изоформы 1 и 2 гольджина-7 включали полную кодирующую последовательность (137 и 154 аминокислотных остатков), изоформы ламинов А и С – фрагменты с аминокислотного остатка 383 до С-конца (192 и 283 аминокислотных остатков), так как N-концевые части обеих изоформ одинаковы, изоформы 1 и 2 Nasp включали фрагменты с аминокислотного остатка 133 до С-конца (316 и 481 аминокислотных остатков) по той же причине. Соответствующие праймеры приведены в табл. 2. Для амплификации использовали универсальную для всех 6 изоформ программу: 3 мин при 95°C, далее 35 циклов: 30 с при 95°C, 30 с при 58°C, 1 мин 50 с при 72°C; после 35 цикла – 5 мин при 72°C. По завершении амплификации специфичность продуктов анализировали электрофорезом в агарозном геле.
Таблица 2.
Праймеры для специфического клонирования сплайсоформ
| Праймеры для амплификации полноразмерных форм с кДНК | |
|---|---|
| название | последовательность |
| LamTotF | 5'-TGCTGGAGGGCGAGGAGG-3' |
| LamAR | 5'-GGAGCAGACAGGAGGTGGC-3' |
| LamCR | 5'-TTTGGCATGGAGGTGGGAG-3' |
| Nasp2F | 5'-AATGTAGATGAGGAAGCAAG-3' |
| Nasp1F | 5'-GATAATGTAGATGAAACTGAAG-3' |
| NaspShR | 5'-GCCTCCTTCATCTGCTCAT-3' |
| NaspLnR | 5'-CTCGCCTCTTAACATGCAG-3' |
| GolTotF | 5'-GGCGGAGCGGCGGGTC-3' |
| Gol1R | 5'-AGGGACACTCTGCCATCGG-3' |
| Gol2R | 5'- ACGAACAGCAACCCTCCAG-3' |
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Выбор кандидатных генов для определения белков, имеющих не синтезируемые в тимусе сплайсоформы с потенциально аутореактивными эпитопами, сделан на основе опубликованных данных по секвенированию генов мыши, обладающих различием паттерна сплайсинга между эпителями тимуса и кожи [6], и списка генов, для которых у человека известны сплайсоформзависимые аутоиммунные заболевания [5]. Три некодирующих гена были исключены из анализа. После объединения двух этих источников данных проведена оценка полученного списка из 339 кандидатных генов на основе трех критериев: 1) ген имеет достаточный уровень экспрессии в тимусе (есть записи об экспрессии и в базе ENCODE, и, по материалам секвенирования эпителия тимуса в работе Sansom и соавт. [1], экспрессия превышает уровень 0.5 RPKM); 2) для гена в базе Uniprot есть несколько сплайсоформ белка; 3) IEDB предсказывает наличие в отличающей сплайсоформы части белка эпитопов для MHC I либо MHC II с рейтингом не ниже 5% в общем рейтинге эпитопов. Последовательное применение трех этих фильтров позволило разделить кандидатные гены на 4 группы (табл. 3), причем в четвертую группу вошли 87 генов, для которых было характерно: а) достаточный уровень экспрессии в тимусе; б) наличие нескольких изоформ белка, возникающих благодаря альтернативному сплайсингу; в) различия между этими изоформами по потенциальным Т-клеточным эпитопам MHC I и MHC II.
Таблица 3.
| Ген не имеет детектируемой экспрессии в тимусе (87 генов) | 1110017D15Rik, 1700001C19Rik, 1700023F06Rik, 1700102H20Rik, 3110009E18Rik, 4930511M06Rik, 4930570G19Rik, Ace2, Adam22, Amy2a5, Ank1, Arhgap6, Armc3, Atp12a, Cabyr, Cacna2d1, Cadps2, Caln1, Camk2a, Ccdc33, Chd5, Chn1, Csrnp3, Csrp3, Cyp17a1, Cyp4f14, Dlgap1, Dusp13, Fam178b, Fam187b, Fam19a5, Fam5c, Fgf12, Folr1, Fxyd2, Gad2, Gm9866, Gpm6b, Golga8b, Grip1, Igsf1, Kcne3, Kcnj6, Kif17, Kng1, Lrrc18, Lrrc36, Map6, Meg3, Mgat4c, Myt1l, Nrap, Nr1h4, Nrcam, Nrxn1, Nrxn3, Olfm3, Oprl1, Osbpl1a, Pex5l, Ptprr, Phactr3, Phyhipl, Pklr, Plcd4, Plch1, Proc, Rbfox1, Ric3, Rims2, Shrpc, Slc12a8, Slc14a2, Slc17a1, Slc22a8, Slc6a18, Spag16, Spag9, Spata17, Spdya, Ssb, Stxbp5l, Tmc5, Tmod2, Wnk3, Zfp57, Zpbp2 |
| В Uniprot есть только одна сплайсоформа белка (130 генов) | 1700028K03Rik, 1700102H20Rik, 4931406C07Rik, A330009N23Rik, A730017C20Rik, AI854517, Abi2, Acr, Aldh3a1, Aldoa, Apol9a, Apol9b, Asb8, Atxn3, Bdnf, Blm, Bnipl, Cacna1a, Cacng5, Caly, Ccdc23, Cd27, Cenpa, Cenpb, Cenpc1, Chd3, Clcn5, Cldn10, Cldn25, Clta, Cnp, Coro2a, Cttnbp2nl, Dlg3, Dph3, Dpy30, Dync1i2, Eif5a, Elavl2, Exosc10, Exosc5, Exosc9, Fbl, Ffar2, Gars, Git2, Gjc2, Gpm6a, Gpr35, Gpd2, Gtf2e2, Gria1, Hars, Hist2h2ac, Hist4h4, Hmox2, Igsf5, Islr2, Kif1a, Ldha, Lmnb1, Ly6e, Lrrc57, Map3k9, Mdk, Mea1, Mmp28, Mpo, Mtfr1, Mtm1, Myadm, Nbr1, Nip7, Nrxn2, Nsun6, Nt5c2, Numa1, Nup35, Pcdh19, Pcna, Pcyt1a, Pdpk1, Pdzrn4, Pex7, Pfkfb2, Pfkfb3, Pigp, Prtn3, Ptprj, Ptprn, Ptpro, Rai14, Rdx, Rgs17, Rhbdd1, Rhbdf2, Rpain, Rpl3l, Rplp2, Sell, Sema4a, Sirt4, Snrpa, Snrpb, Snrpd1, Snrpn, Tbc1d14, Tgm1, Tk1, Tmbim6, Tmem40, Tmsb10, Top1, Top2a, Tpo, Trim13, Trim21, Trim52, Trove2, Tsc22d4, Ttc4, Uba1, Ube2i, Usp14, Vash2, Wdfy1, Wdsub1, Wnt7b, Xrcc5, Xrcc6 |
| Сплайсоформы не различаются по предсказанным эпитопам (35 генов) | Arvcf, Arfgap1, Atp6v0a1, Cacna2d2, Calca, Cdadc1, Clasp1, Cul4b, Dctn1, Dmtn, Dync1i1, Eif4g1, Elavl4, Eya2, Fam105a, Fbxw11, Fgf14, Fchsd2, Fmnl1, Hnrnpa2b1, Lgals9, Nkain4, Ociad1, Pak3, Pifo, Pkib, R3hdm2, Serp2, Sgce, Sla, Slc8a3, Sptan1, Thpo, Tmc8, Tssk4 |
| Ген экспрессируется в тимусе, сплайсоформы различаются по набору эпитопов (87 генов) | Abr, Acbd6, Add2, Ano10, Arpp21, Asap2, Asph, Ate1, Bin1, Brdt, Cacna1c, Cant1, Capzb, Cd44, Cdkn2a, Clip4, Crem, Dbn1, Dclk1, Dclk2, Dlg1, Dlgap4, Dnmt1, Dnmt3b, Dock9, Dst, Ece2, Elmo1, Enah, Fam160a2, Far1, Fn1, Fnbp1l, G6pc2, Gabpb1, Gas7, Golga7, Gtf2i, Hecw2, Igsf9, Il1rap, Il1rl1, Kcnma1, Ldb3, Lmna, Ltbp4, Magi1, Magi2, Map4, Mbp, Mif4gd, Mtcl1, Naca, Nasp, Ncoa6, Nfe2l1, Ogdh, Oxr1, Parp6, Pcdh15, Pde1c, Pdlim5, Pla2g12a, Plec, Pnkd, Pxk, Rara, Rgs3, Rnf20, Rnf44, Rpgr, Rtn3, Sgk1, Sidt1, Slc25a25, Snap91, Speg, Srp54, Taf6l, Tmem107, Tmpo, Tnfrsf18, Tpd52, Tpm1, Ubap2l, Ubtf, Vps26a |
Из четвертой группы далее случайным образом выбрали 10 генов для экспериментального сравнения паттернов их экспрессии в тимусе и других органах мыши; эти характеристики приведены в табл. 4. Для всех этих генов характерны множественные сплайсоформы, большое число потенциальных Т-клеточных аутоантигенов и возможность дифференциального сплайсинга в тимусе.
Таблица 4.
Гены, выбранные для экспериментальной проверки паттерна сплайсинга
| Ген | Название и функции белка | Экспрессия мРНК в тимусе (RPKM) из Sanson/по ENCODE/ максимальная вне тимуса | Число сплайсоформ белка | Размеры сплайсоформ* | Паттерн альтернативного сплайсинга | Расчетное число эпитопов MHC I | Расчетное число эпитопов MHC II |
|---|---|---|---|---|---|---|---|
| Add2 | β-Аддуцин, связь актиновых филаментов со спектрином | 1.72/0.37/33 | 3 | 725/562/476 | Альтернативные C-концы |
6 | 3 |
| Dclk1 | Даблкортинподобная киназа 1, регуляция сборки микротрубочек | 3.32/0.34/35 | 14 | Варьирует от 756 до 56 | Альтернативные участки по всей длине | 6 | 2 |
| Dst | Дистонин, связь между актиновыми и промежуточными филаментами | 11/2/9 | 36 | Варьирует от 7730 до 2639 | Альтернативные участки по всей длине | >100 | >100 |
| G6pc2 | Глюкозо-6-фосфатаза, фермент глюконеогенеза | 0.54/0.004/0.164 | 4 | 355/229/154/169 | Альтернативные C-концы и N-концы | 7 | 4 |
| Golga7 | Гольджин A7, субъединица пальмитоилтрансфера-зы аппарата Гольджи | 23/18/35 | 3 | 137/134/153 | Альтернативные C-концы | 5 | 0 |
| Lmna | Ламины A/C, белки ядерной оболочки | 49/8/181 | 3 | 665/574/462 | Альтернативные C-концы и N-концы | 9 | 5 |
| Magi2 | Мембрансвязанная гуанилаткиназа | 0.78/0.14/7 | 14 | Варьирует от 1414 до 1024 | Альтернативные C-концы и N-концы | 20 | 10 |
| Mbp | Основный белок миелина, аутоантиген | 11/12/425 | 17 | Варьирует от 350 до 123 | Альтернативные участки по всей длине | 11 | 10 |
| Nasp | Ядерный антиген сперматозоидов, транспортер гистона H1 в ядро, аутоантиген | 28/2/98 | 3 | 448/773/421 | Альтернативные середины | 7 | 4 |
| Ubtf | Транскрипционный фактор для РНК-полимеразы I | 22/14/28 | 5 | 764/727/752/795/758 | Альтернативные C-концы | 13 | 8 |
Схема строения различных сплайсоформ белков, характерных для выбранных генов, а также расположение в них потенциальных Т-клеточных эпитопов MHC I и MHC II приведено на рис. 2.
Рис. 2.
Схематичное изображение выровненных последовательностей основных сплайсоформ 10 белков, выбранных для анализа уровня экспрессии методом обратной транскрипции/ПЦР в реальном времени. Для всех белков, кроме дистонина, указано положение основных предсказанных эпитопов для MHC I и MHC II (для дистонина такие эпитопы не приведены по причине его слишком большого размера). Обозначения: И – изоформы, а.к.о. – аминокислотные остатки. Эпитопы MHC I отмечены знаком (*), эпитопы MHC II – знаком (+), если два эпитопа перекрываются, то такая последовательность отмечена (*/+). Числа под выпадающими и альтернативными последовательностями показывают длину таких аминокислотных последовательностей. Для всех перечисленных белков более длинные и более короткие сплайсоформы имеют различия по числу потенциальных аутоантигенных эпитопов.
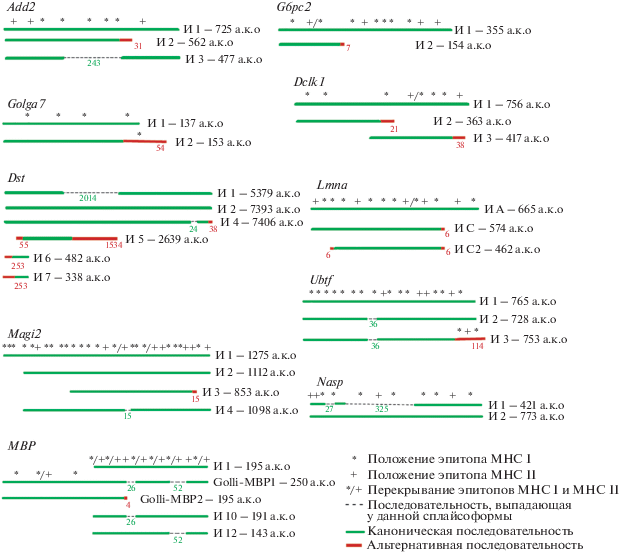
Схема эксперимента по определению соотношения различных сплайсоформ мРНК выбранных кандидатных генов приведена на рис. 3. Следует заметить, что при анализе профиля генной экспрессии в эпителии тимуса Sansom и др. [1] обнаружили, что для клеток mTEC мыши характерна экспрессия 21 763 генов, а для клеток кортикального эпителия – 19 474 генов, причем 45 из них не экспрессируются в mTEC. В результате общее число экспрессирующихся на уровне транскриптома генов в тимусе составляет 21 808. Таким образом, несмотря на общепринятый взгляд о необходимости работы только с популяцией клеток mTEC, мы пришли к выводу, что можно не делить популяцию эпителиальных клеток тимуса, поскольку кортикальный эпителий также обладает весьма широким профилем эктопической экспрессии. По этой причине при сортировке эпителиальных клеток тимуса мы не выделяли отдельно клетки кортикального и медуллярного эпителия.
Уровень экспрессии различных сплайсоформ мРНК кандидатных генов оценивали при помощи сплайсоформспецифической ПЦР в реальном времени (рис. 4). С целью исключить зависимость результатов от соотношений сплайсоформ мРНК в каждом отдельном органе в качестве контроля для эпителия тимуса использовали панель органов, в которую входили в равном соотношении РНК, выделенные из селезенки, головного мозга, печени, почек, легких, семенника и бедренной мышцы. На такой панели органов удалось показать экспрессию всех анализируемых сплайсоформ кандидатных генов, за исключением короткой изоформы 2 глюкозо-6-фосфатазы и длинных изоформ MBP (МВР Голли). В отсортированных эпителиальных клетках тимуса выявлена экспрессия всех 10 генов-мишеней, как и в сборной панели органов.
Рис. 4.
Анализ относительной экспрессии 10 генов-мишеней в тимусе и на панели органов мыши. Выявлено три гена (Golga7, Lmna, Nasp), подходящих для проверки профиля экспрессии сплайсоформ. В качестве 100 единиц относительной экспрессии взято среднее расчетное значение для двух генов нормировки: Actb (β-актин) и Cycs (цитохром с). Достоверные различия уровня экспрессии отдельных сплайсоформ в тимусе отмечены звездочками. Гены Add2 и Dst не были включены в дальнейший анализ по причине слишком низкого уровня экспрессии в тимусе мажорных сплайсоформ, гены G6pc2 и Magi2 – по причине высокого уровня экспрессии полноразмерных сплайсоформ, включающих в себя основные эпитопы. Для генов Dclk1, MBP, Ubtf не выявлено различий между уровнями экспрессии сплайсоформ в тимусе. Сокращения: иф. – изоформа, общ. – амплификация участка, общего для нескольких изоформ.
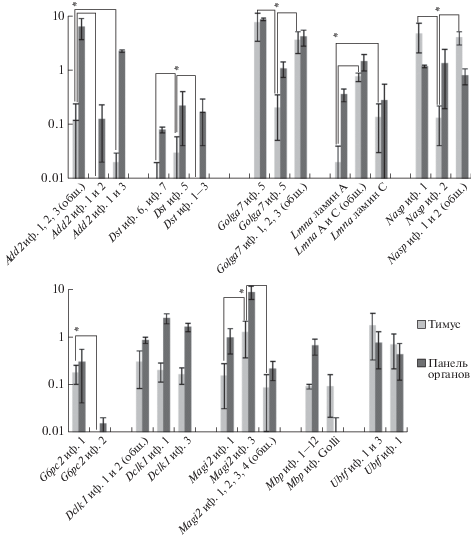
Гены удалось разделить на три группы по сложности паттерна сплайсинга в тимусе относительно других органов. Три гена (Dclk1, Mbp, Ubtf) давали сбалансированный транскриптом, сочетающий проанализированные сплайсоформы в схожих соотношениях в тимусе и других органах тела. Для семи генов (Add2, Dst, G6pc2, Golga7, Lmna, Magi2, Nasp) в эпителиальных клетках тимуса обнаружен дисбаланс в соотношении сплайсоформ мРНК – с дефицитом определенных сплайсоформ. В тоже время для глюкозо-6-фосфатазы (ген G6pc2) и киназы (ген Magi2) хорошо представлены полноразмерные формы, содержащие все потенциальные Т-клеточные эпитопы, что обеспечивает процесс отрицательной селекции в тимусе – несмотря на разницу в соотношении сплайсоформ мРНК. Для пяти генов (Add2, Dst, Golga7, Lmna, Nasp) обнаружены различия в количествах сплайсоформ в тимусе, при этом некоторые из их минорных сплайсоформ имели уникальные эпитопы, которые экспрессировались в панели органов. Мы предполагаем, что для продуктов генов с дисбалансом сплайсоформ в тимусе могут отсутствовать эпитопы, связанные с минорными изоформами более крупного размера, в частности, у аддуцина эпитопы изоформ 1 и 2, отсутствующие у изоформы 3; у дистонина эпитопы изоформ 1–4, которые на порядок крупнее выявленной изоформы 5; у ламина А/С эпитопы изоформы А; у гольджина-7 эпитопы изоформы 2; у транспортера гистона Н1 Nasp эпитопы изоформы 2. Возможно, для генов Add2 и Dst с низким уровнем экспрессии уход аутореактивных Т-лимфоцитов от отрицательной селекции связан не только с различиями в репертуаре их сплайсоформ в тимусе, но и со слишком низким содержанием соответствующих белков и недостаточным уровнем любых их МНС-презентируемых эпитопов, причем не обязательно сплайсингзависимых.
Гены Golga7, Lmna и Nasp были выделены в качестве перспективных для дальнейшего анализа профиля транскрипции в тимусе по причине того, что для них характерен сопоставимый уровень экспрессии всех сплайсоформ в панели тканей, но есть минорные сплайсоформы с достоверно отличающимся и достаточно высоким уровнем экспрессии в эпителии тимуса. Ген Add2 был исключен из дальнейшего анализа по причине низкого уровня экспрессии в эпителии тимуса даже мажорной изоформы 3 – примерно 0.02% от уровня панели нормировки. Ген Dst был исключен из дальнейшего анализа по той же причине – низкого уровня экспрессии мажорной изоформы 5, а также из-за большого числа трудно различимых минорных сплайсоформ белка и их исключительно крупного размера (свыше 5 тыс. аминокислот).
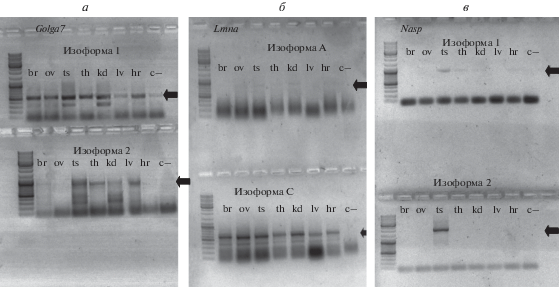
Далее проведен детальный анализ спектра экспрессии сплайсоформ трех выбраных кандидатных генов (Golga7, Lmna и Nasp) в тимусе (тотально, без выделения эпителиальных клеток) и на панели органов (расширенной путем добавления мРНК из яичника, с заменой бедренной мышцы на сердечную). Анализ полноразмерных транскриптов подтвердил перспективность выбора генов Golga7, Lmna и Nasp (рис. 5). Изоформа 1 гольджина-7 и ламин С оказались убиквитарными, в то время как изоформа 2 гольджина-7 экспрессировалась в семеннике, почке, печени и тимусе (слабее, чем изоформа 1), а ламин А – только в яичнике, семеннике и почке. Несмотря на экспрессию изоформы 2 гольджина-7 в тимусе, его следует оставить в числе генов, для которых возможен уход аутореактивных Т-клеток от отрицательной селекции, поскольку анализ полноразмерных мРНК в тимусе подтвердил значительное количественное преобладание изоформы 1 над изоформой 2, в то время как в печени преобладающей изоформой была изоформа 2. Изоформа 1 белка Nasp обнаружена, кроме семенника, в тимусе, изоформа 2 Nasp – только в семеннике, где она превалировала над изоформой 1.
Рис. 5.
Электрофоретический анализ экспрессии полноразмерных сплайсоформ мРНК генов гольджина-7 (а), ламина (б) и Nasp (в) в тимусе и других органах мыши. Сокращения: br – головной мозг, ov – яичник, ts – семенник, th – тимус, kd – почка, lv – печень, hr – сердце, c– –отрицательный контроль обратной транскрипции. Стрелки указывают на положение ПЦР-продуктов ожидаемого размера.
ЗАКЛЮЧЕНИЕ
В результате проведенного анализа выборки из 10 экспрессирующихся в тимусе генов, имеющих множественные сплайсоформы, показано, что набор вариантов мРНК в клетках эпителия тимуса действительно может ограничивать число эпитопов, доступных для отрицательной селекции аутореактивных Т-лимфоцитов. Add2, Dst, Golga7, Lmna и Nasp можно рассматривать в качестве примеров генов мыши, для белковых продуктов которых возможен уход специфических аутореактивных Т-клеток от отрицательной селекции. В то же время с некоторых генов (например, G6pc2 и Magi2) в тимусе транскрибируются наиболее крупные сплайсоформы, содержащие все потенциальные эпитопы, а для других генов (в том числе Dclk1, Mbp и Ubtf) в тимусе представлен, по-видимому, полный набор основных сплайсоформ, покрывающий эпитопы, присутствующие в периферических тканях.
В результате проведенного биоинформатического анализа примерно для 25% белков (87 из 339) выявлена возможность образования скрытых в тимусе эпитопов, поскольку: 1) их гены действительно стабильно детектируемо экспрессировались в тимусе, 2) белки имели несколько альтернативных сплайсоформ, 3) сплайсоформы отличались достаточно сильно, чтобы иметь разные наборы презентируемых MHC эпитопов. Из паттерна сплайсинга 10 экспериментально проверенных генов для 5 можно говорить о дефиците некоторых потенциальных эпитопов в тимусе. Таким образом, число генов с подобной регуляцией сплайсинга, приводящей к появлению аутореактивных Т-клеток, составляет примерно половину от 25% всех генов с альтернативным сплайсингом, то есть около 10–15%.
Работа поддержана грантом РФФИ № 16-04-01188.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Sansom S.N., Shikama-Dorn N., Zhanybekova S., Nusspaumer G., Macaulay I.C., Deadman M.E., Heger A., Ponting C.P., Hollander G.A. (2014) Population and single-cell genomics reveal the Aire dependency, relief from Polycomb silencing, and distribution of self-antigen expression in thymic epithelia. Genome Res. 24(12), 1918–1931.
Bjorses P., Aaltonen J., Horelli-Kuitunen N., Yaspo M.L., Peltonen L. (1998) Gene defect behind APECED: a new clue to autoimmunity. Hum. Mol. Genet. 7(10), 1547–1553.
St-Pierre C., Trofimov A., Brochu S., Lemieux S., Perreault C. (2015) Differential Features of AIRE-induced and AIRE-independent promiscuous gene expression in thymic epithelial cells. J. Immunol. 195, 498–506.
Diez J., Park Y., Zeller M., Brown D., Garza D., Ricordi C., Hutton J., Eisenbarth G.S., Pugliese A. (2001) Differential splicing of the IA-2 mRNA in pancreas and lymphoid organs as a permissive genetic mechanism for autoimmunity against the IA-2 type 1 diabetes autoantigen. Diabetes. 50(4), 895–900.
Ng B., Yang F., Huston D.P., Yan Y., Yang Y., Xiong Z., Peterson L.E., Wang H., Yang X.F. (2004) Increased noncanonical splicing of autoantigen transcripts provides the structural basis for expression of untolerized epitopes. J. Allergy Clin. Immunol. 114(6), 1463–1470.
Keane P., Ceredig R., Seoighe C. (2015) Promiscuous mRNA splicing under the control of AIRE in medullary thymic epithelial cells. Bioinformatics. 31(7), 986–990.
Kim Y., Ponomarenko J., Zhu Z., Tamang D., Wang P., Greenbaum J., Lundegaard C., Sette A., Lund O., Bourne P.E., Nielsen M., Peters B. (2012) Immune epitope database analysis resource. Nucleic Acids Res. 40, W525-30. doi 10.1093/nar/gks438
Wang P., Sidney J., Dow C., Mothé B., Sette A., Peters B. (2008) A systematic assessment of MHC class II peptide binding predictions and evaluation of a consensus approach. PLoS Comput. Biol. 4(4), e1000048.
Chomczynski P., Sacchi N. (1987) Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Anal. Biochem. 162(1), 156–159.
Livak K.J., Schmittgen T.D. (2001) Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25(4), 402–408.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология