Молекулярная биология, 2019, T. 53, № 2, стр. 268-273
Гомеотические гены DUX4, контролирующие развитие эмбриона человека на стадии двух бластомеров, окружены областями, которые образуют контакты с кластерами генов рРНК
О. В. Кретова a, Д. М. Федосеева a, Ю. В. Кравацкий a, И. Р. Алембеков a, И. Ю. Словохотов a, Н. А. Чуриков a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: tchurikov@eimb.ru
Поступила в редакцию 12.07.2018
После доработки 21.08.2018
Принята к публикации 25.09.2018
Аннотация
Многие гены, контролирующие развитие эмбриона и дифференцировку клеток человека, образуют хромосомные контакты с кластерами генов рРНК, вовлеченные в эпигенетическую регуляцию многих генов. Места контактов генов рРНК часто приходятся на протяженные (до 50 т.п.н.) участки, содержащие метку хроматина ‒ гистон H3K27ac, характерный для суперэнхансеров, а также на перицентромерные и субтеломерные области хромосом. Нами обнаружено, что гены DUX4, расположенные в субтеломерной области хромосомы 4 человека, окружены областями, которые особенно часто контактируют с генами рРНК. Показано, что область размером 25 т.п.н. этой хромосомы, представленная в версии hg19 секвенированной части генома человека, содержит несколько копий гена DUX4. Места контактов генов рРНК, расположенные вокруг этой области, содержат сайты метилирования, а также сайты связывания CTCF. Предполагается, что контакты генов рРНК важны для сайленсинга данных копий генов DUX4.
Трехмерные структуры хромосом играют важную роль в эпигенетической регуляции экспрессии генов. В последние годы обнаружено, что кластеры генов рРНК (рДНК) образуют многочисленные контакты с разными районами хромосом [1]. В различных линиях клеток человека эти контакты чаще всего обнаруживаются в участках 5‒50 т.п.н. суперэнхансеров, для которых характерно присутствие гистона H3K27ac ‒ метки активного хроматина [2], а также в перицентромерных, субтеломерных областях хромосом и в областях, содержащих горячие точки двухцепочечных разрывов ДНК [1]. Кроме того, гетерохроматиновые области кластеров рДНК инициируют гетерохроматинизацию в хромосомах эмбриональных стволовых клеток, а ингибирование гетерохроматинизации останавливает дифференцировку этих клеток [3]. Важную роль в этом процессе играет некодирующая, так называемая промоторная РНК (promoter associated RNA), необходимая для запуска гетерохроматинизации рДНК. Затем кластеры рДНК инициируют репрессию или активацию транскрипции во многих хромосомах, что приводит к потере плюрипотентности эмбриональных стволовых клеток [4]. Таким образом, есть все основания полагать, что обнаруженные контакты кластеров рДНК ‒ это причина, а не следствие появления активных и репрессивных меток в хромосомах человека в процессе дифференцировки клеток.
В эмбрионах животных транскрипция сначала неактивна, а затем она запускается с помощью так называемой зиготической активации генома (zygotic genome activation, ZGA). Недавно обнаружили, что к жизненно важным генам, определяющим развитие зиготы человека, относятся еще недостаточно изученные гены семейства DUX, ключевые для индукции активной транскрипции у плацентарных организмов [5]. Известно, что эти гены кодируют факторы транскрипции, содержащие по два гомеобокса, т.е. по две последовательности, кодирующие гомеодомены. Эти домены узнают специфические нуклеотидные последовательности и запускают транскрипцию определенных генов. Кроме того, с этих генов, многие из которых рассматриваются как ретрогены (гены DUX4-like, или DUX4L), т.е. как гены, возникшие в результате ретропозиций предкового гена DUX4, транскрибируются некодирующие РНК, также регулирующие многие гены человека [6]. Недавно обнаружили, что гены DUX4, расположенные на самом кончике хромосомы 4 человека, необходимы для транскрипции на стадии двухклеточного эмбриона [7].
С помощью метода 4С в данной работе выявлены контакты рДНК, локализованные вокруг субтеломерной области хромосомы 4 человека, в которой находятся гены DUX4.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
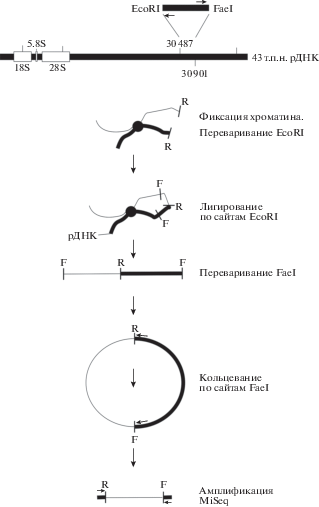
Эксперименты 4С (circular chromosome conformation capture). Препараты ДНК выделяли, как описано ранее [1]. С этой целью линию HEK293T эмбриональных клеток человека фиксировали в 1.5%-ном формальдегиде, затем выделяли препараты ядер. ДНК в фиксированном хроматине обрабатывали избытком рестриктазы EcoRI, после чего разводили препараты ДНК и проводили лигирование соседствующих фрагментов хроматина (рис. 1). Для укорачивания лигированных фрагментов ДНК использовали рестрикцию ферментом FaeI. После кольцевания разбавленных препаратов ДНК по сайтам FaeI проводили гнездовую ПЦР-амплификацию, используя рДНК-специфичные праймеры, расположенные в некодирующей области генов рДНК, как описано ранее: 5'-GCCTAAGCCTGCTGAGAACTTTC-3', 5'-CAGCATTCTGTAGGGAGATCAAATC-3' (для ПЦР-2) и 5'-TCTTTGAAAAAAATCCCAGAAGT-GGT-3', 5'-AAGTCCAGAAATCAACTCGCCAGT-3' (для ПЦР-1) [1]. Амплифицированную ДНК использовали для приготовления библиотек и глубокого секвенирования. “Сырые” данные секвенирования двух биологических реплик помещены в базу данных NCBI (accession number GSE49193).
Рис. 1.
Схема экспериментов 4С. Вверху показан рибосомный повтор длиной 42 999 п.н., содержащий четыре сайта EcoRI (черточки вверху). EcoRI-сайт, расположенный у координаты 30 487, был выбран для экспериментов 4С. Ниже показан ближайший к нему сайт FaeI (координата 30 901). R – EcoRI-сайты. F – FaeI-сайты. На схеме жирной линией выделена последовательность рДНК, а тонкой – любая область генома, которая контактирует с генами рДНК. Схематически показаны праймеры, которые использовали при ПЦР-2.
Картирование чтений 4C-рДНК. Нуклеотидные последовательности, полученные после глубокого секвенирования, фильтровали и обрабатывали с использованием нескольких программ. На первом этапе длину и качество чтений глубокого секвенирования оценивали с помощью программы FastQC [8], после чего данные фильтровали с помощью программы Cutadapt [9]. Места контактов генов рДНК в хромосомах человека, определенные методом 4С-рДНК, просматривали, используя браузеры IGB и UCSC.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Эпигенетические метки области, содержащей гены DUX4 в хромосоме 4
В подробно аннотированных текущих версиях генома человека (hg19 и hg38) субтеломерная область хромосомы 4 человека содержит восемь генов DUX, хотя в аннотациях разных версий генома имеются и заметные разночтения (не указано). На рис. 2 представлена область 100 т.п.н., находящаяся на самом кончике хромосомы 4.
Рис. 2.
Физическая карта района 100 т.п.н. субтеломерной области хромосомы 4 человека (координаты по версии hg19). Вверху указаны места контактов данной области с генами рДНК. По оси Y указано число картированных чтений глубокого секвенирования – 4C-рДНК. 1K – одна тысяча чтений. Гены DUX расположены в районе 25 т.п.н., окруженном местами этих контактов. Названия генов DUX приведены, как указано в браузере UCSC. Ниже приведены эпигенетические метки данной области, включающие H3K27ac, сайты связывания CTCF, а также области метилирования ДНК.
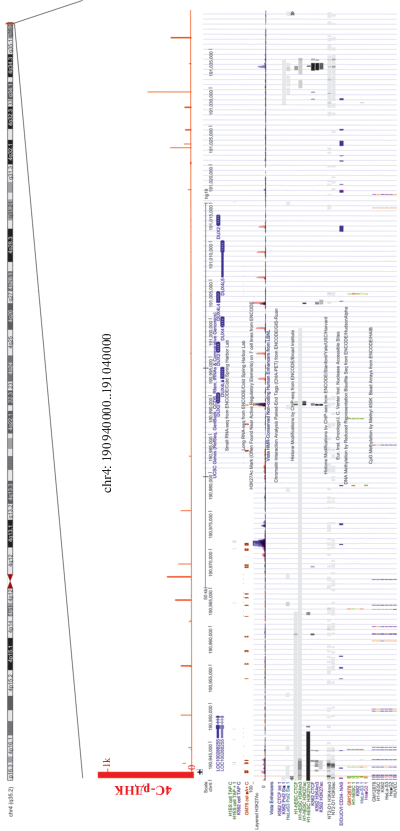
Видно, что генам DUX соответствуют небольшие пики меток активного хроматина H3K27ac на его коротких отрезках. Эти перекрывающиеся пики ацетилированного гистона H3 обнаружены в семи линиях клеток человека разного происхождения: в линии лимфобластомы (GM12878), в эмбриональных стволовых клетках (H1-hESC), клетках скелетных мышц (HSMM), эндотелия пупочной вены (HUVEC), клетках миелогенного лейкоза (K562), кератиноцитах (NHEK) и фибробластах легкого (NHLF). Слабые пики обнаружены в основном в лимфобластах (красный цвет). Они приходятся на 5'-концевые области генов DUX. В остальных линиях пики H3K27ac практически отсутствуют. Такое распределение метки H3K27ac не характерно для активных суперэнхансеров [1]. Кроме того, 5'-концевой ген DUX содержит сайт связывания CTCF и H3K27me3 ‒ метку репрессированного хроматина.
Область, содержащая гены DUX4, окружена местами контактов с генами рДНК
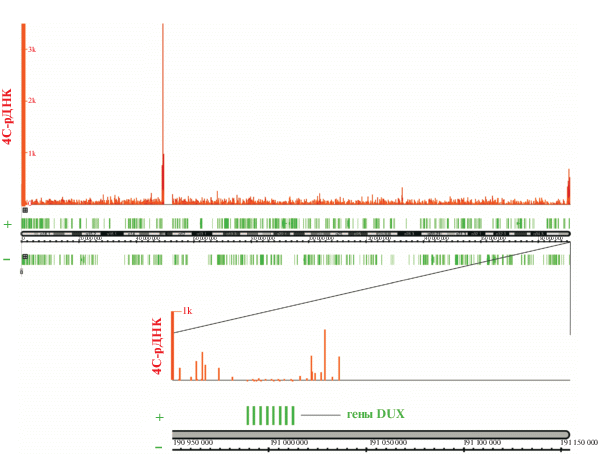
На расстоянии нескольких тысяч пар нуклеотидов вокруг генов DUX находятся участки, контактирующие с генами рДНК. Всего на хромосоме 4 найдены две области наиболее частых контактов с генами рДНК: в перицентрометром районе (около координаты 49 млн.п.н.) и в субтеломерной области [1]. Это хорошо видно на рис. 3, на котором показана вся хромосома 4 и места контактов генов рДНК в ней. На нижней части рис. 3 видны редкие места контактов в области самих генов DUX и несколько сайтов довольно частых контактов вокруг генов. Такой паттерн контактов убедительно свидетельствует о том, что методом 4С картированы именно сближенные петли хроматина, а не произошло случайное лигирование удаленных фрагментов хроматина после переваривания рестриктазой EcoRI. Кластеризация сайтов контактов генов рДНК характерна и для других районов хромосом человека [1].
Рис. 3.
Физическая карта целой хромосомы 4 (версия генома hg19) и места контактов генов рДНК (4C-рДНК). Ниже детально показана субтеломерная область с 5'-концевыми генами на плюс-цепи хромосомы. Видны редкие места контактов в области генов DUX и частые места контактов вокруг них. “+” и “–” – гены, аннотированные на плюс- и минус-цепях хромосомы 4 соответственно.
Интересно, что области контактов генов рДНК, указанные на рис. 2, содержат характерные для них метки, как и описанные ранее в других районах хромосом человека [1] – пики H3K27ac, сигналы ChIA-PET, указывающие на участие соответствующих районов в межхромосомных контактах, а также места связывания CTCF. Однако все эти метки, хотя и присутствуют в указанной субтеломерной области хромосомы 4, но они значительно ослаблены по сравнению с другими районами контактов генов рДНК, в которых расположены транскрипционно активные гены [1]. Кроме того, проксимальная область контактов обогащена сайтами метилирования ДНК. Видно также, что в клетках H1-hESC эта область содержит протяженную метку репрессированного хроматина – H3K27me3. Взятые вместе, эти результаты свидетельствуют о том, что контакты рДНК могут вызывать сайленсинг генов DUX. Это согласуется с данными, согласно которым гены DUX транскрипционно активны лишь в герминальных клетках, тогда как в соматических клетках они подвергаются эпигенетическому сайленсингу [6]. Известно, что гены рДНК могут индуцировать как сайленсинг, так и активацию транскрипции в генах-мишенях [4]. Данные, полученные нами на клетках HEK293T, свидетельствуют о том, что контакты генов рДНК в субтеломерном районе хромосомы 4, содержащем гены DUX, могут быть вовлечены в индукцию сайленсинга.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Гены DUX довольно вариабельны. Число копий тандемных повторов 3.3 т.п.н. (D4Z4-повторы), которые содержат эти гены на конце хромосомы 4, варьирует у разных индивидов в широких пределах ‒ от 11 до 150 [10]. Нарушение регуляции генов DUX4 в мышцах вызывает так называемую плече-лопаточно-лицевую миопатию (facioscapulohumeral dystrophy, FSHD), при которой наблюдается слабость мышц плечевого пояса [11]. Это заболевание обусловлено неполной репрессией повторов D4Z4, содержащих гены DUX4 [12]. Нарушение репрессии связывают с изменениями в экспрессии малых некодирующих РНК [12]. Эти данные согласуются с нашими неопубликованными результатами, согласно которым контакты генов рДНК с генами-мишенями осуществляются с помощью малых РНК разной природы. Становится понятным, почему мы обнаружили контакты рДНК преимущественно вокруг генов DUX – сайленсингу подвергается вся область повторов D4Z4.
Недавно показали, что в регуляции активности генов DUX и генов рДНК в раннем развитии и в определении идентичности эмбриональных стволовых клеток человека участвуют элементы LINE1 [7]. Это свидетельствует о сложности механизмов эпигенетического сайленсинга и активации больших групп генов. Необходимо дальнейшее изучение контактов генов рДНК и генов-мишеней, что важно как для понимания механизмов эпигенетической регуляции, так и для разработки подходов к генотерапии.
Исследование выполнено при поддержке Российского научного фонда (грант № 18-14-00122). Обработка данных глубокого секвенирования поддержана Программой фундаментальных исследований государственных академий на 2013–2020 годы (тема № 0103_2014_0005).
Список литературы
Tchurikov N.A., Fedoseeva D.M., Sosin D.V., Snezhkina A.V., Melnikova N.V., Kudryavtseva A.V., Kravatsky Y.V., Kretova O.V. (2015) Hot spots of DNA double-strand breaks and genomic contacts of human rDNA units are involved in epigenetic regulation. J. Mol. Cell. Biol. 7, 366‒382. doi 10.1093/jmcb/mju038
Hnisz D., Abraham B.J., Lee T.I., Lau A., Saint-André V., Sigova A.A., Hoke H.A., Young R.A. (2013) Super-enhancers in the control of cell identity and disease. Cell. 155, 934–947.
Feinberg A.P. (2014) The nucleolus gets the silent treatment. Cell Stem Cell. 15, 675–676. doi 10.1016/ j.stem.2014.11.017
Savić N., Bär D., Leone S., Frommel S.C., Weber F.A., Vollenweider E., Ferrari E., Ziegler U., Kaech A., Shakhova O., Cinelli P., Santoro R. (2014) lncRNA maturation to initiate heterochromatin formation in the nucleolus is required for exit from pluripotency in ESCs. Cell Stem Cell. 15, 720‒734. doi 10.1016/ j.stem.2014.10.005
De Iaco A., Planet E., Coluccio A., Verp S., Duc J., Trono D. (2017) DUX-family transcription factors regulate zygotic genome activation in placental mammals. Nat. Genet. 49, 941‒945. doi 10.1038/ng.3858
van der Maarel S.M., Tawil R., Tapscott S.J. (2011) Facioscapulohumeral muscular dystrophy and DUX4: breaking the silence. Trends Mol Med. 17, 252–258. doi 10.1016/j.molmed.2011.01.001
Percharde M., Lin C.J., Yin Y., Guan J., Peixoto G.A., Bulut-Karslioglu A., Biechele S., Huang B., Shen X., Ramalho-Santos M. (2018) A LINE1-nucleolin partnership regulates early development and ESC identity. Cell. 174, doi 10.1016/j.cell.2018.05.043
Andrews S. (2010) FastQC: a quality control tool for high throughput sequence data. http://www.bioinformatics.babraham.ac.uk/projects/fastqc.
Martin M. (2011) Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet. J. 17, 10–12.
Vizoso M., Esteller M. (2012) The activatory long non-coding RNA DBE-T reveals the epigenetic etiology of facioscapulohumeral muscular dystrophy. Cell Res. 22, 1413‒1415. doi 10.1038/cr.2012.93
Whiddon J.L., Langford A.T., Wong C.J., Zhong J.W., Tapscott S.J. (2017) Conservation and innovation in the DUX4-family gene network. Nat. Genet. 49, 935‒940. doi 10.1038/ng.3846
Lim J.W., Wong C.J., Yao Z., Tawil R., van der Maa-rel S.M., Miller D.G., Tapscott S.J., Filippova G.N. (2018) Small noncoding RNAs in FSHD2 muscle cells reveal both DUX4- and SMCHD1-specific signatures. Hum. Mol. Genet. 15, 2644–2657. doi 10.1093/hmg/ ddy173
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


