Молекулярная биология, 2019, T. 53, № 2, стр. 274-281
Rpn4-подобный белок Candida glabrata комплементирует делецию гена RPN4 у Saccharomyces cerevisiae
Д. С. Карпов a, b, *, Е. Н. Гринева a, С. В. Киселева a, Е. С. Челарская a, Д. С. Спасская a, В. Л. Карпов a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Научно-исследовательский институт биомедицинской химии им. В.Н. Ореховича
119121 Москва, Россия
* E-mail: aleom@yandex.ru
Поступила в редакцию 03.09.2018
После доработки 06.10.2018
Принята к публикации 08.10.2018
Аннотация
Экспрессия протеасомных генов в клетках дрожжей Saccharomyces cerevisiae координировано регулируется системой, состоящей из фактора транскрипции ScRpn4 и его сайта связывания, названного PACE. Ранее мы показали, что, несмотря на незначительное общее сходство с ScRpn4, Rpn4-подобные белки биотехнологически значимых видов дрожжей Komagataella pfaffii (Pichia pastoris), Yarrowia lipolytica и Debaryomyces hansenii способны комплементировать делецию RPN4 у S. сerevisiae. Условно-патогенные дрожжи Candida glabrata также имеют ген Rpn4-подобного белка, однако экспериментально этот белок не охарактеризован. В настоящей работе показано, что в гетерологичной системе экспрессии S. cerevisiae ортолог ScRpn4 из C. glabrata восстанавливает устойчивость к стрессовым воздействиям и уровень мРНК протеасомных генов у мутантного штамма S. cerevisiae с делецией гена SCRPN4. Кроме того, уникальная N-концевая область необходима для функционирования CgRpn4. Полученные нами данные указывают на то, что CgRpn4 служит активатором транскрипции протеасомных генов, а модельная система S. cerevisiae может использоваться в дальнейшем структурном и функциональном анализе этого белка.
ВВЕДЕНИЕ
Дрожжи Candida glabrata относятся к условно-патогенным микроорганизмам, нередко обитающим на слизистых оболочках в качестве компонента нормальной микрофлоры человека. Тем не менее, в ряде случаев, в частности у пациентов с иммунодефицитом, C. glabrata может вызывать кандидоз. По статистике C. glabrata становится причиной 15‒25% всех случаев кандидозов [1]. C. glabrata, принадлежащие к семейству Saccharomycetaceae класс Saccharomycetes, филогенетически более близки к пекарским дрожжам Saccharomyces cerevisiae, чем к возбудителям кандидоза, например C. albicans [2].
Причины вирулентности C. glabrata, по-видимому, частично связаны с повышенной устойчивостью этих микроорганизмов к действию активных форм кислорода [1]. Также C. glabrata характеризуется устойчивостью к азолам (например флюконазолу) – фунгицидам, нарушающим биосинтез эргостерола, компонента клеточных мембран. Одним из ключевых факторов формирования устойчивости к лекарственным препаратам и окислительному стрессу является Pdr1p [3]. У дрожжей S. cerevisiae экспрессия гена этого фактора транскрипции регулируется Rpn4p, в то же время Pdr1p служит трансактиватором гена RPN4, который отвечает за устойчивость ко многим типам неблагоприятных воздействий – тепловому шоку, окислительному и ДНК-повреждающему стрессу [4].
Фактор транскрипции Rpn4 впервые был охарактеризован нами в сотрудничестве с коллегами из Германии как компонент системы координированной регуляции экспрессии протеасомных генов [5, 6]. 26S протеасома – это АТР-зависимый мультисубъединичный протеазный комплекс, который отвечает за деградацию большинства внутриклеточных белков, в том числе поврежденных и неправильно свернутых. В промоторах почти всех генов, кодирующих субъединицы протеасомы, обнаружен регуляторный элемент PACE (Proteasome Associated Control Element, 5'-GGTGGCAAA-3'), с которым связывается Rpn4. Фактор Rpn4 S. cerevisiae (ScRpn4) регулирует не только протеасомные гены, но и множество других, включая гены компонентов различных антистрессовых ответов [7].
В ходе биоинформатического анализа геномов различных организмов PACE-подобные элементы и гены, кодирующие ScRpn4-подобные белки, были выявлены только у дрожжей класса Saccharomycetes [2]. Ранее мы показали, что ScRpn4-подобные белки таких биотехнологически значимых дрожжей, как Yarrowia lipolytica, Komagataella pfaffii (Pichia pastoris) и Debaryomyces hansenii способны восстанавливать уровень мРНК протеасомных генов и устойчивость к стрессовым воздействиям штамма S. cerevisiae с делецией гена SCRPN4 [8, 9]. Ортолог этого гена найден и у C. glabrata. Показано, что экспрессия CGRPN4 увеличивается в условиях окислительного стресса, в связи с чем возникает предположение о его участии в устойчивости к неблагоприятным условиям по аналогии с белком S. cerevisiae [3, 10]. Вероятно, этот фактор транскрипции вовлечен в активацию программы, направленной на нейтрализацию активных форм кислорода. Однако экспериментально белок CgRpn4 до сих пор не охарактеризован.
В настоящей работе ген ортолога Rpn4 C. glabrata клонирован и экспрессирован в штамме S. cerevisiae с делецией собственного гена RPN4. Установлена способность белка CgRpn4 обеспечивать устойчивость к различным видам стресса, включая окислительный стресс, и восстанавливать экспрессию PRE1, кодирующего важную структурную субъединицу 20S протеасомного субкомплекса S. cerevisiae. Установлено также, что делеция участка гена, кодирующего N-концевую область, приводит к инактивации CgRpn4.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы дрожжей и условия роста клеток. В работе использовали штаммы S. cerevisiae BY4742 (MATα; his3∆1; leu2∆0; lys2∆0; ura3∆0) дикого типа (“Euroscarf”, Германия) и BY4742 rpn4-∆ (Matα; his3∆1; leu2∆0; lys2∆0; ura3∆0; YDL020c::kanMX4) c делецией гена RPN4 (“Euroscarf”). Клетки дрожжей растили при 30°C на среде YPD (2% глюкозы, 2% пептона, 1% дрожжевого экстракта) и трансформировали, используя литий-ацетатный метод [11]. Трансформированные клетки растили на селективной среде, содержащей YNB, смесь аминокислот Dropout media, аденин и 2% глюкозы без урацила (все компоненты производства “Sigma”, США).
Получение плазмидных конструкций. Получение плазмиды YCp-Lac36-CGRPN4. Участок гена CAGL0K01727g, кодирующий предполагаемый ортолог ScRpn4, амплифицировали с геномной ДНК C. glabrata HTL [12], выделенной методом встряхивания со стеклянными шариками (“Sigma”) [13], c праймерами CgRpn4-NcoI-F и CgRpn4-XhoI-R (табл. 1). Амплифицированный фрагмент очищали и клонировали в плазмиду YCpLac36-SCRPN4 (табл. 1) по сайтам NcoI и XhoI вместо гена SCRPN4.
Таблица 1.
Плазмиды, использованные в работе
| Плазмида | Характеристика плазмиды | Источник |
|---|---|---|
| YCpLac36 | Низкокопийный центромерный дрожжевой вектор | [14] |
| YCpLac36-SCRPN4 | Экспрессионная плазмида, производная YСpLac36, кодирующая ген RPN4 S. cerevisiae, под контролем промотора SCRPN4 | [14, 15] |
| YCpLac36-ΔN210-SCRPN4 | Экспрессионная плазмида, производная YСpLac36-SCRPN4, кодирующая ScRpn4 с делецией 210 N-концевых аминокислот | [9] |
| YCp-Lac36-CGRPN4 | Экспрессионная плазмида, производная YСpLac36-SCRPN4, кодирующая Rpn4 C. glabrata | Данная работа |
| YCp-Lac36-ΔN177-CGRPN4 | Экспрессионная плазмида, производная YСpLac36-SCRPN4, кодирующая CgRpn4 с делецией 177 N-концевых аминокислот | » |
Получение плазмиды YCp-Lac36-ΔN177-CGRPN4. Фрагмент гена CAGL0K01727g, кодирующий CgRpn4 без первых 177 N-концевых аминокислотных остатков, амплифицировали с геномной ДНК C. glabrata HTL c праймерами CgRPN4-ΔN177-NcoI-F и CgRpn4-XhoI-R и клонировали в YCpLac36-SCRPN4 по сайтам NcoI и XhoI.
Присутствие клонированных фрагментов ДНК проверяли с помощью ПЦР с колоний Escherichia coli и рестрикционным анализом плазмид, выделенных из ПЦР-положительных колоний. Нуклеотидную последовательность клонированных фрагментов подтверждали секвенированием.
Определение устойчивости дрожжей к стрессу с помощью серии разведений культуры на чашках Петри. Культуры клеток дрожжей, выращенные в течение ночи на жидкой селективной среде, разводили до оптической плотности ОD600 = 1. Далее готовили серию из последовательных десятикратных разведений суспензии клеток в воде и высевали на твердую селективную среду без урацила с добавлением стрессовых агентов: ацетата кадмия, MMS, 4-NQO и циклогексимида. Тест на устойчивость к L-канаванину проводили на селективной среде без урацила и аргинина (все реактивы производства “Sigma”). В контроле штаммы растили на среде без добавления стрессовых агентов. Устойчивость штаммов дрожжей к стрессу определяли качественно по скорости формирования колоний.
Определение относительного уровня мРНК. Культуры клеток дрожжей, выращенные в течение ночи на жидкой селективной среде, разводили до ОD600 = 0.2 и подращивали в течение 4 ч. Стресс индуцировали, добавляя 4-NQO до конечной концентрации 2 мкг/мл и инкубируя в течение еще 45 мин. Клетки осаждали центрифугированием, суммарную РНК выделяли с использованием горячего кислого фенола [16]. кДНК синтезировали с использованием обратной транскриптазы RevertAid H-minus (“Thermo Scientific”, США) по протоколу, рекомендованному производителем, с олиго(dT) в качестве затравки. Относительное количество мРНК протеасомного гена PRE1 определяли методом ПЦР в реальном времени с флуоресцентным красителем Eva Green (“Синтол”, Россия) на приборе LightCycler-480-II (“Roche Life Science”, США) в соответствии с рекомендациями производителя. Референсным геном служил ген актина (ACT1). Первичную обработку данных проводили с помощью программного обеспечения, поставляемого с прибором. Дальнейшую обработку проводили с помощью Microsoft Excel. Олигонуклеотиды, последовательности которых представлены в табл. 2, подбирали с помощью сервера mFold [17].
Таблица 2.
Олигонуклеотиды, использованные в работе
| Олигонуклеотид | Нуклеотидная последовательность 5' → 3' |
|---|---|
| CgRpn4-NcoI-F | GAGAGAGAACCATGGCGTCTATAGATTTGGGACT |
| CgRpn4-XhoI-R | GAGAGAGAACTCGAGTTATGCAGTGACAAATCCGAT |
| CgRPN4-ΔN177-NcoI-F | GAGAGAGAACCATGGACGACGATGACCTGAGTGA |
| PRE1-EX-RT-F | ACTACTGAGGAGGGTTTAG |
| PRE1-EX-RT-R | CTTATGCCATCTTTATCCACGA |
| ACT1-ex-RT-F | CCTTCTGTTTTGGGTTTGGAATC |
| ACT1-ex-RT-R | TGGAGCCAAAGCGGTGATTTCCT |
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
CgRpn4 имеет слабое сходство с ScRpn4 вне ДНК-связывающего домена
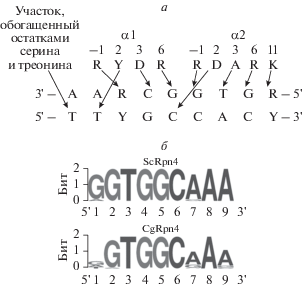
В ходе биоинформатического анализа установлено, что общее сходство аминокислотных последовательностей белков CgRpn4 (XP_448300.1, кодируемого геном CAGL0K01727g), и ScRpn4 составляет 48.1% (рис. 1). К участкам, имеющим наибольшее локальное сходство, можно отнести N-концевой фрагмент (остатки 1‒21), входящий в состав сигнала убиквитиннезависимого протеолиза у ScRpn4 [18], и С-концевую область, содержащую предполагаемый ДНК-связывающий домен, представленный “цинковыми пальцами” (N-ZnF и C-ZnF). В центральной части CgRpn4, как и у ScRpn4, можно выделить два участка, обогащенных остатками глутаминовой и аспарагиновой кислот (так называемые кислые домены). N-концевые кислые домены (NAD) этих белков хорошо сопоставлены в выравнивании, тогда как С-концевые (CAD) не имеют значимого сходства и удалены друг от друга. Отметим, что в целом N-концевая область CgRpn4 не обладает значимым сходством с N-концевой областью ScRpn4, содержащей сильный трансактиваторный домен [19]. Примечательно, что в N-концевой области CgRpn4 обнаружен участок, обогащенный остатками глутамина (QRR), который, возможно, входит в состав потенциального глутамин-богатого трансактиваторного домена.
Рис. 1.
Выравнивание аминокислотных последовательностей CgRpn4 и ScRpn4. Глобальное выравнивание выполняли с помощью программы EMBOSS-Stretcher (http://www.ebi.ac.uk/Tools/psa/emboss_stretcher/) [20] с параметрами по умолчанию. В вычислениях использовали стандартную матрицу сопоставления аминокислотных остатков BLOSUM62. Полужирным выделены биологически значимые участки. Вертикальная черта (|) соответствует идентичным аминокислотным остаткам, двоеточие (:) – сходным (положительное значение в матрице BLOSUM62), точка (.) – несходным остаткам (отрицательное значение в матрице BLOSUM62). Подчеркнуты и пронумерованы аминокислотные остатки распознающих α-спиралей цинковых пальцев ScRpn4.

Одна из особенностей структуры Rpn4-подобных белков – необычный N-ZnF. Этот участок не выявляется онлайн программами, идентифицирующими домены цинковых пальцев, например (http://zf.princeton.edu/ [21]), тогда как стандартный С-концевой цинковый палец типа C2H2 и распознаваемый им фрагмент 3'-GGTG-5' PACE-элемента предсказываются правильно. Предполагаемые контакты между цинковыми пальцами выведены в соответствии с системой правил распознавания ДНК доменами цинковых пальцев, так называемого “кода цинковых пальцев”, которые, в свою очередь, определены в ходе анализа кристаллографических данных комплексов цинковых пальцев с распознаваемыми элементами ДНК [21–23]. Мы предположили, что в распознавании фрагмента 5'-ААА-3' по комплементарной цепи участвуют как N-ZnF, так и прилегающий к нему участок, обогащенный остатками серина и треонина.
Высокое сходство ДНК-связывающих доменов и особенно остатков, предположительно образующих контакты с ДНК (рис. 1, 2а), должно обеспечивать распознавание сходных элементов. Действительно, консенсус элемента, обнаруженного в промоторах протеасомных генов C. glabrata [2], очень похож на PACE-элемент S. cerevisiae (рис. 2б). Отметим, что некоторые отличия в положениях 1 и 7 консенсуса коррелируют с наличием аминокислотных замен возле остатка тирозина в 2-ом положении распознающей спирали N-ZnF и остатка лизина в положении 11 распознающей спирали C-ZnF (рис. 1). Известно, что аминокислотные остатки, находящиеся рядом с распознающими остатками, влияют на специфичность узнавания последовательности ДНК доменами цинковых пальцев [22].
Рис. 2.
Предполагаемые “код цинковых пальцев” ScRpn4 и консенсусы элементов, узнаваемых ScRpn4 и CgRpn4. а – Схема предполагаемых контактов аминокислотных остатков распознающих ДНК α-спиралей цинковых пальцев ScRpn4 с консенсусом его сайта связывания. Указаны ключевые аминокислотные остатки распознающих α-спиралей цинковых пальцев ScRpn4 (см. рис. 1). Контакты предсказаны в соответствии с системой правил распознавания ДНК доменами цинковых пальцев [21–23]. б – Логотип консервативности нуклеотидов регуляторных элементов в промоторных областях протеасомных генов, с которыми взаимодействуют CgRpn4 и ScRpn4, построенный с помощью программы WebLogo [24] на основе последовательностей, приведенных в [2].
CgRpn4 восстанавливает устойчивость штамма S. cerevisiae с делецией гена SCRPN4 к различным видам стресса
Делеция гена SCRPN4 делает клетки дрожжей гиперчувствительными к различным видам стресса (рис. 3а).
Рис. 3.
CgRpn4 комплементирует делецию RPN4 у S. cerevisiae. а – Экспрессия CGRPN4 в штамме rpn4-Δ восстанавливает устойчивость мутантного штамма к действию различных стрессовых агентов. Обозначения токсичных соединений: CAN – канаванин, аналог лизина; Сd2+ – CdCl2; CHX – циклогексимид, ингибитор трансляции; 4-NQO – ДНК-окисляющее соединение; AsIII – метаарсенит натрия NaAsO2, вызывает окислительный стресс. б – Экспрессия CGRPN4 в штамме rpn4-Δ восстанавливает уровень мРНК PRE1 как в норме, так и при действии 4-NQO. Ген ACT1 использовали в качестве референсного. Нормированное значение уровня мРНК PRE1 в штамме rpn4-Δ в нормальных условиях принято за единицу. Представлены средние значения трех независимых опытов. Разброс данных представлен стандартным отклонением. WT – штамм дикого типа; rpn4-Δ – штамм с делецией гена SCRPN4; ScRpn4 – штамм rpn4-Δ, трансформированный плазмидой YCpLac36-SCRPN4; ScRpn4 – штамм rpn4-Δ, трансформированный плазмидой YCpLac36-CGRPN4.
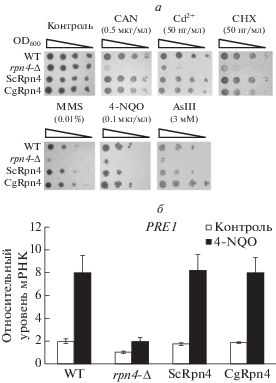
В результате недостаточной активности протеасом в штамме с делецией гена SCRPN4 клетки становятся чувствительными к воздействиям, которые вызывают накопление неправильно свернутых белков – тепловому шоку и токсичным аналогам аминокислот, таким как азетидин (аналог пролина) и канаванин (аналог аргинина), а также к действию мышьяка. Кроме того, в штамме с делецией гена SCRPN4 нарушена работа систем репарации ДНК, что снижает его жизнеспособность при воздействии ДНК-повреждающих агентов, таких как MMS и 4-NQO [14]. Повреждение ДНК тесно связано с окислительным стрессом, так как сопутствующее нарушение работы митохондрий приводит к образованию активных форм кислорода. Свободные радикалы, в свою очередь, также вызывают повреждение ДНК. В отсутствие фактора Rpn4 снижается экспрессия YAP1 – основного сенсора окислительного стресса в клетках дрожжей [7]. Помимо прямых окислителей (пероксидов) окислительный стресс может индуцироваться ионами тяжелых металлов (кадмий) и металлоидов (мышьяк), которые приводят к увеличению концентрации активных форм кислорода в клетке [25, 26].
Комплементация делеции гена SCRPN4 в штамме S. cerevisiae путем введения в плазмиды YCpLac36-SCRPN4, кодирующей полноразмерный белок под контролем собственного промотора, восстанавливает нормальный фенотип клеток в условиях стресса, вызваного канаванином, кадмием, мышьяком, циклогексимидом и ДНК-повреждающими агентами – MMS и 4-NQO (рис. 3а). Трансформация плазмидой YCp-Lac36-CGRPN4, кодирующей ортолог Rpn4 из C. glabrata, также приводит к восстановлению устойчивости к указанным стрессовым агентам (рис. 3а). Таким образом, функциональный анализ указывает на первостепенную роль гена CGRPN4 в обеспечении устойчивости клеток к окислительному, протеотоксическому и ДНК-повреждающему стрессу.
CgRpn4 восстанавливает экспрессию протеасомного гена PRE1 в штамме с делецией SCRPN4
Белок Rpn4 идентифицирован как фактор транскрипции, обеспечивающий координированную экспрессию генов, кодирующих субъединицы 26S протеасомы [5, 6]. В условиях действия стресса, индуцирующего экспрессию SCRPN4, например при повреждении ДНК, происходит накопление белка Rpn4 и последующая активация экспрессии его генов-мишеней, включая протеасомные гены [27]. В штамме с делецией гена SCRPN4 уровень мРНК гена PRE1, кодирующего субъединицу 20S протеолитического субкомплекса протеасомы, снижен в нормальных условиях роста, и наблюдается его слабая индукция в присутствии ДНК-окисляющего агента 4-NQO (рис. 3б). Введение в мутантный штамм полноразмерной копии как собственного гена, так и ортолога из C. glabrata восстанавливает уровень мРНК гена PRE1 как в норме, так и в присутствии 4-NQO (рис. 3б). Это означает, что белок CgRpn4 способен связываться с промоторами генов-мишеней Rpn4, включая протеасомный ген PRE1, и выполнять функцию активатора транскрипции.
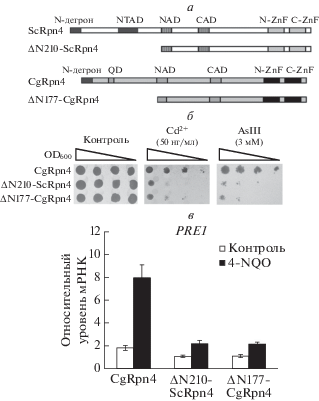
N-концевая область CgRpn4 содержит трансактиваторный домен и необходима для функционирования белка
Основной трансактиваторный домен ScRpn4 находится в пределах 210 N-концевых аминокислот. Делеция этой области приводит к полной утрате активности фактора транскрипции [19]. Соответствующий ей N-концевой участок CgRpn4 продолжается до NAD-домена и составляет 177 аминокислот (рис. 4а). Делеция соответствующего фрагмента гена CGRPN4 также приводит к потере трансактиваторной активности белка CgRpn4. Укороченный белок не способен восстанавливать устойчивость к стрессу и уровень мРНК генов-мишеней в штамме с делецией SCRPN4 (рис. 4б, в). Таким образом, N-концевая область белка CgRpn4 также может содержать весь основной трансактиваторный домен или его часть.
Рис. 4.
N-концевая область необходима для функционирования CgRpn4. а – Схема структуры белков, кодируемых экспрессионными плазмидами. б – CgRpn4 с делецией 177 N-концевых остатков не восстанавливает устойчивость штамма rpn4-Δ к стрессу. в – CgRpn4-Δ177 не восстанавливает уровень экспрессии PRE1 в штамме rpn4-Δ. Ген ACT1 использовали в качестве референсного. Нормированное значение уровня мРНК PRE1 в штамме rpn4-Δ, продуцирующем ΔN210-ScRpn4, в нормальных условиях принято за единицу. Представлены средние значения трех независимых опытов. Разброс данных представлен стандартным отклонением. ΔN210-ScRpn4 – штамм rpn4-Δ, трансформированный плазмидой YCpLac36-ΔN210-SCRPN4; ΔN177-CgRpn4 – штамм rpn4-Δ, трансформированный плазмидой YCpLac36-ΔN177-CgRPN4. Сd2+ – CdCl2; AsIII –NaAsO2, вызывает окислительный стресс.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В настоящей работе впервые клонирован ген Rpn4-подобного белка условно-патогенных дрожжей C. glabrata. В гетерологичной системе экспрессии S. cerevisiae показано, что CgRpn4 способен функционально замещать ScRpn4. Кроме того, уникальная N-концевая область необходима для функционирования CgRpn4.
C. glabrata эволюционно более близкий S. cerevisiae вид, чем K. pfaffii, Y. lipolytica и D. hansenii [2]. В соответствии с этим CgRpn4 обнаруживает большее сходство с ScRpn4 (48.1%), чем KpRpn4 (36.8%), YlRpn4 (36.3%) [8] или DhRpn4 (37.2%) [9]. Как и у других Rpn4-подобных белков, наивысшее локальное сходство выявлено у предполагаемого ДНК-связывающего домена, представленного цинковыми пальцами, тогда как N-концевая область уникальна для CgRpn4. Отметим, что высокое сходство ДНК-связывающих доменов ScRpn4 и CgRpn4 и идентичность аминокислотных остатков, контактирующих, вероятно, с ДНК, соответствует высокому сходству профилей распознаваемых PACE-подобных элементов (рис. 2а). Можно ожидать, что CgRpn4 способен регулировать те же гены, что и ScRpn4. В числе генов, регулируемых Rpn4, можно отметить YAP1, кодирующий фактор транскрипции, действующий как сенсор активных форм кислорода, и как регулятор экспрессии генов, чьи продукты участвуют в ответе на окислительный стресс, например тиоредоксинов [28]. Кроме того, важными мишенями Rpn4 служат факторы транскрипции Pdr1–Pdr3, участвующие в феномене множественной лекарственной устойчивости [4].
Различия же в последовательности N-концевых областей CgRpn4 и ScRpn4 ведут к закономерному вопросу, содержит ли N-концевая область CgRpn4 трансактиваторный домен? Полученные нами данные указывают на то, что N-концевая область CgRpn4 действительно необходима для функционирования белка как фактора транскрипции и вносит основной вклад в формирование устойчивости к стрессу. В N-концевой области CgRpn4 находится короткий участок, обогащенный остатками глутамина, который может быть частью домена бóльшего размера. Обогащенные глутамином домены встречаются у небольшого числа факторов транскрипции и вспомогательных транскрипционных комплексов S. cerevisiae, например, у Нap1p, Нap2p и в субъединице Gal11p одного из медиаторных комплексов [29].
Таким образом, нами впервые показано, что в гетерологичной системе S. cerevisiae Rpn4-подобный белок из C. glabrata служит активатором транскрипции протеасомных генов и участвует в ответе на различные виды стресса, включая окислительный. Можно предположить, что этот белок вносит значительный вклад в устойчивость C. glabrata к окислительному стрессу и фунгицидам, и, опосредованно, в вирулентность микроорганизма. Эта гипотеза требует экспериментального подтверждения.
Авторы сообщают об отсутствии конфликта интересов.
Часть работы выполнена на оборудовании ЦКП “Геном” ИМБ РАН (http://www.eimb.ru/rus/ ckp/ccu_genome_c.php).
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (№ 18-34-00704) (рис. 1), Программы фундаментальных исследований государственных академий наук на 2013–2020 годы (№ 01201363823) (рис. 2) и Российского научного фонда (№ 17-74-30030) (pис. 3, 4).
Список литературы
Gabaldon T., Carrete L. (2016) The birth of a deadly yeast: tracing the evolutionary emergence of virulence traits in Candida glabrata. FEMS Yeast Res. 16, fov110.
Mannhaupt G., Feldmann H. (2007) Genomic evolution of the proteasome system among hemiascomycetous yeasts. J. Mol. Evol. 65, 529–540.
Vermitsky J.-P., Earhart K.D., Smith W.L., Homayouni R., Edlind T.D., Rogers P.D. (2006) Pdr1 regulates multidrug resistance in Candida glabrata: gene disruption and genome-wide expression studies. Mol. Microbiol. 61, 704–722.
Owsianik G., Balzil L., Ghislain M. (2002) Control of 26S proteasome expression by transcription factors regulating multidrug resistance in Saccharomyces cerevisiae. Mol. Microbiol. 43, 1295–1308.
Капранов А.Б., Преображенская О.В., Тютяева В.В., Штука Р., Фельдман Х., Карпов В.Л. (2001) Выделение и идентификация PACE-связывающего белка Rpn4 – нового транскрипционного активатора, участвующего в регуляции 26S протеасомных и других генов. Молекуляр. биология. 35, 420–431.
Mannhaupt G., Schnall R., Karpov V., Vetter I., Feldmann H. (1999) Rpn4 acts as a transcription factor by binding to PACE, a nonamer box found upstream of 26S proteasomal and other genes in yeast. FEBS Lett. 450, 27–34.
Спасская Д.С., Карпов Д.С., Миронов А.С., Карпов В.Л. (2014) Транскрипционный фактор Rpn4 Saccharomyces cerevisiae обеспечивает комплексный антистрессовый ответ при действии метилметансульфоната. Молекуляр. биология. 48, 166–175.
Гринева Е.Н., Лейнсоо А.Т., Спасская Д.С., Карпов Д.С., Карпов В.Л. (2014) Функциональный анализ Rpn4-подобных белков из Komagataella (Pichia) pastoris и Yarrowia lipolytica в гетерологичной системе Saccharomyces cerevisiae. Биотехнология. 6, 8–17.
Karpov D.S., Grineva E.N., Leinsoo A.T., Nadolinskaia N.I., Danilenko N.K., Tutyaeva V.V., Spasskaya D.S., Preobrazhenskaya O.V., Lysov Y.P., Karpov V.L. (2017) Functional analysis of Debaryomyces hansenii Rpn4 on a genetic background of Saccharomyces cerevisiae. FEMS Yeast Res. 17, fow098.
Enjalbert B., Smith D.A., Cornell M.J., Alam I., Nicholls S., Brown A.J.P., Quinn J. (2006) Role of the Hog1 stress-activated protein kinase in the global transcriptional response to stress in the fungal pathogen Candida albicans. Mol. Biol. Cell. 17, 1018‒1032.
Gietz R.D., Woods R.A. (2002) Transformation of yeast by the LiAc/ss carrier DNA/PEG method. Methods Enzymol. 350, 87–96.
Schwarzmuller T., Ma B., Hiller E., Istel F., Tscherner M., Brunke S., Ames L., Firon A., Green B., Cabral V., Marcet-Houben M., Jacobsen I.D., Quintin J., Seider K., Frohner I., Glaser W., Jungwirth H., Bachellier-Bassi S., Chauvel M., Zeidler U., Ferrandon D., Gabaldon T., Hube B., d’Enfert C., Rupp S., Cormack B., Haynes K., Kuchler K. (2014) Systematic phenotyping of a large-scale Candida glabrata deletion collection reveals novel antifungal tolerance genes. PLoS Pathog. 10, e1004211.
Amberg D.C., Burke D.J., Strathern J.N. (2006) Isolation of yeast genomic DNA for southern blot analysis. CSH Protoc. 2006.
Karpov D.S., Spasskaya D.S., Tutyaeva V.V., Miro-nov A.S., Karpov V.L. (2013) Proteasome inhibition enhances resistance to DNA damage via upregulation of Rpn4-dependent DNA repair genes. FEBS Lett. 587, 3108–3114.
Спасская Д.С., Карпов Д.С., Карпов В.Л. (2011) Dam-метилаза Escherichia coli как молекулярный инструмент для картирования сайтов связывания дрожжевого фактора транскрипции Rpn4p. Молекуляр. биология. 45, 642–651.
Schmitt M.E., Brown T.A., Trumpower B.L. (1990) A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucl. Acids Res. 18, 3091–3092.
Zuker M. (2003) Mfold web server for nucleic acid folding and hybridization prediction. Nucl. Acids Res. 31, 3406–3415.
Ha S.W., Ju D., Xie Y. (2012) The N-terminal domain of Rpn4 serves as a portable ubiquitin-independent degron and is recognized by specific 19S RP subunits. Biochem. Biophys. Res. Commun. 419, 226–231.
Karpov D.S., Tutyaeva V.V., Karpov V.L. (2008) Mapping of yeast Rpn4p transactivation domains. FEBS Lett. 582, 3459–3464.
McWilliam H., Li W., Uludag M., Squizzato S., Park Y.M., Buso N., Cowley A.P., Lopez R. (2013) Analysis tool web services from the EMBL-EBI. Nucl. Acids Res. 41, W597–600.
Persikov A., Singh M. (2014) De novo prediction of DNA-binding specificities for Cys2His2 zinc finger proteins. Nucl. Acids Res. 42, 97–108.
Wolfe S.A., Nekludova L., Pabo C.O. (2000) DNA recognition by Cys2His2 zinc finger proteins. Annu. Rev. Biophys. Biomol. Struct. 29, 183–212.
Dreier B., Segal D.J., Barbas C.F. (2000) Insights into the molecular recognition of the 5′-GNN-3′ family of DNA sequences by zinc finger domains. J. Mol. Biol. 303, 489– 502.
Crooks G.E., Hon G., Chandonia J.M., Brenner S.E. (2004) WebLogo: a sequence logo generator. Genome Res. 14, 1188–1190.
Brennan R.J., Schiestl R.H. (1996) Cadmium is an inducer of oxidative stress in yeast. Mutat. Res. 356, 171–178.
Haugen A.C., Kelley R., Collins J.B., Tucker C.J., Deng C., Afshari C.A., Brown J.M., Ideker T., Van Hou-ten B. (2004) Integrating phenotypic and expression profiles to map arsenic-response networks. Genome Biol. 5, R95.
London M.K., Keck B.I., Ramos P.C., R. Dohmen R.J. (2004) Regulatory mechanisms controlling biogenesis of ubiquitin and the proteasome. FEBS Lett. 567, 259–264.
Lee J., Godon C., Lagniel G., Spector D., Garin J., Labarre J., Toledano M.B. (1999) Yap1 and Skn7 control two specialized oxidative stress response regulons in yeast. J. Biol. Chem. 274, 16040–16046.
Mitchell P.J., Tjian R. (1989) Transcriptional regulation in mammalian cells by sequence specific DNA binding proteins. Science. 245, 371–378.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


