Молекулярная биология, 2019, T. 53, № 4, стр. 627-637
Структурно-функциональная организация контрольного района митохондриальной ДНК шерстистого мамонта (Mammuthus primigenius)
И. В. Корниенко a, b, *, Т. Г. Фалеева b, c, Н. В. Орешкова d, e, f, С. Е. Григорьев g, Л. В. Григорьева h, Ю. А. Путинцева e, К. В. Крутовский e, i, j, k
a Федеральный исследовательский центр Южный научный центр Российской академии наук
344006 Ростов-на-Дону, Россия
b Южный федеральный университет, Академия биологии и биотехнологии им. Д.И. Ивановского
344090 Ростов-на-Дону, Россия
c Филиал № 2 федерального государственного казенного учреждения “111 Главный государственный центр судебно-медицинских и криминалистических экспертиз” Министерства обороны Российской Федерации
344000 Ростов-на-Дону, Россия
d Институт леса им. В.Н. Сукачева Сибирского отделения Российской академии наук
660036 Красноярск, Россия
e Сибирский федеральный университет, Научно-образовательный центр геномных исследований
660036 Красноярск, Россия
f Федеральный исследовательский центр “Красноярский научный центр Сибирского отделения Российской академии наук”, Лаборатория геномных исследований и биотехнологии
660036 Красноярск, Россия
g Северо-Восточный федеральный университет, Лаборатория Музей мамонта им. П.А. Лазарева,
Институт прикладной экологии Севера
677980 Якутск, Россия
h Северо-Восточный федеральный университет, Международный центр коллективного пользования
“Молекулярная палеонтология”
677980 Якутск, Россия
i Department of Forest Genetics and Forest Tree Breeding, Georg-August University of Göttingen
37077 Göttingen, Germany
j Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
k Department of Ecosystem Science and Management, Texas A&M University
TX 77843-2138 College, Station, USA
* E-mail: ikornienko@yandex.ru
Поступила в редакцию 07.02.2019
После доработки 04.03.2019
Принята к публикации 13.03.2019
Аннотация
Митохондриальный геном шерстистого мамонта, в том числе и Малоляховского мамонта, был секвенирован ранее, с последующей аннотацией всех генов (MF770243). Впервые на примере Малоляховского мамонта нами описаны функционально-значимые участки контрольного района митогенома шерстистых мамонтов.
ВВЕДЕНИЕ
К настоящему времени полностью прочитаны и аннотированы митохондриальные геномы нескольких шерстистых мамонтов [1–7], однако структурно-функциональная организация контрольного района их мтДНК оставалась неизвестной. Ранее упоминалось об обнаружении тандемных повторов в некодирующих участках контрольного района мтДНК [3], однако сведения о функциональной роли этих повторов или об иных значимых элементах контрольного региона мтДНК шерстистых мамонтов отсутствуют. В настоящей работе полностью аннотированы все ключевые элементы некодирующего района мтДНК Малоляховского шерстистого мамонта.
В конце лета 2012 г. на острове Малый Ляховский (74°07′ с.ш., 140°40′ в.д.) Новосибирского архипелага недалеко от морского побережья на склоне невысокого холма была обнаружена туша шерстистого мамонта (Mammuthus primigenius). По оценке палеонтологов возраст находки составлял 32.5–33 тыс. лет [8]. Главной особенностью Малоляховского мамонта была уникальная сохранность мягких тканей. Обследование туши мамонта показало, что хорошо сохранилась нижняя часть головы, частично оволосенный хобот (единственный в мире сохранившийся хобот взрослого мамонта), области шеи и брюха, полностью передние конечности и дистальная половина правой задней конечности. Верхняя часть трупа – череп, позвонки, дорсальные части ребер, тазовые кости, левая задняя нога были объедены хищниками и скелетированы вследствие того, что после гибели животного эти части оставались на поверхности земли (предположительно, мамонт нижней частью провалился в промоину). При раскопках не удалось обнаружить большинство позвонков и около половины ребер. Вероятно, эти мелкие кости были растащены животными. Отсутствовала также левая ветвь тазовой кости. Череп был фрагментирован на несколько частей. В полной анатомической последовательности сохранился скелет левой задней конечности с остатками шерсти и жировой ткани в области колена и стопы. Нога была вытянута назад в горизонтальной плоскости. Правая задняя конечность от голени и ниже оказалась подвернута под брюхо в неестественной для нормального физиологического состояния позе. Мышечные ткани передних конечностей имели буроватый цвет. Кожа сохранила эластичность, и в целом мумификация туши была минимальной благодаря залеганию нижней части туловища в практически чистом льду.
В процессе раскопок в ледовых пустотах ниже брюха и в подмышечной области в двух местах наблюдалось скопление небольшого количества темно-бурой жидкости, что удивительно, учитывая возраст находки. Большое количество этой замерзшей бурой жидкости окружало всю тушу в самой ее нижней части. Морфологические признаки свидетельствовали, что животное было старой самкой приблизительно 50–60 лет, в частности, судя по относительно небольшому размеру – рост около 230 см в холке, наличию сосков, форме и весу бивней (16 и 17 кг), состоянию последней генерации зубов [8].
В процессе предыдущих исследований был секвенирован митохондриальный геном Малоляховского мамонта [7] с последующей аннотацией всех генов (MF770243), однако функционально-значимые участки некодирующего района этой мтДНК представлены не были.
В настоящей работе описаны результаты расшифровки функционально значимых элементов контрольного района мтДНК Малоляховского мамонта.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Пробоподготовка биологического материала. Для молекулярно-генетического исследования отобраны фрагменты мышечных тканей хобота, плечевой кости и ребра. Пробоподготовку биологического материала и выделение ДНК проводили согласно [7].
ПЦР-амплификация, количественная оценка, секвенирование и видовая идентификация препаратов ДНК. Количество мтДНК, выделенной из фрагментов плечевой кости и ребра, а также из мягких тканей хобота Малоляховского шерстистого мамонта, оценивали с помощью термоциклера с мультиканальным детектором iQ5 (Bio-Rad). С этой целью проводили ПЦР-амплификацию фрагмента гена cytB с использованием праймеров, соответствующих укороченному варианту ранее описанных универсальных праймеров [13] (табл. 1).
Таблица 1.
Укороченные универсальные праймеры, использованные для ПЦР-амплификации фрагмента митохондриального гена cytB из образцов Малоляховского мамонта
| ПЦР-праймеры, прямой (F) и обратный (R) | Нуклеотидные последовательности (5' → 3') |
|---|---|
| cytB_F | CCATCCAACATCTCAGCATGATGAAA |
| cytB_R | CCCCTCAGAATGATATTTGTCCTCA |
ПЦР-амплификацию при конечной концентрации ионов магния 2.5 мМ проводили с использованием следующей программы: предварительная инкубация при 95°С в течение 2.5 мин, затем 40 циклов, каждый из которых включал денатурацию (95°С, 15 с), отжиг (50°С, 1 мин) и элонгацию (72°С, 2 мин).
Регистрацию накопления ампликонов в реальном времени и построение калибровочных кривых проводили по технологии ПЦР в режиме реального времени, основанной на усилении флуоресценции при связывании интеркалятора EVA Green с дцДНК.
ПЦР-амплификацию контрольного района мтДНК Малоляховского мамонта проводили с прямыми праймерами Mom_1F, Mom_2F и Mom_3F в различных сочетаниях с обратными праймерами Mom_1R, Mom_2R, Mom_3R и Mom_4R. Нуклеотидные последовательности праймеров приведены в табл. 2.
Таблица 2.
Праймеры, использованные для ПЦР-амплификации контрольного района мтДНК Малоляховского мамонта
| ПЦР-праймеры, прямые (F) и обратные (R) | Нуклеотидные последовательности (5' → 3') |
|---|---|
| Mom_1F (15377) | CGCTATCAATACCCAAAACTGA |
| Mom_2F (15513) | GACCATACTATGTATAATC |
| Mom_3F (15863) | TCTTACTTCAGGACCATCT |
| Mom_1R (15916) | CTTATTTAAGAGGAAAGAG |
| Mom_2R (16569) | GAAGAGATAGTTACATAGTG |
| Mom_3R (16789) | CAAATCTAGGAACATGGCTCTT |
| Mom_4R (60) | AGTTGGATATACTCGTCTAGG |
ПЦР-амплификацию при конечной концентрации ионов магния 2.5 мМ проводили при помощи термоциклера GeneAmp PCR System 9700 (“Applied Biosystems”, США) в режиме эмуляции 1°С/с с использованием следующей программы: предварительная инкубация при 95°С в течение 2.5 мин; затем 35 циклов, каждый из которых включал денатурацию (95○С, 20 с), отжиг праймеров (55○С, 20 с), и элонгацию (72○С, 2 мин), с последующим завершающим этапом элонгации при 72○С в течение 10 мин.
Продукты ПЦР очищали с помощью колонок GeneJET PCR Purification (“Thermo Scientific”, США).
Амплифицированные участки мтДНК секвенировали с помощью набора реактивов для секвенирования (DNA Sequencing Kit, BigDye Terminator Cycle Sequencing Ready Reaction, “Applied Biosystems”) v. 1.1 (а также v. 3.1). Электрофорез образцов проводили с помощью ДНК-анализатора ABI PRISM 3130xl. Нуклеотидные последовательности мтДНК анализировали с помощью программы Sequencing Analysis (версия 5.3).
ПЦР-амплификацию локуса, содержащего участок STR с повторяющимся мотивом ACGCAT контрольного района мтДНК Малоляховского мамонта, проводили с использованием флуоресцентно меченного красителем FAM прямого праймера Mom_16146F в различных сочетаниях с обратными праймерами Mom_16570R, Mom_16611R, Mom_60R, нуклеотидные последовательности которых приведены в табл. 3.
Таблица 3.
Праймеры, использованные для ПЦР-амплификации STR-локуса контрольного района мтДНК Малоляховского мамонта
| ПЦР- праймеры, прямые (F) и обратные (R) | Нуклеотидные последовательности (5' → 3') |
|---|---|
| Mom_16146F-FAM | TAAATTCCCAGCGTACACGC |
| Mom_16570R | GAAGAGATAGTTACATAGTGG |
| Mom_16611R | AGTTACCCTTGGCTCAATTTC |
| Mom_60R | AGTTGGATATACTCGTCTAGG |
ПЦР-амплификацию при конечной концентрации ионов магния 2.0 мМ проводили при помощи термоциклера GeneAmp PCR System 9700 (“Applied Biosystems”) в режиме эмуляции 1°С/с с использованием следующей программы: предварительная инкубация (95°С, 2.5 мин), затем 32 цикла, каждый из которых включал денатурацию (95°С, 20 с), отжиг праймеров (58°С, 20 с) и элонгацию (72°С, 1 мин), с последующим заверщающим этапом элонгации при 72°С в течение 10 мин.
Электрофорез образцов проводили с помощью ДНК-анализатора ABI PRISM 3130xl. Длину ампликонов определяли с помощью программы GeneMapper ID (версия 3.2) по размерным стандартам ROX500 и ROX1000 с использованием алгоритма расчета Size Calling Method–Local Southern Method.
Анализ нуклеотидных последовательностей ДНК. С целью определения консервативных нуклеотидных последовательностей в контрольном регионе мтДНК Малоляховского мамонта с помощью программ Clustal X v. 2.1 [14] и UGENE v. 1.27 [15] проведены множественные сравнения с аналогичными нуклеотидными последовательностями различных видов млекопитающих [16].
Вторичную структуру фрагментов оцДНК анализировали с помощью программы RNAstructure [17].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Доказательство принадлежности выделенной ДНК шерстистому мамонту представлено в [7]. Необходимо отметить, что в древней ДНК накапливаются различные повреждения, наиболее частое из которых – самопроизвольный гидролиз аминогруппы в положении 4′ С и превращение последнего в U, что приводит к замене C→U→Т в процессе ПЦР. Однако несмотря на большой возраст находки (около 33 тыс. лет [8]), никаких изменений, присущих древней ДНК (признаки дезаминирования остатков цитозина и аденина), в ДНК, выделенной из различных образцов Малоляховского мамонта (хобота, плечевой кости и ребра), не обнаружено, что дополнительно свидетельствует о высокой сохранности тканей и ДНК.
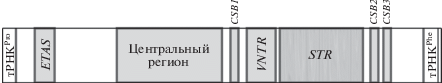
При множественном выравнивании нуклеотидной последовательности мтДНК Малоляховского шерстистого мамонта (MF770243) с контрольными районами мтДНК Homo sapiens (V00662), Mus musculus (V00711), Rattus norvegicus, Oryctolagus cuniculus, Cephalorinchus commersonii, Kogia breviceps, Balaena mysticetus (X14848), Bos taurus (V00654), Ovis aries (L29055), Diceros bicornis (L22010), Equus asinus (X97337), Equus caballus (X79547), Halichoerus grypus (X72004) и Didelphis virginiana (Z29573) [15] идентифицированы консервативные последовательности CSB1, CSB2, CSB3 (conserved sequence block) и ETAS (extended termination associated sequences 1) (табл. 4), ассоциированные с сайтами начала и завершения транскрипции мтДНК соответственно [18].
Таблица 4.
Нуклеотидные последовательности функционально значимых участков контрольного района Малоляховского шерстистого мамонта
| Локус | Нуклеотидная последовательность, 5'→3' | Длина, п.н. |
|---|---|---|
| CSB1 | ATTCAGTCAATGCTCAGAGGACATA | 25 |
| CSB2 | CAAACCCCCCTTACCCCCC | 19 |
| CSB3 | TGTCAAACCCCAAAAGCA | 18 |
| ETAS | CCATACTATGTATAATCGTGCATCACATTATTTACCCCATGCTTATAAGCAAGTA CTGTTTAA | 64 |
| Центральный регион | CTTAACTACCTACCTCCGAGAAACCATCAACCCGCCCATCTTCGTGTCCCTCTTC TCGCTCCGGGCCCATCAATTGTGGGGGTTTCTATACTGGATCTATACCTGGCATC TGGTTCTTACTTCAGGACCATCTCACCTAAAATCGCCCACTCTTTCCTCTTAAAT AAGACATCTCGATGGATTAATGACTAATCAGCCCATGATCATAACATAACTGTG GTGTCATGCATTTGGTATCTTTTTAATTTTGGGGATGCTGTGATTCAGCTATGGC CGTCTGAGGCCCTAACACAGTCAAGCAACTTGTAGCTGAGCTTG | 318 |
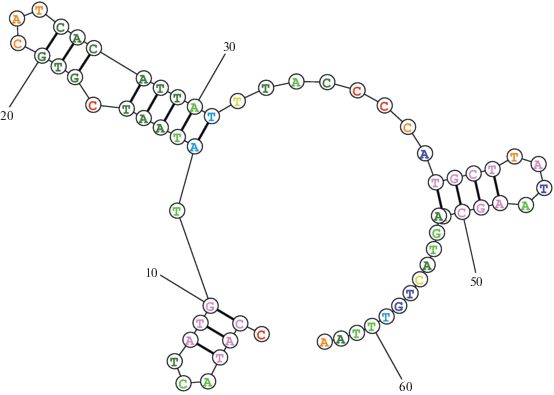
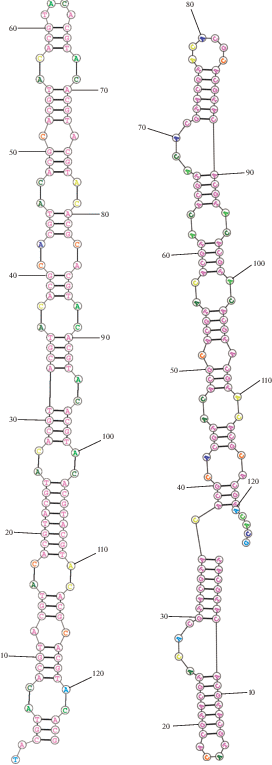
Сравнительный анализ локуса ETAS у различных представителей класса млекопитающих, включая отряд хоботных, показал его консервативность в ходе эволюции (табл. 5), что указывает на важное функциональное значение этого локуса мтДНК. ETAS Малоляховского мамонта содержит три участка, способных к формированию шпилечных структур, которые, возможно, и принимают участие в терминации транскрипции мтДНК (рис. 1 ).
Рис. 1.
Наиболее энергетически выгодная вторичная структура локуса ETAS Малоляховского мамонта, рассчитанная с помощью программы RNAstructure.
Между локусами ETAS и CSB1 контрольного района мтДНК Малоляховского мамонта располагается консервативный центральный район (CR) длиной 318 п.н. (рис. 2 ). У хоботных точечные замены в нем представлены только 15 транзициями (12 из них C→T, две – T→C и одна – A→G), семью трансверсиями A→T и двумя делециями. Причем различия в нуклеотидных последовательностях CR у шерстистых мамонтов практически отсутствовали, за исключением одного представителя вида M. primigenius (JF912200, замена C на T в позиции 102) и M. columbi (замена C на T в позиции 150).
Один из представителей азиатских слонов, E. maximus (AJ428946), имел такую же последовательность CR мтДНК, как и Малоляховский мамонт. У двух других (EF588275 и NC_005129) различия представлены транзициями C→T в положениях 150 и 286. Кроме того, у одного из трех E. smaximus (EF588275) выявлена делеция T и G в позициях 218 и 219 соответственно.
Нуклеотидные замены в CR африканских саванных слонов L. africana (NC_000934, AB443879, DQ316069) представлены транзициями C→T в позициях 102 и 150, а также трансверсией A→T в позиции 119.
Отличия нуклеотидной последовательности CR мтДНК африканских лесных слонов L. cyclotis от Малоляховского мамонта представлены транзициями C→T и T→C в позициях 150 и 240, соответственно, а также трансверсиями A→T в позициях 119 и 244. Отсутствие 35 н. на 3'-конце CR у одного из африканских лесных слонов (KJ557423) [18] можно объяснить только неполной нуклеотидной последовательностью мтДНК этого животного.
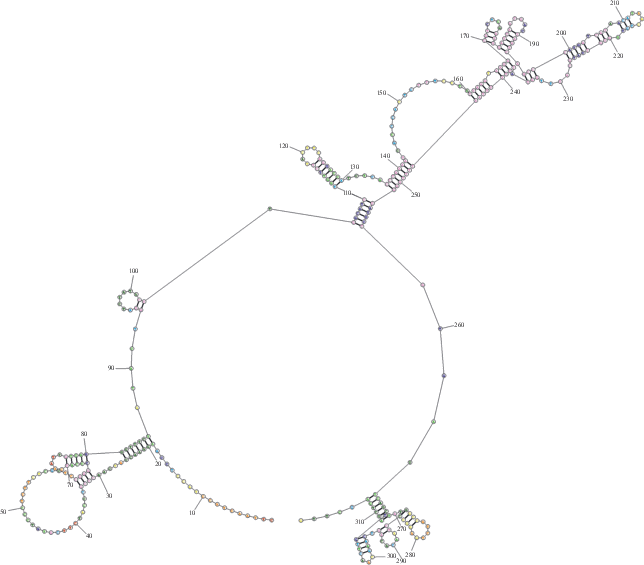
Функциональное значение CR мтДНК до конца не выяснено. Однако способность этого локуса формировать стабильные шпилечные структуры (рис. 3 ) косвенно указывает на его важную роль в транскрипции мтДНК хоботных.
Рис. 3.
Энергетически выгодная вторичная структура центрального района D-петли мтДНК Малоляховского шерстистого мамонта, рассчитанная с помощью программы RNAstructure.
Локусы CSB1, CSB2, CSB3, как и ETAS, высококонсервативны у ныне живущих и вымерших представителей отряда хоботных (табл. 6), что не удивительно, учитывая важную функциональную роль этих участков ДНК в инициации транскрипции мтДНК. Из табл. 6 видно, что из трех локусов CSB1-3 наиболее консервативен CSB3, нуклеотидная последовательность которого практически не менялась в ходе эволюции хоботных.
Таблица 6.
Нуклеотидные последовательности консервативных районов локусов CSB1, CSB2, CSB3 у ряда представителей млекопитающих, в том числе хоботных
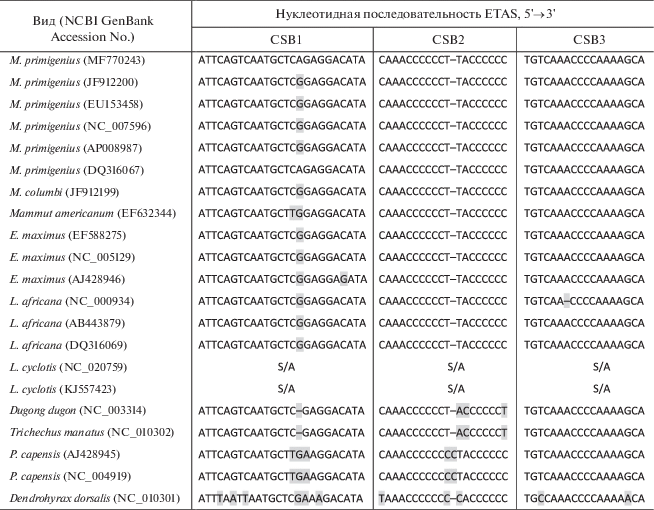
Ввиду исключительной функциональной важности локусов CSB1, CSB2 и CSB3 для транскрипции мтДНК, их отсутствие в митохондриальном геноме лесного слона (L. cyclotis) может указывать на неполную последовательность контрольного района лесного слона (KJ557423 [19] и NC_020759 [20]). Проверить это можно путем амплификации локусов с помощью локус-специфичных праймеров, но это не входило в задачи нашего исследования.
Между участками CSB1 и CSB2 контрольного района мтДНК Малоляховского мамонта расположен VNTR-локус, состоящий из повторяющегося 4 раза мотива ACGTACACGTACGTACACGCACGTACACGTAC (за исключением двух транзиций С↔Т и двух инделей внутри мотивов). Учитывая, что нуклеотидная последовательность VNTR-локуса склонна к образованию множества энергетически выгодных вторичных одноцепочечных структур (рис. 4 ), можно предположить его некую роль в процессах репликации либо транскрипции мтДНК шерстистого мамонта. С другой стороны, сравнительный анализ нуклеотидных последовательностей CR других шерстистых мамонтов (NC_007596 [1], AP008987 и DQ316067 [3]) показал, что эти участки полиморфны, что плохо согласуется с гипотезой о важной роли VNTR-локуса в функционировании митохондриального генома, предполагающей низкую изменчивость таких локусов, особенно внутри вида или между близкими видами.
Рис. 4.
Две энергетически наиболее выгодные вторичные структуры одноцепочечного VNTR-локуса контрольного района мтДНК Малоляховского мамонта, рассчитанные с помощью программы RNAstructure.
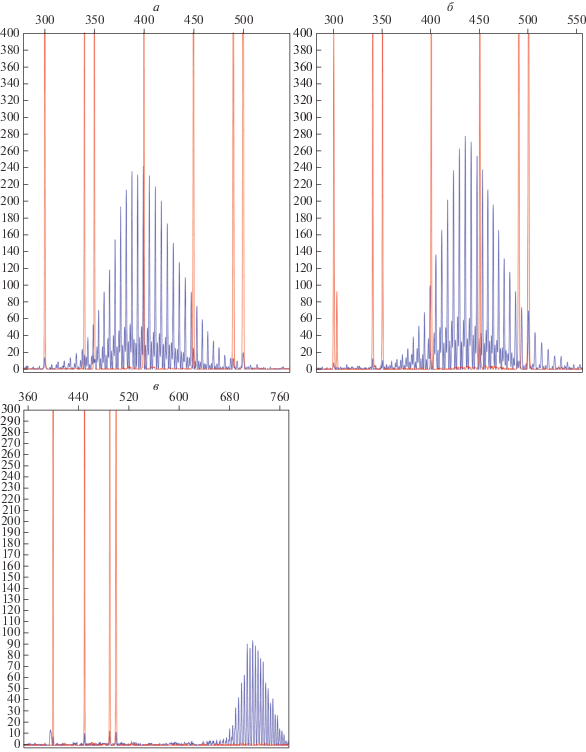
Кроме VNTR-локуса, между CSB1 и CSB2 контрольного района мтДНК Малоляховского шерстистого мамонта локализован протяженный участок STR с повторяющимся мотивом ACGCAT. Количество повторов в этом STR-локусе составляет не менее 40. Более точную цифру определить крайне затруднительно, поскольку “проскальзывание” или “спотыкание” ДНК-полимеразы в процессе репликации приводит либо к пропуску повтора, либо к его лишней вставке. Возникновение такого рода ошибок ДНК-полимеразы при репликации этого STR-локуса может приводить к гетероплазмии по длине мтДНК. Действительно, при проведении ПЦР-амплификации данного участка одновременно получены десятки ампликонов различной длины с шагом 6 п.н. (рис. 5 ). Маловероятно, что наблюдаемая гетероплазмия ложная и возникает из-за ошибок Taq-полимеразы в процессе ПЦР, иногда происходящих при амплификации участков с высокоповторяющимися мотивами. Обычно такие ошибки связаны с гораздо более короткими мотивами, такими как моно- и динуклеотидные повторы. Полиморфизм длины ПЦР-продуктов в пределах одной пробы может зависеть также от того, насколько удачно подобраны праймеры для исследуемого участка мтДНК. Однако появление гетероплазмии мы наблюдали в процессе ПЦР-амплификации независимо от праймеров, с помощью которых получены ампликоны.
Рис. 5.
Гетероплазмия по длине, возникающая в процессе ПЦР-амплификации участка контрольного района мтДНК Малоляховского мамонта, содержащего STR-повторы (ACGCAT)>40. Разница в размерах между ампликонами кратна 6 н. Распределение длин ампликонов, полученных при использовании пары праймеров F16146/R16570 (ожидаемый размер ампликона 425 п.н.) (а), F16146/R16611 (ожидаемый размер ампликона 466 п.н.) (б), и F16146/R60 (ожидаемый размер ампликона 766 п.н.) (в).
Стоит отметить, что гетероплазмия длины контрольного района мтДНК, обусловленная присутствием различного количества тандемных повторов, встречается в разных таксономических группах эукариот, начиная с дрожжей [21] и заканчивая высшими млекопитающими [22, 23], включая африканских слонов [24].
Несмотря на то, что число STR-повторов у хоботных и родственных видов является изменчивым признаком (табл. 7), нуклеотиды основного (“core”) мотива ACGCAT обладают высокой степенью консервативности в процессе эволюции. Так, нуклеотидная последовательность “основного” мотива STR-локуса контрольного района мтДНК Малоляховского мамонта (ACGCAT) идентична нуклеотидным последовательностям мотива этого локусов у других хоботных: шерстистых мамонтов (NC_007596 [1], AP008987, DQ316067 [3]), азиатских (AJ428946 [25], EF588275, NC_005129 [3]) и африканских слонов (NC_000934 [24], DQ316069 [3]). Присутствие STR (ACGCAT)n в контрольном регионе мтДНК характерно не только для хоботных, но и для родственных им видов, таких как дюгонь (NC_003314 [26]), американский ламантин (NC_010302 [25]) и западный даман (D. dorsalis, NC_010301 [25]). Стоит отметить, что в контрольном регионе мтДНК капского дамана (P. capensis, AJ428945 [25] и NC_004919 [27]) вместо тандемных повторов (ACGCAT)n локализованы тандемные повторы с “основным” мотивом GTATACAC. Эти факты указывают на приблизительное время возникновения STR (ACGCAT)n в контрольном регионе мтДНК млекопитающих – не позднее эоцена.
Таблица 7.
Количество повторов ACGCAT в STR-локусе контрольного района мтДНК у ряда представителей млекопитающих, в том числе хоботных
| Вид (NCBI GenBank Accession No.) | Количество повторов ACGCAT |
|---|---|
| M. primigenius (MF770243) M. primigenius (JF912200) M. primigenius (EU153458) M. primigenius (NC_007596) M. primigenius (AP008987) M. primigenius (DQ316067) M. columbi (JF912199) Mammut americanum (EF632344) E. maximus (EF588275) E. maximus (NC_005129) E. maximus (AJ428946) L. africana (NC_000934) L. africana (AB443879) L. africana (DQ316069) L. cyclotis (NC_020759) L. cyclotis (KJ557423) D. dugon (NC_003314) T. manatus (NC_010302) D. dorsalis (NC_010301) |
>40 N N 28 49 50 N N 31 39 30 43 N 38 N N 65 72 37 |
Таким образом, в настоящей работе впервые полностью представлены все функционально значимые участки контрольного района мтДНК шерстистого мамонта, а также показана возможная функциональная роль повторяющихся нуклеотидных мотивов, локализованных между консервативными локусами CSB1 и CSB2, отвечающих за терминацию транскрипции митохондриального генома.
Публикация подготовлена в рамках реализации госзадания ЮНЦ РАН, № гр. Проекта 01201363186.
В настоящей работе соблюдены стандарты работы с животными образцами.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Krause J., Dear P.H., Pollack J.L., Slatkin M., Spriggs H., Barnes I., Lister A.M., Ebersberger I., Pääbo S., Hofreiter M. (2006) Multiplex amplification of the mammoth mitochondrial genome and the evolution of Elephantidae. Nature. 439, 724‒727.
Poinar H.N., Schwarz C., Qi J., Shapiro B., Macp-hee R.D., Buigues B., Tikhonov A., Huson D.H., Tomsho L.P., Auch A., Rampp M., Miller W., Schuster S.C. (2006) Metagenomics to paleogenomics: large-scale sequencing of mammoth DNA. Science. 311, 392‒394.
Rogaev E.I., Moliaka Y.K., Malyarchuk B.A., Kondrashov F.A., Derenko M.V., Chumakov I., Grigorenko A.P. (2006) Complete mitochondrial genome and phylogeny of Pleistocene Mammoth Mammuthus primigenius. PLoS Biol. 4, e73.4a.
Gilbert M.T., Drautz D.I., Lesk A.M., Ho S.Y., Qi J., Ratan A., Hsu C.H., Sher A., Dalén L., Götherström A., Tomsho L.P., Rendulic S., Packard M., Campos P.F., Kuznetsova T.V., Shidlovskiy F., Tikhonov A., Willerslev E., Iacumin P., Buigues B., Ericson P.G., Germonpré M., Kosintsev P., Nikolaev V., Nowak-Kemp M., Knight J.R., Irzyk G.P., Perbost C.S., Fredrikson K.M., Harkins T.T., Sheridan S., Miller W., Schuster S.C. (2008) Intraspecific phylogenetic analysis of Siberian woolly mammoths using complete mitochondrial genomes. Proc. Natl. Acad. Sci. USA. 105, 8327‒8332.
Enk J., Devault A., Debruyne R., King C.E., Treangen T., O’Rourke D., Salzberg S.L., Fisher D., MacPhee R., Poinar H. (2011) Complete Columbian mammoth mitogenome suggests interbreeding with woolly mammoths. Genome Biol. 12, R51.
Chang D., Knapp M., Enk J., Lippold S., Kircher M., Lister A., MacPhee R.D., Widga C., Czechowski P., Sommer R., Hodges E., Stümpel N., Barnes I., Dalén L., Derevianko A., Germonpré M., Hillebrand-Voiculescu A., Constantin S., Kuznetsova T., Mol D., Rathgeber T., Rosendahl W., Tikhonov A.N., Willerslev E., Hannon G., Lalueza-Fox C., Joger U., Poinar H., Hofreiter M., Shapiro B. (2017) The evolutionary and phylogeographic history of woolly mammoths: a comprehensive mitogenomic analysis. Sci Rep. 27, 44585.
Kornienko I.V., Faleeva T.G., Oreshkova N.V., Grigoriev S.E., Grigoreva L.V., Simonov E.P., Kolesnikova A.I., Putintseva Yu.A., Krutovsky K.V. (2018) Complete mitochondrial genome of a woolly mammoth (Mammuthus primigenius) from Maly Lyakhovsky Island (New Siberian Islands, Russia) and its phylogenetic assessment. Mitochondrial DNA Part B. 3, 596‒598.
Grigoriev S.E., Fisher D.C., Obada T., Shirley E.A., Rountrey A.N., Savvinov G.N., Garmaeva D.K., Novgorodov G.P., Cheprasov M.Y., Vasilev S.E. (2017) A woolly mammoth (Mammuthus primigenius) carcass from Maly Lyakhovsky Island (New Siberian Islands, Russian Federation). Quaternary International. 445, 89‒103.
PrepFiler User Guide (2008) Applied Biosystems. 84 p.
Tissue and Hair Extraction Kit (for use with DNA IQ) Protocol (2006) Madison. Promega Corporation. Rev. 5. 11 p.
DNA IQ System – Small Sample Casework Protocol. Technical Bulletin (2006) Madison. Promega Corporation. Rev. 4. 14 p.
Корниенко И.В., Харламов С.Г. (2012) Методы исследования ДНК человека. Выделение ДНК и ее количественная оценка в аспекте судебно-медицинского исследования вещественных доказательств биологического происхождения: Учебно-методическое пособие. Ростов-на-Дону: Изд-во Южного федерального университета, 216 с.
Kocher T.D., Thomas W.K., Meyer A., Edwards S.V., Pääbo S., Villablanca F.X., Wilson A.C. (1989) Dynamics of mitochondrial DNA evolution in animals: Amplification and sequencing with conserved primers. Proc. Natl. Acad. Sci. USA. 86, 6196‒6200.
Larkin M.A., Blackshields G., Brown N.P., Chenna R., McGettigan P.A., McWilliam H., Valentin F., Wallace I.M., Wilm A., Lopez R., Thompson J.D., Gibson T.J., Higgins D.G. (2007) Clustal W and Clustal X version 2.0. Bioinformatics. 23, 2947–2948.
Okonechnikov K., Golosova O., Fursov M., the UGENE team (2012) Unipro UGENE: a unified bioinformatics toolkit. Bioinformatics. 28, 1166–1167.
Sbisa E., Tanzariello F., Reyes A., Pesole G., Saccone C. (1997) Mammalian mitochondrial D-loop region structural analysis: identification of new conserved sequences and their functional and evolutionary implications. Gene. 205, 125‒140.
Reuter J.S., Mathews D.H. (2010) RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129‒137.
Корниенко И.В., Брень А.Б., Войнова Н.В., Гуськов Е.П. (2004) Структурная организация митохондриальной ДНК позвоночных животных. Успехи современ. биологии. 124(1), 17‒27.
Finch T.M., Zhao N., Korkin D., Frederick K.H., Eggert L.S. (2014) Evidence of positive selection in mitochondrial complexes I and V of the African elephant. PLoS One. 9, e92587.
Brandt A.L., Ishida Y., Georgiadis N.J., Roca A.L. (2012) Forest elephant mitochondrial genomes reveal that elephantid diversification in Africa tracked climate transitions. Mol. Ecol. 21, 1175‒1189.
Skelly P.J., Clark-Walker G.D. (1991) Polymorphisms in tandemly repeated sequences of Saccharomyces cerevisiae mitochondrial DNA. J. Mol. Evol. 32, 396‒404.
Dufresne C., Mignotte F., Gueride M. (1996) The presence of tandem repeats and the initiation of replication in rabbit mitochondrial DNA. Eur. J. Biochem. 235, 593‒600.
Lunt D.H., Whipple L.E., Hyman B.C. (1998) Mitochondrial DNA variable number tandem repeats (VNTRs): utility and problems in molecular ecology. Mol. Ecology. 7, 1441‒1455.
Hauf J., Waddell P.J., Chalwatzis N., Joger U., Zimmermann F.K. (2000) The complete mitochondrial genome sequence of the African elephant (Loxodonta africana), phylogenetic relationships of Proboscidea to other mammals and D-loop heteroplasmy. Zoology. 102, 184‒195.
Arnason U., Adegoke J.A., Gullberg A., Harley E.H., Janke A., Kullberg M. (2008) Mitogenomic relationships of placental mammals and molecular estimates of their divergences. Gene. 421, 37‒51.
Arnason U., Adegoke, J.A., Bodin K., Born E.W., Esa Y.B., Gullberg A., Nilsson M., Short R.V., Xu X., Janke A. (2002) Mammalian mitogenomic relationships and the root of the eutherian tree. Proc. Natl. Acad. Sci. USA. 99, 8151‒8156.
MurataY., Nikaido M., Sasaki T., Cao Y., Fukumoto Y., Hasegawa M., Okada N. (2003) Afrotherian phylogeny as inferred from complete mitochondrial genomes. Mol. Phylogenet. Evol. 28, 253‒260.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология