Молекулярная биология, 2019, T. 53, № 4, стр. 600-612
Эволюция и сравнительная геномика плазмиды pSM22 группы IncF/MOBF12
Т. Р. Ясаков a, *, Л. Г. Анисимова a, Н. В. Жарикова a, Е. Ю. Журенко a, В. В. Коробов a, Т. В. Маркушева a
a Уфимский федеральный исследовательский центр Российской академии наук,
Уфимский институт биологии
450054 Уфа, Россия
* E-mail: iasakovtimur@gmail.com
Поступила в редакцию 24.08.2018
После доработки 24.01.2019
Принята к публикации 28.01.2019
Аннотация
Из клеток Serratia marcescens выделена и секвенирована новая плазмида pSM22. Плазмида pSM22 состоит из 43190 п.н., имеет средний GC-состав 58% и содержит 31 открытую рамку считывания, которые образуют модули репликации, конъюгации, стабильности, а также адаптивный модуль. Модуль репликации представлен сайтом начала репликации лидирующей цепи плазмиды, сайтом терминации репликации (terC), генами repA (=repA1) и repA4, а также последовательностью copA, кодирующей антисмысловую РНК (асРНК). Эти структуры функционально объединены в составе репликона FII (группа несовместимости IncFII). Значительные отличия репликона FII от канонических R-плазмид R1 и NR1 (=R100=R222) позволили предположить, что плазмида pSM22 обладает репликоном FII нового подтипа. Модуль конъюгации состоит из 13 генов, обладающих высоким сходством с генами, ответственными за конъюгацию плазмиды F. Сравнительный геномный анализ выявил сходство структуры модулей конъюгации pSM22 и F. Наличие трех консервативных мотивов в релаксазе (TraI) pSM22 позволило классифицировать эту плазмиду по системе конъюгации как представителя типа MOBF клады F12. Модуль стабильности состоит из генов resD и parA, которые отвечают за разрешение мультимерных форм плазмиды и ее последующую сегрегацию между дочерними клетками. Адаптивный модуль содержит гены секреции и процессинга микроцина H47 (MccH47) и УФ-устойчивости. Мозаичная структура pSM22, а также редукционный путь эволюции ее модулей указывают на высокую геномную пластичность бактерий рода Serratia. Анализ организации модулей pSM22 проясняет эволюционные взаимоотношения среди плазмид группы IncF/MOBF12 у бактерий семейства Enterobacteriaceae и открывает перспективы дальнейших сравнительных геномных исследований плазмид бактерий рода Serratia.
ВВЕДЕНИЕ
Плазмиды – это внехромосомные молекулы ДНК, способные к автономной репликации. Посредством горизонтального переноса плазмиды обеспечивают прокариотам адаптивные преимущества в изменяющихся условиях окружающей среды [1]. В число этих преимуществ входят устойчивость к антибиотикам [2], солям тяжелых металлов [3], способность к деградации ксенобиотиков [4, 5], что привлекает к плазмидам неослабевающее внимание.
Существенный прогресс в технологиях секвенирования позволил значительно увеличить количество полностью секвенированных плазмид [6]. В свою очередь, это дало возможность проводить сравнительный геномный анализ, прослеживать распространение плазмид в различных таксонах прокариот, определять направления эволюции и разрабатывать новые системы классификации [7, 8]. Ранее были секвенированы полные нуклеотидные последовательности и проведен сравнительный геномный анализ многих плазмид бактерий семейства Enterobacteriaceae. Подробно описаны плазмиды бактерий родов Klebsiella [9], Salmonella [10, 11], Escherichia [12] и др.
Широко распространенные бактерии рода Serratia относятся к семейству Enterobacteriaceae. Они могут вызывать заболевания человека, животных и насекомых [13, 14], а также быть симбионтами растений [15]. Представители этого рода характеризуются пластичностью, что позволяет им занимать различные ниши в окружающей среде [16], в значительной степени благодаря плазмидам. Например, некоторые плазмиды определяют устойчивость Serratia к антибиотикам [17, 18], другие отвечают за биодеградацию сырой нефти [19]. Однако особенности строения модулей плазмид Serratia и их эволюция еще недостаточно изучены.
Нами выделена и секвенирована новая плазмида группы IncF/MOBF12 бактерий рода Serratia, содержащая гены контроля УФ-устойчивости, с позиций концепции модульного строения проведен ее сравнительный геномный анализ. Определены модули репликации, конъюгации, стабильности и адаптивный (“грузовой”) модуль. В составе адаптивного модуля выявлены области, содержащие гены контроля SOS-ответа, процессинга, секреции микроцина H47 и другие. На основе сравнительного анализа последовательности белка релаксазы TraI показано филогенетическое родство pSM22 с другими плазмидами группы несовместимости IncFII разных представителей семейства Enterobacteriaceae. Выявлены значительные структурные изменения репликона, которые, вероятно, указывают на появление нового подтипа внутри группы FII.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Объекты исследования. В работе использованы следующие бактериальные штаммы: Serratia marcescens 22S (=B-6493) (коллекция УИБ УФИЦ РАН) и Escherichia coli DH5B. Штамм 22S был выделен из загрязненных ксенобиотиками почв г. Уфы. Частичная последовательность гена 16S рРНК штамма 22S депонирована в базу данных GenBank под номером KY563745.
Получение препаратов плазмидной ДНК. Плазмидную ДНК (плазмида pSM22 и векторы библиотеки клональных вставок) выделяли по методу Birnboim H.C. и Doly J. [20] с незначительными модификациями.
ПДРФ-анализ проводили по результатам гель-электрофореза в 1%-ном агарозном геле. Плазмиды расщепляли эндонуклеазами рестрикции KpnI, EcoRI, HincII, PaeI, SalI и HindIII. Реакцию проводили в течение 2 ч при температуре 37оС согласно рекомендациям производителя (“Promega”, США).
ДНК плазмиды pSM22 обрабатывали ультразвуком при 22 кГц на приборе УЗДН-2Т (“Академприбор”, Украина). Ультрализат очищали от посторонних примесей при помощи электрофореза в 0.8%-ном легкоплавком агарозном геле (“Promega”, США) в Трис-ацетатном буфере (ТАЕ), согласно стандартной методике [21]. Полосу фрагментов ДНК размером от 3 до 5 т.п.н. выделяли с помощью набора реактивов Wizard PCR Preps (“Promega”) в соответствии с рекомендациями производителя. Качество препаратов оценивали с использованием спектрофотометра SmartSpec 3000 (“BioRad”, США) по соотношению поглощения при длинах волн 260 и 280 нм (А260/А280).
Фрагменты плазмиды pSM22 клонировали в вектор pGEM®-3zf(+) (“Promega”). С этой целью плазмидную ДНК обрабатывали смесью ДНК-полимеразы фага Т4, ДНК-киназы фага Т4 и ДНК-полимеразы I E. coli (все “Fermentas”, США) для достраивания концов. Для лигирования фрагментов плазмиды pSM22 5 мкг вектора обрабатывали эндонуклеазой рестрикции Ecl136II (“Fermentas”). К полученному гидролизату добавляли щелочную фосфатазу CIAP (“Fermentas”). Затем препарат линеаризованного дефосфорилированного вектора переосаждали этанолом, разводили до концентрации 10 нг/мкл в буфере ТЕ (50 мМ Трис-HCl, pH 8.0, 1 мМ EDТА) и хранили при –20°С. Лигирование проводили в 20 мкл лигазной смеси следующего состава: 1× Т4-ДНК-лигазный буфер (30 мМ Трис-HCl pH 7.8, 10 мМ MgCl2, 10 мМ дитиотреитола (ДТТ), 1 мМ АТР, 5% полиэтиленгликоля (ПЭГ6000)); 50 нг вектора pGEM®-3zf(+) (“Promega”), 3 ед. акт. Т4-ДНК-лигазы, 20 мкг фрагмента ДНК. Смесь тщательно перемешивали и инкубировали в течение 1 ч при 25°С.
Секвенирование клональных вставок библиотеки случайных фрагментов плазмидной ДНК выполняли по методу Сэнгера [22] с использованием набора реактивов Big Dye Terminator v.3.1 (“Applied Biosystems”, США) на автоматическом секвенаторе ABI PRIZM 3730 (“Applied Biosystems”) и универсальных плазмидных праймеров M13F и M13R. Чтение проводили с обоих концов клональной вставки.
Сборку и анализ последовательностей отдельных чтений осуществляли при помощи программного пакета Phred/Phrap/Consed software (http://cmgm.stanford.edu/help/manual/software/ sequence/phred-phrap.html). Аннотацию последовательности плазмиды проводили с помощью NCBI ORFfinder (https://www.ncbi.nlm.nih.gov/ orffinder). Выравнивание нуклеотидных и аминокислотных последовательностей выполняли в программе BioEdit [23]. Филогенетический анализ проводили в программе MEGA7 [24] с помощью метода ближайших соседей [25]. Статистическую значимость ветвления определяли с использованием бутстреп-анализа 1000 альтернативных деревьев [26], а эволюционные расстояния – с использованием поправки Пуассона [27].
Полная последовательность плазмиды pSM22 депонирована в базу данных GenBank под номером HQ896493.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Структура и особенности плазмиды pSM22
Препарат ДНК, выделенный из клеток штамма 22S, для предварительного определения длины плазмиды подвергли ферментативному гидролизу с использованием эндонуклеаз рестрикции KpnI, EcoRI, HincII, SalI и HindIII. В качестве маркера молекулярных масс использовали ДНК фага λ, гидролизованного HindIII. Выявлены сайты рестрикции всех эндонуклеаз за исключением HindIII (рис. 1а). Последующий ПДРФ-анализ, выполненный в программе Plasmid, позволил установить ее длину ~43 т.п.н. Размер pSM22, уточненный после полного секвенирования, составил 43190 п.н. (средний GC-состав – 58%) (рис. 1б).
Рис. 1.
Структура плазмиды pSM22. а – Электрофореграмма продуктов расщепления ДНК плазмиды эндонуклеазами рестрикции. 1 – Маркер молекулярных масс GeneRuler™ (“Thermo Scientific”, США), 2 – KpnI, 3 – EcoRI, 4 – HincII, 5 – SalI, 6 – λ/HindIII, 7 – препарат нативной ДНК плазмиды (суперскрученная и линейная формы). б – Карта плазмиды pSM22 с указанием расположения сайтов соответствующих эндонуклеаз. Стрелками указаны размер и расположение открытых рамок считывания (ОРС) в составе плазмиды. Разными цветами и типом линий выделены ОРС, относящиеся к различным модулям.
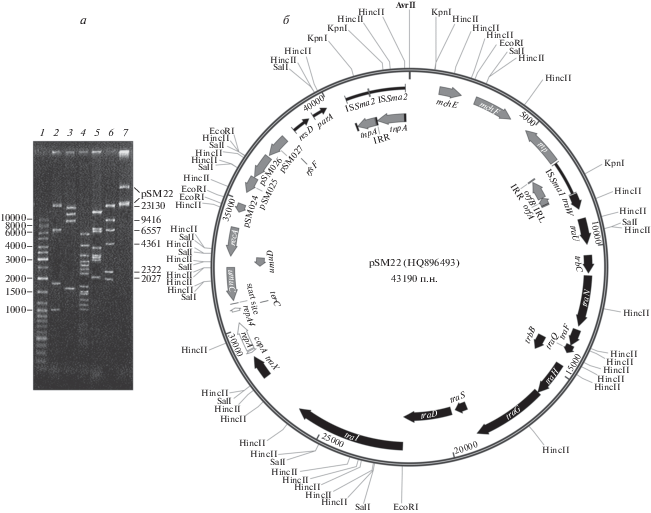
За начало кольцевой последовательности pSM22 принят первый нуклеотид уникального сайта эндонуклеазы AvrII (CCTAGG). Аннотация последовательности позволила выявить 31 возможную ОРС, из которых 19 транскрибируются с лидирующей, а 12 с отстающей цепи ДНК (табл. 1). Большинство из них кодируют белки с известными функциями, представленные в базе данных UniProtKB/Swiss-Prot, или имеют консервативные домены. Среди них белки, ответственные за репликацию (Rep), конъюгативный перенос (Tra), сегрегацию (Par), рекомбинацию и транспозицию, а также ряд белков системы секреции микроцина H47 и УФ-резистентности (табл. 1). В заключение следует отметить, что другие плазмиды в штамме S. marcescens 22S не обнаружены.
Таблица 1.
Открытые рамки считывания плазмиды pSM22
| ОРС | Предложен-ное имя гена | Положение, п.н. | G+C состав, мол. % | Длина, нуклеотид/ аминокислота) | Ближайший гомолог/функция/плазмида или геном | Степень сходства аминокислот, % | Величина E | Иденти-фикатор UniProt |
|---|---|---|---|---|---|---|---|---|
| ОРС1 | mchE | 1159–2019 | 56 | 861/286 | Белок MchE/секреция микроцина H47/Escherichia coli H47 | 76 | 7e-165 | Q9EXN6 |
| ОРС2 | mchF | 2586–4130 | 57 | 1545/514 | АТР-связывающий белок MchF/ секреция (процессинг) микроцина H47/E. coli H47 | 76 | 0.0 | Q9EXN5 |
| ОРС3 | tnp** | 6475–4856 | 60 | 1620/539 | Транспозаза ORF3 IS-элемента ISSfl3 семейства IS66/транспозиция/pWR100 | 70 | 0.0 | – |
| ОРС4 | orfB** | 7505–6672 | 53 | 834/277 | Транспозаза ORF2 IS-элемента ISKpn37 семейства IS3/транспозиция/pKP04VIM | 64 | 7e-131 | – |
| ОРС5 | orfA** | 7837–7520 | 54 | 318/105 | Транспозаза ORF1 IS-элемента ISKpn37 семейства IS3/транспозиция/pKP04VIM | 68 | 4e-45 | – |
| ОРС6 | traW | 7945–8535 | 63 | 591/196 | Белок TraW/конъюгация/F | 59 | 2e-80 | P18472 |
| ОРС7 | traU | 8921–9919 | 62 | 999/332 | Белок TraU/конъюгация/F | 75 | 0.0 | P18471 |
| ОРС8 | trbC | 10 346–11 020 | 64 | 675/224 | Белок TrbC/конъюгация/F | 50 | 8e-53 | P18473 |
| ОРС9 | traN | 11 127–13 094 | 61 | 1968/655 | Белок TraN/конъюгация/F | 47 | 0.0 | P24082 |
| ОРС10 | traF | 13 253—13 885 | 63 | 633/210 | Белок TraF/конъюгация/F | 65 | 3e-92 | P14497 |
| ОРС11 | traQ | 13 896–14 210 | 63 | 315/104 | Белок TraQ/конъюгация/F | 48 | 3e-19 | P18033 |
| ОРС12 | trbB | 14 068—14 733 | 63 | 666/221 | Белок TrbB/конъюгация/F | 58 | 7e-70 | P18035 |
| ОРС13 | traH | 14 730–16 097 | 60 | 1368/455 | Белок TraH/конъюгация/F | 78 | 0.0 | P15069 |
| ОРС14 | traG | 16 099–18 930 | 57 | 2832/943 | Белок TraG/конъюгация/F | 53 | 0.0 | P33790 |
| ОРС15 | traS | 18 927—19 457 | 39 | 531/176 | Белок TraS/конъюгация/F | 34 | 1e-26 | P09129 |
| ОРС16 | traD | 19 660–21 858 | 58 | 2199/732 | Белок TraD/конъюгация/F, p1658/97 | 59 | 0.0 | P09130 |
| ОРС17 | traI | 21 858–26 141 | 64 | 4284/1427 | Белок TraI/конъюгация/F | 55 | 0.0 | P14565 |
| ОРС18 | traX | 27 909–28 805 | 65 | 897/298 | Белок TraX/конъюгация/F | 37 | 4e-24 | P22709 |
| ОРС19 | repA (=repA1)* | 29 126–30 226 | 61 | 1101/366 | Белок семейства IncFII RepA/репликация | – | – | – |
| ОРС20 | repA4 | 30 690–30 815 | 56 | 126/41 | Неохарактеризованный белок repA4/не известна/NR1 (=R100) | 62 | 3e-09 | P03848 |
| ОРС21 | umuС | 32 382–31 108 | 65 | 1275/424 | Белок UmuC/SOS-ответ/E. coli K-12 | 61 | 0.0 | P04152 |
| ОРС22 | umuD | 32 817–32 437 | 66 | 381/126 | Белок UmuD/SOS-ответ/Salmonella typhimurium LT2 | 55 | 1e-43 | P22493 |
| ОРС23 | recA | 33 926–32 817 | 65 | 1110/369 | Белок RecA (рекомбиназа A)/SOS-ответ/Sodalis glossinidius | 64 | 2e-159 | P62220 |
| ОРС24 | pSM024 | 34 861–34 535 | 51 | 327/108 | Отсутствует | – | – | – |
| ОРС25 | pSM025 | 35 983–35 348 | 59 | 636/211 | Отсутствует | – | – | – |
| ОРС26 | pSM026 | 36 943–36 011 | 55 | 933/310 | Нуклеаза/участие в рекомбинации/pSLT | 63 | 1e-144 | P37415 |
| ОРС27 | pSM027 | 37 899–37 015 | 41 | 885/294 | Отсутствует | – | – | – |
| ОРС28 | resD | 38 365–39 108 | 64 | 744/247 | Резольваза (D белок) /стабильность плазмиды/pColBM-Cl139 | 47 | 6e-61 | P18021 |
| ОРС29 | parA* | 39 280–39 903 | 56 | 624/207 | Белок ParA/сегрегация | – | – | – |
| ОРС30 | tnpA** | 41 610–40 762 | 59 | 849/282 | Транспозаза IS-элемента ISVa3 семейства IS91 (частичная последовательность)/транспозиция/pEIB1 | 54 | 7e-92 | – |
| ОРС31 | tnpA** | 42 952–41 708 | 60 | 1245/414 | Транспозаза IS-элемента ISVa3 семейства IS91/транспозиция/pEIB1 | 54 | 1e-154 | – |
* Гены, идентифицированные по наличию консервативных аминокислотных доменов согласно базе данных Conserved Domain Database (CDD). ** Гены, идентифицированные в базе данных инсерционных последовательностей ISFinder (https://www-is.biotoul.fr/index.php).
Модуль репликации
Структуру модуля образуют гены и регуляторные элементы, необходимые для процесса репликации. Функционально они образуют единый репликон, на основе которого плазмиды классифицируют по группам несовместимости. Анализ ОРС плазмиды выявил ген repA (=repA1) (29 126–30 226), что определило присутствие структур модуля репликации на данной цепи плазмиды и позволило рассматривать эту цепь как вероятную лидирующую, а другую, соответственно, как отстающую. Аминокислотная последовательность, кодируемая геном repA, содержала аминокислотные остатки и мотивы, характерные для консервативного домена белков семейства IncFII RepA (рис. 2).
Рис. 2.
Аминокислотные мотивы в составе консервативного домена белка RepA плазмиды pSM22 (выделены черным), характерного для белков семейства IncFII RepA (pfam02387), согласно web-интерфейсу CD-Search (ресурс NCBI) для поиска в базе данных Conserved Domain Database (CDD).

Это дало основания анализировать структуру модуля репликации pSM22 с позиции поиска структурных элементов, характерных для репликона типа FII. При проведении сравнительного анализа в качестве прототипов использовали плазмиды NR1 (=R100=R222) (DQ364638) и R1 (KY749247), имеющие значительный уровень сходства между собой, а также единую и хорошо описанную структуру репликона данного типа [28]. Стоит упомянуть, что NR1 это трансмиссивная R-плазмида размером 94.5 т.п.н., несущая ряд генов устойчивости к различным антибиотикам. Эта плазмида стала архетипом большого числа сходных R-плазмид [29]. В составе репликона типа FII выделяют следующие ОРС: repA2 (ген copB), repA3, repA6 (ген tap), repA1 (=repA) и repA4. На противоположной цепи плазмиды между repA2 и repA6 находится короткая (91 п.н.) последовательность copA, кодирующая антисмысловую РНК (асРНК) CopA. Также между ОРС repA1 и repA4 выделяют область начала репликации ori (89 865–90 013). При этом ОРС repA3 и repA4 не имеют определенной функции и не транскрибируются [29, 30]. На лидирующей цепи плазмиды pSM22 обнаружен не только ген repA, но также сильно редуцированный ген repA4. Кроме того, мы обнаружили copA на отстающей цепи плазмиды (29 085–28 994). Однако в составе репликона отсутствовали рамки repA3 и repA6 (ген tap) (рис. 1б). Предполагаемая область ori между repA и repA4 не имела высокого уровня сходства с ori плазмид R1 и NR1. Тем не менее, идентифицирован in silico возможный сайт начала синтеза лидирующей цепи (3'-GCTCCCTACCCTAT-5') длиной 14 п.н., имеющий 71% сходства с этим сайтом плазмиды R1. Обычно в пределах репликона типа FII содержатся сайты терминации репликации (terC) [31]. В репликоне плазмиды pSM22 идентифицировано вероятное расположение одного из сайтов (30 801–30 814). При этом установлено, что последовательность этого сайта отличалась от R1 и NR1 заменой единственного нуклеотида, а уровень сходства составил 93%.
Таким образом, присутствие основных структурных элементов, характерных для плазмидных репликонов типа FII, позволило классифицировать pSM22 как плазмиду группы несовместимости IncFII. Однако полученные результаты указывают на значительные структурные изменения в репликоне pSM22, отличающие его от канонических плазмид R1 и NR1.
Модуль конъюгации
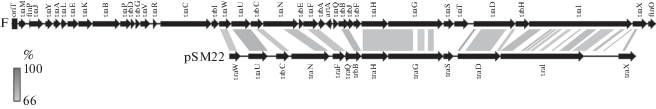
Модуль конъюгации плазмиды pSM22 представлен группой генов, кодирующих совокупность белков, ответственных за реализацию процесса конъюгативного переноса данной плазмиды (Tra-белки). Всего этот модуль насчитывает 13 ОРС, которые располагаются на лидирующей цепи плазмиды. Аминокислотные последовательности, кодируемые этими ОРС, содержат консервативные мотивы доменов, характерных для Tra-белков, а ближайшие гомологи в базе данных UniProtKB/Swiss-Prot найдены среди генов tra-оперона плазмиды F (табл. 1). Известно, что данная плазмида, называемая также F-фактор, длиной чуть более 99 т.п.н. (номер AP001918 в GenBank), содержит полный набор генов, необходимых для осуществления конъюгации. Всего в tra-опероне плазмиды F насчитывается 37 генов. Сравнение генов tra-оперонов плазмид F, R1 и NR1 c генами модуля конъюгации pSM22 показало, что уровень сходства между ними составляет 56, 58 и 57% соответственно. При этом стоит отметить, что плазмида F имеет репликон типа FI в отличие от репликона типа FII плазмид R1, NR1 и pSM22. Также выявлена необычная структура модуля конъюгации плазмиды pSM22. В частности, среди генов, вовлеченных в процесс конъюгации, отсутствуют такие важные гены, как finO, trbh, traT, trbF, trbJ, artA, trbA, trbE и полная группа генов от traC до traM, расположенных с 5'-конца последовательности плазмиды. (рис. 3). Указанные гены ответственны за образование поры спаривания (Mpf) и критически необходимы для процесса конъюгации [32]. Следует отметить, что в плазмиде pSM22 нам также не удалось обнаружить потенциальную область инициации переноса oriT. В плазмиде F эта область находится за геном traM, определяя, в том числе и начало модуля конъюгации. Кроме того, сравнительный анализ модулей конъюгации плазмиды F и pSM22 выявил области различной степени сходства – от 66 до 100%.
Рис. 3.
Сравнение молекулярно-генетических структур модулей конъюгации плазмид F и pSM22. Область начала переноса (oriT) плазмиды F представлена черным прямоугольником. Градациями серого обозначены области различной степени сходства нуклеотидных последовательностей (67–100%).
Один из важнейших генов – traI, кодирующий релаксазу, представлен в pSM22 в редуцированном (4284 п.н.) с 3'-конца виде. Между тем аминокислотная последовательность релаксазы может служить критерием классификации системы конъюгативного переноса [33] плазмиды pSM22. В белке TraI, кодируемом pSM22, идентифицированы три мотива, характерных для плазмид семейства MOBF. Внутри этого семейства плазмида pSM22 классифицирована нами как представитель клады F12 (рис. 4б). Для установления взаимоотношений между известными представителями клады F12 семейства MOBF и плазмиды pSM22 проведен их филогенетический анализ с использованием аминокислотных последовательностей релаксазы TraI. На рис. 3 представлено филогенетическое древо последовательностей релаксаз MOBF12, а также консервативные мотивы в их составе. Результаты филогенетического анализа указывают на то, что релаксазы, кодируемые плазмидами pSM22 и F, произошли от общего предка. Релаксаза pSM22 дивергировала значительно раньше, чем релаксазы плазмид R100 и pSLT, образующих единый кластер с F. Тем не менее, все они сохранили консервативные аминокислотные мотивы семейства MOBF12. Примечательно, что хотя консервативные мотивы релаксазы плазмиды pENTE01 отличаются от других родственных белков единственной заменой валина на изолейцин (V → I) в третьем мотиве (рис. 4б), на филогенетическом дереве она располагается скорее в качестве внешней группы. Этот факт указывает на высокую консервативность функционально важных аминокислотных мотивов релаксаз этой группы по сравнению со всей последовательностью белка.
Рис. 4.
Сравнительный анализ релаксазы, кодируемой плазмидой pSM22. а – Филогенетическое дерево аминокислотных последовательностей релаксаз (TraI) pSM22 и других плазмид семейства MOBF12. Масштаб соответствует замене одного аминокислотного остатка из 10. Цифрами указан показатель статистической значимости порядка ветвления (%) (значащими признаются значения выше 50), рассчитанной с помощью бутстреп-анализа. б – Консервативные мотивы выровненных аминокислотных последовательностей релаксаз плазмид семейства MOBF12 выделены черным.
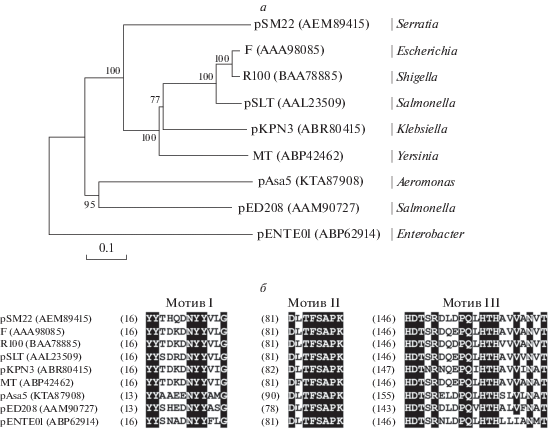
Результаты анализа строения модуля конъюгации pSM22 позволяют сделать вывод о “классической” архитектуре tra-генов, характерной для F-подобных плазмид подгруппы A группы MOBF12 [8]. Таким образом, на основании изучения строения модулей репликации и конъюгации плазмида pSM22 отнесена к группе IncF/MOBF12.
Модуль стабильности
Гены модуля стабильности отвечают за разрешение плазмидных мультимеров резольвазами и равномерное распределение (сегрегацию) плазмид в каждую из дочерних клеток (Par) при делении материнской клетки. Система разрешения мультимерных форм плазмиды pSM22 представлена геном resD (38365–39108), кодирующим резольвазу (D-белок). По результатам BLAST-поиска в базе данных UniProtKB/Swiss-Prot установлено, что наибольшей степенью сходства с белком-продуктом гена resD обладают D-белки плазмид pColBM-Cl139 и F, выполняющие функцию сайт-специфической резольвазы и репрессора транскрипции [34]. Возможная последовательность rfsF – сайта рекомбинации мультимеров pSM22, обладает 75%-ным сходством с rfsF плазмиды pColBM-Cl139 [35]. Анализ ОРС плазмиды pSM22 позволил выявить ген parA (39280–39903), кодирующий АТРазу ParA. Эта ATPаза содержит домены, характерные для ParA-подобных белков. Известно, что ParA вместе с плазмидным сайтом распределения parS и связывающимся с ним белком ParB образуют систему ParA-опосредованного распределения [36]. Однако в составе плазмиды не обнаружено ни гена parB, ни сайта parS (рис. 1б).
Полученные результаты указывают на неконтролируемое распределение pSM22 между дочерними клетками. Однако плазмида имеет все элементы системы разрешения мультимерных форм плазмидной ДНК, что способствует ее сегрегационной устойчивости.
Адаптивный (“грузовой”) модуль
Этот модуль образуют гены, которые могут быть вовлечены в адаптацию бактерий, а также те, чья функция остается неизвестной. У pSM22 они разбиты на несколько групп (табл. 1). Первая группа – гены секреции микроцина H47 (mchE и mchF), расположенные на лидирующей цепи плазмиды pSM22. Эта группа с обоих концов окружена несколькими новыми IS-элементами – ISSma1 и ISSma2 (названия присвоены базой данных ISFinder на основе общепринятых правил), локализованными на отстающей цепи (рис. 1б). С ее 5'-конца находятся два одинаковых ISSma2-элемента семейства IS91 (наиболее близки к ISVa3), причем их вставка в pSM22, по-видимому, произошла последовательно и с наложением друг на друга. А с ее 3'-конца располагается ISSma1-элемент семейства IS3, состоящий из двух ОРС – orfA и orfB. При этом аминокислотные последовательности, кодируемые этими ОРС, наиболее близки к ISKpn37. Интересно, что рядом с ISSma1 обнаружена ОРС3, кодирующая транспозазу. Ее ближайший гомолог обнаруживается в составе ISSfl3 семейства IS66. Согласно информации, представленной в базе данных ISFinder, IS-элементы этого семейства могут содержать три и более ОРС. Однако в плазмиде pSM22 не обнаружено других ОРС семейства IS66, кроме ОРС3. Известно, что помимо mchE и mchF в кластер генов микроцина H47 (MccH47) обычно входят еще пять генов: mchA, mchB, mchC, mchD и mchI, расположенных на хромосоме E. coli [37]. Эти гены не найдены в составе pSM22. Вторая группа включает область плазмиды pSM22 (33926–31108) со средним GС-составом 65%. Она состоит из трех ОРС, расположенных сразу за модулем репликации и аннотированных как гены SOS-системы: recA, umuD и umuC (рис. 1б). Третью группу составляют четыре ОРС (pSM024–pSM027) с GC-составом 51, 59, 55, и 41%, соответственно (рис. 1), гомологи которых не найдены. Значения GC-состава у них значительно ниже, чем у других модулей плазмиды, например конъюгации (60%) и репликации (61%), за исключением pSM025 (59%). Это может указывать на включение данных генов в состав плазмиды из хромосомы бактерии хозяина или другой плазмиды посредством рекомбинации. Дальнейший анализ показал, что эти ОРС кодируют консервативные домены белков с неизвестной функцией.
Расположение групп генов адаптивного модуля, а также присутствие IS-элементов указывают на мозаичную природу pSM22, свойственную большинству плазмид.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Согласно современным представлениям, структурная организация генов плазмид связана с их функциональным назначением. Так, все эти гены можно разделить на несколько групп, которые локализуются в определенной области плазмиды и образуют модули [38]. В некоторых публикациях, посвященных молекулярной организации плазмид, модули объединяют в сектора так называемых общих функций поддержания и конъюгации [39]. Принимая во внимание эти представления, мы описали плазмиду pSM22 как структуру, в которой обнаружены модули репликации, конъюгации, стабильности, а также адаптивный модуль. Модуль, отвечающий за репликацию, обеспечивает само существование плазмиды. Обсуждая его строение, следует напомнить, что в нем отсутствуют важные регуляторные элементы – ген лидерного пептида tap и ген системы отрицательного контроля репликации copB. Известно, что белок CopB и асРНК CopA ограничивают экспрессию repA на транскрипционном и посттранскрипционном уровнях соответственно. Белок CopB осуществляет репрессию транскрипции мРНК гена repA, блокируя его промотор, а асРНК CopA образует РНК-РНК-дуплекс с гомологичной областью мРНК гена лидерного пептида tap, препятствуя его трансляции. Это, в свою очередь, блокирует необходимое соединение мРНК генов tap и repA при трансляции [40]. Эти данные, а также успешная репликация pSM22 делают очевидным, что приобретенные структурные изменения репликона сняли ограничение на копийность плазмиды. Таким образом, на основе строения модуля репликации можно сделать вывод о том, что функционально базовым репликоном pSM22 является область длиной около 2000 п.н. от сайта начала репликации лидирующей цепи плазмиды до последовательности, кодирующей асРНК CopA (рис. 1б). Эти выводы подтверждаются результатами [29], согласно которым ген repA и область ori относятся к элементам, минимально необходимым для осуществления функции репликации NR1. Очевидно, репликон pSM22 претерпел значительные изменения в процессе эволюции. Получив полный набор регуляторных элементов от общей с NR1 предковой плазмиды, pSM22 утратила некритические компоненты модуля репликации после многочисленных генных перестроек и под давлением факторов среды. Оставшиеся критические компоненты эволюционировали в разной степени. Например, белок RepA сохранил мотивы консервативного домена. А обнаруженные сайты начала репликации лидирующей цепи и терминации репликации (terC) сохранили гомологию с соответствующими предковыми регуляторными элементами плазмиды NR1 на уровне 71 и 93% соответственно, в то время как область ori изменилась в значительной степени. Учитывая данные о высокой скорости эволюции репликонов IncF [41], мы предполагаем, что pSM22 может иметь новый подтип репликона типа FII. Однако проверка этой гипотезы требует отдельного исследования.
Так как в модуле конъюгации отсутствует больше половины генов, необходимых для нормального переноса плазмиды между бактериальными клетками, можно предположить, что плазмида pSM22 утратила способность к конъюгации. Следует отметить, что ряд генов, ответственных за ту или иную функцию в процессе конъюгации, может отсутствовать у многих плазмид, при этом они сохраняют способность к конъюгации [8]. Также нельзя отрицать возможность функциональной компенсации отсутствия необходимых генов, путем использования хромосомных генов бактерии-хозяина. Однако, по-видимому, это не касается плазмиды pSM22, утратившей слишком большое число критически важных генов (например, генов контроля образования поры спаривания) и регуляторных элементов (область инициации переноса oriT). Косвенно это подтверждается редуцированностью белка релаксазы TraI, который в силу этого утратил способность к осуществлению своих функций. Результаты сравнительного анализа модуля конъюгации плазмид pSM22 и F указывают на постепенную деградацию этой области, утратившей свою функциональную значимость. Судя по сходному строению модулей репликации и конъюгации плазмид pSM22 и NR1, а также по наличию областей высокой гомологии между ними, можно утверждать, что они произошли от одного предка. В случае модуля конъюгации это подтверждается также результатами филогенетического анализа релаксазы TraI. Известно, что модули могут эволюционировать независимо и иметь разное происхождение [38]. Однако в случае модулей репликации и конъюгации pSM22 и NR1 мы имеем дело с одной предковой плазмидой.
Известно, что функционирование системы сегрегации плазмид обеспечивается генами parA и parB в составе одного оперона, а также сайтом parS [42]. Анализируя строение модуля стабильности pSM22, мы пришли к заключению об отсутствии части системы сегрегации parABS, которая представлена всего одним геном – parA. Поэтому отсутствие гена parB и сайта parS указывает на неконтролируемую сегрегацию pSM22 между дочерними клетками. Мы предполагаем, что ген parB был утрачен, поскольку отпала необходимость в нем для работы системы parABS. Вероятно, предковая плазмида pSM22 имела больший размер и, как следствие, меньшую копийность. В этом случае система контролируемой сегрегации была ей жизненно необходима. При этом следует отметить, что эти предположения согласуются с изменениями в структуре репликона плазмиды, приведшими к редукции структур, ограничивающих ее копийность. В отличие от parABS система разрешения мультимерных форм плазмиды может выполнять свои функции, хотя она также представлена одним геном resD (D-белок), так как в этом случае мы обнаружили сайт-мишень белка D – последовательность rfsF. Известно, что образование мультимеров может приводить к неравномерной сегрегации и, как следствие, к потере плазмид клетками [43]. Поэтому сохранность всех элементов системы разрешения мультимерных форм pSM22 можно объяснить их критической необходимостью в отличие от элементов системы parABS. Исходя из результатов BLAST-поиска гомологов D-белка и особенностей строения модуля конъюгации, можно заключить, что ген этого белка также достался pSM22 от общего с плазмидой F предка в отличие от гена рarA, гомологов которого не было у F-подобных плазмид. Отсутствие гомологов может свидетельствовать о том, что систему parABS плазмида pSM22 могла получить от эволюционно очень далекого не F‑подобного предка до утраты гена parB и сайта parS, не описанного на данный момент. Таким образом произошла не только утрата части модуля стабильности, но и замена генов.
В составе адаптивного (“грузового”) модуля, как показано выше, мы обнаружили ряд генов, вовлеченных в индукцию SOS-ответа, секрецию микроцинов, а также генов с неизвестной функцией. Известно, что инициация SOS-ответа находится под контролем белка-репрессора LexA, который при повреждении ДНК подвергается автокаталитическому протеолизу, индуцируемому белком RecA. Это, в свою очередь, приводит к дерепрессии SOS-генов [44, 45]. Ген lexA, в отличие от гена recA, в плазмиде pSM22 не обнаружен. Однако утверждать, что SOS-гены экспрессируются конститутивно, вряд ли можно в силу нескольких причин. Во-первых, ген lexA всегда представлен на хромосоме бактерии-хозяина. Во-вторых, опубликованы данные, согласно которым кодируемый плазмидой гомолог UmuD (субъединица полимеразы V) регулирует экспрессию SOS-генов [46]. Напомним, что гомолог UmuD кодируется также плазмидой pSM22.
Следует отметить, что SOS-система репарации ДНК включает несколько десятков генов [47], которые отсутствуют в составе pSM22. Однако имеются данные о возникновении SOS-индукции у бактерий в присутствии плазмид [48], а также о повышении радиоустойчивости бактерий, несущих мутантный аллель плазмидного гена recA [49]. Учитывая эти данные, вопрос о контроле экспрессии и работе SOS-системы плазмиды pSM22 и ее влиянии на устойчивость штамма-хозяина к УФ-излучению остается открытым. Тем не менее, полученные результаты позволяют классифицировать pSM22 как плазмиду УФ-резистентности. Считается, что такие плазмиды играют важную роль в экологии природных бактериальных сообществ, подвергающихся воздействию солнечной радиации или различных химических веществ, разрушающих ДНК [50]]. Следует отметить, что именно из популяций, длительно находящихся под влиянием подобных факторов, выделен штамм-деструктор ксенобиотиков (фенола и дихлорфенола) S. marcescens 22S, несущий плазмиду pSM22 [51]. Бактерии, обитающие в таких местах, как правило, содержат плазмиды с генами контроля деградации ксенобиотиков. Значительная их часть представлена плазмидами группы несовместимости IncP [52–54], в то время как плазмиды IncF играют главную роль в устойчивости к антибиотикам [41]. Однако на pSM22 отсутствуют гены устойчивости к антибиотикам. Следует отметить, что детерминанты УФ-резистентности отмечены ранее в составе плазмид групп несовместимости IncB, IncI, IncJ, IncL, IncM, IncN [50, 55]], в то время как у плазмид IncF такие детерминанты описаны впервые в нашей работе. С учетом сказанного можно предполагать, что гены контроля УФ-резистентности достаточно редко присутствуют на плазмидах группы IncFII, особенно на плазмидах, выделенных из бактерий рода Serratia. Следует отметить, что мы не обнаружили генов контроля деградации ксенобиотиков в составе исследуемой плазмиды, вероятно, вследствие их хромосомной локализации. Ранее мы уже сталкивались с этим при анализе плазмиды pCS36-4CPA семейства ColE1 другого штамма-деструктора [56].
Обсуждая систему синтеза микроцина H47 (MccH47), следует обратить внимание на то, что обнаруженные на плазмиде гены – mchE и mchF – контролируют только функции его секреции и процессинга, но не синтез и иммунитет к нему [57]. Учитывая данные о хромосомной локализации генов mchE и mchF E. coli в составе одного кластера, можно предположить, что гены секреции mchE и mchF были “захвачены” плазмидой при ее реинтеграции из хромосомы бактерии-хозяина или в процессе рекомбинации. Отметим, что данные о плазмидной или хромосомной локализации генов системы микроцина H47 у бактерий S. marcescens отсутствовали.
Также в составе “грузового” модуля выявлен ряд ОРС с неизвестной функцией. Так как их GC-состав отличается от состава других “истинно” плазмидных генов, можно предположить, что их происхождение отличается от происхождения остальной части плазмиды.
Структура адаптивного модуля указывает на мозаичную природу последовательности плазмиды pSM22, что характерно и для других плазмид группы IncF/MOBF12, в частности, для F, R1 и NR1 (=R100=R222) [58]. Модули репликации, конъюгации и стабильности унаследованы pSM22 от общего предка с другими F-подобными плазмидами, в то время как адаптивный модуль, как конструктор, собран из разных генных кластеров. Они располагаются в разных частях плазмиды, имеют различный GC-состав и, подобно генам секреции микроцина H47, могут иметь хромосомное происхождение. Таким образом, интенсивные генетические перестройки в адаптивном модуле, на которые указывает также присутствие IS-элементов, обуславливают высокую пластичность генома бактерии-хозяина S. marcescens 22S.
Обобщая сказанное, следует отметить, что почти для всех модулей плазмиды (репликации, конъюгации и стабильности) pSM22 характерно упрощение структуры, что говорит о ее редукционной эволюции. Структура и функционирование pSM22 более сходны с малыми плазмидами с их сравнительно небольшим размером, большим числом копий в клетке и отсутствием способности к конъюгации. Одновременно с этим происходят интенсивные геномные перестройки в модуле адаптивности. Все это указывает на важную роль pSM22 в обеспечении адаптационных преимуществ бактерии-хозяина в изменяющихся условиях окружающей среды.
ЗАКЛЮЧЕНИЕ
Таким образом, при помощи секвенирования и сравнительного геномного анализа нами изучена плазмида pSM22 группы IncF/MOBF12 бактерий рода Serratia, которая, вероятно, обладает новым подтипом репликона типа FII. Раскрыты особенности эволюции pSM22 и филогенетических взаимоотношений с другими плазмидами. Впервые на плазмиде типа FII обнаружены гены контроля SOS-ответа, процессинга и секреции микроцина H47.
Авторы благодарят Б.Б. Кузнецова, Б.К. Бумажкина, Т.В. Колганову и Е.О. Патутину (Институт биоинженерии РАН, Москва) за помощь в проведении экспериментов.
В работе использована инфраструктура и оборудование Центров коллективного пользования “Биоинженерия” (Москва) и “Агидель”(Уфа).
Работа выполнена в рамках Государственного задания (№ гос. регистрации АААА-А18-118022190098-9).
Настоящая статья не содержит каких-либо исследований с использованием животных в качестве объектов.
Настоящая статья не содержит каких-либо исследований с участием людей в качестве объектов исследований.
Список литературы
Norman A., Hansen L.H., Sørensen S.J. (2009) Conjugative plasmids: vessels of the communal gene pool. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 364, 2275–2289.
Petrova M., Kurakov A., Shcherbatova N., Mindlin S. (2014) Genetic structure and biological properties of the first ancient multiresistance plasmid pKLH80 isolated from a permafrost bacterium. Microbiology. 160, 2253–2263.
Zhai Y., He Z., Kang Y., Yu H., Wang J., Du P., Zhang Z., Hu S., Gao Z. (2016) Complete nucleotide sequence of pH11, an IncHI2 plasmid conferring multi-antibiotic resistance and multi-heavy metal resistance genes in a clinical Klebsiella pneumoniae isolate. Plasmid. 86, 26–31.
Филонов А.Е., Ахметов Л.И., Пунтус И.Ф., Есикова Т.З., Гафаров А.Б., Кошелева И.А., Боронин А.М. (2010) Горизонтальный перенос катаболических плазмид и биодеградация нафталина в открытой почве. Микробиология. 79(2), 206–212.
Панов А.В., Волкова О.В., Пунтус И.Ф., Есико-ва Т.З., Кошелева И.А., Боронин А.М. (2013) scpA – новый ген салицилатгидроксилазы, локализованный на плазмидах деградации салицилата/капролактама. Молекуляр. биология. 47, 116–123.
Orlek A., Phan H., Sheppard A.E., Doumith M., Ellington M., Peto T., Crook D., Walker A.S., Woodford N., Anjum M.F., Stoesser N. (2017) Ordering the mob: Insights into replicon and MOB typing schemes from analysis of a curated dataset of publicly available plasmids. Plasmid. 91, 42–52.
Rozhon W., Petutschnig E., Khan M., Summers D.K., Poppenberger B. (2010) Frequency and diversity of small cryptic plasmids in the genus Rahnella. BMC Microbiol. 10, 56.
Fernandez-Lopez R., de Toro M., Moncalian G., Garcillan-Barcia M.P., de la Cruz F. (2016) Comparative genomics of the conjugation region of F-like plasmids: five shades of F. Front. Mol. Biosci. 3, 71.
Ying J., Wu S., Zhang K., Wang Z., Zhu W., Zhu M., Zhang Y., Cheng C., Wang H., Tou H., Zhu C., Li P., Ying J., Xu T., Yi H., Li J., Ni L., Xu Z., Bao Q., Lu J. (2015) Comparative genomics analysis of pKF3-94 in Klebsiella pneumoniae reveals plasmid compatibility and horizontal gene transfer. Front. Microbiol. 6, 831.
Kubasova T, Cejkova D, Matiasovicova J, Sekelova Z., Polansky O., Medvecky M., Rychlik I., Juricova H. (2016) Antibiotic resistance, core-genome and protein expression in IncHI1 plasmids in Salmonella typhi-murium. Genome Biol. Evol. 8, 1661–1671.
Cain A.K., Hall R.M. (2013) Evolution of IncHI1 plasmids: two distinct lineages. Plasmid. 70, 201–208.
Johnson T.J., Shepard S.M., Rivet B., Danzeisen J.L., Carattoli A. (2011) Comparative genomics and phylogeny of the IncI1 plasmids: a common plasmid type among porcine enterotoxigenic Escherichia coli. Plasmid. 66, 144–151.
Mahlen S.D. (2011) Serratia infections: from military experiments to current practice. Clin. Microbiol. Rev. 24, 755–791.
Chen S., Blom J., Walker E.D. (2017) Genomic, physiologic, and symbiotic characterization of Serratia marcescens strains isolated from the mosquito Anopheles stephensi. Front. Microbiol. 8, 1483.
Гарипова С.Р., Гарифуллина Д.В., Баймиев А.Х., Хайруллин Р.М. (2017) Межмикробные взаимоотношения бактерий Serratia sp. Ent16 – симбионта клубенька гороха и колонизация ими эндоризосферы хозяина. Прикл. биохимия и микробиология. 53, 299–307.
Iguchi A., Nagaya Y., Pradel E., Ooka T., Ogura Y., Katsura K., Kurokawa K., Oshima K., Hattori M., Parkhill J., Sebaihia M., Coulthurst S.J., Gotoh N., Thomson N. R., Ewbank J.J., Hayashi T. (2014) Genome evolution and plasticity of Serratia marcescens, an important multidrug-resistant nosocomial pathogen. Genome Biol. Evol. 6, 2096–2110.
Zhao W.H., Hu Z.Q., Chen G., Matsushita K., Fukuchi K., Shimamura T. (2007) Characterization of imipenem-resistant Serratia marcescens producing IMP-type and TEM-type β-lactamases encoded on a single plasmid. Microbiol. Res. 162, 46–52.
da Costa Guimarães A.C., Almeida A.C., Nicoletti A.G., Vilela M.A., Gales A.C., de Morais M.M. (2013) Clonal spread of carbapenem-resistant Serratia marcescens isolates sharing an IncK plasmid containing blaKPC-2. Int. J. Antimicrob. Agents. 42, 369–370.
Kehinde F.O., Isaac S.A. (2017) Chromosomal and plasmid mediated degradation of crude oil by Bacillus coagulans, Citrobacter koseri and Serratia ficaria isolated from the soil. Afr. J. Biotechnol. 16, 1242–1253.
Birnboim H.C., Doly J. (1979) A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucl. Acids Res. 7, 1513–1523.
Ogden R.C., Adams D.A. (1987) Electrophoresis in agarose and acrylamide gels. Methods Enzymol. 152, 61–87.
Sanger F., Nicklen S., Coulson A.R. (1977) DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. USA. 84, 5463–5467.
Hall T.A. (1999) BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids. Symp. 41, 95–98.
Kumar S., Stecher G., Tamura K. (2016) MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 33, 1870–1874.
Saitou N., Nei M. (1987) The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4, 406–425.
Felsenstein J. (1985) Confidence limits on phylogenies: an approach using the bootstrap. Evolution. 39, 783–791.
Zuckerkandl E., Pauling L. (1965) Evolutionary divergence and convergence in proteins. In: Evolving Genes and Proteins. Eds Bryson V., Vogel H.J. N.Y.: Acad. Press, 97–166.
Nordström M., Nordström K. (1985) Control of replication of FII plasmids: comparison of the basic replicons and of the copB systems of plasmids Rl00 and Rl. Plasmid. 13, 81–87.
Womble D.D., Rownd R.H. (1988) Genetic and physical map of plasmid NR1: comparison with other IncFII antibiotic resistance plasmids. Microbiol. Rev. 52, 433–451.
Jiang T., Min Y.-N., Liu W., Womble D.D., Rownd R.H. (1993) Insertion and deletion mutations in the repA4 region of the IncFII plasmid NR1 cause unstable inheritance. J. Bacteriol. 175, 5350–5358.
Bernander R., Krabbe M., Nordström K. (1992) Mapping of the in vivo start site for leading strand DNA synthesis in plasmid R1. EMBO J. 11, 4481–4487.
Schröder G., Lanka E. (2005) The mating pair formation system of conjugative plasmids – a versatile secretion machinery for transfer of proteins and DNA. Plasmid. 54, 1–25.
Garcillán-Barcia M.P., Francia M.V., de la Cruz F. (2009) The diversity of conjugative relaxases and its application in plasmid classification. FEMS Microbiol. Rev. 33, 657–687.
Lane D., de Feyter R., Kennedy M., Phua S.H., Semon D. (1986) D protein of miniF plasmid acts as a repressor of transcription and as a site-specific resolvase. Nucl. Acids Res. 14, 9713–9728.
Thumm G., Olschläger T., Braun V. (1988) Plasmid pColBM-Cl139 does not encode a colicin lysis protein but contains sequences highly homologous to the D protein (resolvase) and the oriV region of the miniF plasmid. Plasmid. 20, 75–82.
Hwang L.C., Vecchiarelli A.G., Han Y.W., Mizuuchi M., Harada Y., Funnell B.E., Mizuuchi K. (2013) ParA-mediated plasmid partition driven by protein pattern self-organization. EMBO J. 32, 1238–1249.
Gaggero C., Moreno F., Laviña M. (1993) Genetic analysis of microcin H47 antibiotic system. J. Bacteriol. 175, 5420–5427.
Garcillán-Barcia M.P., Alvarado A., de la Cruz F. (2011) Identification of bacterial plasmids based on mobility and plasmid population biology. FEMS Microbiol. Rev. 35, 936–956.
Fernández-López, R., Pilar Garcillán-Barcia, M., Revilla, C., Lázaro, M., Vielva, L.,de La Cruz, F. (2006) Dynamics of the IncW genetic backbone imply general trends in conjugative plasmid evolution. FEMS Microbiol. Rev. 30, 942–966.
del Solar G., Giraldo R., Ruiz-Echevarría M.J., Espinosa M., Díaz-Orejas R. (1998) Replication and control of circular bacterial plasmids. Microbiol. Mol. Biol. Rev. 62, 434–464.
Villa L., García-Fernández A., Fortini D., Carattoli A. (2010) Replicon sequence typing of IncF plasmids carrying virulence and resistance determinants. J. Antimicrob. Chemother. 65, 2518–2529.
Surtees J.A., Funnell B.E. (2003) Plasmid and chromosome traffic control: how ParA and ParB drive partition. Curr. Top. Dev. Biol. 56, 145–9180.
Crozat E., Fournes F., Cornet F., Hallet B., Rousseau P. (2014) Resolution of multimeric forms of circular plasmids and chromosomes. Microbiol. Spectr. 2, PLAS-0025-2014.
Little J.W. (1982) Control of the SOS regulatory system by the level of RecA protease. Biochimie. 64, 585–589.
Bell J.C., Kowalczykowski S.C. (2016) RecA: regulation and mechanism of a molecular search engine. Trends. Biochem. Sci. 41, 491–507.
Díaz-Magaña A., Alva-Murillo N., Chávez-Moctezuma M.P., López-Meza J.E., Ramírez-Díaz M.I., Cervantes C. (2015) A plasmid-encoded UmuD homologue regulates expression of Pseudomonas aeruginosa SOS genes. Microbiology. 161, 1516–1523.
Ушаков В.Ю. (2010) SOS-система репарации ДНК у бактерий (обзор). Вест. Пермского ун-та. Сер. Биология. 2, 19–30.
Тиганова И.Г., Русина О.Ю., Андреева И.В. (2006) SOS-индукция в присутствии плазмиды рКМ101 в бактериальных клетках Escherichia coli K12. Молекуляр. генетика, микробиология и вирусология. 1, 11–14.
Вербенко В.Н., Кузнецова Л.В., Крупьян Е.П., Суслов А.В. (2009) Оператор-конститутивная мутация в гене recA повышает радиоустойчивость Escherichia coli. Генетика. 45, 1048–1054.
Kokjohn T.A., Miller R.V. (1994) IncN plasmids mediate UV resistance and error-prone repair in Pseudomonas aeruginosa PAO. Microbiology. 140, 43–48.
Коробов В.В., Маркушева Т.В., Кусова И.В., Журенко Е.Ю., Галкин Е.Г., Жарикова Н.В., Гафиятова Л.Р. (2006) Штамм бактерий Serratia marcescens В-6493 – деструктор фенола и дихлорфенола. Биотехнология. 2, 63–65.
Волкова О.В., Панов А.В., Кошелева И.А., Боронин А.М. (2013) Классификация IncP-7 плазмид, основанная на структурном разнообразии их базовых репликонов. Молекуляр. биология. 47, 232–242.
Волкова О.В., Кошелева И.А., Боронин А.М. (2013) Организация и особенности поддержания базового репликона IncP-7-плазмиды биодеградации нафталина pFME5. Генетика. 49, 558–568.
Сечеников А.А., Ковальчук К.В., Василенко С.Л., Титок М.А. (2013) Селекция мутантов по системе инициации репликации плазмиды pBS267 группы IncP-9. Генетика. 49, 189–195.
Pinney R.J. (1980) Distribution among incompatibility groups of plasmids that confer UV mutability and UV resistance. Mutat. Res. 72, 155–159.
Zharikova N., Iasakov T., Bumazhkin B., Patutina E., Zhurenko E., Korobov V., Sagitova A., Kuznetsov B., Markusheva T. (2018) Isolation and sequence analysis of pCS36-4CPA, a small plasmid from Citrobacter sp. 36-4CPA. Saudi J. Biol. Sci. 25, 660–671.
Azpiroz M.F., Rodríguez E., Laviña M. (2001) The structure, function, and origin of the microcin H47 ATP-binding cassette exporter indicate its relatedness to that of colicin V. Antimicrob. Agents Chemother. 45, 969–972.
Cox K.E.L., Schildbach J.F. (2017) Sequence of the R1 plasmid and comparison to F and R100. Plasmid. 91, 53–60.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


