Молекулярная биология, 2019, T. 53, № 6, стр. 1029-1037
Эволюция системы координированной регуляции экспрессии протеасомных генов у дрожжей-сахаромицетов
Д. С. Карпов a, *, Ю. П. Лысов a, В. Л. Карпов a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: aleom@yandex.ru
Поступила в редакцию 29.05.2019
После доработки 05.06.2019
Принята к публикации 05.06.2019
Аннотация
26S протеасома – это мультисубъединичный АТР-зависимый протеазный комплекс, необходимый для нормального функционирования любой эукариотической клетки и ее выживания в условиях стресса. Двадцать лет назад нами в сотрудничестве с коллегами из Германии была впервые экспериментально описана система координированной регуляции экспрессии протеасомных генов у дрожжей Saccharomyces cerevisiae, состоящая из фактора транскрипции ScRpn4 и его сайта связывания, названного PACE. На основании результатов биоинформатического поиска, проведенного по первым секвенированным геномам дрожжей, постулировано существование Rpn4-подобных белков и PACE-подобных элементов у других видов дрожжей-сахаромицетов (класс Saccharomycetes). Нами экспериментально охарактеризованы Rpn4-подобные белки из биотехнологически значимых видов дрожжей Komagataella pfaffii (Pichia pastoris), Yarrowia lipolytica и Debaryomyces hansenii, а также условно-патогенных Candida glabrata. С накоплением большого количества информации о нуклеотидных последовательностях геномов новых видов и штаммов дрожжей возникает вопрос о разнообразии структур и вероятных механизмов функционирования системы регуляции протеасомных генов. В настоящей работе проведен биоинформатический поиск Rpn4-подобных белков и PACE-подобных элементов у 3111 штаммов, принадлежащих к 427 видам дрожжей-сахаромицетов. Показано, что у Rpn4-подобных белков консервативен только ДНК-связывающий домен, что соответствует консервативности PACE-элементов. Выявлены системы, содержащие более одного Rpn4-подобного белка, отличающиеся по структуре ДНК-связывающих доменов, а также системы с авторегуляцией генов Rpn4-подобных белков. Учитывая роль Rpn4-подобных белков, а также протеасом в клеточном ответе на стресс, можно предположить, что обнаруженное разнообразие систем регуляции протеасомных генов соответствует приспособленности организмов к условиям обитания.
ВВЕДЕНИЕ
26S протеасома – это АТР-зависимый мультисубъединичный протеазный комплекс, состоящий из 20S протеолитического субкомплекса, нековалентно соединенного с 19S регуляторным субкомплексом [1, 2]. 26S протеасома отвечает за деградацию большинства внутриклеточных белков, в том числе регуляторных, поврежденных и неправильно свернутых, контролируя тем самым все основные клеточные процессы и ответ на стресс. Двадцать лет назад в сотрудничестве с коллегами из Германии нами впервые была описана система координированной регуляции экспрессии протеасомных генов у пекарских дрожжей Saccharomyces cerevisiae [3, 4]. Промоторные области большинства генов субъединиц протеасомы содержат элемент PACE (Proteasome Associated Control Element) (5'-GGTGGCAAA-3'), с которым связывается Rpn4. Фактор Rpn4 S. cerevisiae (ScRpn4) регулирует не только протеасомные гены, но и множество других, включая гены, кодирующие белки различных антистрессовых ответов [5].
В ходе биоинформатического анализа геномов различных организмов PACE-подобные элементы и гены, кодирующие Rpn4-подобные белки, выявлены только у дрожжей класса Saccharomycetes [6]. Нами показано, что Rpn4-подобные белки у таких дрожжей, как Candida glabrata, Yarrowia lipolytica, Komagataella pfaffii и Debaryomyces hansenii, несмотря на их низкое общее сходство, способны комплементировать делецию гена RPN4 у S. cerevisiae [7‒9], а также регулировать экспрессию протеасомных генов посредством PACE и подобных ему элементов [8]. Полученные результаты подтверждают родство обнаруженных Rpn4-подобных белков и открывают возможность проведения более широкого биоинформатического поиска PACE-подобных элементов и Rpn4-подобных белков в накопленных к настоящему моменту данных о секвенированных геномах различных дрожжей.
В настоящей работе выполнен биоинформатический поиск и охарактеризованы компоненты системы координированной регуляции экспрессии протеасомных генов более чем у 3000 штаммов, принадлежащих 427 видам дрожжей-сахаромицетов.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Поиск последовательностей белков, похожих на субъединицы протеасомы и Rpn4 S. cerevisiae. Список последовательностей 34 субъединиц протеасом и фактора транскрипции Rpn4 S. cerevisiae получен из базы данных (http://www.yeastgenome.org/, по состоянию на 5.04.2019). Список видов Saccharomycetes с секвенированными геномами (427) получен с сайта www.ncbi.nlm.nih.gov/genome с помощью запроса “saccharomycetes[orgn]” (по состоянию на 13.04.19). Нуклеотидные последовательности геномов 3111 штаммов, принадлежащих этим видам дрожжей, получены из депозитория GenBank (ftp://ftp.ncbi.nlm.nih.gov/genomes/genbank/fungi/) в формате аннотированных файлов, содержащих в названии “_genomic.gbff”. Следует отметить, что первое место в этом списке занимает вид S. cerevisiae (1005 штаммов), вторым по представленности является Candida albicans (93 штамма), замыкает тройку Komagataella pastoris (67 штаммов). Большинство других видов представлено в основном тремя‒пятью штаммами. С помощью скрипта, написанного на языке С, последовательности геномов были виртуально транслированы во всех шести рамках считывания. С помощью локально установленной программы BLAST (версия 2.9.0, ftp://ftp.ncbi.nlm.nih.gov/blast/executables/ blast+/LATEST/) в полученных виртуальных протеомах выполнен поиск последовательностей, наиболее близких (E-value < 10–70) к последовательностям субъединиц протеасом и Rpn4.
Поиск Rpn4-подобных белков у видов, не входящих в род Saccharomyces. Поскольку последовательность фактора транскрипции Rpn4 в целом не консервативна, то поиск Rpn4-подобных белков у дрожжей, не принадлежащих к роду Saccharomyces, выполнен c помощью его наиболее консервативного ДНК-связывающего домена [6] (аминокислотные остатки 439‒531). Поиск осуществляли с помощью PSI-BLAST с параметрами по умолчанию в классе Saccharomycetes, исключив виды Saccharomyces. Полученные последовательности использовали в построении множественного выравнивания с помощью COBALT [10] с параметрами по умолчанию. Результат выравнивания визуализировали с помощью COBALT и Jalview2 [11]. Ключевым критерием включения белка в список Rpn4-подобных белков считали идентичность положения большинства распознающих аминокислотных остатков в обоих доменах “цинковых пальцев”.
Поиск сайтов PACE в промоторных областях генов, близких по последовательности к протеасомным генам и RPN4 S. cerevisiae. Из генома извлекали участки длиной 500 п.н., расположенные левее старт-кодона трансляции в генах, кодирующих сходные белки, отобранные на предыдущих этапах. Внутри этих участков проводили поиск PACE-подобных элементов по обеим цепям. Чтобы выявить ключевые основания PACЕ-подобных элементов, проанализировали результаты экспериментального исследования сродства Rpn4 к PACE-подобным последовательностям, мутированным вариантам PACE [4, 12‒14], а также профили консенсусов PACE-подобных элементов у изученных нами ранее штаммов дрожжей Y. lipolytica, K. phaffii, D. hansenii и C. glabrata [7‒9]. Инвариантными, характеризующими PACE-подобный элемент, приняты гуанин в четвертом положении и аденин в восьмом положении сайта. Поиск PACE-подобных сайтов выполняли с помощью скрипта, написанного на языке С.
Визуализация результатов поиска PACE-подобных сайтов. Профили консервативных нуклеотидов в обнаруженных PACE-подобных элементах строили с помощью WebLogo 3.6.0 (http://weblogo.threeplusone.com/create.cgi) [15]. Тепловые карты нуклеотидных замен в PACE строили с помощью онлайн сервиса Heatmapper (http://www1.heatmapper.ca/) [16]. Входные данные для обработки в указанных сервисах готовили с помощью скриптов, написанных на языке программирования Python.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Распределение и структура PACE и подобных ему элементов в промоторных областях протеасомных генов и Rpn4
PACE – необходимый компонент системы координированной регуляции экспрессии протеасомных генов. Представляло интерес выяснить, существуют ли преимущественные позиции PACE в промоторных областях протеасомных генов дрожжей-сахаромицетов. На рис. 1 представлено распределение положения PACE относительно старт-кодона трансляции протеасомных генов. Показано, что PACE находится преимущественно между 100 и 120 п.н.
Рис. 1.
Распределение положения канонического PACE в промоторных областях протеасомных генов у Saccharomycetes.
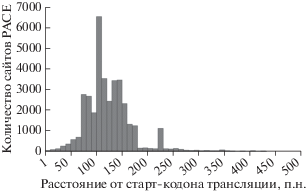
Установлено также, что 96.05% всех обнаруженных сайтов локализуются в участке от 50 до 250 н. влево от старт-кодона. Этот диапазон выбран для анализа положения PACE-подобных сайтов с одним и двумя отличиями от канонического PACE. Согласно полученным результатам (рис. 2а), распределение вариантов PACE с одной заменой в целом повторяет распределение канонического PACE. Варианты сайта с двумя заменами, в любом положении (кроме 4-го и 8-го, см. Экспериментальную часть) и с заменами, которые не являются соседними, распределены более равномерно в пределах анализируемого диапазона, что указывает на присутствие среди таких сайтов большого количества случайных последовательностей. В дальнейшем мы использовали сайты, отличающиеся от PACE заменой только одного нуклеотида.
Рис. 2.
Положение и анализ PACE и подобных ему элементов в промоторных областях протеасомных генов и RPN4. а ‒ Положение PACE и подобных ему элементов в промоторных областях протеасомных генов. б ‒ Положение PACE и подобных ему элементов в промоторных областях генов RPN4. в ‒ Логотипы консервативности нуклеотидов PACE-подобных элементов. г ‒ Тепловые карты позиционных матриц однонуклеотидных замен в PACE-подобных элементах. Значения в ячейках совпадающих нуклеотидов обнулены. Присутствие гуанина в положении 4 и аденина в 8 принято характеристистическим для PACE и ему подобных элементов (см. “Экспериментальную часть”), поэтому замены в этих положениях запрещены. Левая карта соответствует логотипу PACE-подобных элементов S. cerevisiae, центральная – другим видам дрожжей и правая – логотипу элементов в промоторах RPN4-генов.
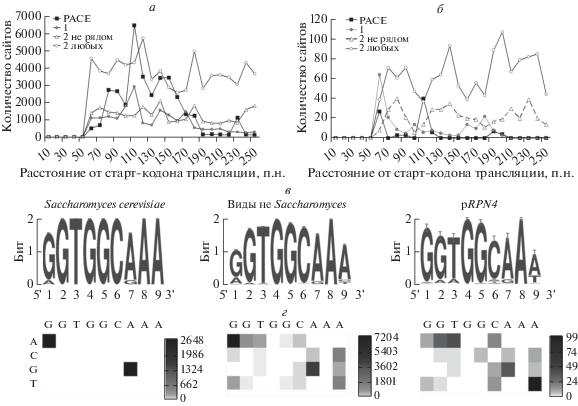
Отметим, что у многих видов дрожжей в промоторных областях протеасомных генов встречается не один, а два и даже три PACE и подобных ему элемента в пределах от 50 до 250 п.н. от старт-кодона трансляции.
Впервые обнаружено, что у 373 штаммов, принадлежащих 61 виду дрожжей, PACE и подобные ему элементы находятся в промоторных областях генов, кодирующих Rpn4-подобные белки (рис. 2б). Можно отметить бимодальное распределение канонического PACE и его вариантов с одной заменой. Основной пик приходится на участок 50‒70 п.н., а второй, как и у промоторов протеасомных генов – на участок 100‒120 п.н. На рис. 2в, г представлены результаты более детального анализа положения и вида однонуклеотидных замен в PACE. У штаммов S. cerevisiae встречаются практически только две замены G → A (G1A) в первом положении и А → G (A7G) в седьмом. Следовательно, консенсусная последовательность PACE-подобных элементов у S. cerevisiae ‒ 5'-RGTGGCRAA-3'. У других видов дрожжей ожидаемо наблюдается большее разнообразие замен. После замен G1A и A7G наиболее часто встречаются замены G2A, T3A, а также A9 на любую другую букву. Согласно экспериментальным данным, замена GC во втором тринуклеотиде GGC резко снижает сродство к нему Rpn4 S. cerevisiae [4]. У других видов дрожжей замены C и особенно G встречаются очень редко. Возможно, сайты с заменами в этих положениях нефункциональны, что требует экспериментальной проверки. Таким образом, консенсусную последовательность PACE-подобных элементов у других видов дрожжей (не S. cerevisiae) можно представить как 5'-RRWGGСRAN-3'. В сайтах в промоторах генов RPN4 замена G1A встречается редко, несколько чаще A7G и наиболее часто ‒ A9T. Следует отметить повышенное количество замен С в тринуклеотиде GGC, что может указывать на более низкое сродство этих сайтов к Rpn4, чем у сайтов в промоторах протеасомных генов. Таким образом, консенсусную последовательность PACE-подобных элементов в промоторах RPN4-генов можно определить как 5'-GRWGGCRAW-3'.
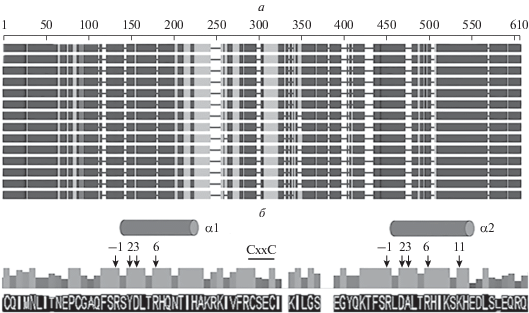
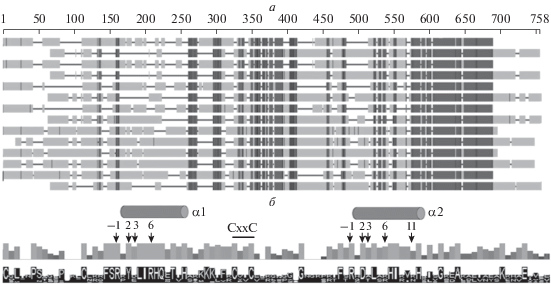
Второй важный компонент системы координированной регуляции протеасомных генов ‒ Rpn4. Ожидаемо, что у видов рода Saccharomyces эти белки в целом похожи (рис. 3а), очень высоким сходством обладает ДНК-связывающий домен, включая распознающие аминокислотные остатки в α-спиралях доменов цинковых пальцев.
Рис. 3.
Множественное выравнивание последовательностей Rpn4-подобных белков у дрожжей Saccharomyces. а ‒ Фрагмент схемы множественного выравнивания. Вверху указана шкала выравнивания (в аминокислотных остатках). Выравнивание выполнено с помощью программы COBALT [10] с параметрами по умолчанию. Более темным цветом указаны наиболее сходные участки. б ‒ Профиль консервативности ДНК-связывающего домена Rpn4-подобных белков, полученный с помощью программы Jalview2 [11]. Показано положение распознающих α-спиралей (α1 и α2) и аминокислотных остатков в этих спиралях, которые, как предполагают, контактируют с ДНК, в доменах N- и С-концевого цинковых пальцев соответственно.
Ожидаемо, что гораздо большее разнообразие последовательностей Rpn4-подобных белков и структуры их ДНК-связывающих доменов наблюдается у других видов Saccharomycetes (рис. 4 и 5). Всего выявлено 120 Rpn4-подобных белков, принадлежащих 80 видам дрожжей, не относящихся к роду Saccharomyces. Следует отметить в целом небольшое сходство Rpn4-подобных белков. Ранее мы показали, что общее сходство между CgRpn4 C. glabrata, DhRpn4 D. hansenii, KpRpn4 K. phaffii и YlRpn4 Y. lipolytica и ScRpn4 S. cerevisiae составляет 48.1 [9], 37.2 [8], 36.8 и 36.3% [7] соответственно. Представленные схемы выравниваний (рис. 4а, б) дают наглядное представление о небольшом сходстве анализируемых последовательностей. Наиболее консервативна лишь область ДНК-связывающего домена, что согласуется с высокой консервативностью PACE-элементов (рис. 2в).
Рис. 4.
Схемы множественных выравниваний последовательностей Rpn4-подобных белков. Сверху указана длина выравнивания (в аминокислотных остатках). Выравнивания выполнены с помощью программы COBALT [10] с параметрами по умолчанию. а ‒ Фрагмент схемы множественного выравнивания белков первой группы. б ‒ Фрагмент схемы выравнивания второй группы. в ‒ Схема множественного выравнивания белков третьей группы. г ‒ Схема множественного выравнивания белков четвертой группы.
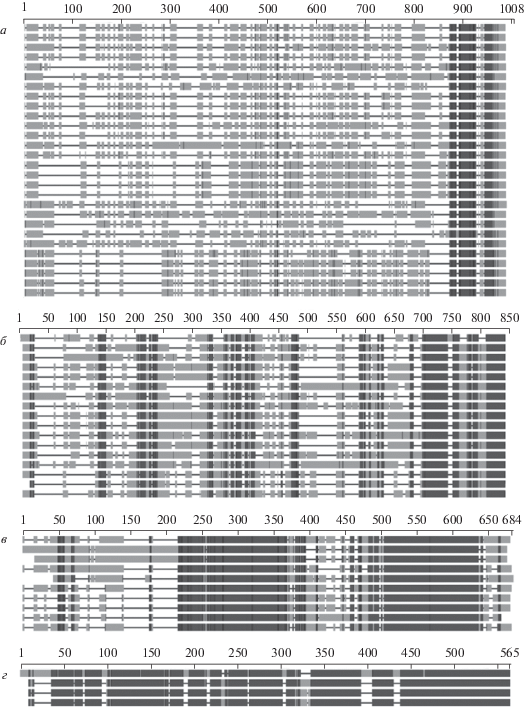
Рис. 5.
Профили выравнивания ДНК-связывающих доменов Rpn4-подобных белков. Выравнивания, выполненные с помощью программы COBALT [10], визуализированы с помощью программы Jalview2 [11]. Показано положение распознающих α1- и α2-спиралей N- и С-концевых доменов цинковых пальцев и аминокислотных остатков в этих спиралях, которые, как предполагается, контактируют с ДНК. 1, 2, 3 и 4 – обозначение групп Rpn4-подобных белков.
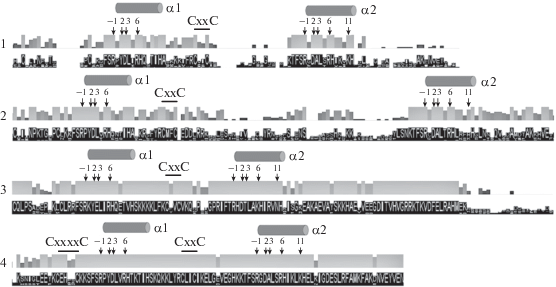
На основании особенностей структуры ДНК-связывающего домена выделены четыре группы Rpn4-подобных белков (рис. 5). ДНК-связывающий домен белков первой группы, состоящей из 89 членов, в основных чертах такой же, как у детально описанного Rpn4 S. cerevisiae [3, 4, 6]. В ДНК-связывающем домене белков второй группы (17 белков, обнаруженных у представителей Brettanomyces, Candida, Cyberlindnera, Ogataea и Pichia) расстояние между остатками цистеина и гистидина, связывающими ион цинка в C-концевом цинковом пальце, сильно увеличено за счет вставки из 31‒50 аминокислотных остатков. Эта вставка не содержит мотивов цинковых пальцев. Кроме того, в положении 11 распознающей спирали α2 помимо остатка лизина встречается также остаток аргинина. В ДНК-связывающем домене третьей группы (10 белков) выявлено удлинение C-концевой части домена. В дополнительном участке заметную долю составляют отрицательно заряженные остатки аминокислот. Кроме того, в третьей позиции распознающей спирали α1 вместо остатка аспарагиновой кислоты находится остаток глутаминовой кислоты, а в положении 11 распознающей спирали α2 вместо остатка лизина находится аспарагин. Наконец, в ДНК-связывающем домене четырех белков четвертой группы, выявленных у видов рода Hanseniaspora, домен N-концевого цинкового пальца содержит классический мотив С(Х)2–4С вместо уникального для Rpn4 S. cerevisiae мотива с сильно разделенными остатками цистеина.
Довольно высокое общее сходство белков внутри третьей и четвертой групп (рис. 4в, г), по-видимому, связано, с их небольшим числом. Можно ожидать, что по мере накопления секвенированных геномов других штаммов дрожжей и, соответственно, увеличения числа членов каждой группы Rpn4-подобных белков, общее сходство последовательностей будет падать при сохранении консервативности ДНК-связывающего домена.
В ходе поиска Rpn4-подобных белков обнаружены семь видов, в геноме которых одновременно присутствуют гены двух Rpn4-подобных белков: Clavispora lusitaniae, виды рода Candida (С. viswanathii, С. parapsilosis, C. orthopsilosis и С. haemulonis) и виды Debaryomyces (D. hansenii и D. fabryi) (рис. 6). Один из этих Rpn4 принадлежит к первой группе, а второй – к третьей. Результаты множественного выравнивания выявили близкое сходство как полных последовательностей этих белков, так и их ДНК-связывающих доменов. Некоторые отличия наблюдаются в распознающих аминокислотных остатках, что соответствует различиям между первой и третьей группами Rpn4-подобных белков.
Рис. 6.
Множественное выравнивание последовательностей паралогичных Rpn4-подобных белков. а ‒ Схема множественного выравнивания. Вверху указана шкала выравнивания в аминокислотных остатках. Выравнивание выполнено с помощью программы COBALT [10] с параметрами по умолчанию. Темным цветом выделены наиболее сходные участки. б ‒ Профиль консервативности ДНК-связывающего домена Rpn4-подобных белков, полученный с помощью программы Jalview2 [11]. Показано положение распознающих α1- и α2-спиралей в N- и С-концевом доменах цинковых пальцев и ДНК-распознающих аминокислотных остатков в этих спиралях.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В настоящей работе показано, что высокая консервативность последовательностей PACE соответствует большому сходству структуры ДНК-связывающих доменов и идентичности большинства ДНК-распознающих аминокислотных остатков у Rpn4-подобных белков, при том что остальная часть этих белков не обнаруживает существенного сходства. Найдены протеасомные гены с двумя и тремя PACE-подобными элементами. Впервые PACE выявлены в промоторах некоторых генов Rpn4-подобных белков, найдены также семь видов, геном которых кодирует два Rpn4-подобных белка, принадлежащих первой и третьей группам, согласно нашей классификации.
Одна из парадигм молекулярной эволюции гласит, что изменения фенотипа и, возможно, образование новых видов обусловлены изменением регуляции, а не функций самих генов [17]. Изменения регуляции экспрессии генов могут быть достигнуты за счет изменения сайтов связывания факторов транскрипции и/или структуры самих факторов. Система регуляции протеасомных генов подтверждает эту парадигму: протеасомные гены очень консервативны, довольно консервативен и PACE, однако структура Rpn4-подобных белков очень разнообразна. N-концевые области Rpn4-подобных белков, где, скорее всего, можно обнаружить регуляторные домены [18], могут быть, судя по множественным выравниваниям (рис. 4а, б), уникальными у каждого вида. Это может указывать на различия в силе и количестве, а также в составе тех белков и белковых комплексов, участвующих в инициации транскрипции, с которыми взаимодействуют разные Rpn4-подобные белки у разных видов. Сложность регуляторной системы увеличивается с появлением нескольких сайтов связывания Rpn4. Ранее мы показали, что в двустороннем промоторе MAG1-DDI1 и в промоторе YAP1 находятся по два PACE-подобных элемента, но в регуляции экспрессии, по крайней мере в нормальных условиях, участвуют только проксимальные сайты [5]. Однако эти результаты не означают, что второй сайт не функционален. Перечисленные гены, включая протеасомные, участвуют в клеточном ответе на стресс (повреждение ДНК, окислительный стресс и т.д.). Нами показано, что индуцированная стрессом экспрессия RPN4, в промоторе которого находятся два сайта связывания факторов транскрипции Pdr1 и Pdr3 [19], дозозависимо увеличивается при действии стрессовых факторов [20]. Аналогично можно предположить, что в условиях сильного стресса Rpn4 может увеличить экспрессию гена-мишени путем использования второго сайта.
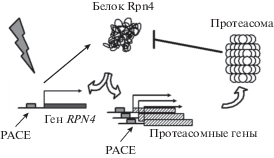
Присутствие PACE-подобных элементов в промоторах генов Rpn4-подобных белков может означать существование петли авторегуляции, которая, взаимодействуя с механизмом отрицательной обратной связи [21, 22], может менять соотношение концентраций Rpn4 и протеасомы. На рис. 7 представлена схема усложненной системы регуляции экспрессии протеасомных генов с участием петли положительной авторегуляции Rpn4. Механизм отрицательной обратной связи реализуется следующим образом: Rpn4 активирует экспрессию протеасомных генов, взаимодействуя с PACE в промоторах, что ведет к синтезу субъединиц и сборке активных протеасом, которые, в свою очередь, распознают Rpn4 и деградируют его. Это снижает количество фактора, а значит, экспрессию протеасомных генов и количество самих протеасом. Последовательности PACE, обнаруженные в промоторах RPN4-подобных генов, могут служить слабыми сайтами связывания, следовательно, они будут задействованы только при высоких концентрациях Rpn4. Тогда, при сильном стрессовом воздействии, из-за гиперактивации гена RPN4 сторонними факторами количество Rpn4 возрастает настолько, что он активирует не только транскрипцию протеасомных генов, но и экспрессию собственного гена, а значит, и синтез белка. Таким образом, включается петля положительной авторегуляции (рис. 7). Можно предположить, что с ее участием система переходит на новый уровень, обеспечивая повышенную продукцию протеасом и других Rpn4-зависимых мишеней в ответ на сильное стрессовое воздействие.
Рис. 7.
Схема регуляции экспрессии протеасомных генов с участием петли положительной авторегуляции Rpn4p.
Следующим уровнем усложнения системы регуляции экспрессии протеасомных генов может быть существование второго Rpn4-подобного белка. В проведенных ранее биоинформатических изысканиях [6] отмечено присутствие в геноме D. hansenii гена второго Rpn4-подобного белка, но высказывалось необоснованное предположение о его нефункциональности. В ходе наших поисков обнаружено семь подобных белков. В соответствующих генах не обнаружено каких-либо мутаций, нарушающих рамку считывания. Кроме того, нами амплифицирован ген второго Rpn4-белка из D. hansenii, т.е. его последовательность не является ошибкой сборки последовательностей геномов. Однако попытки клонировать этот ген без нарушения рамки считывания пока не увенчались успехом, что свидетельствует о его высокой токсичности (а, значит, и о наличии активностей) для E. coli. Два Rpn4-подобных белка обнаружены в геномах родственных групп Clavispora, Candida и Debaryomyces, известных своей способностью выживать в экстремальных условиях. Представители Candida зачастую являются патогенами человека, они сверхустойчивы к окислительному стрессу [23‒25], а Debaryomyces [26] может расти в средах с очень высокой концентрацией солей. Учитывая роль Rpn4 в ответе клеток на стресс, можно предположить, что присутствие двух белков позволяет этим дрожжам эффективно справляться с действием экстремальных факторов окружающей среды.
Часть работы выполнена на оборудовании ЦКП “Геном” Института молекулярной биологии РАН (http://www.eimb.ru/rus/ckp/ccu_genome_c.php).
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (№ 18-04-07021, № 18-04-00692).
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Glickman M.H., Ciechanover A. (2002) The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiol. Rev. 82, 373–428.
Lander G.C., Estrin E., Matyskiela M.E., Bashore C., Nogales E., Martin A. (2012) Complete subunit architecture of the proteasome regulatory particle. Nature. 482, 186–193.
Mannhaupt G., Schnall R., Karpov V., Vetter I., Feldmann H. (1999) Rpn4p acts as a transcription factor by binding to PACE, a nonamer box found upstream of 26S proteasomal and other genes in yeast. FEBS Lett. 450, 27–34.
Капранов А.Б., Преображенская О.В., Тютяева В.В., Штука Р., Фельдман Х., Карпов В.Л. (2001) Выделение и идентификация PACE-связывающего белка Rpn4 – нового транскрипционного активатора, участвующего в регуляции 26S протеасомных и других генов. Молекуляр. биология. 35, 420–431.
Спасская Д.С., Карпов Д.С., Миронов А.С., Карпов В.Л. (2014) Транскрипционный фактор Rpn4 Saccharomyces cerevisiae обеспечивает комплексный антистрессовый ответ при действии метилметансульфоната. Молекуляр. биология. 48, 166–175.
Mannhaupt G., Feldmann H. (2007) Genomic evolution of the proteasome system among hemiascomycetous yeasts. J. Mol. Evol. 65, 529–540.
Гринева Е.Н., Лейнсоо А.Т., Спасская Д.С., Карпов Д.С., Карпов В.Л. (2014) Функциональный анализ Rpn4-подобных белков из Komagataella (Pichia) pastoris и Yarrowia lipolytica в гетерологичной системе Saccharomyces cerevisiae. Биотехнология. 6, 8–17.
Karpov D.S., Grineva E.N., Leinsoo A.T., Nadolinskaia N.I., Danilenko N.K., Tutyaeva V.V., Spasskaya D.S., Preobrazhenskaya O.V., Lysov Y.P., Karpov V.L. (2017) Functional analysis of Debaryomyces hansenii Rpn4 on a genetic background of Saccharomyces cerevisiae. FEMS Yeast Res. 17, fow098.
Карпов Д.С., Гринева Е.Н., Киселева С.В., Челарская Е.С., Спасская Д.С., Карпов В.Л. (2019) Rpn4-подобный белок Candida glabrata комплементирует делецию гена RPN4 у Saccharomyces cerevisiae. Молекуляр. биология. 53, 274–281.
Papadopoulos J.S., Agarwala R. (2007) COBALT: constraint-based alignment tool for multiple protein sequences. Bioinformatics. 23, 1073–1079.
Waterhouse A.M., Procter J.B., Martin D.M., Clamp M., Barton G.J. (2009) Jalview Version 2–a multiple sequence alignment editor and analysis workbench. Bioinformatics. 25, 1189–1191.
Gasch A.P., Moses A.M., Chiang D.Y., Fraser H.B., Berardini M., Eisen M.B. (2004) Conservation and evolution of cis-regulatory systems in ascomycete fungi. PLoS Biol. 2, e398.
Harbison C.T., Gordon D.B., Lee T.I., Rinaldi N.J. Macisaac K.D., Danford T.W., Hannett N.M., Tagne J.B., Reynolds D.B., Yoo J., Jennings E.G., Zeitlinger J., Pokholok D.K., Kellis M., Rolfe P.A., Takusagawa K.T., Lander E.S., Gifford D.K., Fraenkel E., Young R.A. (2004) Transcriptional regulatory code of a eukaryotic genome. Nature. 431, 99–104.
Bulyk M.L., Huang X., Choo Y., Church G.M. (2001) Exploring the DNA-binding specificities of zinc fingers with DNA microarrays. Proc. Natl. Acad. Sci. USA. 98, 7158–7163.
Crooks G.E., Hon G., Chandonia J.M., Brenner S.E. (2004) WebLogo: a sequence logo generator. Genome Res. 14, 1188–1190.
Babicki S., Arndt D., Marcu A., Liang Y., Grant J.R., Maciejewski A., Wishart D.S. (2016) Heatmapper: web-enabled heat mapping for all. Nucl. Acids Res. 44, W147–W153.
Hahn S., Young E.T. (2011) Transcriptional regulation in Saccharomyces cerevisiae: transcription factor regulation and function, mechanisms of initiation, and roles of activators and coactivators. Genetics. 189, 705–736.
Karpov D.S., Tutyaeva V.V., Karpov V.L. (2008) Mapping of yeast Rpn4p transactivation domains. FEBS Lett. 582, 3459–3464.
Owsianik G., Balzi l L., Ghislain M. (2002) Control of 26S proteasome expression by transcription factors regulating multidrug resistance in Saccharomyces cerevisiae. Mol. microbiol. 43, 1295–1308.
Карпов Д.С., Осипов С.А., Преображенская О.В., Карпов В.Л. (2008) Rpn4p – позитивный и негативный регулятор транскрипции убиквитин-протеасомной системы. Молекуляр. биология. 42, 518–525.
Ju D., Wang L., Mao X., Xie Y. (2004) Homeostatic regulation of the proteasome via an Rpn4-dependent feedback circuit. Biochem. Biophys. Res. Commun. 321, 51–57.
Xie Y., Varshavsky A. (2001) RPN4 is a ligand, substrate, and transcriptional regulator of the 26S proteasome: a negative feedback circuit. Proc. Natl. Acad. Sci. USA. 98, 3056–3061.
Dantas Ada S., Day A., Ikeh M., Kos I., Achan B., Quinn J. (2015) Oxidative stress responses in the human fungal pathogen, Candida albicans. Biomolecules. 5, 142–165.
Gabaldon T., Carrete L. (2016) The birth of a deadly yeast: tracing the evolutionary emergence of virulence traits in Candida glabrata. FEMS Yeast Res. 16, fov110.
Pathirana R.U., Friedman J., Norris H.L., Salvatori O., McCall A.D., Kay J., Edgerton M. (2018) fluconazole-resistant Candida auris is susceptible to salivary histatin 5 killing and to intrinsic host defenses. Antimicrobial. Agents Chemotherapy. 62, e01872–17.
Prista C., Michan C., Miranda I.M., Ramos J. (2016) The halotolerant Debaryomyces hansenii, the Cinderella of non-conventional yeasts. Yeast. 33, 523–533.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


