Молекулярная биология, 2019, T. 53, № 6, стр. 1038-1048
Противоположные эффекты гистона H1 и белка HMGN5 на дистанционные взаимодействия в хроматине
Е. В. Низовцева a, Ю. С. Поликанов b, c, О. И. Кулаева a, N. Clauvelin d, Y. V. Postnikov e, W. K. Olson d, В. М. Студитский a, f, *
a Cancer Epigenetics Program, Fox Chase Cancer Center
19422 Philadelphia, USA
b Department of Biological Sciences, University of Illinois at Chicago
IL 60607 Chicago, USA
c Department of Medicinal Chemistry and Pharmacognosy, University of Illinois at Chicago
IL 60607 Chicago, USA
d Department of Chemistry and Chemical Biology, BioMaPS Institute for Quantitative Biology, Rutgers,
the State University of New Jersey
NJ 08854 Piscataway, USA
e Protein Section, Laboratory of Metabolism, Center for Cancer Research, National Cancer Institute
MD 20892 Bethesda, USA
f Биологический факультет Московского государственного университета им. М.В. Ломоносова
119234 Москва, Россия
* E-mail: vasily.studitsky@fccc.edu
Поступила в редакцию 10.05.2019
После доработки 14.06.2019
Принята к публикации 14.06.2019
Аннотация
Транскрипционные энхансеры взаимодействуют с промоторами через протяженные участки хроматина, расположенные между ними. Механизм подобных взаимодействий мало изучен. Ранее мы разработали систему in vitro, позволяющую количественно оценить скорость энхансер-промоторных взаимодействий. С использованием этой системы показано, что фибриллы хроматина поддерживают эффективные взаимодействия между энхансером и промотором. В представленной работе изучена роль линкерного гистона H1 и структурного белка HMGN5 в энхансер-промоторных взаимодействиях. Показан значительный ингибирующий эффект Н1 на эффективность энхансер-промоторных взаимодействий в хроматине, зависящий от присутствия N- и С-концевых участков белка. Ассоциированный с активным хроматином белок HMGN5, меняющий связывание Н1 с нуклеосомой и компактизацию хроматина, ослабляет ингибирующий эффект Н1. Полученные данные свидетельствуют о том, что эффективность взаимодействия энхансера и промотора зависит от структуры и динамики фибриллы хроматина, расположенной между ними, и может регулироваться белками, связанными с хроматином.
ВВЕДЕНИЕ
Взаимодействия между регуляторными участками ДНК и их мишенями происходят в ходе регуляции различных процессов, таких как экспрессия генов, транслокация хромосом и сплайсинг [1, 2]. Показано, что, по крайней мере для регуляции транскрипции, необходим физический контакт между активирующими последовательностями ДНК (энхансерами) и их мишенями ‒ промоторами. Подобные контакты осуществляются через взаимодействия специфических ДНК-связывающих белков и сопровождаются образованием петель хроматина [3–5]. Энхансеры могут эффективно активировать транскрипцию in cis на различных расстояниях (от сотен до сотен тысяч пар нуклеотидов) [6]. Чтобы обеспечить формирование петель такого различного размера, структура хроматина должна быть гибкой и динамичной [7]. Для участия в таких регуляторных взаимодействиях хроматин должен быть активирован и декомпактизован до уровня хроматиновой фибриллы [8, 9].
Хроматин имеет довольно мобильную структуру [10, 11], но ее вклад в эффективность энхансер-промоторных взаимодействий (ЭПВ) недостаточно изучен. Последовательности ДНК, удаленные друг от друга на расстояние более 1 т.п.н., не могут эффективно взаимодействовать in vitro, если между ними располагается участок линейной ДНК, не организованный в хроматин [12, 13]. Упаковка ДНК в хроматин может улучшать взаимодействия участков ДНК как на коротких расстояниях (200 п.н.) [14, 15], так и на протяженных (до 4 т.п.н.), что показано в экспериментах по внутримолекулярному лигированию удаленных последовательностей ДНК [(16] и измерению ЭПВ in vitro [17, 18]. Линкерный гистон Н1 и белок HMGN5 существенно влияют на компактизацию хроматина in vitro [19], и их присутствие коррелирует с активным и неактивным состояниями хроматина in vivo соответственно [20–23].
Поскольку линкерный гистон Н1 и белок HMGN5 существенно меняют структуру и динамику хроматина, можно предположить, что эти белки также влияют на ЭПВ в хроматине. Ранее мы разработали экспериментальную систему, позволяющую количественно оценить эффективность ЭПВ в хроматине in vitro [24]. Показано, что фибрилла хроматина обеспечивает эффективные ЭПВ, и важную роль в этом процессе играют N-концевые участки коровых гистонов [25]. В данной работе мы применили эту in vitro систему для изучения роли линкерного гистона Н1 и белка HMGN5 в ЭПВ в хроматине.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Белки. Все белки транскрипционного комплекса были очищены, как описано ранее [24]. Гистон Н1 выделен согласно [26] и очищен двухэтапной хроматографией с использованием ионообменной колонки Pharmacia Mono S и Phenomenex Jupiter C4. Использовали смесь всех вариантов Н1 тимуса теленка за исключением Н10. HMGN5 очищен, как описано ранее [19]. Рекомбинантный Н1 и его мутанты выделены согласно [27].
Сборка хроматиновых матриц. Донорный хроматин из эритроцитов курицы, не содержащий Н1/Н5, получен согласно [28]. Хроматин с удаленными концевыми доменами получен обработкой трипсином донорного хроматина из эритроцитов курицы [29].
Сборка хроматина на линеаризованной матричной ДНК проведена при различных соотношениях донорного хроматина и матрицы (0.75 : 1; 1 : 1; 1.25 : 1) с использованием градиентного диализа от 1 М до 10 мМ NaCl [28].
Анализ хроматиновых матриц в неденатурирующем геле. Тринуклеосомную матрицу ддя анализа связывания Н1 с хроматином собирали на 5'-радиоактивно меченном рестрикционном ApoI/EcoRI-фрагменте плазмиды pYP07, описанной ранее [25], и содержащей последовательности ДНК 601, позиционирующие нуклеосомы (НПП), на расстоянии 30 п.н. друг от друга. Матрицы инкубировали в транскрипционном буфере с Н1 либо с его мутантными формами в течение 30 мин при 37°С и молярном соотношении линкерного гистона к октамеру 1 : 1. Комплексы разделяли в 0.7%-ном агарозном геле в течение 4 ч при 4°С. Гель высушивали, экспонировали на радиочувствительный экран (“BioRad”, США) и анализировали с помощью фосфоимиджера (“BioRad”, США).
Анализ связывания Н1 с нуклеосомами с помощью нуклеазы микрококков. Связывание Н1 с собранным хроматином проверяли с помощью нуклеазы микрококков. Хроматиновые матрицы (150 нг) обрабатывали нуклеазой микрококков (“New England Biolabs”, CША) в концентрации 3.5 ед./мл в течение 1 мин при 37°С в транскрипционном буфере с добавлением 5 мМ CaCl2. Реакцию останавливали стоп-раствором (300 мМ NaAc (pН 5.3), 20 мМ EDTA pH 8.0). ДНК очищали и радиоактивно метили протеинкиназой Т4 (“New England Biolabs”), далее анализировали электрофорезом в 8%-ном полиакриламидном геле (ПААГ) (39 : 1). Гель высушивали, экспонировали на радиочувствительный экран, экран сканировали с помощью фосфоимиджера (“BioRad”).
Проверка качества сборки хроматиновых матриц. Ожидаемое позиционирование нуклеосом на используемых матрицах проверяли с использованием анализа чувствительности к рестрикционным эндонуклеазам с последующим анализом методом удлинения праймера. Хроматин собирали на линеаризованной плазмиде pYP05 и обрабатывали рестрикционными эндонуклеазами AluI и ScaI. ДНК очищали и использовали в качестве матрицы для ПЦР с одним радиоактивно меченным праймером (метод удлинения праймера) и Taq-ДНК-полимеразой (“New England Biolabs”). В данном эксперименте праймер отжигали непосредственно перед промотором [30].
Транскрипция. Условия для in vitro транскрипции были оптимизированы для максимального использования хроматиновой матрицы. Транскрипцию проводили, как описано ранее [24]. Во всех опытах использовали одинаковую концентрацию матрицы.
Вся матрица была линеаризована. На ней собирали закрытый инициаторный комплекс (РПз). Один раунд транскрипции проводили в реакционной смеси объемом 50 мкл в транскрипционном буфере, содержащем 50 мМ Трис-OAc (pH 8.0), 100 мМ KOAc, 8 мМ Mg(OAc)2, 27 мМ NH4OAc, 0.7% PEG-8000 и 0.2 мМ дитиотреитол (DTT) при концентрации ДНК или хроматина 1 нМ, а также 10 нМ РНК-полимеразы E. coli, 300 нМ σ54, 120 нМ NtrC и 400 нМ NtrB. Сначала все компоненты смешивали вместе в транскрипционном буфере (общий объем 40 мкл), инкубировали в течение 15 мин при 37°С для формирования закрытого комплекса. Далее 5 мкл 40 мМ АТР, разведенного в транскрипционном буфере, добавляли в реакцию до конечной концентрации 4 мМ. Реакцию проводили в течение 2 мин при 37°С для формирования открытого инициаторного комплекса (РПо). Затем смесь из четырех рибонуклеозидтрифосфатов (по 4 мМ каждого) в транскрипционном буфере с 2.5 мкКи [α-32P]-GTP (3000 Ки/мМ) и 2 мг/мл гепарина добавляли в реакцию для инициации элонгации и ограничения циклов транскрипции до одного раунда. Реакцию проводили при 37°С в течение 15 мин и останавливали смесью фенол : хлороформ в соотношении 1 : 1. Радиоактивно меченную РНК очищали и анализировали в денатурирующем ПААГ. Гель высушивали, экспонировали на радиочувствительный экран и сканировали, как описано выше. Данные анализировали с помощью программы OptiQuant.
Гистон Н1 (12 нМ) инкубировали с РПз при 37°С в течение 30 мин, после чего HMGN5 (24 нМ) добавляли к смеси и инкубировали на льду в течение 30 мин перед формированием РПо.
Компьютерное моделирование. Цифровое моделирование ЭПВ на больших расстояниях на полинуклеосомных матрицах (12 × 177 п.н.), содержащих 12 повторов НПП ДНК длиной 177 п.н. проведено по алгоритму, описанному ранее [25]. Вероятность взаимодействия оценивали из расчета расстояний (80‒125 нм) между центрами РНК-полимеразы и белка NtrC, которые связаны со специфическими последовательностями ДНК-матрицы. Предсказанное увеличение взаимодействий определяли как отношение вероятностей контактов полинуклеосомной матрицы и линейной ДНК той же длины. Новое уточнение в методологии заключается в том, что учитывали форму нуклеосомы, форму и электростатический потенциал регуляторного белка NtrC и РНК-полимеразы, а также структуру ДНК в этих комплексах [5]. Подвижность линкерной ДНК моделировали с участием движений, затрагивающих отдельные сегменты ДНК, а исключенный объем детектировали с использованием симулятора твердого тела (OpenDE).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Экспериментальная система для ЭПВ на нуклеосомных матрицах
Для получения хроматиновых матриц с равноудаленными на заданное расстояние друг от друга нуклеосомами, способными спонтанно формировать хроматиновую фибриллу, использовали последовательности ДНК 601. Эти последовательности обладают высокой аффинностью к гистонам и позволяют точно позиционировать нуклеосомы in vitro [31] (рис. 1а). Как показано ранее, сверхспирализация ДНК значительно усиливает ЭПВ [13]. Чтобы исключить эффект сверхспирализации ДНК на ЭПВ в хроматине, использовали только линеаризованные матрицы, которые позволили количественно оценить эффективность ЭПВ по описанной ранее схеме [25, 30].
Рис. 1.
Анализ хроматиновых матриц и связывания с ними гистона Н1. а – Хроматиновые матрицы, полученные с использованием разных соотношений донорного хроматина и ДНК-матрицы, обработаны эндонуклеазами AluI и ScaI, сайты которых расположены внутри позиционирующих нуклеосомы последовательностей и в линкерной ДНК соответственно. б – ДНК после обработки эндонуклеазами была очищена, амплифицирована с использованием одного радиоактивно меченного праймера и проанализирована в денатурирующем ПААГ. в – Связывание гистона Н1 с линеаризованной 13-нуклеосомной матрицей проверено с помощью обработки эндонуклеазой микрококков. Как и ожидалось, при связывании Н1 с хроматином появляется дополнительная защита примерно 20 п.н. ДНК.
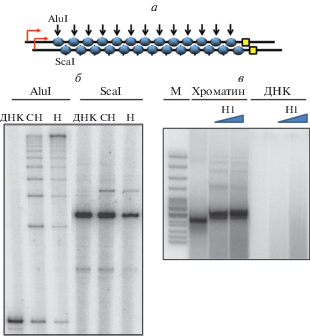
Плазмида pYP05 содержит 13 НПП длиной 147 п.н., расположенных между энхансером и промотором (рис. 1а). Каждая НПП лежит внутри повторяющегося элемента 177 п.н. длиной (601177 × 13). Таким образом, расстояние между соседними НПП составляет 30 п.н., полинуклеосомная матрица 601177 × 13 может спонтанно формировать фибриллу хроматина без линкерных гистонов [32, 33]. Сборка хроматина проведена на линеаризованной плазмиде pYP05 при различном соотношении донорного хроматина и ДНК-матрицы (1 : 1, 1 : 0.75 и 1 : 0.5) путем переноса октамеров гистонов при градиентном диализе, начиная с 1 М NaCl [30].
Полноту сборки хроматиновых матриц и качество позиционирования нуклеосом оценивали с помощью рестрикционного анализа. Показано, что увеличение отношения донорный хроматин : ДНК ведет к улучшению защиты ДНК НПП от разрезания ферментами [25, 30] (рис. 1б). Даже при самом высоком соотношении донорного хроматина и ДНК большинство нуклеосом формируется преимущественно на высокоаффинных НПП, а не на последовательностях энхансера и промотора [30]. Менее 20% не-НПП-ДНК (включая линкерную ДНК, а также энхансер и промотор) покрыто нуклеосомами (рис. 1б). Таким образом, практически все НПП покрыты нуклеосомами, в то время как остальная ДНК плазмиды в основном свободна от нуклеосом.
Линкерный гистон Н1 ингибирует ЭПВ в хроматине
Полинуклеосомные матрицы с длиной нуклеосомного повтора 177 п.н. спонтанно формируют хроматиновые фибриллы даже в отсутствие линкерных гистонов [32, 34]. При связывании линкерных гистонов структура таких матриц не претерпевает сильных изменений [32, 33]. Линкерные гистоны связываются с ДНК на входе и выходе ее из нуклеосомы [27, 35] и уменьшают скорость спонтанного отворачивания нуклеосомной ДНК от октамера гистонов [36, 37]. Предположили, что обратимое отворачивание концов нуклеосомной ДНК от октамера способно увеличивать гибкость фибриллы хроматина и повышать таким образом скорость ЭПВ [18]. Если это предположение верно, то Н1 будет уменьшать скорость ЭПВ в хроматине. Ранее показали, что Н1 ингибирует транскрипцию in vitro [38, 39]. Однако предыдущие исследования были направлены на влияние Н1 на ДНК-белковые взаимодействия и структуру хроматина, а роль Н1 в удаленных взаимодействиях в хроматине не изучали.
Для оценки влияния линкерного гистона Н1 на скорость ЭПВ были сформированы транскрипционные комплексы на 601177 × 13 полинуклеосомных матрицах. Затем эти комплексы преинкубировали с гистоном Н1 (или его укороченными вариантами) и использовали в качестве матриц для транскрипции. Связывание полноразмерного Н1 и его мутантных форм без концевых доменов с нуклеосомами достигает насыщения при молярном соотношении гистона Н1 и нуклеосом 1 : 1 (рис. 1в), тем самым подтверждая, что одна молекула Н1 связывает одну нуклеосому.
Скорость ЭПВ измеряли с использованием плазмиды (pYP05), содержащей NtrC-зависимый энхансер, который активирует промотор glnAp2 [30], а также 13 нуклеосом, расположенных между ними (рис. 2а). Экспериментальный подход к измерению скорости ЭПВ в хроматине путем количественного анализа получаемых транскриптов приведен на рис. 2б. Энхансер на указанной матрице активировался белковым комплексом NtrC, который фосфорилировался киназой NtrB [40]. Фосфорилированный NtrC взаимодействует с холоферментом Eσ54 и стимулирует превращение неактивного закрытого комплекса в открытый, функционально активный инициаторный комплекс [41, 42]. В процессе ЭПВ ДНК образует петлю [43].
Рис. 2.
Эффект гистона Н1 на ЭПВ в хроматине. а – 13-нуклеосомная линейная хроматиновая матрица. Показаны положения РНК-полимеразы и энхансер-связывающего белкового комплекса (NtrC) на промоторе (П) и энхансере (Э) соответственно. б – Схема эксперимента по измерению скорости ЭПВ в хроматине. Формировался закрытый инициаторный комплекс РНК-полимеразы (РПз), переходящий в открытый комплекс (РПо) после добавления АТР. Далее добавляли NTP и гепарин, который позволяет ограничить транскрипцию одним раундом и снимает нуклеосомный барьер. в – Анализ меченых транскриптов в денатурирующем ПААГ после транскрипции при различных молярных соотношениях Н1 к нуклеосоме. г – Количественный анализ транскриптов, показанных на рис. 2в.
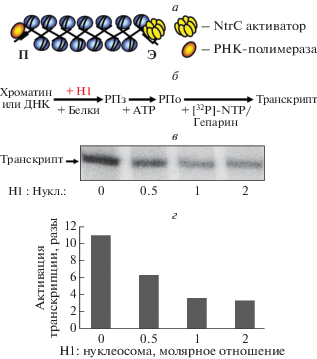
Транскрипционный анализ (рис. 2в) показывает, что связывание Н1 с интактными полинуклеосомными матрицами и такими же матрицами, но с гистонами без концевых доменов, в соотношении 1 : 1 к нуклеосомам приводит к сильному ингибированию ЭПВ (рис. 2в, г). При этом связывание Н1 с фибриллой хроматина не должно изменять ее компактизацию [33]. Поэтому негативный эффект Н1 на ЭПВ может быть объяснен следующим: а) уменьшением скорости отворачивания нуклеосомной ДНК от октамера гистонов [18], так как Н1 связывается и “запирает” ДНК, входящую и выходящую из нуклеосомы (рис. 3а, б), увеличением жесткости фибриллы. Гибкость фибриллы хроматина определяется в основном гибкостью линкерной ДНК, которая, скорее всего, будет уменьшаться в результате связывания Н1 (рис. 3а).
Рис. 3.
Роль различных участков гистона Н1 в ЭПВ в хроматине. а – Схема связывания гистона Н1 с нуклеосомой. Н1 связывается с ДНК на входе и выходе из нуклеосомы, предположительно делая ее менее гибкой. б – Связывание интактного Н1 и его мутантных форм с хроматином. в – Гистон Н1 и его мутантные формы, не имеющие С-концевого участка, либо обоих концевых участков, добавлены к хроматиновой матрице после остальных белков, участвующих в транскрипции. Скорость ЭПВ измерена, как описано на рис. 2б. Анализ меченых транскриптов в денатурирующем ПААГ. г – Количественный анализ транскриптов, показанных на рис. 3в.
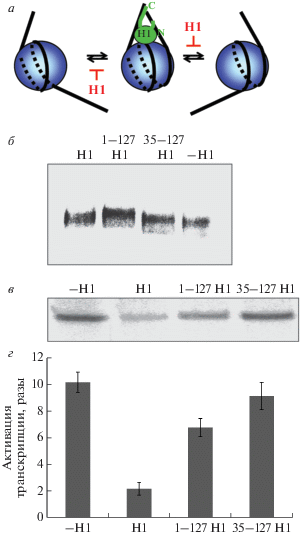
Чтобы исключить какие-либо из этих механизмов, а также определить функцию доменов Н1 в ЭПВ использовали мутантные варианты Н1 с удаленным положительно заряженным С-концевым доменом (1‒127 Н1) и глобулярный Н1 (35‒127 Н1). Обе укороченные формы Н1 связываются с фибриллой хроматина (рис. 3б). Более высокая мобильность Н1-содержащих матриц в геле связана, возможно, с большей жесткостью этих матриц [44].
При помощи транскрипционного анализа показано, что делеция С-концевого домена Н1 ведет к трехкратному уменьшению ингибирующего эффекта Н1 (рис. 3в, г). Таким образом, С-концевой домен Н1 играет ключевую роль в ингибировании ЭПВ гистоном Н1. Этот домен может уменьшать скорость ЭПВ с использованием обоих механизмов, описанных выше. В поддержку первого механизма (разворачивания) свидетельствует тот факт, что скорость ЭПВ на хроматиновых матрицах, лишенных N-концевых доменов коровых гистонов, также ингибируется Н1 (рис. 4). Поскольку хроматиновая фибрилла без концевых доменов гистонов характеризуется минимальными межнуклеосомными взаимодействиями [36, 45] (рис. 4а), наиболее вероятно, что Н1 ингибирует спонтанные разворачивания нуклеосомной ДНК от октамера или уменьшает гибкость линкерной ДНК. Делеция С-концевого домена Н1 приводит к уменьшению ингибирования Н1 на 30%, в то время как делеция обоих концевых доменов Н1 оказывает минимальное влияние на скорость ЭПВ (рис. 3в, г). Негативный эффект С-концевого домена Н1 на ЭПВ связан, возможно, с внутринуклеосомными взаимодействиями, так как длина этого домена слишком мала для взаимодействия с другими нуклеосомами. Хотя глобулярный Н1 взаимодействует с ДНК на входе и выходе ее из нуклеосомы [27], подобные взаимодействия недостаточны для ингибирования ЭПВ ни одним из описанных выше способов.
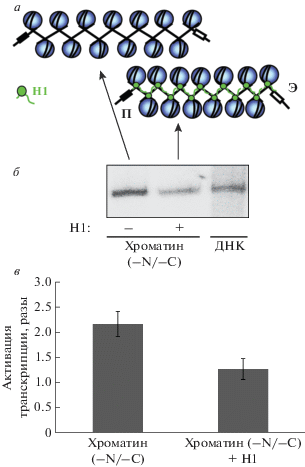
Рис. 4.
Гистон Н1 ингибирует ЭПВ в хроматине, в котором отсутствуют концевые домены коровых гистонов. а – Схема связывания Н1 с хроматином без концевых доменов коровых гистонов (Хроматин (–N/–C)). б – Анализ меченых транскриптов после транскрипции в присутствии Н1 и без него (как описано на рис. 2б) в денатурирующем ПААГ. в – Количественный анализ транскриптов, показанных на рис. 4б.
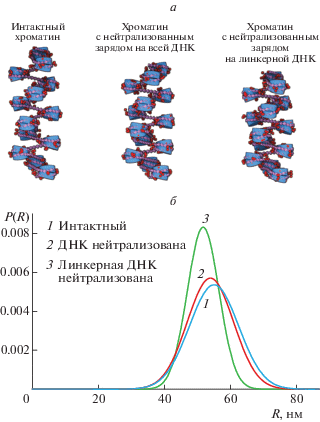
Хотя структуры комплексов Н1-нуклеосомы определены с низким разрешением [22, 46], в отсутствие детальной информации о структуре этого комплекса высокого разрешения весьма сложно понять, как Н1 влияет на ЭПВ. Точное положение глобулярного ДНК-связывающего домена Н1 до сих пор вызывает много вопросов [47]. Еще более непонятно, как изначально неупорядоченные концевые участки линкерного гистона взаимодействуют с ДНК [48]. Более того, расположение глобулярного домена Н1 может зависеть от варианта гистона [48] и от формирования “стебля” ДНК на выходе из нуклеосомного ядра, описаного для тринуклеосомных матриц [27, 49], и может не происходить на более длинных матрицах с варьирующими межнуклеосомными расстояниями. В качестве предварительной аппроксимации взаимодействий Н1–нуклеосома мы оценили как нейтрализация заряда ДНК меняет конфигурацию и деформируемость фибриллы с 13 равноудаленными интактными нуклеосомами (рис. 5а). Хотя полная нейтрализация ДНК оказывает минимальный эффект на моделируемые структуры, нейтрализация только линкерной ДНК заметно меняет компактизацию и пластичность моделируемой фибриллы (рис. 5а). Распределение расстояний между концевыми парами нуклеотидов сдвигается в сторону низких значений, а также сужается более существенно, когда нейтрализована только линкерная ДНК (рис. 5б), показывая тем самым, что связывание Н1 может придать жесткость фибрилле и немного изменить общую геометрию, сделав ее менее гибкой (рис. 5). Выяснение того, какие именно силы определяют снижение скорости ЭПВ в присутствии Н1, станет предметом дальнейших исследований.
Рис. 5.
Компьютерное моделирование фибриллы хроматина: дальние межнуклеосомные взаимодействия усиливаются после нейтрализации заряда ДНК. а – Модель фрагментов хроматина с 13 интактными нуклеосомами, расположенными на полностью заряженной ДНК, полностью нейтрализованной ДНК и ДНК с нейтрализованными линкерными участками, основанная на средней конфигурации центральных участков моделируемых структур. Предполагается, что центральные участки правильно отражают конформационные состояния, принимаемые всей фибриллой с равномерно расположенными нуклеосомами. б – Распределение расстояний между первой и последней парой нуклеотидов на моделируемом хроматине длиной 13 нуклеосом, расположенных на расстоянии 30 п.н (линкеры) друг от друга, на интактной ДНК, полностью нейтрализованной ДНК и ДНК.
Таким образом, наши результаты свидетельствуют о том, что Н1 блокирует ЭПВ в хроматине путем связывания с линкерной ДНК в основном через С-концевой домен с участием N-концевого домена. Связывание с линкерной ДНК также может ингибировать разворачивание ДНК от октамера гистонов и (или) уменьшать пластичность фибриллы хроматина. Удаление концевых доменов гистонов предотвращает формирование стабильной хроматиновой фибриллы и сильно уменьшает ингибирующий эффект Н1 на ЭПВ.
Белок HMGN5 нейтрализует негативное влияние гистона Н1 на ЭПВ
Для дальнейшей оценки роли гистона Н1 в ЭПВ в хроматине, а также возможных белков, которые могут положительно действовать на ЭПВ в Н1-содержащем хроматине, мы проанализировали влияние белка HMGN5, связывающего нуклеосомы, на ЭПВ. HMGN5 связывает Н1-содержащие нуклеосомные матрицы и инициирует частичное разворачивание 30 нм фибриллы с длиной межнуклеосомной ДНК 60 п.н. [19]. Отрицательно заряженный С-концевой домен HMGN5 взаимодействует с положительно заряженным С-концевым доменом Н1, участвующим во взаимодействии Н1–ДНК [19], нарушая тем самым эти взаимодействия. Таким образом, можно ожидать, что HMGN5 будет препятствовать ингибированию ЭПВ гистоном Н1. Когда матрицы содержат более короткую межнуклеосомную линкерную ДНК (30 п.н.), как в используемой здесь экспериментальной системе, связывание Н1 с полинуклеосомными матрицами незначительно меняет структуру фибриллы хроматина [32, 33]. Следовательно, ожидается, что HMGN5 затронет Н1-хроматиновые взаимодействия, но не общую структуру фибриллы.
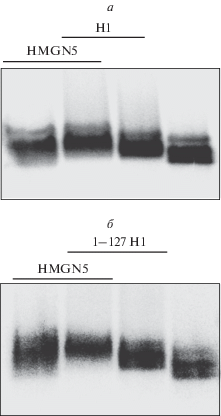
Для оценки влияния HMGN5 на скорость ЭПВ в Н1-содержащем хроматине, а также в хроматине, содержащем Н1 без С-концевого участка (1‒127 Н1), транскрипционные комплексы, сформированые на 601177 × 13 нуклеосомных матрицах, инкубировали с Н1 либо с 1‒127 Н1 и/или HMGN5 и анализировали в неденатурирующем геле (рис. 6). Подвижность Н1-содержащих нуклеосомных матриц в нативном геле изменялась в присутствии HMGN5 и отличалась от подвижности HMGN5-содержащих нуклеосомных матриц, показывая тем самым, что Н1 остается связанным с матрицами в присутствии HMGN5 (рис. 6). Таким образом, Н1 (и 1‒127 Н1) и HMGN5 связываются с хроматином одновременно.
Рис. 6.
Связывание белка HMGN5 с трехнуклеосомными матрицами, содержащими интактный гистон Н1 и его мутантный вариант 1‒127 Н1. Матрицы получены с использованием радиоактивно меченной ДНК и проанализированы в агарозном неденатурирующем геле после инкубации с Н1 (а), 1‒127 Н1 (б) и/или HMGN5.
Как ожидалось, HMGN5 положительно влияет на скорость ЭПВ в Н1-содержащих матрицах и не влияет на ЭПВ в матрицах, лишенных Н1 (рис. 7а–в). Максимальный эффект наблюдается при молярном соотношении HMGN5 : нуклеосома = 2 : 1 (рис. 7в). В присутствии HMGN5 скорость взаимодействий увеличивается примерно в 2 раза по сравнению с Н1-содержащим хроматином, показывая, что HMGN5 отменяет негативное действие Н1 и его мутантной формы с делетированным С-концевым доменом на ЭПВ. Более низкий положительный эффект (примерно 1.5-кратный) HMGN5 на ЭПВ в хроматине, содержащем 1‒127 Н1, можно объяснить взаимодействиями между HMGN5 и N-концевым доменом Н1, который из-за меньшего положительного заряда N-концевого домена имеет более слабые контакты с ДНК, чем С-концевой домен HMGN5. HMGN5 не влияет на ЭПВ на матрицах, содержащих глобулярный Н1 (35‒127), подтверждая тот факт, что основные взаимодействия Н1‒HMGN5 происходят с участием концевых доменов Н1.
Рис. 7.
Белок HMGN5 нейтрализует негативный эффект гистона Н1 на ЭПВ. а, б – Анализ меченых транскриптов в денатурирующем ПААГ после транскрипции нуклеосомных матриц в присутствии гистона Н1, 1‒127 Н1 и/или белка HMGN5. Скорость ЭПВ измеряли, как на рис. 2б. в – Количественный анализ транскриптов, показанных на рис. 7б. г – Схема взаимодействия белков Н1 и HMGN5 с хроматиновой матрицей.

Подводя итог, можно сказать, что белок HMGN5 частично отменяет отрицательное действие Н1 на ЭПВ в хроматине, по-видимому, нарушая контакты Н1‒ДНК, которые осуществляются через положительно заряженные концевые домены гистона Н1 (рис. 7г).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В данной работе мы использовали экспериментальную систему in vitro [30], позволяющую проводить количественный анализ ЭПВ в физиологических условиях на полинуклеосомных матрицах с точно позиционированными нуклеосомами, формирующими хроматиновые фибриллы (рис. 1). Используя эту систему, мы показали следующее. 1) Связывание линкерного гистона Н1 с полинуклеосомными матрицами ведет к сильному снижению скорости ЭПВ на интактном хроматине и хроматине без концевых доменов коровых гистонов (рис. 2‒4). 2) Ингибирование ЭПВ гистоном Н1 обусловлено преимущественно С‑концевым доменом, который, по-видимому, взаимодействует с линкерной ДНК и либо уменьшает гибкость фибриллы, либо ингибирует сворачивание/разворачивание нуклеосомной ДНК от октамера (рис. 3, 5). 3) Белок HMGN5 нейтрализует отрицательный эффект гистона Н1 на ЭПВ (рис. 7), с большой вероятностью нарушая контакты Н1‒ДНК через взаимодействие с концевыми доменами Н1.
Хроматин может влиять на скорость ЭПВ через компактизацию ДНК и/или через дополнительный динамический компонент, облегчающий взаимодействия. Все больше данных свидетельствует о том, что динамический компонент крайне важен для ЭПВ в хроматине [50]. Наши более ранние исследования показывают, что сильный эффект структуры хроматина на скорость ЭПВ невозможно объяснить просто компактизацией ДНК [18]. Идентифицировано несколько факторов, затрагивающих динамические свойства хроматина, независимо от их влияния на компактизацию. 1) Связывание гистона Н1 с интактным хроматином и хроматином с гистонами без концевых доменов существенно снижает скорость ЭПВ (рис. 1‒3). Поскольку Н1 минимально изменяет структуру хроматиновой фибриллы, сформированную на матрицах с повтором из 177 п.н. [32, 33, 36], он должен менять ее динамические свойства, необходимые для эффективной ЭПВ. 2) Белок HMGN5 также лишь минимально влияет на структуру фибриллы хроматина; однако он способен отменять негативный эффект Н1 на ЭПВ (рис. 7). Таким образом, наши результаты свидетельствуют о том, что динамика хроматина и конкурирующие взаимодействия между белками, связывающими нуклеосомы, играют решающую роль в определении скорости ЭПВ.
Связывание линкерного гистона Н1 сильно ингибирует ЭПВ в хроматине (рис. 2‒4). Это ингибирование, вероятно, происходит с участием взаимодействий Н1‒ДНК, так как разрушение этих взаимодействий, будь то удаление концевых доменов гистонов (рис. 4) или добавление HMGN5 (рис. 7), ведет по крайней мере к частичной отмене ингибирующего эффекта Н1. Поскольку связывание Н1 не меняет структуру хроматиновой фибриллы [32], наиболее значимым представляется его влияние на гибкость фибриллы и разворачивание–сворачивание нуклеосомной ДНК от октамера. Связанный с хроматином Н1 уменьшает гибкость фибриллы хроматина [44, 51]. Подобное снижение гибкости согласуется с наблюдаемым уменьшением эффективности ЭПВ в присутствии Н1.
На расстоянии более 1 т.п.н. на линейной или релаксированной ДНК, свободной от гистонов, ЭПВ происходит медленно и является скорость-лимитирующей стадией в процессе транскрипции in vitro [13]. ЭПВ на таком расстоянии в хроматине происходят гораздо быстрее, и могут регулироваться с помощью различных факторов, отраженных в нашей работе. Следовательно, ЭПВ в хроматине может быть новым скорость-лимитирующим этапом в регуляции экспрессии генов. В частности, возможно, что модификации гистонов, дестабилизирующие фибриллу хроматина (такие, как ацетилирование Н4К16) [52], могут снизить эффективность ингибирования гистоном Н1 [53] и увеличить эффективность ЭПВ. Другой возможный сценарий заключается в том, что модификации или замена линкерных гистонов могут быть необходимы для активации доменов хроматина. Так, фосфорилирование линкерных гистонов предотвращает ингибирование АТР-зависимого ремоделирования [54], а деубиквитинирование гистона Н2А усиливает вытеснение Н1 в процессе активации [55]. Белок HMGN5, связанный с активным хроматином [19], улучшает ЭПВ в хроматине in vitro (рис. 7) и может дестабилизировать также фибриллу хроматина in vivo.
Данная работа показывает, что хроматиновая фибрилла представляет собой динамичную структуру, поддерживающую эффективные взаимодействия и имеющую высокую степень компактизации в процессе ЭПВ. Нами идентифицированы факторы, которые сильно влияют на скорость ЭПВ, такие как Н1 и HMGN5. Наши данные свидетельствуют о том, что ЭПВ можно рассматривать как регулируемую стадию экспрессии генов эукариот.
Мы благодарим T. Richmond (ETH Zürich, Institute of Molecular Biology and Biophysics, Zürich, Швейцария) за плазмиду, содержащую последовательности позиционирования нуклеосомы 601177×12, и S. Dimitrova (Université Grenoble Alpes, CNRS UMR 5309, INSERM U1209, Institute for Advanced Biosciences (IAB), Site Santé– Allée des Alpes, Франция) за предоставление интактных и мутантных версий гистона H1.
Эта работа поддержана грантами NIH RO1GM119398 и R21CA220151 для В.М. Студитского, грантом RO1GM34809 для W.K. Olson и грантом Российского научного фонда (14-24-00031). Работа Ю.С. Поликанова спонсирована государственными стартап-фондами штата Иллинойс, США. Работа Ю.В. Постникова спонсирована National Institutes of Health, National Cancer Institute, США.
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
de Laat W., Duboule D. (2013) Topology of mammalian developmental enhancers and their regulatory landscapes. Nature. 502, 499‒506.
Gibcus J.H., Dekker J. (2013) The hierarchy of the 3D genome. Mol. Cell. 49, 773‒782.
Harmston N., Lenhard B. (2013) Chromatin and epigenetic features of long-range gene regulation. Nucl. Acids Res. 41, 7185‒7199.
Krivega I., Dean A. (2012) Enhancer and promoter interactions-long distance calls. Curr. Opin. Genet. Dev. 22, 79‒85.
Nizovtseva E.V., Todolli S., Olson W.K., Studitsky V.M. (2017) Towards quantitative analysis of gene regulation by enhancers. Epigenomics. 9, 1219‒1231.
Noordermeer D., Branco M. R., Splinter E., Klous P., van Ijcken W., Swagemakers S., Koutsourakis M., van der Spek P., Pombo A., de Laat W. (2008) Transcription and chromatin organization of a housekeeping gene cluster containing an integrated beta-globin locus control region. PLoS Genet. 4, e1000016.
Ringrose L., Chabanis S., Angrand P. O., Woodroofe C., Stewart A.F. (1999) Quantitative comparison of DNA looping in vitro and in vivo: chromatin increases effective DNA flexibility at short distances. EMBO J. 18, 6630‒6641.
Ghirlando R., Felsenfeld G. (2008) Hydrodynamic studies on defined heterochromatin fragments support a 30-nm fiber having six nucleosomes per turn. J. Mol. Biol. 376, 1417‒1425.
Gilbert N., Boyle S., Fiegler H., Woodfine K., Carter N.P., Bickmore W.A. (2004) Chromatin architecture of the human genome: gene-rich domains are enriched in open chromatin fibers. Cell. 118, 555‒566.
Platani M., Goldberg I., Lamond A.I., Swedlow J.R. (2002) Cajal body dynamics and association with chromatin are ATP-dependent. Nat. Cell. Biol. 4, 502‒508.
Gartenberg M.R., Neumann F.R., Laroche T., Blaszczyk M., Gasser S.M. (2004) Sir-mediated repression can occur independently of chromosomal and subnuclear contexts. Cell. 119, 955‒967.
Bellomy G.R., Record M.T., Jr. (1990) Stable DNA loops in vivo and in vitro: roles in gene regulation at a distance and in biophysical characterization of DNA. Prog. Nucl. Acid Res. Mol. Biol. 39, 81‒128.
Liu Y., Bondarenko V., Ninfa A., Studitsky V.M. (2001) DNA supercoiling allows enhancer action over a large distance. Proc. Natl. Acad. Sci. USA. 98, 14883‒14888.
Jackson J.R., Benyajati C. (1993) DNA-histone interactions are sufficient to position a single nucleosome juxtaposing Drosophila Adh adult enhancer and distal promoter. Nucl. Acids Res. 21, 957‒967.
Schild C., Claret F.X., Wahli W., Wolffe A.P. (1993) A nucleosome-dependent static loop potentiates estrogen-regulated transcription from the Xenopus vitellogenin B1 promoter in vitro. EMBO J. 12, 423‒433.
Stein A., Dalal Y., Fleury T.J. (2002) Circle ligation of in vitro assembled chromatin indicates a highly flexible structure. Nucl. Acids Res. 30, 5103‒5109.
Laybourn P.J., Kadonaga J.T. (1992) Threshold phenomena and long-distance activation of transcription by RNA polymerase II. Science. 257, 1682‒1685.
Rubtsov M.A., Polikanov Y.S., Bondarenko V.A., Wang Y.H., Studitsky V.M. (2006) Chromatin structure can strongly facilitate enhancer action over a distance. Proc. Natl. Acad. Sci. USA. 103, 17690‒17695.
Rochman M., Postnikov Y., Correll S., Malicet C., Wincovitch S., Karpova T.S., McNally J.G., Wu X., Bubunenko N.A., Grigoryev S., Bustin M. (2009) The interaction of NSBP1/HMGN5 with nucleosomes in euchromatin counteracts linker histone-mediated chromatin compaction and modulates transcription. Mol. Cell. 35, 642‒656.
Woodcock C.L., Skoultchi A.I., Fan Y. (2006) Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17‒25.
Shimada M., Chen W.Y., Nakadai T., Onikubo T., Guermah M., Rhodes D., Roeder R.G. (2019) Gene-specific H1 eviction through a transcriptional activator → p300 → NAP1 → H1 pathway. Mol. Cell. 74, 1‒16.
Garcia-Saez I., Menoni H., Boopathi R., Shukla M.S., Soueidan L., Noirclerc-Savoye M., Le Roy A., Skoufias D.A., Bednar J., Hamiche A., Angelov D., Petosa C., Dimitrov S. (2018) Structure of an H1-bound 6-nucleosome array reveals an untwisted two-start chromatin fiber conformation. Mol. Cell. 72, 902‒915. e7.
Azad G.K., Ito K., Sailaja B.S., Biran A., Nissim-Rafinia M., Yamada Y., Brown D.T., Takizawa T., Meshorer E. (2018) PARP1-dependent eviction of the linker histone H1 mediates immediate early gene expression during neuronal activation. J. Cell Biol. 217, 473‒481.
Polikanov Y.S., Rubtsov M.A., Studitsky V.M. (2007) Biochemical analysis of enhancer-promoter communication in chromatin. Methods. 41, 250‒258.
Kulaeva O.I., Zheng G., Polikanov Y.S., Colasanti A.V., Clauvelin N., Mukhopadhyay S., Sengupta A.M., Studitsky V.M., Olson W.K. (2012) Internucleosomal interactions mediated by histone tails allow distant communication in chromatin. J. Biol. Chem. 287, 20248‒20257.
Goodwin G.H., Johns E.W. (1973) Isolation and characterisation of two calf-thymus chromatin non-histone proteins with high contents of acidic and basic amino acids. Eur. J. Biochem. 40, 215‒219.
Syed S.H., Goutte-Gattat D., Becker N., Meyer S., Shukla M.S., Hayes J.J., Everaers R., Angelov D., Bednar J., Dimitrov S. (2010) Single-base resolution mapping of H1-nucleosome interactions and 3D organization of the nucleosome. Proc. Natl. Acad. Sci. USA. 107, 9620‒9625.
Walter W., Studitsky V.M. (2004) Construction, analysis, and transcription of model nucleosomal templates. Methods. 33, 18‒24.
Polach K.J., Lowary P.T., Widom J. (2000) Effects of core histone tail domains on the equilibrium constants for dynamic DNA site accessibility in nucleosomes. J. Mol. Biol. 298, 211‒223.
Polikanov Y.S., Studitsky V.M. (2009) Analysis of distant communication on defined chromatin templates in vitro. Methods Mol. Biol. 543, 563‒576.
Thastrom A., Lowary P.T., Widlund H.R., Cao H., Kubista M., Widom J. (1999) Sequence motifs and free energies of selected natural and non-natural nucleosome positioning DNA sequences. J. Mol. Biol. 288, 213‒229.
Dorigo B., Schalch T., Kulangara A., Duda S., Schroeder R.R., Richmond T.J. (2004) Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science. 306, 1571‒1573.
Routh A., Sandin S., Rhodes D. (2008) Nucleosome repeat length and linker histone stoichiometry determine chromatin fiber structure. Proc. Natl. Acad. Sci. USA. 105, 8872‒8877.
Dorigo B., Schalch T., Bystricky K., Richmond T.J. (2003) Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J. Mol. Biol. 327, 85‒96.
Caterino T.L., Fang H., Hayes J.J. (2011) Nucleosome linker DNA contacts and induces specific folding of the intrinsically disordered H1 carboxyl-terminal domain. Mol. Cell. Biol. 31, 2341‒2348.
Carruthers L.M., Hansen J.C. (2000) The core histone N termini function independently of linker histones during chromatin condensation. J. Biol. Chem. 275, 37285‒37290.
Brockers K., Schneider R. (2019) Histone H1, the forgotten histone. Epigenomics. 11, 363‒366.
Ding H.F., Bustin M., Hanse U. (1997) Alleviation of histone H1-mediated transcriptional repression and chromatin compaction by the acidic activation region in chromosomal protein HMG-14. Mol. Cell. Biol. 17, 5843‒5855.
O’Neil T.E., Meersseman G., Pennings S., Bradbury E.M. (1995) Deposition of histone H1 onto reconstituted nucleosome arrays inhibits both initiation and elongation of transcripts by T7 RNA polymerase. Nucl. Acids Res. 23, 1075‒1082.
Ninfa A.J., Magasanik B. (1986) Covalent modification of the glnG product, NRI, by the glnL product, NRII, regulates the transcription of the glnALG operon in Escherichia coli. Proc. Natl. Acad. Sci. USA. 83, 5909‒5913.
Buck M., Cannon W. (1992) Activator-independent formation of a closed complex between sigma 54- holoenzyme and nifH and nifU promoters of Klebsiella pneumoniae. Mol. Microbiol. 6, 1625‒1630.
Popham D.L., Szeto D., Keener J., Kustu S. (1989) Function of a bacterial activator protein that binds to transcriptional enhancers. Science. 243, 629‒635.
Su W., Porter S., Kustu S., Echols H. (1990) DNA-looping and enhancer activity: association between DNA-bound NtrC activator and RNA polymerase at the bacterial glnA promoter. Proc. Natl. Acad. Sci. USA. 87, 5504‒5508.
Langowski J., Heermann D.W. (2007) Computational modeling of the chromatin fiber. Semin. Cell. Dev. Biol. 18, 659‒667.
Gordon F., Luger K., Hansen J.C. (2005) The core histone N-terminal tail domains function independently and additively during salt-dependent oligomerization of nucleosomal arrays. J. Biol. Chem. 280, 33701‒33706.
Bednar J., Garcia-Saez I., Boopathi R., Cutter A.R., Papai G., Reymer A., Syed S.H., Lone I.N., Tonchev O., Crucifix C., Menoni H., Papin C., Skoufias D.A., Kurumizaka H., Lavery R., Hamiche A., Hayes J.J., Schultz P., Angelov D., Petosa C., Dimitrov S. (2017) Structure and dynamics of a 197 bp nucleosome in complex with linker histone H1. Mol. Cell. 66, 384‒397. e8.
Zhou B.R., Feng H., Kato H., Dai L., Yang Y., Zhou Y., Bai Y. (2013) Structural insights into the histone H1-nucleosome complex. Proc. Natl. Acad. Sci. USA. 110, 19390‒19395.
Fang H., Clark D.J., Hayes J.J. (2012) DNA and nucleosomes direct distinct folding of a linker histone H1 C‑terminal domain. Nucl. Acids Res. 40, 1475‒1484.
Meyer S., Becker N.B., Syed S.H., Goutte-Gattat D., Shukla M.S., Hayes J.J., Angelov D., Bednar J., Dimitrov S., Everaers R. (2011) From crystal and NMR structures, footprints and cryo-electron-micrographs to large and soft structures: nanoscale modeling of the nucleosomal stem. Nucl. Acids Res. 39, 9139‒9154.
Nizovtseva E.V., Clauvelin N., Todolli S., Polikanov Y.S., Kulaeva O.I., Wengrzynek S., Olson W.K., Studitsky V.M. (2017) Nucleosome-free DNA regions differentially affect distant communication in chromatin. Nucl. Acids Res. 6, 3059‒3067.
Grigoryev S.A., Arya G., Correll S., Woodcock C.L., Schlick T. (2009) Evidence for heteromorphic chromatin fibers from analysis of nucleosome interactions. Proc. Natl. Acad. Sci. USA. 106, 13317‒13322.
Shogren-Knaak M., Ishii H., Sun J.M., Pazin M.J., Davie J.R., Peterson C.L. (2006) Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311, 844‒847.
Robinson P.J., An W., Routh A., Martino F., Chapman L., Roeder R.G., Rhodes D. (2008) 30 nm chromatin fibre decompaction requires both H4-K16 acetylation and linker histone eviction. J. Mol. Biol. 381, 816‒825.
Horn P.J., Carruthers L.M., Logie C., Hill D.A., Solomon M.J., Wade P.A., Imbalzano A.N., Hansen J.C., Peterson C.L. (2002) Phosphorylation of linker histones regulates ATP-dependent chromatin remodeling enzymes. Nat. Struct. Biol. 9, 263‒267.
Zhu P., Zhou W., Wang J., Puc J., Ohgi K.A., Erdjument-Bromage H., Tempst, P., Glass C.K., Rosenfeld M.G. (2007) A histone H2A deubiquitinase complex coordinating histone acetylation and H1 dissociation in transcriptional regulation. Mol. Cell. 27, 609‒621.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


