Молекулярная биология, 2020, T. 54, № 1, стр. 164-176
Качественный протеомный профайлинг сукцинилирования лизина выявил его участие в первичном метаболизме в растениях чая (Camellia sinensis)
C. Qiu a, Y. Wang a, J. H. Sun a, W. J. Qian a, H. Xie a, Y. Q. Ding a, Z. T. Ding a, *
a Tea Research Institute, Qingdao Agricultural University, Chengyang District
266109 Qingdao, Shandong, China
* E-mail: dzttea@163.com
Поступила в редакцию 27.11.2018
После доработки 01.03.2019
Принята к публикации 07.03.2019
Аннотация
Посттрансляционная модификация остатков лизина сукцинатом влияет на структуру и функцию белков. Однако информация о сукцинилировании белков в растениях чая ограничена. Используя метод иммуноблотинга, мы обнаружили сукцинилирование белков чая. Проведен глобальный качественный анализ по идентификации сайтов сукцинилирования белков с использованием высокоточной нано-LC-МС/МС (жидкостной хроматографии и тандемной масс-спектрометрии) в сочетании с аффинной хроматографией. Идентифицировано 142 сайта сукцинилирования лизина в 86 белках чайных листьев. Выявленные сукцинилированные белки вовлечены в различные биологические процессы, причем значительная доля сайтов сукцинилирования обнаружена в ферментах, участвующих в первичном метаболизме, включая метаболизм глиоксилата и дикарбоксилата, цикл Кребса и метаболизм глицина, серина и треонина. Кроме того, 10 новых сайтов сукцинилирования обнаружены в гистонах. На основании полученных результатов можно предполагать, что в растениях чая сукцинилированные белки играют важную регуляторную роль в биологических процессах, особенно в первичном метаболизме. В представленной работе проведен не только всесторонний анализ сукцинилома лизинов в растениях чая, но и получена информация, важная для дальнейшего изучения функций сукцинилированного лизина.
ВВЕДЕНИЕ
Динамические и обратимые посттрансляционные модификации (PTM) играют важную роль в диверсификации функций белков и контроле многих биологических процессов [1‒3]. Недавно список из сотен различных PTM пополнился сукцинилированием лизина, обнаруженным у многих белков, включая гистоны. Сукцинилирование лизина вызывает значительные структурные изменения путем замены положительно заряженного остатка на отрицательно заряженный [3]. По сравнению с ацетилированием (42 Да) или метилированием (14 Да) сукцинилирование вводит большую функциональную группировку (100 Да), что способствует значительным изменениям в структуре и функции белка [4]. Впервые сукцинилированные белки были обнаружены у Escherichia coli [4], а впоследствии идентифицированы и в тканях животных [5]. В последнее время интенсивно исследуют сукцинилирование лизина в различных организмах, включая бактерии (Vibrio parahemolyticus и Mycobacterium tuberculosis), животных (Mus musculus и Homo sapiens) и растениях (томат, пшеница обыкновенная и Dendrobium officinale) [2, 6‒12]. На основании данных проведенных ранее исследований предполагают, что сукцинилирование лизина регулирует структуру и функции гистонов [3]. Более того, недавно показано, что сукцинилирование хроматина может быть механизмом, который модулирует транскрипцию всего генома и активность системы репарации ДНК [13].
Чай (Camellia sinensis (L.) O. Kuntze), вечнозеленое древесное растение, ‒ одна из самых популярных культур в мире. После расшифровки полной геномной последовательности чая возрастает важность исследований протеома и модификаций белков этого растения. Однако о модификациях, особенно сукцинилировании белков чая, известно мало. На сегодняшний день проведено только одно исследование по сукцинилированию лизина в чае Анцзы Байча (Anji Baicha), где показано, что различные сукцинилированные белки в основном представлены в процессах фотосинтеза, фиксации углерода, биосинтеза аминокислот и метаболизма порфиринов и хлорофилла [14]. С целью углубить понимание роли сукцинилирования лизина мы, используя иммуноблотинг, проанализировали белки растения Camellia sinensis (L.) O. Kuntze и показали, что сукцинилирование лизина происходит в основном в листьях этого растения.
В исследовании положено начало систематической идентификации сукцинилома в высокопродуктивном и высококачественном материале сорта QN3. Всего идентифицировано 142 сайта сукцинилирования на 86 белках, включая 10 сайтов сукцинилирования в гистонах чайных листьев. Проведен биоинформационный анализ всех идентифицированных лизинсукцинилированных белков, включающий обогащение по терминам онтологии генов (GO) и Киотской энциклопедии генов и геномов (KEGG), определение субклеточной локализации, идентификацию вероятных белок-белковых взаимодействий (PPI) и т.д. Наконец, обсуждается связь сукцинилирования лизина с метаболизмом глиоксилата и дикарбоксилата, циклом трикарбоновых кислот (ТСА) и метаболизмом глицина, серина и треонина. Таким образом, в исследовании представлена функциональная аннотация сукцинилирования лизина в растениях чая, что, безусловно, углубляет наши знания по PТМ белков.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Растения и условия их роста. В исследовании использованы однолетние саженцы чая сорта QN3. Саженцы выращивали в фитотроне при 12-часовом световом (25°C)/12-часовом темновом (20°C) фотопериоде с интенсивностью потока фотонов света 18 000 люкс/(м2 с2) и влажностью 75%. Растения орошали питательным раствором Plant-Prod 20-20-20 Fertilizer (“Lambrou Agro”, Кипр) с интервалом в 2 суток. Чайные листья собирали через один месяц и хранили при –80°C до экстракции белков. Проводили три биологических повтора.
Иммуноблотинг. Иммуноблотинг проводили после разделения белков лизата с помощью электрофореза в денатурирующем 12%-ном ПААГ. Белки переносили на нитроцеллюлозную мембрану (“Millipore”, США), которую инкубировали сначала в блокирующем буфере (0.05% Твин-20 и 5% обезжиренного сухого молока в PBS), а затем с кроличьими поликлональными антисукциниллизиновыми антителами (“PTM Biolabs”, Китай), разведенными в блокирующем буфере в соотношении 1 : 1000, в течение ночи при 4°C. Мембраны промывали, инкубировали с конъюгированными с пероксидазой хрена козьими антителами против антител кролика (“Sigma”, США), разведенными в соотношении 1 : 2000, снова промывали и инкубировали с хемилюминесцентным субстратом (“Sigma”) для последующей детекции сигнала.
Пробоподготовка. Образцы чайных листьев измельчали в жидком азоте и затем переносили в 5‑мл центрифужную пробирку. К порошку добавляли 4 объема буфера для лизиса, содержащего 8 М мочевину (“Sigma”), 1% Triton X-100, 10 мМ DTT (“Sigma”), 1% коктейля ингибиторов протеаз (“Calbiochem”, США), 3 мкМ трихостатин А (“Sigma”), 50 мМ ниацинамид (“Sigma”) и 2 мМ EDTA (“Sigma”), и обрабатывали ультразвуком (38 × 3 c с перерывами в 5 с) на льду с использованием ультразвукового дезинтегратора высокой интенсивности (“Scientz”, Китай). Осадок отделяли и удаляли центрифугированием при 20 000 × g при 4°C в течение 10 мин. Наконец, белок из полученного лизата осаждали предварительно охлажденной 20%-ной трихлоруксусной кислотой в течение 2 ч при –20°С и отделяли от супернатанта центрифугированием при 12 000 × g в течение 3 мин при 4°C. Осадок трижды промывали предварительно охлажденным ацетоном и повторно растворяли в 8 М мочевине. Для определения концентрации белка использовали набор BCA kit (“Beyotime”) в соответствии с инструкциями производителя.
Перед протеолитическим гидролизом раствор белка обрабатывали 5 мМ DTT в течение 30 мин при 56°С (для восстановления S-S-связей), а затем алкилировали с использованием 11 мМ йод-ацетамида (“Sigma”) в течение 15 мин при комнатной температуре. В полученный раствор добавляли 100 мМ NH4HCO3 до конечной концентрации мочевины менее 2 М. Наконец, добавляли трипсин (“Promega”), в массовом соотношении 1 : 50 к суммарному белку, и проводили первый гидролиз в течение ночи при 37°С. С целью добиться полного расщепления белка еще раз добавляли трипсин в массовом соотношении 1 : 100 к суммарному белку и проводили инкубацию в течение 4 ч.
Аффинное обогащение. Для обогащения смеси сукцинилированными по лизину пептидами триптические пептиды, растворенные в буфере NETN, содержащем 100 мМ NaCl, 1 мМ EDTA, 50 мМ Трис-HCl (pH 8.0) и 0.5% NP-40, инкубировали с предварительно отмытыми гранулами агарозы, на которых иммобилизованы антисукциниллизиновые антитела (Cat. No PTM-402, “PTM Biolabs”), при легком встряхивании в течение ночи при 4°C. Затем гранулы четырежды промывали буфером NETN и дважды деионизированной водой. Связавшиеся пептиды элюировали с гранул 0.1%-ной трифторуксусной кислотой. Фракции элюатов объединяли и высушивали в вакуумном испарителе. Для анализа методом жидкостной хроматографии и тандемной масс-спектрометрии (LC-MS/MS) полученные пептиды обессоливали с помощью колонок C18 ZipTips (“Millipore”) в соответствии с инструкциями производителя.
LC-MS/MS. Триптические пептиды растворяли в 0.1%-ной муравьиной кислоте (“Fluka”) (растворитель A) и загружали в аналитическую колонку с обращенной фазой (длина 15 см, диаметр 75 мкм) собственного производства. С помощью растворителя B (0.1%-ная муравьиная кислота в 98%-ном ацетонитриле) создавали градиент с увеличением содержания ацетонитрила: от 6 до 23% в течение 26 мин, от 23 до 35% в течение 8 мин, повышение до 80% в течение 3 мин и удерживание при 80% в последние 3 мин ‒ при постоянной скорости потока на всех этапах в 400 нл/мин в хроматографической системе EASY-nLC 1000 UPLC (“Thermo Fisher Scienti-fic”, США).
Пептиды подвергали воздействию наноэлектронного источника ионизирующего излучения с последующей тандемной масс-спектрометрией (MS/MS) на приборе Q ExactiveTM Plus (“Thermo Fisher Scientific”), подключенном в режиме онлайн к хроматографу UPLC. Подаваемое напряжение распыления электронов составляло 2.0 кВ. Диапазон сканирования m/z составлял от 350 до 1800 для полного сканирования, и неповрежденные пептиды детектировали в Orbitrap с разрешением 70 000. Пептиды выбирали для последующей МС/МС при значении нормализованной энергии столкновения равной 28, и фрагменты детектировали в Orbitrap с разрешением 17 500. Определяемую данными процедуру чередования между одним сканированием MS с последующим сканированием 20 MS/MS для 20 лучших предшественников проводили с динамическим исключением 15.0 с. Автоматическую регулировку усиления устанавливали на 5E4, пороговое значение сигнала ‒ на уровне 10 000 ионов/с, а максимальное время ввода пробы ‒ на 100 мс.
Поиск по белковой базе данных. Идентификацию белков и сайтов сукцинилирования выполняли с помощью поисковой системы Maxquant (v.1.5.2.8). Тандемные масс-спектры анализировали с помощью белковой базы данных, построенной по последовательности генома Camellia si-nensis (http://www.plantkingdomgdb.com/tea_tree/), объединенной с базой обратных белковых последовательностей. Трипсин/Р указывали в качестве фермента гидролиза и допускали максимум 4 пропущенных сайта расщепления. В первичном поиске погрешность изменения массы ионов-предшественников устанавливали на уровне 20 ppm и 5 ppm ‒ в основном поиске [15]. Разброс по массе фрагментных ионов указывали равным 0.02 Да. Карбамидометилирование цистеина указывали как постоянную модификацию, а модификацию сукцинилирования и окисление метионина ‒ как вариабельные модификации. Частоту ложных обнаружений ограничивали значением < 0.01, а минимальный счет для модифицированных пептидов устанавливали на уровне > 40.
Биоинформационный анализ. Модификации аминокислот проверяли по базе данных Unimod (http://www.unimod.org/). Элементы вторичной структуры белков предсказывали с помощью NetSurfP. Вероятность образования элемента вторичной структуры данного типа с модифицированными остатками лизина сравнивали с контрольной последовательностью без модификаций лизина для всех сукцинилированных белков, идентифицированных в этом исследовании, и p-value рассчитывали, как описано ранее. GO-аннотацию результатов протеомного анализа проводили с помощью базы данных UniProt-GOA (http:// www.ebi.ac.uk/GOA/) и классифицировали белки по трем категориям: биологический процесс, клеточный компонент и молекулярная функция. Базу данных KEGG использовали при аннотировании биохимических путей, в которых участвуют идентифицированные белки. Белки аннотировали также с помощью программы InterProScan (http://www.ebi.ac.uk/interpro/scan.html), сканирующей заданную последовательность на соответствие белковым структурам в базе данных InterPro. Коррекцию при проверке множественных гипотез проводили с использованием стандартных методов контроля уровня ложных обнаружений. Аннотации GO и биохимических путей с корректированным p-value < 0.05 считали значимыми. Анализ сети PPI для идентифицированных сукцинилированных белков проводили с использованием программного обеспечения Cytoscape, а входную сеть PPI получали из базы данных STRING версии 10.5. Алгоритм теоретической кластеризации графов, MCODE, использовали при анализе плотно связанных узлов сети PPI.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Детекция белков и сайтов сукцинилированного лизина
С целью охарактеризовать степень сукцинилирования лизина в белках растений чая мы провели анализ Вестерн-блот белковых лизатов листьев чая с антителами против сукциниллизина. Как и ожидалось, обнаружено множество белковых полос, что указывает на присутствие различных сукцинилированных белков (рис. 1а).
Рис. 1.
Контроль качества данных LC-MS/MS. а ‒ Анализ сукцинилирования белков методом вестерн-блот. Время экспозиции 3 с. На дорожки 1‒3 нанесены три биологических повтора. б ‒ Пептидная шкала для всех сукцинилированных пептидов построена как функция погрешностей массы откалиброванных пептидов, выраженных в частях на миллион (ppm). в ‒ Распределение длин идентифицированных пептидов. г ‒ Распределение сайтов сукцинилирования в белках.
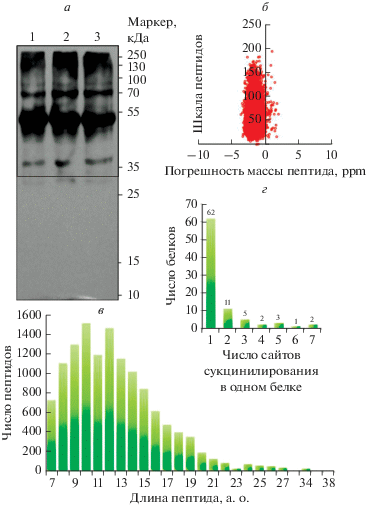
С помощью высокопроизводительного качественного протеомного анализа идентифицировано в общей сложности 142 сайта сукцинилирования лизина на 86 белках, что представляет собой существенный объем данных по сукцинилому лизина в растениях чая. Изменение массы составило 100.0186 кДа. Масс-спектрометрические данные доступны в базе данных Proteome Xchange (номер проекта PXD011717). Распределение погрешности массы было около нуля и большинство значений меньше 10 ppm (рис. 1б), что соответствует требованиям по точности массы в данных масс-спектрометрии. Кроме того, длины большинства пептидов варьировали от 8 до 20 аминокислотных остатков (рис. 1в), что соответствует типичной длине триптических пептидов. Среди идентифицированных белков около 72.1% (62) имели один сайт сукцинилирования, тогда как 27.9% (24) ‒ несколько сайтов (рис. 1г). Наибольшее число сайтов сукцинилирования обнаружено на двух белках: DLD и rbcL. Подробная информация по всем идентифицированным сукцинилированным белкам представлена в Приложении 1 (см. сайт http://www.molecbio.ru/ downloads/2020/1/supp_Qiu_rus.pdf).
Функциональная аннотация и субклеточная локализация сукцинилированных белков
GO-анализ служит основным инструментом биоинформатики, позволяющим проводить сравнение генов или белков между различными видами [16]. С целью выяснить функциональную роль сукцинилирования белков чая все идентифицированные белки проанализировали по трем категориям: биологический процесс, клеточный компонент и молекулярная функция (рис. 2, Приложение 2 , см. сайт http://www.molecbio.ru/ downloads/2020/1/supp_Qiu_rus.pdf). Большинство сукцинилированных белков оказалось вовлеченным в процессы метаболизма (34%) и единого организма (30%), в клеточные механизмы (25%) и локализацию (7%) (рис. 2а). Что касается клеточных компонентов, большое число сукцинилированных белков распределено в клетке (36%), макромолекулярном комплексе (24%) и органелле (21%) (рис. 2б). Что касается молекулярных функций, то около половины лизинсукцинилированных белков классифицировано как ассоциированные с каталитической активностью (45%) и менее 40% классифицированы как связывающие белки (39%) (рис. 2в). Это значит, что сукцинилированные белки в основном аннотированы в биологических процессах и молекулярных функциях.
Рис. 2.
Функциональная аннотация и клеточная локализация сукцинилированных белков. Классификация сукцинилированных белков по биологическим процессам (а), клеточным компонентам (б) и молекулярной функции (в). г ‒ Субклеточная локализация сукцинилированных по лизину белков чая. д ‒ Соотношение сукцинилированных белков в хлоропластах, цитоплазме и митохондриях Triticum asetivum, Dendrobium officinale, Solanum lycopersicum, Brachypodium distachyon и Taxus × media на основании данных, полученных в работах [10‒12, 17, 18]).
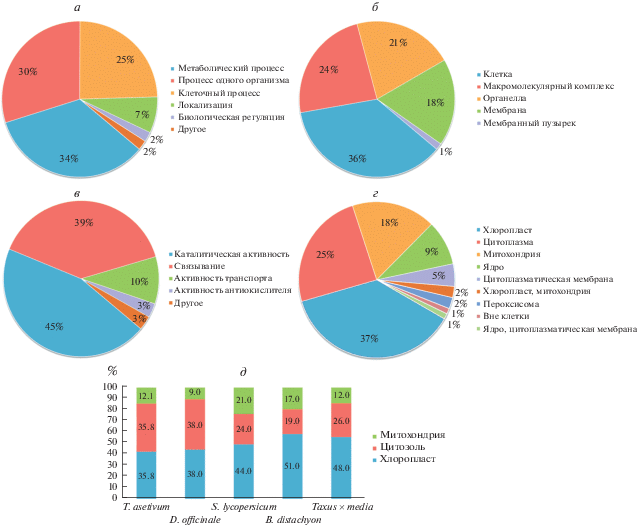
Предсказание субклеточной локализации белка служит важным этапом в предсказании функции белка и аннотации генома в биоинформационном анализе. С целью лучше понять функции сукцинилированных белков проведено исследованы их возможной субклеточной локализации. Как представлено на рис. 2г, 32 из идентифицированных сукцинилированных белков могут находиться в хлоропласте (37%), 21 в цитоплазме (25%) и 15 в митохондриях (18%). В соответствии с ранее полученными данными по Triticum asetivum, Dendrobium officinale, Solanum lycopersicum, Brachypodium distachyon и Taxus × media [10‒12, 17, 18], сукцинилированные белки встречаются в основном в хлоропласте, цитоплазме и митохондриях растений (рис. 2д). На основании этих результатов можно сделать вывод о том, что лизинсукцинилированные белки чая выполняют широкий спектр биологических функций.
Анализ обогащения GO терминами, состава белковых доменов и путей KEGG с участием сукцинилированных белков
С целью глубже понять биологические функции сукцинилированных белков мы провели анализ обогащения GO-терминами и состава белковых доменов. В категории клеточного компонента сукцинилированные белки в основном относятся к митохондриям (p-value = 8.98 × 10–7), протон-транспортному комплексу АТP-синтазы (p-value = 3.44 × 10–6) и внутриклеточной части (p-value = 4.15 × 10–6) (зеленая полоса на рис. 3а, Приложение 3 , см. сайт http://www.molecbio.ru/ downloads/2020/1/supp_Qiu_rus.pdf). В категории молекулярной функции многим модифицированным белкам приписано связывание кофакторов (p-value = 9.04 × 10–10), оксидоредуктазная активность (p-value = 1.06 × 10–5) и связывание коферментов (p-value = 1.54 × 10–5) (синяя полоса на рис. 3а, Приложение 3 ). В категории биологического процесса идентифицированные белки картированы в метаболическом процессе низкомолекулярных соединений (p-value = 5.59 × 10–19), метаболическом процессе в едином организме (p‑value = 2.25 × 10–12), метаболическом процессе карбоновых кислот (p-value = 4.3 × 10–11), в процессе метаболизма оксикислот (p-value = 6.42 × 10–11) и процессе метаболизма органических кислот (p‑value = 6.61 × 10–11) (красная полоса на рис. 3а, Приложение 3 ). Таким образом, сукцинилирование лизина может играть важную роль в метаболизме и регуляции растительных клеток. Для понимания регуляторной роли сукцинилирования необходимы специальные эксперименты. Между тем, в ходе анализа состава белковых доменов обнаружено значительное обогащение сукцинилированных белков доменами для присоединения биотина/липоильной группы, с единичным гибридным мотивом, с гистоновой укладкой и с гистонами H2A/H2B/H3 (рис. 3б, Приложение 3 ).
Рис. 3.
Классификация сукцинилированных по лизину белков по биологическим терминам. Обогащение сукцинилированных белков по GO-терминам (а), доменам (б) и KEGG (в). Размерность горизонтальной оси –lg (p-value по точному критерию Фишера).
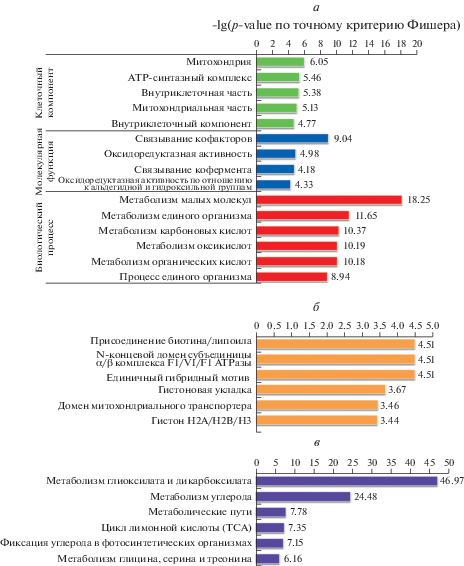
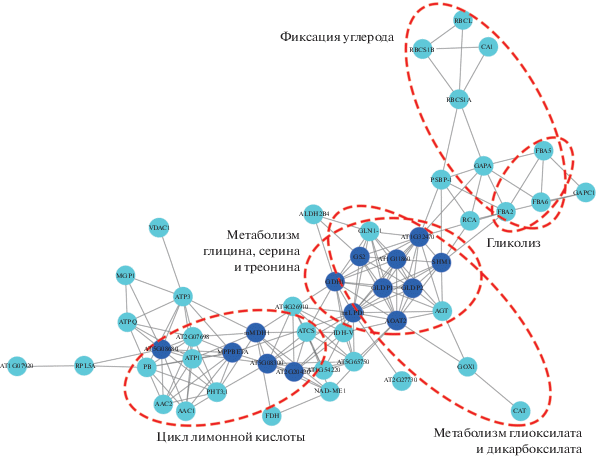
Чтобы получить более подробную информацию о метаболических путях, в которые вовлечены лизинсукцинилированные белки, мы провели анализ обогащения путей KEGG. Всего идентифицировано 16 значимо обогащенных путей KEGG, включая метаболизм глиоксилата и дикарбоксилата, метаболизм углерода, цикл ТСА, фиксацию углерода в фотосинтезирующих организмах и метаболизм глицина, серина и треонина (рис. 3в, Приложение 4 , см. сайт http:// www.molecbio.ru/downloads/2020/1/supp_Qiu_ rus.pdf). Среди них метаболизм глиоксилата и дикарбоксилата представлен наибольшим числом белков. К этому пути отнесены такие белки как HAO, katE, rbcL, glnA, GLDC, GGAT, glyA и AGXT (рис. 4a), в которых суммарно идентифицировано 20 сайтов сукцинилирования лизина (табл. 1). Как показано на рис. 4а, 7 сукцинилированных белков, включая gcvT, AGXT, glyA, GGAT, GLDC, DLD и gcvH, вовлечены в метаболизм глицина, серина и треонина. В этих белках обнаружено 19 сайтов сукцинилирования (табл. 1). Три репрезентативных LC-MS/MS-спектра сукцинилированных пептидов, относящихся к белкам glyA, AGXT и GGAT из метаболизма глиоксилата и дикарбоксилата и метаболизма глицина, серина и треонина, представлены в Приложении 5 и на рис. S1‒S3 (см. сайт http://www.molecbio.ru/ downloads/2020/1/supp_Qiu_rus.pdf).
Рис. 4.
Биохимические пути с участием сукцинилированных по лизину ферментов. a ‒ Метаболизм глиоксилата и дикарбоксилата, метаболизм глицина, серина и треонина. б ‒ Цикл TCA. Сукцинилированные белки отмечены синим цветом. Рибулозо-1,5-PP ‒ рибулозо-1,5-дифосфат, Lip-Pr ‒ липоилированный белок, dHLip-Pr ‒ дигидролипоилированный белок, S-AMdHLip-Pr ‒ S-аминометилдигидролипоилированный белок, THF ‒ тетрагидрофолат, katЕ ‒ изофермент каталазы, HAO ‒ пероксисомальная оксидаза (S)-2-гидроксикислот, rbcL ‒ большая субъединица рибулозобисфосфаткарбоксилазы, glnА ‒ глутаминсинтетаза, GLDC ‒ глициндегидрогеназа, GGAT ‒ глутамат‒глиоксилат-аминотрансфераза, glyA ‒ серин-гидроксиметилтрансфераза, AGXT ‒ серин-глиоксилат-аминотрансфераза, gcvT ‒ аминометилтрансфераза, DLD ‒ дигидролипоилдегидрогеназа, gcvH ‒ белок H системы расщепления глицина, LSC1 ‒ α-субъединица сукцинил-КоА-синтетазы, LSC2 ‒ β-субъединица сукцинил-КоА-синтетазы, OGDH ‒ компонент E1 2-оксоглутаратдегидрогеназы, DLST ‒ компонент Е2 2-оксоглутаратдегидрогеназы, CS ‒ цитратсинтаза, IDH3 ‒ дегидрогеназа (NAD+), ThPP ‒ тиаминдифосфат, dHLipAm-E ‒ дигидролипоамид Е, S-Su-dHLipAm-E ‒ S-сукцинил-дигидролипоамид Е.
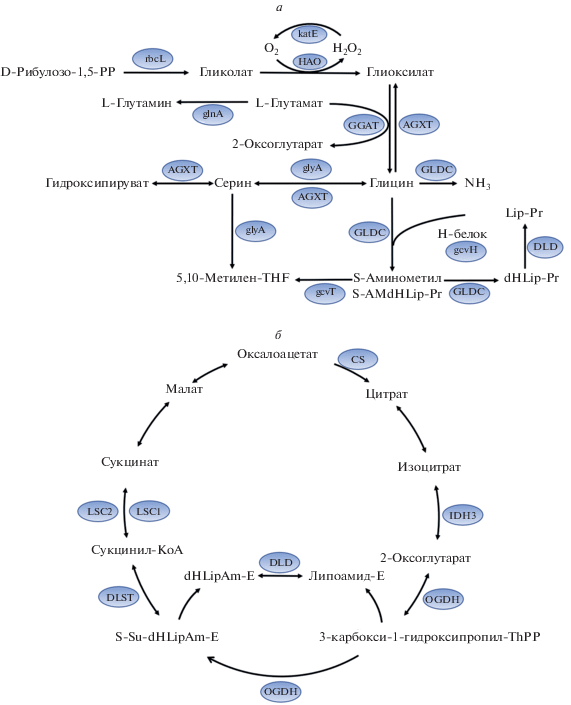
Таблица 1.
Позиции сукцинилированного лизина в белках
| ID белка | Описание | Позиция |
|---|---|---|
| CSA010160.1 | Дигидролипоилдегидрогеназа 1, митохондриальная [Ricinus communis] | K149, K334, K431, K154, K446, K411, K146 |
| CSA017504.1 | Цитратсинтаза, митохондриальная [Juglansregia] | K81, K165, K370, K372 |
| CSA032333.1 | 2-оксоглутаратдегидрогеназа, митохондриальная [Vitis vinifera] | K531, K600, K604 |
| CSA028548.1 | Субъединица α-1 cукцинил-КоА-лигазы [ADP-образующей], митохондриальная [Solanum tuberosum] | K67, K90 |
| CSA019997.1 | Компонент сукцинилтрансферазы остатка дигидролипоиллизина из 2-оксоглутаратдегидрогеназного комплекса 2, подобный митохондриальному [Nelumbonucifera] | K369 |
| CSA028417.1 | Компонент сукцинил-трансферазы остатка дигидролипоиллизина из 2-оксоглутаратдегидрогеназного комплекса 2, подобный митохондриальному [Nelumbonucifera] | K334 |
| CSA016078.1 | Каталитическая субъединица 5 изоцитратдегидрогеназы [NAD], подобная митохондриальной [Malus domestica] | K139 |
| CSA035317.1 | Серин-глиоксилат-аминотрансфераза [Capsicum annuum] | K25, K353, K223 |
| CSA025613.1 | Серин-гидроксиметилтрансфераза, митохондриальная [Nicotiana attenuata] | K407 |
| CSA026298.1 | Серин-гидроксиметилтрансфераза, митохондриальная [Vitis vinifera] | K488 |
| CSA000918.1 | Глутамат-глиоксилат-аминотрансфераза 2 [Jatropha curcas] | K35 |
| CSA015996.1 | Глициндегидрогеназа (декарбоксилирующая), митохондриальная [Nelumbo nucifera] | K459 |
| CSA020117.1 | Глициндегидрогеназа (декарбоксилирующая), митохондриальная [Nelumbo nucifera] | K454 |
| CSA019996.1 | Аминометилтрансфераза, митохондриальная [Nelumbo nucifera] | K395, K168 |
| CSA029723.1 | H-белок 3 системы расщепления глицина, митохондриальный [Juglans regia] | K152, K156 |
| CSA035939.1 | Изоформа X1 пероксисомальной оксидазы GLO1 (S)-2-гидроксикислот [Cajanus cajan] | K347 |
| CSA001272.1 | Изофермент 3 каталазы [Nicotiana tabacum] | K495 |
| CSA020605.1 | Большая цепь рибулозобисфосфаткарбоксилазы (хлоропласт) [Camellia crapnelliana] | K60, K62, K68, K150, K139, K190, K300 |
| CSA036938.1 | Большая цепь рибулозобисфосфаткарбоксилазы (хлоропласт) [Camellia crapnelliana] | K21, K25, K198, K267 |
| CSA001114.1 | Изофермент глутаминсинтетазы в листьях (хлоропласт) [Juglans regia] | K355 |
| CSA010785.1 | Цитоплазматический изофермент 1 глутаминсинтетазы [Gossypium raimondii] | K272, K267 |
| CSA034848.1 | β-субъединица сукцинил-КоА-лигазы [ADP-образующей] [Morus notabilis] | K89 |
К основным производным цикла ТСА относятся сукцинат и сукцинил-КоА. Из участвующих в этом цикле ферментов 7 модифицированы сукцинилированием, включая LSC1, LSC2, DLD, OGDH, DLST, CS и IDH3 (рис. 4б). Двадцать сайтов сукцинилирования идентифицировано в этих белках (табл. 1). Три репрезентативных LC-MS/MS-спектра сукцинилированных пептидов, полученных из ферментов цикла TCA: LSC1, LSC2 и DLD, ‒ представлены в Приложении 5 , рис. S4‒S6 . На основании полученных результатов мы предположили, что в чае сукцинилирование лизина может быть вовлечено в регуляцию цикла TCA, метаболизм глицина, серина и треонина и метаболизм глиоксилата и дикарбоксилата.
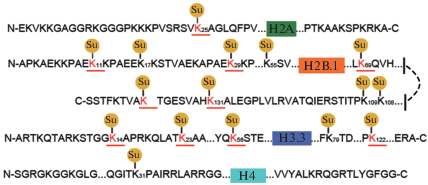
Сукцинилирование лизинов в гистонах
Посттрансляционные модификации гистонов важны для экспрессии генов и репарации ДНК [3]. В соответствии с анализом состава белковых доменов, значительное количество сукцинилированных лизинов обнаружено в гистонах. Всего в гистонах обнаружено 16 сайтов сукцинилирования лизина (рис. 5, Приложение 6 , см. сайт http://www.molecbio.ru/downloads/2020/1/supp_ Qiu_rus.pdf). По одному сайту сукцинилирования обнаружено в гистонах H2A (K25) и H4 (K31), 9 сайтов сукцинилирования обнаружено в гистоне H2B (K11, K17, K29, K55, K69, K108, K109, K131, K139) и 5 сайтов – в гистоне H3 (K14, K23, K56, K79, K122). По сравнению с данными для Anji Baicha [14], впервые в чае идентифицировано 10 сайтов, включая H2AK25, H2BK11, K2BK29, H2BK69, H2BK131, H2BK139, H3K14, H3K23, H3K56 и H3K122. Более того, H3K14, H3K56, H3K122 и H4K31 ранее обнаружены у человека [6], Drosophila melanogaster [3] и Toxoplasma gondii [19], что указывает на консервативность этих сайтов у животных и растений.
Сеть PPI по белкам с сукцинилированным лизином
В ходе дальнейшего изучения сукцинилированных белков чая с помощью базы данных STRING и программного обеспечения Cytoscape сконструированы сети PPI. В результате обнаружена тесная взаимосвязь для 59 сукцинилированных белков (рис. 6). Подробная информация о белках, вовлеченных в сеть, представлена в Приложении 7 (см. сайт http://www.molecbio.ru/ downloads/2020/1/supp_Qiu_rus.pdf). Используя MCODE, из полной сети взаимодействий мы извлекли несколько высокообогащенных кластеров взаимодействия. Три наиболее обогащенные взаимодействиями группы связаны с первичным метаболизмом, что согласуется с анализом путей KEGG (рис. S7 Приложения 5 ). С целью оценить важность сукцинилированных белков, находящихся в узлах полной сети, мы рассчитали степень каждого белка. Выявлено 14 лизинсукцинилированных белков, имеющих степень не менее 10. Интересно, что эти белки вовлечены в метаболизм глиоксилата и дикарбоксилата, глицина, серина и треонина и цикл ТСА. Таким образом, сукцинилирование лизина может играть важную роль в регуляции первичного метаболизма.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В работе изучен сукцинилом лизина в листьях чая QN3. Для характеристики сукцинилированных белков и биохимических путей с их участием использованы методы биоинформатики. В результате обнаружено 142 лизина со сдвигом массы 100.0186 Да. Согласно аннотации Unimod, наиболее вероятной структурой для этого сдвига масс считается сукцинильная группа или ее изомер ‒ метилмалонильная группа. Стоит отметить, что антитела, использованные в работе, специфически узнают сукцинилирование. Об использовании этих высокоспецифичных антител уже сообщалось ранее [4, 10‒12, 14]. Это главная причина, по которой есть основание считать, что определенная нами модификация ‒ сукцинилирование.
Сайты сукцинилирования обнаружены в 86 белках. Эти белки ассоциированы с метаболизмом глиоксилата и дикарбоксилата, метаболизмом глицина, серина и треонина и циклом ТСА. Анализ сети PPI показал, что сукцинилированные белки этих трех путей метаболизма находятся в тесном взаимодействии. На основании полученных данных можно говорить о том, что сукцинилирование лизина играет важную роль в метаболических путях в листьях чая. Метаболизм глиоксилата и дикарбоксилата тесно связан с метаболизмом глицина, серина и треонина, так как последний обеспечивает первый гидроксипируватом и глиоксилатом. Обнаружено 34 сайта сукцинилирования в 11 участвующих в обоих процессах белках, включая glyA, AGXT, GGAT, GLDC и другие.
Среди этих белков glyA (EC 2.1.2.1) представляет собой пиридоксальфосфат (витамин B6)-зависимый фермент, который катализирует превращение серина в глицин и наоборот [20]. У растений активность glyA обнаружена в цитозоле [21‒23], митохондриях [21‒25], пластидах [21, 23, 26] и ядре [25]. В отсутствие glyA происходит повреждение ДНК растений; останавливается ряд реакций, таких как фотосинтез, что ведет к возникновению соответствующих заболеваний [27, 28]. Сообщалось, что рецессивная мутация glyA 1-1 вызывала нарушение регуляции гибели клеток и, как следствие, приводила к образованию хлоротических и некротических повреждений в различных условиях окружающей среды [29]. На сегодняшний день нет сообщений, в которых изучена функциональная значимость сукцинилирования glyA в растениях. Нами идентифицировано 2 сайта сукцинилирования glyA (K407 в CSA025613.1, K488 в CSA026298.1) в пиридоксальфосфатзависимом трансферазном домене. Это значит, что сукцинилирование glyA может играть важную роль в регуляции конверсии серина в глицин. Кроме того, в одном из исследований показано, что glyA важен в одноуглеродном метаболизме и фотодыхании у высших растений, то есть он вовлечен в процессы роста и развития растений и устойчивости к биотическим и абиотическим стрессам [30]. Это говорит о том, что сукцинилирование glyA может также существенно влиять на рост и развитие, а также на устойчивость к биотическим и абиотическим стрессам у растений чая.
AGXT (EC 2.6.1.44/2.6.1.45/2.6.1.51) и GGAT (EC 2.6.1.4/2.6.1.2/2.6.1.44) ‒ это два ключевых фермента процесса фотодыхания, которые катализируют реакцию трансаминирования [31, 32]. Функция AGXT ‒ катализ конверсии серина и глиоксилата в гидроксипируват и глицин [33, 34]. Ранее показано, что физиологическая функция AGXT заключается в преодолении таких стрессов, как инфекция патогенами, засоление и засуха [35]. Например, сверхэкспрессия AGXT в Cucumis melo приводила к значительному повышению устойчивость растения к болезням [36]. Сверхэкспрессия гена AtAGT1 Arabidopsis thaliana, кодирующего AGXT, значительно повышала солеустойчивость трансгенной ряски [37]. Аналогично, GGAT катализирует превращение глутамата и глиоксилата в α-кетоглутарат и глицин [38, 39]. Ранее показано, что фотореспираторные аминотрансферазные реакции, катализируемые GGAT и AGXT, важны для регуляции уровня содержания аминокислот [40]. Нами в AGXT идентифицировано три сайта сукцинилирования (K52, K353, K223 в CSA035317.1) и один сайт в GGAT (K35 в CSA000918.1). Следовательно, активности AGXT и GGAT могут регулироваться сукцинилированием лизина. Однако регуляция фотодыхания под действием AGXT и GGAT изучена не до конца [31, 32]. Проведенное нами исследование открывает новые возможности для понимания механизмов регуляции AGXT и GGAT.
В ходе анализа нами обнаружены сукцинилированные белки, вовлеченные в цикл TCA. Ранее сукцинилирование лизина картировано в ферментах цикла TCA в Brachypodium distachyon L. [17] и Taxus × media [18]. Нами идентифицировано 20 сайтов сукцинилирования на семи белках, функционирующих в цикле TCA, что предполагает участие сукцинилирования лизина в регуляции цикла TCA. Интересно, что два сайта сукцинилирования (K67 и K90 в CSA028548.1) найдены в LSC1 (EC 6.2.1.4/6.2.1.5) и один (K89 в CSA034848.1) в LSC2 (EC 6.2.1.4/6.2.1.5). Функция LSC заключается в катализе нуклеотидзависимой конверсии сукцинил-КоА в сукцинат [41]. По-видимому, сукцинилирование лизина, влияя на ферментативную активность LSC, опосредованно регулирует выработку сукцинил-КоА и, как следствие, сукцинилирование. Кроме того, белок DLD (EC 1.8.1.4), который наиболее интенсивно сукцинилирован по остаткам лизина (K146, K149, K154, K334, K431 и K446 в CSA010160.1), также участвует в цикле TCA. Ранее установлено, что сверхэкспрессия DLD повышает фотосинтез и, в свою очередь, накопление биомассы арабидопсиса [42]. Однако, насколько нам известно, пока нет сообщений о функциональной роли сукцинилирования DLD в растениях чая. Может ли сукцинилирование DLD регулировать фотосинтез, еще предстоит выяснить.
Работа выполнена при финансовой поддержке Significant Application Projects of Agriculture Technology Innovation in Shandong Province, the Technology System of Modern Agricultural Industry in Shandong Province (SDAIT-19-01) и the Special Foundation for Distinguished Taishan Scholar of Shangdong Province (no. ts201712057).
Статья не содержит исследований с участием людей или животных, выполненных кем-либо из авторов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Witze E.S., Old W.M., Resing K.A., Ahn N.G. (2007) Mapping protein post-translational modifications with mass spectrometry. Nat. Methods. 4(10), 798‒806.
Pan J., Chen R., Li C., Li W., Ye Z. (2015) Global analysis of protein lysine succinylation profiles and their overlap with lysine acetylation in the marine bacterium Vibrio parahemolyticus. J. Proteome Res. 14(10), 4309‒4318.
Xie Z., Dai J., Dai L., Tan M., Cheng Z., Wu Y., Boeke J.D., Zhao Y. (2012) Lysine succinylation and lysine malonylation in histones. Mol. Cell. Proteomics. 11(5), 100‒107.
Zhang Z., Tan M., Xie Z., Dai L., Chen Y., Zhao Y. (2011) Identification of lysine succinylation as a new post-translational modification. Nat. Chem. Biol. 7(1), 58‒63.
Du J., Zhou Y., Su X., Yu J.J., Khan S., Jiang H., Kim J., Woo J., Kim J.H., Choi B.H. (2011) Sirt5 is an NAD-dependent protein lysine demalonylase and desuccinylase. Science. 334(6057), 806‒809.
Weinert B., Schölz C., Wagner S., Iesmantavicius V., Su D., Daniel J., Choudhary C. (2013) Lysine succinylation is a frequently occurring modification in prokaryotes and eukaryotes and extensively overlaps with acetylation. Cell Rep. 4(4), 842‒851.
Xie L., Liu W., Li Q., Chen S., Xu M., Huang Q., Zeng J., Zhou M., Xie J. (2014) First succinyl-proteome profiling of extensively drug-resistant Mycobacterium tuberculosis revealed involvement of succinylation in cellular physiology. J. Proteome Res. 14(1), 107‒119.
Cheng Y., Hou T., Ping J., Chen G., Chen J. (2016) Quantitative succinylome analysis in the liver of non-alcoholic fatty liver disease rat model. Proteome Sci. 14(1), 3.
Xie L., Li J., Deng W., Yu Z., Fang W., Chen M., Liao W., Xie J., Pan W. (2017) Proteomic analysis of lysine succinylation of the human pathogen Histoplasma capsulatum. J. Proteomics. 154, 109‒117.
Jin W., Wu F. (2016) Proteome-wide identification of lysine succinylation in the proteins of tomato (Solanum lycopersicum). PLoS One. 11(2), e0147586.
Zhang Y., Wang G., Song L., Mu P., Wang S., Liang W., Lin Q. (2017) Global analysis of protein lysine succinylation profiles in common wheat. BMC Genomics. 18(1), 309.
Feng S., Jiao K., Guo H., Jiang M., Hao J., Wang H., Shen C. (2017) Succinyl-proteome profiling of Dendrobium officinale, an important traditional Chinese orchid herb, revealed involvement of succinylation in the glycolysis pathway. BMC Genomics. 18(1), 598.
Smestad J., Erber L., Chen Y., Maher L.J. 3rd. (2018) Chromatin succinylation correlates with active gene expression and is perturbed by defective TCA cycle metabolism. iSience. 2, 63–75.
Xu Y.X., Shen C.J., Ma J.Q., Chen W., Mao J., Zhou Y.Y., Chen L. (2017) Quantitative succinyl-proteome profi-ling of Camellia sinensis cv. “Anji Baicha” during periodic albinism. Sci. Rep. 7(1), 1873.
Tyanova S., Temu T., Cox J. (2016) The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat. Protoc. 11(12), 2301‒2319.
Young M.D., Wakefield M.J., Smyth G.K., Oshlack A. (2010) Gene ontology analysis for RNA-seq: accounting for selection bias. Genome Biol. 11(2), R14.
Zhen S., Deng X., Wang J., Zhu G., Cao H., Yuan L., Yan Y. (2016) First comprehensive proteome analyses of lysine acetylation and succinylation in seedling leaves of Brachypodium distachyon L. Sci. Rep. 6, 31576.
Shen C., Xue J., Sun T., Guo H., Zhang L., Meng Y., Wang H. (2016) Succinyl-proteome profiling of a high taxol containing hybrid Taxus species (Taxus × media) revealed involvement of succinylation in multiple me-tabolic pathways. Sci. Rep. 6, 21764.
Li X., Hu X., Wan Y., Xie G., Li X., Chen D., Cheng Z., Yi X., Liang S., Tan F. (2014) Systematic identification of the lysine succinylation in the protozoan parasite Toxoplasma gondii. J. Proteome Res. 13(12), 6087‒6095.
Bhavani B.S. (2009) Structure-function relationships in serine hydroxymethyltransferase. Thesis for the degree of PhD in Biochemistry to the University of Misore. Mysore, India.
Besson V., Neuburger M., Rebeille F., Douce R. (1995) Evidence for three serine hydroxymethyltransferases in green leaf cells. Purification and characterization of the mitochondrial and chloroplastic isoforms. Plant Physiol. Biochem. 33(6), 665‒673.
Gardeström P., Edwards G.E., Henricson D., Ericson I. (2010) The localization of serine hydroxymethyltransferase in leaves of C3 and C4 species. Physiol. Plant 64(1), 29‒33.
Neuburger M., Rébeillé F., Jourdain A., Nakamura S., Douce R. (1996) Mitochondria are a major site for folate and thymidylate synthesis in plants. J. Biol. Chem. 271(16), 9466‒9472.
Turner SR., Ireland RJ., Morgan C., Rawsthorne S. (1992) Identification and localization of multiple forms of serine hydroxymethyl transferase in pea (Pisum sativum) and characterization of a cDNA encoding a mitochondrial isoform. J. Biol. Chem. 267(19), 13528‒13534.
Rebeille F., Neuburger M., Douce R. (1994) Interaction between glycine decarboxylase, serine hydroxymethyl-transferase and tetrahydrofolate polyglutamates in pea leaf mitochondria. Biochem. J. 302(1), 223‒228.
Zhang Y., Sun K., Sandoval FJ., Santiago K., Roje S. (2010) One-carbon metabolism in plants: characterization of a plastid serine hydroxymethyltransferase. Biochem. J. 430(1), 97‒105.
Mouillon JM., Aubert S., Bourguignon J., Gout E., Douce R., Rebeille F. (2010) Glycine and serine catabolism in non-photosynthetic higher plant cells: their role in C1 metabolism. Plant J. 20(2), 197‒205.
Li M., Limei C. (2008) The research advances on serine hydroxymethyltransferase gene in plants. Biotechnology Bulletin. 2, 15‒19.
Moreno J.I., Martín R., Castresana C. (2010) Arabidopsis SHMT1, a serine hydroxymethyltransferase that functions in the photorespiratory pathway influences resistance to biotic and abiotic stress. Plant J. 41(3), 451‒463.
Wang D., Liu H., Li S., Zhai G., Shao J., Tao Y. (2015) Characterization and molecular cloning of a serine hydroxymethyltransferase 1 (OsSHM1) in rice. J. Integr. Plant Biol. 57(9), 745‒756.
Zhang Z., Mao X., Ou J., Ye N., Zhang J., Peng X. (2015) Distinct photorespiratory reactions are preferentially catalyzed by glutamate:glyoxylate and serine:glyoxylate aminotransferases in rice. J. Photochem. Photobiol. B. 142(142C), 110‒117.
Liang Y., Zeng X., Peng X., Hou X. (2018) Arabidopsis glutamate:glyoxylate aminotransferase 1 (Ler) mutants generated by CRISPR/Cas9 and their characteristics. Transgenic Res. 27(1), 1‒14.
Liepman AH., Olsen L.J. (2010) Peroxisomal alanine:glyoxylate aminotransferase (AGT1) is a photorespiratory enzyme with multiple substrates in Arabidopsis thaliana. Plant J. 25(5), 487‒498.
Zhang Q., Lee J., Pandurangan S., Clarke M., Pajak A., Marsolais F. (2013) Characterization of Arabidopsis serine:glyoxylate aminotransferase, AGT1, as an asparagine aminotransferase. Phytochemistry. 85(2), 30‒35.
Abogadallah G.M. (2011) Differential regulation of photorespiratory gene expression by moderate and severe salt and drought stress in relation to oxidative stress. Plant Sci. 180(3), 540‒547.
Taler D., Galperin M., Benjamin I., Cohen Y., Kenigsbuch D. (2004) Plant eR genes that encode photorespiratory enzymes confer resistance against disease. Plant Cell. 16(1), 172‒184.
Yang L., Han H., Liu M., Zuo Z., Zhou K., Lü J., Zhu Y., Bai Y., Wang Y. (2013) Overexpression of the Arabidopsis photorespiratory pathway gene, serine:glyoxylate aminotransferase (AtAGT1), leads to salt stress tolerance in transgenic duckweed (Lemna minor). Plant Cell Tiss. Organ Cult. 113(3), 407‒416.
Igarashi D., Miwa T., Seki M., Kobayashi M., Kato T., Tabata S., Shinozaki K., Ohsumi C. (2010) Identification of photorespiratory glutamate:glyoxylate aminotransferase (GGAT) gene in Arabidopsis. Plant J. 33(6), 975‒987.
Liepman A.H., Olsen L.J. (2003) Alanine aminotransferase homologs catalyze the glutamate:glyoxylate aminotransferase reaction in peroxisomes of Arabidopsis. Plant Physiol. 131(1), 215‒227.
Igarashi D., Tsuchida H., Miyao M., Ohsumi C. (2006) Glutamate:glyoxylate aminotransferase modulates amino acid content during photorespiration. Plant Physiol. 142(3), 901‒910.
Przybyla-Zawislak B., Dennis R.A., Zakharkin S.O., McCammon M.T. (1998) Genes of succinyl-CoA ligase from Saccharomyces cerevisiae. Eur. J. Biochem. 258(2), 736‒743.
Timm S., Wittmiß M., Gamlien S., Ewald R., Florian A., Frank M., Wirtz M., Hell R., Fernie A.R., Bauwe H. (2015) Mitochondrial dihydrolipoyl dehydrogenase activity shapes photosynthesis and photorespiration of Arabidopsis thaliana. Plant Cell. 27(7), 1968‒1984.
Дополнительные материалы
- скачать ESC1.xls
- Таблица 1
- скачать ESC2.xls
- Таблица 2
- скачать ESC3.xls
- Таблица 3
- скачать ESC4.xls
- Таблица 4
- скачать ESC5.doc
- Рис. 1-7
- скачать ESC6.xls
- Таблица 5
- скачать ESC7.xls
- Таблица 6
Инструменты
Молекулярная биология