Молекулярная биология, 2020, T. 54, № 4, стр. 603-615
Геномные маркеры, ассоциированные с устойчивостью к низким температурам у Brassica rapa L.
А. В. Амосова a, Т. Е. Саматадзе a, Г. В. Мозгова b, В. Н. Кипень b, А. Г. Дубовская c, А. М. Артемьева c, О. Ю. Юркевич a, С. А. Зощук a, В. А. Лемеш b, О. В. Муравенко a, *
a Институт молекулярной биологии имени В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Беларусь
c Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
* E-mail: olgmur1@yandex.ru
Поступила в редакцию 05.03.2020
После доработки 05.03.2020
Принята к публикации 17.03.2020
Аннотация
Возделывание хозяйственно ценного вида Brassica rapa L. в регионах рискованного земледелия требует получения сортов, устойчивых к низким температурам. Механизм низкотемпературного стрессового ответа представляет собой сложную сеть процессов, которая затрагивает экспрессию многих генов, включая рибосомные, а также коррелирует с хромосомной изменчивостью растений. В представленной работе методами FISH- и ПЦР-анализа впервые проведено изучение внутривидовой изменчивости хромосом по числу и локализации кластеров 45S и 5S рДНК генов, а также комплекса молекулярных маркеров холодоустойчивости у озимых сортов B. rapa, районированных в регионах рискованного земледелия. В результате исследования выявлены SSR-маркеры (Na10-CO3, BrgMS5339-1) и SCAR-маркеры (BoCCA1-F/BoCCA1-1R1, BoCCA1-F/BoCCA1-2R1), пригодные для диагностики устойчивых и восприимчивых к холоду генотипов B. rapa. У масличных и листовых сортов обнаружено большое число маркеров холодоустойчивости и высокий уровень полиморфизма по распределению на хромосомах кластеров 45S и 5S рДНК, в том числе наличие гетероморфных вариантов хромосом. У кормовых сортов выявлены менее устойчивые к холоду генотипы и меньше полиморфных вариантов хромосом с сайтами рДНК. Таким образом, показано, что наименее устойчивые генотипы обладают меньшим хромосомным полиморфизмом по распределению кластеров рибосомных генов и наоборот. Полученные результаты могут представлять интерес для практической селекции при выведении новых, устойчивых к низким температурам сортов B. rapa.
ВВЕДЕНИЕ
Brassica rapa L. (Brassicaceae) – широко распространенный вид семейства капустных, который включает в себя разнообразные хозяйственно ценные листовые и корнеплодные культуры масличного, овощного и кормового назначения [1]. Считается, что геномы предковых форм B. rapa (2n = 2x = 20, геном АА) и родственного вида Brassica oleracea L. (капуста огородная) (2n = 2x = 18, геном СС) стали природными донорами аллополиплоидного генома еще одной хозяйственно ценной культуры ‒ рапса Brassica napus L. (2n = 4x = 38, геном AACC) [2, 3]. В ходе эволюции и доместикации появилось большое число морфотипов вида B. rapa, которые возделывают в районах с различными климатогеографическими условиями [1, 4‒6]. Культивация B. rapa в регионах рискованного земледелия требует получения и использования сортов и форм, устойчивых к низким температурам, что считается одним из определяющих факторов успешного роста, развития и высокой продуктивности сельскохозяйственной культуры.
Механизм низкотемпературного стрессового ответа представляет собой сложную сеть процессов, которая изменяет метаболизм и экспрессию генов растений [7, 8]. Факторы транскрипции и эффекторные гены, участвующие в реакции растений на холодовой стресс, в совокупности называются холодорегулируемыми генами (COR, cold-regulated) [9]. В настоящее время гены COR выделены и идентифицированы у нескольких видов растений, в том числе у видов, родственных B. rapa: Arabidopsis thaliana и B. napus [10‒12]. Показано, что эти гены могут индуцироваться и другими факторами абиотического стресса, в том числе обезвоживанием и засолением почвы [13, 14]. У устойчивых растений при холодовом стрессе активируется синтез криопротекторов [15] и фосфатидной кислоты, которая входит в состав липидов клеточных мембран [16‒18]. Обнаружено, что при высоком содержании малонового диальдегида (MDA, malondialdehyde) в мембранах растений происходит ускорение перекисного окисления липидов, в результате чего снижается устойчивость растения к холоду [19]. У рапса, B. napus, выявлена отрицательная корреляция относительной проводимости мембран с устойчивостью к холоду [20]. Обнаружено, что у B. rapa степень повреждения растений отрицательными температурами также коррелирует с содержанием MDA и относительной проводимостью мембран, а также выявлены ДНК-маркеры, основанные на полиморфизме микросателлитных последовательностей генома (SSR-маркеры): Na10-CO3, BrGMS4511 и BrGMS397, ‒ пригодные для диагностики устойчивых и восприимчивых к холоду генотипов [21]. При оценке устойчивости к холоду B. napus по SSR-маркерам идентифицировано три локуса количественных признаков (QTL, Quantitative Trait Loci): qSPADYL-6, qSPADYS-6 и qMADYS-6, ‒ ассоциированных с содержанием MDA и расположенных в одной области группы сцепления ‒ LG6. BLAST-анализ показал, что последовательности маркеров, сцепленных c этими QTL, коллинеарны с последовательностями ДНК хромосомы A08 у B. rapa [22]. Также у B. napus выявлены два гена, BnaA08g05330D и BnaA08g15470D, кодирующие COR-белки, связанные с формированием холодостойких генотипов. Кроме того, обнаружено высокое сходство последовательностей генов COR у B. napus с генами Bra039858 и Bra01579 у B. rapa [22].
В регуляции многих физиологических и метаболических процессов растений важную роль играют циркадные ритмы. Показано, что ассоциированный с циркадными ритмами белок CCA1 (транскрипционный фактор семейства MYB), участвует в позитивной регуляции холодового ответа растений путем индукции экспрессии генов CBF (C-repeat-binding factor), что определяет различия сельскохозяйственных культур по степени устойчивости к низким температурам [23]. У B. oleracea в гене BoCCA1 обнаружены многочисленные инсерции, делеции и однонуклеотидные полиморфизмы (SNP) и установлены последовательности фрагментов геномной ДНК в этом локусе, которые идентифицируют с помощью ПЦР (SCAR-маркеры, Sequence Characterized Amplified Region), что позволяет выявлять их наличие в двух аллелях гена (BoCCA1-1 и BoCCA1-2), ассоциированных с устойчивостью и восприимчивостью растений к низким температурам [24].
Воздействия неблагоприятных факторов окружающей среды могут проявляться на уровне структурно-функциональной организации хроматина и коррелируют с хромосомной изменчивостью растений [25]. Одним из самых нестабильных геномных регионов является рДНК [26]. У некоторых видов растений (A. thaliana, Phaseolus vulgaris, виды рода Capsicum и др.), произрастающих в разных природно-климатических условиях, выявлены значительные различия в размере генома из-за вариации числа копий 45S рДНК [27‒29]. Показано также, что хромосомное распределение и число кластеров 45S и 5S рДНК, а также транскрипционная активность рибосомных генов у особей одного и того же вида растений может меняться под влиянием различных факторов окружающей среды [30‒33]. У видов рода Brassica выявлен внутривидовой полиморфизм по хромосомной локализации кластеров 45S и 5S рДНК [34‒38]. В кариотипах озимых сортов рапса обнаружена сортовая специфичность сочетаний полиморфных вариантов хромосом и особенности хромосомного распределения 45S и 5S рДНК [39, 40].
Вместе с тем комплексного изучения хромосомной изменчивости рибосомных генов и молекулярных маркеров холодоустойчивости среди различных подвидов полиморфного вида B. rapa с широким ареалом распространения не проводилось. Такие исследования могут предоставить новые сведения о формировании геномных механизмов стрессового ответа в процессе эволюции и селекции.
Целью работы было изучение внутривидовой изменчивости хромосомного распределения кластеров генов 45S и 5S рРНК, а также комплекса молекулярных SSR- и SCAR-маркеров холодоустойчивости у масличных, листовых и кормовых озимых сортов B. rapa, районированных в регионах рискованного земледелия.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалом исследования служили образцы восьми морозостойких сортов Brassica rapa L. из разных мест произрастания (табл. 1). Семена получены из коллекции ФИЦ Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР), Санкт-Петербург, Россия.
Таблица 1.
Список изученных сортов B. rapa из разных мест произрастания
| № | Номер по каталогу ВИР | Название вида | Название сорта | Страна происхождения |
|---|---|---|---|---|
| 1 | к-45 | Капуста полеваяBrassica rapa ssp. oleifera (DC.) Metzg. (syn. Brassica campestris L.; Brassica rapa subsp. campestris (L.) A.R. Clapham) | Gruber | Финляндия |
| 2 | к-44 | Капуста полеваяBrassica rapa ssp. oleifera (DC.) Metzg. (syn. Brassica campestris L.; Brassica rapa subsp. campestris (L.) A.R. Clapham) | Rapido | Финляндия |
| 3 | к-371 | Капуста полеваяBrassica rapa ssp. oleifera (DC.) Metzg. (syn. Brassica campestris L.; Brassica rapa subsp. campestris (L.) A.R. Clapham) | Злата | Россия |
| 4 | к-109 | Капуста полеваяBrassica rapa ssp. oleifera (DC.) Metzg. (syn. Brassica campestris L.; Brassica rapa subsp. campestris (L.) A.R. Clapham) | Латвийская | Латвия |
| 5 | к-1406 | Репа кормовая – турнепс, Brassica rapa L. ssp. rapa Metzg. | Brunstadnepe | Норвегия |
| 6 | к-1405 | Репа кормовая – турнепс, Brassica rapa L. ssp. rapa Metzg. | Ova Daehnfeldt | Дания |
| 7 | к-242 | Репа листовая форма комацуна, Brassica rapa L. ssp. rapa var. komatsuna Makino | Goseki Late | Япония |
| 8 | к-217 | Репа листовая форма сирона, Brassica rapa L. ssp. rapa f. shirona Kitam | Okute Oosaka Shirona | Япония |
Митотические хромосомные препараты готовили из корневой меристемы проростков семян с использованием ДНК-интеркалирующего агента 9-аминоакридина (9-AMA) по методу, разработанному ранее для растений с хромосомами небольших размеров [41].
Для проведения FISH-анализа использовали следующие пробы ДНК:
1) pTa71, содержащую 18S-5.8S-26S (45S) последовательность рДНК мягкой пшеницы длиной 9 т.п.н. [42];
2) pTa794, содержащую последовательность 5S рДНК пшеницы длиной 420 п.н. [43].
Эти пробы ДНК метили (прямое мечение) флуорохромами SpectrumAqua и SpectrumRed (“Abbott Molecular”, Германия) с использованием метода ник-трансляции в соответствии с протоколами фирмы-производителя.
Процедуру FISH с комбинацией меченных проб 45S и 5S рДНК, а также отмывку хромосомных препаратов проводили по ранее описанному методу [44].
После процедуры FISH хромосомные препараты окрашивали флуоресцентным красителем DAPI (4',6-диамидин-2-фенилиндол), растворенным в среде Vectashield в конечной концентрацией 0.1 мкг/мл (“Vector Laboratories”, Великобритания).
Хромосомные препараты анализировали с использованием эпифлуоресцентного микроскопа Olympus BX61 (“Olympus”, Япония), соединенного с монохромной CCD-камерой (“Roper Scientific, Inc. Cool Snap”, США). Полученные изображения обрабатывали с использованием стандартного программного обеспечения Adobe Photoshop 10.0 (“Adobe”, США). Для каждого сорта анализировали не менее пяти растений и 15 метафазных пластинок. Идентификацию хромосомных пар в кариотипах проводили на основе анализа размеров и морфологии хромосом, а также распределения хромосомных маркеров в соответствии с цитологической номенклатурой, предложенной ранее [45].
Геномную ДНК выделяли из 100‒300 мг свежих листьев индивидуальных растений образцов B. rapa с помощью набора реактивов для выделения и очистки ДНК Genomic DNA Purification Kit согласно инструкции фирмы-производителя (“Thermo Fisher Scientific”, США). Концентрацию ДНК и степень ее очистки определяли с использованием спектрофотометра Implen Nano Photometer N50 (“Implen”, Германия). Для оценки генетической гетерогенности исследовали не менее 15 индивидуальных растений каждого сорта.
Целевые фрагменты ДНК амплифицировали с локусспецифическими парами праймеров, фланкирующими участки последовательностей генов, детерминирующих различия в устойчивости к пониженным отрицательным температурам и устойчивости к замораживанию у семейства Brassicaceae.
ПЦР проводили на амплификаторе SimpliAmp Thermal Cycler (“Applied Biosystems”) со следующими условиями реакции амплификации для ДНК-маркеров (табл. 2): 5 мин при 95°С; 35 циклов: 15 с при 95°С, 30 с при температуре отжига (см. табл. 2, колонка Та), 45 с при 72°С; и 10 мин при 72°С.
Таблица 2.
Олигонуклеотиды, использованные для амплификации генетических маркеров
| Генетический маркер | Последовательность, 5' → 3' | Ta, °C | Ссылка |
|---|---|---|---|
| Na 10-CO3 | F: 5'-TTGGGTGTCTTTGTTACCCC-3' R: 5'-ACCGAGAAGACTGATACGGG-3' |
58.0 | [46] |
| CB10578 | F: 5'-TGTCCACTCACTCTCTTTGTT-3' R: 5'-AGGCTAAGTTGAAGTGCAAG-3' |
56.0 | [46] |
| BrgMS653 | F: 5'-AGGAAGGAAATTGTGATTGCTC-3' R: 5'-TGTAAAAGAGGTGTGGATTTGC-3' |
57.0 | [47] |
| BrgMS609 | F: 5'-ATGGCTGTGAACTTGTGATGTG-3' R: 5'-CGGATGTTGGAAAGATGAGATT-3' |
59.0 | [47] |
| BrgMS5339 | F: 5'-CTACCTGAAGATGACCCAGACG-3' R: 5'-GCATACAAACCTCGTCCTAAGC-3' |
60.0 | [47] |
| BoCCA1 (F/1R1) | F: 5'-TGCCTTGAGGCTTTACGGTAGAG-3' R: 5'-CTGAGCAGTTGTCTTCCTGCAGA-3' |
60.0 | [24] |
| BoCCA1 (F/2R1) | F: 5'-TGCCTTGAGGCTTTACGGTAGAG-3' R: 5'-CAGTTCCGGTCTTTCTAGGATAAGGG-3' |
60.0 | [24] |
| BoCCA1 (1F2/1R2) | F: 5'-AACGTCTCTGTCACATGCTCCTC-3' R: 5'-CCATTCTCTTGCCTCTCCACCAAAG-3' |
62.0 | [24] |
| BoCCA1 (2F2/2R2) | F: 5'-GGACAAGGTCTCTATCACATGCTCCTA-3' R: 5'-CTGCCTCAACATCATCACTACTCGAA-3' |
62.0 | [24] |
ПЦР проводили в 25 мкл реакционной смеси, содержащей буфер Taq Turbo (“Евроген”, Россия): 2.5 мM MgCl2, 0.40 мкМ каждого праймера, 0.1 мкM каждого dNTPs, 0.04 ед. HS Taq ДНК-полимеразы (“Евроген”) и 50‒100 нг суммарной ДНК.
ПЦР-продукты разделяли электрофорезом в 2.0%-ном агарозном геле с добавлением раствора бромида этидия до конечной концентрации 0.5 мкг/мл при напряжении 100 В в течение 60 мин. Результаты электрофореза документировали с использованием системы E-Box CX5 (“Vilber”, Германия), программное обеспечение E-Box CX5 TS Edge 18.01.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Структурные варианты хромосом
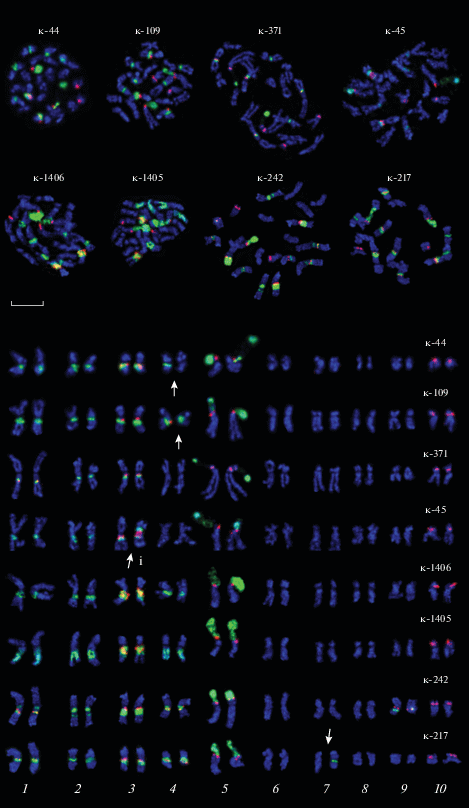
В результате проведенного FISH-анализа проб 45S и 5S рДНК в кариотипах изученных образцов B. rapa обнаружено от 8 до 12 кластеров 45S рДНК и 6‒10 кластеровов 5S рДНК (табл. 3, рис. 1). При визуальном анализе выявлена вариабельность по размерам (интенсивности сигналов гибридизации) кластеров 45S и 5S рДНК на хромосомах 1, 2, 3, 4 и 5 (табл. 3, рис. 1). Кроме того, обнаружен полиморфизм по числу кластеров 45S рДНК на хромосомах образцов масличного сорта Rapido к-44 (8‒9‒10 сайтов) и листового сорта Okute Oosaka Shirona к-217 (10‒11‒12 сайтов). В кариотипах образца масличного сорта Латвийская к-109 обнаружен полиморфизм по числу кластеров 5S рДНК (6‒7‒8). В кариотипе одного растения образца масличного сорта Gruber к-45 выявлена перестройка (изохромосома) одного из гомологов пары хромосомы 3. Кроме того, обнаружены дополнительные кластеры 45S и 5S рДНК в кариотипах образца листового сорта Goseki Late к-242 (хромосома 9L), а также сайт 45S рДНК у образца листового сорта Okute Oosaka Shirona к-217 (хромосома 7L). Следует заметить, что в кариотипах образцов масличных сортов Rapido к-44, Gruber к-45, Латвийская к-109 и листового сорта Okute Oosaka Shirona к-217 идентифицированы как гомоморфные, так и гетероморфные сочетания гомологов хромосом 3, 4 и 7 (табл. 3, рис. 1). В кариотипах кормовых сортов Brunstadnepe к-1406 и Ova Daehnfeldt к-1405 внутрисортовой полиморфизм по размерам и локализации кластеров 45S и 5S рДНК не выявлен. Между собой кариотипы этих сортов различались только размерами кластеров 45S рДНК на длинном плече хромосом 2 и 4.
Таблица 3.
Распределение кластеров рДНК на хромосомах изученных сортов B. rapa
| Образец | Хромосомы с 45S (/двойным сайтом рДНК) | Хромосомы с 5S рДНК | Хромосомы с колокализованными сайтами 45S (/двойным сайтом) + 5S рДНК, с указанием полиморфных или перестроенных вариантов хромосом | Общее число хромосом с 45S/5S рДНК (сортовые особенности) | |||
|---|---|---|---|---|---|---|---|
| 2 L ПЦ/ПК | 10 S СТ |
1 L ПЦ/ПК + ПК | 3 L ПЦ/ПК + ПК |
4 L ПЦ/ПК + МД |
5 S ЯОР |
||
| Rapido к-44 |
К/К | С | К/К + 0 | К/К + К | К/0 + 0 0/0 + 0 |
К + С | 8‒9‒10/6 (гетероморфизм по 45S рДНК на хромосоме 4) |
| Латвйская к-109 |
К/0 | С | К/К + 0 | К/К + С | К/К + С К/К + 0 |
К + С | 10/6‒7‒8 (гетероморфизм по 5S рДНК на хромосоме 4) |
| Злата к-371 |
К/M | С | C/К + C | С/С + С | 0/0 + 0 | К + К | 8/8 |
| Gruber к-45 |
К/C | С | С/К + 0 | С/С + К i С/С + К |
0/0 + 0 | К + С | 8/6 (гетероморфизм, изохромосома 3L) |
| Ova Daehnfeldt к-1406 |
К/С | С | К/К + 0 | К/К + К | C/C + 0 | К + С | 10/6 |
| Brunstadnepe к-1405 | К/К | С | К/К + 0 | К/К + К | К/К + 0 | К + С | 10/6 |
| Goseki Late к-242 |
К /С | С | С/К + С | К/К + К | С/К + С | К + С | 12/10 (на хромосоме 9L дополнительные сайты 45S и 5S рДНК) |
| Okute Oosaka Shirona к-217 |
К/С | С | С/К + С | К/К + К | К/К + С | К + С | 10‒11‒12/8 (на хромосоме 7L дополнительный сайт 45S рДНК, гетероморфизм) |
SSR-анализ
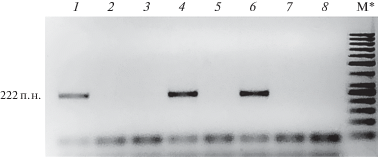
При анализе с использованием SSR-маркера Na10-CO3 выявлен амплифицируемый фрагмент размером 222 п.н. у масличных сортов Rapido к-44 и Злата к-371, а также у листового сорта Goseki Late к-242. У остальных сортов (масличных cортов Gruber к-45 и Латвийская к-109, листового сорта Okute Oosaka Shirona к-217 и двух кормовых сортов Brunstadnepe к-1406 и Ova Daenfeldht к-1405) эта последовательность не обнаружена (рис. 2).
Рис. 2.
Электрофоретическое разделение ампликонов для SSR-маркера Na10-CO3. Генотипы сортов B. rapa: 1 – Rapido, 2 – Gruber, 3 – Латвийская, 4 – Злата, 5 – Okute Oosaka Shirona, 6 – Goseki Late, 7 – Ova Daehnfeldt, 8 – Brunstadnepe. М* ‒ маркер длин ДНК (здесь и далее).
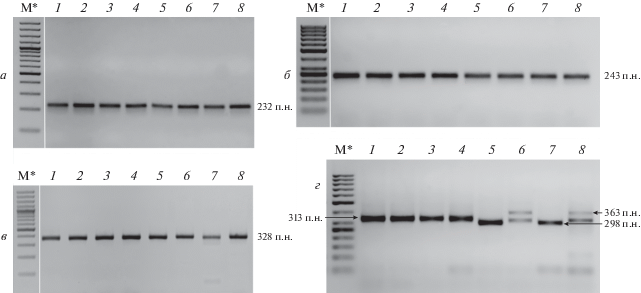
Исследован полиморфизм SSR-маркеров CB10578-1, BrgMS653-2, BrgMS609-1 и BrgMS5339-1 из группы сцепления LG6 B. napus, ассоциированных с QTL-областями qSPADYL-6, qSPADYS-6 и qMDAYS-6. Установлено, что SSR-маркеры CB10578-1, BrgMS653-2 и BrgMS609-1 электрофоретически мономорфны, поэтому дифференцировать по ним изученные сорта B. rapa не представлялось возможным (рис. 3а‒в). Для SSR-маркера BrgMS5339-1, фланкирующего области qSPADYS-6 и qMDAYS-6, определены полиморфные фрагменты. Последовательность размером 313 п.н. детектировали у масличных сортов Gruber к-45, Rapido к-44, Злата к-371 и Латвийская к-109. У листового сорта Goseki Late к-242 и кормового сорта Brunstadnepe к-1406 идентифицированы полиморфные фрагменты размером 313 п.н. и 363 п.н., а у листового сорта Okute Oosaka Shirona к-217 и кормового сорта Ova Daehnfeldt к-1405 ‒ последовательность размером 298 п.н. (рис. 3г).
Анализ с помощью SCAR-маркеров
У всех исследованных масличных и листовых сортов ПЦР-анализ с использованием праймеров BoCCA1-F/BoCCA1-1R1 выявил фрагмент размером 987 п.н., соответствующий аллелю BoCCA1-1, который связан с формированием холодоустойчивости у B. oleracea и B. napus. Для сортов репы кормовой Ova Daehnfeldt к-1405 и Brunstadnepe к-1406 идентифицирован фрагмент размером 752 п.н., который соответствовал аллелю BoCCA1-2, характерному для восприимчивых к холоду форм B. napus (рис. 4а). ПЦР-анализ с праймерами BoCCA1-1F2/BoCCA1-1R2 выявил у масличного сорта Злата к-371 амплифицируемый фрагмент размером 528 п.н., а для остальных изученных сортов B. rapa фрагмент размером 555 п.н. (рис. 4б).
Рис. 4.
Электрофоретическое разделение ампликонов для аллеля BOCCA1-1 (F/1R1) (а), BOCCA1-1 (1F2/1R2) (б), BOCCA1-2 (F/2R1) (в). Генотипы сортов B. rapa: 1 – Rapido, 2 – Gruber, 3 – Латвийская, 4 – Злата, 5 – Okute Oosaka Shirona, 6 – Goseki Late, 7 – Ova Daehnfeldt, 8 – Brunstadnepe.
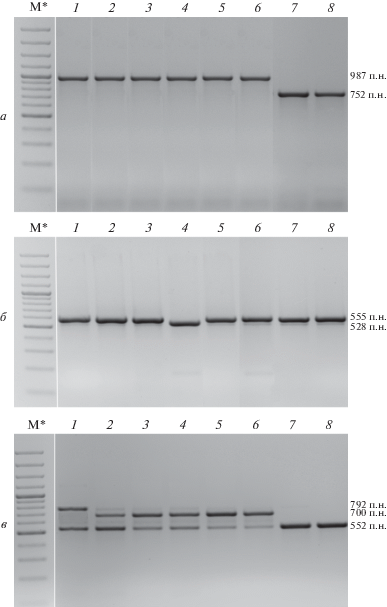
В результате ПЦР-анализа с праймерами BoCCA1-F/BoCCA1-2R1 последовательность аллеля BoCCA1-2 обнаружена у всех исследованных масличных и листовых сортов. Так, у масличного сорта Rapido к-44 выявлен фрагмент размером 792 п.н.; у масличных сортов Gruber к-45, Латвийская к-109, Злата к-371 и листовых сортов Okute Oosaka Shirona к-217 и Goseki Late к-242 ‒ размером 700 п.н. У всех этих сортов также идентифицирован фрагмент размером 552 п.н., а у кормовых сортов репы Ova Daehnfeldt к-1406 и Brunstadnepe к-1405 детектировали только этот фрагмент (рис. 4в). При использовании праймеров BoCCA1-2F2/BoCCA1-2R2 у всех изученных сортах выявлен фрагмент размером 531 п.н.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В регионах рискованного земледелия экстремальные погодные условия представляют серьезную проблему для сельского хозяйства, что вызывает необходимость получения новых сортов, устойчивых к абиотическому и в частности к холодовому стрессу [13, 48]. Установлено, что растения отвечают на холодовой стресс путем индукции экспрессии факторов транскрипции, регулирующих гены даунстрим, что обеспечивает им устойчивость к низким температурам. [49]. В этом случае белки CBF распознают и связываются с последовательностью CRT/ DRE (C-repeat/dehydration responsive element) в промоторе многих COR-генов, таким образом регулируя транскрипцию генов даунстрим [50]. Показано, что при холодовом стрессе у растений белок STCH4 (sensitive to chilling 4) модулирует процессинг рРНК и, будучи фактором биогенезиса рибосом, модулирует их формирование и запускает процесс устойчивости к холоду путем стимуляции трансляции белков CBF, необходимых для выживания растений при низких температурах [51]. Семейство генов рРНК занимает большую часть ядерного генома и отличается, с одной стороны, высокой степенью сохранности последовательностей и функций, а с другой ‒ широким диапазоном вариабельности по числу копий (CNV) и их локализации в геноме [26]. Показано, что число копий рДНК может варьировать как между видами, так и внутри видов [28, 42, 52, 53]. Например, в геноме L. usitatissimum число лабильных последовательностей ДНК, таких как гены рРНК, может изменяться в зависимости от условий окружающей среды [54, 55]. При абиотическом (холодовом) стрессе в геноме Solanum tuberosum CNV наблюдали для генов, связанных с адаптацией и устойчивостью растений; причем в этот процесс было вовлечено около 30% генома, в том числе рДНК [56]. Понятно, что это привело к высокой гетерогенности генома.
В результате проведенного нами FISH-анализа с использованием зондов 45S и 5S рДНК выявлен значительный полиморфизм по числу и распределению кластеров рибосомных генов на хромосомах озимых сортов B. rapa, районированных в регионах рискованного земледелия. Как правило, в кариотипах B. rapa кластеры 45S рДНК локализованы на парах хромосом 1, 2, 3, 4 и 5, а кластеры 5S рДНК – на парах хромосом 1, 3, 4, 5 и 10, причем на четырех парах хромосом (1, 3, 4 и 5) сайты 45S и 5S рДНК обычно расположены совместно [38, 45]. Сходное распределение кластеров рДНК наблюдали на хромосомах изучаемых нами кормовых сортов, за исключением отсутствия сигналов 5S рДНК на хромосомах 1 и 4. Кроме того, эти сорта были наименее полиморфными по локализации кластеров рДНК на хромосомах среди всех изученных нами сортов. У масличных сортов часто встречался гетероморфизм гомологов по локализации кластеров 45S и 5S рДНК, а у масличного сорта Gruber к-45 была обнаружена изохромосома по длинному плечу хромосомы 3 с удвоением всех сайтов рДНК. У изученных листовых сортов, как и у масличных сортов, мы наблюдали гетероморфизм гомологов по локализации сайтов рДНК, а также редкие полиморфные варианты наличия кластеров 45S и 5S рДНК на хромосоме 9 и 45S рДНК на хромосоме 7. Ранее подобная локализация сайтов рДНК на хромосомах 7 и 9 не была описана для B. rapa и других видов рода Brassica, содержащих A-субгеном [38, 45]. Наличие полиморфизма по числу и активности рибосомных генов считается одним из генетических показателей, отражающих устойчивость к абиотическому стрессу. Известно, что большое число генов, участвующих в различных функционально значимых клеточных механизмах, в том числе холодоустойчивости, тесно связаны с экспрессией рРНК [51, 56, 57]. При изучении популяций некоторых видов хвойных растений обнаружено влияние стрессовых факторов среды на уровень внутривидового полиморфизма по числу копий рДНК [58, 59], а также на изменение числа локусов 45S и 5S рДНК, выявляемое в кариотипах методом FISH [60, 61]. Изменение числа локусов 45S рДНК в зависимости от мест произрастания популяций наблюдали в кариотипах и других видов растений, в том числе Phaseolus vulgaris [27] и видов рода Capsicum [29]. Внутривидовая изменчивость числа сайтов 45S и 5S рДНК обнаружена в кариотипах сортов озимого и ярового рапса, адаптированных к определенным климатическим зонам [39]. В результате проведенного нами исследования показано, что в кариотипах изученных масличных, листовых и кормовых озимых сортов B. rapa повышен уровень хромосомного полиморфизма по числу и распределению сайтов рибосомных генов. Таким образом, подтверждена отмеченная другими авторами адаптивная значимость этого показателя.
Ранее отмечено, что в формировании устойчивости B. rapa и родственных видов B. oleracea и B. napus к низким температурам задействовано множество генов, запускающих ответ на холодовой стресс [21, 22, 24]. Для анализа сортов B. rapa мы использовали различные молекулярные маркеры, выявляющие холодоустойчивость генотипов у всех трех родственных видов. Так, Huang и др. [21] идентифицировали SSR-маркер Na10-CO3, ассоциированный с пониженным содержанием MDA и относительной проводимостью клеточной мембраны у растений B. rapa. Этот ДНК-маркер позволял достоверно дифференцировать устойчивые и неустойчивые к холоду растений F2 популяции B. rapa, полученной от скрещивания холодостойкого сорта Longyou 6 и не устойчивого к холоду сорта Tianyou 4. Использование маркера Na10-CO3 позволило нам обнаружить последовательность размером 222 п.н., определяющую устойчивость к пониженным температурам, только у двух масличных сортов и одного листового сорта. У кормовых сортов устойчивость по этому маркеру не обнаружена. Учитывая, что кормовую репу традиционно выращивают в районах рискованного земледелия, нами подтверждено, что устойчивость и чувствительность к холодовому стрессу обеспечивается комплексом генов.
Нами исследован полиморфизм четырех маркерных микросателлитных локусов из группы сцепления LG6 B. napus, сцепленных с QTL, ассоциированными с холодостойкостью. Ранее установлено, что гены устойчивости к пониженным температурам могут располагаться в области 1.069–15.652 M на хромосоме A08. Выявлено два гена, BnaA08g05330D и BnaA08g15470D, кодирующих COR-белки, и установлено, что эти гены определяют формирование холодостойких генотипов у B. napus. Замечено высокое сходство последовательностей генов COR B. napus с генами Bra039858 и Bra01579 B. rapa [22]. Нами установлено, что три SSR-маркера: CB10578-1, BrgMS653-2 и BrgMS609-1 ‒ мономорфны и не дифференцируют изученные сорта B. rapa. Для SSR-маркера BrgMS5339-1 идентифицированы полиморфные фрагменты размером 313 п.н. и 363 п.н. у листового сорта Goseki Late к-242 и кормового сорта Brunstadnepe к-1406. У всех исследованных масличных сортов детектировали только последовательность размером 313 п.н. Используя этот маркер, мы обнаружили один фрагмент размером 298 п.н. у листового сорта Okute Oosaka Shirona и кормового сорта Ova Daehnfeldt. Можно предположить, что выявленные различия по длине фрагментов связаны с микроделециями, инсерциями или точечными мутациями в геномах изученных сортов.
Известно, что циркадные ритмы влияют на многие физиологические и метаболические процессы у растений. Белок CCA1 играет важную роль в фитохромзависимой индукции фотосинтетических генов и регуляции циркадных ритмов. В условиях воздействия пониженных температур CCA1 определяет различия растений по степени устойчивости к холоду [23]. У B. oleracea обнаружены два аллеля гена BoCCA1: BoCCA1-1 (тип BN106, ассоциированный с морозостойкостью) и BoCCA1-2 (тип BN106, определяющий чувствительность растений к отрицательным температурам) [24]. Учитывая близкое родство геномов B. rapa и B. oleracea, мы провели поиск гомеологичных аллелей гена BoCCA1 у изученных сортов.
Ранее показано, что пары праймеров BoCCA1-F/BoCCA1-1R1 и BoCCA1-1F2/BoCCA1-1R2 позволяют выявлять последовательности, связанные с формированием холодоустойчивости у B. oleracea и B. napus. Первая пара праймеров фланкирует аллель BoCCA1-1 и позволяет амплифицировать фрагмент размером 987 п.н. у устойчивых к холоду форм B. oleracea и B. napus [24, 40]. Кроме того, у восприимчивых к холоду форм B. napus эта пара праймеров также выявляет фрагмент размером 752 п.н., который соответствует размеру последовательности аллеля BoCCA1-2 [40]. Вторая пара праймеров позволяет амплифицировать фрагмент размером 555 п.н. у устойчивых к холоду форм B. oleracea [24]. Проведенный нами ПЦР-анализ с использованием пары праймеров BoCCA1-F/BoCCA1-1R1 позволил выявить у изученных масличных и листовых сортов аллель BOCCA1-1, характерный для холодостойких форм (фрагмент размером 987 п.н.). Заметим, что только у сортов репы кормовой амплифицировался фрагмент размером 752 п.н., характерный для чувствительных к холоду форм и соответствующий аллелю BoCCA1-2. Учитывая тот факт, что размеры амплифицируемых фрагментов у изученных сортов B. rapa соответствовали размерам гомеологичных фрагментов у B. oleracea и B. napus, использование пары праймеров BoCCA1-F/BoCCA1-1R1 можно считать перспективным для эффективного выявления SCAR-маркера, дифферецирующего аллели BoCCA1-1 и BoCCA1-2 у B. rapa. Третья пара праймеров, BoCCA1-1F2/BoCCA1-1R2, выявляла у изученных сортов фрагмент размером 555 п.н., характерный для устойчивых к холоду форм B. oleracea [24]. Однако размеры амплифицируемых фрагментов практически не отличались у изучаемых сортов B. rapa, что не позволило нам дифференцировать сорта по этой последовательности.
Известно, что пары праймеров BoCCA1-F/ BoCCA1-2R1 и BoCCA1-2F2/BoCCA1-2R2 могут выявлять последовательности, характерные для восприимчивых к холоду генотипов B. oleracea и B. napus. Первая пара, фланкирующая аллель BoCCA1-2, позволяет амплифицировать у восприимчивых к холоду образцов фрагмент размером 792 п.н., а вторая пара определяет фрагмент размером 531 п.н. [24, 40]. Использование первой пары праймеров позволило нам выявить аллель BoCCA1-2 у всех изученных масличных и листовых сортов; при этом, у масличного сорта Rapido к-44 обнаружен фрагмент размером 792 п.н., соответствующий аналогичному фрагменту у B. napus, а у масличных сортов Gruber к-45, Латвийская к-109, Злата к-371 и листовых сортов Okute Oosaka Shirona к-217 и Goseki Late к-242 ‒ фрагмент меньшего размера (700 п.н). Также у всех этих сортов выявлен фрагмент размером 552 п.н. Ранее в последовательности гена ССA1 выявили многочисленные делеции, инсерции и точечные мутации [24]. из чего можно предположить, что у ряда сортов B. rapa произошла делеция в последовательности аллеля BoCCA1-2, но это пока только гипотеза. Использование второй пары праймеров, BoCCA1-2F2/BoCCA1-2R2, позволило нам выявить у всех масличных, листовых и кормовых сортов последовательность размером 531 п.н., которая также связана с чувствительностью образцов к холоду [24, 40]. Одинаковый размер фрагмента, амплифицируемого этими праймерами, не представляет возможности дифференцирования изучаемых сортов по этому параметру.
Таким образом, в представленной работе определены пары праймеров, выявляющие полиморфные фрагменты ДНК, которые позволяют дифференцировать генотипы B. rapa по устойчивости и восприимчивости к низким температурам и выявлять аллели BoCCA1-1 и BoCCA1-2, ассоциированные с этими особенностями генотипов.
В результате проведенного нами сравнительного анализа полученных результатов показано, что для кормовых сортов ‒ у них выявлено наименьшее число полиморфных вариантов хромосом с сайтами 45S и 5S рДНК ‒ характерны наименее устойчивые к холоду генотипы. У масличных и листовых сортов B. rapa обнаружено наибольшее число маркеров, связанных с холодоустойчивостью; при этом в кариотипах этих сортов выявлен высокий уровень полиморфизма по числу и локализации на хромосомах кластеров рДНК, в том числе наличие гетероморфных вариантов хромосом.
Таким образом, в результате комплексного изучения хромосомной изменчивости рибосомных генов и молекулярных маркеров холодоустойчивости, проведенного среди масличных, листовых и кормовых сортов высокополиморфного вида B. rapa, обнаружено, что наименее устойчивые генотипы обладают меньшим хромосомным полиморфизмом по числу и хромосомной локализации кластеров рибосомных генов и наоборот. Полученные в настоящей работе результаты могут представлять интерес для практической селекции при выведении новых, устойчивых к низким температурам сортов B. rapa.
Работа выполнена при финансовой поддержке Российского Фонда Фундаментальных Исследований (проект № 18-54-00027 Бел_а), а также в рамках договора с Белорусским Республиканским Фондом Фундаментальных Исследований (№ Б18Р-108 от 30.06.2018 г. “Комплексное изучение формирования морозостойкости на уровне геномов у рапса и сурепицы”) и Программы фундаментальных исследований государственных академий наук на 2013‒2020 годы (тема № 0120136 3824).
Статья не содержит каких-либо исследований с использованием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Артемьева А.М., Чесноков Ю.В., Клоке Э. (2008) Генетическое разнообразие и внутривидовые филогенетические взаимоотношения культур вида Brassica rapa L. по результатам анализа микросателлитов. Информ. вестник ВОГиС. 12, 608–619.
Nagaharu U., Nagaharu N. (1935) Genome analysis of Brassica with special reference to the experimental formation of B. napus and peculiar mode of fertilization. J. Japan. Bot. 7, 389–453.
Lu K., Wei L., Li X., Wang Y., Wu J., Liu M., Zhang C., Chen Z., Xiao Z., Jian H., Cheng F., Zhang K., Du H., Cheng X., Qu C., Qian W., Liu L., Wang R., Zou Q., Ying J., Xu X., Mei J., Liang Y., Chai Y.-R., Tang Z., Wan H., Ni Y., He Y., Lin N., Fan Y., Sun W., Li N.-N., Zhou G, Zheng H., Wang X., Paterson A.H., Li J. (2019) Whole-genome resequencing reveals Brassica napus origin and genetic loci involved in its improvement. Nat. Commun. 10(1), 1154.
Bailey L.H. (1940) Certain noteworthy Brassicas. Genets Herb. 4(9), 318–330.
Olsson G. (1954) Crosses within the campestris group of the genus Brassica. Hereditas. 40, 398–418.
Синская Е.Н. (1969) Историческая география культурной флоры. Ленинград: Наука.
Tomashow M.F. (1999) Plant cold acclimation: freezing tolerance genes and regulatory mechanisms. Annu. Rev. Plant Physiol. Plant Mol. Biol. 50, 571–599.
Cook G. (2004) Genetically Modified Language. The Discourse of Arguments for GM Crops and Food. London, UK: Routledge.
Thomashow M.F. (1998) Role of cold-responsive genes in plant freezing tolerance. Plant Physiol. 118, 1–7.
Cui B.M., Li Y.X., Le J.H., Zheng M.G., Bao H.F. (2003) Cloning and sequence analysis of COR15a gene from Arabidopsis thaliana. J. Shihezi University. 7, 87–89.
Zhong K.Y., Ye M.S., Hu X.W., Guo J.C. (2006) Role of the transcription factors CBF in plant cold tolerance. Yi Chuan. 28, 249–254.
Zhang T.G., Mao Y.S., Chen Q.Q., Zhou K., Sun W.C. (2016) Cloning and expression analysis of COR15-like gene in Brassica campestris ‘Longyou 6’. Acta Agric. Boreali-Occident. Sin. 25, 707–714.
Mickelbart M.V., Hasegawa P.M., Bailey-Serres J. (2015) Genetic mechanisms of abiotic stress tolerance that translate to crop yield stability. Nat. Rev. Genet. 16, 237–251.
Zhu J.K. (2016) Abiotic stress signaling and responses in plants. Cell. 167, 313–324.
Ananga A.O., Cebert E., Ochieng J.W., Kumar S., Kambiranda D., Vasanthaiah H, Tsolova V., Senwo Z., Konan K., Anike F.N. (2012) Prospects for transgenic and molecular breeding for cold tolerance in canola (Brassica napus L.). In: Oilseeds. Ed. Akpan U.G. Croatia: InTech, 1–32.
Smolenska-Sym G., Kacperska A. (1994) Phosphatidylinositol metabolism in low temperature-affected winter oilseed rape leaves. Physiol. Plant. 91, 1–8.
Smolenska-Sym G., Kacperska A. (1996) Inositol 1,4,5triphosphate formation in leaves of winter oil seed rape plants in response to freezing tissue water potential and abscisic acid. Physiol. Plant. 96, 692–698.
Testerink C., Munnik T. (2005) Phosphatidic acid: a multifunctional stress signaling lipid in plants. Trends Plant Sci. 10, 368–375.
Lin Y., Guo W., Xu Z., Jia Z. (2012) Cold resistance and changes in MDA and soluble sugar of lives of Ligustrum lucidum Ait in winter. Chin. Agric. Sci. Bull. 25, 68–72. https://www.oriprobe.com/journals/zgnxtb/ 2012_25.html
Huang H.L., Cao Z.Y., Tang B., Ning Z.L., Cui Z.B., Zhou Y.P. (2014) Electrical conductivity analysis of 17 rapeseed (Brassica napus L.) varieties’ cold resistance. Hunan Agric. Sci. 21, 1–3.
Huang Z., Zhang X., Jiang S., Qin M., Zhao N., Lang L., Liu Y., Tian Z., Liu X., Wang Y., Zhang B., Xu A. (2017) Analysis of cold resistance and identification of SSR markers linked to cold resistance genes in Brassica rapa L. Breed Sci. 67, 213–220.
Huang Z., Zhao N., Qin M., Xu A. (2018) Mapping of quantitative trait loci related to cold resistance in Brassica napus L. J. Plant Physiol. 231, 147‒154.
Green R.M., Tobin E.M. (1999) Loss of the circadian clock-associated protein 1 in Arabidopsis results in altered clock-regulated gene expression. Proc. Natl. Acad. Sci. USA. 96, 4176–4179.
Song H., Yi H., Han C.-T., Hur Y. (2018) Allelic variation in Brassica oleracea CIRCADIAN CLOCK ASSOCIATED 1 (BoCCA1) is associated with freezing tolerance. Hortic. Environ. Biotechnol. 59, 423–434.
Cattivell L., Baldi P., Crosatti C., Di Fonzo N., Faccioli P., Grossi M., Mastrangelo A.M., Pecchioni N., Stanca A.M. (2002) Chromosome regions and stress-related sequences involved in resistance to abiotic stress in Triticeae. Plant Mol. Biol. 48, 649–665.
Kobayashi T. (2008) A new role of the rDNA and nucleolus in the nucleus – rDNA instability maintains genome integrity. BioEssays. 30, 267–272.
Pedrosa-Harand A., de Almeida C.C., Mosiolek M., Blair M.W., Schweizer D., Guerra M. (2006) Extensive ribosomal DNA amplification during Andean common bean (Phaseolus vulgaris L.) evolution. Theor. Appl. Genet. 112, 924–933.
Long Q., Rabanal F.A., Meng D., Huber C.D., Farlow A., Platzer A., Zhang Q., Vilhjalmsson B.J., Korte A., Nizhynska V., Voronin V., Korte P., Sedman L., Mandáková T., Lysak M.A., Seren Ü., Hellmann I., Nordborg M. (2013) Massive genomic variation and strong selection in Arabidopsis thaliana lines from Sweden. Nat. Genet. 45, 884–890.
Scaldaferro M.A., da Cruz M.V., Cecchini N.M., Moscone E.A. (2016) FISH and AgNor mapping of the 45S and 5S rRNA genes in wild and cultivated species of Capsicum (Solananceae). Genome. 59, 95–113.
Cullis C.A. (1979) Quantitative variations of ribosomal RNA genes in flax genothrophs. Heredity. 42, 237–246.
Sobol’ M.A. (2001) Role of the nucleolus in plant cell response to environmental physical factors. Tsitol. Genet. 35, 72–84.
Boulon S., Westman B.J., Hutten S., Boisver F-M., Lamond A.I. (2010) The nucleolus under stress. Mol. Cell. 40, 216–227.
Goryachkina O.V., Badaeva E.D., Muratova E.N., Zelenin A.V. (2013) Molecular cytogenetic analysis of Siberian Larix species by fluorescence in situ hybridization. Plant Syst. Evol. 299, 471–479.
Kamisugi Y., Nakayama S., O’Neil C.M., Mathias R.J., Trick M., Fukui K. (1998) Visualization of the Brassica self-incompatibility S-locus on identified oilseed rape chromosomes. Plant Mol. Biol. 38, 1081–1087.
Schrader O., Budahn H., Ahne R. (2000) Detection of 5S and 25S rRNA genes in Sinapis alba, Raphanus sativus and Brassica napus by double fluorescence in situ hybridization. Theor. Appl. Genet. 100, 665–669.
Snowdon J., Friedt W. (2004) Molecular markers in Brassica oilseed breeding: current status and future possibilities. Plant Breed. 123, 1‒8.
Hasterok R., Wolny E., Hosiawa M., Kowalczyk M., Kulak-Ksiazczyk S., Ksiazczyk T., Heneen W.K., Maluszynska J. (2006) Comparative analysis of rDNA distribution in chromosomes of various species of Brassicaceae. Ann. Bot. 97, 205–216.
Zheng J.S., Sun C.Z., Xiao D., Zhang S.N., Bonnema G., Hou X.L. (2015) Karyotype variation and conservation in morphotypes of non-heading Chinese cabbage. Plant Syst. Evol. 301, 1781–1791.
Amosova A.V., Zemtsova L.V., Grushetskaya Z.E., Samatadze T.E., Mozgova G.V., Pilyuk Y.E., Volovik V.T., Melnikova N.V., Zelenin A.V., Lemesh V.A., Muravenko O.V. (2014) Intraspecific chromosomal and genetic polymorphism in Brassica napus L. detected by cytogenetic and molecular markers. J. Genet. 93, 133–143.
Мозгова Г.В., Хоружий Н.Е., Амосова А.В., Пилюк Я.Э., Белявский В.М., Лемеш В.А., Муравенко О.В. (2019) Генетический полиморфизм рапса (Brassica napus) в связи с морозостойкостью. Молекулярная и прикладная генетика. 26, 34–44.
Muravenko O.V., Amosova A.V., Samatadze T.E., Popov K.V., Poletaev A.I., Zelenin A.V. (2003) 9-aminoacridin: an efficient reagent to improve human and plant chromosome banding patterns and to standardize chromosome image analysis. Cytometry A. 51, 52–57.
Gerlach W.L., Bedbrook J.R. (1979) Cloning and characterization of ribosomal RNA genes from wheat and barley. Nucleic Acids Res. 7, 1869–1885.
Gerlach W.L., Dyer T.A. (1980) Sequence organization of the repeating units in the nucleus of wheat which contain 5S rRNA genes. Nucleic Acids Res. 8, 4851–4865.
Muravenko O.V., Yurkevich O.Yu., Bolsheva N.L., Samatadze T.E., Nosova I.V., Zelenina D.A. Volkov A.A., Popov K.V., Zelenin A.V. (2009) Comparison of genomes of eight species of sections Linum and Adenolinum from the genus Linum based on chromosome banding, molecular markers and RAPD analysis. Genetica. 135, 245–255.
Amosova A.V., Zemtsova L.V., Yurkevich O.Yu., Zhidkova E.N., Shostak N.G., Muravlev A.A., Artemyeva A.M., Samatadze T.E., Zoshchuk S.A., Muravenko O.V. (2017) Genomic changes in generations of synthetic rapeseed-like allopolyploid grown under selection. Euphytica. 213, 217–229.
Piquemal J., Cinquin E., Couton F., Rondeau C., Seignoret E., Doucet I., Perret D., Villeger M.J., Vincourt P., Blanchard P. (2005) Construction of an oilseed rape (Brassica napus L.) genetic map with SSR markers. Theor. Appl. Genet. 111, 1514–1523.
Xu J., Qian X., Wang X., Li R., Cheng X., Yang Y., Fu J., Zhang S., King G.J., Wu J., Liu K. (2010) Construction of an integrated genetic linkage map for the A genome of Brassica napus using SSR markers derived from sequenced BACs in B. rapa. BMC Genomics. 11, 594.
Porter J.R., Semenov M.A. (2005) Crop responses to climatic variation. Philos. Trans. R. Soc. Lond. B Biol. Sci. 360, 2021–2035.
Chinnusamy V., Zhu J., Zhu J.K. (2007) Cold stress regulation of gene expression in plants. Trends Plant Sci. 12, 444–451.
Zhao C., Lang Z., Zhu J.K. (2015) Cold responsive gene transcription becomes more complex. Trends Plant Sci. 20, 466–468.
Yu H., Kong X., Huang H., Wu W., Park J., Yun D.J., Lee B.H., Shi H., Zhu J.K. (2020) STCH4/REIL2 confers cold stress tolerance in Arabidopsis by promoting rRNA processing and CBF protein translation. Cell Reports. 30, 229–242.
Rogers S.O., Bendich A.J. (1987) Ribosomal RNA genes in plants: variability in copy number and in intergenic spacer. Plant Mol. Biol. 9, 509–520.
Bustamante F.O., Rocha L.C., Torres G.A., Davide L.C., Mittelmann A., Techio V.H. (2014) Distribution of rDNA in diploid and polyploid Lolium multiflorum Lam. and fragile sites in 45S rDNA regions. Crop Sci. 54, 617–625.
Oh T.J., Cullis C.A. (2003) Labile DNA sequences in flax identified by combined sample representational difference analysis (csRDA). Plant Mol. Biol. 52, 527–536.
Cullis C.A. (2005) Mechanisms and control of rapid genomic changes in flax. Ann. Bot. 95, 201–206.
Hardigan M.A., Crisovan E., Hamilton J.P. Kim J., Laimbeer P., Leisner C.P., Manrique-Carpintero N.C., Newton L., Pham G.M., Vaillancourt B., Yang X., Zeng Z., Douches D.S., Jiang J., Veilleux R.E., Buell C.R. (2016) Genome reduction uncovers a large dispensable genome and adaptive role for copy number variation in asexually propagated Solanum tuberosum. Plant Cell. 28, 388–405.
Li B., Kremling K.A.G., Wu P., Bukowski R., Romay M.C., Xie E., Buckler E.S., Chen M. (2018) Coregulation of ribosomal RNA with hundreds of genes contributes to phenotypic variation. Genome Res. 28, 1555–1565.
Govindraju D.R., Cullis C.A. (1982) Ribosomal DNA variation among populations of Pinus rigida Mill. (pitch pine) ecosystem. I. Distribution of copy numbers. Heredity. 69, 133–140.
Bobola M.S., Smith D.E., Klein A.S. (1992) Five major nuclear chromosome repeats represent a large and variable fraction of the genomic DNA of Picea rubens and P. mariana. Mol. Biol. Evol. 9, 125–137.
Bogunić F., Muratović E., Ballian D., Siljak-Yakovlev S., Brown S. (2007) Genome size stability among five subspecies of Pinus nigra Arnold s.l. Environ. Exp. Bot. 59, 354–360.
Bogunić F., Siljak-Yakovlev S., Muratović E., Ballian D. (2011) Different karyotype patterns among allopatric Pinus nigra (Pinaceae) populations revealed by molecular cytogenetics. Plant Biol. 13, 194–200.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология