Молекулярная биология, 2021, T. 55, № 5, стр. 748-771
Структурные, функциональные и эволюционные характеристики белков с повторами
Е. И. Дерюшева a, *, А. В. Мачулин b, О. В. Галзитская c, d, **
a Институт биологического приборостроения Российской академии наук, Федеральный исследовательский центр “Пущинский научный центр биологических исследований Российской академии наук”
142290 Пущино, Россия
b Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина Российской академии наук, Федеральный исследовательский центр “Пущинский научный центр биологических исследований
Российской академии наук”
142290 Пущино, Россия
c Институт белка Российской академии наук
142290 Пущино, Россия
d Институт теоретической и экспериментальной биофизики Pоссийской академии наук
142290 Пущино, Россия
* E-mail: evgenia.deryusheva@gmail.com
** E-mail: ogalzit@vega.protres.ru
Поступила в редакцию 16.03.2021
После доработки 29.03.2021
Принята к публикации 29.03.2021
Аннотация
В обзоре обобщены и систематизированы данные по классификации, таксономическому распределению, особенностям структуры и функционирования белков со структурными повторами. Рассмотрены современные подходы к идентификации структурных повторов в белках, в том числе особенности специализированных баз данных белковых доменов. Обсуждается роль белков со структурными повторами в патогенезе различных заболеваний, а также перспективы их использования в качестве каркасов для создания лекарственных препаратов. Проанализированы современные подходы к пониманию механизмов эволюционного развития белков со структурными повторами.
ВВЕДЕНИЕ
За последние 20 лет интерес к белкам со структурными повторами заметно увеличился в связи с их распространенностью, уникальностью функционирования и взаимосвязью с патогенезом различных заболеваний. Структурные повторы очень разнообразны как по длине (от нескольких аминокислот до структурных доменов из 100 и более остатков), так и по аминокислотному составу [1]. Предполагается, что до 25% всех белков содержат какой-либо повтор [1–3], при этом такие повторы находят почти в каждом третьем белке человека [1, 4].
Белки со структурными повторами участвуют, как правило, в выполнении различных функций, например, в регуляции функционирования клеточных органелл [5], связывании нуклеотидов [6], формировании антивирусного ответа [7], в механизмах клеточной вирулентности [8], передаче сигналов [9] и многих других.
Современные исследования указывают на существование связи между структурными повторами в белках и различными заболеваниями [10–12]. Например, патогенез таких заболеваний, как рак предстательной железы, доброкачественная гиперплазия предстательной железы и ревматоидный артрит связывают с полиморфизмом длины глутаминовых и глициновых повторов андрогенного рецептора [13, 14].
Кроме того, белки с повторами интересны для специалистов в области белковой инженерии и дизайна синтетических белков. Они рассматриваются в качестве перспективных стабильных каркасов для конструирования белков, которые способны распознавать мишени со сродством и специфичностью в ряде случаев большими, чем у антител [15, 16].
Такие белки используют и для проверки теорий эволюционной молекулярной биологии. Повторы принято рассматривать как удачную эволюционную стратегию, поскольку регулярность вторичной структуры и разнообразие трехмерной сборки приводят к существованию молекул разного размера со множеством значимых функций [17]. Как правило, структурные повторы (структурные домены, мотивы) имеют высокую степень идентичности, что свидетельствует в пользу гипотезы об общем предке, содержащем один структурный повтор или мотив [17]. При этом предполагается, что на ранних этапах эволюции эффективность функциональной активности достигалась путем олигомеризации одиночных повторов [18]. Однако есть примеры белковых комплексов с повторами, которые образуются как из олигомеров с единичными повторами, так и из одной цепи с множественными структурными повторами. К тому же процесс кооперативного сворачивания белка с множественными повторами термодинамически более выгоден, чем сворачивание гомоолигомерного белка из мономеров [18].
В нашем обзоре обобщены современные взгляды на структурные характеристики белков с повторами, механизмы их функционирования, особенности эволюционного развития, взаимосвязь с патогенезом заболеваний, а также оценены перспективы их использования в качестве каркасов для создания высокоэффективных лекарственных препаратов.
МЕХАНИЗМ ВОЗНИКНОВЕНИЯ ПОВТОРОВ
Структурные повторы представляют собой множественные копии участков различной длины белковой цепи. Повторы часто формируют домены или структурные мотивы, определяющие функционирование всего белка. Многочисленные исследования изменений в расположении концевых доменов (т.е. перестановки N- и C-концевых доменов в белках) показали, что основная роль в таких перестановках отводится механизму дублирования генов, кодирующих белки, слиянию и потере концевых доменов, а не появлению новых структурных доменов [19–30].
Поскольку предполагается, что повторы структурных доменов образуются за счет внутреннего дублирования, то при тандемном дублировании в гене копия встраивается рядом с ее источником [17, 31]. При этом сходство аминокислотных последовательностей может отражать информацию о недавних дублированиях [32].
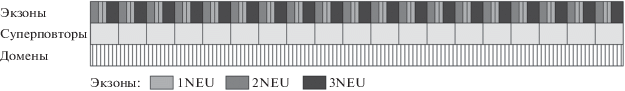
Формирование коротких белковых повторов может быть обусловлено образованием ДНК-шпилек при гипермутабильности минисателлитных локусов (повторяющихся единиц длиной более 10 нуклеотидов) в процессе рекомбинации [1, 33]. При этом число доменов может увеличиваться способом, аналогичным дублированию минисателлитных локусов, т.е. существуют “горячие точки”, которые фланкируют повторяющиеся регионы [34]. Если такие рекомбинационные мотивы расположены в интронах, то дупликацию можно рассматривать как перетасовку экзонов. Однако перетасовка экзонов не может объяснить эволюцию всех доменных повторов, так как в некоторых случаях перестановки повторов обнаруживаются и в отдельных экзонах. Следовательно, если такие мотивы существуют, то некоторые специфичные структурные повторы кодируются экзонами, в то время как другие повторы закодированы в основном в интронах [32]. Различие в размерах повторяющихся белковых структурных единиц соотносят с размером дублирующейся области ДНК. При этом принято различать микросателлитные [35, 36], минисателлитные [1, 33] и сателлитные повторы ДНК [31]. Пример кодирования структурных повторов в белке небулине [32, 37] показан на рис. 1 .
Анализ 24 эукариотических протеомов [32] позволил утверждать, что формирование повторяющихся областей обусловлено одновременным дублированием сразу нескольких доменов: при этом дублирование одного домена встречается реже. Кроме того, дублирование встречается в основном в середине белковой цепи между другими повторами [32]. Количество доменов, участвующих в каждом дублировании, может значительно различаться в пределах семейств доменов [32, 38, 39]. Дублирование определенного количества доменов в некоторых семействах обусловлено функциональными или структурными ограничениями, как это показано для небулина [32, 37]. Тенденция к дублированию определенного числа доменов найдена в некоторых семействах доменов [32]. Также утверждается [32], что отсутствует корреляция между размером домена и количеством повторов, т.е. более крупные структурные повторы дублируются с той же частотой, что и малые. Следовательно, можно предположить, что механизм, лежащий в основе дупликации повторов, не зависит от размера дублированной области. Дублирование внеклеточных доменов частично объясняется перетасовкой экзонов [32].
Опираясь на результаты сопоставления белковых повторов с кодирующей последовательностью ДНК, изучили распределение шаблонов интронов/экзонов у нескольких видов организмов и предложили разделить эти гены на два класса [40]. Гены первого класса имеют случайную длину экзона, который формируется за счет накопления интронов через случайные вставки внутри повторяющихся единиц. Гены второго класса состоят исключительно из экзонов, соответствующих повторяющимся единицам, за счет чего образуются локальные повторы интронов/экзонов [40].
Однако несмотря на активный поиск принципов возникновения структурных повторов в белках, единый механизм этого явления до сих пор не установлен.
КЛАССИФИКАЦИЯ БЕЛКОВ С ПОВТОРАМИ
Большое количество белков, содержащих повторяющиеся элементы, привело к необходимости их классификации с целью понимания взаимосвязи между аминокислотной последовательностью, структурой и функцией, а также их эволюционного развития.
В основе одной из первых классификаций белков с повторами лежали структурные различия таких повторов, при этом выделяли β-пропеллеры, β‑трилистники, анкириновые повторы (АнкП), лейцин-богатые повторы (ЛБП), тетратрикопептидные (ТПП) и армадилловые повторы (АрмП) [17].
В настоящее время выделяют пять основных классов белков со структурными повторами, которые различаются длиной повторяющихся единиц и общей структурной организацией. Часть классов делят на подклассы на основе структурных и функциональных особенностей молекул белков [41] (рис. 2).
Рис. 2.
Структурная классификация белков с повторами, основанная на различиях в длине повторяющихся единиц и общей структурной организации. Приведены примеры типичных белков каждого класса с указанием PDB-кодов их структур.
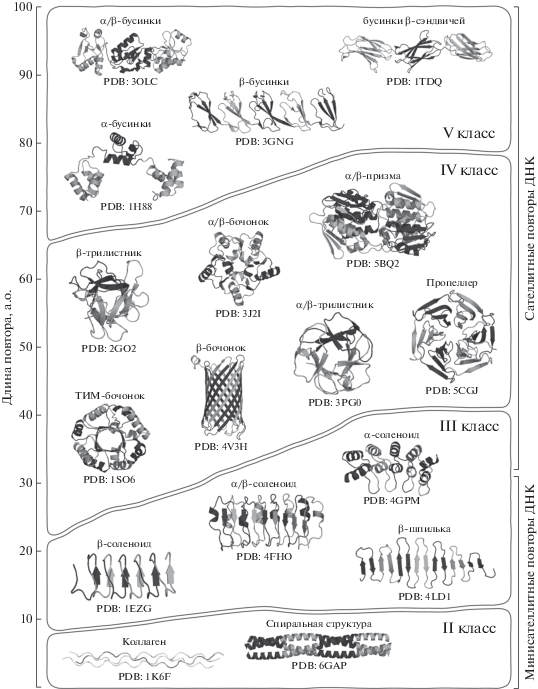
К классу I относятся белки и пептиды, образованные повторами из одного или двух аминокислотных остатков, которые могут формировать кристаллиты (мелкие кристаллы, не имеющие ясно выраженной ограненной формы), в большинстве случаев вредоносные для живых организмов. Структуры класса I соответствуют микросателлитным повторам ДНК [35, 36]. Неограниченный размер и высокая стабильность кристаллитов препятствуют применению к ним существующих методов деградации. Именно эти свойства принято рассматривать как основу цитотоксичности таких повторов [42], что подтверждено многочисленными экспериментами по влиянию таких агрегатов на живые организмы [43–47]. Участки с такого рода повторами преимущественно гидрофильны и имеют высокую степень неупорядоченности [48, 49]. Примером структуры класса I является кристаллит, образованный глутаминовым полипептидом [13, 50, 51], для которого характерна ассоциация с рядом нейродегенеративных заболеваний, в том числе с болезнью Хантингтона [45, 52]. В настоящее время примеры таких структур отсутствуют в PDB, доступны только смоделированные структуры некоторых представителей этого класса [50, 53].
К классу II относятся фибриллярные структуры с повторами из трех–семи аминокислотных остатков, стабилизированные межцепочечными взаимодействиями. К основным представителям этого класса относятся коллаген [54] и спиральные (двух- и более) структуры полипептидов [55]. Первичная структура коллагена состоит из трипептидного повтора GlyXY, где X и Y – любые остатки (в основном, пролин или его гидроксилированная форма, гидроксипролин). Трехмерные структуры коллагена представляют собой протяженные полипролиновые тройные спирали [54, 56, 57]. На сегодняшний день выделяют 29 различных типов коллагенов [58].
Спиральные (двух- и более) структуры полипептидов различаются гептадными повторами, представленными консенсусной последовательностью (abcdefg)n, обычно с гидрофобными остатками в положениях a и d и полярными остатками в других положениях [59, 60]. При этом структуры более высоких порядков, образованные такими спиралями, могут сильно отличаться по периодичности [55]. В отличие от класса I, структуры класса II имеют определенный размер и стабильность. Структуры класса II соответствуют минисателлитным повторам ДНК [33, 36].
К классу III принято относить белки с повторами из 5–40 остатков [61], в которых один повторяющийся элемент определяет формирование конечной складчатой структуры. В составе этого класса выделяют два подкласса белков – с соленоидными и несоленоидными структурами. Соленоидные структуры представляют собой особым образом свернутые полипептидные цепи, из которых формируются белки с массивными удлиненными структурами, отличающиеся от большинства глобулярных белков [61]. Повторяющейся структурной единицей соленоидных белков является специфический сегмент, состоящий обычно из 12–45 а.о. При этом структура белка формируется из одной, двух, трех или даже четырех сегментных конструкций, соединенных петлями. Сегментные конструкции в таких белках могут быть представлены α-спиралями, β-листами, полипролиновыми спиралями. По специфичности сегментных конструкций различают α-спиральные или β-структурные соленоиды, α/β-соленоиды [61–64]. К соленоидным белкам класса III относят группу белков с ЛБП (α/β-соленоид) [65–67]¸ белки-антифризы (antifreeze proteins, AFP) (β-структурные соленоиды) [68], белки с АнкП-, АрмП-, ТПП- и HEAT-повторами (HEAT repeats) (α-спиральные соленоиды) [69–72]. К несоленоидным структурам класса III относятся молекулы, имеющие сложную укладку, в центре которой находятся протяженные β-листы, формирующиеся за счет межцепочечных водородных связей и взаимодействия неполярных боковых цепей структуры с повторяющимися элементами [73]. В качестве примера таких белков можно привести, например, люминальный домен кальнексина [74], P-домен кальретикулина [75], поверхностный белок A Borrelia burgdorferi [76]. Как и структуры класса II, структуры класса III соответствуют минисателлитным повторам ДНК [33, 36].
К белкам класса IV относятся белки с “закрытыми” (не удлиненными) структурами с повторами из 30–60 аминокислотных остатков [77, 78]. Отдельные подклассы этого класса представлены белками со структурами α/β-бочонков (α/β barrel), ТИМ-бочонков (triosephosphate isomerase barrel, TIM barrel), пропеллеров (propeller), β-трилистников (β-trefoil) и α/β-трилистников (α/β-trefoil), α/β-призм (α/β-prism) и трансмембранных β-бочонков (transmembrane β-barrels). ТИМ-бочонки являются самой распространенной структурной повторяющейся единицей каталитических доменов, они присутствуют примерно в 10% всех известных ферментов [79, 80]. β-Пропеллеры представляют собой структуру, сформированную 4–8 высокосимметричными β-листами в форме лопастей, расположенных тороидально вокруг центральной оси. При этом образуется активный сайт белка, похожий на воронку [81]. Так, например, нейраминидаза вируса гриппа представляет собой шестилопастный β-пропеллерный белок, активный в форме тетрамера. Этот белок катализирует отщепление фрагментов сиаловой кислоты от белков клеточной мембраны, что способствует нацеливанию вновь продуцируемых вирионов на неинфицированные клетки [82]. Повторы β-трансдуцина (WD40) представляют собой β-пропеллерные структуры с 4–16 лопастями. Такие белки выполняют множество функций, включая передачу сигналов, регуляцию транскрипции и регуляцию клеточного цикла [83, 84]. β-Пропеллерные фитазы, содержащие шестилопастный β-винт, способны гидролизовать сложноэфирные связи в молекуле фитата, основной форме накопления фосфатов в растениях [85]. β-Трилистники представляют собой структуру, состоящую из шести β-шпилек, каждая из которых образована двумя β-листами. Вместе они образуют β-бочонок с треугольной “шапочкой”, состоящей из трех β-шпилек [86, 87]. Как структурная единица β-трилистник был идентифицирован в ингибиторе трипсина Кунитца (Kunitz STI protease inhibitor) некоторых растений [88, 89], в интерлейкинах-1α и -1β [90, 91], в факторах роста фибробластов 1 и 2 [18]. Структуры α/β-трилистников образованы β-шпильками, в структуре которых есть периферические α‑спирали. В качестве примера белков с такими повторами можно привести белки семейства лектинов [92].
Структура α/β-призмы представляет собой, как следует из ее названия, треугольную призму, каждая сторона которой содержит повторы из пары α-спиралей и антипараллельного β-листа, состоящего из трех β-участков. При этом α-спирали расположены внутри структуры, а β-листы – преимущественно снаружи. В качестве примера белков с α/β-призмами можно привести семейство карбоксивинилтрансфераз [93].
Белки с трансмембранными β-бочонками встречаются только во внешних мембранах грамотрицательных бактерий, в клеточных стенках грамположительных бактерий и наружных мембранах митохондрий и хлоропластов [78]. В число этих белков входят белки внешней мембраны OmpA и OmpX [94, 95], порины OmpF и PhoE [96], субстрат-специфичные порины LamB (Laminin subunit beta-1) и ScrY (Sucrose porin) [97], TonB-зависимые транспортеры сидерофоров железа FhuA и FepA [98]. Структуры класса IV соответствуют сателлитным повторам ДНК [31].
Белки класса V – это белки с большими повторяющимися единицами, способными независимо сворачиваться в стабильные домены. Размер отдельных структурных единиц составляет 50–60 а.о. Структуру молекулы белков с такими повторами можно представить как “бусинки на нити” (“beads on a string”), в которой отдельные бусинки соответствуют глобулярным доменам. Классическим примером таких доменов служит домен “цинковый палец” (Zn-finger) [99, 100] – наиболее распространенный ДНК-связывающий мотив, стабилизированный одним или двумя ионами цинка. В отдельный подкласс этого класса принято выделять молекулы, имеющие протяженную и полужесткую структуру за счет плотного соединения между повторяющимися модулями. Такие конструкции представляют собой спектриноподобные повторы (spectrin-like repeats), образованные 100–130 а.о. Каждый повтор при этом сформирован пучком из трех–пяти α-спиралей, расположенных вдоль оси молекулы. Некоторые α-спирали этих связок длиннее других, что позволяет соседним повторяющимся модулям взаимодействовать друг с другом вдоль оси в стержневой конструкции [101]. К белкам с такими структурами относятся спектрины [101], белок клеточной адгезии Ebh (cell-wall-associated adhesion protein Ebh) [102], семейство куллинов (сullin) [103]. В отдельный подкласс выделяют полужесткие конструкции, образованные β-структурными доменами длиной около 60 а.о., например, белки системы комплемента (Sushi repeats or complement control protein (CCP)), такие как селектины [104] и кадгерины [105]. Структуры класса V, как и структуры класса IV соответствуют сателлитным повторам ДНК [31].
Таким образом, существующая классификация белков со структурными повторами позволяет утверждать, что длина 20–50 а.о. соответствует “горячей точке” структурного разнообразия. Повторы именно такой длины могут укладываться в соленоиды, спирали, “бусинки на нитке” или в разные типы закрытых конструкций. Кроме того, повторы, в основе которых лежат β-листы, формируют более разнообразные структуры, чем структуры, содержащие α-спирали. Так, на основе β-листов в качестве повторяющихся структур могут формироваться β-соленоиды, β-спиральные складки, однослойные антипараллельные β-структуры, β-трилистники или β-пропеллеры. При этом α-спиральные структуры в основном ограничиваются α-спиральными катушками и α-соленоидами [41].
В дополнение отметим, что в ряде работ повторы делят на идеальные, почти идеальные и неидеальные. За основу такого деления приняты значении расстояния Хэмминга (число позиций, в которых соответствующие символы двух последовательностей одинаковой длины различны) между консенсусной последовательностью и выровненными последовательностями областей с повторами [49, 106, 107]. Среди белков с известной трехмерной структурой практически отсутствуют белки с идеальными повторами, поскольку они приобретают “неидеальность” из-за мутаций (вставки, делеции) в процессе эволюции. При этом идеальные повторы занимают особое место среди белковых повторов, поскольку каждый остаток в повторе обладает высокой структурной и функциональной значимостью, как, например, в структуре коллагена или некоторых структурах, содержащих повторы, сформированные β-листами [54, 56, 57, 108].
ТАКСОНОМИЧЕСКАЯ РАСПРОСТРАНЕННОСТЬ БЕЛКОВ С ПОВТОРАМИ
Структурные повторы в белках встречаются во всех царствах живых организмов. Количество структурных повторов в различных семействах белков может сильно варьировать. В среднем, у эукариотических организмов оно выше, чем у прокариотических, что связано с их полифункциональностью [32, 109, 110]. Высказываются также предположения, что такие повторы служат дополнительным источником изменчивости эукариотических организмов для компенсации низких скоростей их генерации (биологической продуктивности) [1]. Считается, что не менее 25% всех белков и треть всех белков из протеома человека содержат структурные повторы [1, 2].
Разбивка структурных повторов по длине на четыре группы: гомоповторы (длина 1 а.о.), микроповторы (1–3 а.о.), маленькие повторы (4–15 а.о.), доменные повторы (>15 а.о.) позволила проанализировать распределение содержания повторов различной длины во всех белках базы UniProt [111] (табл. 1).
Таблица 1.
Распределение количества записей в базе UniProt, содержащих гомоповторы, микроповторы, короткие повторы и доменные повторы*
| Доля и характеристика повтора | Археи | Бактерии | Эукариоты |
|---|---|---|---|
| Все белки | |||
| Количество записей в UniProt | 19 370 | 332 327 | 181 814 |
| Средняя длина | 288 | 313 | 436 |
| Число повторов | 6420 | 103 842 | 92 472 |
| Доля всех повторов | 0.331 | 0.312 | 0.509 |
| Доля гомоповторов | 0.006 | 0.006 | 0.086 |
| Доля микроповторов | 0.117 | 0.109 | 0.245 |
| Доля коротких повторов | 0.217 | 0.208 | 0.328 |
| Доля доменных повторов | 0.051 | 0.049 | 0.143 |
| Белки с повторами | |||
| Средняя длина | 355 | 404 | 572 |
| Доля гомоповторов | 0.019 | 0.019 | 0.169 |
| Доля микроповторов | 0.354 | 0.350 | 0.482 |
| Доля коротких повторов | 0.654 | 0.667 | 0.644 |
| Доля доменных повторов | 0.154 | 0.157 | 0.281 |
Аналогичный анализ по выявлению различий в длине структурных повторов у эволюционно различных групп организмов, включая вирусы, проведен в [113] (табл. 2).
Таблица 2.
Различия длины структурных повторов у организмов разных эволюционных групп*
| Таксономическая группа | Число последова- тельностей |
Число последова- тельностей с повторами |
Белки, содержащие повторы, % | Средняя длина повтора |
|---|---|---|---|---|
| Всего | 554 241 | 28 003 | 5 | 15 |
| Археи | 19 525 | 351 | 2 | 11 |
| Бактерии | 333 691 | 6794 | 2 | 17 |
| Эукариоты | ||||
| Грибы | 33 613 | 3996 | 12 | 14 |
| Растения | 42 101 | 3601 | 12 | 13 |
| Позвоночные | 18 292 | 1461 | 8 | 14 |
| Беспозвоночные | 27 607 | 3372 | 12 | 19 |
| Вирусы | 16 852 | 889 | 5 | 14 |
* Данные из [113].
В целом утверждается [111], что чуть более 50% белков содержат хотя бы один структурный повтор, при этом, как правило, в белках эукариот число повторов больше, чем у архей и бактерий. Отмечена также положительная корреляция [111] между длиной аминокислотной последовательности и количеством структурных повторов в молекуле белка. Эукариотические белки содержат, как правило, более одного конкретного повтора. Это согласуется с данными об участии структурных повторов в регуляции экспрессии генов и передаче сигналов [114, 115], т.е. более сложным организмам для выполнения большего числа функций требуется больше повторов. Отметим также, что короткие структурные повторы встречаются чаще, чем длинные [111, 116, 117]. Согласно [113], структурные повторы у эукариот встречаются чаще по сравнению с бактериями и археями.
Проведен анализ появления 11780 мотивов из шести аминокислотных остатков, состоящих из двух случайно расположенных аминокислот (повторы вида ASSSSS, RGGRGG, PAPAPA), в 97 эукариотических и 25 бактериальных протеомах [118]. Сравнение частоты встречаемости таких мотивов в разных протеомах позволило оценить возможное филогенетическое родство различных таксономических царств. Так, отряд протистов Diplomonadida филогенетически более близок к бактериям, чем к эукариотам, а таксономические группы Stramenopiles и Amoebozoa ближе друг к другу, чем к другим царствам эукариот [118]. Кроме того, неупорядоченные гомоповторы (повторы, состоящие из аминокислотных остатков, способствующих неупорядоченности) чаще встречаются в эукариотических, чем в бактериальных протеомах [119, 120].
И хотя структурные повторы более представлены у эукариот, чем у прокариот [121], изучение специфичных повторов в белках позволяет выявить особенности их таксономического распределения. Так, масштабные исследования различных таксонов показали, что 98% протеомов бактерий и 78% протеомов архей содержат хотя бы один белок с ТПП [122]. Специфическая функция отдельного ТПП-содержащего белка, вероятно, определяет уровень вирулентности микроорганизма [8, 123]. Белки хотя бы с одним АнкП идентифицированы в 57% бактериальных и 9% архейных протеомах, при этом АрмП найден в 77% случаев [122]. Кроме того, как показано недавно, почти 85.6% видов бактерий (набор данных из [122]) содержат не менее двух повторов, а три повтора найдены более чем у половины (52%) из них [122].
ИДЕНТИФИКАЦИЯ ПОВТОРОВ В БЕЛКАХ: АЛГОРИТМЫ И БАЗЫ ДАННЫХ
Алгоритмы идентификации повторов в белках
Учитывая распространенность, уникальность функционирования и связь повторов с патогенезом различных заболеваний, распознавание структурных повторов в белках представляет актуальную задачу. Структурные повторы часто “не идеальны”, содержат ряд эволюционных мутаций (замены, вставки, делеции), поэтому некоторые из них трудны для идентификации. Разработаны специальные алгоритмы и программы поиска структурных повторов в белках. По алгоритму, лежащему в основе работы существующих программ поиска и идентификации повторов в белках, их можно разделить на пять основных групп (табл. 3).
Таблица 3.
Алгоритмы идентификации повторов в белках
| Алгоритм | Характерис- тика повтора |
Пример ресурса * |
|---|---|---|
| Фурье-анализ (Fourier transform analysis) | Длинные повторы без инделей | REPPER (https://toolkit.tuebingen.mpg.de/tools/repper) |
| Выравнивание последовательностей самих на себя (sequence self-alignment (SSA)) | Более 15 а.о., без инделей | RADAR (https://www.ebi.ac.uk/Tools/pfa/radar/) TRUST (https://www.ibi.vu.nl/programs/trustwww/) REPRO (https://www.ibi.vu.nl/programs/reprowww/) |
| Профиль скрытых марковских моделей (СММ) (Hidden Markov Models, HHMs)) | Длинные “неидеальные” повторы | PFAM (http://pfam.xfam.org/) SMART (http://smart.embl-heidelberg.de/) PROSITE (https://prosite.expasy.org/) TPRPRED (https://toolkit.tuebingen.mpg.de/tools/tprpred) |
| Сравнение профилей
СММ-СММ (HMM-HMM or profile-profile comparisons) |
Длинные “неидеальные” повторы | HHREPID (https://toolkit.tuebingen.mpg.de/) TRAL (https://www.vital-it.ch/software/tral) |
| Кластеризация коротких строк (Short string extension algorithms) | Менее 15–20 а.о., с инделями | T-REKS (https://bioinfo.crbm.cnrs.fr/index.php?route= tools&tool=3) XSTREAM (https://amnewmanlab.stanford.edu/xstream/) |
Использование Фурье-анализа для поиска структурной периодичности в белках не требует предварительных данных о наличии самих повторов. Такой способ подходит для идентификации длинных повторов без вставок (в фибриллярных белках, таких как коллаген или спиральные (двух- и более) структуры полипептидов, класс II) и инделей, и является ab initio методом [124, 125]. Методы кластеризации, включающие алгоритмы расширения коротких строк, позволяют идентифицировать тандемные повторы с инделями и дают хорошие результаты при идентификации коротких (менее 15–20 а.о.) повторов [126, 127]. Алгоритмы выравнивания последовательностей самих на себя эффективны для обнаружения длинных повторов (более ~ 10 а.о.), однако они часто не идентифицируют короткие повторы и не различают тандемные и чередующиеся повторы [128, 129]. Подходы, основанные на профилях скрытых марковских моделей (СММ и сравнение профилей СММ–СММ), идеальны для обнаружения длинных “неидеальных” повторов [130, 131]. Однако они требуют заранее сформированного выравнивания предполагаемых повторов и, следовательно, не подходят для автоматизированного крупномасштабного анализа ab initio. Кроме того, результативность этих методов зависит от качества выравнивания последовательностей, используемых в качестве основы и полноты СММ-профилей.
В настоящее время для поиска некоторых специфичных повторов применяют также комбинированные и специализированные методы идентификации периодичности в белках. Так, метод поиска внутренней симметрии в белках (detection of internal symmetry) реализован в программе CE-Symm (https://github.com/rcsb/symmetry/releaseshttps://github.com/rcsb/symmetry) для поиска ТИМ-бочонков и β-пропеллеров [132]. Программа AnkPred (http://bioinf.iiit.ac.in/AnkPred/) использует подход на основе применения графов, правила на основе вторичной структуры для идентификации АнкП в белках [133]. Программы ProSTRIP (http://cluster.physics.iisc.ernet.in/prostrip/https://bio.tools/prostrip ) [134] и Swelfe (https://bioserv.rpbs.univ-paris-diderot.fr/cgi-bin/ Swelfehttps://bioserv.rpbs.univ-paris-diderot.fr/cgi-bin/Swelfe ) [135] используют комбинированные алгоритмы специализированного конформационного алфавита (conformational alphabet analysis) и динамического машинного обучения для поиска повторов на всех уровнях белковой организации молекул. Метод TAPO (https://bioinfo.crbm.cnrs.fr/index.php?route=tools&tool=2) для поиска повторов в трехмерных структурах белков в дополнение к атомным координатам анализа использует периодичность в конформационном алфавите, распределении вторичной структуры, картах контактов аминокислотных остатков и расположении направления элементов вторичной структуры [136]. Сервер Reptile (http://reptile.unibe.ch) позволяет производить протеомно-вероятностный поиск идеальных повторов в белках паразитических и других организмов на основе сравнительной геномики [137]. Для поиска структурных повторов в белках также можно использовать метод разбиения структурного пространства с последующим анализом энергетического ландшафта [138]. Отметим, что на сегодняшний день наиболее правильным для выявления повторов в белках считается использование комбинации доступных программных продуктов.
Базы данных структурных повторов в белках
На основе протеомных данных можно проводить систематический масштабный анализ аминокислотных последовательностей белков с целью идентификации в них специфических мотивов и повторов и проведения дальнейшего анализа их изменчивости, структуры, функции и эволюционного развития. Необходимо с высокой точностью на уровне аминокислотной последовательности установить границы доменов для последующего множественного выравнивания. Данных о третичной структуре для визуального определения границ доменов и наличия повторов во многих белковых семействах не существует. Для решения таких задач с помощью различных биоинформатических подходов созданы специализированные базы данных структурных мотивов. Сведения о структурных повторах, их количестве и границах отдельных доменов можно найти также в базах данных белковых доменов и семейств, представляющих интегрированные и хорошо аннотированные ресурсы (табл. 4).
Таблица 4.
Базы данных белковых семейств, доменов и структурных повторов
| Тип структурного повтора | База данных | Основные характеристики** | Адрес сервера* |
|---|---|---|---|
| Белковые семейства и домены | Pfam [139] | 18259 белковых семейств | http://pfam.xfam.org/ |
| SMART [140] | 1300 белковых доменов | http://smart.embl-heidelberg.de/ | |
| InterPro [141] | Интеграция профилей и характеристик из баз данных CATH, CDD, HAMAP, MobiDB Lite, Panther, Pfam, PIRSF, PRINTS, Prosite, SFLD, SMART, SUPERFAMILY и TIGRfams | https://www.ebi.ac.uk/interpro/ | |
| PROSITE [142] | 311 моделей; 1296 профилей | https://prosite.expasy.org/ | |
| SUPFAM [143] | Аннотации 63 244 последовательностей из UniProt | http://supfam.org/ | |
| UniProt [144] | 563 972 аннотированных последовательностей; интеграция профилей и характеристик других баз данных | https://www.uniprot.org/ | |
| Структурные повторы | Protein Repeat DataBase (PRDB) [145] | 2 380 528 повторов в 836 670 белках | https://bioinfo.crbm.cnrs.fr/index.php? route=databases&tool=17 |
| RepeatsDB [146] | Аннотация и классификация 100 941 повторов в 14 072 отдельных белковых цепях, доступных в банке данных трехмерных структур белков PDB (https://www.rcsb.org/) [147] | https://repeatsdb.org/ | |
| LRRfinder [148] | Поиск и анализ ЛБП; более 4000 уникальных природных ЛБП | http://www.lrrfinder.com/lrrfinder.php | |
| HRaP [149] | Поиск гомоповторов и повторяющихся неструктурированных мотивов в эукариотических и бактериальных протеомах | http://bioinfo.protres.ru/hrap/ |
В основе каждой базы данных, содержащей информацию о структуре белков, их укладке, доменной организации, лежат определенные алгоритмы работы. Поскольку эти алгоритмы в ряде случаев различаются, а в базы данных, имеющих один и тот же алгоритм, обычно введены дополнительные условия или ограничения (например, набор правил ProRule в PROSITE; дополнительный анализ отсутствия схожих участков, вставок и делеций в Pfam), выходные данные для одного и того же объекта могут сильно различаться [113, 150].
Тем не менее, описанные базы данных содержат сведения и подходы к проведению биоинформатического анализа структурных повторов с целью оценки их распределения в протеомах, специфичности длин и аминокислотного состава, изучения функциональных и структурных данных. Так, например, база данных PRDB включает инструменты для оценки тенденции белков со структурными повторами быть неструктурированными и позволяет анализировать консервативные неповторяющиеся домены, примыкающие по аминокислотной последовательности к таким повторам [145]. В базе данных RepeatsDB приведена аннотированная иерархическая классификация, кластеризующая уровни по структурному сходству (класс > топология > укладка), дополненная классификацией род (clan) > семейство на основе гомологии последовательностей и присутствии специфичных мотивов (на основе данных базы Pfam) [146]. Некоторые репрезентативные базы данных содержат структурно-функциональную информацию о подробно охарактеризованных семействах белков со структурными повторами, например таких, как ЛБП [148]. База HRaP [149] позволяет идентифицировать гомоповторы и повторяющиеся неструктурированные мотивы в эукариотических и бактериальных протеомах.
Имеющиеся данные, несмотря на большое разнообразие подходов к идентификации структурных повторов в белках и доступных баз данных, охватывают только некоторые типы повторов и/или исследуются на отдельных геномах/протеомах. Таким образом, создание всеобъемлющего ресурса данных по-прежнему представляет собой перспективное направление для улучшения понимания значимости структурных повторов в белках.
СТРУКТУРНЫЕ ПОВТОРЫ И ПАТОГЕНЕЗ ЗАБОЛЕВАНИЙ
Многочисленные исследования доказывают существование связи между белками со структурными повторами и различными заболеваниями (табл. 5 ).
Таблица 5.
Заболевания, ассоциированные с некоторыми специфичными структурными повторами
| Структурный повтор | Характеристика нарушения | Ассоциированное заболевание | Ccылка |
|---|---|---|---|
| Полиглутаминовый и полиглициновый повторы | Полиморфизм длины повторов андрогенного рецептора | Рак предстательной железы, доброкачественная гиперплазия предстательной железы, ревматоидный артрит, болезнь Хантингтона | [151–153], [45, 52] |
| β-Пропеллеры | Гетерозиготные или гомозиготные мутации гена Х-хромосомы, кодирующего повтор WD40 | Нейродегенерация, связанная с β-пропеллерными белками (β-propeller protein-associated neurodegeneration, BPAN) рассеянность, миоклония, эпилеп-тические спазмы, эпилептические синдромы (синдром Веста и синдром Леннокса–Гасто) | [154–156] |
| ЛБП | Мутации гена, кодирующего белки, содержащие ЛБП | Миопия (близорукость), митохондриальная энцефаломиопатия, болезнь Крона | [157–159] |
| ТИМ-повторы | Мутации белка β-D-галактозидазы, содержащего ТИМ-повторы | GM1-ганглиозидозы, мукополисахаридоз типа IV или синдром Моркио | [160, 161] |
| АнкП | Низкая экспрессия мышечных белков, содержащих АнкП | Мышечные заболевания, рабдомиосаркома | [162–164] |
| АрмП | Мутации гена, кодирующего белок с АрмП | Опухоли кишечника, прогрессия | [165, 166] |
| β-Трилистники | Мутации генов, кодирующих белки, содержащие β-трилистник | Врожденные нарушения гликозилирования (опухолевый кальциноз) (FTC) и синдром гипер-фосфатемии–гиперостоза (HHS) | [167–170] |
| Спектриноподобные повторы | Низкий уровень дистрофина, содержащего спектриноподобные повторы | Атрофия мышц мышечная дистрофия Дюшенна | [171, 172] |
Так, например, нейродегенерация, связанная с β-пропеллерным белком BPAN (β-propeller protein-associated neurodegeneration), характеризуется ранним началом судорог, задержкой развития и умственной отсталостью [156]. Полиморфизм гена WDR45 идентифицирован как у мужчин, так и у женщин с этим заболеванием [155, 156]. Наследственные мутации в гене рецептора липопротеинов низкой плотности – белка, в структуре которого содержится по крайней мере один β-пропеллер, приводят к развитию семейной гиперхолестеринемии. При этом заболевании повышается концентрация липопротеинов низкой плотности и холестерина, что приводит к развитию коронарного атеросклероза и ишемической болезни сердца [156, 173]. При наследственных формах болезни Паркинсона часто обнаруживаются мутации в гене LRRK2, кодирующем протеинкиназу 2, которая содержит ЛБП, однако механизм возникновения заболевания у носителей мутаций в этом гене не установлен [174]. Одной из возможных причин болезни Крона – аутоиммунного воспалительного заболевания желудочно-кишечного тракта, считаются мутации в гене CARD15, влияющие на аминокислотные последовательности в ЛБП домена активации белка каспазы (caspase recruitment domain-containing protein 15) [159]. Болезнь Хантингтона – аутосомно-доминантное нейродегенеративное заболевание, обусловленное увеличением числа копий тринуклеотидных повторов CAG в гене белка хантингтина [175, 176]. Ряд заболеваний, включая рак предстательной железы, доброкачественную гиперплазию предстательной железы, мужское бесплодие и ревматоидный артрит также связывают с полиморфизмом длины глутаминовых и глициновых повторов в молекулах рецепторов андрогенов [153]. К наследственным заболеваниям из группы лизосомных болезней накопления (lysosomal storage diseases) относится мукополисахаридоз типа IV, или синдром Моркио, который характеризуется значительными деформациями скелета, особенно грудной клетки [177], а также GM1-ганглиозидозы. Эти заболевания связывают с мутациями в β-D-галактозидазе, содержащей в своей структуре ТИМ-повторы [160, 161]. GM1-ганглиозидозы обусловлены дефектом или недостатком β-галактозидазы, что приводят к нарушению метаболизма человека и накоплению субстратов (ганглиозида GM1, гликопротеинов и кератансульфата) главным образом в центральной и периферической нервной системе [178]. Функциональная недостаточность АнкП-содержащих белков в скелетных мышцах приводит к развитию различных заболеваний [162, 164]. Мутации гена APC (adenomatous polyposis coli), кодирующего большой белок с множеством функциональных АрмП, приводят к развитию опухолей кишечника [165]. Мутации генов, кодирующих GalNAc-трансферазу, содержащую в своей структуре повторы β-трилистника, приводят к врожденным дефектам гликозилирования, при которых нарушается синтез гликанов, их присоединение к гликопротеинам и гликолипидам, а также синтез гликозилфосфатидилинозита [167–170]. Низкая экспрессия белка дистрофина, содержащего 24 спектриноподобных повтора, приводит к развитию мышечной дистрофии Дюшенна [171, 172].
Отметим также, что многие белки, содержащие структурные повторы, характеризуются высокими уровнями внутренней неупорядоченности [12, 107, 179–181], что приводит к их структурно-функциональной гетерогенности. Подобная неупорядоченность считается одним из факторов вовлеченности таких белков в патогенез различных заболеваний человека [12, 49, 106]. Так, показано [182, 183], что доля неупорядоченных остатков увеличивается в белках, содержащих гомоповторы, состоящие из заряженных и полярных аминокислотных остатков, и уменьшается в повторах из гидрофобных остатков. Анализ 122 различных протеомов показал, что максимальная доля неупорядоченных остатков характерна для белков, содержащих гомоповторы лизина и аргинина, минимальная – для гомоповторов из валина и лейцина [182–184]. При этом гомоповторы, состоящие из аминокислотных остатков E, S, Q, G, L, P, D, A и H, ассоциированы с патогенезом ряда болезней человека. На основе проведенного анализа создан ресурс HRaDis (HomoRepeats and human Diseases http://bioinfo.protres.ru/hradis/) для изучения взаимосвязи гомоповторов с патологиями человека [183]. Приведенные примеры показывают, что различные структурные повторы, обнаруженные во множестве белков в различных количествах, играют важную роль в патогенезе ряда серьезных заболеваний.
БЕЛКИ С ПОВТОРАМИ В КАЧЕСТВЕ КАРКАСОВ ДЛЯ ПРОЕКТИРОВАНИЯ НОВЫХ МОЛЕКУЛ
Топологическая сложность и кооперативность, возникающая из особенностей типичных глобулярных белковых структур, часто представляет проблему при работе в области белкового дизайна. Белки с повторами, такими как АнкП, ТПП и ЛБП, в отличие от глобулярных белков имеют регулярные, линейно расположенные структурные блоки, что делает их идеальными объектами для анализа и дальнейшего модулирования свойств [185–189].
Так, предложен список уникальных повторов с указанием их основных структурных характеристик [190]. При этом одновременно с общими принципами проектирования белков на основе специфичных структурных повторов выделены определенные классы повторов, наиболее перспективных в качестве белковых каркасов, основ новых связывающих реагентов или биоматериалов для биомедицинских и нанотехнологических приложений [190].
На начальных этапах разработки новых искусственных белков на основе структурных повторов в каждом повторе идентифицируют остатки, определяющие специфичность укладки. Обычно с этой целью, используя базы данных белковых структур (PDB (https://www.rcsb.org/), PDBe-KB (https://www.ebi.ac.uk/pdbe/node/1)), проводят поиск консенсусной аминокислотной последовательности. При этом, чем более консервативен аминокислотный остаток в данном положении, тем более вероятно, что в конечной синтезируемой молекуле укладка сохранится [187, 191]. Предложено применять “модульный” подход при использовании белков с повторами в качестве каркасов для проектирования новых молекул [192]. В этом случае модули, состоящие из повторяющихся структурных единиц, выравниваются для идентификации специфических особенностей, которые впоследствии предполагается закладывать в шаблоны проектируемых белков. Реализация предлагаемого подхода показана на примере белков, содержащих ТПП и АрмП [192]. Разработан вычислительный подход [193], который объединяет информацию о специфичности аминокислотной последовательности белка и структурные особенности белков с повторами (на основе Rosetta de novo [194]), для создания “идеальных” искусственных стабильных модулей. Данный подход реализован для шести специфичных повторов (АнкП, АрмП, ТПП, HEAT, ЛБП и WD40).
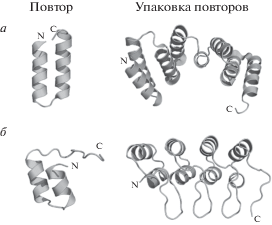
Примеры использования ТПП и АнкП в качестве белковых каркасов показаны на рис. 3.
Рис. 3.
Представление использования ТПП и АнкП в качестве белковых каркасов. а – Структура одного ТПП-повтора, и кристаллическая структура искусственного белка с четырьмя ТПП-повторами (PDB: 2AVP) [195]. б – Структура одного АнкП, и кристаллическая структура искусственного белка, содержащего четыре АнкП (PDB: 6MOG) [196].
В настоящее время на основе некоторых повторов разработано несколько библиотек белковых семейств, активно использующихся в дизайне новых лекарственных препаратов и бионанотехнологических продуктов (табл. 6).
Таблица 6.
Примеры использования некоторых повторов в качестве каркасов семейств искусственных белков
| Повтор | Семейство белков | Характеристика/применение |
|---|---|---|
| АнкП | DARPins | Повышенные эпитопные характеристики, микробициды против ВИЧ, диагностика рака |
| LoopDARPins | ||
| АрмП | dArmRP | Связывание пептидов модульным способом |
| β-Пропеллерные повторы | Pizza Tako Cake |
Строительные блоки для бионанотехнологичных продуктов |
| ЛБП | Хорошая растворимость при физиологических значениях pH, продуктивность, протеолитическая стабильность | |
| HEAT-повторы | αRep | Термостабильность, хорошая экспрессия |
| ТПП | CTPR | Термостабильность, противоопухолевые агенты, нановолокна, монослои и наноструктурированные тонкие пленки, подложка для синтеза наночастиц и нанокластеров |
| Двухспиральные повторы | TALE | Редактирование генома |
Белок DARPins (Designed Ankyrin Repeat Proteins) – один из примеров белка, сконструированного на основе АнкП-повтора (рис. 3б) [197, 198]. DARPins считается перспективным стабильным каркасом для конструирования белков, распознающих мишени со сродством и специфичностью (способностью связываться только с определенным антигеном) большей, чем у антител [197–200]. На основе белка DARPins разрабатывается также новое поколение белков LoopDARPins с расширенными эпитопными характеристиками [199]. DARPins также рассматривают как микробициды (антисептические лекарственные средства) против ВИЧ [201]. Кроме того, специфичный для опухолевых клеток антиген DARPins, слитый с белковым токсином, используют для диагностики онкологических заболеваний [202].
Аналогичные исследования проводятся также на белках, содержащих АрмП [203–206], и белках с ЛБП [207–209]. Так, белки dArmRP (Designed armadillo repeat proteins [206]) в настоящее время рассматриваются в качестве каркасов, связывающих пептиды модульным способом [210, 211].
На основе белков, содержащих ЛБП, предложена библиотека каркасов [209], с помощью которой можно осуществить дизайн молекул и решить проблемы их низкой растворимости при физиологических значениях pH, низкой продуктивности и протеолитической нестабильности [212]. На основе ТПП-повторов разработаны библиотеки консенсусных белков CTPR (consensus tetratricopeptide repeat proteins) для специфического связывания с белками, взаимодействующими с супрессорами различных опухолевых образований [213–215] (рис. 3а). Модульные свойства CTPR и особенности их самосборки позволяют разрабатывать различные надмолекулярные архитектуры, такие как нановолокна, упорядоченные монослои, наноструктурированные тонкие пленки [216–218]. Кроме того, особенности поверхностей CTPR позволяют использовать их в качестве подложки для синтеза металлических наночастиц и формирования металлических нанокластеров [216, 219, 220].
Белки, содержащие β-пропеллерные структуры, также часто применяют в белковой инженерии [156]. Так, глюкозодегидрогеназу (GDH), содержащую шестилопастную β-пропеллерную структуру, использовали для создания биодатчика глюкозы [221]. В результате получен химерный белок GDH, имеющий более высокую термостабильность, более высокую стабильность связывания кофакторов и повышенную специфичность к субстрату [221]. Эти свойства были приписаны усилению гидрофобных взаимодействий из-за мутаций на С-конце β-пропеллерного домена. β-Пропеллерный домен нейраминидазы вируса гриппа часто используется при разработке лекарственных средств [222–224]. Получены эффективные ингибиторы нейраминидазы гриппа, которые замедляют или останавливают прогрессирование гриппозной инфекции [224].
На основе β-пропеллерных повторов реконструированы идеально симметричные 6- и 8-лопастные белки – Pizza6 [225] и Tako8 [226] соответственно. Искусственные белки предлагается использовать в качестве строительных блоков в бионанотехнологических разработках [226, 227].
Обсуждается также перспектива использования белков с HEAT-повторами в качестве каркаса для искусственных белков [228–230]. Описано новое семейство искусственных белков αRep, созданных на основе термостабильных HEAT-подобных повторов, которые хорошо экспрессируются и обладают стабильностью [228–230].
Архитектура эффекторных белков, подобных активатору транскрипции (transcription activator-like effector, TALE), основана на канонических двухспиральных повторах, каждый из которых участвует в распознавании одной конкретной цепочки ДНК [231, 232]. Спирали, расположенные вокруг центральной оси, формируют общую сверхспиральную структуру белка для связывания дуплекса ДНК [232, 233]. Сконструированные таким образом молекулы, нацеленные на определенные последовательности ДНК, предлагается использовать в редактировании генома [234, 235].
Таким образом, белки со структурными повторами представляют интерес в качестве строительных блоков для инженерии новых лекарственных препаратов, белковых наноструктур и наноматериалов, а также для разработки новых биоматериалов и функциональных гибридных материалов.
ЭВОЛЮЦИОННОЕ РАЗВИТИЕ БЕЛКОВ С ПОВТОРАМИ
Возникновение структурных повторов в белках считается удачной эволюционной стратегией, так как регулярность вторичной структуры и разнообразие трехмерной сборки приводят к существованию молекул разного размера с множеством значимых функций [17].
Эволюционный анализ белков человека с повторами, проведенный на 61 эукариотическом протеоме [116], показал, что большинство повторов являются древними, при этом их число и порядок копирования сохраняются в процессе видоизменений организма. Эволюция повторов в белках (не H. sapiens) отражает процессы дублирования и появления делеций в ходе эволюции организмов. Сходные результаты получены и при изучении протеомов растений [236].
Анализ идентичности аминокислотных последовательностей в белках со структурными повторами различных видов показал, что число повторов увеличивается за счет одновременной дупликации сразу нескольких доменов [32, 237]. Кроме того, такие повторяющиеся единицы часто дублируются в центр региона с повторами. Такой механизм противоречит эволюционному развитию других белков, увеличение структуры которых происходит за счет добавления доменов к концевым участкам.
Предложена гипотеза, согласно которой повторы с высокой степенью гомологичности имеют общего предка, содержащего один повтор [17]. На ранних этапах эволюции эффективность функциональной активности может достигаться путем олигомеризации одиночных повторов [18]. Однако есть примеры белковых комплексов с повторами, которые образуются как из олигомеров с единичными повторами, так и из одной цепи с множественными повторами. При этом процесс кооперативного сворачивания белка с множественными повторами термодинамически более выгоден, чем сворачивание гомоолигомерного белка из мономеров [17].
Некоторые повторы в белках гипервариабельны, т.е. количество повторяющихся модулей внутри повторяющейся области сильно изменяется в пределах эволюционно короткого времени за счет вставок (расширения) или удаления (сокращения) структурных единиц. В некоторых белках эти изменения происходят так быстро, что их можно наблюдать не только у разных видов, но и в одной популяции при адаптивных [238] и нейтральных условиях [239].
Эволюционный анализ набора данных для 109 полностью секвенированных геномов многоклеточных животных [240] показал, что белки с независимо сворачивающимися повторами имеют меньше ограничений на изменение числа повторов по сравнению с белками, повторяющиеся единицы которых сворачиваются кооперативно. Это показывает, что вставки и делеции, влияющие на структуры повторяющихся единиц, обычно негативно влияют на взаимосвязь белок–функция. Кроме того, предполагается, что согласованная эволюция, т.е. эволюционное развитие, при котором паралогичные гены внутри одного вида более тесно связаны друг с другом, чем с членами одного и того же семейства генов близкородственных видов, является одной из положительных эволюционных стратегий для ряда белковых семейств с повторами [240]. Повторы в таких белковых семействах содержат больше инделей, что приводит к появлению дополнительных функций, связанных с совместной эволюцией паразита и хозяина, реакцией на стресс и развитием нервной системы [240]. Адаптация организмов при этом ускоряет изменение числа повторов, уменьшая вариабельность последовательностей между отдельными единицами. Если же вид начинает участвовать в эволюционной гонке с паразитом, то согласованная эволюция обеспечивает более высокую частоту повторений инделей с целью увеличения генетической изменчивости, направленной против паразитирующего организма [240].
Исследование и реконструкция изменений некоторых белковых семейств позволяет судить об особенностях эволюционного развития специфических повторов в белках. Так, например, предполагается, что специфичная архитектура ТИМ-повтора могла возникнуть на ранних этапах эволюции биосинтеза белка в качестве идеального каркаса для метаболического перехода от рибозимов, пептидов и геохимических катализаторов к современным белкам-ферментам [80]. Предполагаемая функция мышечного белка небулина, содержащего до 20 суперповторов (один такой суперповтор содержит семь повторяющихся доменов небулина), − регуляция длины миофибрилл [241]. Следовательно, можно предположить, что увеличение длины небулина за счет большего числа структурных повторов связано с увеличением длины миофибрилл в некоторых тканях [37]. Несмотря на широкий диапазон размеров пропеллерных повторов в белках, их лопасти часто имеют сходные последовательности, что указывает на общее происхождение [242]. Анализ работ [227, 242, 243] позволяет предположить, что большинство β-пропеллерных повторов возникло в результате дивергентной эволюции за счет разнообразия лопастей у более древних организмов.
Однако, несмотря на большое количество исследований, посвященных эволюции белков, содержащих специфические структурные повторы, однозначного понимания механизма формирования таких белков на сегодняшний день не существует.
ЗАКЛЮЧЕНИЕ
В нашем обзоре систематизированы данные по классификации белков со структурными повторами, их таксономическому распределению, а также особенностям структуры и функционирования. Объяснены механизмы возникновения структурных повторов. Рассмотрены подходы к идентификации повторов (биоинформатические алгоритмы поиска и специализированные базы данных), а также основные проблемы данной задачи и способы ее решения. Систематизированы данные о связи белков с повторами с патогенезом различных заболеваний, описано использование белков с повторами в качестве объектов исследований в области белковой инженерии и дизайна синтетических белков.
За последние два десятилетия идентифицировано множество глобулярных и неглобулярных белков, содержащих структурные повторы [17, 41]. Традиционные биоинформатические подходы, разработанные для анализа глобулярных доменов, не всегда могут применяться к белкам с повторами. И хотя наблюдается устойчивый прогресс в разработке новых инструментов прогнозирования и исследования как последовательностей, так и структур таких белков, лишь небольшая доля таких исследований посвящена сравнительным или комплексным интегрированным данным. Структурные повторы обнаруживаются в белках с разнообразными функциями. На сегодняшний день предпринято только несколько попыток классифицировать биологические роли таких белков с целью поиска взаимосвязи повтор–функция [1, 4, 244]. Отметим, что большинство исследований посвящено отдельно взятым протеомам или только нескольким специфическим повторам.
Таким образом, исследование структурных повторов в белках, понимание взаимосвязи между особенностями их последовательности и структурно-функциональными свойствами, выявление механизмов их эволюции представляется перспективным направлением современной молекулярной, структурной и эволюционной биологии. Полученные при этом результаты будут способствовать успешному конструированию новых молекул для применения в медицине, нанотехнологиях и создания новых биоматериалов.
Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований в рамках научного проекта № 20-14-50211.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Marcotte E.M., Pellegrini M., Yeates T.O., Eisenberg D. (1999) A census of protein repeats. J. Mol. Biol. 293, 151–160. https://doi.org/10.1006/jmbi.1999.3136
Pellegrini M., Renda M.E., Vecchio A. (2012) Ab initio detection of fuzzy amino acid tandem repeats in protein sequences. BMC Bioinformatics. 13, S8. https://doi.org/10.1186/1471-2105-13-S3-S8
Pellegrini M., Marcotte E.M., Yeates T.O. (1999) A fast algorithm for genome-wide analysis of proteins with repeated sequences. Proteins. 35, 440–446. PMID:10382671
Jorda J., Kajava A.V. (2010) Protein homorepeats sequences, structures, evolution, and functions. Adv. Protein Chem. Struct. Biol. 79, 59–88. https://doi.org/10.1016/S1876-1623(10)79002-7
Schmitz-Linneweber C., Small I. (2008) Pentatricopeptide repeat proteins: a socket set for organelle gene expression. Trends Plant Sci. 13, 663–670. https://doi.org/10.1016/j.tplants.2008.10.001
Renault L., Nassar N., Vetter I., Becker J., Klebe C., Roth M., Wittinghofer A. (1998) The 1.7 A crystal structure of the regulator of chromosome condensation (RCC1) reveals a seven-bladed propeller. Nature. 392, 97–101. https://doi.org/10.1038/32204
Varela M., Diaz-Rosales P., Pereiro P., Forn-Cuní G., Costa M.M., Dios S., Romero A., Figueras A., Novoa B. (2014) Interferon-induced genes of the expanded IFIT family show conserved antiviral activities in non-mammalian species. PLoS One. 9, e100015. https://doi.org/10.1371/journal.pone.0100015
Cerveny L., Straskova A., Dankova V., Hartlova A., Ceckova M., Staud F., Stulik J. (2013) Tetratricopeptide repeat motifs in the world of bacterial pathogens: role in virulence mechanisms. Infect. Immun. 81, 629–635. https://doi.org/10.1128/IAI.01035-12
Jacobsen S.E., Binkowski K.A., Olszewski N.E. (1996) SPINDLY, a tetratricopeptide repeat protein involved in gibberellin signal transduction in Arabidopsis. Proc. Natl. Acad. Sci. USA. 93, 9292–9296. https://doi.org/10.1073/pnas.93.17.9292
Baxa U., Cassese T., Kajava A.V., Steven A.C. (2006) Structure, function, and amyloidogenesis of fungal prions: filament polymorphism and prion variants. Adv. Protein Chem. 73, 125–180. https://doi.org/10.1016/S0065-3233(06)73005-4
Kajava A.V, Squire J.M., Parry D.A.D. (2006) Beta-structures in fibrous proteins. Adv. Protein Chem. 73, 1–15. https://doi.org/10.1016/S0065-3233(06)73001-7
Darling A.L., Uversky V.N. (2017) Intrinsic disorder in proteins with pathogenic repeat expansions. Molecules. 22, 2027. https://doi.org/10.3390/molecules22122027
Sikorski P., Atkins E. (2005) New model for crystalline polyglutamine assemblies and their connection with amyloid fibrils. Biomacromolecules. 6, 425–432. https://doi.org/10.1021/bm0494388
Den Dunnen W.F.A. (2017) Trinucleotide repeat disorders. Handb. Clin. Neurol. 145, 383–391. https://doi.org/10.1016/B978-0-12-802395-2.00027-4
Шилова О.Н., Деев С.М. (2019) Дарпины – перспективные адресные белки для тераностики. Acta Naturae. 11, 42–53.
Mittl P.R., Ernst P., Plückthun A. (2020) Chaperone-assisted structure elucidation with DARPins. Curr. Opin. Struct. Biol. 60, 93–100. https://doi.org/10.1016/j.sbi.2019.12.009
Andrade M.A., Perez-Iratxeta C., Ponting C.P. (2001) Protein repeats: structures, functions, and evolution. J. Struct. Biol. 134, 117–131. https://doi.org/10.1006/jsbi.2001.4392
Ponting C.P., Russell R.B. (2000) Identification of distant homologues of fibroblast growth factors suggests a common ancestor for all beta-trefoil proteins. J. Mol. Biol. 302, 1041–1047. https://doi.org/10.1006/jmbi.2000.4087
Apic G., Huber W., Teichmann S.A. (2003) Multi-domain protein families and domain pairs: comparison with known structures and a random model of domain recombination. J. Struct. Funct. Genomics. 4, 67–78. https://doi.org/10.1023/a:1026113408773
Ye Y., Godzik A. (2004) Comparative analysis of protein domain organization. Genome Res. 14, 343–353. https://doi.org/10.1101/gr.1610504
Moore A.D., Bornberg-Bauer E. (2012) The dynamics and evolutionary potential of domain loss and emergence. Mol. Biol. Evol. 29, 787–796. https://doi.org/10.1093/molbev/msr250
Kersting A.R., Bornberg-Bauer E., Moore A.D., Grath S. (2012) Dynamics and adaptive benefits of protein domain emergence and arrangements during plant genome evolution. Genome Biol. Evol. 4, 316–329. https://doi.org/10.1093/gbe/evs004
Kummerfeld S.K., Teichmann S.A. (2005) Relative rates of gene fusion and fission in multi-domain proteins. Trends Genet. 21, 25–30. https://doi.org/10.1016/j.tig.2004.11.007
Weiner J., Bornberg-Bauer E. (2006) Evolution of circular permutations in multidomain proteins. Mol. Biol. Evol. 23, 734–743. https://doi.org/10.1093/molbev/msj091
Weiner J., Beaussart F., Bornberg-Bauer E. (2006) Domain deletions and substitutions in the modular protein evolution. FEBS J. 273, 2037–2047. https://doi.org/10.1111/j.1742-4658.2006.05220.x
Wang M., Caetano-Anollés G. (2009) The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world. Structure. 17, 66–78. https://doi.org/10.1016/j.str.2008.11.008
Zmasek C.M., Godzik A. (2011) Strong functional patterns in the evolution of eukaryotic genomes revealed by the reconstruction of ancestral protein domain repertoires. Genome Biol. 12, R4. https://doi.org/10.1186/gb-2011-12-1-r4
Zmasek C.M., Godzik A. (2012) This Déjà vu feeling – analysis of multidomain protein evolution in eukaryotic genomes. PLoS Comput. Biol. 8, e1002701. https://doi.org/10.1371/journal.pcbi.1002701
Forslund S.K., Kaduk M., Sonnhammer E.L.L. (2019) Evolution of protein domain architectures. Methods Mol. Biol. 1910, 469–504. https://doi.org/10.1007/978-1-4939-9074-0_15
Moore A.D., Grath S., Schüler A., Huylmans A.K., Bornberg-Bauer E. (2013) Quantification and functional analysis of modular protein evolution in a dense phylogenetic tree. Biochim. Biophys. Acta. 1834, 898–907. https://doi.org/10.1016/j.bbapap.2013.01.007
Garrido-Ramos M.A. (2017) Satellite DNA: an evolving topic. Genes (Basel). 8, 230. https://doi.org/10.3390/genes8090230
Björklund A.K., Ekman D., Elofsson A. (2006) Expansion of protein domain repeats. PLoS Comput. Biol. 2, e114. https://doi.org/10.1371/journal.pcbi.0020114
Buard J., Vergnaud G. (1994) Complex recombination events at the hypermutable minisatellite CEB1 (D2S90). EMBO J. 13, 3203–3210. https://doi.org/10.1002/j.1460-2075.1994.tb06619.x
Djian P. (1998) Evolution of simple repeats in DNA and their relation to human disease. Cell. 94, 155–160. https://doi.org/10.1016/s0092-8674(00)81415-4
Ellegren H. (2000) Microsatellite mutations in the germline: implications for evolutionary inference. Trends Genet. 16, 551–558. https://doi.org/10.1016/s0168-9525(00)02139-9
Kruglyak S., Durrett R.T., Schug M.D., Aquadro C.F. (1998) Equilibrium distributions of microsatellite repeat length resulting from a balance between slippage events and point mutations. Proc. Natl. Acad. Sci. USA. 95, 10774–10778. https://doi.org/10.1073/pnas.95.18.10774
Björklund A.K., Light S., Sagit R., Elofsson A. (2010) Nebulin: a study of protein repeat evolution. J. Mol. Biol. 402, 38–51. https://doi.org/10.1016/j.jmb.2010.07.011
Deryusheva E.I., Machulin A. V., Selivanova O.M., Galzitskaya O.V. (2017) Taxonomic distribution, repeats, and functions of the S1 domain-containing proteins as members of the OB-fold family. Proteins. 85, 602–613. https://doi.org/10.1002/prot.25237
Machulin A. V, Deryusheva E.I., Selivanova O.M., Galzitskaya O.V. (2019) The number of domains in the ribosomal protein S1 as a hallmark of the phylogenetic grouping of bacteria. PLoS One. 14, e0221370. https://doi.org/10.1371/journal.pone.0221370
Sokol D., Benson G., Tojeira J. (2007) Tandem repeats over the edit distance. Bioinformatics. 23, e30-35. https://doi.org/10.1093/bioinformatics/btl309
Kajava A.V. (2012) Tandem repeats in proteins: from sequence to structure. J. Struct. Biol. 179, 279–288. https://doi.org/10.1016/j.jsb.2011.08.009
Perutz M.F. (1999) Glutamine repeats and neurodegenerative diseases: molecular aspects. Trends Biochem. Sci. 24, 58–63. https://doi.org/10.1016/s0968-0004(98)01350-4
Fan X. (2001) Oligomerization of polyalanine expanded PABPN1 facilitates nuclear protein aggregation that is associated with cell death. Hum. Mol. Genet. 10, 2341–2351. https://doi.org/10.1093/hmg/10.21.2341
Strømme P., Mangelsdorf M.E., Shaw M.A., Lower K.M., Lewis S.M.E., Bruyere H., Lütcherath V., Gedeon A.K., Wallace R.H., Scheffer I.E., Turner G., Partington M., Frints S.G.M., Fryns J.-P., Sutherland G.R., Mulley J.C., Gécz J. (2002) Mutations in the human ortholog of Aristaless cause X-linked mental retardation and epilepsy. Nat. Genet. 30, 441–445. https://doi.org/10.1038/ng862
Orr H.T., Zoghbi H.Y. (2007) Trinucleotide repeat disorders. Annu. Rev. Neurosci. 30, 575–621. https://doi.org/10.1146/annurev.neuro.29.051605.113042
Mosbach V., Poggi L., Richard G.-F. (2019) Trinucleotide repeat instability during double-strand break repair: from mechanisms to gene therapy. Curr. Genet. 65, 17–28. https://doi.org/10.1007/s00294-018-0865-1
Mosbach V., Poggi L., Viterbo D., Charpentier M., Richard G.-F. (2018) TALEN-induced double-strand break repair of CTG trinucleotide repeats. Cell Rep. 22, 2146–2159. https://doi.org/10.1016/j.celrep.2018.01.083
Faux N.G., Bottomley S.P., Lesk A.M., Irving J.A., Morrison J.R., de la Banda M.G., Whisstock J.C. (2005) Functional insights from the distribution and role of homopeptide repeat-containing proteins. Genome Res. 15, 537–551. https://doi.org/10.1101/gr.3096505
Jorda J., Xue B., Uversky V.N., Kajava A.V. (2010) Protein tandem repeats – the more perfect, the less structured. FEBS J. 277, 2673–2682. https://doi.org/10.1111/j.1742-464X.2010.07684.x
Healy E.F., Little C., King P.J. (2014) A model for small heat shock protein inhibition of polyglutamine aggregation. Cell Biochem. Biophys. 69, 275–281. https://doi.org/10.1007/s12013-013-9795-1
Gruber A., Hornburg D., Antonin M., Krahmer N., Collado J., Schaffer M., Zubaite G., Lüchtenborg C., Sachsenheimer T., Brügger B., Mann M., Baumeister W., Hartl F.U., Hipp M.S., Fernández-Busnadiego R. (2018) Molecular and structural architecture of polyQ aggregates in yeast. Proc. Natl. Acad. Sci. USA. 115, E3446–E3453. https://doi.org/10.1073/pnas.1717978115
Lyubchenko Y.L., Krasnoslobodtsev A. V, Luca S. (2012) Fibrillogenesis of huntingtin and other glutamine containing proteins. Subcell. Biochem. 65, 225–251. https://doi.org/10.1007/978-94-007-5416-4_10
Christie N.T.M., Lee A.L., Fay H.G., Gray A.A., Kikis E.A. (2014) Novel polyglutamine model uncouples proteotoxicity from aging. PLoS One. 9, e96835. https://doi.org/10.1371/journal.pone.0096835
Sorushanova A., Delgado L.M., Wu Z., Shologu N., Kshirsagar A., Raghunath R., Mullen A.M., Bayon Y., Pandit A., Raghunath M., Zeugolis D.I. (2019) The collagen suprafamily: from biosynthesis to advanced biomaterial development. Adv. Mater. 31, e1801651. https://doi.org/10.1002/adma.201801651
Lupas A.N., Bassler J., Dunin-Horkawicz S. (2017) The structure and topology of α-helical coiled coils. Subcell. Biochem. 82, 95–129. https://doi.org/10.1007/978-3-319-49674-0_4
Hennet T. (2019) Collagen glycosylation. Curr. Opin. Struct. Biol. 56, 131–138. https://doi.org/10.1016/j.sbi.2019.01.015
Berisio R., Vitagliano L., Mazzarella L., Zagari A. (2009) Crystal structure of the collagen triple helix model [(Pro-Pro-Gly)10]3. Protein Sci. 11, 262–270. https://doi.org/10.1110/ps.32602
Gordon M.K., Hahn R.A. (2010) Collagens. Cell Tissue Res. 339, 247–257. https://doi.org/10.1007/s00441-009-0844-4
Lupas A.N., Gruber M. (2005) The structure of alpha-helical coiled coils. Adv. Protein Chem. 70, 37–78. https://doi.org/10.1016/S0065-3233(05)70003-6
Gromiha M.M., Parry D.A. (2004) Characteristic features of amino acid residues in coiled-coil protein structures. Biophys. Chem. 111, 95–103. https://doi.org/10.1016/j.bpc.2004.05.001
Kobe B., Kajava A.V. (2000) When protein folding is simplified to protein coiling: the continuum of solenoid protein structures. Trends Biochem. Sci. 25, 509–515. https://doi.org/10.1016/s0968-0004(00)01667-4
Groves M.R., Barford D. (1999) Topological characteristics of helical repeat proteins. Curr. Opin. Struct. Biol. 9, 383–389. https://doi.org/10.1016/s0959-440x(99)80052-9
Kajava A.V., Steven A.C. (2006) Beta-rolls, beta-helices, and other beta-solenoid proteins. Adv. Protein Chem. 73, 55–96. https://doi.org/10.1016/S0065-3233(06)73003-0
Hennetin J., Jullian B., Steven A.C., Kajava A.V. (2006) Standard conformations of beta-arches in beta-solenoid proteins. J. Mol. Biol. 358, 1094–1105. https://doi.org/10.1016/j.jmb.2006.02.039
Kobe B., Deisenhofer J. (1996) Mechanism of ribonuclease inhibition by ribonuclease inhibitor protein based on the crystal structure of its complex with ribonuclease A. J. Mol. Biol. 264, 1028–1043. https://doi.org/10.1006/jmbi.1996.0694
Peters J.W., Stowell M.H., Rees D.C. (1996) A leucine-rich repeat variant with a novel repetitive protein structural motif. Nat. Struct. Biol. 3, 991–994. https://doi.org/10.1038/nsb1296-991
Huizinga E.G., Tsuji S., Romijn R.A.P., Schiphorst M.E., de Groot P.G., Sixma J.J., Gros P. (2002) Structures of glycoprotein Ibalpha and its complex with von Willebrand factor A1 domain. Science. 297, 1176–1179. https://doi.org/10.1126/science.107355
Liou Y.C., Tocilj A., Davies P.L., Jia Z. (2000) Mimicry of ice structure by surface hydroxyls and water of a beta-helix antifreeze protein. Nature. 406, 322–324. https://doi.org/10.1038/35018604
Fournier D., Palidwor G.A., Shcherbinin S., Szengel A., Schaefer M.H., Perez-Iratxeta C., Andrade-Navarro M.A. (2013) Functional and genomic analyses of alpha-solenoid proteins. PLoS One. 8, e79894. https://doi.org/10.1371/journal.pone.0079894
Cho U.S., Xu W. (2007) Crystal structure of a protein phosphatase 2A heterotrimeric holoenzyme. Nature. 445, 53–57. https://doi.org/10.1038/nature05351
Xing Y., Takemaru K.-I., Liu J., Berndt J.D., Zheng J.J., Moon R.T., Xu W. (2008) Crystal structure of a full-length beta-catenin. Structure. 16, 478–487. https://doi.org/10.1016/j.str.2007.12.021
Hast M.A., Beese L.S. (2008) Structure of protein geranylgeranyltransferase-I from the human pathogen Candida albicans complexed with a lipid substrate. J. Biol. Chem. 283, 31933–31940. https://doi.org/10.1074/jbc.M805330200
Mitraki A., Papanikolopoulou K., Van Raaij M.J. (2006) Natural triple beta-stranded fibrous folds. Adv. Protein Chem. 73, 97–124. https://doi.org/10.1016/S0065-3233(06)73004-2
Schrag J.D., Bergeron J.J.M., Li Y., Borisova S., Hahn M., Thomas D.Y., Cygler M. (2001) The structure of calnexin, an ER chaperone involved in quality control of protein folding. Mol. Cell. 8, 633–644. https://doi.org/10.1016/s1097-2765(01)00318-5
Ellgaard L., Riek R., Herrmann T., Güntert P., Braun D., Helenius A., Wüthrich K. (2001) NMR structure of the calreticulin P-domain. Proc. Natl. Acad. Sci. USA. 98, 3133–3138. https://doi.org/10.1073/pnas.051630098
Makabe K., Biancalana M., Yan S., Tereshko V., Gawlak G., Miller-Auer H., Meredith S.C., Koide S. (2008) High-resolution structure of a self-assembly-competent form of a hydrophobic peptide captured in a soluble beta-sheet scaffold. J. Mol. Biol. 378, 459–467. https://doi.org/10.1016/j.jmb.2008.02.051
Alvarez M., Zeelen J.P., Mainfroid V., Rentier-Delrue F., Martial J.A., Wyns L., Wierenga R.K., Maes D. (1998) Triose-phosphate isomerase (TIM) of the psychrophilic bacterium Vibrio marinus. Kinetic and structural properties. J. Biol. Chem. 273, 2199–2206. https://doi.org/10.1074/jbc.273.4.2199
Koebnik R., Locher K.P., Van Gelder P. (2000) Structure and function of bacterial outer membrane proteins: barrels in a nutshell. Mol. Microbiol. 37, 239–253. https://doi.org/10.1046/j.1365-2958.2000.01983.x
Wierenga R.K. (2001) The TIM-barrel fold: a versatile framework for efficient enzymes. FEBS Lett. 492, 193–198. https://doi.org/10.1016/s0014-5793(01)02236-0
Goldman A.D., Beatty J.T., Landweber L.F. (2016) The TIM barrel architecture facilitated the early evolution of protein-mediated metabolism. J. Mol. Evol. 82, 17–26. https://doi.org/10.1007/s00239-015-9722-8
Chen C.K.-M., Chan N.-L., Wang A.H.-J. (2011) The many blades of the β-propeller proteins: conserved but versatile. Trends Biochem. Sci. 36, 553–561. https://doi.org/10.1016/j.tibs.2011.07.004
Pitt J.J., Da Silva E., Gorman J.J. (2000) Determination of the disulfide bond arrangement of Newcastle Disease virus hemagglutinin neuraminidase. J. Biol. Chem. 275, 6469–6478. https://doi.org/10.1074/jbc.275.9.6469
Schapira M., Tyers M., Torrent M., Arrowsmith C.H. (2017) WD40 repeat domain proteins: a novel target class? Nat. Rev. Drug Discov. 16, 773–786. https://doi.org/10.1038/nrd.2017.179
Jain B.P., Pandey S. (2018) WD40 repeat proteins: signalling scaffold with diverse functions. Protein J. 37, 391–406. https://doi.org/10.1007/s10930-018-9785-7
Kumar V., Yadav A.N., Verma P., Sangwan P., Saxena A., Kumar K., Singh B. (2017) β-Propeller phytases: diversity, catalytic attributes, current developments and potential biotechnological applications. Int. J. Biol. Macromol. 98, 595–609. https://doi.org/10.1016/j.ijbiomac.2017.01.134
Murzin A.G., Lesk A.M., Chothia C. (1992) beta-Trefoil fold. Patterns of structure and sequence in the Kunitz inhibitors interleukins-1 beta and 1 alpha and fibroblast growth factors. J. Mol. Biol. 223, 531–543. https://doi.org/10.1016/0022-2836(92)90668-a
Gosavi S., Whitford P.C., Jennings P.A., Onuchic J.N. (2008) Extracting function from a beta-trefoil folding motif. Proc. Natl. Acad. Sci. USA. 105, 10384–19389. https://doi.org/10.1073/pnas.0801343105
Bendre A.D., Ramasamy S., Suresh C.G. (2018) Analysis of Kunitz inhibitors from plants for comprehensive structural and functional insights. Int. J. Biol. Macromol. 113, 933–943. https://doi.org/10.1016/j.ijbiomac.2018.02.148
Zhou J., Li C., Chen A., Zhu J., Zou M., Liao H., Yu Y. (2020) Structural and functional relationship of Cassia obtusifolia trypsin inhibitor to understand its digestive resistance against Pieris rapae. Int. J. Biol. Macromol. 148, 908–920. https://doi.org/10.1016/j.ijbiomac.2020.01.193
Giri Rao V.V.H., Gosavi S. (2015) Structural perturbations present in the folding cores of interleukin-33 and interleukin-1β correlate to differences in their function. J. Phys. Chem. B. 119, 11203–11214. https://doi.org/10.1021/acs.jpcb.5b03111
Hailey K.L., Capraro D.T., Barkho S., Jennings P.A. (2013) Allosteric switching of agonist/antagonist activity by a single point mutation in the interluekin-1 receptor antagonist, IL-1Ra. J. Mol. Biol. 425, 2382–2392. https://doi.org/10.1016/j.jmb.2013.03.016
Liao J.-H., Chien C.-T.H., Wu H.-Y., Huang K.-F., Wang I., Ho M.-R., Tu I.-F., Lee I.-M., Li W., Shih Y.-L., Wu C.-Y., Lukyanov P.A., Hsu S.-T.D., Wu S.-H. (2016) A multivalent marine lectin from Crenomytilus grayanus possesses anti-cancer activity through recognizing globotriose Gb3. J. Am. Chem. Soc. 138, 4787–4795. https://doi.org/10.1021/jacs.6b00111
Bensen D.C., Rodriguez S., Nix J., Cunningham M.L., Tari L.W. (2012) Structure of MurA (UDP-N-acetylglucosamine enolpyruvyl transferase) from Vibrio fischeri in complex with substrate UDP-N-acetylglucosamine and the drug fosfomycin. Acta Crystallogr. Sect. F. Struct. Biol. Cryst. Commun. 68, 382–385. https://doi.org/10.1107/S1744309112006720
Pautsch A., Schulz G.E. (1998) Structure of the outer membrane protein A transmembrane domain. Nat. Struct. Biol. 5, 1013–1017. https://doi.org/10.1038/2983
Kim K., Kim K.-P., Choi J., Lim J.-A., Lee J., Hwang S., Ryu S. (2010) Outer membrane proteins A (OmpA) and X (OmpX) are essential for basolateral invasion of Cronobacter sakazakii. Appl. Environ. Microbiol. 76, 5188–5198. https://doi.org/10.1128/AEM.02498-09
Balasubramaniam D., Arockiasamy A., Kumar P.D., Sharma A., Krishnaswamy S. (2012) Asymmetric pore occupancy in crystal structure of OmpF porin from Salmonella typhi. J. Struct. Biol. 178, 233–244. https://doi.org/10.1016/j.jsb.2012.04.005
Kim B.-H., Andersen C., Kreth J., Ulmke C., Sch-mid K., Benz R. (2002) Site-directed mutagenesis within the central constriction site of ScrY (sucroseporin): effect on ion transport and comparison of maltooligosaccharide binding to LamB of Escherichia coli. J. Membr. Biol. 187, 239–253. https://doi.org/10.1007/s00232-001-0167-1
Ferguson A.D., Deisenhofer J. (2002) TonB-dependent receptors – structural perspectives. Biochim. Biophys. Acta – Biomembr. 1565, 318–332. https://doi.org/10.1016/S0005-2736(02)00578-3
Oteiza P.I., Mackenzie G.G. (2005) Zinc, oxidant-triggered cell signaling, and human health. Mol. Aspects Med. 26, 245–255. https://doi.org/10.1016/j.mam.2005.07.012
García C.C., Damonte E.B. (2007) Zn finger containing proteins as targets for the control of viral infections. Infect. Disord. Drug Targets. 7, 204–212. https://doi.org/10.2174/187152607782110004
Kusunoki H., Minasov G., Macdonald R.I., Mondragón A. (2004) Independent movement, dimerization and stability of tandem repeats of chicken brain alpha-spectrin. J. Mol. Biol. 344, 495–511. https://doi.org/10.1016/j.jmb.2004.09.019
Tanaka Y., Sakamoto S., Kuroda M., Goda S., Gao Y.-G., Tsumoto K., Hiragi Y., Yao M., Watanabe N., Ohta T., Tanaka I. (2008) A helical string of alternately connected three-helix bundles for the cell wall-associated adhesion protein Ebh from Staphylococcus aureus. Structure. 16, 488–496. https://doi.org/10.1016/j.str.2007.12.018
Zheng N., Schulman B.A., Song L., Miller J.J., Jeffrey P.D., Wang P., Chu C., Koepp D.M., Elledge S.J., Pagano M., Conaway R.C., Conaway J.W., Harper J.W., Pavletich N.P. (2002) Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416, 703–709. https://doi.org/10.1038/416703a
Lukacik P., Roversi P., White J., Esser D., Smith G.P., Billington J., Williams P.A., Rudd P.M., Wormald M.R., Harvey D.J., Crispin M.D.M., Radcliffe C.M., Dwek R.A., Evans D.J., Morgan B.P., Smith R.A.G., Lea S.M. (2004) Complement regulation at the molecular level: the structure of decay-accelerating factor. Proc. Natl. Acad. Sci. USA. 101, 1279–1284. https://doi.org/10.1073/pnas.0307200101
Harrison O.J., Jin X., Hong S., Bahna F., Ahlsen G., Brasch J., Wu Y., Vendome J., Felsovalyi K., Hampton C.M., Troyanovsky R.B., Ben-Shaul A., Frank J., Troyanovsky S.M., Shapiro L., Honig B. (2011) The extracellular architecture of adherens junctions revealed by crystal structures of type I cadherins. Structure. 19, 244–256. https://doi.org/10.1016/j.str.2010.11.016
Van Bibber N.W., Haerle C., Khalife R., Xue B., Uversky V.N. (2020) Intrinsic disorder in tetratricopeptide repeat proteins. Int. J. Mol. Sci. 21, 3709. https://doi.org/10.3390/ijms21103709
Machulin A., Deryusheva E., Lobanov M., Galzits-kaya O. (2019) Repeats in S1 proteins: flexibility and tendency for intrinsic disorder. Int. J. Mol. Sci. 20, 2377. https://doi.org/10.3390/ijms20102377
Aachmann F.L., Svanem B.I.G., Güntert P., Petersen S.B., Valla S., Wimmer R. (2006) NMR structure of the R-module: a parallel beta-roll subunit from an Azotobacter vinelandii mannuronan C-5 epimerase. J. Biol. Chem. 281, 7350–7356. https://doi.org/10.1074/jbc.M510069200
Apic G., Gough J., Teichmann S.A. (2001) Domain combinations in archaeal, eubacterial and eukaryotic proteomes. J. Mol. Biol. 310, 311–325. https://doi.org/10.1006/jmbi.2001.4776
Ekman D., Björklund A.K., Frey-Skött J., Elofsson A. (2005) Multi-domain proteins in the three kingdoms of life: orphan domains and other unassigned regions. J. Mol. Biol. 348, 231–243. https://doi.org/10.1016/j.jmb.2005.02.007
Delucchi M., Schaper E., Sachenkova O., Elofsson A., Anisimova M. (2020) A new census of protein tandem repeats and their relationship with intrinsic disorder. Genes (Basel). 11, 407. https://doi.org/10.3390/genes11040407
Schaper E., Korsunsky A., Pečerska J., Messina A., Murri R., Stockinger H., Zoller S., Xenarios I., Anisimova M. (2015) TRAL: tandem repeat annotation library. Bioinformatics. 31, 3051–3053. https://doi.org/10.1093/bioinformatics/btv306
Tørresen O.K., Star B., Mier P., Andrade-Navarro M.A., Bateman A., Jarnot P., Gruca A., Grynberg M., Kajava A.V., Promponas V.J., Anisimova M., Jakobsen K.S., Linke D. (2019) Tandem repeats lead to sequence assembly errors and impose multi-level challenges for genome and protein databases. Nucl. Acids Res. 47, 10994–11006. https://doi.org/10.1093/nar/gkz841
Bilgin Sonay T., Koletou M., Wagner A. (2015) A survey of tandem repeat instabilities and associated gene expression changes in 35 colorectal cancers. BMC Genomics. 16, 702. https://doi.org/10.1186/s12864-015-1902-9
Theriot J.A. (2013) Why are bacteria different from eukaryotes? BMC Biol. 11, 119. https://doi.org/10.1186/1741-7007-11-119
Schaper E., Gascuel O., Anisimova M. (2014) Deep conservation of human protein tandem repeats within the eukaryotes. Mol. Biol. Evol. 31, 1132–1148. https://doi.org/10.1093/molbev/msu062
Schaper E., Kajava A.V., Hauser A., Anisimova M. (2012) Repeat or not repeat? – Statistical validation of tandem repeat prediction in genomic sequences. Nucl. Acids Res. 40, 10005–10017. https://doi.org/10.1093/nar/gks726
Galzitskaya O.V., Lobanov M.Y. (2015) Phyloproteomic analysis of 11780 six-residue-long motifs occurrences. Biomed. Res. Int. 2015, 208346. https://doi.org/10.1155/2015/208346
Lobanov M.Y., Galzitskaya O.V. (2011) Disordered patterns in clustered Protein Data Bank and in eukaryotic and bacterial proteomes. PLoS One. 6, e27142. https://doi.org/10.1371/journal.pone.0027142
Lobanov M.Y., Galzitskaya O.V. (2012) Occurrence of disordered patterns and homorepeats in eukaryotic and bacterial proteomes. Mol. Biosyst. 8, 327–337. https://doi.org/10.1039/c1mb05318c
Kajava A.V. (2001) Review: proteins with repeated sequence–structural prediction and modeling. J. Struct. Biol. 134, 132–144. https://doi.org/10.1006/jsbi.2000.4328
Jernigan K.K., Bordenstein S.R. (2015) Tandem-repeat protein domains across the tree of life. Peer. J. 3, e732. https://doi.org/10.7717/peerj.732
D’Andrea L.D., Regan L. (2003) TPR proteins: the versatile helix. Trends Biochem. Sci. 28, 655–662. https://doi.org/10.1016/j.tibs.2003.10.007
Gruber M., Söding J., Lupas A.N. (2005) REPPER-repeats and their periodicities in fibrous proteins. Nucl. Acids Res. 33, W239–W243. https://doi.org/10.1093/nar/gki405
Taylor W.R., Heringa J., Baud F., Flores T.P. (2002) A Fourier analysis of symmetry in protein structure. Protein Eng. 15, 79–89. https://doi.org/10.1093/protein/15.2.79
Newman A.M., Cooper J.B. (2007) XSTREAM: a practical algorithm for identification and architecture modeling of tandem repeats in protein sequences. BMC Bioinformatics. 8, 382. https://doi.org/10.1186/1471-2105-8-382
Jorda J., Kajava A.V. (2009) T-REKS: identification of tandem REpeats in sequences with a K-meanS based algorithm. Bioinformatics. 25, 2632–2638. https://doi.org/10.1093/bioinformatics/btp482
Heger A., Holm L. (2000) Rapid automatic detection and alignment of repeats in protein sequences. Proteins. 41, 224–237. https://doi.org/10.1002/1097-0134(20001101)41:2<224:: aid-prot70>3.0.co;2-z
Szklarczyk R., Heringa J. (2004) Tracking repeats using significance and transitivity. Bioinformatics. 20 (Suppl 1), i311–i317. https://doi.org/10.1093/bioinformatics/bth911
Bucher P., Karplus K., Moeri N., Hofmann K. (1996) A flexible motif search technique based on generalized profiles. Comput. Chem. 20, 3–23. https://doi.org/10.1016/s0097-8485(96)80003-9
Biegert A., Söding J. (2008) De novo identification of highly diverged protein repeats by probabilistic consistency. Bioinformatics. 24, 807–814. https://doi.org/10.1093/bioinformatics/btn039
Bliven S.E., Lafita A., Rose P.W., Capitani G., Prlić A., Bourne P.E. (2019) Analyzing the symmetrical arrangement of structural repeats in proteins with CE-Symm. PLoS Comput. Biol. 15, e1006842. https://doi.org/10.1371/journal.pcbi.1006842
Chakrabarty B., Parekh N. (2014) Identifying tandem ankyrin repeats in protein structures. BMC Bioinformatics. 15, 6599. https://doi.org/10.1186/s12859-014-0440-9
Sabarinathan R., Basu R., Sekar K. (2010) ProSTRIP: a method to find similar structural repeats in three-dimensional protein structures. Comput. Biol. Chem. 34, 126–130. https://doi.org/10.1016/j.compbiolchem.2010.03.006
Abraham A.-L., Rocha E.P.C., Pothier J. (2008) Swelfe: a detector of internal repeats in sequences and structures. Bioinformatics. 24, 1536–1537. https://doi.org/10.1093/bioinformatics/btn234
Do Viet P., Roche D.B., Kajava A.V. (2015) TAPO: A combined method for the identification of tandem repeats in protein structures. FEBS Lett. 589, 2611–2619. https://doi.org/10.1016/j.febslet.2015.08.025
Fankhauser N., Nguyen-Ha T.-M., Adler J., Mäser P. (2007) Surface antigens and potential virulence factors from parasites detected by comparative genomics of perfect amino acid repeats. Proteome Sci. 5, 20. https://doi.org/10.1186/1477-5956-5-20
Parra R.G., Espada R., Sánchez I.E., Sippl M.J., Ferreiro D.U. (2013) Detecting repetitions and periodicities in proteins by tiling the structural space. J. Phys. Chem. B. 117, 12887–12897. https://doi.org/10.1021/jp402105j
Mistry J., Chuguransky S., Williams L., Qureshi M., Salazar G.A., Sonnhammer E.L.L., Tosatto S.C.E., Paladin L., Raj S., Richardson L.J., Finn R.D., Bateman A. (2021) Pfam: the protein families database in 2021. Nucl. Acids Res. 49, D412–D419. https://doi.org/10.1093/nar/gkaa913
Letunic I., Khedkar S., Bork P. (2021) SMART: recent updates, new developments and status in 2020. Nucl. Acids Res. 49, D458–D460. https://doi.org/10.1093/nar/gkaa937
Blum M., Chang H.-Y., Chuguransky S., Grego T., Kandasaamy S., Mitchell A., Nuka G., Paysan-Lafosse T., Qureshi M., Raj S., Richardson L., Salazar G.A., Williams L., Bork P. Bridge A., Gough J., Haft D.H., Letunic I., Marchler-Bauer A., Mi H., Natale D.A., Necci M., Orengo C.A., Pandurangan A.P., Rivoire C., Sigrist C.J.A., Sillitoe I., Thanki N., Thomas P.D., Tosatto S.C.E., Wu C.H., Bateman A., Finn R.D. (2021) The InterPro protein families and domains database: 20 years on. Nucl. Acids Res. 49, D344–D354. https://doi.org/10.1093/nar/gkaa977
Sigrist C.J.A., De Castro E., Cerutti L., Cuche B.A., Hulo N., Bridge A., Bougueleret L., Xenarios I. (2013) New and continuing developments at PROSITE. Nucl. Acids Res. 41, D344–D347. https://doi.org/10.1093/nar/gks1067
Pandurangan A.P., Stahlhacke J., Oates M.E., Smithers B., Gough J. (2019) The SUPERFAMILY 2.0 database: a significant proteome update and a new webserver. Nucl. Acids Res. 47, D490–D494. https://doi.org/10.1093/nar/gky1130
UniProt Consortium (2021) UniProt: the universal protein knowledgebase in 2021. Nucl. Acids Res. 49, D480–D489. https://doi.org/10.1093/nar/gkaa1100
Jorda J., Baudrand T., Kajava A.V. (2012) PRDB: Protein Repeat DataBase. Proteomics. 12, 1333–1336. https://doi.org/10.1002/pmic.201100534
Paladin L., Bevilacqua M., Errigo S., Piovesan D., Mičetić I., Necci M., Monzon A.M., Fabre M.L., Lopez J.L., Nilsson J.F., Rios J., Menna P.L., Cabrera M., Buitron M.G., Kulik M.G., Fernandez-Alberti S., Fornasari M.S., Parisi G., Lagares A., Hirsh L., Andrade-Navarro M.A., Kajava A.V., Tosatto S.C.E. (2021) RepeatsDB in 2021: improved data and extended classification for protein tandem repeat structures. Nucl. Acids Res. 49, D452–D457. https://doi.org/10.1093/nar/gkaa1097
Burley S.K., Bhikadiya C., Bi C., Bittrich S., Chen L., Crichlow G. V, Christie C.H., Dalenberg K., Di Costanzo L., Duarte J.M., Dutta S., Feng Z., Ganesan S., Goodsell D.S., Ghosh S., Green R.K., Guranović V., Guzenko D., Hudson B.P., Lawson C.L., Liang Y., Lowe R., Namkoong H., Peisach E., Persikova I., Randle C., Rose A., Rose Y., Sali A., Segura J., Sekharan M., Shao C., Tao Y.P., Voigt M., Westbrook J.D., Young J.Y., Zardecki C., Zhuravleva M. (2021) RCSB Protein Data Bank: powerful new tools for exploring 3D structures of biological macromolecules for basic and applied research and education in fundamental biology, biomedicine, biotechnology, bioengineering and energy sciences. Nucl. Acids Res. 49, D437–D451. https://doi.org/10.1093/nar/gkaa1038
Offord V., Werling D. (2013) LRRfinder2.0: a webserver for the prediction of leucine-rich repeats. Innate Immun. 19, 398–402. https://doi.org/10.1177/1753425912465661
Lobanov M.Y., Sokolovskiy I.V, Galzitskaya O.V. (2014) HRaP: database of occurrence of HomoRepeats and patterns in proteomes. Nucl. Acids Res. 42, D273–D278. https://doi.org/10.1093/nar/gkt927
Дерюшева E.И., Мачулин А.В., Селиванова О.М., Сердюк И.Н. (2010) Семейство рибосомных белков S1 содержит уникальный консервативный домен. Молекуляр. биология. 44, 728–734.
Orafidiya F.A., McEwan I.J. (2015) Trinucleotide repeats and protein folding and disease: the perspective from studies with the androgen receptor. Futur. Sci. OA. 1, FSO47. https://doi.org/10.4155/fso.15.47
Walcott J.L., Merry D.E. (2002) Trinucleotide repeat disease. The androgen receptor in spinal and bulbar muscular atrophy. Vitam. Horm. 65, 127–147. https://doi.org/10.1016/s0083-6729(02)65062-9
McEwan I.J. (2001) Structural and functional alterations in the androgen receptor in spinal bulbar muscular atrophy. Biochem. Soc. Trans. 29, 222–227. https://doi.org/10.1042/0300-5127:0290222
Hor C.H.H., Tang B.L. (2019) Beta-propeller protein-associated neurodegeneration (BPAN) as a genetically simple model of multifaceted neuropathology resulting from defects in autophagy. Rev. Neurosci. 30, 261–277. https://doi.org/10.1515/revneuro-2018-0045
Mollereau B., Walter L. (2019) Is WDR45 the missing link for ER stress-induced autophagy in beta-propeller associated neurodegeneration?. Autophagy. 15, 2163–2164. https://doi.org/10.1080/15548627.2019.1668229
Pons T., Gómez R., Chinea G., Valencia A. (2003) Beta-propellers: associated functions and their role in human diseases. Curr. Med. Chem. 10, 505–524. https://doi.org/10.2174/0929867033368204
Matsushima N., Takatsuka S., Miyashita H., Kretsinger R.H. (2019) Leucine rich repeat proteins: sequences, mutations, structures and diseases. Protein Pept. Lett. 26, 108–131. https://doi.org/10.2174/0929866526666181208170027
Matsushima N., Tachi N., Kuroki Y., Enkhbayar P., Osaki M., Kamiya M., Kretsinger R.H. (2005) Structural analysis of leucine-rich-repeat variants in proteins associated with human diseases. Cell. Mol. Life Sci. 62, 2771–2791. https://doi.org/10.1007/s00018-005-5187-z
Hugot J.P., Chamaillard M., Zouali H., Lesage S., Cézard J.P., Belaiche J., Almer S., Tysk C., O’Morain C.A., Gassull M., Binder V., Finkel Y., Cortot A., Modigliani R., Laurent-Puig P., Gower-Rousseau C., Macry J., Colombel J.F., Sahbatou M., Thomas G. (2001) Association of NOD2 leucine-rich repeat variants with susceptibility to Crohn’s disease. Nature. 411, 599–603. https://doi.org/10.1038/35079107
Shimizu T. (2013) Structural basis for β-galactosidase associated with lysosomal disease. Yakugaku Zasshi. 133, 509–517. https://doi.org/10.1248/yakushi.13-00001-1
Ohto U., Usui K., Ochi T., Yuki K., Satow Y., Shimizu T. (2012) Crystal structure of human β-galactosidase: structural basis of Gm1 gangliosidosis and morquio B diseases. J. Biol. Chem. 287, 1801–1812. https://doi.org/10.1074/jbc.M111.293795
Ishiguro N., Motoi T., Osaki M., Araki N., Minamizaki T., Moriyama M., Ito H., Yoshida H. (2005) Immunohistochemical analysis of a muscle ankyrin-repeat protein, Arpp, in paraffin-embedded tumors: evaluation of Arpp as a tumor marker for rhabdomyosarcoma. Hum. Pathol. 36, 620–625. https://doi.org/10.1016/j.humpath.2005.04.014
Ishiguro N., Baba T., Ishida T., Takeuchi K., Osaki M., Araki N., Okada E., Takahashi S., Saito M., Watanabe M., Nakada C., Tsukamoto Y., Sato K., Ito K., Fukayama M., Mori S., Ito H., Moriyama M. (2002) Carp, a cardiac ankyrin-repeated protein, and its new homologue, Arpp, are differentially expressed in heart, skeletal muscle, and rhabdomyosarcomas. Am. J. Pathol. 160, 1767–1778. https://doi.org/10.1016/S0002-9440(10)61123-6
Tee J.-M., Peppelenbosch M.P. (2010) Anchoring skeletal muscle development and disease: the role of ankyrin repeat domain containing proteins in muscle physiology. Crit. Rev. Biochem. Mol. Biol. 45, 318–330. https://doi.org/10.3109/10409238.2010.488217
Crist R.C., Roth J.J., Baran A.A., McEntee B.J., Siracusa L.D., Buchberg A.M. (2010) The armadillo repeat domain of Apc suppresses intestinal tumorigenesis. Mamm. Genome. 21, 450–457. https://doi.org/10.1007/s00335-010-9288-0
Li D., Song H., Mei H., Fang E., Wang X., Yang F., Li H., Chen Y., Huang K., Zheng L., Tong Q. (2018) Armadillo repeat containing 12 promotes neuroblastoma progression through interaction with retinoblastoma binding protein 4. Nat. Commun. 9, 2829. https://doi.org/10.1038/s41467-018-05286-2
Topaz O., Shurman D.L., Bergman R., Indelman M., Ratajczak P., Mizrachi M., Khamaysi Z., Behar D., Petronius D., Friedman V., Zelikovic I., Raimer S., Metzker A., Richard G., Sprecher E. (2004) Mutations in GALNT3, encoding a protein involved in O‑linked glycosylation, cause familial tumoral calcinosis. Nat. Genet. 36, 579–581. https://doi.org/10.1038/ng1358
Duncan E.L., Danoy P., Kemp J.P., Leo P.J., McCloskey E., Nicholson G.C., Eastell R., Prince R.L., Eisman J.A., Jones G., Sambrook P.N., Reid I.R., Dennison E.M., Wark J., Richards J.B., Uitterlinden A.G., Spector T.D., Esapa C., Cox R.D., Brown S.D., Thakker R.V., Addison K.A., Bradbury L.A., Center J.R., Cooper C., Cremin C., Estrada K., Felsenberg D., Glüer C.C., Hadler J., Henry M.J., Hofman A., Kotowicz M.A., Makovey J., Nguyen S.C., Nguyen T.V., Pasco J.A., Pryce K., Reid D.M., Rivadeneira F., Roux C., Stefansson K., Styrkarsdottir U., Thorleifsson G., Tichawangana R., Evans D.M., Brown M.A. (2011) Genome-wide association study using extreme truncate selection identifies novel genes affecting bone mineral density and fracture risk. PLoS Genet. 7, e1001372. https://doi.org/10.1371/journal.pgen.1001372
Esapa C.T., Head R.A., Jeyabalan J., Evans H., Hough T.A., Cheeseman M.T., McNally E.G., Carr A.J., Thomas G.P., Brown M.A., Croucher P.I., Brown S.D.M., Cox R.D., Thakker R.V. (2012) A mouse with an N-Ethyl-N-nitrosourea (ENU) Induced Trp589Arg Galnt3 mutation represents a model for hyperphosphataemic familial tumoural calcinosis. PLoS One. 7, e43205. https://doi.org/10.1371/journal.pone.0043205
Lorenz V., Cejas R.B., Bennett E.P., Nores G.A., Irazoqui F.J. (2017) Functional control of polypeptide GalNAc-transferase 3 through an acetylation site in the C-terminal lectin domain. Biol. Chem. 398, 1237–1246. https://doi.org/10.1515/hsz-2017-0130
Percival J.M. (2018) Perspective: spectrin-like repeats in dystrophin have unique binding preferences for syntrophin adaptors that explain the mystery of how nNOSμ localizes to the sarcolemma. Front. Physiol. 9, 1369. https://doi.org/10.3389/fphys.2018.01369
Thomas G.D. (2013) Functional muscle ischemia in Duchenne and Becker muscular dystrophy. Front. Physiol. 4, 381. https://doi.org/10.3389/fphys.2013.00381
Dušková L., Nohelová L., Loja T., Fialová J., Zapletalová P., Réblová K., Tichý L., Freiberger T., Fajkusová L. (2020) Low density lipoprotein receptor variants in the beta-propeller subdomain and their functional impact. Front. Genet. 11, 691. https://doi.org/10.3389/fgene.2020.00691
Cogo S., Manzoni C., Lewis P.A., Greggio E. (2020) Leucine-rich repeat kinase 2 and lysosomal dyshomeostasis in Parkinson disease. J. Neurochem. 152, 273–283. https://doi.org/10.1111/jnc.14908
Lee J.-M., Correia K., Loupe J., Kim K.-H., Barker D., Hong E.P., Chao M.J., Long J.D., Lucente D., Vonsattel J.P.G., Pinto R.M., Abu Elneel K., Ramos E.M., Mysore J.S., Gillis T., Wheeler V.C., MacDonald M.E., Gusella J.F., McAllister B., Massey T., Medway C., Stone T.C., Hall L., Jones L., Holmans P., Kwak S., Ehrhardt A.G., Sampaio C., Ciosi M., Maxwell A., Chatzi A., Monckton D.G., Orth M., Landwehr-meyer G.B., Paulsen J.S., Dorsey E.R., Shoulson I., Myers R.H. (2019) CAG repeat not polyglutamine length determines timing of Huntington’s disease onset. Cell. 178, 887–900, e14. https://doi.org/10.1016/j.cell.2019.06.036
Bates G.P., Dorsey R., Gusella J.F., Hayden M.R., Kay C., Leavitt B.R., Nance M., Ross C.A., Scahill R.I., Wetzel R., Wild E.J., Tabrizi S.J. (2015) Huntington disease. Nat. Rev. Dis. Prim. 1, 15005. https://doi.org/10.1038/nrdp.2015.5
Prat C., Lemaire O., Bret J., Zabraniecki L., Fournié B. (2008) Morquio syndrome: diagnosis in an adult. Joint. Bone Spine. 75, 495–498. https://doi.org/10.1016/j.jbspin.2007.07.021
Bley A.E., Giannikopoulos O.A., Hayden D., Kubilus K., Tifft C.J., Eichler F.S. (2011) Natural history of infantile G(M2) gangliosidosis. Pediatrics. 128, e1233–1241. https://doi.org/10.1542/peds.2011-0078
Saravanan K.M., Ponnuraj K. (2019) Sequence and structural analysis of fibronectin-binding protein reveals importance of multiple intrinsic disordered tandem repeats. J. Mol. Recognit. 32, e2768. https://doi.org/10.1002/jmr.2768
Li X., Tao Y., Murphy J.W., Scherer A.N., Lam T.T., Marshall A.G., Koleske A.J., Boggon T.J. (2017) The repeat region of cortactin is intrinsically disordered in solution. Sci. Rep. 7, 16696. https://doi.org/10.1038/s41598-017-16959-1
Roberts S., Dzuricky M., Chilkoti A. (2015) Elastin-like polypeptides as models of intrinsically disordered proteins. FEBS Lett. 589, 2477–2486. https://doi.org/10.1016/j.febslet.2015.08.029
Lobanov M.Y., Galzitskaya O.V. (2015) How common is disorder? Occurrence of disordered residues in four domains of life. Int. J. Mol. Sci. 16, 19490–19507. https://doi.org/10.3390/ijms160819490
Lobanov M.Y., Klus P., Sokolovsky I.V., Tartaglia G.G., Galzitskaya O.V. (2016) Non-random distribution of homo-repeats: links with biological functions and human diseases. Sci. Rep. 6, 26941. https://doi.org/10.1038/srep26941
Lobanov M.Y., Furletova E.I., Bogatyreva N.S., Roytberg M.A., Galzitskaya O.V (2010) Library of disordered patterns in 3D protein structures. PLoS Comput. Biol. 6, e1000958. https://doi.org/10.1371/journal.pcbi.1000958
Forrer P., Binz H.K., Stumpp M.T., Plückthun A. (2004) Consensus design of repeat proteins. ChemBioChem. 5, 183–189. https://doi.org/10.1002/cbic.200300762
Forrer P., Stumpp M.T., Binz H.K., Plückthun A. (2003) A novel strategy to design binding molecules harnessing the modular nature of repeat proteins. FEBS Lett. 539, 2–6. https://doi.org/10.1016/s0014-5793(03)00177-7
Main E.R.G., Jackson S.E., Regan L. (2003) The folding and design of repeat proteins: reaching a consensus. Curr. Opin. Struct. Biol. 13, 482–489. https://doi.org/10.1016/s0959-440x(03)00105-2
Main E.R.G., Lowe A.R., Mochrie S.G.J., Jackson S.E., Regan L. (2005) A recurring theme in protein engineering: the design, stability and folding of repeat proteins. Curr. Opin. Struct. Biol. 15, 464–471. https://doi.org/10.1016/j.sbi.2005.07.003
Javadi Y., Itzhaki L.S. (2013) Tandem-repeat proteins: regularity plus modularity equals design-ability. Curr. Opin. Struct. Biol. 23, 622–631. https://doi.org/10.1016/j.sbi.2013.06.011
Stumpp M.T., Forrer P., Binz H.K., Pluckthun A. (2015) Repeat protein from collection of repeat proteins comprising repeat modules. US Patent 9,006,389. https://patents.google.com/patent/US9006389B2/en
Glasgow A.A., Huang Y.-M., Mandell D.J., Thompson M., Ritterson R., Loshbaugh A.L., Pellegrino J., Krivacic C., Pache R.A., Barlow K.A., Ollikainen N., Jeon D., Kelly M.J.S., Fraser J.S., Kortemme T. (2019) Computational design of a modular protein sense-response system. Science. 366, 1024–1028. https://doi.org/10.1126/science.aax8780
Sawyer N., Chen J., Regan L. (2013) All repeats are not equal: a module-based approach to guide repeat protein design. J. Mol. Biol. 425, 1826–1838. https://doi.org/10.1016/j.jmb.2013.02.013
Parmeggiani F., Huang P.-S., Vorobiev S., Xiao R., Park K., Caprari S., Su M., Seetharaman J., Mao L., Janjua H., Montelione G.T., Hunt J., Baker D. (2015) A general computational approach for repeat protein design. J. Mol. Biol. 427, 563–575. https://doi.org/10.1016/j.jmb.2014.11.005
Leaver-Fay A., Tyka M., Lewis S.M., Lange O.F., Thompson J., Jacak R., Kaufman K., Renfrew P.D., Smith C.A., Sheffler W., Davis I.W., Cooper S., Treuille A., Mandell D.J., Richter F., Ban Y.E., Fleishman S.J., Corn J.E., Kim D.E., Lyskov S., Berrondo M., Mentzer S., Popović Z., Havranek J.J., Karanicolas J., Das R., Meiler J., Kortemme T., Gray J.J., Kuhlman B., Baker D., Bradley P. (2011) ROSETTA3: an object-oriented software suite for the simulation and design of macromolecules. Methods Enzymol. 487, 545–574. https://doi.org/10.1016/B978-0-12-381270-4.00019-6
Kajander T., Cortajarena A.L., Mochrie S., Regan L. (2007) Structure and stability of designed TPR protein superhelices: unusual crystal packing and implications for natural TPR proteins. Acta Crystallogr. Sect. D. Biol. Crystallogr. 63, 800–811. https://doi.org/10.1107/S0907444907024353
Mohan K., Ueda G., Kim A.R., Jude K.M., Fallas J.A., Guo Y., Hafer M., Miao Y., Saxton R.A., Piehler J., Sankaran V.G., Baker D., Garcia K.C. (2019) Topological control of cytokine receptor signaling induces differential effects in hematopoiesis. Science. 364, eaav7532. https://doi.org/10.1126/science.aav7532
Plückthun A. (2015) Designed ankyrin repeat proteins (DARPins): binding proteins for research, diagnostics, and therapy. Annu. Rev. Pharmacol. Toxicol. 55, 489–511. https://doi.org/10.1146/annurev-pharmtox-010611-134654
Boersma Y.L. (2018) Advances in the application of designed ankyrin repeat proteins (DARPins) as research tools and protein therapeutics. Methods Mol. Biol. 1798, 307–327. https://doi.org/10.1007/978-1-4939-7893-9_23
Schilling J., Schöppe J., Plückthun A. (2014) From DARPins to LoopDARPins: novel LoopDARPin design allows the selection of low picomolar binders in a single round of ribosome display. J. Mol. Biol. 426, 691–721. https://doi.org/10.1016/j.jmb.2013.10.026
Stumpp M.T., Amstutz P. (2007) DARPins: a true alternative to antibodies. Curr. Opin. Drug Discov. Dev. 10, 153–159. PMID:17436550
Schweizer A., Rusert P., Berlinger L., Ruprecht C.R., Mann A., Corthésy S., Turville S.G., Aravantinou M., Fischer M., Robbiani M., Amstutz P., Trkola A. (2008) CD4-specific designed ankyrin repeat proteins are novel potent HIV entry inhibitors with unique characteristics. PLoS Pathog. 4, e1000109. https://doi.org/10.1371/journal.ppat.1000109
Zahnd C., Kawe M., Stumpp M.T., de Pasquale C., Tamaskovic R., Nagy-Davidescu G., Dreier B., Schibli R., Binz H.K., Waibel R., Plückthun A. (2010) Efficient tumor targeting with high-affinity designed ankyrin repeat proteins: effects of affinity and molecular size. Cancer Res. 70, 1595–1605. https://doi.org/10.1158/0008-5472.CAN-09-2724
Reichen C., Madhurantakam C., Plückthun A., Mittl P.R.E. (2014) Crystal structures of designed armadillo repeat proteins: implications of construct design and crystallization conditions on overall structure. Protein Sci. 23, 1572–1583. https://doi.org/10.1002/pro.2535
Madhurantakam C., Varadamsetty G., Grütter M.G., Plückthun A., Mittl P.R.E. (2012) Structure-based optimization of designed Armadillo-repeat proteins. Protein Sci. 21, 1015–1028. https://doi.org/10.1002/pro.2085
Reichen C., Madhurantakam C., Hansen S., Grütter M.G., Plückthun A., Mittl P.R.E. (2016) Structures of designed armadillo-repeat proteins show propagation of inter-repeat interface effects. Acta Crystallogr. Sect. D. Struct. Biol. 72, 168–175. https://doi.org/10.1107/S2059798315023116
Ernst P., Honegger A., van der Valk F., Ewald C., Mittl P.R.E., Plückthun A. (2019) Rigid fusions of designed helical repeat binding proteins efficiently protect a binding surface from crystal contacts. Sci. Rep. 9, 16162. https://doi.org/10.1038/s41598-019-52121-9
Park K., Shen B.W., Parmeggiani F., Huang P.-S., Stoddard B.L., Baker D. (2015) Control of repeat-protein curvature by computational protein design. Nat. Struct. Mol. Biol. 22, 167–174. https://doi.org/10.1038/nsmb.2938
Rämisch S., Weininger U., Martinsson J., Akke M., André I. (2014) Computational design of a leucine-rich repeat protein with a predefined geometry. Proc. Natl. Acad. Sci. USA. 111, 17875–17880. https://doi.org/10.1073/pnas.1413638111
Stumpp M.T., Forrer P., Binz H.K., Plückthun A. (2003) Designing repeat proteins: modular leucine-rich repeat protein libraries based on the mammalian ribonuclease inhibitor family. J. Mol. Biol. 332, 471–487. https://doi.org/10.1016/s0022-2836(03)00897-0
Ernst P., Plückthun A. (2017) Advances in the design and engineering of peptide-binding repeat proteins. Biol. Chem. 398, 23–29. https://doi.org/10.1515/hsz-2016-0233
Reichen C., Hansen S., Plückthun A. (2014) Modular peptide binding: from a comparison of natural binders to designed armadillo repeat proteins. J. Struct. Biol. 185, 147–162. https://doi.org/10.1016/j.jsb.2013.07.012
Schlehuber S., Skerra A. (2002) Tuning ligand affinity, specificity, and folding stability of an engineered lipocalin variant – a so-called “anticalin” – using a molecular random approach. Biophys. Chem. 96, 213–228. https://doi.org/10.1016/s0301-4622(02)00026-1
Horibe T., Kohno M., Haramoto M., Ohara K., Kawakami K. (2011) Designed hybrid TPR peptide targeting Hsp90 as a novel anticancer agent. J. Transl. Med. 9, 8. https://doi.org/10.1186/1479-5876-9-8
Cortajarena A.L., Yi F., Regan L. (2008) Designed TPR modules as novel anticancer agents. ACS Chem. Biol. 3, 161–166. https://doi.org/10.1021/cb700260z
Horibe T., Torisawa A., Kohno M., Kawakami K. (2012) Molecular mechanism of cytotoxicity induced by Hsp90-targeted Antp-TPR hybrid peptide in glioblastoma cells. Mol. Cancer. 11, 59. https://doi.org/10.1186/1476-4598-11-59
Mejias S.H., Aires A., Couleaud P., Cortajarena A.L. (2016) Designed repeat proteins as building blocks for nanofabrication. Adv. Exp. Med. Biol. 940, 61–81. https://doi.org/10.1007/978-3-319-39196-0_4
Grove T.Z., Regan L., Cortajarena A.L. (2013) Nanostructured functional films from engineered repeat proteins. J. R. Soc. Interface. 10, 20130051. https://doi.org/10.1098/rsif.2013.0051
Carter N.A., Grove T.Z. (2015) Repeat-proteins films exhibit hierarchical anisotropic mechanical properties. Biomacromolecules. 16, 706–714. https://doi.org/10.1021/bm501578j
Mejías S.H., López-Andarias J., Sakurai T., Yoneda S., Erazo K.P., Seki S., Atienza C., Martín N., Cortaja-rena A.L. (2016) Repeat protein scaffolds: ordering photo- and electroactive molecules in solution and solid state. Chem. Sci. 7, 4842–4847. https://doi.org/10.1039/c6sc01306f
Couleaud P., Adan-Bermudez S., Aires A., Mejías S.H., Sot B., Somoza A., Cortajarena A.L. (2015) Designed modular proteins as scaffolds to stabilize fluorescent nanoclusters. Biomacromolecules. 16, 3836–3844. https://doi.org/10.1021/acs.biomac.5b01147
Masakari Y., Hara C., Araki Y., Gomi K., Ito K. (2020) Improvement in the thermal stability of Mucor prainii-derived FAD-dependent glucose dehydrogenase via protein chimerization. Enzyme Microb. Technol. 132, 109387. https://doi.org/10.1016/j.enzmictec.2019.109387
Crennell S.J., Garman E.F., Laver W.G., Vimr E.R., Taylor G.L. (1993) Crystal structure of a bacterial sialidase (from Salmonella typhimurium LT2) shows the same fold as an influenza virus neuraminidase. Proc. Natl. Acad. Sci. USA. 90, 9852–9856. https://doi.org/10.1073/pnas.90.21.9852
Glanz V.Y., Myasoedova V.A., Grechko A.V., Orekhov A.N. (2018) Inhibition of sialidase activity as a therapeutic approach. Drug Des. Devel. Ther. 12, 3431–3437. https://doi.org/10.2147/DDDT.S176220
Sacramento C.Q., Jordão A.K., Abrantes J.L., Alves C.M., Marttorelli A., Fintelman-Rodrigues N., de Freitas C.S., de Melo G.R., Cunha A.C., Ferreira V.F., Souza T.M.L. (2020) Neuraminidase from influenza A and B viruses is susceptible to the compound 4-(4-phenyl-1H-1,2,3-triazol-1-yl)-2,2,6,6-tetramethylpipe-ridine-1-oxyl. Curr. Top. Med. Chem. 20, 132–139. https://doi.org/10.2174/1568026620666191227142433
Voet A.R.D., Noguchi H., Addy C., Simoncini D., Terada D., Unzai S., Park S.-Y., Zhang K.Y.J., Tame J.R.H. (2014) Computational design of a self-assembling symmetrical β-propeller protein. Proc. Natl. Acad. Sci. USA. 111, 15102–15107. https://doi.org/10.1073/pnas.1412768111
Noguchi H., Addy C., Simoncini D., Wouters S., Mylemans B., Van Meervelt L., Schiex T., Zhang K.Y.J., Tame J.R.H., Voet A.R.D. (2019) Computational design of symmetrical eight-bladed β-propeller proteins. IUCrJ. 6, 46–55. https://doi.org/10.1107/S205225251801480X
Mylemans B., Laier I., Kamata K., Akashi S., Noguchi H., Tame J.R.H., Voet A.R.D. (2021) Structural plasticity of a designer protein sheds light on β-propeller protein evolution. FEBS J. 288, 530–545. https://doi.org/10.1111/febs.15347
Urvoas A., Guellouz A., Valerio-Lepiniec M., Gra-ille M., Durand D., Desravines D.C., van Tilbeurgh H., Desmadril M., Minard P. (2010) Design, production and molecular structure of a new family of artificial alpha-helicoidal repeat proteins (αRep) based on thermostable HEAT-like repeats. J. Mol. Biol. 404, 307–327. https://doi.org/10.1016/j.jmb.2010.09.048
Guellouz A., Valerio-Lepiniec M., Urvoas A., Chevrel A., Graille M., Fourati-Kammoun Z., Desmadril M., van Tilbeurgh H., Minard P. (2013) Selection of specific protein binders for pre-defined targets from an optimized library of artificial helicoidal repeat proteins (alphaRep). PLoS One. 8, e71512. https://doi.org/10.1371/journal.pone.0071512
Valerio-Lepiniec M., Urvoas A., Chevrel A., Guellouz A., Ferrandez Y., Mesneau A., de la Sierra-Gallay I.L., Aumont-Nicaise M., Desmadril M., van Tilbeurgh H., Minard P. (2015) The αRep artificial repeat protein scaffold: a new tool for crystallization and live cell applications. Biochem. Soc. Trans. 43, 819–824. https://doi.org/10.1042/BST20150075
Deng D., Yan C., Pan X., Mahfouz M., Wang J., Zhu J.-K., Shi Y., Yan N. (2012) Structural basis for sequence-specific recognition of DNA by TAL effectors. Science. 335, 720–723. https://doi.org/10.1126/science.1215670
Mak A.N.-S., Bradley P., Cernadas R.A., Bogda-nove A.J., Stoddard B.L. (2012) The crystal structure of TAL effector PthXo1 bound to its DNA target. Science. 335, 716–719. https://doi.org/10.1126/science.1216211
Flechsig H. (2014) TALEs from a spring–superelasticity of Tal effector protein structures. PLoS One. 9, e109919. https://doi.org/10.1371/journal.pone.0109919
Bogdanove A.J., Voytas D.F. (2011) TAL effectors: customizable proteins for DNA targeting. Science. 333, 1843–1846. https://doi.org/10.1126/science.1204094
Scholze H., Boch J. (2011) TAL effectors are remote controls for gene activation. Curr. Opin. Microbiol. 14, 47–53. https://doi.org/10.1016/j.mib.2010.12.001
Schaper E., Anisimova M. (2015) The evolution and function of protein tandem repeats in plants. New Phytol. 206, 397–410. https://doi.org/10.1111/nph.13184
Moore A.D., Björklund A.K., Ekman D., Bornberg-Bauer E., Elofsson A. (2008) Arrangements in the modular evolution of proteins. Trends Biochem. Sci. 33, 444–451. https://doi.org/10.1016/j.tibs.2008.05.008
Verstrepen K.J., Jansen A., Lewitter F., Fink G.R. (2005) Intragenic tandem repeats generate functional variability. Nat. Genet. 37, 986–990. https://doi.org/10.1038/ng1618
Chevanne D., Saupe S.J., Clavé C., Paoletti M. (2010) WD-repeat instability and diversification of the Podospora anserina hnwd non-self recognition gene family. BMC Evol. Biol. 10, 134. https://doi.org/10.1186/1471-2148-10-134
Schüler A., Bornberg-Bauer E. (2016) Evolution of protein domain repeats in metazoa. Mol. Biol. Evol. 33, 3170–3182. https://doi.org/10.1093/molbev/msw194
McElhinny A.S., Kazmierski S.T., Labeit S., Gregorio C.C. (2003) Nebulin: the nebulous, multifunctional giant of striated muscle. Trends Cardiovasc. Med. 13, 195–201. https://doi.org/10.1016/s1050-1738(03)00076-8
Chaudhuri I., Söding J., Lupas A.N. (2008) Evolution of the beta-propeller fold. Proteins. 71, 795–803. https://doi.org/10.1002/prot.21764
Kopec K.O., Lupas A.N. (2013) β-Propeller blades as ancestral peptides in protein evolution. PLoS One. 8, e77074. https://doi.org/10.1371/journal.pone.0077074
Tompa P. (2003) Intrinsically unstructured proteins evolve by repeat expansion. Bioessays. 25, 847–855. https://doi.org/10.1002/bies.10324
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология