Молекулярная биология, 2021, T. 55, № 5, стр. 772-795
Гуаниновые квадруплексы в метаболизме клеточного ядра
Е. В. Мариловцева a, b, *, В. М. Студитский a, c
a Биологический факультет Московского государственного университета им. М.В. Ломоносова
119234 Москва, Россия
b Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова
Российской академии наук
117997 Москва, Россия
c Cancer Epigenetics Program, Fox Chase Cancer Center
PA 19111 Philadelphia, USA
* E-mail: ekaterina.marilovtseva@hotmail.com
Поступила в редакцию 09.02.2021
После доработки 29.03.2021
Принята к публикации 31.03.2021
Аннотация
Жизнедеятельность клетки в значительной степени зависит от точной регуляции экспрессии генов. Одним из механизмов такой регуляции является формирование нуклеиновыми кислотами вторичных структур, среди которых особое место занимают гуаниновые квадруплексы (G-квадруплексы, или G4). G-квадруплексы – динамические структуры, стабильность которых определяется их размером, ионным составом и природой образующих их нуклеиновых кислот, и регулируется разнообразными белковыми факторами. Гуаниновые квадруплексы играют важную роль в регуляции многих процессов, происходящих на ДНК и РНК, – от поддержания гомеостаза теломер до определения сайта посадки рибосомы на мРНК. Благодаря этому G-квадруплексы считаются перспективной мишенью для противоопухолевой терапии, а их детальное изучение представляет важную задачу современной биологии. В обзоре рассмотрена структура и термодинамические свойства G-квадруплексов, их биологические функции, а также их взаимодействие с некоторым ядерными белками.
ВВЕДЕНИЕ
Дезоксирибонуклеиновая кислота (ДНК) – полимерная молекула спиральной структуры, состоящая из двух закрученных друг вокруг друга антипараллельных комплементарных полинуклеотидных цепей. Удержание цепей осуществляется за счет водородных связей, образуемых, в основном, комплементарными пуриновыми и пиримидиновыми азотистыми основаниями с формированием пар аденин–тимин и гуанин–цитозин [1]. Этот тип взаимодействия, названный уотсон–криковским, лежит в основе большинства конформаций ДНК, в частности, правозакрученных B-ДНК и A-ДНК [2–4] и левозакрученной Z-ДНК [5]. Кроме того, уотсон–криковские взаимодействия обеспечивают около 60% взаимодействий внутри вторичных структур РНК [6].
В то же время, азотистые основания могут вступать не только в уотсон-криковские, но и в другие взаимодействия, формируя ряд неканонических структур, в том числе гуаниновые (G) структуры, среди которых главное место занимают G-квадруплексы [7]. В настоящее время известно, что квадруплексы, способные формироваться как на ДНК, так и на РНК, выполняют в клетке ряд функций: поддерживают структуру теломер, вносят вклад в компартментализацию хроматина, координируют работу транскрипционных факторов и модификаторов ДНК и гистонов, а также принимают участие в регуляции трансляции. Таким образом, G-квадруплексы играют важную роль в поддержании гомеостаза, регуляции метаболизма и пролиферации клетки, что делает их привлекательными мишенями для противоопухолевых, противовирусных и антибактериальных препаратов и, в перспективе, медикаментов, нацеленных на лечение некоторых генетических заболеваний. Стоит отметить, что за последние десятилетия синтезировано множество лигандов, специфически связывающих гуаниновые квадруплексы и снижающих пролиферативную активность клеток [8]; некоторые лиганды уже проходят клинические испытания в качестве противоопухолевых средств [9]. Гуаниновые квадруплексы применяют также в клинической диагностике [10, 11]. Интересной представляется возможность использования гуаниновых структур на ДНК в молекулярной электронике и проектировании нанопроводов и наносенсоров. Показано, что гуаниновые квадруплексы, абсорбированные на слюдяном субстрате, переносят заряд на расстояния до нескольких сотен нанометров, причем регистрируемая сила тока составляет от десятков до сотен пикоампер [12]. Наконец, недавно обнаружили, что гуаниновые квадруплексы можно использовать в качестве компонента молекулярной системы выявления патогенной кишечной палочки Escherichia coli K88 в продуктах питания [13].
Учитывая многообразие вариантов применения гуаниновых квадруплексов, более детальное изучение особенностей их динамики и функций крайне важно не только для более полного понимания механизмов регуляции экспрессии генов и молекулярных основ патогенеза различных заболеваний, но и для разработки новых подходов к их лечению, а также для развития нанотехнологий.
В нашем обзоре рассмотрены особенности структуры G-квадруплексов, их разновидности, биологические функции и факторы, влияющие на стабильность этих структур in vitro, in cellulo и in vivo.
СТРУКТУРЫ ГУАНИНСОДЕРЖАЩИХ НУКЛЕИНОВЫХ КИСЛОТ
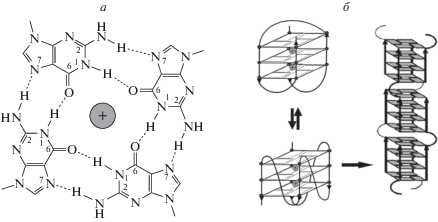
Нуклеотидные последовательности типа Gx1NL1Gx2NL2Gx3NL3Gx4 (где x > 3) (табл. 1) могут образовывать гуаниновые тетрады (гуаниновые квартеты) за счет Хугстиновского спаривания четырех входящих в их состав гуанинов [14–16]. Гуаниновая тетрада стабилизирована восемью водородными связями – четырьмя N2–H⋅⋅⋅N7 (2.87 Å) и четырьмя N1–H⋅⋅⋅O6 (2.84 Å) [17] (рис. 1а). Три и более G-тетрады могут собираться в “стопки”, образуя правозакрученные или, реже, левозакрученные спиральные структуры – G-квадруплексы, или G4, стабилизированные одновалентными катионами [18, 19] (рис. 1б). Расстояние между двумя соседними гуаниновыми тетрадами в G4 составляет (в зависимости от конформации входящих в их состав гуанинов) 3.39–3.55 Å, что близко по значению к расстоянию между двумя парами оснований в B-ДНК (3.4 Å) [20]. Молекулярно-динамические расчеты показывают, что катионы, стабилизирующие гуаниновые квадруплексы, участвуют в самых ранних этапах их формирования [21].
Таблица 1.
Некоторые последовательности, формирующие гуаниновый квадруплекс, в геноме человека
| Ген | Последовательности | Нуклеотидная последовательность |
|---|---|---|
| hTELO | (TTAGGG)3 | |
| CMYC | Pu27 | TTATGGGGAGGGTGGGGAGGGTGGGGAAGG |
| CKIT | ckit1 | CAGAGGGAGGGCGCTGGGAGGAGGGGCTG |
| ckit2 | CCCCGGGCGGGCGCGAGGGGAGGGGAGGC | |
| VEGF | CCCGGGGCGGGCCGGGGGCGGGGTCCCGGCGGGGCGGAG | |
| HIF-1α | GCGAGGGCGGGGGAGAGGGGAGGGGCGCG | |
| BCL2 | Pu39 | GTCGGGGCGAGGGCGGGGGAAGGAGGGCGCGGGCGGGGA |
| KRAS | GGGAGGGAGGGAAGGAGGGAGGGAGGGA |
Рис. 1.
Неканонические гуаниновые структуры ДНК. а – Строение гуаниновой тетрады: четыре гуанина образуют квартет, стабилизированный Хугстиновскими взаимодействиями. б – Три гуаниновых тетрады образуют стабилизированный ионами щелочного или щелочноземельного металла гуаниновый квадруплекс с антипараллельной (слева вверху) или параллельной (слева внизу) топологией, причем эти два вида топологии могут переходить друг в друга в зависимости от условий среды.
Несколько молекул ДНК, содержащих гуаниновые треки, могут образовывать так называемые гуаниновые нанопроволоки (G-wires) с параллельной укладкой (рис. 1б), что впервые показали в 1994 году для теломерной последовательности d(G4T2G4) тетрахимены [22]. Согласно данным атомно-силовой микроскопии и кристаллографического анализа, на всем своем протяжении G-нанопроволока имеет единообразную высоту и ширину, близкую к диаметру G-тетрады, и может изгибаться и перекручиваться [23]. Позднее с использованием атомно-силовой микроскопии высокого разрешения выявили необычные свойства нанопроволок, образованных последовательностью d(G4T2G4) в водном растворе. Оказалось, что данные структуры могут отличаться периодичностью расположения входящих в их состав квадруплексов (4.3, 2.2 и 0.9 нм) и способом их укладки – левозакрученной (4.3 и 2.2 нм), зигзагообразной (4.3 нм) и, в редких случаях, правозакрученной (0.9 нм). Также показано, что гуаниновые нанопроволоки обладают повышенной жесткостью по сравнению с ДНК-дуплексами [24].
Гуаниновые квадруплексы могут включать одну, две или четыре молекулы нуклеиновой кислоты. В соответствии с этим выделяют две основные группы квадруплексов – внутримолекулярные или межмолекулярные. Поскольку в последовательностях нуклеиновых кислот, образующих G-квадруплексы, гуаниновые треки чередуются с другими нуклеотидами, последние формируют петли – латеральные, диагональные и пропеллерные (двойные возвратные) – в соответствии с их расположением внутри квадруплекса. В основе разнообразия структур гуаниновых квадруплексов лежит их способность комбинировать петли разного типа [25].
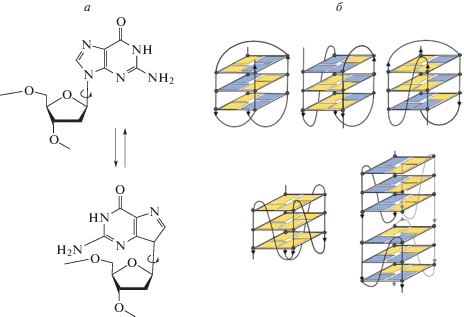
В зависимости от топологии – укладки молекул нуклеиновой кислоты – гуаниновые квадруплексы подразделяются на параллельные, антипараллельные и гибридные; топология же прямо зависит от гуанин-гликозидного торсионного угла, определяющего конформацию (син- или анти-) гуаниновых оснований, образующих G-квадруплекс (рис. 2а). Например, тетрамолекулярный квадруплекс может иметь только параллельную топологию, так как все составляющие его гуанины имеют анти-конформацию. В то же время, до 50% гуанинов, входящих в состав моно- и бимолекулярных G-квадруплексов, могут иметь син-конформацию, поэтому такие G4 могут быть как параллельными, так и антипараллельными (содержащими комбинацию гуанинов с син- и анти-конформацией) (рис. 2б) [17].
Рис. 2.
Зависимость особенностей топологии гуаниновых квадруплексов от конформации входящих в их состав гуаниновых оснований. а – Анти- и син-конформация гуанинового основания. б – Некоторые типы топологии гуаниновых квадруплексов, образованных комбинациями гуаниновых оснований, имеющих разную конформацию: внутримолекулярный G-квадруплекс с антипараллельной топологией “корзина” (basket) с диагональной петлей (верхний ряд, слева); внутримолекулярный G-квадруплекс с гибридной топологией (верхний ряд, центр); внутримолекулярный G-квадруплекс с топологией смешанного типа “3+1” (верхний ряд, справа); внутримолекулярный G-квадруплекс с параллельной топологией и пропеллерными петлями (нижний ряд, слева); межмолекулярный G-квадруплекс с параллельной топологией и пропеллерными петлями, образованный двумя цепями нуклеиновой кислоты (нижний ряд, справа). Желтые элементы соответствуют анти-конформации, голубые – син-конформации гуанинового основания.
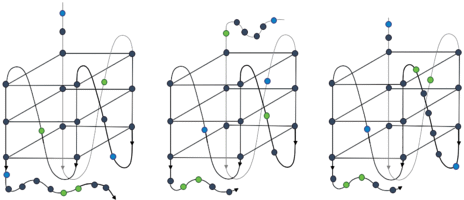
Богатые гуанином последовательности полиморфны и, как правило, могут образовывать несколько структурно различающихся квадруплексов. Например, теломерная ДНК человека, состоящая из пяти (TTAGGG) повторов, способна образовывать, как минимум, два разных внутримолекулярных G4 с разной длиной петель [26]. Стоит отметить, что длина петель является важной характеристикой G-квадруплекса, влияющей на его стабильность. Эксперименты по УФ-плавлению, проведенные для 21 библиотеки G-квадруплексов на ДНК, имеющих одинаковую параллельную топологию типа “двойная возвратная петля”, но петли разной длины (1–3 н.), показали, что увеличение общей длины петель на один нуклеотид вызывает падение температуры плавления (Tm) и изменение свободной энергии при температуре 37°C (ΔGVH) [27]. Наглядным примером формирования различных структур на одной нуклеотидной последовательности могут служить два гуаниновых квадруплекса, формируемых последовательностью Pu27 (5'-TGGGGAGGGTGGGGAGGGTGGGGAAGG-3') гена CMYC – myc-1245 и myc-2345 (рис. 3). Показано, что квадруплекс myc-1245 типа L161 (длины петель (L) составляют 1, 6 и 1 н. соответственно) имеет температуру плавления на 15°C ниже, чем myc-2345 типа L121 [28].
Рис. 3.
Некоторые варианты укладки последовательности Pu27 (5′-TGGGGAGGGTGGGGAGGGTGGGGAAGG-3′), расположенной в промоторе NHEIII1 гена CMYC, в гуаниновый квадруплекс. Слева направо: myc-1234 (TGGGGAGGGTGGGGAGGGTGGGGAAGG), myc-2345 (TGGGGAGGGTGGGGAGGGTGGGGAAGG), myc-1245 (TGGGGAGGGTGGGGAGGGTGGGGAAGG); кружками на схеме отмечены гуаниновые (серые), адениновые (зеленые) и тиминовые (синие) основания.
Изучено также влияние длины петель на термодинамические свойства и топологию гуаниновых квадруплексов, образованных РНК-последовательностями. С помощью библиотеки гуаниновых квадруплексов, формируемых на 18 РНК-последовательностях генома человека, проведен сравнительный анализ стабильности G4 на РНК с длинами петель от 1 до 15 н. в растворе с концентрацией KCl от 1 до 100 мМ. Оказалось, что, как и в случае с G4 на ДНК, стабильность гуаниновых квадруплексов РНК обратно пропорциональна длине петель [29].
ЯМР-анализ и спектрометрия кругового дихроизма (КД-спектрометрия) гуаниновых квадруплексов myc-2345 (GGG-T-GGG-GA-GGG-T-GGG) и myc-1234 (GGG-A-GGG-TG-GGG-A-GGG), имеющих идентичную топологию и длины петель, показали, что на стабильность и термодинамические характеристики G4 влияет не только длина петель, но и их нуклеотидный состав. Оказалось, что аденины внешних мононуклеотидных петель myc-1234 выталкиваются из бороздки и повышают степень обводненности G4, снижая его стабильность, в то время, как более компактные тимины тех же петель myc-2345, напротив, располагаются в плоскости бороздки, что делает этот вариант myc-G4 энергетически более выгодным и, следовательно, повышает его стабильность. Поэтому в 20 мМ растворе K+Tm myc-2345 на 14°C выше, чем у myc-1234 [30].
ТИПЫ ГУАНИНОВЫХ КВАДРУПЛЕКСОВ
В геноме человека методами секвенирования нового поколения детектируется более 700 000 гуаниновых квадруплексов [31]. In cellulo (и, по-видимому, in vivo) G-квадруплексы имеют преимущественно параллельную топологию, что отчасти обусловлено особенностями ионного состава клетки (139 мМ K+, 12 мМ Na+, 0.8 мМ Mg2+ и т.д) [32].
Гуаниновые квадруплексы на ДНК
Согласно результатам ChIP-секвенирования (проведенного с использованием клеточной линии HaCaTa), гуаниновые квадруплексы на ДНК в клетках человека преимущественно образуются в районах, свободных от нуклеосом [33]. Кроме того, ряд исследований показал, что в геноме человека мотивы, потенциально способные образовывать гуаниновые квадруплексы, располагаются в G/C-богатых, сверхчувствительных к нуклеазам участках промоторов >40% генов [34], большинство из которых являются протоонкогенами [35]. Кроме того, гуаниновые квадруплексы могут формироваться на ДНК в области теломерных повторов [36].
В лабораторных условиях in vitro G-квадруплексы на молекуле ДНК получают в основном путем термической денатурации богатой гуанином последовательности с ее последующим медленным охлаждением в присутствии соли калия или другого щелочного металла. В клетке G4 в ДНК формируются в ходе транскрипции богатых гуанином последовательностей (РНК-полимеразный комплекс разрывает водородные связи между цепями ДНК, позволяя “распаренным” гуанинам вступать во взаимодействие друг с другом и образовывать неканонические структуры). Эта гипотеза подтверждена данными транскрипции in vitro в комплексе с атомной силовой микроскопией [37] и in cellulo (в B-лимфоцитах мыши) – результатами полногеномного футпринтинга нуклеазой S1, узнающей одноцепочечные неканонические области ДНК [38].
Также показано, что G-квадруплексы могут формироваться в процессе репликации. Окрашивание синхронизированных культур раковых клеток человека антителами BG4 к гуаниновым квадруплексам показало, что в наименьшей степени эти структуры образуются в G0/G1-фазе, когда репликация не происходит. В контрольной точке G1/S количество G-квадруплексов возрастает примерно в 2.5 раза, а в S-фазе достигает максимальных значений, увеличиваясь примерно в 4.8 раза. Блокирование репликации афидиколином – ингибитором ДНК-полимеразы α – приводит к двукратному снижению количества сигналов от BG4, что подтверждает важность репликации для формирования гуаниновых квадруплексов in cellulo [39]. Формирование G4 на ДНК после прохождении ДНК-полимеразного комплекса обеспечивает дополнительный уровень защиты ДНК от повторного раунда репликации и способствует постепенному подавлению транскрипции и переходу клетки к делению.
Диаметр гуаниновых квадруплексов на ДНК составляет 24.3–28.7 Å. Благодаря этому, такие G4 обладают достаточно высоким дестабилизирующим потенциалом в отношении ДНК-дуплексов. Биофизический анализ G4-образующей последовательности регуляторной области гена BCL2 показывает, что формирование квадруплекса дестабилизирует участки дцДНК, расположенные в пределах 5 п.н. от него [40].
Гуаниновые квадруплексы на РНК
Как сказано выше, гуаниновые квадруплексы могут формироваться не только на ДНК, но и на РНК; структура G4 на РНК расшифрована с разрешением 0.61 Å [41]. В основе формирования гуаниновых квадруплексов на ДНК и РНК лежат одни и те же процессы, однако, в силу особенностей состава и структуры дезоксирибонуклеиновой и рибонуклеиновой кислот стабильность и структура этих двух типов квадруплексов неодинаковы.
Во-первых, согласно данным КД-спектрометрии и калийзависимого нативного электрофореза, полученных для РНК-последовательности (GGAA)10, для формирования G4 на РНК достаточно всего двух G-тетрад [42]. Во-вторых, благодаря тому, что тимин в РНК замещен урацилом, у которого отсутствует метильная группа в положении C5, степень обводненности петель в G4 на РНК ниже, чем в G4 на ДНК, поэтому первые отличаются более высокой Tm и большей стабильностью [43]. Кроме того, наличие в бороздках РНК упорядоченных 2′-гидроксогрупп остатков рибозы обеспечивает образование дополнительных водородных связей внутри квадруплексов. Наконец, 2′-OH-группы ограничивают торсионный угол остатка рибозы, что препятствует формированию син-конформации (и, соответственно, способствует формированию анти-конформации) азотистых оснований, образующих G4, поэтому РНК (кроме искусственно созданного РНК-аптамера Spinach) может формировать только параллельные G-квадруплексы в отличие от ДНК, способной образовывать G4 с различной топологией [44].
Межмолекулярные гуаниновые квадруплексы, образованные цепями ДНК и РНК
В процессе транскрипции синтезируемая РНК может комплементарно связывать ДНК-матрицу, образуя гибридную структуру ДНК:РНК – R‑петлю. Если обе нуклеиновые кислоты, вовлеченные в формирование R-петли, содержат по два близлежащих G-трека, то возможно формирование межмолекулярного гуанинового квадруплекса ДНК:РНК, поэтому соответствующие пары G-треков на ДНК называют мнимыми G4-формирующими последовательностями (PHQS). Биоинформатический анализ показал, что в геномах эукариотических организмов, начиная с амфибий, PHQS сконцентрированы в пределах 1000 н. ниже сайта начала транскрипции [45]. Интересно, что транскрибируемые области генома содержат больше PHQS, чем классических G4-формирующих последовательностей: >97% генов человека содержат PHQS и могут формировать G4 на гибридных молекулах ДНК:РНК ниже точки начала транскрипции в приблизительном соотношении 73 G4 на один ген [46]. Благодаря отрицательному суперскручиванию, создаваемому РНК-полимеразой II и частично распространяющемуся вверх по цепи ДНК, в присутствии РНК-транскриптов, несущих необходимое число G-треков, гуаниновые квадруплексы ДНК:РНК могут также образовываться выше точки начала транскрипции. Этот феномен был открыт в результате анализа ДНК, состоящей из T7-промотора, фланкированного парой идентичных олигонуклеотидов (G4TG4 или G4TG4TG4), с помощью in vitro транскрипции [47].
Кроме того, in cellulo в клетках HeLa показано формирование теломерных G-квадруплексов на молекулах ДНК:РНК с параллельной топологией, обладающих достаточно высокой стабильностью (Tm = 51.7°C) [48]; такие ДНК:РНК G4 повышают устойчивость теломер к экзонуклеазам [49].
ВЛИЯНИЕ ИОННОГО СОСТАВА НА ФОРМИРОВАНИЕ И СТАБИЛЬНОСТЬ ГУАНИНОВЫХ КВАДРУПЛЕКСОВ
В силу особенностей строения гуаниновых квадруплексов, их стабильность во многом определяется природой входящих в их состав одновалентных катионов и уменьшается в следующем порядке: K+$ \gg $ Rb+ ≥ Na+ > Cs+ ≥ Li+. Таким образом, наиболее выгодными для формирования гуаниновых квадруплексов являются ионы калия, что хорошо соотносится с их высокой (по сравнению с другими ионами) концентрацией в клетке. Разный эффект различных катионов на стабильность G4 может объясняться их диаметром и энергией обводнения [50]. Так, ион K+ имеет относительно большой радиус (1.52 Å) и потому располагается между двумя тетрадами, что снижает степень его обводненности и позволяет образовывать координационные связи со всеми восемью O6-атомами гуанинов обеих тетрад, в то время как ион Na+ в силу меньших размеров (1.16 Å) умещается внутри одной тетрады, образуя связи только с четырьмя O6 [51, 52]. Ионы лития имеют еще меньший радиус, чем ионы натрия, и сильно обводнены, в связи с чем гуаниновые квадруплексы на основе Li+ практически не образуются. В то же время, присутствие ионов лития влияет на общий выход гуаниновых квадруплексов, образующихся в растворе ионов K+ или Na+. В первом случае образуется больше квадруплексов, т.е. ионы Li+ потенцируют эффект ионов K+. Во втором случае наблюдается противоположный эффект [53].
Ионы двухвалентных металлов также могут участвовать в регуляции формирования и динамики неканонических гуаниновых структур. В частности, ионы металлов подгруппы IIa, как и одновалентные катионы, могут выступать в роли стабилизаторов гуаниновых тетрад, составляющих квадруплекс, взаимодействуя с O6-атомами гуанинов. При этом, как и в ситуации с одновалентными ионами, стабильность квадруплексов коррелирует с диаметром иона и падает в ряду Sr2+$ \gg $ Ba2+ > Ca2+ > Mg2+ [54, 55]. Интересно также отметить, что G-квадруплексы на ДНК, образованные при участии ионов Sr2+ в концентрациях, не превышающих 30 мМ, обладают гораздо большей стабильностью и прочностью, чем, например, G4(K+) [56]. Достаточно сильным стабилизирующим эффектом по отношению к гуаниновым квадруплексам обладают (даже в небольших концентрациях) ионы бария [57], которые также могут индуцировать формирование G4 из ДНК-декамеров, содержащих гуанин, что подтверждено с помощью кристаллографического анализа [58].
Некоторые двухвалентные катионы способны вызывать структурные переходы внутри гуаниновых квадруплексов. Например, ионы Sr2+ [59] и Mg2+ (в комплексе со спермидином) [22] способны индуцировать формирование G-нанопроволок на последовательности типа d((G4T2)3G4). Открыт также интересный эффект кальция. На примере гуанинового квадруплекса, сформированного на последовательности d(G4T4G4), показано, что ионы Ca2+ в концентрации 1 мМ вызывают дестабилизацию антипараллельных G4, а постепенное повышение концентрации кальция индуцирует изменение топологии – переход антипараллельных квадруплексов в параллельные с дальнейшим формированием G-нанопроволок [60, 61]. Аналогичные структурные переходы вызывают ионы никеля; при этом эффект Ni2+ можно отменить добавлением EDTA [62]. Однако поскольку ионы никеля способны образовывать координационные связи с N7-атомами гуанинов, их присутствие препятствует образованию водородных связей между гуанинами и ингибирует формирование G4 de novo [63].
В 2011 году, исследуя G-квадруплекс PS2.M, Liu и соавт. показали, что ионы свинца способны вытеснять K+ и Na+ из гуаниновых квадруплексов, не вызывая их структурных переходов. Каждый образовавшийся таким образом квадруплекс стабилизируется двумя ионами Pb2+, расположенными между парами соседних тетрад, что делает G4(Pb2+) компактным и очень стабильным – температура плавления такого квадруплекса достигает 72°C. Дальнейший анализ показал, что ионы свинца эффективнее замещают ионы натрия, чем ионы калия, что легко объясняется разницей в уровне стабильности G4(K+) и G4(Na+) [64]. Интересно также отметить влияние ртути на гуаниновые квадруплексы: ионы этого токсичного элемента с высокой аффинностью связывают тимин [65], образуют связи (T–Hg2+–T), препятствуют сгибанию ДНК и, следовательно, образованию G4. Кроме того, Hg2+, как и Ni2+, способны изменять топологию гуаниновых квадруплексов [66]. В то же время, согласно некоторым данным, ионы ртути могут оказывать стабилизирующий эффект на уже сформированные гуаниновые квадруплексы и триплексы [67, 68].
Многие двухвалентные и трехвалентные катионы дестабилизируют гуаниновые квадруплексы либо ингибируют их формирование. Это, в частности, касается ионов Al3+, Cd2+, Co2+, Cu2+, Fe2+ и Fe3+. Так, к примеру, ионы меди и кобальта, подобно ионам никеля и ртути, взаимодействуют с атомами N7 гуанинов, препятствуя образованию G-квадруплексов, хотя присутствие высоких концентраций K+ до определенной степени ослабляет этот эффект и делает возможным образование гуаниновых триплексов [63]. Кроме того, добавление ионов меди дестабилизирует уже сформированные гуаниновые квадруплексы, причем этот эффект нивелируется добавлением EDTA. Это наблюдение справедливо и в отношении других перечисленных двух- и трехвалентных катионов, дестабилизирующих G4, включая, как сказано выше, ионы Ni2+. Интересно отметить, что дестабилизирующий эффект ионов меди полностью ингибируется не только EDTA, но и ионами бария в молярном соотношении 1 : 10 (Ba2+ : Cu2+), так как в отличие от многих других двухвалентных катионов ионы меди не влияют на топологию G-квадруплексов [57].
Рассматривая влияние различных ионов на стабильность и топологию гуаниновых квадруплексов, стоит отметить, что G4 на РНК преимущественно схожи с G4 на ДНК. Тем не менее, на примере мРНК NRAS и теломерной РНК TERRA показано, что только ионы K+, Sr2+ и, до некоторой степени, Ba2+, способны эффективно стабилизировать РНК-квадруплексы; в то же время, ионы Na+, Ca2+ и Mg2+ не оказывают подобного эффекта [69]. Двухвалентные ионы тяжелых металлов (Pb2+, Cd2+ и Hg2+) связывают G4 на РНК и нарушают их структуру [70].
РЕГУЛЯТОРНЫЕ ФУНКЦИИ ГУАНИНОВЫХ КВАДРУПЛЕКСОВ НА ДНК
Подобно посттрансляционным модификациям гистонов [71, 72], гуаниновые квадруплексы, формирующиеся на ДНК, выполняют роль регуляторов репликации, транскрипции и рекомбинации, а образование G4 в молекулах РНК влияет на синтез белка. Поэтому, а также благодаря высокой частоте встречаемости GC-богатых последовательностей в геноме эукариот, гуаниновые квадруплексы имеют важное значение для поддержания нормального метаболизма клетки, а нарушение регуляции их образования может приводить к развитию различных заболеваний, нарушению эмбрионального развития или преждевременному старению.
В этом разделе обзора рассмотрены биологические функции гуаниновых квадруплексов и их роль в регуляции экспрессии генов.
Регуляция репликации
Известно, что повышение концентрации ионов K+, стимулирующее формирование G-квадруплексов, ингибирует репликацию ДНК in vitro [73], указывая на антагонизм между этими двумя процессами. Показано также, что и сами гуаниновые квадруплексы оказывают влияние на активность репликации. Так, используя FISH-гибридизацию и иммунофлуоресцентное окрашивание антителами к фосфорилированному γH2AX, показали, что обработка культуры иммортализованных фибробластов человека G4-стабилизирующим лигандом RHPS4 вызывает образование разрывов теломерной ДНК, причем практически исключительно в S-фазе, в которой имеет место репликация [74]. Аналогичные данные получены и для G4-стабилизирующих лигандов 360A [75] и PhenDC3 [76].
“Выключение” одной или нескольких хеликаз, обладающих G4-резольвазной (т.е., разворачивающей) активностью, как и использование G4-стабилизирующих лигандов, приводит к торможению процесса репликации и образованию соответствующих повреждений ДНК. FISH-анализ отдельных молекул реплицируемой ДНК показал, что in cellulo в фибробластах мышей, гомозиготных по нулевому аллелю гена, кодирующего ДНК-хеликазу BLM (Blmm3/m3), скорость движения репликационной вилки через теломеры существенно ниже, чем в контрольных фибробластах мышей дикого типа [76]. Этот эффект описан и для ДНК-хеликазы FANCJ в культуре клеток DT40 курицы [77] и ДНК-хеликазы WRN в клетках HeLa и культуре первичных фибробластов IMR90 [78].
Таким образом, G-квадруплексы могут, выступая в качестве естественных препятствий на пути движения ДНК-полимеразного комплекса, тормозить репликативную вилку. Это приводит к возникновению делеций в одной из цепей теломерной ДНК и образованию так называемых хрупких теломер. Для предотвращения этого в клетках существует механизм высвобождения ДНК-полимеразных комплексов, блокированных G-квадруплексами, с помощью экзонуклеазы 1 (EXO1): EXO1, обладающая 5' → 3'-экзонуклеазной и РНКазной (РНКаза H) активностями, удаляет несколько нуклеотидов из дочерней ДНК вблизи G4, высвобождая ДНК-полимеразный комплекс [79].
В то же время, согласно полногеномному картированию, у многоклеточных организмов некоторые ориджины репликации (от 67% у Drosophila melanogaster до 90% у Homo sapiens) предваряются элементами OGRE (Origin G-rich Repeated Element), представляющие собой G/C-богатые последовательности, способные формировать G-квадруплексы [80]. Ориджины этого класса располагаются в некодирующих областях генома [81] и, как следствие, в отличие от ориджинов, локализованных вблизи точек начала транскрипции генов, их активация не ассоциирована с транскрипцией и осуществляется по иному механизму. Показано, что в геноме курицы гуаниновые квадруплексы, формирующиеся в элементах OGRE, участвуют в поддержании оптимальной активности двух ассоциированных с ними ориджинов (med14 и βA) [82]. Оказалось, что in cellulo в эмбриональных стволовых клетках мыши (линия CGR8) формирование гуаниновых квадруплексов в OGRE также необходимо для включения ассоциированных с ними ориджинов репликации, чья активность не зависит от транскрипции. Более того, введение с помощью системы CRISPR-Сas9 OGRE одного из ориджинов репликации в репликационно и транскрипционно неактивный участок генома, не содержащий G4-формирующих последовательностей, приводит к образованию нового функционального ориджина репликации на модифицированном участке генома. Вместе с тем, удаление элемента OGRE из ориджина в значительной степени снижает его активность, не влияя, однако, на активность транскрипции ассоциированного с ним гена. Обработка клеток G4-стабилизирующим лигандом PhenDC3 способна вызывать активацию OGRE-ассоциированных ориджинов репликации и инактивацию ориджинов, не содержащих G4 в регуляторном элементе и активируемых транскрипцией близлежащих генов (что логично следует из того факта, что G4, расположенные в промоторной области гена, преимущественно ингибируют транскрипцию (см. ниже)) [81] Поскольку OGRE расположены на расстоянии 160–280 п.н от ассоциированных с ними ориджинов [80], можно предположить, что роль G-квадруплексов в их активации обусловлена тем обстоятельством, что данные структуры служат мишенями, с которыми связывается множество белков, включая некоторые факторы репликации, например ORC [83].
Гуаниновые квадруплексы принимают участие в патогенезе заболеваний, связанных с нарушением репликации митохондриального генома и дефектами дыхательной функции клетки. Известно, что тяжелая цепь мтДНК обогащена последовательностями, формирующими G4. При этом образование гуаниновых квадруплексов на мтДНК можно рассматривать как одно из следствий малигнизации клетки [84]. В исследовании Falabella и соавт., опубликованном в 2019 году, показано, что in vitro низкие дозы G4-стабилизирующего лиганда RHPS4 не оказывают влияния на G-квадруплексы ядерной ДНК, но вызывают резкое ингибирование элонгации митохондриального репликона и транскрипта и делетирование области генома, кодирующей белки дыхательного комплекса. Роль G4 в данном процессе подтверждена введением в мтДНК мутации, способствующей образованию антипараллельного G-квадруплекса повышенной стабильности, что вызывало серьезные дефекты в дыхательной системе клетки [85].
Кроме того, известно, что потенциальные G4-формирующие последовательности содержатся в ориджинах репликации некоторых вирусов, например, вируса саркомы Капоши, где данные последовательности ассоциированы с комплексами репликации ORC и MCM и необходимы для стабильного поддержания вирусной эписомы в клетке хозяина [86]. Поэтому предполагается, что гуаниновые квадруплексы на ДНК принимают участие в регуляции репликации вирусных геномов.
Поддержание структуры и функции теломер
Теломеры играют важнейшую роль в поддержании стабильности генома, процессах старения и в развитии опухолей. По своей природе теломеры представляют собой ДНК-белковые комплексы, защищающие концы хромосом от слипания и деградации. Теломеры человека состоят из многократных тандемных TTAGGG-повторов общей длиной 5000–20 000 п.н., оканчивающихся так называемой T-петлей – структурой, образованной 3′-концевыми участками оцДНК длиной 30–500 н. В ряду клеточных делений в силу особенностей репликации некодирующей цепи происходит постепенное укорочение теломерной ДНК. По достижении теломерами критической длины пролиферация прекращается, однако в активно делящихся эмбриональных клетках длина теломер поддерживается за счет активности теломеразы – РНК-зависимой обратной транскриптазы TERT. Дифференцировка клеток характеризуется, помимо прочего, подавлением экспрессии генов плюрипотентности, в том числе теломеразы, но в процессе малигнизации клеток и дальнейшего образования стволовых опухолевых клеток ген теломеразы реактивируется [87].
Благодаря своему нуклеотидному составу, терминирующая оцДНК теломер формирует гуаниновые квадруплексы Htel. С помощью КД-спектрометрии и ЯМР показано, что теломерные гуаниновые квадруплексы, образованные in vitro, представляют собой смесь параллельных и антипараллельных структур с короткими (>3 н.) петлями [88], а данные иммунопреципитации хроматина с антителами D1, полученными методом фагового дисплея, указывают на параллельную топологию теломерных G4, формирующихся in cellulo в клетках SiHa [89]. Методами рентгеноструктурного анализа и молекулярного моделирования показали, что в теломере в области перехода 3′-концевого G4 в ДНК-дуплекс второй (считая от G4) атом тимина в дцДНК выступает наружу, что делает возможным образование триады TAT, защищающей область между квадруплексом и дуплексом от связывания низкомолекулярными лигандами, потенциально способными нарушить структуру теломер [90].
Теломерные гуаниновые квадруплексы Htel на ДНК в целом очень стабильны (Tm = 65°C) [91]. Однако данные ИК-спектроскопии in vitro выявили обратно пропорциональную зависимость между стабильностью теломерных G4 и их концентрацией: в присутствии переизбытка Htel, обычно стабильных, степень их разворачивания изменяется с −19 на 7 мл/моль. Этот эффект может быть объяснен тем обстоятельством, что немногие развернувшиеся G4 в системе с переизбытком ДНК повышают общую обводненность системы, приводя к разворачиванию еще большего числа квадруплексов и так далее. Поскольку концентрация ДНК в ядре клетки крайне высока, описанный эффект может играть важную роль в регуляции теломерных гуаниновых квадруплексов in vivo [92]. Кроме того, G-квадруплексы Htel могут связываться и разворачиваться RecQ-подобной хеликазой StyRecQL [93], РНК-хеликазой DHX36 [94] и другими белками, обладающими G4-резольвазной активностью, что необходимо для репликации теломерной ДНК.
Теломерная последовательность человека дает начало двум некодирующим РНК-транскриптам – TERC, длиной 11 н. (5'-CUAACCCUAAC-3'), служащему матрицей для синтеза теломерной последовательности теломеразой [95], и TERRA, содержащему 96 UUAGGG-повторов. Изучение молекулярной динамики и устойчивости TERRA к РНКазе T1 показало, что ее зрелая структура включает три уникальных элемента – гуаниновые квадруплексы, образованные группами повторов ((UUAGGG)4U) и ((UUAGGG)8U), и оцРНК длиной 25 н. [96]. G4-квадруплексы TERRA могут связываться РНК-хеликазами [97], а также комплексами белков, содержащих домены RGG (FMRP) [98] или GAR (TRF2) [99]. Кроме того, TERRA связывается с теломеразой независимо от TERC, выступая, таким образом, в качестве конкурентного ингибитора активности теломеразы [100].
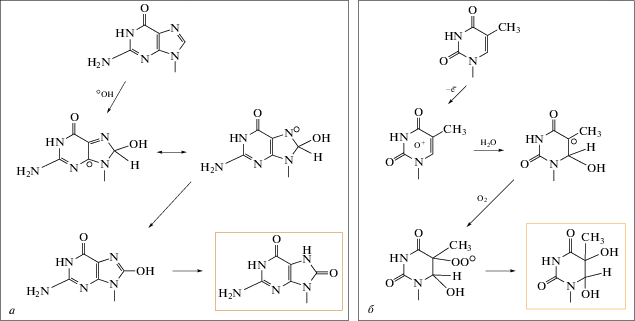
Свободные радикалы кислорода могут повреждать азотистые основания, составляющие гуаниновый квадруплекс, в результате чего образуются 8-оксогуанин (8oxoG) и 5,6-дигидрокси-5,6-дигидротимин (тимингликоль (Tg)) (рис. 4), которые в норме становятся мишенями для системы эксцизионной репарации оснований (base excision repair (BER)) [101, 102]. Поскольку теломеры расположены на концах хромосом и содержат одноцепочечные участки ДНК и большое количество остатков гуанина, они особенно уязвимы для свободных радикалов кислорода. Из-за того, что активность 8-оксогуанингликозилазы (OGG1) в контексте гуаниновых квадруплексов и оцДНК крайне низка, BER-репарация на теломерах протекает очень медленно, ввиду чего со временем в теломерах происходит накопление продуктов окисления азотистых оснований, что пагубно сказывается на их структуре и функции [103]. С помощью smFRET-микроскопии Lee и соавт. детально изучили механизм воздействия азотистых оснований, поврежденных свободными радикалами кислорода, на структуру гуаниновых квадруплексов теломер человека. Оказалось, что окисление центральных гуанинов теломерного G-квадруплекса приводит к его разворачиванию, а также препятствует формированию G4 de novo. Нарушение структуры G4 делает теломерную ДНК более доступной для взаимодействия с теломеразой и POT1 [104, 105]. Как следствие, происходит активация теломеразы [106] и POT1-опосредованная инициация формирования комплекса шелтеринов, который (как показано in vivo в культуре Schizosaccharomyces pombe) препятствует посадке на теломеру ферментов системы репарации [107]. Как результат, происходит удлинение теломеры и накопление в ней продуктов окисления свободными радикалами. Кроме того, полноразмерный POT1 связывается in vitro и in cellulo с хеликазой BLM и, в еще большей степени, с хеликазой WRN [108], разворачивающими гуаниновые квадруплексы, что приводит к усилению разрушительного эффекта окисления гуанина на гомеостаз теломер.
Рис. 4.
Повреждение азотистых оснований в ДНК под воздействием активных форм кислорода. а – Образование 8‑оксогуанина (8oxoG): гидроксильный радикал атакует связь N7=C8 с образованием интермедиата, который в процессе дальнейшего окисления может трансформироваться в 8-гидроксогуанин (8-OHGua); способный, в свою очередь, переходить в 8-оксогуанин. б – Образование тимингликоля (Tg): гидроксильный радикал атакует связь C5=C6, при этом образуется гидроксигидропироксид тимина, который далее восстанавливается до тимингликоля.
Образование Tg в петле гуанинового квадруплекса поблизости от образующих его G-тетрад также нарушает структуру G4. Это происходит из-за того, что в теломерном G-квадруплексе тимин принимает участие в укладке петель ДНК, а неплоская конформация Tg нарушает укладку соседних оснований. Две дополнительные OH-группы в положениях C5 и C6 в молекуле Tg не только создают дополнительное стерическое препятствие для образования тесных контактов внутри петли, но и повышают гидрофильность основания и степень обводнения квадруплекса, что вызывает его дестабилизацию. Поскольку Tg в меньшей степени, чем 8-оксогуанин, дестабилизирует G-квадруплекс, то и связывание Tg-модифицированной теломеры белком POT1 менее выражено, чем в случае 8oxoG-модифицированных теломер. Противоположная картина наблюдается в случае теломеразы, по-видимому, из-за того, что РНК-компонент теломеразы связывается с ДНК эффективнее, чем с POT1 [104].
Показано, что синтетические последовательности (TTAGGG)5, содержащие 8oxoG, в растворе K+/Na+ образуют гибридные G-квадруплексы с выпетливанием области поврежденного гуанина [105]. Теоретически это должно нивелировать эффект мутации. Однако поскольку теломеры человека содержат огромное количество (TTAGGG)-повторов [109], окисление содержащихся в них гуанинов и тиминов на глобальном уровне рано или поздно приводит к дестабилизации гуаниновых квадруплексов и вызывает вышеозначенные эффекты. Кроме того, продукты окисления азотистых оснований больше подвержены дальнейшему окислению, чем нативные пурины и пиримидины, поэтому повышение концентрации 8-оксогуанина в теломерах приводит со временем к накоплению в них гидантоина, а постоянная репарация гликозилазами NEIL (которые активно колокализуются с G-квадруплексами) с течением времени приводит к истощению теломер и нарушению регуляции их функции [105].
Регуляция рекомбинации
В клетках дрожжей Saccharomyces cerevisiae G-богатая последовательность 5′-GAGGGGAGGGGAAGGGGAGGGGAA-3′ хромосомы IV, ассоциированная с введением двухцепочечных разрывов в ДНК в процессе мейоза, образует G-квадруплекс. Согласно данным, полученным с помощью КД-спектрометрии, атомно-силовой микроскопии и анализа сдвига электрофоретической подвижности, этот и другие G4 узнаются и связываются с C-концевым доменом компонента синаптонемного комплекса in vitro и in vivo [110, 111], поэтому предполагается, что формирование G-квадруплексов необходимо для нормального формирования синаптонемного комплекса и успешного кроссинговера в процессе мейоза.
In silico анализ генома человека позволил выявить 32996 “горячих точек” рекомбинации, содержащих G4-формирующие последовательности, что косвенно указывает на участие гуаниновых квадруплексов в регуляции мейотической рекомбинации у высших эукариот [112]. Также показано, что Blm–/– эмбриональные стволовые клетки человека и мыши характеризуются большим количеством спонтанных обменов между сестринскими хроматидами (SCE) в районе G4-формирующих последовательностей. Это наблюдение подтверждает роль гуаниновых квадруплексов в активации рекомбинации у высших эукариот [113], поскольку, как сказано выше, хеликаза BLM обладает G4-резольвазной активностью [114].
Формирование 3D-структуры хроматина
Относительно недавно продемонстрировали роль гуаниновых квадруплексов в формировании 3D-структуры хроматина. Так, Hou и соавт. [115] показали, что расположенные на границах топологически ассоциированных доменов (ТАДов) генома последовательности ДНК, обогащенные G4-образующими мотивами, перекрываются с большим количеством сайтов посадки архитектурных белков CTCF и SMC3, а образуемые этими мотивами гуаниновые квадруплексы могут (подобно сайтам связывания CTCF) выполнять функцию инсуляторов, способствуя разделению и выпетливанию соседних ТАДов, связанных комплексом когезинов. Кроме того, с помощью атласа ReMap выявлен высокий уровень перекрывания последовательностей, образующих гуаниновые квадруплексы, с сайтами связывания регуляторов хроматина, в частности, гистоновой деметилазы PHF8 [115]. Это наблюдение свидетельствует о том, что формирование гуаниновых квадруплексов ассоциировано с локальной декомпактизацией хроматина. Об этом же свидетельствуют и другие данные. Так, показано, что in cellulo в геноме человека гуаниновые квадруплексы, образующиеся в области островков CpG (CGI) активного хроматина, колокализованы с ДНК-(цитозин-5)-метилтрансферазой DNMT1 и окружены гипометилированными участками ДНК [116]. Эти наблюдения позволили сделать вывод, что образование гуаниновых квадруплексов в районе CpG-островков приводит к связыванию и ингибированию цитозин-5-метилтрансферазы DNMT1 и способствует потере метильной метки на C5 в регуляторной области генов [116]. G4 на РНК также принимают участие в регуляции структуры хроматина. В частности, теломерная РНК TERRA взаимодействует с гистоновой деметилазой LSD1, удаляющей метильные группы с молекул гистонов H3, моно- и диметилированных по остаткам Lys4 и Lys9 [117], причем TERRA может выполнять роль аллостерического регулятора деметилирующей активности LSD1 [118]. LSD1 предпочтительно связывает гуаниновые квадруплексы, образованные элементом GG(UUAGGG)8UUA РНК TERRA. Сайт связывания LSD1 с гуаниновыми квадруплексами в TERRA отличается от сайта связывания гистона H3, но перекрывается с сайтом связывания нуклеосомной ДНК, располагаясь между аминокислотными остатками 227 и 251 SWIRM/аминооксидазного домена белка [96].
Оказалось также, что области образования гуаниновых квадруплексов перекрываются с сайтами посадки факторов транскрипции, таких как E2F4 и регулятор экспрессии инсулина NEUROD1. Иными слова, многие энхансерные области генома способны формировать эти вторичные структуры [115]. Более того, в экспериментах in silico и in cellulo показано, что одноцепочечные петли соседних квадруплексов, формирующихся на ассоциированных с энхансерами длинных (500–1500 п.н.) G4-образующих последовательностях (Long G4-capable regions – LG4), часто комплементарны друг другу, благодаря чему LG4 могут формировать особую третичную структуру – G4K (“G4 Kiss”) [119]. Наличие же G4-образующих последовательностей в промоторах предполагает возможность образования гибридных энхансер-промоторных гуаниновых квадруплексов, т.е. гуаниновые квадруплексы потенциально способны регулировать активность экспрессии генов на уровне формирования и поддержания контакта между его регуляторными областями [119, 120].
Таким образом, гуаниновые квадруплексы могут играть важную роль в регуляции формирования хроматинового ландшафта на разных его уровнях, начиная с образования контактов между энхансерами и промоторами и заканчивая компартментализацией хроматид. В то же время, по-прежнему малоизученным остается влияние гуаниновых квадруплексов (и других вторичных структур ДНК) на динамику формирования нуклеосом и наоборот.
Регуляция транскрипции
Как сказано выше, гуаниновые квадруплексы обнаружены в промоторных областях протоонкогенов, где они, как правило, формируются на кодирующей цепи после первого раунда транскрипции и создают препятствие для связывания транскрипционных факторов и продвижения РНК-полимеразы II [121]. Наиболее хорошо изучена роль гуаниновых квадруплексов в регуляции транскрипции протоонкогена CMYC, играющего важнейшую роль в эмбриогенезе, генерировании плюрипотентных стволовых клеток и патогенезе онкологических заболеваний. В промоторной области гена CMYC, в положении –142…–115 п.н. (выше промотора P1) (рис. 5) расположена регуляторная область NHEIII1, содержащая на 5′-конце богатую гуанином последовательность Pu27 (5'-TGGGGAGGGTGGGGAGGGTGGGGAAGG-3'). Эта область чувствительна к ДНКазе I и S1-нуклеазам и регулирует до 80% активности гена in cellulo [122]. Согласно данным ЯМР, эта последовательность может образовывать несколько внутримолекулярных гуаниновых квадруплексов с параллельной топологией и разной длиной петель, наиболее стабильный из которых – myc-2345 (GGG-T-GGG-GA-GGG-T-GGG) [28, 30]. В состоянии дуплекса NHEIII1 поддерживает активное состояние CMYC, сворачивание же указанного участка ДНК в гуаниновый квадруплекс приводит к подавлению экспрессии гена in cellulo. G4 блокирует связывание с регуляторной областью CMYC факторов транскрипции, например, Sp1, инициирующего сборку CMYC-активирующего комплекса FUSE/FBP/FIR/TFIIH [123]. Структура G4, расположенного в CMYC-NHEIII1, термодинамически очень стабильна и при физиологических концентрациях ионов калия их температура плавления может достигать 90°C [124].
Рис. 5.
Схема строения гена CMYC. Ген CMYC человека содержит несколько промоторов (P0, P1, P2, P3). Перед промотором P1 (в положении –142…–115) расположен элемент NHEIII1, включающий последовательность Pu27 (в рамке), способную образовывать гуаниновые квадруплексы с параллельной топологией, отличающиеся длиной, нуклеотидной последовательностью петель и термодинамическими характеристиками [126].
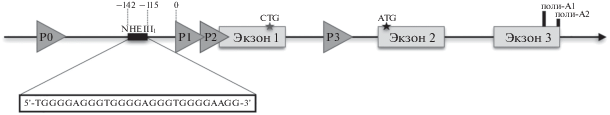
Последовательности, гомологичные Pu27 промоторной области гена CMYC, найдены также в регуляторных областях SOX2, CDH4, BRD7 и еще 13 генов [125].
Другой ген, регуляция активности которого во многом определяется присутствием в его промоторной области гуанинового квадруплекса, – протоонкоген BCL2, неконтролируемое повышение экспрессии которого приводит к дерегуляции системы апоптоза и малигнизации клеток [127]. G-богатая область Pu39, расположенная в положении –58…–19 п.н. (выше промотора P1) регуляторной области гена BCL2, может формировать 15 разных гуаниновых квадруплексов, однако только три из них обладают достаточно высокой стабильностью [128]. Стабилизация гуаниновых квадруплексов в BCL2-Pu39 лигандами, например PDF, приводит к подавлению транскрипции гена [129]. Среди других генов, в промоторах которых могут формироваться гуаниновые квадруплексы, подавляющие их транскрипцию, – гены рецепторной тирозинкиназы CKIT [130, 131], эндотелиального фактора роста VEGF [132], гомеобоксa HOXC10 [133], индуцируемого гипоксией фактора HIF1 [134] и других.
Интересно, что формирование гуаниновых квадруплексов в промоторе гена не всегда приводит к снижению уровня его экспрессии. Примером этого является OCT4, кодирующий фактор транскрипции, поддерживающий плюрипотентность эмбриональных стволовых клеток и участвующий в дедифференцировке опухолевых клеток. С использованием эмбриональных клеток человека (CCTL14) и клеток остеосаркомы (U2OS) показано, что in cellulo богатая гуанином последовательность, расположенная вблизи сайта начала транскрипции OCT4, образует параллельный G-квадруплекс, необходимый для поддержания высокого уровня экспрессии гена. Введение точечной мутации G18A в указанную область дестабилизирует G4 и приводит к заметному снижению активности гена, которая восстанавливается до исходного уровня при обработке клеток N-мезопорфирином IX (NMM) – стабилизирующим лигандом гуаниновых квадруплексов [135]. Гуаниновые квадруплексы в промоторе положительно влияют на транскрипцию генов релаксина [136], инсулина [137, 138], а также тирозингидроксилазы [139].
Наконец, известно, что на активность транскрипции влияет формирование на ее начальных этапах межмолекулярных гуаниновых квадруплексов ДНК : РНК. Так, изучение гена NRAS с использованием in vitro транскрипции РНК-полимеразой T7, сайт-специфического мутагенеза и люциферазного анализа в клетках HEK293, показало, что формирование таких G4 подавляет транскрипцию гена in vitro и in cellulo [46]. Считается, что квадруплексы ДНК : РНК участвуют также в регуляции терминации транскрипции, поскольку могут формироваться сразу после прохождения РНК-полимеразой II обогащенных гуанином участков, расположенных после сайта полиаденилирования. Эти 3′-концевые G4, образованные ДНК и РНК, привлекают хеликазу сенатаксин (SETX), которая разворачивает R-петли, открывая доступ 5' → 3'-экзонуклеазе Xrn2 к 3'-терминирующему сайту транскрипции, что приводит к высвобождению синтезированной РНК [140].
Роль в патогенезе при экспансии нуклеотидных повторов
В основе патогенеза ряда неврологических заболеваний лежит неконтролируемое увеличение (экспансия) числа идентичных олигонуклеотидных повторов в регуляторных областях генов. К примеру, при боковом амиотрофическом склерозе (ALS) и лобно-височной деменции (FTD) наблюдается экспансия гексануклеотидного повтора HRE (GGGGCC) в некодирующей области гена C9ORF72. В норме C9ORF72 содержит не более 25 HRE; симптомы заболевания начинают проявляться, когда количество HRE достигает нескольких сотен [141]. Тандемы повторов (G4C2)n могут образовывать гуаниновые квадруплексы. Топология G-квадруплексов, сформированных последовательностью (G4C2)n, зависит от количества повторов (G4C2): G-квадруплексы d(G4C2)4 в 100% случаев антипараллельны; d(G4C2)2, d(G4C2)3 и d(G4C2)5 образуют как параллельные, так и антипараллельные G-квадруплексы; G4, образованные d(G4C2)n другой длины, имеют в основном параллельную топологию. С помощью КД-спектроскопии и ЯМР показано, что G-квадруплексы d(G4C2)2 с параллельной топологией представляют собой симметричный тетрамер, а с антипараллельной – асимметричный димер [142]. Формирование (G4C2)n-G4 в некодирующей области C9ORF72 приводит к обрыву транскрипции, а на его транскрипте – к некорректной инициации трансляции. В результате в клетке накапливаются укороченные РНК-продукты и дипептиды, оказывающие цитотоксический эффект и усугубляющие патологический процесс.
Гуаниновые квадруплексы, образуемые в области экспансии олигонуклеотидов, участвуют в патогенезе и других заболеваний, таких как болезнь Альцгеймера [143] и синдром ломкой X‑хромосомы [144].
ВЗАИМОДЕЙСТВИЕ ГУАНИНОВЫХ КВАДРУПЛЕКСОВ С ЯДЕРНЫМИ БЕЛКАМИ
В связи с многообразием функций гуаниновых квадруплексов, точная регуляция их формирования необходима для поддержания гомеостаза клетки. Важную роль в этом процессе играют особенности белкового состава клетки, зависящего, например, от стадии клеточного цикла [145].
Ниже рассмотрены ядерные белки, участвующие в регуляции формирования и разрушения гуаниновых квадруплексов в ДНК.
Хеликазы нуклеиновых кислот
Выявлено более 20 ДНК- и РНК-хеликаз, обладающих G4-резольвазной активностью in vitro, in cellulo и in vivo. Среди них упомянутые выше эукариотические ДНК-хеликазы суперсемейства RecQ (Pif1, BLM, WRN), содержащие железо-серный кластер (XPD, FANCJ, DDX11, RTEL1), DEAH-box-содержащие РНК-хеликазы DHX9 и DHX36 (RHAU) и другие [146].
Хеликазы XPB и XPD, элементы комплекса TFIIH эукариот, необходимого для инициации транскрипции РНК-полимеразой II и репарации ДНК [147], связывают гуаниновые квадруплексы в промоторных областях активно транскрибируемых генов: С использованием ChIP-seq показано, что около 40% пиков XPB и XPD соответствуют нуклеотидным последовательностям, образующим G-квадруплексы. Биохимический анализ подтвердил взаимодействие XPB и XPD с квадруплексами, а также способность XPD разворачивать G4 на ДНК. Предполагается, что гуаниновые квадруплексы связываются XPB, для чего необходима АТPазная активность белка, после чего к образовавшемуся комплексу присоединяется XPD, вызывающая дестабилизацию и разворачивание G4 [148]. XPB и XPD связывают преимущественно межмолекулярные гуаниновые квадруплексы на ДНК, которые могут образовываться в ориджинах репликации [149].
Хеликаза FANCJ (Fanconi anemia complementation group J protein) принимает участие в репарации межцепочечных сшивок ДНК в клетках человека, а ее мутации ассоциированы с анемией Фанкони, характеризующейся прогрессирующим нарушением функции костного мозга и предрасположенностью к онкологическим заболеваниям [150]. Хеликаза FANCJ может также связывать и разворачивать G-квадруплексы [151], благодаря чему играет важную роль в репликации формирующих их последовательностей [152]. Согласно результатам анализа мутаций K141A/K142A хеликазы FANCJ, за связывание с гуаниновыми квадруплексами ответственен ее N-концевой пептид AKKQ [153]. FANCJ-AKKA связывает также TTA-петли гуаниновых квадруплексов теломер, содержащих 8-оксогуанин, и укорочение этих петель до длины в один тимин приводит к снижению аффинности FANCJ к теломерным G4 [154]. По-видимому, взаимодействие FANCJ с 8oxoG играет роль в разворачивании поврежденных гуаниновых квадруплексов, расположенных на теломерной ДНК, необходимой для их дальнейшей репарации. Поскольку, благодаря домену PIP-box, хеликаза FANCJ может связываться с PCNA и REV-1, в настоящее время изучается вовлеченность указанных белков в процесс FANCJ-опосредованного разворачивания гуаниновых квадруплексов [154]. В связывании G-квадруплексов FANCJ важную роль играет железосерный кластер белка: варианты хеликазы, несущие мутации C283R, C283S и A349P, и, следовательно, лишенные способности координировать FeS-кластер, обладают пониженным сродством к G4 в реплисоме [155].
РНК-хеликазы DEAH-box могут разворачивать как G4 на ДНК, так и G4 на РНК в направлении 3′ → 5′. Это касается, в частности, хеликаз DHX9 и DHX36. In silico показано, что DHX9 и DHX36 связывают и разворачивают гуаниновые квадруплексы, формирующиеся в 5′-НТО мРНК транскрипционных факторов и эпигенетических регуляторов вблизи старт-кодонов. Разворачивание G4 способствует сборке 80S рибосомы на канонических кодонах и активации трансляции основной изоформы белка [156]. DDX36 преимущественно разворачивает G-квадруплексы с параллельной топологией [157], причем этот процесс происходит быстрее, если одноцепочечная последовательность богата аденином (показано для одномолекулярных G4 на ДНК и тетрамолекулярных G4 на РНК) [158]. N-концевой домен DDX36 участвует в связывании данного белка с гуаниновым квадруплексом, образованным на ДНК [159].
DEAD-box-содержащая РНК-хеликаза DDX5, играющая важную роль в патогенезе онкологических заболеваний (повышение уровня ее экспрессии характерно для большинства видов опухолей человека [160–162]), также обладает G4-резольвазной активностью в отношении внутримолекулярных гуаниновых квадруплексов, сформированных как на РНК, так и на ДНК. Так, показано, что in cellulo DDX5 узнает и разворачивает G-квадруплексы в NHEIII1-области промотора CMYC, а также G-квадруплексы в мРНК CMYC. Несмотря на то что DDX5, как и другие хеликазы, обладает АТРазной активностью, DDX5-опосредованное разворачивание гуаниновых квадруплексов не требует гидролиза АТР [163].
Поли(АDP-рибоза)-полимеразы
Поли(ADP-рибоза)-полимеразы (PARP) – семейство ДНК-связывающих белков, содержащих два мотива типа цинковых пальцев и катализирующих реакцию переноса ADP-рибозы на белки-мишени (поли(ADP)рибозилирование). Семейство PARP насчитывает, как минимум, 18 белков, содержащих консервативный каталитический домен. Основная функция белков PARP (в частности, PARP1, PARP2 и PARP3) – узнавание и связывание одноцепочечных разрывов ДНК, что является первым этапом сборки системы эксцизионной репарации. Большинство субстратов PARP – это белки систем репарации, ферменты синтеза ДНК, регуляторы хроматина и гистоны; кроме того, в присутствии разрывов ДНК PARP подвергаются авто-поли(ADP)рибозилированию in vitro, in cellulo и in vivo [164].
PARP1 может связывать гуаниновые квадруплексы, формируемые теломерной ДНК и последовательностью промотора гена CKIT человека in vitro [165]. In cellulo показано, что PARP1 связывает гуаниновые квадруплексы, образующиеся в процессе транскрипции богатой гуанином последовательности в соотношении две–три молекулы к одному G4. При этом происходит активация и авто-поли(ADP)рибозилирование PARP1 [37]. В присутствии 100 мМ KCl PARP1 человека связывает гуаниновые квадруплексы на промоторе CMYC in vitro, а также in cellulo. Это связывание повышает ферментативную активность PARP1 (хотя и в меньшей степени, чем разрывы ДНК) и, в конечном итоге, приводит к разворачиванию G4; важнейшую роль в данном процессе играет первый цинковый палец PARP1, а G4-стабилизирующий лиганд TMPyP4 снижает PARP1-опосредованное связывание и разворачивание G-квадруплексов промотора CMYC [166]. В то же время, стабилизация G-квадруплексов в клетках лигандом RHPS4, вызывающая ингибирование теломеразы [167] и остановку репликации теломер [74], способствует рекрутированию PARP1 (но не PARP2) в область теломер, где данный белок, активируясь, катализирует поли(ADP)рибозилирование POT1, что приводит к диссоциации комплекса шелтеринов. Для связывания PARP1 с теломерой необходимо присутствие белка TRF2 и гуаниновых квадруплексов [168]. В промоторе PARP1 также присутствует богатая гуанином последовательность, образующая внутримолекулярный (3+1) гибридный гуаниновый квадруплекс длиной 23 н., благодаря чему данная последовательность может обеспечивать саморегуляцию PARP1 на уровне экспрессии его гена [169].
PARP3, как и PARP1, способен узнавать и связывать гуаниновые квадруплексы, образующиеся в области двухцепочечных разрывов ДНК, причем это взаимодействие приводит к разворачиванию G4. Этот процесс важен для инициации репарации ДНК как путем негомологичного соединения концов, так и с помощью гомологической рекомбинации, и значимость его подтверждается тем, что обработка клеток PARP3–/– G4-стабилизирующими лигандами PhenDC3 и пиридостатином останавливает репарацию и вызывает гибель клеток [170].
Нуклеолин
Нуклеолин (NCL) – фосфопротеин, выполняющий в клетке множество функций. Во-первых, NCL может играть роль шаперона гистонов и коремоделера хроматина так как, стимулируя диссоциацию гистонового димера H2A–H2B и дестабилизируя тем самым гистоновый октамер, данный белок способствует смещению нуклеосом ремоделерами SWI/SNF и ACF [171]. Также нуклеолин может выступать в роли транскрипционного фактора [172]. Кроме того, сайты связывания нуклеолина содержатся в пре-рРНК, а также в 18S и 28S РНК, что подразумевает участие данного белка в биогенезе рибосом [173].
Нуклеолин индуцирует формирование G-квадруплекса in vitro на одноцепочечной последовательности промотора CMYC; in cellulo нуклеолин с высокой аффинностью и селективностью связывает и стабилизирует параллельные гуаниновые квадруплексы, формирующиеся в области NHEIII1 промотора CMYC. В свою очередь, стабилизация гуаниновых квадруплексов в промоторе CMYC ингибирует транскрипцию его мРНК. Таким образом, нуклеолин, экспрессия которого контролируется фактором CMYC, может действовать в петле отрицательной обратной связи, подавляя экспрессию CMYC [174]. Нуклеолин связывает и стабилизирует гуаниновые квадруплексы, расположенные в промоторе рецептора андрогена (AR) [175]. Наконец, нуклеолин связывает эндогенный элемент HRE, расположенный в некодирующей области гена C9ORF72, стабилизируя сформировавшиеся там гуаниновые квадруплексы и инициируя тем самым молекулярные процессы, которые приводят к развитию нейродегенеративного заболевания ALS/FTD [176].
Узнавание нуклеолином гуанинового квадруплекса не зависит от его топологии или особенностей состава G4-образующей последовательности и со стороны квадруплекса определяется исключительно длиной его петель; NCL человека предпочтительно связывает G4 с длиной петель ≥3 н. [177].
Нуклеолин в основном локализуется в ядре клетки, но выявляется также на клеточных мембранах и в цитоплазме [178], где принимает участие в регуляции G4 на РНК, что играет особую роль в репликации генома РНК-содержащих вирусов, о чем будет рассказано ниже.
Вирус Эпштейна–Барр (EBV) инфицирует клетки иммунной системы. Ядерный антиген 1 (Ebna1), играющий важную роль в репликации этого вируса и поддержании его генома, является сильным антигеном, поэтому для успешного размножения вируса экспрессия гена Ebna1 должна поддерживаться на низком уровне. В этом процессе важную роль играет богатая гуанином последовательность мРНК Ebna1, кодирующая функциональные глицин-аланиновые повторы (GAr) и образующая G-квадруплекс, снижающий активность ее трансляции. Стабилизация G4 обеспечивается нуклеолином клетки хозяина, поэтому in cellulo эктопическая экспрессия NCL ингибирует синтез Ebna1. Снижение же экспрессии нуклеолина, равно как и обработка клеток лигандом гуаниновых квадруплексов PhenDC3, который препятствует связыванию нуклеолина с G4 на мРНК Ebna1, вызывает противоположный эффект [179].
Коровая РНК вируса гепатита C (HCV) образует гуаниновый квадруплекс, преимущественно параллельной топологии, в положении между нуклеотидами 267 и 285, соответствующем области, кодирующей домен D1 корового белка [180], который, обладая положительным зарядом (благодаря большому количеству основных аминокислот), связывает вирусную РНК и индуцирует формирование нуклеокапсида [181]. В клетке хозяина указанный гуаниновый квадруплекс стабилизируется нуклеолином, чья экспрессия повышается в ответ на инфицирование вирусом. Стабилизация же G-квадруплекса приводит, в свою очередь, к ингибированию транскрипции РНК и репликации генома HCV [182].
Промотор LTR HIV-1 содержит несколько G-треков, которые могут формировать разные варианты гуаниновых квадруплексов. Нуклеолин клетки хозяина связывает и стабилизирует большинство (хотя и не все) вариантов этих G-квадруплексов, что вызывает снижение эффективности экспрессии генома HIV-1 [183].
Опухолевый супрессор P53
Опухолевый супрессор P53 играет ключевую роль в регуляции пролиферации клетки. Одним из основных генов-мишеней P53 является протоонкоген CMYC: известно, что связывание P53 с областью NHEIII1 промотора CMYC приводит к снижению уровня экспрессии гена [184]. В то же время, связывание P53 с промотором CMYC нарушается в отсутствие C-концевого домена белка, а также при дестабилизации гуаниновых квадруплексов, в норме формируемых последовательностью NHEIII1. Таким образом, G4 играют ключевую роль в P53-опосредованной регуляции экспрессии CMYC и, вероятно, других протоонкогенов [185].
Эксперименты in vitro показали, что связывание P53 человека с теломерной последовательностью G3(TTAG3)n, участвующее в защите теломер, также как и в случае промотора CMYC, требует присутствия в ДНК гуаниновых квадруплексов, что может косвенно свидетельствовать о стабилизирующем эффекте P53 в отношении гуаниновых квадруплексов. Увеличение длины теломерной последовательности (и, как следствие, числа гуаниновых квадруплексов в системе), а также введение G4-стабилизирующего лиганда N-метилмезопорфирина IX (NMM) усиливают взаимодействие P53 с теломерой. Аффинность P53 к гуаниновым квадруплексам теломер значительно выше, если в качестве стабилизирующих катионов выступают ионы K+, а не Na+ [186].
Комплексы Polycomb
Репрессирующие комплексы Polycomb – PRC2, триметилирующий гистон H3 по остатку Lys27, и PRC1, моноубиквитинирующий гистон H2A по остатку Lys119 в клетках млекопитающих (и Lys118 в клетках D. melanogaster) – необходимы для подавления транскрипции и формирования гетерохроматина, потому они играют важную роль в эмбриогенезе, клеточной дифференцировке и онкогенезе [187].
Недавно Hanna и соавт. показали, что BMI1, белок PRC1-подобного комплекса группы Polycomb, ассоциированный с ранней спорадической формой болезни Альцгеймера, сконцентрирован в участках гетерохроматина, обогащенных мнимыми G4-формирующими последовательностями PHQS, а клетки с пониженной экспрессией BMI1 и релаксированным хроматином характеризуются накоплением гуаниновых квадруплексов. Таким образом, сделан вывод, что формирование гетерохроматина, обеспечиваемое прежде всего комплексами Polycomb, препятствует формированию G‑квадруплексов. По-видимому, белки комплексов PRC узнают и связывают PHQS, ассоциированные с активными генами, создавая, тем самым, “центры” гетерохроматинизации и ингибируя процесс формирования гуаниновых квадруплексов [188].
Помимо ДНК, комплексы Polycomb млекопитающих – прежде всего PRC2 – неспецифически связывают различные РНК [189]. In vitro очищенный PRC2 человека узнает моноосновные одноцепочечные РНК в следующем порядке убывания сродства к ним: G > C, U $ \gg $ A. In cellulo SUZ12, субъединица PRC2, преимущественно связывает РНК-мотивы, состоящие из коротких повторов последовательно расположенных гуанинов, причем формирование этими повторами гуаниновых квадруплексов существенно повышает сродство SUZ12 к РНК. В то же время, дцРНК не являются мишенями для PRC2 [42].
Важно отметить, что in vivo G-треки, потенциально способные формировать гуаниновые квадруплексы, в большом количестве представлены на участках генома, содержащих гистон H3, триметилированный по остатку Lys27 (H3K27me3), т.е. присутствуют на генах-мишенях PRC2. мРНК этих генов также содержат G-треки и способны образовывать G4, что делает их мишенями для PRC2. Так, в эмбриональных клетках мыши PRC2 присутствует в промоторных областях большого количества активных генов. При этом в процессе транскрипции синтезируемая мРНК связывает PRC2, который перераспределяется с промотора [190] в соответствии со следующей закономерностью в отношении эффективности связывания комплекса PRC2 с нуклеиновыми кислотами и белками: РНК $ \gg $ нуклеосомы с длинным ДНК-линкером ≈ ДНК $ \gg $ гистоновые хвосты [191]. Таким образом, РНК-транскрипты генов-мишеней PRC2 конкурируют за комплекс с хроматином, препятствуя триметилированию H3K27 отдельных областей генома и осуществляя положительную обратную связь в отношении регуляции уровня экспрессии кодирующих их генов. При этом в процессе клеточной дифференцировки пул РНК-транскриптов, связывающих Polycomb, постепенно уменьшается вслед за изменением соотношения транскрипционных факторов, активирующих и ингибирующих кодирующие их гены [192].
ЗАКЛЮЧЕНИЕ
Многообразие механизмов регуляции экспрессии генов обеспечивает тонкую настройку клеточных процессов и поддержание гомеостаза, а их детальное изучение необходимо не только для понимания функционирования организма и отдельных его элементов, но и для разработки новых методов диагностики и лечения онкологических и других заболеваний. Один из таких регуляторных механизмов – формирование нуклеиновыми кислотами вторичных структур, к которым, в частности, относятся гуаниновые квадруплексы (G4).
Благодаря быстрому развитию методической базы, за последние десятилетия детально описаны строение и термодинамические характеристики различных G-квадруплексов [193], определена их роль в регуляции экспрессии генов на разных ее этапах, выявлены эндогенные клеточные факторы [194] и синтезированы лиганды [195], влияющие на динамику данных структур. Кроме того, благодаря своей термодинамической и химической стабильности, а также низкой иммуногенности, гуаниновые квадруплексы могут быть использованы в диагностических целях [10]. Так, G4-содержащий тромбиновый аптамер TBA может использоваться для связывания и визуализации наномолярных концентраций тромбина. Образованный РНК G4-аптамер Spinach применяется для детекции 3,5-дифторо-4-гидроксибензилиден имидазолина (DFHBI) и некоторых других соединений. G4-аптамер PS2.M при связывании с гемином проявляет каталитические свойства, идентичные свойствам пероксидазы хрена, окисляя в присутствии пероксида водорода колориметрические субстраты 3,3′,5,5′-тетраметилбензидин и 2,2′-азино-бис(3-этилбензотиазолин-6-сульфоновую) кислоту, и благодаря этому может использоваться для детекции катионов, органических молекул, белков и нуклеиновых кислот [11]. Кроме того, на основе лигандов гуаниновых квадруплексов разрабатываются новые противоопухолевые средства; например, кварфлоксин (CX-3543) прошел фазу I клинических испытаний и показал эффективность в терапии солидных опухолей и лимфом, и фазу II в случае нейроэндокринной карциномы [9]. Помимо этого, поскольку гуаниновые квадруплексы участвуют в регуляции жизнедеятельности и размножения бактерий, простейших и вирусов, связывающие их лиганды показали себя как потенциальные препараты для борьбы с Salmonella enterica [196], лечения малярии [197], герпеса [198] и других инфекционных заболеваний. Наконец, в 2020 году предложен метод выявления патогенной кишечной палочки E. coli K88 в продуктах питания, в основе которого лежит молекулярная система, одним из основных компонентов которой является гуаниновый квадруплекс [13].
Стоит также отметить, что благодаря своей электропроводности гуаниновые квадруплексы и образуемые ими гуаниновые нанопроволоки представляют собой перспективный материал для создания нанопроводов и наноприборов с новыми характеристиками [12].
Несмотря на обилие данных, касающихся свойств гуаниновых квадруплексов и применения G4-содержащих молекул и G4-лигандов в медицине, наши знания об этих структурах все еще остаются неполными. Во-первых, по-прежнему малоизученными остаются межмолекулярные гуаниновые квадруплексы, хотя биоинформатические исследования показывают, что именно этот тип G4 наиболее широко представлен в клетках эукариот [46]. В контексте межмолекулярных G-квадруплексов особый интерес представляет изучение динамики их конформационного перехода во внутримолекулярные G4, а также другие канонические и неканонические структуры ДНК и РНК. Не менее интересен вопрос о существовании белков, селективно узнающих и связывающих только межмолекулярные или только внутримолекулярные G-квадруплексы. Наконец, остается открытым вопрос о возможности существования межмолекулярных гуаниновых квадруплексов РНК:РНК. Помимо этого, достаточно мало известно о взаимной регуляции нуклеосом и G-квадруплексов. Основная масса исследований гуаниновых квадруплексов проводится in vitro и in cellulo или с использованием методов in silico, поэтому данные о специфике их структуры, пространственно-временных особенностях формирования, динамике и регуляции in vivo практически отсутствуют. Причем это касается как межмолекулярных, так и более хорошо изученных внутримолекулярных гуаниновых квадруплексов. Прежде всего, это обусловлено техническими трудностями, такими как невозможность селективного маркирования и инициации формирования и разворачивания единичных гуаниновых квадруплексов в заданном месте генома, поэтому дизайн и синтез лигандов, распознающих не только структуру, но и последовательность, формирующую G4, абсолютно необходимы для дальнейшего развития данного направления.
Таким образом, изучение гуаниновых квадруплексов по-прежнему остается важной задачей не только молекулярной биологии, биофизики и химии, но и медицины, и в перспективе может внести существенный вклад в решение проблемы лечения многих заболеваний, а также в развитие нанотехнологий.
Работа выполнена при финансовой поддержке Российского научного фонда (проект № 19-74-30003).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Watson J.D., Crick F.H. (1953) Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature. 171, 737–738.
Barone G., Fonseca Guerra C., Bickelhaupt F.M. (2013) B-DNA structure and stability as function of nucleic acid composition: dispersion-corrected DFT study of dinucleoside monophosphate single and double strands. Chem. Open. 2, 186–193.
Franklin R.E., Gosling R.G. (1953) The structure of sodium thymonucleate fibres. I. The influence of water content. Acta Crystallogr. 6, 673–677.
Dickerson R.E. (1992) DNA structure from A to Z. Methods Enzymol. 211, 67–111.
Wang A.H.J., Quigley G.J., Kolpak F.J., Crawford J.L., Vanboom J.H., Vandermarel G., Rich A. (1979) Molecular-structure of a left-handed double helical DNA fragment at atomic resolution. Nature. 282, 680–686.
Leontis N.B., Westhof E. (2001) Geometric nomenclature and classification of RNA base pairs. RNA. 7, 499–512.
Gellert M., Lipsett M.N., Davies D.R. (1962) Helix formation by guanylic acid. Proc. Natl. Acad. Sci. USA. 48, 2013–2018.
Duarte A.R., Cadoni E., Ressurreicao A.S., Moreira R., Paulo A. (2018) Design of modular G-quadruplex ligands. Chem. Med. Chem. 13, 869–893.
Information N.C.f.B. (2020) National Center for Biotechnology Information. https://go.drugbank.com/ drugs/DB06638.
Viglasky V., Hianik T. (2013) Potential uses of G-quadruplex-forming aptamers. Gen. Physiol. Biophys. 32, 149–172.
Kwok C.K., Merrick C.J. (2017) G-quadruplexes: prediction, characterization, and biological application. Trends Biotechnol. 35, 997–1013.
Livshits G.I., Stern A., Rotem D., Borovok N., Eidelshtein G., Migliore A., Penzo E., Wind S.J., Di Felice R., Skourtis S.S., Cuevas J.C., Gurevich L., Kotlyar A.B., Porath D. (2014) Long-range charge transport in single G-quadruplex DNA molecules. Nat. Nanotechnol. 9, 1040–1046.
Wang Z., Lu Q., Xu T., Wang F., Huang F., Peng Y., Deng L. (2020) G-quadruplex-based assay combined with aptamer and gold nanoparticles for Escherichia coli K88 determination. Mikrochim. Acta. 187, 308.
Burge S., Parkinson G.N., Hazel P., Todd A.K., Neidle S. (2006) Quadruplex DNA: sequence, topology and structure. Nucl. Acids Res. 34, 5402–5415.
Lane A.N., Chaires J.B., Gray R.D., Trent J.O. (2008) Stability and kinetics of G-quadruplex structures. Nucl. Acids Res. 36, 5482–5515.
Puig Lombardi E., Londono-Vallejo A. (2020) A guide to computational methods for G-quadruplex prediction. Nucl. Acids Res. 48, 1–15.
Haider S.M., Parkinson G.N., Neidle S. (2003) Structure of a G-quadruplex–ligand complex. J. Mol. Biol. 326, 117–125.
Chung W.J., Heddi B., Schmitt E., Lim K.W., Mechulam Y., Phan A.T. (2015) Structure of a left-handed DNA G-quadruplex. Proc. Natl. Acad. Sci. USA. 112, 2729–2733.
Fu B., Huang J., Chen Y., Wang Y., Xue T., Xu G., Wang S., Zhou X. (2016) Right-handed and left-handed G-quadruplexes have the same DNA sequence: distinct conformations induced by an organic small molecule and potassium. Chem. Commun. (Camb.). 52, 10052–10055.
Lech C.J., Heddi B., Phan A.T. (2013) Guanine base stacking in G-quadruplex nucleic acids. Nucl. Acids Res. 41, 2034–2046.
Štefl R., Cheatham T.E., Špačková N.A., Fadrná E., Berger I., Koča J., Šponer J. (2003) Formation pathways of a guanine-quadruplex DNA revealed by molecular dynamics and thermodynamic analysis of the substates. Biophys. J. 85, 1787–1804.
Marsh T.C., Henderson E. (1994) G-wires: self-assembly of a telomeric oligonucleotide, d(GGGGTTGGGG), into large superstructures. Biochemistry. 33, 10718–10724.
Marsh T.C., Vesenka J., Henderson E. (1995) A new DNA nanostructure, the G-wire, imaged by scanning probe microscopy. Nucl. Acids Res. 23, 696–700.
Bose K., Lech C.J., Heddi B., Phan A.T. (2018) High-resolution AFM structure of DNA G-wires in aqueous solution. Nat. Commun. 9, 1959.
Williamson J.R. (1994) G-quartet structures in telomeric DNA. Annu. Rev. Biophys. Biomol. Struct. 23, 703–730.
Parkinson G.N., Lee M.P., Neidle S. (2002) Crystal structure of parallel quadruplexes from human telomeric DNA. Nature. 417, 876–880.
Bugaut A., Balasubramanian S. (2008) A sequence-independent study of the influence of short loop lengths on the stability and topology of intramolecular DNA G-quadruplexes. Biochemistry. 47, 689–697.
Phan A.T., Modi Y.S., Patel D.J. (2004) Propeller-type parallel-stranded G-quadruplexes in the human c-myc promoter. J. Am. Chem. Soc. 126, 8710–8716.
Pandey S., Agarwala P., Maiti S. (2013) Effect of loops and G-quartets on the stability of RNA G-quadruplexes. J. Phys. Chem. B. 117, 6896–6905.
Mathad R.I., Hatzakis E., Dai J., Yang D. (2011) c-MYC promoter G-quadruplex formed at the 5'-end of NHE III1 element: insights into biological relevance and parallel-stranded G-quadruplex stability. Nucl. Acids Res. 39, 9023–9033.
Chambers V.S., Marsico G., Boutell J.M., Di Antonio M., Smith G.P., Balasubramanian S. (2015) High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat. Biotechnol. 33, 877–881.
Lodish H., Berk A., Zipursky S.L., Matsudaira P., Baltimore D., Darnell J. (2000) Molecular Cell Biology, 4 Ed. New York: W.H. Freeman and Company, p. 1184.
Hansel-Hertsch R., Beraldi D., Lensing S.V., Marsico G., Zyner K., Parry A., Di Antonio M., Pike J., Kimura H., Narita M., Tannahill D., Balasubramanian S. (2016) G-quadruplex structures mark human regulatory chromatin. Nat. Genet. 48, 1267–1272.
Huppert J.L., Balasubramanian S. (2007) G-quadruplexes in promoters throughout the human genome. Nucl. Acids Res. 35, 406–413.
Eddy J., Maizels N. (2006) Gene function correlates with potential for G4 DNA formation in the human genome. Nucl. Acids Res. 34, 3887–3896.
Zhang X.Y., Cao E.H., Zhang Y., Chou C., Bai C. (2003) K+ and Na+-induced self-assembly of telomeric oligonucleotide d(TTAGGG)n. J. Biomol. Struct. Dyn. 20, 693–702.
Mela I., Kranaster R., Henderson R.M., Balasubramanian S., Edwardson J.M. (2012) Demonstration of ligand decoration, and ligand-induced perturbation, of G-quadruplexes in a plasmid using atomic force microscopy. Biochemistry. 51, 578–585.
Kouzine F., Wojtowicz D., Baranello L., Yamane A., Nelson S., Resch W., Kieffer-Kwon K.R., Benham C.J., Casellas R., Przytycka T.M., Levens D. (2017) Permanganate/S1 nuclease footprinting reveals non-B DNA structures with regulatory potential across a mammalian genome. Cell Syst. 4, 344–356. e7.
Biffi G., Tannahill D., McCafferty J., Balasubramanian S. (2013) Quantitative visualization of DNA G-quadruplex structures in human cells. Nat. Chem. 5, 182–186.
Konig S.L., Huppert J.L., Sigel R.K., Evans A.C. (2013) Distance-dependent duplex DNA destabilization proximal to G-quadruplex/i-motif sequences. Nucl. Acids Res. 41, 7453–7461.
Deng J., Xiong Y., Sundaralingam M. (2001) X-ray analysis of an RNA tetraplex (UGGGGU)(4) with divalent Sr(2+) ions at subatomic resolution (0.61 A). Proc. Natl. Acad. Sci. USA. 98, 13665–13670.
Wang X., Goodrich K.J., Gooding A.R., Naeem H., Archer S., Paucek R.D., Youmans D.T., Cech T.R., Davidovich C. (2017) Targeting of Polycomb repressive complex 2 to RNA by short repeats of consecutive guanines. Mol. Cell. 65, 1056–1067. e5.
Olsen C.M., Marky L.A. (2009) Energetic and hydration contributions of the removal of methyl groups from thymine to form uracil in G-quadruplexes. J. Phys. Chem. B. 113, 9–11.
Zhang D.H., Fujimoto T., Saxena S., Yu H.Q., Miyoshi D., Sugimoto N. (2010) Monomorphic RNA G-quadruplex and polymorphic DNA G-quadruplex structures responding to cellular environmental factors. Biochemistry. 49, 4554–4563.
Xiao S., Zhang J.Y., Zheng K.W., Hao Y.H., Tan Z. (2013) Bioinformatic analysis reveals an evolutional selection for DNA:RNA hybrid G-quadruplex structures as putative transcription regulatory elements in warm-blooded animals. Nucl. Acids Res. 41, 10379–10390.
Zheng K.W., Xiao S., Liu J.Q., Zhang J.Y., Hao Y.H., Tan Z. (2013) Co-transcriptional formation of DNA:RNA hybrid G-quadruplex and potential function as constitutional cis element for transcription control. Nucl. Acids Res. 41, 5533–5541.
Zhang J.Y., Xia Y., Hao Y.H., Tan Z. (2020) DNA:RNA hybrid G-quadruplex formation upstream of transcription start site. Sci. Rep. 10, 7429.
Bao H.L., Xu Y. (2020) Telomeric DNA-RNA-hybrid G-quadruplex exists in environmental conditions of HeLa cells. Chem. Commun. (Camb). 56, 6547–6550.
Xu Y., Suzuki Y., Ishizuka T., Xiao C.D., Liu X., Hayashi T., Komiyama M. (2014) Finding a human telomere DNA-RNA hybrid G-quadruplex formed by human telomeric 6-mer RNA and 16-mer DNA using click chemistry: a protective structure for telomere end. Bioorg. Med. Chem. 22, 4419–4421.
Hud N.V., Smith F.W., Anet F.A., Feigon J. (1996) The selectivity for K+ versus Na+ in DNA quadruplexes is dominated by relative free energies of hydration: a thermodynamic analysis by 1H NMR. Biochemistry. 35, 15383–15390.
Shannon R.D. (1976) Revised effective ionic radii and systematic studies of interatomic distances in halides and chalcogenides. Acta Crystallogr. A. 32, 751–767.
Phillips K., Dauter Z., Murchie A.I., Lilley D.M., Luisi B. (1997) The crystal structure of a parallel-stranded guanine tetraplex at 0.95 A resolution. J. Mol. Biol. 273, 171–182.
You J., Li H., Lu X.M., Li W., Wang P.Y., Dou S.X., Xi X.G. (2017) Effects of monovalent cations on folding kinetics of G-quadruplexes. Biosci. Rep. 37, BSR20170771
Venczel E.A., Sen D. (1993) Parallel and antiparallel G-DNA structures from a complex telomeric sequence. Biochemistry. 32, 6220–6228.
Halder K., Hartig J.S. (2011) RNA quadruplexes. Met. Ions Life Sci. 9, 125–139.
Wlodarczyk A., Grzybowski P., Patkowski A., Dobek A. (2005) Effect of ions on the polymorphism, effective charge, and stability of human telomeric DNA. Photon correlation spectroscopy and circular dichroism studies. J. Phys. Chem. B. 109, 3594–3605.
Lu H., Li S., Chen J., Xia J., Zhang J., Huang Y., Liu X., Wu H.C., Zhao Y., Chai Z., Hu Y. (2015) Metal ions modulate the conformation and stability of a G-quadruplex with or without a small-molecule ligand. Metallomics. 7, 1508–1514.
Zhang D., Huang T., Lukeman P.S., Paukstelis P.J. (2014) Crystal structure of a DNA/Ba2+ G-quadruplex containing a water-mediated C-tetrad. Nucl. Acids Res. 42, 13422–13429.
Chen F.M. (1992) Sr2+ facilitates intermolecular G-quadruplex formation of telomeric sequences. Biochemistry. 31, 3769–3776.
Miyoshi D., Nakao A., Toda T., Sugimoto N. (2001) Effect of divalent cations on antiparallel G-quartet structure of d(G4T4G4). FEBS Lett. 496, 128–133.
Miyoshi D., Nakao A., Sugimoto N. (2003) Structural transition from antiparallel to parallel G-quadruplex of d(G4T4G4) induced by Ca2+. Nucl. Acids Res. 31, 1156–1163.
Doluca O., Withers J.M., Filichev V.V. (2013) Molecular engineering of guanine-rich sequences: Z-DNA, DNA triplexes, and G-quadruplexes. Chem. Rev. 113, 3044–3083.
Blume S.W., Guarcello V., Zacharias W., Miller D.M. (1997) Divalent transition metal cations counteract potassium-induced quadruplex assembly of oligo(dG) sequences. Nucl. Acids Res. 25, 617–625.
Liu W., Zhu H., Zheng B., Cheng S., Fu Y., Li W., Lau T.C., Liang H. (2012) Kinetics and mechanism of G-quadruplex formation and conformational switch in a G-quadruplex of PS2.M induced by Pb2+. Nucl. Acids Res. 40, 4229–4236.
Sheng Z., Han J., Zhang J., Zhao H., Jiang L. (2011) Method for detection of Hg2+ based on the specific thymine-Hg2+-thymine interaction in the DNA hybridization on the surface of quartz crystal microbalance. Colloids Surf. B. 87, 289–292.
Karimata H., Miyoshi D., Fujimoto T., Koumoto K., Wang Z.M., Sugimoto N. (2007) Conformational switch of a functional nanowire based on the DNA G‑quadruplex. Nucleic Acids Symp. Ser. (Oxf.). (51), 251–252.
Kong R.M., Ma L., Han X., Ma C., Qu F., Xia L. (2020) Hg2+-mediated stabilization of G-triplex based molecular beacon for label-free fluorescence detection of Hg2+, reduced glutathione, and glutathione reductase activity. Spectrochim. Acta A Mol. Biomol. Spectrosc. 228, 117855.
Guo L., Nie D., Qiu C., Zheng Q., Wu H., Ye P., Hao Y., Fu F., Chen G. (2012) A G-quadruplex based label-free fluorescent biosensor for lead ion. Biosens. Bioelectron. 35, 123–127.
Guiset Miserachs H., Donghi D., Borner R., Johannsen S., Sigel R.K. (2016) Distinct differences in metal ion specificity of RNA and DNA G-quadruplexes. J. Biol. Inorg. Chem. 21, 975–986.
Fay M.M., Lyons S.M., Ivanov P. (2017) RNA G‑quadruplexes in biology: principles and molecular mechanisms. J. Mol. Biol. 429, 2127–2147.
Bannister A.J., Kouzarides T. (2011) Regulation of chromatin by histone modifications. Cell Res. 21, 381–395.
Maleki S., Keeney S. (2004) Modifying histones and initiating meiotic recombination; new answers to an old question. Cell. 118, 404–406.
Weitzmann M.N., Woodford K.J., Usdin K. (1996) The development and use of a DNA polymerase arrest assay for the evaluation of parameters affecting intrastrand tetraplex formation. J. Biol. Chem. 271, 20958–20964.
Rizzo A., Salvati E., Porru M., D’Angelo C., Stevens M.F., D’Incalci M., Leonetti C., Gilson E., Zupi G., Biroccio A. (2009) Stabilization of quadruplex DNA perturbs telomere replication leading to the activation of an ATR-dependent ATM signaling pathway. Nucl. Acids Res. 37, 5353–5364.
Gauthier L.R., Granotier C., Hoffschir F., Etienne O., Ayouaz A., Desmaze C., Mailliet P., Biard D.S., Boussin F.D. (2012) Rad51 and DNA-PKcs are involved in the generation of specific telomere aberrations induced by the quadruplex ligand 360A that impair mitotic cell progression and lead to cell death. Cell Mol. Life Sci. 69, 629–640.
Drosopoulos W.C., Kosiyatrakul S.T., Schildkraut C.L. (2015) BLM helicase facilitates telomere replication during leading strand synthesis of telomeres. J. Cell Biol. 210, 191–208.
Schwab R.A., Nieminuszczy J., Shin-ya K., Niedzwiedz W. (2013) FANCJ couples replication past natural fork barriers with maintenance of chromatin structure. J. Cell Biol. 201, 33–48.
Crabbe L., Verdun R.E., Haggblom C.I., Karlseder J. (2004) Defective telomere lagging strand synthesis in cells lacking WRN helicase activity. Science. 306, 1951–1953.
Stroik S., Kurtz K., Lin K., Karachenets S., Myers C.L., Bielinsky A.K., Hendrickson E.A. (2020) EXO1 resection at G-quadruplex structures facilitates resolution and replication. Nucl. Acids Res. 48, 4960–4975.
Cayrou C., Coulombe P., Puy A., Rialle S., Kaplan N., Segal E., Mechali M. (2012) New insights into replication origin characteristics in metazoans. Cell Cycle. 11, 658–667.
Prorok P., Artufel M., Aze A., Coulombe P., Peiffer I., Lacroix L., Guedin A., Mergny J.L., Damaschke J., Schepers A., Cayrou C., Teulade-Fichou M.P., Ballester B., Mechali M. (2019) Involvement of G-quadruplex regions in mammalian replication origin activity. Nat. Commun. 10, 3274.
Valton A.L., Hassan-Zadeh V., Lema I., Boggetto N., Alberti P., Saintome C., Riou J.F., Prioleau M.N. (2014) G4 motifs affect origin positioning and efficiency in two vertebrate replicators. EMBO J. 33, 732–746.
Hoshina S., Yura K., Teranishi H., Kiyasu N., Tominaga A., Kadoma H., Nakatsuka A., Kunichika T., Obuse C., Waga S. (2013) Human origin recognition complex binds preferentially to G-quadruplex-preferable RNA and single-stranded DNA. J. Biol. Chem. 288, 30161–30171.
Bharti S.K., Sommers J.A., Zhou J., Kaplan D.L., Spelbrink J.N., Mergny J.L., Brosh R.M., Jr. (2014) DNA sequences proximal to human mitochondrial DNA deletion breakpoints prevalent in human disease form G-quadruplexes, a class of DNA structures inefficiently unwound by the mitochondrial replicative Twinkle helicase. J. Biol. Chem. 289, 29975–29993.
Falabella M., Kolesar J.E., Wallace C., de Jesus D., Sun L., Taguchi Y.V., Wang C., Wang T., Xiang I.M., Alder J.K., Maheshan R., Horne W., Turek-Herman J., Pagano P.J., St Croix C.M., Sondheimer N., Yatsu-nyk L.A., Johnson F.B., Kaufman B.A. (2019) G-quadruplex dynamics contribute to regulation of mitochondrial gene expression. Sci. Rep. 9, 5605.
Madireddy A., Purushothaman P., Loosbroock C.P., Robertson E.S., Schildkraut C.L., Verma S.C. (2016) G-quadruplex-interacting compounds alter latent DNA replication and episomal persistence of KSHV. Nucl. Acids Res. 44, 3675–3694.
O'Sullivan R.J., Karlseder J. (2010) Telomeres: protecting chromosomes against genome instability. Nat. Rev. Mol. Cell Biol. 11, 171–181.
Ambrus A., Chen D., Dai J., Bialis T., Jones R.A., Yang D. (2006) Human telomeric sequence forms a hybrid-type intramolecular G-quadruplex structure with mixed parallel/antiparallel strands in potassium solution. Nucl. Acids Res. 34, 2723–2735.
Liu H.Y., Zhao Q., Zhang T.P., Wu Y., Xiong Y.X., Wang S.K., Ge Y.L., He J.H., Lv P., Ou T.M., Tan J.H., Li D., Gu L.Q., Ren J., Zhao Y., Huang Z.S. (2016) Conformation selective antibody enables genome profiling and leads to discovery of parallel G-quadruplex in human telomeres. Cell Chem. Biol. 23, 1261–1270.
Russo Krauss I., Ramaswamy S., Neidle S., Haider S., Parkinson G.N. (2016) Structural insights into the quadruplex-duplex 3' interface formed from a telomeric repeat: a potential molecular target. J. Am. Chem. Soc. 138, 1226–1233.
Bielskute S., Plavec J., Podbevsek P. (2019) Impact of oxidative lesions on the human telomeric G-quadruplex. J. Am. Chem. Soc. 141, 2594–2603.
Somkuti J., Adanyi M., Smeller L. (2019) Self-crowding influences the temperature - pressure stability of the human telomere G-quadruplex. Biophys. Chem. 254, 106248.
McRae E.K.S., Davidson D.E., Dupas S.J., McKenna S.A. (2018) Insights into the RNA quadruplex binding specificity of DDX21. Biochim. Biophys. Acta, Gen. Subj. 1862, 1973–1979.
Smaldino P.J., Routh E.D., Kim J.H., Giri B., Creacy S.D., Hantgan R.R., Akman S.A., Vaughn J.P. (2015) Mutational dissection of telomeric DNA binding requirements of G4 resolvase 1 shows that G4‑structure and certain 3'-tail sequences are sufficient for tight and complete binding. PLoS One. 10, e0132668.
Feng J., Funk W.D., Wang S.S., Weinrich S.L., Avilion A.A., Chiu C.P., Adams R.R., Chang E., Allsopp R.C., Yu J., Le S., West M.D., Harley C.B., Andrews W.H., Greider C.W., Villeponteau B. (1995) The RNA component of human telomerase. Science. 269, 1236–1241.
Hirschi A., Martin W.J., Luka Z., Loukachevitch L.V., Reiter N.J. (2016) G-quadruplex RNA binding and recognition by the lysine-specific histone demethylase-1 enzyme. RNA. 22, 1250–1260.
Tippana R., Hwang H., Opresko P.L., Bohr V.A., Myong S. (2016) Single-molecule imaging reveals a common mechanism shared by G-quadruplex-resolving helicases. Proc. Natl. Acad. Sci. USA. 113, 8448–8453.
Majumder M., House R., Palanisamy N., Qie S., Day T.A., Neskey D., Diehl J.A., Palanisamy V. (2016) RNA-binding protein FXR1 regulates p21 and TERC RNA to bypass p53-mediated cellular senescence in OSCC. PLoS Genet. 12, e1006306.
Deng Z., Norseen J., Wiedmer A., Riethman H., Lieberman P.M. (2009) TERRA RNA binding to TRF2 facilitates heterochromatin formation and ORC recruitment at telomeres. Mol. Cell. 35, 403–413.
Redon S., Reichenbach P., Lingner J. (2010) The non-coding RNA TERRA is a natural ligand and direct inhibitor of human telomerase. Nucl. Acids Res. 38, 5797–5806.
Ravanat J.L., Douki T., Cadet J. (2001) Direct and indirect effects of UV radiation on DNA and its components. J. Photochem. Photobiol. B. 63, 88–102.
Starikov E., Lewis J., Tanaka S. (2006) Enzymatic recognition of radiation-produced oxidative DNA lesion. In: Modern Methods for Theoretical Physical Chemistry of Biopolymers, 1 Ed. Elsevier Science, pp. 604.
Dou H., Mitra S., Hazra T.K. (2003) Repair of oxidized bases in DNA bubble structures by human DNA glycosylases NEIL1 and NEIL2. J. Biol. Chem. 278, 49679–49684.
Lee H.T., Bose A., Lee C.Y., Opresko P.L., Myong S. (2017) Molecular mechanisms by which oxidative DNA damage promotes telomerase activity. Nucl. Acids Res. 45, 11752–11765.
An N., Fleming A.M., Burrows C.J. (2016) Human telomere G-quadruplexes with five repeats accommodate 8-oxo-7,8-dihydroguanine by looping out the DNA damage. ACS Chem. Biol. 11, 500–507.
Fouquerel E., Lormand J., Bose A., Lee H.T., Kim G.S., Li J., Sobol R.W., Freudenthal B.D., Myong S., Opresko P.L. (2016) Oxidative guanine base damage regulates human telomerase activity. Nat. Struct. Mol. Biol. 23, 1092–1100.
Theruvathu J.A., Darwanto A., Hsu C.W., Sowers L.C. (2014) The effect of Pot1 binding on the repair of thymine analogs in a telomeric DNA sequence. Nucl. Acids Res. 42, 9063–9073.
Opresko P.L., Mason P.A., Podell E.R., Lei M., Hickson I.D., Cech T.R., Bohr V.A. (2005) POT1 stimulates RecQ helicases WRN and BLM to unwind telomeric DNA substrates. J. Biol. Chem. 280, 32069–32080.
Sadava D., Hills D., Heller, C., Berenbaum M. (2009) Life: The science of biology, 9 Ed. Sunderland, MA: Sinauer Associated Inc., pp. 389.
Khan K., Madhavan T.P., Kshirsagar R., Boosi K.N., Sadhale P., Muniyappa K. (2013) N-terminal disordered domain of Saccharomyces cerevisiae Hop1 protein is dispensable for DNA binding, bridging, and synapsis of double-stranded DNA molecules but is necessary for spore formation. Biochemistry. 52, 5265–5279.
Kshirsagar R., Khan K., Joshi M.V., Hosur R.V., Muniyappa K. (2017) Probing the potential role of non-B DNA structures at yeast meiosis-specific DNA double-strand breaks. Biophys. J. 112, 2056–2074.
Mani P., Yadav V.K., Das S.K., Chowdhury S. (2009) Genome-wide analyses of recombination prone regions predict role of DNA structural motif in recombination. PLoS One. 4, e4399.
van Wietmarschen N., Merzouk S., Halsema N., Spierings D.C.J., Guryev V., Lansdorp P.M. (2018) BLM helicase suppresses recombination at G-quadruplex motifs in transcribed genes. Nat. Commun. 9, 271.
Wu W.Q., Hou X.M., Li M., Dou S.X., Xi X.G. (2015) BLM unfolds G-quadruplexes in different structural environments through different mechanisms. Nucl. Acids Res. 43, 4614–4626.
Hou Y., Li F., Zhang R., Li S., Liu H., Qin Z.S., Sun X. (2019) Integrative characterization of G-quadruplexes in the three-dimensional chromatin structure. Epigenetics. 14, 894–911.
Mao S.Q., Ghanbarian A.T., Spiegel J., Martinez Cuesta S., Beraldi D., Di Antonio M., Marsico G., Hansel-Hertsch R., Tannahill D., Balasubramanian S. (2018) DNA G-quadruplex structures mold the DNA methylome. Nat. Struct. Mol. Biol. 25, 951–957.
Rudolph T., Beuch S., Reuter G. (2013) Lysine-specific histone demethylase LSD1 and the dynamic control of chromatin. Biol. Chem. 394, 1019–1028.
Porro A., Feuerhahn S., Lingner J. (2014) TERRA-reinforced association of LSD1 with MRE11 promotes processing of uncapped telomeres. Cell Rep. 6, 765–776.
Williams J.D., Houserova D., Johnson B.R., Dyniewski B., Berroyer A., French H., Barchie A.A., Bilbrey D.D., Demeis J.D., Ghee K.R., Hughes A.G., Kreitz N.W., McInnis C.H., Pudner S.C., Reeves M.N., Stahly A.N., Turcu A., Watters B.C., Daly G.T., Langley R.J., Gillespie M.N., Prakash A., Larson E.D., Kasukurthi M.V., Huang J., Jinks-Robertson S., Borchert G.M. (2020) Characterization of long G4-rich enhancer-associated genomic regions engaging in a novel loop:loop ‘G4 Kissing’ interaction. Nucl. Acids Res. 48, 5907–5925.
Hegyi H. (2015) Enhancer-promoter interaction facilitated by transiently forming G-quadruplexes. Sci. Rep. 5, 9165.
Eddy J., Vallur A.C., Varma S., Liu H., Reinhold W.C., Pommier Y., Maizels N. (2011) G4 motifs correlate with promoter-proximal transcriptional pausing in human genes. Nucl. Acids Res. 39, 4975–4983.
Siddiqui-Jain A., Grand C.L., Bearss D.J., Hurley L.H. (2002) Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. Proc. Natl. Acad. Sci. USA. 99, 11593–11598.
Brooks T.A., Hurley L.H. (2009) The role of supercoiling in transcriptional control of MYC and its importance in molecular therapeutics. Nat. Rev. Cancer. 9, 849–861.
Hatzakis E., Okamoto K., Yang D. (2010) Thermodynamic stability and folding kinetics of the major G‑quadruplex and its loop isomers formed in the nuclease hypersensitive element in the human c-Myc Promoter: effect of loops and flanking segments on the stability of parallel-stranded intramolecular G-quadruplexes. Biochemistry. 49, 9152–9160.
Rezzoug F., Thomas S.D., Rouchka E.C., Miller D.M. (2016) Discovery of a family of genomic sequences which interact specifically with the c-MYC promoter to regulate c-MYC expression. PLoS One. 11, e0161588.
Dexheimer T.S., Carey S.S., Zuohe S., Gokhale V.M., Hu X., Murata L.B., Maes E.M., Weichsel A., Sun D., Meuillet E.J., Montfort W.R., Hurley L.H. (2009) NM23-H2 may play an indirect role in transcriptional activation of c-myc gene expression but does not cleave the nuclease hypersensitive element III(1). Mol. Canc. Therapeut. 8, 1363–1377.
Hardwick J.M., Soane L. (2013) Multiple functions of BCL-2 family proteins. Cold Spring Harb. Perspect Biol. 5, a008722.
Dexheimer T.S., Sun D., Hurley L.H. (2006) Deconvoluting the structural and drug-recognition complexity of the G-quadruplex-forming region upstream of the bcl-2 P1 promoter. J. Am. Chem. Soc. 128, 5404–5415.
Feng Y., Yang D., Chen H., Cheng W., Wang L., Sun H., Tang Y. (2016) Stabilization of G-quadruplex DNA and inhibition of Bcl-2 expression by a pyridostatin analog. Bioorg. Med. Chem. Lett. 26, 1660–1663.
Gunaratnam M., Swank S., Haider S.M., Galesa K., Reszka A.P., Beltran M., Cuenca F., Fletcher J.A., Neidle S. (2009) Targeting human gastrointestinal stromal tumor cells with a quadruplex-binding small molecule. J. Med. Chem. 52, 3774–3783.
Rankin S., Reszka A.P., Huppert J., Zloh M., Parkinson G.N., Todd A.K., Ladame S., Balasubramanian S., Neidle S. (2005) Putative DNA quadruplex formation within the human c-kit oncogene. J. Am. Chem. Soc. 127, 10584–10589.
Sun D., Liu W.J., Guo K., Rusche J.J., Ebbinghaus S., Gokhale V., Hurley L.H. (2008) The proximal promoter region of the human vascular endothelial growth factor gene has a G-quadruplex structure that can be targeted by G-quadruplex-interactive agents. Mol. Canc. Therapeut. 7, 880–889.
Zhang X., Zhao B., Yan T., Hao A., Gao Y., Li D., Sui G. (2018) G-quadruplex structures at the promoter of HOXC10 regulate its expression. Biochim. Biophys. Acta, Gene Regul. Mech. 1861, 1018–1028.
De Armond R., Wood S., Sun D., Hurley L.H., Ebbinghaus S.W. (2005) Evidence for the presence of a guanine quadruplex forming region within a polypurine tract of the hypoxia inducible factor 1alpha promoter. Biochemistry. 44, 16341–16350.
Renciuk D., Rynes J., Kejnovska I., Foldynova-Trantirkova S., Andang M., Trantirek L., Vorlickova M. (2017) G-quadruplex formation in the Oct4 promoter positively regulates Oct4 expression. Biochim. Biophys. Acta, Gene Regul. Mech. 1860, 175–183.
Gu H.P., Lin S., Xu M., Yu H.Y., Du X.J., Zhang Y.Y., Yuan G., Gao W. (2012) Up-regulating relaxin expression by G-quadruplex interactive ligand to achieve antifibrotic action. Endocrinology. 153, 3692–3700.
Connor A.C., Frederick K.A., Morgan E.J., McGown L.B. (2006) Insulin capture by an insulin-linked polymorphic region G-quadruplex DNA oligonucleotide. J. Am. Chem. Soc. 128, 4986–4991.
Kennedy G.C., Rutter W.J. (1992) Pur-1, a zinc-finger protein that binds to purine-rich sequences, transactivates an insulin promoter in heterologous cells. Proc. Natl. Acad. Sci. USA. 89, 11498–11502.
Farhath M.M., Thompson M., Ray S., Sewell A., Balci H., Basu S. (2015) G-quadruplex-enabling sequence within the human tyrosine hydroxylase promoter differentially regulates transcription. Biochemistry. 54, 5533–5545.
Skourti-Stathaki K., Proudfoot N.J., Gromak N. (2011) Human senataxin resolves RNA/DNA hybrids formed at transcriptional pause sites to promote Xrn2-dependent termination. Mol. Cell. 42, 794–805.
Babic Leko M., Zupunski V., Kirincich J., Smilovic D., Hortobagyi T., Hof P.R., Simic G. (2019) Molecular mechanisms of neurodegeneration related to C9orf72 hexanucleotide repeat expansion. Behav. Neurol. 2019, 2909168.
Zhou B., Geng Y., Liu C., Miao H., Ren Y., Xu N., Shi X., You Y., Lee T., Zhu G. (2018) Characterizations of distinct parallel and antiparallel G-quadruplexes formed by two-repeat ALS and FTD related GGGGCC sequence. Sci. Rep. 8, 2366.
Fisette J.F., Montagna D.R., Mihailescu M.R., Wolfe M.S. (2012) A G-rich element forms a G-quadruplex and regulates BACE1 mRNA alternative splicing. J. Neurochem. 121, 763–773.
Fry M., Loeb L.A. (1994) The fragile X syndrome d(CGG)n nucleotide repeats form a stable tetrahelical structure. Proc. Natl. Acad. Sci. USA. 91, 4950–4954.
Di Antonio M., Ponjavic A., Radzevicius A., Ranasinghe R.T., Catalano M., Zhang X., Shen J., Needham L.M., Lee S.F., Klenerman D., Balasubramanian S. (2020) Single-molecule visualization of DNA G-quadruplex formation in live cells. Nat. Chem. 12, 832–837.
Mendoza O., Bourdoncle A., Boule J.B., Brosh R.M., Jr., Mergny J.L. (2016) G-quadruplexes and helicases. Nucl. Acids Res. 44, 1989–2006.
Fuss J.O., Tainer J.A. (2011) XPB and XPD helicases in TFIIH orchestrate DNA duplex opening and damage verification to coordinate repair with transcription and cell cycle via CAK kinase. DNA Repair (Amst.). 10, 697–713.
Gray L.T., Vallur A.C., Eddy J., Maizels N. (2014) G quadruplexes are genomewide targets of transcriptional helicases XPB and XPD. Nat. Chem. Biol. 10, 313–318.
Kudlicki A.S. (2016) G-quadruplexes involving both strands of genomic DNA are highly abundant and colocalize with functional sites in the human genome. PLoS One. 11, e0146174.
Brosh R.M., Jr., Cantor S.B. (2014) Molecular and cellular functions of the FANCJ DNA helicase defective in cancer and in Fanconi anemia. Front. Genet. 5, 372.
Wu Y., Shin-ya K., Brosh R.M., Jr. (2008) FANCJ helicase defective in Fanconi anemia and breast cancer unwinds G-quadruplex DNA to defend genomic stability. Mol. Cell. Biol. 28, 4116–4128.
Castillo Bosch P., Segura-Bayona S., Koole W., van Heteren J.T., Dewar J.M., Tijsterman M., Knipscheer P. (2014) FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33, 2521–2533.
Wu C.G., Spies M. (2016) G-quadruplex recognition and remodeling by the FANCJ helicase. Nucl. Acids Res. 44, 8742–8753.
Lowran K., Campbell L., Popp P., Wu C.G. (2019) Assembly of a G-quadruplex repair complex by the FANCJ DNA helicase and the REV1 polymerase. Genes (Basel). 11, 5.
Odermatt D.C., Lee W.T.C., Wild S., Jozwiakowski S.K., Rothenberg E., Gari K. (2020) Cancer-associated mutations in the iron-sulfur domain of FANCJ affect G-quadruplex metabolism. PLoS Genet. 16, e1008740.
Murat P., Marsico G., Herdy B., Ghanbarian A.T., Portella G., Balasubramanian S. (2018) RNA G-quadruplexes at upstream open reading frames cause DHX36- and DHX9-dependent translation of human mRNAs. Genome Biol. 19, 229.
Chen M.C., Tippana R., Demeshkina N.A., Murat P., Balasubramanian S., Myong S., Ferre-D’Amare A.R. (2018) Structural basis of G-quadruplex unfolding by the DEAH/RHA helicase DHX36. Nature. 558, 465–469.
Yangyuoru P.M., Bradburn D.A., Liu Z., Xiao T.S., Russell R. (2018) The G-quadruplex (G4) resolvase DHX36 efficiently and specifically disrupts DNA G4s via a translocation-based helicase mechanism. J. Biol. Chem. 293, 1924–1932.
Tippana R., Chen M.C., Demeshkina N.A., Ferre-D’Amare A.R., Myong S. (2019) RNA G-quadruplex is resolved by repetitive and ATP-dependent mechanism of DHX36. Nat. Commun. 10, 1855.
Causevic M., Hislop R.G., Kernohan N.M., Carey F.A., Kay R.A., Steele R.J., Fuller-Pace F.V. (2001) Overexpression and poly-ubiquitylation of the DEAD-box RNA helicase p68 in colorectal tumours. Oncogene. 20, 7734–7743.
Hashemi V., Masjedi A., Hazhir-Karzar B., Tanomand A., Shotorbani S.S., Hojjat-Farsangi M., Ghalamfarsa G., Azizi G., Anvari E., Baradaran B., Jadidi-Niaragh F. (2019) The role of DEAD-box RNA helicase p68 (DDX5) in the development and treatment of breast cancer. J. Cell. Physiol. 234, 5478–5487.
Xing Z., Russon M.P., Utturkar S.M., Tran E.J. (2020) The RNA helicase DDX5 supports mitochondrial function in small cell lung cancer. J. Biol. Chem. 295, 8988–8998.
Wu G., Xing Z., Tran E.J., Yang D. (2019) DDX5 helicase resolves G-quadruplex and is involved in MYC gene transcriptional activation. Proc. Natl. Acad. Sci. USA. 116, 20453–20461.
Morales J., Li L., Fattah F.J., Dong Y., Bey E.A., Patel M., Gao J., Boothman D.A. (2014) Review of poly (ADP-ribose) polymerase (PARP) mechanisms of action and rationale for targeting in cancer and other diseases. Crit. Rev. Eukaryot. Gene Expr. 24, 15–28.
Soldatenkov V.A., Vetcher A.A., Duka T., Ladame S. (2008) First evidence of a functional interaction between DNA quadruplexes and poly(ADP-ribose) polymerase-1. ACS Chem. Biol. 3, 214–219.
Fekete A., Kenesi E., Hunyadi-Gulyas E., Durgo H., Berko B., Dunai Z.A., Bauer P.I. (2012) The guanine-quadruplex structure in the human c-myc gene’s promoter is converted into B-DNA form by the human poly(ADP-ribose)polymerase-1. PLoS One. 7, e42690.
Salvati E., Leonetti C., Rizzo A., Scarsella M., Mottolese M., Galati R., Sperduti I., Stevens M.F., D’Incalci M., Blasco M., Chiorino G., Bauwens S., Horard B., Gilson E., Stoppacciaro A., Zupi G., Biroccio A. (2007) Telomere damage induced by the G-quadruplex ligand RHPS4 has an antitumor effect. J. Clin. Investig. 117, 3236–3247.
Salvati E., Botta L., Amato J., Di Leva F.S., Zizza P., Gioiello A., Pagano B., Graziani G., Tarsounas M., Randazzo A., Novellino E., Biroccio A., Cosconati S. (2017) Lead discovery of dual G-quadruplex stabilizers and poly(ADP-ribose) polymerases (PARPs) inhibitors: a new avenue in anticancer treatment. J. Med. Chem. 60, 3626–3635.
Sengar A., Vandana J.J., Chambers V.S., Di Antonio M., Winnerdy F.R., Balasubramanian S., Phan A.T. (2019) Structure of a (3+1) hybrid G-quadruplex in the PARP1 promoter. Nucl. Acids Res. 47, 1564–1572.
Day T.A., Layer J.V., Cleary J.P., Guha S., Stevenson K.E., Tivey T., Kim S., Schinzel A.C., Izzo F., Doench J., Root D.E., Hahn W.C., Price B.D., Weinstock D.M. (2017) PARP3 is a promoter of chromosomal rearrangements and limits G4 DNA. Nat. Commun. 8, 15110.
Angelov D., Bondarenko V.A., Almagro S., Menoni H., Mongelard F., Hans F., Mietton F., Studitsky V.M., Hamiche A., Dimitrov S., Bouvet P. (2006) Nucleolin is a histone chaperone with FACT-like activity and assists remodeling of nucleosomes. EMBO J. 25, 1669–1679.
Hanakahi L.A., Dempsey L.A., Li M.J., Maizels N. (1997) Nucleolin is one component of the B cell-specific transcription factor and switch region binding protein, LR1. Proc. Natl. Acad. Sci. USA. 94, 3605–3610.
Serin G., Joseph G., Faucher C., Ghisolfi L., Bouche G., Amalric F., Bouvet P. (1996) Localization of nucleolin binding sites on human and mouse pre-ribosomal RNA. Biochimie. 78, 530–538.
Gonzalez V., Guo K., Hurley L., Sun D. (2009) Identification and characterization of nucleolin as a c-myc G-quadruplex-binding protein. J. Biol. Chem. 284, 23622–23635.
Miranti C.K., Moore S., Kim Y., Chappeta V.R., Wu K., De B., Gokhale V., Hurley L.H., Reyes-Reyes E.M. (2020) Nucleolin represses transcription of the androgen receptor gene through a G-quadruplex. Oncotarget. 11, 1758–1776.
Haeusler A.R., Donnelly C.J., Periz G., Simko E.A., Shaw P.G., Kim M.S., Maragakis N.J., Troncoso J.C., Pandey A., Sattler R., Rothstein J.D., Wang J. (2014) C9orf72 nucleotide repeat structures initiate molecular cascades of disease. Nature. 507, 195–200.
Lago S., Tosoni E., Nadai M., Palumbo M., Richter S.N. (2017) The cellular protein nucleolin preferentially binds long-looped G-quadruplex nucleic acids. Biochim. Biophys. Acta, Gen. Subj. 1861, 1371–1381.
Berger C.M., Gaume X., Bouvet P. (2015) The roles of nucleolin subcellular localization in cancer. Biochimie. 113, 78–85.
Lista M.J., Martins R.P., Billant O., Contesse M.A., Findakly S., Pochard P., Daskalogianni C., Beauvineau C., Guetta C., Jamin C., Teulade-Fichou M.P., Fahraeus R., Voisset C., Blondel M. (2017) Nucleolin directly mediates Epstein–Barr virus immune evasion through binding to G-quadruplexes of EBNA1 mRNA. Nat. Commun. 8, 16043.
Wang S.R., Min Y.Q., Wang J.Q., Liu C.X., Fu B.S., Wu F., Wu L.Y., Qiao Z.X., Song Y.Y., Xu G.H., Wu Z.G., Huang G., Peng N.F., Huang R., Mao W.X., Peng S., Chen Y.Q., Zhu Y., Tian T., Zhang X.L., Zhou X. (2016) A highly conserved G-rich consensus sequence in hepatitis C virus core gene represents a new anti-hepatitis C target. Sci. Adv. 2, e1501535.
Boulant S., Vanbelle C., Ebel C., Penin F., Lavergne J.P. (2005) Hepatitis C virus core protein is a dimeric alpha-helical protein exhibiting membrane protein features. J. Virol. 79, 11353–11365.
Bian W.X., Xie Y., Wang X.N., Xu G.H., Fu B.S., Li S., Long G., Zhou X., Zhang X.L. (2019) Binding of cellular nucleolin with the viral core RNA G-quadruplex structure suppresses HCV replication. Nucl. Acids Res. 47, 56–68.
Tosoni E., Frasson I., Scalabrin M., Perrone R., Butovskaya E., Nadai M., Palu G., Fabris D., Richter S.N. (2015) Nucleolin stabilizes G-quadruplex structures folded by the LTR promoter and silences HIV-1 viral transcription. Nucl. Acids Res. 43, 8884–8897.
Ho J.S., Ma W., Mao D.Y., Benchimol S. (2005) p53-Dependent transcriptional repression of c-myc is required for G1 cell cycle arrest. Mol. Cell. Biol. 25, 7423–7431.
Petr M., Helma R., Polaskova A., Krejci A., Dvorakova Z., Kejnovska I., Navratilova L., Adamik M., Vorlickova M., Brazdova M. (2016) Wild-type p53 binds to MYC promoter G-quadruplex. Biosci. Rep. 36, e00397.
Adamik M., Kejnovska I., Bazantova P., Petr M., Renciuk D., Vorlickova M., Brazdova M. (2016) p53 binds human telomeric G-quadruplex in vitro. Biochimie. 128-129, 83–91.
Chittock E.C., Latwiel S., Miller T.C.R., Müller C.W. (2017) Molecular architecture of polycomb repressive complexes. Biochem. Soc. Trans. 45, 193–205.
Hanna R., Flamier A., Barabino A., Bernier G. (2021) G-quadruplexes originating from evolutionary conserved L1 elements interfere with neuronal gene expression in Alzheimer’s disease. Nat. Commun. 12, 1828.
Deckard C.E., Sczepanski J.T. (2018) Polycomb repressive complex 2 binds RNA irrespective of stereochemistry. Chem. Commun. 54, 12061–12064.
Kaneko S., Son J., Shen S.S., Reinberg D., Bonasio R. (2013) PRC2 binds active promoters and contacts nascent RNAs in embryonic stem cells. Nat. Struct. Mol. Biol. 20, 1258–1264.
Wang X., Paucek R.D., Gooding A.R., Brown Z.Z., Ge E.J., Muir T.W., Cech T.R. (2017) Molecular analysis of PRC2 recruitment to DNA in chromatin and its inhibition by RNA. Nat. Struct. Mol. Biol. 24, 1028–1038.
Kaneko S., Son J., Bonasio R., Shen S.S., Reinberg D. (2014) Nascent RNA interaction keeps PRC2 activity poised and in check. Genes Dev. 28, 1983–1988.
Harkness R.W.T., Mittermaier A.K. (2017) G-quadruplex dynamics. Biochim. Biophys. Acta, Proteins Proteomics. 1865, 1544–1554.
Rhodes D., Lipps H.J. (2015) G-quadruplexes and their regulatory roles in biology. Nucl. Acids Res. 43, 8627–8637.
Sun Z.Y., Wang X.N., Cheng S.Q., Su X.X., Ou T.M. (2019) Developing novel G-quadruplex ligands: from interaction with nucleic acids to interfering with nucleic acid-protein interaction. Molecules. 24, 396.
Jain N., Mishra S.K., Shankar U., Jaiswal A., Sharma T.K., Kodgire P., Kumar A. (2020) G-quadruplex stabilization in the ions and maltose transporters gene inhibit Salmonella enterica growth and virulence. Genomics. 112, 4863–4874.
Calvo E.P., Wasserman M. (2016) G-quadruplex ligands: potent inhibitors of telomerase activity and cell proliferation in Plasmodium falciparum. Mol. Biochem. Parasitol. 207, 33–38.
Artusi S., Nadai M., Perrone R., Biasolo M.A., Palu G., Flamand L., Calistri A., Richter S.N. (2015) The herpes simplex virus-1 genome contains multiple clusters of repeated G-quadruplex: implications for the antiviral activity of a G-quadruplex ligand. Antiviral Res. 118, 123–131.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


