Молекулярная биология, 2021, T. 55, № 5, стр. 846-857
Репортерная система люцифераза-EGFP для оценки метилирования ДНК в клетках млекопитающих
X. X. Wang a, *, H. J. Jia b, Y. R. Lv a, b, H. H. Sun a, b, X. L. Wei c, J. Y. Tan c, Z. Z. Jing b, **
a School of Public Health, Lanzhou University
730000 Lanzhou, China
b State Key Laboratory of Veterinary of Etiological Biology, Key Laboratory of Veterinary Public Health of Agricultural Ministry, Lanzhou Veterinary Research Institute, Chinese Academy of Agricultural Sciences
730000 Lanzhou, China
c Institute of Immunology, School of Basic Medical Sciences, Lanzhou University
730000 Lanzhou, China
* E-mail: wangxiaoxia@lzu.edu.cn
** E-mail: jingzhizhong@caas.cn
Поступила в редакцию 06.06.2020
После доработки 22.10.2020
Принята к публикации 03.11.2020
Аннотация
Метилирование – эпигенетическая модификация ДНК, играющая важную роль во многих биологических процессах. Представлена система pLTR-Luc2P-EGFP для оценки метилирования ДНК в клетках млекопитающих. В этой системе экспрессия репортерного гена люциферазы Luc2P-EGFP находится под контролем 5'-длинного концевого повтора (LTR) HIV-1, который содержит несколько CpG-сайтов. Метилирование этих CpG-сайтов приводит к прекращению экспрессии Luc2P-EGFP, что можно визуализировать с помощью флуоресцентной микроскопии с последующей количественной оценкой в тесте определения люциферазной активности. Для проверки действия системы конструкцию pLTR-Luc2P-EGFP метилировали in vitro, трансфицировали в линию клеток 293T и наблюдали снижение экспрессии Luc2P-EGFP. Уровень метилирования ДНК определяли, используя предварительно смешанные образцы репортерной ДНК с уровнями метилирования от 0 до 100%. Показано, что точность определения люциферазной активности превышает точность вестерн-блотинга против EGFP. Анализ методом Bland–Altman показал, что результаты определения люциферазной активности хорошо согласуются с фактическими уровнями метилирования ДНК. Нами создана репортерная система, способная эффективно и надежно выявлять количественные изменения в метилировании ДНК в клетках млекопитающих. Эту систему можно использовать в качестве инструмента для высокопроизводительного скрининга молекул, модулирующих метилирование ДНК.
ВВЕДЕНИЕ
Эпигенетика описывает фенотипические изменения, которые влияют на экспрессию генов, не затрагивая нуклеотидные последовательности ДНК [1]. Эпигенетические изменения, в число которых входят метилирование ДНК, модификации гистонов и микроРНК, наследуемы и обратимы [1, 2]. С тех пор, как более 40 лет назад Riggs A.D. [3] и Holliday R. [4] предложили убедительную модель молекулярного механизма наследования, метилирование ДНК рассматривали как парадигму передачи эпигенетической информации [5]. В настоящее время очевидно, что метилирование ДНК ‒ это распространенная эпигенетическая модификация, которая играет важную регуляторную роль в таких процессах, как сайленсинг транспозонов, геномный импринтинг, инактивация Х-хромосомы и процессы развития [6, 7]. Нарушения метилирования часто ассоциированы с повышенной частотой возникновения некоторых заболеваний [8].
В клетках млекопитающих метилирование в основном происходит по С5 цитозинового кольца (5mC) [9–11]. Метилцитозин содержится преимущественно в CpG-динуклеотидах, которые составляют только 1% генома человека. Однако богатые CpG участки, так называемые CpG-островки, расположены в промоторных областях более 70% всех известных генов человека [12–14]. 5mC, особенно кластеризованные в сайтах CpG, являются важными сайленсерами транскрипции c промоторов генов и эндогенных ретротранспозонов в геноме [15–17]. Многие исследования показали, что эпигенетический сайленсинг множества генов, вызванный гиперметилированием CpG-островков, ассоциированных с промотором, часто связан с развитием конкретных заболеваний [18–20]. В связи с этим разработка простого, надежного и чувствительного метода обнаружения метилирования ДНК и его изменений (например, гипер- или гипометилирования) представляет большой интерес.
Традиционные методы анализа метилирования ДНК основаны преимущественно на обработке бисульфитом натрия, при которой неметилированный цитозин превращается в урацил, в то время как метилированный цитозин устойчив к бисульфиту и не подвергается превращению [21]. Это позволяет различить метилированную и неметилированную ДНК, для чего используют метил-специфичную ПЦР, секвенирование ДНК или комбинированный бисульфитный рестрикционный анализ. Благодаря своей надежности и точности эти методы широко используются для количественной оценки сайт-специфического метилирования ДНК. Однако они требуют проведения таких сложных процедур, как клонирование и секвенирование, что ограничивает их использование в высокопроизводительном анализе [22, 23]. Вместе с тем, разработаны методы, основанные на высокоэффективной жидкостной хроматографии [24], ПЦР с рестрикционными ферментами [25] и газовая хроматография/масс-спектрометрия [26]. Эти методы сложны, трудоемки и дороги. В связи с этим возникает потребность в более удобном и простом методе определения метилирования ДНК.
Люциферазы, выделенные из некоторых видов животных, – это ферменты, катализирующие реакцию, которая сопровождается испусканием света (биолюминесценцией). Люцифераза часто используется в качестве репортера для мониторинга активности гена/промотора гена из-за ее высокой чувствительности, надежности сигнала и удобства анализа. К примеру, Sanchez и соавт. использовали репортерный ген люциферазы для анализа взаимосвязи между структурой и биологической активностью стриголактонов [27]. Solberg и соавт. с помощью люциферазного теста охарактеризовали активность 5′-фланкирующего промотора гена Tcf3 мыши [28]. Более того, комбинация двух люцифераз – светлячковой и Renilla – повысила точность и воспроизводимость анализа; при этом люцифераза Renilla служит внутренним контролем при мониторинге количества и жизнеспособности клеток. Таким образом, репортерный люциферазный тест можно рассматривать как мощный инструмент для изучения регуляторных элементов генов. Так, для изучения регуляции транскрипции гена Sox2 человека создан репортерный люциферазный тест на основе клеточной линии 293-Sox2-Luciferase [29]. Недавно для тестирования перекрестной нейтрализации коронавирусов сыворотками пациентов с SARS и COVID-19 получен репортерный люциферазный вирус icSARS-CoV-2-nLuc-GFP [30].
Теория эпигенетической регуляции предполагает, что активность промотора согласуется с уровнем метилирования ДНК [31, 32]. Поэтому уровень люциферазной активности в дальнейшем использовали в качестве показателя статуса метилирования и/или изменений метилирования промотора гена. Так, по уровню активности люциферазы светлячка оценили влияние Gadd45a на метилирование ДНК [33]. Определение метилирования участков CpG с использованием плазмиды, содержащей HIV LTR-люциферазу, упомянуто в небольшом отчете [34]. Однако количественную точность и надежность детекции, включая дозовую зависимость между уровнями метилирования и активностью люциферазы, еще предстоит оценить.
Чтобы полностью охарактеризовать свойства репортерной системы на основе люциферазы, в качестве репортера для мониторинга активности промотора мы использовали модифицированный ген люциферазы светлячка (Luc2P), слитый с EGFP, и создали клеточную систему для измерения изменений метилирования ДНК. Результат анализа активности люциферазы подтвержден путем сравнения с данными, полученными с помощью анализа чувствительности к HpaΙΙ [35] и вестерн-блотинга [36, 37]. Нами доказано, что эта репортерная система позволяет надежно и точно измерять изменения метилирования ДНК в живых клетках.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Конструкции ДНК. Экспрессионную плазмиду pEGFP-N1, приобретенную у “BD Biosciences Clontech” (CША), использовали в качестве основы для конструирования pLTR-Luc2P-EGFP. 5'‑LTR HIV-1 амплифицировали с помощью ПЦР из HIV-1 pNL4.3 и использовали для замены цитомегаловирусного промотора (CMV) в плазмиде pEGFP-N1 по сайтам AseI и NheI. Luc2P амплифицировали из pGL4.32 [luc2P/NF-kB-RE/Hygro] (“Promega”, США) и клонировали в pEGFP-N1 по сайтам HindIII и BamHI. Правильность структуры плазмиды pLTR-Luc2P-EGFP проверена секвенированием. Для ПЦР-амплификации использовали праймеры:
5'-AseI-LTR – TCGTATTAATTGGAAGGGCTAATTTGGTC;
3'-NheI-LTR – CTAGCTAGCTGCTAGAGATTTTCCACACTGAC;
5'-HindIII-luc2P – CCCAAGCTTATGGAAGATGCCAAAAACATTA;
3'-BamHI-luc2P – CGGGATCCGACGTTGATCCTGGCGCTGG.
Метилирование ДНК плазмиды in vitro. Плазмиду pLTR-Luc2P-EGFP обрабатывали CpG-метилазой M.SssI (“Zymo Res”, США). Плазмиду (2 мкг) инкубировали с 0.6 мМ S-аденозилметионина и 4 ед. M.SssI при 30°C в течение 6 ч. Затем в реакционную систему добавляли 2 ед. M.SssI и продолжали инкубировать при 30°C в течение 6–8 ч. Обработанную плазмиду концентрировали с использованием 3 М ацетата натрия. Полное CpG-метилирование плазмиды pLTR-Luc2P-EGFP подтверждено расщеплением эндонуклеазами (HpaII и MspI, “Thermo Fisher Scientific”, США) и бисульфит-опосредованным картированием остатков метилцитозина.
Бисульфит-опосредованное картирование метилцитозина. После бисульфитной конверсии (“Zymo Res.”) интересующие области плазмиды pmeLTR-Luc2P-EGFP, обработанной M.SssI, и неметилированной плазмиды pLTR-Luc2P-EGFP амплифицировали с помощью ПЦР (праймеры для LTR: LTR-BSP-F, 5'-TATGAGTTAGTATGGATGGG-3', и LTR-BSP-R, 5'-AATCTAACCAAAAAAACCCAATACA-3'). Продукты ПЦР выделяли из геля с помощью набора для экстракции ДНК (“Axygen”, США) и клонировали в вектор pJET1.2 для секвенирования. Данные анализировали с помощью онлайн-сервиса программного обеспечения QUantification Tool for Methylation Analysis (http://quma.cdb.riken.jp/) [38].
Культуры клеток и транзиентная трансфекция. Клетки линий 293T (эмбриональная почка человека, HEK) и HeLa культивировали в среде DMEM (“Gibco”, США) с добавлением 100 ед./мл пенициллина, 0.1 мг/мл стрептомицина и 10% фетальной сыворотки крупного рогатого скота (FBS, “Gibco”). Клетки культивировали при 37°C в инкубаторе с содержанием 5% CO2.
Клетки 293Т для проведения транзиентной трансфекции высевали в 12-луночные планшеты. По достижении 80% монослоя клетки трансфицировали неметилированной плазмидой pLTR-Luc2P-EGFP или метилированной pmeLTR-Luc2P-EGFP с использованием реагента FuGENE® HD (“Promega”), следуя протоколу производителя. Контрольный вектор pGL4.74 с геном люциферазы Renilla [hRluc/TK] (“Promega”) котрансфицировали с репортерным вектором в соотношении 1 : 10.
Иммунофлуоресцентный анализ. Для определения 5mC клетки HeLa, выращенные на покровных стеклах, трансфицировали неметилированной и метилированной плазмидой pLTR-Luc2P-EGFP (0.2 мкг/лунку) в течение 36 ч и фиксировали в 4%-ном параформальдегиде в фосфатно-солевом буфере (PBS) в течение 15 мин при комнатной температуре. Клетки трижды промывали PBS и для блокирования инкубировали в PBS, содержащем 5% FBS и 0.3% Triton X-100, затем инкубировали в течение ночи при 4°C с моноклональными антителами к 5mC (1 : 100, “EpiGentek”, США). На следующий день после повторного выдерживания при комнатной температуре в течение 1 ч клетки трижды промывали PBS и инкубировали с антителами козы к IgG (H + L) мыши, конъюгированными с красителем Alexa Fluor 555 (1 : 500, “Thermo Fisher”) в течение 1 ч при 37°C. После трех отмывок в PBS клетки инкубировали с раствором DAPI (“Beyotime”, Китай) в течение 3–5 мин. Образцы промывали PBS, затем помещали на предметные стекла со средой для фиксации флуоресценции (“Beyotime”) и исследовали с помощью конфокального микроскопа (“Leica Inc”, Германия).
Для обнаружения EGFP клетки 293T исследовали через 36–48 ч после трансфекции с помощью флуоресцентного инвертированного микроскопа Nikon (Nikon ECLIPSE Ti, Япония), изображения фотографировали и обрабатывали с помощью NIS-Elements D.
Вестерн-блотинг. Через 48 ч после трансфекции клетки линии 293Т лизировали на льду в буфере RIPA с добавлением ингибиторов протеаз (“Beyotime”). Лизаты фракционировали с помощью электрофореза в денатурирующем полиакриламидном геле (SDS-PAGE) и переносили на нитроцеллюлозную (NC) мембрану (“Millipore”, США). Мембраны блокировали 5%-ным обезжиренным сухим молоком в PBS, инкубировали с антителами кролика к GFP (разведение 1 : 400 в PBS, “Santa Cruz”, США) и снова инкубировали со вторичными антителами козы к IgG кролика, конъюгированными с пероксидазой хрена (IgG-HRP, разведение 1 : 10 000 в PBS, “Elabscience”, США), иммуноблотинг GAPDH выполняли с антителом к GAPDH человека (1 : 10 000, “Sigma”, США) в качестве контроля. Детектировали с использованием усиленной хемилюминесценции (“Bio-Rad”, США).
Анализ активности люциферазы. Активность люциферазы светлячков и Renilla измеряли с использованием системы анализа репортеров люциферазы Dual-Glo® (“Promega”). Через 48 ч после трансфекции в лунки планшета добавляли реагент Dual-Glo® в объеме, равном объему культуральной среды. Планшет переворачивали в течение 20 мин для достижения полного лизиса. Люминесценцию люциферазы светлячка (Fluc) измеряли на люминометре для микропланшетов GloMax®-96 (“Promega”). Затем добавляли равное количество реагента Dual-Glo® Stop & Glo® и через 20 мин измеряли люминесценцию Renilla (Rluc). Относительную активность люциферазы (RLA, Relative Luciferase Activity) рассчитывали по соотношению активностей Fluc/Rluc. Относительный коэффициент ответа (RRR, Relative Response Ratio) рассчитывали по формуле [(экспериментальный RLA) – (RLA клеток, трансфицированных полностью метилированной ДНК)]/[(RLA клеток, трансфицированных неметилированной ДНК)-(RLA клеток, трансфицированных полностью метилированной ДНК)].
Анализ чувствительности к HpaII. Уровни CpG-метилирования 5'-LTR в pLTR-Luc2P-EGFP определяли с помощью количественной ПЦР в реальном времени (кПЦР) после расщепления HpaII, которое блокируется метилированием CpG [35]. Суммарную ДНК выделяли из трансфицированных клеток с помощью набора Quick-DNATM Miniprep Plus (“Zymo”). ДНК (0.5 мкг) инкубировали с 10 ед. HpaII или в контрольной реакции без HpaII при 37°C в течение 4 ч и инактивировали в течение 20 мин при 80°C. В количественной ПЦР с SYBR Premix Ex Taq (Tli RNaseH Plus, “Takara”, Китай) использовали равные количества как в контрольной реакции, так и в реакции с добавлением HpaII. Затем амплифицировали 5'-LTR, фланкирующий два сайта расщепления HpaII. В качестве внутреннего стандарта использовали конститутивный ген GAPDH. ПЦР проводили на приборе Applied Biosystems 7500 Real-Time PCR в следующих условиях: 95°C в течение 30 с, затем 40 циклов – 95°C, 5 с и 60°C, 30 с. Последовательности праймеров для 5'-LTR: прямой – GCCAATGAAGGAGAGAACAACA и обратный – AGCGGAAAGTCCCTTGTA; для GAPDH: прямой – GAAGGTGAAGGTCGGAGTCAAC и обратный – CAGAGTTAAAAGCAGCCCTGGT. Реакцию проводили в трех повторностях. Чувствительность сайта CCGG к HpaII рассчитывали как [1–2Ct (пустой) – Ct (HpaII)] ×100%.
Статистический анализ. Данные представлены как среднее значение ± стандартное отклонение, вычисленное по данным по крайней мере двух независимых экспериментов; каждое повторение состоит не менее чем из трех технических повторов. Двусторонний t-критерий Стьюдента и однофакторный дисперсионный анализ (ANOVA) выполнены с использованием SPSS (версия 21, IBM). Статистически значимым считали P < 0.05.
Гистограмма получена с помощью Graphpad Prism 6 для наиболее наглядного отображения данных. Стандартную кривую строили с помощью Excel 2013, чтобы показать корреляцию между результатами трех анализов метилирования ДНК и стандартом. График Bland–Аltman получен с помощью программного обеспечения MedCalc для оценки соответствия трех анализов метилирования ДНК.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Конструирование и метилирование репортерного вектора pLTR-Luc2P-EGFP
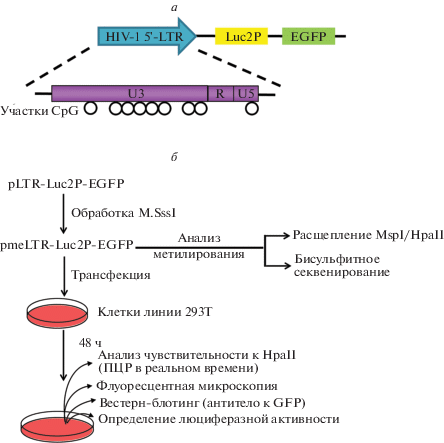
В качестве основы для создания репортерного вектора использовали pEGFP-N1. Модифицированную люциферазу светлячка Luc2P встраивали в сайт множественного клонирования, в качестве репортера использовали слитый белок Luc2P-EGFP. Исходный промотор CMV был заменен на 5'-LTR HIV-1, который содержит восемь сайтов CpG и легко метилируется [34, 39] (рис. 1а). После метилирования in vitro с помощью M.SssI вектор pmeLTR-Luc2P-EGFP может быть доставлен в клетки млекопитающих. Затем статус метилирования промотора 5'-LTR можно отслеживать, анализируя чувствительность к HpaII (ПЦР в реальном времени) прямо [35] или косвенно за счет измерения уровня экспрессии репортерного гена Luc2P-EGFP. В трансфицированных клетках экспрессию Luc2P-EGFP можно оценивать качественно с помощью флуоресцентной микроскопии, полуколичественно – с помощью вестерн-блотинга (антитела к GFP) или определять количественно по активности люциферазы (рис. 1б).
Рис. 1.
Описание репортерного вектора и схема эксперимента. а – Схематическое изображение конструкции pLTR-Luc2P-EGFP. Показаны ключевые компоненты, в которых экспрессия слитого белка Luc2P-EGFP находится под контролем промотора 5'-LTR HIV-1. б – Схематическая иллюстрация экспериментальных процедур.
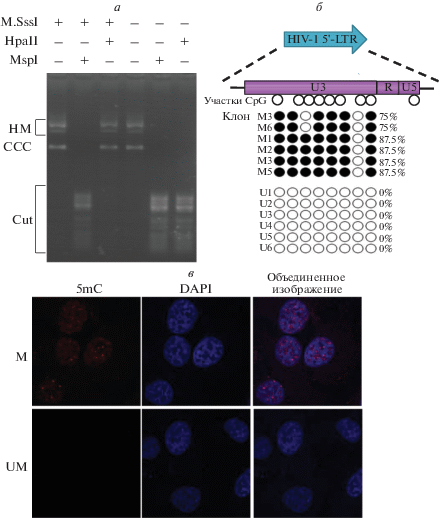
Вектор pmeLTR-Luc2P-EGFP после обработки M.SssI анализировали с использованием рестриктаз HpaII/MspI. Оба фермента распознают один и тот же сайт CCGG, однако расщепление HpaII может быть заблокировано метилированием CpG [40]. На рис. 2а приведена картина расщепления метилированной и неметилированной ДНК рестриктазой MspI, показывающая присутствие сайтов CCGG в векторе pLTR-Luc2P-GFP. Однако устойчивой к действию HpaII была только ДНК, обработанная M.SssI, что указывает на полное метилирование CpG-сайтов в pmeLTR-Luc2P-EGFP после обработки M.SssI. Эффективность метилирования подтверждена также бисульфитным секвенированием области LTR. Как показано на рис. 2б, в четырех из шести плазмидных клонов, обработанных M.SssI (M1–M6), степень метилирования CpG-сайтов составляла 87.5%, а в остальных двух – 75%. Напротив, во всех шести неметилированных клонах (U1–U6) метилированы были 0% сайтов CpG. Затем pmeLTR-Luc2P-EGFP трансфицировали в клетки линии HeLa, 5mC определяли с помощью иммунофлуоресцентного анализа. В клетках, трансфицированных неметилированной ДНК, 5mC не обнаружен (рис. 2в, нижняя панель). В клетках, трансфицированных pmeLTR-Luc2P-EGFP, 5mC обнаружен и в основном локализован в ядре (рис. 2в, верхняя панель). По-видимому, pmeLTR-Luc2P-EGFP остается метилированным и стабильным после доставки в живые клетки.
Рис. 2.
Анализ метилирования ДНК, опосредованного M.SssI. а – Расщепление ДНК рестрикционной эндонуклеазой. Метилированные (M.SssI+) и неметилированные (M.SssI-) расщепляли ДНК-субстраты pLTR-Luc2P-GFP, как показано. HM, CCC и Cut – более высокая масса, ковалентно замкнутая кольцевая и расщепленная плазмида соответственно. б – Бисульфит-опосредованное картирование pLTR-Luc2P-GFP. Каждая строка представляет собой молекулу ДНК, независимо клонированную из метилированных (M1–M6) или неметилированных (U1–U6) клонов pLTR-Luc2P-EGFP. Каждый столбец представляет один CpG-сайт в области LTR. Белые кружки указывают на неметилированные CpG-сайты, закрашенные – на метилированные. Уровень метилирования ппоказан как процент метилированных CpG в каждом клоне. в – Иммунофлуоресцентный анализ 5mC в метилированных (M) или неметилированных (UM) клетках, трансдуцированных pLTR-Luc2P-EGFP. Клетки HeLa, трансдуцированные репортерной плазмидой, окрашивали антителом к 5mC и DAPI. Показаны репрезентативные результаты трех экспериментов.
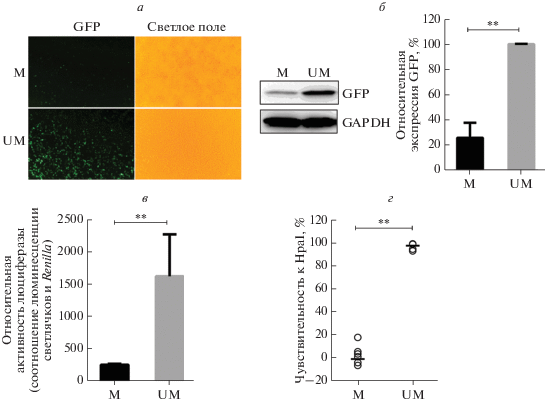
Оценка репортерной системы
Для оценки репортерной системы равные количества репортерных плазмид, обработанных или необработанных M.SssI, вводили в клетки линии 293T. Через 48 ч после трансфекции клетки анализировали согласно схеме, представленной на рис. 1б. Методами флуоресцентной микроскопии и вестерн-блотинга показано более сильное подавление экспрессии Luc2P-GFP в клетках, трансфицированных pmeLTR-Luc2P-EGFP, чем в клетках, трансфицированных неметилированной плазмидой (рис. 3а, б). Аналогичный результат получен с помощью анализа активности люциферазы, т.е. относительная активность люциферазы в клетках, трансфицированных pmeLTR-Luc2P-EGFP, была значительно снижена (рис. 3в, P < 0.01). Более того, чувствительность ДНК, выделенной из клеток, трансфицированных pmeLTR-Luc2P-EGFP, к HpaII была значительно снижена (рис. 3г, P < 0.01), т.е. подавление экспрессии гена коррелирует с метилированием промотора. Таким образом, показано, что эти три метода (обнаружение EGFP, Luc2P, 5mC в области LTR) позволяют отличать метилированные образцы ДНК от неметилированных.
Рис. 3.
Оценка репортерной системы pLTR-Luc2P-EGFP в клетках линии 293T. а – Изображение флуоресценции Luc2P-EGFP, полученное методом флуоресцентной микроскопии. Интенсивность флуоресценции EGFP в клетках линии 293T, трансфицированных репортерными векторами M или UM (слева). Клетки высевали примерно с одинаковой плотностью, о чем свидетельствуют данные фазово-контрастной микроскопии (справа). б – Вестерн-блотинг Luc2P-EGFP в клетках, трансдуцированных векторами M и UM. Репрезентативное изображение блота получено из трех экспериментов. На гистограмме представлены данные, определенные по относительной интенсивности флуоресценции с использованием анализа изображений (среднее ± стандартное отклонение из трех экспериментов. **P < < 0.01, t-критерий Стьюдента). в – Относительная активность люциферазы в клетках линии 293T, трансфицированных плазмидами M и UM. Эксперименты проводили 3 раза, по три повторности каждый. Данные представлены как среднее ± стандартное отклонение. **P < 0.01, t-критерий Стьюдента. г – Чувствительность области LTR к HpaII в клетках, трансфицированных плазмидами M и UM. Открытые кружки – данные отдельного эксперимента, короткая линия – средние значения. **P < 0.01, t-критерий Стьюдента.
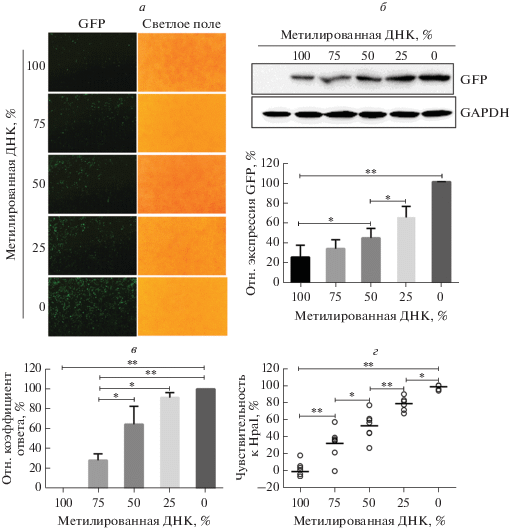
Количественная оценка статуса метилирования с помощью репортерной системы
Позволяет ли наша система количественно определять метилирование ДНК? Чтобы понять это, мы провели эксперимент с разведением. Полностью метилированную плазмиду (pmeLTR-Luc2P-EGFP) принимали за 100%, а неметилированную (pLTR-Luc2P-EGFP) – за 0%. Плазмиды смешивали в различных соотношениях (3 : 1, 2 : 2, 1 : 3) с получением 75, 50 и 25% метилированной плазмиды. Смесью ДНК с разными градиентами метилирования (100, 75, 50, 25, 0%) трансфицировали клетки линии 293Т и определяли статус метилирования. Как следует из рис. 4, снижение уровней метилирования приводит к постепенному увеличению экспрессии Luc2P-GFP; на это указывает интенсивность флуоресценции, данные вестерн-блотинга, относительный коэффициент ответа (RRR) люциферазного теста. Та же тенденция показана при анализе чувствительности к HpaII. Тем самым мы подтвердили, что представленная репортерная система может найти применение в количественных измерениях метилирования ДНК.
Рис. 4.
Количественная оценка точности репортерной системы. Клетки линии 293T, трансфицированные pLTR-Luc2P-EGFP, метилированными на 100, 75, 50, 25, 0%, изучали с помощью флуоресцентной микроскопии (а), вестерн-блотинга (б), определения люциферазной активности, представленного в виде относительного коэффициента ответа (в) и анализа чувствительности к HpaII (г). Эксперименты проводили 3 раза, культуры получали в трех повторностях. Результаты представлены как среднее ± стандартное отклонение. Репрезентативное флуоресцентное изображение и изображение блота получены из результатов трех экспериментов. Светлые кружки (г) – данные отдельных экспериментов, линии – средние значения. *P < 0.05, **P < 0.01, однофакторный дисперсионный анализ (ANOVA).
Оценка точности и надежности методов определения метилирования ДНК
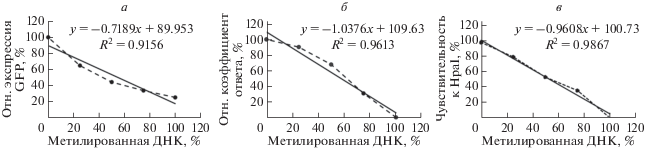
Чтобы оценить точность методов определения метилированния ДНК, проведен линейный регрессионный анализ данных, представленных на рис. 4, отложенных в зависимости от процента метилирования. На рис. 5 показана сильная регрессионная зависимость между нормированными значениями и процентом метилированния ДНК (R2 = 0.9156 для вестерн-блотинга; R2 = 0.9613 для люциферазного теста; R2 = 0.9867 для анализа чувствительности к HpaII), что свидетельствует о хорошей корреляции между измеренным значением и фактическим уровнем метилирования.
Рис. 5.
Анализ линейной регрессии между уровнем метилирования трансфицированной плазмиды и нормированными значениями, полученными методом вестерн-блотинга (а), определения люциферазной активности (б) и чувствительности к HpaII (в), представленными на рис. 4. Сплошной кружок – средние значения, прямая линия – соответствующая стандартная кривая, пунктиром показана связь между фактическими значениями. Приведено уравнение кривой линейной регрессии и коэффициент корреляции (R2).
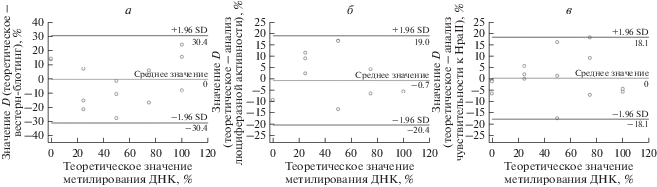
Соответствие результатов измерений проанализировано также с помощью графика Bland–Altman [41, 42]. Все значения, полученные с помощью вестерн-блотинга, люциферазного теста и анализа чувствительности к HpaII, находятся рамках соответствия, представленного на рис. 6; в эти рамки попадают все 100% различий. Считается, что если в пределы соответствия попадают более 95% различий, то соответствие считают хорошим [42]. Таким образом, результаты, полученные с помощью трех методов, хорошо соответствуют фактическому уровню 5mC pLTR-Luc2P-EGFP.
Рис. 6.
Графики Bland–Altman для вестерн-блотинга (а), анализа люциферазной активности (б) и анализа чувствительности к HpaII (в). Значения D представляют разницу между полученными результатами и соответствующим процентом метилирования. Пунктирные линии соответствуют среднему значению (%), сплошные линии – среднему значению (%) ± 1.96 стандартного отклонения, полые кружки – различия в величинах D.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Нами представлена репортерная система, позволяющая оценивать модификации 5mC в клетках млекопитающих вместе с точной и надежной визуализацией и количественным определением событий метилирования, включая ПЦР в реальном времени, вестерн-блотинг и анализ люциферазной активности. Первые два метода, несмотря на то что для них требуются только реактивы, доступные в молекулярно-биологических лабораториях, более сложны и трудоемки, чем люциферазный тест. Для анализа люциферазной активности требуется специальное оборудование и реагенты, однако отсутствует этап подготовки образца. Таким образом, это простой, быстрый, высоко чувствительный метод, позволяющий получать точные результаты с минимальными усилиями.
Люциферазу впервые обнаружили у светлячка (Photinus pyralis) в 1985 г. В настоящее время метод определения люциферазы широко используется в науке и промышленности [43]. Репортерные системы, содержащие люциферазу, применяют в функциональной геномике (например, в скрининге РНК-интерференции) для изучения сигнальных путей, молекулярных механизмов и определения биологической активности. Начиная с 1987 г., термин “люциферазный репортерный тест” встретился более чем в 20 000 публикаций [44]. Репортерные гены люциферазы обладают следующими важными характеристиками: исключительной чувствительностью (в 10–1000 раз большей, чем у таких флуоресцентных репортеров, как GFP), широким динамическим диапазоном, отсутствием эндогенной активности в клетках-хозяевах, мешающей, как правило, количественному определению, и моментальным измерением [45].
Таким образом, мы использовали двойной люциферазный тест для оценки метилирования 5'-LTR HIV-1 в трансфицированных клетках линии 293T. Люцифераза светлячка Luc2P отслеживает транскрипционную активность промотора LTR, в то время как люцифераза Renilla служит внутренним контролем для минимизации изменчивости из-за ошибок пипетирования, разной жизнеспособности клеток и эффективности трансфекции [46]. Соотношение двух сигналов люциферазы (относительная активность люциферазы) является эквивалентом относительной экспрессии Luc2P-EGFP и косвенно отражает статус метилирования промотора. В нашем исследовании экспрессию Luc2P-EGFP также выявляли с помощью вестерн-блотинга. Показано, что с помощью обоих методов можно измерять уровень экспрессии белка в линейном диапазоне от 0 до 100% метилированной ДНК. Люциферазный тест имеет более высокую точность (R2 = = 0.9613), чем вестерн-блотинг (R2 = 0.9156). В то же время для непосредственного изучения метилирования LTR мы применяли анализ чувствительности к HpaII, который основан на комбинации чувствительной к метилированию рестриктазы HpaII и количественной ПЦР [35, 47]. Показано, что чувствительность к HpaII также отрицательно коррелирует с уровнем метилирования pLTR-Luc2P-EGFP. Анализ Bland–Altman показал, что люциферазный тест сопоставим с анализом чувствительности к HpaII и хорошо согласуется с фактическим уровнем метилирования ДНК. Таким образом, доказано, что определение люциферазной активности вполне может использоваться для детектирования метилирования ДНК в живых клетках.
Более того, простота и чувствительность делает люциферазный тест высокопроизводительным инструментом для поиска новых соединений, которые модулируют метилирование ДНК. Действительно, люциферазные тесты использовали для скрининга противомикробных агентов против Mycobacterium tuberculosis и иммуносупрессоров еще в 1990-х гг. [48, 49]. В дальнейшем подтвердили потенциальную пользу люциферазного теста при проведении скрининговых исследований, поскольку этот метод может не только снизить стоимость скрининга лекарственных средств, но и повысить надежность и предсказуемость результатов [50].
Что касается нашей репортерной системы, в которой экспрессия люциферазы управляется промотором HIV-1, то ее можно использовать для поиска соединений, направленных на реактивацию латентного HIV-1. Латентный период HIV-1 представляет основное препятствие для полного удаления этого вируса из организма инфицированных. Реактивация латентного вируса – это первый шаг в стратегии “бей и убей”, новом направлении в терапии HIV-1 [51]. Показано, что метилирование ДНК в области LTR тесно связано с регуляцией латентности вируса [52]. Гиперметилирование подавляет экспрессию вирусных генов и стабилизирует латентное состояние HIV-1. Соединения, деметилирующие LTR, реактивируют латентный вирус и могут в дальнейшем использоваться для устранения или уменьшения вирусных резервуаров [51, 53]. По этой причине вместо использования HIV-инфицированных клеточных линий или первичных культур клеток, где латентность может быть достигнута с помощью более чем одного механизма, соединения, реактивирующие вирус исключительно через деметилирование ДНК, могут быть проверены в нашей репортерной системе, что также применимо к изучению метилирования LTR в латентном периоде HIV-1. Тем самым можно расширить скрининг соединений против заболеваний, вызванных аномальными изменениями метилирования [54].
В заключение необходимо отметить, что разработана и проверена репортерная система pLTR-Luc2P-EGFP на основе люциферазы. Доказана пригодность и надежность этой системы оценки изменений метилирования. Наше исследование продвигает использование биолюминесценции в области эпигенетики и предлагает новый метод скрининга соединений, метилирующих/деметилирующих ДНК.
Исследование поддержано Национальным фондом естественных наук Китая (№ 81501734, 81803308), Фондом естественных наук провинции Gansu Китая (№ 20JR5RA215), а также Фондом фундаментальных исследований для центральных университетов (№ lzujbky-2015-260).
В настоящей работе не использовали людей или животных в качестве объектов исследования.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kanwal R., Gupta S. (2012) Epigenetic modifications in cancer. Clin. Genet. 81(4), 303–311.
Ellis L., Atadja P.W., Johnstone R.W. (2009) Epigenetics in cancer: targeting chromatin modifications. Mol. Cancer Ther. 8(6), 1409–1420.
Riggs A.D. (1975) X inactivation, differentiation, and DNA methylation. Cytogenet. Cell Genet. 14, 9–25.
Holliday R., Pugh J.E. (1975) DNA modification mechanisms and gene activity during development. Science. 187, 226–232.
Jeltsch A., Jurkowska R.Z. (2014) New concepts in DNA methylation. Trends Biochem. Sci. 39(7), 310–318.
Wu S.C., Zhang Y. (2010) Active DNA demethylation: many roads lead to Rome. Nat. Rev. Mol. Cell Biol. 11(9), 607–620.
Jaenisch R., Bird A. (2003) Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33(Suppl), 245–254.
Nadasi E., Clark J.S., Szanyi I., Varjas T., Ember I., Baliga R., Arany I. (2009) Epigenetic modifiers exacerbate oxidative stress in renal proximal tubule cells. Anticancer Res. 29(6), 2295–2299.
Rodriguez-Paredes M., Esteller M. (2011) Cancer epigenetics reaches mainstream oncology. Nat. Med. 17(3), 330–339.
Zhu J.K. (2009) Active DNA demethylation mediated by DNA glycosylases. Annu. Rev. Genet. 43, 143–166.
Suzuki M.M., Bird A. (2008) DNA methylation landscapes: provocative insights from epigenomics. Nat. Rev. Genet. 9(6), 465–476.
Momparler R.L. (2003) Cancer epigenetics. Oncogene. 22(42), 6479–6483.
Plass C. (2002) Cancer epigenomics. Hum. Mol. Genet. 11(20), 2479–2488.
Tsai A.G., Lu H., Raghavan S.C., Muschen M., Hsieh C.L., Lieber M.R. (2008) Human chromosomal translocations at CpG sites and a theoretical basis for their lineage and stage specificity. Cell. 135(6), 1130–1142.
Varley K.E., Gertz J., Bowling K.M., Parker S.L., Reddy T.E., Pauli-Behn F., Cross M.K., Williams B.A., Stamatoyannopoulos J.A., Crawford G.E., Absher D.M., Wold B.J., Myers R.M. (2013) Dynamic DNA methylation across diverse human cell lines and tissues. Genome Res. 23(3), 555–567.
Baylin S.B., Jones P.A. (2011) A decade of exploring the cancer epigenome – biological and translational implications. Nat. Rev. Cancer. 11(10), 726–734.
Berman B.P., Weisenberger D.J., Aman J.F., Hinoue T., Ramjan Z., Liu Y., Noushmehr H., Lange C.P., van Dijk C.M., Tollenaar R.A., van Den Berg D., Laird P.W. (2011) Regions of focal DNA hypermethylation and long-range hypomethylation in colorectal cancer coincide with nuclear lamina-associated domains. Nat. Genet. 44(1), 40–46.
Xiwei W., Rauch T.A., Xueyan Z., Bennett W.P., Latif F., Krex D., Pfeifer G.P. (2010) CpG island hypermethylation in human astrocytomas. Cancer Res. 70(7), 2718–2727.
Tao L.H., Wang W., Li L., Kramer P.K., Pereira M.A. (2005) DNA hypomethylation induced by drinking water disinfection by-products in mouse and rat kidney. Toxicol. Sci. 87(2), 344–352.
Boultwood J., Wainscoat J.S. (2007) Gene silencing by DNA methylation in haematological malignancies. Br. J. Haematol. 138(1), 3–11.
Fuso A., Scarpa S., Grandoni F., Strom R., Lucarelli M. (2006) A reassessment of semiquantitative analytical procedures for DNA methylation: comparison of bisulfite- and HpaII polymerase-chain-reaction-based methods. Analyt. Biochem. 350(1), 24–31.
Aichinger E., Köhler C. (2010) Bisulphite sequencing of plant genomic DNA. Methods Mol. Biol. 655, 433–443.
Docherty S.J., Davis O.S.P., Haworth C.M.A., Plomin R., Mill J. (2010) DNA methylation profiling using bisulfite-based epityping of pooled genomic DNA. Methods. 52(3), 255–258.
Kuo K.C., McCune R.A., Gehrke C.W., Midgett R., Ehrlich M. (1980) Quantitative reversed-phase high performance liquid chromatographic determination of major and modified deoxyribonucleosides in DNA. Nucl. Acids Res. 8(20), 4763–4776.
Boers R., Boers J., de Hoon B., Kockx C., Ozgur Z., Molijn A., van IjckenW., Laven J., Gribnau J. (2018) Genome-wide DNA methylation profiling using the methylation-dependent restriction enzyme LpnPI. Genome Res. 28(1), 88–99.
Tang Y., Gao X.-D., Wang Y.S., Yuan B.-F., Feng Y.-Q. (2012) Widespread existence of cytosine methylation in yeast DNA measured by gas chromatography/mass spectrometry. Analyt. Chem. 84(16), 7249–7255.
Sanchez E., Artuso E., Lombardi C., Visentin I., Lace B., Saeed W., Lolli M.L, Kobauri P., Ali Z., Spyrakis F., Cubas P., Cardinale F., Prandi C. (2018) Structure-activity relationships of strigolactones via a novel, quantitative in planta bioassay. J. Exp. Botany. 69(9), 2333–2343.
Solberg N., Machon O., Krauss S. (2012) Characterization and functional analysis of the 5'-flanking promoter region of the mouse Tcf3 gene. Mol. Cell. Biochem. 360, 289–299.
Xiao D., Zhang W., Li Y., Liu K., Zhao J., Sun X., Shan L., Mao Q., Xia H. (2016) A novel luciferase knock-in reporter system for studying transcriptional regulation of the human Sox2 gene. J. Biotechnol. 219, 110–116.
Hou Y.J., Okuda K., Edwards C.E., Martinez D.R., Asakura T., Dinnon K.H., 3rd, Kato T., Lee R.E., Yount B.L., Mascenik T.M., Chen G., Olivier K.N., Ghio A., Tse L.V., Leist Sarah R., Gralinski L.E., Schäfer A., Dang H., Gilmore R., Nakano S., Sun L., Fulcher M.L., Livraghi-Butrico A., Nicely N.I.,. Cameron M., Cameron C., Kelvin D.J., de Silva A., Margolis D.M., Markmann A., Bartelt L., Zumwalt R., Martinez F.J., Salvatore S.P., Borczuk A., Tata P.R., Sontake V., Kimple A., Jaspers I., O’Neal W.K., Randell S.H., Boucher R.C., Baric R.S. (2020) SARS-CoV-2 reversegenetics reveals a variable infection gradient in the respiratory tract. Cell. 182(2), 429–446 e414.
Antequera F. (2003) Structure, function and evolution of CpG island promoters. Cell. Mol. Life Sci. 60(8), 1647–1658.
Jones P.A. (2012) Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat. Rev. Genet. 13(7), 484–492.
Li Z., Gu T.P., Weber A.R., Shen J.Z., Li B.Z., Xie Z.G., Yin R., Guo F., Liu X., Tang F., Wang H., Schar P., Xu G.-L. (2015) Gadd45a promotes DNA demethylation through TDG. Nucl. Acids Res. 43(8), 3986–3997.
Ishida T., Hamano A., Koiwa T., Watanabe T. (2006) 5' Long terminal repeat (LTR)-selective methylation of latently infected HIV-1 provirus that is demethylated by reactivation signals. Retrovirology. 3, 69.
Guo J.U., Su Y., Zhong C., Ming G.L., Song H. (2011) Hydroxylation of 5-methylcytosine by TET1 promotes active DNA demethylation in the adult brain. Cell. 145(3), 423–434.
Hirano S. (2012) Western blot analysis. Methods Mol. Biol. 926, 87–97.
Jiang C., Wu D., Haacke E.M. (2017) Ferritin-EGFP. Chimera as an endogenous dual-reporter for both fluorescence and magnetic resonance imaging in human glioma U251 cells. Tomography. 3(1), 1–8.
Kumaki Y., Oda M., Okano M. (2008) QUMA: quantification tool for methylation analysis. Nucl. Acids Res. 36(Web Server issue), W170–175.
Power D., Santoso N., Dieringer M., Yu J., Huang H., Simpson S., Seth I., Miao H., Zhu J. (2015) IFI44 suppresses HIV-1 LTR promoter activity and facilitates its latency. Virology. 481, 142–150.
Fouse S.D., Nagarajan R.O., Costello J.F. (2010) Genome-scale DNA methylation analysis. Epigenomics. 2(1), 105–117.
Du J., Gay M.C.L., Lai C.T., Trengove R.D., Hartmann P.E., Geddes D.T. (2017) Comparison of gravimetric, creamatocrit and esterified fatty acid methods for determination of total fat content in human milk. Food Chem. 217, 505–510.
Euser A.M., Dekker F.W., le Cessie S. (2008) A practical approach to Bland–Altman plots and variation coefficients for log transformed variables. J. Clin. Epidemiol. 61(10), 978–982.
Thorne N., Inglese J., Auld D.S. (2010) Illuminating insights into firefly luciferase and other bioluminescent reporters used in chemical biology. Chem. Biol. 17(6), 646–657.
Yun C., Dasgupta R. (2014) Luciferase reporter assay in Drosophila and mammalian tissue culture cells. Curr. Protoc. Chem. Biol. 6(1), 7–23.
Xie W., Silvers R., Ouellette M., Wu Z., Lu Q., Li H., Gallagher K., Johnson K., Montoute M. (2016) A luciferase reporter gene system for high-throughput screening of gamma-globin gene activators. Methods Mol. Biol. 1439, 207–226.
Stables J., Scott S., Brown S., Roelant C., Burns D., Lee M.G., Rees S. (1999) Development of a dual glow-signal firefly and Renilla luciferase assay reagent for the analysis of G-protein coupled receptor signalling. J. Receptor Signal Transduction Res. 19, 395–410.
Gadkar V., Filion M. (2014) New developments in quantitative real-time polymerase chain reaction technology. Curr. Issues Mol. Biol. 16, 1–6.
Cooksey R.C., Crawford J.T., Jacobs W.R., Jr., Shin-nick T.M. (1993) A rapid method for screening antimicrobial agents for activities against a strain of Mycobacterium tuberculosis expressing firefly luciferase. Antimicrobial Agents Chemotherapy. 37(6), 1348–1352.
Baldari C.T., Di Somma M.M., Majolini M.B., Ulivieri C., Milia E., Telford J.L. (1998) NF-AT-luciferase reporter T cell lines as tools to screen immunosuppressive drugs. Biol. J. Internat. Assoc. Biol. Standardization. 26(1), 1–5.
Michelini E., Cevenini L., Calabretta M.M., Calabria D., Roda A. (2014) Exploiting in vitro and in vivo bioluminescence for the implementation of the three Rs principle (replacement, reduction, and refinement) in drug discovery. Analyt. Bioanalyt. Chem. 406(23), 5531–5539.
Henderson L.J., Reoma L.B., Kovacs J.A., Nath A. (2020) Advances toward curing HIV-1 infection in tissue reservoirs. J. Virol. 94(3), e00375–19.
Kauder S.E., Bosque A., Lindqvist A., Planelles V., Verdin E. (2009) Epigenetic regulation of HIV-1 latency by cytosine methylation. PLoS Pathogens. 5(6), 1–15.
Bouchat S., Delacourt N., Kula A., Darcis G., van Driessche B., Corazza F., Gatot J.S., Melard A., Vanhulle C., Kabeya K., Pardons M., Avettand-Fenoel V., Clumeck N., De Wit S., Rohr O., RouziouxC., Van Lint C. (2016) Sequential treatment with 5-aza-2'-deoxycytidine and deacetylase inhibitors reactivates HIV-1. EMBO Mol. Med. 8(2), 117–138.
Greenberg M.V.C., Bourc’his D. (2019) The diverse roles of DNA methylation in mammalian development and disease. Nat. Rev. Mol. Cell Biol. 20(10), 590–607.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


