Молекулярная биология, 2022, T. 56, № 1, стр. 83-102
Генно-инженерные системы для изучения вирусных патогенов человека из семейства Coronaviridae
С. О. Галкин a, b, А. Н. Анисенко a, b, c, О. А. Шадрина b, c, М. Б. Готтих b, c, *
a Факультет биоинженерии и биоинформатики Московского государственного университета
им. М.В. Ломоносова
119991 Москва, Россия
b Химический факультет Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
c Научно-исследовательский институт физико-химической биологии им. А.Н. Белозерского Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: gottikh@belozersky.msu.ru
Поступила в редакцию 12.05.2021
После доработки 09.06.2021
Принята к публикации 14.06.2021
- EDN: WVMETT
- DOI: 10.31857/S0026898422010025
Аннотация
Охватившая весь мир пандемия коронавирусного инфекционного заболевания (COVID-19), которое вызвано ранее неизвестным бетакоронавирусом SARS-CoV-2, сделала крайне актуальной разработку простых и безопасных клеточных систем, позволяющих манипулировать с геномом вируса, а также проводить высокопроизводительный скрининг его потенциальных ингибиторов. В этом обзоре мы постарались обобщить существующие на настоящий момент данные о генно-инженерных системах для исследования не только SARS-CoV-2, но и других вирусов из семейства Coronaviridae. Кроме того, в обзоре кратко представлены основные сведения о строении и жизненном цикле коронавирусов.
ВВЕДЕНИЕ
COVID-19 (Coronavirus infectious disease 2019) ‒ инфекционное заболевание, вызванное новым штаммом бетакоронавируса, вирусом-2 острого респираторного синдрома (SARS-CoV-2), ‒ уже унесло миллионы жизней по всему миру. В связи с высокой патогенностью и контагиозностью возбудителя Всемирной организацией здравоохранения была объявлена пандемия COVID-19. В научных центрах по всему миру активно идут исследования SARS-CoV-2, проведены масштабные работы по созданию вакцин, существенные усилия направлены на поиск лекарственных средств для эффективной терапии и профилактики COVID-19.
За последние 20 лет коронавирусы были этиологическими агентами трех масштабных вспышек тяжелых респираторных инфекций человека: тяжелый острый респираторный синдром (SARS) в 2002 году, ближневосточный респираторный синдром (MERS) в 2012 году и коронавирусная инфекция 2019 года (COVID-19), которая затронула практически все страны и вызвала общемировой кризис здравоохранения [1, 2]. SARS-CoV-2 отличается от SARS-CoV-1 и MERS-CoV продолжительным инкубационным периодом, большим процентом бессимптомных носителей, а также способностью передаваться от человека к человеку при отсутствии клинических симптомов у инфицированных индивидов [3]. Такой набор особенностей SARS-CoV-2 привел к тому, что локальная вспышка в китайском Ухане переросла в пандемию. Летальность COVID-19 сильно зависит от возраста и пола человека: наиболее высока вероятность летального исхода у пожилых людей, особенно мужского пола [4]. Выявлена существенная корреляция между тяжестью заболевания COVID-19 и сопутствующими хроническими заболеваниями или избыточной массой тела [5].
Успех в исследовании высокопатогенных коронавирусов и, в частности, в поиске потенциальных препаратов для терапии вызываемых ими заболеваний во многом зависит от понимания особенностей инфекционного цикла вируса, а, следовательно, и развития технологий, используемых для его исследования. В основном это генно-инженерные системы; их много, они отличаются большим разнообразием, и каждая имеет свои преимущества и недостатки. В представленном обзоре предпринята попытка систематизировать существующие на сегодняшний день системы для изучения коронавирусов, подробно обозначив сильные и слабые стороны каждой из них.
БИОЛОГИЯ КОРОНАВИРУСОВ
Представители семейства Coronaviridae инфицируют млекопитающих и птиц, вызывая респираторные заболевания различной степени тяжести. До 2019 года в человеческой популяции циркулировало 6 коронавирусов, из которых Betacoronavirus 1 (HCoV-OC43), HCoV-229E, HCoV-NL63 и HCoV-HKU1 считаются сезонными респираторными вирусами. Появившийся в 2002 году коронавирус, ассоциированный с тяжелым острым респираторным синдромом (SARS-CoV, рекомендуемое с 2020 года название SARS-CoV-1; род Betacoronavirus, подрод Sarbecovirus), и коронавирус ближневосточного респираторного синдрома (MERS-CoV; род Betacoronavirus, подрод Merbecovirus) оказались высокопатогенными для человека. В 2019 году человечество столкнулось с еще одним штаммом подрода Sarbecovirus, получившим название SARS-CoV-2 [6]. В данном разделе описываются основные детали строения и жизненного цикла представителей семейства Coronaviridae в целом и семи человеческих патогенов в частности.
СТРОЕНИЕ ВИРИОНА
Коронавирусы ‒ это оболочечные, содержашие (+)-цепь РНК вирусы IV группы по классификации Балтимора [7]. Средний диаметр вирионов составляет 80‒120 нм. Вирионы имеют сферическую форму с характерными шипами, формируемыми S-белком, которые напоминают солнечную корону. Схематичное строение вириона коронавируса приведено на рис. 1. Снаружи вирион покрыт суперкапсидом ‒ производным клеточной мембраны с интегрированными вирусными белками. В суперкапсиде большинства коронавирусов человека можно обнаружить следующие вирусные белки: формирующий шип гликопротеин (S) взаимодействует с поверхностью хозяйской клетки и инициирует проникновение компонентов вириона внутрь нее; мембранный белок (М) обеспечивает взаимодействие компонентов вириона между собой, что важно для успешной сборки вирусных частиц [8], а также белок оболочки (E), который формирует пентамерные трансмембранные каналы и выполняет роль виропорина [9]. Включение E-белка в вирион не строго обязательно для образования инфекционных вирусных частиц, однако такие вирусы аттенуированы относительно вирусов дикого типа [8]. Вирусы HCoV-OC43 и HCoV-HKU1 дополнительно содержат в составе оболочки белок гемагглютинин-эстеразу (HE), который разрушает рецептор S-белка и тем самым способствует более эффективному отпочковыванию новых вирионов [10].
Рис. 1.
Схематичное строение вириона представителей семейства Coronaviridae. Спирально-симметричный нуклеокапсид, состоящий из белка нуклеокапсида (N) и одной молекулы РНК-генома, окружен суперкапсидом клеточного происхождения с встроенными вирусными белками: S, M и E. Белок гемагглютинин-эстераза (HE) входит в состав вирионов коронавирусов человека HCoV-OC43 и HCoV-HKU1, но не HCoV-229E, HCoV-NL63, MERS-CoV, SARS-CoV-1 и SARS-CoV-2. Вирион коронавирусов содержит около 300 копий S-белка, 2000 копий М-белка, 1000 копий N-белка и 100 копий E-белка [11].
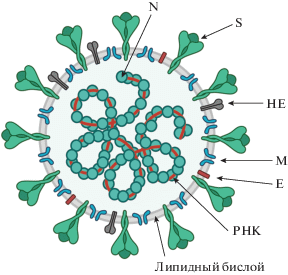
Под внешней оболочкой расположен спирально-симметричный нуклеокапсид, состоящий из N-белка и геномной (+)-цепи РНК. N-белок связан с вирусной РНК и выполняет структурную функцию: упаковки и стабилизации вирусного генома. Кроме того, N-белок модулирует клеточный иммунный ответ хозяина на вирусную инфекцию [8], а также способствует более эффективному синтезу 3'-дистальных генов [12].
СТРОЕНИЕ ГЕНОМА
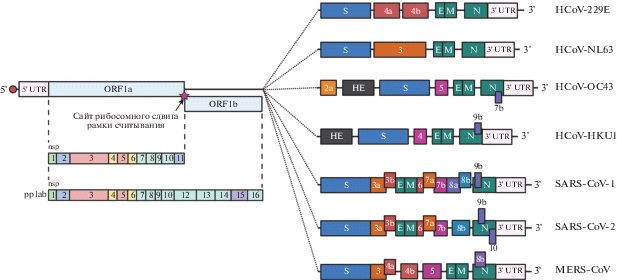
Геномная РНК коронавирусов не сегментирована, кепирована по 5'-концу и полиаденилирована по 3'-концу. Особенность генома Coronaviridae ‒ рекордная для РНК-содержащих вирусов длина, около 26‒32 тыс. оснований [13], в то время как средний размер одноцепочечных несегментированных РНК вирусов человека в среднем не превышает 10‒12 тыс. оснований [14]. Предполагают, что увеличенный по сравнению с другими вирусами размер генома коронавирусов обусловлен наличием редактирующей активности в комплексе РНК-зависимой РНК-полимеразы (RdRp), которая снижает мутационную нагрузку [15].
Принципиально весь геном коронавирусов можно разделить на две смысловые части. Первая содержит открытую рамку считывания (ORF) ORF1a/b, кодирующую белки, отвечающие главным образом за процессы репликации и транскрипции. Вторая часть кодирует структурные и вспомогательные белки, отвечающие за взаимодействие с поверхностью клетки и проникновение в нее, а также адаптацию к хозяину. Результаты сравнительного анализа последовательностей геномов различных коронавирусов подтверждают функциональное значение этого разделения: консервативность ORF1a/b выше среди коронавирусов, чем консервативность остальной части генома [16].
Число ORF в геноме отличается у разных видов коронавирусов и составляет от 6 до 11 [17]. Первая ORF, ORF1a/b, составляет около 2/3 генома и кодирует два полипротеина: pp1a и pp1ab. Полипротеин pp1ab образуется в результате рибосомного сдвига рамки считывания на один нуклеотид назад (‒1) перед терминирующим кодоном ORF1a. В состав pp1a/pp1ab входят неструктурные белки (non-structural proteins, NSPs), ответственные за процессы репликации и транскрипции, подавление синтеза клеточных мРНК и модуляцию клеточного иммунного ответа [18].
При анализе большого массива данных выявлено, что многие NSPs одновременно выполняют несколько функций в репликативном цикле вируса, что позволяет вирусу справиться с широким спектром задач ‒ от собственной репликации до противодействия клеточному иммунному ответу, ‒ дополнительно не увеличивая геном [19–22]. В табл. 1 приведена актуальная информация о наиболее изученных функциях NSPs. Среди них можно выделить несколько функциональных блоков: белки, отвечающие за формирование RdRp-комплекса (NSP7‒NSP16); белки, вовлеченные в образование двумембранных репликативных органелл (NSP3‒NSP6), процессинг полипротеинов pp1a и pp1ab (NSP3, NSP5), а также в модуляцию клеточных процессов (NSP1, NSP2). Остальная часть генома кодирует необходимые структурные белки: S, N, E, M, (HE), ‒ а также ряд вспомогательных белков, характерных для каждого конкретного вида [23, 24]. Считается, что вспомогательные белки не обязательны для репликации вируса, хотя способствуют более эффективной сборке вирионов как за счет подавления внутриклеточного иммунитета, так и выполняя роль скаффолд-белков при сборке вирионов [25].
Таблица 1.
Функции неструктурных белков коронавирусов
| Название белка | Основные функции |
|---|---|
| NSP1 | Подавляет трансляцию клеточных мРНК, связываясь с 18S рРНК 40S субчастицы рибосомы [26]; способствует разрезанию клеточных мРНК в районе 5'UTR [27] |
| NSP2 | Возможно, регулирует сигнальные пути выживания клетки; взаимодействует с клеточными белками prohibitin 1 и prohibitin 2 [28]; не обязателен для успешной инфекции [29] |
| NSP3 | Осуществляет процессинг pp1a и pp1ab, а именно NSP1–NSP4, за счет наличия домена папаинподобной протеазы-2 [30]; компонент поры в двумембранных органеллах, осуществляющей экспорт вирусных геномных и субгеномных РНК в цитоплазму [31]; обладает деубиквитинирующей активностью; отщепляет от белков моно-ADP-рибозу, подавляя клеточный интерфероновый ответ [30] |
| NSP4 | Участвует в образовании двумембранных репликативных органелл [32] |
| NSP5 | 3-химотрипсинподобная протеаза, осуществляет процессинг pp1a и pp1ab, а именно, NSP4–NSP11/ NSP4–NSP16 [33] |
| NSP6 | Предотвращает слияние везикул, содержащих вирусные белки, с лизосомами; участвует в образовании двумембранных репликативных органелл [34] |
| NSP7 | Формирует гексадекамерный комплекс с NSP8, который, вероятно, функционирует как фактор процессивности [35] и праймаза [36, 37] |
| NSP8 | Формирует гексадекамерный комплекс с NSP7, который, вероятно, функционирует как фактор процессивности [35] и праймаза [36, 37]; подавляет встраивание белков в клеточную мембрану, формируя комплекс с 7SL РНК сигналраспознающих частиц (signal recognition particle, SRP) и 28S рРНК [26] |
| NSP9 | Подавляет встраивание белков в клеточную мембрану, формируя комплекс с 7SL РНК SRP [26]; компонент РНК-полимеразного комплекса [19] |
| NSP10 | Важен для синтеза (‒)-цепи РНК в процессе вирусной репликации [38]; вовлечен в процессинг pp1a/ab [39]; регулирует активность компонентов RdRp-комплекса: NSP14 и NSP16 [40, 41] |
| NSP11 | Короткий пептид, который образуется при процессинге pp1a. Функции неизвестны [19] |
| NSP12 | Компонент RdRp, обладающий полимеразной активностью [19] |
| NSP13 | Обладает хеликазной активностью [42] |
| NSP14 | Способствует повышению точности синтеза вирусной РНК за счет наличия редактирующей 3' → 5'-экзонуклеазной активности [43]; участвует в 5'-кепировании вирусной РНК, обладает N7-метилтрансферазной активностью [44] |
| NSP15 | Подавляет активацию клеточных сенсоров двухцепочечных РНК (дцРНК); обладает функционально значимой эндорибонуклеазной активностью, хотя точный механизм неизвестен [45] |
| NSP16 | Нарушает сплайсинг, связываясь с U1/U2 малой ядерной РНК [26]; участвует в 5'-кепировании вирусной РНК, обладает 2'-O-метилтрансферазной активностью [40] |
Схема строения генома представителей семейства Coronaviridae, инфицирующих человека, приведена на рис. 2. Стоит отметить, что для всех коронавирусов порядок следования генов структурных белков: ORF1a/b → S → E → M → N ‒ консервативен, но, по-видимому, не несет функциональной нагрузки, так как при их перестановке инфекционность полученных вирусов не изменялась [46].
Рис. 2.
Схематичное строение геномов коронавирусов, циркулирующих в популяции человека. На схеме справа прописными буквами указаны кодируемые вирусные белки, цифрами и строчными буквами – номера вирусспецифичных ORF. UTR ‒ нетранслируемая область. Цифрами слева обозначены номера закодированных NSPs.
ЖИЗНЕННЫЙ ЦИКЛ
Проникновение в клетку
Жизненный цикл коронавирусов начинается со связывания вириона с рецепторами на поверхности клетки-хозяина (рис. 3). Ключевую роль в этом процессе играет вирусный поверхностный гликопротеин S. Именно этот белок, изменчивость которого выше, чем других вирусных белков [16], определяет видовой и тканевой тропизм патогена. Со стороны клетки во взаимодействии с вирусными частицами принимают участие различные поверхностные компоненты [47]. Коронавирус HCoV-229E в качестве рецептора использует аминопептидазу N (ANPEP). Это металлопротеиназа, представленная на поверхности эпителиальных клеток кишечника, легких и почек. В кишечнике этот фермент участвует в гидролизе пептидов, образующихся под действием желудочных и панкреатических протеаз. Функция ANPEP в легких и почках установлена не до конца ‒ скорее всего, она метаболизирует различные регуляторные пептиды [48]. Для вируса HCoV-NL63 ситуация с клеточными рецепторами неоднозначна. Показано, что S-белок этого вируса взаимодействует с ангиотензинконвертирующим ферментом-2 (ACE2) [49], а M-белок связывается с гепарансульфатированными белками на поверхности клеток [50]. Это взаимодействие М-белка может быть вспомогательным фактором для проникновения вируса или повышать локальную концентрацию вируса на поверхности клетки. При направленном удалении S-белка из состава исследуемых вирионов они сохраняли способность инфицировать клетки, даже в условиях заблокированного антителами ACE2, из чего можно сделать вывод о существовании альтернативных путей проникновения HCoV-NL63 в клетку, например через вышеуказанное взаимодействие М-белка с гепарансульфатированными белками [50]. Вирусы HCoV-OC43 и HCoV-HKU1 в качестве рецептора используют гликопротеины, содержащие 9-О-ацетилированную нейраминовую кислоту [25]. Эти же вирусы, как упоминалось ранее, имеют в своем составе белок гемагглютинин-эстеразу, которая способствует более эффективному взаимодействию с клеточной поверхностью и отпочковыванию новых вирусных частиц от зараженной клетки [51]. MERS-CoV в качестве рецептора использует дипептидилпептидазу-4 (DPP4) ‒ поверхностный димерный белок, который в норме процессирует гормоны и хемокины [52]. SARS-CoV-1 и SARS-CoV-2 в качестве основного рецептора используют ACE2 [25], хотя SARS-CoV-2 в качестве кофактора также использует гепарансульфатированные белки: взаимодействие с ними вызывает конформационные перестройки в S-белке, которые способствуют его более эффективному связыванию с ACE2 [53].
Рис. 3.
Схема жизненного цикла вирусов сем. Coronaviridae. Инфекция начинается со связывания вирусной частицы с рецептором на поверхности клетки-хозяина, что приводит к слиянию вирусной и клеточной мембран (ранний путь проникновения) или захвату вирионов в состав эндосом и последующему слиянию вирусной и клеточной мембраны (поздний путь проникновения). Вирусная РНК в комплексе с N-белком проникает в цитоплазму. В результате трансляции первой открытой рамки считывания ORF1a/b образуются полипротеины pp1a и pp1ab, которые подвергаются автопротеолизу с образованием NSPs. Из NSPs формируется комплекс RdRp, который осуществляет репликацию и транскрипцию вирусной РНК. Этот процесс происходит в двумембранных органеллах, которые образуются из шероховатого эндоплазматического ретикулума под действием компонентов pp1a и pp1ab. В процессе репликации/транскрипции образуются полноразмерные и субгеномные вирусные РНК. С субгеномных РНК транслируются структурные и вспомогательные белки. Структурные белки собираются в интермедиат между эндоплазматическим ретикулумом и аппаратом Гольджи (ERGIC) и вместе с комплексом из N-белка и геномной вирусной РНК формируют вирионы. Собранные вирионы накапливаются во внутриклеточных везикулах, а затем выходят из зараженной клетки за счет экзоцитоза [модифицировано из работы 54].
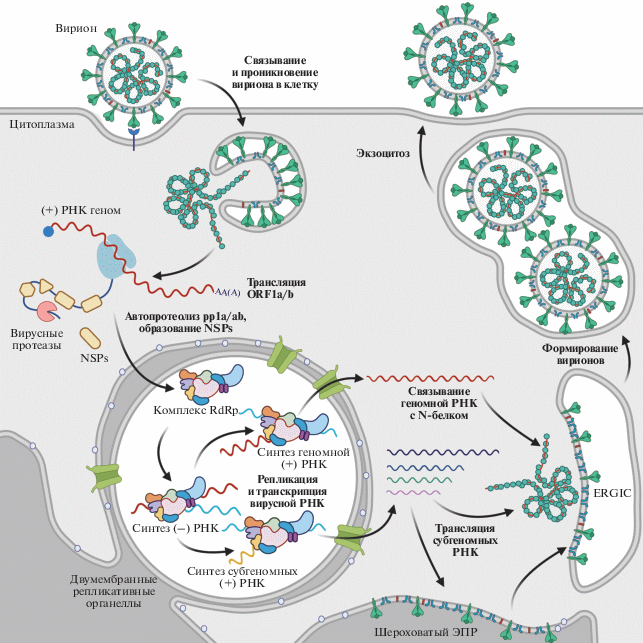
После связывания с рецептором S-белок под действием поверхностных клеточных протеаз процессируется и претерпевает конформационные перестройки, осуществляя слияние вирусной и клеточной мембран. Такой способ проникновения называется ранним. В случае, если поверхностные протеазы по какой-либо причине недоступны, активируется поздний путь проникновения в клетку. В этом случае вирус сначала подвергается эндоцитозу. В эндосоме, под действием пониженного pH, активируется клеточная протеаза катепсин L, которая подвергает S-белок протеолизу, вызывая слияние вирусной и эндосомной мембран [55]. После этого вирусный нуклеокапсид, состоящий из геномной РНК и N-белка, оказывается в цитоплазме и начинаются процессы вирусной транскрипции и репликации.
Репликация и транскрипция
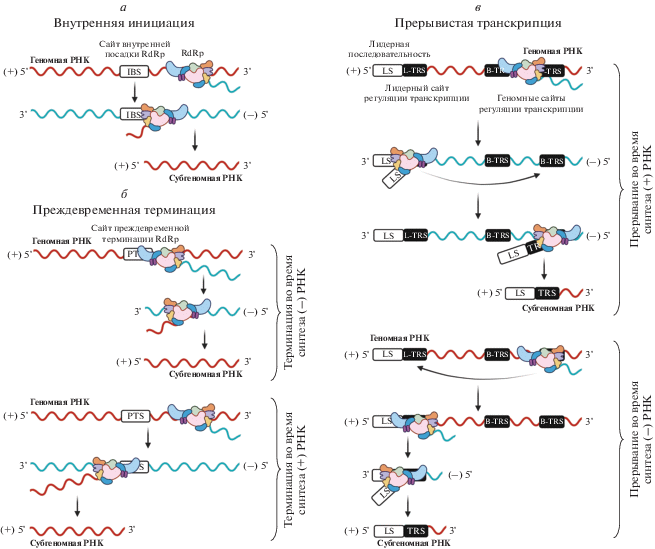
Как упомянуто выше, РНК коронавируса содержит большое число ORF (от 6 до 11) [24], что приводит к проблеме в трансляции 3'-дистальных генов. Коронавирусы решают эту задачу за счет образования субгеномных РНК [56, 57]. Среди (+)РНК-вирусов встречаются два основных механизма их синтеза: за счет наличия в геноме последовательностей внутренней посадки RdRp (cем. Caliciviridae, сем. Bromoviridae, сем. Virgaviridae, сем. Togaviridae) или сайтов преждевременной терминации транскрипции (сем. Nodaviridae, cем. Togaviridae, сем. Roniviridae, сем. Tombusviridae) (рис. 4а, б) [58]. Однако в случае коронавирусов процесс синтеза субгеномных РНК носит прерывистый характер [40, 41, 43]: в процессе синтеза (‒)-цепи РНК RdRp-комплекс диссоциирует от (+)РНК-матрицы в определенных сайтах, связывается в регионе 5'UTR и продолжает синтез РНК (рис. 4б) [59]. Позиции диссоциации и последующего связывания RdRp определяются последовательностями, регулирующими транскрипцию (transcription regulation sequence, TRS). В некоторых случаях “перескок” RdRp возможен при синтезе (+)РНК-цепи: в этом случае RdRp диссоциирует от TRS, расположенной в 5' UTR, и вновь гибридизуется с TRS перед началом белоккодирующей последовательности (рис. 4б) [60]. Полученные в процессе прерывистой транскрипции укороченные РНК служат матрицей для последующей трансляции вирусных белков. Интересно, что именно процесс прерывистой транскрипции позволяет вирусу использовать специфичные механизмы подавления клеточной трансляции, не влияя на трансляцию вирусных мРНК. Считается, что наличие лидерной последовательности в составе всех вирусных мРНК защищает их от деградации NSP1-белком, в то время как клеточные мРНК разрезаются им вблизи 5'-конца [56]. В случае, если RdRp-комплекс в процессе удлинения цепи РНК не диссоциирует, происходит синтез полноразмерной цепи РНК, комплементарной геномной, а на ее матрице синтезируются геномные (+)РНК, которые используются для сборки новых вирусных частиц и синтеза полипротеинов pp1a и pp1ab. Показано, что активный синтез вирусных белков начинается уже через пять часов после инфекции [61]. Одновременно с этим существенно повышается содержание вирусных РНК в клетке, что обусловлено не только репликацией и прерывистой транскрипцией, но и подавлением синтеза клеточных мРНК вирусным белком NSP1 [62].
Рис. 4.
Схематичное изображение стратегий синтеза субгеномных РНК коронавирусов. а ‒ Модель внутренней инициации. IBS – внутренний сайт посадки RdRp. б ‒ Модель преждевременной терминации транскрипции. Терминация может происходить как во время синтеза (+)-цепи, так и во время синтеза (‒)-цепи РНК. PTS – сайт преждевременной терминации RdRp. в ‒ Модель прерывистой транскрипции. Прерывание может происходить как во время синтеза (+)-цепи, так и во время синтеза (‒)-цепи РНК [58]. LS – лидерная последовательность; TRS – последовательность, регулирующая транскрипцию; L-TRS – лидерная TRS; B-TRS – TRS кодирующей части.
Сборка вириона и отпочковывание
Отличительная черта репликативного цикла коронавирусов ‒ место сборки вирионов. Сборка большей части оболочечных вирусов происходит в плазматической мембране клетки; в то время как вирионы коронавирусов собираются в компартменте, переходном между эндоплазматическим ретикулумом и аппаратом Гольджи (endoplasmic-reticulum–Golgi intermediate compartment, ERGIC). Показано, что после трансляции структурные белки S, M, E и N аккумулируются в ERGIC [63]. Геномная (+)РНК связывается с N-белком, формируя нуклеокапсид. В узнавании специального сигнала упаковки генома (package sequence, PS) в составе РНК участвует M-белок [64]. Способность N-белка узнавать PS зависит от вида вируса: например, упаковка геномной РНК мышиного коронавируса (mouse hepatitis virus, MHV) происходит по N-независимому механизму [65], а упаковка генома SARS-CoV-1 ‒ по N-зависимому [66]. Сигнал упаковки находится в пределах ORF1a/b, что обеспечивает включение в вирион только полноразмерных РНК [67, 68].
Центром сборки новых вирусных частиц выступает M-белок, который взаимодействует с белками S, N и E и геномной (+)РНК [69]. После того, как белки M, S, E и комплекс N-белка с РНК оказываются в ERGIC, собираются и отпочковываются зрелые вирусные частицы. Собранные вирионы накапливаются во внутриклеточных везикулах, которые, в свою очередь, высвобождаются во внеклеточное пространство в результате экзоцитоза или лизиса зараженной клетки [70].
СИСТЕМЫ ДЛЯ ИЗУЧЕНИЯ РЕПЛИКАЦИИ КОРОНАВИРУСОВ
Успех в изучении высокопатогенных вирусов, к которым относятся некоторые коронавирусы, и в поиске потенциальных препаратов для их терапии зависит от развития технологий, позволяющих манипуляции с вирусами и в частности с их геномом. Традиционно для изучения вирусной репликации используют полноразмерные вирусные системы, то есть инфекционные вирусные частицы. К преимуществам таких систем можно отнести максимальное соответствие реальным условиям заражения. Во время инфекции вирусные белки контактируют между собой и со множеством клеточных факторов, формируя сложные сети белковых взаимодействий, которые реализуются в указанных системах. Однако применение инфекционных вирусов сопряжено с высоким риском инфицирования оператора, поэтому работа с ними допускается лишь в сертифицированных лабораториях, оснащенных соответствующим оборудованием. Более того, в случае РНК-содержащих вирусов при работе с полноразмерными вирусными системами достаточно сложно получать варианты вирусов, мутантных по исследуемым генам. В случае коронавирусов манипуляции с их геномами крайне затруднены еще и большим размеров их геномных РНК, который, как указано выше, варьирует от 26 до 32 тыс. оснований [13]. В этой связи большое внимание уделяется как модификации полноразмерных вирусных систем, так и разработке более простых и безопасных систем, позволяющих изучать отдельные этапы жизненного цикла коронавирусов и проводить поиск их ингибиторов. Далее мы подробно рассмотрим существующие на данный момент варианты модифицированных полноразмерных вирусных систем, при работе с которыми можно получать вирусные РНК, содержащие необходимые мутации, а также более безопасные, чем вирусы дикого типа, хотя зачастую и менее информативные псевдовирусные системы и репликоны.
МОДИФИЦИРОВАННЫЕ ПОЛНОРАЗМЕРНЫЕ ВИРУСНЫЕ СИСТЕМЫ
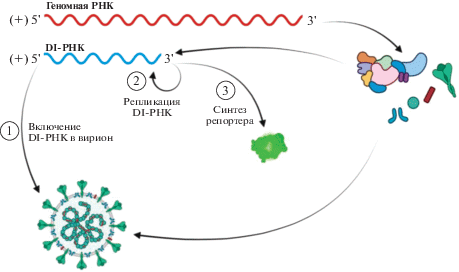
Для получения различным образом измененного вирусного генома коронавирусов применяют два подхода: непосредственная модификация геномной РНК (defective interfering RNA, DI-РНК) или получение кДНК, содержащей необходимые модификации, которую затем используют для синтеза геномной РНК и последующей сборки вирусных частиц.
DI-РНК-системы
Исторически изучение механизмов репликации коронавирусов начиналось с использования вирусных DI-РНК, содержащих лишь части вирусного генома: регуляторные вирусные последовательности, необходимые для поддержания репликации РНК, и последовательности, кодирующие части вирусных белков. Такие РНК способны реплицироваться, но исключительно в клетках, инфицированных соответствующим коронавирусом, а в ряде случаев и упаковываться в неинфекционные вирусоподобные частицы (VLP) [67, 71, 72] (рис. 5). Таким образом можно получать новые варианты вирусных частиц, несущих необходимые модификации. DI-РНК могут также содержать репортерный ген и, если происходит репликация и транскрипция генома, с помощью таких систем можно оценивать эффективность работы вирусных ферментов. Это одни из первых и наиболее примитивных систем, с помощью которых был получен большой массив данных о механизмах репликации и упаковки коронавирусных геномов. Например, с помощью DI-РНК были определены сигналы упаковки MHV [73–75], вируса трансмиссивного гастроэнтерита свиней (transmissible gastroenteritis virus, TGEV) [67] и SARS-CoV-2 [68]. Для MHV в этой системе продемонстрировано взаимодействие М-белка с PS РНК [64]. На примере SARS-CoV-2 показано, что при трансфекции зараженных вирусом клеток дефектной РНК она начинает активно реплицироваться и включаться в вирионы, что приводит к снижению продукции инфекционных вирусных частиц приблизительно вдвое через 24 ч после введения DI-РНК. На основании этих данных было сделано предположение, что DI-РНК можно использовать в качестве терапевтического агента для снижения вирусной нагрузки [68].
Инфекционная кДНК, встроенная в искусственную бактериальную хромосому
Развитие методов молекулярного клонирования позволило собирать полноразмерные вирусные кДНК по частям. Впервые полноразмерная кДНК коронавирусного генома была получена для TGEV методом клонирования в искусственную бактериальную хромосому (bacterial artificial chromosome, BAC) [76]. В составе BAC вирусная кДНК находилась под контролем цитомегаловирусного (CMV) промотора, а с 3′-конца была ограничена поли(А)-участком, последовательностью рибозима вируса гепатита дельта (HDV) и терминатором транскрипции BGH (рис. 6а). Рибозим HDV катализирует разрыв цепи РНК между последним нуклеотидом генома коронавируса и первым нуклеотидом рибозима, что позволяет получить РНК, содержащую 3'-концевую последовательность, полностью идентичную вирусной [77]. Подобное соответствие необходимо в силу того, что 3'UTR последовательность коронавирусов крайне важна для успешной репликации и транскрипции вирусной РНК [78]. После трансфекции ВАС в клетках синтезируется полногеномная вирусная РНК и белки, которые собираются в инфекционные вирусные частицы. Использование BAC для работы с кДНК коронавирусных геномов обладает рядом преимуществ. Во-первых, становится возможным эффективно наработать неограниченное количество необходимой кДНК в Escherichia coli. Во-вторых, можно с высокой эффективностью трансфицировать клетки млекопитающих этой бактериальной хромосомой, что позволяет осуществлять внутриклеточную экспрессию вирусной РНК и не прибегать к более сложным методикам ее получения, например in vitro транскрипции. В-третьих, последовательность BAC относительно легко модифицировать, используя Red-рекомбиназу и эндонуклеазу рестрикции I‑SceI, что позволяет вносить изменения в изучаемые гены вируса [79].
Рис. 6.
Схема получения полноразмерной вирусной кДНК с использованием искусственной бактериальной хромосомы (а), метода TAR-клонирования (б), метода in vitro лигирования (в) или с использованием рекомбинантных поксвирусов (г). CMV – промотор цитомегаловируса; pA – поли(А)-хвост; Rz – рибозим HDV; MCS – полилинкер.
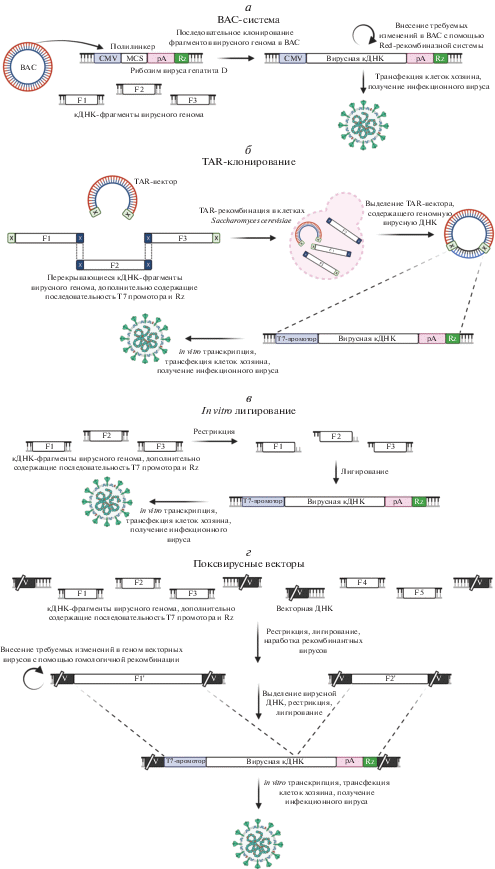
В последующем подход клонирования вирусной кДНК в BAC, помимо TGEV, был применен для создания инфекционных клонов человеческих патогенов: HCoV-OC43 [80], MERS-CoV [81], SARS-CoV-1 [82] и SARS-CoV-2 [83] (табл. 2).
Таблица 2.
Способы получения полноразмерных кДНК коронавирусов, способных инфицировать человека
Инфекционная кДНК, полученная с помощью TAR-клонирования
Ассоциированная с трансформацией рекомбинация (transformation-associated recombination, TAR) ‒ метод клонирования, использующий особенность клеток Saccharomyces cerevisiae, в которых с высокой частотой происходит гомологичная рекомбинация перекрывающихся фрагментов ДНК (рис. 6б) [91]. Использование клеток дрожжей для получения и наработки полноразмерных коронавирусных кДНК обладает рядом преимуществ перед бактериальной системой. Во-первых, длинные последовательности ДНК в целом менее стабильны в бактериях, чем в дрожжах. Во-вторых, показано, что фрагменты последовательности гена, кодирующего ORF1a/b, могут быть токсичны для бактерий [76]. В-третьих, высокая эффективность TAR-клонирования позволяет быстро и без большого труда внести мутации в геном вируса: для этого достаточно всего лишь внести изменения в один или несколько трансформируемых фрагментов. Этот метод был использован для сборки и модификации длинных геномов ДНК-вирусов: CMV (размер генома ~236 т.п.н.) [92, 93] и вируса простого герпеса (размер генома ~152 т.п.н.) [94, 95]. Из человеческих патогенов из семейства Coronaviridae методом TAR-клонирования были получены кДНК геномов MERS-CoV, HCoV-229E, HCoV-HKU1 и SARS-CoV-2 (табл. 2) [84]. Заметим, что этим методом кДНК SARS-CoV-2 была получена всего за два месяца [84].
Инфекционная кДНК, полученная in vitro лигированием
Еще один подход к получению геномов коронавирусов ‒ сборка полноразмерной вирусной кДНК из фрагментов меньшего размера, которые фланкированы уникальными сайтами рестрикции. В качестве таких сайтов могут быть использованы последовательности, узнаваемые эндонуклеазами рестрикции I или III типа. В последнем случае после лигирования теряются сайты рестрикции, в связи с чем становится невозможной дальнейшая модификация уже полученной кДНК, так как сайт узнавания и сайт внесения разрыва в случае эндонуклеаз рестрикции III типа разнесены в пространстве. Для in vitro лигирования можно использовать как сайты рестрикции, которые уже существуют в исследуемом геноме, так и искусственно созданные. В этом случае сайты рестрикции вводят при помощи синонимичных замен, которые не изменяют аминокислотную последовательность вирусных белков. Также в ходе подготовки кДНК в начало собираемого таким образом генома вводится последовательность T7-промотора, необходимая для наработки РНК в бесклеточной системе с использование T7-РНК-полимеразы [88, 96] (рис. 6в). Полученная геномная РНК доставляется в целевые клетки трансфекцией или электропорацией, после чего в них идут процессы вирусной транскрипции и репликации, а также сборки инфекционных вирусных частиц. К недостаткам этого метода можно отнести необходимость в некоторых случаях изменять нативную нуклеотидную последовательность вирусного генома для введения дополнительных сайтов для эндонуклеаз рестрикции или для удаления случайных последовательностей Т7-терминатора, встречающихся в геноме коронавирусов. Однако использование эндонуклеаз рестрикции III типа, которые узнают ассиметричные последовательности ДНК и делают разрезы на определенном расстоянии от сайта посадки, позволяет минимизировать необходимость введения синонимичных замен. Несомненным плюсом такого подхода считается возможность фрагментировать вирусный геном таким образом, чтобы разбить на отдельные фрагменты гены белков, которые проявляют токсичность при наработке в бактериях. С использованием in vitro лигирования получены полноразмерные кДНК коронавирусов человека HCoV-NL63 [86], SARS-CoV-1 [88], MERS-CoV [87] и SARS-CoV-2 [90] (табл. 2).
Инфекционная кДНК, полученная с использованием векторов на основе поксвирусов
Альтернативной стратегий для получения и модификации инфекционных кДНК может быть использование векторов на основе вируса осповакцины (сем. Poxviridae). Полноразмерную кДНК коронавирусов, размер которой порядка 30 т.п.н., можно легко нарабатывать в составе генома вируса осповакцины, имеющего экстремально большой размер (около 200 т.п.н.). В рамках этого подхода на первом этапе с помощью in vitro лигирования перекрывающихся фрагментов кДНК коронавируса получают полноразмерную кДНК, которую после стадии очистки встраивают в поксвирусный вектор (рис. 6г). Однако в некоторых случаях возможно клонирование нескольких больших фрагментов кДНК в отдельные векторы [85]. Например, в случае получения кДНК SARS-CoV-1 из восьми исходных фрагментов получили два поксвирусных вектора, которые содержали в себе фрагменты 1‒20288 и 20272‒29727 генома SARS-CoV-1; причем в состав первого вектора также входила последовательность Т7-промотора. После этого проводили еще одно in vitro лигирование ДНК полученных рекомбинантных вирусов с последующей in vitro транскрипцией и трансфекцией эукариотических клеток наработанной РНК с целью получения полноценных инфекционных вирусных частиц SARS-CoV-1 [89]. Введение дополнительного этапа с использованием поксвирусных векторов, хотя и усложняет описанный подход in vitro лигирования, все же имеет ряд преимуществ. Так, геномы рекомбинантных поксвирусов стабильны, эффективно реплицируются в клеточных культурах, что позволяет преодолеть проблему токсичности некоторых коронавирусных последовательностей для бактерий [97]. Важно также, что есть подходы, позволяющие эффективно вносить мутации в геном рекомбинантных поксвирусов с помощью гомологичной рекомбинации, и это открывает широкие возможности для модификации изучаемых вирусных генов. С использованием рекомбинантных поксвирусов получены кДНК коронавирусов HCoV-229E [85] и SARS-CoV-1 [89] (табл. 2). Однако этот подход достаточно сложен и, возможно, именно этим можно объяснить отсутствие статей, в которых используются векторы на основе поксвирусов для получения полноразмерной кДНК SARS-CoV-2.
ПСЕВДОВИРУСНЫЕ СИСТЕМЫ
В качестве альтернативы полноразмерным вирусам в исследованиях часто используют так называемые псевдовирусы. Псевдовирусы, они же VLP, ‒ это репликативно некомпетентные варианты исходных вирусов. В общем виде псевдовирусная система состоит как минимум из двух фрагментов ДНК: один кодирует вирусный геном, из состава которого удален один или несколько генов важных структурных вирусных белков, а вместо него встроен репортерный ген, позволяющий в клетках оценивать эффекты воздействий на вирусную репликацию; второй фрагмент ДНК кодирует недостающий сегмент генома. Совместная экспрессия двух фрагментов в специальных упаковывающих клетках приводит к сборке вирусных частиц, способных инфицировать целевые клетки и воспроизводить отдельные этапы жизненного цикла коронавирусов, но неспособных продуцировать новые вирусные частицы в трансдуцированных клетках, так как они содержат в своем составе укороченный геном. Несомненное и главное преимущество псевдовирусов заключается в их безопасности: для работы с ними не требуется специальных разрешений и соблюдения соответствующих уровней защиты.
Псевдовирусы используют как в фундаментальных, так и в прикладных исследованиях. В настоящее время уже разработаны противовирусные вакцины на основе VLP (см. в обзоре [98]). Их также используют в качестве векторов для таргетной доставки лекарств и генной терапии [99], в качестве положительного контроля в различных тест-системах для диагностики вирусных инфекций [100], как модели для исследования различных этапов жизненного цикла вируса в условиях, приближенных к физиологическим.
Для патогенов из семейства Coronaviridae разработано много разнообразных псевдовирусных систем. Большая часть работ, в которых используются VLP, посвящена изучению различных аспектов строения коронавирусов, а также механизмов проникновения вирионов в клетку и отпочковывания от нее. Пионерские исследования были проведены на MHV [101]. Показано, что для сборки и эффективного отпочковывания псевдовирусных частиц необходима экспрессия всего двух вирусных белков: M и E. S-белок, хотя и включался в состав VLP, не был необходим для продукции вирусных частиц. Удивительным было то, что N-белок не только не влиял на сборку псевдовирусных частиц, но и не включался в их состав при коэкспрессии с M- и E-белками [65]. Стоит заметить, что столь необычные результаты согласуются с данными о том, что сигнал упаковки геномной РНК MHV взаимодействует с M-, а не с N-белком [64]. VLP также были получены для HCoV-NL63. Минимальный набор структурных белков для формирования псевдовирусов и в этом случае включал только M- и E-белки [102]. В случае SARS-CoV-1 данные о минимальном наборе белков для формирования псевдовирусных частиц весьма противоречивы. Согласно одному из исследований, для формирования в клетке псевдовирусных частиц необходима экспрессия двух белков: M и N. Однако эффективное отпочковывание псевдовирусов происходило лишь при коэкспрессии уже трех белков: S, M и N. Белок E, несмотря на включение в VLP при коэкспрессии с M- и N-белками, не был необходим для формирования псевдовирусов [103]. Согласно другому исследованию, для эффективной сборки и отпочковывания псевдовирусов на основе SARS-CoV-1 необходима экспрессия белков M, E и N [104]. Подобная разница, вероятно, связана с различиями в используемых упаковочных клетках: HEK-293 и Vero E6 соответственно. В случае SARS-CoV-2 для сборки VLP в клетках линии HEK-293T необходимы белки M, S и E [105]. Интересно, но для SARS-CoV-2 также наблюдаются различия в сборке VLP в клетках линий HEK-293T и Vero E6: при коэкспрессии белков S, E, M и N в клетках Vero E6 S-белок более эффективно включался в состав VLP, чем в клетках HEK-293T [106].
Меньшее число работ посвящено созданию VLP для изучения амплификации и транскрипции РНК коронавирусов. Например, для SARS-CoV-2 разработана специальная клеточная система, которая позволяет исследовать многие фундаментальные аспекты биологии вируса [107]. В рамках этой работы ген N-белка в геноме SARS-CoV-2 заменяли на ген зеленого флуоресцентного белка (GFP). Для сборки VLP N-белок был экспрессирован эктопически в клетках-упаковщиках. В результате такого подхода VLP не могут нарабатываться в клетках, не экспрессирующих N-белок. Культивирование клеток-упаковщиков, экспрессирующих N-белок, в течение месяца не приводило к актам рекомбинации, способствующим образованию полноразмерного генома SARS-CoV-2 и продукции инфекционных вирусов, что подтверждает безопасность таких систем. Тем не менее, были предложены дальнейшие меры по повышению безопасности созданной псевдовирусной системы. Для этого ген N-белка “разбили” на две части и “разнесли” по разным векторам. Полноценный N-белок образовывался в результате опосредованного интеином белкового сплайсинга [107]. Созданные псевдовирусные частицы могут быть использованы для самого широкого круга задач: фундаментального исследования биологии SARS-CoV-2, скрининга потенциальных ингибиторов проникновения, транскрипции и репликации вируса, создания вакцин и детекции нейтрализующих антител у пациентов. Благодаря использованию этой системы, уже удалось многое узнать о SARS-CoV-2. Так, обнаружено, что N-белок контактирует с компонентами клеточных стресс-гранул G3BP1 и G3BP2, а также найдены потенциальные ингибиторы репликации этого вируса [107].
Использование псевдовирусов на основе сем. Coronaviridae имеет важное практическое приложение. Большое число работ посвящено разработке вакцин на основе VLP [102, 106‒108]. На основе нейротропного коронавируса HCoV-229E [109] создан VLP вектор, способный трансдуцировать дендритные клетки человека [110]. РНК созданного псевдовируса кодирует полный ген ORF1a/b, а также три репортерных гена, которые впоследствии можно заменить на целевые. За счет прерывистой транскрипции все три гена могут быть экспрессированы в трансдуцированных клетках. Уникальность созданного вектора заключается в том, что в его состав помещают сразу несколько репортерных генов и суммарная длина генома составляет около 6 т.п.н. Однако такая система имеет и существенный недостаток: помимо целевых генов происходит экспрессия всего полипротеина pp1a/ab, некоторые фрагменты которого проявляют цитотоксичность [110].
РЕПЛИКОНЫ
Репликоны ‒ это самоподдерживающиеся РНК, содержащие все регуляторные элементы вирусного генома. При наличии вирусных неструктурных белков, обеспечивающих вирусную репликацию и транскрипцию, с репликонов синтезируются новые молекулы РНК [111]. Необходимые неструктурные белки могут быть закодированы в самом репликоне или экспрессироваться в клетке с дополнительных векторов. Репликоны не содержат структурных белков и не продуцируют инфекционных вирусных частиц, поэтому представляют собой безопасную альтернативу полноразмерным вирусным системам. Часто в составе репликонов закодированы репортерные гены, что позволяет относительно легко оценивать эффективность синтеза РНК в соответствующих условиях. Для изучения отдельных этапов жизненного цикла коронавирусов, в том числе репликации генома, транскрипции, трансляции, хорошо подходят и псевдовирусы, но тем не менее именно репликоны широко используют в подобного рода исследованиях, поскольку они намного проще в обращении и еще более безопасны, чем VLP, хотя и менее приближены к условиям реальной инфекции.
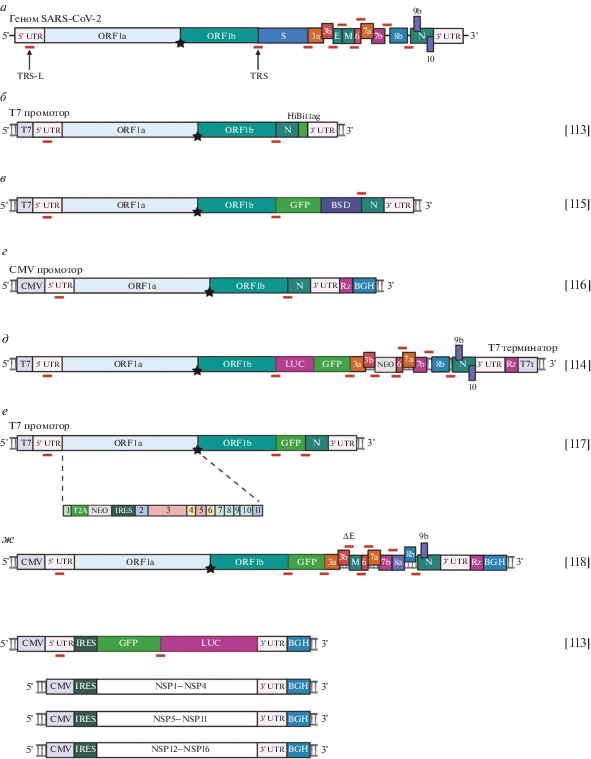
Принципиально все репликоны коронавирусов устроены одинаково: 5′- и 3′-концевые последовательности репликонов полностью совпадают соответственно с 5'- и 3′UTR коронавирусов. При изучении прерывистой транскрипции внутри репликона помещают последовательность, соответствующую межгенному участку исследуемого коронавируса и регулирующую процесс перескока RdRp-комплекса [112–118]. Как видно из рис. 7, в подавляющем большинстве случаев репликоны содержат большую рамку считывания ORF1a и 1b, которая кодирует неструктурные белки, участвующие в репликации вирусной РНК, а также последовательность гена белка N, необходимого для эффективного синтеза вирусных РНК [119, 120]. В состав репликонов на место генов разных структурных белков вируса часто встраивают гены белков-репортеров. Такие репортеры находятся под контролем вирусных регуляторных последовательностей TRS, взятых от генов соответствующих структурных белков TRS-S (от белка S), TRS-N (от белка N) и TRS-M (от белка M). В случае минимальных репликонов (рис. 7з), не кодирующих pp1a/b и N-белок, вирусные белки, обеспечивающие репликацию/транскрипцию, необходимо экспрессировать в исследуемых клетках с дополнительных векторов.
Рис. 7.
Основные структурные элементы репликонов коронавирусов. а ‒ Схема строения генома SARS-CoV-2 (приведена в качестве референсной структуры). б‒з ‒ Структура различных репликонов [112–118]; в репликоне всегда закодированы регуляторные элементы (5'UTR, 3'UTR, TRS), компоненты pp1a/b отсутствуют только в составе минимальных репликонов (з) [118]. Тело репликона может находиться под контролем либо T7-промотора, либо клеточного промотора. В первом случае можно проводить как in vitro транскрипцию с последующей трансфекцией клеток наработанной РНК, так и трансфицировать клетки ДНК-конструкцией при параллельной экспрессии T7 РНК-полимеразы [112–118]. На схеме буквами указаны кодируемые белки. Красной чертой обозначены TRS. GFP – зеленый флуоресцентный белок; LUC – люцифераза; BSD – белок устойчивости к бластицидину; NEO – белок устойчивости к неомицину; IRES – участок внутренней посадки рибосомы.
Репликоны часто используются для высокопроизводительного скрининга ингибиторов вирусной репликации и транскрипции [81, 112, 113, 115, 117, 118, 121–125]. Репликоны на основе SARS-CoV-1 нашли применение при тестировании ингибиторов вирусных протеаз 3CLpro и PLpro, хеликазы и сдвига рамки считывания при трансляции [126–128]. При проведении скрининга в некоторых исследованиях получали стабильные репликонсодержащие клеточные линии, для чего помимо репортерных генов в репликон встраивали ген устойчивости к антибиотику ‒ чаще всего к бластицидину и зеоцину [115, 123]. С помощью репликонов удобно оценивать влияние экспрессии различных белков на процессы репликации и транскрипции вирусного генома. Использование репликонов позволяет достоверно показать, что тот или иной эффект на жизненный цикл вируса связан именно с влиянием на процессы репликации/транскрипции, а не сборки и отпочковывания вириона. Например, с помощью репликонов J.-M. Wang и соавт. [117] показали, что для эффективной репликации РНК SARS-CoV-1 необходима экспрессия NSP16, но не NSP1 и NSP2. А недавно Y. Luo и др. [118] обнаружили, что в присутствии продукта экспрессии с ORF6 значительно повышается эффективность репликации РНК SARS-CoV-2.
ЗАКЛЮЧЕНИЕ
В представленном обзоре мы попытались систематизировать данные по разработанным к настоящему времени генно-инженерным системам для изучения патогенов человека из семейства Coronaviridae. Для каждого подхода приведены примеры возможного использования, обозначены достоинства и недостатки каждой системы. Мы не можем претендовать на полный охват всех используемых для изучения коронавирусов систем и полученных с их помощью результатов, поскольку исследования коронавирусов, и в первую очередь SARS-CoV-2, активно развиваются во всем мире и каждый день появляются новые данные по механизмам патогенеза новой коронавирусной инфекции, ингибиторам репликации SARS-CoV-2, разрабатываемым лекарственным средствам и вакцинам. Надеемся, что приведенные в обзоре данные заинтересуют тех, кто занимается исследованием коронавирусов, и сориентируют их в выборе именно той системы, которая подходит для решения конкретной научной задачи.
Работа поддержана грантом РФФИ № 20-04-60216_вирусы.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Все иллюстрации были созданы с помощью программного обеспечения BioRender.
Список литературы
Yin Y., Wunderink R.G. (2018) MERS, SARS and other coronaviruses as causes of pneumonia. Respirology. 23, 130–137.
Wu F., Zhao S., Yu B., Chen Y.-M., Wang W., Song Z.-G., Hu Y., Tao Z.-W., Tian J.-H., Pei Y.-Y., Yuan M.-L., Zhang Y.-L., Dai F.-H., Liu Y., Wang Q.-M., Zheng J.-J., Xu L., Holmes E.C., Zhang Y.-Z. (2020) A new coronavirus associated with human respiratory disease in China. Nature. 579, 265–269.
Rothe C., Schunk M., Sothmann P., Bretzel G., Froeschl G., Wallrauch C., Zimmer T., Thiel V., Janke C., Guggemos W., Seilmaier M., Drosten C., Vollmar P., Zwirglmaier K., Zange S., Wölfel R., Hoelscher M. (2020) Transmission of 2019-nCoV infection from an asymptomatic contact in Germany. N. Engl. J. Med. 382, 970–971.
Perez-Saez J., Lauer S.A., Kaiser L., Regard S., Delaporte E., Guessous I., Stringhini S., Azman A.S., Serocov-POP Study Group. (2021) Serology-informed estimates of SARS-CoV-2 infection fatality risk in Geneva, Switzerland. Lancet Infect. Dis. 21, e69–e70.
Rebello C. J., Kirwan J.P., Greenway F.L. (2020) Obesity, the most common comorbidity in SARS-CoV-2: is leptin the link? Int. J. Obes. (Lond.). 44, 1810–1817.
Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. (2020) The species severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat. Microbiol. 5, 536–544.
Baltimore D. (1971) Expression of animal virus genomes. Bacteriol. Rev. 35, 235–241.
Schoeman D., Fielding B.C. (2019) Coronavirus envelope protein: current knowledge. Virol. J. 16, 69.
Sarkar M., Saha S. (2020) Structural insight into the role of novel SARS-CoV-2 E protein: a potential target for vaccine development and other therapeutic strategies. PLoS One. 15, e0237300.
Lang Y., Li W., Li Z., Koerhuis D., van den Burg A.C.S., Rozemuller E., Bosch B.-J., van Kuppeveld F.J. M., Boons G.-J., Huizinga E.G., van der Schaar H.M., de Groot R.J. (2020) Coronavirus hemagglutinin-esterase and spike proteins coevolve for functional balance and optimal virion avidity. Proc. Natl. Acad. Sci. USA. 117, 25759–25770.
Bar-On Y.M., Flamholz A., Phillips R., Milo R. (2020) SARS-CoV-2 (COVID-19) by the numbers. eLife. 9, e57309.
Almazán F., Galán C., Enjuanes L. (2004) The nucleoprotein is required for efficient coronavirus genome replication. J. Virol. 78, 12683–12688.
Masters P.S. (2006) The molecular biology of coronaviruses. Adv. Virus Res. 66, 193‒292.
Belshaw R., Pybus O.G., Rambaut A. (2007) The evolution of genome compression and genomic novelty in RNA viruses. Genome Res. 17, 1496–1504.
Sanjuán R., Nebot M.R., Chirico N., Mansky L.M., Belshaw R. (2010) Viral mutation rates. J. Virol. 84, 9733–9748.
Chen Y., Liu Q., Guo D. (2020) Emerging coronaviruses: genome structure, replication, and pathogenesis. J. Med. Virol. 92, 418–423.
Song Z., Xu Y., Bao L., Zhang L., Yu P., Qu Y., Zhu H., Zhao W., Han Y., Qin C. (2019) From SARS to MERS, thrusting coronaviruses into the spotlight. Viruses. 11, 59.
Sa Ribero M., Jouvenet N., Dreux M., Nisole S. (2020) Interplay between SARS-CoV-2 and the type I interferon response. PLoS Pathog. 16(7), e1008737.
Snijder E.J., Decroly E., Ziebuhr J. (2016) The nonstructural proteins directing coronavirus RNA synthesis and processing. Adv. Virus Res. 96, 59–126.
Raj R. (2021) Analysis of non-structural proteins, NSPs of SARS-CoV-2 as targets for computational drug designing. Biochem. Biophys. Rep. 25, 100847.
Khan M.T., Irfan M., Ahsan H., Ahmed A., Kaushik A.C., Khan A.S., Chinnasamy S., Ali A., Wei D.-Q. (2021) Structures of SARS-CoV-2 RNA-binding proteins and therapeutic targets. Intervirology. 64, 55–68.
Yoshimoto F.K. (2020) The proteins of severe acute respiratory syndrome coronavirus-2 (SARS CoV-2 or n-COV19), the cause of COVID-19. Protein J. 39, 198–216.
Guo Y.-R., Cao Q.-D., Hong Z.-S., Tan Y.-Y., Chen S.-D., Jin H.-J., Tan K.-S., Wang D.-Y., Yan Y. (2020) The origin, transmission and clinical therapies on coronavirus disease 2019 (COVID-19) outbreak – an update on the status. Mil. Med. Res. 7, 11.
Cui J., Li F., Shi Z.-L. (2019) Origin and evolution of pathogenic coronaviruses. Nat. Rev. Microbiol. 17, 181–192.
Artika I.M., Dewantari A.K., Wiyatno A. (2020) Molecular biology of coronaviruses: current knowledge. Heliyon. 6, e04743.
Banerjee A.K., Blanco M.R., Bruce E.A., Honson D.D., Chen L.M., Chow A., Bhat P., Ollikainen N., Quinodoz S.A., Loney C., Thai J., Miller Z.D., Lin A.E., Schmidt M.M., Stewart D.G., Goldfarb D., De Lorenzo G., Rihn S.J., Voorhees R.M., Botten J.W., Majumdar D., Guttman M. (2020) SARS-CoV-2 disrupts splicing, translation, and protein trafficking to suppress host defenses. Cell. 183, 1325‒1339.e21.
Gaglia M M., Covarrubias S., Wong W., Glaunsin-ger B.A. (2012) A common strategy for host RNA degradation by divergent viruses. J. Virol. 86, 9527–9530.
Cornillez-Ty C.T., Liao L., Yates J.R., Kuhn P., Buchmeier M.J. (2009) Severe acute respiratory syndrome coronavirus nonstructural protein 2 interacts with a host protein complex involved in mitochondrial biogenesis and intracellular signaling. J. Virol. 83, 10314–10318.
Graham R.L., Sims A.C., Brockway S.M., Bari, R.S., Denison M.R. (2005) The nsp2 replicase proteins of murine hepatitis virus and severe acute respiratory syndrome coronavirus are dispensable for viral replication. J. Virol. 79, 13399–13411.
Lei J., Kusov Y., Hilgenfeld R. (2018) Nsp3 of coronaviruses: structures and functions of a large multi-domain protein. Antiviral Res. 149, 58–74.
Wolff G., Limpens R.W.A.L., Zevenhoven-Dobbe J.C., Laugks U., Zheng S., de Jong A.W.M., Koning R.I., Agard D.A., Grünewald K., Koster A.J., Snijder E.J., Bárcena M. (2020) A molecular pore spans the double membrane of the coronavirus replication organelle. Science. 369, 1395–1398.
Oostra M., te Lintelo E.G., Deijs M., Verheije M.H., Rottier P.J.M., de Haan C.A.M. (2007) Localization and membrane topology of coronavirus nonstructural protein 4: involvement of the early secretory pathway in replication. J. Virol. 81, 12323–12336.
Tomar S., Johnston M.L., St John S.E., Osswald H.L., Nyalapatla P.R., Paul L.N., Ghosh A.K., Denison M.R., Mesecar A.D. (2015) Ligand-induced dimerization of middle east respiratory syndrome (MERS) coronavirus nsp5 protease (3CLpro): implications for nsp5 regulation and the development of antivirals. J. Biol. Chem. 290, 19403–19422.
Cottam E.M., Whelband M.C., Wileman T. (2014) Coronavirus NSP6 restricts autophagosome expansion. Autophagy. 10, 1426–1441.
Zhai Y., Sun F., Li X., Pang H., Xu X., Bartlam M., Rao Z. (2005) Insights into SARS-CoV transcription and replication from the structure of the nsp7-nsp8 hexadecamer. Nat. Struct. Mol. Biol. 12, 980–986.
Imbert I., Guillemot J.-C., Bourhis J.-M., Bussetta C., Coutard B., Egloff M.-P., Ferron F., Gorbalenya A.E., Canard B. (2006) A second, non-canonical RNA-dependent RNA polymerase in SARS coronavirus. EMBO J. 25, 4933–4942.
te Velthuis A.J.W., van den Worm S.H.E., Snijder E.J. (2012) The SARS-coronavirus nsp7+nsp8 complex is a unique multimeric RNA polymerase capable of both de novo initiation and primer extension. Nucleic Acids. Res. 40, 1737–1747.
Sawicki S.G., Sawicki D.L., Younker D., Meyer Y., Thiel V., Stokes H., Siddell S.G. (2005) Functional and genetic analysis of coronavirus replicase-transcriptase proteins. PLoS Pathog. 1, e39.
Donaldson E.F., Sims A.C., Graham R.L., Denison M.R., Baric R.S. (2007) Murine hepatitis virus replicase protein nsp10 is a critical regulator of viral RNA synthesis. J. Virol. 81, 6356–6368.
Bouvet M., Debarnot C., Imbert I., Selisko B., Snijder E.J., Canard B., Decroly E. (2010) In vitro reconstitution of SARS-coronavirus mRNA cap methylation. PLoS Pathog. 6, e1000863.
Bouvet M., Imbert I., Subissi L., Gluais L., Canard B., Decroly E. (2012) RNA 3'-end mismatch excision by the severe acute respiratory syndrome coronavirus nonstructural protein nsp10/nsp14 exoribonuclease complex. Proc. Natl. Acad. Sci. USA. 109, 9372–9377.
Shu T., Huang M., Wu D., Ren Y., Zhang X., Han Y., Mu J., Wang R., Qiu Y., Zhang D.-Y., Zhou X. (2020) SARS-coronavirus-2 Nsp13 possesses NTPase and RNA helicase activities that can be inhibited by bismuth salts. Virol. Sin. 35, 321–329.
Minskaia E., Hertzig T., Gorbalenya A.E., Campanacci V., Cambillau C., Canard B., Ziebuhr J. (2006) Discovery of an RNA virus 3′→5′ exoribonuclease that is critically involved in coronavirus RNA synthesis. Proc. Natl. Acad. Sci. USA. 103, 5108–5113.
Chen Y., Cai H., Pan J., Xiang N., Tien P., Ahola T., Guo D. (2009) Functional screen reveals SARS coronavirus nonstructural protein nsp14 as a novel cap N7 methyltransferase. Proc. Natl. Acad. Sci. USA. 106, 3484–3489.
Deng X., Hackbart M., Mettelman R.C., O’Brien A., Mielech A.M., Yi G., Kao C.C., Baker S.C. (2017) Coronavirus nonstructural protein 15 mediates evasion of dsRNA sensors and limits apoptosis in macrophages. Proc. Natl. Acad. Sci. USA. 114, E4251–E4260.
Forni D., Cagliani R., Clerici M., Sironi M. (2017) Molecular evolution of human coronavirus genomes. Trends Microbiol. 25, 35–48.
Li F. (2016) Structure, function, and evolution of coronavirus spike proteins. Annu. Rev. Virol. 3, 237–261.
Yeager C.L., Ashmun R.A., Williams R.K., Cardellichio C.B., Shapiro L.H., Look A.T., Holmes K.V. (1992) Human aminopeptidase N is a receptor for human coronavirus 229E. Nature. 357, 420–422.
Wu K., Li W., Peng G., Li F. (2009) Crystal structure of NL63 respiratory coronavirus receptor-binding domain complexed with its human receptor. Proc. Natl. Acad. Sci. USA. 106, 19970–19974.
Naskalska A., Dabrowska A., Szczepanski A., Milewska A., Jasik K.P., Pyrc K. (2019) Membrane protein of human coronavirus NL63 is responsible for interaction with the adhesion receptor. J. Virol. 93, e00355-19.
Desforges M., Desjardins J., Zhang C., Talbot P.J. (2013) The acetyl-esterase activity of the hemagglutinin-esterase protein of human coronavirus OC43 strongly enhances the production of infectious virus. J. Virol. 87, 3097–3107.
Raj V.S., Mou H., Smits S.L., Dekkers D.H.W., Müller M.A., Dijkman R., Muth D., Demmers J.A.A., Zaki A., Fouchier R.A.M., Thiel V., Drosten C., Rottier P.J.M., Osterhaus A.D.M.E., Bosch B.J., Haagmans B.L. (2013) Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC. Nature. 495, 251–254.
Kalra R.S., Kandimalla R. (2021) Engaging the spikes: heparan sulfate facilitates SARS-CoV-2 spike protein binding to ACE2 and potentiates viral infection. Sig. Transduct. Target. Ther. 6(1), 39.
Hartenian E., Nandakumar D., Lari A., Ly M., Tucker J.M., Glaunsinger B.A. (2020) The molecular viro-logy of coronaviruses. J. Biol. Chem. 295, 12910–12934.
Tang T., Bidon M., Jaimes J.A., Whittaker G.R., Daniel S. (2020) Coronavirus membrane fusion mechanism offers a potential target for antiviral deve-lopment. Antiviral. Res. 178, 104792.
Sola I., Almazán F., Zúñiga S., Enjuanes L. (2015) Continuous and discontinuous RNA synthesis in coronaviruses. Annu. Rev. Virol. 2, 265–288.
Sawicki S.G., Sawicki D.L., Siddell S.G. (2007) A contemporary view of coronavirus transcription. J. Virol. 81, 20–29.
Sztuba-Solińska J., Stollar V., Bujarski J.J. (2011) Subgenomic messenger RNAs: mastering regulation of (+)-strand RNA virus life cycle. Virology. 412, 245–255.
Zúñiga S., Sola I., Alonso S., Enjuanes L. (2004) Sequence motifs involved in the regulation of discontinuous coronavirus subgenomic RNA synthesis. J. Virol. 78, 980–994.
Wu H.-Y., Brian D.A. (2007) 5'-proximal hot spot for an inducible positive-to-negative-strand template switch by coronavirus RNA-dependent RNA polymerase. J. Virol. 81, 3206–3215.
Bojkova D., Klann K., Koch B., Widera M., Krause D., Ciesek S., Cinatl J., Münch C. (2020) Proteomics of SARS-CoV-2-infected host cells reveals therapy targets. Nature. 583, 469‒472.
Finkel Y., Gluck A., Nachshon A., Winkler R., Fisher T., Rozman B., Mizrahi O., Lubelsky Y., Zuckerman B., Slobodin B., Yahalom-Ronen Y., Tamir H., Ulitsky I., Israely T., Paran N., Schwartz M., Stern-Ginossar N. (2021) SARS-CoV-2 uses a multipronged strategy to impede host protein synthesis. Nature. 594, 240–245.
Woo J., Lee E.Y., Lee M., Kim T., Cho Y.-E. (2019) An in vivo cell-based assay for investigating the specific interaction between the SARS-CoV N-protein and its viral RNA packaging sequence. Biochem. Biophys. Res. Commun. 520, 499–506.
Narayanan K., Makino S. (2001) Cooperation of an RNA packaging signal and a viral envelope protein in coronavirus RNA packaging. J. Virol. 75, 9059–9067.
Vennema H., Godeke G.J., Rossen J.W., Voorhout W.F., Horzinek M.C., Opstelten D.J., Rottier P.J. (1996) Nucleocapsid-independent assembly of coronavirus-like particles by co-expression of viral envelope protein genes. EMBO J. 15, 2020–2028.
Hsieh P.-K., Chang S.C., Huang C.-C., Lee T.-T., Hsiao C.-W., Kou Y.-H., Chen I.-Y., Chang C.-K., Huang T.-H., Chang M.-F. (2005) Assembly of severe acute respiratory syndrome coronavirus RNA packaging signal into virus-like particles is nucleocapsid dependent. J. Virol. 79, 13848–13855.
Masters P.S. (2019) Coronavirus genomic RNA packaging. Virology. 537, 198–207.
Yao S., Narayanan A., Majowicz S., Jose J., Archetti M. (2021) A synthetic defective interfering SARS-CoV-2. Peer J. 9, e11686.
Kuo L., Hurst-Hess K.R., Koetzner C.A., Masters P.S. (2016) Analyses of coronavirus assembly interactions with interspecies membrane and nucleocapsid protein chimeras. J. Virol. 90, 4357–4368.
Orenstein J.M., Banach B., Baker S. (2008) Morphogenesis of coronavirus HCoV-NL63 in cell culture: a transmission electron microscopic study. Open Infect. Dis. J. 2, 52–58.
Makino S., Lai M.M. (1990) Studies of coronavirus DI RNA replication using in vitro constructed DI cDNA clones. Adv. Exp. Med. Biol. 276, 341–347.
Kim K.H., Narayanan K., Makino S. (1998) Characterization of coronavirus DI RNA packaging. Adv. Exp. Med. Biol. 440, 347–353.
Bos E.C., Dobbe J.C., Luytjes W., Spaan W.J. (1997) A subgenomic mRNA transcript of the coronavirus mouse hepatitis virus strain A59 defective interfering (DI) RNA is packaged when it contains the DI packa-ging signal. J. Virol. 71, 5684–5687.
Makino S., Keck J.G., Stohlman S.A., Lai M.M. (1986) High-frequency RNA recombination of murine coronaviruses. J. Virol. 57, 729–737.
Williams G.D., Chang R.-Y., Brian D.A. (1999) A phylogenetically conserved hairpin-type 3′ untransla-ted region pseudoknot functions in coronavirus RNA replication. J. Virol. 73, 8349–8355.
Almazán F., González J.M., Pénzes Z., Izeta A., Calvo E., Plana-Durán J., Enjuanes L. (2000) Engineering the largest RNA virus genome as an infectious bacterial artificial chromosome. Proc. Natl. Acad. Sci. USA. 97, 5516–5521.
Strauss-Soukup J.K., Strobel S.A. (2001) Ribozyme enzymology. In: RNA. Eds Söll D., Nishimura S., Moore P. Pergamon, Oxford, pp. 187–206.
Yang D., Leibowitz J.L. (2015) The structure and functions of coronavirus genomic 3' and 5' ends. Virus Res. 206, 120–133.
Yang J., Sun B., Huang H., Jiang Y., Diao L., Chen B., Xu C., Wang X., Liu J., Jiang W., Yang S. (2014) High-efficiency scarless genetic modification in Escherichia coli by using lambda red recombination and I-SceI cleavage. Appl. Environ. Microbiol. 80, 3826–3834.
St-Jean J.R., Desforges M., Almazán F., Jacomy H., Enjuanes L., Talbot P.J. (2006) Recovery of a neurovirulent human coronavirus OC43 from an infectious cDNA clone. J. Virol. 80, 3670–3674.
Almazán F., DeDiego M.L., Sola I., Zuñiga S., Nieto-Torres J.L., Marquez-Jurado S., Andrés G., Enjuanes L. (2013) Engineering a replication-competent, propagation-defective middle east respiratory syndrome coronavirus as a vaccine candidate. mBio, 4, e00650-13.
Pfefferle S., Krähling V., Ditt V., Grywna K., Mühlberger E., Drosten C. (2009) Reverse genetic characterization of the natural genomic deletion in SARS-coronavirus strain Frankfurt-1 open reading frame 7b reveals an attenuating function of the 7b protein in-vitro and in-vivo. Virol. J. 6, 131.
Ye C., Chiem K., Park J.-G., Oladunni F., Platt R. N., Anderson T., Almazan F., de la Torre J.C., Martinez-Sobrido L. (2020) Rescue of SARS-CoV-2 from a single bacterial artificial chromosome. mBio. 11(5), e02168-20. https://doi.org/10.1128/mBio.02168-20
Thi Nhu Thao T., Labroussaa F., Ebert N., V’kovski P., Stalder H., Portmann J., Kelly J., Steiner S., Holwerda M., Kratzel A., Gultom M., Schmied K., Laloli L., Hüsser L., Wider M., Pfaender S., Hirt D., Cippà V., Crespo-Pomar S., Schröder S., Muth D., Niemeyer D., Corman V.M., Müller M.A., Drosten C., Dijkman R., Jores J., Thiel V. (2020) Rapid reconstruction of SARS-CoV-2 using a synthetic genomics platform. Nature. 582, 561–565.
Eriksson K.K., Makia D., Thiel V. (2008) Generation of recombinant coronaviruses using vaccinia virus as the cloning vector and stable cell lines containing coronaviral replicon RNAs. Methods Mol. Biol. 454, 237–254.
Donaldson E.F., Yount B., Sims A.C., Burkett S., Pickles R.J., Baric R.S. (2008) Systematic assembly of a full-length infectious clone of human coronavirus NL63. J. Virol. 82, 11948–11957.
Scobey T., Yount B.L., Sims A.C., Donaldson E.F., Agnihothram S.S., Menachery V.D., Graham R.L., Swanstrom J., Bove P.F., Kim J.D., Grego S., Randell S.H., Baric R.S. (2013) Reverse genetics with a full-length infectious cDNA of the Middle East respiratory syndrome coronavirus. Proc. Natl. Acad. Sci. USA. 110, 16157–16162.
Yount B., Curtis K.M., Fritz E.A., Hensley L.E., Jahrling P.B., Prentice E., Denison M.R., Geisbert T.W., Baric R.S. (2003) Reverse genetics with a full-length infectious cDNA of severe acute respiratory syndrome coronavirus. Proc. Natl. Acad. Sci. USA. 100, 12995–13000.
van den Worm S.H.E., Eriksson K.K., Zevenhoven J.C., Weber F., Züst R., Kuri T., Dijkman R., Chang G., Siddell S.G., Snijder E. J., Thiel V., Davidson A.D. (2012) Reverse genetics of SARS-related coronavirus using vaccinia virus-based recombination. PLoS One. 7, e32857.
Xie X., Muruato A., Lokugamage K.G., Narayanan K., Zhang X., Zou J., Liu J., Schindewolf, C., Bopp N.E., Aguilar P.V., Plante K.S., Weaver S.C., Makino S., LeDuc J.W., Menachery V.D., Shi P.Y. (2020) An infectious cDNA clone of SARS-CoV-2. Cell Host Microbe. 27, 841–848.e3.
Kouprina N., Larionov V. (2016) Transformation-associated recombination (TAR) cloning for genomics studies and synthetic biology. Chromosoma. 125, 621.
Cunningham C., Gatherer D., Hilfrich B., Baluchova K., Dargan D.J., Thomson M., Griffiths P.D., Wilkinson G.W.G., Schulz T.F., Davison A.J. (2010) Sequences of complete human cytomegalovirus genomes from infected cell cultures and clinical specimens. J.Gen. Virol. 91, 605.
Vashee S., Stockwell T.B., Alperovich N., Denisova E.A., Gibson D.G., Cady K.C., Miller K., Kannan K., Malouli D., Crawford L.B., Voorhies A.A., Bruening E., Caposio P., Früh K. (2017) Cloning, assembly, and modification of the primary human cytomegalovirus isolate Toledo by yeast-based transformation-associated recombination. mSphere. 2, e00331-17.
Oldfield L.M., Grzesik P., Voorhies A.A., Alperovich N., MacMath D., Najera C.D., Chandra D.S., Prasad S., Noskov V.N., Montague M. G., Friedman R.M., Desai P.J., Vashee S. (2017) Genome-wide engineering of an infectious clone of herpes simplex virus type 1 using synthetic genomics assembly methods. Proc. Natl. Acad. Sci. USA. 114, E8885–E8894.
Smith S., Reuven N., Mohni K.N., Schumacher A.J., Weller S.K. (2014) Structure of the herpes simplex virus 1 genome: manipulation of nicks and gaps can abrogate infectivity and alter the cellular DNA damage response. J. Virol. 88, 10146–10156.
Yount B., Curtis K.M., Baric R.S. (2000) Strategy for systematic assembly of large rna and dna genomes: transmissible gastroenteritis virus model. J. Virol. 74, 10600–10611.
Merchlinsky M., Moss B. (1992) Introduction of foreign DNA into the vaccinia virus genome by in vitro ligation: recombination-independent selectable cloning vectors. Virology. 190, 522–526.
Mohsen M.O., Zha L., Cabral-Miranda G., Bachmann M.F. (2017) Major findings and recent advances in virus-like particle (VLP)-based vaccines. Semin. Immunol. 34, 123–132.
Nooraei S., Bahrulolum H., Hoseini Z.S., Katalani C., Hajizade A., Easton A.J., Ahmadian G. (2021) Virus-like particles: preparation, immunogenicity and their roles as nanovaccines and drug nanocarriers. J. Nanobiotechnology. 19, 59.
Chan S.K., Du P., Ignacio C., Mehta S., Newton I.G., Steinmetz N.F. (2021) Biomimetic virus-like particles as severe acute respiratory syndrome coronavirus 2 diagnostic tools. ACS Nano. 15, 1259–1272.
Bos E.C., Luytjes W., Spaan W.J. (1997) The function of the spike protein of mouse hepatitis virus strain A59 can be studied on virus-like particles: cleavage is not required for infectivity. J. Virol. 71, 9427–9433.
Naskalska A., Dabrowska A., Nowak P., Szczepanski A., Jasik K., Milewska A., Ochman M., Zeglen S., Rajfur Z., Pyrc K. (2018) Novel coronavirus-like particles targeting cells lining the respiratory tract. PLoS One. 13, e0203489.
Huang Y., Yang Z., Kong W., Nabel G.J. (2004) Ge-neration of synthetic severe acute respiratory syndrome coronavirus pseudoparticles: implications for assembly and vaccine production. J. Virol. 78, 12557–12565.
Siu Y.L., Teoh K.T., Lo J., Chan C.M., Kien F., Escriou N., Tsao S.W., Nicholls J.M., Altmeyer R., Peiris J.S., Bruzzone R., Nal B. (2008) The M, E, and N structural proteins of the severe acute respiratory syndrome coronavirus are required for efficient assembly, trafficking, and release of virus-like particles. J. Virol. 82, 11318–11330.
Swann H., Sharma A., Preece B., Peterson A., Eldridge C., Belnap D.M., Vershinin M., Saffarian S. (2020) Minimal system for assembly of SARS-CoV-2 virus like particles. Sci. Rep. 10, 21877.
Xu R., Shi M., Li J., Song P., Li N. (2020) Construction of SARS-CoV-2 virus-like particles by mammalian expression system. Front. Bioeng. Biotechnol. 8, 862.
Ju X., Zhu Y., Wang Y., Li J., Zhang J., Gong M., Ren W., Li S., Zhong J., Zhang L., Zhang Q.C., Zhang R., Ding Q. (2021) A novel cell culture system modeling the SARS-CoV-2 life cycle. PLoS Pathog. 17, e1009439.
Wang C., Zheng X., Gai W., Zhao Y., Wang H., Wang H., Feng N., Chi H., Qiu B., Li N., Wang T., Gao Y., Yang S., Xia X. (2016) MERS-CoV virus-like particles produced in insect cells induce specific humoural and cellular imminity in rhesus macaques. Oncotarget. 8, 12686–12694.
Talbot P.J., Ekandé S., Cashman N.R., Mounir S., Stewart J.N. (1993) Neurotropism of human coronavirus 229E. Adv. Exp. Med. Biol. 342, 339–346.
Thiel V., Karl N., Schelle B., Disterer P., Klagge I., Siddell S.G. (2003) Multigene RNA vector based on coronavirus transcription. J. Virol. 77, 9790–9798.
Khromykh A.A. (2000) Replicon-based vectors of positive strand RNA viruses. Curr. Opin. Mol. Ther. 2, 555–569.
Kotaki T., Xie X., Sh, P.-Y., Kameoka M. (2021) A PCR amplicon-based SARS-CoV-2 replicon for antiviral evaluation. Sci. Rep. 11, 2229.
He X., Quan S., Xu M., Rodriguez S., Goh S.L., Wei J., Fridman A., Koeplinger K.A., Carroll S.S., Grobler J.A., Espeseth A.S., Olsen D.B., Hazuda D.J., Wang D. (2021) Generation of SARS-CoV-2 reporter replicon for high-throughput antiviral screening and testing. Proc. Natl. Acad. Sci. USA. 118, e2025866118.
Ge F., Luo Y., Liew P.X., Hung E. (2007) Derivation of a novel SARS–coronavirus replicon cell line and its application for anti-SARS drug screening. Virology. 360, 150–158.
Almazán F., DeDiego M.L., Galán C., Escors D., Álvarez E., Ortego J., Sola I., Zuñiga S., Alonso S., Moreno J.L., Nogales A., Capiscol C., Enjuanes L. (2006) Construction of a severe acute respiratory syndrome coronavirus infectious cDNA clone and a replicon to study coronavirus RNA synthesis. J. Virol. 80, 10900–10906.
Hertzig T., Scandella E., Schelle B., Ziebuhr J., Siddell S.G., Ludewig B., Thiel V. (2004) Rapid identification of coronavirus replicase inhibitors using a selectable replicon RNA. J. Gen. Virol. 85, 1717–1725.
Wang J.-M., Wang L.-F., Shi Z.-L. (2008) Construction of a non-infectious SARS coronavirus replicon for application in drug screening and analysis of viral protein function. Biochem. Biophys. Res. Commun. 374, 138–142.
Luo Y., Yu F., Zhou M., Liu Y., Xia B., Zhang X., Liu J., Zhang J., Du Y., Li R., Wu L., Zhang X., Pan T., Guo D., Peng T., Zhang H. (2021) Engineering a reliable and convenient SARS-COV-2 replicon system for analysis of viral RNA synthesis and screening of antiviral inhibitors. mBio. 12, e02754-20. https://doi.org/10.1128/mBio.02754-20
McBride R., van Zyl M., Fielding B.C. (2014) The coronavirus nucleocapsid is a multifunctional protein. Viruses. 6, 2991–3018.
Dutta N.K., Mazumdar K., GordyJ.T. (2020) The nucleocapsid protein of SARS-COV-2: a target for vaccine development. J. Virol. 94, e00647-20.
Ge F., Xiong S., Lin F.S., Zhang Z.P., Zhang X.E. (2008) High-throughput assay using a GFP-expressing replicon for SARS-CoV drug discovery. Antiviral Res. 80, 107–113.
Pfefferle S., Schöpf J., Kögl M., Friedel C.C., Müller M.A., Carbajo-Lozoya J., Stellberger T., von Dall’Armi E., Herzog P., Kallies S., Niemeyer D., Ditt V., Kuri T., Züst R., Pumpor K., Hilgenfeld R., Schwarz F., Zimmer R., Steffen I., Weber F., Thiel V., Herrler G., Thiel H.J., Schwegmann-Wessels C., Pöhlmann S., Haas J., Drosten C., von Brunn A. (2011) The SARS-coronavirus-host interactome: identification of cyclophilins as target for pan-coronavirus inhibitors. PLoS Pathog. 7, e1002331.
Jin Y.-Y., Lin H., Cao L., Wu W.-C., Ji Y., Du L., Jiang Y., Xie Y., Tong K., Xing F., Zheng F., Shi M., Pan J.A., Peng X., Guo D. (2021) A convenient and biosafe replicon with accessory genes of SARS-CoV-2 and its potential application in antiviral drug disco-very. Virol. Sin. 36(5), 913–923. https://doi.org/10.1007/s12250-021-00385-9
Wang B., Zhang C., Lei X., Ren L., Zhao Z., Wang J., Huang H. (2021) Construction of non-infectious SARS-CoV-2 replicons and their application in drug evaluation. Virol. Sin. 36, 890–900.https://doi.org/10.1007/s12250-021-00369-9
Zhang Q.-Y., Deng C.-L., Liu J., Li J.-Q., Zhang H.-Q., Li N., Zhang Y.-N., Li X.-D., Zhang B., Xu Y., Ye H.-Q. (2021) SARS-CoV-2 replicon for high-throughput antiviral screening. J. Gen. Virol. 102, 001583.
Ahn D.-G., Lee W., Choi J.-K., Kim S.-J., Plant E.P., Almazán F., Taylor D.R., Enjuanes L., Oh J.-W. (2011) Interference of ribosomal frameshifting by antisense peptide nucleic acids suppresses SARS coronavirus replication. Antiviral Res. 91, 1–10.
Adedeji A.O., Marchand B., te Velthuis A.J.W., Snijder E.J., Weiss S., Eoff R.L., Singh K., Sarafianos S.G. (2012) Mechanism of nucleic acid unwinding by SARS-CoV helicase. PLoS One. 7, e36521.
Ivanov K.A., Hertzig T., Rozanov M., Bayer S., Thiel V., Gorbalenya A.E., Ziebuhr J. (2004) Major genetic marker of nidoviruses encodes a replicative endoribonuclease. Proc. Natl. Acad. Sci. USA. 101, 12694–12699.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология