Молекулярная биология, 2022, T. 56, № 1, стр. 103-117
Взаимодействие MxyR Mycobacterium tuberculosis c ксиланами: необычные лиганды транскрипционных регуляторов семейства MarR
S. Mauran a, N. T. Perera b, I. C. Perera a, *
a Synthetic Biology Laboratory, Department of Zoology and Environment Sciences, Faculty of Science,
University of Colombo
00700 Colombo 03, Sri Lanka
b Department of Chemistry, Faculty of Applied Sciences, University of Sri Jayewardenepura
Nugegoda, Sri Lanka
* E-mail: icperera@sci.cmb.ac.lk
Поступила в редакцию 19.02.2021
После доработки 12.04.2021
Принята к публикации 28.04.2021
- EDN: OLVEUZ
- DOI: 10.31857/S0026898422010074
Аннотация
Ген rv3095 Mycobacterium tuberculosis, принадлежащий семейству транскрипционных регуляторов MarR, кодирует фактор MxyR (Mycobacterium xylanase regulator – регулятор ксиланазы Mycobacterium). Этот ген расположен дивергентно по отношению к генам, кодирующим гидролазу (rv3094c), оксидоредуктазу (rv3093c), белок-транспортер семейства ABC (rv3092c), и конвергентно по отношению к гену ксиланазы (rv3096). Ксиланаза широко используется микробными патогенами растений для деградации ксиланов – основного компонента гемицеллюлозы. В работе методом сдвига электрофоретической подвижности исследованы молекулярные взаимодействия очищенного транскрипционного регулятора MxyR. Этот белок взаимодействует с расположенной выше межгенной областью mxyO с высокой специфичностью и константой диссоцииации, равной 5.01 ± 0.017 нМ. Примечательно, что это связывание ослабляется специфическими углеводными лигандами, такими как ксилан, L-арабиноза и D-галактоза, со значениями IC50, равными 22.7 ± 1.02 нг/мкл, 360.8 ± 24.25 нг/мкл и 2320.0 ± 96.97 мкг/мкл соответственно. Очевидно, эта ассоциация изменяет конформацию ДНК-связывающей спирали α4, вследствие чего MxyR теряет способность связываться с операторной ДНК, тем самым не препятствуя транскрипции регулируемых им генов ксиланазы и других белков. В результате проведенного исследования идентифицированы природные лиганды MxyR M. tuberculosis, что углубляет наши знания относительно метаболической регуляции углевода ксилана.
Туберкулез ‒ основная причина смертности и заболеваемости в развивающихся странах, отрицательно сказывающаяся на их экономической и социальной динамике. Согласно статистике Всемирной организации здравоохранения (ВОЗ) за 2019 год, 10 млн человек было инфицированы туберкулезом, из которых 12% ‒ дети [1 ] . В программе ВОЗ, названной “The End TB strategy”, поставлена задача победить это заболевание к 2030 году [1 ] . В рамках реализации этой стратегии изучение биологии Mycobacterium tuberculosis важно для поиска новых подходов для борьбы с заболеванием.
Изучение регуляции транскрипции ‒ одна из жизненно важных областей в изучении физиологии микроорганизмов. Ключевым механизмом регуляции выступают транскрипционные факторы, динамически связывающиеся с геномной ДНК и контролирующие транскрипцию генов [2, 3 ] . Семейство регуляторов транскрипции множественной антибиотикорезистентности (MarR) представляет значительный интерес, поскольку сообщалось о том, что они контролируют в том числе и вирулентность патогенных бактерий [2, 4‒7]. Регуляторные пути бактериальной вирулентности довольно сложны, так как несколько регуляторов могут действовать на экспрессию отдельного гена или один регулятор может действовать на экспрессию многих регуляторных доменов. Помимо вирулентности, члены регуляторов семейства MarR контролируют ряд адаптивных реакций бактерии, таких как ответ на окислительный стресс [8–11 ] , устойчивость к антибиотикам [12 ] , термочувствительность [13 ] и деградация фенольных соединений [3, 14, 15 ] .
Регуляторы транскрипции семейства MarR в M. tuberculosis могут выступать как специфическими, так и глобальными регуляторами, участвующими в ответе на лечение антибиотиками и внешнее воздействие. MosR (Rv1049), регулятор чувствительности к окислительному стрессу, активирует ген rv1050 M. tuberculosis, что позволяет бактерии выживать в окружающей среде, вызывающей окислительный стресс, например в макрофагах [16, 17 ] . Белок ответа на низкий pH, кодируемый rv1404; чувствительный к агенту на основе имидазо-[1,2-a]-пиридин-4-карбонитрила (MP-III-71) белок, кодируемый rv2887, и регулятор мембранных эффлюксных насосов Rv0678 считаются глобальными регуляторами. В регулон Rv1404 входит более тридцати генов, включая гены метилтрансфераз Rv1405c и Rv1403, участвующих в адаптивных процессах [18 ] . Белок Rv2887, определяющий чувствительность к MP-III-71, регулирует экспрессию гена rv0560c, кодирующего S-аденозил-L-метионинзависимую метилтрансферазу, и генов, участвующих в биосинтезе бензохинона и менохинона [19, 20 ] . Мембранный белок микобактерий, регулирующий экспрессию rv0678, связывается с промоторами оперонов mmpS2-mmpL2, mmpS4-mmpL4, rv0991 и mmpS5 [21 ] .
Транскрипционный фактор Rv3095 M. tuberculosis, который по результатам данного исследования предлагается переименовать в MxyR (Mycobacterium xylanase regulator – регулятор ксиланазы Mycobacterium), относится к семейству MarR, и его роль и физиологические функции мало изучены. Нами была подтверждена его регуляторная функция и описаны его природные лиганды. Кластер близкорасположенных генов включает предполагаемый ген ксиланазы, который обычно находят в патогенных бактериях растений и сапрофитах, ‒ фермента, расщепляющего ксилан на ксилозу и олигосахариды [22 ] . Ксилан, основной компонент гемицеллюлозы, представляет собой полисахарид, состоящий из линейной цепи ксилозы с замещениями в боковой цепи. Остов полисахарида в основном состоит из остатков 1,4-β-ксилопиранозида, которые могут содержать в боковой цепи ацетильную, арабинофуранозильную и 4-O-метилглюкуронильную группы ‒ в зависимости от источника полисахарида. Микроорганизмы, разлагающие ксилан, “снабжены” набором соответствующих ферментов: эндоксиланаз, β-ксилозидаз, α-L-арабинофуранозидаз, α-глюкуронидаз и эстераз [22, 23 ] , ‒ а также регуляторами транскрипции, такими как XynR и XlnR, для контроля биосинтеза ферментов, участвующих в процессе деградации ксилана [24–26]. Присутствие гена-гомолога ксиланаз в патогенной для человека бактерии M. tuberculosis объяснить трудно, исходя из известной функции этого фермента, но наводит на мысль о его пока неизвестной биологической роли. В этом исследовании в клетках M. tuberculos проанализирована регуляция экспрессии гипотетического гена ксиланазы транскрипционным фактором MxyR.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Определение структуры генетического локуса и моделирование структуры белка. Структура MxyR была смоделирована в программе SWISS-MODEL с использованием структуры PA1607 в качестве шаблона. Для валидации модели полученные углы всех пептидных связей наносили на диаграмму Рамачандрана [27]. Топологический порядок α-спиралей и β-листов определяли выравниванием моделируемой структуры MxyR по отношению к PA1607. Для анализа генетического локуса использовали карты депонированных геномов NCBI и программу NCBI BLASTp. Промоторные области, включая ‒35 и ‒10 и сайты начала транскрипции, были предсказаны с помощью программы BPROM из панели инструментов Softberry (“Softberry Inc.”, США; http://www.softberry.com/berry.phtml?topic=bprom&group=programs&subgroup=gfindb) [28].
Филогенетический анализ. Для поиска гомологов с помощью программы BLAST использовали пептидную последовательность MxyR из штамма M. tuberculosis H37Rv (Uniprot No. P9WMG3). Белковые последовательности с уровнем идентичности выше 60% отфильтровывали для исключения удвоений и сохраняли с NCBI-сервера локально в формате “fasta”. Полученные последовательности выравнивали в программе MEGA 6.06 с использованием алгоритма выравнивания ClustalW. Филогенетическое дерево выровненных последовательностей строили методом ближайших соседей (neighbor-joining). При валидации дерева методом бутстрепа использовали 1000 реплик. Дерево построено в масштабе, а эволюционные расстояния рассчитаны с использованием метода поправки Пуассона в единицах числа аминокислотных замен.
Выделение геномной ДНК. Культуру Mycobacterium tuberculosis H37Rv выращивали на скошенной среде Левенштейна–Йенсена (LJ) и использовали для выделения геномной ДНК. Колонии собирали в отдельный флакон с буфером для экстракции (50 мМ Трис-HCl, pH 7.4, и 20 мМ EDTA, pH 8.0) и прогревали при 80°C в течение 20 мин. Твердую клеточную стенку разрушали с помощью стеклянных шариков и инкубировали с лизоцимом в течение 2 ч при 37°C. Лизированные клетки обрабатывали 20 мг/мл протеиназы K в протеиназном буфере (100 мМ Трис-HCl, pH 7.8, 50 мМ EDTA и 5% SDS) и инкубировали при 45°C в течение ночи. Клеточный дебрис осаждали центрифугированием при 3000 g в течение 20 мин, а супернатант экстрагировали смесью фенол‒хлороформ‒изоамиловый спирт (25 : 24 : 1) с последующим осаждением этанолом. Выделенную ДНК анализировали с помощью горизонтального электрофореза в агарозном геле.
Клонирование и выделение белка. Ген rv3095, кодирующий регуляторный белок MxyR (новое обозначение введено на основании результатов проведенного исследования), амплифицировали с использованием праймеров MxyR_Fw и MxyR_Rv (табл. S1 , см. Приложение на сайте http://www. molecbio.ru/downloads/2022/1/supp_Mauran_rus.pdf). ПЦР проводили в объеме 25 мкл, содержащем 70 нг геномной ДНК, 10 мМ Трис-HCl (рН 8.8), 50 мМ KCl, 2 мМ MgCl2, 20 мкМ каждого dNTP, 20 мкМ праймеров и 2.5 единицы ДНК-полимеразы Taq (“Sigma”, США). Реакцию проводили в следующем режиме: начальная денатурация при 95°C в течение 5 мин, затем 30 циклов, включающих денатурацию при 95°C в течение 1 мин, отжиг при 55°С в течение 30 с и элонгацию при 72°C в течение 1 мин; и в конце ‒ финальная элонгация в течение 10 мин.
Амплифицированный продукт клонировали в экспрессионный вектор pET28a c введением гистидиновой метки (His-tag) на N-конец белка и трансформировали в Escherichia coli TOP10F'. Отсутствие ошибок в генетической конструкции подтверждали секвенированием по Сэнгеру. Экспрессионный штамм E. coli BL21 (DE3) PlysS трансформировали рекомбинантной плазмидой для наработки белка. Ночную культуру разбавляли в 500 раз и выращивали в 2 л среды LB (содержащей 0.5 мкг/мл канамицина) до оптической плотности 0.5 при длине волны 600 нм, соответствующей экспоненциальной фазе роста. Для индукции экспрессии добавляли 0.5 мМ изопропил-β-D-1-тиогалактопиранозид (IPTG) и продолжали культивирование в течение 2 ч, после чего охлаждали на льду в течение 20 мин. Бактериальные клетки собирали центрифугированием при 4500 g в течение 20 мин при 4°C и хранили при ‒80°C.
Для выделения целевого белка замороженную биомассу оттаивали, ресуспендировали в буфере для лизиса (LB): 20 мМ HEPES, pH 7.4, 50 мМ KCl, 4.5% глицерин, 20 мМ имидазол, 0.15 мМ PMSF, 10 мМ β-меркаптоэтанол ‒ и инкубировали с 0.5 мкг/мкл лизоцима в течение 1 ч при 4°C, периодически встряхивая. Лизис останавливали добавлением 0.05% Triton Х-100 в 500 мМ NaCl. Полученную суспензию обрабатывали ультразвуком в течение 5 мин (импульс 10 с с интервалом 10 с) при 4°C, центрифугировали и супернатант инкубировали с уравновешенными в LB гранулами Ni-NTA при 4°C в течение 1 ч при встряхивании; промывали тем же буфером и элюировали целевой белок ступенчатым градиентом имидазола, начиная с 20 мМ до 1 М. Наиболее чистые фракции собирали и проводили диализ против буфера: 20 мМ HEPES, pH 7.4, 50 мМ KCl, 4.5% глицерин, 0.15 мМ PMSF, 10 мМ β-меркаптоэтанол ‒ до разведения 1 : 1000. Диализованный очищенный белок доводили до концентрации 1 мг/мл с помощью концентраторов с отсечкой по молекулярной массе (MWCO) (“Thermo Fisher Scientific”, США). В наиболее чистых фракциях содержание глицерина доводили до 20% (v/v) и помещали их на хранение при ‒80°C.
Степень очистки выделенного белка анализировали электрофорезом в SDS-ПААГ с использованием бычьего сывороточного альбумина (BSA) в качестве стандарта. Для количественного определения белка гель, окрашенный Coomassie Brilliant Blue, анализировали с помощью программного обеспечения ImageJ.
Сшивание белков. Олигомерное состояние белка определяли по образованию белок-белковых сшивок в буфере, содержащем 20 мМ HEPES, рН 6.0, и 50 мМ NaCl, в присутствии 0.00125% глутаральдегида. Инкубацию проводили с белком и комплексом белок‒ДНК в течение 2 мин, реакцию останавливали добавлением 1 М Трис-HCl (pH 8.0) с последующим добавлением SDS-содержащего буфера для нанесения образцов.
Влияние ДНК на олигомерное состояние MxyR определяли по содержанию олигомера, образующегося в результате обработки глутаральдегидом MxyR, предварительно проинкубированного с операторной ДНК mxyO в концентрации 10, 50, 100 и 200 нМ.
Сдвиг электрофоретический подвижности. Межгенную область mxyO, которая, как предполагается, является промоторной областью, амплифицировали с помощью ПЦР из геномной ДНК M. tuberculosis H37Rv с праймерами mxyO_Fw и mxyO_Rv (табл. S1 ). ПЦР проводили в тех же условиях, которые описаны выше, за исключением стадии отжига, проводившейся при 58°C в течение 1 мин. Амплифицированный продукт фрагмента ДНК mxyO размером 81 п.н. очищали пассивной элюцией и/или электроэлюированием с последующей экстракцией смесью фенол‒хлороформ и осаждением этанолом.
Реакции связывания проводили в объеме 20 мкл, где очищенный белок MxyR в возрастающих концентрациях (4.66, 5.85, 7.26, 8.67, 13.88, 15.18 и 16.5 нМ) инкубировали с 9.5 нМ межгенной области mxyO в буфере для связывания (20 мМ Тris-HCl, pH 8.0, 0.06% Triton X-100, 1.5% глицерин) при 25°C в течение 30 мин. Комплексы разделяли в 12%-ном ПААГ в TAE-буфере (40 мМ Трис, 20 мМ AcOH, 1 мМ EDTA, pH 8.3) при 7.5 В/см в течение 3 ч при 4°C. Продукты визуализировали в ультрафиолетовом свете после окрашивания геля SYBR Green. Для количественного определения материала в полосах, соответствующих свободной ДНК и комплексам белок‒ДНК, использовали программу ImageJ; полученные данные анализировали и наносили на график с использованием программы GraphPad Prism 7.00 и аппроксимировали по уравнению Хилла для специфического связывания лигандов [29, 30]:
Специфичность связывания. Специфичность взаимодействия между MxyR и его промоторной областью mxyO определяли путем воздействия на комплекс белок‒ДНК конкурентной ДНК в избыточных молярных концентрациях (0.72, 1.43, 2.87, 4.3, 5.74 и 7.17 нМ) по сравнению с mxyO. В качестве конкурентной ДНК использовали линейную плазмиду pET28a.
Стехиометрия ДНК-белкового комплекса. Анализ сдвига электрофоретической подвижности белка при связывании ДНК проводили в трех повторах в вышеуказанных условиях при избыточных молярных концентрациях ДНК. Для количественного определения ДНК, как свободной, так и в комплексе с белком, в полосах геля использовали программу ImageJ [30]. Полученные данные наносили на график зависимости процентного содержания комплекса от соотношения концентраций MxyR в комплексе и в межгенной области mxyO и анализировали с помощью GraphPad Prism 7.00 [29]. Значение x на пересечении между касательными к наклону и плато кривой использовали для расчета стехиометрии комплекса MxyR‒mxyO при насыщении.
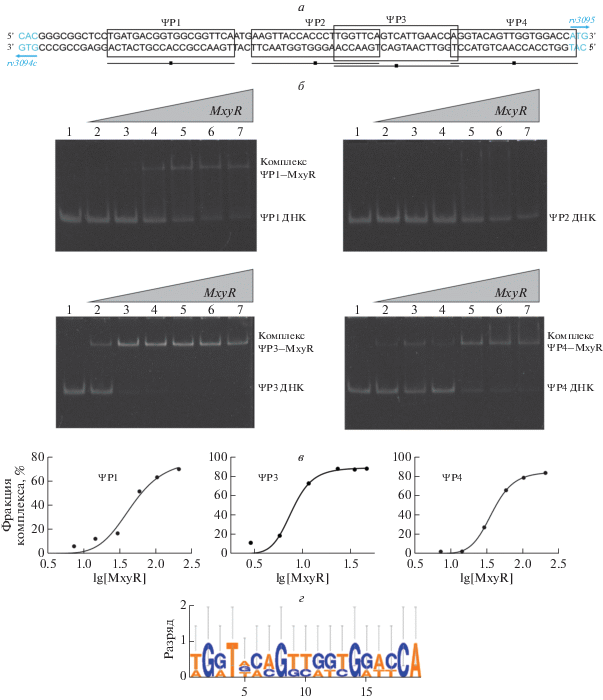
Связывание MxyR с псевдопалиндромными последовательностями. На основании стехиометрических результатов было предсказано четыре псевдопалиндромные последовательности в межгенной области mxyO. Псевдопалиндромные последовательности размером 19 п.н. предсказаны с использованием алгоритма EMBOSS:Palindrome, допускающего до 10 несовпадений для каждой последовательности (http://emboss.bioinformatics.nl/ cgi-bin/emboss/palindrome). Олигомерные последовательности предсказанных связывающихся псевдопалиндромов (табл. S1 ) были синтезированы в компании “Intergrated DNA Technologies” (США). Олигонуклеотиды растворяли в буфере для отжига дуплексов, свободном от нуклеаз (100 мМ KOAc, 30 мМ HEPES, pH 7.5). Равные молярные количества прямой и обратной цепей каждого псевдопалиндромного сайта смешивали и нагревали до 94°C в течение 2 мин, после чего оставляли при комнатной температуре. Полученные двухцепочечные палиндромы очищали электроэлюцией. Значения сдвига электрофоретической подвижности и константы диссоциации (Kd) рассчитывали для каждого сайта отдельно, как указано выше. Консенсусную операторную последовательность длиной 19 п.н. сгенерировали из предсказанных палиндромов с использованием программы WebLogo 3.7 (http://weblogo.threeplusone.com/create.cgi) [31].
Анализ in vitro связывания с предсказанными природными лигандами. Присутствие предполагаемого гена ксиланазы в опероне предполагает, что ксилан или любые продукты деградации ксилана могут действовать как естественные лиганды для комплекса белок‒ДНК. Две структурно разные молекулы ксилана: ксилан с боковыми группами арабинозы и глюкуроновой кислоты, экстрагированный из кукурузного початка (“Sisco”, США), ксилан с 4-O-метил-глюкуроновой кислотой, присоединенной к каждому 10 остатку ксилозы, экстрагированный из букового дерева (“Sigma”, США), ‒ а также мономер ксилана D(+)-ксилозу, структурно отличающийся пентозный сахар D(‒)-арабинозу и гексозный сахар галактозу тестировали против комплекса MxyR–mxyO. Выбранные лиганды растворяли в 50 мМ Na-цитратном буфере (pH 5.3) и инкубировали с MxyR, который брали в концентрации, сравнимой с Kd для комплекса MxyR–mxyO, в буфере, содержащем 20 мM Трис-HCl (pH 8.0), 0.06% Triton X-100 и 1.5% глицерина, при 25°C в течение 30 мин. Связывание с ДНК проводили в буфере, содержащем 20 мМ Трис-HCl, pH 8.0, 50 мМ NaCl и 1.5% глицерина, при 25°C в течение 30 мин. Реакционную смесь наносили на 12%-ный ПААГ гель в TAE-буфере (pH 8.3) и разделяли при 7.5 В/см в течение 3 ч при 4°C. Гель анализировали в ультрафиолетовом свете после окрашивания SYBR Green.
Кроме того, исследована аттенуация комплекса ДНК‒белок продуктами расщепления арабиноксилана и глюкуроноксилана. Для этого анализа использовали внеклеточную ксиланазу из Tricoderma virens. Фермент ксиланазу добавляли к ксилану в буфере, содержащем Трис-HCl (pH 8.0), и инкубировали при 37°C в течение ночи. Реакцию останавливали нагреванием при 70°C в течение 10 мин. Анализ связывания обработанных и необработанных ксиланов проводили аналогично описанному выше.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Структура генетического локуса
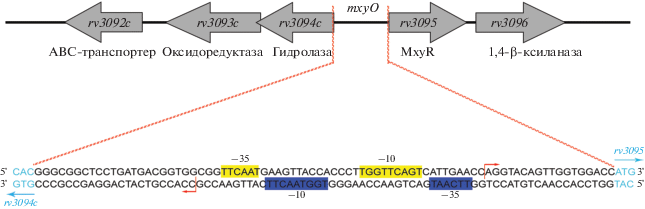
Ген rv3095 M. tuberculosis H37Rv кодирует транскрипционный регулятор семейства MarR длиной 158 аминокислотных остатков, который в этой работе упоминается как MxyR. Межгенная область mxyO длиной 81 п.н. разделяет гены rv3095 и rv3094c. Фланкирующие mxyO гены rv3095 и rv3096 ориентированы в одном направлении, тогда как rv3092c, rv3093c и rv3094c ориентированы в противоположном направлении (рис. 1). Межгенная область mxyO действует как общая промоторная, включающая регионы ‒35 и ‒10 дивергентно ориентированных промоторов и предполагаемые сайты связывания MxyR (рис. 1). Функцию белков, кодируемых генами rv3094c и rv3096, определяли путем поиска гомологов (BLASTp). Можно предположить, что гены rv3094c и rv3096 кодируют гидролазу (84% идентичности при 100%-ном покрытии) и 1,4-β-ксиланазу (83% идентичности при 98%-ном охвате). Наиболее близкие гомологи с охарактеризованной функцией обнаружены у нетуберкулезной микобактерии M. kansasii.
Рис. 1.
Генетическая организация области, прилегающей к mxyO и содержащей предполагаемые сайты связывания MxyR. Стрелками показаны точки старта и направления транскрипции. MxyR, 1,4-β-ксиланаза, гидролаза, оксидоредуктаза и ABC-транспортер кодируются генами rv3095, rv3096, rv3094c, rv3093c и rv3092c соответственно. Межгенный участок mxyO длиной 81 п.о., разделяющий гены, кодирующие MxyR и гидролазу, действует как общая промоторная область, включающая регионы ‒35 и ‒10, выделенные желтым и темно-синим цветом. Сайты начала транскрипции в обоих направлениях показаны красными изогнутыми стрелками, тогда как начало ORF MxyR и гидролазы указаны голубыми стрелками. Области промоторов и точки начала транскрипции предсказаны с помощью программы BPROM.
Филогенетический анализ
Последовательности результатов поиска Protein Blast (BLASTp) с более чем 60%-ным сходством были выровнены с ClustalW, и филогенетическое дерево было построено по методу ближайших соседей. Дерево было укоренено с помощью MarR Escherichia coli, при этом в качестве контроля использовали хорошо охарактеризованный гомолог из Pseudomonas aeruginosa Q9I3B4. При выравнивании MxyR с другими гомологами семейства MarR выявлено, что ДНК-узнающие α-спирали есть только в подмножестве гомологов, которое в основном включает MarR из видов микобактерий (рис. 2a и 2б). Помимо микобактерий, большинство других видов, которые, как предполагается, кодируют гомолог MxyR, относится к актиномицетам.
Рис. 2.
Филогенетический анализ белка MxyR. а ‒ Выравнивание последовательностей MxyR M. tuberculosis и его гомологов. б ‒ Эволюционные отношения MxyR и регуляторов транскрипции, для которых выявлена тесная связь с MxyR. Кластер, содержащий виды микобактерий, выделен синим цветом, MxyR ‒ красным. Дерево укоренено с помощью MarR E. coli в качестве внешней группы. Для каждого гомолога приведено видовое название и идентификатор записи в Uniprot. в ‒ Модель белка MxyR с выделением α-спиралей и β-листов. Каждый мономер (синий и серый) моделируется цепью A и B PA1607. ДНК-связывающие домены обоих мономеров обозначены желтым цветом.
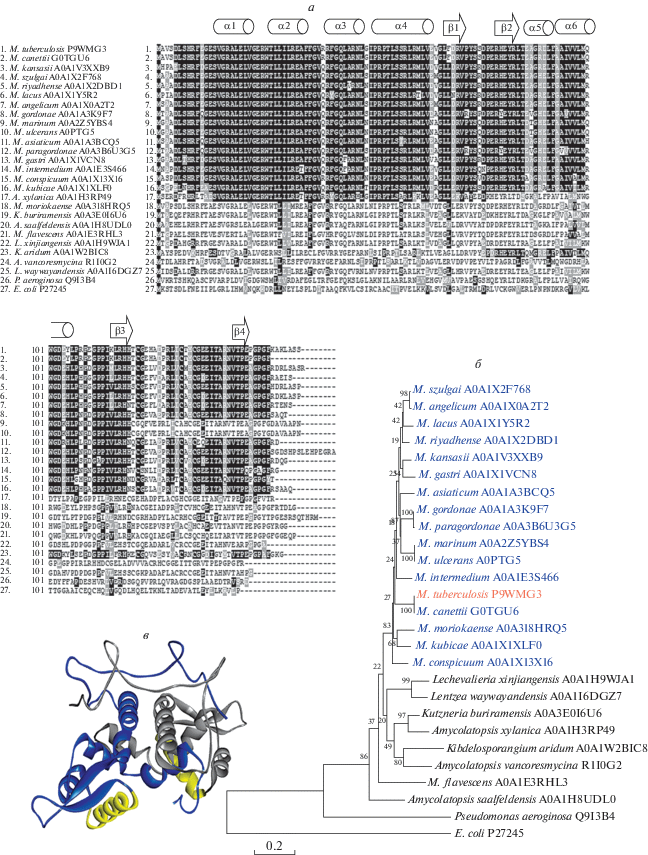
Структура MxyR смоделирована в программе SWISS с использованием PA1607 в качестве шаблона и подтверждена анализом с использованием графика Рамачандрана (рис. 2в и рис. S1 ). Построение показывает, что большинство остатков (91.5%) находится в предпочитаемой области и смоделированная структура имеет большое число параллельных β-листов и правосторонних α-спиралей (рис. S1 , см. Приложение на сайте http:// www.molecbio.ru/downloads/2022/1/supp_Mauran_rus.pdf).
Мономерная форма моделируемой структуры MxyR состоит из шести α-спиралей и четырех β-листов в топологическом порядке: α1‒ α2‒α3‒α4‒β1‒β2‒α5‒α6‒β3‒β4 (рис. 2в). Спирали α5 и α6 образуют длинную непрерывную спираль. Антипараллельные β-листы между спиралями α4 и α5 формируют мотив спираль-поворот-спираль (wHTH), тогда как другие два β-листа, β3 и β4, принимают участие в димеризации субъединиц. Димеризация в основном происходит через спирали α1, α2, α5 и α6 и стабилизируется ими, поскольку антипараллельное образование длинной спирали α5 и α6 важно для образования димера. Согласно структурному выравниванию MxyR с PA1607, спираль α4 идентифицирована как ДНК-узнающая, которая напрямую взаимодействует с операторной областью ДНК.
MxyR связывается с вышележащей промоторной областью mxyO
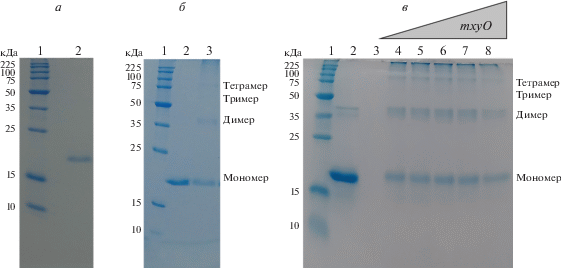
Для исследования связывания MxyR с mxyO ген rv3095, кодирующий MxyR, клонировали в экспрессионный вектор pET28a. Штамм E. coli BL21 (DE3) PlysS трансформировали рекомбинантной плазмидой и индуцировали сверхэкспрессию целевого белка, который затем удалось очистить почти до гомогенного состояния (рис. 3а). Олигомерную форму целевого белка анализировали через образованные глутаральдегидом сшивки (рис. 3б). Показано, что MxyR (Mr мономера ~21 кДа) образует димер (~42 кДа) в качестве основной фракции. Влияние ДНК на олигомерное состояние белка анализировали, используя увеличивающиеся концентраций mxyO: 10 → 50 → 100 → 200 нМ – против обработанного глутаральдегидом белка (рис. 3в). Показано, что связывание с ДНК не влияет на олигомерное состояние MxyR.
Рис. 3.
MxyR M. tuberculosis H37Rv преимущественно находится в виде димера. а ‒ Электрофоретический анализ очищенного белка MxyR (дорожка 2). б ‒ Анализ обработанного глутаральдегидом очищенного MxyR: MxyR (2), обработанный глутаральдегидом MxyR (3). в ‒ Сшивание MxyR глутаральдегидом в присутствии mxyO: MxyR (концентрация мономера 5 нМ) (2), ДНК mxyO (концентрация 200 нМ) (3), обработанный глутаральдегидом MxyR (4), обработанный глутаральдегидом MxyR в присутствии 10 (5), 50 (6), 100 (7) и 200 (8) нМ mxyO. Везде на дорожку 1 нанесен маркер молекулярной массы белков.
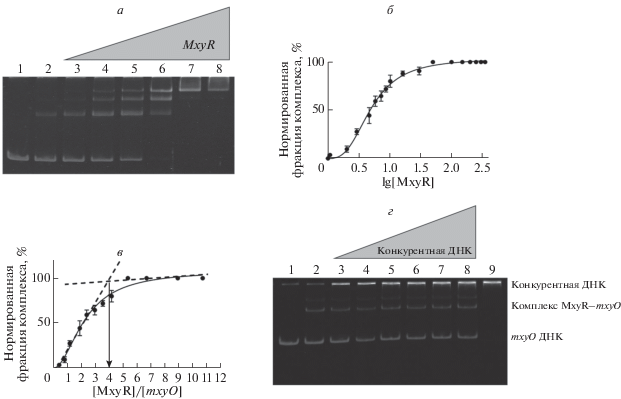
Связывание MxyR с областью mxyO исследовали по сдвигу электрофоретической подвижности. Межгенная область вместе с дополнительными 23 и 24 основаниями фланкирующих генов использована в качестве целевой ДНК mxyO (область между 3 463 867 и 3 463 995). Анализ сдвига и стехиометрические расчеты показали, что при увеличении концентрации белка образуется четыре комплекса (рис. 4a и 4в). Графическую зависимость образования комплекса MxyR‒mxyO от концентрации MxyR аппроксимировали по уравнению Хилла и рассчитали константу диссоциации (Kd 5.01 ± ± 0.017 нМ) (рис. 4б). На положительную кооперативность указывает коэффициент Хилла (n), равный 2.8 ± 0.19. В результате анализа образования комплекса белок‒ДНК в присутствии увеличивающихся концентраций ДНК pET28a подтверждена специфичность связывания MxyR с последовательностью mxyO (рис. 4г).
Рис. 4.
Связывание MxyR с ДНК mxyO. a ‒ Анализ сдвига электрофоретической подвижности MxyR с ДНК mxyO: свободная ДНК mxyO (1), продукты взаимодействия ДНК mxyO (9.5 нМ) с 4.66 (2), 5.85 (3), 7.26 (4), 8.67 (5), 13.88 (6), 15.18 (7) и 16.5 (8) нМ белка MxyR. б ‒ Нормализованная концентрация комплекса MxyR‒mxyO как функция концентрации MxyR (lg[MyxR]). Приведено стандартное отклонение (SD) по результатам трех независимых повторов. в ‒ Определение стехиометрии комплекса MxyR‒mxyO. График построен как зависимость образования комплекса в процентах от соотношения концентраций белка MxyR и ДНК mxyO. г ‒ Анализ специфичности связывания MxyR с ДНК: только ДНК mxyO (1); примерно 50% ДНК mxyO (5.01 нМ) находится в комплексе с MxyR (9.5 нМ) (2); к комплексу белок‒ДНК добавляли 0.72 (3), 1.43 (4), 2.87 (5), 4.3 (6), 5.74 (7) и 7.17 (8) нМ линейной ДНК pET28a в качестве потенциального конкурентного ингибитора; только ДНК pET28a (9).
MxyR связывается с псевдопалиндромными последовательностями
Псевдопалиндромы ΨP1, ΨP2, ΨP3 и ΨP4 (рис. 5а) в межгенной области mxyO были исследованы на потенциальные сайты связывания MxyR. В связи с тем, что MxyR ‒ регулятор транскрипции типа MarR, было предсказано, что он связывается с инвертированным повтором длиной 19 п.н. в области между mxyR и геном, кодирующим гидролазу. Показано, что среди предсказанных сайтов MxyR связывается с ΨP1, ΨP3 и ΨP4, причем с высоким сродством к ΨP3 (Kd = 7.7 ± ± 1.08 нМ) (рис. 5б и 5в). Для псевдопалиндромов ΨP1 и ΨP4 значения Kd были выше: 44.4 ± 1.48 нМ и 37.0 ± 1.02 нМ соответственно. Псевдопалиндромы ΨP3 и ΨP4 перекрываются друг с другом одной парой оснований. Выровненные последовательности использовали для создания консенсусной последовательности с помощью инструмента WebLogo (рис. 5г). Рассматривая консенсусную последовательность, можно заметить ее асимметричность с наличием нескольких высококонсервативных областей (G2T4G8 и G14C18A19).
Рис. 5.
Связывание MxyR с четырьмя псевдопалиндромными последовательностями. а ‒ Последовательность mxyO и предполагаемые связываемые MxyR псевдопалиндромные последовательности ΨP1, ΨP2, ΨP3 и ΨP4. б ‒ Анализ сдвига электрофоретической подвижности MxyR при взаимодействии с псевдопалиндромными последовательностями: свободная ДНК (1); к палиндромной ДНК добавляли белок MxyR в концентрации 15 (2), 30 (3), 60 (4), 100 (5), 200 (6) или 300 (7) нМ. в ‒ Зависимость образования комплексов MxyR‒ДНК от lg[MxyR]. г ‒ Консенсусная последовательность, с которой связывается MxyR. Высота букв оснований соответствует частоте встречаемости в каждой позиции.
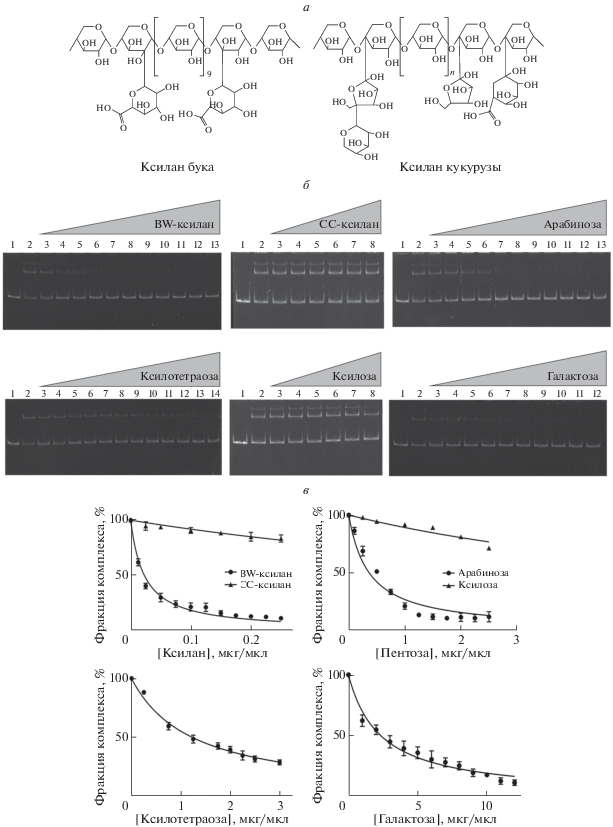
Связывание углеводов
Присутствие предполагаемого гена ксиланазы в исследованном кластере привело нас к исследованию промежуточных продуктов пути деградации ксилана в качестве потенциальных природных лигандов MxyR. Нами проанализировано два структурно различных варианта ксилана: арабиноксилан и глюкуроноксилан, ‒ субстраты фермента ксиланазы. Кроме того, в качестве контролей использовали D-ксилозу как сахар-пентозу, образующуюся после утилизации ксиланов ксиланазой; L-арабинозу как изомер D-ксилозы и D-галактозу как сахар-гексозу. Два структурно различных варианта ксилана происходят из двух разных растительных источников. Так, ксилан с боковыми группами 4-O-метилглюкуроновой кислоты, присоединенной к каждому 10 остатку ксилозы (далее: BW-ксилан), был получен из двудольного растения бука, а ксилан с боковыми группами арабинозы и глюкуроновой кислоты (далее: CC-ксилан) был получен из однодольного растения кукурузы (рис. 6а). Обнаружено, что растворимость CC-ксилана выше, чем BW-ксилана. Среди протестированных углеводов только BW-ксилан, L-арабиноза и D-галактоза ослабляли связывание MxyR с ДНК (рис. 6б и 6в). Хотя L‑арабиноза в высоких концентрациях действует как лиганд MxyR, содержащий L-арабинозу CC‑ксилан не “работает” ни в одной из исследованных концентраций. Оказалось, что комплекс MxyR‒mxyO с самой высокой электрофоретической подвижностью проявлял наибольшую стабильность при добавлении лигандов, то есть обладал наибольшей аффинностью связывания. Мы также исследовали стабильность комплекса MxyR‒mxyO в присутствии трех ксилоолигосахаридов: ксилобиозы, ксилотриозы и ксилотетраозы (рис. S2 и 6 б). Среди них только ксилотетраоза ослабляла связывание MxyR с ДНК (рис. 6б и 6в). Наибольшее ослабление связывания MxyR с ДНК наблюдали для BW-ксилана ‒ с IC50 22.74 ± ± 1.02 нг/мкл. Для других лигандов: L-арабинозы, D-галактозы и ксилотетраозы ‒ значения IC50 были гораздо выше: 360.8 ± 24.25, 2320 ± 96.97 и 1241 ± 35.24 нг/мкл соответственно (рис. 6в).
Рис. 6.
Кинетика связывания MxyR с углеводами. a ‒ Структура ксилана кукурузного початка с боковыми группами ксилоарабинозы и глюкуроновой кислоты (CC-ксилан) и ксилана бука с 4-O-метилглюкуроновой кислотой, присоединенной к каждому 10 остатку ксилозы (BW-ксилан). б ‒ Влияние углеводов на комплекс MxyR‒mxyO: ДНК mxyO (1), примерно 50% ДНК mxyO находится в комплексе с MxyR (2), комплекс белок‒ДНК в присутствии возрастающих указанных концентраций углеводов. в ‒ Количественный анализ содержания комплекса MxyR‒mxyO в зависимости от концентрации лиганда. Результаты представлены как среднее значение ± SD, рассчитанное на основании трех независимых экспериментов.
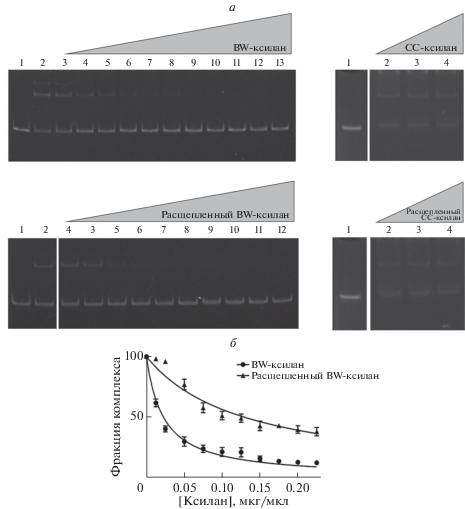
Кроме того, мы исследовали аттенуацию комплекса ДНК‒белок продуктами расщепления ксилана. Для этого использовали внеклеточную ксиланазу из Trichoderma virens. CC-ксилан ни в нативной, ни в расщепленной формах не влиял на стабильность комплекса, в то время как BW-ксилан “срабатывал” в обеих формах (рис. 7a и 7б); при этом нативный BW-ксилан в 6 раз эффективнее дестабилизировал комплекс MxyR‒mxyO по сравнению с обработанным ксиланазой препаратом (IC50 22.5 ± 1.04 и 130.0 ± 6.09 нг/мкл соответственно).
Рис.7.
Влияние расщепленных и нативных ксиланов на стабильность комплекса MxyR‒mxyO. а ‒ Анализ сдвига электрофоретической подвижности с расщепленными и нативными BW- и CC-ксиланами: ДНК mxyO (1), комплекс MxyR‒mxyO в равновесных условиях (2), на остальные дорожки нанесены образцы комплекса MxyR‒mxyO в присутствии возрастающих концентраций СС- или BW-ксилана. б ‒ Зависимость содержания комплекса MxyR‒mxyO от концентрации нативного или обработанного ксиланазой BW-ксилана.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Понимание регуляции экспрессии генов на транскрипционном уровне имеет решающее значение для анализа метаболизма любого организма. Уже много исследований посвящено изучению транскрипционных факторов M. tuberculosis, среди которых факторы семейства MarR привлекают большое внимание. Белки Rv1049 (MosR) [16 ] , Rv0880, Rv2887 [20, 32], Rv1404 [18 ] и Rv0678 [21 ] исследовали в аспекте их участия в ответе на окислительный и кислотный стрессы, реакции на воздействие лекарственных средств и глицериновых эфиров жирных кислот. Однако на факторы транскрипции семейства MarR, реагирующие на углеводы, обращали внимание редко, поэтому обнаружение их гомолога у M. tuberculosis было неожиданным ‒ обычно их находят в сапрофитах и патогенах растений [24, 26, 33]. Здесь интересующий кластер генов включает предполагаемый ген ксиланазы, который может быть функционально неактивен по отношению к хозяину патогена, человеку, в связи с отсутствием в его организме субстрата ‒ ксилана. 1,4-β-ксиланаза участвует в пути разложения ксилана и расщепляет его до ксилозы и/или ксилоолигосахаридов [34]. Присутствие предполагаемого гена ксиланазы в генном кластере предполагает, что на регуляцию транскрипции этого кластера влияет ксилан и/или интермедиаты, образующиеся при его деградации.
MxyR связывается с mxyO, который расположен между собственным геном (rv3095) и геном rv3094, кодирующим гидролазу, что позволяет предположить, что на регуляцию транскрипции может влиять связывание сигма-факторов с общей промоторной областью. Динамическое связывание MxyR с операторной областью может обеспечивать базальный уровень экспрессии mxyR. Благодаря оперонной структуре кластера, транскрипционная регуляция может распространяться не только на “собственную” экспрессию, но и на другие белки, кодируемые генами этого кластера, включая предполагаемый фермент ксиланазу. In vitro добавление BW-ксилана к комплексу MxyR‒mxyO значительно снижало его содержание. В клетке ксилан действует как лиганд MxyR и ослабляет его связывание с ДНК, что влияет на транскрипцию. В связи с тем, что связывание MxyR с промоторной областью может либо активировать, либо супрессировать транскрипцию гена ксиланазы, оно будет напрямую влиять на гомеостаз уровня ксилана в клетке.
Хотя ксилан в большом количестве содержится в растительном материале как неотъемлемая часть гемицеллюлозы, можно полагать, что M. tuberculosis не сталкивается с этим соединением в больших количествах в течение своего жизненного цикла. Возможно, что гипотетический ген ксиланазы происходит от предковых почвенных микобактерий. Филогенетический анализ выявил, что большинство гомологов MxyR принадлежат почвенным и другим бактериям окружающей среды (рис. 2a и 2б). Гомологи MxyR в основном связаны с условно-патогенными бактериями, при полном сходстве с транскрипционным фактором из M. canettii – вида, относящегося к комплексу Mycobacterium tuberculosis, ‒ а также с некоторыми другими нетуберкулезными бактериями, поражающими пациентов с ослабленным иммунитетом. Хотя эти бактерии поражают легочную систему человека, некоторые из этих видов были первоначально обнаружены и выделены из окружающей среды: M. szulgai и M. angelicum из пресноводных источников и пресноводных рыб [35, 36], M. marinum из морской воды и морских рыб [37], M. conspicuum и M. moriokaense из почвы [38, 39].
Помимо микобактерий, большинство других видов, содержащих гомолог MxyR, относится к актиномицетам, выделенными из почвы. Amycolaptosis vancoresmycina, Kutzneria buriramensis, A. xylanica и A. saalfeldensis относятся к семейству Pseudonocardiaceae. Это семейство актиномицетов имеет в составе клеточной стенки типа IV мезо-диаминопимелиновую кислоту, арабинозу и галактозу; MK-9(H4) в качестве основного менахинона и отсутствие миколовых кислот [40–43]. Lechevalieria xinjiangensis и Lentzea waywayandensis, принадлежащие к семейству Actinosynnemataceae, также кодируют гомолог MxyR. Семейство Actinosynnemataceae имеет состав клеточной стенки типа III с мезо-диаминопимелиновой кислотой, галактозой и маннозой или рамнозой [44, 45]. Почвенные бактерии A. saalfeldensis и A. xylanica используют L-арабинозу, D-фруктозу, D-глюкозу и D-ксилозу в качестве единственного источника углерода [40–43]. Присутствие гомологов MxyR в почвенных бактериях можно расценивать как подтверждение гипотезы о том, что этот регулятор пути деградации ксилана в M. tuberculosis, сохранился от предковых почвенных микобактерий, поскольку организм хозяина (человека) не продуцирует и не накапливает ксиланы в течение жизни. Однако, по всей видимости, у M. tuberculosis MxyR сохранился в активной форме для выполнения той же и/или другой физиологической функции.
В настоящее время нет сообщений о наличии у человека ксилана, ксилозы или родственных соединений. Однако в капсуле M. tuberculosis обнаружены следовые количества ксилана [46]. Присутствие гена ксиланазы в патогене, который не сталкивается с ксиланом, конечно, интригует. Между тем гены, кодирующие предполагаемые переносчики углеводов в M. tuberculosis, играют важную роль в использовании углеводов в качестве источника углерода только в течение первой недели заражения. Благодаря наличию адаптивной иммунной системы, этот организм может использовать липиды в качестве основного источника углерода на последующих стадиях своего инфекционного цикла [47]. Сапрофитные микобактерии используют более широкий спектр углеводов в качестве источника углерода и имеют большое число систем утилизации углеводов, тогда как в M. tuberculosis найдено только пять из них [44]. Однако эти углеводы не могут быть использованы в качестве источника углерода, поскольку они являются структурными компонентами клеточной оболочки микобактерий. Хотя M. tuberculosis не имеет доступа к ксилозе в естественной среде, эта бактерия обладает способностью синтезировать молекулы сахаров, которые необходимы для формирования ее структуры и/или физиологии, из других углеводов ‒ тех, которые могут быть ею усвоены [44]. Действительно, большая часть компонентов клеточной оболочки микобактерий синтезируется самим организмом [45, 48]. Следовательно, присутствие ксилана в капсуле M. tuberculosis и синтез этой бактерией необходимых углеводов из других усваиваемых ею сахаров позволяют говорить о возможности ею и синтеза ксилана. Таким образом, мы предполагаем, что клетки M. tuberculosis могут усваиваемые ими углеводы конвертировать в ксилан.
Нами показано, что ксилан с 4-O-метил-глюкуроновой кислотой, присоединенной к каждому 10 остатку ксилозы, действует как лиганд MxyR, ослабляя его связывание с ДНК (рис. 6). Помимо этого соединения, L-арабиноза и галактоза также действуют как лиганды MxyR, но пути утилизации всех этих соединений M. tuberculosis неизвестны. Есть собщения о значительном содержании производных арабинозы в оболочке микобактериальных клеток в виде арабиноманнана в капсуле и арабиногалактана, соединяющего миколовые кислоты с клеточной стенкой [46, 49].
Остается немало вопросов на пути к полному пониманию метаболизма M. tuberculosis. Выявление транскрипционных цепочек ‒ важнейшая задача, и проведенное исследование вносит вклад в понимание регуляторной функции необычных для этого патогена углеводов
В итоге определена роль белка MxyR как регулятора транскрипции из семейства MarR и идентифицированы его природные лиганды. Кроме того, изучен механизм регуляции экспрессии гена ксиланазы M. tuberculosis, высказано предположение о его происхождении от предковых почвенных бактерий. Исследованы природные лиганды MxyR, а для углеводов, которые присутствуют в клеточной стенке микобактерий: ксиланов с 4-O-метил-глюкуроновой кислотой, присоединенной к каждому 10 остатку ксилозы, L-арабинозы и галактозы, ‒ выявлена способность ослаблять связывание MxyR с ДНК, а значит их можно рассматривать как потенциальные лиганды MxyR. Полученные результаты ставят много вопросов и задают направления дальнейших исследований по физиологии M. tuberculosis.
Работа поддержана Национальным исследовательским советом Шри-Ланки (National Research Council, Sri Lanka; Grant No: NRC 15-063).
Cтатья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
World Health Organization. (2020) Global tuberculosis report 2020.
Perera I.C., Grove A. (2010) Urate is a ligand for the transcriptional regulator PecS. J. Mol. Biol. 402(3), 539–551.
Wilkinson S.P., Grove A. (2004) HucR, a novel uric acid-responsive member of the MarR family of transcriptional regulators from Deinococcus radiodurans. J. Biol. Chem. 279(49), 51442–51450.
Stapleton M.R., Norte V.A., Read R.C., Green J. (2002) Interaction of the Salmonella typhimurium transcription and virulence factor SlyA with target DNA and identification of members of the SlyA regulon. J. Biol. Chem. 277(20), 17630–17637.
Ellison D.W., Miller V.L. (2006) Regulation of virulence by members of the MarR/SlyA family. Curr. Opin. Microbiol. 9, 153–159.
Chang Y.M., Jeng W.Y., Ko T.P., Yeh Y.J., Chen C.K., Wang A.H. (2010) Structural study of TcaR and its complexes with multiple antibiotics from Staphylococcus epidermidis. Proc. Natl. Acad. Sci. USA. 107(19), 8617–8622.
Michaux C., Sanguinetti M., Reffuveille F., Auffray Y., Posteraro B., Gilmore M.S., Hartke A., Giard J.C. (2011) SlyA is a transcriptional regulator involved in the virulence of Enterococcus faecalis. Infect. Immun. 79(7), 2638–2645.
Birukou I., Seo S.M., Schindler B.D., Kaatz G.W., Brennan R.G. (2014) Structural mechanism of transcription regulation of the Staphylococcus aureus multidrug efflux operon mepRA by the MarR family repressor MepR. Nucleic Acids Res. 42(4), 2774–2788.
Kim H., Choe J. (2013) The X-ray crystal structure of PA1374 from Pseudomonas aeruginosa, a putative oxidative-stress sensing transcriptional regulator. Biochem. Biophys. Res. Commun. 431(3), 376–381.
Palm G.J., Khanh Chi B., Waack P., Gronau K., Becher D., Albrecht D., Hinrichs W., Read R.J., Antelmann H. (2012) Structural insights into the redox-switch mechanism of the MarR/DUF24-type regulator HypR. Nucleic Acids Res. 40(9), 4178–4192.
Aoki R., Takeda T., Omata T., Ihara K., Fujita Y. (2012) MarR-type transcriptional regulator ChlR activates expression of tetrapyrrole biosynthesis genes in response to low-oxygen conditions in cyanobacteria. J. Biol. Chem. 287(16), 13500–13507.
Seoane A.S., Levy S.B. (1995) Characterization of MarR, the repressor of the multiple antibiotic resistance (mar) operon in Escherichia coli. J. Bacteriol. 177(12), 3414–3419.
Quade N., Mendonca C., Herbst K., Heroven A.K., Ritter C., Heinz D.W., Dersch P. (2012) Structural basis for intrinsic thermosensing by the master virulence regulator RovA of Yersinia. J. Biol. Chem. 287(43), 35796–35803.
Martin R.G., Rosner J.L. (1995) Binding of purified multiple antibiotic-resistance repressor protein (MarR) to mar operator sequences. Proc. Natl. Acad. Sci. USA. 92(12), 5456–5460.
Alekshun M.N., Levy S.B., Mealy T.R., Seaton B.A., Head J.F. (2001) The crystal structure of MarR, a regulator of multiple antibiotic resistance, at 2.3 Å resolution. Nat. Struct. Biol., 8, 710–714.
Brugarolas P., Movahedzadeh F., Wang Y., Zhang N., Bartek I.L., Gao Y.N., Voskuil M.I., Franzblau S.G., He C. (2012) The Oxidation-sensing regulator (MosR) is a new redox- dependent transcription factor in Mycobacterium tuberculosis. J. Biol. Chem. 287(45), 37703–37712.
Camara A.S., Horjales E. (2018) Computer simulations reveal changes in the conformational space of the transcriptional regulator MosR upon the formation of a disulphide bond and in the collective motions that regulate its DNA-binding affinity. PLoS One. 13(2), 1–23.
Healy C., Golby P., Machugh D.E., Gordon S.V. (2016) The MarR family transcription factor Rv1404 coordinates adaptation of Mycobacterium tuberculosis to acid stress via controlled expression of Rv1405c, a virulence-associated methyltransferase. Tuberculosis. 97, 154–162.
Winglee K., Lun S., Pieroni M., Kozikowski A., Bishai W. (2015) Mutation of Rv2887, a marR-like gene, confers Mycobacterium tuberculosis resistance to an imidazopyridine-based agent. Antimicrob. Agents Chemother. 59(11), 6873–6881.
Warrier T., Kapilashrami K., Argyrou A., Ioerger T.R., Little D., Murphy K.C. (2016) N-methylation of a bactericidal compound as a resistance mechanism in Mycobacterium tuberculosis. Proc. Natl. Acad. Sci. USA. 113(31), E4523–E4530.
Radhakrishnan A., Kumar N., Wright C.C., Chou T.H, Tringides M.L., Bolla J.R., Lei H.T, Rajashankar K.R, Su C.C, Purdy G.E., Yu E.W. (2014) Crystal structure of the transcriptional regulator Rv0678 of Mycobacterium tuberculosis. J. Biol. Chem. 289(23), 16526–16540.
Yeoman C.J., Han Y., Dodd D., Schroeder C.M., Mackie R.I., Cann I.K.O. (2010) Thermostable enzymes as biocatalysts in the biofuel industry. Adv. Appl. Microbiol. 70, 1–55.
Schyns P. (1997) The bacterial degradation of xylan. In: Xylan Degradation by the Anaerobic Bacterium Bacteroides xylanolyticus. 1–44.
Han Y., Agarwal V., Dodds D., Kims J., Bae B., Mackie R.I., Nair S.K., Cann I.K.O. (2012) Biochemical and structural insights into xylan utilization by the thermophilic bacterium Caldanaerobius polysaccharolyticus. J. Biol. Chem. 287(42), 34946–34960.
Johnsen U., Dambeck M., Zaiss H., Fuhrer T., Soppa J., Sauer U., Schönheit P. (2009) D-xylose degradation pathway in the halophilic archaeon Haloferax volcanii. J. Biol. Chem.284, 27290–27303.
Hasper A.A., Visser J., De Graaff L.H. (2000) The Aspergillus niger transcriptional activator XlnR, which is involved in the degradation of the polysaccharides xylan and cellulose, also regulates D-xylose reductase gene expression. Mol. Microbiol. 36(1), 193–200.
Lovell S.C., Davis I.W., Arendall III W.B., de Bak-ker P.I.W., Word J.M., Prisant M.G., Richardson J.S., Richardson D.C. (2003) Structure validation by C alpha geometry: Phi, Psi and C beta deviation. Proteins Struct. Funct. Genet. 50(3), 437–450.
Solovyev V.V., Salamov A. (2011) Automatic annotation of microbial genomes and metagenomic sequences. In: Metagenomics and its Applications in Agriculture, Biomedicine and Environmental Studies. 61–78.
Specific binding with Hill’s slope performed using GraphPad Prism version 7.00 for Windows, GraphPad Software, San Diego, California USA, www.graphpad.com
Schneider C.A., Rasband W.S., Eliceiri K.W., Instrumentation C. (2012) NIH Image to ImageJ : 25 years of Image Analysis. Nat. Methods. 9(7), 671–675.
Crooks G.E., Hon G., Chandonia J.M., Brenner S.E. (2004) WebLogo: a sequence logo generator. Genome Res. 14(6), 1188–1190.
Peterson E.J.R., Ma S., Sherman D.R., Baliga N.S. (2016) Network analysis identifies Rv0324 and Rv0880 as regulators of bedaquiline tolerance in Mycobacterium tuberculosis. Nat. Microbiol. 1(8), 1–7.
Häkkinen M., Sivasiddarthan D., Aro N., Saloheimo M., Pakula T.M. (2015) The effects of extracellular pH and of the transcriptional regulator PACI on the transcriptome of Trichoderma reesei. Microb. Cell Fact. 14(63), 1–15.
Topakas E., Panagiotou G., Christakopoulos P. (2013) Xylanases: characteristics, sources, production, and applications. In: Bioprocessing Technologies in Biorefinery for Sustainable Production of Fuels, Chemicals, and Polymers. 147–165.
Marumo K., Nakamura H., Tazawa S., Kazumi Y., Kawano R., Shirata C., Taguchi K. (2010) Isolation of novel mycobacteria contaminating an aquarium fish tank in a Japanese university hospital. J. Appl. Microbiol. 109(2), 558–566.
Pourahmad F., Pate M., Borroni E., Cabibbe A.M., Capitolo E., Cittaro D., Frizzera E., Mariottini A., Marumo K., Cirillo D.M., Tortoli E. (2015) Mycobacterium angelicum sp. nov., a non-chromogenic, slow-growing species isolated from fish and related to Mycobacterium szulgai. Int. J. Syst. Evol. Microbiol. 65(12), 4724–4729.
Aronson J.D. (1926) Spontaneous tuberculosis in salt water fish. J. Infect. Dis. 39(4), 315–320.
Tsukamura M., Yan I., Imaeda T. (1986) Mycobacterium moriokaense sp. nov., a rapidly growing, nonphotochromogenic mycobacterium. Internl. J. Syst. Bacteriol. 36(2), 333–338.
Kopecky J., Kyselkova M., Omelka M., Cermak L., Novotna J., Grundmann G., Moënne-loccoz Y., Sagova-Mareckova M. (2011) Soil biology and biochemistry environmental mycobacteria closely related to the pathogenic species evidenced in an acidic forest wetland. Soil Biol. Biochem. 43(3), 697–700.
Suriyachadkun C., Ngaemthao W., Chunhametha S., Tamura T., Sanglier J.J. (2013) Kutzneria buriramensis sp. nov., isolated from soil, and emended description of the genus Kutzneria. Int. J. Syst. Evol. Microbiol. 63(1), 47–52.
Chen J., Su J.J., Wei Y.Z., Li Q.P., Yu L.Y., Liu H.Y., Zhang Y.Q., Zhang Y.Q. (2010) Amycolatopsis xylanica sp. nov., isolated from soil. Int. J. Syst. Evol. Microbiol. 60(9), 2124–2128.
Kaur N., Kumar S., Mayilraj S. (2014) Genomics data genome sequencing and annotation of Amycolatopsis vancoresmycina. Genomics Data. 2, 16–17.
Carlsohn M.R., Groth I., Tan G.Y.A., Schütze B., Saluz H.P., Munder T., Yang J., Wink J., Goodfellow M. (2007) Amycolatopsis saalfeldensis sp. nov., a novel actinomycete isolated from a medieval alum slate mine. Int. J. Syst. Evol. Microbiol. 57(7), 1640–1646.
Titgemeyer F., Amon J., Parche S., Mahfoud M., Bail J., Schlicht M., Rehm N., Hillmann D., Stephan J., Walter B., Burkovski A., Niederweis M. (2007) A genomic view of sugar transport in Mycobacterium smegmatis and Mycobacterium tuberculosis. J. Bacteriol. 189(16), 5903–5915.
Hanks J.H. (1961) Demonstration of capsules on M. leprae during carbol-fuchsin staining mechanism of the Ziehl-Neelsen stain. Int. J. Lepr. Other Mycobact. Dis. 29(2), 179–182.
Ortalo-Magné A., Dupont M.A., Lemassu A., Andersen A.B., Gounon P., Daffé M. (1995) Molecular composition of the outermost capsular material of the tubercle bacillus. Microbiology. 141(7), 1609–1620.
Sassetti C.M., Rubin E.J. (2003) Genetic requirements for mycobacterial survival during infection. Proc. Natl. Acad. Sci. USA. 100(22), 12989–12994.
Hanks J.H. (1961) The origin of the capsules on Mycobacterium leprae and other tissue-grown Mycobacteria. Int. J. Lepr. Other Mycobact. Dis. 29(2), 172–174.
Chatterjee D. (1997) The mycobacterial cell wall: structure, biosynthesis and sites of drug action. Curr. Opin. Chem. Biol. 1(4), 579–588.
Дополнительные материалы
- скачать EMS.docx
- Table S1. sequences of synthetic oligonucleotides used in the current study.
Figure S1. MxyR Model validation by Ramachandran’s plot. It shows that majority of the residues (91.5%) are in the favored region of the plot.
Figure S2. Binding of xylooligosaccharides towards MxyR.
Инструменты
Молекулярная биология


