Молекулярная биология, 2022, T. 56, № 3, стр. 451-464
Динамика структуры мРНК эукариот в ходе трансляции
Н. С. Бизяев a, Т. В. Егорова a, b, Е. З. Алкалаева a, b, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Центр высокоточного редактирования и генетических технологий для биомедицины
119991 Москва, Россия
* E-mail: alkalaeva@eimb.ru
Поступила в редакцию 02.11.2021
После доработки 07.12.2021
Принята к публикации 07.12.2021
- EDN: OPVYIR
- DOI: 10.31857/S002689842203003X
Аннотация
На настоящий момент нет единого представления о том, какую пространственную структуру образует мРНК в ходе трансляции. Известно, что множество белков, связанных с 5′-концом мРНК, взаимодействуют с белками, связанными с 3′-концом. Более того, такие белки часто влияют на активность друг друга. В пределах одной молекулы мРНК это возможно лишь тогда, когда мРНК образует кольцевую структуру, в которой ее концы сближены. В 90-х годах XX века изучение таких белков привело к появлению гипотезы о существовании структуры трансляционно активной мРНК, представляющей собой замкнутую петлю (closed-loop), в которой концы молекулы зафиксированы рядом друг с другом с помощью белковых и/или РНК-взаимодействий. Однако затем стали появляться свидетельства того, что сближение концов мРНК и формирование closed-loop-структуры не обязательны для трансляции некоторых мРНК. Более того, в некоторых работах утверждалось, что транслируемая мРНК, напротив, должна быть развернута в линейную структуру. Таким образом, пространственная структура транслируемой мРНК не обязательно должна быть универсальной для всех мРНК и может динамически меняться, что влияет на ее функциональную активность. В рамках данного обзора мы обобщили разнообразие экспериментальных данных и гипотез о связи пространственной структуры мРНК с ее трансляционной активностью.
ВВЕДЕНИЕ
Пространственная организация биологических макромолекул важна для их функционирования. Эукариотические мРНК в основном моноцистронны, т.е. кодируют один белок, и имеют в своем составе следующие структурные элементы (рис. 1а): 5′-конец, защищенный кеп-структурой; 5′-нетранслируемую область (5′-НТО); последовательность, кодирующую белок, и 3′-НТО, которая оканчивается поли(А)-хвостом. Необходимо отметить однако, что не все мРНК полиаденилированы, например, гистоновые мРНК часто не содержат поли(А) [1, 2].
Рис. 1.
Представления о структуре мРНК эукариот. а – Функциональные элементы мРНК эукариот. С 5′-конца мРНК защищена кеп-структурой, с 3′-конца – поли(А)-хвостом. Кодирующая последовательность фланкирована нетранслируемыми областями (НТО), UAA – пример стоп- кодона, которым заканчивается кодирующая последовательность. б – Представления о различных пространственных структурах трансляционно активных мРНК. I ‒ Трансляционно активна мРНК со сближенными 5′- и 3′-концами (структура closed-loop). II ‒ Трансляционно активна линейная мРНК. III ‒ Структура мРНК динамична. Создано с помощью BioRender.com.
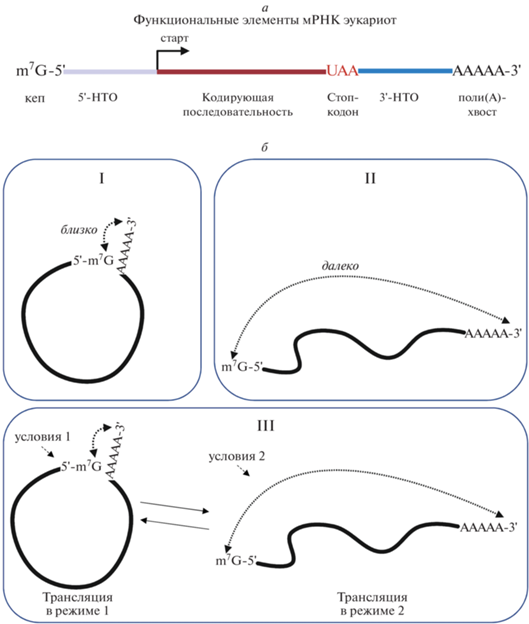
В ходе взаимодействия с рибосомой и трансляции молекула мРНК, а также связанные с ней белковые комплексы претерпевают значительные пространственные перестройки. Поэтому для понимания особенностей трансляции мРНК мало лишь изучить ее структуру in vitro, необходимо получить информацию о функционально активной конформации этой молекулы in vivo. В данном обзоре мы проанализируем разнообразие взглядов на пространственную организацию трансляционно активной мРНК. На сегодняшний день можно выделить три основных гипотезы (рис. 1б), каждую из них мы рассмотрим в соответствующем разделе. Согласно первой гипотезе трансляционно активна та мРНК, в структуре которой 5′- и 3′-концы молекулы сближены. Согласно второй, напротив, трансляционно активна развернутая мРНК, в которой 5′- и 3′-концы удалены друг о друга. Третья гипотеза предполагает, что структура мРНК динамична, поэтому трансляционная активность мРНК имеет сложную зависимость от ее пространственной организации и иных факторов.
I. ТРАНСЛЯЦИОННО АКТИВНА мРНК СО СБЛИЖЕННЫМИ 5′- И 3′-КОНЦАМИ (CLOSED-LOOP-СТРУКТУРА)
В середине 60-х годов XX века с развитием методов определения пространственной структуры больших молекул стало возможным визуализировать полисомы – несколько рибосом, одновременно транслирующих белок с одной молекулы мРНК. Обнаружено, что линия из визуализированных рибосом в полисоме может загибаться, в результате чего сближаются ее концы, что может свидетельствовать об аналогичном сближении пространственно удаленных участков самой мРНК [3]. Такие структуры также можно интерпретировать как следствие сближения 5′-конца мРНК и района стоп-кодона, так как в 3′-нетранслируемой области рибосомы в норме отсутствуют [4]. При этом установить точную конформацию мРНК в экспериментах с полисомами не представляется возможным, так как мРНК такими методами не визуализируется. Уже в 90-х годах с помощью атомно-силовой микроскопии было показано, что в растворе в присутствии эукариотического кеп-связывающего фактора инициации 4Е (eIF4E), поли(А)-связывающего белка (PABP) и eIF4G11, который связывает оба этих белка, мРНК фиксируется в циркуляризованном состоянии, тогда как в отсутствие какого-либо из указанных белков этого не происходит [5]. Однако в этой работе для увеличения персистентной длины мРНК использовали длинную антисмысловую ДНК, комплементарную 82% ее полной последовательности, что, как указывают другие авторы [6], препятствует спонтанной циркуляризации молекулы и, возможно, значительно усиливает необходимость PABP, а также комплекса eIF4F, состоящего из факторов инициации eIF4E, eIF4G и eIF4A, для стабилизации замкнутой структуры. В другой работе с помощью формальдегида проводили быструю сшивку компонентов клетки, находящихся рядом друг с другом. Обнаружено, что eIF4F, связанный на 5′-кепе, часто сшивается с 3′-последовательностями мРНК, а PABP – с 5′-концевыми последовательностями (но в меньшей степени). Это указывает на физическое сближение eIF4F с 3′-концом мРНК, а PABP – с 5′-концом [7]. Параллельно стали появляться биохимические свидетельства взаимодействия PABP и eIF4F, которое приводит к костимуляции активации трансляции (синергии) в модельных in vitro системах [5, 7–15]. В присутствии в системе данных белков проявляется также синергия в активации трансляции такими структурными элементами мРНК, как поли(А)-хвост и кеп [12, 14–18]. В отсутствие поли(А) последовательности eIF4G и PABP взаимодействуют слабо [19], что указывает на необходимость связывания PABP с концом мРНК для активации трансляции. Все эти свидетельства привели к формированию гипотезы, согласно которой трансляционно активна мРНК, имеющая структуру со сближенными 5′- и 3′-концами. Эта структура фиксируется взаимодействующими друг с другом белками, которые связаны с разными концами мРНК, или непосредственным взаимодействием участков РНК. Такая структура получила название замкнутой петли (closed-loop). Классическим примером такой структуры считается closed-loop-структура мРНК, зафиксированная взаимодействиями белков eIF4E, eIF4G, eIF4B, eIF3 и PABP (табл. 1) (рис. 2а).
Таблица 1.
Разнообразие closed-loop-структур мРНК
| Организм | 5′-Концевые элементы | Связующие элементы | 3′-Концевые элементы | Ссылка |
|---|---|---|---|---|
| Эукариоты | eIF4E (на кепе) | eIF4G; eIF4G-eIF4A-eIF4B; eIF4G-eIF4A/ eIF3-PAIP1 | PABP (на поли(А)-хвосте) | [1, 5, 8–15, 20, 21, 27] |
| Вирус гриппа | eIF4E (на кепе); NS1 (на сайте в 5′-НТО) | eIF4G; NS1; NS1-eIF4G | PABP (на поли(А)-хвосте) | [37, 42, 43] |
| Ротавирусы | eIF4E (на кепе) | eIF4G | NSP3 (на GACC в 3′-НТО) | [19, 22, 44–47] |
| Tombusviridae и Luteoviridae | Шпилька в мРНК | – | 3'-CITE структура мРНК | [1, 51–54] |
| Эукариоты | eIF4E (на кепе) | (4E-HP)-Bicoid | BBR-элемент (в 3′-НТО) | [55, 56] |
| Maskin-CREB | CRE-элемент (в 3′-НТО) | [58, 61] | ||
| eIF4G-eIF3-SLIP | SLBP (на SL петле в 3′-НТО) | [1, 65–68] | ||
| eIF4G-eIF3-METTL3 | Gm6AGG (в 3′-НТО) | [70] | ||
| Человек | Ago2 (на кепе) | – | микроРНК, комплементарная участку в 3′-НТО | [63] |
| Млекопитающие | eIF4E (на кепе) | eIF4G-GAIT комплекс | GAIT-связывающий участок в 3′-НТО | [64] |
Рис. 2.
Разнообразие циркуляризованных структур мРНК. а – Модели closed-loop-структур мРНК. Серым обозначена 5′-НТО, красным – основная кодирующая последовательность, синим – 3′-НТО. В кольце кеп-связывающий eIF4E взаимодействует с eIF4G, который также взаимодействует с PABP. За счет этого в мРНК пространственно сближаются 5′- и 3′-концы мРНК. В восьмерке PABP не только взаимодействует с eIF4G-eIF4E, но также связывает eRF3, находящийся в комплексе с eRF1. Во время терминации комплекс eRF1-eRF3 связывает рибосому на стоп-кодоне. За счет таких взаимодействий стоп-кодон (UAA), поли(А)-хвост и кеп взаимно сближаются. Для этого должна формироваться первая петля (из 5′-НТО и кодирующей области) и вторая петля (из 3′-НТО). б – Возможные типы циркуляризации мРНК. Создано с помощью BioRender.com.
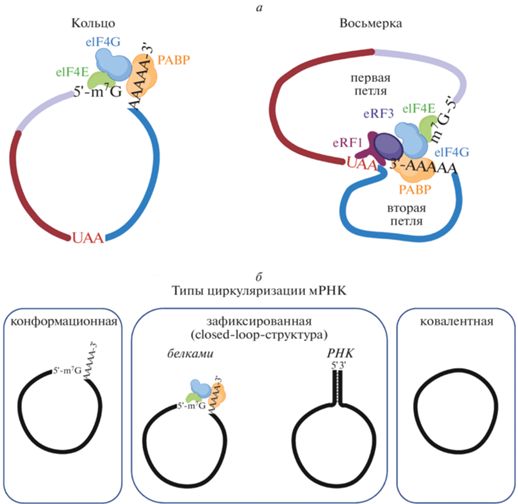
Взаимодействие в растворе показано как для пар белков инициаторного комплекса eIF4E, eIF4G, eIF4B и eIF3, так и для белков eIF4G и PABP млекопитающих, земноводных, насекомых, дрожжей и растений [9, 10, 13, 19–27]. Так как эти трансляционные факторы обнаружены у подавляющего большинства эукариот [28], можно предположить, что модель closed-loop является универсальной. Впоследствии обнаружили множество других примеров взаимного влияния связанных с мРНК факторов трансляции различных эукариотических организмов. Так, например, белок PAIP1 взаимодействует как с PABP, так и с факторами инициации трансляции eIF3 и eIF4A, связывающими eIF4G. Показано, что взаимодействие PAIP1 и eIF3 активирует инициацию трансляции. Считается, что через взаимодействие с eIF3 PAIP1 дополнительно стабилизирует структуру closed-loop, образованную eIF4F-PABP [1, 20, 21].
Обнаружено взаимодействие между PABP и фактором терминации трансляции 3 (eRF3) у дрожжей и человека, что указывает на возможное пространственное сближение поли(А)-хвоста и района стоп-кодона [29–33]. Также показана консервативность взаимодействия гомологов eRF3 и PABP у эукариот [29]. Кроме того, eRF3 может формировать комплекс с PABP, еще одним белком комплекса closed-loop, PAIP1 [34]. Обнаружено, что трансляция и, в частности, стадия терминации активируются в результате взаимодействия eRF3 с PABP [31–33]. При этом ингибирование взаимодействия eRF3 и PABP в системе из ретикулоцитов кролика не влияет на формирование 80S инициаторных комплексов, но понижает общий уровень трансляции [32]. Имеются также экспериментальные свидетельства того, что связанный с поли(А) PABP может оказывать положительное воздействие на трансляцию и в отсутствие связи c eIF4F [31, 35–37], что также говорит в пользу функциональной значимости кеп-независимых замкнутых структур мРНК (например, при образовании комплекса поли(А)–PABP–eRF3–стоп-кодон). Более того, внутриклеточная концентрация PABP в 3–10 раз превышает концентрацию eIF4G в клетках млекопитающих и дрожжей, что также указывает на возможность существования таких структур [38, 39]. Все это позволило предложить более сложную пространственную модель мРНК, имеющую структуру в виде восьмерки, в которой одновременно сближены 5′- и 3′-концы мРНК, а также район стоп-кодона (рис. 2а) [32]. В такой структуре можно выделить первую петлю, начинающуюся от 5′-конца и заканчивающуюся стоп-кодоном, и вторую петлю – от стоп-кодона до 3′-конца. Модель формирования второй петли объясняет как связанный с поли(А)-хвостом PABP стимулирует терминацию трансляции на стоп-кодоне, который в последовательности мРНК обычно значительно удален от поли(А)-хвоста. Эта модель подтверждается еще и тем, что в клетках дрожжей PABP-зависимая супрессия сквозного чтения стоп-кодонов происходит даже в отсутствие eRF3 [33], а значит, независимо от терминации трансляции, но с сохранением возможности пространственного сближения со стоп-кодоном. Стоит отметить, что в активации терминации трансляции может участвовать и PAIP1, который входит в состав первой петли мРНК и в присутствии eRF1 и eRF3 стабилизирует постерминационные комплексы рибосом, если в смеси отсутствует PABP [34]. Помимо этого, пространственное сближение PABP с природным стоп-кодоном также подавляет на нем процесс нонсенс-опосредованного распада мРНК (NMD), конкурирующего с терминацией [40, 41].
Гипотеза формирования closed-loop-структур также прекрасно объясняет взаимное влияние на трансляцию других белков и РНК-последовательностей, находящихся в противоположных участках мРНК (табл. 1). К примеру, белок NS1 вируса гриппа связывается со специфичным сайтом в 5'-НТО полиаденилированной вирусной мРНК и взаимодействует одновременно со связанными на ней же eIF4G и PABP, что специфически повышает эффективность трансляции вирусных мРНК [37, 42, 43] (табл. 1). Однако многие вирусы используют стратегию разрушения closed-loop-структуры, образованной eIF4G и PABP, для подавления трансляции клеточных мРНК. Так, в мРНК ротавирусов, содержащей 5′-кеп, в отсутствие поли(А)-хвоста closed-loop-структура может формироваться посредством взаимодействия комплекса eIF4E-eIF4G с вирусным белком NSP3, связывающимся с последовательностью GACC в 3′-НТО. NSP3 конкурирует с PABP за связывание с eIF4G, что также ведет к переходу PABP из цитоплазмы в ядро. Под действием белка NSP3 происходит разрушение классических closed-loop-структур, вызванных взаимодействием eIF4F-PABP. Все это ведет к супрессии трансляции мРНК хозяина и, напротив, к активации трансляции вирусных мРНК [19, 22, 44–47] (табл. 1). Более того, целевыми последовательностями многих протеаз других вирусов служат белки eIF4G и PABP, что во многих случаях нарушает их взаимодействие [37, 48–50]. Нацеленность вирусов на разрушение взаимодействия eIF4G и PABP свидетельствует о важности структуры closed-loop для инициации трансляции мРНК хозяина. Альтернативная closed-loop-структура образуется при трансляции одноцепочечных РНК(+) вирусов, относящихся к семействам Tombusviridae и Luteoviridae. В 3′-НТО мРНК этих вирусов содержатся последовательности, формирующие кеп-независимые трансляционные элементы (CITE). Эти последовательности связывают комплекс eIF4F и рекрутируют его в область, расположенную перед старт-кодоном, инициируя трансляцию мРНК. CITE сближается с 5′‑НТО за счет РНК-РНК-взаимодействий, формируя closed-loop-структуру, необходимую для активации трансляции. Экспериментально показано, что нарушение таких взаимодействий приводит к ингибированию инициации трансляции [1, 51–54] (табл. 1). Кроме того, обнаружено, что часть собственных клеточных систем репрессии трансляции функционирует аналогично вирусным, действуя через конкуренцию за взаимодействие с компонентами, образующими closed-loop-структуру eIF4E–eIF4G–PABP. Так, в регуляции развития дрозофилы и лягушек происходит связывание белков-регуляторов с 3′-НТО, что ведет к привлечению 4E-связывающих белков (4E-BP), которые взаимодействуют с eIF4E, вытесняя eIF4G. К примеру, белок Bicoid связывает BBR-элемент в 3′-НТО и 4E-связывающий белок 4E-HP, который, в свою очередь, связывает eIF4E, вытесняя его из комплекса с eIF4G (табл. 1). Однако в большинстве комплексов (4E-BP)–eIF4E аффинность последнего к кепу меньше, чем в комплексе с eIF4G, так что не ясно, возможно ли формирование closed-loop-структур через 4E-BP как альтернативы разрушения closed-loop-структуры PABP–eIF4G–eIF4E. Стоит отметить, что часть 4E-BP функционирует именно путем разрушения такой closed-loop-структуры [23, 55–60]. Интересно, что у земноводных образование closed-loop-структуры 3′-НТО–CREB–Maskin–eIF4E–кеп ведет к маскировке мРНК в процессе эмбриогенеза и сопряжено с деаденилированием, что исключает формирование трансляционно активной closed-loop-структуры с участием PABP. В процессе демаскировки, напротив, происходит разрушение closed-loop-структуры с участием CREB, а также аденилирование мРНК [58, 61]. У мышей обнаружена CREB-зависимая циркадная ритмичность процессов деаденилирования и аденилирования отдельных мРНК, что позволяет контролировать трансляцию [62] (табл. 1). В клетках человека ингибиторный комплекс может образовываться посредством связывания микроРНК с участком 3′-НТО, с которой может взаимодействовать белок аргонавт (Ago2), имеющий кеп-связывающий домен [63] (табл. 1). Интересно, что появление дополнительной петли в closed-loop-структуре может приводить к подавлению трансляции, формируя альтернативные структуры мРНК, организованные в виде восьмерки. Так, под воздействием интерферона-γ в макрофагах собирается комплекс GAIT, состоящий из тРНК-синтетазы, глицеральдегид-3-фосфат-дегидрогеназы, рибосомного белка L13a и некоторых других. Этот комплекс связывается как со специальным участком посадки в 3′-НТО, так и с eIF4G, находящимся в составе closed-loop-структуры с eIF4E и PABP. Образование такой структуры позволяет пространственно сблизить комплекс GAIT с 5′-НТО, что препятствует сборке на нем 43S преинициаторного комплекса [64] (табл. 1). Еще один случай образования альтернативной структуры closed-loop – трансляция гистоновых мРНК. Показано, что у значительного количества гистоновых мРНК многоклеточных животных отсутствует поли(А)-хвост, но в 3′-НТО находится SL-петля. Closed-loop-структура таких мРНК формируется за счет последовательного взаимодействия SL-петли мРНК и 5′-кепа через белки SLBP-SLIP-eIF3-eIF4G-eIF4E. Показано, что взаимодействия SL-петли и SLBP играют ключевую роль в созревании, деградации и трансляции гистоновых мРНК. Предполагается, что описанная closed-loop-структура, как и образованная eIF4G–PABP, активирует инициацию трансляции [1, 65–68] (табл. 1). Гомологичные SL-петле структуры гистоновых мРНК и гомологи SLBP обнаружены у многих протистов, что говорит об эволюционной древности данной системы [69]. Наконец, недавно показали, что метилтрансфераза METTL3 и фактор инициации eIF3 солокализованы в цитоплазме и в их присутствии полисомы формируют преимущественно компактные кольцеобразные структуры, что не наблюдается в отсутствие METTL3. При дальнейшем изучении обнаружили, что METTL3 одновременно связывается как c GGAC-сайтом в 3′-НТО, так и с eIF3, связанным с белками на кепе. При этом METTL3 метилирует аденозин в консенсусной последовательности GGAC. В присутствии eIF3 и METTL3 повышается эффективность трансляции мРНК, содержащих этот сайт, даже в отсутствие поли(А)-хвоста. Это не наблюдается при ингибировании взаимодействия METTL3 и eIF3. METTL3 – это N6-аденозин-метилтрансфераза, активность которой вовлечена в регуляцию трансляции многих онкогенов (в частности, рецептора EGFR в клетках легких) и генов, связанных с апоптозом. Сверхэкспрессия гена, кодирующего этот белок, стимулирует развитие рака легкого, тогда как сверхэкспрессия гена, кодирующего мутантный METTL3, не способный связывать eIF3, не вызывает такого эффекта. Все это свидетельствует о ключевой роли именно closed-loop-структуры, а не только паттерна метилирования [70] в онкогенной активности METTL3 (табл. 1).
В поддержку существования и функциональной значимости closed-loop-структур мРНК свидетельствует феномен самопроизвольной циркуляризации мРНК в отсутствие специализированных белков (рис. 2б). In silico расчеты предсказывают, что концы большинства мРНК в растворе конформационно сближены (<10 нм) [71–75], что подтверждается in vitro экспериментами в отсутствие белков, способных стабилизировать такую структуру [76, 77]. Дистанция между концами не зависит от длины мРНК и от ее последовательности [72, 73, 77]. Таким образом, циркуляризация мРНК – это свойство, присущее большинству мРНК, а не результат отбора определенной последовательности [72, 74, 76–78]. Конечно, на это можно возразить, что с началом трансляции рибосомы будут непрерывно плавить вторичную структуру мРНК и этот фактор спонтанного сближения концов перестанет существовать. Однако неясно, каким образом происходит инициация трансляции в closed-loop-структуре и имеется ли там стадия сканирования лидерной последовательности мРНК, в ходе которой будет происходить плавление вторичной структуры. Внедрение в мРНК неструктурированных последовательностей (к примеру, поли(CA)) приводит к расхождению концов мРНК [77]. Соответственно, присутствие комплекса из белков, связанных на разных концах РНК, по-видимому, не является необходимым условием циркуляризации РНК (рис. 2б) [6]. Напротив, способность РНК к циркуляризации может быть использована для организации на ее концах регуляторных белковых комплексов [78]. Добавление поли(А)-хвоста к РНК ведет к увеличению расстояния между ее концами: так, поли(А)-хвост из 30 н. увеличивает его на 5 нм. Таким образом, поли(А)-хвост, не участвуя во взаимодействии с 5′-концом, образует выпетливание из циркуляризованной РНК [77], которое затем может быть исправлено взаимодействиями белков в составе closed-loop-структуры. In vivo картирование внутримолекулярных взаимодействий РНК выявило активное спаривание регионов, удаленных друг от друга в первичной структуре [79–81], что может в целом стабилизировать циркуляризованную структуру мРНК. Таким образом, конформация мРНК в closed-loop-моделях близка к ее конформации в отсутствие каких-либо связанных с ней белков, а значит, сворачивание и поддержание closed-loop-структуры мРНК не должны быть энергозатратными для клетки процессами.
Все эти данные позволяют сделать вывод, что формирование closed-loop-структур мРНК играет значительную роль в активации как трансляции в целом, так и ее отдельных этапов. Эта гипотеза успешно предсказывает взаимное влияние на трансляцию белков, связывающихся в противоположных частях мРНК, что отражено в публикациях последних лет [31, 34, 70, 82].
Модель мРНК, имеющей структуру в виде восьмерки (рис. 2а), наиболее полно объясняет вовлечение рибосом в новые раунды трансляции после терминации, наблюдаемое в ряде работ [83, 84]. Согласно этой модели, районы стоп-кодона и 5′-кепа пространственно сближены, что повышает локальную концентрацию компонентов терминации, рециклинга и инициации, а также позволяет 5′-кеп-связанным белкам напрямую взаимодействовать с рибосомой, находящейся в районе стоп-кодона. Предполагается, что такая closed-loop-структура активирует инициацию трансляции путем рекрутирования субъединиц рибосом, формируя 48S инициаторный комплекс [1, 9, 85].
Однако при обсуждении вклада closed-loop-структуры в регуляцию трансляции следует учитывать, что образующие ее белки могут взаимодействовать и активировать друг друга не только в составе такой структуры, но и в свободном состоянии в растворе [1]. Например, если отсутствие белков eIF4G и PABP в клетках человека или дрожжей летально, то отсутствие взаимодействий между ними не летально [86–88]. Также возможно и взаимное влияние белков, связанных с разными молекулами мРНК. К примеру, мРНК, содержащая поли(А)-хвост, стимулирует как трансляцию в целом, так и эффективность терминации трансляции на другой молекуле мРНК без поли(А)-хвоста [31, 89]. Поэтому вопрос о реальном вкладе взаимодействия белков, находящихся на одной молекуле мРНК, в активность трансляции остается открытым.
С другой стороны, циркуляризованная мРНК может быть зафиксирована не только посредством белков (рис. 2б). Например, трансляция мРНК с комплементарными друг другу 5′-НТО и 3′-НТО длиной 30 н., но без 5′-кепа и поли(А)-хвоста, в лизате протекает не менее эффективно, чем трансляция мРНК с той же кодирующей последовательностью, имеющей 5′-кеп и поли(А), и способной образовывать closed-loop-структуру с участием eIF4G и PABP. При этом контрольная мРНК без таких структур транслируется в 3 раза хуже [90]. Стоит упомянуть однако, что у этой работы есть существенные недостатки, которые могут влиять на интерпретацию данных. Так, трансляцию проводили в лизате ретикулоцитов кролика (RRL), где, как известно, плохо воспроизводится как зависимость трансляции от m7G-кепа, так и синергия между кепом и поли(А)-хвостом. В другой работе показано, что кодирующая последовательность в составе ковалентно циркуляризованной РНК (рис. 2б) может транслироваться в культуре клеток человека и дрозофилы за счет внутреннего сайта посадки рибосомы (IRES). При добавлении поли(А) или поли(Т)-последовательностей в эту мРНК эффективность трансляции, в отличие от мРНК в closed-loop-структуре, понижается [91]. Интересно, что удалось добиться IRES-зависимой трансляции ковалентно циркуляризованных мРНК по механизму катящегося кольца (рибосома непрерывно транслирует циркуляризованную мРНК по кругу) [92]. В дальнейшем in vitro в том же лизате и in vivo в культуре клеток HeLa удалось провести трансляцию по механизму катящегося кольца с ковалентно циркуляризованной мРНК, не содержащей IRES, но содержащей неструктурированный участок. Более того, трансляция мРНК размером 129–380 н. происходит значительно лучше в ковалентно циркуляризованной структуре по сравнению с линейной молекулой [93]. Согласно последним исследованиям, клетки эукариот содержат значимое количество ковалентно циркуляризованых РНК, большая часть которых не транслируется, но участвует в различных клеточных процессах (к примеру, в привлечении микроРНК для подавления трансляции). Так что пространственное сближение (а в данном случае даже соединение) концов РНК играет роль не только в функционировании мРНК [94, 95].
Подводя итог, можно сказать, что модель closed-loop-структуры трансляционно активной мРНК имеет множество экспериментальных подтверждений, что делает ее (на сегодняшний момент) наиболее вероятной.
II. ТРАНСЛЯЦИОННО АКТИВНА ЛИНЕЙНАЯ мРНК
Несмотря на множество данных, подтверждающих существование и функциональность closed-loop-структуры, в последнее время стали появляться факты, не укладывающиеся в эту гипотезу. Так недавнее изучение динамики пространственной структуры РНК в клетках методами одномолекулярной флуоресцентной in situ гибридизации (smFISH) и микроскопии с помощью отжига коротких антисмысловых флуоресцентных нуклеотидов, комплементарных различным регионам мРНК, показало, что расстояние между концами мРНК повышается с увеличением плотности рибосом, и что активно транслируемые мРНК находятся в конформации, в которой их концы значительно удалены друг от друга [24, 96]. Обработка клеток как пуромицином, вызывающим диссоциацию рибосом с мРНК, так и гомохаррингтонином, фиксирующим инициирующие рибосомы, приводит к увеличению количества циркуляризованной мРНК. Ингибирование взаимодействия eIF4G и PABP в активно транслирующих клетках никак не влияет на количество циркуляризованной мРНК в сравнении с клетками, где такое взаимодействие не подавлено. Авторы этих исследований заключили, что наблюдаемый эффект прямо противоположен ожидаемому согласно гипотезе о closed-loop-структуре и, таким образом, опровергает ее. Они считают, что данные in vivo более приближены к реальности, чем полученные in vitro результаты, на которых базируется первая гипотеза. На основании этого предложена альтернативная гипотеза, согласно которой в клетках функционально активна линейная мРНК [24, 96]. Однако стоит отметить, что структура РНК в этих исследованиях была визуализирована отжигом множества антисмысловых олигонуклеотидов, что могло снизить эффективность образования циркуляризованной мРНК и, соответственно, перевести ее в расплетенную форму, поэтому говорить о полном соответствии модели условиям в клетке не приходится.
В других экспериментах, проведенных на дрожжах, где изучали эффективность трансляции большого количества различных клеточных мРНК (а не единичных модельных мРНК, как делали ранее), обнаружили, что трансляция мРНК с длинными кодирующими последовательностями, в отличие от коротких, относительно слабо или вообще не зависела от eIF4F и PABP, т.е. не была связана с формированием closed-loop-структуры [87, 97]. Эти данные заставляют задуматься о реальном вкладе образования closed-loop-структур мРНК в активацию трансляции.
Тем не менее, представления о трансляционно активной линейной мРНК не объясняют, почему множество элементов, находящихся в одном регионе мРНК, влияет на эффективность элементов, находящихся в других регионах. Эта гипотеза выглядит слишком строгой, а ее выводы пока плохо предсказывают ожидаемую эффективность трансляции мРНК. В настоящий момент имеется альтернативная гипотеза о структуре функциональной мРНК в клетке, подразумевающая синтез двух описанных выше гипотез.
III. СТРУКТУРА мРНК ДИНАМИЧНА
В последнее время появляются свидетельства того, что структура трансляционно активной мРНК динамична, а значит, простой связи между структурой мРНК и ее функцией, как предполагают предыдущие гипотезы, может и не быть [1, 6].
Так, показано, что в клетке различные мРНК имеют разную пространственную структуру. Методами электронной и криоэлектронной микроскопии полисом (как свободных, так и связанных с эндоплазматическим ретикулумом) обнаружены различные формы их пространственной организации, лишь часть из которых образует кольцеобразную структуру [3, 4, 35, 98–102]. Более того, пространственная организация транслирующих полисом динамична: первые связавшиеся с мРНК рибосомы в основном формируют кольцеобразную или линейную полисому, связывание большего количества рибосом (до плотности 1 рибосома на 100 н.) приводит к преимущественной линеаризации полисом и дальнейшему формированию более сложных компактных трехмерных петлеобразных структур. Однако кольцеобразные и линейные формы встречаются на любых этапах [100]. Показано также, что в бесклеточной системе трансляции доля кольцеобразных полисом почти не зависит от наличия 5′-кепа, поли(А)-хвоста или их обоих и составляет около 40−50% [102].
В серии исследований на дрожжах обнаружено, что трансляция разных мРНК по-разному зависит от функционирования белков, участвующих в формировании closed-loop-структуры. Так мРНК, содержащие короткие кодирующие последовательности (около 500 н.), в отличие от мРНК с длинными кодирующими последовательностями, преимущественно связаны с eIF4F и PABP, что значительно повышает плотность рибосом на них и стимулирует их активную трансляцию. Значительная часть генов “домашнего хозяйства” (гистоны, рибосомные и митохондриальные белки) транслируются с таких мРНК [36, 103–105]. Обнаружено, что отсутствие дрожжевого белка ASC1 40S субъединицы рибосом (RACK1 у человека) приводит к снижению эффективности трансляции мРНК с короткими кодирующими последовательностями. Предполагается, что этот белок участвует в модуляции трансляции таких мРНК, влияя на формирование closed-loop-структур, что подтверждается его связыванием с eIF4G и 40S субъединицей рибосом [104, 105]. Это согласуется с результатами, согласно которым эффективная трансляция дрожжевых мРНК с короткой кодирующей последовательностью зависит от eIF4G и, как следствие, от формирования closed-loop-структуры, но слабо зависит от хеликазы eIF4A и ее партнера eIF4B, так как такие мРНК имеют неструктурированную 5′-НТО. При этом eIF4A и eIF4B важны для трансляции мРНК, которые не зависят от eIF4G [87, 97]. Во всех исследованиях трансляция мРНК с длинными кодирующими последовательностями относительно слабо или вообще не зависела от eIF4F и PABP. Однако показано, что длинные мРНК также могут образовывать closed-loop-структуру, но, в целом, не удалось обнаружить универсальные корреляции между closed-loop-структурой на мРНК и эффективностью трансляции, длиной кодирующей последовательности и поли(А)-хвоста, а также общей длиной РНК [7]. Здесь необходимо отметить, что эти работы проведены на клетках дрожжей, трансляция в которых может существенно отличаться от трансляции в клетках высших эукариот.
Вариации структуры самой молекулы мРНК, регуляция активности факторов, фиксирующих ее пространственную структуру, регуляция активности рибосомных комплексов, пространственная сегрегация мРНК также открывают широкие и гибкие возможности для регуляции трансляции [1, 6]. Получены данные, показывающие, что структура мРНК динамично изменяется. Например, измерение расстояния между концами мРНК методом smFRET обнаружило флуктуации между различными состояниями молекулы [77]. Также различными методами показано, что эффективность взаимодействия eIF4E c кепом зависит как от этапа инициации, так и от длины мРНК [106], а расстояние между кепом и 3′-НТО изменяется во времени [107]. В процессе инициации у дрожжей обнаруживаются две closed-loop-структуры: первая формируется при сборке 48S преинициаторного комплекса и нуждается в eIF4E, eIF4G, PABP и eIF3, а вторая – при сборке 80S инициаторного комплекса, для нее необходимы дополнительно eRF1 и eRF3, но не стоп-кодон [8]. Причем первого набора белков достаточно для формирования closed-loop-структуры в виде кольца, а второго – для формирования восьмерки.
На структуру молекулы мРНК может влиять динамика длины поли(А)-хвоста, которая может изменяться у одной мРНК с течением времени [108–110]. От длины поли(А)-хвоста зависит, в частности, сколько молекул PABP могут с ним связаться [111]. А от этого, в свою очередь, может зависеть, будет ли на мРНК формироваться стабильная closed-loop-структура.
Более того, длина поли(А)-хвоста не всегда критична для определения трансляционной активности. Так показано, что у земноводных и рыб эффективность трансляции сильно коррелирует с длиной поли(А)-хвоста лишь в раннем эмбриональном периоде [108]. Показано также, что распределение длин поли(А)-хвостов в мРНК незрелых ооцитов и в активированных яйцеклетках дрозофилы сильно отличается, и лишь в яйцеклетках эффективность трансляции коррелирует с длиной поли(А)-хвоста [109]. Это указывает на то, что вклад closed-loop-структуры в регуляцию трансляции мРНК может сложным образом зависеть и от клеточного контекста в целом. Так, нуклеазная обработка RRL приводит к исчезновению эффекта синергии кепа и поли(А)-хвоста, который не восстанавливается при последующем добавлении клеточных РНК [18].
Регуляция аффинности и концентрации белков, образующих или разрушающих разные closed-loop-структуры, может вести к динамической смене структур мРНК. В частности, многие факторы трансляции имеют сайты фосфорилирования, а некоторые взаимодействуют с дополнительными белками, конкурирующими за их связывание (например, 4E-BP) [1, 23, 55–60, 105, 112].
Суммируя эти данные, едва ли стоит рассматривать closed-loop или линейные структуры мРНК как статичные и единственно необходимые для эффективной трансляции любой мРНК. В публикациях последнего времени преобладает представление, что пространственная организация мРНК может различаться на разных этапах трансляции, в меняющихся условиях (в частности, на разных стадиях клеточного цикла и при стрессе), а также зависеть от состава мРНК. Однако какие-либо универсальные закономерности динамики транслируемых мРНК пока не установлены.
Согласно одним данным, максимальная синергия между 5′-кепом и поли(А)-хвостом и, следовательно, closed-loop-организация мРНК наблюдется при ненасыщающей концентрации факторов и мРНК в цитоплазме, когда эффективность трансляции далека от максимальной, а также при агрегации в условиях стресса. Связывание большого количества рибосом при активной трансляции приводит к разворачиванию мРНК из компактной структуры, что согласуется с исследованиями, рассмотренными в гипотезе II, наблюдавшими линейную мРНК при эффективной трансляции [12, 15, 24, 96] (рис. 3а).
Рис. 3.
Модели динамичной структуры мРНК. а – Модель по Khong и соавт. [96]: closed-loop структура связана с неактивно транслируемой или агрегируемой мРНК. б – Модель по Thompson и соавт. [105]: closed-loop-структура связана с активно транслируемой мРНК, преимущественно с короткой основной кодирующей последовательностью. Создано с помощью BioRender.com.
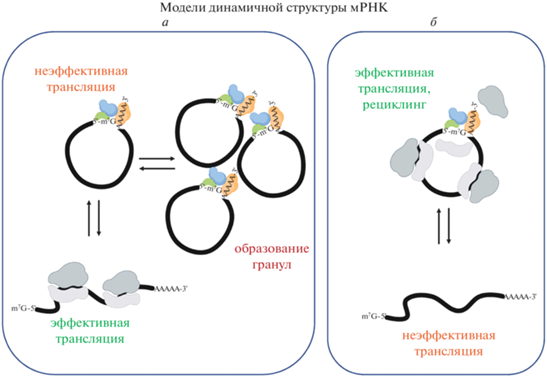
Согласно другим данным, формирование closed-loop-структуры, напротив, повышает эффективность трансляции и плотность рибосом на ней, причем преимущественно на мРНК с короткими кодирующими последовательностями [36, 87, 97, 103–105] (рис. 3б). Это согласуется с тем, что в составе кольцеобразных полисом скорость реинициации повышена, что подтверждает гипотезу closed-loop-зависимого рециклинга [100]. Более того, в in vitro трансляции показано, что эффективность трансляции первых связавшихся с кепированной мРНК рибосом не зависит от наличия поли(А)-хвоста, тогда как эффективность последующих раундов трансляции заметно повышается в присутствии поли(А)-хвоста. Такое усиление блокируется добавлением в реакционную смесь как поли(А)-олигонуклеотидов, так и аналога кепа m7GpppG. Первый конкурирует с мРНК за связывание PABP, тогда как второй – за eIF4E. При этом оба компонента значительно слабее влияют на кинетику первых раундов трансляции, не зависящей от поли(А)-хвоста. Соответственно, формирование closed-loop-структуры мРНК увеличивает эффективность трансляции только после первых раундов трансляции [17].
Несмотря на то что третья гипотеза наиболее широко и полно описывает имеющиеся разнообразные и противоречивые данные, единая непротиворечивая модель транзиентной closed-loop-структуры мРНК и ее регуляции в настоящий момент отсутствует. Эта гипотеза пока не имеет конкретных универсальных обобщений, не обладает предсказательной силой и лишь констатирует, что любая структура мРНК может играть какую-то особую роль.
ЗАКЛЮЧЕНИЕ
Мы рассмотрели гипотезы, предлагаемые в настоящий момент для объяснения связи между эффективной трансляцией и структурой мРНК. Первые две гипотезы предлагают строгие критерии определения функциональной структуры мРНК, однако каждая из них сталкивается с противоречащими им данными, которые сложно поддаются объяснению в рамках данных гипотез. Третья не определяет единственно правильную структуру мРНК, а напротив, описывает их разнообразие и динамичность. Какая бы из описанных гипотез не оказалась верной, очевидно, что элементы, находящиеся на противоположных концах мРНК, могут влиять на активность друг друга, а значит, каким-то образом взаимодействовать.
Работа выполнена при поддержке Российского научного фонда (№ 14-14-00487, анализ влияния closed-loop-структур на трансляцию) и Российского научного фонда (№ 19-74-10078, изучение значения динамичности структуры мРНК в трансляции).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Fakim H., Fabian M.R.F. (2019) Communication is key: 5′–3′ interactions that regulate mRNA translation and turnover. In: The Biology of mRNA: Function and Structure. Advances in Experimental Medicine and Biology. Eds Oeffinger M., Zenklusen D. Cham: Springer Nature Switzerland, 1203. 149–165.
Marzluff W.F. (1992) Histone 3′ ends: essential and regulatory functions. Gene Expr. 2, 93–97.
Mathias A.P., Williamson R., Huxley H.E., Page S. (1964) Occurrence and function of polysomes in rabbit reticulocytes. J. Mol. Biol. 9, 154–IN11.
Christensen A.K., Kahn L.E., Bourne C.M. (1987) Circular polysomes predominate on the rough endoplasmic reticulum of somatotropes and mammotropes in the rat anterior pituitary. Am. J. Anatomy. 178, 1–10.
Wells S.E., Hillner P.E., Vale R.D., Sachs A.B. (1998) Circularization of mRNA by eukaryotic translation initiation factors. Mol. Cell. 2, 135–140.
Vicens Q., Kieft J.S., Rissland O.S. (2018) Revisiting the closed-loop model and the nature of mRNA 5′–3′ communication. Mol. Cell. 72, 805–812.
Archer S.K., Shirokikh N.E., Hallwirth C.V., Beilharz T.H., Preiss T. (2015) Probing the closed-loop model of mRNA translation in living cells. RNA Biol. 12, 248–254.
Amrani N., Ghosh S., Mangus D.A., Jacobson A. (2008) Translation factors promote the formation of two states of the closed-loop mRNP. Nature. 453, 1276–1280.
Kahvejian A., Svitkin Y.V., Sukarieh R., M’Boutchou M.N., Sonenberg N. (2005) Mammalian poly(A)-binding protein is a eukaryotic translation initiation factor, which acts via multiple mechanisms. Genes Dev. 19, 104–113.
Le H., Tanguay R.L., Balasta M.L., Wei C.C., Browning K.S., Metz A.M., Goss D.J., Gallie D.R. (1997) Translation initiation factors eIF-iso4G and eIF-4B interact with the poly(A)-binding protein and increase its RNA binding activity. J. Biol. Chem. 272, 16247–16255.
Gross J.D., Moerke N.J., Von Der Haar T., Lugovskoy A.A., Sachs A.B., McCarthy J.E.G., Wagner G. (2003) Ribosome loading onto the mRNA cap is driven by conformational coupling between eIF4G and eIF4E. Cell. 115, 739–750.
Borman A.M., Yanne M., Kean K.M. (2000) Biochemical characterisation of cap-poly(A) synergy in rabbit reticulocyte lysates: the eIF4G-PABP interaction increases the functional affinity of eIF4E for the capped mRNA 5′-end. Nucl. Acids Res. 28, 4068–4075.
Von Der Haar T., Ball P.D., McCarthy J.E.G. (2000) Stabilization of eukaryotic initiation factor 4E binding to the mRNA 5′-cap by domains of eIF4G. J. Biol. Chem. 275, 30551–30555.
Machida K., Shigeta T., Yamamoto Y., Ito T., Svitkin Y., Sonenberg N., Imataka H. (2018) Dynamic interaction of poly(A)-binding protein with the ribosome. Sci. Rep. 8, 17435.
Michel Y.M., Poncet D., Piron M., Kean K.M., Borman A.M. (2000) Cap-poly(A) synergy in mammalian cell-free extracts. Investigation of the requirements for poly(A)-mediated stimulation of translation initiation. J. Biol. Chem. 275, 32268–32276.
Gallie D.R. (1991) The cap and poly(A) tail function synergistically to regulate mRNA translational efficiency. Genes Dev. 5, 2108–2116.
Alekhina O.M., Terenin I.M., Dmitriev S.E., Vassilenko K.S. (2020) Functional cyclization of eukaryotic mRNAs. Internat. J. Mol. Sci. 21, 1–18.
Rifo R.S., Ricci E.P., Decimo D., Moncorge O., Ohlmann T. (2007) Back to basics: the untreated rabbit reticulocyte lysate as a competitive system to recapitulate cap/poly(A) synergy and the selective advantage of IRES-driven translation. Nucl. Acids Res. 35, e121–e121.
Safaee N., Kozlov G., Noronha A.M., Xie J., Wilds C.J., Gehring K. (2012) Interdomain allostery promotes assembly of the poly(A) mRNA complex with PABP and eIF4G. Mol. Cell. 48, 375–386.
Craig A.W.B., Haghighat A., Yu A.T.K., Sonenberg N. (1998) Interaction of polyadenylate-binding protein with the eIF4G homologue PAIP enhances translation. Nature. 392, 520–523.
Martineau Y., Derry M.C., Wang X., Yanagiya A., Berlanga J.J., Shyu A.-B., Imataka H., Gehring K., Sonenberg N. (2008) Poly(A)-Binding protein-interacting protein 1 binds to eukaryotic translation initiation factor 3 to stimulate translation. Mol. Cell. Biol. 28, 6658–6667.
Groft C.M., Burley S.K. (2002) Recognition of eIF4G by rotavirus NSP3 reveals a basis for mRNA circularization. Mol. Cell. 9, 1273–1283.
Nelson M.R., Leidal A.M., Smibert C.A. (2004) Drosophila cup is an eIF4E-binding protein that functions in Smaug-mediated translational repression. EMBO J. 23, 150–159.
Adivarahan S., Livingston N., Nicholson B., Rahman S., Wu B., Rissland O.S., Zenklusen D. (2018) Spatial organization of single mRNPs at different stages of the gene expression pathway. Mol. Cell. 72, 727–738.e5.
Haghighat A., Sonenberg N. (1997) eIF4G dramatically enhances the binding of eIF4E to the mRNA 5′-cap structure. J. Biol. Chem. 272, 21677–21680.
Tarun S.Z., Sachs A.B. (1996) Association of the yeast poly(A) tail binding protein with translation initiation factor elF-4G. EMBO J. 15, 7168–7177.
Bushell M., Wood W., Carpenter G., Pain V.M., Morley S.J., Clemens M.J. (2001) Disruption of the interaction of mammalian protein synthesis eukaryotic initiation factor 4B with the poly(A)-binding protein by caspase- and viral protease-mediated cleavages. J. Biol. Chem. 276, 23922–23928.
Bannerman B.P., Kramer S., Dorrell R.G., Carrington M. (2018) Multispecies reconstructions uncover widespread conservation, and lineage-specific elaborations in eukaryotic mRNA metabolism. PLoS One. 13, 1–23.
Cosson B., Berkova N., Couturier A., Chabelskaya S., Philippe M., Zhouravleva G. (2002) Poly(A)-binding protein and eRF3 are associated in vivo in human and Xenopus cells. Biol. Cell. 94, 205–216.
Hoshino S., Imai M., Kobayashi T., Uchida N., Katada T. (1999) The eukaryotic polypeptide chain releasing factor (eRF3/GSPT) carrying the translation termination signal to the 3′-poly(A) tail of mRNA. J. Biol. Chem. 274, 16677–16680.
Ivanov A., Mikhailova T., Eliseev B., Yeramala L., Sokolova E., Susorov D., Shuvalov A., Schaffitzel C., Alkalaeva E. (2016) PABP enhances release factor recruitment and stop codon recognition during translation termination. Nucl. Acids Res. 44, 7766–7776.
Uchida N., Hoshino S. ichi, Imataka H., Sonenberg N., Katada T. (2002) A novel role of the mammalian GSPT/eRF3 associating with poly(A)-binding protein in cap/poly(A)-dependent translation. J. Biol. Chem. 277, 50286–50292.
Cosson B., Couturier A., Chabelskaya S., Kiktev D., Inge-Vechtomov S., Philippe M., Zhouravleva G. (2002) Poly(A)-binding protein acts in translation termination via eukaryotic release factor 3 interaction and does not influence [PSI(+)] propagation. Mol. Cell. Biol. 22, 3301–3315.
Ivanov A., Shuvalova E., Egorova T., Shuvalov A., Sokolova E., Bizyaev N., Shatsky I., Terenin I., Alkalaeva E. (2019) Polyadenylate-binding protein-interacting proteins PAIP1 and PAIP2 affect translation termination. J. Biol. Chem. 294, 8630–8639.
Bonderoff J.M., Lloyd R.E. (2010) Time-dependent increase in ribosome processivity. Nucl. Acids Res. 38, 7054–7067.
Costello J., Castelli L.M., Rowe W., Kershaw C.J., Talavera D., Mohammad-Qureshi S.S., Sims P.F.G., Grant C.M., Pavitt G.D., Hubbard S.J., Ashe M.P. (2015) Global mRNA selection mechanisms for translation initiation. Genome Biol. 16, 1–21.
Smith R.W.P., Gray N.K. (2010) Poly(A)-binding protein (PABP): a common viral target. Biochem. J. 426, 1–12.
Kulak N.A., Pichler G., Paron I., Nagaraj N., Mann M. (2014) Minimal, encapsulated proteomic-sample processing applied to copy-number estimation in eukaryotic cells. Nat. Methods. 11, 319–324.
Merrick W.C., Pavitt G.D. (2018) Protein synthesis initiation in eukaryotic cells. Cold Spring Harb. Perspect. Biol. 10, a033092.
Singh G., Rebbapragada I., Lykke-Andersen J. (2008) A competition between stimulators and antagonists of Upf complex recruitment governs human nonsense-mediated mRNA decay. PLoS Biol. 6, 860–871.
Ivanov P.V., Gehring N.H., Kunz J.B., Hentze M.W., Kulozik A.E. (2008) Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. EMBO J. 27, 736–747.
Aragón T., de la Luna S., Novoa I., Carrasco L., Ortín J., Nieto A. (2000) Eukaryotic translation initiation factor 4GI is a cellular target for NS1 protein, a translational activator of influenza virus. Mol. Cell. Biol. 20, 6259–6268.
Burgui I., Aragón T., Ortín J., Nieto A. (2003) PABP1 and eIF4GI associate with influenza virus NS1 protein in viral mRNA translation initiation complexes. J. Gen. Virol. 84, 3263–3274.
Vende P., Piron M., Castagne N., Poncet D. (2000) Efficient translation of rotavirus mrna requires simultaneous interaction of NSP3 with the eukaryotic translation initiation factor eIF4G and the mRNA 3′ end. J. Virol. 74, 7064–7071.
Gratia M., Sarot E., Vende P., Charpilienne A., Baron C.H., Duarte M., Pyronnet S., Poncet D. (2015) Rotavirus NSP3 is a translational surrogate of the poly(A) binding protein-poly(A) complex. J. Virol. 89, 8773–8782.
Harb M., Becker M.M., Vitour D., Baron C.H., Vende P., Brown S.C., Bolte S., Arold S.T., Poncet D. (2008) Nuclear localization of cytoplasmic poly(A)-binding protein upon rotavirus infection involves the interaction of NSP3 with eIF4G and RoXaN. J. Virol. 82, 11283–11293.
Piron M., Vende P., Cohen J., Poncet D. (1998) Rotavirus RNA-binding protein NSP3 interacts with eIF4GI and evicts the poly(A) binding protein from eIF4F. EMBO J. 17, 5811–5821.
Joachims M., Van Breugel P.C., Lloyd R.E. (1999) Cleavage of poly(A)-binding protein by enterovirus proteases concurrent with inhibition of translation in vitro. J. Virol. 73, 718–727.
Kuyumcu-Martinez N.M., Van Eden M.E., Younan P., Lloyd R.E. (2004) Cleavage of poly(A)-binding protein by poliovirus 3C protease inhibits host cell translation: a novel mechanism for host translation shutoff. Mol. Cell. Biol. 24, 1779–1790.
Prévôt D., Darlix J.L., Ohlmann T. (2003) Conducting the initiation of protein synthesis: the role of eIF4G. Biol. Cell. 95, 141–156.
Guo L., Allen E.M., Miller W.A. (2001) Base-pairing between untranslated regions facilitates translation of uncapped, nonpolyadenylated viral RNA. Mol. Cell. 7, 1103–1109.
Fabian M.R., White K.A. (2006) Analysis of a 3′-translation enhancer in a tombusvirus: a dynamic model for RNA-RNA interactions of mRNA termini. RNA. 12, 1304–1314.
Nicholson B.L., White K.A. (2011) 3′ Cap-independent translation enhancers of positive-strand RNA plant viruses. Curr. Opin. Virol. 1, 373–380.
Simon A.E., Miller W.A. (2013) 3′ Cap-independent translation enhancers of plant viruses. Annu. Rev. Microbiol. 67, 21–42.
Sonenberg N., Hinnebusch A.G. (2009) Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136, 731–745.
Cho P.F., Poulin F., Cho-Park Y.A., Cho-Park I.B., Chicoine J.D., Lasko P., Sonenberg N. (2005) A new paradigm for translational control: inhibition via 5′-3′ mRNA tethering by Bicoid and the eIF4E cognate 4EHP. Cell. 121, 411–423.
Cho P.F., Gamberi C., Cho-Park Y.A., Cho-Park I.B., Lasko P., Sonenberg N. (2006) Cap-dependent translational inhibition establishes two opposing morphogen gradients in Drosophila embryos. Curr. Biol. 16, 2035–2041.
Stebbins-Boaz B., Cao Q., de Moor C.H., Mendez R., Richter J.D. (1999) Maskin Is a CPEB-associated factor that transiently interacts with eIF-4E. Mol. Cell. 4, 1017–1027.
Minshall N., Reiter M.H., Weil D., Standart N. (2007) CPEB interacts with an ovary-specific eIF4E and 4E-T in early Xenopus oocytes. J. Biol. Chem. 282, 37389–37401.
Nakamura A., Sato K., Hanyu-Nakamura K. (2004) Drosophila cup is an eIF4E binding protein that associates with bruno and regulates oskar mRNA translation in oogenesis. Dev. Cell. 6, 69–78.
Richter J.D. (2007) CPEB: a life in translation. Trends Biochem. Sci. 32, 279–285.
Kojima S., Sher-Chen E.L., Green C.B. (2012) Circadian control of mRNA polyadenylation dynamics regulates rhythmic protein expression. Genes Dev. 26, 2724–2736.
Kiriakidou M., Tan G.S., Lamprinaki S., De Planell-Saguer M., Nelson P.T., Mourelatos Z. (2007) An mRNA m7G cap binding-like motif within human Ago2 represses translation. Cell. 129, 1141–1151.
Kapasi P., Chaudhuri S., Vyas K., Baus D., Komar A.A., Fox P.L., Merrick W.C., Mazumder B. (2007) L13a blocks 48S assembly: role of a general initiation factor in mRNA-specific translational control. Mol. Cell. 25, 113–126.
Wang Z.F., Whitfield M.L., Ingledue T.C., Domin-ski Z., Marzluff W.F. (1996) The protein that binds the 3′ end of histone mRNA: a novel RNA-binding protein required for histone pre-mRNA processing. Genes Dev. 10, 3028–3040.
Neusiedler J., Mocquet V., Limousin T., Ohlmann T., Morris C., Jalinot P. (2012) INT6 interacts with MIF4GD/SLIP1 and is necessary for efficient histone mRNA translation. RNA. 18, 1163–1177.
Tan D., Marzluff W.F., Dominski Z., Tong L. (2013) Structure of histone mRNA stem-loop, human stem-loop binding protein, and 3′ hExo ternary complex. Science. 339, 318–321.
Von Moeller H., Lerner R., Ricciardi A., Basquin C., Marzluff W.F., Conti E. (2013) Structural and biochemical studies of SLIP1-SLBP identify DBP5 and eIF3g as SLIP1-binding proteins. Nucl. Acids Res. 41, 7960–7971.
Samuelsson T., Da M. (2008) Early evolution of histone mRNA 3′ end processing. RNA. 14, 1–10.
Choe J., Lin S., Zhang W., Liu Q., Wang L., Ramirez-Moya J., Du P., Kim W., Tang S., Sliz P., Santisteban P., George R.E., Richards W.G., Wong K.-K., Locker N., Slack F.J., Gregory R.I. (2018) mRNA circularization by METTL3–eIF3h enhances translation and promotes oncogenesis. Nature. 561, 556–560.
Fang L.T., Gelbart W.M., Ben-Shaul A. (2011) The size of RNA as an ideal branched polymer. J. Chem. Physics. 135, 19–21.
Yoffe A.M., Prinsen P., Gelbart W.M., Ben-Shaul A. (2011) The ends of a large RNA molecule are necessarily close. Nucl. Acids Res. 39, 292–299.
Clote P., Ponty Y., Steyaert J.M. (2012) Expected distance between terminal nucleotides of RNA secondary structures. J. Math. Biol. 65, 581–599.
Fang L.T. (2011) The end-to-end distance of RNA as a randomly self-paired polymer. J. Theor. Biol. 280, 101–107.
Han H.S.W., Reidys C.M. (2012) The 5′-3′ distance of RNA secondary structures. J. Comput. Biol. 19, 867–878.
Leija-Martínez N., Casas-Flores S., Cadena-Nava R.D., Roca J.A., Mendez-Cabañas J.A., Gomez E., Ruiz-Garcia J. (2014) The separation between the 5′-3′ ends in long RNA molecules is short and nearly constant. Nucl. Acids Res. 42, 13963–13968.
Lai W.-J.C., Kayedkhordeh M., Cornell E.V., Farah E., Bellaousov S., Rietmeijer R., Salsi E., Mathews D.H., Ermolenko D.N. (2018) mRNAs and lncRNAs intrinsically form secondary structures with short end-to-end distances. Nat. Commun. 9, 4328.
Ermolenko D.N., Mathews D.H. (2021) Making ends meet: new functions of mRNA secondary structure. Wiley Interdiscip. Rev. RNA. 12, 1–14.
Aw J.G.A., Shen Y., Wilm A., Sun M., Lim X.N., Boon K.L., Tapsin S., Chan Y.S., Tan C.P., Sim A.Y.L., Zhang T., Susanto T.T., Fu Z., Nagarajan N., Wan Y. (2016) In vivo mapping of eukaryotic RNA interactomes reveals principles of higher-order organization and regulation. Mol. Cell. 62, 603–617.
Lu Z., Zhang Q.C., Lee B., Flynn R.A., Smith M.A., Robinson J.T., Davidovich C., Gooding A.R., Goodrich K.J., Mattick J.S., Mesirov J.P., Cech T.R., Chang H.Y. (2016) RNA duplex map in living cells reveals higher-order transcriptome structure. Cell. 165, 1267–1279.
Sharma E., Sterne-Weiler T., O′Hanlon D., Blencowe B.J. (2016) Global mapping of human RNA–RNA interactions. Mol. Cell. 62, 618–626.
Egorova T., Biziaev N., Shuvalov A., Sokolova E., Mukba S., Evmenov K., Zotova M., Kushchenko A., Shuvalova E., Alkalaeva E. (2021) eIF3j facilitates loading of release factors into the ribosome. Nucl. Acids Res. 49, 11181–11196.
Skabkin M.A., Skabkina O.V., Hellen C.U.T., Pestova T.V. (2013) Reinitiation and other unconventional posttermination events during eukaryotic translation. Mol. Cell. 51, 249–264.
Rajkowitsch L., Vilela C., Berthelot K., Ramirez C.V., McCarthy J.E.G. (2004) Reinitiation and recycling are distinct processes occurring downstream of translation termination in yeast. J. Mol. Biol. 335, 71–85.
Tarun S.Z., Sachs A.B. (1995) A common function for mRNA 5′ and 3′ ends in translation initiation in yeast. Genes Dev. 9, 2997–3007.
Kessler S.H., Sachs A.B. (1998) RNA recognition motif 2 of yeast PAB1p is required for its functional interaction with eukaryotic translation initiation factor 4G. Mol. Cell. Biol. 18, 51–57.
Park E.-H., Zhang F., Warringer J., Sunnerhagen P., Hinnebusch A.G. (2011) Depletion of eIF4G from yeast cells narrows the range of translational efficiencies genome-wide. BMC Genomics. 12, 68.
Altmann M., Linder P. (2010) Power of yeast for analysis of eukaryotic translation initiation. J. Biol. Chem. 285, 31907–31912.
Borman A.M., Michel Y.M., Malnou C.E., Kean K.M. (2002) Free poly(A) stimulates capped mRNA translation in vitro through the eIF4G-poly(A)-binding protein interaction. J. Biol. Chem. 277, 36818–36824.
Kawaguchi D., Shimizu S., Abe N., Hashiya F., Tomoike F., Kimura Y., Abe H. (2020) Translational control by secondary-structure formation in mRNA in a eukaryotic system. Nucleosides, Nucleotides Nucl. Acids. 39, 195–203.
Wang Y., Wang Z. (2015) Efficient backsplicing produces translatable circular mRNAs. RNA. 21, 172–179.
Chen C.Y., Sarnow P. (1995) Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs. Science. 268, 415–417.
Abe N., Matsumoto K., Nishihara M., Nakano Y., Shibata A., Maruyama H., Shuto S., Matsuda A., Yoshida M., Ito Y., Abe H. (2015) Rolling circle translation of circular RNA in living human cells. Sci. Rep. 5, 16435. https://doi.org/10.1038/srep16435
Liu J., Liu T., Wang X., He A. (2017) Circles reshaping the RNA world: from waste to treasure. Mol. Cancer. 16, 1–12.
Cortés-López M., Miura P. (2016) Focus: epigenetics: emerging functions of circular RNAs. Yale J. Biol. Med. 89, 527.
Khong A., Parker R. (2018) mRNP architecture in translating and stress conditions reveals an ordered pathway of mRNP compaction. J. Cell Biol. 217, 4124–4140.
Sen N.D., Zhou F., Harris M.S., Ingolia N.T., Hinnebusch A.G. (2016) eIF4B stimulates translation of long mRNAs with structured 5′ UTRs and low closed-loop potential but weak dependence on eIF4G. Proc. Natl. Acad. Sci. USA. 113, 10464–10472.
Christensen A.K., Bourne C.M. (1999) Shape of large bound polysomes in cultured fibroblasts and thyroid epithelial cells. Anat. Rec. 255, 116–129.
Shelton E., Kuff E.L. (1966) Substructure and configuration of ribosomes isolated from mammalian cells. J. Mol. Biol. 22, 23–31.
Afonina Z.A., Myasnikov A.G., Shirokov V.A., Klaholz B.P., Spirin A.S. (2015) Conformation transitions of eukaryotic polyribosomes during multi-round translation. Nucl. Acids Res. 43, 618–628.
Brandt F., Carlson L.A., Hartl F.U., Baumeister W., Grünewald K. (2010) The three-dimensional organization of polyribosomes in intact human cells. Mol. Cell. 39, 560–569.
Afonina Z.A., Myasnikov A.G., Shirokov V.A., Klaholz B.P., Spirin A.S. (2014) Formation of circular polyribosomes on eukaryotic mRNA without cap-structure and poly(A)-tail: a cryo electron tomography study. Nucl. Acids Res. 42, 9461–9469.
Arava Y., Wang Y., Storey J.D., Liu C.L., Brown P.O., Herschlag D. (2003) Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proc. Natl. Acad. Sci. USA. 100, 3889–3894.
Thompson M.K., Rojas-Duran M.F., Gangaramani P., Gilbert W.V. (2016) The ribosomal protein Asc1/RACK1 is required for efficient translation of short mRNAs. ELife. 5, 1–22.
Thompson M.K., Gilbert W.V. (2017) mRNA length-sensing in eukaryotic translation: reconsidering the “closed loop” and its implications for translational control. Curr. Genet. 63, 613–620.
Çetin B., Leary S.E.O. (2021) mRNA- and factor-driven dynamic variability controls eIF4F-cap recognition for translation initiation. BioRxiv. 2021.06.17.448745; https://doi.org/10.1101/20
Koch A., Aguilera L., Morisaki T., Munsky B., Stasevich T.J. (2020) Quantifying the dynamics of IRES and cap translation with single-molecule resolution in live cells. Nat. Struct. Mol. Biol. 27, 1095–1104.
Subtelny A.O., Eichhorn S.W., Chen G.R., Sive H., Bartel D.P. (2014) Poly(A)-tail profiling reveals an embryonic switch in translational control. Nature. 508, 66–71.
Lim J., Lee M., Son A., Chang H., Kim V.N. (2016) MTAIL-seq reveals dynamic poly(A) tail regulation in oocyte-to-embryo development. Genes Dev. 30, 1671–1682.
Park J.-E., Yi H., Kim Y., Chang H., Kim V.N. (2016) Regulation of poly(A) tail and translation during the somatic cell cycle. Mol. Cell. 62, 462–471.
Елисеева И.А., Лябин Д.Н., Овчинников Л.П. (2013) Поли(А)-связывающие белки: строение, функции и регуляция активности. Успехи биол. химии. 53, 3–34.
Hay N. (2004) Upstream and downstream of mTOR. Genes Dev. 18, 1926–1945.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


