Молекулярная биология, 2022, T. 56, № 4, стр. 628-641
Rpn4p без ДНК-связывающего домена обеспечивает устойчивость Saccharomyces cerevisiae к окислительному стрессу и циклогексимиду
Д. С. Карпов a, *, Д. С. Спасская a, В. В. Тютяева a, В. Л. Карпов b
a Центр высокоточного редактирования и генетических технологий для биомедицины, Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: aleom@yandex.ru
Поступила в редакцию 12.02.2022
После доработки 09.03.2022
Принята к публикации 09.03.2022
- EDN: NLGPSJ
- DOI: 10.31857/S002689842204005X
Аннотация
Убиквитин-протеасомная система участвует в контроле всех основных молекулярных процессов в норме и реакции клеток на стрессовые воздействия. Rpn4p служит ключевым транскрипционным регулятором протеасомы у дрожжей класса Saccharomycetes и участвует также в ответе клеток на различные стрессовые воздействия. Помимо протеасомных генов, Rpn4 влияет на экспрессию нескольких сотен других, включая гены, вовлеченные в репарацию ДНК и ответ на окислительный стресс. В то же время молекулярные механизмы, используемые Rpn4 в контроле генов-мишеней и функционировании как регулятора ответа клеток на стрессовые условия, остаются во многом неясными. Цель работы состояла в определении доменов Rpn4, необходимых для обеспечения устойчивости клеток к стрессовым воздействиям. Показано, что N-концевая и центральная части белка, содержат участки, необходимые для устойчивости ко всем видам стресса. Предполагаемый сигнал ядерной локализации не влияет на функционирование Rpn4. Неожиданно белок с делецией обоих мотивов “цинковых пальцев”, образующих ДНК-связывающий домен, обеспечивал устойчивость дрожжей к окислительному стрессу и циклогексимиду. Более того, нами показано, что Rpn4 может привлекаться к промоторным областям регулируемых им генов даже в том случае, если они не содержат его сайтов связывания. На основании полученных данных можно предполагать, что Rpn4 участвует в регуляции генов и ответе клеток на стресс за счет белок-белковых взаимодействий.
Убиквитин-протеасомная система (УПС) включает в себя семейство ферментов, распознающих белковые субстраты и модифицирующих их ковалентно цепями небольшого белка убиквитина (масса около 8.5 кДа), и мультисубъединичный ATP-зависимый протеиназный комплекс – протеасому, куда доставляются полиубиквитинированые субстраты и где происходит отщепление от них полиубиквитиновых цепей, разворачивание белков и их расщепление до пептидов [1]. Основные функции УПС заключаются в контроле активности регуляторных белков и расщеплении белков, структура которых нарушена вследствие ошибок в экспрессии генетической информации или под действием стрессовых условий [2]. УПС участвует в протекании всех основных клеточных процессов и необходима в клеточном ответе на различные виды стресса. Нарушение функционирования УПС влечет развитие тяжелых нейродегенеративных заболеваний, дистрофию мышц, нарушение иммунного ответа и т.д. [2]. Активность УПС контролируется на различных уровнях, включая экспрессию генов. Так, активность протеасом у всех эукариот регулируется по механизму отрицательной обратной связи с участием Rpn4-подобных факторов транскрипции у низших эукариот (дрожжей) [3, 4] или факторов транскрипции высококонсервативного семейства Cap-N-Collar у высших эукариот [5]. Следует отметить, что экспрессия протеасомных генов регулируется координированно и бóльшая часть промоторных областей этих генов содержит сайты связывания соответствующих факторов [6]. У дрожжей класса Saccharomycetes Rpn4-подобные факторы транскрипции взаимодействуют с довольно консервативными элементами, называемыми PACE (Proteasome-Associated Control Element) за счет С-концевого ДНК-связывающего домена, содержащего два “цинковых пальца” [3, 4, 7]. В N-концевой и центральной части Rpn4p нами и другими авторами картированы трансактиваторные домены [8‒10]. Ранее M. Nelson и соавт. [11] предположили, что фрагмент полипептидной цепи 381‒398 этого белка представляет собой сигнал ядерной локализации Rpn4p и экспериментально показали локализацию Rpn4p в ядре.
Помимо протеасомных генов Rpn4p участвует в регуляции экспрессии нескольких сотен других дрожжевых генов [12], по-видимому, используя при этом как PACE, так и PACE-подобные элементы, обозначенные как MACE (MAG1-associated control element) и RACE (RPN8-associated control element) [13], а также укороченную GC-богатую часть сайта связывания ‒ так называемый PACE-core [14]. В частности, нами показана Rpn4-зависимая регуляция экспрессии генов нескольких путей репарации ДНК [15, 16], а также генов, вовлеченных в ответ клеток на окислительный стресс [17]. Нами и другими авторами показано, что Rpn4 участвует в клеточном ответе на различные виды стресса, включая ДНК-повреждающие агенты, тяжелые металлы, аналоги аминокислот, циклогексимид, окислительные агенты [10, 16‒18]. Несмотря на успехи в изучении структуры и функций Rpn4p, до сих пор не выяснены молекулярные механизмы, которые этот фактор задействует для выполнения роли регулятора транскрипции, а также регулятора клеточного ответа на стрессовые воздействия.
Цель представленной работы заключалась в идентификации доменов Rpn4p, необходимых для обеспечения устойчивости клетки к действию солей тяжелых металлов, окислительному стрессу и циклогексимиду.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы и условия их выращивания. В работе использовали штаммы Saccharomyces cerevisiae 334t rpn4-Δ (MATα, pep4-3, prb1-1122, ura3-52, leu2-3,112, reg1-501, gal1, rpn4::G418) с делецией гена RPN4 [8], штамм дикого типа BY4742 (MATα, his3D1, leu2D0, lys2D0, ura3D0) (“Euroscarf”, Германия) и производные от него мутантные штаммы rpn4-∆ (BY4742 YDL020c::kanMX4) (“Euroscarf”) и YPL (BY4742 pre1-8 nat+) [15], а также штамм PJ69-4A (MATα, trp1-901, leu2-3,112, ura3-52, his3-200, gal4-Δ, gal80-Δ, LYS2::GAL1-HIS3, GAL2-ADE2, met2::GAL7-lacZ) [19]. Дрожжевые культуры растили при 30°C на среде YPD (2% глюкозы, 2% пептона, 1% дрожжевого экстракта) и трансформировали, используя литий-ацетатный метод [20]. Штаммы, трансформированные экспрессионными плазмидами, выращивали в минимальной среде, содержащей YNB (6.7 г/л), смесь 20 аминокислот, глюкозу (2%), а также козаминовые кислоты (0.5%) без добавления урацила. В случае двугибридной системы дрожжевые трансформанты растили на селективной среде без триптофана, лейцина, гистидина и аденина. Использовали компоненты питательных сред производства “Sigma” (США). Плазмиды, использованные в работе, описаны в табл. 1.
Таблица 1.
Плазмиды, использованные в работе
| Название плазмиды | Описание плазмиды | Источник |
|---|---|---|
| pRS426 | Высококопийный (2μ) шаттл-вектор для клонирования | [21] |
| YCpLac33 | Низкокопийный (CEN) шаттл-вектор для клонирования | [22] |
| Ycp-Ub-dam | Вектор YCpLac33, кодирующий Dam-метилазу Escherichia coli с присоединенным убиквитинзависимым дегроном Rpn4p | [23] |
| YCp-GDR | Вектор YCpLac33, кодирующий Rpn4p, к которому с N-конца присоединена Dam-метилаза; экспрессия химерного гена контролируется промотором GAL1 | [23] |
| YCp-GDR-C/A | Вектор YCpLac33, кодирующий мутантный Rpn4p с заменой кодонов цистеина на кодоны аланина в позициях 450, 477, 480, к которому с N-конца присоединена Dam-метилаза | [23] |
| YCp-GDR-∆NCZnF | Вектор YCpLac33, кодирующий мутантный Rpn4p с делецией ДНК-связывающего домена, к которому с N-конца присоединена Dam-метилаза | Данная работа |
| pPDX2-GAL | Высококопийный дрожжевой экспрессионный шаттл-вектор | [24] |
| pPRPN4 | Вектор pPDX2-GAL, кодирующий полноразмерный белок Rpn4p под контролем GAL1 промотора | [8] |
| pPRPN4∆56 | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией 56 N-концевых а.о., под контролем GAL1 промотора | [8] |
| pPRPN4∆109 | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией 109 N-концевых а.о., под контролем GAL1 промотора | [8] |
| pPRPN4∆151 | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией 151 N-концевых а.о., под контролем GAL1 промотора | [8] |
| pPRPN4∆180 | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией 180 N-концевых а.о., под контролем GAL1 промотора | [8] |
| pPRPN4Δ210 | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией 210 N-концевых а.о., под контролем GAL1 промотора | [8] |
| pPRPN4ΔNAD | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией N-кислого домена под контролем GAL1 промотора | [8] |
| pPRPN4ΔCAD | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией С-кислого домена под контролем GAL1 промотора | [8] |
| pPRPN4ΔNCAD | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией обоих кислых доменов, включая участок между ними, под контролем GAL1 промотора | [8] |
| pPRPN4ΔNLS | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией предполагаемого сигнала ядерной локализации под контролем GAL1 промотора | Данная работа |
| pPRPN4ΔNZnF | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией предполагаемого N-цинкового пальца под контролем GAL1 промотора | Данная работа |
| pPRPN4ΔNCZnF | Вектор pPDX2-GAL, кодирующий Rpn4p с делецией обоих цинковых пальцев под контролем GAL1 промотора | [25] |
| pPRPN4(4K-R) | Вектор pPDX2-GAL, кодирующий Rpn4p с заменой четырех остатков лизина на остатки аргинина в главном N-концевом трансактиваторном домене | [8] |
| pGBT9 | Дрожжевой вектор двугибридной системы, кодирующий ДНК-связывающий домен фактора транскрипции Gal4p | “Takara Bio”, Saint-Germain-en-Laye, Франция |
| pGAD424 | Дрожжевой вектор двугибридной системы, кодирующий трансактиваторный домен фактора транскрипции Gal4p | “Takara Bio”, Saint-Germain-en-Laye, Франция |
| pGBT-Su(Hw) | Вектор pGBT9 с геном белка Su(Hw) Drosophila melanogaster, клонированным в одной рамке считывания с участком, кодирующим ДНК-связывающий домен Gal4p | [26] |
| pGAD-mod | Вектор pGAD424 с геном белка Mod(mdg4)-67.2 D. melanogaster, клонированным в одной рамке считывания с участком, кодирующим трансактиваторный домен Gal4p | [26] |
| pGBT10 | Вектор, производный от pGBT9 и содержащий сайт NcoI | Данная работа |
| pGAD425 | Вектор, производный от pGAD424 и содержащий сайт NcoI | Данная работа |
| pGBT-Rpn2 | Вектор pGBT9 с геном RPN2, клонированным в одной рамке считывания с участком, кодирующим ДНК-связывающий домен Gal4p | Данная работа |
| pGAD-Rpn2 | Вектор pGAD424 с геном RPN2, клонированным в одной рамке считывания с участком, кодирующим трансактиваторный домен Gal4p | Данная работа |
| pGBT-Nas2 | Вектор pGBT10 с геном NAS2, клонированным в одной рамке считывания с участком, кодирующим ДНК-связывающий домен Gal4p | Данная работа |
| pGAD-Nas2 | Вектор pGAD425 с геном NAS2, клонированным в одной рамке считывания с участком, кодирующим трансактиваторный домен Gal4p | Данная работа |
| pGBT-Rpn4 | Вектор pGBT10 с геном RPN4, клонированным в одной рамке считывания с участком, кодирующим ДНК-связывающий домен Gal4p | Данная работа |
| pGAD-Rpn4 | Вектор pGAD425 с геном RPN4, клонированным в одной рамке считывания с участком, кодирующим трансактиваторный домен Gal4p | Данная работа |
Получение плазмид pPRPN4ΔNLS и pPRPN4ΔNZnF. Варианты гена RPN4, содержащие делецию предполагаемого сигнала ядерной локализации и N-концевого “цинкового пальца”, получали путем двухстадийного сайт-направленного мутагенеза с помощью высокоточной ДНК-полимеразы KAPA HiFi (“Roche Molecular Systems, Inc.”, США), используя пары праймеров RPN4-NcoI-F/RPN4-ΔNLS-R и RPN4-ΔNLS-F/Rpn4-XhoI-R и RPN4-NcoI-F/RPN4-ΔNZnF-R и RPN4-ΔNZnF-F/Rpn4-XhoI-R (табл. 2) соответственно. Полученные ПЦР-фрагменты очищали c использованием набора GeneJET PCR Purification Kit (“Thermo Fisher Scientific”, США) и клонировали в вектор pPDX2 по сайтам эндонуклеаз BamHI и XhoI. Отсутствие посторонних мутаций и наличие целевых делеций в последовательностях клонированных фрагментов проверяли секвенированием.
Таблица 2.
Олигонуклеотиды, использованные в работе
| Олигонуклеотид | Нуклеотидная последовательность 5' → 3' | Источник |
|---|---|---|
| Клонирование делеционных форм RPN4 | ||
| RPN4-NcoI-F RPN4-XhoI-R RPN4-ΔNLS-F RPN4-ΔNLS-R RPN4-ΔNZnF-F RPN4-ΔNZnF-R |
AGAGAGAGACCATGGCTTCTACGGAACTTAGTа ACACACAGACCTCGAGACGGGGGGTAAGTATATAATATAAT AACGGCACATTAGAAAATTATACTAAGAAGCACTTA CTTAGTATAATTTTCTAATGTGCCGTTTTCATT GATAATGAATGCTAAAAGGAAGATTGTCTTC TCCTTTTAGCATTCATTATCTGACACGTAA |
Данная работа |
| RPN4-ΔNCZnF-R | CGACTGACGCTCGAGTTAATTCATTATCTGACACGTAA | [25] |
| Клонирование генов в векторы дрожжевой двугибридной системы | ||
| RPN2-EcoRI-F RPN2-XhoI-R NAS2-NcoI-F NAS2-XhoI-R pGAD-GBT-iMCS-F pGAD-GBT-iMCS-R |
GAGAGAGAAGAATTCTCTTTGACGACTGCTGCTCCT GAGAGAGAACTCGAGCTAAGCACTAGGGAAGTCGAC CAGAGACCATGGAGGAAGAAGAATTAAGT TACTGCCTCGAGTTATAGCTCTTGTATCCTACAACC AATTGACCATGGCACATATGACACCACAACTCGAGG AATTCCTCGAGTTGTGGTGTCATATGTGCCATGGTC |
Данная работа |
| DamIDb | ||
| RPT6-ND-F RPT6-ND-R ACT1+880-F ACT1+880-R ADH1-251-F ADH1-251-R RPT6-545-F RPT6-545-R |
TAAACGTGAAGGACTTAAAG CTTTTTCCACCATCATCAAC GATGTCACGGACAATTTCTC CTGGTTTCTCTCTACCTCAC CAAGAACCGTTCAACCATTTG CGTTAAATACTCTGGTGTCTG CTACAGTTCTGAATTTGAGC GACCAAATCTGAATAATCACG |
[23] |
| CLN1+300-F CLN1+300-R CLN2+319-F CLN2+319-R |
GTATCAACTTTCAGTGATGAC CTTGGTCCTTTAACACTACTC GCATAATAGTTACCAGAGTCG CCTACTGAAAAACGGAAAGG |
Данная работа |
| ПЦР в реальном времени | ||
| PDA1-EX-RT-F PDA1-EX-RT-R CLN2+319-F CLN2+319-R |
AGTTTGCTAAGGACTGGTGTC AATTGGATCGTTCTTGGATC GCATAATAGTTACCAGAGTCG CCTACTGAAAAACGGAAAGG |
Данная работа |
Двугибридная дрожжевая система. Гены RPN2, NAS2 и RPN4 амплифицировали с геномной ДНК дрожжей с помощью высокоточной ДНК-полимеразы KAPA HiFi (“Roche Molecular Systems, Inc.”), используя пары праймеров RPN2-EcoRI-F/RPN2-XhoI-R, NAS2-NcoI-F/XhoI-R и RPN4-NcoI-F/RPN4-XhoI-R. Полученные ПЦР-фрагменты очищали с помощью набора GeneJET PCR Purification Kit (“Thermo Fisher Scientific”). ПЦР-фрагмент гена RPN2 обрабатывали эндонуклеазами EcoRI и XhoI и клонировали в векторы pGBT9 и pGAD424, гидролизованные по сайтам эндонуклеаз EcoRI и SalI. Сайты XhoI и SalI после гидролиза соответствующими эндонуклеазами образуют комплементарные липкие концы. Отсутствие посторонних мутаций в последовательностях клонированных фрагментов проверяли секвенированием. Полученные плазмиды назвали pGBT-Rpn2 и pGAD-Rpn2 (табл. 1).
Перед клонированием генов NAS2 и RPN4 в векторы двугибридной системы ввели сайт NcoI. Векторы pGAD424 и pGBT9 обрабатывали эндонуклеазой EcoRI и лигировали с двухцепочечным олигонуклеотидом, имеющим липкие концы, комплементарные EcoRI, и образованным путем отжига олигонуклеотидов – pGAD-GBT-iMCS-F/pGAD-GBT-iMCS-R. Наличие вставки олигонуклеотида проверяли путем рестрикционного анализа по сайтам NcoI и PvuI с последующим секвенированием. Полученные плазмиды назвали pGAD425 и pGAD10. ПЦР-фрагменты генов NAS2 и RPN4 очищали набором GeneJET PCR Purification Kit (“Thermo Fisher Scientific”), обрабатывали эндонуклезами NcoI и XhoI и клонировали в модифицированные векторы двугибридной системы, обработанные эндонуклезами NcoI и SalI. Отсутствие посторонних мутаций в последовательностях клонированных фрагментов проверяли секвенированием. Полученные плазмиды назвали pGBT-Rpn4, pGBT-Nas2, pGAD-Rpn4 и pGAD-Nas2 (табл. 1).
Штамм PJ69-4a котрансформировали парой плазмид, производных векторов pGBT9 и pGAD424, с помощью стандартного литий-ацетатного метода [20]. Колонии выращивали на селективной среде без триптофана и лейцина. Затем колонии рассевали штрихом на чашках Петри с агаризованной селективной средой без триптофана, лейцина, гистидина и аденина в присутствии 5 мМ 3‑амино-1,2,4-триазола (3-AT) в качестве ингибитора фоновой активации экспрессии гена HIS3. Чашки инкубировали при 30°С в течение 2–3 суток.
Определение устойчивости дрожжей к стрессовым условиям путем выращивания серийных разведений дрожжевой культуры на чашках Петри. Дрожжевые культуры растили до стационарной фазы в течение ночи при 30°С. Культуры разводили до конечной оптической плотности OD600 = 1, готовили пятикратные разведения и высевали на твердую селективную среду с добавлением стрессовых агентов в виде капель объемом 5 мкл. В контроле штаммы растили на среде без добавления стрессовых агентов. Устойчивость к стрессу исследуемых дрожжевых штаммов определяли качественно по скорости формирования колоний при 30°С.
Определение активности Dam-метилазы (DamID). Плазмиду YCp-GDR-∆NCZnF, кодирующую химерный белок Dam-Rpn4∆NCZnF с делецией ДНК-связывающего домена, получали путем клонирования ПЦР-фрагмента гена RPN4, полученного с праймерами RPN4-NcoI-F и RPN4-∆NCZnF-R, в плазмиду YCp-GDR по сайтам NcoI и XhoI. Отсутствие посторонних мутаций в последовательностях клонированных фрагментов проверяли секвенированием.
Клетки штамма rpn4-∆, продуцирующие Dam-метилазу, соединенную с убиквитинзависимым дегроном Rpn4 (U-dam), гибридный белок Dam-Rpn4 или гибридные белки Dam-Rpn4-C/A и Dam-Rpn4∆NCZnF с нарушенной ДНК-связывающей активностью Rpn4, растили при 30°С до оптической плотности 1‒1.5 при длине волны 600 нм. Из полученной культуры выделяли геномную ДНК с использованием смеси фенол-хлороформ [27]. Далее 1 мкг геномной ДНК обрабатывали эндонуклеазами рестрикции MboΙ (“Thermo Fisher Scientific”) в концентрации 1 ед./мкл не менее 18 ч при 37°С. Обработанную ДНК амплифицировали методом ПЦР в реальном времени с праймерами, фланкирующими исследуемый GATC-сайт. Референтной последовательностью служил фрагмент гена RPT6, не содержащий сайтов GATC. Использованные в работе праймеры указаны в табл. 2. Каждую реакцию проводили в трех технических повторах на приборе LightCycler 480-II (“Roche Diagnostics”, США) в присутствии флуоресцентного красителя Eva Green (“Синтол”, Россия). Метилирующую активность (R) определяли по соотношению сигналов от обработанной и необработанной ферментом пробы. Относительную метилирующую активность (Rrel) определяли как соотношение метилирующих активностей Dam-Rpn4, или Dam-Rpn4-C/A, или Dam-Rpn4∆NCZnF к U‑dam. Разброс данных рассчитывали по ранее описанной методике [23].
ПЦР в реальном времени с обратной транскрипцией (ОТ). Относительные уровни мРНК интересующих генов определяли с помощью ОТ-ПЦР в реальном времени, как описано ранее [8].
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Картирование доменов Rpn4p, обеспечивающих устойчивость дрожжей к стрессовым факторам
Ранее нами проведен делеционный анализ RPN4 и оценена роль участков этого фактора в активации транскрипции протеасомных генов [8, 25]. Теперь полученный ранее набор мутантных производных RPN4 дополнен вариантами с делецией предполагаемого сигнала ядерной локализации (участок, кодирующий аминокислотные остатки 381‒398) и N-концевого домена “цинковых пальцев” (участок, кодирующий аминокислотные остатки 444‒468) (рис. 1а). В предыдущих работах картирование важных участков Rpn4p проводили в нормальных условиях роста дрожжей. Теперь основной задачей было определение участков Rpn4p, необходимых для обеспечения устойчивости дрожжевых клеток к различным стрессовым воздействиям, а именно: окислительному стрессу, вызванному действием перекиси водорода, ацетату кадмия и ингибитору трансляции – циклогексимиду. Согласно полученным данным (рис. 1б), как и ожидалось, продукция полноразмерного белка Rpn4p, его более активного производного Rpn4(4K-R) с заменой лизинов на аргинины в N-концевом трансактиваторном домене (NTAD) [8] или варианта с делецией N-концевого кислого домена (NAD), служащего убиквитинзависимым сигналом деградации Rpn4p [28], восстанавливает устойчивость мутантного штамма с делецией гена RPN4, ко всем стрессовым воздействиям. Делеция участка, состоящего из первых 56 аминокислот (Rpn4∆56), который входит в состав N-концевого сигнала убиквитиннезависимой деградации Rpn4p [29], лишь немного снижает защитную функцию Rpn4p.
Рис. 1.
Определение участков Rpn4p, обеспечивающих устойчивость дрожжей к стрессу. a – Схема положения делеций в Rpn4p. б – Результаты теста на устойчивость к стрессовым агентам штамма 334t rpn4-Δ, синтезирующего делеционные формы Rpn4p. Представлены фотографии репрезентативных чашек Петри после инкубации в течение 2 или 3 суток. В качестве положительного контроля использовали мутантный штамм rpn4-Δ, трансформированный плазмидой, кодирующей полноразмерный ген RPN4 (самый верхний ряд разведений), а в качестве негативного контроля – штамм rpn4-Δ, трансформированный вектором pPDX2 (второй ряд разведений сверху). CHX – циклогексимид.

Неожиданно дальнейшая делеция N-концевого домена Rpn4p привела к полной потере устойчивости дрожжей ко всем видам стресса, несмотря на то, что форма Rpn4∆109 сохраняла довольно высокую транскрипционную активность в отношении протеасомных генов [8]. Далее, хотя делеция 210 N-концевых а.о. полностью инактивировала Rpn4p в отношении транскрипции протеасомных генов [8, 25], мутантный вариант защищал клетки от действия окислительного стресса и кадмия. Следует отметить, что вариант с делецией 180 а.о. не обеспечивал защиту дрожжей от действия кадмия и окислительного стресса. Эти данные указывают на то, что между 180 и 210 а.о. может находится репрессорный домен, действующий в отношении непротеасомных генов-мишеней Rpn4p. Ранее нами и другими авторами выявлена способность Rpn4p функционировать в качестве репрессора транскрипции [12, 30]. Делеция С-концевого кислого домена (CAD), функционирующего как трансактиваторный домен [8], ожидаемо подавляла устойчивость дрожжей к окислительному стрессу и кадмию, но при этом защитные свойства в отношении циклогексимида сохранялись. Делеция центральной части белка, включающей оба кислых домена и участок между ними, инактивировала фактор в случае действия кадмия и значительно ослабляла при воздействии циклогексимида и окислительного стресса. Можно предположить, что полной инактивации не происходит из-за наличия NTAD у этих делеционных вариантов белка. На основании этих данных можно говорить о том, что делеция предполагаемого сигнала ядерной локализации Rpn4p не влияет на его функции, а также предположить наличие в его структуре еще одного сигнала ядерной локализации или возможность его транспорта в ядро в качестве белка, ассоциированного с протеасомой [4, 29]. Наиболее значимым и неожиданным результатом оказалось то, что при полной делеции ДНК-связывающего домена Rpn4p дрожжи сохраняют устойчивость к циклогексимиду и перекиси водорода, но становятся чувствительными к кадмию. Следует отметить, что при этом удаление N-концевого “цинкового пальца” с сохранением С-концевого “цинкового пальца” инактивировало Rpn4p в отношении всех видов стресса. Таким образом, для функционирования Rpn4p как антистрессорного фактора необязательно наличие ДНК-связывающего домена. Возможно, в этом случае Rpn4p вовлечен в белок-белковые взаимодействия.
Мутантный штамм с нарушенной экспрессией субъединицы 20S протеасомы проявляет повышенную устойчивость к циклогексимиду
Ранее при поиске мутаций, вызывающих сверхустойчивость к циклогексимиду, выявлены штаммы с мутациями в генах 20S протеолитического субкомплекса протеасомы [31]. Поскольку Rpn4p c делецией ДНК-связывающего домена не активирует транскрипцию протеасомных генов [8, 25], интересно было выяснить, способен ли мутантный штамм YPL с нарушенной Rpn4-зависимой регуляцией гена субъединицы PRE1 [15] проявлять сверхустойчивость к циклогексимиду. Согласно полученным результатам (рис. 2), штамм YPL проявлял повышенную устойчивость к циклогексимиду по сравнению со штаммом дикого типа при концентрации циклогексимида 50 нг/мкл. Однако при более высокой концентрации циклогексимида, 75 нг/мкл, разница в скорости формирования колоний мутанта и дикого типа отсутствовала. Ранее U. Gerlinger и соавт. сообщали [31], что не все мутации, в частности в гене PRE1, ведут к четко выраженному фенотипу сверхустойчивости к циклогексимиду. Как правило, таким фенотипом обладают штаммы, несущие мутации одновременно в двух генах (например, pre1-1 pre4-1), что соответствует очень сильному нарушению протеасомзависимого протеолиза. Можно отметить, что в штамме 334t rpn4-∆ нарушена деградация белков в двух внутриклеточных протеолитических системах: УПС (отсутствие трансактиватора) и автофагии (отсутствие ключевой протеазы Prb1p, обеспечивающей активацию других протеаз в аутофагосомах [32]). Возможно, такое серьезное нарушение внутриклеточного протеолиза функционально соответствует двойным мутациям в протеасомных генах и вносит вклад в наблюдаемый фенотип устойчивости к циклогексимиду в присутствие Rpn4∆210 и Rpn4∆NCZnF.
Рис. 2.
Проверка устойчивости к циклогексимиду (CHX) штаммов дикого типа и мутантов с нарушенной функцией протеасомы. Представлены фотографии репрезентативных чашек Петри после инкубации в течение 2 или 3 суток при 30°С. В эксперименте использованы штамм дикого типа BY4742 (WT), производный от него вариант с делецией гена RPN4 (rpn4-Δ) и две независимые колонии штамма YPL с нарушенной Rpn4-зависимой регуляцией экспрессии гена PRE1, кодирующего субъединицу 20S протеолитического комплекса протеасомы.

Rpn2p и Nas2p не взаимодействуют с Rpn4 в двугибридной дрожжевой системе
Полученные нами данные указывают на то, что нарушение экспрессии протеасомных генов вряд ли служит основным механизмом, посредством которого мутант Rpn4∆NCZnF обеспечивает устойчивость дрожжей к стрессу. Логично предположить, что в отсутствие ДНК-связывающей активности мутантный Rpn4p функционирует за счет взаимодействия с белками-партнерами и белковыми комплексами. Ранее показано, что Rpn4p может очищаться вместе с протеасомой [33] и способен связываться с отдельными субъединицами 19S регуляторного комплекса протеасомы, включая Rpn2p [4, 29]. Кроме того, в масштабных исследованиях белок-белковых взаимодействий показано, что Rpn4p физически взаимодействует с Nas2p [34], который служит одним из специализированных шаперонов, обеспечивающих сборку 19S регуляторного субкомплекса протеасомы [35]. Чтобы проверить полученные ранее данные о взаимодействии Rpn4p с Rpn2p и Nas2p, мы использовали дрожжевую двугибридную систему. Положительным контролем служили конструкции, продуцирующие белки Su(Hw) и Mod(mdg4), взаимодействие между которыми в двугибридной системе показано ранее [26]. Согласно полученным данным (рис. 3a), Rpn4p, будучи соединенным с ДНК-связывающим доменом Gal4p, может активировать экспрессию репортерных генов и в отсутствие белка-партнера с трансактиваторным доменом. Очевидно, это происходит благодаря наличию собственных трансактиваторных доменов Rpn4p. В парах pGBT-Rpn2/pGAD425-Rpn4 и pGBT-Nas2/pGAD425-Rpn4 не выявлено активации репортерных генов, что выражается в отсутствии роста дрожжевых колоний на селективной среде. Следует отметить, что дрожжи растили в присутствии 5 мМ 3-AT, ‒ чтобы надежно подавлять фоновую активацию репортерных генов. Полученные нами данные согласуются с результатами систематичного анализа, проведенного G. Cagney и др. [36], по взаимодействию между субъединицами протеасомы в дрожжевой двугибридной системе. Авторы тоже не обнаружили продуктивного взаимодействия между Rpn4p и Rpn2p при использовании 3-AT, но выявили взаимодействие Rpn4p со структурной субъединицей 20S протеасомы Pre7p. Однако позже S. Ha и соавт. [29] с помощью высокочувствительного метода переноса биотиновой метки показали взаимодействие Rpn4p in vitro с такими субъединицами 19S регуляторного комплекса протеасомы, как Rpn2p, Rpn5p, Rpt1p и Rpt6. Можно предположить, что выявленные взаимодействия Rpn4p с протеасомными субъединицами кратковременные и служат “инструментом” деградации Rpn4p в протеасоме.
Рис. 3.
Анализ взаимодействия Rpn4p с субъединицей 19S регуляторного комплекса протеасомы Rpn2p и шапероном Nas2p в дрожжевой двугибридной системе. а – Фотография репрезентативной чашки Петри. б – Схема комбинаций плазмид, использованных при трансформации дрожжей. Штамм PJ69-4a, трансформированный комбинацией плазмид pGBT-Su(Hw) и pGAD-mod, служил положительным контролем наличия белок-белковых взаимодействий.
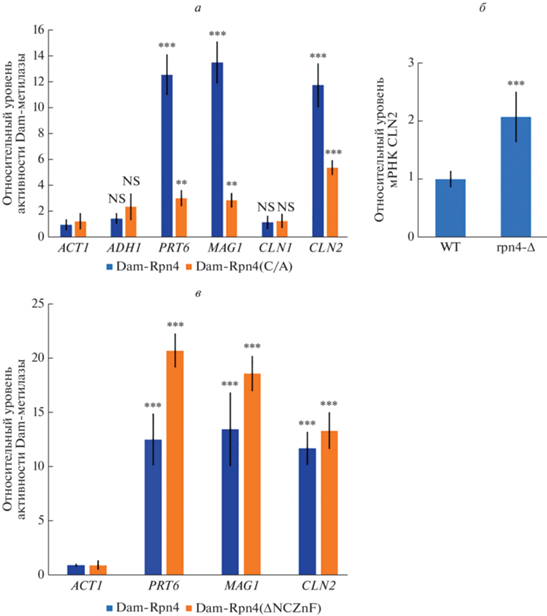
Rpn4p может привлекаться к промоторным областям генов за счет белок-белковых взаимодействий
В ходе исследования взаимодействия Rpn4p с промоторными областями генов, как содержащих его сайты связывания (PACE или MACE), так и не содержащих их, нами неожиданно обнаружено, что Rpn4p может привлекаться к промотору гена циклина CLN2, который не содержит сайтов связывания фактора. Более того, мутантный Rpn4p с ДНК-связывающим доменом, инактивированным путем внесения замен остатков цистеина на аланины, также привлекается к промотору CLN2, хотя и с меньшей силой (рис. 4а). Полученные данные указывают на то, что Rpn4p может привлекаться к регуляторным областям генов, по-видимому, за счет белок-белковых взаимодействий. Из литературных данных известно, что привлечение фактора транскрипции к гену может быть непродуктивным, то есть не приводить к изменению экспрессии гена [37]. С целью выяснить, продуктивно ли привлечение Rpn4p к CLN2, мы определили уровень мРНК CLN2 в штамме BY4742 с мутацией rpn4-∆ относительно изогенного штамма дикого типа. Оказалось, что в мутантном штамме уровень мРНК CLN2 повышен в среднем в 2 раза (рис. 4б). Из этого следует, что Rpn4p служит репрессором гена CLN2. Кроме того, проверили происходит ли взаимодействие мутантного Rpn4p с делецией ДНК-связывающего домена, Rpn4∆NCZnF, с геном CLN2. Согласно полученным данным (рис. 4в), взаимодействие Rpn4∆NCZnF усилилось и превысило уровень, наблюдаемый в случае белка дикого типа. Взаимодействие Rpn4∆NCZnF усилилось также и с промоторными областями Rpn4p-зависимых генов: RPT6, содержащего PACE, и MAG1, содержащего MACE. Эти результаты можно объяснить полученными нами ранее данными о том, что уровень экспрессии мутантного белка Rpn4∆NCZnF значительно выше, чем белка дикого типа [25]. Таким образом, показано, что Rpn4p может привлекаться к регулируемым им генам за счет белок-белковых взаимодействий и этот процесс стимулируется в случае делеции ДНК-связывающего домена.
Рис. 4.
Rpn4p может привлекаться к промоторным областям регулируемых им генов за счет белок-белковых взаимодействий. а – Привлечение Rpn4p к промоторным областям генов-мишеней и генов, не регулируемых Rpn4p. В анализе DamID за единицу активности принят сигнал, полученный при метилировании гена ACT1 химерным белком Dam-Rpn4. б – Анализ относительного уровня мРНК гена CLN2 с помощью ОТ-ПЦР в реальном времени. В качестве референсного гена использован PDA1. в – Результаты анализа DamID с использованием химерного белка Dam-Rpn4∆NCZnF. Данные представлены в виде среднего для трех независимых повторов, разброс данных представлен стандартным отклонением. В экспериментах использованы штаммы BY4742 и производный от него BY4742 rpn4-∆. Статистическая значимость различий в средних определена с помощью t-критерия Стьюдента. NS – отличия статистически незначимы; **0.001 < p-value < 0.01; ***p-value < 0.001.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В этой работе мы использовали набор делеционных производных Rpn4p, чтобы картировать функционально важные участки этого транскрипционного фактора, ответственные за его “работу” в условиях воздействия различных видов стресса. Установлено, что бóльшая часть N-концевой области, содержащая трансактиваторный домен NTAD, а также кислый трансактиваторный домен CAD нужны для устойчивости дрожжей ко всем видам стресса. В то же время без NAD, предполагаемого сигнала ядерной локализации или ДНК-связывающего домена Rpn4p может продолжать обеспечивать защиту дрожжей от стрессовых воздействий. Результаты, полученные с помощью DamID-анализа, указывают на возможность Rpn4p, лишенного ДНК-связывающего домена, участвовать в регуляции экспрессии генов за счет белок-белковых взаимодействий.
Отметим, что мутантные варианты Rpn4p без NAD и NCZnF наиболее активны в отношении устойчивости дрожжей к циклогексимиду и окислительному стрессу (рис. 1б). Эти данные можно объяснить повышением содержания соответствующих мутантных вариантов Rpn4p, которые наблюдали ранее [25]. В случае делеции NAD стабилизация Rpn4p связана, по-видимому, с нарушением его деградации по убиквитинзависимому пути [28]. На данный момент неясно, по какой причине происходит стабилизация Rpn4p при делеции ДНК-связывающего домена.
Отсутствие активности предполагаемого сигнала ядерной локализации можно рассматривать как указание на наличие, как минимум, еще одного сигнала транспорта Rpn4p в ядро, который компенсирует отсутствие этого участка. Таким сигналом может служить N-концевой дегрон, который распознается α-кариоферином Srp1p в комплексе с Sts1p [38]. Это предположение согласуется с полученными нами данными, указывающими на то, что делеция первых 56 а.о., включающих значительную часть N-дегрона, незначительно влияет на активность Rpn4p. Однако делеция 109 а.о., включающая в себя полный N-дегрон, подавляет способность Rpn4 обеспечивать устойчивость дрожжей к стрессу (рис. 1б). Отметим, что мутантный белок сохраняет некоторую активность активатора транскрипции протеасомных генов [8]. Это не исключает того, что делеция 109 а.о. может подавлять способность Rpn4p регулировать экспрессию непротеасомных генов. Не исключено также, что предполагаемый сигнал ядерной локализации менее активен, чем N-концевой дегрон.
Полученные нами и другими авторами данные позволяют сформулировать несколько предположений о том, каким образом Rpn4p, лишенный ДНК-связывающей функции, обеспечивает устойчивость дрожжей к стрессу.
Первое предположение заключается в том, что Rpn4p без ДНК-связывающего домена может нарушать деградацию субстратов протеасомы, включая белки, обеспечивающие устойчивость к рассматриваемым видам стресса. Эта гипотеза основана на данных о том, что делеция ДНК-связывающего домена повышает содержание Rpn4 [25], а так как при этом сохранена N-концевая область Rpn4p, обеспечивающая взаимодействие с 19S регуляторным комплексом протеасомы [29], то мутантный Rpn4p продолжает более интенсивно связываться с протеасомой, конкурируя с другими ее субстратами. Кроме того, мутантный Rpn4p сохраняет также Ubr2-зависимый дегрон [28], благодаря чему может конкурировать за связывание и полиубиквитинирование с другими субстратами Ubr2-лигазы, вызывая их стабилизацию.
Второе предположение заключается в привлечении Rpn4p в качестве трансактиваторного домена к генам, не имеющим его сайтов связывания. В отсутствие ДНК-связывающего домена происходит диссоциация мутантного Rpn4p от участков его связывания в геноме и тогда, взаимодействуя с другими факторами транскрипции, он становится участником регуляции другого набора генов, в том числе вовлеченных в устойчивость к исследуемым видам стресса.
И, наконец, третье предположение состоит в том, что Rpn4p связывается с другими белками, не имеющими отношение к регуляции транскрипции генов, но вовлеченными в экспрессию генетической информации на других этапах, в том числе и в устойчивость дрожжей к циклогексимиду и окислительному стрессу. Так, согласно информации, представленной в базе данных Saccharomyces cerevisiae database (https://www.yeastgenome.org/ посещение 06.02.2022), обнаружено физическое взаимодействие Rpn4p с 29 белками. Почти половину из этих белков составляют субъединицы протеасомы или ее шапероны (Nas2p, Pre2p, Pre7p, Rpn2p, Rpn5p, Rpn6p, Rpn10p, Rpn12p, Sem1p, Rpt1p, Rpt2p, Rpt3p, Rpt5p, Rpt6p), а также ферменты системы полиубиквитинирования белков (Ubr1p и Mub1p) и Hsp40p (Apj1p), вовлеченный в SUMO-опосредованную деградацию белковых агрегатов. Взаимодействие с этими белками, по-видимому, функционально ведет к деградации Rpn4p. К этой же группе можно отнести Srp1p, гомолог кариоферина-α, который, помимо импорта белков в ядро, взаимодействует с рибосомой и протеасомой и опосредует деградацию новосинтезированных белков [38]. Показано, что Srp1p взаимодействует с областью N-концевого дегрона Rpn4p и обеспечивает его деградацию в протеасоме, а нарушение функционирования Srp1p повышает чувствительность дрожжей к циклогексимиду и другим стрессовым факторам, вызывающим накопление неправильно свернутых белков [38]. Помимо Srp1p в связывании и транспорте Rpn4p в ядро может участвовать Mog1p, который, в свою очередь, связывается с Ran-GTPазой [39]. Другая часть белков, взаимодействующих с Rpn4, вовлечена в регуляцию экспрессии генов на уровне организации хроматина (Ccr4p, Esa1p, Isw1p), метаболизма мРНК (Nab2p, Dhh1p, Puf3p), регуляции трансляции (Caf20p, Hek2p) и cворачивания новосинтезированных белков (Ssb2p). Отдельно нужно отметить взаимодействие Rpn4p c Dpb2p – второй большой субъединицей ДНК-полимеразы II, участвующей в репликации и репарации ДНК [40]. Известно, что ДНК-полимераза II, участвует в репаративном синтезе ДНК в ходе устранения повреждений ДНК, вызванных окислительным стрессом [41].
Полученные нами и другими авторами [36] данные указывают на то, что взаимодействие Rpn4p с компонентами УПС имеет временный характер и ведет к расщеплению Rpn4p. Возможно, такое же значение имеют взаимодействия Rpn4p с шаперонами, участвующими в сворачивании новосинтезированных белков. Взаимодействие Rpn4p (прямое или опосредованное) с белками, участвующими в регуляции экспрессии генов на уровне организации хроматина, по-видимому, связано с его функциями как регулятора экспрессии генов. На сегодняшний день неясно, имеет ли функциональное значение взаимодействие Rpn4p с регуляторами трансляции и ДНК-полимеразой II, которые, в свою очередь, имеют отношение к ответу дрожжей на действие циклогексимида и окислительный стресс. Очевидно, что взаимодействие с этими белками не требует участия ДНК-связывающего домена Rpn4p. Таким образом, имеющиеся на сегодняшний день данные указывают на то, что Rpn4p может быть вовлечен в белок-белковые взаимодействия, которые выходят за рамки его функций как субстрата протеасомы или фактора транскрипции. Необходимы дальнейшие исследования, чтобы верифицировать обнаруженные белки-партнеры и установить значение взаимодействий Rpn4p с ними.
Авторы выражают благодарность Центру высокоточного редактирования генома и генетических технологий для биомедицины ИМБ РАН за предоставленные вычислительные мощности и методики для анализа данных.
Работа выполнена при поддержке Российского Научного Фонда (грант № 17-74-30030).
Работа выполнена с использованием оборудования центра коллективного пользования “Геном” ИМБ РАН (http://www.eimb.ru/ru1/ckp/ccu_genome_ce.php).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Glickman M.H., Ciechanover A. (2002) The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiol. Rev. 82(2), 373‒428.
Lecker S.H., Goldberg A.L., Mitch W.E. (2006) Protein degradation by the ubiquitin-proteasome pathway in normal and disease states. J. Am. Soc. Nephrol. 17(7), 1807‒1819.
Карпов Д.С., Лысов Ю.П., Карпов В.Л. (2019) Эволюция системы координированной регуляции экспрессии протеасомных генов у дрожжей-сахаромицетов. Молекуляр. биология. 53(6), 1029‒1037.
Xie Y., Varshavsky A. (2001) RPN4 is a ligand, substrate and transcriptional regulator of the 26S proteasome: a negative feedback circuit. Proc. Natl. Acad. Sci. USA. 98(6), 3056‒3061.
Xie Y. (2010) Feedback regulation of proteasome gene expression and its implications in cancer therapy. Cancer Metastasis Rev. 29(4), 687‒693.
Mannhaupt G., Feldmann H. (2007) Genomic evolution of the proteasome system among Hemiascomycetous yeasts. J. Mol. Evol. 65(5), 529‒540.
Mannhaupt G., Schnall R., Karpov V., Vetter I., Feldmann H. (1999) Rpn4 acts as a transcription factor by binding to PACE, a nonamer box found upstream of 26S proteasomal and other genes in yeast. FEBS Lett. 450, 27‒34.
Karpov D.S., Tutyaeva V.V., Karpov V.L. (2008) Mapping of yeast Rpn4p transactivation domains. FEBS Lett. 582(23–24), 3459‒3464.
Ju D., Xu H., Wang X., Xie Y. (2010) The transcription activation domain of Rpn4 is separate from its degrons. Int. J. Biochem. Cell Biol. 42(2), 282‒286.
Karpov D.S., Grineva E.N., Leinsoo A.T., Nadolinskaia N.I., Danilenko N.K., Tutyaeva V.V., Spasskaya D.S., Preobrazhenskaya O.V., Lysov Y.P., Karpov V.L. (2017) Functional analysis of Debaryomyces hansenii Rpn4 on a genetic background of Saccharomyces cerevisiae. FEMS Yeast Res. 17(1), fow098.
Nelson M.K., Kurihara T., Silver P.A. (1993) Extragenic suppressors of mutations in the cytoplasmic C terminus of SEC63 define five genes in Saccharomyces cerevisiae. Genetics. 134(1), 159‒173.
Jelinsky S.A., Estep P., Church G.M., Samson L.D. (2000) Regulatory networks revealed by transcriptional profiling of damaged Saccharomyces cerevisiae cells: Rpn4 links base excision repair with proteasomes. Mol. Cell Biol. 20, 8157‒8167.
Капранов А.Б., Преображенская О.В., Тютяева В.В., Штука Р., Фельдман Х., Карпов В.Л. (2001) Выделение и идентификация PACE-связывающего белка Rpn4 ‒ нового транскрипционного активатора, участвующего в регуляции 26S протеасомных и других генов. Молекуляр. биология. 35, 420‒431.
Shirozu R., Yashiroda H., Murata S. (2015) Identification of minimum Rpn4-responsive elements in genes related to proteasome functions. FEBS Lett. 589(8), 933‒940.
Karpov D.S., Spasskaya D.S., Tutyaeva V.V., Miro-nov A.S., Karpov V.L. (2013) Proteasome inhibition enhances resistance to DNA damage via upregulation of Rpn4-dependent DNA repair genes. FEBS Lett. 587(18), 3108‒3114.
Spasskaya D.S., Nadolinskaia N.I., Tutyaeva V.V., Lysov Y.P., Karpov V.L., Karpov D.S. (2020) Yeast Rpn4 links the proteasome and DNA repair via RAD52 regulation. Int. J. Mol. Sci. 21(21), 8097.
Karpov D.S., Spasskaya D.S., Nadolinskaia N.I., Tutyaeva V.V., Lysov Y.P., Karpov V.L. (2019) Deregulation of the 19S proteasome complex increases yeast resistance to 4-NQO and oxidative stress via upregulation of Rpn4- and proteasome-dependent stress responsive genes. FEMS Yeast Res. 19(2), foz002.
Teixeira M.C., Dias P.J., Simoes T., Sa-Correia I. (2008) Yeast adaptation to mancozeb involves the up-regulation of FLR1 under the coordinate control of Yap1, Rpn4, Pdr3, and Yrr1. Biochem. Biophys. Res. Commun. 367(2), 249‒255.
James P., Halladay J., Craig E.A. (1996) Genomic libraries and a host strain designed for highly efficient two-hybrid selection in yeast. Genetics. 144(4), 1425‒1436.
Gietz R.D., Woods R.A. (2002) Transformation of yeast by the LiAc/ss carrier DNA/PEG method. Methods Enzymol. 350, 87–96.
Christianson T.W., Sikorski R.S., Dante M., Shero J.H., Hieter P. (1992) Multifunctional yeast high-copy-number shuttle vectors. Gene. 110(1), 119‒122.
Gietz R.D., Sugino A. (1988) New yeast-Escherichia coli shuttle vectors constructed with in vitro mutage-nized yeast genes lacking six-base pair restriction sites. Gene. 74(2), 527‒534.
Спасская Д.С., Карпов Д.С., Карпов В.Л. (2011) Dam-метилаза Escherichia coli как молекулярный инструмент для картирования сайтов связывания дрожжевого фактора транскрипции Rpn4p. Молекуляр. биология. 45(4), 642‒651.
Козлов Д.Г., Чеперегин С.Э., Честков А.В., Крылов В.Н., Цыганков Ю.Д. (2010) Клонирование и экспрессия гена лизоцима бактериофага FMV в клетках дрожжей Saccharomyces cerevisiae и Pichia pastoris. Генетика. 46(3), 340‒348.
Карпов Д.С., Тютяева В.В., Берестень С.Ф., Карпов В.Л. (2008) Картирование участков Rpn4p, ответственных за активацию транскрипции протеасомных генов. Молекуляр. биология. 42(3), 526‒532.
Melnikova L., Kostyuchenko M., Parshikov A., Georgiev P., Golovnin A. (2018) Role of Su(Hw) zinc finger 10 and interaction with CP190 and Mod(mdg4) proteins in recruiting the Su(Hw) complex to chromatin sites in Drosophila. PLoS One. 13(2), e0193497.
Ausubel F.M., Brent R., Kingston R.E., Moore D.D., Smith J.A., Seidman J.G., Struhl K. (1998) Current Protocols in Molecular Biology. New York: Wiley-Interscience.
Ju D., Xie Y. (2006) Identification of the preferential ubiquitination site and ubiquitin-dependent degradation signal of Rpn4. J. Biol. Chem. 281, 10657‒10662.
Ha S.W., Ju D., Xie Y. (2012) The N-terminal domain of Rpn4 serves as a portable ubiquitin-independent degron and is recognized by specific 19S RP subunits. Biochem. Biophys. Res. Commun. 419(2), 226‒231.
Карпов Д.С., Осипов С.А., Преображенская О.В., Карпов В.Л. (2008) Rpn4p ‒ позитивный и негативный регулятор транскрипции убиквитин-протеасомной системы. Молекуляр. биология. 42(3), 518‒525.
Gerlinger U.M., Guckel R., Hoffmann M., Wolf D.H., Hilt W. (1997) Yeast cycloheximide-resistant crl mutants are proteasome mutants defective in protein degradation. Mol. Biol. Cell. 8(12), 2487‒2499.
He C., Klionsky D.J. (2009) Regulation mechanisms and signaling pathways of autophagy. Annu. Rev. Genet. 43, 67‒93.
Fujimoro M., Tanaka K., Yokosawa H., Toh-e A. (1998) Son1p is a component of the 26S proteasome of the yeast Saccharomyces cerevisiae. FEBS Lett. 423, 149‒154.
Drees B.L., Sundin B., Brazeau E., Caviston J.P., Chen G.C., Guo W., Kozminski K.G., Lau M.W., Moskow J.J., Tong A., Schenkman L.R., McKenzie A., 3rd, Brennwald P., Longtine M., Bi E., Chan C., Novick P., Boone C., Pringle J.R., Davis T.N., Fields S., Drubin D.G. (2001) A protein interaction map for cell polarity development. J. Cell Biol. 154(3), 549‒571.
Funakoshi M., Tomko R.J., Jr., Kobayashi H., Hochstrasser M. (2009) Multiple assembly chaperones govern biogenesis of the proteasome regulatory particle base. Cell. 137(5), 887‒899.
Cagney G., Uetz P., Fields S. (2001) Two-hybrid ana-lysis of the s 26S proteasome. Physiol. Genomics. 7(1), 27‒34.
Lickwar C.R., Mueller F., Hanlon S.E., McNally J.G., Lieb J.D. (2012) Genome-wide protein-DNA binding dynamics suggest a molecular clutch for transcription factor function. Nature. 484(7393), 251‒255.
Ha S.W., Ju D., Xie Y. (2014) Nuclear import factor Srp1 and its associated protein Sts1 couple ribosome-bound nascent polypeptides to proteasomes for cotranslational degradation. J. Biol. Chem. 289(5), 2701‒2710.
Baker R.P., Harreman M.T., Eccleston J.F., Corbett A.H., Stewart M. (2001) Interaction between Ran and Mog1 is required for efficient nuclear protein import. J. Biol. Chem. 276(44), 41255‒41262.
Jain R., Aggarwal A.K., Rechkoblit O. (2018) Eukaryotic DNA polymerases. Curr. Opin. Struct. Biol. 53, 77‒87.
Chalissery J., Jalal D., Al-Natour Z., Hassan A.H. (2017) Repair of oxidative DNA damage in Saccharomyces cerevisiae. DNA Repair (Amst.). 51, 2‒13.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


