Молекулярная биология, 2022, T. 56, № 5, стр. 860-880
Биочип для генотипирования полиморфизмов, ассоциированных с цветом глаз, волос, кожи, группой крови, половой принадлежностью, основной гаплогруппой Y-хромосомы, и его использование для исследования славянской популяции
Д. О. Фесенко a, *, И. Д. Ивановский b, П. Л. Иванов c, Е. Ю. Земскова c, А. С. Агапитова b, С. А. Поляков a, О. Е. Фесенко d, М. А. Филиппова a, А. С. Заседателев a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b ООО “Центр исследования ДНК”
141402 Химки, Московская обл., Россия
c Российский центр судебно-медицинской экспертизы Министерства здравоохранения
Российской Федерации
125284 Москва, Россия
d Научно-исследовательский институт физики Южного федерального университета
344090 Ростов-на-Дону, Россия
* E-mail: deferos@yandex.ru
Поступила в редакцию 04.04.2022
После доработки 20.04.2022
Принята к публикации 20.04.2022
- EDN: SSMGEC
- DOI: 10.31857/S0026898422050056
Аннотация
В работе представлен метод генотипирования панели из 60 однонуклеотидных полиморфизмов (SNP) с помощью одностадийной ПЦР с последующей гибридизацией на гидрогелевом биочипе. Пул анализируемых полиморфизмов состоит из 41 SNP, входящих в панель HIrisPlex-S, 4 SNP гена АВ0 (261G>Del, 297A>G, 657C>T, 681G>A), маркеров генов AMELX и AMELY и 14 SNP-маркеров гаплогрупп Y-хромосомы: B (M60), C (M130), D (CTS3946), E (M5388), G (P257), H (M2920), I (U179), J (M304), L (M185), N (M231), O (M175), Q (M1105), R (P224) и T (M272). Получаемые генетические данные позволяют прогнозировать фенотип искомого лица по признакам цвета глаз, волос, кожи, группе крови АВ0, половой принадлежности, геногеографического происхождения по мужской линии. Протокол постановки максимально упрощен для облегчения внедрения метода в практику. Установлено распределение частот аллелей исследуемых полиморфизмов, а также групп крови АВ0 среди славян (N = 482), происхождением преимущественно из центральной России.
Преступность сопровождает человечество на протяжении всей его истории и поимка преступника всегда будет оставаться актуальной задачей для общества. Именно поэтому криминалистика ‒ одна из первых областей, в которой достижения молекулярной биологии нашли практическое применение [1]. Запрос экспертов-криминалистов стимулирует исследования, направленные на поиск любой полезной для следствия информации, которая может быть извлечена из генома. В настоящее время можно разделить получаемую информацию на два типа: идентифицирующая и поисковая.
Идентифицирующая информация представляет собой уникальный, в масштабе популяции, генетический профиль человека. Пионерские работы в области молекулярно-генетической экспертизы были посвящены идентификации, или геномной дактилоскопии. Наибольшее практическое развитие геномная дактилоскопия получила на своей родине, в Великобритании: на июнь 2021 года национальная база этой страны (The United Kingdom National DNA Database; NDNAD) содержала 5.7 млн генетических профилей (для сравнения, в национальной базе биометрической информации IDENT1 находится немногим больше – всего 7.1 млн человек). Учитывая, что по данным NDNAD более 15% профилей задублировано, в базе оказалось не более 7.3% всего населения страны. Несмотря на то, что в NDNAD попадают наименее законопослушные граждане, вероятность идентификации преступника по совпадению с профилем в базе, как можно видеть, невелика даже в наиболее развитых в этом отношении странах.
В случае отсутствия профиля разыскиваемого лица в базе данных на первый план выходят методы получения поисковой информации, позволяющие сформировать круг подозреваемых лиц. Свидетельские показания относительно внешности преступника субъективны и не всегда достоверны вследствие стресса, плохого зрения и зрительной памяти, слабой освещенности. В последние годы, благодаря развитию генетики, в криминалистике появился новый термин “genetic witness” – генетический свидетель. Это ДНК преступника, содержащая объективную информацию о нем. Подход, направленный на извлечение этой информации, получил название ДНК-фенотипирования. К настоящему моменту установлены генетические ассоциации для многих фенотипических признаков: пола [2], пигментации (цвет глаз волос и кожи) [3], наличия веснушек [4], склонности к раннему поседению [5] и облысению [6], морфологии волос (прямые/вьющиеся) [7], роста [8], индекса массы тела [9], возраста [10], биогеографического происхождения [11], черт лица [12]. Большинство из этих признаков относится к полигенным и не для всех обнаруженные детерминанты позволяют прогнозировать фенотип с достаточной для следствия достоверностью. В связи с этим мы отобрали только те признаки, для которых ассоциация надежно подтверждена. В последнее десятилетие наиболее исследуемые поисковые признаки связаны с пигментацией: цвет глаз, волос и кожи. Из нескольких сотен опубликованных работ наиболее достоверно этот признак прогнозирует панель HirisPlex-S, предложенная Манфредом Кайзером и Сьюзан Уолш [3], поэтому 41 полиморфизм (SNP)11, входящий в их панель, мы включили в нашу работу.
Группа крови АВ0, несмотря на то, что не является фенотипическим признаком внешности, исторически входит в круг поисковых характеристик, значимых для следствия. Ранее нами опубликован протокол определения группы крови и пола методом генотипирования на биочипах [13], который уже успешно внедрен в экспертную практику [14]. В представленной здесь работе мы использовали те же полиморфизмы, однако существенно улучшили методику их генотипирования.
Половая принадлежность, как один из наиболее важных фенотипических признаков, входит во все наборы для геномной дактилоскопии по локусам коротких тандемных повторов (STR-локусам). В качестве маркера чаще всего используется ген AMEL, распложенный на обеих половых хромосомах, но отличающийся по последовательности на X- и Y-хромосомах. Мы сочли целесообразным также включить его в свою панель.
Последняя группа признаков, внесенная в это исследование ‒ основные, или кóровые (от англ. core), гаплогруппы Y-хромосомы. В связи с неоднородностью распределения гаплогрупп внутри разных этносов знание гаплогруппы в образце в ряде случаев может дать информацию о геногеографическом происхождении человека. При формировании перечня гаплогрупп, которые будут включены в исследование, мы опирались на разработанную YCC (Y Chromosome Consortium) филогенетическую структуру Y-хромосомы, состоящую из 20 основных гаплогрупп, обозначаемых латинскими буквами от A до Т [15, 16]. Мы исключили 6 гаплогрупп, так как определение четырех из них не будет давать дополнительной информации (А – предковая для всех современных мужчин, F – предковая для гаплогрупп G‒T, K – для L‒T, P – для Q и R), а S и M встречаются только на островах Юго-Восточной Азии и Океании и не актуальны для Евразийского континента. В итоге, в панель вошло 14 из 20 основных гаплогрупп, образующих, согласно классификации YCC, самостоятельные стволы эволюционного древа Y-хромосомы: B, C, D, E, G, H, I, J, L, N, O, Q, R и T.
Как уже было сказано, большинство фенотипических признаков, формирующих поисковую информацию, являются полигенными. Это означает, что для их определения с приемлемой для практического применения точностью требуется генотипирование большого числа полиморфизмов. В современном молекулярно-биологическом арсенале можно найти достаточно методов мультиплексного генотипирования: это и секвенирование нового поколения (NGS), и микрочипы высокой плотности (например, Identitas [17]), и другие подходы. Однако все они успешно работают только на высоких концентрациях ДНК хорошей сохранности [18]. В экспертной практике типичный образец ДНК ‒ это 10‒100 пкг/мкл, причем нередко в деградированном состоянии, поэтому, при разработке метода генотипирования на первом месте находится его применимость для таких сложных образцов.
Перспектива внедрения любого метода в широкую практику зависит от его сложности, поэтому еще одна важная задача разработчика ‒ максимальное упрощение протокола постановки и снижение требований к квалификации эксперта. Возвращаясь к панели HIrisPlex-S, следует отметить, что предлагаемый ее авторами способ генотипирования основан на технологии SNaPshot с регистрацией результатов капиллярным электрофорезом. Несмотря на то, что это удобно, так как необходимое оборудование уже имеется во многих современных экспертных генетических лабораториях, недостаток кроется в громоздком протоколе исследования образца. В него включено семь стадий: две мультиплексные ПЦР на 17 и 24 SNP, очистка ПЦР-продукта на колонках, две мультиплексные реакции однонуклеотидной пролонгации (SBE), обработка щелочной фосфатазой, проведение капиллярного электрофореза. Некоторые из этих стадий предполагают открытие пробирки с образцом, что создает риск контаминации. Такой метод может быть освоен в крупных центрах для резонансных преступлений, но для повседневных экспертиз в рядовых региональных лабораториях он явно не предназначен. Резюмируя, можно сказать, что перед нами стояла задача создания простого в постановке, чувствительного метода с высокой мультиплексностью реакции и максимальной невосприимчивостью к деградации исследуемой ДНК.
Ранее нами описаны отдельные методы получения фенотипической информации о группе крови и половой принадлежности [13] и основных гаплогруппах Y-хромосомы [19]. Их недостатком была малая информативность и использование гнездной ПЦР, усложняющей постановку и создающей риск контаминации. Теперь мы предлагаем метод, лишенный вышеуказанных недостатков, созданный с учетом сложности исследуемых образцов ДНК и позволяющий одновременно генотипировать панель из 60 полиморфизмов, которую мы назвали Phenotype Expert и включили в нее маркеры пигментации (41 SNP), основные гаплогруппы Y-хромосомы (14 SNP), пол (AMELX/Y) и группу крови АВ0 (4 SNP). Метод имеет максимально упрощенный протокол проведения и представляет собой одностадийную 52-плексную ПЦР с последующей гибридизацией на биологических микрочипах. Для оценки распределения аллелей в популяции мы провели генотипирование 482 образцов ДНК, полученных от славян, рожденных в городах преимущественно Центральной России и проживающих в Москве, Московской области и Санкт-Петербурге.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Образцы ДНК. В работе использованы образцы ДНК 482 взрослых доноров (254 мужчин и 228 женщин). Для повышения однородности выборки в исследование были включены только доноры, идентифицирующие себя русскими или славянами. Образцы биологического материала были собраны в Москве и Санкт-Петербурге. По месту рождения доноры распределились следующим образом: Центральный федеральный округ ‒ 304 (из них 187 ‒ Москва, 65 ‒ Московская область), Северо-Западный федеральный округ ‒ 75 (из них 59 ‒ Санкт-Петербург и Ленинградская область), Приволжский федеральный округ ‒ 28, Южный и Северо-Кавказский федеральные округа ‒ 21, Уральский, Сибирский и Дальневосточный федеральные округа ‒ 20, Украина ‒ 8, Казахстан ‒ 7, Азербайджан ‒ 4, Беларусь ‒ 3, Узбекистан ‒ 3, Киргизия ‒ 2, Туркменистан ‒ 1, Польша ‒ 1, Германия ‒ 1, Литва ‒ 1, Чехия ‒ 1, не указано ‒ 1.
Выбор полиморфных локусов. Перечень и ряд характеристик полиморфизмов, включенных в исследование, приведены в табл. 1. Слева указана группа фенотипических признаков, к которой относятся соответствующие SNP. Также в таблице приведены координаты SNP в геноме, ген, в котором он расположен (либо гаплогруппа Y-хромосомы, к которой он относится), аллельные варианты и длины ПЦР-продуктов, содержащих исследуемые SNP. Полиморфизмы №№ 1‒41 объединены в общую группу “Пигментация радужной оболочки, волос и кожи”, так как часть из них влияет перекрестно на несколько признаков (более подробно см. в работе [3]). Для определения группы крови использованы четыре позиции в гене AB0: 261 (C>del), 297 (A>G), 657 (G>A) и 681 (C>T) [20], ‒ для определения пола ‒ различающиеся фрагменты гена AMEL на X- и Y-хромосомах [13]. При выборе SNP, ассоциированных с гаплогруппами Y-хромосомы, мы руководствовались данными ISOGG (Y-DNA Haplogroup Tree 2019‒2020, ver. 15.73). Для гаплогрупп, имеющих несколько синонимичных биаллельных маркеров, были выбраны наиболее подходящие с точки зрения конструирования зондов и подбора праймеров и обеспечивающие наиболее короткие ампликоны.
Таблица 1.
Полиморфизмы, входящие в панель Phenotype Expert
| Признак | № | SNP | Хромосома, позиция | Ген, гаплогруппа | Аллелиa | Длина продукта, п.н. |
|---|---|---|---|---|---|---|
| Пигментация радужной оболочки, волос и кожи | 1 | rs312262906 | 16:89919343 | MC1R | A/Del | 76 |
| 2 | rs11547464 | 16:89919683 | MC1R | G/A | 130b | |
| 3 | rs885479 | 16:89919746 | MC1R | G/Aa | 130b | |
| 4 | rs1805008 | 16:89919736 | MC1R | C/T | 130b | |
| 5 | rs1805005 | 16:89919436 | MC1R | G/T | 58 | |
| 6 | rs1805006 | 16:89919510 | MC1R | C/A | 92c | |
| 7 | rs1805007 | 16:89919709 | MC1R | C/T | 130b | |
| 8 | rs1805009 | 16:89920138 | MC1R | G/C | 103 | |
| 9 | rs201326893 | 16:89919714 | MC1R | C/A | 130b | |
| 10 | rs2228479 | 16:89919532 | MC1R | G/A | 92c | |
| 11 | rs1110400 | 16:89919722 | MC1R | T/C | 130b | |
| 12 | rs28777 | 5:33958854 | SLC45A2 | А/С | 77 | |
| 13 | rs16891982 | 5:33951588 | SLC45A2 | G/C | 57 | |
| 14 | rs12821256 | 12:88934558 | KITLG | T/Ca | 98 | |
| 15 | rs4959270 | 6:457748 | LOC105374875 | C/A | 85 | |
| 16 | rs12203592 | 6:396321 | IRF4 | C/T | 90 | |
| 17 | rs1042602 | 11:89178528 | TYR | C/Aa | 73 | |
| 18 | rs1800407 | 15:27985172 | OCA2 | C/Ta | 79 | |
| 19 | rs2402130 | 14:92334859 | SLC24A4 | A/G | 66 | |
| 20 | rs12913832 | 15:28120472 | HERC2 | G/Aa | 59 | |
| 21 | rs2378249 | 20:34630286 | PIGU | A/Ga | 60 | |
| 22 | rs12896399 | 14:92307319 | LOC105370627 | G/T | 70 | |
| 23 | rs1393350 | 11:89277878 | TYR | G/Aa | 85 | |
| 24 | rs683 | 9:12709305 | TYRP1 | A/Ca | 97 | |
| 25 | rs3114908 | 16:89317317 | ANKRD11 | C/T | 49 | |
| 26 | rs1800414 | 15:27951891 | OCA2 | T/C | 53 | |
| 27 | rs10756819 | 9:16858086 | BNC2 | A/G | 87 | |
| 28 | rs2238289 | 15:28208069 | HERC2 | A/Ga | 105 | |
| 29 | rs17128291 | 14:92416482 | SLC24A4 | A/Ga | 78 | |
| 30 | rs6497292 | 15:28251049 | HERC2 | A/Ga | 58 | |
| 31 | rs1129038 | 15:28111713 | HERC2 | T/Ca | 74 | |
| 32 | rs1667394 | 15:28285036 | HERC2 | T/C | 65 | |
| 33 | rs1126809 | 11:89284793 | TYR | G/A | 76 | |
| 34 | rs1470608 | 15:28042975 | OCA2 | C/Ta | 90 | |
| 35 | rs1426654 | 15:48134287 | SLC24A5 | A/G | 70 | |
| 36 | rs6119471 | 20:34197406 | ASIP | C/G | 76 | |
| 37 | rs1545397 | 15:27942626 | OCA2 | A/T | 138 | |
| 38 | rs6059655 | 20:34077942 | RALY | G/Aa | 61 | |
| 39 | rs12441727 | 15:28026629 | OCA2 | G/A | 63 | |
| 40 | rs3212355 | 16:89917970 | MC1R | C/Ta | 63 | |
| 41 | rs8051733 | 16:89957798 | DEF8 | A/Ga | 68 | |
| Группа крови | 42 | rs8176719 | 9:133257521 | AB0 (6 ex) | G/Del | 130 |
| 43 | rs8176720 | 9:133257486 | AB0 (6 ex) | A/G | ||
| 44 | rs8176742 | 9:133256050 | AB0 (7 ex) | G/A | 83 | |
| 45 | rs8176741 | 9:133256074 | AB0 (7 ex) | C/T | ||
| Пол | 46 | – | Y:68687721 X:11298267 |
AMELX/Y | – | 66/70 |
| Гаплогруппы Y-хромосомы | 47 | rs2032623 | Y:19716186 | B (M60) | Del/T | 74 |
| 48 | rs35284970 | Y:2866813 | C (M130) | C/T | 83 | |
| 49 | rs202084622 | Y:13170094 | D (CTS3946) | G/A | 71 | |
| 50 | rs9786534 | Y:7184761 | E (M5388) | A/T | 86 | |
| 51 | rs2740980 | Y:12312201 | G (P257) | G/A | 68 | |
| 52 | rs576940616 | Y:15364331 | H (M2920) | G/T | 84 | |
| 53 | rs2319818 | Y:14242828 | I (U179) | G/A | 117 | |
| 54 | rs13447352 | Y:20587967 | J (M304) | A/C | 73 | |
| 55 | rs2032607 | Y:12792926 | L (M185) | C/T | 85 | |
| 56 | rs9341278 | Y:13357844 | N (M231) | G/A | 86 | |
| 57 | rs2032678 | Y:13396820 | O (M175) | In/Del | 83 | |
| 58 | rs563604826 | Y:13022263 | Q (M1105) | G/A | 69 | |
| 59 | rs17307398 | Y:15174113 | R (P224) | C/T | 65 | |
| 60 | rs9341308 | Y:20576889 | T (M272) | A/G | 60 |
a Так обозначены аллели, номенклатура которых в оригинальной панели HIrisPlex-S, соответствует противоположной цепи ДНК. b Эти шесть позиций амплифицируются с общей пары праймеров и находятся внутри одного ПЦР-продукта длиной 130 п.н. c Эти две позиции амплифицируются с общей пары праймеров и находятся внутри одного ПЦР-продукта длиной 92 п.н.
Мультиплекс. На стадии подбора ПЦР-праймеров основной задачей была минимизация размера ампликона. В табл. 1 приведены значения для каждого локуса. Средняя длина ампликонов составила 79 п.н. В ходе экспериментальной оценки работоспособности праймеров и подбора условий ПЦР использовали несколько образцов свежевыделенной ДНК в концентрации 5, 0.5 и 0.05 нг/мкл каждый. После достижения стабильной работы мультиплексной ПЦР исследовали 482 образца ДНК коллекции. В ходе генотипирования мы сталкивались с эпизодическим выпадением ряда локусов. Для обеспечения их более стабильной амплификации концентрации соответствующих пар праймеров в смеси корректировали. Повторно образцы с неполным профилем не генотипировали, поэтому объем данных для отдельных полиморфизмов может быть меньше 482. Последовательности праймеров доступны по запросу.
Таблица 2.
Частоты аллелей и генетическое разнообразие полиморфизмов панели Phenotype Expert в российской популяции (по результатам исследования) и в пяти основных популяциях (по данным 1000 Genome Project)
| № | Ген, полиморфизм | Генотип и минорный аллель | Частоты и генетическое разнообразие в российской популяцииa | AFRb | AMRc | EASd | EURe | SASf | |||
|---|---|---|---|---|---|---|---|---|---|---|---|
| NO | NE | FO | χ2 | FO в других популяциях | |||||||
| 1 | MC1R rs312262906 Del>A |
del del | 462 | 462.001 | 0.998 | 0.001 | 1 | 0.097 | 1 | 0.994 | 1 |
| del A | 1 | 0.999 | 0.002 | 0 | 0.003 | 0 | 0.006 | 0 | |||
| AA | 0 | 0.001 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| A | 0.001 | 0 | 0.001 | 0 | 0.003 | 0 | |||||
| 2 | MC1R rs11547464 G>A |
GG | 423 | 423.058 | 0.977 | 0.059 | 0.998 | 0.988 | 1 | 0.982 | 1 |
| GA | 10 | 9.885 | 0.023 | 0.002 | 0.012 | 0 | 0.018 | 0 | |||
| AA | 0 | 0.058 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| A | 0.012 | 0.001 | 0.006 | 0 | 0.009 | 0 | |||||
| 3 | MC1R rs885479 C>T |
CC | 345 | 344.669 | 0.845 | 0.049 | 0.988 | 0.516 | 0.125 | 0.871 | 0.926 |
| CT | 60 | 60.662 | 0.149 | 0.011 | 0.337 | 0.518 | 0.119 | 0.067 | |||
| TT | 3 | 2.669 | 0.007 | 0.002 | 0.147 | 0.357 | 0.01 | 0.006 | |||
| T | 0.081 | 0.007 | 0.316 | 0.616 | 0.07 | 0.04 | |||||
| 4 | MC1R rs1805008 C>T |
CC | 392 | 390.271 | 0.839 | 1.089 | 0.992 | 0.994 | 1 | 0.881 | 0.992 |
| CT | 68 | 71.458 | 0.154 | 0.008 | 0.006 | 0 | 0.115 | 0.008 | |||
| TT | 5 | 3.271 | 0.007 | 0 | 0 | 0 | 0.004 | 0 | |||
| T | 0.084 | 0.004 | 0.003 | 0 | 0.062 | 0.004 | |||||
| 5 | MC1R rs1805005 G>T |
GG | 383 | 382.916 | 0.872 | 0.004 | 0.989 | 0.856 | 1 | 0.793 | 0.986 |
| GT | 54 | 54.169 | 0.123 | 0.011 | 0.144 | 0 | 0.189 | 0.014 | |||
| TT | 2 | 1.916 | 0.004 | 0 | 0 | 0 | 0.018 | 0 | |||
| T | 0.066 | 0.005 | 0.072 | 0 | 0.112 | 0.007 | |||||
| 6 | MC1R rs1805006 C>A |
CC | 481 | 481.000 | 1.000 | – | 0.998 | 0.997 | 1 | 0.98 | 0.998 |
| CA | 0 | 0.000 | 0.000 | 0.002 | 0.003 | 0 | 0.02 | 0.002 | |||
| AA | 0 | 0.000 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| A | 0.000 | 0.001 | 0.001 | 0 | 0.01 | 0.001 | |||||
| 7 | MC1R rs1805007 C>T |
CC | 393 | 391.569 | 0.928 | 3.875 | 0.994 | 0.968 | 0.998 | 0.865 | 0.99 |
| CT | 27 | 29.861 | 0.071 | 0.006 | 0.032 | 0.002 | 0.127 | 0.01 | |||
| TT | 2 | 0.569 | 0.001 | 0 | 0 | 0 | 0.008 | 0 | |||
| T | 0.037 | 0.003 | 0.016 | 0.001 | 0.072 | 0.005 | |||||
| 8 | TUBB3 rs1805009 G>C |
GG | 463 | 463.001 | 0.998 | 0.001 | 0.997 | 0.98 | 1 | 0.984 | 1 |
| GC | 1 | 0.999 | 0.002 | 0.003 | 0.02 | 0 | 0.016 | 0 | |||
| CC | 0 | 0.001 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| C | 0.001 | 0.002 | 0.1 | 0 | 0.008 | 0 | |||||
| 9 | MC1R rs201326893 C>A |
CC | 482 | 482.000 | 1.000 | – | 1 | 0.997 | 1 | 1 | 1 |
| CA | 0 | 0.000 | 0.000 | 0 | 0.003 | 0 | 0 | 0 | |||
| AA | 0 | 0.000 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| A | 0.000 | 0 | 0.005 | 0 | 0 | 0 | |||||
| 10 | MC1R rs2228479 G>A |
GG | 396 | 397.771 | 0.830 | 1.001 | 0.992 | 0.954 | 0.498 | 0.869 | 0.963 |
| GA | 81 | 77.458 | 0.162 | 0.008 | 0.046 | 0.427 | 0.125 | 0.037 | |||
| AA | 2 | 3.771 | 0.008 | 0 | 0 | 0.075 | 0.006 | 0 | |||
| A | 0.089 | 0.004 | 0.023 | 0.289 | 0.069 | 0.018 | |||||
| 11 | MC1R rs1110400 T>C |
TT | 429 | 429.069 | 0.975 | 0.071 | 1 | 0.983 | 1 | 0.984 | 0.996 |
| TC | 11 | 10.863 | 0.025 | 0 | 0.017 | 0 | 0.016 | 0.004 | |||
| CC | 0 | 0.069 | 0.000 | 0 | 0 | 0 | 0 | 0 | |||
| C | 0.013 | 0 | 0.009 | 0 | 0.008 | 0.002 | |||||
| 12 | SLC45A2 rs28777 А>С |
AA | 429 | 429.512 | 0.891 | 0.195 | 0.047 | 0.305 | 0.012 | 0.917 | 0.051 |
| AC | 52 | 50.975 | 0.106 | 0.297 | 0.375 | 0.21 | 0.08 | 0.344 | |||
| CC | 1 | 1.512 | 0.003 | 0.657 | 0.32 | 0.778 | 0.004 | 0.605 | |||
| C | 0.056 | 0.805 | 0.507 | 0.883 | 0.044 | 0.777 | |||||
| 13 | SLC45A2 rs16891982 G>C |
GG | 411 | 410.919 | 0.876 | 0.004 | 0.005 | 0.282 | 0 | 0.885 | 0.006 |
| GC | 56 | 56.162 | 0.120 | 0.062 | 0.363 | 0.012 | 0.107 | 0.106 | |||
| CC | 2 | 1.919 | 0.004 | 0.933 | 0.354 | 0.988 | 0.008 | 0.888 | |||
| C | 0.064 | 0.964 | 0.536 | 0.994 | 0.062 | 0.941 | |||||
| 14 | KITLG rs12821256 A>G |
AA | 370 | 367.560 | 0.810 | 1.613 | 0.995 | 0.948 | 1 | 0.771 | 0.967 |
| AG | 77 | 81.880 | 0.180 | 0.005 | 0.052 | 0 | 0.215 | 0.033 | |||
| GG | 7 | 4.560 | 0.010 | 0 | 0 | 0 | 0.014 | 0 | |||
| G | 0.100 | 0.002 | 0.026 | 0 | 0.121 | 0.016 | |||||
| 15 | LOC105374875 rs4959270 C>A |
CC | 150 | 152.576 | 0.330 | 0.240 | 0.648 | 0.277 | 0.433 | 0.282 | 0.515 |
| CA | 231 | 225.847 | 0.489 | 0.303 | 0.507 | 0.454 | 0.483 | 0.405 | |||
| AA | 81 | 83.576 | 0.181 | 0.05 | 0.216 | 0.113 | 0.235 | 0.08 | |||
| A | 0.425 | 0.201 | 0.47 | 0.34 | 0.476 | 0.282 | |||||
| 16 | IRF4 rs12203592 C>T |
CC | 403 | 405.094 | 0.846 | 1.676 | 0.985 | 0.862 | 1 | 0.785 | 0.988 |
| CT | 75 | 70.811 | 0.148 | 0.014 | 0.133 | 0 | 0.197 | 0.012 | |||
| TT | 1 | 3.094 | 0.006 | 0.002 | 0.006 | 0 | 0.018 | 0 | |||
| T | 0.080 | 0.008 | 0.072 | 0 | 0.116 | 0.006 | |||||
| 17 | TYR rs1042602 G>T |
GG | 262 | 259.167 | 0.697 | 1.133 | 0.976 | 0.597 | 0.998 | 0.417 | 0.877 |
| GT | 97 | 102.665 | 0.276 | 0.024 | 0.331 | 0.002 | 0.421 | 0.119 | |||
| TT | 13 | 10.167 | 0.027 | 0 | 0.072 | 0 | 0.161 | 0.004 | |||
| T | 0.165 | 0.012 | 0.238 | 0.001 | 0.372 | 0.063 | |||||
| 18 | OCA2 rs1800407 G>A |
GG | 426 | 424.423 | 0.894 | 1.955 | 0.997 | 0.937 | 0.998 | 0.859 | 0.947 |
| GA | 46 | 49.154 | 0.103 | 0.003 | 0.063 | 0.002 | 0.131 | 0.053 | |||
| AA | 3 | 1.423 | 0.003 | 0 | 0 | 0 | 0.01 | 0 | |||
| A | 0.055 | 0.002 | 0.032 | 0.001 | 0.076 | 0.027 | |||||
| 19 | SLC24A4 rs2402130 A>G |
AA | 340 | 343.539 | 0.714 | 1.520 | 0.179 | 0.648 | 0.881 | 0.626 | 0.748 |
| AG | 133 | 125.922 | 0.262 | 0.443 | 0.288 | 0.117 | 0.322 | 0.227 | |||
| GG | 8 | 11.539 | 0.024 | 0.378 | 0.063 | 0.002 | 0.052 | 0.025 | |||
| G | 0.155 | 0.6 | 0.207 | 0.061 | 0.213 | 0.138 | |||||
| 20 | HERC2 rs12913832 C>T |
CC | 290 | 281.955 | 0.589 | 4.237 | 0.003 | 0.058 | 0 | 0.451 | 0.004 |
| CT | 155 | 171.091 | 0.357 | 0.05 | 0.288 | 0.004 | 0.37 | 0.133 | |||
| TT | 34 | 25.955 | 0.054 | 0.947 | 0.654 | 0.996 | 0.179 | 0.863 | |||
| T | 0.233 | 0.972 | 0.798 | 0.998 | 0.364 | 0.929 | |||||
| 21 | PIGU rs2378249 T>C |
TT | 340 | 341.388 | 0.736 | 0.279 | 0.714 | 0.89 | 0.677 | 0.777 | 0.378 |
| TC | 116 | 113.224 | 0.244 | 0.259 | 0.098 | 0.292 | 0.211 | 0.479 | |||
| CC | 8 | 9.388 | 0.020 | 0.027 | 0.012 | 0.032 | 0.012 | 0.143 | |||
| C | 0.142 | 0.157 | 0.061 | 0.178 | 0.117 | 0.382 | |||||
| 22 | LOC105370627 rs12896399 G>T |
GG | 172 | 174.619 | 0.368 | 0.253 | 0.958 | 0.559 | 0.44 | 0.342 | 0.454 |
| GT | 232 | 226.762 | 0.477 | 0.042 | 0.378 | 0.425 | 0.455 | 0.438 | |||
| TT | 71 | 73.619 | 0.155 | 0 | 0.063 | 0.135 | 0.203 | 0.108 | |||
| T | 0.394 | 0.021 | 0.252 | 0.347 | 0.43 | 0.327 | |||||
| 23 | TYR rs1393350 C>T |
CC | 293 | 293.921 | 0.611 | 0.061 | 0.853 | 0.767 | 0.994 | 0.559 | 0.894 |
| CT | 166 | 164.158 | 0.341 | 0.135 | 0.222 | 0.006 | 0.396 | 0.104 | |||
| TT | 22 | 22.921 | 0.048 | 0.012 | 0.012 | 0 | 0.046 | 0.002 | |||
| T | 0.218 | 0.008 | 0.122 | 0.003 | 0.244 | 0.054 | |||||
| 24 | TYRP1 rs683 T>G |
GG | 172 | 166.160 | 0.345 | 1.204 | 0.787 | 0.378 | 0.978 | 0.159 | 0.642 |
| TG | 222 | 233.680 | 0.485 | 0.203 | 0.476 | 0.022 | 0.427 | 0.297 | |||
| TT | 88 | 82.160 | 0.170 | 0.011 | 0.147 | 0 | 0.414 | 0.061 | |||
| T | 0.413 | 0.112 | 0.385 | 0.011 | 0.627 | 0.21 | |||||
| 25 | ANKRD11 rs3114908 C>T |
CC | 201 | 205.632 | 0.462 | 1.018 | 0.439 | 0.438 | 0.552 | 0.473 | 0.468 |
| CT | 203 | 193.736 | 0.435 | 0.46 | 0.435 | 0.397 | 0.431 | 0.413 | |||
| TT | 41 | 45.632 | 0.103 | 0.101 | 0.127 | 0.052 | 0.095 | 0.119 | |||
| T | 0.320 | 0.331 | 0.344 | 0.25 | 0.311 | 0.325 | |||||
| 26 | OCA2 rs1800414 T>C |
TT | 451 | 451.027 | 0.985 | 0.027 | 0.998 | 1 | 0.149 | 1 | 0.992 |
| TC | 7 | 6.947 | 0.015 | 0.002 | 0 | 0.51 | 0 | 0.008 | |||
| CC | 0 | 0.027 | 0.000 | 0 | 0 | 0.341 | 0 | 0 | |||
| C | 0.008 | 0.001 | 0 | 0.596 | 0 | 0.004 | |||||
| 27 | BNC2 rs10756819 A>G |
GG | 130 | 134.907 | 0.280 | 0.805 | 0.847 | 0.184 | 0.192 | 0.099 | 0.202 |
| AG | 250 | 240.187 | 0.498 | 0.144 | 0.501 | 0.502 | 0.449 | 0.495 | |||
| AA | 102 | 106.907 | 0.222 | 0.009 | 0.314 | 0.306 | 0.451 | 0.303 | |||
| A | 0.471 | 0.081 | 0.565 | 0.557 | 0.676 | 0.55 | |||||
| 28 | HERC2 rs2238289 T>C |
TT | 362 | 358.685 | 0.749 | 1.689 | 0.101 | 0.401 | 0.089 | 0.672 | 0.337 |
| TC | 105 | 111.629 | 0.233 | 0.393 | 0.47 | 0.438 | 0.258 | 0.487 | |||
| CC | 12 | 8.685 | 0.018 | 0.505 | 0.13 | 0.472 | 0.07 | 0.176 | |||
| C | 0.135 | 0.702 | 0.365 | 0.691 | 0.199 | 0.419 | |||||
| 29 | SLC24A4 rs17128291 T>C |
TT | 362 | 363.252 | 0.755 | 0.251 | 0.982 | 0.795 | 0.825 | 0.708 | 0.83 |
| TC | 112 | 109.497 | 0.228 | 0.018 | 0.19 | 0.169 | 0.268 | 0.16 | |||
| CC | 7 | 8.252 | 0.017 | 0 | 0.014 | 0.006 | 0.024 | 0.01 | |||
| C | 0.131 | 0.009 | 0.11 | 0.09 | 0.158 | 0.09 | |||||
| 30 | HERC2 rs6497292 T>C |
TT | 427 | 424.805 | 0.881 | 3.026 | 0.145 | 0.755 | 0.379 | 0.835 | 0.607 |
| TC | 51 | 55.389 | 0.115 | 0.433 | 0.228 | 0.466 | 0.149 | 0.325 | |||
| CC | 4 | 1.805 | 0.004 | 0.422 | 0.017 | 0.155 | 0.016 | 0.067 | |||
| C | 0.061 | 0.638 | 0.131 | 0.388 | 0.09 | 0.23 | |||||
| 31 | HERC2 rs1129038 A>G |
AA | 289 | 279.706 | 0.589 | 5.707 | 0.003 | 0.058 | 0 | 0.447 | 0.004 |
| AG | 151 | 169.588 | 0.357 | 0.05 | 0.291 | 0.002 | 0.376 | 0.131 | |||
| GG | 35 | 25.706 | 0.054 | 0.947 | 0.651 | 0.998 | 0.177 | 0.865 | |||
| G | 0.233 | 0.972 | 0.797 | 0.999 | 0.365 | 0.93 | |||||
| 32 | HERC2 rs1667394 T>C |
TT | 336 | 329.028 | 0.696 | 5.364 | 0.011 | 0.32 | 0.071 | 0.6 | 0.143 |
| TC | 117 | 130.944 | 0.277 | 0.153 | 0.49 | 0.399 | 0.312 | 0.45 | |||
| CC | 20 | 13.028 | 0.028 | 0.837 | 0.19 | 0.53 | 0.087 | 0.407 | |||
| C | 0.166 | 0.913 | 0.435 | 0.729 | 0.244 | 0.632 | |||||
| 33 | TYR rs1126809 G>A |
GG | 281 | 282.722 | 0.590 | 0.195 | 0.985 | 0.761 | 0.998 | 0.545 | 0.896 |
| GA | 174 | 170.555 | 0.356 | 0.012 | 0.228 | 0.002 | 0.406 | 0.1 | |||
| AA | 24 | 25.722 | 0.054 | 0.003 | 0.012 | 0 | 0.05 | 0.004 | |||
| A | 0.232 | 0.009 | 0.125 | 0.001 | 0.252 | 0.054 | |||||
| 34 | OCA2 rs1470608 C>A |
CC | 331 | 327.669 | 0.681 | 1.110 | 0.006 | 0.441 | 0.024 | 0.692 | 0.358 |
| CA | 132 | 138.661 | 0.288 | 0.091 | 0.467 | 0.187 | 0.278 | 0.46 | |||
| AA | 18 | 14.669 | 0.030 | 0.903 | 0.092 | 0.79 | 0.03 | 0.182 | |||
| A | 0.175 | 0.949 | 0.326 | 0.883 | 0.169 | 0.412 | |||||
| 35 | SLC24A5 rs1426654 A>G |
AA | 480 | 479.002 | 0.996 | 481.0 | 0.008 | 0.401 | 0 | 0.994 | 0.507 |
| AG | 0 | 1.996 | 0.004 | 0.133 | 0.378 | 0.024 | 0.006 | 0.356 | |||
| GG | 1 | 0.002 | 0.000 | 0.859 | 0.222 | 0.976 | 0 | 0.137 | |||
| G | 0.002 | 0.926 | 0.411 | 0.988 | 0.003 | 0.315 | |||||
| 36 | ASIP rs6119471 G>C |
CC | 480 | 480.001 | 0.998 | 0.001 | 0.097 | 0.873 | 1 | 0.998 | 1 |
| GC | 1 | 0.999 | 0.002 | 0.434 | 0.115 | 0 | 0.002 | 0 | |||
| GG | 0 | 0.001 | 0.000 | 0.469 | 0.012 | 0 | 0 | 0 | |||
| G | 0.001 | 0.686 | 0.069 | 0 | 0.001 | 0 | |||||
| 37 | OCA2 rs1545397 A>T |
AA | 381 | 375.901 | 0.790 | 5.578 | 0.971 | 0.53 | 0.01 | 0.851 | 0.783 |
| AT | 84 | 94.197 | 0.198 | 0.027 | 0.343 | 0.214 | 0.143 | 0.198 | |||
| TT | 11 | 5.901 | 0.012 | 0.002 | 0.127 | 0.776 | 0.006 | 0.018 | |||
| T | 0.111 | 0.015 | 0.298 | 0.883 | 0.078 | 0.118 | |||||
| 38 | RALY rs6059655 C>T |
CC | 458 | 458.299 | 0.951 | 0.314 | 0.994 | 0.986 | 1 | 0.92 | 0.975 |
| CT | 24 | 23.402 | 0.049 | 0.006 | 0.014 | 0 | 0.076 | 0.025 | |||
| TT | 0 | 0.299 | 0.001 | 0 | 0 | 0 | 0.004 | 0 | |||
| T | 0.025 | 0.003 | 0.007 | 0 | 0.042 | 0.012 | |||||
| 39 | OCA2 rs12441727 G>A |
GG | 353 | 353.780 | 0.786 | 0.134 | 0.985 | 0.617 | 0.601 | 0.767 | 0.654 |
| GA | 92 | 90.440 | 0.201 | 0.014 | 0.346 | 0.353 | 0.213 | 0.307 | |||
| AA | 5 | 5.780 | 0.013 | 0.002 | 0.037 | 0.046 | 0.02 | 0.039 | |||
| A | 0.113 | 0.008 | 0.21 | 0.222 | 0.126 | 0.192 | |||||
| 40 | MC1R rs3212355 G>A |
GG | 469 | 469.000 | 1.000 | – | 0.811 | 0.98 | 1 | 1 | 1 |
| GA | 0 | 0.000 | 0.000 | 0.179 | 0.02 | 0 | 0 | 0 | |||
| AA | 0 | 0.000 | 0.000 | 0.011 | 0 | 0 | 0 | 0 | |||
| A | 0.000 | 0.1 | 0.01 | 0 | 0 | 0 | |||||
| 41 | DEF8 rs8051733 T>C |
TT | 183 | 185.440 | 0.497 | 0.369 | 0.431 | 0.683 | 0.518 | 0.527 | 0.564 |
| TC | 160 | 155.121 | 0.416 | 0.44 | 0.288 | 0.409 | 0.396 | 0.366 | |||
| CC | 30 | 32.440 | 0.087 | 0.129 | 0.029 | 0.073 | 0.078 | 0.07 | |||
| C | 0.295 | 0.349 | 0.173 | 0.278 | 0.275 | 0.253 | |||||
| 42 | AB0 (6 exon) rs8176719 C>del |
CC | 129 | 133.163 | 0.280 | 0.588 | 0.098 | 0.075 | 0.145 | 0.167 | 0.143 |
| C del | 245 | 236.675 | 0.498 | 0.392 | 0.314 | 0.47 | 0.455 | 0.515 | |||
| del del | 101 | 105.163 | 0.221 | 0.51 | 0.611 | 0.385 | 0.378 | 0.342 | |||
| Del | 0.471 | 0.706 | 0.768 | 0.62 | 0.605 | 0.599 | |||||
| 43 | AB0 (6 exon) rs8176720 T>C |
TT | 183 | 180.451 | 0.377 | 0.241 | 0.263 | 0.248 | 0.264 | 0.453 | 0.313 |
| TC | 222 | 227.098 | 0.474 | 0.495 | 0.516 | 0.506 | 0.419 | 0.446 | |||
| CC | 74 | 71.451 | 0.149 | 0.242 | 0.236 | 0.23 | 0.127 | 0.241 | |||
| C | 0.386 | 0.489 | 0.494 | 0.483 | 0.337 | 0.464 | |||||
| 44 | AB0 (7 exon) rs8176742 T>C |
TT | 310 | 315.758 | 0.661 | 2.995 | 0.015 | 0.184 | 0.073 | 0.054 | 0.057 |
| TC | 157 | 145.484 | 0.304 | 0.242 | 0.473 | 0.431 | 0.332 | 0.323 | |||
| CC | 11 | 16.758 | 0.035 | 0.743 | 0.343 | 0.496 | 0.614 | 0.62 | |||
| C | 0.187 | 0.864 | 0.579 | 0.711 | 0.78 | 0.781 | |||||
| 45 | AB0 (7 exon) rs8176741 A>G |
GG | 326 | 327.814 | 0.680 | 0.327 | 0.705 | 0.908 | 0.647 | 0.843 | 0.597 |
| AG | 143 | 139.372 | 0.289 | 0.251 | 0.089 | 0.317 | 0.145 | 0.339 | |||
| AA | 13 | 14.814 | 0.031 | 0.044 | 0.003 | 0.036 | 0.012 | 0.063 | |||
| A | 0.175 | 0.169 | 0.048 | 0.194 | 0.084 | 0.233 | |||||
| 46 | B (M60) rs2032623 del>T | del | 254 | 1 | – | – | 0.98 | 0.998 | 1 | 1 | 1 |
| T | 0 | 0.000 | – | 0.02 | 0.002 | 0 | 0 | 0 | |||
| 47 | C (M130) rs35284970 C>T | C | 252 | 0.992 | – | – | 1 | 1 | 0.951 | 1 | 0.931 |
| T | 2 | 0.008 | – | 0 | 0 | 0.049 | 0 | 0.069 | |||
| 48 | D (CTS3946) rs202084622 G>A | G | 254 | 1.000 | – | – | 1 | 1 | 0.918 | 1 | 1 |
| A | 0 | 0.000 | – | 0 | 0 | 0.082 | 0 | 0 | |||
| 49 | E (M5388) rs9786534 A>T | A | 244 | 0.961 | – | – | 0.103 | 0.824 | 1 | 0.975 | 1 |
| T | 10 | 0.039 | – | 0.897 | 0.176 | 0 | 0.025 | 0 | |||
| 50 | G (P257) rs2740980 G>A | G | 246 | 0.969 | – | – | 0.995 | 0.948 | 1 | 0.967 | 0.973 |
| A | 8 | 0.031 | – | 0.005 | 0.052 | 0 | 0.033 | 0.027 | |||
| 51 | H (M2920) rs576940616 G>T | G | 254 | 1.000 | – | – | 1 | 0.999 | 1 | 0.999 | 0.836 |
| T | 0 | 0.000 | – | 0 | 0.001 | 0 | 0.001 | 0.164 | |||
| 52 | I (U179) rs2319818 G>A | G | 206 | 0.811 | – | – | 0.972 | 0.965 | 1 | 0.862 | 1 |
| A | 48 | 0.189 | – | 0.028 | 0.035 | 0 | 0.138 | 0 | |||
| 53 | J (M304) rs13447352 A>C | A | 240 | 0.945 | – | – | 1 | 0.918 | 1 | 0.887 | 0.865 |
| C | 14 | 0.055 | – | 0 | 0.082 | 0 | 0.113 | 0.135 | |||
| 54 | L (M185) rs2032607 C>T | C | 254 | 1.000 | – | – | 1 | 1 | 1 | 1 | 0.896 |
| T | 0 | 0.000 | – | 0 | 0 | 0 | 0 | 0.104 | |||
| 55 | N (M231) rs9341278 G>A | G | 223 | 0.878 | – | – | 1 | 1 | 0.967 | 0.904 | 0.996 |
| A | 31 | 0.122 | – | 0 | 0 | 0.033 | 0.096 | 0.004 | |||
| 56 | O (M175) rs2032678 in>del | In | 254 | 1.000 | – | – | 1 | 1 | 0.184 | 1 | 0.985 |
| del | 0 | 0.000 | – | 0 | 0 | 0.816 | 0 | 0.015 | |||
| 57 | Q (M1105) rs563604826 G>A | G | 252 | 0.992 | – | – | 0.995 | 0.895 | 0.995 | 0.989 | 0.975 |
| A | 2 | 0.008 | – | 0.005 | 0.105 | 0.005 | 0.011 | 0.025 | |||
| 58 | R (P224) rs17307398 C>T | C | 118 | 0.465 | – | – | 0.962 | 0.565 | 0.996 | 0.417 | 0.612 |
| T | 136 | 0.535 | – | 0.038 | 0.435 | 0.004 | 0.583 | 0.388 | |||
| 59 | T (M272) rs9341308 A>G | A | 251 | 0.988 | – | – | 1 | 0.976 | 1 | 0.983 | 1 |
| G | 3 | 0.012 | – | 0 | 0.024 | 0 | 0.017 | 0 | |||
a NО – наблюдаемое число генотипов, NE – ожидаемое число генотипов, Fо – ожидаемая частота аллеля или генотипа. b AFR (African) – африканская популяция; c AMR (Ad Mixed American) – смешанная американская популяция; d EAS (East Asian) – восточноазиатская популяция; e EUR (European) – европейская популяция; f SAS (South Asian) ‒ южноазиатская популяция.
Схема биочипа. Биочип наносили по схеме, приведенной на рис. 1а. В него входит четыре блока, соответствующих группе определяемых признаков: пигментация, группа крови АВ0, пол и гаплогруппа Y-хромосомы. Для всех полиморфизмов было подобрано по паре олигонуклеотидов, комплементарных соответствующим аллельным вариантам. ПЦР-праймеры и ДНК-зонды произведены компанией “Lumiprobe” (Россия). Изготовление гидрогелевых биочипов проводили по оригинальной технологии Института молекулярной биологии им. В.А. Энгельгардта Российской академии наук (ИМБ РАН), в беспылевых помещениях, с использованием пинового робота Qarray2 (“Genetix”, Великобритания), согласно ранее описанной методике [19].
Рис. 1.
Биологический микрочип Phenotype Expert и примеры его использования для генотипирования образцов ДНК человека. Схема расположения ДНК-зондов на биочипе Phenotype Expert (а). Цифры над каждой парой ячеек, соответствуют номеру полиморфизма из табл. 1. Обозначения внутри ячеек соответствуют аллелям полиморфизмов. Три ячейки “М” необходимы для контроля работы портативного анализатора, содержат флуорофор Cy5 и позволяют корректно позиционировать матрицу для обсчета сигналов ячеек. В нижней части рисунка приведены примеры флуоресцентного изображения биочипа, полученные в результате генотипирования женского (б) и трех мужских (в‒д) образцов ДНК.

Протокол генотипирования. Для сокращения протокола постановки мы применили модификацию полимеразной цепной реакции, впервые описанную под названием LATE-PCR (linear-after-the-exponential PCR) [21], или ЛПЭ-ПЦР, ‒ линейная после экспоненциальной ПЦР. Все обратные праймеры по 5'-концу содержат универсальную вставку, с которой при понижении температуры отжига на линейной стадии ЛПЭ-ПЦР связывается флуоресцентно меченный универсальный праймер. На этом этапе происходит асимметричная амплификация с накоплением меченой, преимущественно одноцепочечной ДНК, необходимой для последующей гибридизации. Данный вариант ЛПЭ-ПЦР упрощен в сравнении с ранее опубликованным [22] за счет исключения универсальной вставки в прямом праймере. Ход экспериментального подбора условий амплификации мы не приводим в данной работе, ниже указана уже оптимизированная программа и состав реакционной смеси.
ПЦР проводили на амплификаторе SpeedCycler (“Analytik Jena”, Германия) в объеме 25 мкл реакционной смеси: ПЦР-буфер с полимеразой HotTaq Multi, 4 е.а. (“Асфоген”, Россия), 5 мМ MgSO4, 0.2 мМ каждого из dNTP (“Сибэнзим”, Россия), смесь праймеров; 200 пкмоль универсального праймера Cy5-TCATTGGATCTCATTA, 0.1‒500 нг геномной ДНК. Полимеразу активировали 2 мин при 95°С и на первом этапе проводили 50 циклов амплификации (94°С – 30 с, 65°С – 40 с, 72°С – 30 с), затем на втором этапе ‒ 50 циклов амплификации (94°С – 20 с, 52°С – 30 с, 72°С – 30 с).
Гибридизация. Гибридизационную камеру биочипа заполняли смесью следующего состава: 25% формамид, 5× SSPE, 50 об% ПЦР-продукта. После инкубации (10 ч, 37°С) и отмывки (10 мин в SSPE при комнатной температуре) биочипы промывали дистиллированной водой, высушивали сжатым воздухом, помещали в портативный анализатор (ООО “БИОЧИП-ИМБ”, Россия) и регистрировали с экспозицией 0.5‒2 с флуоресценцию в формате SPE. На рис. 1 приведены примеры таких флуоресцентных изображений для женского (рис. 1б) и мужских (рис. 1 в‒д) образцов ДНК. Анализ изображения проводили с использованием программ ImaGel 2.0 и ImaGel Studio (ИМБ РАН). Результатом автоматической обработки каждого биочипа был текстовый файл с генотипом образца. Равновесие Харди‒Вайнберга рассчитывали общеизвестным способом.
Чувствительность. Для оценки чувствительности метода провели генотипирование трех образцов ДНК, каждый из которых был взят в следующих концентрациях: 500, 250, 100, 50, 25 и 10 пг на реакцию.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
При составлении перечня определяемых поисковых признаков мы исходили из их практической значимости для следственной работы и потенциальной возможности установления этих признаков по генотипу. В результате нами были сформированы четыре кластера: пигментация, группа крови АВ0, пол и гаплогруппа Y-хромосомы.
Пигментация
Информация о пигментации глаз, волос и кожи, безусловно, представляет поисковую ценность. Сотни научных работ, посвященных обнаружению и верификации генетических детерминант этих признаков, вышли в свет за последние два десятилетия. Мы остановили свой выбор на панели HIrisPlex-S, так как на данный момент она наиболее точно прогнозирует фенотип пигментации. Для этой панели авторы не только опубликовали математическую модель прогнозирования фенотипа, но и оформили ее в виде удобного онлайн-ресурса (https://hirisplex.erasmusmc.nl) на сайте Департамента генетической идентификации Erasmus MC (Нидерланды). Интерфейс ресурса позволяет внести результаты генотипирования образца и получить вероятностный результат по трем признакам пигментации. Для цвета глаз предлагается три категории: серо-голубой, карий и смешанный (зеленые, болотные). Цвет волос оценивается в категориях светлые/темные, а также в четырех градациях: блондин, шатен, брюнет, рыжий. Цвет кожи подразделяется на пять оттенков: очень бледная, светлая, смуглая, темная, черная. В ряде исследований показана высокая степень достоверности прогнозирования фенотипа по данной модели не только в западноевропейских и североамериканских [23], но и в более актуальных для нас восточноевропейских популяциях [24].
Из опубликованных исследований народов, населяющих Россию, помимо вышеуказанной работы на белорусской популяции [24], еще одна была выполнена на выборке из 144 образцов 14 народностей (в том числе 8 образцов русских) по 29 маркерам из 41 [25] и другая – на выборке из 286 образцов 38 народностей (в том числе 50 образцов русских) по 24 маркерам [26], хотя данные по частотам аллелей в этих работах не представлены.
Здесь мы приводим результаты генотипирования 482 образцов (табл. 2). Как видно из правой части табл. 2, аллельные частоты ряда полиморфизмов проявляют геногеографическую специфичность. Для более наглядного представления результатов мы сопоставили на графике (рис. 2) частоту минорного аллеля каждого полиморфизма, полученную нами в ходе исследования (RU), с частотами в трех наиболее близких к ней макропопуляций: европейской (EUR), восточноазиатской (EAS) и южноазиатской (SAS). Из рис. 2 видно, что частоты аллелей для четверти полиморфизмов существенно отличаются для европейской (EUR) и азиатских (EAS и SAS) популяций. Принимая во внимание этот факт, а также то, что российская популяция занимает промежуточное положение между европейскими и азиатскими народами, для получения осмысленных данных необходимо было уделить особое внимание составлению однородной выборки. Первичные результаты генотипирования, которые мы здесь не приводим, были получены для 555 жителей Москвы и Санкт-Петербурга без учета их этнической принадлежности. При их обработке мы получили существенное отклонение от равновесия Харди‒Вайнберга для полиморфизмов, имеющих геногеографическую этноспецифичность. После исключения из выборки всех доноров с неславянской самоидентификацией нам удалось получить данные, характерные для более однородной популяции.
Рис. 2.
График частот минорных аллелей 41 полиморфизма, относящегося к панели пигментации. Полиморфизмы расположены по мере возрастания частоты минорного аллеля в исследованной нами выборке (RU). Также для каждого полиморфизма приведены частоты в европейской (EUR), восточноазиатской (EAS) и южноазиатской (SAS) популяциях, согласно данным 1000 Genome Project.
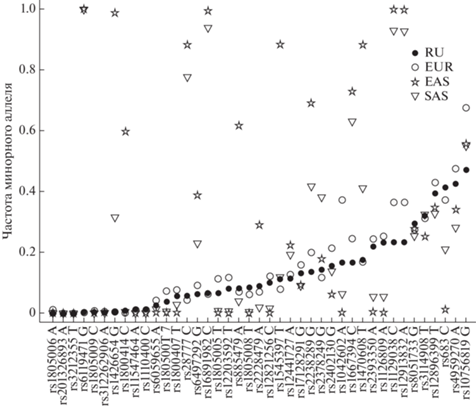
Тем не менее сочетание таких факторов, как различающееся для ряда полиморфизмов аллельное распределение в EUR- и EAS- популяциях, условность понятия “этническая принадлежность”, а также метисация лиц, самоопределяющих себя славянами или русскими, усложняет формирование однородной выборки в исторически гетерогенных популяциях типа российской. Мы полагаем, что повышенные значения χ2 для некоторых полиморфизмов обусловлены этой неоднородностью.
Кроме того, идентифицировать себя славянами или русскими могут лица любого этногеографического происхождения. Ярким подтверждением этого служит следующий эпизод: полиморфизм rs1426654 (№ 35 в табл. 2) имеет полярное распределение частот аллеля G в популяциях EUR (0.003) и EAS (0.988). В нашем исследовании распределение частот его аллелей значительно отклоняется от равновесия Харди‒Вайнберга: АА ‒ 480, AG ‒ 0, GG ‒ 1, χ2 = 481. Очевидной причиной этого стал единственный образец с генотипом G/G, наиболее характерным для восточноазиатских популяций. И действительно, на фотографии донора этого образца (часть коллекции охарактеризована фотографически) мы обнаружили девушку с типичными восточноазиатскими чертами лица; при этом она идентифицирует себя русской и имеет славянские фамилию, имя и отчество. Нельзя исключить, что она приемный ребенок в русской семье. Этот случай подчеркивает неравнозначность происхождения и этнической самоидентификации. Поисковая ценность в такой ситуации зависит от того, какие методы использует следствие: при работе с паспортными данными такой индивид был бы пропущен, а при поиске по базе фотографий подозреваемых или при поквартирном обходе типичная внешность привлекла бы внимание.
К настоящему времени опубликованы десятки работ по определению биогеографического происхождения по аутосомным маркерам. Методы, описанные в этих работах, позволяют не только выявить принадлежность к макропопуляции (европейская, азиатская, африканская), но и установить степень метисации индивида [27‒29]. Мы полагаем возможным и целесообразным в дальнейшем расширить панель Phenotype Expert за счет актуальных для Евразии аутосомных маркеров биогеографического происхождения, которые дополнят получаемую поисковую информацию.
Группа крови АВ0
Серологическое определение группы крови АВ0 очень часто дает ошибочные результаты на экспертных образцах, что связано с их бактериальным загрязнением и деградацией агглютиногенов. Кроме того, не все биологические объекты пригодны для серологического анализа. Гораздо более достоверно группу крови определяют генотипированием полиморфизмов в 6 и 7 экзонах гена АВ0 [30], причем объектом исследования может быть любой материал, содержащий геномную ДНК.
В табл. 3 приведены сочетания аллелей по четырем полиморфизмам в гене AB0, образующих (благодаря выделению трех вариантов нулевого аллеля: О1, О1v и О2) 15 генетических групп и 4 серологические. Это позволяет различать шесть I(0) групп, четыре II(А), четыре III(В) и одну IV(АВ). В результате генотипирования определены частоты аллелей, генетических и серологических групп в исследуемой выборке (табл. 4), что не только дает возможность делать вероятностные расчеты при вынесении экспертного заключения, но и пополняет знания в области популяционной генетики.
Таблица 3.
Аллельспецифичные полиморфные позиции в экзонах 6 и 7 гена AB0, используемые для генотипирования в тест-системе Phenotype Expert
| Позиция SNP, аллельa |
Экзон 6 | Экзон 7 | ||
|---|---|---|---|---|
| 261 | 297 | 681 | 657 | |
| A | G | A | G | C |
| B | G | T | ||
| O1 | del | |||
| O1v | del | G | A | |
| O2 | G | |||
Таблица 4.
Частоты аллелей, генотипов и групп крови AB0 по результатам генотипирования коллекции образцов славянской популяции (N = 482) в тест-системе Phenotype Expert
| Группа | Аллель | Генотип | Частота, (N)a | Частота группы крови, (N)a |
|---|---|---|---|---|
| A | 0.263 (254) | |||
| B | 0.174 (168) | |||
| O1 | 0.348 (335) | |||
| O1v | 0.193 (186) | |||
| O2 | 0.022 (21) | |||
| I(0) | O1/O1 | 0.112 (54) | 0.307 (148) | |
| O1/O1v | 0.149 (72) | |||
| O1v/O1v | 0.290 (14) | |||
| O2/O1 | 0.010 (5) | |||
| O2/O1v | 0.004 (2) | |||
| O2/O2 | 0.002 (1) | |||
| II(A) | A/A | 0.075 (36) | 0.371 (179) | |
| A/O1 | 0.191 (92) | |||
| A/O1v | 0.093 (45) | |||
| A/O2 | 0.012 (6) | |||
| III(B) | B/O1 | 0.120 (58) | 0.241 (116) | |
| B/O1v | 0.081 (39) | |||
| B/O2 | 0.012 (6) | |||
| B/B | 0.027 (13) | |||
| IV(AB) | A/B | 0.081 (39) | 0.81 (39) |
Определение гаплогруппы Y-хромосомы
Значимость генетической информации, расположенной на Y-хромосоме, для практической криминалистики подтверждается не только множеством научных публикаций, но и наличием широкого спектра коммерческих наборов, таких, как Yfiler® Plus PCR Amplification Kit (“Applied Biosystems”, США), PowerPlex® Y23 System (“Promega”, США), AmpFLSTR® Yfiler™ PCR Amplification Kit (“Applied Biosystems”), COrDIS Ystr (ООО “Гордиз”, Россия) и других. Эти наборы, в первую очередь, предназначены для идентификации личности по аллелям STR-локусов, расположенных на Y-хромосоме, однако они применяются и для определения гаплогруппы непрямым методом ‒ с помощью программ-предикторов.
Для определения гаплогруппы Y-хромосомы STR- и SNP-маркеры имеют свои преимущества и недостатки. С точки зрения однозначности установления гаплогруппы более достоверными считаются SNP, так как имеют частоту спонтанных мутаций существенно ниже (2.5 × 10–8 [31]), чем STR (10–2–10–3 [32, 33]). С одной стороны, высокая частота мутаций в STR-локусах служит причиной гомоплазии, которая, в свою очередь, может приводить к ошибкам определения гаплогрупп с помощью предикторов. С другой стороны, при верной предикции STR-гаплотип обладает большей информативностью, так как для получения столь же детальной информации по SNP-маркерам потребуется анализ существенно большего числа полиморфизмов. С позиции криминалистической практики предпочтение находится на стороне SNP-маркеров, так как небольшая протяженность амплифицируемого для их определения фрагмента обеспечивает преимущества при исследовании деградированной ДНК в сравнении с более длинными фрагментами при анализе STR-маркеров.
Данные о распределении гаплогрупп в различных этносах позволяют выполнить вероятностную оценку этногеографического происхождения носителя ДНК. Особая ценность информации о гаплогруппе Y-хромосомы связана с традицией передачи фамилии по мужской линии. Можно сказать, что гаплогруппа и фамилия ‒ это два сцепленных между собой признака с разным способом наследования: генетическим и культурным соответственно. Заметим, что даже при значительном нивелировании в смешанных браках характерных антропологических признаков предка по мужской линии его фамилия будет передана искомому носителю ДНК. Учитывая наличие этнической специфичности фамилий, мы можем говорить о возможности использования информации о гаплогруппе в образце с места преступления для ранжирования подозреваемых по их фамилиям. Фокусировка дальнейшей экспертизы на наиболее вероятных подозреваемых позволит сократить сроки и затраты на расследование.
Ярким примером, демонстрирующим поисковые возможности этого подхода, может послужить наш опыт ‒ когда при генотипировании коллекции был обнаружен единственный образец с гаплогруппой О, практически не встречающейся у европейцев, но распространенной в странах Восточной Азии. Этот образец принадлежал донору с характерной корейской фамилией Югай. Несмотря на то, что такие контрастные примеры на практике встречаются нечасто, они наглядно демонстрируют возможности метода. Для расширенного внедрения этого поискового подхода требуется его глубокая проработка в тесном сотрудничестве с ономатологами.
Как отмечено в вводной части, нами проанализированы все базовые гаплогруппы, представляющие самостоятельные стволы древа Y-хромосомы, за исключением M и S, которые практически не встречаются за пределами островов Юго-Восточной Азии и Океании. Тем не менее предлагаемый метод позволяет косвенно установить наличие M- или S-гаплогрупп в образце: в этом крайне маловероятном случае по всем 14 Y-маркерам будет наблюдаться гибридизация только верхнего ряда ячеек, содержащих ДНК-зонды к аллелям предкового типа.
Более полно раскрыть потенциал Y-хромосомы как источника геногеографической информации позволит дальнейшая детализация каждой из гаплогрупп до нескольких десятков субкладов (субгаплогрупп). Существующий научно-технический уровень не позволяет создать метод для одновременного генотипирования сотен маркеров, который при этом характеризуется высокой чувствительностью и практической простотой. В этой ситуации оптимальным подходом может быть двухэтапное исследование, в котором поиск на начальном этапе сужается до одной базовой гаплогруппы, а на втором расширяется до исследования ее субкладов. Для развития этого направления ведется работа по созданию отдельных биочипов, идентифицирующих субклады каждой из базовых гаплогрупп.
Оценка метода генотипирования
Как сказано выше, к методам генотипирования в криминалистике предъявляются повышенные требования, связанные с низким качеством и другими особенностями исследуемого материала. Наиболее полно они изложены в “Руководстве по валидации методов анализа ДНК” Международной рабочей группы судебных экспертов в SWGDAM (Scientific Working Group on DNA Analysis Methods; https:// www.swgdam.org/_files/ugd/4344b0_813b241e8944497e99b9c45b163b76bd.pdf). Мы учитывали требования SWGDAM в ходе создания инструмента генотипирования, однако его экспериментальная валидация по всему перечню SWGDAM ‒ это объемное исследование, составляющее предмет отдельной публикации.
Для увеличения шансов успешного генотипирования фрагментированной ДНК при дизайне праймеров стояла задача получения максимально коротких ампликонов. В рамках метода ПЦР с последующей гибридизацией минимальная длина фрагмента, который можно получить, зависит от нуклеотидного состава локуса и в идеальном случае (при соотношении AT/GC ≈ 1) составляет около 50 п.н. Для большинства SNP были подобраны индивидуальные праймеры, однако в генах MC1R и AB0 часть анализируемых полиморфизмов оказалась расположена достаточно компактно и их целесообразно было амплифицировать в пределах одного ПЦР-продукта: №№ 2, 3, 4, 7, 9, 11 ‒ 130 п.н.; №№ 6, 10 ‒ 92 п.н.; №№ 42, 43 ‒ 130 п.н. и №№ 44, 45 ‒ 83 п.н. (табл. 1). Это увеличило среднюю длину ампликонов, однако снизило мультиплексность ПЦР с 60 до 52. В итоге нам удалось добиться средней длины ПЦР-продукта 79 п.н., что не только существенно меньше, чем в ближайшем аналоге (133 п.н. [3]), но и приближается к пределу возможностей метода гибридизационного анализа.
Для понимания границ применимости метода в криминалистической практике требуется оценка его чувствительности. Снижение концентрации ДНК в анализируемом образце повышает вероятность выпадения одного из аллелей либо всего локуса. Выпадение одного из аллелей при гомозиготном генотипе не визуализируется на гибридизационной картине и не влияет на результат интерпретации. В случае же гетерозиготного генотипа выпадение аллеля создает ложную гомозиготность и может повлиять на прогнозируемый фенотип.
Мы провели предварительную оценку чувствительности разработанного метода на трех образцах, содержание ДНК которых составляло 10, 25, 50, 100, 250 и 500 пг в реакционной смеси. Для образцов 50‒500 пг на реакцию был получен полноценный профиль по всем локусам; при 25 пг выпадения аллелей наблюдали в двух локусах одного из образцов; при 10 пг в двух образцах было потеряно по одному аллелю и в третьем образце выпали аллели в пяти локусах. Учитывая, что 10 пг ДНК соответствует менее чем двум полным геномам человека, можно предположить, что выпадение связано с физическим отсутствием в реакции хромосомы или ее фрагмента, содержащего второй аллель некорректно прогенотипированных полиморфизмов.
Характерный уровень чувствительности большинства мультиплексных наборов для криминалистики составляет 50‒100 пг геномной ДНК на ПЦР, а для наиболее близкого к разработанному нами набору для генотипирования ‒ панели HIrisPlex-S методом SNaPShot [34] ‒ она составляет 63 пкг для каждой из двух мультиплексных реакций, то есть для получения полного профиля по 41 маркеру потребуется 125 пкг ДНК, что и следует считать истинной чувствительностью этого метода. Полученные предварительные результаты позволяют нам удовлетворительно оценить инструмент Phenotype Expert по параметру чувствительности.
Подводя итог, можно сказать, что представленная в работе панель маркеров для ДНК-фенотипирования превосходит имеющиеся аналоги по объему получаемой поисковой информации, а разработанный метод ориентирован на исследование деградированных объектов и позволяет получать весь заявленный объем генетических данных из образцов с низкой концентрацией ДНК, характерной для экспертной практики. Предельно простой протокол генотипирования позволяет обеспечить успешное внедрение метода в широкую практику, а полученные новые данные о распределении частот аллелей используемых полиморфизмов впервые установлены для российской популяции и использованы для валидации метода.
Написание настоящей статьи не потребовало специального финансирования.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики. От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Jeffreys A.J., Wilson V., Thein S.L. (1985) Hypervariable 'minisatellite' regions in human DNA. Nature. 314(6006), 67–73.
Mannucci A., Sullivan K.M., Ivanov P.L., Gill P. (1994) Forensic application of a rapid and quantitative DNA sex test by amplification of the X-Y homologous gene amelogenin. Int. J. Legal Med. 106(4), 190‒193.
Chaitanya L., Breslin K., Zuñiga S., Wirken L., Pośpiech E., Kukla-Bartoszek M., Sijen T., Knijff P., Liu F, Branicki W., Kayser M., Walsh S. (2018) The HIrisPlex-S system for eye, hair and skin colour prediction from DNA: introduction and forensic developmental validation. Forensic Sci. Int. Genet. 35, 123‒135.
Kukla-Bartoszek M., Pośpiech E., Woźniak A., Boroń M., Karłowska-Pik J., Teisseyre P., Zubańska M., Bronikowska A., Grzybowski T., Płoski R., Spólnicka M., Branicki W. (2019) DNA-based predictive models for the presence of freckles. Forensic Sci. Int. Genet. 42, 252‒259
Pośpiech E., Kukla-Bartoszek M., Karłowska-Pik J., Zieliński P., Woźniak A., Boroń M., Dąbrowski M., Zubańska M., Jarosz A., Grzybowski T., Płoski R., Spólnicka M., Branicki W. (2020) Exploring the possibility of predicting human head hair greying from DNA using whole-exome and targeted NGS data. BMC Genomics. 21(1), 538.
Marcińska M., Pośpiech E., Abidi S., Andersen J. D., van den Berge M., Carracedo Á., Eduardoff M., Marczakiewicz-Lustig A., Morling N., Sijen T., Skowron M., Söchtig J., Syndercombe-Court D., Weiler N., EUROFORGEN-NoE Consortium, Schneider P.M., Ballard D., Børsting C., Parson W., Phillips C., Branicki W. (2015) Evaluation of DNA variants associated with androgenetic alopecia and their potential to predict male pattern baldness. PLoS One. 10(5), e0127852.
Pośpiech E., Karłowska-Pik J., Marcińska M., Abidi S., Andersen J.D., Berge M., Carracedo Á., Eduardoff M., Freire-Aradas A., Morling N., Sijen T., Skowron M., Söchtig J., Syndercombe-Court D., Weiler N., Schneider P. M., Ballard D., Børsting C., Parson W., Phillips C., EUROFORGEN-NoE Consortium (2015) Evaluation of the predictive capacity of DNA variants associated with straight hair in Europeans. Forensic Sci. Int. Genet. 19, 280‒288.
Aulchenko Y.S., Struchalin M.V., Belonogova N.M., Axenovich T.I., Weedon M.N., Hofman A., Uitterlinden A.G., Kayser M., Oostra B.A., van Duijn C.M., Janssens A.C., Borodin P.M. (2009) Predicting human height by Victorian and genomic methods. Eur. J. Hum. Genet. 17(8), 1070–1075.
Cho S., Lee E. H., Kim H., Lee J.M., So M.H., Ahn J.J., Lee H.Y. (2021) Validation of BMI genetic risk score and DNA methylation in a Korean population. Int. J. Legal Med. 135(4), 1201–1212.
Vidaki A., Ballard D., Aliferi A., Miller T.H., Barron L.P., Syndercombe Court D. (2017) DNA methylation-based forensic age prediction using artificial neural networks and next generation sequencing. Forensic Sci. Int. Genet. 28, 225–236.
Frudakis T.N. (2010) Molecular Photofitting: Predicting Ancestry and Phenotype Using DNA. Elsevier.
Claes P., Hill H., Shriver M.D. (2014) Toward DNA-based facial composites: preliminary results and validation. Forensic Sci. Int. Genet. 13, 208‒216.
Фесенко Д.О., Митяева О.Н., Наседкина Т.В., Рубцов П.М., Лысов Ю.П., Заседателев А.С. (2010) Генотипирование биологического материала по локусам HLA-DQA1, AB0, AMEL с помощью биочипов. Молекуляр. биология, 44(3), 456‒462.
Фесенко Д.О., Иванов П.Л., Корниенко А.Е., Земскова Е.Ю., Заседателев А.С., Наседкина Т.В. (2011) Оптимизация биологического микрочипа для генотипирования локуса АВ0: аналитические аспекты. Судебно-медицинская экспертиза. 54, 30‒33.
Y Chromosome Consortium. (2002) A nomenclature system for the tree of human Y-chromosomal binary haplogroups. Genome Res. 12(2), 339‒348.
Karafet T.M., Mendez F.L., Meilerman M.B., Underhill P.A., Zegura S.L., Hammer M.F. (2008). New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree. Genome Res. 18(5), 830–838.
Keating B., Bansal A.T., Walsh S., Millman J., Newman J., Kidd K., Budowle B., Eisenberg A., Donfack J., Gasparini P., Budimlija Z., Henders A.K., Chandrupatla H., Duffy D.L., Gordon S.D., Hysi P., Liu F., Medland S.E., Rubin L., Martin N.G., International Visible Trait Genetics (VisiGen) Consortium. (2013) First all-in-one diagnostic tool for DNA intelligence: genome-wide inference of biogeographic ancestry, appearance, relatedness, and sex with the Identitas v1 Forensic Chip. Int. J. Legal Med. 127(3), 559–572.
Alvarez-Cubero M.J., Saiz M., Martínez-García B., Sayalero S.M., Entrala C., Lorente J.A., Martinez-Gonzalez L.J. (2017) Next generation sequencing: an application in forensic sciences? Ann. Hum. Biol. 44(7), 581–592.
Fesenko D.O., Chudinov A.V., Surzhikov S.A., Zasedatelev A.S. (2014) Biochip for genotyping SNPs defining core Y-chromosome haplogroups in Russian population groups. BioChip J. 8, 171‒178.
Yip S.P. (2000) Single-tube multiplex PCR-SSCP analysis distinguishes 7 common ABO alleles and readily identifies new alleles. Blood. 95(4), 1487–1492.
Sanchez J.A., Pierce K.E., Rice J.E., Wangh L.J. (2004) Linear-After-The-Exponential (LATE)-PCR: an advanced method of asymmetric PCR and its uses in quantitative real-time analysis. Proc. Natl. Acad. USA. 101(7), 1933–1938.
Фесенко Д.О., Корниенко А.Е., Чудинов А.В., Наседкина Т.В. (2011) Получение одноцепочечной ДНК в одноэтапной ПЦР с легкоплавким избыточным праймером для гибридизации на биочипах. Молекуляр. биология. 45(2), 267‒271.
Chen Y., Branicki W., Walsh S., Nothnagel M., Kayser M., Liu F. (2021) The impact of correlations between pigmentation phenotypes and underlying genotypes on genetic prediction of pigmentation traits. Forensic Sci. Int. Genet. 50, 102395.
Середенко (Шинкевич) М.В., Вакула C.И., Шаптуренко М.Н., Кондратюк А.В., Боровко С.Р., Луговнев А.В., Гудиевская И.Г., Скрыпник О.В., Марченко Л.Н., Кильчевский А.В. (2021) Прогностическая способность системы генетического фенотипирования HIrisPlex в белорусской популяции. Экологическая генетика. 19(1), 67–76.
Балановский О.П, Петрушенко В.С., Горин И.О., Кагазежева Ж.А., Маркина Н.В., Кострюкова Е.С., Лейбова Н.А., Маурер А.М., Балановская Е.В. (2019) Точность предикции пигментации волос и глаз по генетическим маркерам для популяций России. Вестник РГМУ. 5, 25‒41.
Balanovska E., Lukianova E., Kagazezheva J., Maurer A., Leybova N., Agdzhoyan A., Gorin I., Petrushenko V., Zhabagin M., Pylev V., Kostryukova E., Balanovsky O. (2020) Optimizing the genetic prediction of the eye and hair color for North Eurasian populations. BMC Genomics. 21(1), 527.
Halder I., Shriver M., Thomas M., Fernandez J. R., Frudakis T. (2008) A panel of ancestry informative markers for estimating individual biogeographical ancestry and admixture from four continents: utility and applications. Hum. Mutat. 29(5), 648–658.
Phillips C., Santos C., Fondevila M., Carracedo Á., Lareu M.V. (2016) Inference of ancestry in forensic analysis I: autosomal ancestry-informative marker sets. Methods Mol. Biol. 1420, 233–253.
Setser C.H., Planz J.V., Barber R.C., Phillips N.R., Chakraborty R., Cross D.S. (2020) Differentiation of Hispanic biogeographic ancestry with 80 ancestry informative markers. Sci. Rep. 10(1), 7745.
Doi Y., Yamamoto Y., Inagaki S., Shigeta Y., Miyaishi S., Ishizu H. (2004) A new method for ABO genotyping using a multiplex single-base primer extension reaction and its application to forensic casework samples. Legal Med. 6(4), 213–223.
Reich D.E., Schaffner S.F., Daly M.J., McVean G., Mullikin J.C., Higgins J.M., Richter D.J., Lander E.S., Altshuler D. (2002) Human genome sequence variation and the influence of gene history, mutation and recombination. Nat. Genet. 32(1), 135–142.
Aşicioglu F., Oguz-Savran F., Ozbek U. (2004) Mutation rate at commonly used forensic STR loci: paternity testing experience. Dis. Markers. 20(6), 313–315.
Hohoff C., Dewa K., Sibbing U., Hoppe K., Forster P., Brinkmann B. (2007) Y-chromosomal microsatellite mutation rates in a population sample from northwestern Germany. Int. J. Legal Med. 121(5), 359–363.
Walsh S., Liu F., Wollstein A., Kovatsi L., Ralf A., Kosiniak-Kamysz A., Branicki W., Kayser M. (2013) The HIrisPlex system for simultaneous prediction of hair and eye colour from DNA. Forensic Sci. Int. Genet. 7(1), 98–115.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


