Молекулярная биология, 2023, T. 57, № 1, стр. 56-70
Структура и эволюция гена AqE у насекомых
Л. В. Пузакова a, *, М. В. Пузаков a
a Институт биологии южных морей им. А.О. Ковалевского Российской академии наук
299011 Севастополь, Россия
* E-mail: kvluda@yandex.ru
Поступила в редакцию 21.04.2022
После доработки 07.07.2022
Принята к публикации 24.07.2022
- EDN: AXMDWN
- DOI: 10.31857/S0026898423010135
Аннотация
Ген AqE, кодирующий сульфолактатдегидрогеназа-подобный фермент семейства LDH2/MDH2-оксидоредуктаз, найден в геномах бактерий и грибов, животных и растений, образ жизни которых связан с водной средой, а также членистоногих, в частности, насекомых, которые ведут преимущественно сухопутный образ жизни. Чтобы проследить эволюционную судьбу гена AqE, мы изучили распространенность этого гена и его структуру в классе насекомых. Обнаружено, что ген AqE представлен не во всех отрядах/подотрядах насекомых, наблюдается потеря гена. В некоторых отрядах он дуплицирован или мультиплицирован. Установлена вариабельность гена как по длине, так и по экзон-интронной структуре ‒ от безинтронной до многоинтронной. Обнаружено, что мультипликация гена AqE насекомых имеет древнюю природу, однако есть и “молодые” дупликации. Не исключено, что в связи с появлением паралогов ген приобрел новую функцию.
ВВЕДЕНИЕ
Семейство LDH2/MDH2-оксидоредуктаз (LDH/MDH-оксидоредуктазы типа 2) включает ферменты, которые участвуют в разнообразных метаболических процессах и на основании филогенетического анализа подразделяются на восемь групп [1‒4]. Охарактеризованы функции ферментов клады ComC (L-сульфолактатдегидрогеназа, SLDH), клады LDH (лактатдегидрогеназа типа 2), клады AllD (уреидогликолатдегидрогеназа), клады YiaK (2,3-дикето-L-гулонатредуктаза) и клады DpkA (Δ1-пиперидин-2-карбоксилат/Δ1-пирролин-2-карбоксилатредуктаза), в то время как функция белков трех других клад: термофильных архей, YbiC и YlbC, остается неизвестной. Поскольку в филогенетический анализ было включено только 20 аминокислотных последовательностей [4], то, возможно, в дальнейшем такая классификация будет пересмотрена.
Функция LDH2/MDH2-оксидоредуктаз на данный момент охарактеризована только у архей и бактерий [3, 4]. Недавно у эукариот выявили гены, которые кодируют белки, гомологичные SLDH архей [5]. У архей и бактерий этот фермент кодируется геном comC [6‒8]. ComC-подобные гены найдены в геномах грибов, растений и животных. Однако в царстве растений гомологи comC обнаружены только у водорослей, тогда как у мхов и семенных растений они отсутствуют. Гомологи гена выявлены у преобладающего числа эволюционных групп животных, за исключением четвероногих (земноводных, рептилий, птиц и млекопитающих) [5]. ComC-подобные гены эукариот условно названы AqE (aquatic enzyme, водный фермент) в связи с его встречаемостью преимущественно в тех таксонах, образ жизни или стадии онтогенеза которых ассоциированы с водной (жидкой) средой. Подробный анализ встречаемости и структуры гена AqE у костистых рыб показал, что в большинстве исследованных отрядов сохраняется идентичная экзон-интронная организация (11 экзонов) и характерные консервативные мотивы этого гена [9]. Однако у карпообразных (Cypriniformes) этот ген утерян полностью, а у представителей лососеобразных (Salmoniformes) претерпел значительные делеции и, по всей видимости, нефункционален [9]. Ранее предполагалось, что SLDH-подобный белок, кодируемый геном AqE, участвует в анаэробном энергообмене и его утрата отдельными таксонами как животных, так и растений связана с сопутствующей выходу на сушу перестройкой метаболизма, обусловленной насыщенностью среды кислородом и отсутствием естественной гипоксии, характерной для гидробионтов [5]. Утрата гена AqE у карпообразных и лососеобразных могла произойти из-за индивидуальной эволюции этих таксонов и соответствующей реструктуризации метаболических путей. Известно, что у предков Cypriniformes и Salmoniformes происходили независимые полногеномные дупликации (так называемая четвертая полногеномная дупликация позвоночных) [10, 11]. Избыток оксидоредуктаз, возникший в результате дупликации генома, мог стать причиной потери значимости гена AqE для метоболизма, так как появился большой “ресурс” для негомологичной замены или появления новых ферментов и новых метаболических путей [9]. Анализ дифференциальной экспрессии AqE желтого горбыля (Larimichthys crocea) показал, что уровень активности этого гена высок в печени, жабрах и коже, тогда как в других тканях он низкий. В связи с этим предположили, что функция SLDH-подобного белка рыб может быть связана с выведением продуктов метаболизма [12].
Среди таксонов, представители которых сохранили ген AqE, большой интерес представляют насекомые, поскольку они являются сухопутными организмами. Насекомые ‒ очень разнообразный класс организмов, обитающих в различных условиях, в том числе в средах, где могут сталкиваться с дефицитом кислорода. Так, например, представители отрядов Trichoptera, Megaloptera, Odonata проходят личиночную стадию развития в водной среде и дышат растворенным в воде кислородом. Также представители некоторых отрядов насекомых развиваются в почве ‒ Auchenorrhyncha, Isoptera, Diplura, Protura, в древесине ‒ Zoraptera, Hymenoptera (например Orussus abietinus) или Polyphaga (например Dendroctonus ponderosae, Anoplophora glabripennis), в разлагающихся органических субстратах ‒ некоторые представители отряда Diptera. Паразитические виды, такие как Hymenoptera, Strepsiptera, живут в тканях животных, используя для дыхания кислород, растворенный в гемолимфе. Есть среди насекомых и экстремофилы, обитающие в холодных условиях, около ледников ‒ Grylloblattodea.
Нами изучено разнообразие, структура и эволюция гена AqE в классе насекомых с таксономическим разрешением до отряда. Присутствие гена AqE в геноме насекомых, в частности комара Aedes albopictus (отряд Diptera), было отмечено ранее [5], однако детально этот ген изучен впервые.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Поиск генов AqE у насекомых. Поиск гомологов гена AqE у насекомых проводили с использованием алгоритма локального выравнивания tBLASTn [13]. В качестве референсной последовательности выбрали аминокислотную последовательность, кодируемую AqE комара A. albopictus. Эта последовательность была идентифицирована при первичном исследовании генов AqE [5]. Проанализированы полногеномные ДНК представителей 17 отрядов. Еще 10 отрядов исследованы с помощью поиска гомологий в базе данных транскриптомов. Использованы геномные и транскриптомные последовательности, размещенные на серверах NCBI. Уникальные идентификаторы полногеномных и транскриптомных сборок представителей класса насекомых, а также таксонов Diplura и Protura приведены в табл. 1.
Таблица 1.
Характеристики вариантов гена AqE у представителей подтипа Hexapoda
| Таксон | Отряд | Представитель/идентификатор геномной или транcкриптомной сборки | Ген | Количество экзонов | Белок, а.о. | Перекрытие с РП, % | Идентичность к РП, % | Дополнительная информация | Транскрипт* | |
|---|---|---|---|---|---|---|---|---|---|---|
| Monocondylia | Archaeognatha | Machilis hrabei/ GAUM00000000 |
AqE1 | ND | 363 | 97 | 59.89 | Есть | ||
| Meinertellus cundinamarcensis/ GAUG00000000 |
AqE1 | ND | 356 | 96 | 56.57 | Есть | ||||
| Polyneoptera | Orthoptera | Xenocatantops brachycerus/ GCA_900249655 |
AqE1 | 1 | 364 | 96 | 61.02 | НО | ||
| Laupala kohalensis/ GCA_002313205 |
AqE1 | 5 | 313 | 94 | 50.74 | ДТ1 | НО | |||
| AqE2 | 6 | 309 | 96 | 42.65 | Делеция С-конца, 10 а.о.ДТ1Стоп-кодон в ОРС | НТOrthoptera ‒ есть | ||||
| AqE3 | 1 | 48 | 12 | 55.56 | Сохранился короткий фрагмент | |||||
| Blattodea | Blattella germanica/ GCA_003018175 |
AqE1 | 1 | 351 | 94 | 33.33 | Есть полиА-хвост и прямые повторы | НО | ||
| AqE2 | 1 | 350 | 96 | 37.7 | Старт-кодон не обнаружен | НО | ||||
| AqE3 | 1 | 363 | 97 | 39 | НО | |||||
| AqE4 | 1 | 328 | 89 | 33.6 | Старт-кодон не обнаружен. Делеция N-конца, 30 а.о. | НО | ||||
| AqE5 | 4 | 336 | 97 | 50.7 | ДТ1 | Есть | ||||
| AqE6 | 4 | 358 | 98 | 62.64 | Старт-кодон не обнаружен | Есть | ||||
| AqE7 | 6 | 343 | 91 | 57.83 | Есть | |||||
| AqE8 | 3 | 150 | 41 | 42.86 | Протяженная делеция N-конца , 158 а.о. Делеция С-конца, 55 а.о.Старт- и стоп-кодоны не обнаружены | Есть | ||||
| AqE9 | 5 | 276 | 82 | 47 | ДТ1Делеция С-конца, 60 а.о | Есть | ||||
| Isoptera | Cryptotermes secundus/ GCA_002891405 |
AqE1 isozyme 1 | 10 | 381 | 96 | 65.34 | Удлинение N-конца на 18 а.о. | НТIsoptera – НО | ||
| AqE1 isozyme 2 | 9 | 354 | 96 | 65.34 | НТIsoptera – НО | |||||
| Zootermopsis nevadensis/ GCA_000696155 |
AqE1 | 9 | 354 | 96 | 64 | НТIsoptera – НО | ||||
| Mantodea | Metallyticus splendidus/GATB00000000 | AqE1 | ND | 360 | 96 | 59.71 | Есть | |||
| Stagmatoptera biocellata/GDBT00000000 | AqE1 | ND | 365 | 96 | 61.36 | Есть | ||||
| Plasmatodea | Нет данных по таксону | НТ | ||||||||
| Mantophasmatodea | Нет данных по таксону | НО | ||||||||
| Grylloblattode | Нет данных по таксону | НО | ||||||||
| Dermaptera | Diplatys sp./GDCS01000000 | AqE1 | ND | 355 | 96 | 58.36 | Есть | |||
| Forficula auricularia/GAYQ00000000 | AqE1 | ND | 355 | 98 | 51.12 | Есть | ||||
| Paraneoptera/Condylognatha | Hemiptera | Heteroptera | Cimex lectularius/ GCA_000648675 |
AqE1 isozyme 1 | 10 | 368 | 96 | 56.82 | Есть | |
| AqE1 isozyme 2 | 9 | 356 | 96 | 56.82 | Есть | |||||
| Sternorrhyncha | НО | НО | ||||||||
| Coleorrhyncha | Нет данных по таксону | НО | ||||||||
| Auchenorrhyncha | Philaenus spumarius/ GCZA01000000 |
AqE1 | ND | 356 | 96 | 66 | Есть | |||
| Homalodisca
vitripennis/ GCA_000696855 |
AqE1 | 8 | 337 | 92 | 64.29 | НТAuchenorrhyncha ‒ есть | ||||
| AqE2 | 6 | 301 | 95 | 47.67 | ДТ1 | НТAuchenorrhyncha ‒ есть | ||||
| Psocoptera | Badonnelia titei/ GDFG01000000 | AqE1 | ND | 361 | 97 | 67.8 | Есть | |||
| Elipsocus kuriliensis/ GDFL01000000 | AqE1 | ND | 358 | 98 | 61.58 | Есть | ||||
| AqE2 | ND | 357 | 97 | 61.13 | Есть | |||||
| Phthiraptera | Pediculus humanus/GCA_000006295 | AqE1 | 6 | 345 | 92 | 64.09 | Старт-кодон не обнаружен | НТPhthiraptera ‒ есть | ||
| Thysanoptera | Frankliniella occidentalis/ GCA_000697945 |
AqE1 | 8 | 344 | 96 | 59.26 | Старт-кодон не обнаружен | Есть | ||
| Holometabola | Trichoptera | Glossosoma conforme/GCA_003347265 | AqE1 | 7 | 360 | 96 | 56.23 | Делеция в ОРС, 16 а.о | НТTrichoptera ‒ есть | |
| Lepidoptera | Spodoptera litura/GCA_002706865 | AqE1 | 8 | 360 | 96 | 56 | Есть | |||
| AqE2 | 8 | 356 | 97 | 56.09 | Есть | |||||
| Diptera | Drosophila busckii/GCA_001277935 | AqE1 | 3 | 437 | 98 | 69.47 | Удлинение N-конца на 80 а.о | НТDiptera ‒ есть | ||
| AqE2 | 3 | 437 | 98 | 69.47 | Удлинение N-конца на 80 а.о | НТDiptera ‒ есть | ||||
| AqE3 | 3 | 437 | 98 | 69.47 | Удлинение N-конца на 80 а.о | НТDiptera ‒ есть | ||||
| AqE4 | 1 | 396 | 99 | 38.89 | Удлинение N-конца на 30 а.о.. | НТDiptera ‒ есть | ||||
| AqE5 | 1 | 474 | 95 | 35.80 | Удлинение N-конца на 112 а.о.Есть полиА-хвост и прямые повторы | НТDiptera ‒ есть | ||||
| Drosophila miranda/GCA_000269505 | AqE1 | 3 | 440 | 98 | 69.92 | Удлинение N-конца на 81 а.о. | Есть | |||
| AqE2 | 1 | 384 | 97 | 40.23 | Удлинение N-конца на 25 а.о. | Есть | ||||
| AqE3 | 1 | 464 | 98 | 38.44 | Удлинение N-конца на 103 а.о.Есть короткий полиА-хвост и прямые повторы | Есть | ||||
| Aedes aegypti/GCA_002204515 | AqE1 | 5 | 486 | 100 | 98.62 | Удлинение N-конца на 123 а.о. | Есть | |||
| Aedes albopictus/GCA_001444175 | AqE1 | 5 | 487 | 100 | 99.72 | Удлинение N-конца на 124 а.о. | Есть | |||
| AqE2 | 4 | 363 | 100 | 100 | Есть | |||||
| AqE3 | 5 | 443 | 100 | 99.72 | Удлинение N-конца на 80 а.о. | Есть | ||||
| Anopheles gambiae/GCA_000005575 | AqE1 | 5 | 501 | 98 | 83.57 | Удлинение N-конца на 139 а.о.. | НО | |||
| AqE2 | 4 | 364 | 98 | 83.57 | НО | |||||
| Siphonaptera | Ctenocephalides felis/GCA_003426905 | AqE1 | 5 | 403 | 97 | 55 | Удлинение N-конца на 37 а.о. | Есть | ||
| AqE2 | 4 | 297 | 68 | 59 | Делеция С-конца 104 а.о. Удлинение N-конца на 37 а.о.a. | Есть | ||||
| AqE3 | 5 | 391 | 97 | 51 | Удлинение N-конца на 39 а.о.Делеция в ОРС ‒ 16 а.о | Есть | ||||
| Mecoptera | Nannochorista philpotti/GADB00000000 | AqE1 | ND | 368 | 97 | 72.47 | Есть | |||
| Boreus hyemalis/GAYK00000000 | AqE1 | ND | 370 | 98 | 68.27 | Есть | ||||
| Megaloptera | Corydalus cornutus/GATG00000000 | AqE1 | ND | 366 | 96 | 62.50 | Есть | |||
| Neuroptera | Osmylus fulvicephalus/GAYC00000000 | AqE1 | ND | 365 | 96 | 61.25 | Есть | |||
| AqE2 | ND | 387 | 96 | 53.85 | Удлинение N-конца на 27 а.о. Старт- и стоп-кодоны не обнаружены | Есть | ||||
| Pseudomallada prasinus/GAVV00000000 | AqE1 | ND | 355 | 95 | 40.57 | Есть | ||||
| Raphidioptera | Xanthostigma xanthostigma/GCSF00000000 | AqE1 | ND | 365 | 98 | 58.99 | Есть | |||
| Inocellia crassicornis/GAZH00000000 | AqE1 | ND | 364 | 98 | 57.58 | Есть | ||||
| Coleoptera | Adephaga | НО | НО | |||||||
| Archostemata | НО | НО | ||||||||
| Polyphaga | Tribolium castaneum/ GCA_000002335 |
AqE1 isozyme 1 | 4 | 358 | 98 | 53.76 | ДТ2 | НТColeoptera ‒ есть | ||
| AqE1 isozyme 2 | 4 | 322 | 85 | 54.19 | ДТ2Делеция С-конца, 53 а.о | НТColeoptera ‒ есть | ||||
| AqE2 | 4 | 352 | 96 | 55.97 | ДТ2 | НТColeoptera ‒ есть | ||||
| AqE4 | 7 | 391 | 97 | 60.97 | Удлинение N-конца на 31 а.о. | НТColeoptera ‒ есть | ||||
| Dendroctonus
ponderosae/ GCA_000355655 |
AqE1 | 8 | 414 | 96 | 56.7 | Удлинение N-конца на 42 а.о. | Есть | |||
| Anoplophora
glabripennis/ GCA_000390285 |
AqE1 | 8 | 412 | 96 | 60.51 | Удлинение N-конца на 47 а.о. | Есть | |||
| AqE2 | 3 | 147 | 43 | 44.55 | Делеция С-конца, 244 а.о. Удлинение N-конца на 26 а.о. | Есть | ||||
| AqE3 | 5 | 264 | 63 | 41.99 | Делеция N-конца, 125 а.о. Удлинение С-конца на 19 а.о. | Есть | ||||
| AqE4 | 8 | 363 | 96 | 54.13 | Есть | |||||
| AqE5 isozyme 1 | 8 | 363 | 96 | 56.53 | Есть | |||||
| AqE5 isozyme 2 | 7 | 312 | 88 | 57.14 | Делеция N-конца, 52 а.о | Есть | ||||
| Leptinotarsa decemlineata/GCA_000500325 | AqE1 | 3 | 166 | 45 | 44.58 | Делеция N-конца, 193 а.о | Есть | |||
| AqE2 | 7 | 409 | 59 | 52.94 | Негомологичный фрагмент на N-конце, 97 а.о.Негомологичный фрагмент на С-конце ‒ 125 а.о | Есть | ||||
| AqE3 | 7 | 302 | 71 | 65.61 | Негомологичный фрагмент на N-конце, 56 а.о. Делеция в ОРС, 64 а.о. | Есть | ||||
| Strepsiptera | Mengenilla moldrzyki | НО | НО | |||||||
| Hymenoptera | Nasonia vitripennis/GCA_000002325 | AqE1 | 2 | 412 | 97 | 54.24 | Удлинение N-конца на 49 а.о. | Есть | ||
| Apis mellifera/GCA_000002195 | AqE1 | 2 | 434 | 97 | 48.59 | Удлинение N-конца на 72 а.о. | Есть | |||
| Cephus cinctus/GCA_000341935 | AqE1 | 2 | 395 | 97 | 53.26 | Удлинение N-конца на 32 а.о. | Есть | |||
| Orussus abietinus/GCA_000612105 | AqE1 | 2 | 403 | 97 | 51.13 | Удлинение N-конца на 32 а.о. | Есть | |||
| Neodiprion lecontei/GCA_001263575 | AqE1 | 2 | 392 | 98 | 53.98 | Негомологичный фрагмент на N-конце, 26 а.о. | Есть | |||
| Polyneoptera | Embioptera | Data on taxon unavailable | НО | |||||||
| Plecoptera | Leuctra sp/GAUF00000000 | AqE1 | ND | 359 | 96 | 64.10 | Есть | |||
| Brachyptera vera/GDBN00000000 | AqE1 | ND | 359 | 97 | 64.02 | Старт-кодон не обнаружен | Есть | |||
| Perla marginata/GATV00000000 | AqE1 | ND | 357 | 96 | 62.39 | Короткий скаффолд – С-конец не обнаружен | Есть | |||
| Zoraptera | Zorotypus caudelli/GAYA00000000 | AqE1 | ND | 382 | 96 | 57.67 | Удлинение С-конца на 20 а.о. | Есть | ||
| AqE2 | ND | 370 | 98 | 42.62 | Есть | |||||
| AqE3 | ND | 380 | 97 | 35.03 | Удлинение N-конца на 19 а.о., С-конца на 12 а.о. | Есть | ||||
| Palaeoptera | Odonata | Calopteryx splendens/GCA_002093875 | AqE1 | 7 | 334 | 97 | 54.81 | ДТ1,Делеция С-конца, 17 а.о. | Есть | |
| AqE2 | 6 | 306 | 94 | 46.65 | Старт-кодон не обнаружен.ДТ1,Делеция С-конца, 19 а.о. | Есть | ||||
| Ephemenoptera | Ephemera danica/GCA_000507165 | AqE1 | 8 | 347 | 94 | 60.82 | Делеция С-конца, 19 а.о. | Есть | ||
| Diplura | Occasjapyx japonicus/GAXJ00000000 | AqE1 | ND | 358 | 96 | 53.28 | Есть | |||
| AqE2 | ND | 357 | 97 | 53.26 | Естьs | |||||
| Protura | Acerentomon sp./GAXE00000000 | AqE1 | ND | 367 | 97 | 51.97 | Есть | |||
* По результатам анализа базы данных транскриптомов. Примечания. РП – референсная последовательность; НД – нет данных (ген найден через анализ транскриптома); НО – не обнаружено; НТ– нет транскриптома; ДТ1 – делеция типа 1: “KRSNHYGIAGWYTLRAMNAGCVGMSMTNTSPLASPTRSKEA”; ДТ2 – делеция типа 2: “PTGHPTTDANLAF”; ОРС – открытая рамка считывания.
Границы экзонов у обнаруженных генов AqE уточняли визуально по наибольшему сходству последовательностей между референсной и исследуемой последовательностями и наличию 5'- и 3'-границы сайта сплайсинга. Кодирующие последовательности AqE, полученные в результате анализа, использовали для поиска транскрибируемых последовательностей РНК. Копиям AqE в случае выявления более одной в геноме, присваивали порядковый номер в соответствии с очередностью обнаружения.
Филогенетический анализ. Множественное выравнивание аминокислотных последовательностей выполняли с помощью MUSCLE [14], а полученные данные использовали для построения филогенетического дерева с использованием программного обеспечения MEGA X [15] и метода максимального правдоподобия (ML). Использовали следующие параметры: бутстреп, 100 повторов; модель замещения аминокислот Ле-Гаскуэль-2008; и гамма-распределение. Анализ включал 91 аминокислотную последовательность AqE подтипа Hexapoda.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Представленность гена AqE в геномах насекомых
В ходе исследования гена AqE мы проанализировали 51 вид насекомых, представляющих 27 отрядов. Полногеномные последовательности ДНК на момент исследования были получены только у представителей 17 анализируемых отрядов. Другие 10 отрядов мы смогли проанализировать, используя базу данных транскриптомов. В качестве референсного белка (шаблона) мы использовали аминокислотную последовательность, кодируемую AqE комара A. albopictus. Этот белок был выбран в качестве референсного, поскольку экзон-интронная организация его гена имеет высокое сходство с классическим AqE [5, 9]. В результате установлена значительная вариабельность присутствия, структуры и количества копий AqE у представителей насекомых (табл. 1). Нами определены три основные тенденции: (1) сохранение единственной копии AqE; (2) полная потеря AqE; и (3) мультипликация AqE, варьирующая у разных видов от 2 до 9 копий.
Группы, которые потеряли весь ген, представлены в меньшинстве (табл. 1). Это отряд Strepsiptera (Holometabola), подотряды Adephaga, Archostemata (Coleoptera, Holometabola) и подотряд Sternorrhyncha (Hemiptera, Condylognatha). В подотряде Sternorrhyncha в 11 просмотренных сборках не выявлено гомологий c AqE ‒ среди них Aphidomorpha, Aleyrodoidea, Psylloidea. У Dactylopius coccus, относящегося к Dactylopiidae, обнаружены два гена AqE, но при дополнительной проверке выяснилось, что оба гена – это фрагменты бактериальной ДНК, вероятно попавшей в пробу. Этот факт подтверждается высоким сходством кодируемых белков D. coccus с белками бактерий: AqE1 D. coccus с AqE Bosea sp. (идентичность 72.93% (CP014301.1)) и AqE2 D. coccus с AqE Ochrobactrum anthropi (идентичность 98.50% (LT671862.1)). При этом наблюдается низкая идентичность к референсному AqE комара A. albopictus: AqE1 (29.29%) и AqE2 (29.64%).
У представителей отрядов Mantophasmatodea, Grylloblattodea, Embioptera и подотряда Coleorrhyncha изучены только последовательности транскриптомов. Не выявлено транскриптов, сходных с транскриптом, кодирующим референсный белок AqE. Однако в этом случае нельзя сделать однозначный вывод о потере гена.
Остальные группы распределились следующим образом ‒ представители 10 отрядов и одного подотряда содержат единственную копию гена, в 15 отрядах и одном подотряде ген мультиплицирован (табл. 1). Разные виды тех отрядов, где AqE мультиплицирован, имеют разное число копий гена. Так, например, у представителя отряда Orthoptera (Polyneoptera) Xenocatantops brachycerus одна копия гена AqE, а у Laupala kohalensis ‒ три. В отряде Diptera A. aegypti имеет одну копию гена, а A. albopictus ‒ три. В отряде Coleoptera (Polyphaga) у Dendroctonus ponderosae одна копия AqE, у Tribolium castaneum и Leptinotarsa decemlineata ‒ три, у Anoplophora glabripennis ‒ пять. Некоторые виды содержат значительное число копий гена AqE, так в геноме Blattella germanica число копий достигает 9.
Анализ транскриптомных баз данных насекомых
Для качественной оценки потенциальной функциональности генов AqE насекомых проведен анализ транскриптомных баз данных NCBI. Транскрипты гена AqE обнаружены в большинстве случаев. У представителей некоторых отрядов транскрипты гена AqE не были найдены. Поэтому предприняли попытки обнаружить транскрипты гена AqE у других представителей этих отрядов и в ряде случаев получили положительный результат. В некоторых случаях транскрипты не обнаружены ни у анализируемых видов, ни в таксоне в целом. Данные для некоторых организмов в базе ТSА отсутствовали (табл. 1).
Рассмотрение структуры гена AqE показывает, что в большинстве случаев этот ген потенциально функционален. В одном случае (L. kohalensis AqE2) открытая рамка считывания (ОРС) содержит стоп-кодон, следовательно, ген нефункционален. В некоторых случаях AqE представлен сильно делетированной копией, либо небольшим фрагментом (L. kohalensis AqE3 (144 п.н./48 а.о.) или B. germanica AqE8 (450 п.н./150 а.о.)). Очевидно, что такие фрагменты не могут быть функциональными. Однако не исключена возможность присутствия транскриптов даже при нефункциональности гена. Так, в базах TSA обнаружены транскрипты An. glabripennis AqE2 протяженностью 441 п.н./47 а.о. или An. glabripennis AqE3 (738 п.н./246 а.о.). Известно еще несколько подобных случаев (табл. 1). Кроме того, отсутствие старт-кодона, выявленное в некоторых случаях, мы связываем с ошибками при поиске (низкая идентичность). Например, у B. germanica AqE6 не обнаружен метионин, однако транскрипты представлены в базе TSA (табл. 1).
Филогенетические взаимоотношения генов AqE насекомых
Чтобы установить взаимосвязь между генами AqE насекомых, проведен филогенетический анализ, основанный на методе максимального правдоподобия, в который была включена 91 аминокислотная последовательность, кодируемая этими генами. Полученное дерево не позволило выявить четкие эволюционные отношения между различными вариантами AqE, потому что статистическая значимость превалирующей доли визуально отличных клад не превышала 70% (рис. 1). Тем не менее, мы разделили дерево по топологии на восемь условных кластеров и установили некоторые закономерности.
Рис. 1.
Эволюционные взаимоотношения генов AqE насекомых. Филогенетический анализ выполнен в программе MEGA X c помощью метода максимального правдоподобия. Приведены только бутстреп-значения, превышающие 50%. Справа от дендрограммы указаны отряды, к которым принадлежат соответствующие организмы.

Так, например, в каждом из кластеров I–IV и VI–VIII сгруппированы гены организмов, не встречающиеся ни в одном из остальных шести кластеров. (кроме B. germanica). При этом одна группа (кластер V) отличается визуально более длинными эволюционными расстояниями и содержит гены AqE организмов, которые представлены также в других группах (кластеры I, II, VI, VII). Единственное исключение ‒ это ген Pseudomallada prasinus (отряд Neuroptera). Гены AqE этого насекомого не попадают ни в один из других семи кластеров (I–IV и VI–VIII). Однако у одного представителя отряда Neuroptera (Osmylus fulvicephalus) гены AqE представлены как в кластере V, так и в кластере VI.
Кластеры I–IV и VI–VIII включают AqE, которые эволюционируют медленнее и присутствуют практически у всех изученных организмов, а кластер V содержит гены, эволюционирующие с более высокой скоростью, и многие группы, по-видимому, утратили этот вариант AqE. Гомологи гена AqE из кластера V выявлены у 10 видов: T. castaneum (AqE1), B. germanica (AqE1, AqE2, AqE3, AqE4, AqE5, AqE8 и AqE9), O. fulvicephalus (AqE2), An. glabripennis (AqE2 и AqE3), Leptinotarsa decemlineata (AqE1 и AqE2), L. kohalensis (AqE2 и AqE3), P. prasinus (AqE1), Zorotypus caudelli (AqE2 и AqE3), Drosophila busckii (AqE4 и AqE5) и D. miranda (AqE2 и AqE3).
Другое важное явление ‒ молодые дупликации, которые хорошо видны на филогенетическом дереве (рис. 1) и выявлены у четырех видов: A. albopictus, An. gambiae, Ctenocephalides felis и D. busckii. Так, например, у комара A. albopictus выявлены три копии AqE. Сходство между аминокислотными последовательностями, кодируемыми этими генами, превышает 99%. Белковый продукт гена AqE2 отличается одной аминокислотной заменой от белков, кодируемых AqE1 и AqE3. Однако белки AqE1 и AqE3 содержат на N-конце дополнительный фрагмент длиной 124 и 80 а.о. соответственно. Еще один комар (An. gambiae) имеет две копии гена, идентичные примерно на 100%, при этом белковый продукт гена AqE1 также содержит дополнительный N-концевой фрагмент (139 а.о.). У блохи C. felis обнаружены три гомолога гена AqE. Идентичность белков, кодируемых этими генами, колеблется от 93 до 97%, при этом присутствуют также вставки и делеции (табл. 1). У мухи D. busckii белковые продукты трех из пяти выявленных копий гена сходны на 99.77‒100%.
Структурные различия генов AqE насекомых
Определена экзон-интронная структура ОРС каждого выявленного гена AqE. Результаты оказались неожиданными – гены AqE насекомых существенно варьировали по данному признаку. Количество экзонов у разных представителей колеблется от 1 до 10, при этом в геноме одного организма могут одновременно присутствовать гены AqE с разным количеством экзонов. Например, один из трех генов D. miranda трехэкзонный (входит в кластер I), а два одноэкзонных (кластер V). Или у T. castaneum два гена содержат четыре экзона (AqE1, AqE2), а один семь (AqE4), при этом AqE1 входит в филогенетический кластер V, а AqE2 и AqE4 – в кластер VI.
Основная тенденция, наблюдаемая у насекомых, – сокращение числа интронов в гене AqE, согласуется с общей тенденцией в эволюции геномов. В большинстве линий эукариот эволюция связана, главным образом, с потерей интронов [16]. Классический AqE у рыб имеет 11 экзонов, причем это довольно консервативный признак [9]. Возможно, у рыб, которые обитают в более однородных условиях, чем насекомые, продукт этого гена участвует в некоторых ключевых реакциях обмена и подвержен более строгому отбору. У насекомых строгость отбора была ослаблена, за короткий срок накопились значительные изменения в структуре этого гена. Это может быть связано с преобразованием функции фермента в тех условиях, в которых обитает и развивается конкретный вид, а разнообразие условий обитания насекомых очень велико: от жидкой среды (в которой личинка дышит кислородом, растворенным в воде, субстрате или межклеточной жидкости) и почвы до атмосферной среды, где нет дефицита кислорода.
Кроме того, встречаются варианты AqE с одним экзоном. Этот процесс может быть связан с ретрогенизацией – вставкой в геном ДНК, полученной в процессе обратной транскрипции с мРНК исходного гена и, соответственно, свободной от интронов. Проверка возможной ретрогенизации у видов, имеющих одноэкзонные копии AqE, показала следующее: у B. germanica только один (AqE1) из четырех одноэкзонных генов имеет полиА-хвост и прямые повторы, которые обычно свидетельствуют о произошедшем событии ретрогенизации. Остальные три одноэкзонные копии B. germanica таких структур не имеют. У одноэкзонного AqE4 D. busckii нет ни полиА-хвоста, ни повторов, в отличие от AqE5, имеющего обе эти структуры. У AqE2 D. miranda нет ни п-олиА-хвоста, ни повторов, у AqE3 – повторы есть, но полиА-хвост короткий. Таким образом, AqE, имеющие прямые повторы и полиА-хвост, подверглись, очевидно, событию ретрогенизации. Что касается других безинтронных копий, то прямые повторы и полиА-хвост могут отсутствовать у более старых ретрогенов, но одновременное присутствие в геноме копий и с интронами, и без интронов предполагает, что безинтронные гены созданы путем ретрогенизации [17].
Модификации в аминокислотных последовательностях AqE
Множественное выравнивание белков AqE насекомых показало, что некоторые гены в процессе эволюции претерпели существенные модификации – делеции крайних или центральных зон, а также вставки добавочных участков. Протяженность AqE значительно варьирует, что связано, в основном, с делециями или вставками. Есть случаи, когда ген фрагментирован, но здесь мы, скорее всего, имеем дело с псевдогенизацией и остатком гена.
Копии AqE, имеющие дополнительный фрагмент, кодирующий 25–139 а.о. (табл. 1), представлены у всех просмотренных нами представителей Diptera. У D. busckii все пять копий AqE, а у D. miranda все три копии AqE содержат дополнительный фрагмент. У A. albopictus и An. gambiae наряду с удлиненными копиями AqE есть и копии без вставок. У представителя C. felis (Siphonaptera) все три гена AqE кодируют дополнительные фрагменты из 37 а.о. У O. fulvicephalus (Neuroptera) в начале гена AqE2 находится участок, кодирующий 27 добавочных аминокислот. Вставки найдены и в AqE4 T. castaneum (Coleoptera), и в AqE1 Dendroctonus ponderosae (Coleoptera). У An. glabripennis в трех из пяти генов AqE (AqE1, AqE2, AqE3) также есть удлинения (табл. 1).
В некоторых отрядах выявлены гены AqE с делециями, локализованными в основном в двух участках (табл. 1). Так, первый вариант делеции в центральной части гена представлен в двух из трех генов AqE L. kohalensis (Orthoptera), в двух из девяти генов B. germanica (Blattodea), в одной из двух копий у Homalodisca vitripennis (Hemiptera). Эту же делецию содержат оба варианта AqE у Calopteryx splendens (Odonata). Все эти элементы распределены по дереву без всякой взаимосвязи друг с другом, следовательно, делеция, по-видимому, возникала независимо.
Второй тип делеции встречается в генах AqE1 и AqE2 T. castaneum (Coleoptera), тогда как AqE4 не содержит такой делеции. При этом копии расположены на дереве без корреляции с делециями. Таким образом, варианты AqE с делециями не имеют общего корня; делеция, по всей видимости, возникала независимо всякий раз в каждом конкретном случае.
Есть также и фланкирующие делеции, но они встречаются в единичных случаях.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Известно, что гены остаются стабильными, если не происходит каких-то существенных изменений во внешних условиях и стабилизирующий отбор “выбрасывает” любые мутации, изменяющие или нарушающие функцию белка. Ген AqE присутствует почти во всех таксонах водных хордовых [9]. Его протяженность, интрон-экзонная структура и консервативные домены в целом достаточно постоянны, а присутствие транскриптов свидетельствует о “работоспособности” гена. У представителей класса насекомых ситуация противоположная – наблюдается выраженная неоднородность в наличии/отсутствии гена в геноме, в числе копий гена, его размере и экзон-интронной структуре (табл. 1). Причем эта неоднородность проявляется вплоть до вида, что наталкивает на мысль, что у насекомых, оказавшихся в новых условиях обитания, ген AqE попал под действие движущего отбора и активно эволюционировал/эволюционирует вплоть до настоящего момента, так как относительно недавно дивергировавшие группы организмов имеют значительные различия.
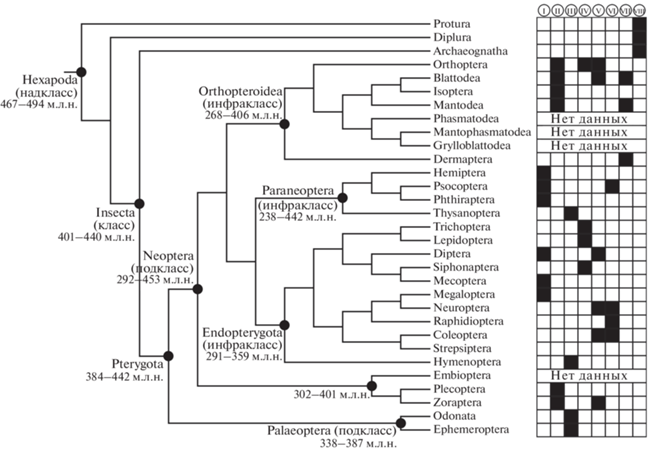
Филогенетический анализ показал, что все разнообразие AqE у насекомых делится на восемь кластеров (рис. 1). При этом статистическая значимость обособления кластеров ниже 50%. Если рассматривать распределение отрядов по кластерам, то видна выраженная мозаичность, которая не позволяет предположить пути эволюции гена AqE у насекомых (рис. 2). Возможно, что активная мультипликация AqE началась еще при обособлении эволюционной группы Pterygota около 384–442 млн лет назад. Новые копии эволюционировали независимо, а в дальнейшем возникали как новые дупликации, так и потери уже существующих генов, в связи с чем и сформировалось мозаичное распределение без корреляции филогении AqE с филогенией видов. Множественные события дупликаций и потери гена, а также быстрой эволюции свидетельствуют о том, что AqE у насекомых утратил свою консервативность и значимость в метаболических процессах. Так, например, еще одна подробно изученная группа животных Actinopterygii (костистые рыбы) дивергировала от других позвоночных примерно в тот же период: 364–409 млн лет назад. Однако ген AqE у этих животных строго консервативен [9].
Рис. 2.
Присутствие разных филогенетических кластеров гена AqE среди подтипа Hexapoda. Филогенетические отношения между отрядами и данные о времени дивергенции взяты из базы данных TimeTree (https://timetree.org/). м.л.н. ‒ миллионы лет назад.
Дупликация ‒ один из основных источников изменений, способствующих быстрой эволюции генома [17, 18]. Судьба большинства дуплицированных генов – псевдогенизация и/или элиминация из генома [19, 20]. У насекомых встречаются случаи псевдогенизации – у L. kohalensis в гене AqE2 рамка считывания нарушена стоп-кодоном, ген AqE3 остался в виде фрагмента, кодирующего продукт длиной 48 а.о. У B. germanica AqE8 представляет собой фрагмент, кодирующий пептид длиной 150 а.о. Продукт гена AqE2 An. glabripennis состоит из 147 а.о. (при этом выявлены транскрипты). У Leptinotarsa decemlineata все три гена AqE неполноценны (также выявлены транскрипты). В большинстве же случаев ген остается визуально функциональным, имеет неповрежденную ОРС, а зачастую и транскрипты. Следовательно, в подавляющем числе случаев мы имеем дело с дупликацией, которая успешно закрепилась в геноме насекомых. Ведущий эволюционный биохимик Shelley Copley предполагает, что средний фермент может иметь до 10 разных функций [21]. Соответственно, при дупликации гена любая смежная активность может быть отправной точкой для эволюции нового фермента, функция которого сходна с оригинальной или довольно сильно от нее отличается.
Дуплицированные гены могут закрепиться в геноме в результате процессов: а) неофункционализации – приобретения возникшим паралогом новой функции; б) ухода от адаптивного конфликта, когда исходный ген выполняет в организме две функции или более, а после дупликации бифункционального гена происходит разделение функций между возникшими копиями; в) субфункционализации, при которой из-за накопления мутаций оба паралога становятся необходимыми для выполнения функции, которую ранее обеспечивал предковый ген; г) консервации или мутации типа CNV (вариации числа копий), при которой копии сохраняются в неизменном состоянии, но увеличение их количества дает организму возможность синтеза большего количества специфических РНК или белков [17, 22, 23].
Мы предполагаем, что дупликации закрепились в геноме насекомых как результат неофункционализации или ухода от адаптивного конфликта. Если бы у предка насекомых дупликация привела к субфункционализации, то в настоящий момент среди потомков не должно быть организмов с одной копией гена AqE, так как при субфункционализации оба паралога необходимы для выполнения исходной функции гена. Нами же обнаружены организмы, имеющие единственный функциональный ген AqE (табл. 1). Вариация числа копий предполагает простое увеличение копийности, мы же имеем дело с очень измененными копиями AqE.
На рис. 1 видно, что AqE кластера V имеют более длинные эволюционные расстояния, чем другие. Это может быть косвенным свидетельством того, что эта группа AqE могла приобрести новую функцию. Известно, что по мере того, как организмы приобретали большее количество специализированных типов клеток, дупликация генов способствовала образованию совершенно разных ферментов, приспособленных к этим клеткам, а также увеличивала регуляторную специфичность в отношении времени их экспрессии в развитии [24‒26]. Новые ферменты могут образовываться, поскольку избыточность копий позволяет одной последовательности свободно мутировать и приобретать новые каталитические функции. Таким образом, возможно, произошло формирование большого разнообразия дегидрогеназ [27].
В ходе эволюции конкретных видов насекомых возникало множество дупликаций, которые успешно закреплялись в геноме. Среди них, помимо случаев неофункционализации или ухода от адаптивного конфликта, может быть и субфункционализация, например, у B. germanica все 9 дупликаций гена накопили достаточно мутаций и имеют давнее происхождение, судя по их расхождению на филогенетическом древе. Не исключена и возможность консервации, так у D. busckii идентичность копий 1, 2 и 3 (всего их пять) гена составляет 99.77–100%. Однако сказать наверняка вследствие чего закрепилась дупликация в каждом конкретном случае мы не можем из-за недостаточности данных.
Десять отрядов насекомых сохранили единственную копию гена, потеряв дуплицированную. Но есть случаи и полной потери гена – четыре отряда вообще не имеют гена AqE (табл. 1). Существуют различные сценарии эволюционной инактивации и/или потери гена: медленное накопление мутационных изменений в гене и превращение его в псевдоген с дальнейшей постепенной деградацией (фрагментация), или же внезапная и полная потеря гена – делеция вследствие неравного кроссинговера при мейозе или перемещения мобильных генетических элементов. Мы не обнаружили никаких остатков гена в исследованных нами геномах в группах, потерявших ген AqE, поэтому более достоверным представляется, что делеция, а, возможно, также и псевдогенизация привела к такой сильной деградации, что ген не обнаруживается при поиске.
Потеря генов – широко распространенный в эволюции феномен. Показано, что потеря генов может быть адаптивной или нейтральной. Если ген становится избыточным, то его потеря оказывает нейтральное влияние на жизнедеятельность [28, 29]. С другой стороны, потеря гена может существенно повышать адаптивный потенциал вида. Мутации, приводящие к потере функциональности гена, происходят чаще, чем мутации, которые приводят к приобретению новой функции [30]. Следовательно, потери гена сильнее влияют на адаптивную эволюцию, особенно во время быстрой адаптации к резким изменениям окружающей среды [31].
Мы не можем делать выводы об адаптивном характере потери гена у насекомых из-за отсутствия данных, свидетельствующих в пользу такой возможности. Вероятнее всего, виды, потерявшие ген, приобрели иные пути метаболизма, вследствие чего AqE утратил свою значимость и элиминировался из генома, или же аналогичные гены взяли на себя его функцию. Известны примеры негомологичной замены генов. Так, например, ферменты SLDH, которые используют оксалоацетат в качестве субстрата с относительно высокой эффективностью [32], могут действовать в качестве аналога MDH, компенсирующего отсутствие специфической MDH у метаногенной археи [6].
Насекомые – это единственный класс животных, обитающий на суше и сохранивший ген AqE, кодирующий SLDH-подобный белок [5]. Не исключено, что у насекомых AqE включен в некие метаболические пути, которые оказались полезными в условиях их обитания. Личинки насекомых, а иногда и имаго, зачастую сталкиваются с недостатком кислорода, избытком сероводорода, метана и иных газов, образующихся при брожении и разложении органических веществ. В таких стрессовых условиях, возможно, необходимы новые метаболические пути, способные обеспечить существование организма в подобных агрессивных средах. Наиболее важная функция оксидоредуктаз связана с их эколого-биохимической ролью в развитии адаптивных реакций, выражающихся, как правило, в регуляции баланса аэробных и анаэробных процессов [33]. Показано, что оксидоредуктазы могут повышать устойчивость растений к холодовому и солевому стрессу [34–36].
Функция гена AqE пока не установлена, но, возможно, его продукт является резервным метаболитом, который включается в анаэробный обмен в экстремальных ситуациях.
ЗАКЛЮЧЕНИЕ
В результате изучения распространенности и разнообразия гена AqE установлено, что этот ген представлен не во всех отрядах/подотрядах насекомых, при том что у некоторых видов ген AqE имеет два и более гомолога. При этом даже в пределах одного отряда/подотряда наблюдается гетерогенность как в количестве гомологов, так и в потере генов. Предполагается, что мультипликация гена AqE насекомых имеет древнюю природу, однако возможны и молодые дупликации. Более того, установлена вариабельность как длины гена, так и числа экзонов в нем (от 1 до 10). Не исключено, что в связи с появлением паралогов ген приобрел новую функцию. Такое разнообразие в представленности и в структуре создает большие сложности с идентификацией паралогов и установлением эволюционных взаимосвязей. Возможно, при более детальном (на уровне семейств и родов) исследовании разнообразия AqE удастся получить более точные данные. Дополнить картину эволюции позволит также изучение этого гена у всех членистоногих или более древних таксонов.
Предполагается, что необычное поведение гена AqE, может быть связано как с потерей его значимости для метаболизма насекомых, так и с приобретением новых функций и/или с необходимостью адаптации к разнообразным условиям, в которых обитают представители этого класса.
Исследование проведено в рамках государственного задания ФГБУН ИМБИ “Функциональные, метаболические и токсикологические аспекты существования гидробионтов и их популяций в биотопах с различным физико-химическим режимом” (номер гос. регистрации 121041400077-1).
Настоящая статья не содержит исследований с использованием животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Honka E., Fabry S., Niermann T., Palm P., Hensel R. (1990) Properties and primary structure of the L-malate dehydrogenase from the extremely thermophilic archaebacterium Methanothermus fervidus. Eur. J. Biochem. 188, 623–632. https://doi.org/10.1111/j.1432-1033.1990.tb15443.x
Jendrossek D., Kratzin H.D., Steinbuchel A. (1993) The Alcaligenes eutrophus ldh structural gene encodes a novel type of lactate dehydrogenase. FEMS Microbiol. Lett. 112, 229–235. https://doi.org/10.1111/j.1574-6968.1993.tb06453.x
Muramatsu H., Mihara H., Kakutani R., Yasuda M., Ueda M., Kurihara T., Esaki N. (2005) The putative malate/lactate dehydrogenase from Pseudomonas putida is an NADPH-dependent delta1-piperideine-2-carboxylate/delta1-pyrroline-2-carboxylate reductase involved in the catabolism of D-lysine and D-proline. J. Biol. Chem. 280(7), 5329‒5335. https://doi.org/10.1074/jbc.M411918200
Muramatsu H., Mihara H., Goto M., Miyahara I., Hirotsu K., Kurihara T., Esaki N. (2005) A new family of NAD(P)H-dependent oxidoreductases distinct from conventional Rossmann-fold proteins. J. Biosci. Bioeng. 99, 541‒754. https://doi.org/10.1263/jbb.99.541
Puzakova L.V., Puzakov M.V., Soldatov A.A. (2019) Gene encoding a novel enzyme of LDH2/MDH2 family is lost in plant and animal genomes during transition to land. J. Mol. Evol. 87, 52‒59. https://doi.org/10.1007/s00239-018-9884-2
Irimia A., Madern D., Zaccaï G., Vellieux F.M. (2004) Methanoarchaeal sulfolactate dehydrogenase: prototype of a new family of NADH-dependent enzymes. EMBO J. 23, 1234‒1244. https://doi.org/10.1038/sj.emboj.7600147
Denger K., Cook A.M. (2010) Racemase activity effected by two dehydrogenases in sulfolactate degradation by Chromohalobacter salexigens: purification of (S)-sulfolactate dehydrogenase. Microbiology (Reading). 156, 967‒974. https://doi.org/10.1099/mic.0.034736-0
Zhang Y., Schofield L.R., Sang C., Dey D., Ronimus R.S. (2017) Expression, purification, and characterization of (R)-sulfolactate dehydrogenase (ComC) from the rumen methanogen Methanobrevibacter millerae SM9. Archaea. 6, 5793620. https://doi.org/10.1155/2017/5793620
Puzakova L.V., Puzakov M.V., Gostyukhina O.L. (2021) Newly discovered AqE gene is highly conserved in non-tetrapod vertebrates. J Mol Evol. 89, 214‒224. https://doi.org/10.1007/s00239-021-09997-x
Berthelot C., Brunet F., Chalopin D., Juanchich A., Bernard M., Noël B., Bento P., Da Silva C., Labadie K., Alberti A., Aury J. M., Louis A., Dehais P., Bardou P., Montfort J., Klopp C., Cabau C., Gaspin C., Thorgaard G.H., Boussaha M., Quillet E., Guymard R., Galiana D., Bobe J., Volff J.N., Genêt C., Wincker P., Jaillon O., Roest Crollius H., Guiguen Y. (2014) The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates. Nat. Commun. 5, 3657.
Petit J., David L., Dirks R., Wiegertjes G.F. (2017) Genomic and transcriptomic approaches to study immunology in cyprinids: what is next? Devel. Compar. Immunol. 75, 48‒62.
Пузакова Л.В., Пузаков М.В. (2022) Тканеспецифичность активности гена AqE у желтого горбыля Larimichthys crocea. Генетика. 58, 540–549.
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucl. Acids Res. 25, 3389‒3402. https://doi.org/10.1093/nar/25.17.3389
Edgar R.C. (2004) MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucl. Acids Res. 32, 1792‒1797. https://doi.org/10.1093/nar/gkh340
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. (2018) MEGA X: molecular evolutionary genetics analysis across computing platforms. Mol. Biol. Evol. 35(6), 1547‒1549. https://doi.org/10.1093/molbev/msy096
Rogozin I.B., Carmel L., Csuros M., Koonin E.V. (2012) Origin and evolution of spliceosomal introns. Biol. Direct. 7, 11. https://doi.org/10.1186/1745-6150-7-11
Cardoso-Moreira M., Long M. (2012) The origin and evolution of new genes. In: Evolutionary Genomics. Methods Mol. Biol. (Methods and Protocols). Ed. Anisimova M. 856. Humana Press, 161–186. https://doi.org/10.1007/978-1-61779-585-5_7
Taylor J.S., Raes J. (2004) Duplication and divergence: the evolution of new genes and old ideas. Annu. Rev. Genet. 38, 615‒643. https://doi.org/10.1146/annurev.genet.38.072902.092831
Lynch M., Conery J.S. (2000) The evolutionary fate and consequences of duplicate genes. Science. 290, 1151‒1155. https://doi.org/10.1126/science.290.5494.1151
Журавлева Г.А., Инге-Вечтомов С.Г. (2009) Возникновение новых белков за счет дупликации генов ‒ что общего в эволюции зрительных цветочувствительных белков и факторов терминации трансляции. Молекуляр. биология. 43, 759‒771.
Copley S.D. (2017) Shining a light on enzyme promiscuity. Curr. Opin. Struct. Biol. 47, 167‒175. https://doi.org/10.1016/j.sbi.2017.11.001
Ohno S. (1970) Introduction. In: Evolution by Gene Duplication. Berlin, Heidelberg: Springer, https://doi.org/10.1007/978-3-642-86659-3_1
Hahn M.W. (2009) Distinguishing among evolutionary models for the maintenance of gene duplicates. J. Hered. 100, 605‒617. https://doi.org/10.1093/jhered/esp047
Markert C.L. (1971) Developmental Genetics. Heinrich Ursprung. 214 p.
Markert C.L., Shaklee J.B., Whitt G.S. (1975) Evolution of a gene. Multiple genes for LDH isozymes provide a model of the evolution of gene structure, function and regulation. Science. 189, 102‒114. https://doi.org/10.1126/science.1138367
Zuckerkandl E. (1978) Multilocus enzymes, gene regulation, and genetic sufficiency. J. Mol. Evol. 12, 57‒89. https://doi.org/10.1007/BF01732545
Eventhoff W., Rossman M. G. (1975) The evolution of the dehydrogenases and kinases. CRC Crit. Rev. Biochem. 3, 111–140.
Moreau R., Dabrowski K. (1998) Body pool and synthesis of ascorbic acid in adult sea lamprey (Petromyzon marinus): an agnathan fish with gulonolactone oxidase activity. Proc. Natl. Acad. Sci. USA. 95, 10279‒10282. https://doi.org/10.1073/pnas.95.17.10279
Drouin G., Godin J.R., Pagé B. (2011) The genetics of vitamin C loss in vertebrates. Curr Genomics. 12, 371‒378. https://doi.org/10.2174/138920211796429736
Albalat R., Cañestro C. (2016) Evolution by gene loss. Nat. Rev. Genet. 17, 379‒391. https://doi.org/10.1038/nrg.2016.39
Greenberg A.J., Moran J.R., Coyne J.A., Wu C.I. (2003) Ecological adaptation during incipient speciation revealed by precise gene replacement. Science. 302, 1754‒1757. https://doi.org/10.1126/science.1090432
Graupner M., Xu H., White R.H. (2000) Identification of an archaeal 2-hydroxy acid dehydrogenase catalyzing reactions involved in coenzyme biosynthesis in methanoarchaea. J. Bacteriol. 182, 3688–3692.
Мещерякова О.В. (2004) Динамика активности изоферментов лактатдегидрогеназы, малатдегидрогеназы и α-глицерофосфатдегидрогеназы в процессе адаптации рыб к различным факторам окружающей среды. Автореферат дисс. канд. биол. наук. Петрозаводск. 24 с.
Kandoi D., Mohanty S., Tripathy B.C. (2018) Overexpression of plastidic maize NADP-malate dehydrogenase (ZmNADP-MDH) in Arabidopsis thaliana confers tolerance to salt stress. Protoplasma. 255, 547‒563. https://doi.org/10.1007/s00709-017-1168-y
Wang Q.J., Sun H., Dong Q.L., Sun T.Y., Jin Z.X., Hao Y.J., Yao Y.X. (2016) The enhancement of tolerance to salt and cold stresses by modifying the redox state and salicylic acid content via the cytosolic malate dehydrogenase gene in transgenic apple plants. Plant Biotechnol. J. 14, 1986–1997.
Yao Y.X., Dong Q.L., Zhai H., You C.X., Hao Y.J. (2011) The functions of an apple cytosolic malate dehydrogenase gene in growth and tolerance to cold and salt stresses. Plant Physiol. Bioch. 49, 257–264.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


