Молекулярная биология, 2023, T. 57, № 2, стр. 269-284
Механизмы специфичности системы CRISPR/Cas9 в геномном редактировании
Л. М. Кулишова a, И. П. Вохтанцев a, Д. В. Ким a, b, Д. О. Жарков a, b, *
a Институт химической биологии и фундаментальной медицины, Сибирское отделение Российской академии наук
630090 Новосибирск, Россия
b Новосибирский государственный университет
630090 Новосибирск, Россия
* E-mail: dzharkov@niboch.nsc.ru
Поступила в редакцию 15.08.2022
После доработки 03.10.2022
Принята к публикации 06.10.2022
- EDN: EFDQPQ
- DOI: 10.31857/S0026898423020155
Аннотация
Не так давно открытая система CRISPR/Cas9, основанная на действии комплементарно адресуемых нуклеаз и изначально предназначенная для защиты бактерий от чужеродных генетических элементов, стала удобным инструментом в руках ученых для манипуляции геномами живых клеток. Технология геномного редактирования CRISPR/Cas9 вышла за пределы лаборатории и уже находит применение в биотехнологии и сельском хозяйстве. Однако использование этого метода для редактирования клеток человека в медицинских целях ограничено внеадресной активностью системы CRISPR/Cas9, которая может привести к появлению онкогенных мутаций. В связи с этим ведутся активные исследования с целью разработки вариантов системы CRISPR/Cas9 с повышенной точностью. В обзоре освещены механизмы точного и ошибочного действия рибонуклеопротеина Cas9, природные и искусственно созданные варианты РНК-адресуемых нуклеаз, способы модуляции специфичности за счет модификации направляющей РНК и другие подходы к увеличению точности системы CRISPR/Cas9 в геномном редактировании.
В связи с огромным интересом к редактированию генома с помощью технологии CRISPR/Cas9 (англ. Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR-ASsociated protein 9; сгруппированные короткие палиндромные повторы, разделенные регулярными промежутками/CRISPR-ассоциированный белок-9) большое внимание в настоящее время уделяется различным аспектам специфичности этой системы, которая дает возможность точного редактирования генома ‒ внесения специфичных замен путем гомологичной рекомбинации с низким фоном негомологичного соединения концов. Технологию CRISPR/Cas9 активно используют для создания клеточных линий, моделирующих заболевания, и рассматривают потенциал ее применения для терапевтического вмешательства в геном человека. Однако для этого необходимо снизить неадресное действие системы, в идеале ‒ до такого значения, которое не превышало бы нормальный уровень мутаций ДНК в клетках человека (~10−10 мутаций на пару нуклеотидов на одно клеточное деление) [1‒3].
Белок Cas9 (в дальнейшем, если нет дополнительных уточнений, рассматривается Cas9 из Streptococcus pyogenes, SpCas9) состоит из нескольких доменов и обладает РНК-зависимой ДНК-эндонуклеазной активностью, локализованной в двух из них: HNH-домене (домен, содержащий высококонсервативные остатки His–Asn–His), который расщепляет цепь ДНК, комплементарную направляющей РНК (адресуемая цепь), и RuvC-подобном домене, гидролизующем цепь ДНК, совпадающую с последовательностью направляющей РНК (неадресуемая цепь) [4, 5]. Для расщепления ДНК необходимо наличие в ней протоспейсера ‒ участка двухцепочечной ДНК (дцДНК), одна из цепей которой образует комплементарные связи с направляющей РНК, и примыкающего непосредственно к протоспейсеру мотива 5′-NGG-3′ (PAM, англ. Protospacer-Adjacent Motif) (рис. 1а).
Рис. 1.
Схема организации фермент-субстратных комплексов РНК-адресуемых нуклеаз Cas9 (а) и Cas12a (б).

В природе действие Cas9 адресуется короткой направляющей CRISPR-РНК (crРНК), а для каталитической активности необходима трансактивирующая CRISPR-РНК (tracrРНК). Для применения в геномном редактировании зачастую эти две РНК интегрируют в единую направляющую РНК (далее использованы аббревиатуры: sgРНК для единой направляющей РНК и gРНК для направляющей РНК вне зависимости от ее природы). sgРНК содержит вариабельный участок узнавания целевой последовательности (20 нуклеотидов для SpCas9) и минимально необходимый фрагмент tracrРНК. Рибонуклеопротеиновый комплекс (RNP, англ. RiboNucleoProtein) Cas9 расщепляет обе цепи ДНК между третьим и четвертым нуклеотидами с 5'-стороны от PAM [4, 5]. Образовавшийся двухцепочечный разрыв далее подвергается репарации клеточными системами гомологичной рекомбинации (HDR, англ. Homology-Directed Recombination) или воссоединения негомологичных концов (NHEJ, англ. Non-Homologous End Joining) [6, 7]. В первом случае при наличии подходящего донора генетической информации редактирование приводит к точной замене исходной последовательности на донорную, а во втором обычно происходят небольшие делеции или инсерции на месте разрыва (рис. 2).
Рис. 2.
Пути репарации двухцепочечного разрыва в клетках с образованием точной замены (гомологичная рекомбинация) или инсерции/делеции (воссоединение негомологичных концов). Донор генетического материала для гомологичной рекомбинации обозначен серым цветом.

МОЛЕКУЛЯРНЫЕ МЕХАНИЗМЫ ОШИБОЧНОГО ДЕЙСТВИЯ БЕЛКА Cas9
Несмотря на кажущуюся простоту и специфичность комплементарной адресации нуклеазы Cas9 при помощи направляющих РНК, в первых попытках редактирования клеток человека система CRISPR/Cas9 оказалась недостаточно специфичной, так как вносила в геном множественные неадресные изменения [8‒11]. В работах по определению основных детерминант комплементарной специфичности комплекса Cas9–РНК показано, что 6–8 пар оснований ДНК-мишени, ближайших к PAM, как правило, должны точно соответствовать последовательности gРНК, однако в условиях избытка фермента это требование становится менее жестким [12, 13]. Исчерпывающий анализ последствий наличия однонуклеотидных несоответствий (мисматчей) между несколькими sgРНК и ДНК-мишенью в клеточных линиях HEK293 и K562 подтвердил, что нуклеаза Cas9 менее чувствительна к мисматчам в дальней от PAM области по сравнению с расположенными вблизи PAM [9, 10]. Чувствительность к единичным заменам максимальна в пределах 8–14 нуклеотидов, ближайших к РАМ. Эта область, определяющая специфичность узнавания, получила название “затравочной последовательности” (seed sequence). Впоследствии методами одномолекулярной микроскопии удалось установить, что при узнавании ДНК-мишени в затравочной последовательности быстро образуется гетеродуплекс, и, если он стабилен, комплементарные связи РНК–ДНК распространяются далее по всему узнаваемому участку [14]. Однако оказалось, что мисматчи разных типов влияют на специфичность системы в разной степени. При детальном анализе 11 дополнительных локусов генома обнаружили многочисленные исключения из принципа полной комплементарности затравочной последовательности: толерантность к мисматчам варьировала в зависимости от конкретной пары оснований, при этом мисматчи rC:dC сильнее всего снижали нуклеазную активность Cas9 [10].
Последствия множественных мисматчей между gРНК и ДНК-мишенью также исследовали как в отношении числа мисматчей в последовательности, так и их взаимного расположения [9]. Выяснилось, что ключевым фактором для потери активности Cas9 служит число мисматчей, но также важно их непосредственное соседство и близость к РАМ. Два, а тем более три мисматча, особенно в прилежащей к PAM области, значительно снижают активность Cas9 независимо от их взаимного расположения, но при удалении от PAM наибольшее влияние оказывают соседние друг с другом мисматчи. Однако в редких случаях фермент Cas9 способен продуктивно узнавать последовательности, содержащие до 7 мисматчей [15].
На основании недавно определенных структур Cas9 в комплексе с ДНК и РНК, отражающих разные стадии узнавания субстрата, стало возможным установить структурные особенности динамического процесса узнавания при точном и неточном соответствии gРНК и ДНК-мишени [16, 17] (рис. 3). Связываясь с дцДНК, комплекс Cas9–sgРНК изгибает ее примерно на 50°, что приводит к выворачиванию из дуплекса трех оснований, непосредственно прилегающих к PAM. Образования гетеродуплекса в этом участке оказывается достаточным для дальнейшего раскрытия дцДНК. При наличии 3-нуклеотидного мисматча в центральной части ДНК-мишени гетеродуплекс образуется полностью, но такого изгиба не происходит, что не позволяет HNH-домену занять положение, необходимое для катализа. Однако ДНК, содержащая 3-нуклеотидный мисматч в удаленных от PAM участках, способна изгибаться и тем самым индуцировать каталитически компетентную конформацию Cas9. Это обусловлено наличием в гетеродуплексе позиций, не образующих связей с белком в промежуточных конформациях, предшествующих образованию каталитически компетентного фермент-субстратного комплекса.
Рис. 3.
Схема многоэтапного узнавания субстрата рибонуклеопротеином Cas9. ДНК обозначена черными линиями, РНК ‒ серой. (1) ‒ узнавание PAM и образование первичного комплекса; (2) ‒ конформационное изменение фермента и изгибание ДНК вблизи от PAM, облегчающее раскрытие дуплекса; (3) ‒ образование гетеродуплекса в затравочной области, при наличии в затравочной области мисматча этот шаг не реализуется; (4) ‒ образование полного гетеродуплекса с ДНК-мишенью; (5) ‒ изгибание гетеродуплекса с образованием дополнительных связей с белком, приводящих активные центры доменов HNH и RuvC (обозначены треугольниками) в контакт с расщепляемыми фосфодиэфирными связями, при наличии мисматча в дальней от PAM области этот шаг не реализуется.
Кроме мисматчей потенциальным источником неадресного узнавания служит возможность комплементарных взаимодействий с образованием небольших петель. При исчерпывающем анализе влияния инсерций и делеций в sgРНК на нуклеазную активность Cas9 оказалось, что система толерантна к ДНК-выпетливаниям размером в 1 нуклеотид и к РНК-выпетливаниям до 4 нуклеотидов длиной, причем степень толерантности зависит от положения лишних нуклеотидов относительно РАМ [18].
Анализ влияния нуклеотидных замен в РАМ выявил, что при замене последовательности NGG на NAG или NGA сохраняется до 20% нуклеазной активности Cas9 [10, 19], а для PAM NGT активность составляет около 10% [20]. Способность Cas9 расщеплять мишени вблизи неканонических PAM впоследствии была подтверждена данными по полногеномному секвенированию после трансфекции клеток человека библиотеками sgРНК [21]. Как известно, метилирование ДНК не влияет на активность Cas9, поэтому способность фермента узнавать неканонические PAM ведет к тому, что потенциально адресуемые участки в среднем встречаются 1 раз на 4 п.н. генома человека [10].
Наблюдаемый уровень неадресных изменений генома вполне приемлем для исследований потери функции генов на эукариотических клеточных линиях [22‒25]. В этих случаях клетки обычно трансфицируют конструкциями на основе вирусных векторов, экспрессирующих Cas9 и sgРНК на низком уровне ‒ вплоть до 1 конструкции на клетку. В одном из широкомасштабных анализов Cas9-опосредованных нокаутов генов в клетках млекопитающих [22] уровень расщепления в локусе-мишени составил 97%, а в 13 потенциальных неадресных участках, отличающихся от мишени не более чем на 3 п.н., ‒ менее 2.5%. Единственный сайт с высоким уровнем неадресного расщепления был полностью комплементарен sgРНК по 8 п.н., прилегающим к РАМ. Подобные сайты встречаются с частотой ~2 на геном человека и почти всегда находятся в некодирующей ДНК [22].
ПОДБОР САЙТОВ-МИШЕНЕЙ in silico ДЛЯ ПОВЫШЕНИЯ ТОЧНОСТИ РЕДАКТИРОВАНИЯ
Одним из первых шагов для повышения точности геномного редактирования стало создание компьютерных алгоритмов для выбора последовательности gРНК, поскольку вероятность неадресных изменений можно с некоторой точностью предсказать, исходя из экспериментально установленного влияния мисматчей на активность системы CRISPR/Cas9. В целом, gРНК должны направлять белок Cas9 к такой мишени, последовательность которой имеет минимальное сходство с другими участками генома, а похожие на нее участки либо не находятся рядом с PAM, либо отличаются от адресной последовательности в ближней к PAM части. Применение алгоритмов первого поколения сразу же позволило повысить специфичность редактирования на 50% [10]. Более современные алгоритмы учитывают энергию образования гетеродуплексов ДНК/РНК [26]. К настоящему времени создано несколько десятков программ для предсказания наиболее выгодных последовательностей-мишеней [27, 28].
Новое поколение подходов к селекции мишеней стало возможно после разработки эффективных и быстрых методов детекции неадресных мутаций во всем геноме. В первых исследованиях применялось глубокое полногеномное секвенирование, позволяющее в том числе отличать индивидуальные вариации генома от неадресных мутаций. Специально адаптированный для выявления сайтов неадресного редактирования метод Digenome-Seq основан на полногеномном секвенировании исходной и отредактированной ДНК после ее расщепления Cas9 с нужной gРНК [29]. Однако разработаны и многочисленные менее затратные методы, комбинирующие захват, фиксацию, амплификацию и секвенирование именно измененных участков генома: BLESS [30], NTGTS [31], GUIDE-Seq [15], SITE-Seq [32], CircleSeq [33], CHANGE-seq [34] и другие. Большие наборы данных, получаемые из таких экспериментов, позволили использовать методы машинного обучения для предсказания неадресных модификаций и оптимизации подбора сайтов-мишеней [34‒36]. Накопленные экспериментальные данные для различных видов живых организмов: человека, мыши, Danio rerio, Drosophila melanogaster, Arabidopsis thaliana и многих других ‒ позволяют учитывать видоспецифичные паттерны неадресных изменений для дальнейшего повышения точности геномного редактирования [37‒41].
Аналогичным образом в алгоритмах второго поколения были использованы данные по иммунопреципитации каталитически неактивного Cas9 (D10A H840A; dCas9) c последующим высокопараллельным секвенированием связанных фрагментов геномной ДНК [11, 42‒44]. Хотя специфичность связывания dCas9 не вполне точно отражает специфичность расщепления активным ферментом, такие алгоритмы успешно используют в дизайне последовательностей gРНК для регуляции транскрипции, эпигенетической модификации, редактирования оснований и прайм-редактирования, где направляющий модуль представляет собой каталитически неактивную форму Cas9 [45‒49]. В новых вариантах алгоритмов машинного обучения для редактирования оснований используют непосредственно данные по адресным и неадресным мутациям из полногеномного секвенирования [50].
ВАРИАНТЫ Cas9 С ПОВЫШЕННОЙ ТОЧНОСТЬЮ
В свете описанных проблем со специфичностью внесения двухцепочечных разрывов нуклеазой Cas9 практически сразу начались попытки модификации фермента для повышения точности системы. Первая использованная стратегия связана с применением Cas9-никаз и по сути аналогична популярной схеме применения химерных белков, сочетающих домены узнавания на основе цинковых пальцев или TAL-эффекторов (англ. Transcription Activator-Like; подобные активаторам транскрипции) и димеризующийся эндонуклеазный домен рестриктазы FokI [51]. Замены D10A в домене RuvC или Н840А в домене HNH превращают Cas9 в никазы, которые вносят в ДНК только одноцепочечные разрывы. При использовании Cas9-никаз с двумя правильно подобранными gРНК можно получать два близко расположенных одноцепочечных разрыва, которые вместе дадут двухцепочечный; при этом специфичность его образования будет значительно повышена из-за необходимости одновременного узнавания двух последовательностей. Увеличение специфичности в случае применения парных комплексов Cas9 D10A‒gРНК составляет около 2 порядков для клеточной линии HEK-293FT [52], а в ряде исследований находится ниже уровня детекции [13, 53, 54]. На практике этот подход был использован, в частности, для получения особей крупного рогатого скота с точечной заменой в гене NRAMP1, придающей врожденный иммунитет к туберкулезу [55]. Известны также попытки использовать химерную конструкцию dCas9–FokI [56‒58]. В клетках HEK293 специфичность такого белка была в 140 раз выше, чем Cas9, и в 1.3–8.8 превосходила пару Cas9-никаз.
Установление пространственной структуры эндонуклеазы Cas9 и ее комплексов с ДНК и РНК, в том числе разных конформеров, образуемых в ходе узнавания мишени [16, 17, 59‒68], позволило применить для повышения специфичности фермента методы рационального дизайна (табл. 1). При сканирующем аланиновом мутагенезе ДНК-связывающего канала Cas9 было получено 11 вариантов с улучшенной специфичностью, которая, по всей видимости, объясняется уменьшением вклада ДНК-белковых контактов в стабилизацию комплекса Cas9–gРНК–ДНК и соответствующим увеличением вклада комплементарных взаимодействий ДНК–РНК [69]. Скрининг комбинаторной библиотеки этих замен позволил идентифицировать варианты с сохранившейся активностью и повышенной специфичностью: eSpCas(1.0) (K810A K1003A R1060A) и eSpCas(1.1) (K848A K1003A R1060A). Они не вносили нецелевых изменений в 22 из 24 предсказанных наиболее вероятных неадресных сайтов и были чувствительны к единственному мисматчу в gРНК за пределами затравочной последовательности [69].
Таблица 1.
Генно-инженерные варианты Cas9 из Streptococcus pyogenes с повышенной специфичностью
| Название | Принцип создания | Замены по сравнению с SpCas9 дикого типа | Ссылка |
|---|---|---|---|
| eSpCas(1.0) | Рациональный дизайн: дестабилизация электростатических контактов ДНК с белком | K810A K1003A R1060A | [69] |
| eSpCas(1.1) | Рациональный дизайн: дестабилизация электростатических контактов ДНК с белком | K848A K1003A R1060A | [69] |
| SpCas9-HF1 | Рациональный дизайн: дестабилизация водородных связей ДНК с белком | N497A R661A Q695A Q926A | [70] |
| HypaCas9 | Рациональный дизайн: облегчение конформационной коррекции в ходе узнавания субстрата | N692A M694A Q695A H698A | [71] |
| SuperFi-Cas9 | Рациональный дизайн: дестабилизация комплекса белка с гетеродуплексом, содержащим мисматчи | Y1010D Y1013D Y1016D V1018D R1019D Q1027D K1031D | [16] |
| evoCas9 | Направленная эволюция: селекция на адресную инактивацию токсичного гена и отсутствие неадресной инактивации геномного локуса в дрожжах | M495V Y515N K526E R661Q | [72] |
| Sniper-Cas9 | Направленная эволюция: селекция на адресную инактивацию токсичного гена и отсутствие неадресной инактивации геномного локуса в Escherichia coli | F539S M763I K890N | [73] |
| HiFi Cas9 | Направленная эволюция: селекция на адресную инактивацию токсичного гена и отсутствие неадресной инактивации геномного локуса в E. coli | R691A | [74] |
| xCas9-3.6 | Рациональный дизайн: расширение спектра узнаваемых PAM, увеличение специфичности как побочный эффект | E108G S217A A262T S409I E480K E543D M694I E1219V | [20] |
| xCas9-3.7 | Рациональный дизайн: расширение спектра узнаваемых PAM, увеличение специфичности как побочный эффект | A262T R324L S409I E480K E543D M694I E1219V | [20] |
Аналогичный подход был использован для повышения специфичности Cas9 за счет дестабилизации водородных связей между белком и ДНК. В структуре комплекса есть 4 водородных связи между фосфатами ДНК-мишени и остатками Asn497, Arg661, Gln695 и Gln926. Скрининг библиотеки, содержащей варианты с заменой четырех этих остатков на Ala во всех возможных комбинациях, привел к обнаружению высокоспецифичных вариантов: R661A Q695A Q926A и N497A R661A Q695A Q926A (SpCas9-HF1) [70]. Частоты нецелевых мутаций, индуцированных SpCas9-HF1, в 34 из 36 предсказанных неадресных сайтов были статистически неотличимы от фонового уровня.
Первоначально предполагали, что замена остатков, находящихся на ДНК-белковом интерфейсе, приводит в целом к снижению аффинности связывания Cas9 с ДНК. Однако более детальные исследования с использованием динамического ферстеровского резонансного переноса энергии не подтвердили эту гипотезу [71]. Оказалось, что повышение специфичности связано с механизмом “конформационной коррекции” (conformational proofreading) в ходе последовательных изменений пространственной ориентации доменов REC3 (англ. RECognition domain 3; узнающий домен-3), REC2 и HNH при связывании фермента с субстратом (рис. 3). Домен REC3 действует как аллостерический эффектор, который узнает гетеродуплекс РНК/ДНК, чтобы обеспечить активацию нуклеазного домена HNH, а домен REC2 препятствует доступу каталитических остатков к гидролизуемым фосфодиэфирным связям при наличии мисматчей [16, 71]. На основе этих экспериментов был сконструирован вариант HypaCas9 (N692A M694A Q695A H698A) с мутациями в домене REC3, более специфичный по сравнению даже с eSpCas(1.1) и SpCas9-HF1 при схожей эффективности [71]. Другой вариант, SuperFi-Cas9 (Y1010D Y1013D Y1016D V1018D R1019D Q1027D K1031D), в котором все аминокислотные остатки, стабилизирующие комплекс с мисматчем, заменены на Asp, показал 500-кратное повышение специфичности in vitro [16], но действие его в клетках пока не исследовано.
Помимо рационального дизайна, неоднократно делались попытки получения улучшенных вариантов белка Cas9 методами направленной эволюции (табл. 1). Для этого создано несколько систем селекции in vivo с одновременным отбором на адресную инактивацию токсичного гена и отсутствие неадресной инактивации геномного локуса. Одна из первых таких успешных систем на основе клеток дрожжей позволяет одновременно оценивать как адресную, так и неадресную активность Cas9 [72]. Скрининг библиотеки вариантов Cas9 со случайными мутациями в домене REC3 выявил варианты, повышающие точность редактирования при сохранении его эффективности. По своей точности наилучший из них фермент ‒ evoCas9 (M495V Y515N K526E R661Q) ‒ превышает как белок дикого типа, так и рационально сконструированные варианты Cas9 (в среднем 4-кратное улучшение по сравнению с eSpCas и SpCas9-HF1), сохраняя при этом адресную активность, близкую к белку дикого типа [72]. В другой системе, основанной на селекции в клетках Escherichia coli, был получен вариант Sniper-Cas9 (F539S M763I K890N), который показал высокую специфичность без уменьшения целевой активности в клетках человека [73]. По сравнению с eSpCas9(1.1), SpCas9-HF1, evoCas9 и HypaCas9 фермент Sniper-Cas9 проявлял самую высокую специфичность в 10 из 12 сайтов клеток HEK-293T и HeLa. Похожий метод селекции в E. coli позволил получить вариант HiFi Cas9 (R691A), который по сравнению с другими улучшенными вариантами показывал наивысшую активность в первичных гемопоэтических клетках человека при использовании в виде предсформированных RNP [74]. Повышенная точность SpCas9-HF1, HypaCas9 и HiFi Cas9 в клетках может быть обусловлена тем, что у этих эндонуклеаз резко снижена способность образовывать двухцепочечный разрыв, отчего они частично действуют как никазы [75].
В отличие от протоспейсера, узнавание PAM основано не на принципе комплементарности, а исключительно на взаимодействии обоих оснований G в последовательности NGG ДНК с аминокислотными остатками белка [59]. Попытки модифицировать узнавание PAM белком Cas9 в основном были направлены на расширение набора PAM-последовательностей, а не на повышение точности узнавания PAM. Любопытно, что некоторые полученные методом направленной эволюции варианты со множественными аминокислотными заменами (xCas9), узнающие PAM NG, NNG, GAA, GAT и CAA (xCas9-3.6 E108G S217A A262T S409I E480K E543D M694I E1219V и xCas9-3.7 A262T R324L S409I E480K E543D M694I E1219V), дополнительно проявляли и в 10–100 раз более высокую специфичность к мишени в клеточных линиях HEK-293T и U2OS [20].
ТОЧНОСТЬ НУКЛЕАЗ Cas ИЗ ДРУГИХ ВИДОВ БАКТЕРИЙ
Помимо SpCas9, достаточно детально исследованы некоторые РНК-адресуемые CRISPR-ассоциированные эндонуклеазы из других микроорганизмов, принадлежащих к различным типам системы CRISPR. В числе этих ферментов удалось обнаружить нуклеазы с высокой специфичностью и с расширенным спектром РАМ. В пределах системы CRISPR типа II интерес для практического геномного редактирования представляют ферменты из Staphylococcus aureus (SaCas9), Francisella novicida (FnCas9) и Neisseria meningitidis (NmCas9). SaCas9 привлекает небольшим размером (примерно на 25% короче, чем SpCas9) и повышенным в сравнении с SpCas9 числом оборотов в каталитической реакции [76, 77]. Специфичность действия SaCas9 в клетках человека зависит от длины gРНК: она несколько ниже, чем у SpCas9, с оптимальной для активности gРНК (21–23 н.), но при укорочении gРНК до 20 н. значительно повышается из-за снижения толерантности к мисматчам [76, 78‒80]. Методами рационального дизайна были получены варианты SaCas9 с повышенной точностью, аналогичные SpCas9-HF1 [81]. Активность FnCas9 составляет ~70% активности SpCas9, а специфичность в несколько раз выше [82, 83]. Однако по неясным до конца причинам фермент FnCas9 весьма чувствителен к структуре хроматина и неактивен по многим локусам в геноме человека [82]. Несколько меньшая активность и заметно более высокая точность по сравнению с SpCas9 характерна и для NmCas9 [84, 85].
Большой интерес вызывают также белки Cas12a (Cpf1) ‒ представители CRISPR-систем типа V (рис. 1б). В отличие от Cas9, они содержат только RuvC-подобный домен и для проявления эндонуклеазной активности требуют только достаточно короткой crРНК (~42 н.), причем длина протоспейсера составляет 23‒24 н. [86]. PAM находится с 5'-стороны от протоспейсера и имеет последовательность (Т)2–3N, а гидролиз дцДНК происходит по фосфодиэфирным связям между нуклеотидами 23–24 адресуемой цепи (считая от PAM) и 18–19 неадресуемой цепи, образуя липкие концы. Многие ферменты Cas12a неактивны при синтезе в клетках млекопитающих, хотя представители этого класса из Acidaminococcus sp. (AsCas12a) и Lachnospiraceae (LbCas12a) проявляют нуклеазную активность [86]. В клеточных линиях U2OS и HEK293 эффективность AsCas12a и LbCas12a была сравнима с таковой для Cas9, при этом 17 из 20 crРНК в сочетании с AsCas12a и 12 из 20 в случае LbCas12a не индуцировали неадресных мутаций [87, 88]. Специфичность к РАМ у AsCas12a выше, чем у LbCas12a. Систематический анализ мисматчей gРНК выявил, что система частично толерантна к единичным несоответствиям последовательности ДНК-мишени, но уже два мисматча практически полностью подавляют ее активность [87].
ВЛИЯНИЕ СТРУКТУРЫ НАПРАВЛЯЮЩЕЙ РНК НА ТОЧНОСТЬ РЕДАКТИРОВАНИЯ
Дизайн РНК-компонента системы CRISPR/ Cas9 также вносит заметный вклад в точность геномного редактирования. Так, специфичность редактирования значительно увеличивается при присоединении двух G на 5′-конец sgРНК (GG-X20): в клетках миелоидного лейкоза K562 sgРНК стандартного дизайна проявляла неадресную активность по всем 7 тестируемым сайтам, а sgРНК GG-X20 ‒ только по одному [13]. Сходное действие оказывает добавление структурированных участков, например G-квадруплексов, на 3′-конец sgРНК [89]. Укорочение sgРНК на 1–3 нуклеотида повышает специфичность, увеличивая чувствительность к мисматчам, и при этом незначительно снижает активность Cas9 [12, 90]. Для эффективного функционирования комплекса Cas9–sgРНК в клетках человека длина адресующей части РНК должна составлять минимум 17 н.: при этой длине ни активность системы, измеренная в процентах измененных клеток, ни соотношение HDR : NHEJ не отличаются от таковых для полноразмерной sgРНК (20 н.) [90]. В целом, укороченные sgРНК могут увеличивать специфичность системы более чем на три порядка в клетках человека [15, 90, 91] и дрожжей [92]. Интересно, что в системе in vitro из нескольких олигонуклеотидных библиотек sgРНК и ДНК-мишеней укороченные sgРНК (17–18 н.) и полноразмерная sgРНК (20 н.) обладают сходными профилями специфичности к мишеням [92]. Таким образом, структура хроматина, вероятно, также играет роль в точности редактирования. При использовании комбинации tracrРНК и синтетической crРНК вместо sgРНК наблюдаемый уровень неадресных изменений в клеточных линиях K562 и HeLa был низким [13].
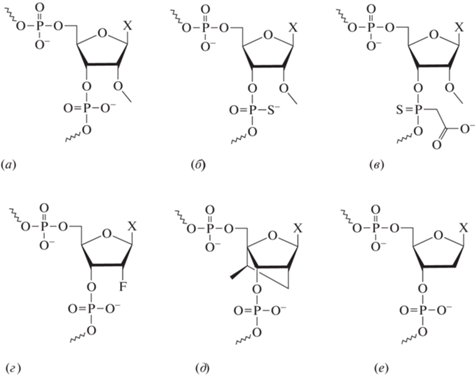
Введение химических модификаций в РНК (рис. 4) также может влиять на активность и специфичности системы CRISPR/Cas9. Первые работы в этом направлении имели своей целью повысить устойчивость gРНК к внутриклеточным нуклеазам. Например, редактирование линии клеток K562 с использованием sgРНК, в которой три 5'-концевых и три 3'-концевых позиции несли нуклеотиды, содержащие 2'-O-метил-3'-тиофосфат или 2'-O-метил-3'-тиофосфоноацетат, происходило значительно эффективнее, чем с немодифицированной sgРНК, хотя неадресная активность CRISPR-системы была немного выше [93]. Специфичность при этом возрастала в несколько раз, если Cas9 вводили в клетки не в виде экспрессионной плазмиды, а как рекомбинантный фермент в комплексе с sgРНК. Аналогичную стратегию использовали для системы crРНК/tracrРНК, в которой комбинированная модификация crРНК по ряду критических позиций остатками 2'-фтор-, 2'-O-метил- и закрытых (2'–4'-мостиковых) нуклеотидов позволила в несколько раз увеличить и активность, и специфичность редактирования в клетках HEK-293T [94]. Внедрение мостиковых нуклеиновых кислот в структуру gРНК снижает неадресную активность за счет ускорения динамических переходов между открытой и закрытой конформациями гетеродуплексов с мисматчами [95].
Рис. 4.
Некоторые модификации, вносимые в структуру gРНК. а ‒ 2'-O-метилрибонуклеотиды; б ‒ 2'-O-метилрибонуклеотиды с 3'-тиофосфатной связью; в ‒ 2'-O-метилрибонуклеотиды с 3'-тиофосфонацетатной связью; г ‒ 2'-фторнуклеотиды; д ‒ 2'-4'-мостиковые нуклеотиды; е ‒ 2'-дезоксирибонуклеотиды.
Много внимания было уделено редактированию при помощи гибридных направляющих нуклеиновых кислот, в которых сочетались остатки рибо- и дезоксирибонуклеотидов, что также позволяло повысить специфичность узнавания мишеней за счет меньшей энергии взаимодействия в парах dNMP:dNMP по сравнению с rNMP:dNMP [96‒98]. Одновременное использование нескольких типов модификаций в разных позициях, как правило, позволяет повысить эффективность редактирования за счет синергичных эффектов [98]. Разработка системы скрининга активных химически модифицированных crРНК и tracrРНК в совокупности с рациональным дизайном сайтов модификации с сохранением контактирующих с белком 2′-OH-групп в структуре комплекса Cas9–РНК–ДНК привела к созданию высокомодифицированных “улучшенных” (enhanced) sgРНК (e-sgРНК), в которых более половины всех рибонуклеотидов заменены на 2'-фтор, 2'-O-метил- или тиофосфатные производные [99, 100]. Такие e-sgРНК были успешно применены для редактирования гена Pcsk9 у мышей [99].
ВЛИЯНИЕ СИСТЕМ ДОСТАВКИ НА ТОЧНОСТЬ РЕДАКТИРОВАНИЯ
Для нокаута генов в эукариотическом геноме методом CRISPR/Cas9 в клетку необходимо доставить два компонента системы: Cas9 и sgРНК (либо Cas9, crРНК и tracrРНК). Для точной замены путем HDR дополнительно необходим донор для рекомбинации. Доставка компонентов возможна в виде кодирующей ДНК, РНК (мРНК Cas9 и sgРНК) либо рекомбинантного белка и РНК, синтезированной химическим или ферментативным способом.
В первых работах по редактированию генома клеток человека упор был сделан на кодирующие ДНК-конструкции, поскольку гомогенность синтетической РНК была недостаточной. Стандартные методы трансфекции до сих пор работают вполне удовлетворительно для решения многих исследовательских задач, однако для потенциальных терапевтических приложений и скрининга РНК-библиотек разработаны специализированные векторы на основе лентивирусов, аденовирусов и аденоассоциированных вирусов [101, 102]. Вне зависимости от способа трансфекции доставка внутрь клетки плазмид, кодирующих Cas9 и РНК, зачастую ведет к полной или частичной интеграции плазмидной ДНК в геном хозяина по целевому или нецелевым сайтам [103, 104]. Кроме того, продукция элементов системы с плазмидной матрицы поддерживается в течение нескольких суток, что повышает вероятность внесения в геном неадресных изменений [103]. Вследствие этого внутриклеточная доставка векторов, кодирующих элементы системы CRISPR/Cas9, на сегодня считается неприемлемой для терапевтического использования, а организмы, созданные с ее помощью, законодательством большинства стран рассматриваются как генетически модифицированные.
Усовершенствование методов синтеза РНК позволило непосредственно доставлять в клетку мРНК Cas9 и нужную sgРНК [103, 105]. Точность редактирования при таком способе доставки как минимум не уступает точности при доставке кодирующих конструкций [106, 107]. Замечено, что при этом совместная доставка мРНК Cas9 и немодифицированной sgРНК дает невысокую частоту событий редактирования, но химические модификации sgРНК позволяют в несколько раз увеличить как эффективность, так и специфичность [93].
Доставка в клетку готового RNP, сформированного из рекомбинантного белка Cas9 и нужной sgРНК, стала очень популярна из-за хороших показателей эффективности и специфичности редактирования. Так, электропорация или липофекция комплекса RNP в клетки человека позволяет повысить специфичность редактирования примерно на порядок по сравнению с трансфекцией экспрессирующими плазмидами [103, 105, 108]. Высокая специфичность, по всей вероятности, связана с тем, что время существования RNP в клетке составляет часы, в то время как экспрессия плазмидных генов может продолжаться несколько суток. Из менее стандартных подходов можно упомянуть метод доставки RNP в виде конъюгата с интернализуемыми пептидами разнообразной структуры, способными проникать в клетку по разным зависимым и независимым от эндоцитоза механизмам [109]. При конъюгации таких пептидов с Cas9 через тиоэфирную связь обработка клеток RNP позволяла достичь эффективности редактирования 3‒16% для разных клеточных линий, а специфичность по сравнению с плазмидным контролем повышалась в 2.2–4.1 раза [110]. Наконец, весьма многообещающей оказалась доставка с помощью золотых наночастиц, конъюгированных с ДНК и покрытых катионным полимером поли-{N-[N-(2-аминоэтил)-2-аминоэтил]аспартамидом}, способствующим интернализации [111]. Эффективность HDR на панели трансформированных и первичных клеток человека и мыши составила 3–6%, а in vivo на мышиной модели миодистрофии Дюшенна при внутримышечном введении удалось достичь эффективности редактирования ~5.4% для гена в мышечных клетках при неадресных событиях редактирования по 21 вероятному сайту в диапазоне 0.005–0.2% [111].
УСЛОВИЯ ВНУТРИКЛЕТОЧНОЙ ЭКСПРЕССИИ КОМПОНЕНТОВ СИСТЕМЫ CRISPR/Cas9
Длительное присутствие компонентов системы CRISPR/Cas9 внутри клеток и их высокая концентрация способствуют неадресному редактированию. Одной из первых идей для достижения оптимального соотношения между эффективностью и специфичностью системы стало применение индуцируемых промоторов для экспрессии Cas9 (iCas9) [112‒114]. Для строгой проверки специфичности одной из таких систем на панели человеческих клеток (293T, HeLa и SK-BR-3) было исследовано редактирование системой iCas9 с sgРНК (полностью комплементарных или с мисматчем), нацеленными на гены KDM5C, EMX1 и VEGFA. По сравнению с нерегулируемой продукцией индуцированная экспрессия Cas9 приводила к задержке редактирования не полностью комплементарных мишеней на несколько десятков часов, в то время как кинетика редактирования точных мишеней была сходна для обоих способов экспрессии, а процент неадресных изменений для всех локусов снижался на порядок и более [114]. В лабораторных исследованиях оказался полезным подход со стабильной интеграцией гена белка Cas9 в хромосому под контролем индуцируемого промотора, что далее позволяет проводить модификации с доставкой лишь sgРНК и, при необходимости, донора для рекомбинации [112, 115]. При реализации такой схемы в плюрипотентных стволовых клетках человека уровень модификации по неадресным сайтам был ниже порога детекции [112].
Внутриклеточную активность Cas9 можно контролировать и на посттрансляционном уровне, например, ограничивая время существования активного фермента. Так, в системе split-Cas9 С‑ и N-концевой домены нуклеазы синтезируются раздельно в виде химерных полипептидов с белком FKBP и FRB-доменом белка mTOR, которые могут димеризоваться в присутствии рапамицина [116]. Однако самопроизвольная димеризация Cas9 не позволяет полностью свести на нет активность такого комплекса, хотя удалось снизить неадресную модификацию до уровня 4–20% от Cas9 [116]. Использование в аналогичной системе N- и C‑концевого фрагментов Cas9, соединенных с фотодимеризуемыми Magnet-доменами, полученными из фоторецептора VVD Neurospora, позволило снизить фоновый уровень событий редактирования до недетектируемого [117]. В качестве еще одного метода посттрансляционного контроля в последовательность Cas9 включали 4-гидрокситамоксифензависимый интеин ‒ домен, способный в определенных условиях катализировать собственное удаление из белка [118]. При редактировании локусов EMX, VEGFA и CLTA в культуре клеток HEK293 в присутствии индуктора эффективность была сравнима с таковой для Сas9, а специфичность увеличена в 25 раз [118].
Ограничить активность Cas9 можно за счет экспрессии вместе с двумя sgРНК: нацеленной на заданный локус и направленной против самого Cas9. Таким способом удалось значительно сократить период активной экспрессии Cas9 и повысить специфичность редактирования в 4.0–7.5 раз [119, 120]. Наконец, при введении замен путем HDR можно включить в состав донора генетического материала фенотипически нейтральную замену, которая в случае удачной рекомбинации ликвидирует PAM или изменяет затравочную последовательность, гибридизующуюся с gРНК. Такой подход позволил увеличить точность редактирования локусов APP и PSEN1 в индуцированных плюрипотентных стволовых клетках и в линии HEK293 в 2–10 раз [121, 122].
Наконец, в связи с сопряжением механизмов репарации двухцепочечных разрывов с фазами клеточного цикла рассматривают возможность его синхронизации с редактированием. В качестве основной системы доставки в этом случае предпочтителен RNP, так как его действие начинается сразу после введения в клетки. При трансфекции RNP синхронизация клеток HEK-293T, первичных фибробластов человека и эмбриональных стволовых клеток человека нокодазолом на границе фаз G2/M повышала частоту событий HDR примерно в 3 раза, а уровень неадресных событий, определенных полногеномным секвенированием, не превышал фонового [123]. При редактировании гетерозиготных мутаций в зиготе человека введение RNP и ДНК-донора рекомбинации в S-фазе позволяло добиться преимущественного использования экзогенной ДНК, а не хромосомной копии как матрицы для рекомбинации [124].
Таким образом, в настоящее время активно исследуют несколько разных путей повышения точности геномного редактирования, основанного на комплементарной адресации, то есть в большинстве случаев ‒ на системе CRISPR/Cas9. Позволит ли какой-то из этих путей или какая-либо их комбинация достичь точности, приемлемой для терапевтического применения геномного редактирования, ‒ вопрос, который зависит не только от манипуляций с системами редактирования, но и от точности методов определения нецелевых событий. Современные методы высокопроизводительного секвенирования позволяют детектировать небольшие изменения в геноме лучше, чем крупные перестройки, и дискуссии о вероятности крупных перестроек как нецелевых событий редактирования не утихают до сих пор [125‒127]. Ситуация, возможно, изменится с массовым внедрением нанопорного секвенирования, которое дает гораздо более длинные участки прочтения. В любом случае приемлемая безопасность технологии при применении in vivo может быть достигнута только при уровне неадресных изменений (причем для любой gРНК), сравнимом с фоновым уровнем соматического мутагенеза. Общепринятое значение точности репликации в нераковых клетках человека в культуре составляет ~10−10 мутаций на пару нуклеотидов на одно клеточное деление [1], что достаточно хорошо совпадает с недавно полученными оценками в экспериментах по секвенированию единичных клеток клональных линий из разных тканей [128‒130]. Такая точность остается пока недостижимой даже для “сверхточных” вариантов Cas9. Для терапевтических манипуляций ex vivo (например, при редактировании с последующей аутотрансплантацией) точность действия системы редактирования может быть несколько ниже, но это компенсируется необходимостью полногеномного секвенирования для поиска клонов, содержащих только адресные мутации, и чем ниже точность системы, тем больше нужно попыток секвенирования. В целом, повышение точности останется в ближайшие годы одним из главных направлений исследований в области геномного редактирования.
Работа поддержана грантом Российского научного фонда 21-64-00017. Д.В. Ким выражает благодарность Российскому фонду фундаментальных исследований (проект 20-34-90092-Аспиранты; анализ клеточных механизмов специфичности). Д.О. Жарков выражает благодарность Министерству высшего образования и науки Российской Федерации (государственное задание ИХБФМ СО РАН № 121031300056-8; структурный анализ специфичности ферментов).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kunkel T.A. (2009) Evolving views of DNA replication (in)fidelity. Cold Spring Harb. Symp. Quant. Biol. 74, 91‒101.
Supek F., Lehner B. (2019) Scales and mechanisms of somatic mutation rate variation across the human genome. DNA Repair. 81, 102647.
Balmain A. (2020) The critical roles of somatic mutations and environmental tumor-promoting agents in cancer risk. Nat. Genet. 52, 1139‒1143.
Wright A.V., Nuñez J.K., Doudna J.A. (2016) Biology and applications of CRISPR systems: Harnessing Nature’s toolbox for genome engineering. Cell. 164, 29‒44.
Jiang F., Doudna J.A. (2017) CRISPR–Cas9 structures and mechanisms. Annu. Rev. Biophys. 46, 505‒529.
Ceccaldi R., Rondinelli B., D’Andrea A.D. (2016) Repair pathway choices and consequences at the double-strand break. Trends Cell Biol. 26, 52‒64.
Xue C., Greene E.C. (2021) DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37, 639‒656.
Cradick T.J., Fine E.J., Antico C.J., Bao G. (2013) CRISPR/Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity. Nucleic Acids Res. 41, 9584‒9592.
Fu Y., Foden J.A., Khayter C., Maeder M.L., Reyon D., Joung J.K., Sander J.D. (2013) High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822‒826.
Hsu P.D., Scott D.A., Weinstein J.A., Ran F.A., Konermann S., Agarwala V., Li Y., Fine E.J., Wu X., Shalem O., Cradick T.J., Marraffini L.A., Bao G., Zhang F. (2013) DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827‒832.
Kuscu C., Arslan S., Singh R., Thorpe J., Adli M. (2014) Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat. Biotechnol. 32, 677‒683.
Pattanayak V., Lin S., Guilinger J.P., Ma E., Doudna J.A., Liu D.R. (2013) High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat. Biotechnol. 31, 839‒843.
Cho S.W., Kim S., Kim Y., Kweon J., Kim H.S., Bae S., Kim J.-S. (2014) Analysis of off-target effects of CRISPR-/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24, 132‒141.
Ivanov I.E., Wright A.V., Cofsky J.C., Palacio Aris K.D., Doudna J.A., Bryant Z. (2020) Cas9 interrogates DNA in discrete steps modulated by mismatches and supercoiling. Proc. Natl. Acad. Sci. USA. 117, 5853‒5860.
Tsai S.Q., Zheng Z., Nguyen N.T., Liebers M., Topkar V.V., Thapar V., Wyvekens N., Khayter C., Iafrate A.J., Le L.P., Aryee M.J., Joung J.K. (2015) GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat. Biotechnol. 33, 187‒197.
Bravo J.P.K., Liu M.-S., Hibshman G.N., Dangerfield T.L., Jung K., McCool R.S., Johnson K.A., Taylor D.W. (2022) Structural basis for mismatch surveillance by CRISPR–Cas9. Nature. 603, 343‒347.
Cofsky J.C., Soczek K.M., Knott G.J., Nogales E., Doudna J.A. (2022) CRISPR–Cas9 bends and twists DNA to read its sequence. Nat. Struct. Mol. Biol. 29, 395‒402.
Lin Y., Cradick T.J., Brown M.T., Deshmukh H., Ranjan P., Sarode N., Wile B.M., Vertino P.M., Stewart F.J., Bao G. (2014) CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Res. 42, 7473‒7485.
Zhang Y., Ge X., Yang F., Zhang L., Zheng J., Tan X., Jin Z.-B., Qu J., Gu F. (2014) Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells. Sci. Rep. 4, 5405.
Hu J.H., Miller S.M., Geurts M.H., Tang W., Chen L., Sun N., Zeina C.M., Gao X., Rees H.A., Lin Z., Liu D.R. (2018) Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556, 57‒63.
Kim N., Kim H.K., Lee S., Seo J.H., Choi J.W., Park J., Min S., Yoon S., Cho S.-R., Kim H.H. (2020) Prediction of the sequence-specific cleavage activity of Cas9 variants. Nat. Biotechnol. 38, 1328‒1336.
Wang T., Wei J.J., Sabatini D.M., Lander E.S. (2014) Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343, 80‒84.
Wang T., Birsoy K., Hughes N.W., Krupczak K.M., Post Y., Wei J.J., Lander E.S., Sabatini D.M. (2015) Identification and characterization of essential genes in the human genome. Science. 350, 1096‒1101.
Wang T., Yu H., Hughes N.W., Liu B., Kendirli A., Klein K., Chen W.W., Lander E.S., Sabatini D.M. (2017) Gene essentiality profiling reveals gene networks and synthetic lethal interactions with oncogenic Ras. Cell. 168, 890‒903.e15.
Park R.J., Wang T., Koundakjian D., Hultquist J.F., Lamothe-Molina P., Monel B., Schumann K., Yu H., Krupzcak K.M., Garcia-Beltran W., Piechocka-Trocha A., Krogan N.J., Marson A., Sabatini D.M., Lander E.S., Hacohen N., Walker B.D. (2017) A genome-wide CRISPR screen identifies a restricted set of HIV host dependency factors. Nat. Genet. 49, 193‒203.
Zhang D., Hurst T., Duan D., Chen S.-J. (2019) Unified energetics analysis unravels SpCas9 cleavage activity for optimal gRNA design. Proc. Natl. Acad. Sci. USA. 116, 8693‒8698.
Newman A., Starrs L., Burgio G. (2020) Cas9 cuts and consequences; detecting, predicting, and mitigating CRISPR/Cas9 on- and off-target damage: techniques for detecting, predicting, and mitigating the on- and off-target effects of Cas9 editing. Bioessays. 42, 2000047.
Li C., Chu W., Gill R.A., Sang S., Shi Y., Hu X., Yang Y., Zaman Q.U., Zhang B. (2022) Computational tools and resources for CRISPR/Cas genome editing. Genomics Proteomics Bioinformatics. S1672-0229(22)00027-4. https://doi.org/10.1016/j.gpb.2022.02.006
Kim D., Bae S., Park J., Kim E., Kim S., Yu H.R., Hwang J., Kim J.-I., Kim J.-S. (2015) Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat. Methods. 12, 237‒243.
Crosetto N., Mitra A., Silva M.J., Bienko M., Dojer N., Wang Q., Karaca E., Chiarle R., Skrzypczak M., Ginalski K., Pasero P., Rowicka M., Dikic I. (2013) Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat. Methods. 10, 361‒365.
Frock R.L., Hu J., Meyers R.M., Ho Y.-J., Kii E., Alt F.W. (2015) Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases. Nat. Biotechnol. 33, 179‒186.
Cameron P., Fuller C.K., Donohoue P.D., Jones B.N., Thompson M.S., Carter M.M., Gradia S., Vidal B., Garner E., Slorach E.M., Lau E., Banh L.M., Lied A.M., Edwards L.S., Settle A.H., Capurso D., Llaca V., Deschamps S., Cigan M., Young J.K., May A.P. (2017) Mapping the genomic landscape of CRISPR–Cas9 cleavage. Nat. Methods. 14, 600‒606.
Tsai S.Q., Nguyen N.T., Malagon-Lopez J., Topkar V.V., Aryee M.J., Joung J.K. (2017) CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR–Cas9 nuclease off-targets. Nat. Methods. 14, 607‒614.
Lazzarotto C.R., Malinin N.L., Li Y., Zhang R., Yang Y., Lee G., Cowley E., He Y., Lan X., Jividen K., Katta V., Kolmakova N.G., Petersen C.T., Qi Q., Strelcov E., Maragh S., Krenciute G., Ma J., Cheng Y., Tsai S.Q. (2020) CHANGE-seq reveals genetic and epigenetic effects on CRISPR–Cas9 genome-wide activity. Nat. Biotechnol. 38, 1317‒1327.
Listgarten J., Weinstein M., Kleinstiver B.P., Sousa A.A., Joung J.K., Crawford J., Gao K., Hoang L., Elibol M., Doench J.G., Fusi N. (2018) Prediction of off-target activities for the end-to-end design of CRISPR guide RNAs. Nat. Biomed. Eng. 2, 38‒47.
Kirillov B., Savitskaya E., Panov M., Ogurtsov A.Y., Shabalina S.A., Koonin E.V., Severinov K.V. (2022) Uncertainty-aware and interpretable evaluation of Cas9–gRNA and Cas12a‒gRNA specificity for fully matched and partially mismatched targets with Deep Kernel Learning. Nucleic Acids Res. 50, e11.
Bortesi L., Zhu C., Zischewski J., Perez L., Bassié L., Nadi R., Forni G., Lade S.B., Soto E., Jin X., Medina V., Villorbina G., Muñoz P., Farré G., Fischer R., Twyman R.M., Capell T., Christou P., Schillberg S. (2016) Patterns of CRISPR/Cas9 activity in plants, animals and microbes. Plant Biotechnol. J. 14, 2203‒2216.
Doench J.G., Fusi N., Sullender M., Hegde M., Vaimberg E.W., Donovan K.F., Smith I., Tothova Z., Wilen C., Orchard R., Virgin H.W., Listgarten J., Root D.E. (2016) Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 34, 184‒191.
Liang S.-Q., Liu P., Smith J.L., Mintzer E., Maitland S., Dong X., Yang Q., Lee J., Haynes C.M., Zhu L.J., Watts J.K., Sontheimer E.J., Wolfe S.A., Xue W. (2022) Genome-wide detection of CRISPR editing in vivo using GUIDE-tag. Nat. Commun. 13, 437.
Uribe-Salazar J.M., Kaya G., Sekar A., Weyenberg K., Ingamells C., Dennis M.Y. (2022) Evaluation of CRISPR gene-editing tools in zebrafish. BMC Genomics. 23, 12.
Zirin J., Bosch J., Viswanatha R., Mohr S.E., Perrimon N. (2022) State-of-the-art CRISPR for in vivo and cell-based studies in Drosophila. Trends Genet. 38, 437‒453.
Wu X., Scott D.A., Kriz A.J., Chiu A.C., Hsu P.D., Dadon D.B., Cheng A.W., Trevino A.E., Konermann S., Chen S., Jaenisch R., Zhang F., Sharp P.A. (2014) Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 32, 670‒676.
O’Geen H., Henry I.M., Bhakta M.S., Meckler J.F., Segal D.J. (2015) A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res. 43, 3389‒3404.
Boyle E.A., Andreasson J.O.L., Chircus L.M., Sternberg S.H., Wu M.J., Guegler C.K., Doudna J.A., Greenleaf W.J. (2017) High-throughput biochemical profiling reveals sequence determinants of dCas9 off-target binding and unbinding. Proc. Natl. Acad. Sci. USA. 114, 5461‒5466.
Qi L.S., Larson M.H., Gilbert L.A., Doudna J.A., Weissman J.S., Arkin A.P., Lim W.A. (2013) Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173‒1183.
Komor A.C., Kim Y.B., Packer M.S., Zuris J.A., Liu D.R. (2016) Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533, 420‒424.
Vojta A., Dobrinić P., Tadić V., Bočkor L., Korać P., Julg B., Klasić M., Zoldoš V. (2016) Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Res. 44, 5615‒5628.
Gaudelli N.M., Komor A.C., Rees H.A., Packer M.S., Badran A.H., Bryson D.I., Liu D.R. (2017) Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551, 464‒471.
Anzalone A.V., Randolph P.B., Davis J.R., Sousa A.A., Koblan L.W., Levy J.M., Chen P.J., Wilson C., Newby G.A., Raguram A., Liu D.R. (2019) Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576, 149‒157.
Arbab M., Shen M.W., Mok B., Wilson C., Matuszek Ż., Cassa C.A., Liu D.R. (2020) Determinants of base editing outcomes from target library analysis and machine learning. Cell. 182, 463‒480.e30.
Kim Y.-G., Cha J., Chandrasegaran S. (1996) Hybrid restriction enzymes: zinc finger fusions to FokI cleavage domain. Proc. Natl. Acad. Sci. USA. 93, 1156‒1160.
Ran F.A., Hsu P.D., Lin C.-Y., Gootenberg J.S., Konermann S., Trevino A.E., Scott D.A., Inoue A., Matoba S., Zhang Y., Zhang F. (2013) Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell. 154, 1380‒1389.
Chiang T.-W.W., le Sage C., Larrieu D., Demir M., Jackson S.P. (2016) CRISPR-Cas9D10A nickase-based genotypic and phenotypic screening to enhance genome editing. Sci. Rep. 6, 24356.
Gopalappa R., Suresh B., Ramakrishna S., Kim H.H. (2018) Paired D10A Cas9 nickases are sometimes more efficient than individual nucleases for gene disruption. Nucleic Acids Res. 46, e71.
Gao Y., Wu H., Wang Y., Liu X., Chen L., Li Q., Cui C., Liu X., Zhang J., Zhang Y. (2017) Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects. Genome Biol. 18, 13.
Guilinger J.P., Thompson D.B., Liu D.R. (2014) Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat. Biotechnol. 32, 577‒582.
Tsai S.Q., Wyvekens N., Khayter C., Foden J.A., Thapar V., Reyon D., Goodwin M.J., Aryee M.J., Joung J.K. (2014) Dimeric CRISPR RNA-guided Fok-I nucleases for highly specific genome editing. Nat. Biotechnol. 32, 569‒576.
Wyvekens N., Topkar V.V., Khayter C., Joung J.K., Tsai S.Q. (2015) Dimeric CRISPR RNA-guided Fok-I-dCas9 nucleases directed by truncated gRNAs for highly specific genome editing. Hum. Gene Ther. 26, 425‒431.
Anders C., Niewoehner O., Duerst A., Jinek M. (2014) Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513, 569‒573.
Jinek M., Jiang F., Taylor D.W., Sternberg S.H., Kaya E., Ma E., Anders C., Hauer M., Zhou K., Lin S., Kaplan M., Iavarone A.T., Charpentier E., Nogales E., Doudna J.A. (2014) Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Sc-ience. 343, 1247997.
Nishimasu H., Ran F.A., Hsu P.D., Konermann S., Shehata S.I., Dohmae N., Ishitani R., Zhang F., Nureki O. (2014) Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156, 935‒949.
Jiang F., Zhou K., Ma L., Gressel S., Doudna J.A. (2015) A Cas9–guide RNA complex preorganized for target DNA recognition. Science. 348, 1477‒1481.
Jiang F., Taylor D.W., Chen J.S., Kornfeld J.E., Zhou K., Thompson A.J., Nogales E., Doudna J.A. (2016) Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science. 351, 867‒871.
Huai C., Li G., Yao R., Zhang Y., Cao M., Kong L., Jia C., Yuan H., Chen H., Lu D., Huang Q. (2017) Structural insights into DNA cleavage activation of CRISPR-Cas9 system. Nat. Commun. 8, 1375.
Chen W., Zhang H., Zhang Y., Wang Y., Gan J., Ji Q. (2019) Molecular basis for the PAM expansion and fidelity enhancement of an evolved Cas9 nuclease. PLoS Biol. 17, e3000496.
Guo M., Ren K., Zhu Y., Tang Z., Wang Y., Zhang B., Huang Z. (2019) Structural insights into a high fidelity variant of SpCas9. Cell Res. 29, 183‒192.
Zhu X., Clarke R., Puppala A.K., Chittori S., Merk A., Merrill B.J., Simonović M., Subramaniam S. (2019) Cryo-EM structures reveal coordinated domain motions that govern DNA cleavage by Cas9. Nat. Struct. Mol. Biol. 26, 679‒685.
East K.W., Newton J.C., Morzan U.N., Narkhede Y.B., Acharya A., Skeens E., Jogl G., Batista V.S., Palermo G., Lisi G.P. (2020) Allosteric motions of the CRISPR–Cas9 HNH nuclease probed by NMR and molecular dynamics. J. Am. Chem. Soc. 142, 1348‒1358.
Slaymaker I.M., Gao L., Zetsche B., Scott D.A., Yan W.X., Zhang F. (2016) Rationally engineered Cas9 nucleases with improved specificity. Science. 351, 84‒88.
Kleinstiver B.P., Pattanayak V., Prew M.S., Tsai S.Q., Nguyen N.T., Zheng Z., Joung J.K. (2016) High-fidelity CRISPR–Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529, 490‒495.
Chen J.S., Dagdas Y.S., Kleinstiver B.P., Welch M.M., Sousa A.A., Harrington L.B., Sternberg S.H., Joung J.K., Yildiz A., Doudna J.A. (2017) Enhanced proofreading governs CRISPR–Cas9 targeting accuracy. Nature. 550, 407‒410.
Casini A., Olivieri M., Petris G., Montagna C., Reginato G., Maule G., Lorenzin F., Prandi D., Romanel A., Demichelis F., Inga A., Cereseto A. (2018) A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nat. Biotechnol. 36, 265‒271.
Lee J.K., Jeong E., Lee J., Jung M., Shin E., Kim Y.-H., Lee K., Jung I., Kim D., Kim S., Kim J.-S. (2018) Directed evolution of CRISPR-Cas9 to increase its specificity. Nat. Commun. 9, 3048.
Vakulskas C.A., Dever D.P., Rettig G.R., Turk R., Jacobi A.M., Collingwood M.A., Bode N.M., McNeill M.S., Yan S., Camarena J., Lee C.M., Park S.H., Wiebking V., Bak R.O., Gomez-Ospina N., Pavel-Dinu M., Sun W., Bao G., Porteus M.H., Behlke M.A. (2018) A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nat. Med. 24, 1216‒1224.
Murugan K., Suresh S.K., Seetharam A.S., Severin A.J., Sashital D.G. (2021) Systematic in vitro specificity profiling reveals nicking defects in natural and engineered CRISPR–Cas9 variants. Nucleic Acids Res. 49, 4037‒4053.
Ran F.A., Cong L., Yan W.X., Scott D.A., Gootenberg J.S., Kriz A.J., Zetsche B., Shalem O., Wu X., Makarova K.S., Koonin E.V., Sharp P.A., Zhang F. (2015) In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520, 186‒191.
Yourik P., Fuchs R.T., Mabuchi M., Curcuru J.L., Robb G.B. (2019) Staphylococcus aureus Cas9 is a multiple-turnover enzyme. RNA. 25, 35‒44.
Friedland A.E., Baral R., Singhal P., Loveluck K., Shen S., Sanchez M., Marco E., Gotta G.M., Maeder M.L., Kennedy E.M., Kornepati A.V.R., Sousa A., Collins M.A., Jayaram H., Cullen B.R., Bumcrot D. (2015) Characterization of Staphylococcus aureus Cas9: a smaller Cas9 for all-in-one adeno-associated virus delivery and paired nickase applications. Genome Biol. 16, 257.
Tycko J., Barrera L.A., Huston N.C., Friedland A.E., Wu X., Gootenberg J.S., Abudayyeh O.O., Myer V.E., Wilson C.J., Hsu P.D. (2018) Pairwise library screen systematically interrogates Staphylococcus aureus Cas9 specificity in human cells. Nat. Commun. 9, 2962.
Huston N.C., Tycko J., Tillotson E.L., Wilson C.J., Myer V.E., Jayaram H., Steinberg B.E. (2019) Identification of guide-intrinsic determinants of Cas9 specificity. CRISPR J. 2, 172‒185.
Tan Y., Chu A.H.Y., Bao S., Hoang D.A., Kebede F.T., Xiong W., Ji M., Shi J., Zheng Z. (2019) Rationally engineered Staphylococcus aureus Cas9 nucleases with high genome-wide specificity. Proc. Natl. Acad. Sci. USA. 116, 20969‒20976.
Chen F., Ding X., Feng Y., Seebeck T., Jiang Y., Davis G.D. (2017) Targeted activation of diverse CRISPR-Cas systems for mammalian genome editing via proximal CRISPR targeting. Nat. Commun. 8, 14958.
Acharya S., Mishra A., Paul D., Ansari A.H., Azhar M., Kumar M., Rauthan R., Sharma N., Aich M., Sinha D., Sharma S., Jain S., Ray A., Jain S., Ramalingam S., Maiti S., Chakraborty D. (2019) Francisella novicida Cas9 interrogates genomic DNA with very high specificity and can be used for mammalian genome editing. Proc. Natl. Acad. Sci. USA. 116, 20959‒20968.
Lee C.M., Cradick T.J., Bao G. (2016) The Neisseria meningitidis CRISPR-Cas9 system enables specific genome editing in mammalian cells. Mol. Ther. 24, 645‒654.
Amrani N., Gao X.D., Liu P., Edraki A., Mir A., Ibraheim R., Gupta A., Sasaki K.E., Wu T., Donohoue P.D., Settle A.H., Lied A.M., McGovern K., Fuller C.K., Cameron P., Fazzio T.G., Zhu L.J., Wolfe S.A., Sontheimer E.J. (2018) NmeCas9 is an intrinsically high-fidelity genome-editing platform. Genome Biol. 19, 214.
Zetsche B., Gootenberg J.S., Abudayyeh O.O., Slaymaker I.M., Makarova K.S., Essletzbichler P., Volz S.E., Joung J., van der Oost J., Regev A., Koonin E.V., Zhang F. (2015) Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163, 759‒771.
Kim D., Kim J., Hur J.K., Been K.W., Yoon S.-h., Kim J.-S. (2016) Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nat. Biotechnol. 34, 863‒868.
Kleinstiver B.P., Tsai S.Q., Prew M.S., Nguyen N.T., Welch M.M., Lopez J.M., McCaw Z.R., Aryee M.J., Joung J.K. (2016) Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nat. Biotechnol. 34, 869‒874.
Nahar S., Sehgal P., Azhar M., Rai M., Singh A., Sivasubbu S., Chakraborty D., Maiti S. (2018) A G-quadruplex motif at the 3′ end of sgRNAs improves CRISPR–Cas9 based genome editing efficiency. Chem. Commun. 54, 2377‒2380.
Fu Y., Sander J.D., Reyon D., Cascio V.M., Joung J.K. (2014) Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat. Biotechnol. 32, 279‒284.
Morgens D.W., Wainberg M., Boyle E.A., Ursu O., Araya C.L., Tsui C.K., Haney M.S., Hess G.T., Han K., Jeng E.E., Li A., Snyder M.P., Greenleaf W.J., Kundaje A., Bassik M.C. (2017) Genome-scale measurement of off-target activity using Cas9 toxicity in high-throughput screens. Nat. Commun. 8, 15178.
Fu B.X.H., St. Onge R.P., Fire A.Z., Smith J.D. (2016) Distinct patterns of Cas9 mismatch tolerance in vitro and in vivo. Nucleic Acids Res. 44, 5365‒5377.
Hendel A., Bak R.O., Clark J.T., Kennedy A.B., Ryan D.E., Roy S., Steinfeld I., Lunstad B.D., Kaiser R.J., Wilkens A.B., Bacchetta R., Tsalenko A., Dellinger D., Bruhn L., Porteus M.H. (2015) Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells. Nat. Biotechnol. 33, 985‒989.
Rahdar M., McMahon M.A., Prakash T.P., Swayze E.E., Bennett C.F., Cleveland D.W. (2015) Synthetic CRISPR RNA-Cas9–guided genome editing in human cells. Proc. Natl. Acad. Sci. USA. 112, E7110‒E7117.
Cromwell C.R., Sung K., Park J., Krysler A.R., Jovel J., Kim S.K., Hubbard B.P. (2018) Incorporation of bridged nucleic acids into CRISPR RNAs improves Cas9 endonuclease specificity. Nat. Commun. 9, 1448.
Yin H., Song C.-Q., Suresh S., Kwan S.-Y., Wu Q., Walsh S., Ding J., Bogorad R.L., Zhu L.J., Wolfe S.A., Koteliansky V., Xue W., Langer R., Anderson D.G. (2018) Partial DNA-guided Cas9 enables genome edi-ting with reduced off-target activity. Nat. Chem. Bio-l. 14, 311‒316.
Rueda F.O., Bista M., Newton M.D., Goeppert A.U., Cuomo M.E., Gordon E., Kröner F., Read J.A., Wrigley J.D., Rueda D., Taylor B.J.M. (2017) Mapping the sugar dependency for rational generation of a DNA-RNA hybrid-guided Cas9 endonuclease. Nat. Commun. 8, 1610.
O’Reilly D., Kartje Z.J., Ageely E.A., Malek-Adamian E., Habibian M., Schofield A., Barkau C.L., Rohilla K.J., DeRossett L.B., Weigle A.T., Damha M.J., Gagnon K.T. (2019) Extensive CRISPR RNA modification reveals chemical compatibility and structure–activity relationships for Cas9 biochemical activity. Nucleic Acids Res. 47, 546‒558.
Yin H., Song C.-Q., Suresh S., Wu Q., Walsh S., Rhym L.H., Mintzer E., Bolukbasi M.F., Zhu L.J., Kauffman K., Mou H., Oberholzer A., Ding J., Kwan S.-Y., Bogorad R.L., Zatsepin T., Koteliansky V., Wolfe S.A., Xue W., Langer R., Anderson D.G. (2017) Structure-guided chemical modification of guide RNA enables potent non-viral in vivo genome editing. Nat. Biotechnol. 35, 1179‒1187.
Mir A., Alterman J.F., Hassler M.R., Debacker A.J., Hudgens E., Echeverria D., Brodsky M.H., Khvorova A., Watts J.K., Sontheimer E.J. (2018) Heavily and fully modified RNAs guide efficient SpyCas9-mediated genome editing. Nat. Commun. 9, 2641.
Nelson C.E., Gersbach C.A. (2016) Engineering delivery vehicles for genome editing. Annu. Rev. Chem. Biomol. Eng. 7, 637‒662.
Xu C.L., Ruan M.Z.C., Mahajan V.B., Tsang S.H. (2019) Viral delivery systems for CRISPR. Viruses. 11, 28.
Kim S., Kim D., Cho S.W., Kim J., Kim J.-S. (2014) Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24, 1012‒1019.
Hanlon K.S., Kleinstiver B.P., Garcia S.P., Zaborowski M.P., Volak A., Spirig S.E., Muller A., Sousa A.A., Tsai S.Q., Bengtsson N.E., Lööv C., Ingelsson M., Chamberlain J.S., Corey D.P., Aryee M.J., Joung J.K., Breakefield X.O., Maguire C.A., György B. (2019) High levels of AAV vector integration into CRISPR-induced DNA breaks. Nat. Commun. 10, 4439.
Liang X., Potter J., Kumar S., Zou Y., Quintanilla R., Sridharan M., Carte J., Chen W., Roark N., Ranganathan S., Ravinder N., Chesnut J.D. (2015) Rapid and highly efficient mammalian cell engineering via Cas9 protein transfection. J. Biotechnol. 208, 44‒53.
Wang H., Yang H., Shivalila C.S., Dawlaty M.M., Cheng A.W., Zhang F., Jaenisch R. (2013) One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910‒918.
Yang H., Wang H., Shivalila C.S., Cheng A.W., Shi L., Jaenisch R. (2013) One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370‒1379.
Zuris J.A., Thompson D.B., Shu Y., Guilinger J.P., Bessen J.L., Hu J.H., Maeder M.L., Joung J.K., Chen Z.-Y., Liu D.R. (2015) Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo. Nat. Biotechnol. 33, 73‒80.
Peraro L., Kritzer J.A. (2018) Emerging methods and design principles for cell-penetrant peptides. Angew. Chem. Int. Ed. 57, 11868‒11881.
Ramakrishna S., Dad A.-B.K., Beloor J., Gopalappa R., Lee S.-K., Kim H. (2014) Gene disruption by cell-penetrating peptide-mediated delivery of Cas9 protein and guide RNA. Genome Res. 24, 1020‒1027.
Lee K., Conboy M., Park H.M., Jiang F., Kim H.J., Dewitt M.A., Mackley V.A., Chang K., Rao A., Skinner C., Shobha T., Mehdipour M., Liu H., Huang W.-c., Lan F., Bray N.L., Li S., Corn J.E., Kataoka K., Doudna J.A., Conboy I., Murthy N. (2017) Nanoparticle delivery of Cas9 ribonucleoprotein and donor DNA in vivo induces homology-directed DNA repair. Nat. Biomed. Eng. 1, 889‒901.
González F., Zhu Z., Shi Z.-D., Lelli K., Verma N., Li Q.V., Huangfu D. (2014) An iCRISPR platform for rapid, multiplexable, and inducible genome editing in human pluripotent stem cells. Cell Stem Cell. 15, 215‒226.
Dow L.E., Fisher J., O’Rourke K.P., Muley A., Kastenhuber E.R., Livshits G., Tschaharganeh D.F., Socci N.D., Lowe S.W. (2015) Inducible in vivo genome editing with CRISPR-Cas9. Nat. Biotechnol. 33, 390‒394.
Cao J., Wu L., Zhang S.-M., Lu M., Cheung W.K.C., Cai W., Gale M., Xu Q., Yan Q. (2016) An easy and efficient inducible CRISPR/Cas9 platform with improved specificity for multiple gene targeting. Nucleic Acids Res. 44, e149.
Castaño J., Bueno C., Jiménez-Delgado S., Roca-Ho H., Fraga M.F., Fernandez A.F., Nakanishi M., Torres-Ruiz R., Rodríguez-Perales S., Menéndez P. (2017) Generation and characterization of a human iPSC cell line expressing inducible Cas9 in the “safe harbor” AAVS1 locus. Stem Cell Res. 21, 137‒140.
Zetsche B., Volz S.E., Zhang F. (2015) A split-Cas9 architecture for inducible genome editing and transcription modulation. Nat. Biotechnol. 33, 139‒142.
Nihongaki Y., Kawano F., Nakajima T., Sato M. (2015) Photoactivatable CRISPR-Cas9 for optogenetic genome editing. Nat. Biotechnol. 33, 755‒760.
Davis K.M., Pattanayak V., Thompson D.B., Zuris J.A., Liu D.R. (2015) Small molecule–triggered Cas9 protein with improved genome-editing specificity. Nat. Chem. Biol. 11, 316‒318.
Chen Y., Liu X., Zhang Y., Wang H., Ying H., Liu M., Li D., Lui K.O., Ding Q. (2016) A self-restricted CRISPR system to reduce off-target effects. Mol. Ther. 24, 1508‒1510.
Petris G., Casini A., Montagna C., Lorenzin F., Prandi D., Romanel A., Zasso J., Conti L., Demichelis F., Cereseto A. (2017) Hit and go CAS9 delivered through a lentiviral based self-limiting circuit. Nat. Commun. 8, 15334.
Paquet D., Kwart D., Chen A., Sproul A., Jacob S., Teo S., Olsen K.M., Gregg A., Noggle S., Tessier-Lavigne M. (2016) Efficient introduction of specific homozygous and heterozygous mutations using CRISPR/Cas9. Nature. 533, 125‒129.
Kwart D., Paquet D., Teo S., Tessier-Lavigne M. (2017) Precise and efficient scarless genome editing in stem cells using CORRECT. Nat. Protoc. 12, 329‒354.
Lin S., Staahl B.T., Alla R.K., Doudna J.A. (2014) Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766.
Ma H., Marti-Gutierrez N., Park S.-W., Wu J., Lee Y., Suzuki K., Koski A., Ji D., Hayama T., Ahmed R., Darby H., Van Dyken C., Li Y., Kang E., Park A.-R., Kim D., Kim S.-T., Gong J., Gu Y., Xu X., Battaglia D., Krieg S.A., Lee D.M., Wu D.H., Wolf D.P., Heitner S.B., Izpisua Belmonte J.C., Amato P., Kim J.-S., Kaul S., Mitalipov S. (2017) Correction of a pathogenic gene mutation in human embryos. Nature. 548, 413‒419.
Kosicki M., Tomberg K., Bradley A. (2018) Repair of double-strand breaks induced by CRISPR–Cas9 leads to large deletions and complex rearrangements. Nat. Biotechnol. 36, 765‒771.
Iyer V., Boroviak K., Thomas M., Doe B., Riva L., Ryder E., Adams D.J. (2018) No unexpected CRISPR-Cas9 off-target activity revealed by trio sequencing of gene-edited mice. PLoS Genet. 14, e1007503.
Höijer I., Emmanouilidou A., Östlund R., van Schendel R., Bozorgpana S., Tijsterman M., Feuk L., Gyllensten U., den Hoed M., Ameur A. (2022) CRISPR-Cas9 induces large structural variants at on-target and off-target sites in vivo that segregate across generations. Nat. Commun. 13, 627.
Li R., Di L., Li J., Fan W., Liu Y., Guo W., Liu W., Liu L., Li Q., Chen L., Chen Y., Miao C., Liu H., Wang Y., Ma Y., Xu D., Lin D., Huang Y., Wang J., Bai F., Wu C. (2021) A body map of somatic mutagenesis in morphologically normal human tissues. Nature. 597, 398‒403.
Abascal F., Harvey L.M.R., Mitchell E., Lawson A.R.J., Lensing S.V., Ellis P., Russell A.J.C., Alcantara R.E., Baez-Ortega A., Wang Y., Kwa E.J., Lee-Six H., Cagan A., Coorens T.H.H., Spencer Chapman M., Olafsson S., Leonard S., Jones D., Machado H.E., Davies M., Øbro N.F., Mahubani K.T., Allinson K., Gerstung M., Saeb-Parsy K., Kent D.G., Laurenti E., Stratton M.R., Rahbari R., Campbell P.J., Osborne R.J., Martincorena I. (2021) Somatic mutation landscapes at single-molecule resolution. Nature. 593, 405‒410.
Cagan A., Baez-Ortega A., Brzozowska N., Abascal F., Coorens T.H.H., Sanders M.A., Lawson A.R.J., Harvey L.M.R., Bhosle S., Jones D., Alcantara R.E., Butler T.M., Hooks Y., Roberts K., Anderson E., Lunn S., Flach E., Spiro S., Januszczak I., Wrigglesworth E., Jenkins H., Dallas T., Masters N., Perkins M.W., Deaville R., Druce M., Bogeska R., Milsom M.D., Neumann B., Gorman F., Constantino-Casas F., Peachey L., Bochynska D., Smith E.S.J., Gerstung M., Campbell P.J., Murchison E.P., Stratton M.R., Martincorena I. (2022) Somatic mutation rates scale with lifespan across mammals. Nature. 604, 517‒524.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


