Молекулярная биология, 2023, T. 57, № 2, стр. 254-268
Поли(АDP-рибоза)полимеразы 1 и 2: классические функции и взаимодействие с HPF1 ‒ новым фактором поли(ADP-рибозил)ирования гистонов
Т. А. Кургина a, О. И. Лаврик a, b, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
b Новосибирский национальный исследовательский государственный университет
630090 Новосибирск, Россия
* E-mail: lavrik@niboch.nsc.ru
Поступила в редакцию 09.08.2022
После доработки 07.09.2022
Принята к публикации 07.09.2022
- EDN: EFQJGH
- DOI: 10.31857/S0026898423020167
Аннотация
Поли(ADP-рибоза) (PAR) – отрицательно заряженный полимер, линейный и разветвленный, состоящий из мономеров АDP-рибозы. Синтез этого полимера катализируют ферменты поли(ADP-рибоза)полимеразы (PARP) при активации на повреждениях ДНК, используя в качестве субстрата никотинамидадениндинуклеотид (NAD+). Наиболее изучены два члена семейства PARP: PARP1 и PARP2. Эти важнейшие ядерные белки участвуют во многих клеточных процессах, в том числе в регуляции репарации ДНК. PARP1 и PARP2 катализируют как синтез, так и перенос поли(ADP-рибозы) на аминокислотные остатки белков-мишеней, в том числе проводят эффективное ауто-PARилирование. Ввиду ключевой роли в регуляции процесса репарации ДНК PARP1 и PARP2 считают перспективными мишенями для химиотерапии. Недавно открыт новый фактор PARилирования гистонов ‒ HPF1, ‒ который модулирует активность PARP1/2, образуя временный совместный активный центр с PARP1/2. В присутствии HPF1 модификация гистонов происходит по остаткам серина. Общий механизм взаимодействия HPF1 с PARP1/2 в настоящее время интенсивно исследуют. В представленном обзоре мы рассматриваем открытие и классический механизм PARилирования у высших эукариот, а также роль в данном процессе HPF1 – нового фактора PARилирования гистонов.
ВВЕДЕНИЕ
Поли(ADP-рибозил)ирование (PARилирование) представляет собой посттрансляционную модификацию белков, катализируемую ферментами поли(ADP-рибоза)полимеразами (PARP). Эта посттрансляционная модификация регулирует ключевые клеточные процессы, такие как репарация, репликация ДНК, организация структуры хроматина, экспрессия генов, процессинг РНК, биогенез рибосом и трансляция [1–10]. Известно, что PARилирование регулирует как функции белков, так и их внутриклеточную локализацию, а также участвует в формировании немембранных клеточных компартментов [4, 11–13]. Таким образом, ферменты PARP и катализируемая ими реакция синтеза поли(ADP-рибозы) (PAR) и ADP-рибозилирования белков осуществляют ключевую регуляцию процессов жизнедеятельности клеток высших эукариот. Именно поэтому PARP1 ‒ первоочередная мишень, уничтожаемая клеткой в процессе апоптоза [14, 15].
Основные ферменты, катализирующие PARилирование в ядре и синтез протяженного полимера PAR, ‒ два ДНК-зависимых фермента семейства PARP: PARP1 и PARP2 [5, 16]. Функции PARP1 и PARP2 в регуляции репарации ДНК перекрываются [16]. Оба фермента вовлечены в процесс эксцизионной репарации оснований (base excision repair, BER) [17–19] и взаимодействуют с молекулами ДНК, имитирующими интермедиаты коротко- и длиннозаплаточного путей BER [20–24]. Были показаны и количественно оценены белок-белковые взаимодействия PARP1 и PARP2 с такими участниками процесса BER, как ДНК-полимераза β (Polβ), белок-1 из группы комплементации (XRCC1) и ДНК-лигазой III [18, 19, 25]. Показано влияние PARP1 и PARP2 на процесс BER с использованием синтетических ДНК-дуплексов, содержащих повреждения, а также нуклеосом [17–19, 26]. Кроме того, установлена роль PARP1 в регуляции процессов репарации двухцепочечных разрывов, а также процесса удаления из ДНК объемных повреждений [27–29].
В связи с ключевой ролью в регуляции репарации ДНК и других клеточных процессов PARP1 и PARP2 считают перспективными мишенями для поиска новых противораковых препаратов, а также лекарств для борьбы с нейродегенеративными заболеваниями [30–34]. Следует отметить, что только число обзоров, касающихся разработки ингибиторов PARP как потенциальных лекарственных средств, за 2021 год составило 365, а число статей по этой теме за тот же период превысило 1400. Ежегодно проводятся международные симпозиумы, посвященные исследованиям ферментов PARP и реакции ADP-рибозилирования в связи с бурным развитием в мире этого направления исследований и их исключительной важности для медицины. На данный момент многие ингибиторы PARP1 находятся в стадии клинических и доклинических испытаний, а олапариб, рукапариб, нирапариб и талазопариб уже используют в клинике для лечения онкологических заболеваний. Ингибиторы PARP1 наиболее интенсивно используют как химиотерапевтические препараты при лечении рака яичников и молочной железы, преимущественно в случае мутации в генах BRCA1/2. Несколько ингибиторов PARP в настоящее время находятся в стадии доклинических и клинических испытаний в качестве препаратов для лечения других типов злокачественных опухолей, а также для противовоспалительной терапии инфаркта миокарда, инсультов и других заболеваний [30, 35, 36].
Несмотря на то, что история изучения синтеза PAR и ферментов PARP началась еще в 60-е годы XX века, совсем недавно обнаружен новый белковый кофермент PARP1 и PARP2 ‒ фактор PARилирования гистонов 1 (histone PARylation factor 1, HPF1) [37]. Этот белок регулирует активность и специфичность PARP1 и PARP2 и образует с ними временный совместный активный центр [38]. Роль такого взаимодействия заключается в переключении специфичности PARилирования с остатков аспартата, глутамата и других аминокислот на остатки серина [39]. HPF1 играет важную роль в катализируемом PARP1 и PARP2 PARилировании гистонов [40–42]. В настоящий момент механизм взаимодействия PARP1/PARP2 с HPF1 и функции этого механизма в контексте хроматина только начинают изучать и это направление исследований ферментов PARP вызывает большой интерес.
ИСТОРИЯ ИЗУЧЕНИЯ ПОЛИ(ADP-РИБОЗИЛ)ИРОВАНИЯ)
Первым указанием на процесс PARилирования стало открытие группы Поля Манделя (P. Mandel, P. Chambon, J.D. Weill), работавшей в Страсбурге (Франция), в 1963 г. [43]. Авторы показали, что включение [14C]-аденина из АТP в кислотонерастворимую фракцию препарата ядер из куриной печени усиливалось в 1000 раз в присутствии никотинамидмононуклеотида (NMN). Сначала было высказано предположение, что кислотонерастворимый продукт реакции – полиадениловая кислота, то есть поли(А). Однако впоследствии было установлено, что в этих условиях образуется PAR [44]. В это же время были начаты исследования PAR в Национальном онкологическом центре в Токио [45]. Японские ученые подтвердили результаты группы французских исследователей по NMN-индуцированному накоплению нерастворимого в кислоте полимера в печени крысы и, кроме того, в клетках гепатомы. Интересно, что уже в это время появилась гипотеза, что образование этого продукта связано с ростом раковых клеток. В результате биохимических исследований установили структуру полимера и “участников” его биосинтеза. Показано, что в ядре под действием NAD-пирофосфорилазы из NMN и ATP образуется NAD+, а фрагмент ADP-рибозы из NAD+ затем используется для синтеза PAR с высвобождением никотинамида [45]. Таким образом, первым импульсом для исследований PAR стала работа группы P. Mandel, опубликованная в 1963 году [43], которая получила развитие и в других лабораториях мира.
Долгое время PARP1 считали единственным ферментом, обладающим PARилирующей активностью в клетках млекопитающих [1]. Однако в течение нескольких лет интенсивных исследований идентифицировали пять различных генов, кодирующих другие поли- и моно(ADP-рибоза)полимеразы. В настоящее время семейство PARP включает 17 различных белков [46–49].
Установлено, что PAR представляет собой линейный и разветвленный полимер, состоящий из ADP-рибозных звеньев, связанных гликозидными связями [16]. Для синтеза PAR необходима молекула NAD+ в качестве предшественника и непосредственного субстрата PARP. Конститутивные уровни PAR обычно довольно низки в нестимулированных клетках [50]. Однако в ответ на генотоксический стресс (то есть при появлении разрывов в ДНК) активность PARP и уровни PAR могут возрасти в 10‒500 раз. Структура PAR хорошо охарактеризована. В полимере PAR ADP-рибозные звенья связаны гликозидными рибозо-рибозными 1'-2'-связями. Длина цепи полимеров неоднородна и может достигать 200‒400 мономеров in vitro и in vivo [51, 52]. Большинство свободных или связанных с белком полимеров (ADP-рибозы), синтезируемых при генотоксическом стрессе, быстро гидролизуется под действием поли(ADP-рибоза)гликогидролазы (PARG) с периодом полураспада от 40 с до 6 мин in vivo, что указывает на динамическую природу этого процесса в живых клетках [53]. Эффективный синтез и последующий быстрый гидролиз PAR определяют динамическую природу регуляции процессов, зависимых от PARP.
СТРУКТУРА ФЕРМЕНТОВ PARP1 И PARP2 И ОСОБЕННОСТИ ИХ ВЗАИМОДЕЙСТВИЯ С ДНК
Суперсемейство PARP охватывает по меньшей мере 17 ферментов, вовлеченных во множество биологических процессов, таких как регуляция транскрипции, репарации ДНК, репликации, клеточного цикла и других [10, 47, 48]. Представителей суперсемейства PARP объединяет наличие консервативного каталитического домена (CAT), который содержит высококонсервативную последовательность, называемую “PARP signature” и образующую активный центр ферментов семейства PARP [49, 54]. Некоторые представители PARP-семейства катализируют моно-, а не поли(ADP-рибозил)ирование [48]. Наиболее изученный член семейства PARP ‒ PARP1 ‒ служит сенсором повреждений ДНК, обычно вызванных ионизирующим излучением и окислительным стрессом, и инициирует рекрутирование соответствующих белков в места повреждения ДНК [55, 56]. PARP2 также был обнаружен как фермент, катализирующий синтез PAR в ядре [57]. Проводятся интенсивные исследования по выяснению роли PARP2 и его кооперации с PARP1 [10, 47, 58, 59].
Структура PARP1 достаточно консервативна – у различных видов позвоночных идентичность аминокислотной последовательности составляет около 62%. Белок PARP1 человека имеет молекулярную массу 113 кДа. Фермент состоит из трех структурно-функциональных доменов: N-концевой ДНК-связывающий домен (NTR-домен), сайт аутомодификации и С-концевой каталитический домен (рис. 1) [60, 61]. ДНК-связывающий домен включает так называемые “цинковые пальцы”: гомологичные ZnF1 и ZnF2 и отличающийся от них ZnF3. Мутационный анализ ZnF1 и ZnF2 выявил ключевую роль ZnF1 в ДНК-зависимой активности PARP1 in vitro, в то время как ZnF2 оказался несущественным, хотя может играть важную роль в связывания PARP1 с определенными повреждениями ДНК [62]. ZnF3 обеспечивает контакт разных доменов при организации функциональной структуры белка [63]. Кроме этого, NTR-домен содержит сигнал ядерной локализации (NLS), который имеет сайт протеолитического расщепления PARP1 каспазами в процессе апоптоза. В состав центральной области входит BRCT-мотив, участвующий в формировании белок-белковых контактов, и сайт аутомодификации, который содержит потенциальные акцепторы PAR: 15 остатков глутамата и три остатка серина [64–66]. С-концевой домен состоит из каталитического домена (ART), регуляторного спирального домена (HD) и домена WGR [61]. Именно каталитический домен почти полностью идентичен у различных видов [49].
Рис. 1.
Доменное строение PARP1 и PARP2. Функции доменов обозначены сверху и снизу от схемы; звездочкой отмечено три остатка серина, которые служат преимущественными мишенями PARилирования PARP1 in vivo.
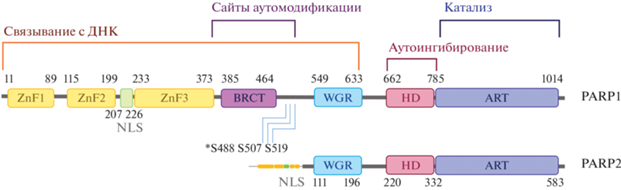
PARP2 имеет массу 66 кДа. Несмотря на значительную гомологию в структуре С-концевого каталитического домена, PARP1 и PARP2 значительно различаются по N-концевым участкам: в то время как PARP1 содержит три цинковых пальца (ZnF) и домен BRCT, NTR-домен PARP2 намного короче и его вторичная структура неупорядочена [61, 67, 68]. Такие структурные различия NTR влияют на взаимодействие ферментов с ДНК. PARP1 связывает поврежденную ДНК преимущественно цинковыми пальцами [61]. Домены WGR и BRCT также участвуют во взаимодействии с неповрежденной ДНК благодаря механизму “monkey barr”, который опосредует миграцию PARP1 по ДНК [65, 69]. PARP2 из-за отсутствия цинковых пальцев взаимодействует с ДНК-повреждениями доменами NTR и WGR [70]. Различия в доменной структуре и механизмах связывания с ДНК могут объяснить как различное сродство PARP1 и PARP2 к тем или иным повреждениям ДНК, так и отличающуюся каталитическую активность [20, 26, 71]. Кроме того, PARP1 и PARP2 имеют разное сродство к неповрежденной ДНК [71].
В экспериментах in vitro каталитическая активность фермента PARP2 слабее, чем PARP1, и синтезируемые им цепи PAR короче. In vitro PARP1 эффективно связывает ДНК и активируется широким спектром повреждений ДНК, в то время как PARP2, как предполагается, более специфично активируется ДНК, содержащей разрывы, фланкированные 5'-фосфатом (включая расщепленные апуриновые (АP) сайты) [70, 72–74]. В целом, сродство PARP1 к поврежденной, а также к неповрежденной ДНК выше, чем PARP2, но PARP2 с большей эффективностью, чем PARP1, связывает фланкированные 5'-фосфатами однонитевые разрывы ДНК [62, 75‒77]. PARP1 взаимодействует с ранними интермедиатами BER, тогда как PARP2 наиболее эффективно “работает” на однонитевых разрывах ‒ субстратах финальной стадии лигирования [56‒58, 61, 76].
НЕКОТОРЫЕ АСПЕКТЫ ФУНКЦИОНАЛЬНОЙ РОЛИ PARP1 И PARP2 В КЛЕТКЕ
В клетках человека в ответ на повреждение ДНК бóльшая часть активности PARP обеспечивается PARP1 (около 90%), в то время как на долю PARP2 приходится 10–15% [12]. Из-за гораздо более медленной аутомодификации PARP2 часто рассматривают как каталитически менее активный аналог PARP1. Примечательно, что, несмотря на существенно меньшую каталитическую активность PARP2, а также различное сродство PARP1 и PARP2 к повреждениям ДНК [71], нокаут PARP1 (как и нокаут PARP2) не летален для клеток, в то время как двойной нокаут этих ферментов вызывает эмбриональную летальность. Из-за генетической нестабильности клетки PARP1–/– и PARP2–/– обладают более высокой чувствительностью к агентам, повреждающим ДНК [78]. Более того, нокаут гена PARP2 на мышиных моделях приводит к дефектам в развитии Т-клеток [79], эритропоэза [80] и сперматогенеза [81], которые не проявляются у мышей, лишенных PARP1, что указывает на уникальные функции PARP2. Предполагается, что PARP2 компенсирует недостаток PARP1 в ответ на генотоксический стресс, хотя до сих пор непонятно, как это происходит при относительно меньшей активности PARP2 и различиях в сродстве к повреждениям ДНК у этих двух ферментов. Для PARP1 показана стимуляция активности РНК-связывающими белками, такими как YB1 [82, 83], SAM68 [84], и взаимодействие с Fus [85]. Для PARP2 взаимодействие с белками, которые модулируют его активность, только начинают изучать.
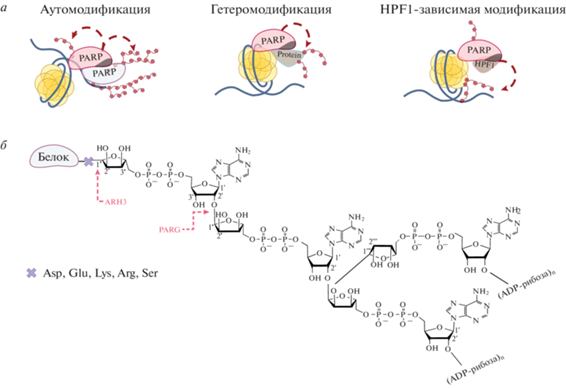
Поврежденная ДНК служит кофактором-активатором для PARP1 и PARP2 [61, 68, 70, 86, 87]. Связывание ДНК-повреждения индуцирует цепь междоменных перестроек PARP1 и PARP2, что в конечном итоге приводит к изменению конформации аутоингибиторного спирального домена (HD) [61, 86, 88, 89]. Этот домен закрывает собой активный центр PARP1/2. Его реорганизация каталитически активирует ферменты и облегчает связывание NAD+ в активном центре. Активированные PARP1 и PARP2 синтезируют и ковалентно присоединяют PAR к аминокислотным остаткам самих PARP и различных белков-мишеней. В этом контексте рассматривают реакции ауто-PARилирования (аутомодификация, при которой молекула PAR ковалентно присоединена к PARP) и гетеро-PARилирования (гетеромодификация, когда PAR присоединяется к другим белкам-мишеням) (рис. 2). В настоящий момент остается открытым вопрос о механизме ауто-PARилирования. С одной стороны, есть доказательства аутомодификации PARP1 по цис-механизму ‒ когда молекула фермента служит акцептором PAR, который она же и синтезирует [90, 91]. С другой стороны, сообщалось о способности PARP1 и PARP2 образовывать димеры, ускоряющие PARилирование в связи с вкладом транс-механизма модификации [92, 93]. Например, по данным светорассеяния, PARP1 и PARP2 в растворе находятся преимущественно в виде димеров [93]. Предположение о том, что аутомодификация PARP1 и PARP2 происходит межмолекулярным образом в составе димера, состоящего из двух субъединиц, согласуется со способностью PARP1 и PARP2 ковалентно модифицировать другие белки [52, 83]. В качестве дополнительного аргумента в пользу бимолекулярной природы реакции ауто-PARилирования можно привести стехиометрию комплексов PARP1 : ДНК и PARP2 : ДНК, которая составляет 2 : 1 для достижения оптимальной ферментативной активности [94, 95].
Рис. 2.
Структура возможных комплексов PARP (а) и структура PAR (б). PAR-модификация PARP (аутомодификация), белков-мишеней (гетеромодификация) и HPF1-зависимая модификация остатков серина. ARH3 ‒ ADP-рибозилгидролаза-3; PARG ‒ поли(ADP-рибоза)гликогидролаза.
Интенсивно изучалась роль PARP1 и PARP2 в механизмах BER и репарации одноцепочечных разрывов [17, 18, 23, 24, 26]. Функции PARP1 и PARP2 в регуляции этих процессов установлены с использованием модельных дуплексов ДНК, а также нуклеосом [17, 26, 72]. Сейчас мы знаем, что PARP1 и PARP2 могут выполнять как общие, так и специфические функции в поддержании стабильности генома, кооперируясь друг с другом. Возможно, что разделение функций PARP1 и PARP2 в различных процессах происходит в том числе при участии их белков-партнеров, поэтому открытие белковых коферментов PARP1 и PARP2 и изучение их функций представляет особенный интерес.
HPF1 ‒ НОВЫЙ КОФАКТОР ФЕРМЕНТОВ PARP1 И PARP2
Первоначально было установлено, что PARP1 и PARP2 модифицируют в белках преимущественно остатки глутамата и аспартата, а также лизина и аргинина [96–98]. Однако, недавно показано, что остатки серина также являются распространенными акцепторами PAR в клетках человека, в том числе в контексте репарации двунитевых разрывов [39–41]. Установлено, что PARP1 и PARP2 необходимы, но недостаточны для PARилирования остатков серина [42]. Фактор PARилирования гистонов-1 (HPF1) образует как с PARP1, так и с PARP2 временный непрочный комплекс, судя по высоким значениям констант диссоциации: Kd комплексов PARP1/2:HPF1:нуклеосома составляют 790 и 280 нМ соответственно [99, 100]. HPF1 формирует с этими ферментами совместный активный центр, который и модифицирует остатки серина [37]. Позже показали, что HPF1 человека специфически взаимодействует с PARP1 и PARP2, тем самым облегчая реакцию PARилирования гистонов (в отсутствие HPF1 PARP1/2 не модифицируют гистоны in vitro) [39].
Взаимодействие HPF1 с PARP1 усиливается присутствием ДНК и NAD+. Как описано выше, PARP1 и PARP2 имеют спиральный субдомен HD, который быстро разворачивается при распознавании повреждения ДНК, открывая сайт связывания NAD+ [87]. Удаление HD усиливает взаимодействие HPF1:PARP1/2 in vitro [38]. Предполагается, что этот субдомен ингибирует связывание ферментов с HPF1, а для реализации их взаимодействия необходимо ДНК-индуцированное развертывание HD.
Недавно методом рентгеноструктурного анализа и криоэлектронной микроскопии разрешена структура HPF1 в комплексе с каталитическим доменом PARP2 [38, 101]. Обнаружено, что для взаимодействия HPF1 с PARP1/2 важен консервативный остаток Asp283 в С-концевой области HPF1, который контактирует с His826 в PARP1 (His381 в PARP2). Важно отметить, что эти остатки гистидина в PARP1/2 критичны для элонгации реакции PARилирования и удлинения цепи PAR [102].
Структурный и мутационный анализ комплекса HPF1:PARP2 показал, что опосредованное HPF1 переключение аминокислотной специфичности PARP1/2 можно объяснить введением в общий активный центр каталитического остатка Glu284 HPF1 [38]. PARP1 и PARP2 содержат один каталитический остаток глутамата (Glu988 и Glu545 соответственно), каждый из которых, как было установлено, имеет решающее значение для инициации PARилирования [103]. Однако одного остатка глутамата в активных центрах PARP1 и PARP2 оказалось недостаточно для ADP-рибозилирования остатков серина [39]. Взаимодействие HPF1 и PARP1/2 располагает Glu284 HPF1 рядом с каталитическим глутаматом PARP1/2 и молекулой NAD+, что позволяет сформировать активный центр, способный катализировать эффективный перенос ADP-рибозы на серин. Мутантный HPF1 с заменой Glu284 на Ala теряет способность переключать аминокислотную специфичность PARP1/2 на остатки серина [38]. Депротонированный Glu284 HPF1 может действовать как основание в этой реакции, оттягивая протон от акцепторного остатка серина и тем самым повышая его нуклеофильность. По сути, HPF1 способствует увеличению нуклеофильности серина для эффективного катализа, как это происходит в активных центрах сериновых протеаз, эстераз и липаз, с той разницей, что в случае протеаз повышение нуклеофильности серина обеспечивает каталитическая триада Ser-His-Asp [104]. Заметим, что формирование объединенного активного центра, в котором присутствуют субстратсвязывающие и каталитические остатки PARP1/2 и HPF1, напоминает аналогичные функции белков, активирующих GТFазу, ‒ они тоже вводят необходимый для каталитиза аминокислотный остаток в активный центр фермента [38].
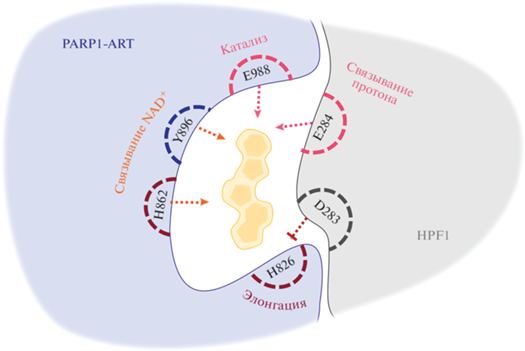
Интересно, что связывание с HPF1 приводит к образованию более коротких полимеров PAR [37]. Показано, что Asp283 HPF1 взаимодействует с His381 PARP2 (и аналогичным His826 PARP1) и занимает отрицательно заряженный связывающий карман, который во время реакции удлинения цепи PAR нужен для узнавания пирофосфатной группы акцепторной единицы ADP-рибозы (рис. 3) [38]. Аналогичные результаты получены в отношении His826 PARP1: мутант этого белка с реверсированием заряда в позиции 826 (His826Glu) терял способность формировать совместный активный центр с HPF1. Этот мутант катализировал PARилирование остатков аспартата и глутамата, но синтезировал только короткие полимеры PAR. Таким образом, связывание HPF1 с PARP1/2 блокирует ответственный за удлинение полимера остаток гистидина (His826 и His381 в активных центрах PARP1 и PARP2 соответственно) и исключает тем самым элонгацию PAR.
Рис. 3.
Схематическое изображение совместного активного центра PARP1 и HPF1 с описанием функций аминокислотных остатков. Триада H-Y-E (H826, Y896, E988) задействована в позиционировании NAD+ в ADP-рибозилтрансферазном центре (ART) и катализе. HPF1 предоставляет остаток глутамата (E284), обеспечивающий депротонирование остатка серина, что повышает его нуклеофильность. Остаток E988 PARP1 участвует в нуклеофильной атаке связи никотинамид-рибоза депротонированным остатком серина, завершая стадию инициации. В совместном активном центре остаток H826 PARP1, необходимый для элонгации, экранирован остатком D283 HPF1.
Недавно две группы исследователей, независимо друг от друга, пришли к выводу, что HPF1 стимулирует стадию инициации PARилирования [105, 106]. Обнаружено увеличение начальной скорости аутомодификации PARP1 и PARP2 и увеличение уровня синтезированной PAR в присутствии субэквимолярных концентраций HPF1. В то же время высокие (микромолярные) концентрации HPF1 ингибировали синтез PAR [105]. Это объясняется подавлением элонгации в ситуации, когда центр элонгации занят HPF1 при его высокой концентрации, соответствующей насыщающей [99, 100]. По всей видимости, при концентрации HPF1 ниже насыщающей, которая составляет 2‒5 мкМ, после инициации PARилирования в объединенном активном центре PARP:HPF1 HPF1 диссоциирует из комплекса с PARP в силу низкого сродства. Это подтверждено в экспериментах, в которых продемонстрирована элонгация синтеза PAR свободным PARP [64, 105]. Таким образом, насыщающие концентрации HPF1 затрудняют стадию элонгации в активном центре PARP, что выражается в снижении количества синтезируемого полимера, а низкие концентрации HPF1 стимулируют синтез PAR. Это очень интересный пример регуляции синтеза PAR.
C эффектами, оказываемыми HPF1 на синтез PAR, согласуется его влияние на NAD+-гидролазную активность PARP1 и PARP2. Способность PARP1 гидролизовать NAD+ без сопутствующего синтеза PAR обнаружена ранее и в отсутствие HPF1 [107]. HPF1 в высоких концентрациях по отношению к PARP1 (20-кратный избыток) стимулирует эту активность [102]. Авторы объясняют переключение PARP1 на гидролиз NAD+ с образованием свободной ADP-рибозы действием двух факторов: при насыщающих концентрациях HPF1 элонгация PAR невозможна, а когда сайты для инициации синтеза “исчерпаны”, PARP использует воду в качестве акцептора ADP-рибозы, гидролизуя NAD+. Однако нами показано, что в условиях, обеспечивающих стимуляцию PARилирования, происходит HPF1-зависимое уменьшение “холостого” гидролиза NAD+ [105]. По всей видимости, субэквимолярные концентрации HPF1 не насыщают все активные центры PARP. Свободные от HPF1 молекулы PARP могут как предоставлять сайты для PARилирования, так и катализировать элонгацию [105]. Следует отметить, что концентрация HPF1 в клетке значительно ниже концентрации PARP1 и сопоставима с концентрацией PARP2 [37]. Таким образом, относительная концентрация HPF1 гораздо ниже концентрации, необходимой для насыщения активных центров PARP1/2, в соответствии с найденными величинами констант диссоциации для комплексов PARP1/2:HPF1:нуклеосома (790 и 280 нМ соответственно) [99, 100]. Это обеспечивает оптимальные условия для стимуляции инициации без подавления элонгации и усиления гидролиза NAD+ и в очередной раз позволяет предположить, что в присутствии HPF1 аутомодификация PARP1/2 происходит в димере, при этом одна из субъединиц связана с HPF1 и образует временный совместный активный центр, а вторая служит акцептором PAR.
Таким образом, HPF1 оказывает противоположные эффекты на инициацию и элонгацию синтеза PAR. Стимуляция инициации, по всей видимости, опосредована введением дополнительного каталитического глутамата в активный центр фермента. Подавление элонгации связано с взаимодействием HPF1 с остатком His826 активного центра PARP1, важного для элонгации. В экспериментах in vitro и in vivo баланс между этими двумя эффектами преимущественно определяется соотношением концентраций HPF1:PARP [99, 100].
ПРИВЛЕЧЕНИЕ PARP1, PARP2 И HPF1 НА САЙТЫ ПОВРЕЖДЕНИЯ ДНК
На основании данных, полученных на начальных этапах изучения HPF1, стали считать, что этот белок вместе с PARP1 вовлечен в репарацию ДНК. Так, I. Gibbs-Seymour и соавт. [37] показали, что HPF1 ограничивает гипераутомодификацию PARP1 in vivo и in vitro и рекрутируется к местам повреждения ДНК. Нокаут HPF1 значительно повышает чувствительность клеток к обработке агентами, алкилирующими ДНК, такими как метилметансульфонат (MMS), и повышает чувствительность клеток к ингибированию PARP. Оказалось, что двойной нокаут HPF1‒/‒PARP1‒/‒ приводит к некоторому снижению чувствительности клеток к MMS и ингибиторам PARP по сравнению с одиночными нокаутами, то есть частично восстанавливает фенотип дикого типа [37].
Сообщалось, что после рекрутирования PARP1 в комплексе с HPF1 к участкам повреждения ДНК последний остается на поврежденных сайтах дольше, чем сам PARP1 [37, 38, 64]. В течение первых 30 с PARP1 рекрутируется в места повреждения. HPF1 привлекается на сайты повреждения вместе с PARP1, при этом динамика привлечения HPF1 не зависит от PARилирования и опосредуется, по всей видимости, белок-белковыми взаимодействиями [37]. В течение 2 мин PARP1, по всей видимости, покидает сайты повреждения [64], в то время как HPF1 находится там до 5 мин [37]. Обработка ингибиторами PARP1 приводит к задержке и PARP1, и HPF1 на сайтах повреждений [37, 108]. Аналогично на длительность удержания PARP1 на сайтах повреждения ДНК действует нокаут гена HPF1: в этом случае время увеличивается [64]. Это, по-видимому, влияет на динамику рекрутирования белков к сайтам повреждений, что подтверждает роль HPF1 в передаче сигнала о повреждении ДНК. На основании этих фактов можно предположить, что каталитическая активность PARP1 необходима для его диссоциации с сайтов повреждения, а HPF1 ускоряет этот процесс, скорее всего, за счет стимуляции инициации PARилирования [105].
PARP2 рекрутируется на сайты повреждения ДНК позже PARP1 [108, 109]. Так, показано, что каталитическая активация PARP1 ускоряет привлечение PARP2, но некритичнo, так как в отсутствие PARP1 PARP2 все же привлекается на сайты повреждения ДНК, хотя и с некоторой задержкой [109]. Можно предположить, что PARP1 в комплексе с HPF1 катализирует PARилирование, а синтезированный PAR ускоряет привлечение PARP2 к поврежденной ДНК. Таким образом, PARP1, HPF1 и затем PARP2 совместно локализуются в местах повреждения ДНК в составе хроматина. В этом контексте исследования in vitro, в которых продемонстрирована более эффективная HPF1-зависимая модификация гистонов именно при участии PARP2 в сравнении с PARP1, можно трактовать как косвенное доказательство участия HPF1 в ответе на повреждение ДНК на уровне хроматина [105].
ДИНАМИКА ВОЗНИКНОВЕНИЯ И ДЕГРАДАЦИИ ПОЛИ(ADP-РИБОЗЫ), СВЯЗАННОЙ С ОСТАТКАМИ СЕРИНА
Как сказано выше, PARилирование ‒ это обратимая посттрансляционная модификация. Наряду с ферментами, синтезирующими PAR, и взаимодействующими с этим полимером белками есть и ферменты, деградирующие PAR [6]. Среди последних в первую очередь следует упомянуть PARG. Этот PAR-деградирующий фермент, в силу механизма его действия, не удаляет первый остаток ADP-рибозы, связанный с аминокислотным остатком белка-мишени [110, 111]. Продукт реакции, катализируемой PARG, ‒ белок, модифицированный моно-ADP-рибозой (MAR), который служит субстратом ферментов, удаляющих MAR. Функция PARG критична для клетки, так как нокаут гена PARG приводит к накоплению PAR, раннему апоптозу в клеточных линиях человека и эмбриональной летальности у мышей [112]. Таким образом, эффективный PARG-опосредованный оборот PAR необходим для функционирования клеточных систем после повреждения ДНК [17, 85].
MAR возникает как продукт MARилирования или действия PARG и удаляется несколькими ферментами, принадлежащими к семейству ADP-рибозилгидролаз (ARH1, ARH2 и ARH3) и макродоменсодержащих ADP-рибозилгидролаз (MacroD1, MacroD2 и TARG1) [113–115]. Среди них только ARH3 представляет собой ADP-рибозилгидролазу, удаляющую MAR с остатков серина [116, 117], остальные гидролизуют моно(ADP-рибозу), связанную с аспартатом, глутаматом или аргинином.
По всей видимости, синтезированный полимер PAR быстро деградирует под действием PARG. Стабильность оставшейся ковалентно присоединенной к PARP1/2 MAR определяется в первую очередь природой аминокислотного остатка, с которым она связана. В протеомных исследованиях сайтов PAR-модификации обнаружили, что модифицированные остатки серина не только преобладали, но и были самыми стабильными in vivo. Это подтверждается тем фактом, что рибозилирование по остаткам аспартата и глутамата обнаруживают только в клетках, нокаутных по PARG [97]. В то же время MARилирование серина достоверно регистрируют в клетках дикого типа [112]. Стабильность этой модификации регулируется преимущественно ARH3 [112]. Таким образом, каталитической активности комплекса HPF1:PARP1/2, синтезирующего PAR, противодействуют ферменты, его удаляющие: PARG и ARH3 [116–118].
ПОЛИ(ADP-РИБОЗИЛ)ИРОВАНИЕ ГИСТОНОВ И КАНОНИЧЕСКИЕ ГИСТОНОВЫЕ МЕТКИ
Функциональная значимость PARилирования гистонов ‒ контроль состояния хроматина. Показано, что предпочтительными субстратами PARP1/2:HPF1 служат гистоны H2B и H3, а именно их аминокислотные остатки H2BS6, H3S28 и H3S10 [39]; причем последний – более эффективный акцептор ADP-рибозы по сравнению с H3S28 [40]. Заметим, что указанные сайты PARилирования гистонов служат и сайтами фосфорилирования [41, 119]. Остаток H2BS6 фосфорилируется в начале митоза и полностью дефосфорилируется после его завершения. Фосфорилирование H2BS6 особенно выражено в центромерных и прицентромерных областях, а его блокирование приводит к нарушению анафазы и неполному цитокинезу [120]. Фосфорилирование H3S10 инициируется в перицентромерном гетерохроматине в клетках поздней интерфазы G2. После инициации фосфорилирование H3, по-видимому, распространяется по всему хроматину. В целом, существует точная пространственная и временнáя корреляция между фосфорилированием H3 и начальными стадиями конденсации хроматина [121]. PARилирование H2BS6 и H3S10 может оказывать регуляторное действие и вызывать остановку клеточного цикла в случае повреждения ДНК. Эту гипотезу подтверждают результаты исследования N. Hananya и др. [122], в котором показано, что PARилирование H2BS6 ингибирует конденсацию хроматина и организацию структур более высокого порядка до завершения репарации.
Фосфорилирование H3S28 вместе с H3S10 вовлечено в регуляцию транскрипции [123]. По всей видимости, в контексте регуляции транскрипции важную роль играет сочетание фосфорилирования H3S10 с ацетилированием остатков H3K9 или H3K14 [124–126]. В частности, быстрое и временное фосфорилирование гистона H3 по остаткам Ser10 и Ser28 под действием киназ MSK1 и MSK2 (митоген- и стрессзависимые киназы) наблюдается при транскрипционной активации генов раннего ответа [125]. Показано, что MSK1/2-опосредованное фосфорилирование H3S28 на стрессзависимых промоторах способствует диссоциации корепрессорных комплексов HDAC и тем самым усилению локального ацетилирования гистонов и последующей активации транскрипции стресс-индуцированных генов [123]. На данный момент показано, что PARилирование гистона H3 по серинам препятствует его ацетилированию и, наоборот, ацетилировние H3K9 достаточно для блокирования PARилирования H3S10 [127]. Иными словами, фосфорилирование и ацетилирование супрессируют HPF1-зависимое PARилирование гистонов. Это объясняет вклад гистоновых деацетилаз в регуляцию репарации ДНК [128, 129]. В ответ на повреждение ДНК наблюдали быстрое деацетилирование H3K9 и H3K14 [130, 131]. В то же время ацетилирование ограничивало распространение PARилирвания гистонов, препятствуя чрезмерному расширению сигнала о повреждении, а восстановление ацетилирования происходило после завершения процесса репарации ДНК. Установлено, что в клетках, нокаутных по ARH3, уровень ацетилирования H3K9 в ответ на повреждение ДНК снижен более продолжительное время, чем в клетках дикого типа. По всей видимости, стабильное ADP-рибозилирование гистонов препятствует восстановлению уровня их ацетилирования [112]. Это может приводить к взаимному исключению ацетилирования H3K9 и H3K14 и PARилирования гистонов (рис. 4).
Рис. 4.
Изменение паттерна посттрансляционной модификации гистонов при продвижении по клеточному циклу и повреждении ДНК.
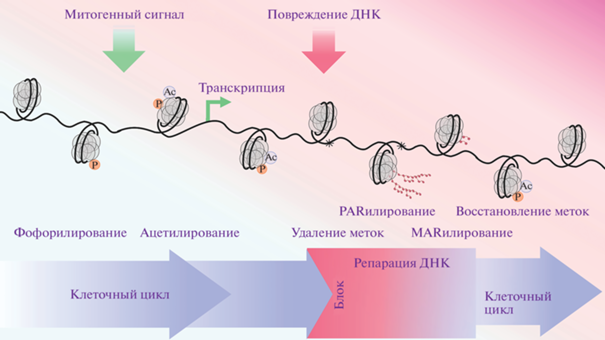
Таким образом, баланс между PARилированием и фосфорилированием остатков серина регулируется киназами, деацетилазами и ферментами, удаляющими PAR. В то время как фосфорилирование и ацетилирование считаются маркерами транскрипции и продвижения клетки по клеточному циклу, PARилирование, по всей видимости, регулирует остановку этих процессов для того, чтобы предоставить клетке время для восстановления структуры ДНК в случае ее повреждения.
ЗАКЛЮЧЕНИЕ
ADP-рибозилирование представляет собой посттрансляционную модификацию, которая реализуется как в мономерной (MAR), так и в полимерной (PAR) формах. Несмотря на то, что исследования PAR длятся уже 60 лет, новые детали механизма этой посттрансляционной модификации продолжают выяснять. Вслед за открытием ферментов, ответственных за синтез и деградацию PAR, обнаружили множество белков, взаимодействующих с этим полимером. С каждым годом расширяется спектр клеточных процессов, в которых показано участие PARилирования; при этом репарация ДНК, конечно, остается главным направлением.
Исследования PARP проводят как на модельных ДНК, так и в системе нуклеосом. В последнем случае можно обнаружить взаимодействие PARP с гистонами и его влияние на структуру нуклеосомы [26, 132‒134]. Открытие PARилирования остатков серина и вместе с ним кофактора этого процесса ‒ HPF1 ‒ ознаменовало новый виток в исследованиях этой реакции; поэтому вряд ли дальнейшие исследования функционирования ферментов PARP в составе хроматина стоит проводить в отсутствие этого важного кофактора. На сегодняшний день уже известны некоторые закономерности взаимодействия HPF1 с PARP1/2, хотя есть и противоречащие друг другу данные, которые необходимо уточнить для понимания механизма действия HPF1. Примечательно, что делеция гена HPF1 не отменяет PARилирования остатков серина [40]. Это дает основание предположить существование других, еще неизвестных кофакторов PARP1/2, меняющих специфичность PARилирования по аналогии с HPF1. Влияние HPF1 на активность PARP1/2, показанное in vivo и in vitro, важно понимать в контексте исследования ингибиторов PARP1/2, некоторые из которых уже используют как антираковые препараты в клинике. Недавно обнаружено вовлечение HPF1 в клеточный ответ при обработке такими ингибиторами [100]. Таким образом, последующий поиск ингибиторов PARP1/2 необходимо проводить с учетом влияния взаимодействий PARP1/2 c HPF1.
Авторы выражают благодарность Речкуновой Надежде Ивановне за обсуждения при написании обзора.
Работа выполнена при поддержке Российского научного фонда (грант № 22-14-00112) и в рамках Проекта государственного задания ИХБФМ СО РАН № 121031300041-4 (глава “Некоторые аспекты клеточной роли PARP1 и PARP2”).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kraus W.L. (2015) PARPs and ADP-ribosylation: 50 years… and counting. Mol. Cell. 58, 902–910.
Gibson B.A., Kraus W.L. (2012) New insights into the molecular and cellular functions of poly(ADP-ribose) and PARPs. Nat. Rev. Mol. Cell Biol. 13, 411–424.
Wei H., Yu X. (2016) Functions of PARylation in DNA damage repair pathways. Genomics Proteomics Bioinformatics. 14, 131–139.
Cohen M.S., Chang P. (2018) Insights into the biogenesis, function, and regulation of ADP-ribosylation. Nat. Chem. Biol. 14, 236–243.
Kraus W.L. (2020) PARPs and ADP-ribosylation: 60 years on. Genes Dev. 34, 251–253.
O’Sullivan J., Tedim Ferreira M., Gagné J.P., Sharma A.K., Hendzel M.J., Masson J.Y., Poirier G.G. (2019) Emerging roles of eraser enzymes in the dynamic control of protein ADP-ribosylation. Nat. Commun. 10(1), 1182.
Perina, D., Mikoč A., Ahel J., Ćetković H., Žaja R., Ahel I. (2014) Distribution of protein poly(ADP-ribosyl)ation systems across all domains of life. DNA Repair (Amst.). 23, 4–16.
Žaja R., Mikoč A., Barkauskaite E., Ahel I. (2012) Molecular insights into poly(ADP-ribose) recognition and processing. Biomolecules. 3, 1–17.
Hanzlikova H., Kalasova I., Demin A.A., Pennicott L.E., Cihlarova Z., Caldecott K.W. (2018) The importance of poly(ADP-ribose) polymerase as a sensor of unligated Okazaki fragments during DNA replication. Mol. Cell. 71, 319‒331.
Hanzlikova H., Gittens W., Krejcikova K., Zeng Z., Caldecott K.W. (2017) Overlapping roles for PARP1 and PARP2 in the recruitment of endogenous XRCC1 and PNKP into oxidized chromatin. Nucleic Acids Res. 45, 2546–2557.
Buch-Larsen S.C., Rebak A.K.L.F.S., Hendriks I.A., Nielsen M.L. (2021) Temporal and site-specific ADP-ribosylation dynamics upon different genotoxic stresses. Cells. 10(11), 2927.
Hoch N.C., Polo L.M. (2019) ADP-ribosylation: from molecular mechanisms to human disease. Genet. Mol. Biol. 43, e20190075.
Ray Chaudhuri A., Nussenzweig A. (2017) The multifaceted roles of PARP1 in DNA repair and chromatin remodelling. Nat. Rev. Mol. Cell Biol. 18, 610–621.
Soldani C., Scovassi A.I. (2002) Poly(ADP-ribose) polymerase-1 cleavage during apoptosis: an update. Apoptosis. 7, 321–328.
D’Amours D., Sallmann F.R., Dixit V.M., Poirier G.G. (2001) Gain-of-function of poly(ADP-ribose) polymerase-1 upon cleavage by apoptotic proteases: implications for apoptosis. J. Cell Sci. 114(Pt. 20), 3771–3778.
D’Amours D., Desnoyers S., D’Silva I., Poirier G.G. (1999) Poly(ADP-ribosyl)ation reactions in the regulation of nuclear functions. Biochem. J. 342(Pt. 2), 249–268.
Lavrik O.I. (2020) PARPs’ impact on base excision DNA repair. DNA Repair (Amst.). 93, 102911.
Vasil’eva I., Moor N., Anarbaev R., Kutuzov M., Lavrik O. (2021) Functional roles of PARP2 in assembling protein‒protein complexes involved in base excision DNA repair. Int. J. Mol. Sci. 22, 4679.
Ходырева С.Н., Лаврик О.И. (2016) Поли(ADP-рибоза)полимераза 1 – ключевой регулятор репарации ДНК. Молекуляр. биология. 50, 655–673.
Sukhanova M.V., Hamon L., Kutuzov M.M., Joshi V., Abrakhi S., Dobra I., Curmi P.A., Pastre D., Lavrik O.I. (2019) A single-molecule atomic force microscopy study of PARP1 and PARP2 recognition of base excision repair DNA intermediates. J. Mol. Biol. 431, 2655–2673.
Moor N.A., Vasil’eva I.A., Kuznetsov N.A., Lavrik O.I. (2020) Human apurinic/apyrimidinic endonuclease 1 is modified in vitro by poly(ADP-ribose) polymerase 1 under control of the structure of damaged DNA. Biochimie. 168, 144–155.
Суханова М.В. Лаврик О.И., Ходырева С.Н. (2004) Поли(ADP-рибозо)полимераза-1 ‒ регулятор белково-нуклеиновых взаимодействий в процессах, возникающих при генотоксическом воздействии. Молекуляр. биология. 38, 834‒847.
Sukhanova M.V., Khodyreva S.N., Lebedeva N.A., Prasad R., Wilson S.H., Lavrik O.I. (2005) Human base excision repair enzymes apurinic/apyrimidinic endonuclease1 (APE1), DNA polymerase β and poly(ADP-ribose) polymerase 1: interplay between strand-displacement DNA synthesis and proofreading exonuclease activity. Nucleic Acids Res. 33, 1222–1229.
Sukhanova M., Khodyreva S., Lavrik O. (2010) Poly(ADP-ribose) polymerase 1 regulates activity of DNA polymerase β in long patch base excision repair. Mutat. Res. 685, 80–89.
Моор Н.А., Лаврик О.И. (2018) Белок-белковые взаимодействия системы эксцизионной репарации оснований ДНК. Биохимия. 83, 564‒576.
Kutuzov M.M., Belousova E.A., Kurgina T.A., Ukraintsev A.A., Vasil’eva I.A., Khodyreva S.N., Lavrik O.I. (2021) The contribution of PARP1, PARP2 and poly(ADP-ribosyl)ation to base excision repair in the nucleosomal context. Sci. Rep. 11, 4849.
Caron M.C., Sharma A.K., O’Sullivan J., Myler L.R., Ferreira M.T., Rodrigue A., Coulombe Y., Ethier C., Gagné J.P., Langelier M.F., Pascal J.M., Finkelstein I.J., Hendzel M.J., Poirier G.G., Masson J.Y. (2019) Poly(ADP-ribose) polymerase-1 antagonizes DNA resection at double-strand breaks. Nat. Commun. 10, 2954.
Krasikova Y., Rechkunova N., Lavrik O. (2021) Nucleotide excision repair: from molecular defects to neurological abnormalities. Int. J. Mol. Sci. 22, 6220.
Maltseva E.A., Rechkunova N.I., Sukhanova M.V., Lavrik O.I. (2015) Poly(ADP-ribose) polymerase 1 modulates interaction of the nucleotide excision repair factor XPC-RAD23B with DNA via poly(ADP-ribosyl)ation. J. Biol. Chem. 290, 21811–21820.
Flippot R., Patrikidou A., Aldea M., Colomba E., Lavaud P., Albigès L., Naoun N., Blanchard P., Terlizzi M., Garcia C., Bernard-Tessier A., Fuerea A., Di Palma M., Escudier B., Loriot Y., Baciarello G., Fizazi K. (2022) PARP inhibition, a new therapeutic avenue in patients with prostate cancer. Drugs. 82(7), 719‒733.
Spiegel J.O., van Houten B., Durrant J.D. (2021) PARP1: structural insights and pharmacological targets for inhibition. DNA Repair (Amst.). 103, 103125.
Dizdar O., Arslan C., Altundag K. (2015) Advances in PARP inhibitors for the treatment of breast cancer. Exp. Opin. Pharmacother. 16, 2751–2758.
Curtin N.J., Szabo C. (2020) Poly(ADP-ribose) polymerase inhibition: past, present and future. Nat. Rev. Drug Discov. 19, 711–736.
Schreiber V., Illuzzi G., Héberlé E., Dantzer F. (2015) From poly(ADP-ribose) discovery to PARP inhibitors in cancer therapy. Bull. Cancer. 102, 863–873.
Sim H.W., Galanis E., Khasraw M. (2022) PARP inhibitors in glioma: a review of therapeutic opportunities. Cancers (Basel). 14. 1003.
Sonnenblick A., de Azambuja E., Azim H.A., Piccart M. (2015) An update on PARP inhibitors‒moving to the adjuvant setting. Nat. Rev. Clin. Oncol. 12, 27–41.
Gibbs-Seymour I., Fontana P., Rack J.G.M., Ahel I. (2016) HPF1/C4orf27 is a PARP-1-interacting protein that regulates PARP-1 ADP-ribosylation activity. Mol. Cell. 62, 432–442.
Suskiewicz M.J., Zobel F., Ogden T.E.H., Fontana P., Ariza A., Yang J.C., Zhu K., Bracken L., Hawthorne W.J., Ahel D., Neuhaus D., Ahel I. (2020) HPF1 completes the PARP active site for DNA damage-induced ADP-ribosylation. Nature. 579, 598.
Bonfiglio J.J., Fontana P., Zhang Q., Colby T., Gibbs-Seymour I., Atanassov I., Bartlett E., Zaja R., Ahel I., Matic I. (2017) Serine ADP-ribosylation depends on HPF1. Mol. Cell. 65, 932‒940.
Palazzo L., Leidecker O., Prokhorova E., Dauben H., Matic I., Ahel I. (2018) Serine is the major residue for ADP-ribosylation upon DNA damage. Elife. 7, e34334.
Leidecker O., Bonfiglio J.J., Colby T., Zhang Q., Atanassov I., Zaja R., Palazzo L., Stockum A., Ahel I., Matic I. (2016) Serine is a new target residue for endogenous ADP-ribosylation on histones. Nat. Chem. Biol. 12, 998–1000.
Sun F.H., Zhao P., Zhang N., Kong L.L., Wong C.C.L., Yun C.H. (2021) HPF1 remodels the active site of PARP1 to enable the serine ADP-ribosylation of histones. Nat. Commun. 12, 1028.
Chambon P., Weill J.D., Mandel P. (1963) Nicotinamide mononucleotide activation of new DNA-dependent polyadenylic acid synthesizing nuclear enzyme. Biochem. Biophys. Res. Commun. 11, 39–43.
Chambon P., Weill J.D., Doly J., Strosser M.T., Mandel P. (1966) On the formation of a novel adenylic compound by enzymatic extracts of liver nuclei. Biochem. Biophys. Res. Commun. 25, 638–643.
Fujimura S., Hasegawa S., Shimizu Y., Sugimura T. (1967) Polymerization of the adenosine 5'-diphosphate-ribose moiety of nicotinamide-adenine dinucleotide by nuclear enzyme. I. Enzymatic reactions. Biochim. Biophys. Acta. 145, 247–259.
Sugimura T., Miwa M. (1994) Poly(ADP-ribose): historical perspective. Mol. Cell. Biochem. 138, 5–12.
Langelier M.F., Eisemann T., Riccio A.A., Pascal J.M. (2018) PARP family enzymes: regulation and catalysis of the poly(ADP-ribose) posttranslational modification. Curr. Opin. Struct. Biol. 53, 187–198.
Amé J.C., Spenlehauer C., de Murcia G. (2004) The PARP superfamily. BioEssays. 26, 882–893.
Otto H., Reche P.A., Bazan F., Dittmar K., Haag F., Koch-Nolte F. (2005) In silico characterization of the family of PARP-like poly(ADP-ribosyl)transferases (pARTs). BMC Genomics. 6, 139.
Ferro A.M., Minaga T., Piper W.N., Kun E. (1978) Analysis of larger than tetrameric poly(adenosine diphosphoribose) by a radioimmunoassay in nuclei separated in organic solvents. Biochim. Biophys. Acta. 519, 291–305.
Hassa P.O., Haenni S.S., Elser M., Hottiger M.O. (2006) Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going? Microbiol. Mol. Biol. Rev. 70, 789–829.
Alemasova E.E., Lavrik O.I. (2019) Poly(ADP-ribosyl)ation by PARP1: reaction mechanism and regulatory proteins. Nucleic Acids Res. 47, 3811–3827.
Koh D.W., Dawson V.L., Dawson T.M. (2005) The road to survival goes through PARG. Cell Cycle. 4, 397–399.
Han S., Tainer J.A. (2001) The ARTT motif and a unified structural understanding of substrate recognition in ADP-ribosylating bacterial toxins and eukaryotic ADP-ribosyltransferases. Int. J. Med. Microbiol. 291, 523–529.
Schreiber V., Dantzer F., Amé J.C., de Murcia G. (2006) Poly(ADP-ribose): novel functions for an old molecule. Nat. Rev. Mol. Cell. Biol. 7, 517–528.
Pascal J.M., Ellenberger T. (2015) The rise and fall of poly(ADP-ribose): an enzymatic perspective. DNA Repair (Amst.). 32, 10–16.
Amé J.C., Rolli V., Schreiber V., Niedergang C., Apiou F., Decker P., Muller S., Höger T., Ménissier-de Murcia J., de Murcia G. (1999) PARP-2, a novel mammalian DNA damage-dependent poly(ADP-ribose) polymerase. J. Biol. Chem. 274, 17860–17868.
Eisemann T., Pascal J.M. (2020) Poly(ADP-ribose) polymerase enzymes and the maintenance of genome integrity. Cell. Mol. Life Sci. 77, 19–33.
Ghosh R., Roy S., Kamyab J., Dantzer F., Franco S. (2016) Common and unique genetic interactions of the poly(ADP-ribose) polymerases PARP1 and PARP2 with DNA double-strand break repair pathways. DNA Repair (Amst.). 45, 56–62.
Ruf A., de Murcia J.M., de Murcia G.M., Schulz G.E. (1996) Structure of the catalytic fragment of poly(AD-ribose) polymerase from chicken. Proc. Natl. Acad. Sci. USA. 93, 7481–7485.
Langelier M.F., Planck J.L., Roy S., Pascal J.M. (2012) Structural basis for DNA damage-dependent poly(ADP-ribosyl)ation by human PARP-1. Science. 336, 728–732.
Langelier M.F., Ruhl D.D., Planck J.L., Kraus W.L., Pascal J.M. (2010) The Zn3 domain of human poly(ADP-ribose) polymerase-1 (PARP-1) functions in both DNA-dependent poly(ADP-ribose) synthesis activity and chromatin compaction. J. Biol. Chem. 285, 18877–18887.
Langelier M.F., Planck J.L., Roy S., Pascal J.M. (2011) Crystal structures of poly(ADP-ribose) polymerase-1 (PARP-1) zinc fingers bound to DNA: structural and functional insights into DNA-dependent PARP-1 activity. J. Biol. Chem. 286, 10690–10701.
Prokhorova E., Zobel F., Smith R., Zentout S., Gibbs-Seymour I., Schützenhofer K., Peters A., Groslambert J., Zorzini V., Agnew T., Brognard J., Nielsen M.L., Ahel D., Huet S., Suskiewicz M.J., Ahel I. (2021) Serine-linked PARP1 auto-modification controls PARP inhibitor response. Nat. Commun. 12, 4055.
Rudolph J., Muthurajan U.M., Palacio M., Mahadevan J., Roberts G., Erbse A.H., Dyer P.N., Luger K. (2021) The BRCT domain of PARP1 binds intact DNA and mediates intrastrand transfer. Mol. Cell. 81, 4994‒5006.
Tao Z., Gao P., Liu H.W. (2009) Identification of the ADP-ribosylation sites in the PARP-1 automodification domain: analysis and implications. J. Am. Chem. Soc. 131, 14258–14260.
Obaji E., Haikarainen T., Lehtiö L. (2018) Structural basis for DNA break recognition by ARTD2/PARP2. Nucleic Acids Res. 46, 12154–12165.
Obaji E., Haikarainen T., Lehtiö L. (2016) Characterization of the DNA dependent activation of human ARTD2/PARP2. Sci. Rep. 6, 34487.
Rudolph J., Mahadevan J., Dyer P., Luger K. (2018) Poly(ADP-ribose) polymerase 1 searches DNA via a “monkey bar” mechanism. Elife. 7, e37818.
Riccio A.A., Cingolani G., Pascal J.M. (2016) PARP-2 domain requirements for DNA damage-dependent activation and localization to sites of DNA damage. N-ucleic Acids Res. 44, 1691–1702.
Sukhanova M.V., Abrakhi S., Joshi V., Pastre D., Kutuzov M.M., Anarbaev R.O., Curmi P.A., Hamon L., Lavrik O.I. (2015) Single molecule detection of PARP1 and PARP2 interaction with DNA strand breaks and their poly(ADP-ribosyl)ation using high-resolution AFM imaging. Nucleic Acids Res. 44, e60.
Kutuzov M.M., Khodyreva S.N., Amé J.C., Ilina E.S., Sukhanova M.V., Schreiber V., Lavrik O.I. (2013) Interaction of PARP-2 with DNA structures mimicking DNA repair intermediates and consequences on activity of base excision repair proteins. Biochimie. 95, 1208–1215.
D’Silva I., Pelletier J.D., Lagueux J., D’Amours D., Chaudhry M.A., Weinfeld M., Lees-Miller S.P., Poirier G.G. (1999) Relative affinities of poly(ADP-ribose) polymerase and DNA-dependent protein kinase for DNA strand interruptions. Biochim. Biophys. Acta. 1430, 119–126.
Langelier M.F., Riccio A.A., Pascal J.M. (2014) PARP-2 and PARP-3 are selectively activated by 5' phosphorylated DNA breaks through an allosteric regulatory mechanism shared with PARP-1. Nucleic Acids Res. 42, 7762–7775.
Cistulli C., Lavrik O.I., Prasad R., Hou E., Wilson S.H. (2004) AP endonuclease and poly(ADP-ribose) polymerase-1 interact with the same base excision repair intermediate. DNA Repair (Amst.). 3, 581–591.
Khodyreva S.N., Prasad R., Ilina E.S., Sukhanova M.V., Kutuzov M.M., Liu Y., Hou E.W., Wilson S.H., Lavrik O.I. (2010) Apurinic/apyrimidinic (AP) site recognition by the 5'-dRP/AP lyase in poly(ADP-ribose) polymerase-1 (PARP-1). Proc. Natl. Acad. Sci. USA. 107, 22090–22095.
Lavrik O.I., Prasad R., Sobol R.W., Horton J.K., Ackerman E.J., Wilson S.H. (2001) Photoaffinity labeling of mouse fibroblast enzymes by a base excision repair intermediate. Evidence for the role of poly(ADP-ribose) polymerase-1 in DNA repair. J. Biol. Chem. 276, 25541–25548.
Ménissier de Murcia J., Ricoul M., Tartier L., Niedergang C., Huber A., Dantzer F., Schreiber V., Amé J.C., Dierich A., LeMeur M., Sabatier L., Chambon P., de Murcia G. (2003) Functional interaction between PARP-1 and PARP-2 in chromosome stability and embryonic development in mouse. EMBO J. 22, 2255–2263.
Nicolás L., Martínez C., Baró C., Rodríguez M., Baroja-Mazo A., Sole F., Flores J.M., Ampurdanés C., Dantzer F., Martin-Caballero J., Aparicio P., Yelamos J. (2010) Loss of poly(ADP-ribose) polymerase-2 leads to rapid development of spontaneous T-cell lymphomas in p53-deficient mice. Oncogene. 29, 2877–2883.
Farreś J., Martín-Caballero J., Martínez C., Lozano J. J., Llacuna L., Ampurdanés C., Ruiz-Herguido C., Dantzer F., Schreiber V., Villunger A., Bigas A., Yélamos J. (2013) Parp-2 is required to maintain hematopoiesis following sublethal γ-irradiation in mice. Blood. 122, 44–54.
Farrés J., Llacuna L., Martin-Caballero J., Martínez C., Lozano J.J., Ampurdanés C., López-Contreras A.J., Florensa L., Navarro J., Ottina E., Dantzer F., Schreiber V., Villunger A., Fernández-Capetillo O., Yélamos J. (2015) PARP-2 sustains erythropoiesis in mice by limiting replicative stress in erythroid progenitors. Cell Death Differ. 22, 1144–1157.
Alemasova E.E., Naumenko K.N., Kurgina T.A., Anarbaev R.O., Lavrik O.I. (2018) The multifunctional protein YB-1 potentiates PARP1 activity and decreases the efficiency of PARP1 inhibitors. Oncotarget. 9, 23349-23365.
Naumenko K.N., Sukhanova M.V., Hamon L., Kurgina T.A., Alemasova E.E., Kutuzov M.M., Pastré, D., Lavrik O.I. (2020) Regulation of poly(ADP-ribose) polymerase 1 activity by Y-box-binding protein 1. Biomolecules. 10, 1325.
Sun X., Fu K., Hodgson A., Wier E.M., Wen M.G., Kamenyeva O., Xia X., Koo L.Y., Wan F. (2016) Sam68 is required for DNA damage responses via regulating poly(ADP-ribosyl)ation. PLoS Biol. 14, e1002543.
Sukhanova M.V., Singatulina A.S., Pastré D., Lavrik O.I. (2020) Fused in sarcoma (FUS) in DNA repair: tango with poly(ADP-ribose) polymerase 1 and compartmentalisation of damaged DNA. Int. J. Mol. Sci. 21, 7020.
Obaji E., Maksimainen M.M., Galera-Prat A., Lehtiö L. (2021) Activation of PARP2/ARTD2 by DNA damage induces conformational changes relieving enzyme autoinhibition. Nat. Communs. 12, 3479.
Dawicki-McKenna J.M., Langelier M.F., DeNizio J.E., Riccio A.A., Cao C.D., Karch K.R., McCauley M., Steffen J.D., Black B.E., Pascal J.M. (2015) PARP-1 activation requires local unfolding of an autoinhibitory domain. Mol. Cell. 60, 755–768.
Langelier M.F., Pascal J M. (2013) PARP-1 mechanism for coupling DNA damage detection to poly(ADP-ribose) synthesis. Curr. Opin. Struct. Biol. 23, 134–143.
Ogden T.E.H., Yang J.C., Schimpl M., Easton L.E., Underwood E., Rawlins P.B., McCauley M.M., Langelier M.F., Pascal J.M., Embrey K.J., Neuhaus D. (2021) Dynamics of the HD regulatory subdomain of PARP-1; substrate access and allostery in PARP activation and inhibition. Nucleic Acids Res. 49, 2266–2288.
Bauer P.I., Buki K.G., Hakam A., Kun E. (1990) Macromolecular association of ADP-ribosyltransferase and its correlation with enzymic activity. Biochem. J. 270, 17–26.
Eustermann S., Videler H., Yang J.C., Cole P.T., Gruszka D., Veprintse D., Neuhaus D. (2011) The DNA-binding domain of human PARP-1 interacts with DNA single-strand breaks as a monomer through its second zinc finger. J. Mol. Biol. 407, 149–170.
Buki K.G., Bauer P.I., Hakam A., Kun E. (1995) Identification of domains of poly(ADP-ribose) polymerase for protein binding and self-association. J. Biol. Chem. 270, 3370–3377.
Vasil’eva I.A., Anarbaev R.O., Moor N.A., Lavrik O.I. (2019) Dynamic light scattering study of base excision DNA repair proteins and their complexes. Biochim. Biophys. Acta. 1867, 297–305.
Panzeter P.L., Althaus F.R. (1994) DNA strand break-mediated partitioning of poly(ADP-ribose) polymerase function. Biochemistry. 33, 9600–9605.
Pion E., Ullmann G.M., Amé J.C., Gérard D., de Murcia G., Bombarda E. (2005) DNA-induced dimerization of poly(ADP-ribose) polymerase-1 triggers its activation. Biochemistry. 44, 14670–14681.
Li P., Zhen Y., Yu Y. (2019) Site-specific analysis of the Asp- and Glu-ADP-ribosylated proteome by quantitative mass spectrometry. Methods Enzymol. 626, 301–321.
Zhang Y., Wang J., Ding M., Yu Y. (2013) Site-specific characterization of the Asp- and Glu-ADP-ribosylated proteome. Nat. Methods. 10, 981–984.
Martello R., Leutert M., Jungmichel S., Bilan V., Larsen S.C., Young C., Hottiger M.O., Nielsen M.L. (2016) Proteome-wide identification of the endogenous ADP-ribosylome of mammalian cells and tissue. Nat. Commun. 7, 12917.
Gaullier G., Roberts G., Muthurajan U.M., Bowerman S., Rudolph J., Mahadevan J., Jha A., Rae P.S., Luger K. (2020) Bridging of nucleosome-proximal DNA double-strand breaks by PARP2 enhances its interaction with HPF1. PLoS One. 15, e0240932.
Rudolph J., Roberts G., Luger K. (2021) Histone PARylatio-n factor 1 contributes to the inhibition of PARP1 by cancer drugs. Nat. Commun. 12, 736.
Bilokapic S., Suskiewicz M.J., Ahel I., Halic M. (2020) Bridging of DNA breaks activates PARP2-HPF1 to modify chromatin. Nature. 585, 609–613.
Rudolph J., Roberts G., Muthurajan U.M., Luger K. (2021) HPF1 and nucleosomes mediate a dramatic switch in activity of PARP1 from polymerase to hydrolase. Elife. 10, e65773.
Marsischky G.T., Wilson B.A., Collier R.J. (1995) Role of glutamic acid 988 of human poly-ADP-ribose polymerase in polymer formation: evidence for active site similarities to the ADP-ribosylating toxins. J. Biol. Chem. 270, 3247–3254.
Dodson G., Wlodawer A. (1998) Catalytic triads and their relatives. Trends Biochem. Sci. 23, 347–352.
Kurgina T.A., Moor N.A., Kutuzov M.M., Naumenko K.N., Ukraintsev A.A., Lavrik O.I. (2021) Dual function of HPF1 in the modulation of PARP1 and PARP2 activities. Commun. Biol. 4, 1259.
Langelier M.F., Billur R., Sverzhinsky A., Black B.E., Pascal J.M. (2021) HPF1 dynamically controls the PARP1/2 balance between initiating and elongating ADP-ribose modifications. Nat. Commun. 12, 6675.
Desmarais Y., Ménard L., Lagueux J., Poirier G.G. (1991) Enzymological properties of poly(ADP-ribose)polymerase: characterization of automodification sites and NADase activity. Biochim. Biophys. Acta. 1078, 179–186.
Mortusewicz O., Amé J.C., Schreiber V., Leonhardt H. (2007) Feedback-regulated poly(ADP-ribosyl)ation by PARP-1 is required for rapid response to DNA damage in living cells. Nucleic Acids Res. 35, 7665–7675.
Lin X., Jiang W., Rudolph J., Lee B.J., Luger K., Zha S. (2022) PARP inhibitors trap PARP2 and alter the mode of recruitment of PARP2 at DNA damage sites. Nucleic Acids Res. 50, 3958–3973.
Crawford K., Bonfiglio J.J., Mikoč A., Matic I., Ahel I. (2018) Specificity of reversible ADP-ribosylation and regulation of cellular processes. Crit. Rev. Biochem. Mol. Biol. 53, 64–82.
Uchida K., Suzuki H., Maruta H., Abe H., Aoki K., Miwa M., Tanuma S.-I. (1993) Preferential degradation of protein-bound (ADP-ribose), by nuclear poly(ADP-ribose) glycohydrolase from human placenta. J. Biol. Chem. 268, 3194–3200.
Hendriks I.A., Buch-Larsen S.C., Prokhorova E., Elsborg J.D., Rebak A.K.L.F.S., Zhu K., Ahel D., Lukas C., Ahel I., Nielsen M.L. (2021) The regulatory landscape of the human HPF1- and ARH3-dependent ADP-ribosylome. Nat. Commun. 12, 5893.
Sharifi R., Morra R., Appel C.D., Tallis M., Chioza B., Jankevicius G., Simpson M.A., Matic I., Ozkan E., Golia B., Schellenberg M.J., Weston R., Williams J.G., Rossi M.N., Galehdari H., Krahn J., Wan A., Trembath R.C., Crosby A.H., Ahel D., Hay R., Ladurner A.G., Timinszky G., Williams R.S., Ahel I. (2013) Deficiency of terminal ADP-ribose protein glycohydrolase TARG1/C6orf130 in neurodegenerative disease. EMBO J. 32, 1225–1237.
Mashimo M., Kato J., Moss J. (2014) Structure and function of the ARH family of ADP-ribosyl-acceptor hydrolases. DNA Repair. 23, 88–94.
Oka S., Kato J., Moss J. (2006) Identification and characterization of a mammalian 39-kDa poly(ADP-ribose) glycohydrolase. J. Biol. Chem. 281, 705–713.
Fontana P., Bonfiglio J.J., Palazzo L., Bartlett E., Matic I., Ahel I. (2017) Serine ADP-ribosylation reversal by the hydrolase ARH3. Elife. 6, e28533.
Abplanalp J., Leutert M., Frugier E., Nowak K., Feurer R., Kato J., Kistemaker H.V.A., Filippov D.V., Moss J., Caflisch A., Hottiger M.O. (2017) Proteomic analyses identify ARH3 as a serine mono-ADP-ribosylhydrolase. Nat. Commun. 8, 2055.
Slade D., Dunstan M.S., Barkauskaite E., Weston R., Lafite P., Dixon N., Ahel M., Leys D., Ahel I. (2011) The structure and catalytic mechanism of a poly(ADP-ribose) glycohydrolase. Nature. 477, 616–622.
Huang H., Lin S., Garcia B.A., Zhao Y. (2015) Quantitative proteomic analysis of histone modifications. Chem. Rev. 115, 2376.
Seibert M., Krüger M., Watson N.A., Sen O., Daum J.R., Slotman J.A., Braun T., Houtsmuller A.B., Gorbsky G.J., Jacob R., Kracht M., Higgins J.M.G., Schmitz M.L. (2019) CDK1-mediated phosphorylation at H2B serine 6 is required for mitotic chromosome segregation. J. Cell Biol. 218, 1164.
Hendzel M.J., Wei Y., Mancini M.A., van Hooser A., Ranalli T., Brinkley B.R., Bazett-Jones D.P., Allis C.D. (1997) Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348–360.
Hananya N., Daley S.K., Bagert J.D., Muir T.W. (2021) Synthesis of ADP-ribosylated histones reveals site-specific impacts on chromatin structure and function. J. Am. Chem. Soc. 143, 10847–10852.
Sawicka A., Hartl D., Goiser M., Pusch O., Stocsits R.R., Tamir I.M., Mechtler K., Seiser C. (2014) H3S28 phosphorylation is a hallmark of the transcriptional response to cellular stress. Genome Res. 24, 1808.
Cheung P., Tanner K.G., Cheung W.L., Sassone-Corsi P., Denu J.M., Allis C.D. (2000) Synergistic coupling of histone H3 phosphorylation and acetylation in response to epidermal growth factor stimulation. Mol. Cell. 5, 905–915.
Clayton A.L., Mahadevan L.C. (2003) MAP kinase-mediated phosphoacetylation of histone H3 and inducible gene regulation. FEBS Lett. 546, 51–58.
Simboeck E., Sawicka A., Zupkovitz G., Senese S., Winter S., Dequiedt F., Ogris E., di Croce L., Chiocca S., Seiser C. (2010) A phosphorylation switch regulates the transcriptional activation of cell cycle regulator p21 by histone deacetylase inhibitors. J. Biol. Chem. 285, 41062–41073.
Liszczak G., Diehl K.L., Dann G.P., Muir T.W. (2018) Acetylation blocks DNA damage-induced chromatin ADP-ribosylation. Nat. Chem. Biol. 14, 837–840.
Miller K.M., Tjeertes J.V., Coates J., Legube G., Polo S.E., Britton S., Jackson S.P. (2010) Human HDAC1 and HDAC2 function in the DNA-damage response to promote DNA nonhomologous end-joining. Nat. Struct. Mol Biol. 17, 1144–1151.
Mao Z., Hine C., Tian X., van Meter M., Au M., Vaidya A., Seluanov A., Gorbunova V. (2011) SIRT6 promotes DNA repair under stress by activating PARP1. Science. 332, 1443–1446.
Michishita E., McCord R.A., Berber E., Kioi M., Padilla-Nashm H., Damian M., Cheung P., Kusumoto R., Kawahara T.L.A., Barrett J.C., Chang H.Y., Bohr V.A., Ried T., Gozani O., Chua K.F. (2008) SIRT6 is a histone H3 lysine 9 deacetylase that modulates telomeric chromatin. Nature. 452, 492.
Tjeertes J.V., Miller K.M., Jackson S.P. (2009) Screen for DNA-damage-responsive histone modifications identifies H3K9Ac and H3K56Ac in human cells. EMBO J. 28, 1878–1889.
Maluchenko N.V., Sultanov D.S., Kotova E.Y., Kirpichnikov M.P., Studitsky V.M., Feofanov A.V. (2019) Histone tails promote PARP1-dependent structural rearrangements in nucleosomes. Dokl. Biochem. Biophys. 489, 377–379.
Maluchenko N.V., Nilov D.K., Pushkarev S.V., Kotova E.Y., Gerasimova N.S., Kirpichnikov M.P., Langelier M.-F., Pascal J.M., Akhtar M.S., Feofanov A.V., Studitsky V.M. (2021) Mechanisms of nucleosome reorganization by PARP1. Int. J. Mol. Sci. 22, 12127.
Kotova E.Y., Hsieh F.-K., Chang H.-W., Maluchenko N.V., Langelier M.-F., Pascal J.M., Luse D.S., Feofanov A.V., Studitsky V.M. (2022) Human PARP1 facilitates transcription through a nucleosome and histone displacement by Pol II in vitro. Int. J. Mol. Sci. 23, 7107.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


