Российские нанотехнологии, 2023, T. 18, № 3, стр. 302-310
Национальная база генетической информации
И. А. Колесников 1, *, М. В. Николенко 1, А. В. Ермаков 1, А. А. Корженков 1, А. А. Заикин 1, В. Е. Велихов 1, С. А. Бобков 1, Ф. С. Шарко 1, З. Б. Намсараев 1, М. В. Патрушев 1
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
* E-mail: ivan.a.kolesnikov@gmail.com
Поступила в редакцию 21.04.2023
После доработки 21.04.2023
Принята к публикации 24.04.2023
Аннотация
В соответствии с поручением Президента Российской Федерации НИЦ “Курчатовский институт” создает Национальную базу генетической информации (НБГИ). Согласно поправкам в Федеральный закон от 29 декабря 2022 г. организации, осуществляющие генно-инженерную деятельность, производство или поставку генно-инженерно-модифицированных организмов или продукции, полученной с их применением, должны в обязательном порядке депонировать информацию в НБГИ. Планируется, что НБГИ станет ключевым элементом инфраструктуры генетических исследований и разработок в Российской Федерации, обеспечивающим хранение, интеграцию и анализ генетических данных. Представлена информация о вычислительной и сетевой инфраструктуре, программном обеспечении, депонировании данных и аналитических возможностях НБГИ.
ОГЛАВЛЕНИЕ
Введение
1. Вычислительная и сетевая инфраструктура, хранилище данных
2. Программное обеспечение и интерфейс
3. Депонируемые данные
4. Аналитические функциональные возможности
5. Макет НБГИ
Заключение
ВВЕДЕНИЕ
Ключевым объектом научной инфраструктуры являются базы генетических данных, которые обеспечивают хранение и анализ генетических данных для науки и различных отраслей экономики. В настоящее время в мире насчитывается более полутора тысяч баз данных генетической информации, включающих в себя как специализированные базы данных для проведения исследований, так и базы данных генетической информации населения [1].
В соответствии с поручением Президента Российской Федерации № Пр-920 от 4 июня 2020 г. НИЦ “Курчатовский институт” создает Национальную базу генетической информации (НБГИ). В 2022 г. были приняты поправки в Федеральный закон от 29 декабря 2022 г. № 643-ФЗ “О государственном регулировании в области генно-инженерной деятельности”, согласно которому в НБГИ “информация в обязательном порядке предоставляется обладателями генетических данных, осуществляющими генно-инженерную деятельность, производство и (или) поставку генно-инженерно-модифицированных организмов, производство и (или) поставку продукции, полученной с применением генно-инженерно-модифицированных организмов или содержащей такие организмы, государственными учреждениями, иными юридическими лицами и индивидуальными предпринимателями, осуществляющими молекулярно-генетический анализ в целях проведения экспертиз, испытаний и научно-исследовательских работ” [2].
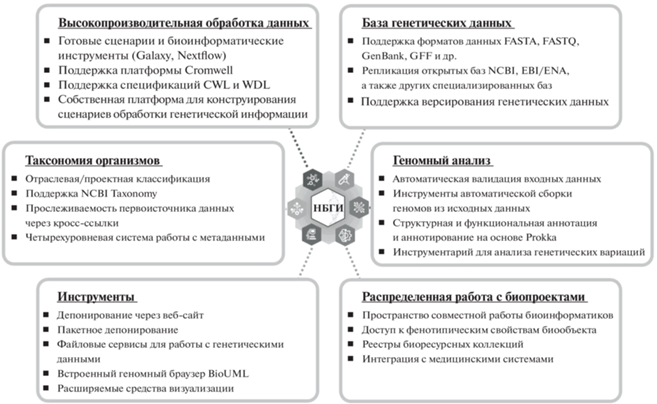
НБГИ должна решать следующие задачи:
– хранение генетической информации всего многообразия биообразцов, включая растения, животных, микроорганизмы дикой природы и метагеномы экосистем, растения и животных для сельского хозяйства, микроорганизмы для промышленности, человека, а также патогенные микроорганизмы;
– классификация генетической информации;
– поиск (по метаданным и по гомологии);
– обеспечение визуализации и анализа генетических данных в интегрированном геномном браузере;
– предоставление доступа к высокопроизводительной и облачной вычислительной инфраструктуре для обработки и анализа генетических данных;
– предоставление возможности конструирования средств анализа (“конвейеров”);
– обеспечение работы с инструментами анализа на основе технологий машинного обучения;
– обеспечение среды коммуникации (социальной сети) для профессиональных сообществ;
– обеспечение публикации научных статей;
– интеграция с ведомственными системами в части предоставления доступа к генетическим данным;
– интеграция с международными базами данных;
– накопление данных, получаемых в ходе реализации “Федеральной научно-технической программы развития генетических технологий на 2019–2027 годы” (рис. 1).
Планируется, что НБГИ станет ключевым элементом инфраструктуры генетических исследований и разработок в Российской Федерации, обеспечивающим хранение, интеграцию и анализ генетических данных, получаемых отечественными и зарубежными организациями и исследователями.
1. ВЫЧИСЛИТЕЛЬНАЯ И СЕТЕВАЯ ИНФРАСТРУКТУРА, ХРАНИЛИЩЕ ДАННЫХ
Вычислительная инфраструктура
Архитектура НБГИ и средства программно-технического и аппаратного обеспечения должны обеспечивать приемлемую скорость выполнения операций, связанных с поиском, визуализацией, анализом и высокопроизводительной обработкой генетической информации с применением технологий машинного обучения и суперкомпьютерных вычислений. Для обеспечения защиты информации в НБГИ выделены три контура обработки информации: открытый, конфиденциальный и специальный. Программно-аппаратные архитектуры каждого из контуров схожи, однако имеются различия, обусловленные выдвигаемыми требованиями по защите информации. В основе информационно-вычислительной инфраструктуры НБГИ лежат следующие решения:
– распределенная система хранения для работы с генетическими данными;
– вычислительный кластер для работы приложений и сервисов на основе контейнеров;
– система управления вычислительными ресурсами, хранилищем данных и вычислительными кластерами;
– ленточная система долговременного хранения.
Распределенная система хранения обеспечивает работу файловой системы и объектного хранилища. Объектное хранилище основано на неиерархической структуре хранения с доступом к объектам через уникальные идентификаторы, в которой данные хранятся в виде объектов. Такое решение предоставляет широкие возможности для хранения метаданных, организации доступа к данным и обеспечивает эффективное масштабирование. В объектном хранилище НБГИ будут размещены генетические данные, депонированные пользователями, а также данные, полученные в ходе импорта и обмена информацией с внешними базами.
Для управления вычислительными ресурсами, кластерами и хранилищем данных в открытом и конфиденциальном контурах применяется облачная технология. Данное решение позволяет гибко управлять инфраструктурой вычислительного кластера, реализовать повышенную изоляцию сегментов обработки информации и балансировать нагрузку между кластерами. В открытом и конфиденциальном контурах НБГИ применяется адаптированное окружение для работы с контейнеризованными приложениями в облачной инфраструктуре, сертифицированное на соответствие стандартам CNCF (Cloud Native Computing Fondation) и требованиям по защите информации согласно действующему законодательству. Такое окружение будет предоставлять расширенный прикладной программный интерфейс для решения следующих задач: создание, конфигурирование, удаление дисков, балансировка нагрузки, управление внешними сетями, настройка групп безопасности и др. Данное решение позволяет упростить техническое обслуживание вычислительных средств. Оценка производительности общих вычислительных ресурсов с учетом рекомендаций к ресурсам для высокопроизводительного секвенирования на основе замера времени для типовых операций обработки составляет 30 000 вычислительных ядер [3]. Для использования аналитических программ, имеющих повышенные требования к вычислительным ресурсам, в частности для вычислений на базе графических ускорителей и операций, требующих большого объема памяти, в составе открытого контура НБГИ предусмотрен кластер высокоинтенсивных вычислений (High Performance Computing). Для обеспечения сохранности данных на длительном временном периоде будет использоваться ленточная система хранения данных.
В качестве высокопроизводительной вычислительной инфраструктуры используются суперкомпьютерные ресурсы. Для обеспечения хранения генетических данных в состав системы входят две системы хранения: система хранения для задач с интенсивным вводом–выводом и распределенная система долговременного хранения для архива геномных данных, построенная на базе накопителей на жестких дисках. При создании вычислительной инфраструктуры основной упор делается на обеспечение отказоустойчивой работы НБГИ и сокращение времени обслуживания. Компоненты инфраструктуры НБГИ на всех уровнях используют горячее резервирование, применяется контейнеризация прикладного программного обеспечения (ПО) [4].
Планируемые объемы хранения информации
Суммарный размер хранилища НБГИ определен исходя из размеров хранилищ действующих зарубежных баз данных. Согласно данным European Bioinformatics Institute (EMBL-EBI) в 2021 г. объем хранимых данных превысил 390 ПБ и продолжает увеличиваться [5]. На сегодня в международных базах данных International Nucleotide Sequence Database Collaboration (INSDC, Международная коллаборация баз данных нуклеотидных последовательностей) опубликовано около 10 ПБ открытых генетической данных, сообщается о хранении сравнимого объема закрытых данных, а также несколько петабайт новых данных публикуется ежегодно [6]. Принимая во внимание, что объем общедоступных генетических данных, которые могут быть использованы широким кругом исследователей, значительно выше (для NCBI SRA объем общедоступных данных составляет около 45 ПБ) и необходимо обеспечить избыточность при хранении данных, для НБГИ в настоящее время предусмотрено хранилище с полезным объемом 50 ПБ [7].
Важной особенностью генетических данных является маленький средний размер файла, ~1 Мбайта. Так, 1 ПБ генетических данных включает в себя около миллиарда файлов, что значительно превышает возможности современных систем хранения. Поэтому при разработке НБГИ были приняты меры для решения данной проблемы: генетические данные объединяются в архивы перед размещением в системе хранения, сервисы по работе с данными извлекают файлы из архивов напрямую, используется объектная система хранения, позволяющая разместить множество файлов без падения производительности.
Модель данных
Для организации данных используется модифицированный подход, реализованный ранее на примере INSDC, которая представляет собой одну из самых известных глобальных инициатив в области обмена генетическими данными, сформировавшаяся в начале 1980-х гг. [6]. Участниками INSDC была создана глобальная всеобъемлющая коллекция нуклеотидных последовательностей и связанных с ними метаданных, находящихся в открытом доступе. Данные варьируются от необработанных прочтений, геномных сборок и выравниваний до разнообразной функциональной аннотации. Регулярный обмен данными, стандартизированные форматы и все чаще обмен технологиями обеспечивают глобальную синхронность в рамках сотрудничества. За счет указанных выше достоинств иерархия метаданных и их стандарты применяются в базах, не входящих в INSDC, в частности в NGDC (National Genomics Data Center, Национальный центр геномных данных, Китай) [8].
Верхним уровнем организации данных является биопроект (BioProject), упрощающий классификацию и систематизацию данных и метаданных для всех участников INSDC. Биопроект – это главным образом метаданные об исследовательских проектах, которые позволяют объединить большие объемы научной информации, упростить ее поиск, повысить доступность для пользователей баз данных, а также объединить участников исследования в едином информационном контексте. База данных биообразцов (BioSample) была разработана для хранения описательной информации (метаданных) непосредственно биологических образцов, из которых в дальнейшем были получены депонируемые генетические данные [9]. Следующим (нижним) уровнем организации в базах INSDC является уровень описания генетических данных – метаданные о геномных сборках и данных секвенирования.
В НБГИ сохраняются уровни метаданных, представленные в базах INSDC, и добавляется новый уровень метаданных – биообъект, располагающийся в иерархии между биопроектом и биообразцом (рис. 2). Биообъект позволяет связать несколько биообразцов, отобранных у одного живого организма или одной культуры клеток, но при различных условиях, в разные моменты времени или из разных тканей. Например, биообъектом могут являться метаданные по конкретной лабораторной крысе (R. norvegicus) с описанием общих характеристик этого животного, в то время как биообразцами будут метаданные по каждому отдельному забору биоматериала в разные промежутки времени после воздействия лекарственными веществами. Введение биообъекта сохраняет совместимость с метаданными баз INSDC, так как при импорте данный уровень может быть восстановлен на основании метаданных биообразца.
Рис. 2.
Иерархия метаданных макета НБГИ. Серым цветом выделены уровни метаданных, непосредственно связанные с генетическими данными.
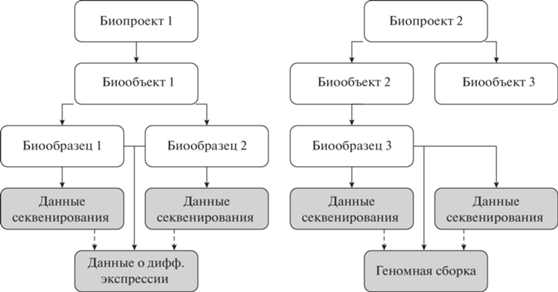
Таким образом, каждый из уровней иерархии метаданных позволяет решать отдельную задачу пользователя: биопроект – поиск исследований по тематике и задачам, а также структурирование и связывание объектов более низких уровней, биообъект – сохранение метаданных по конкретному объекту исследования с возможностью поиска по атрибутам и структурирование образцов, полученных из этого объекта различным способом и/или при различных условиях, биообразец – сохранение метаданных по конкретному образцу объекта исследования, включая все нюансы пробоотбора и воздействий на организм с учетом возможных динамических изменений.
Одним из важнейших атрибутов метаданных является таксономический идентификатор. За основы таксономии в НБГИ была взята система NCBI Taxonomy – курируемая база данных, организованная в виде направленного графа и систематизирующая информацию о всех доменах живых организмов [10]. На начало 2023 г. NCBI Taxonomy включала в себя информацию более чем о 100 000 родов и 2 млн. видов, причем база данных динамично изменяется как за счет пополнений (в 2022 г. было добавлено 80 тыс. новых видов и почти 4 тыс. новых родов), так и за счет адаптации изменений, вносимых в основные таксономические кодексы [11]. В таксономию включены как семь основных рангов: домен или надцарство (эукариоты, археи, бактерии, вирусы), филум или тип, класс, порядок, семейство, род и вид, так и промежуточные, которые позволяют более точно классифицировать организмы. Стоит учесть, что таксономия NCBI не является единственной используемой в биоинформатических базах данных, так, большую популярность имеют таксономические классификации в рамках проекта Silva-LTP (All-species Living Tree Project) и база данных геномной таксономии GTDB (Genome taxonomy database), классифицирующая прокариот на основании их геномных последовательностей и филогении маркерных генов [12, 13]. В НБГИ обеспечивается поддержка нескольких вариантов таксономической классификации с возможностью дальнейшей их взаимной интеграции.
2. ПРОГРАММНОЕ ОБЕСПЕЧЕНИЕ И ИНТЕРФЕЙС
Архитектура программного решения
Архитектура ПО предусматривает выполнение следующих задач:
– высокопроизводительную обработку генетической информации с использованием суперкомпьютерных ресурсов НИЦ “Курчатовский институт”;
– загрузку генетической информации, поиск (по метаданным, гомологический, BLAST и его варианты) и доступ к информации;
– таксономическую и фенотипическую классификацию биообразцов;
– геномный анализ;
– совместную работу с биопроектами, включая среду коммуникаций, единое информационное пространство для разработчиков и исследователей.
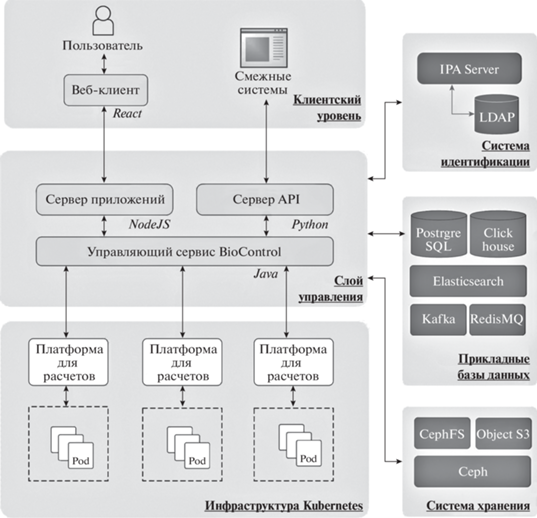
Анализ данных проводится с использованием гетерогенной инфраструктуры, включающей в себя облачную платформу и систему высокопроизводительных вычислений. Вычислительные возможности НБГИ будут предоставляться исследователям для коллективного пользования в рамках доступных квот. Аналитические конвейеры формируются из программных обработчиков, представленных в НБГИ. Пользователь может разработать и передать для использования в НБГИ свой обработчик, описав его в соответствии с заданными правилами. Прикладной программный интерфейс (API) НБГИ будет обеспечивать поддержку широкого спектра прикладных программных пакетов и платформ для геномного анализа. Система контейнеризации содержит прикладные программы вместе с их средами выполнения, необходимыми для каждой прикладной программы, что позволит организовывать конвейеры для анализа данных как на суперкомпьютерных, так и на облачных ресурсах. Архитектура НБГИ представлена на рис. 3.
Также в системе предусмотрена интеграция с популярными биоинформатическими платформами, которая обеспечивает бесшовный запуск пользовательских сценариев на одной из платформ из веб-портала или через общедоступный API. Поддержка данных платформ обеспечивает доступность большого набора готовых инструментов и сценариев. Для бесшовного взаимодействия с платформами разработан управляющий сервис BioControl, обеспечивающий унифицированный интерфейс для взаимодействия с биоинформатическими платформами, в том числе для автоматического создания системного окружения для вызова сценариев, а также мониторинга и управления вычислительными задачами.
Пользователи будут взаимодействовать с НБГИ через единый информационный портал (web portal), а также с использованием программного интерфейса. Для центров секвенирования с большим объемом секвенируемых данных целесообразно создание выделенных каналов связи.
Интерфейс
Основными разделами веб-портала НБГИ являются:
− библиотека данных, содержащая депонированные данные, аннотирование, метаданные, реверсные данные;
− сервисы личного кабинета пользователя;
− консоль администрирования, включающая в себя средства управления НБГИ, мониторинга и диагностики информационно-вычислительной инфраструктуры.
Основные сервисы будут доступны пользователям после авторизации и включают в себя:
− депонирование и валидацию генетических данных;
− анализ генетических данных, включая преобразование форматов и предварительную обработку;
− поиск и визуализацию генетических данных, включая средства геномного браузера и 3D-визуализации;
− мониторинг качества данных, обеспечение средств для работы кураторов данных;
− систему уведомлений, обеспечивающую отправку сообщений по коммуникационным средствам.
3. ДЕПОНИРОВАНИЕ ДАННЫХ
В ходе процесса депонирования данных осуществляются ввод метаданных, загрузка генетических данных и их проверка. Метаданные создаются или выбираются из уже созданных в концепции уровней метаданных. В НБГИ предусмотрены обязательные поля для метаданных в зависимости от уровня метаданных и типа биобразца, а также могут быть добавлены группы полей, из которых требуется заполнить не все, а хотя бы одно поле. Пользовательский ввод в поля метаданных проверяется на соответствие типу поля (число, строка, дата и т.д.), а также правилам заполнения, если они указаны (например, ограничение возраста образца от 0 до 200 лет). Одним из ключевых полей при заполнении метаданных является выбор таксона, который осуществляется поиском узла в существующей в системе таксономии. После прохождения автоматических проверок на корректность введенных метаданных пользователю предоставляется возможность загрузить генетические данные следующими способами:
− через веб-интерфейс портала НБГИ;
− через загрузку данных клиентским ПО (например, ftp/webdav-клиенты, rsync);
− с помощью API.
В НБГИ запланирована поддержка следующих типов и форматов данных:
− данные секвенирования, в том числе высокопроизводительного (ab1, fastq, h5, fast5);
− данные секвенирования, картированные на референсный геном: (BAM, SAM, CRAM);
− нуклеотидные и аминокислотные последовательности (fasta);
− аннотированные нуклеотидные и аминокислотные последовательности (gbk, gff, gtf);
− структуры биомолекул;
− данные о структурных вариациях в геноме (vcf);
− данные о треках (bed, bigwig, bedgraph);
− данные геномного и транскриптомного профилирования при помощи чипов (mictoarray);
− данные трехмерной структуры молекул, включая белки и нуклеиновые кислоты (PDB, PDBx, mmCIF).
В НБГИ реализована пакетная загрузка генетических данных, обеспечивающая автоматизированную загрузку и позволяющая проводить загрузку файлов большого размера или большого количества файлов. Пакетная загрузка представляет собой загрузку данных из файла, сформированного в текстовом формате с указанием перечня файлов и необходимых метаданных. Также в НБГИ разработаны протоколы обмена генетическими и метаданными с другими базами данных генетической информации.
4. АНАЛИТИЧЕСКИЕ ФУНКЦИОНАЛЬНЫЕ ВОЗМОЖНОСТИ
Поиск гомологичных последовательностей
Поиск гомологичных последовательностей реализуется при помощи программ для парного выравнивания как нуклеотидных (ДНК и РНК), так и аминокислотных (белки) последовательностей и включает в себя несколько режимов работы: поиск в нуклеотидной базе данных по нуклеотидному запросу; поиск в белковой базе данных по аминокислотному запросу; поиск в белковой базе данных по нуклеотидному запросу, выравнивание формируется на основе шести вариантов трансляции запроса; поиск в нуклеотидной базе данных по аминокислотному запросу, выравнивание формируется на основе шести вариантов трансляции референсных данных; поиск в нуклеотидной базе данных по нуклеотидному запросу, выравнивание формируется на основе 36 вариантов трансляции запроса и референсных данных.
Задание запроса и указание параметров пользователем включает в себя три этапа:
– формирование запроса осуществляется вставкой искомой последовательности в специальное окно интерфейса или загрузкой файла;
– выбор базы данных в соответствии с задачами пользователя и типом последовательности;
– указание параметров – необязательный шаг, так как параметры по умолчанию, подходящие для типовых задач уже предустановлены.
Просмотр поисковой выдачи – это краткая таблица результатов, включающая в себя основные характеристики выравниваний, графическое отображение выравниваний и просмотр выравниваний. Из окна просмотра поисковой выдачи возможен переход на соответствующую запись в НБГИ. Для удобства пользователей может быть реализована функция скачивания последовательности или последовательностей непосредственно из выдачи инструмента поиска по гомологии.
Геномный браузер
Пользователь может визуализировать геномные данные в геномном браузере, поддерживающем аннотации в стандартных биоинформатических форматах, в том числе в форматах, поддерживаемых обработчиками НБГИ. Геномный браузер ориентирован на изучение структуры генома, выбор определенных геномных регионов и работы с ними, включая отдельные последовательности, размеченные в данных регионах. Вторым приложением для пользователя является визуальный осмотр структуры гена, включая расположения доменов, наличие специфических подпоследовательностей, вторичной и третичной структуры гена. Геномный браузер обеспечивает пользователю доступ как к публичным данным, так и к данным ограниченного доступа, депонированных этим пользователем.
Типовые сценарии обработки данных
Валидация данных в процессе депонирования обеспечивает их целостность, соответствие заявленному биоинформатическому формату и возможность дальнейшей обработки и использования пользователями НБГИ. Депонирование данных в НБГИ осуществляется в различных форматах, некоторые из которых могут иметь специфические особенности. Например, валидация парности применяется для проверки парных прочтений в формате FASTQ – обычно такие данные представлены в виде пары файлов и должны иметь одинаковое количество записей. При этом их предварительная обработка пользователем может привести к нарушению соответствия записей в файлах и привести к ошибкам или невозможности дальнейшей обработки.
Типовые сценарии обработки генетических данных могут быть реализованы как индивидуальными обработчиками, так и программными конвейерами. К типовым сценариям, выполняемым элементарными обработчиками, относятся:
− получение количественных статистических данных о геномных сборках: суммарная длина, число и медианная длина контигов, Г+Ц-состав;
− оценка качества и статистические характеристики данных секвенирования: число прочтений, их средняя длина и ее стандартное отклонение, распределение значений качества и Г+Ц-состава, определение доли служебных последовательностей;
− таксономическая классификация прочтений и геномных сборок;
− оценка полноты и контаминации геномных сборок прокариот и эукариот на основании поиска и подсчета маркерных генов;
− картирование прочтений на референсный геном;
− быстрый поиск гомологичных генов;
− поиск последовательностей, соответствующих скрытым марковским моделям и ковариационным моделям.
Инструменты обработки данных, представляющие собой комбинацию элементарных обработчиков и при необходимости баз данных, могут выполнять следующие задачи, например сборка геномов с предварительной обработкой прочтений, предсказание белок-кодирующих генов, аннотация геномов прокариот и низших эукариот, анализ полнометагеномных данных. Также будет доступно значительное количество программных конвейеров с модульной архитектурой, позволяющих проводить: поиск мутаций в соматических и зародышевых клетках на основании полногеномного или таргетного секвенирования; анализ данных транскриптомного секвенирования с подсчетом числа генов и изоформ и обширным контролем качества; анализ метилирования ДНК на основании данных бисульфитного секвенирования; анализ данных ампликонного секвенирования для метагеномных исследований; анализ древней ДНК с высокой воспроизводимостью; сборку метагеномов и реконструкцию геномов некультивируемых организмов; сборку и аннотацию бактериальных геномов; филогенетический анализ бактериальных геномов с использованием данных секвенирования.
Для НБГИ были проведены разработка и тестирование программных конвейеров для следующих задач: генотипирование посредством полногеномного или ампликонного секвенирования; анализ качества и обработка данных секвенирования; сборка и аннотация прокариотического генома; филогенетический анализ с множественным выравниванием последовательностей с дальнейшей автоматической обработкой выравнивания.
Предлагаемые пользователям возможности анализа генетических данных подходят для использования как в крайне широком круге задач (биотехнология, биомедицина, молекулярная эволюционная биология, сельское хозяйство, животноводство, микробиология, экология и др.), так и на различных этапах анализа – от предварительной обработки данных до получения финальных результатов.
5. МАКЕТ НБГИ
С целью апробации технических решений, принимаемых в ходе технического проектирования НБГИ, в НИЦ “Курчатовский институт” создан макет системы. Макет НБГИ представляет собой программно-аппаратный комплекс, развернутый на доступных вычислительных ресурсах НИЦ “Курчатовский институт” и включающий в себя как вновь разрабатываемое ПО, так и заимствованное открытое ПО. Макет НБГИ обеспечивает возможность одновременной работы до 20 пользователей; организовано хранилище с суммарным полезным объемом 1 ПБ.
Комплекс прикладного ПО реализует следующие функциональные возможности:
– депонирование генетических данных. Поддерживаются форматы .fasta, .fastq. Реализована возможность пакетной загрузки данных путем предварительной загрузки описания в формате .tsv и самих файлов данных в рабочую область хранилища;
– валидация генетических данных. Автоматически проводится проверка форматов данных и аннотирование нуклеотидных последовательностей;
– хранение и организация доступа с учетом разграничения прав доступа к генетическим данным. Объектно-ориентированная модель данных включает в себя следующую структуру: биопроект → биообразец → биообъект;
– поиск генетических данных как на основе указанного описания (метаданных), так и на основе схожести нуклеотидных последовательностей;
– работа с данными из внешних источников с сохранением кросс-связей между данными и индексацией для быстрого поиска;
– анализ данных с использованием биоинформатических конвейеров. Реализована интеграция популярных платформ, предоставляющих широкий набор готовых сценариев;
– работа с системой возможна в защищенной сети посредством разработанного тонкого-клиента – веб-портала.
Пилотная эксплуатация подтвердила корректность заложенных технических решений в архитектуру системы, а также подтвердила возможность масштабирования вычислительной платформы и инфраструктуры хранения данных. В настоящее время НБГИ доступна в НИЦ “Курчатовский институт”, внешним пользователям может быть предоставлен удаленный доступ по согласованию с разработчиками. Сформированные на основе опыта использования макета НБГИ предложения по улучшению функциональных характеристик и повышению удобства работы будут учтены при создании системы в промышленном исполнении.
ЗАКЛЮЧЕНИЕ
В настоящее время система доступна для тестирования в режиме пилотной (экспериментальной) эксплуатации. Поправки в Федеральный закон 2022 от 29 декабря г. № 643-ФЗ “О государственном регулировании в области генно-инженерной деятельности”, согласно которому в НБГИ должны в обязательном порядке депонироваться данные, полученные в ходе генно-инженерной деятельности и молекулярно-генетического анализа, вступают в силу 1 сентября 2024 г. При этом не позднее 31 декабря 2025 г. в НБГИ должна быть депонирована генетическая информация, полученная государственными корпорациями, компаниями, бюджетными и муниципальными учреждениями, а также хозяйственными обществами, в уставном капитале которых доля участия Российской Федерации, субъектов РФ и муниципальных образований составляет не менее 50% [2].
Работы по cозданию НБГИ ведутся во исполнение Перечня поручений Президента Российской Федерации по итогам совещания по вопросам развития генетических технологий в Российской Федерации от 14 мая 2020 г. № Пр-920 (№ Пр-920 от 04.06.2020г.)
Список литературы
Rigden D.J., Fernández X.M. // Nucleic Acids Res. 2022. V. 50. № D1. P. D1.https://doi.org/10.1093/nar/gkab1195
Федеральный закон № 643-ФЗ от 29.12.2022 “О внесении изменений в Федеральный закон “О государственном регулировании в области генно-инженерной деятельности”.
https://www.strand-ngs.com/support/ngs-data-storage-requirements
https://kubernetes.io/
Cantelli G., Bateman A., Brooksbank C. et al. // Nucleic Acids Res. 2022. V. 50. № D1. P. D11. https://doi.org/10.1093/nar/gkab1127
Arita M., Karsch-Mizrachi I., Cochrane G. et al. // Nucleic Acids Res. 2021. V. 49. № D1. P. D121. https://doi.org/10.1093/nar/gkaa967
Ogle C., Reddick D., McKnight C. et al. // Frontiers in Big Data. 2021. V. 4. P. 582468. https://doi.org/10.3389/fdata.2021.582468
National Genomics Data Center Members and Partners // Nucleic Acids Res. 2020. V. 48. № D1. P. D24. https://doi.org/10.1093/nar/gkz913
Barrett T., Clark K., Gevorgyan R. et al. // Nucleic Acids Res. 2012. V. 40. № D1. P. D57. https://doi.org/10.1093/nar/gkr1163
Schoch C.L., Ciufo S., Domrachev M. et al. // Database. 2020. V. 2020. P. baaa062. https://doi.org/10.1093/database/baaa062
https://www.ncbi.nlm.nih.gov/Taxonomy/taxonomyhome.html/index.cgi?chapter=statistics&?&m=0
Yilmaz P., Parfrey L.W., Yarza P. et al. // Nucleic Acids Res. 2014. V. 42. № D1. P. D643. https://doi.org/10.1093/nar/gkt1209
Parks D.H., Chuvochina M., Rinke C. et al. // Nucleic Acids Res. 2022. V. 50. № D1. P. D785. https://doi.org/10.1093/nar/gkab776
Дополнительные материалы отсутствуют.
Инструменты
Российские нанотехнологии