Журнал общей биологии, 2019, T. 80, № 5, стр. 364-371
Использование транскрибируемых спейсеров ITS1 и ITS2 для идентификации беззубок родов Anodonta и Pseudanodonta (Bivalvia: Unionidae: Anodontinae)
А. А. Томилова 1, *, А. В. Кондаков 1, 2, О. Я. Кисиль 2
1 Федеральный исследовательский центр комплексного изучения Арктики им. акад. Н.П. Лаверова РАН
163000 Архангельск, наб. Северной Двины, 23, Россия
2 Северный (Арктический) федеральный университет им. М.В. Ломоносова
163002 Архангельск, наб. Северной Двины, 17, Россия
* E-mail: tomilova_alyona@mail.ru
Поступила в редакцию 17.05.2018
После доработки 14.01.2019
Принята к публикации 24.06.2019
Аннотация
В настоящий момент существуют трудности в идентификации пресноводных моллюсков родов Anodonta и Pseudanodonta при использовании морфологических методов. В публикации рассмотрена возможность применения для этих целей транскрибируемых спейсеров ITS1 и ITS2 в качестве молекулярно-генетических маркеров. Было подтверждено, что данный молекулярный подход совместно с морфологическими признаками позволяет установить видовую принадлежность представителей родов Anodonta и Pseudanodonta.
Моллюски родов Anodonta (Lamarck, 1799) и Pseudanodonta (Bourguignat, 1877) относятся к классу пресноводных двустворчатых, или пластинчатожаберных, подсем. Anodontinae (беззубки) из сем. Unionidae. Представители этих родов повсеместно распространены и могут быть найдены как в водотоках, так и стоячих водах. Они выполняют одну из основных ролей в пресноводных экосистемах и являются важным звеном комплекса организмов, участвующем в самоочищении водных объектов (Шаплыгина и др., 2016).
Для идентификации двустворчатых моллюсков используют преимущественно морфологический метод, основанный на оценке общей формы раковины, ее выпуклости, положения наиболее выступающей точки боковой поверхности створки, в некоторых случаях анализируют мягкое тело (Богатов и др., 2005). Для моллюсков родов Anodonta и Pseudanodonta характерна значительная морфологическая изменчивость (Klishko et al., 2018), поэтому ошибки в идентификации представителей этих родов всегда были проблемой для исследователей. В таких случаях в дополнение к общепринятым компараторным методам целесообразно применять молекулярно-генетические подходы. При исследовании большинства биологических объектов необходимым этапом является определение систематического положения особей, так как неправильное определение видовой принадлежности может повлечь за собой ошибочные выводы (Ворошилова, 2016).
В настоящее время существует широкий спектр разнообразных молекулярных маркеров, которые применяются для исследования полиморфизма нуклеотидных последовательностей. Для митохондриальной ДНК (мтДНК) эукариот характерна высокая скорость накопления генетических замен как у близкородственных видов, так и в пределах одного вида (Ballard, Whitlock, 2004). На сегодняшний день нуклеотидные последовательности участка первой субъединицы цитохром c-оксидазы (COI) применяются в ДНК-баркодинге для идентификации животных чаще, чем любой другой митохондриальный ген (Hebert et al., 2003).
Митохондриальная ДНК эволюционирует (накапливает генетические замены) быстрее, чем ядерная. При этом в ядерной ДНК стоит обратить внимание на межгенные спейсеры ITS (Internal Transcribed Spacers), которые являются некодирующими последовательностями и эволюционируют существенно быстрее по сравнению с участками рибосомального кластера (Baldwin et al., 1995; Alvarez, Wendel, 2003). Различия длин внутренних транскрибируемых спейсеров ITS1 и ITS2 связаны с присутствием в них нуклеотидных вставок и делеций, что является удобным для исследований дивергенции близкородственных организмов (Bargues, Mas-Coma, 2005).
Ранее на основе участков ITS1 и ITS2 были созданы молекулярные определительные ключи для пресноводных моллюсков Северо-Западной Европы (Källersjö et al., 2005). Кроме того, была выполнена молекулярная идентификации видов сем. Unionidae при помощи анализа длин последовательностей ITS1 после обработки специально подобранными рестриктазами (Gerke, Tiedemann, 2001; Zieritz et al., 2012).
Актуальность данной работы связана с тем, что идентификация моллюсков родов Anodonta и Pseudanodonta с помощью сравнения длин молекулярных маркеров ITS1 и ITS2 существенно облегчит и ускорит этот процесс, и исключит необходимость дорогостоящих операций по определению нуклеотидных последовательностей (секвенирование).
Задачами данной работы были оценка пригодности использования длин нуклеотидных последовательностей транскрибируемых спейсеров ITS1 и ITS2 для идентификации двустворчатых моллюсков родов Anodonta и Pseudanodonta и сравнение длин участков ITS1 и ITS2 для некоторых видов родов Anodonta, Pseudanodonta, Unio и Sinanodonta.
МАТЕРИАЛЫ И МЕТОДЫ
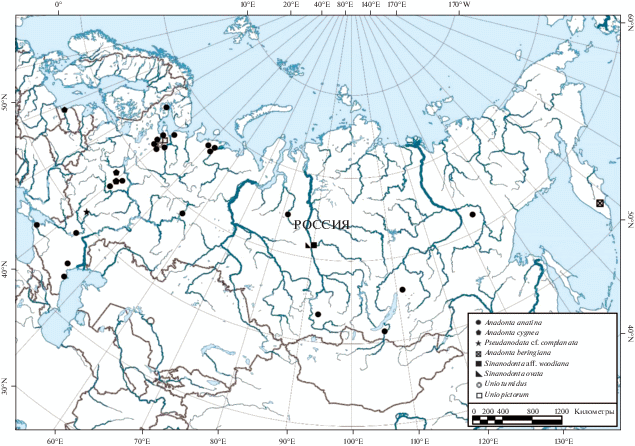
В работе использованы сборы моллюсков отряда Unionoida, выполненные в период с 2013 по 2016 год в водоемах различных бассейнов рек России (табл. 1, рис. 1). В качестве объекта исследования выступали двухстворчатые моллюски Anodonta anatina (L., 1758), A. cygnea (L., 1758), Pseudanodonta cf. complanata (Rossmässler, 1835) из трибы Anodontini (Lopes-Lima et al., 2017). Для сравнения были использованы виды A. beringiana (Middendorf, 1851), Sinanodonta aff. woodiana (Lea, 1834), S. ovata (Bogatov et Starobogatov, 1996) из того же подсем. Anodontinae, но из другой трибы Cristariini (Lopes-Lima et al., 2017). Кроме того, для сравнения использованы представители соседнего подсем. Unionidae: Unio tumidus (Retzius, 1788) и U. pictorum (L., 1758). Ткани исследуемых образцов зафиксированы в 96%-ном этиловом спирте и хранятся в коллекции “Российского музея центров биоразнообразия” на базе ФИЦКИА РАН. Выборка представителей A. anatina более многочисленная, включает 26 особей; также исследованы шесть особей A. cygnea. Вид P. cf. complanata – редкий и охраняемый, поэтому в данном исследовании представлен только двумя особями. В качестве дополнения данных по длине транскрибируемых спейсеров ITS1 и ITS2 включены данные из базы NCBI GenBank11.
Таблица 1.
Список секвенированных и проанализированных последовательностей COI двустворчатых моллюсков
| Вид | Бассейн реки | Номер образца по каталогу | Номер в GenBank |
|---|---|---|---|
| Anodonta anatina | Индига | biv 156/1 | KY328448 |
| biv 156/2 | KY328449 | ||
| Волга | biv 157/1 | KY328453 | |
| Верхняя Ангара | biv 158/1 | KY328455 | |
| Юг | biv 162 | KY328458 | |
| Онега | biv 164/1 | KY328459 | |
| Северная Двина | biv 168/2 | KY328461 | |
| Таз | biv 173/1 | KY328466 | |
| Волга | biv 174/1 | KY328469 | |
| Кереть | biv 189/1 | KY328471 | |
| Селенга | biv 190/2 | KY328474 | |
| Кубань | biv 192 | KY328479 | |
| Москва | biv 194/4 | KY328480 | |
| biv 194/5 | KY328481 | ||
| Обь | biv 198/1 | KY328483 | |
| biv 198/2 | KY328484 | ||
| Лена | biv 199/1 | KY328488 | |
| biv 199/2 | KY328489 | ||
| Печора | biv 206/1 | KY328493 | |
| Дон | biv 208/3 | KY328496 | |
| Ока | biv 209 | KY328497 | |
| Печора | biv 210/1 | KY328498 | |
| biv 210/3 | KY328499 | ||
| Северная Двина | IEPN 669/2 | KY328502 | |
| Кулой | IEPN 670/1 | KY328505 | |
| Северная Двина | 1HH | KY328509 | |
| Anodonta cygnea | Москва | biv 194/1 | MK034153 |
| biv 194/2 | MK034154 | ||
| biv 204/1 | MK034157 | ||
| biv 204/3 | MK034158 | ||
| Преголя | biv 207/1 | MK034159 | |
| biv 207/3 | MK034160 | ||
| Pseudanodonta cf. complanata | Хопер | biv 195/1 | MK034155 |
| biv 195/2 | MK034156 | ||
| Anodonta beringiana | Авача | biv 169 | MK034152 |
| Sinanodonta aff. woodiana | Енисей | biv 191/2 | KY978735 |
| biv 191/5 | KY978738 | ||
| Sinanodonta ovata | biv 191/1 | KY561633 | |
| Unio tumidus | Северная Двина | IEPN 669/6 | MK034161 |
| Unio pictorum | 3НН | MK034162 |
Выделение тотальной клеточной ДНК из тканей пресноводных моллюсков осуществлено с применением фенол-хлороформной экстракции (Sambrook et al., 1989). Амплификация последовательностей проведена при помощи праймеров ITS1F и ITS1R (White et al., 1996) для ITS1, LT1F (Bargues et al., 2001) и ITS4R (White et al., 1996) для ITS2, LCO1490 и HCO2198 (Folmer et al., 1994) для COI. ПЦР-смесь содержала около 200 нг ДНК, 10 пмоль каждого из праймеров, 2.5 мкл Taq-буфера (с 10 × × 2 ммоль MgCl2), 200 мкмоль каждого из dNTP, 0.8 единиц Taq ДНК-полимеразы (НПО “СибЭнзим”, Россия). Реакционная смесь была доведена деионизированной водой до объема 25 мкл. Амплификация участков включала в себя этап первоначальной денатурации ДНК – 95°С, 5 мин; затем 31–37 циклов синтеза фрагмента ДНК: 95°С – 45 с, 57°С (для ITS1) и 50°С (для ITS2 и COI) – 40 с, 72°С – 50 с; а также этап окончательной элонгации цепи – 72°С, 5 мин.
Определение генетических последовательностей фрагмента гена COI у всех исследуемых образцов двустворчатых моллюсков осуществлено при помощи автоматического секвенатора (ABI PRISM® 3730, Applied Biosystems) с использованием набора реагентов ABI PRISM® BigDye™ Terminator v. 3.1. (ЦКП “Геном”, ИМБ РАН). Полученные последовательности гена COI были проанализированы в программе BLAST NCBI для определения видовой принадлежности образцов моллюсков.
Рибосомальный кластер эукариот имеет в своем составе два внутренних транскрибируемых спейсера – ITS1 и ITS2, которые разделяют в рибосомной ДНК гены 18S, 5.8S и 28S РНК (White et al., 1996; Källersjö et al., 2005). Данные последовательности представляют собой объединение ядерных генов, которые располагаются в виде тандемных повторов (рис. 2).
Рис. 2.
Локализация праймеров на рибосомальном кластере и размер рибосомальных генов, амплифицированных совместно с ITS1 и ITS2.
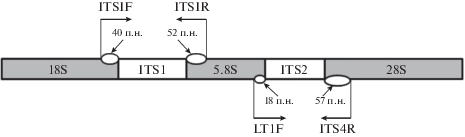
Поскольку подобранные праймеры отжигаются на последовательностях рибосомальных генов (ITS1F на 18S рРНК, ITS1R на 5.8S рРНК, а LT1F на 5.8S рРНК, ITS4R на 28S рРНК), то амплифицированные последовательности ITS1 и ITS2 содержат участки рибосомальных генов.
Определение длин нуклеотидных последовательностей ITS1 и ITS2 осуществлено с помощью системы автоматического капиллярного электрофореза Experion™ (Bio-Rad, США) с использованием чипа 1K Analysis Kit согласно прилагаемым инструкциям. В него вносили по 11 образцов совместно с маркерной линейкой, имеющей диапазон 15–1500 п.н.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Для того чтобы найти истинный размер амплифицированных участков ITS1 и ITS2 для каждого отдельного вида, был определен размер рибосомальных генов 18S, 5.8S и 28S рДНК путем сравнения с нуклеотидными последовательностями JX081669, GU471863, KJ740491 из базы GenBank. Из полученных последовательностей ITS1 и ITS2 вычли длину рибосомальных генов, которые были амплифицированы совместно с внутренними транскрибируемыми спейсерами. Длина данных участков составила: для ITS1 – 40 п.н. при отжиге праймера ITS1F на 18S рРНК и 52 п.н. при отжиге праймера ITS1R на 5.8S рРНК; для ITS2 – 18 п.н. при отжиге праймера LT1F на 5.8S рРНК и 57 п.н. при отжиге праймера ITS4R на 28S рРНК (рис. 2).
Из анализа амплифицированных спейсеров ITS1 и ITS2 следует, что эти последовательности варьируют по длине (табл. 2). Можно заметить, что диапазоны длин последовательностей ITS2 P. cf. complanata (от 310 до 321 п.н.), A. cygnea (от 315 до 329 п.н.), A. anatina (от 321 до 343 п.н.) имеют перекрывающие области, однако длина ITS1 значительно отличается: P. cf. complanata (от 462 до 464 п.н.), A. cygnea (от 448 до 449 п.н., от 470 до 472 п.н.), A. anatina (от 495 до 527 п.н.) – что позволяет отличить данные виды. По длине ITS1 для пресноводных моллюсков видов U. tumidus (315 п.н.) и U. pictorum (333 п.н.) можно предположить, что особи первого вида относятся либо к P. cf. complanata (от 310 до 321 п.н.), либо к A. cygnea (от 310 до 329 п.н.), а второго вида – к A. anatina (от 321 до 343 п.н.), однако данное предположение опровергает интервал последовательностей ITS2. А длина транскрибируемого спейсера ITS2 представителей вида S. ovata (273 п.н.) входит в диапазон аналогичного участка для S. aff. woodiana (от 273 до 282 п.н.), но отличается по участку ITS1: S. ovata (496 п.н.), S. aff. woodiana (502 п.н.).
Таблица 2.
Каталожные номера образцов двустворчатых моллюсков и длина амплифицированных последовательностей ITS1 и ITS2
| Вид | Номер образца по каталогу |
ITS1 (п.н.) | ITS2 (п.н.) | ||
|---|---|---|---|---|---|
| длина | диапазон | длина | диапазон | ||
| Anodonta anatina | biv 156/1 | 497 | 495–527 | 338 | 321–343 |
| biv 156/2 | 495 | 338 | |||
| biv 157/1 | 499 | 342 | |||
| biv 158/1 | 502 | 321 | |||
| biv 162 | 504 | 338 | |||
| biv 164/1 | 504 | 335 | |||
| biv 168/2 | 504 | 335 | |||
| biv 173/1 | 509 | 333 | |||
| biv 174/1 | 508 | 334 | |||
| biv 189/1 | 524 | 333 | |||
| biv 190/2 | 527 | 334 | |||
| biv 192/1 | 514 | 339 | |||
| biv 194/4 | 518 | 336 | |||
| biv 194/5 | 521 | 343 | |||
| biv 198/1 | 509 | 338 | |||
| biv 198/2 | 504 | 338 | |||
| biv 199/1 | 506 | 336 | |||
| biv 199/2 | 512 | 338 | |||
| biv 206/1 | 512 | 338 | |||
| biv 208/3 | 517 | 343 | |||
| biv 209 | 517 | 340 | |||
| biv 210/1 | 507 | 340 | |||
| biv 210/3 | 505 | 339 | |||
| IEPN 669/2 | 499 | 337 | |||
| IEPN 670/1 | 517 | 335 | |||
| 1HH | 508 | 341 | |||
| Anodonta cygnea | biv 194/1 | 449 | 448–449 470–472 |
325 | 315–329 |
| biv 194/2 | 448 | 320 | |||
| biv 204/1 | 472 | 327 | |||
| biv 204/3 | 470 | 329 | |||
| biv 207/1 | 471 | 325 | |||
| biv 207/3 | 470 | 315 | |||
| Pseudanodonta cf. complanata | biv 195/1 | 462 | 462–464 | 321 | 310–321 |
| biv 195/2 | 464 | 310 | |||
| Anodonta beringiana | biv 169 | 479 | 479 | 266 | 266 |
| Sinanodonta aff. woodiana | biv 191/2 | 502 | 502 | 273 | 273–282 |
| biv 191/5 | 502 | 282 | |||
| Sinanodonta ovata | biv 191/1 | 496 | 496 | 273 | 273 |
| Unio tumidus | IEPN 669/6 | 448 | 448 | 315 | 315 |
| Unio pictorum | 3НН | 416 | 416 | 333 | 333 |
Если принимать во внимание перекрывание длин последовательностей ITS1, то можно предположить, что особи вида S. ovata (496 п.н.) и S. aff. woodiana (502 п.н.) могут относиться к виду A. anatina (от 495 до 527 п.н.), однако это утверждение не верно, поскольку длины последовательностей ITS2 варьируют у пресноводных моллюсков вида A. anatina в других пределах (от 321 до 343 п.н.), что не характерно для особей вида S. ovata (273 п.н.) и S. aff. woodiana (от 273 до 282 п.н.).
Наши данные по длинам последовательностей спейсеров ITS1 и ITS2 позволяют расширить интервалы значений, установленные в предыдущем исследовании этих видов (Källersjö et al., 2005). В ходе исследования нами было выявлено и учтено, что полученные ранее последовательности спейсеров (Källersjö et al., 2005) были выравнены некорректно и, следовательно, имеют другую длину. Согласно современным данным, путем сравнения с последовательностями JX081669, GU471863, KJ740491 из базы GenBank нами были рассчитаны интервалы транскрибируемых спейсеров ITS1 и ITS2 для идентификации двустворчатых моллюсков (табл. 3). Полученные данные были объединены с результатами предыдущих исследований (Källersjö et al., 2005) и представлены в сводной табл. 4 и на рис. 3. При анализе последовательностей спейсера ITS2 для моллюсков родов Anodonta и Sinanodonta видно, что длины амплифицированных последовательностей существенно отличаются, что позволяет их идентифицировать (рис. 3). Моллюски вида P. cf. complanata достоверно отличаются от особей других видов длиной спейсера ITS1, а интервалы, полученные для моллюсков P. complanata, пересекаются с таковыми у A. anatina (по ITS1 и ITS2), что свидетельствует о том, что использование спейсеров не во всех случаях позволяет корректно определить видовую принадлежность этих моллюсков. В таких ситуациях для точной идентификации вида необходимо осуществлять ДНК-баркодинг или рестрикционный анализ с использованием эндонуклеазы рестрикции Hinfl (Zieritz et al., 2012).
Таблица 3.
Данные для длин амплифицированных последовательностей ITS1 и ITS2 согласно современным данным JX081669, GU471863, KJ740491 из GenBank
| Вид | Номер в GenBank |
ITS1 (п.н.) | ITS2 (п.н.) | ||
|---|---|---|---|---|---|
| длина | диапазон | длина | диапазон | ||
| Anodonta anatina | DQ060177 | 511 | 511–513 | 345 | 345 |
| DQ060178 | 511 | 345 | |||
| DQ060179 | 511 | 345 | |||
| DQ060180 | 513 | 345 | |||
| DQ060181 | 512 | 345 | |||
| Anodonta cygnea | DQ060185 | 486 | 480–486 | 335 | 335–339 |
| DQ060186 | 480 | 339 | |||
| Pseudanodonta complanata |
DQ060182 | 494 | 494–495 | 335 | 335 |
| DQ060183 | 495 | 335 | |||
| DQ060184 | 494 | 335 | |||
| Unio tumidus | DQ060191 | 450 | 450 | 341 | 341 |
| DQ060192 | 450 | 341 | |||
| Unio pictorum | DQ060189 | 434 | 434 | 335 | 335 |
| DQ060190 | 434 | 335 | |||
| Unio crassus | DQ060187 | 423 | 423–424 | 323 | 323–324 |
| DQ060188 | 424 | 324 | |||
Таблица 4.
Сводные данные для длин амплифицированных последовательностей ITS1 и ITS2
| Вид | ITS1 (п.н.) | ITS2 (п.н.) | ||
|---|---|---|---|---|
| Anodonta anatina | 495–527 | 495–527 | 321–343 | 321–345 |
| 511–513* | 345* | |||
| Anodonta cygnea | 448–449 470–472 |
448–449 470–486 |
315–329 | 315–339 |
| 480–486* | 335–339* | |||
| Anodonta beringiana | 479 | 479 | 266 | 266 |
| Pseudanodonta complanata | 494–495* | 494–495* | 335* | 335* |
| Pseudanodonta cf. complanata | 462–464 | 462–464 | 310–321 | 310–321 |
| Sinanodonta aff. woodiana | 502 | 502 | 273–282 | 273–282 |
| Sinanodonta ovata | 496 | 496 | 273 | 273 |
Рис. 3.
Схема длин транскрибируемых спейсеров ITS1 и ITS2 для представителей родов Anodonta, Pseudanodonta и Sinanodonta.
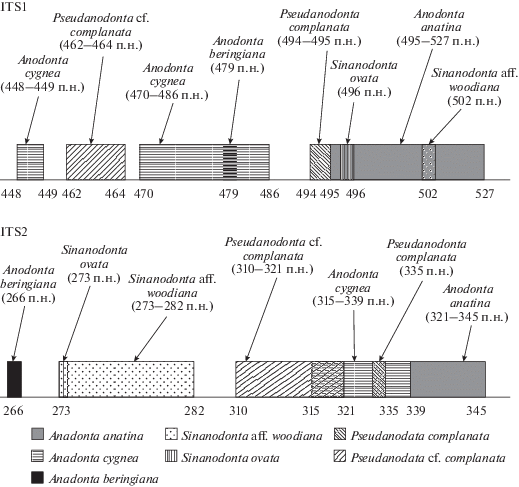
Полученные данные по транскрибируемым спейсерам ITS1 и ITS2 показывают, что у пресноводных моллюсков родов Anodonta и Pseudanodonta в разных частях ареала наблюдается не только морфологическая изменчивость, но и генетический полиморфизм.
Для подтверждения достоверности предлагаемого метода идентификации у всех исследуемых образцов двустворчатых моллюсков предварительно были определены генетические последовательности фрагмента гена COI. Различия полученных последовательностей, в сравнении с представленными в базе GenBank, не превысили 0.5% для всех исследуемых образцов моллюсков, кроме особей P. cf. complanata. Исследуемые образцы моллюсков P. cf. complanata, собранные в р. Хопер (бассейн р. Дон), отличаются от последовательностей гена COI P. complanata, задепонированных в базе данных GenBank, на 2%. Это свидетельствует о том, что данный вид является сестринским видом по отношению к P. complanata. Данные по депонированию последовательностей гена COI в базе GenBank представлены в табл. 1.
ЗАКЛЮЧЕНИЕ
Проведенное исследование показало, что совместное определение длин транскрибируемых спейсеров ITS1 и ITS2 может служить качественным маркером для определения видового статуса представителей родов Anodonta и Pseudanodonta, за исключением случаев идентификации видов P. complanata и A. anatina, которые легко могут быть идентифицированы морфологически или с применением рестрикционного анализа. Следовательно, молекулярно-генетические методы способны эффективно дополнить классические подходы в определении двустворчатых моллюсков.
Авторы благодарны И.Н. Болотову, И.В. Вихреву, М.В. Винарскому, М.Ю. Гофарову, А.А. Махрову, С.Е. Соколовой, О.В. Аксёновой, В.С. Артамоновой и другим коллегам, которые в разные годы участвовали в проведении полевых работ и сборе моллюсков.
Полевые работы по сбору материала и молекулярно-генетические исследования выполнены при поддержке государственного задания № 0409-2019-0041 и гранта РФФИ № 17-45-290066_р_а. Камеральная обработка собранных материалов и подготовка рукописи статьи выполнены при поддержке гранта РНФ № 18-77-00058.
Список литературы
Богатов В.В., Старобогатов Я.И., Прозорова Л.А., 2005. Моллюски рода Colletopterum (Anodontinae, Bivalvia) России и сопредельных территорий // Зоол. журн. Т. 84. № 9. С. 1050–1063.
Ворошилова И.С., 2016. Проблемы и перспективы применения молекулярно-генетических методов для таксономической ревизии пресноводных моллюсков // Тр. Ин-та биол. внутр. вод РАН. № 73. С. 12–24.
Шаплыгина Ю.Н., Курочкина Т.Ф., Насибулина Б.М., 2016. Интегральная значимость пресноводных моллюсков в самоочищении воды дельты реки Волги // Естеств. науки. Экология. Т. 55. № 2. С. 27–32.
Alvarez I., Wendel J.F., 2003. Ribosomal ITS sequences and plant phylogenetic inference // Mol. Phylogenet. Evol. V. 29. № 3. P. 417–434.
Baldwin B.G., Sanderson M.J., Porter J.M. et al., 1995. The ITS region of nuclear ribosomal DNA: A valuable source of evidence on angiosperm phylogeny // Ann. Mo. Bot. Gard. V. 82. № 2. P. 247–277.
Ballard J.W., Whitlock M.C., 2004. The incomplete natural history of mitochondria // Mol. Ecol. V. 13. № 4. P. 729–744.
Bargues M.D., Mas-Coma S., 2005. Reviewing lymnaeid vectors of fascioliasis by ribosomal DNA sequence analyses // J. Helminthol. V. 79. № 3. P. 257–267.
Bargues M.D., Vigo M., Horak P. et al., 2001. European Lymnaeidae (Mollusca: Gastropoda), intermediate hosts of trematodiases, based on nuclear ribosomal DNA ITS-2 sequences // Infect. Genet. Evol. V. 1. № 2. P. 87–107.
Folmer O., Black M., Hoeh W. et al., 1994. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates // Mol. Mar. Biol. Biotechnol. V. 3. № 5. P. 294–299.
Gerke N., Tiedemann R., 2001. A PCR-based molecular identification key to the glochidia of European freshwater mussels (Unionidae) // Conserv. Genet. V. 2. № 3. P. 285–287.
Hebert P.D., Cywinska A., Ball S.L. et al., 2003. Biological identifications through DNA barcodes // Proc. Roy. Soc. B. Biol. Sci. V. 270. № 1512. P. 313–321.
Källersjö M., Proschwitz T., von, Lundberg S. et al., 2005. Evaluation of ITS rDNA as a complement to mitochondrial gene sequences for phylogenetic studies in freshwater mussels: An example using Unionidae from north-western Europe // Zool. Scr. V. 34. № 4. P. 415–424.
Klishko O.K., Lopes-Lima M., Bogan A.E. et al., 2018. Morphological and molecular analyses of Anodontinae species (Bivalvia, Unionidae) of Lake Baikal and Transbaikalia // PloS One. V. 13. № 4. P. e0194944.
Lopes-Lima M., Froufe E., Do V.T. et al., 2017. Phylogeny of the most species-rich freshwater bivalve family (Bivalvia: Unionida: Unionidae): Defining modern subfamilies and tribes // Mol. Phylogenet. Evolut. V. 106. P. 174–191.
Sambrook J., Fritsch E.F., Maniatis T., 1989. Molecular Cloning: A Laboratory Manual. 2nd ed. N.Y.: Cold Spring Harbor Laboratory Press. 1626 p.
White L.R., McPheron B.A., Stauffer J.R., 1996. Molecular genetic identification tools for the unionids of French Creek, Pennsylvania // Malacologia. V. 38. № 1–2. P. 181–202.
Zieritz A., Gum B., Kuehn R. et al., 2012. Identifying freshwater mussels (Unionoida) and parasitic glochidia larvae from host fish gills: A molecular key to the North and Central European species // Ecol. Evol. V. 2. № 4. P. 740–750.
Дополнительные материалы отсутствуют.
Инструменты
Журнал общей биологии