Онтогенез, 2020, T. 51, № 6, стр. 403-416
Гистоновые шапероны семейства NAP: характеристика и роль в онтогенезе
А. А. Акишина a, Е. Е. Куваева a, Ю. Е. Воронцова a, О. Б. Симонова a, *
a Институт биологии развития им. Н.К. Кольцова РАН
119334 Москва, ул. Вавилова, 26, Россия
* E-mail: osimonova@hotmail.com
Поступила в редакцию 16.05.2020
После доработки 10.06.2020
Принята к публикации 15.06.2020
Аннотация
Гистоновые шапероны – это класс белков, которые связывают и транспортируют гистоны, предотвращая их хаотичное агрегирование при образовании нуклеосом. Гистоновые шапероны семейства NAP (Nucleosome Assembly Protein) содержат высоко консервативный центральный домен NAP, необходимый для связывания гистонов и сборки нуклеосом. Они являются неотъемлемым компонентом в создании и поддержании динамики эукариотического хроматина, от которой зависит транскрипция многих генов. В обзоре рассматривается семейство белков NAP и его конкретные представители: NAP1, NAP2 и CG5017/Hanabi. Будучи каноническими переносчиками гистонов, и обеспечивая эффективный ремоделлинг хроматина, белки семейства NAP участвуют в нейрональной дифференцировке, сперматогенезе и формировании долговременной памяти, что указывает на важность этого семейства в онтогенезе.
ВВЕДЕНИЕ
Сборка и разборка хроматина – это динамический биологический процесс, который обуславливается конформационной подвижностью нуклеосом. Благодаря ему осуществляется доступность эукариотического генома для репликации, транскрипции, репарации ДНК и клеточного цикла. Основной структурной единицей эукариотического хроматина является нуклеосома, в которой нить ДНК длиной 146 п.н. обернута вокруг гистонового октамера, состоящего из двух тетрамеров – каждый из которых складывается из гистоновых белков H2A, H2B, H3 и H4 (Luger et al., 1997; Hammond et al., 2017).
Сборка хроматина представляет собой поэтапный процесс, который начинается с присоединения гистонового тетрамера (H3-H4)2 к ДНК и последующим включением димеров H2A-H2B для образования нуклеосомы. В физиологических условиях гистоны и ДНК не способны самостоятельно собираться в нуклеосомы. Гистоновые шапероны – это класс белков, которые связывают гистоны, предотвращая формирование неспецифических агрегатов при образовании нуклеосомной частицы. Они являются ключевыми элементами, участвующими в поддержании стабильности и динамики гистоновых белков в клетке. Разнообразные структурные мотивы и олигомерные состояния обеспечивают электростатические и конформационно-специфические взаимодействия между контактными поверхностями гистонов и гистоновыми шаперонами, что приводит к перемещению последних от одного гистона к другому (“туда-обратно”) и упорядочиванию процесса сборки и разборки нуклеосом. Ковалентные модификации сдвигают гистон-связывающее равновесие шаперонов в сторону тех или иных партнеров до тех пор, пока не будет достигнуто наиболее стабильное состояние – собранная и позиционированная нуклеосома. Таким образом, гистоновые шапероны играют важную роль, обеспечивая эффективность процессов сборки и разборки нуклеосом, замещая гистоны новыми каноническими или вариантными формами. К таким шаперонам относят антисайленсинговый фактор 1 (Asf1), гистоновый регулятор (HIR), фактор сборки хроматина 1 (CAF-1), белки сборки нуклеосом (NAP – Nucleosome Assembly Protein) и др. (Elsässer, D’Arcy, 2012; Mattiroli et al., 2015; Valieva et al., 2016).
В последнее время накапливаются факты о разнообразии функций гистоновых шаперонов, которое они приобрели в ходе эволюции (Okuwaki et al., 2010). Однако основной функцией белков семейства NAP остается их контакт с гистоновыми димерами H2A-H2B, в результате чего обеспечивается правильная сборка и разборка базовых частиц нуклеосом. Недавно в бесклеточной системе было показано, что NAP1 мыши участвует в генерировании интермедиатных частиц – гексасом, вытесняя из нуклеосомы один из димеров H2A-H2B, содержащий убиквитинилированный гистон H2B (Krajewski, 2020). Кроме того, в последнее время особый интерес вызывает вовлеченность белков семейства NAP в процессы развития нервной системы (Qiao et al., 2018), поддержания пролиферации гемопоэтических стволовых клеток (Heshmati et al., 2018) у животных и развития различных органо-тканевых структур у растений (Zhou et al., 2015; Zhu et al., 2017; Barna et al., 2018). Понимание связи между разнообразием белков семейства NAP и специфичностью их взаимодействия с основными и вариантными гистонами важно для изучения ключевых клеточных процессов. Обзор посвящен характеристике семейства белков гистоновых шаперонов NAP (Nucleosome Assembly Protein) и его отдельным представителям – NAP1, NAP2 и CG5017.
СЕМЕЙСТВО БЕЛКОВ NAP (NUCLEOSOME ASSEMBLY PROTEIN)
В семейство NAP входят белки, содержащие высоко консервативный центральный домен NAP, состоящий примерно из 300 аминокислот. Основной функцией этого домена является связывание гистонов. В работе, посвященной описанию структуры дрожжевого белка NAP1 (yNAP1), дана характеристика домена NAP (Park, Luger, 2006). Этот белок имеет два домена. Домен I состоит из протяженной α-спирали, фланкированной двумя короткими α-спиралями. Он отвечает за димеризацию. Главной структурной особенностью домена II (домен взаимодействия с белками) является β-слой, состоящий из четырех антипараллельных β-цепей, который прикрыт α-спиралями с нижней стороны. В структуре этого домена присутствует небольшой выступающий участок, состоящий из антипараллельной β-шпильки – сайта ядерной локализации NLS (nuclear localization sequence) (Мosammaparast, 2002). Каждый домен поделен на два субдомена: A (домен I), B (домен I), C (домен II) и D (домен II). Субдомен А (показан синим цветом на рис. 1) представляет собой α-спираль, на конце которой находится последовательность NES (nuclear export sequence), отвечающая за ядерный экспорт (рис. 1). Далее следует субдомен B, контролирующий доступ к NЕS. Субдомен С формирует амфипатический β-слой, который снизу защищается субдоменом D.
Рис. 1.
Структурная организация белков семейства NAP и домена NAP. (a) Модель третичной структуры (контактная поверхность) дрожжевого NAP1. Субдомены A, B, C и D представлены в синем, желтом, красном и зеленом цветах соответственно. (б) Доменная организация членов семейства NAP: дрожжей – yNAP1, мыши – mNAP1L3 и человека – hSET и hTSPY. Фиолетовым цветом указано положение кислого С-концевого домена. Повторяющийся мотив у mNAP1L3 является частью субдомена B и обозначен штриховкой. Пунктирной линией обозначено отсутствие субдомена B. NES − последовательность ядерного экспорта, NLS – последовательность ядерной локализации.
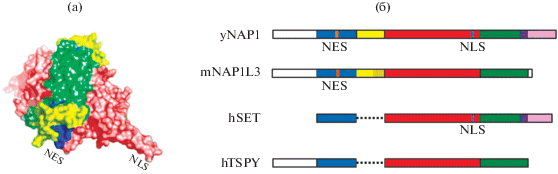
Отличительной чертой подсемейства NAP1 является кислый C-концевой домен (рис. 1б), необходимый для кратковременного вытеснения димеров H2A/H2B, что обеспечивает скольжение нуклеосом. Несмотря на варьирующий размер и непостоянное присутствие, С-концевой домен белков разных подсемейств имеет консервативный аминокислотый состав. Так у белков подсемейства SET кислый С-концевой домен есть; у белков других подсемейств NAP он иногда присутствует, но его длина варьируется, например, у NAP1L5 он короче, а у NAP1L3 и TSPY он практически отсутствует. Предполагают, что именно кислый С-концевой домен способствует контакту белков NAP с гистонами (Park, Luger, 2006; Liu et al., 2019).
Длина аминокислотной последовательности субдомена В является наиболее вариабельной частью у разных белков. Этот домен у yNAP1 Парк и Лагер (Park, Luger, 2006) обозначили как домен “доступности” к NES (accessory domain), или “дополнительный” домен, поскольку он иногда отсутствует у других представителей семейства NAP. Тем не менее, Парк и Лагер предположили, что субдомен B может обеспечить свободный доступ к NES и ядерный экспорт или, наоборот, может маскировать NES, предотвращая перенос белка NAP1 из ядра в цитоплазму. Полностью принять эту гипотезу затрудняет факт того, что у белков SET субдомен В отсутствует. В специфичном для мозга белке NAP1L3 длина субдомена B составляет примерно 181 аминокислотных остатков (рис. 1б). У NAP1L3 субдомен B содержит повторяющийся мотив неизвестной функции, первоначально идентифицированный в белке нейрофиламента Н (NF-H) (Shen et al., 2001). Эти различия в последовательностях, вероятно, определяют специфические функции белков семейства NAP.
У разных организмов члены белкового семейства NAP были идентифицированы по наличию домена NAP на основании сравнительного анализа гомологии последовательностей, но без учета выполняемых ими функций. Поэтому классификация “NAP”-белков остается запутанной. Для дрожжей характерен один член семейства − NAP1, высшие эукариоты, как правило, имеют несколько гомологов NAP1. Семейство NAP можно разбить на подсемейства: NAP1, транслокация SE (SET), NAP1-подобные белки (NAP1L), семенник-специфичные белки Y (TSPY) и специфичные для семенников Y-подобные белки (TSPYL) (von Lindern et al., 1992; Schnieders et al., 1996; Vogel et al., 1998; Attia et al., 2013).
SET – консервативные белки семейства NAP, включают белки дрозофилы и человека. Кристаллическая структура SET, состоящая из N-концевой спирали, основной спирали (backbone helix) и домена “earmuff”, который отвечает за формирование димера, формирующего структуру, напоминающую наушники, сходную со структурой димеров NAP1 (рис. 2). Белки NAP1 и дрожжевой белок Vps75 (гомолог SET, идентифицирован сначала как белок подсемейства NAP1) сходны с SET по доменной структуре и по способности специфически связываться с гистонами. В результате тщательного анализа было показано, что SET и NAP1-белки относятся к гистоновым шаперонам. Одни могут формировать комплексы с гистоновыми ацетилтрансферазами (Vps75), другие – с гистоновыми деацетилазами (Nap1). SET является компонентом белкового комплекса INHAT, участвующего в процессе ингибирования гистоновых ацетилтрансфераз (Eitoku et al., 2008; Moshkin et al., 2009; Das et al., 2010).
Рис. 2.
Структура “наушники” димеров гистоновых шаперонов: дрожжевого yNAP1 (слева) и hSET человека (справа).
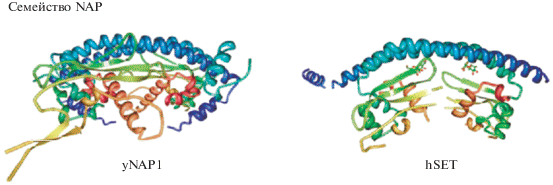
Среди функций SET − участие в функционировании гистонов, взаимодействие с ДНК-связывающими факторами и протеазами, а также регуляция транскрипции, обеспечение стабильности мРНК и участие в процессе апоптоза (Kawase et al., 1996; Brennan, Stetez, 2001; Fan et al., 2003; Gamble et al., 2005; Haruki et al., 2006). В основе многофункциональности белка SET лежит его значимость в сборке и разборке нуклеосом. Также согласно недавним исследованиям SET участвует в вытеснении линкерного гистона H1, способствуя декомпактизации хроматина, формированию преинициаторного комплекса в районе промотора гена и инициации транскрипции. Все эксперименты проводили in vitro, тем не менее, предполагают, что белки SET служат важными регуляторами активации генов и in vivo (Zhang et al., 2015).
TSPY был идентифицирован как член семейства NAP главным образом на основании гомологии аминокислотных последовательностей. Предполагается, что TSPY выполняет функцию, связанную с пролиферацией сперматогониев (Schnieders et al., 1996), но его конкретная роль в клетке не была охарактеризована. Последующая идентификация многих специфичных для семенников Y-подобных белков (TSPYL, TSPYL4) (Ozbun et al., 2001) и продуктов альтернативного сплайсинга (TSPY-S и TSPY-L) в других тканях говорит о том, что функции TSPY тканеспецифичны (Ozbun et al., 2001; Krick et al., 2003; Puffenberger et al., 2004). Результаты некоторых исследований показали, что TSPY активирует гены, контролирующие клеточный цикл и жизнеспособность клеток, и подавляет гены, контролирующие апоптоз (Kido, Lau, 2019).
Экспрессия белков NAP высокоспецифична для многих типов клеток и тканей (Kellogg et al., 1995; Hu et al., 1996; Zhou et al., 2015). Известно, что белки семейства NAP через ремоделирование структуры хроматина участвуют в разнообразных клеточных событиях, они регулируют экспрессию генов на эпигенетическом уровне, смещая гистоны, открывая тем самым доступ регуляторным факторам к местам связывания на ДНК (Kellogg et al., 1995; Compagnone et al., 2000; Chai et al., 2001; Canela et al., 2003; Oram et al., 2006; Okuwaki et al., 2010; Moshkin et al., 2013; Zhou et al., 2016).
Поскольку белки семейства NAP вовлечены во множество жизненно важных для организма процессов, рассмотрим подсемейства NAP1 и NAP1-подобных белков подробнее.
Подсемейство NAP1
Первоначально NAP1 был идентифицирован у Xenopus laevis как кислый белок, который участвует в сборке нуклеосом (Laskey et al., 1978). Поскольку NAP1 является одним из самых первых исследованных представителей семейства NAP, его структура взята за образец.
NAP1 является консервативным белком у всех эукариот, от дрожжей до человека (Ishimi et al., 1983; Ishimi, Kikuchi, 1991; Ito et al., 1996; Steer et al., 2003) и, кроме участия в процессах, отвечающих за сборку хроматина, выполняет множество других функций in vivo.
Дрожжевой NAP1 (возможно, наиболее охарактеризованный из всех гомологов NAP1) представляет собой полипептид с молекулярной массой 48 кДа, который связывает гистоны H2A-H2B, H3-H4 и линкерный гистон H1 и может обеспечивать сборку нуклеосом in vitro и in vivo (рис. 3) (Ishimi, Kikuchi, 1991; McBryant et al., 2003; Kepert et al., 2005; Aguilar-Gurrieri et al., 2016). Структурный и функциональный анализ центрального домена yNAP1 (с 74 по 365 аминокислотный остаток) показал, что именно эта область сохраняет нативную структуру и функционирует при сборке нуклеосом (Fujii-Nakata et al., 1992; McBryant et al., 2003).
Рис. 3.
Механизм ступенчатой сборки нуклеосом, с участием NAP1. Первый этап (I) сборки нуклеосомы заключается в связывании двух комплексов NAP12(H3-H4) с ДНК, вследствие чего, формируется частица тетрасомы и два свободных димера NAP1. Второй этап (II) заключается в том, что тетрасомная частица взаимодействует с комплексом NAP12(H2A-H2B), образуется гексасомная частица и димер NAP1. На третьем этапе (III) происходит увеличение гексасомы до нуклеосомы при помощи еще одного комплекса NAP12(H2A-H2B), в итоге получается полная нуклеосома и свободный димер NAP1. Четвертый, но не обязательный этап (IV) − это добавление в нуклеосому линкерного гистона H1.
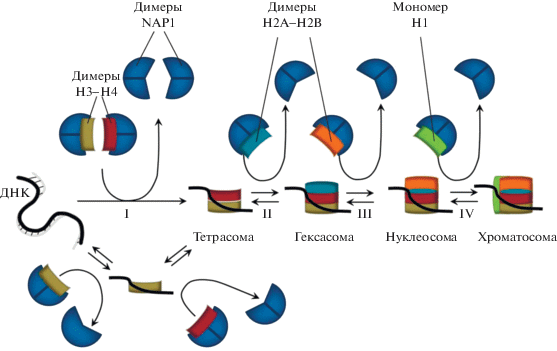
В клетках HeLa NAP1 взаимодействует с вновь синтезированными гистонами H2A и H2B, что указывает на его участие в сборке хроматина de novo (Chang et al., 1997).
Кроме того, в экспериментах in vitro было показано, что NAP1 обладает способностью удалять димеры H2A-H2B из нуклеосом и заменять их либо основными типами димеров, либо альтернативными димерами – H2A.X-H2B, H2A.Z-H2B или макро-H2A1.2-H2B (Park et al., 2005; Chen et al., 2016). Также в бесклеточной системе было показано, что NAP1 мыши стабилизирует гексасомы – функционально-активные интермедиаты нуклеосом, содержащие только один димер H2A-H2B. Особенно сильно это проявляется в случае убиквитинилирования гистона Н2В, которое существенно увеличивает мобильность димеров H2A-H2B даже в отсутствии ремоделирующих хроматин комплексов и положительно влияет на процесс элонгации транскрипции (Krajewski, 2020).
Таким образом, NAP1 участвует как в репликативной, так и в независимой от репликации сборке хроматина, обеспечивая скольжение нуклеосом вдоль ДНК, которое происходит в следствие динамического обмена гистоновых димеров (Park et al., 2005). В результате такого скольжения открывается или закрывается доступ к регуляторным элементам (промоторы, энхансеры, сайленсеры и т.п.), которые влияют на транскрипционный статус соседних генов (Kawase et al., 1996; Ito et al., 2000; Shikama et al., 2000). Так общегеномный анализ, проведенный на дрожжевом штамме с дефицитом экспрессии Nap1 у Saccharomyces cerevisiae, показал нарушение экспрессии 10% всех генов (Ohkuni et al., 2003). У мышей нокаутирование нейрон-специфического гена Nap1l2 (представитель семейства NAP1-подобных белков) приводило к эмбриональной летальности примерно на средней стадии беременности (Rodriguez et al., 1997).
NAP1 может не только связывать гистоны и участвовать в сборке нуклеосом, регулируя транскрипцию, он также задействован в регуляции клеточного цикла. Например, дрожжевой NAP1 специфически взаимодействует с регуляторами митоза: циклинами типа B (clb2), киназой Gin4 и NBP (NAP1 связывающий белок) (Kellogg, Murray, 1995; Altman, Kellogg, 1997; Shimizu et al., 2000; Mortensen et al., 2002). Ядерно-цитоплазматический транспорт канонических и вариантных гистонов в ответ на сигнал, полученный клеткой, является важным этапом в активации сборки хроматина и регуляции экспрессии генов (Park, Luger, 2006). Микроскопия флуоресцентно-меченого NAP1 показала, что в S-фазе клеточного цикла он находится в ядре, а во время фазы G2 он присутствует и в цитоплазме. Локализация NAP1 в цитоплазме у различных видов организмов предполагает его вовлеченность в механизм миграции гистонов между цитоплазмой и ядром (Marheineke, Krude, 1998). Это предположение подтверждается тем, что NAP1 взаимодействует с Kap114p – членом семейства белков кариоферинов (импортинов), ответственных за транспорт в ядро гистонов H2A и H2B (Miyaji-Yamaguchi et al., 2003).
Представляет интерес вовлеченность NAP1 в эпигенетические процессы, связанные с модификацией гистонов. Исследования, проведенные на D. melanogaster, показали, что NAP1 участвует в регуляции транскрипции целевых генов сигнального Notch-пути, обеспечивая: 1) связывание белкового комплекса RLAF с комплексом Su(H)/H, 2) эффективное деметилирование гистона H3K4 гистоновой деметилазой LID, 3) эффективное деацетилирование H3 гистоновой деацетилазой RPD3, осуществляя таким образом контроль плотности нуклеосом в репрессированных локусах – целевых генах сигнального Notch-пути. Участие шаперона NAP1 в деметилировании и деацетилировании гистона H3 является примером эпигенетической регуляции генов, контролирующих развитие, через активность гистон-модифицирующих ферментов (Moshkin et al., 2009, 2013).
Также NAP1 вовлечен в процессы клеточной пролиферации (Kellogg et al., 1995), рекомбинации ДНК (Gao et al., 2012; Machida et al., 2014; Zhou et al., 2016), расхождении хромосом в митозе (Higgins, Herbert, 2013; Tachiwana et al., 2013; Shintomi et al., 2015) и репарации ДНК (Lankenau et al., 2003; Liu et al., 2009; Moshkin et al., 2013). Кроме того, гомологи NAP1 играют важную роль в развитии различных организмов. Например, в развитии листа и корневых волосков у Arabidopsis (Galichet, Gruissem, 2006; Zhu et al., 2017; Barna et al., 2018), а также в процессе формирования эпителия в эмбриогенезе у C. elegans (Patel et al., 2008), в сперматогенезе, миогенезе и нейрональном развитии у Drosophila (Bogdan et al., 2004; Schröter et al., 2004; Kimura, 2013), в дифференцировке и функционировании нервной системы у мышей (Attia et al., 2007).
Подсемейство NAP1-подобных белков
Количество представителей подсемейства NAP1-подобных белков (NAP1-like proteins), которые выявляют по гомологии их аминокислотных последовательностей с NAP1, постоянно растет. Функция многих из них до сих пор остается неизвестной. Есть доказательства того, что, по крайней мере, некоторые из них играют важную роль в регуляции пролиферации нейрональных клеток и формировании нервной системы (Okuwaki et al., 2010; Attia et al., 2013).
Подсемейство белков NAP1L включает: NAP1L1, NAP1L2, NAP1L3, NAP1L4, NAP1L5 и NAP1L6 (табл. 1) (Attia et al., 2013). Было обнаружено, что белковые последовательности самых древних членов семейства NAP1L1 и NAP1L4 идентичны на 64%. NAP1L1 и NAP1L4 экспрессируются повсеместно в тканях человека, тогда как NAP1L2, NAP1L3 и NAP1L5 экспрессируются в мозге (Attia et al., 2013). Результаты недавних исследований показывают, что NAP1L1 и NAP1L4 играют основную роль в сборке и разборке базовых частиц нуклеосом, а также связываются с димерами H2A-H2B, регулируя тем самым механизм транскрипции (Okuwaki et al., 2010). Также они могут быть вовлечены в регуляцию судьбы клеток, контролируя экспрессию p53-зависимых генов, останавливающих клеточный цикл и активирующих апоптоз, посредством ацетилирования специфических сайтов p53 в ходе поддержания клеточного гомеостаза или в ответ на стресс (Tanaka et al., 2019).
Таблица 1.
Представители семейства NAP1-подобных белков у человека
| Название белка/альтернативное название | ID белка в базах данных | Известные функции | Ссылки на первое упоминание |
|---|---|---|---|
| Nucleosome assembly protein 1-like 1/NRP | NP_631946.1 P55209 | Регулирует транспорт гистоновых димеров H2A-H2B и вариантного H2A (H2A.X, H2A.Z и макро-H2A1.2)-H2B и участвует в ассоциации этих гетеродимеров с ДНК, задействован в процессах разборки нуклеосом и замены гистонов | Simon et al., 1994 |
| Nucleosome assembly protein 1-like 2/BPX | NP_068798.1 Q9ULW6 |
Контролирует экспрессию генов-мишеней, таких как регулятор клеточного цикла Cdkn1c, посредством воздействия на ацетилирование гистонов | Rougeulle, Avner, 1996 |
| Nucleosome assembly protein 1-like 3/HB20 | NP_004529.2 Q99457.2 |
Регулирует клеточный цикл, в частности арест в фазе G0 клеточного цикла | Watanabe et al., 1996 |
| Nucleosome assembly protein 1-like 4/NAP2 | NP_005960.1 Q9ULW6 |
Регулирует транспорт гистоновых димеров H2A-H2B и вариантного H2A (H2A.X, H2A.Z и макро-H2A1.2)-H2B и участвует в ассоциации этих гетеродимеров с ДНК, задействован в процессах разборки нуклеосом и замены гистонов | Prawitt et al., 1996 обнаружили ген
и указали на гомологию с NAP Hu et al., 1996 – впервые упоминается как NAP2 |
| Nucleosome assembly protein 1-like 5/DRLM | NP_715638.1 Q96NT1.1 |
Прекращение или экстремально низкая экспрессия вызывает онкогенез в тканях печени | Harada et al., 2002 |
| Nucleosome assembly protein 1-like 6, pseudogene/NAP1L6 | A6NFF2 | Предположительно продукт псевдогена | Luo et al., 2009 |
У человека и мыши семейство NAP1-подобных белков состоит как минимум из пяти членов. Два из них (NAP1L1 и NAP1L4) были охарактеризованы как повсеместно экспрессирующиеся гистоновые шапероны, функции которых, за редким исключением, похожи (Simon et al., 1994; Rodriguez et al., 1997, 2000; Okuwaki et al., 2010). NAP1L1 человека (аналогично мышиному) играет ключевую роль в регуляции эмбрионального нейрогенеза, способствует пролиферации нейрональных предшественников и ингибирует дифференцировку нейронов во время развития коры мозга (Qiao et al., 2018). Остальные три члена NAP1-подобных белков (NAP1L2, NAP1L3 и NAP1L5) являются нейрон-специфическими (Rougeulle, Avner, 1996; Watanabe et al., 1996; Shen et al., 2001; Smith et al., 2003). Биологическое значение нейрон-специфического белка NAP1L2 определили по дефектам нервной трубки, вызванной избыточной пролиферацией нейрональных стволовых клеток, и по снижении дифференцировки нейронов, возникающих во время эмбрионального развития у мышей-мутантов по гену Nap1l2. Другие члены подсемейства NAP1L также участвуют в раннем развитии нервной системы и важны для формирования мозга, поскольку демонстрируют различные специфичные для мозга паттерны экспрессии (Watanabe et al., 1996; Smith et al., 2003; Steer et al., 2003). Было показано, что NAP1L2 контролирует в нейрогенезе экспрессию регулятора клеточного цикла Cdkn1c (cyclin-dependent kinase inhibitor 1c), воздействуя на ацетилирование гистонов (Attia et al., 2007). Исследования в дрожжевой двугибридной системе, коиммунопреципитация и компьютерный анализ структурных последовательностей всех пяти NAP1-подобных белков показали, что они способны напрямую взаимодействовать друг с другом через свои высококонсервативные α-спирали. Коэкспрессия всех NAP1-подобных белков в нейронах и влияние делеции Nap1l2 на паттерны ядерно-цитоплазматического распределения белков NAP1L1 и NAP1L4 в клетке, позволяют предположить, что разнообразие взаимодействий между вышеупомянутыми белками необходимы для подготовки клеток к специфическим условиям нейрональной дифференцировки, таким как: межклеточное перераспределение определенных белков, модификация хроматина, регуляция транскрипции или рекрутирование на ДНК специфических факторов транскрипции (Attia et al., 2011).
NAP2 (NAP1L4)
При сравнении первичных последовательностей белков Nucleosome assembly protein-2 (NAP2 или NAP1L4) человека, мыши, дрожжей и дрозофилы было обнаружено несколько сходных областей. Участки, состоящие из кислых аминокислот, подобные тем, которые обнаруживаются в других белках NAP, могут облегчать контакт этих молекул с гистонами или другими основными белками (Stein et al., 1979; Jantzen et al., 1990; Sarkar et al., 2019). Кластеры отрицательно заряженных областей также обнаружены в гистон-связывающем белке N1 (histone-binding protein N1), нуклеоплазмине и негистоновом белке с высокой электрофоретической подвижностью (High-mobility-group protein-1) (Lapeyre et al., 1987; Schmidt-Zachmann et al., 1987; Wen et al., 1989). Делеционный анализ NAP1 дрожжей показал, что не все кислые домены необходимы для NAP-стимулированного переноса гистонов на ДНК (Fujii-Nakata et al., 1992). Спектр их функций в составе NAP значительно шире. В отличие от других белков NAP млекопитающих, которые имеют три кислых домена, NAP2 имеет только два таких домена. Как и NAP1, NAP2 переносит гистоны на “оголенную” (депротеинизированную) ДНК, хотя и с меньшей эффективностью. Делеция его С‑концевого богатого кислыми аминокислотами региона останавливает перенос гистонов, что говорит о важности этого домена для активности NAP2.
Размер мРНК гена Nap2 составляет 2.6–3.0 т.п.н. Она присутствует во всех проанализированных тканях человека, демонстрируя в три раза более высокий уровень в семенниках (Hu et al., 1996). Хотя позже было выяснено, что у свиньи NAP2 в наибольшей степени представлен в плаценте (Li et al., 2012).
Nap2 был впервые идентифицирован с помощью позиционного клонирования области 11p15.5, нарушение которой приводит к различным заболеваниям, включая опухоль Вильмса (Prawitt et al., 1996). Функциональный анализ рекомбинантного белка NAP2 человека показал, что он может взаимодействовать как с коровыми, так и с линкерными гистонами (Rodriguez et al., 1997). Последние исследования демонстрируют, что NAP2 регулирует пролиферацию клеток и апоптоз путем контроля экспрессии p53-зависимых генов (Tanaka et al., 2019).
Было показано, что NAP2 мигрирует между цитоплазмой и ядром. Его концентрация увеличивается при переходе клеток от пограничной фазы G0/G1 к фазе М (Rodriguez et al., 1997, 2000). В фазе G0/G1 он присутствует в цитоплазме, а затем начинает транслоцироваться в ядро, где и находится на протяжении всей S-фазы. Эти результаты аналогичны результатам, полученным для NAP1 дрозофилы (dNAP1). Отсюда сделали вывод, что белок присутствует в ядре во время S-фазы и является преимущественно цитоплазматическим во время фазы G2. Ито и соавторы (Ito et al., 1996) первыми предположили, что dNAP1 может функционировать в качестве шаперона для доставки гистонов из места их синтеза (цитоплазмы) в место использования (ядро), где они затем включаются в состав нуклеосом. NAP2 также способен связываться с коровыми и линкерными гистонами, транспортировать их, и его локализация зависит от фазы клеточного цикла, то есть этот шаперон функционирует аналогично dNAP1. Предполагаемый сигнал ядерной локализации высоко консервативен у NAP2, hNAP1L1 и mNAP1L1. Эта последовательность характерна ядерным белкам, но не было показано, что именно она определяет ядерную локализацию шаперона NAP2 (Dingwall, Laskey, 1991; Rodriguez et al., 1997).
Максимальная концентрация гистонов, связанных с NAP2, характерна для фазы G1/S, когда идет усиленный синтез гистонов для упаковки реплицированной ДНК (Rodriguez et al., 2000). Количество NAP2 в фазах G1/S и G2/M одинаково, но во время фазы митоза количество гистонов, связанных с NAP2, меньше, что свидетельствует о существовании NAP2, свободных от нехромосомной фракции гистонов, которые возможно участвуют в других процессах (Kellogg, Murray, 1995). Несколько работ решительно поддерживают идею фосфорилирования белка NAP2 во время его миграции из цитоплазмы в ядро. В этом отношении фосфорилирование может вызывать противоположные эффекты: либо это активация сигнала старта транспорта NAP2 в ядро (Jans, Hübner, 1996), либо маскировка сигнала ядерной локализации, которая предотвращает транспорт NAP2 в ядро (Jans, Hübner, 1996; Schwab, Dreyer, 1997). Кроме того, фосфорилирование NAP1 и NAP2 в экстрактах клеток HeLa устраняется гепарином, специфическим ингибитором протеиновой киназы CKII (Casein Kinase 2), функция которой заключается в фосфорилировании различных субстратов. Добавление коровых гистонов in vitro может стимулировать фосфорилирование dNAP1 дрозофилы и NAP2 человека киназой CKII, в результате чего эти шапероны остаются в ядре и не выходят в цитоплазму. Наконец, в экспериментах in vivo было показано, что NAP2 является фосфопротеином (Rodriguez et al., 2000).
NAP2 образует комплекс как с коровыми, так и с линкерными гистонами на протяжении всего клеточного цикла. Связь с линкерным гистоном H1 белок NAP2 осуществляет эффективнее, нежели NAP1 (Rodriguez et al., 2000). Селективное связывание NAP1 с гистонами H2A/H2B (Ishimi et al., 1987), а NAP2 с гистоном H1 предполагает участие этих шаперонов в дополнительной стадии образования нуклеосом (этап IV) (рис. 3). Дальнейшие исследования на культурах клеток HeLa показали, что NAP2 является частью мультибелковых комплексов, которые, возможно, определяют специфичность связывания различных белков NAP c их гистоновым пулом (Rodriguez et al., 2004).
Позже Окуваки и соавторы (Okuwaki et al., 2010) обнаружили, что hNAP1L1 и NAP2 способны связываться с гистонами H2A-H2B и различными димерами вариантного H2A с H2B в клеточных экстрактах, где нет репликации ДНК. Этот факт позволил предположить, что оба hNAP1L играют решающую роль в независимой от репликации ДНК сборке хроматина (Okuwaki et al., 2010). В этой же работе было показано, что активность hNAP1L1 в плане разборки нуклеосом значительно выше, активности NAP2, что предполагает разные роли шаперонов hNAP1L1 и NAP2 в регуляции динамики гистонов. Было обнаружено, что N- и C-концевые области hNAP1L1 больше необходимы для эффективного вытеснения димеров H2A/H2B из базовой частицы нуклеосомы, чем для переноса гистонов на ДНК. Более того, области N- и C-концов NAP2 у химерных белков, полученных в результате совмещения их с последовательностью hNAP1L1, функционировали независимо друг от друга при вытеснении димеров H2A/H2B из базовой частицы нуклеосомы. Из этого следует, что для вытеснения гистонов необходимо совместное участие N- и C-концов обоих hNAP1L. Авторы сделали вывод, что N- и C-концевые области определяют специфичность hNAP1L к гистонам (Okuwaki et al., 2010).
Таким образом, шаперон NAP2 помимо участия в образовании нуклеосом играет важную роль в связывании с линкерным гистоном H1. Кроме того, NAP2 и hNAP1L1 связывают разные вариантные гистоны H2A, образующие димеры с H2B, а особенности структуры этих белков, вероятно, определяют их специфическую роль в регуляции динамики гистонов в ходе различных клеточных процессов.
CG5017 (HANABI, MILKAH, TNAP)
Изучение гена CG5017 началось с получения мутации, которая вызывала усиление проявления аллеля ssa гена spineless у Drosophila melanogaster (Kuzin et al., 1991). Мутация была получена путем мутагенеза после скрещивания мух линии ssa40a с мухами дисгенной линии w oc/FM4. Индуцированный мутагенез вызвал инсерцию мобильного генетического Р-элемента в регуляторную область гена CG5017. Полученная линия была обозначена ssaSc. Мутантные особи линии ssaSc отличались от мух исходной линии ssa40a более выраженной трансформацией дистальных сегментов антенны в тарзус, слиянием тарсальных сегментов, уменьшением размера щетинок и увеличением количества зубцов полового гребешка (Sex comb – Sc) на передней ноге у самцов. Это была первая мутация гена CG5017 с видимым фенотипическим проявлением, которое выражалась в усилении проявления мутации ssa40a (Kuzin et al., 2010; Vorontsova et al., 2012).
Другими исследователями, в экспериментах по поиску мутаций, нарушающих память дрозофилы, были получены и две гипоморфные мутации гена CG5017 (линии milkah-1 и milkah-2). Оказалось, что они не приводят к морфологическим дефектам, но влияют на формирование долгосрочной памяти у дрозофил (Dubnau et al., 2003). Интересно, что эти две линии (и другие мутантные линии, полученные Дубнау и др.) были так названы в честь собак Павлова. Имена и фотографии 40 собак были обнаружены в 1992 году Тимом Талли (Tim Tally), одним из соавторов, во время его экскурсии по домашней резиденции Ивана Петровича Павлова в Санкт-Петербурге. Поиск генов, влияющих на долгосрочную и краткосрочную память дрозофилы, навело исследователя на мысль присвоить найденным мутантам имена подопытных собак Павлова.
Дубнау с соавторами продемонстрировал влияние гена CG5017 на память и обучение у мух (Dubnau et al., 2003). Данные этих исследователей позднее были подтверждены в независимых экспериментах с использованием гипоморфного аллеля CG5017 – ssaSc (Kuzin et al., 2014). У самцов дрозофил, несущих этот мутантный аллель, была нарушена долговременная память и обучение. Таким образом, было показано, что мутации гена CG5017 влияют не только на морфогенез, но и на тонкие физиологические процессы формирования памяти.
Ген CG5017 был картирован в районе 98B-5 хромосомы 3R. Последующие исследования показали, что открытая рамка считывания гена CG5017 дрозофилы кодирует белок длиной 283 аминокислот, большая часть которого (аминокислоты с 46 по 252) представляет собой домен NAP. Выравнивание белковой последовательности CG5017 и NAP1 выявило их сходство на 52%, в следствии чего, его отнесли к семейству NAP (Kuzin et al., 2010). На данный момент, ген имеет несколько названий: CG5017, milkah (Dubnau et al., 2003), Hanabi (Kimura, 2013) и tNAP – testis-specific NAP (Doyen et al., 2015).
Чтобы оценить физиологическую роль белка CG5017, была создана линия, несущая нулевую мутацию по этому гену (Kimura, 2013). Оказалось, что нуль-мутация CG5017 нарушает процесс элонгации сперматид. Японский исследователь Кимура (Kimura, 2013) присвоил гену CG5017 имя Hanabi, что в переводе значит “фейерверк”, поскольку фенотип сперматид в семеннике, в норме собранных в пачки на стадии их удлинения в ходе сперматогенеза, напоминал фейерверк из-за отсутствия их правильной кластеризации. Было показано, что мРНК CG5017/Hanabi присутствует в семенниках и теле самцов. Однако его мРНК не детектируется у дрозофил с удаленными семенниками, также CG5017 не экспрессируется у самок (Kimura, 2013). Взрослые самцы, гомозиготные по нулевому аллелю CG5017, были полностью стерильны и не имели другого фенотипа; с другой стороны, самки были фертильны. Таким образом, было показано, что белок CG5017 необходим для нормального протекания сперматогенеза у дрозофилы (Kimura, 2013) и является семенник-специфическим NAP (tNAP). Действительно, анализ данных, полученных методом RNA-seq, показал, что экспрессия CG5017 крайне тканеспецифична в отличие от экспрессии NAP1, который широко экспрессируется во всех тканях на всех стадиях развития. мРНК гена CG5017 в основном присутствует в семенниках и придаточных железах у самцов, однако она также детектируется в имагинальных дисках на третьей личиночной стадии и в жировом теле на стадии предкуколки (Brown et al., 2014). Кроме того, было показано, что CG5017/tNAP связывается с белком MST77F, который является обязательным компонентом хроматина сперматозоидов и необходим для мужской фертильности (Doyen et al., 2015).
К сожалению, белковый продукт гена CG5017 пока остается практически не изученным. Однако можно предположить, что белок CG5017 является важным эпигенетическим фактором, контролирующим работу гена ss во время развития (в делящихся клетках имагинальных дисков и в мозге), а также участвующим в организации цитоскелета сперматид в семенниках у D. melanogaster. Его гомология с NAP1 указывает на возможное участие в таких процессах, как сборка нуклеосом и регуляция генетической экспрессии через контроль эффективности механизма ремоделирования хроматина. Эта гипотеза была проверена при помощи гуманизированных дрозофил, экспрессирующих ген Арил-гидрокарбонового рецептора (AhR) человека.
AhR является лиганд-активируемым транскрипционным фактором (Busbee et al., 2013; Larigot et al., 2018). При воздействии определенными веществами (ксенобиотиками) на клетку, AhR активирует экспрессию своих целевых генов. Для изучения последствий активации AhR ксенобиотиками in vivo была разработана оригинальная модельная система на основе трансформированных дрозофил, экспрессирующих AhR в клетках семенников, гомологичных клеткам Сертоли человека. Однако во время экспериментов экспрессия некоторых тестируемых целевых генов AhR не повышалась. Поскольку AhR является гомологом белка дрозофилы Spineless, который в свою очередь взаимодействует с CG5017 (Kuzin et al., 1991), было выдвинуто предположение, что мутация гена CG5017 может повлиять на транскрипционный статус целевых генов AhR при внесении ее в геном гуманизированных дрозофил. И действительно, активации AhR на фоне сниженной экспрессии CG5017 привела к дерепрессии его генов-мишеней, участвующих в поддержании клеточного гомеостаза (Cyp6g1, Mgat1, GstT4, Csas и Nans) (Akishina et al., 2019).
Таким образом, независимые эксперименты Кимура (Kimura, 2013), Доен (Doyen et al., 2015) и Акишиной с соавторами (Akishina et al., 2019) показали участие нового белка семейства NAP – CG5017/Hanabi/tNAP – в сперматогенезе дрозофилы, где по данным Кимура (Kimura, 2013) CG5017, вероятно, взаимодействует со специфичным для семенников β-тубулином и актином, регулируя процессы полимеризации и деполимеризации белков цитоскелета, по данным Доен с соавторами (Doyen et al., 2015) формирует комплекс с семенник-специфическим белком MST77F и по данным Акишиной с соавторами (Akishina et al., 2019) может участвовать в эпигенетической репрессии ряда целевых генов AhR в клетках семенника, выполняющих трофическую функцию.
Известно, что другой член семейства NAP – TSPY является мультикопийным геном, продукт которого, также участвует в сперматогенезе у человека и крупного рогатого скота (Hamilton et al., 2012; Shen et al., 2013). В этом отношении белок дрозофилы CG5017 может стать полезной моделью для понимания функции семенник-специфических белков семейства NAP, включая TSPY, в сперматогенезе человека.
ЗАКЛЮЧЕНИЕ
С момента открытия гистоны впечатляют своей уникальной и необычной биохимией. Знания, полученные в ходе изучения этих белков, в течение последнего десятилетия превратили их из статичных белков упаковки ДНК, в динамические модуляторы практически всех процессов, протекающих с участием ДНК. С гистонами тесно связана сеть гистоновых шаперонов. Благодаря своему структурному и функциональному разнообразию, гистоновые шапероны облегчают сборку нуклеосом. Они участвуют во всех аспектах биологии гистонов, от фолдинга в цитоплазме до импорта в ядро и формирования нуклеосом.
Подводя итоги, необходимо сказать, что почти все рассмотренные нами белки-шапероны (NAP1, NAP1L1, NAP1L2, NAP1L3, NAP1L4/NAP2, NAP1L5 и CG5017/Hanabi), взаимодействуя с гистонами, участвуют в регуляции клеточного цикла и ремоделировании хроматина. Идентификация семейства родственных гистон-связывающих белков-шаперонов с одинаковыми аминокислотными последовательностями в ряду от дрожжей до человека, говорит о том, что сборка хроматина является эволюционно консервативным процессом. Будучи каноническими переносчиками гистонов, и обеспечивая эффективный ремоделлинг хроматина, белки семейства NAP участвуют в нейрональной дифференцировке, трофических процессах в сперматогенезе и формировании долговременной памяти, что указывает на важность этого семейства в онтогенезе.
Снижение нормальной экспрессии шаперонов семейcтва NAP или потеря их функциональности делает организм уязвимым к условиям внешней среды, нарушает фертильность, процесс запоминания и может стать причиной летального исхода. Таким образом, трудно переоценить важность гистоновых шаперонов для развития и нормальной жизнедеятельности организма, поэтому поиск и характеристика подобных белков важны не только для фундаментальной науки, но также будут иметь прикладное значение.
Однако ряд вопросов, касающихся механизма действия гистоновых шаперонов, остается пока без ответа. Является ли предложенная модель последовательной сборки нуклеосом реальной в условиях in vivo? Какие структурные особенности характерны для еще неописанных гистоновых шаперонов? Что способствует образованию отдельных специфических комплексов шаперонов с гистонами? Каково назначение регионов, богатых кислыми аминокислотами, присутствующих в последовательности большинства гистоновых шаперонов? Является ли функция шаперонов простой защитой гистонов от их неспецифических взаимодействий in vivo? Как регулируется активность гистоновых шаперонов? Одинаков ли механизм их взаимодействия с коровыми и линкерными гистонами?
Отсутствие полного списка комплексов, которые могут формировать разные шапероны с разными гистонами, трудности интерпретации результатов, полученных in vitro, к ситуации in vivo затрудняют понимание механизма функционирования гистоновых шаперонов. Несмотря на это, в процессе изучения термодинамики сборки и разборки нуклеосом предлагаются гипотетические модели действия гистоновых шаперонов. Эти модели объясняют, как они связываются с гистонами, обеспечивая их специфическое взаимодействие и как сохраняют баланс процессов сборки и разборки нуклеосом. Доказательство этих гипотетических моделей должно стать ключевым в будущих исследованиях механизма действия гистоновых шаперонов.
Список литературы
Aguilar-Gurrieri C., Larabi A., Vinayachandran V. et al. Structural evidence for Nap1-dependent H2A-H2B deposition and nucleosome assembly // EMBO J. 2016. V. 2035. № 13. P. 1465–1482.
Akishina A.A., Vorontsova J.E., Cherezov R.O. et al. NAP family CG5017 chaperone pleiotropically regulates human AHR target genes expression in Drosophila testis // Int. J. Mol. Sci. 2019. V. 20. № 1. P. 118.
Altman R., Kellogg D. Control of mitotic events by Nap1 and the Gin4 kinase // J. Cell Biol. 1997. V. 138. № 1. P. 119–130.
Attia M., Förster A., Rachez C. et al. Interaction between nucleosome assembly protein 1-like family members // J. Mol. Biol. 2011. V. 407. № 5. P. 647–660.
Attia M., Rachez C., Avner P. et al. Nucleosome assembly proteins and their interacting proteins in neuronal differentiation // Arch. Biochem. Biophys. 2013. V. 534. № 1. P. 20–26.
Attia M., Rachez C., De Pauw A. et al. Nap1l2 promotes histone acetylation activity during neuronal differentiation // Mol. Cell. Biol. 2007. V. 27. № 17. P. 6093–6102.
Barna B., Gemes K., Domoki M. et al. Arabidopsis NAP-related proteins (NRPs) contribute to the coordination of plant growth, developmental rate, and age-related pathogen resistance under short days // Plant Sci. 2018. V. 267. P. 124–134.
Bogdan S., Grewe O., Strunk M. et al. Sra-1 interacts with Kette and Wasp and is required for neuronal and bristle development in Drosophila // Development. 2004. V. 131. № 16. P. 3981–3989.
Brennan C.M., Steitz J.A. HuR and mRNA stability // Cell. Mol. Life Sci. 2001. V. 58. № 2. P. 266–277.
Brown J.B., Boley N., Eisman R. et al. Diversity and dynamics of the Drosophila transcriptome // Nature. 2014. V. 512. № 7515. P. 393–399.
Busbee P.B., Rouse M., Nagarkatti M. et al. Use of natural AhR ligands as potential therapeutic modalities against inflammatory disorders // Nutr. Rev. 2013. V. 71. № 6. P. 353–369.
Canela N., Rodriguez-Vilarrupla A., Estanyol J.M. et al. The SET protein regulates G2/M transition by modulating cyclin B-cyclin-dependent kinase 1 activity // J. Biol. Chem. 2003. V. 278. № 2. P. 1158–1164.
Chai Z., Sarcevic B., Mawson A. et al. SET-related cell division autoantigen-1 (CDA1) arrests cell growth // J. Biol. Chem. 2001. V. 276. № 36. P. 33665–33674.
Chang L., Loranger, S.S., Mizzen C. et al. Histones in transit: cytosolic histone complexes and diacetylation of H4 during nucleosome assembly in human cells // Biochemistry. 1997. V. 36. № 3. P. 469–480.
Chen X., D’Arcy S., Radebaugh C.A. et al. Histone chaperone Nap1 is a major regulator of histone H2A-H2B dynamics at the inducible GAL locus // Mol. Cell. Biol. 2016. V. 36. № 8. P. 1287–1296.
Compagnone N.A., Zhang P., Vigne J.L. et al. Novel role for the nuclear phosphoprotein set in transcriptional activation of p450c17 and initiation of neurosteroidogenesis // Mol. Endocrinol. 2000. V. 14. № 6. P. 875–888.
Das C., Tyler J.K., Churchill M.E. The histone shuffle: histone chaperones in an energetic dance // Trends Biochem. Sci. 2010. V. 35. № 9. P. 476–489.
Dingwall C., Laskey R.A. Nuclear targeting sequences – a consensus? // Trends Biochem. Sci. 1991. V. 16. № 12. P. 478–481.
Doyen C.M., Chalkley G.E., Voets O. et al. A testis-specific chaperone and the chromatin remodeler ISWI mediate repackaging of the paternal genome // Cell Rep. 2015. V. 13. № 7. P. 1310–1318.
Dubnau J., Chiang A.S., Grady L. et al. The staufen/pumilio pathway is involved in Drosophila long-term memory // Curr. Biol. 2003. V. 13. № 4. P. 286–296.
Eitoku M., Sato L., Senda T. et al. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly // Cell. Mol. Life Sci. 2008. V. 65. № 3. P. 414–444.
Elsässer S.J., D’Arcy S. Towards a mechanism for histone chaperones // Biochim. Biophys. Acta. 2012. V. 1819. № 3–4. P. 211–221.
Fan Z., Beresford P.J., Oh D.Y. et al. Tumor suppressor NM23–H1 is a granzyme A-activated DNase during CTL-mediated apoptosis, and the nucleosome assembly protein SET is its inhibitor // Cell. 2003. V. 112. № 5. P. 659–672.
Fujii-Nakata T., Ishimi Y., Okuda A. et al. Functional analysis of nucleosome assembly protein, NAP-1. The negatively charged COOH-terminal region is not necessary for the intrinsic assembly activity // J. Biol. Chem. 1992. V. 267. № 29. P. 20980–20986.
Galichet A., Gruissem W. Developmentally controlled farnesylation modulates AtNAP1;1 function in cell proliferation and cell expansion during Arabidopsis leaf development // Plant Physiol. 2006. V. 142. № 4. P. 1412–1426.
Gamble M.J., Erdjument-Bromage H., Tempst P. et al. The histone chaperone TAF-I/SET/INHAT is required for transcription in vitro of chromatin templates // Mol. Cell. Biol. 2005. V. 25. № 2. P. 797–807.
Gao J., Zhu Y., Zhou W. et al. NAP1 family histone chaperones are required for somatic homologous recombination in Arabidopsis // The Plant Cell. 2012. V. 24. № 4. P. 1437–1447.
Hamilton C.K., Verduzco-Gómez A.R., Favetta L.A. et al. Testis-specific protein Y-encoded copy number is correlated to its expression and the field fertility of Canadian Holstein bulls // Sex. Dev. 2012. V. 6. № 5. P. 231–239.
Hammond C.M., Strømme C.B., Huang H. et al. Histone chaperone networks shaping chromatin function // Nat. Rev. Mol. Cell Biol. 2017. V. 18. № 3. P. 141–158.
Harada H., Nagai H., Ezura Y. et al. Down-regulation of a novel gene, DRLM, in human liver malignancy from 4q22 that encodes a NAP-like protein // Gene. 2002. V. 296. № 1–2. P. 171–177.
Haruki H., Okuwaki M., Miyagishi M. et al. Involvement of template-activating factor I/SET in tran-scription of adenovirus early genes as a positive-acting factor // J. Virol. 2006. V. 80. № 2. P. 794–801.
Heshmati Y., Kharazi S., Türköz G. et al. The histone chaperone NAP1L3 is required for haematopoietic stem cell maintenance and differentiation // Sci. Rep. 2018. V. 8. № 1. P. 11202.
Higgins J.M.G., Herbert M. Nucleosome assembly proteins get SET to defeat the guardian of chromosome cohesion // PLoS Genet. 2013. V. 9. № 9. P. e1003829.
Hu R.J., Lee M.P., Johnson L.A. et al. A novel human homologue of yeast nucleosome assembly protein, 65 kb centromeric to the p57KIP2 gene, is biallelically expressed in fetal and adult tissues // Hum. Mol. Genet. 1996. V. 5. № 11. P. 1743–1748.
Ishimi Y., Kikuchi A. Identification and molecular cloning of yeast homolog of nucleosome assembly protein I which facilitates nucleosome assembly in vitro // J. Biol. Chem. 1991. V. 266. № 11. P. 7025–7029.
Ishimi Y., Kojima M., Yamada M. et al. Binding mode of nucleosome-assembly protein (AP-I) and histones // Eur. J. Biochem. 1987. V. 162. № 1. P. 19–24.
Ishimi Y., Yasuda H., Hirosumi J. et al. A protein which facilitates assembly of nucleosome-like structures in vitro in mammalian cells // J. Biochem. 1983. V. 94. № 3. P. 735–744.
Ito T., Bulger M., Kobayashi R. et al. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays // Mol. Cell. Biol. 1996. V. 16. № 6. P. 3112–3124.
Ito T., Ikehara T., Nakagawa T. et al. p300-Mediated acetylation facilitates the transfer of histone H2A-H2B dimers from nucleosomes to a histone chaperone // Genes Dev. 2000. V. 14. № 15. P. 1899–1907.
Jans D.A., Hübner S. Regulation of protein transport to the nucleus: central role of phosphorylation // Physiol. Rev. 1996. V. 76. № 3. P. 651–685.
Jantzen H.M., Admon A., Bell S.P. et al. Nucleolar transcription factor hUBF contains a DNA-binding motif with homology to HMG proteins // Nature. 1990. V. 344. № 6269. P. 830–836.
Kawase H., Okuwaki M., Miyaji M. et al. NAP-I is a functional homologue of TAF-I that is required for replication and transcription of the adenovirus genome in a chromatin-like structure // Genes Cells. 1996. V. 1. № 12. P. 1045–1056.
Kellogg D.R., Kikuchi A., Fujii-Nakata T. et al. Members of the NAP/SET family of proteins interact specifically with B-type cyclins // J. Cell Biol. 1995. V. 130. № 3. P. 661–673.
Kellogg D.R., Murray A.W. NAP1 acts with Clb1 to perform mitotic functions and to suppress polar bud growth in budding yeast // J. Cell Biol. 1995. V. 130. № 3. P. 675–685.
Kepert J.F., Mazurkiewicz J., Heuvelman G.L. et al. NAP1 modulates binding of linker histone H1 to chromatin and induces an extended chromatin fiber conformation // J. Biol. Chem. 2005. V. 280. № 40. P. 34063–34072.
Kido T., Lau Y.C. The Y-linked proto-oncogene TSPY contributes to poor prognosis of the male hepatocellular carcinoma patients by promoting the pro-oncogenic and suppressing the anti-oncogenic gene expression // Cell Biosci. 2019. V. 9. P. 22.
Kimura S. The Nap family proteins, CG5017/Hanabi and Nap1, are essential for Drosophila spermiogenesis // FEBS Lett. 2013. V. 587. № 7. P. 922–929.
Krajewski W.A. The intrinsic stability of H2B-ubiquitylated nucleosomes and their in vitro assembly/disassembly by histone chaperone NAP1 // Biochim. Biophys. Acta. Gen. Subj. 2020. V. 1864. № 3. P. 129497.
Krick R., Jakubiczka S., Arnemann J. Expression, alternative splicing and haplotype analysis of transcribed testis specific protein (TSPY) genes // Gene. 2003. V. 302. № 1–2. P. 11–19.
Kuzin B.A., Doszhanov K.T., Simonova O.B. et al. A new allele variant of ssa and its participation in regulating the proliferation of the stem elements of the leg and antenna imaginal disks in Drosophila melanogaster // Ontogenez. 1991. V. 22. № 2. P. 212–216.
Kuzin B.A., Modestova E.A., Vorontsova Y.E. et al. Interaction of the ss and CG5017 genes in the regulation of morphogenesis of limbs in Drosophila melanogaster // Russ. J. Dev. Biol. 2010. V. 41. № 5. P. 312–317.
Kuzin B.A., Nikitina E.A., Cherezov R.O. et al. Combination of hypomorphic mutations of the Drosophila homologues of aryl hydrocarbon receptor and nucleosome assembly protein family genes disrupts morphogenesis, memory and detoxification // PLoS One. 2014. V. 9. № 4. P. e94975.
Lankenau S., Barnickel T., Marhold J. et al. Knockout targeting of the Drosophila nap1 gene and examination of DNA repair tracts in the recombination products // Genetics. 2003. V. 163. № 2. P. 611–623.
Lapeyre B., Bourbon H., Amalric F. Nucleolin, the major nucleolar protein of growing eukaryotic cells: an unusual protein structure revealed by the nucleotide sequence // Proc. Natl. Acad. Sci. USA. 1987. V. 84. № 6. P. 1472–1476.
Larigot L., Juricek L., Dairou J. et al. AhR signaling pathways and regulatory functions // Biochim. Open. 2018. V. 7. P. 1–9.
Laskey R.A., Honda B.M., Mills A.D. et al. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA // Nature. 1978. V. 275. № 5679. P. 416–420.
Li S., Li J., Tian J. et al. Characterization, tissue expression, and imprinting analysis of the porcine CDKN1C and NAP1L4 genes // J. Biomed. Biotechnol. 2012. V. 2012. P. 946527.
von Lindern M., van Baal S., Wiegant J. et al. Can, a putative oncogene associated with myeloid leukemogenesis, may be activated by fusion of its 3' half to different genes: characterization of the set gene // Mol. Cell. Biol. 1992. V. 12. № 8. P. 3346–3355.
Liu Y., Xu L., Xie C. et al. Structural insights into ceNAP1 chaperoning activity toward ceH2A-H2B // Structure. 2019. V. 27. № 12. P. 1798–1810. e3.
Liu Z., Zhu Y., Gao J. et al. Molecular and reverse genetic characterization of NUCLEOSOME ASSEMBLY PROTEIN1 (NAP1) genes unravels their function in transcription and nucleotide excision repair in Arabidopsis thaliana // Plant J. 2009. V. 59. № 1. P. 27–38.
Luger K., Mäder A.W., Richmond R.K. et al. Crystal structure of the nucleosome core particle at 2.8 A resolution // Nature. 1997. V. 389. № 6648. P. 251–260.
Luo J., Emanuele M.J., Li D. et al. A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene // Cell. 2009. V. 137. № 5. P. 835–848.
Machida S., Takaku M., Ikura M. et al. Nap1 stimulates homologous recombination by RAD51 and RAD54 in higher-ordered chromatin containing histone H1 // Sci. Rep. 2014. V. 4. P. 4863.
Marheineke K., Krude T. Nucleosome assembly activity and intracellular localization of human CAF-1 changes during the cell division cycle // J. Biol. Chem. 1998. V. 273. № 24. P. 15279–15286.
Mattiroli F., D’Arcy S., Luger K. The right place at the right time: chaperoning core histone variants // EMBO Rep. 2015. V. 16. № 11. P. 1454–1466.
Mazurkiewicz J., Kepert J.F., Rippe K. On the mechanism of nucleosome assembly by histone chaperone NAP1 // J. Biol. Chem. 2006. V. 281. № 24. P. 16462–16472.
McBryant S.J., Park Y.J., Abernathy S.M. et al. Preferential binding of the histone (H3-H4)2 tetramer by NAP1 is mediated by the amino-terminal histone tails // J. Biol. Chem. 2003. V. 278. № 45. P. 44574–44583.
Miyaji-Yamaguchi M., Kato K., Nakano R. et al. Involvement of nucleocytoplasmic shuttling of yeast Nap1 in mitotic progression // Mol. Cell. Biol. 2003. V. 23. № 18. P. 6672–6684.
Mortensen E.M., McDonald H., Yates J. 3rd et al. Cell cycle-dependent assembly of a Gin4-septin complex // Mol. Biol. Cell. 2002. V. 13. № 6. P. 2091–2105.
Mosammaparast N., Ewart C.S., Pemberton L.F. A role for nucleosome assembly protein 1 in the nuclear transport of histones H2A and H2B // EMBO J. 2002. V. 21. № 23. P. 6527–6538.
Moshkin Y.M., Doyen C.M., Kan T.W. et al. Histone chaperone NAP1 mediates sister chromatid resolution by counteracting protein phosphatase 2A // PLoS Genet. 2013. V. 9. № 9. P. e1003719.
Moshkin Y.M., Kan T.W., Goodfellow H. et al. Histone chaperones ASF1 and NAP1 differentially modulate removal of active histone marks by LID-RPD3 complexes during NOTCH silencing // Mol. Cell. 2009. V. 35. № 6. P. 782–793.
Ohkuni K., Shirahige K., Kikuchi A. Genome-wide expression analysis of NAP1 in Saccharomyces cerevisiae // Biochem. Biophys. Res. Commun. 2003. V. 306. № 1. P. 5–9.
Okuwaki M., Kato K., Nagata K. Functional characterization of human nucleosome assembly protein 1-like proteins as histone chaperones // Genes Cells. 2010. V. 15. № 1. P. 13–27.
Oram S.W., Liu X.X., Lee T.L. et al. TSPY potentiates cell proliferation and tumorigenesis by promoting cell cycle progression in HeLa and NIH3T3 cells // BMC Cancer. 2006. V. 6. P. 154.
Ozbun L.L., You L., Kiang S. et al. Identification of differentially expressed nucleolar TGF-β1 target (DENTT) in human lung cancer cells that is a new member of the TSPY/SET/NAP-1 superfamily // Genomics. 2001. V. 73. № 2. P. 179–193.
Park Y.J., Chodaparambil J.V., Bao Y. et al. Nucleosome assembly protein 1 exchanges histone H2A-H2B dimers and assists nucleosome sliding // J. Biol. Chem. 2005. V. 280. № 3. P. 1817–1825.
Park Y.J., Luger K. The structure of nucleosome assembly protein 1 // Proc. Natl. Acad. Sci. U. S. A. 2006. V. 103. № 5. P. 1248–1253.
Patel F.B., Bernadskaya Y.Y., Chen E. et al. The WAVE/SCAR complex promotes polarized cell movements and actin enrichment in epithelia during C. elegans embryogenesis // Dev. Biol. 2008. V. 324. № 2. P. 297–309.
Prawitt D., Munroe D.J., Pelletier J. et al. Loss of heterozygosity at Identification of a NAP related gene in the Wilms’ tumor candidate 11p15 in malignant glioma // Am. J. Hum. Genet. 1996. V. 59. P. A79.
Puffenberger E.G., Hu-Lince D., Parod J.M. et al. Mapping of sudden infant death with dysgenesis of the testes syndrome (SIDDT) by a SNP genome scan and identification of TSPYL loss of function // Proc. Natl. Acad. Sci. U.S.A. 2004. V. 101. № 32. P. 11689–11694.
Qiao H., Li Y., Feng C. et al. Nap1l1 controls embryonic neural progenitor cell proliferation and differentiation in the developing brain // Cell Rep. 2018. V. 22. № 9. P. 2279–2293.
Rodriguez P., Munroe D., Prawitt D. et al. Functional characterization of human nucleosome assembly protein-2 (NAP1L4) suggests a role as a histone chaperone // Genomics. 1997. V. 44. № 3. P. 253–265.
Rodriguez P., Pelletier J., Price G.B. et al. NAP-2: histone chaperone function and phosphorylation state through the cell cycle // J. Mol. Biol. 2000. V. 298. № 2. P. 225–238.
Rodriguez P., Ruiz M.T., Price G.B. et al. NAP-2 is part of multi-protein complexes in HeLa cells // J. Cell. Biochem. 2004. V. 93. № 2. P. 398–408.
Rougeulle C., Avner P. Cloning and characterization of a murine brain specific gene Bpx and its human homologue lying within the Xic candidate region // Hum. Mol. Genet. 1996. V. 5. № 1. P. 41–49.
Sarkar P., Zhang N., Bhattacharyya S. et al. Characterization of Caenorhabditis elegans nucleosome assembly protein 1 uncovers the role of acidic tails in histone binding // Biochemistry. 2019. V. 58. № 2. P. 108–113.
Schmidt-Zachmann M.S., Hügle-Dörr B., Franke W.W. A constitutive nucleolar protein identified as a member of the nucleoplasmin family // EMBO J. 1987. V. 6. № 7. P. 1881–1890.
Schnieders F., Dörk T., Arnemann J. et al. Testis-specific protein, Y-encoded (TSPY) expression in testicular tissues // Hum. Mol. Genet. 1996. V. 5. № 11. P. 1801–1807.
Schröter R.H., Lier S., Holz A. et al. Kette and blown fuse interact genetically during the second fusion step of myogenesis in Drosophila // Development. 2004. V. 131. № 18. P. 4501–4509.
Schwab M.S., Dreyer C. Protein phosphorylation sites regulate the function of the bipartite NLS of nucleolin // Eur. J. Cell. Biol. 1997. V. 73. № 4. P. 287–297.
Shen H.H., Huang A.M., Hoheisel J. et al. Identification and characterization of a SET/NAP protein encoded by a brain-specific gene, MB20 // Genomics. 2001. V. 71. № 1. P. 21–33.
Shen Y., Yan Y., Liu Y. et al. A significant effect of the TSPY1 copy number on spermatogenesis efficiency and the phenotypic expression of the gr/gr deletion // Hum. Mol. Genet. 2013. V. 22. № 8. P. 1679–1695.
Shikama N., Chan H.M., Krstic-Demonacos M. et al. Functional interaction between nucleosome assembly proteins and p300/CREB-binding protein family coactivators // Mol. Cell. Biol. 2000. V. 20. № 23. P. 8933–8943.
Shimizu Y., Akashi T., Okuda A. et al. NBP1 (Nap1 binding protein 1), an essential gene for G2/M transition of Saccharomyces cerevisiae, encodes a protein of distinct sub-nuclear localization // Gene. 2000. V. 246. № 1–2. P. 395–404.
Shintomi K., Takahashi T.S., Hirano T. Reconstitution of mitotic chromatids with a minimum set of purified factors // Nat. Cell Biol. 2015. V. 17. № 8. P. 1014–1023.
Simon H.U., Mills G.B., Kozlowski M. et al. Molecular characterization of hNRP, a cDNA encoding a human nucleosome-assembly-protein-I-related gene product involved in the induction of cell proliferation // Biochem. J. 1994. V. 297. P. 389–397.
Smith R.J., Dean W., Konfortova G. et al. Identification of novel imprinted genes in a genome-wide screen for maternal methylation // Genome Res. 2003. V. 13. № 4. P. 558–569.
Steer W.M., Abu-Daya A., Brickwood S.J. et al. Xenopus nucleosome assembly protein becomes tissue-restricted during development and can alter the expression of specific genes // Mech. Dev. 2003. V. 120. № 9. P. 1045–1057.
Stein A., Whitlock J.P., Bina M. Acidic polypeptides can assemble both histones and chromatin in vitro at physiological ionic strength // Proc. Nat. Acad. Sci. U. S. A. 1979. V. 76. № 10. P. 5000–5004.
Tachiwana H., Miya Y., Shono N. et al. Nap1 regulates proper CENP-B binding to nucleosomes // Nucleic Acids Res. 2013. V. 41. № 5. P. 2869–2880.
Tanaka T., Hozumi Y., Martelli A.M. et al. Nucleosome assembly proteins NAP1L1 and NAP1L4 modulate p53 acetylation to regulate cell fate // Biochim. Biophys. Acta. Mol. Cell Res. 2019. V. 1866. № 5. 12. P. 118560.
Valieva M.E., Feofanov A.V., Studitsky V.M. Histone chaperones: Variety and functions // Moscow Univ. Biol. Sci. Bull. 2016. V. 71. P. 165–169.
Vogel T., Dittrich O., Mehraein Y. et al. Murine and human TSPYL genes: novel members of the TSPY-SET-NAP1L1 family // Cytogenet. Cell Genet. 1998. V. 81. № 3–4. P. 265–270.
Vorontsova Y.E., Cherezov R.O., Zatsepina O.G. et al. Gene expression modulation is an evolutionary resource of adaptive alterations in the morphogenesis of insect limbs // Biol. Bull. Russ. Acad. Sci. 2012. V. 39. P. 186–193.
Watanabe T.K., Fujiwara T., Nakamura Y. et al. Cloning, expression pattern and mapping to Xq of NAP1L3, a gene encoding a peptide homologous to human and yeast nucleosome assembly proteins // Cytogenet. Cell Genetics. 1996. V. 74. № 4. P. 281–285.
Wen L., Huang J.K., Johnson B.H. et al. A human placental cDNA clone that encodes nonhistone chromosomal protein HMG-1 // Nucleic Acids Res. 1989. V. 17. № 3. P. 1197–1214.
Zhang Q., Giebler H.A., Isaacson M.K. et al. Eviction of linker histone H1 by NAP-family histone chaperones enhances activated transcription // Epigenetics Chromatin. 2015. V. 8. P. 30.
Zhou W., Gao J., Ma J. et al. Distinct roles of the histone chaperones NAP1 and NRP and the chromatin-remodeling factor INO80 in somatic homologous recombination in Arabidopsis thaliana // Plant J. 2016. V. 88. № 3. P. 397–410.
Zhou W., Zhu Y., Dong A. et al. Histone H2A/H2B chaperones: from molecules to chromatin-based functions in plant growth and development // Plant J. 2015. V. 83. № 1. P. 78–95.
Zhu Y., Rong L., Luo Q. et al. The histone chaperone NRP1 interacts with WEREWOLF to activate GLABRA2 in Arabidopsis root hair development // Plant Cell. 2017. V. 29. № 2. P. 260–276.
Дополнительные материалы отсутствуют.
Инструменты
Онтогенез


