Онтогенез, 2021, T. 52, № 3, стр. 170-194
Геномные дупликации как основа эволюционного успеха позвоночных
А. В. Байрамов a, *, Г. В. Ермакова a, А. В. Кучерявый b, А. Г. Зарайский a
a Федеральное государственное учреждение науки Институт биоорганической химии
им. акад. М.М. Шемякина и Ю.А. Овчинникова РАН
117997 Москва, ул. Миклухо-Маклая, 16/10, Россия
b Федеральное государственное учреждение науки Институт проблем экологии и эволюции им. А.М. Северцова РАН
119071 Москва, Ленинский проспект, 33, Россия
* E-mail: andrbayr@gmail.com
Поступила в редакцию 08.12.2020
После доработки 08.12.2020
Принята к публикации 15.01.2021
Аннотация
Позвоночные, часто рассматриваемые как наиболее сложно организованные представители животного мира, обладают целым рядом уникальных морфологических особенностей, обеспечивших их эволюционную устойчивость и успех. Согласно наиболее распространенной точке зрения, генетической основой этих инноваций были полногеномные дупликации, произошедшие на ранних этапах эволюции позвоночных. Одним из результатов таких дупликаций стало возникновение дополнительных копий регуляторных генов. Будучи выведенными из-под сдерживающего давления естественного отбора, эти копии получили возможность относительно свободно модифицировать свою структуру и функции, оказывая влияние на индивидуальное развитие и строение организмов. Следствием таких древних геномных дупликаций является, например, то, что около 35% генов человека представлены, как минимум, двумя гомологичными копиями. Идея о дупликациях генома на ранних этапах эволюции позвоночных была впервые выдвинута в 1970 г., однако, вопросы о количестве раундов прошедших дупликаций, их масштабе (полногеномные или локальные) и о том, на каком этапе эволюции они происходили, активно обсуждаются до сих пор. В последние годы (2018–2020 гг.) благодаря бурному развитию методов обработки больших объемов данных, получаемых в результате высокопроизводительного секвенирования геномов представителей разных групп позвоночных и их ближайших родственников, бесчерепных и оболочников, был проведен целый ряд широкоформатных сравнительных исследований, направленных на выявление групп синтении в геномах разных эволюционных линий и реконструкцию предковых хромосом. В результате были предложены несколько моделей, описывающих возможные базовые сценарии геномных дупликаций в ранней эволюции позвоночных. Параллельно активно проводятся лабораторные исследования, ориентированные на детальное изучение особенностей экспрессии и функциональных свойств отдельных семейств регуляторных генов у разных групп позвоночных. И здесь открылось много новой информации о представителях эволюционно древних животных, которые еще совсем недавно не входили в число традиционных лабораторных объектов (круглоротые, хрящевые рыбы, хрящевые ганоиды). В нашей обзорной статье мы делаем попытку рассмотреть и осмыслить современные представления о механизмах и эволюционных последствиях геномных дупликаций, а также соотнести свежие данные, полученные в ходе лабораторных исследований, с предлагаемыми на сегодняшний день моделями формирования генотипа позвоночных.
ВВЕДЕНИЕ
Разнообразие жизненных форм и высокий уровень приспособленности живых организмов к особенностям среды обитания являются результатом эволюционных преобразований их геномов и механизмов индивидуального развития. В основе адаптации к внешним условиям и освоения новых экологических ниш лежит приобретение организмами новых признаков. Вопросы появления этих признаков, их закрепления и последующих преобразований являются одними из центральных в эволюционной биологии (Moriyama, Koshiba-Takeushi, 2018). Геномные дупликации могут выступать в качестве фундамента, обеспечивающего появление нового генетического материала, своего рода поставщиком “генетического сырья”, при переработке которого, в ходе последующих эволюционных изменений под воздействием естественного отбора, происходит развитие и усложнение геномов организмов как на структурном, так и на регуляторном уровнях.
Предположение о том, что полногеномные дупликации (ПГД) могут являться двигателем эволюционного прогресса, впервые было высказано в 1970 г. американским исследователем японского происхождения Сусуму Оно (Susumo Ohno) в ставшей впоследствии классической монографии “Эволюция путем дупликации генов” (Evolution by Genes Duplications, Ohno, 1970).
Полногеномные дупликации, как отражено уже в самом их названии, в отличие от локальных удвоений отдельных генов или более протяженных фрагментов генома, приводят к полиплоидии – появлению в организме двойного набора всех хромосом. Такие дупликации могут происходить вследствие нарушений редукции в мейозе или за счет митотических удвоений в родительских половых клетках или на ранних этапах развития зародыша (Van de Peer, 2009, 2017; Moriyama, Koshiba-Takeushi, 2018). С точки зрения эволюционных перспектив удвоившихся генов (подробно спектр возможных сценариев мы рассмотрим ниже), важным результатом ПГД является то, что они приводят к удвоению не только белок-кодирующих последовательностей (экзома организма), но и к дупликации некодирующих участков генома, содержащих регуляторные последовательности.
После ПГД полиплоидия, как правило, элиминируется, за счет того, что рано или поздно парные хромосомы накапливают критическую массу изменений, не позволяющую им оставаться квадривалентными в ходе мейоза (MacKintosh, Ferrier, 2018). В итоге, когда восстанавливается исходная парность всех имеющихся хромосом, их носитель вновь становится диплоидным, то есть происходит его ре-диплоидизация. И, хотя организмы, прошедшие ре-диплоидизацию, больше не являются полиплоидными, они, как правило, сохраняют признаки своего полиплоидного прошлого, одним из которых являются дуплицированные копии отдельных генов.
ПОЛНОГЕНОМНЫЕ ДУПЛИКАЦИИ В ЖИВОЙ ПРИРОДЕ – РЕДКО, НО МЕТКО
Полногеномных дупликаций, последствия которых закрепились и сохранились на эволюционном пути, произошло, по оценкам исследователей, не так много, и чаще они происходили у растений, чем у животных (MacKintosh, Ferrier, 2018). В качестве возможной причины этой разницы рассматривается нарушение механизма формирования половой принадлежности у двуполых животных в случае их полиплоидизации (Mable, 2004; Orr, 1990). Также отмечается, что оплодотворение одной яйцеклетки двумя сперматозоидами или мейотические нарушения имеют, как правило, летальные последствия у млекопитающих и птиц (Sahoo et al., 2017; Forstmeier et al., 2010). Подробнее современные взгляды на проблему геномных дупликаций у позвоночных мы рассмотрим ниже, здесь лишь отметим, что большинство исследователей сходится во мнении, что на ранних этапах эволюции позвоночных (около 500–600 млн лет назад) произошли одна или две ПГД, а позже дополнительные дупликации происходили у рыб: у осетровых, у общего предка костистых рыб, а в последующем и у отдельных представителей этой группы (Meyer et al., 2005; Dehal, Boore, 2005; Du et al., 2020).
Среди представителей беспозвоночных на сегодняшний день две геномные дупликации описаны у паукообразных и одна у коловраток (Flot et al., 2013; Nossa et al., 2014; Kenny et al., 2016; Schwager, 2017).
Для одноклеточных эукариот отмечается, что современные диплоидные пивные дрожжи Saccharomyces cerevisiae, а также представители пяти близких к ним родов произошли после дупликации генома их общего предка около 100 млн лет назад (Scannell, 2007).
У цветковых растений обнаружены следы четырех ПГД, произошедших ориентировочно 150–200 млн лет назад (Soltis et al., 2008, 2009).
У грибов есть свидетельства только одной ПГД (Scannell et al., 2007).
Очевидно, что сведения о дупликациях будут дополняться, но и имеющиеся данные показывают, что ПГД происходили в разные исторические периоды в самых разных группах живых организмов. С учетом рассматриваемых временных масштабов приходится признать, что успешные ПГД на эволюционном пути случались достаточно редко – это единичные события на горизонтах в десятки и сотни миллионов лет.
Бросается в глаза, что оставившие свой след ПГД происходили в таких эволюционно успешных и экологически разнообразных линиях как позвоночные животные и цветковые растения (Jaillon et al., 2004; Meyer et al., 2005; Dehal, Boore, 2005; Tang et al., 2008; Putnam et al., 2008; Moriyama, Koshiba-Takeushi, 2018). Возможно, что бурное эволюционное развитие и возможность освоения широкого спектра экологических ниш представителями этих групп во многом были обеспечены именно дупликациями геномов, произошедшими у их предков.
Макинтош и Ферье делают попытку выявления и описания общих черт успешных ПГД (MacKintosh, Ferrier, 2018).
Авторы отмечают, что большинство, если не все, хорошо описанные ПГД были аллоплоидными событиями (объединением в одном организме геномов разных видов), однако ПГД у костистых рыб и последующая дополнительная дупликация у лососевых описаны как автоплоидные (удвоение собственного генома вида) (Martin, Holland, 2014; Christensen, Davidson, 2017; MacKintosh, Ferrier, 2018). Возможно, имеющиеся существенные различия в родительских хромосомах при аллоплоидизации способствуют скорейшему восстановлению диплоидного статуса потомков – ре-диплоидизации. При автоплоидизации возврат к диплоидности имеет более растянутый во времени, градиентный характер (Otto, 2007).
Ван де Пеером отмечено, что между дупликацией и последующим всплеском видового разнообразия часто присутствует временной лаг и эти события, как правило, сопряжены с существенными изменениями условий окружающей среды и климата. Это позволяет предположить, что полиплоидные организмы, как растения, так и животные могут быть более устойчивы в стрессовые периоды (рис. 1, Van de Peer et al., 2017). Некоторые ПГД у покрытосеменных датированы границей мелового периода и палеогена (так называемое, мел-палеогеновое вымирание около 66 млн лет назад), а дупликация у лососевых, произошедшая 88 млн лет назад, впоследствии привела к развитию способности этих рыб мигрировать между пресными и морскими водами после похолодания на границе эоцена и олигоцена (Vanneste et al., 2014; Macqueen, Johnston, 2014).
Рис. 1.
Полногеномные дупликации могут влиять на устойчивость и жизнеспособность видов в периоды стрессовых изменений окружающих условий (по Van de Peer et al., 2017).

Оценить точное число генов, кодирующих белки даже при наличии качественно отсеквенированного генома сложно. Это связано с трудностями распознавания генов, кодирующих короткие пептидные последовательности, выявления нефункциональных псевдогенов и сборки участков хромосом, содержащих повторы и дупликации (Holland et al., 2016). Даже у эволюционно близких видов количество предполагаемых генов может различаться: согласно данным NCBI, актуальный список белок-кодирующих генов у человека Homo sapiens насчитывает 19 116 генов, у мыши Mus musculus – 23 051 ген, а в целом у позвоночных число генов оценивается в диапазоне от 16 до 26 тысяч (Holland et al., 2016; Piovestan et al., 2019; http://www.informatics.jax.org/mgihome/homepages/stats/all_stats.shtml). При рассмотрении более далеких групп разброс еще больше возрастает: у оболочника Ciona intestinalis – 13 648 генов; у дрозофилы Drosophila melanogaster – 13 919; у нематоды Сaenorhabditis elegans – 20 269.
Наблюдаемое у организма количество генов представляет собой точку динамического равновесия между непрерывными процессами их появления и исчезновения. К сегодняшнему дню описаны возможности появления новых генов вследствие полногеномных, хромосомных, тандемных, локальных дупликаций, ретропозиций и комбинации копирования экзонов с последующим de novo включением некодирующих последовательностей в сочетании с мобильными элементами (Holland et al., 2016). Исследования показывают, что менее 1% генов человека произошли в результате ретротранспозиции (Pan, Zhang, 2009; Ciomborowska et al., 2013).
ПГД, как правило, сопровождаются последующей масштабной утратой генов, по отдельным оценкам достигающей 85% дуплицированных копий (Brunet et al., 2006). В то же время сама по себе “столь удручающая” статистика “выживаемости” генов после ПГД не свидетельствует о незначительности вклада этих дупликаций в судьбу организмов. Детальное сопоставление показывает, что в среднем у позвоночных, прошедших через ПГД, обнаруживается большее число генов, чем у их ближайших беспозвоночных родственников, и это обогащение функционально не равномерно. Оказывается, что по сравнению с ланцетником Branchiostoma floridae, геном позвоночных обогащен генами транскрипционных факторов и генами, вовлеченными в развитие нервной системы (Putnam et al., 2008). Таким образом, даже если дупликации и последующая редукция не приводят к кратному возрастанию общего числа генов, эти события могут обогащать отдельные подмножества генов участвующих в развитии определенных структур и особенностей организмов (Brunet et al., 2006).
Предполагается, что основной вклад в увеличение разброса между группами по числу генов вносят локальные дупликации. Описан целый ряд случаев увеличения числа генов у отдельных животных, связанный с особенностями их структуры, физиологии и поведенческими паттернами. Например, у пчел Apis mellifera увеличено количество генов обонятельных рецепторов (Robertson, Wanner, 2006), у стрекоз – генов опсина, связанных с остротой зрения (Futahashi et al., 2015), у устриц Crassostrea gigas – генов, кодирующих белки теплового шока и экспрессирующихся во время стресса при отливах (Zhang et al., 2012). Среди головоногих моллюсков у осьминога Octopus bimaculoides обнаружена экспансия генов, кодирующих протокадгерины и генов, кодирующих транскрипционные факторы C2H2, участвующие в нейральном развитии (Albertin et al., 2015).
ГЕНЫ НА РАСПУТЬЕ – ЕСТЬ ЛИ ЖИЗНЬ ПОСЛЕ ДУПЛИКАЦИЙ?
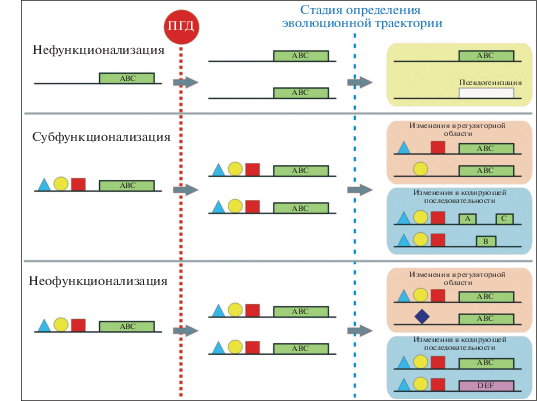
Каждое генетическое изменение, на своем пути от появления к эволюционному сохранению проходит три основных этапа: 1) происхождение (появление мутации), 2) фаза фиксации, когда изменение распространяется в популяции, и 3) фаза сохранения, когда фиксированное изменение сохраняется и поддерживается в популяции. Особенностью дупликаций в рамках этой траектории является возможность возникновения генетических различий между двумя появившимися копиями, что может привести к сохранению их обеих. И, судя по всему, как раз эта фаза установления различий между копиями является определяющей для их дальнейшей эволюционной траектории или, как говорят, “судьбоносной” (от английского “fate-determining stage”) (рис. 2, Innan, Kondrashov, 2010).
Копии генов, появившиеся в результате ПГД, были названы онологами, в честь японского исследователя Сусумо Оно.
В своей классической модели, положившей начало теоретическому обсуждению проблемы дупликации генов, С. Оно предположил, что наиболее вероятный постдупликационный сценарий – потеря функциональности одной из дочерних копий за ненадобностью, или нефункционализация (Ohno, 1970). Сразу после ПГД дочерние аллели предкового гена идентичны, а значит функционально избыточны, поскольку для поддержания функции материнского гена достаточно и одной копии. В силу этого, давление естественного отбора на поддержание обеих копий оказывается низким (Nei, 1969; Ohno, 1970). Исходя из предположения, что вредные мутации случаются гораздо чаще полезных (ломать – не строить!), они неизбежно будут накапливаться в кодирующей области одного из дубликатов и рано или поздно приведут к потере ее функциональности. В результате функциональный в прошлом ген становится псевдогеном, поэтому этот процесс также называют псевдогенизацией (рис. 3, Moriyama, Koshiba-Takeushi, 2018). Ряд экспериментальных исследований действительно подтвердил, что нефункционализация является наиболее распространенным сценарием эволюции дуплицированных генов (Kimura, King, 1979; Li, 1980; Stoltzfus, 1999; Jaillon et al., 2004; Woods et al., 2005; Brunet et al., 2006). Исследования семейств генов после геномной дупликации костистых рыб (ПГДКР – полногеномная дупликация костистых рыб, “калька” с принятой англоязычной аббревиатуры “TS-WGD” – Teleostei-specific Whole Genome Duplication) свидетельствуют о сохранении у рыб Danio rerio около 20% дуплицированных генов, что рассматривается в качестве максимальной (!) оценки степени сохранности дуплицированных генов после этой полногеномной дупликации (Postlethwait et al., 2000, 2004; Woods et al., 2005).
Рис. 3.
Варианты эволюционных траекторий дуплицированных генов (по Moriyama, Koshiba-Takeushi, 2018).
В то же время, С. Оно предположил, что в редких случаях накопленные дополнительные замены могут быть полезными и приводить к обретению новой функции гена, которая будет поддерживаться отбором.
В предложенной модели, однако, не вполне ясно, каким образом естественный отбор может различать новую и исходную копии генов, а ведь для них предполагается разная скорость изменений: быстрое накопление мутаций в свободном от давления отбора “дубликате” и стабильность в “оригинале”, продолжающем выполнять материнскую функцию под давлением отбора (Innan, Kondrashov, 2010).
Для разрешения этого противоречия, в русле развития идей С. Оно, Форс c коллегами предложили модель, получившую название дупликация-вырождение-дополнение (DDC от Duplication–Degeneration–Complementation) (Force et al., 1999). Поскольку, как правило, гены выполняют не одну, а несколько функций, которые при этом могут по-разному проявляться в различных типах клеток на разных стадиях, эти функции или области активности предкового гена могут быть поделены между его дочерними копиями (Glasauer, Neuhauss, 2014). Согласно предложенной модели, мутации могут накапливаться в обеих дочерних (или по отношению друг к другу – сестринских) копиях, снижая их функциональную эффективность. В результате каждая из копий поотдельности оказывается недостаточной для выполнения функции материнского гена и тогда сохранение обоих паралогов может поддерживаться отбором (Force et al., 1999; Lynch, Force, 2000; Lynch et al., 2001). Такое разделение труда между сестринскими копиями может быть связано с изменениями в регуляторных участках и в этом случае оно будет отражаться в изменениях паттернов экспрессии и разделении области экспрессии материнского гена между дочерними копиями. Это называют регуляторной субфункционализацией (рис. 3, Joyner et al., 1987; Woolfe, Elgar, 2007). Примером, иллюстрирующим такой сценарий, может служить экспрессия двух паралогов гена Engrailed1 – En1a и En1b у рыбы D. rerio. Ген En1a экспрессируется в зачатках грудных плавников, в то время как En1b – в нейронах заднего и спинного мозга (Force et al., 1999). При этом единственный ген En1 обнаруженный у мышей экспрессируется как в зачатках передних конечностей, так и в области заднего и спинного мозга (Joyner, Martin, 1987; Davis et al., 1991). Если мутационные изменения происходят в белок-кодирующей последовательности гена, они могут привести к распределению между дочерними копиями функциональных свойств предкового гена (рис. 3, Innan, Kondrashov, 2010).
Еще один из возможных путей субфункционализации был предложен Хьюзом в 1994 г., а позже назван Де Марэ и Раушером “уходом от адаптивного конфликта” (EAC – Escape from Adaptive Control) (Hughes, 1994; DesMarais, Rausher 2008). Эта модель рассматривает ситуацию, когда ген имеет две функции, которые конкурируют между собой и, в силу этого не могут быть одновременно оптимизированы (улучшение одной из функций приводит к ухудшению другой). В этом случае дупликация снимает имеющиеся ограничения и каждый из паралогов получает возможность специализироваться на одной из функций предкового гена, развивая и оптимизируя именно ее. Предполагается, что такой механизм скорее может реализовываться для генов, которые не являются критически важными для выживания, но могут повышать приспособленность организма (Sikosek et al., 2012). Изначально эта модель была предложена в качестве теоретической, однако впоследствии был описан ряд примеров ее реализации в природе (DesMarais, Rausher, 2008; Deng et al., 2010; Huang et al., 2012; Glasauer, Neuhauss, 2014).
Возможность обретения геном новой функции после дупликации, или неофункционализации, впервые так же была высказана С. Оно (Ohno, 1970). Этот механизм еще называют моделью “мутации при нефункциональности” (MDN-mutation during non-functionality) (Hughes, 1994; Conant, Wolfe, 2008). Название отражает основную идею – один из паралогов продолжает поддерживать функции предкового гена, в то время как второй, будучи свободным от функциональных обязательств, может свободно меняться. В данном случае, в отличие от описанных выше моделей, предполагается появление в мутирующем паралоге “полезной” мутации, придающей ему возможность выполнения новой функции (рис. 3). Поскольку такие мутации происходят редко, этот сценарий считается маловероятным по сравнению с не- или субфункционализацией (Glasauer, Neuhauss, 2014). В то же время, поскольку в данном случае происходит появление новой функции, именно этот сценарий рассматривается как потенциальный механизм появления новых признаков в эволюции.
Как и в случае субфункционализации, мутация может происходить в регуляторой (чаще) или кодирующей (реже) последовательностях (Braasch et al., 2006; Douard et al., 2008; Kassahn et al., 2009). Показано, что изменения паттернов экспрессии вследствие мутаций в регуляторной области могут приводить к морфологическим изменениям на эволюционном пути (Guerreiro et al., 2013; Vuolo et al., 2016; Moriyama, Koshiba-Takeushi, 2018).
Другим механизмом, обеспечивающим сохранение обоих паралогов может быть так называемый “эффект дозировки”. Если возрастание уровня экспрессии гена выгодно, его дупликация будет поддерживаться естественным отбором. Считается, что эта модель применима к трем группам генов. В первую очередь – к генам, участвующим в обеспечении взаимодействия организма с внешней средой, генам, регулирующим ответ на стрессы, генам, выполняющим сенсорные и транспортные функции и к генам, связанным с метаболизмом (Kondrashov et al., 2002). Во вторую очередь – к генам, эффективность которых зависит от концентрации (Kondrashov et al., 2004; Veitia et al., 2005). И, наконец, к генам, которые всегда нужны в клетке в больших количествах, например, кодирующих рибосомные белки и гистоны (Kondrashov et al., 2002; Sugino, 2006).
Согласно гипотезе генного баланса, в регуляторных сетях и каскадах важно сохранение определенного соотношения уровней экспрессии участвующих в них генов. Необходимость соблюдения этого условия может приводить к сценариям, в которых после дупликаций все гены определенного сигнального каскада остаются дуплицированными для поддержания его функционирования (Conant, Wolfe, 2007). Поскольку ПГД приводят к удвоению всего генома, сами по себе они не приводят к концентрационным дисбалансам, в то время как дупликации отдельных генов могут имеющийся баланс нарушать. В подтверждение этой идеи и у растений, и у животных было показано, что паралоги генов, функционально тесно связанных между собой, например, генов, участвующих в транскрипционных и сигнальных каскадах, или кодирующих белки, активно включенные в межбелковые взаимодействия, имеют тенденцию к сохранению дуплицированных копий после ПГД, но не после локальных дупликаций (Blomme et al., 2006; Freeling, 2008; Hufton et al., 2009).
Приходится отметить, что иногда идентификация дуплицированных генов может быть затруднена в силу характера и масштаба произошедших в них изменений. Согласно базовой модели, появившиеся в результате дупликации два сестринских аллеля будут равноудалены от предкового гена и оба будут ортологами недуплицированных генов других групп (Sharman, 1999; Holland, 1999). В ходе эволюции дуплицированные гены будут меняться, накапливая изменения, уникальные для каждой из копий. Если скорость изменчивости обоих генов сопоставима (абсолютно идентичной она в любом случае не будет), можно говорить о симметричной дивергенции. Подобная картина наблюдается, например, для четырех кластеров Hox-генов млекопитающих, ортологичных единственному Hox-кластеру ланцетника и сопоставимо равноудаленных от него. Вдобавок все они унаследовали от предковых генов общие черты экспрессии в центральной нервной системе, дополняемые различающимися паттернами в других тканях – соматической и висцеральной мезодерме (Holland et al., 2016). Аналогично, три гена Cdx шпорцевой лягушки Xenopus tropicalis ортологичные единственному Cdx гену ланцетника, имеют общие с ним функциональные черты (Faas, Isaaks, 2009). Однако, кроме подобной симметричной дивергенции может наблюдаться и крайне неравномерное накопление изменений у дочерних копий, когда один из аллелей изменяется быстрее другого, и в этом случае говорят об ассиметричной дивергенции. Примеры такого сценария после ПГД также были описаны у позвоночных. Было показано, что ген Crx является ортологом генов Otx1 и Otx2 млекопитающих. Это удалось установить путем сопоставления расположения трех генов в хромосомах, при том, что по характеру экспрессии ген Crx, экспрессирующийся в глазной сетчатке, отличается как от Otx1 и Otx2, так и от ортологов у беспозвоночных – Otx ланцетника и otd дрозофилы (Plouhinec et al., 2003). Похожая ситуация описана и для гена Pax6, который активно изучался у позвоночных и беспозвоночных в качестве одного из ключевых ранних регуляторов нормального развития зрительных структур (Quiring et al., 1994). Как и в случае с генами Otx, только сравнительно недавно, благодаря анализу хромосомного расположения было установлено, что ген Pax4 является паралогом гена Pax6. За время своей эволюции, в отличие от Pax6, последовательность и регуляторная область гена Pax4, участвующего в развитии печени у позвоночных, сильно изменились (Matsushita et al., 1998). Изменения нуклеотидной последовательности Pax4 настолько существенные, что при филогенетическом анализе для него не обнаруживается гомологов у беспозвоночных и на филогенетическом дереве формально он выглядит как новый ген позвоночных, хотя в действительности является паралогом Pax6 (Holland et al., 2016).
ПОЗВОНОЧНЫЕ – ТРИУМФ ИННОВАТОРОВ
Эволюционным инновациям трудно дать строгое определение, но, как правило, под ними подразумевают качественно новые адаптивные особенности организмов. Зачастую они представлют собой основу, на базе которой могут развиваться последующие эволюционные изменения. Так, развитие зрительной системы, появление цветка или возможности полета, открыли перед их носителями возможности освоения новых экологических ниш. При этом, как отмечал еще в 1905 г. один из пионеров исследований полиплоидии генетик Хьюго де Фриз “естественный отбор может объяснить выживание наиболее приспособленных, но он не может объяснить появление наиболее приспособленных” (de Vries, 1905). Последующее развитие биологии показало, что все фенотипические инновации в итоге имеют молекулярную основу. Они являются результатом генетических изменений, влияющих на биологическую функцию и регуляцию активности как отдельных биологических макромолекул, так и на образуемых этими макромолекулами сетей взаимодействия (Wagner, 2011).
План строения позвоночных можно рассматривать как цельную мозаику, состоящую из филогенетически взаимосвязанных составляющих. Одни могут быть прослежены на глубину общего предка позвоночных или даже всех вторичноротых, и послужили основой для формирования характерных черт современных групп (York, McCauley, 2020). Другие не имеют очевидных предшественников у вымерших или ныне живущих представителей беспозвоночных (Lowe et al., 2015; Satoh, 2016).
Список морфологических особенностей позвоночных внушителен и среди наиболее общепризнанных нужно упомянуть: развитый головной мозг, состоящий из трех основных отделов, включая уникальный для позвоночных конечный мозг, сложно устроенную периферическую нервную систему с парными сенсорными органами, внутреннее ухо с вестибулярным аппаратом и полукруглыми каналами, мышечный дыхательный аппарат, череп, сформированный хрящевой и/или костной тканью; наджаберную, поджаберную и глазную мускулатуры; многокамерное сердце с сердечной мускулатурой; глоточные артерии с эндотелием; сегментированную систему почечной фильтрации (Janvier, 2003; Gee, 2018; York, McCauley, 2020).
Одной из ярких особенностей позвоночных является то, что многие их морфологические инновации развиваются при непосредственном участии одной популяции эмбриональных клеток – клеток нервного гребня (КНГ) (Sauka-Spengler et al., 2007; Hall, 2008; Trainor, 2013). Иногда эту клеточную популяцию даже называют четвертым зародышевым листком, по аналогии и в дополнение к трем классическим типами зародышевых тканей – эктодерме, мезодерме и эндодерме.
Нервный гребень уникален для позвоночных, и он представляет собой популяцию мигрирующих стволовых клеток, дающих начало множеству внешне разнородных структур и черт, во многом и определяющих строение и облик позвоночных. В числе производных нервного гребня можно назвать значительную часть периферической нервной системы, пигментные клетки, элементы сердца и зубов, а также, так называемую, “новую голову” позвоночных – хрящевые и костные структуры формирующие черепно-лицевой скелет (Gans, Northcutt, 1983; Northcutt, 2005; Green et al., 2015). У челюстноротых скелет головы дополняется челюстными структурами, несущими ряды зубов (Northcutt, 1983; Kuratani, 2004; Northcutt, 2005; Gans, Green et al., 2015; Brazeau, Friedman, 2015; Miyashita, 2016).
Все эти инновации, включающие развитие нервной и сенсорной, дыхательной и кровеносной систем, скелета и мускулатуры позволили ранним позвоночным перейти к более активным образу жизни и питания и, в итоге, освоить новые места обитания и экологические ниши (York, McCauley, 2020). Ряд исследований показывает, что и в наши дни КНГ продолжают участвовать в непрерывной морфологической эволюции современных видов, например, у домашних животных (Wilkins et al., 2014; Prescott et al., 2015; Sánchez-Villagra et al., 2016). Это свидетельствует о том, что КНГ не только участвуют в формировании многих уже состоявшихся структур позвоночных, но и остаются источником развития новых признаков и особенностей животных (York, McCauley, 2020).
Значительная часть наших знаний об эмбриональном развитии позвоночных получена в работе с традиционными модельными объектами, такими как зародыши мышей, кур, рыб, шпорцевых лягушек (Gilbert, 2006).
Достоинствами этих объектов являются: возможность получения живых зародышей для проведения экспериментов практически в любое время года, возможность создания генетических линий, возможность применения имеющихся лабораторных методик и подходов, детально разработанные протоколы для работы и содержания как эмбрионов, так и взрослых особей в лабораторных условиях, качественно отсеквенированные и проанализированные геномы и транскриптомы. Эти особенности, безусловно, играют важную роль для успешного проведения функциональных экспериментов. В то же время, для исследования эволюционной истории позвоночных особую ценность приобретают исследования так называемых “немодельных” объектов. Не обладая набором качеств, делающих их удобными для комфортной лабораторной работы, эти животные, однако, занимают важные филогенетические узлы на эволюционном пути позвоночных, что объясняет возрастающий в последние годы интерес к их исследованиям. К таким объектам можно отнести представителей хрящевых и лучеперых рыб, а также бесчелюстных (Dahn et al., 2007; Ota, Kuratani, 2007; Oisi et al., 2007, 2013; Green, Bronner, 2014; Braasch et al., 2015; McCauley et al., 2015; Pasquier et al., 2017). Исследования хрящевых рыб могут помочь ответить на вопросы появления и эволюции кожного скелета и появления парных плавников. Архаичные представители лучеперых, такие как осетровые рыбы и панцирная щука – вопросы трансформации плавников в конечности и эволюции сенсорных органов (York, McCauley, 2020). В своей монографии С. Оно отдельно акцентировал контринтуитивный тезис о том, что структуры более эволюционно продвинутых животных, как правило, берут свое начало от базовых, а не от продвинутых представителей предковой группы. В связи с этим, автор предлагает понимать термин “примитивный” как “незафиксированный и обобщенный”, а “продвинутый” – как “специализированный” (Ohno, 1970).
В рамках исследований истории и механизмов формирования генотипа позвоночных путем геномных дупликаций упомянутые представители рыб интересны тем, что у осетровых обнаружена полиплоидия, а панцирная щука, напротив, отделилась от общего ствола современных костистых рыб до произошедшей у предка этой группы ПГД (Sacerdot et al., 2018; Du et al., 2020).
Круглоротые, в контексте исследования вопросов ранней эволюционной истории позвоночных, заслуживают отдельного рассмотрения.
КРУГЛОРОТЫЕ – ТЕНИ ЗАБЫТЫХ ПРЕДКОВ
В силу своего филогенетического положения, круглоротые, отделившиеся от общего ствола на самых ранних этапах эволюции позвоночных являются очень удачным модельным объектом для исследования происхождения позвоночных и их уникальных особенностей (Shimeld, Donoghue, 2012). Современные круглоротые включают в себя миног и миксин, значительно отличающихся между собой по внешнему виду и образу жизни (Bayramov, 2018). В связи с этим, филогенетическое родство между миногами, миксинами и челюстноротыми долгое время оставалось предметом дискуссий. Одна гипотеза предполагала, что сестринскими группами являются миноги и челюстноротые, а миксины рассматривались в качестве внешней группы (парафилетическое происхождение круглоротых). Ей противостоял взгляд на круглоротых, как на единую группу монофилетического происхождения, в полном составе сестринскую челюстноротым (Janvier, 1996; Heimberg et al., 2010). В последнее время более уверенное подтверждение получила вторая гипотеза об эволюционном единстве современных круглоротых, произошедших от разнообразной группы бесчелюстных рыб – одних из первых представителей позвоночных, появившихся около 500 млн лет назад (Janvier, 2015; Onimaru, Kuraku, 2018; York, McCauley, 2020). К сожалению, миноги и миксины являются единственными ныне живущими представителями этой группы, а остальные бесчелюстные, как и переходные формы от бесчелюстных к челюстноротым, вымерли около 300 млн лет назад (Donoghue, Purnell, 2005; Donoghue, Keating, 2014). Являясь сестринской для челюстноротых группой, современные круглоторые занимают важное филогенетическое положение, позволяя путем сравнительного анализа их строения со строением челюстноротых реконструировать черты общего предка позвоночных. При том, что с эволюционной точки зрения, миноги и миксины в равной степени интересны для исследования базовых черт позвоночных, в лабораторных условиях миноги, при всех имеющихся технических ограничениях, все же имеют существенное преимущество перед миксинами. Взрослые особи миксин труднодоступны, поскольку живут и нерестятся на больших глубинах, зародыши развиваются несколько месяцев и для содержания этих животных требуются специальные лабораторные условия (Ota et al., 2007; Kuratani, Ota, 2008; Shimeld, Donoghue, 2012). Работа Оты и коллег, описывающая развитие нервного гребня у миксины – пиявкорота Бургера Eptatretus burgeri, опубликованная в 2007 г., стала первым исследованием развития миксин за более чем 100 лет (Ota et al., 2007). Миноги же являются наиболее доступными для исследований представителей круглоротых (Bayramov et al., 2018).
Попытки реконструкции облика и строения общего предка позвоночных неизбежно сталкиваются с рядом трудностей, таких как отсутствие надежных внешних групп для современных позвоночных и недостаток информации о филогенетически важных древних представителях круглоротых, в силу своего строения плохо сохраняющихся в палеонтологической летописи (Onimaru, Kuraku, 2018).
Ближайшими родственниками позвоночных являются бесчерепные (ланцетники) и оболочники (асцидии), которые по своему строению сильно отличаются от позвоночных. Внутри эволюционной линии позвоночных круглоротые традиционно рассматриваются в качестве внешней группы по отношению к челюстноротым, однако нельзя исключаться, что отдельные морфологические особенности их современных представителей – миног и миксин – имеют вторичное происхождение. Эти обстоятельства снижают надежность применения традиционных филогенетических методов, основанных на анализе синапоморфий (общих признаков предковой и дочерних групп) и придерживающихся принципа максимальной парсимонии (экономности числа событий).
Масштабная работа Кураку и коллег, включавшая анализ 55 семейств генов позвоночных, как и ряд последующих исследований, свидетельствовали в пользу гипотезы о двух общих для всех позвоночных раундах полногеномной дупликации их общего предка (Kuraku et al., 2009; Lagman et al., 2013; Campanini et al., 2015; Gutierrez-Mazariegos et al., 2016). В то же время, при работе с бесчелюстными, исследователи неизбежно обращали внимание, что при анализе филогении и построении филогенетических деревьев, гены бесчелюстных зачастую уверенней группируются друг с другом, чем с ортологами челюстноротых. При этом общее число генов у бесчелюстных и челюстноротых часто совпадает (Qui et al., 2011). Для понимания этого феномена был проведен анализ гомео-боксных генов Emx, для которых была ранее установлена дупликация в линии миног (Tank et al., 2009). Было показано, что столь часто наблюдаемая кластеризация множественных миножьих генов может быть связана со вторичными изменениями их последовательностей, происходящими независимо у разных паралогов (Noro et al., 2015). Эта особенность генов миног даже получила название “миножий диалект” (Manousaki et al., 2011; Smith et al., 2013).
Для реконструкции предкового фенотипа позвоночных был предолжен подход, основанный на установлении паттернов экспрессии генов до полногеномных дупликаций – метод хронологической реконструкции функции онологов (CHROF от chronological reconstruction of ohnolog functions) (Onimaru, Kuraku, 2018). Этот подход позволяет восстановить паттерн экспрессии предкового гена опираясь на паттерны его дочерних генов, прошедших путь субфункционализации. Предполагается, что регуляторная субфункционализация онологов после ПГД отражается в сходстве паттернов их экспрессии. В основе субфункционализации, согласно упоминавшейся выше модели дупликации-вырождения-дополнения, лежат комплементарные изменения регуляторных элементов онологов. На основе анализа паттернов онологов реконструируется эволюционный путь их паттернов и паттерн предкового гена. Если наблюдается сходство экспрессии онологов в определенной морфологической структуре у нескольких современных видов, история этой структуры может быть прослежена до этапа, предшествующего появлению современных онологов в результате ПГД. Примером подобной реконструкции может выступать анализ паттернов экспрессии генов Pax2, Pax5 и Pax8, появившихся в результате двух раундов ПГД (Kozmik, 1999; Bassham et al., 2008; Goode, Elgar, 2009). У мышей эти гены экспрессируются на границе среднего и заднего мозга, а Pax2 и Pax8 также экспрессируются в пронефросе. У миног наблюдается экспресия Pax2 в тех же структурах (McCauley, Bronner-Fraser, 2002), что позволяет предположить наличие у предкового позвоночного наличие структур гомологичных границе среднего и заднего отделов мозга и пронефроса, с экспрессией в них гомолога Pax2/5/8. В поддержку этой гипотезы говорит то, что экспрессия гена Pax2/5/8 у ланцетника имеет сходства с экспрессией гомологов у позвоночных (Holland, 2013). Такой же подход был применен к анализу экспрессии генов Tbx5 и Tbx4 в передних и задних парных конечностях позвоночных регулируемых специфическими для каждого вида конечности энхансерами (Menke et al., 2008; Minguillon et al., 2012; Adachi et al., 2016). Предполагается, что предковый ген Tbx4/5 экспрессировался в единственной паре конечностей предкового позвоночного, а после дупликации произошла субфункционализация дочерних генов со специализацией каждого из них на экспрессии в отдельной паре конечностей. Остается, правда, открытым вопрос являлась ли эта субфункционализация причиной или следствием появления у потомков двух пар конечностей (Ruvinsky, Gibson-Brown, 2000). К сожалению, палеонтологические исследования ранних позвоночных метасприггины Metaspriggina walcotti и хайкоуихтиса Haikouichthis ercaicunensis, живших до разделения бесчелюстных и челюстноротых пока не обнаружили у них следов парных конечностей (Shu et al., 1999; Shu et al., 2003; Zhang, Hou, 2004; Morris et al., 2014). В то же время у ряда ранних представителей бесчелюстных, таких как Euphaneropidae, анаспиды Anaspida, телодонты Thelodonti и щитковые Ostracodermi описаны парные плавники, хотя их гомология с конечностями челюстноротых небесспорна, и является предметом дискуссий (Ruvinsky, Gibson-Brown, 2000; Coates et al., 2003; Sansom et al., 2010, 2013).
Уязвимостью предложенного подхода является принимаемое в нем допущение, что субфункционализация является более распространенным явлением, чем неофункционализация, по крайней мере, предложенный алгоритм позволяет реконструировать предковые паттерны только в этом случае. Также при отождествлении морфологической эволюции с изменениями паттернов экспрессии генов не учитывается возможность того, что новая структура может появиться не вследствие появления новых, а путем перераспределения активности уже имевшихся у организма генов, их функционального переключения. В целом авторы предлагаемого подхода выражают надежду, что его применение позволит найти эволюционные корни многих уникальных структур позвоночных у ранних представителей группы (Onimaru, Kuraku, 2018).
HOX-ГЕНЫ – НА ПЛЕЧАХ ГИГАНТОВ
Исторически, импульсом к появлению гипотезы о полногеномных дупликациях у позвоночных во многом послужило описание и исследование Hoх-генов, которые представляют собой семейство высококонсервативных гомеобоксных генов обнаруженных в геномах многих групп животных. Они кодируют транскрипционные факторы, участвующие в дифференцировке вдоль передне-задней оси тела (Graham et al., 1989; Carroll, 1995). В геноме Hoх-гены сгруппированы в кластеры. У млекопитающих описано четыре паралогичных Hox-кластера, которые возникли в результате дупликаций от общего предкового кластера на ранних этапах эволюции позвоночных (рис. 4, Duboule, Dolle, 1989; Pascual-Anaya et al., 2013; Parker, Krumlauf, 2017). В отдельных линиях позвоночных можно наблюдать как дополнительные дупликации, так и частичные утраты Hox-генов (Kuraku, Meyer, 2009; Pascual-Anaya et al., 2013). На основе сходства последовательностей и положения в кластерах Hox-гены позвоночных подразделяются на 14 групп паралогов (ГП) (Krumlauf, 1994). Яркой особенностью Hox-кластеров позвоночных является их коллинеарность – в ходе онтогенеза пространственно-временная экспрессия Hox-генов вдоль передне-задней оси коррелирует с их расположением в кластере. При этом ориентация (расположение 5' и 3' концов) у всех генов в кластере совпадает. Первыми экспрессируются антериорные гены, относящиеся к ГП1 и расположенные на 3' конце кластера, то есть коллинеарность носит как временной, так и пространственный характер (Duboule, Dolle, 1989; Kmita, Duboule, 2003; Duboule, 2007). Это проявляется в наблюдаемом наборе экспрессионных доменов Hox-генов, иногда называемой Hox-кодом, который отражает особенности стратификации разных тканей зародыша вдоль передне-задней оси тела (Mallo et al., 2010).
Рис. 4.
Кластеры Hox-генов, обнаруженные у ланцетника, миног и млекопитающих (по Parker et al., 2019a).

Hox-гены связаны с модуляцией регуляторных сигналов в заднем отделе мозга и клетках нервного гребня. Задний мозг позвоночных имеет выраженную сегментную структуру, включающую 7–8 ромбомеров и влияющую на дифференцировку черепно-лицевых структур через КНГ, мигрирующие из нервной трубки в жаберные дуги. Как показали функциональные исследования у разных групп челюстноротых, роль Hox-генов в развитии заднего мозга и КНГ, по-видимому, является высококонсервативной и может быть прослежена по меньшей до уровня общего предка группы (Parker et al., 2019a). Столь древнее происхождение подтверждается и консервативностью регуляторных элементов – Hox-энхансеров, определяющих пространственную сегментацию экспрессии и Hox-чувствительных энхансерных элементов, относящимся к регулируемым ими генам-мишеням (Kim et al., 2000; McEwen et al., 2009; Ravi et al., 2009; Parker et al., 2011, 2014b; Parker, Krumlauf, 2017).
Анализ геномов двух видов миног – морской миноги (Petromyzon marinus) и тихоокеанской миноги (Lethenteron camtschaticum) показал наличие у них шести Hox-кластеров (в полтора раза больше, чем у челюстноротых!), что свидетельствует о дополнительных дупликациях у миног (рис. 4, Kuraku, Kuratani, 2006; Mehta et al., 2013; Pascual-Anaya et al., 2018; Smith et al., 2018). При этом потеря части Hox-генов в кластерах уменьшила количественную разницу между миногами и челюстноротыми – у миног обнаружено 42 Hox-гена, а у мышей 39 – разница отнюдь не в полтора раза (рис. 4). В наиболее антериорных ГП1–4, которые и связаны с экспрессией в заднем мозге и КНГ, в силу чего приковывают к себе максимальноое внимание исследователей, у миног описано 14 генов, а у мышей – 12 (Parker et al., 2019a). Проведенный недавно анализ генома и транскриптома миксины Eptatretus burgeri обнаружил у этого вида 40 Hox-генов, предположительно распределенных по 6 кластерам (Pascual-Anaya et al., 2018). Сравнение их с Hox-кластерами миног выявило ряд общих черт. Каждый вид имеет одинаковое количество Hox-паралогов в ГП1–4, и это указывает, на то, что, вероятно, разделение Hox-кластеров бесчелюстных произошло до дивергенции миног и миксин – более 400 миллионов лет назад (Kuraku, Kuratani, 2006).
Хотя шесть Hox-кластеров круглоротых не получается однозначно филогенетически соотнести с четырьмя кластерами челюстноротых (Pascual-Anaya et al., 2018), это не свидетельствует однозначно об их независимом происхождении. Произошедшее после предковой дупликации быстрое разделение групп с последующей их дивергенцией в том числе и по нуклеотидным/аминокислотным предпочтениям могут существенно ослабить возможность их уверенного филогенетического объединения на основе доступных сегодня алгоритмов (Qiu et al., 2011). В свою очередь, реконструкции, основанные на сопоставлении порядка генов в хромосомах у разных групп позвоночных, соответствуют модели, в которой у общего предка круглоротых и челюстных было четыре Hox-кластера (Smith et al., 2018).
Анализ экспрессии Hox-генов миног выявил у них много общих черт с Hox-генами челюстноротых, прежде всего касающихся экспрессии в ромбомерах заднего мозга и клетках нервного гребня (Parker et al., 2019a). Отличия наблюдались в деталях поздней экспрессии в области заднего мозга, эндостиле и мезодерме первой жаберной дуги. Сопоставление характера экспрессии и кластерной организации Hox-генов у миног и челюстноротых позволяет заключить, что предковые функции Hox-генов в этих группах во многом сохраняются, но по-разному распределяются между дочерними кластерами. Другими словами, Hox-кластеры миног и челюстноротых, имея общее происхождение, прошли на своем эволюционном пути разные варианты субфункционализации.
У беспозвоночных хордовых – ланцетников и оболочников – также наблюдается коллинеарность единственных Hox-кластеров вдоль передне-задней нейральной оси, хотя, у этих групп не обнаруживается характерной для позвоночных сегментации ромбомеров и клеток нервного гребня (Parker, 2019b).
ПОЛНОГЕНОМНЫЕ ДУПЛИКАЦИИ У ПОЗВОНОЧНЫХ – ОДНА ХОРОШО, А ДВЕ ЛУЧШЕ
Основные предполагаемые сценарии полногеномных дупликаций у позвоночных во многом опираются на присутствие одного кластера Hox-генов у их ближайших родственников – ланцетников и оболочников, и разное количество кластеров в двух рано разделившихся ветвях позвоночных – бесчелюстных (шесть Hox-кластеров) и челюстных (четыре Hox-кластера). Исходя из такого количества Hox-кластеров понятно, что в истории позвоночных имели место как минимум два раунда дупликаций, а у бесчелюстных, судя по всему, и больше. Исследователи, как правило, сходятся во мнении, что один раунд произошел на уровне общего предка позвоночных (Holland, Ocampo Daza, 2018). А вот по поводу второго раунда (и возможных дополнительных) – ведутся дискуссии.
Первой была выдвинута гипотеза о двух раундах у общего предка позвоночных до разделения линий бесчелюстных и челюстноротых (Mehta et al., 2013), а основанием для последующих пересмотров и дискуссий во многом стал проведенный подробный анализ секвенирования полного генома морской миноги.
Получение полноценного сиквенса генома миноги оказалось непростой задачей. Дело в том, что одной из особенностей миног, наряду со способностью функциональной регенерации спинного мозга даже во взрослом состоянии, эволюционно независимого развития адаптивного иммунитета и ряда других, является физическая плановая перестройка генома на ранних стадиях развития (PGR от Programmed Genome Rearrangement). Эта геномная перестройка состоит в редукции примерно 20% генома (около 0.5 млрд оснований из 2.3 млрд оснований всего генома) (Smith et al., 2018). Начинается она на седьмом клеточном делении и длится около трех суток, после чего только геномы клеток зародышевой линии несут полный геном, а соматические клетки – редуцированный. Очевидно, что полный геномный сиквенс в таком случае можно получить только путем анализа геномов клеток зародышевой линии, что было сделано в 2018 г. (Smith et al., 2018). Проведение предварительного картирования распределения контигов по супер-скэффолдам (super-scaffolds – собранные после секвенирования геномные протяженные фрагменты, содержащие в среднем около 10–20 млн оснований, которые в дальнейшем предполагается объединять в хромосомы) позволило сопоставить расположение генов у миног и некоторых челюстноротых. Благодаря развитию алгоритмов обработки больших массивов данных, в основе этого сопоставления лежало уже выявление не отдельных генов-ортологов, а целых групп ортологии между разными геномами.
Сопоставление генома морской миноги с геномами курицы и панцирной щуки (представитель костных ганоидов, у которых, в отличие от более эволюционно продвинутых костистых рыб, не было дополнительной дупликации) позволил выдвинуть гипотезу об одном раунде ПГД у общего предка позвоночных, с предшествующими им локальными дупликациями отдельных участков и последующими дополнительными дупликациями хромосом у бесчелюстных (рис. 5, Smith et al., 2015, 2018). Это предположение основано на том, что больше половины (6 из 11) миножьих ортологических групп соответствуют двум куриным хромосомам. Еще две группы не находят четкой гомологии. Оставшиеся три группы имеют гомологию с четырьмя хромосомами. Что, казалось бы, свидетельствует в пользу двух общих раундов у птиц и миног, причем, эти хромосомы несут как раз хорошо охарактеризованные гены – одна Hox и MHC, вторая – ParaHox и NPYR, третья – RAR и ALDH1. Однако Смит и коллеги предполагают, что такая картина могла возникнуть в результате трех независимых хромосомных дупликаций, предшествовавших ПГД. Таким образом, согласно предлагаемой модели, до разделения бесчелюстных и челюстноротых у их общего предка сначала произошли отдельные дупликации трех хромосом, а затем одна полногеномная дупликация (рис. 5, Smith et al., 2018). Хотя, авторы полностью не исключают вероятности того, что нынешний кариотип миног и челюстноротых мог сформироваться в результате двух раундов полногеномной дупликации с последующей элиминацией значительного числа хромосом, они оценивают такой сценарий как менее вероятный.
Рис. 5.
Схемы рассмотренных моделей геномных дупликаций на ранних этапах развития позвоночных. ПГД – полногеномные дупликации, ЛД – локальные дупликации.

Авторы полагают, что у миног есть определенные биологические особенности, которые могут способствовать фиксации хромосомных дупликаций в этой группе. Кариотип миног (основным объектом Смита и коллег является морская минога Petromyzon marinus) состоит примерно из 100 хромосом, среди которых много, так называемых, малых хромосом, дупликация которых будет затрагивать меньшее число генов, и с большей вероятностью пройдет успешно для организма, чем аналогичная дупликация крупных хромосом у человека. Проведенное в 2020 г. секвенирование генома “дальневосточной ручьевой” миноги Lethenteron reissneri выявило у этого вида 72 хромосомы (Zhu et al., 2020). С другой стороны, миноги могут быть очень плодовиты (продуктивность самки морской миноги может достигать 300 000 икринок), в силу чего носитель одной мутации может распространить ее на большое число потомков в популяции (Smith et al., 2015, 2018). Причем, есть основания полагать, что эти характеристики были присущи миногам на протяжении сотен миллионов лет их эволюционной истории, в силу чего они представляют собой очень релевантную модель исследования особенностей предковых форм позвоночных, вполне вероятно обладавших высокой плодовитостью и большим числом малых хромосом (Smith et al., 2018).
Первые попытки реконструкции предкового кариотипа были проведены Накатани и коллегами в 2007 г., когда исследователи выделили в геноме человека консервативные для позвоночных группы связанных генов (Nakatani et al., 2007). Сравнение этих групп с группами других позвоночных (рыб) и оболочников позволило выделить онологи и предположить наличие у предковых позвоночных от 10 до 13 хромосом. По мере накопления данных секвенирования представителей разных групп позвоночных возросли возможности их широкоформатного сравнения и более тонких реконструкций. В 2018 г. Сасердот с коллегами при помощи разработанного ими алгоритма AGORA (Algorithm for Gene order Reconstruction in Ancestors) на основе анализа 61 генома современных позвоночных провели реконструкцию предкового генома амниот – организмов с ориентировочным возрастом 326 млн лет. Выборка включала 40 видов млекопитающих, 3 вида птиц, 2 рептилии, 1 амфибию, 8 костистых рыб, кистеперую рыбу, 2 представителей оболочников, нематоду и дрозофилу (Sacerdot et al., 2018). В результате в предковом геноме амниот предположили наличие 19 786 генов. Были определены предполагаемые пары онологов и выявлены 51 протяженная предковая область (CAR – Contiguous Ancestral Regions) – фрагменты генома, содержащие более 50 генов каждый и по размеру соответствующие хромосомам. Полученные фрагменты группируются в 17 тетрад (17 групп по 4 CAR), что наиболее вероятно соответствует двум раундам полногеномных дупликаций. Причем, выявленные авторами 17 тетрад соответствуют 17 предковым связкам (CLG – Chordate Linkage Group) выявленным ранее при сравнении синтении генов в хромосомах человека и скэффолдах генома ланцетника (Putnam et al., 2008). Количественное отклонение от точного ожидаемого числа хромосом, кратного четырем может быть объяснено исчезновением отдельных хромосом или объединением двух хромосом в одну. Эти явления можно было бы проследить путем анализа геномов внешних групп, но в случае позвоночных геномы ланцетников и оболочников для этих целей, к сожалению, не подходят, поскольку оболочники существенно дивергировали от своего предкового состояния, а у ланцетников геном слишком фрагментирован. Оба этих обстоятельства затрудняют установление хромосомной гомологии. В рамках своей модели Сасердот и коллеги предполагают наличие у предковых позвоночных 17 хромосом, которые при первом раунде дупликации дали начало 34 хромосомам. В дальнейшем произошло 7 объединений хромосом и общее их число редуцировалось до 27, которые во втором раунде, произошедшем до разделения линий бесчелюстных и челюстноротых удвоились с образованием 54 хромосом, часть из которых впоследствии также объединялись. Не исключая теоретические вероятности и других сценариев, полемизируя со Смитом и коллегами, авторы полагают, что сравнение генома миног с реконструированным геномом предка амниот дает более корректные результаты, чем сопоставление с отдельными представителями челюстноротых. В рамках предложенного Сасердот и коллегами анализа группы ортологов в супер-скэффолдах миног в большинстве случаев соответствуют четырем хромосомам у предка амниот (в отличие от модели Смита и коллег, в которой доминировало соотношение 1 : 2), что поддерживает гипотезу о двух общих раундах полногеномной дупликации до разделения бесчелюстных и челюстноротых (рис. 5).
Еще одна модель геномной дупликации у позвоночных была предложена Симаковым и коллегами в 2020 г. В ней, с учетом обновленных данных, авторы рассматривают ланцетника как наиболее близкую к предковым позвоночным группу. Построение модели опирается на те же 17 выявленных консервативных от ланцетника до человека связок (CLG). Авторы предлагают рассматривать синтению групп генов без учета порядка генов в них, который, по их мнению, мог сравнительно легко меняться в ходе эволюции (Simakov et al., 2020). Кариотип ланцетника состоит из 19 хромосом, которые, как предполагается, произошли от 17 предковых CLG, претерпев некоторые локальные преобразования. Часть хромосом позвоночных, в свою очередь, также произошли от одного CLG, а некоторые – в результате слияния двух или трех CLG с последующими изменениями. Малые хромосомы миног, панцирной щуки и курицы размером менее 15 млн пар оснований считаются потомками одной из CLG (Simakov et al., 2020). Согласно предложенной модели, 10 из 17 CLG представлены в виде тетрад у челюстноротых, а остальные – в виде триплетов. Это свидетельствует в пользу двух раундов дупликаций с последующей утратой части хромосом. Анализ слияния хромосом челюстноротых показывает, что большинство этих событий произошло между первым и вторым раундами дупликаций. В то же время, у круглоротых последствий этих слияний не наблюдается и хромосомы миног, судя по всему, напрямую произошли от одиночных CLG. На основе этих данных авторы выдвигают гипотезу об одном раунде ПГД до разделения бесчелюстных и челюстноротых, с последующим вторым раундом в линии челюстноротых (рис. 5, Simakov et al., 2020).
В отличие от более ранних моделей, в которых оба раунда ПГД предполагали автотетраплоидизацию (удвоение диплоидного генома одного организма) (Furlong, Holland, 2002) или, напротив, аллотетраплоидизацию (слияние двух диплоидных геномов разных организмов) (Spring, 1997), в Симаков и коллеги предполагают, что первый раунд был автотетраплоидизацией, а второй – аллотетраплоидизацией. У миног, после отделения от общего ствола позвоночных, в модели предполагаются дополнительные независимые дупликации.
А ЧТО НА ЭТО СКАЖУТ ГЕНЫ?
Описанные модели, разработанные в последние годы, опираются на широкоформатный анализ данных высокопроизводительного секвенирования, объем которых существенно растет в последнее время. Преимуществом такого подхода, безусловно, является масштаб взгляда на проблему в силу количества анализируемой информации. В то же время, для выявления функциональных основ появления и развития отдельных структур в эволюции и онтогенезе позвоночных, ведутся исследования отдельных семейств регуляторных генов и видится важным сопоставление получаемых лабораторных данных с выдвигаемыми гипотезами. В рамках уточнения количества и времени полногеномных дупликаций в ранней эволюции позвоночных интересен обзор данных по сравнительному анализу ортологов регуляторных генов в линиях бесчелюстных и челюстноротых. Благодаря секвенированию генома морской миноги, в последние годы повышается и уровень лабораторных исследований отдельных семейств генов у этой группы. Рассмотрим поподробней некоторые примеры.
При исследовании геноархитектуры конечного мозга миног, в его дорсальной части была обнаружена экспрессия генов Pax6 (Murakami et al., 2001) и Emx (Tank et al., 2009), а в вентральной – генов Dlx (Miojin et al., 2001; Murakami et al., 2001; Neidert et al., 2002). Обнаружение зон экспрессии Pax6 и Dlx и стало на ранних этапах исследований основным свидетельcтвом наличия у миног структур, гомологичных конечному мозгу челюстноротых. Впоследствии, у миног были обнаружены три ортолога гена Pax6. Все они экспрессируются в формирующихся глазных структурах и мозге, что соответствует экспрессии гена Pax6 у челюстноротых, у которых этот ген является ключевым регулятором развития глаз и ЦНС (Osumi et al., 2008; Klimova, Kozmik, 2014). При этом паттерн экспрессии генов Pax6 миног в других структурах различается, что может служить примером пространственной субфункционализации. Экспрессия Pax6α обнаруживается в печени, указывая на то, что у предков позвоночных ген Pax6, по всей видимости, участвовал в формировании этого органа (Ravi et al., 2019). Анализ синтении (набора соседних генов) показал, что Pax6α имеет общие соседние гены (Eif3m и Caluα на 5' конце и Elp4 на 3' конце) с геном Pax6.1 челюстноротых и геном PAX6 человека, что указывает на их ортологию. Локус миножего Pax6β содержит гены Caluβ и Fbxo47, которые также обнаруживаются в окружении гена Pax6.2 у некоторых представителей челюстноротых. Третий ген Pax6 миног, Pax6γ, не обнаруживает синтении с Pax генами челюстноротых. Обнаружение у миног трех ортологов гена Pax6, очевидно, указывает как минимум на два раунда дупликаций, приведших к их появлению. Поскольку только два из них имеют синтению с генами Pax6 челюстноротых, авторы заключают, что их данные вполне могут соответствовать гипотезе Смита и коллег об одном общем раунде полногеномной дупликации для бесчелюстных и челюстноротых, а третий ортолог Pax6 появился у миног в результате локальной дупликации (Ravi et al., 2019). В то же время, как уже упоминалось выше, исследования геномного расположения и особенностей экспрессии показали, что гены Pax4 и Pax6 челюстноротых являются онологами, произошедшими в результате ПГД и последующей асимметричной дивергенции (Manousaki et al., 2011). Показано, что их экспрессия наблюдается в печени, сетчатке и шишковидной железе, что указывает на общность происхождения регуляторных элементов. Есть и отличия, в частности, Pax4 не экспрессируется в центральной нервной системе, включая глаза и обонятельные плакоды. Анализ аминокислотных последовательностей показал, что Pax4 претерпел более глубокие изменения своей структуры, что и позволяет говорить об асимметричной дивергенции онологов (Manousaki et al., 2011; Holland et al., 2016). В сочетании с данными по генам Pax6 у миног (ортологи Pax4 у них пока не описаны), на наш взгляд, в этом случае можно говорить о двух раундах дупликаций в истории Pax генов – в результате первого произошло разделение линий Pax4 и Pax6, а в результате второго – Pax6α и Pax6β. Такой сценарий вполне соответствует модели двух раундов ПГД до разделения бесчелюстных и челюстноротых.
Еще одним примером выступает описание и исследование у миног и челюстноротых генов семейства Noggin, которые являются важными регуляторами раннего развития структур переднего мозга позвоночных. Noggin стал первым известным фактором, в норме секретирующимся в области Шпеманновского организатора, для которого была показана способность вызывать формирование дополнительных осей тела в случаях его экспериментальной эктопической экспрессии на вентральной стороне зародыша амфибий (Dale, Slack, 1987; Smith, Harland, 1992; Lamb et al., 1993; Smith et al., 1993; Slack, Tannahill, 1993). Впоследствии были описаны два гомолога гена Noggin – Noggin2 и Noggin4 (Fletcher et al., 2004; Eroshkin et al., 2006). Долгое время считалось, что основной (по сути – единственной описанной) функцией Noggin1 в развитии позвоночных является подавление сигнального каскада BMP (bone morphogenetic proteins) – группы ростовых факторов из суперсемейства TGF-beta. Модуляция активности BMP каскада является необходимым условием для формирования нервной ткани и дифференцировки дорсальной мезодермы (Xanthos et al., 2002; Moreau, Leclerc, 2004). При исследовании гена Noggin2 было показано, что он обладает способностью подавлять не только BMP, но также и Nodal/Activin и Wnt/beta-cathenin сигнальные каскады, играющих ключевые роли в клеточной дифференцировке и развитии головных структур позвоночных. Оверэкспрессия мРНК Noggin2 в зародышах X. laevis приводит к формированию дополнительного комплекса осевых структур, в которых наблюдается экспрессия переднеголовных генов (Bayramov et al., 2011). У челюстных, за исключением костистых рыб, было описано 3 гена семейства Noggin – Noggin1, Noggin2 и Noggin4. У миног были обнаружены 4 гена (NogginA, NogginB, NogginC и NogginD) (Ermakova et al., 2020, 2021). Гены NogginA, NogginB, NogginC продемонстрировали сходство аминокислотных последовательностей и синтению с генами Noggin1, Noggin2 челюстноротых, а ген NogginD – с геном Noggin4. Оказалось, что гены NogginB и Noggin2 имеют сходный специфический паттерн экспрессии в области конечного мозга (рис. 6а). Ген Noggin2 позвночных уже был описан ранее в качестве важного регулятора формирования переднеголовных структур у амфибий (Bayramov et al., 2011). Экспрессия NogginA в нервной системе и мезодермальных структурах, в свою очередь, похожа на экспрессию Noggin1, а экспрессия NogginC имеет общие черты как с экспрессией Noggin1, так и Noggin2. Диффузный характер экспрессии NogginD сходен с экспрессией Noggin4 челюстноротых. Функциональный анализ способности генов Noggin миног индуцировать формирование дополнительного комплекса осевых структур подтвердил эти гомологии (рис. 6б). В совокупности, если исключить возможность независимого появления столь сходных характеристик генов семейства Noggin у бесчелюстных и челюстноротых, можно заключить, что к моменту разделения их линий у их общего предка уже присутствовали как минимум три разных гена Noggin. Поскольку у ближайших родственников позвоночных, ланцетников и асцидий, описано по одному гену Noggin, появление трех копий предполагает, как минимум, два раунда дупликаций. В связи с этим, исходя из совокупного анализа структуры, экспрессии и функциональных свойств генов Noggin можно говорить о двух раундах дупликаций предкового генома, произошедших в период между появлением общего предка позвоночных и дивергенцией линий бесчелюстных и челюстноротых. Приходится признать, что на данном этапе у нас нет возможности уверенно установить были эти дупликации полногеномными или дупликациями крупных фрагментов генома. В этом случае эволюционная история генов Noggin может описываться как моделью Смита и коллег, так и модель Сасердот и коллег (рис. 6в).
Рис. 6.
а, б – ген NogginB речной миноги Lampetra fluviatilis экспрессируется в зачатке переднего мозга и может индуцировать формирование дополнительного комплекса осевых структур у зародышей шпорцевой лягушки Xenopus laevis (консерватизм экспрессии и функциональных свойств). as – anterior intraencephalic sulcus; ch – optic chiasma; di – diencephalon; hb – habenula; hpt – hypothalamus; mes – mesencephalon; pg – pineal gland; tel – telencephalon. в – предполагаемая схема появления генов семейства Noggin в эволюции позвоночных (по Ermakova et al., 2020).
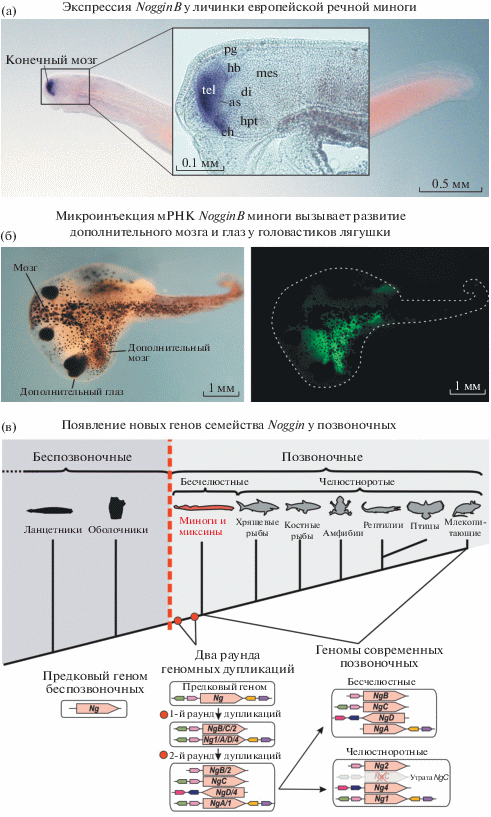
Модель Симакова и коллег, предполагающая один общий раунд дупликации у предковых позвоночных, может описать появление имеющихся Noggin генов при принятии одного из допущений: 1) исходно у позвоночных было два предковых гена Noggin (в этом случае, очевидно, NogginA/B/C/1/2 и NogginD/4) или 2) независимой дивергенции NogginA/B/C и Noggin1/2 у миног и челюстноротых. Контраргументы в первом случае – один Noggin ген у ланцетников и оболочников, а во втором – попарное сходство паттернов экспрессии NogginA и Noggin1 с одной стороны и NogginB и Noggin2 с другой. Безусловно, полностью исключить вероятность реализации таких сценариев невозможно, но они кажутся менее вероятными и требующими больших допущений, чем упомянутые выше два раунда предковой дупликации (Ermakova et al., 2020).
Похожую картину продемонстрировало исследование генов семейства CRD (corticotropin-releasing hormone) (Cardoso et al., 2020). Ранее у челюстноротых было описано пять генов этого семейства (CRH1/CRH2/UCN1 и UCN2/UCN3), произошедших от двух предковых генов в результате двух раундов дупликаций с последующими частичными редукциями (Cardoso et al., 2016). У миног также были обнаружены пять генов этого семейства, которые при филогенетическом анализе последовательностей не продемонстрировали прямой 1 : 1 ортологии с генами челюстноротых. При построении филогенетических деревьев три миножьих гена группируются с генами CRH1/CRH2/UCN1, а два других – с генами UCN2/CN3 челюстноротых (полагаем, как и в случае генов Noggin, это можно назвать “облачной ортологией”). Это позволяет предположить происхождение CRH генов миног от тех же двух предковых генов, что и CRH генов челюстноротых, а отсутствие прямой ортологии объяснить упоминавшимся выше “миножьим диалектом”. Проведенный анализ окружающих генов показал, что некоторые из них представлены у миног четырьмя копиями (тетрадами). Наиболее простым объяснением такого распределения CRH генов у миног является предположение о том, что в своей эволюции они прошли те же два раунда дупликаций, что и гены челюстноротых, что соответстувет двухраундовым моделям дупликации у предка позвоночных (Cardoso et al., 2020).
Гены, входящие в подсемейство FoxP, играют важную роль в развитии и поддержании многих структур и функциональных систем организма – легких, сердца, ЦНС, иммунной системы (Song et al., 2016). Белки FoxP содержат в своей структуре три важных домена – цинковый палец, лейциновую молнию и ДНК-связывающий forkhead-домен, характерный для всех Fox белков (Takahashi et al., 2009). У ланцетников и оболочников описано по одному гену FoxP, в то время как у большинства позвоночных – четыре (FoxP1–4) (Takahashi et al., 2009; Santos et al., 2011), хотя есть и исключения. Так, у птиц отсутствует FoxP3, а у костистых рыб, вследствие дополнительного раунда полногеномной дупликации, напротив, количество генов семейства увеличено (Yang et al., 2010; Andersen et al., 2012; Song et al., 2013). У млекопитающих FoxP гены располагаются в паралогичных хромосомах, что свидетельствует об их происхождении в результате крупномасштабных дупликаций (Dehal, Boore, 2005). Вполне органичной в таком контексте выглядит гипотеза о двух раундах полногеномной дупликации. Подтверждением этой гипотезы стало описание четырех FoxP генов у миног (Song et al., 2016). Причем, аналогичная картина была описана и для других генов, связанных с FoxP как расположением в хромосомах, так, судя по всему, и функционально – генами семейств MIT, mGluR и PLXNA (Song et al., 2016).
Гены ParaHox представляют собой кластер древних регуляторных гомеобоксных генов, эволюционно родственных с Hox-генами (Zhang et al., 2017). Кластер ParaHox включает три семейства транскрипционных факторов – Gsx (у человека – GSH), Pdx (синонимы Xlox, Ipf1), и Cdx, участвующих в развитии нервной и пищеварительной систем (Valerius et al., 1995). У ланцетника описан один ParaHox кластер, включающий все три гена. У позвоночных ParaHox гены распределены по четырем и более локусам, каждый из которых далеко не всегда содержит полный набор из генов. К примеру, у человека кластер в 13-ой хромосоме содержит все три гена (GSX1, PDX1, CDX2), а остальные три локуса – лишь по одному гену – GSH2 в 4-ой хромосоме, CDX1 в 5-ой хромосоме и CDX4 в X хромосоме. Анализ дупликаций генов, окружающих ParaHox кластеры в четырех хромосомах человека, свидетельствует о появлении этих кластеров в результате двух раундов ПГД (Polard, Holland, 2000). Для уточнения ранних этапов эволюции ParaHox генов, внимание исследователей традиционно обратилось к круглоротым, как эволюционно древней группе. Анализ геномных и транскриптомных сиквенсов показал наличие у миног пяти ParaHox генов, организованных в два кластера Gsxα-Pdxα-Cdxα и Gsxβ-Cdxβ. Филогенетический анализ, как это часто случается при анализе генов миног, оказался не очень информативным для поиска прямой ортологии с генами челюстноротых, поскольку гены миног, в силу особенностей состава (уже неоднократно упоминавшийся ранее “миножий диалект”) уверенней кластеризуются друг с другом, чем с ортологами в других группах. Подобный эффект неоднократно наблюдался и при анализах других семейств генов – Hox, KCNA, p53 (Qiu et al., 2011; Mehta et al., 2013; Coffill et al., 2016). Поскольку у миног описано 6 кластеров Hox-генов, вероятно, дупликации, приведшие к их появлению, не затронули ParaHox гены, свидетельствуя о локальности этих дупликаций. Альтернативным сценарием для ParaHox генов является общая c Hox’ами дупликация с последующей утратой дуплицировавшихся кластеров и редукцией их количества до двух (Zhang et al., 2017). В поддержку такого сценария выступает обнаруженние у тихоокеанской миноги (L. camtshchaticum) гомологов генов, соседствующих с ParaHox кластерами у челюстноротых. Гены Pdgfra и Kit связаны с ParaHox B-кластером, а Pdgfrb и Csf1r – c C-кластером. И если ген Kit у миног связан с b-кластером ParaHox генов, то Csf1r расположен отдельно, в участке генома, не несущем ParaHox гены. Два гена Pdgfr миног также не соседствуют с ParaHox кластерами. Присутствие в геноме миног четырех генов-соседей может свидетельствовать о том, что в прошлом ParaHox кластеров также было больше, но часть из них была утрачена (Zhang et al., 2017). Это допущение позволяет соотнести имеющиеся у современных миног два кластера ParaHox генов с гипотезой о двух раундах полногеномных дупликаций у предков позвоночных.
ДУПЛИКАЦИИ У РЫБ – ОТ ЧАСТНОГО К ОБЩЕМУ
Кроме описанных выше ПГД на ранних этапах эволюции позвоночных, внесших вклад в формирование генома всех представителей этого подтипа, свидетельства более поздних дупликаций были обнаружены и в отдельных эволюционных линиях позвоночных. Очевидно, что исследование особенностей и значения таких дупликаций по сравнительно “горячим следам”, может дать уникальную информацию о базовых механизмах и вкладе полногеномных дупликаций в эволюционную историю живых организмов в целом.
Наиболее описанными такими событиями на сегодяшний день являются дупликации у осетровых рыб, дупликации у предка костистых рыб, а также дополнительные раунды дупликаций у отдельных представителей этого класса – лососевых и карповых рыб (Brunet et al., 2006; Glasauer, Neuhauss, 2014; Du et al., 2020).
Осетровые рыбы, хрящевые ганоиды, отделились от общего эволюционного ствола позвоночных около 345 млн лет назад, вскоре после расхождения линий лучеперых и двоякодышащих рыб. Осетровых отличает низкая скорость эволюционных изменений, а раннее развитие икры внешне больше похоже на развитие икры амфибий, чем рыб, что свидетельствует об их архаичности. Особенностью кариотипа осетровых является наблюдаемая у них полиплоидия, с числом хромосом от 120 до 360–380 у разных видов (Havelka et al., 2013). Такая склонность к полиплоидизации может объясняться, хотя бы отчасти, тем, что анализ генома не выявил у представителей осетровых (стерляди) выраженных половых хромосом, наличие которых, как указывалось ранее, может служить ограничителем дупликационных изменений (Du et al., 2020). Исследователи склоняются к тому, что полиплоидия осетров носит автоплоидный характер. При этом, неожиданным оказался очень высокий процент сохранения генов после дупликаций – в среднем около 70%, а для отдельных групп генов, таких как гены глутаматных рецепторов – до 88.5%. Восемь кластеров Hox-генов у стерляди насчитывают 88 генов. При этом отмечается, что, в отличие от костистых рыб, утрата генов у осетров как правило происходит вследствии утраты крупных участков генома – плеча хромосомы или даже хромосомы целиком без заметных перестроек в геноме, вследствие чего хромосомы осетров в основном имеют дискретно диплоидный или тетраплоидный статус.
Первые гипотезы о дополнительной дупликации у костистых рыб были выдвинуты для объяснения обнаружения у представителей этой группы двух ортологов многих генов наземных позвоночных, хотя масштабы этой дупликации не были очевидны изначально (Wittbrodt et al., 1998; Taylor et al., 2001). У рыбы Danio rerio, а впоследствии и у эволюционно наиболее ранних представителей групп Elopomorpha и Osteoglossomorpha, были описаны 7 кластеров Hox-генов (Amores et al., 1998; Prince et al., 1998; Chambers et al., 2009; Guo et al., 2009; Henkel et al., 2012). Открытие двойного набора Hox-генов у ранних представителей костистых рыб стало свидетельством дупликации у предка группы, подтвержденного впоследствии результатами полногеномного секвенирования многих ее представителей (Glasauer, Neuhauss, 2014).
Дополнительные дупликации геномов происходили позже и у отдельных представителей костистых рыб. На сегодняшний день описаны дупликации у предков лососевых и карповых рыб (Alexandrou et al., 2013; Zhang et al., 2013) и, не исключено, что это число будет расти.
Однозначно судить о вкладе полногеномной дупликации в развитие костистых рыб сложно, но можно отметить, что эта группа, представители которой освоили самые разнообразные места обитания и экологические ниши, на сегодняшний день включает в себя примерно половину всех описанных видов позвоночных (около 32 000 из 64 000–68 000) (Glasauer, Neuhauss, 2014; Ravi et al., 2018). Такая статистика, вкупе с общим разнообразием позвоночных позволяет отметить корреляцию предковых геномных дупликаций с последующим морфологическим разнообразием и эволюционными успехами группы, хотя механизмы влияния дупликаций на эти процессы еще только предстоит изучить (Ravi et al., 2018).
ЗАКЛЮЧЕНИЕ
Подытоживая рассмотренные в обзоре модели и примеры, можно отметить, что, несмотря на все ограничения и сложности однозначного решения вопроса о количестве и времени раундов полногеномных дупликаций на ранних этапах эволюции позвоночных, и неизбежных допущениях принимаемых в каждом из рассматриваемых сценариев, “классическая” модель, предполагающая два раунда полногеномных дупликаций у общего предка всей группы не теряет своей актуальности и выглядит убедительно во многих случаях.
Беспрецедентные прорывы последних лет в повышении доступности методов высокопроизводительного секвенирования и обработки больших объемов получаемой информации, в сочетании с возрастающим интересом исследователей к не вполне традиционным, но крайне важным с филогенетической точки зрения модельным объектам, позволяют уже в ближайшей перспективе рассчитывать на существенное сокращение числа “белых пятен” в нашем понимании фундаментальных основ эволюционной истории позвоночных.
Список литературы
Adachi N., Robinson M., Goolsbee A. et al. Regulatory evolution of Tbx5 and the origin of paired appendages // Proc. Natl. Acad. Sci. USA. 2016. V. 113(36). P. 10115–10120.
Albertin C.B., Simakov O., Mitros T. et al. The octopus genome and the evolution of cephalopod neural and morphological novelties // Nature. 2015. V. 524. P. 220–224.
Alexandrou M.A., Swartz B.A., Matzke N.J. et al. Genome duplication and multiple evolutionary origins of complex migratory behavior in Salmonidae // Mol. Phylogenet. Evol. 2013. V. 69. P. 514–523.
Amores A., Force A., Yan Y.L. et al. Zebrafish hox clusters and vertebrate genome evolution // Sciense. 1998. 282:1711–1714.
Andersen K.G., Nissen J.K., Betz A.G. Comparative genomics reveals key gain-of-function events in Foxp3 during regulatory T cell evolution // Front. Immunol. 2012. V. 3. P. 113.
Bassham S., Canestro C., Postlethwait J.H. Evolution of developmental roles of Pax2/5/8 paralogs after independent duplication in urochordate and vertebrate lineages // BMC Biol. 2008. V. 6(1). P. 35.
Bayramov A.V., Eroshkin F.M., Martynova N.Y. et al. Novel functions of Noggin proteins: inhibition of Activin/Nodal and Wnt signaling // Development. 2011. V. 138. P. 5345–5356.
Blomme T., Vandepoele K., de Bodt S. et al. The gain and loss of genes during 600 million years of vertebrate evolution // Genome Biol. 2006. V. 7. P. R43.
Braasch I., Salzburger W., Meyer A. Asymmetric evolution in two fish-specifically duplicated receptor tyrosine kinase paralogons involved in teleost coloration // Mol. Biol. Evol. 2006. V. 23. P. 1192–1202.
Braasch I., Gehrke A.R., Smith J.J. et al. The spotted gar genome illuminates vertebrate evolution and facilitates human-teleost comparisons // Nature Genet. 2015. V. 47. P. 427–437.
Brazeau M.D., Friedman M. The origin and early phylogenetic history of jawed vertebrates // Nature. 2015. V. 520. P. 490–497.
Brunet F.G., Roest Crollius H., Paris M. et al. Gene loss and evolutionary rates following whole-genome duplication in teleost fishes // Mol. Biol. Evol. 2006. V. 23. P. 1808–1816.
Campanini E.B., Vandewege M.W., Pillai N.E. et al. Early evolution of vertebrate Mybs: an integrative perspective combining synteny, phylogenetic, and gene expression analyses // Genome Biol. Evol. 2015. V. 7(11). P. 3009–3021.
Cardoso J.C.R., Bergqvist C.A., Larhammar D. Corticotropin-releasing hormone (CRH) gene family duplications in lampreys correlate with two early vertebrate genome doublings // Front. Neurosci. 2020. V. 14. P. 672.
Cardoso J.C.R., Bergqvist C.A., Félix R.C. et al. Corticotropin-releasing hormone family evolution: five ancestral genes remain in some lineages // J. Mol. Endocrinol. 2016. V. 57. P. 73–86.
Carroll S.B. Evo-devo and an expanding evolutionary synthesis: a genetic theory of morphological evolution // Cell. 2008. V. 134(1). P. 25–36.
Carroll S.B. Homeotic genes and the evolution of arthropods and chordates // Nature. 1995. V. 376. P. 479–485.
Chambers K.E., McDaniell R., Raincrow J.D. et al. Hox cluster duplication in the basal teleost Hiodon alosoides (Osteoglossomorpha) // Theory Biosci. 2009. V. 128. P. 109–120.
Christensen K.A., Davidson W.S. Autopolyploidy genome duplication preserves other ancient genome duplications in Atlantic salmon (Salmo salar) // PLoS One. 2017. V. 12(2). P. e0173053.
Ciomborowska J., Rosikiewicz W., Szklarczyk D. et al. ‘Orphan’ retrogenes in the human genome // Mol. Biol. Evol. 2013. V. 30. P. 384–396.
Coates M. The evolution of paired fins // Theory Biosci. 2003. V. 122(2–3). P. 266–287.
Coffill C.R., Lee A.P., Siau J.W. et al. The p53-Mdm2 interaction and the E3 ligase activity of Mdm2/Mdm4 are conserved from lampreys to humans // Genes Dev. 2016. V. 30(3). P. 281–292.
Conant G.C., Wolfe K.H. Increased glycolytic flux as an outcome of whole-genome duplication in yeast // Mol. Syst. Biol. 2007. V. 3. P. 129.
Dahn R.D., Davis M.C., Pappano W.N. et al. Sonic hedgehog function in chondrichthyan fins and the evolution of appendage patterning // Nature. 2007. V. 445. P. 311–314.
Dale L., Slack J.M.W. Regional specification within the mesoderm of early embryos of Xenopus laevis // Deve-lopment. 1987. V. 100. P. 279–295.
Davis C.A., Holmyard D.P., Millen K.J. et al. Examining pattern formation in mouse, chicken and frog embryos with an En-specific antiserum // Development. 1991. V. 111(2). P. 287–298.
Dehal P., Boore J.L. Two rounds of whole genome duplication in the ancestral vertebrate // PLoS Biol. 2005. V. 3(10). P. e314.
DesMarais D.L., Rausher M.D. Escape from adaptive conflict after duplication in an anthocyanin pathway gene // Nature. 2008. V. 454. P. 762–765.
Donoghue P.C., Keating J.N. Early vertebrate evolution // Palaeontology. 2014. V. 57. P. 879–893.
Donoghue P.C.J., Purnell M.A. Genome duplication, extinction and vertebrate evolution // Trends Ecol. Evol. 2005. V. 20. P. 312–319.
Douard V., Brunet F., Boussau B. et al. The fate of the duplicated androgen receptor in fishes: a late neofunctionalization event? // BMC Evol. Biol. 2008. V. 8. P. 336.
Du K., Stöck M., Kneitz S. et al. The sterlet sturgeon genome sequence and the mechanisms of segmental rediploidization // Nat. Ecol. Evol. 2020. V. 4(6). P. 841–852.
Duboule D. The rise and fall of Hox gene clusters // Development. 2007. V. 134. P. 2549–2560.
Duboule D., Dolle P. The structural and functional organization of the murine HOX gene family resembles that of Drosophila homeotic genes // EMBO J. 1989. V. 8. P. 1497–1505.
Ermakova G.V., Kucheryavyy A.V., Zaraisky A.G. et al. Discovery of four Noggin genes in lampreys suggests two rounds of ancient genome duplication // Communication Biology. 2020. V. 3(1). P. 532.
Ermakova G.V., Kucheryavyy A.V., Zaraisky A.G. et al. Comparative analysis of expression patterns of genes of the Noggin family at early stages of development of the head structures of the European river lamprey Lampetra fluviatilis // Russ. J. Dev. Biol. 2021. V. 52(1). P. 1–10.
Eroshkin F.M., Ermakova G.V., Bayramov A.V. et al. Multiple noggins in vertebrate genome: cloning and expression of noggin2 and noggin4 in Xenopus laevis // Gene Expr. Patterns. 2006. V. 6. P. 180–186.
Faas L., Isaacs H.V. Overlapping functions of Cdx1, Cdx2, and Cdx4 in the development of the amphibian Xenopus tropicalis // Dev. Dyn. 2009. V. 238. P. 835–852.
Fletcher R.B., Watson A.L., Harland R.M. Expression of Xenopus tropicalis noggin1 and noggin2 in early development: two noggin genes in a tetrapod // Gene Expr. Patterns. 2004. V. 5. P. 225–230.
Flot J.-F., Hespeels B., Li X. et al. Genomic evidence for ameiotic evolution in the bdelloid rotifer Adineta vaga // Nature. 2013. V. 500. P. 453–457.
Force A., Lynch M., Pickett F.B. et al. Preservation of duplicate genes by complementary, degenerative mutations // Genetics. 1999. V. 151. P. 1531–1545.
Forstmeier W., Ellegren H. Trisomy and triploidy are sources of embryo mortality in the zebra finch // Proc. Biol. Sci. 2010. V. 277(1694). P. 2655–2660.
Freeling M. The evolutionary position of subfunctionalization, downgraded // Genome Dyn. 2008. V. 4. P. 25–40.
Furlong R.F., Holland P.W.H. Were vertebrates octoploid? // Phil. Trans. R. Soc. Lond. B Biol. Sci. 2002. V. 357. P. 531–544.
Futahashi R., Kawahara-Miki R., Kinoshita M. et al. Extraordinary diversity of visual opsin genes in dragonflies // Proc. Natl Acad. Sci. USA. 2015. V. 112. P. E1247–E1256.
Gans C., Northcutt R.G. Neural crest and the origin of vertebrates: a new head // Science. 1983. V. 220. P. 268–274.
Gee H. Across the Bridge: Understanding the Origin of the Vertebrates. Chicago: University of Chicago Press, 2018.
Gilbert S.F. Developmental Biology. Sunderland: Sinauer Associates, 2006. 9th ed.
Glasauer S.M., Neuhauss S.C. Whole-genome duplication in teleost fishes and its evolutionary consequences // Mol. Genet. Genomics. 2014. V. 289(6). P. 1045–1060.
Goode D.K., Elgar G. The PAX258 gene subfamily: a comparative perspective // Dev Dyn. 2009. V. 238(12). P. 2951–2974.
Graham A., Papalopulu N., Krumlauf R. The murine and Drosophila homeobox gene complexes have common features of organization and expression // Cell. 1989. V. 57. P. 367–378.
Green S.A., Bronner M.E. The lamprey: A jawless vertebrate model system for examining origin of the neural crest and other vertebrate traits // Differentiation. 2014. V. 87. P. 44–51.
Green S.A., Simoes-Costa M., Bronner M. Evolution of vertebrates as viewed from the crest // Nature. 2015. V. 520. P. 474–482.
Guerreiro I., Nunes A., Woltering J.M. et al. Role of a polymorphism in a Hox/Pax-responsive enhancer in the evolution of the vertebrate spine // Proc. Natl. Acad. Sci. USA. 2013. V. 110(26). P. 10682–10686.
Guo B., Gan X., He S. Hox genes of the Japanese eel Anguilla japonica and Hox cluster evolution in teleosts // J. Exp. Zool. 2009. V. 15. 314(2). P. 135–147.
Gutierrez-Mazariegos J., Kumar E., Studer R.A. et al. Evolutionary diversification of retinoic acid receptor ligandbinding pocket structure by molecular tinkering // R. Soc. Open Sci. 2016. V. 3(3). P. 150484.
Hall B.K. The Neural Crest and Neural Crest Cells in Vertebrate Development and Evolution // N.Y.: Springer, 2008.
Havelka M., Hulák M., Bailie D. et al. Extensive genome duplications in sturgeons: new evidence from microsatellite data // J. Appl. Ichthyol. 2013. V. 29. P. 704–708.
Heimberg A.M., Cowper-Sal-Lari R., Sémon M. et al. microRNAs reveal the interrelationships of hagfish, lampreys, and gnathostomes and the nature of the ancestral vertebrate // Proc. Natl. Acad. Sci. USA. 2010. V. 107. P. 19379–19383.
Henkel C.V., Burgerhout E., de Wijze D.L. et al. Primitive duplicate hox clusters in the European eel’s genome // PLoS One. 2012. V. 7. P. e32231.
Holland L.Z., Ocampo Daza D. A new look at an old question: when did the second whole genome duplication occur in vertebrate evolution? // Genome Biol. 2018. V. 19(1). P. 209.
Holland L.Z. Evolution of new characters after whole genome duplications: insights from amphioxus // Semin. Cell. Dev. Biol. 2013. V. 24(2). P. 101–109.
Holland P. Gene duplication: past, present and future // Semin. Cell. Biol. 1999. V. 10. P. 541–547.
Hufton A.L., Mathia S., Braun H. et al. Deeply conserved chordate noncoding sequences preserve genome synteny but do not drive gene duplicate retention // Genome Res. 2009. V. 19. P. 2036–2051.
Hughes A.L. The evolution of functionally novel proteins after gene duplication // Proc. Biol. Sci. 1994. V. 256. P. 119–124.
Innan H., Kondrashov F. The evolution of gene duplications: classifying and distinguishing between models // Nat. Rev. Genet. 2010. V. 11(2). P. 97–108.
Jaillon O., Aury J.M., Brunet F. et al. Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype // Nature. 2004. V. 431(7011). P. 946–957.
Janvier P. Facts and fancies about early fossil chordates and vertebrates // Nature. 2015. V. 520(7548). P. 483–489.
Janvier P. The dawn of the vertebrates: characters versus common ascent in the rise of current vertebrate phylogenies // Palaeontology. 1996. V. 39. P. 259–287.
Janvier P. Vertebrate characters and the Cambrian vertebrates // C.R. Palevol. 2003. V. 2. P. 523–531.
Joyner A.L., Martin G.R. En-1 and En-2, two mouse genes with sequence homology to the Drosophila engrailed gene: expression during embryogenesis // Genes Dev. 1987. V. 1(1). P. 29–38.
Kassahn K.S., Dang V.T., Wilkins S.J. et al. Evolution of gene function and regulatory control after wholegenome duplication: comparative analyses in vertebrates // Genome Res. 2009. V. 19. P. 1404–1418.
Kenny N.J., Chan KW., Nong W. et al. Ancestral whole-genome duplication in the marine chelicerate horseshoe crabs // Heredity (Edinb). 2016. V. 116(2). P. 190–199.
Kim C.B., Amemiya C., Bailey W. et al. Hox cluster genomics in the horn shark, Heterodontus francisci // Proc. Natl. Acad. Sci. USA. 2000. V. 97. P. 1655–1660.
Kimura M., King J.L. Fixation of a deleterious allele at one of two “duplicate” loci by mutation pressure and random drift // Proc. Natl Acad. Sci. USA. 1979. V. 76. P. 2858–2861.
Klimova L., Kozmik Z. Stage-dependent requirement of neuroretinal Pax6 for lens and retina development // Development. 2014. V. 141. P. 1292–1302.
Kmita M., Duboule D. Organizing axes in time and space; 25 years of collinear tinkering // Science. 2003. V. 301. P. 331–333.
Kondrashov F.A., Koonin E.V. A common framework for understanding the origin of genetic dominance and evolutionary fates of gene duplication // Trends Genet. 2004. V. 20. P. 287–291.
Kondrashov F.A., Rogozin I.B., Wolf Y.I. et al. Selection in the evolution of gene duplications // Genome Biol. 2002. V. 3. research0008.1–0008.9.
Kozmik Z., Holland N.D., Kalousova A. et al. Characterization of an amphioxus paired box gene, AmphiPax2/5/8: developmental expression patterns in optic support cells, nephridium, thyroid-like structures and pharyngeal gill slits, but not in the midbrain-hindbrain boundary region // Development. 1999. V. 126. P. 1295–1304.
Krumlauf R. Hox genes in vertebrate development // Cell. 1994. V. 78. P. 191–201.
Kuraku S., Meyer A., Kuratani S. Timing of genome duplications relative to the origin of the vertebrates: did cyclostomes diverge before or after? // Mol. Biol. Evol. 2009. V. 26(1). P. 47–59.
Kuraku S., Kuratani S. Time scale for cyclostome evolution inferred with a phylogenetic diagnosis of hagfish and lamprey cDNA sequences // Zool. Sci. 2006. V. 23. P. 1053–1064.
Kuraku S., Meyer A. The evolution and maintenance of Hox gene clusters in vertebrates and the teleost-specific genome duplication // Int. J. Dev. Biol. 2009. V. 53. P. 765–773.
Kuratani S. Evolution of the vertebrate jaw: comparative embryology and molecular developmental biology reveal the factors behind evolutionary novelty // J. Anat. 2004. V. 205. P. 335–347.
Kuratani S., Ota K.G. Hagfish (Cyclostomata, Vertebrata): searching for the ancestral developmental plan of vertebrates // BioEssays. 2008. V. 30. P. 167–172.
Lagman D., Ocampo Daza D., Widmark J. et al. The vertebrate ancestral repertoire of visual opsins, transducin alpha subunits and oxytocin/vasopressin receptors was established by duplication of their shared genomic region in the two rounds of early vertebrate genome duplications // BMC Evol Biol. 2013. V. 13(1). P. 238.
Lamb T.M., Knecht A.K., Smith W.C. et al. Neural induction by secreted polypeptide noggin // Science. 1993. V. 262. P. 713–718.
Li W.H. Rate of gene silencing at duplicate loci: a theoretical study and interpretation of data from tetraploid fishes // Genetics. 1980. V. 95. P. 237–258.
Lowe C.J., Clarke D.N., Medeiros D.M. et al. The deuterostome context of chordate origins // Nature. 2015. V. 520. P. 456–465.
Lynch M., Force A. The probability of duplicate gene preservation by subfunctionalization // Genetics. 2000. V. 154. P. 459–473.
Lynch M., O’Hely M., Walsh B. et al. The probability of preservation of a newly arisen gene duplicate // Genetics. 2001. V. 159. P. 1789–1804.
Mable B. “Why polyploidy is rarer in animals than in plants”: myths and mechanisms // Biol. J. Linn. Soc. 2004. V. 82. P. 453–466.
Macqueen D.J., Johnston I.A. A well-constrained estimate for the timing of the salmonid whole genome duplication reveals major decoupling from species diversification // Proc. Biol. Sci. 2014. V. 281(1778). P. 20132881.
Mallo M., Wellik D.M., Deschamps J. Hox genes and regional patterning of the vertebrate body plan // Dev. Biol. 2010. V. 344. P. 7–15.
Manousaki T., Feiner N., Begemann G. et al. Co-orthology of Pax4 and Pax6 to the fly eyeless gene: molecular phylogenetic, comparative genomic, and embryological analyses // Evol. Dev. 2011. V. 13. P. 448–459.
Manousaki T., Qiu H., Noro M. et al. Molecular evolution in the lamprey genomes and its relevance to the timing of whole genome duplications // Jawless Fishes of the World. Vol. 1. Newcastle upon Tyne / Eds. Orlov A., Beamish R. Cambridge Scholars Publishing, 2011. P. 2–16.
Martin K.J., Holland P.W. Enigmatic orthology relationships between Hox clusters of the African butterfly fish and other teleosts following ancient whole-genome duplication // Mol. Biol. Evol. 2014. V. 31(10). P. 2592–2611.
Matsushita T., Yamaoka T., Otsuka S. et al. Molecular cloning of mouse paired-box-containing gene (Pax)-4 from an islet b cell line and deduced sequence of human Pax-4 // Biochem. Biophys. Res. Commun. 1998. V. 242. P. 176–180.
McCauley D.W., Bronner-Fraser M. Conservation of Pax gene expression in ectodermal placodes of the lamprey // Gene. 2002. V. 287(1–2). P. 129–139.
McCauley D.W., Docker M.F., Whyard S. et al. Lampreys as diverse model organisms in the genomics era // Bioscience. 2015. V. 65. P. 1046–1056.
McEwen G.K., Goode D.K., Parker H.J. et al. Early evolution of conserved regulatory sequences associated with development in vertebrates // PLoS Genet. 2009. V. 5. P. e1000762.
Mehta T.K., Ravi V., Yamasaki S. et al. Evidence for at least six Hox clusters in the Japanese lamprey (Lethenteron japonicum) // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 16044–16049.
Menke D.B., Guenther C., Kingsley D.M. Dual hindlimb control elements in the Tbx4 gene and region-specific control of bone size in vertebrate limbs // Development. 2008. V. 135(15). P. 2543–2553.
Meyer A., Van de Peer Y. From 2R to 3R: evidence for a fishspecific genome duplication (FSGD) // Bioessays. 2005. V. 27(9). P. 937–945.
Minguillon C., Nishimoto S., Wood S. et al. Hox genes regulate the onset of Tbx5 expression in the forelimb // Development. 2012. V. 139(17). P. 3180–3188.
Miyashita T. Fishing for jaws in early vertebrate evolution: a new hypothesis of mandibular confinement // Biol. Rev. 2016. V. 91. P. 611–657.
Moreau M., Leclerc C. The choice between epidermal and neural fate: a matter of calcium // Int. J. Dev. Biol. 2004. V. 48(2–3). P. 75–84.
Moriyama Y., Koshiba-Takeuchi K. Significance of whole-genome duplications on the emergence of evolutionary novelties // Briefings in Functional Genomics. 2018. V. 17(5). P. 329–338.
Morris S.C., Caron J.-B. A primitive fish from the Cambrian of North America // Nature. 2014. V. 512(7515). P. 419–422.
Muller G.B. Evo-devo: extending the evolutionary synthesis // Nat. Rev. Genet. 2007. V. 8. P. 943–949.
Murakami Y., Ogasawara M., Sugahara F. et al. Identification and expression of the lamprey Pax6 gene: evolutionary origin of the segmented brain of vertebrates // Development. 2001. V. 128(18). P. 3521–3531.
Myojin M., Ueki T., Sugahara F. et al. Isolation of Dlx and Emx gene cognates in an agnathan species, Lampetra japonica, and their expression patterns during embryonic and larval development: conserved and diversified regulatory patterns of homeobox genes in vertebrate head evolution // J. Exp. Zool. 2001. V. 15; 291(1). P. 68–84.
Nakatani Y., Takeda H., Kohara Y. et al. Reconstruction of the vertebrate ancestral genome reveals dynamic genome reorganization in early vertebrates // Genome Res. 2007. V. 17. P. 1254–1265.
Nei M. Gene duplication and nucleotide substitution in evolution // Nature. 1969. V. 221. P. 40–42.
Neidert A.H., Virupannavar V., Hooker G.W. et al. Lamprey Dlx genes and early vertebrate evolution // Proc. Natl. Acad. Sci. USA. 2001. V. 13; 98(4). P. 1665–1670.
Noro M., Sugahara F., Kuraku S. Reevaluating Emx gene phylogeny: homopolymeric amino acid tracts as a potential factor obscuring orthology signals in cyclostome genes // BMC Evol. Biol. 2015. V. 15(1). P. 78.
Northcutt R.G. The new head hypothesis revisited // J. Exp. Zool. 2005. V. 304B. P. 274–297.
Nossa C.W., Havlak P., Yue J.-X. et al. Joint assembly and genetic mapping of the Atlantic horseshoe crab genome reveals ancient whole genome duplication // Gigascience. 2014. V. 3. P. 708–721.
Oisi Y., Ota K.G., Kuraku S. Craniofacial development of hagfishes and the evolution of vertebrates // Nature. 2013. V. 493. P. 175–180.
Onimaru K., Kuraku S. Inference of the ancestral vertebrate phenotype through vestiges of the whole-genome duplications // Brief Funct. Genomics. 2018. V. 17(5). P. 352–361.
Orr H.A. ‘Why polyploidy is rarer in animals than in plants’ revisited // Am. Nat. 1990. V. 136. P. 759–770.
Osumi N., Shinohara H., Numayama-Tsuruta K. et al. Concise review: Pax6 transcription factor contributes to both embryonic and adult neurogenesis as a multifunctional regulator // Stem. Cells. 2008. V. 26. P. 1663–1672.
Ota K.G., Kuraku S., Kuratani S. Hagfish embryology with reference to the evolution of the neural crest // Nature. 2007. V. 446. P. 672–675.
Otto S.P. The evolutionary consequences of polyploidy // Cell. 2007. V. 131(3). P. 452–462.
Pan D., Zhang L. Burst of young retrogenes and independent retrogene formation in mammals // PLoS One. 2009. V. 4. P. e5040.
Parker H.J., Bronner M.E., Krumlauf R. An atlas of anterior hox gene expression in the embryonic sea lamprey head: Hox-code evolution in vertebrates // Dev. Biol. 2019a. V. 453(1). P. 19–33.
Parker H.J., De Kumar B., Green S.A. et al. A Hox-TALE regulatory circuit for neural crest patterning is conserved across vertebrates // Nat. Commun. 2019b. V. 10. P. 1189.
Parker H.J., Krumlauf R. Segmental arithmetic: summing up the Hox gene regulatory network for hindbrain development in chordates // Wiley Interdiscip Rev. Dev. Biol. 2017. V. 6(6). P. e286.
Parker H.J., Piccinelli P., Sauka-Spengler T. et al. Ancient Pbx- Hox signatures define hundreds of vertebrate developmental enhancers // BMC Genomics. 2011. V. 12. P. 637.
Parker H.J., Sauka-Spengler T., Bronner et al. A reporter assay in lamprey embryos reveals both functional conservation and elaboration of vertebrate enhancers // PLoS One. 2014. V. 9. P. e85492.
Pascual–Anaya J., D’Aniello S., Kuratani S. et al. Evolution of Hox gene clusters in deuterostomes// BMC Dev. Biol. 2013. V. 13. P. 26.
Pascual-Anaya J., Sato I., Sugahara F. et al. Hagfish and lamprey Hox genes reveal conservation of temporal colinearity in vertebrates // Nat. Ecol. Evol. 2018. V. 2. P. 859–866.
Pasquier J., Braasch I., Batzel P. et al. Evolution of gene expression after whole-genome duplication: new insights from the spotted gar genome // J. Exp. Zool. Part B Mol. Dev. Evol. 2017. V. 328. P. 709–721.
Piovesan A., Antonaros F., Vitale L. et al. Human protein-coding genes and gene feature statistics // BMC Res. Notes. 2019. V. 12. P. 315.
Plouhinec J.L., Sauka-Spengler T., Germot A. et al. The mammalian Crx genes are highly divergent representatives of the Otx5 gene family, a gnathostome orthology class of orthodenticle-related homeogenes involved in the differentiation of retinal photoreceptors and circadian entrainment // Mol. Biol. Evol. 2003. V. 20. P. 513–521.
Pollard S.L., Holland P.W. Evidence for 14 homeobox gene clusters in human genome ancestry // Curr. Biol. 2000. V. 10(17). P. 1059–1062.
Postlethwait J., Amores A., Cresko W. et al. Subfunction partitioning, the teleost radiation and the annotation of the human genome // Trends Genet. 2004. V. 20(10). P. 481–490.
Postlethwait J.H., Woods I.G., Ngo-Hazelett P. et al. Zebra-fish comparative genomics and the origins of vertebrate chromosomes // Genome Res. 2000. V. 10(12). P. 1890–1902.
Prescott S.L., Srinivasan R., Marchetto M.C. et al. Enhancer divergence and cis-regulatory evolution in the human and chimp neural crest // Cell. 2015. V. 163. P. 68–83.
Prince Y., Joly L., Ekker M. et al. Zebrafish hox genes: genomic organization and modified colinear expression patterns in the trunk // Development. 1998. V. 125. P. 407–420.
Putnam N.H., Butts T., Ferrier D.E. et al. The amphioxus genome and the evolution of the chordate karyotype // Nature. 2008. V. 453. P. 1064–1071.
Qiu H., Hildebrand F., Kuraku S. et al. Unresolved orthology and peculiar coding sequence properties of lamprey genes: the KCNA gene family as test case // BMC Genomics. 2011. V. 12(1). P. 325.
Quiring R., Walldorf U., Kloter U. et al. Homology of the eyeless gene of Drosophila to the small eye gene in mice and Aniridia in humans // Science. 1994. V. 265. P. 785–789.
Ravi V., Bhatia S., Shingate P. et al. Lampreys, the jawless vertebrates, contain three Pax6 genes with distinct expression in eye, brain and pancreas // Sci. Rep. 2019. V. 9(1). P. 19559.
Ravi V., Venkatesh B. The divergent genomes of teleosts // Annu. Rev. Anim. Biosci. 2018. V. 6. P. 47–68.
Ravi V., Lam K., Tay B.H. et al. Elephant shark (Callorhinchus milii) provides insights into the evolution of Hox gene clusters in gnathostomes // Proc. Natl. Acad. Sci. USA. 2009. V. 106. P. 16327–16332.
Robertson H.M., Wanner K.W. The chemoreceptor superfamily in the honey bee, Apis mellifera: expansion of the odorant, but not gustatory, receptor family // Genome Res. 2006. V. 16. P. 1395–1403.
Ruvinsky I., Gibson-Brown J.J. Genetic and developmental bases of serial homology in vertebrate limb evolution // Development. 2000. V. 127(24). P. 5233–5244.
Sacerdot C., Louis A., Bon C. et al. Chromosome evolution at the origin of the ancestral vertebrate genome // Genome Biol. 2018. V. 19(1). P. 166.
Sahoo T., Dzidic N., Strecker M.N. et al. Comprehensive genetic analysis of pregnancy loss by chromosomal microarrays: outcomes, benefits, and challenges // Genet. Med. 2017. V. 19(1). P. 83–89.
Sánchez-Villagra M.R., Geiger M., Schneider R.A. The taming of the neural crest: a developmental perspective on the origins of morphological covariation in domesticated mammals // Royal Soc. Open Sci. 2016. V. 3. P. 160107.
Sansom R.S., Freedman K., Gabbott S.E. et al. Taphonomy and affinity of an enigmatic Silurian vertebrate, Jamoytius kerwoodi White // Palaeontology. 2010. V. 53(6). P. 1393–1409.
Sansom R.S., Gabbott S.E., Purnell M.A. Unusual anal fin in a Devonian jawless vertebrate reveals complex origins of paired appendages // Biol. Lett. 2013. V. 9. P. 20130002.
Santos M.E., Athanasiadis A., Leitao A.B. et al. Alternative splicing and gene duplication in the evolution of the FoxP gene subfamily // Mol. Biol. Evol. 2011. V. 28. P. 237–247.
Satoh N. Chordate Origins and Evolution: The Molecular Evolutionary Road to Vertebrates. San Diego: Academic Press. 2016.
Sauka-Spengler T., Bronner-Fraser M. Evolution of the neural crest viewed from a gene regulatory perspective // Genesis. 2008. V. 46. P. 673–682.
Scannell D.R., Butler G., Wolfe K.H. Yeast genome evolution – the origin of the species // Yeast. 2007. V. 24(11). P. 929–942.
Scannell D.R., Frank A.C., Conant G.C. et al. Independent sorting-out of thousands of duplicated gene pairs in two yeast species descended from a whole-genome duplication // Proc. Natl. Acad. Sci. USA. 2007. V. 104(20). P. 8397–8402.
Schwager E.E., Sharma P.P., Clarke T. et al. The house spider genome reveals an ancient whole-genome duplication during arachnid evolution // BMC Biol. 2017. V. 15(1). P. 62.
Sharman A.C. Some new terms for duplicated genes // Semin. Cell Dev. Biol. 1999. V. 10. P. 561–563.
Shimeld S.M., Donoghue P.C.J. Evolutionary crossroads in developmental biology: cyclostomes (lamprey and hagfish) // Development. 2012. V. 139. P. 2091–2099.
Shu D.-G., Luo H.-L., Conway Morris S. et al. Lower Cambrian vertebrates from south China // Nature. 1999. V. 402(6757). P. 42–46.
Shu D.-G., Morris S.C., Han J. et al. Head and backbone of the early Cambrian vertebrate Haikouichthys // Nature. 2003. V. 421(6922). P. 526–529.
Sikosek T., Chan H.S., Bornberg-Bauer E. Escape from adaptive conflict follows from weak functional trade-offs and mutational robustness // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. 14888–14893.
Simakov O., Marlétaz F., Yue J.X. et al. Deeply conserved synteny resolves early events in vertebrate evolution // Nat. Ecol. Evol. 2020. V. 4(6). P. 820–830.
Slack J.M., Tannahill D. Noggin the dorsalizer // Nature. 1993. V. 361. P. 498–499.
Smith J.J., Keinath M.C. The sea lamprey meiotic map improves resolution of ancient vertebrate genome duplications // Genome Res. 2015. V. 25. P. 1081–1090.
Smith J.J., Kuraku S., Holt C. et al. Sequencing of the sea lamprey (Petromyzon marinus) genome provides insights into vertebrate evolution // Nat. Genet. 2013. V. 45. P. 415–421.
Smith W.C., Knecht A.K., Wu M. et al. Secreted noggin protein mimics the Spemann organizer in dorsalizing Xenopus mesoderm // Nature. 1993. V. 361. P. 547–549.
Smith J.J., Timoshevskaya N., Ye C. et al. The sea lamprey germline genome provides insights into programmed genome rearrangement and vertebrate evolution // Nat. Genet. 2018. V. 50. P. 270–277.
Smith W.C., Harland R.M. Expression cloning of noggin, a new dorsalizing factor localized to the Spemann orginizer in Xenopus embryos // Cell. 1992. V. 70. P. 829–840.
Soltis D.E., Albert V.A., Leebens-Mack J. et al. Polyploidy and angiosperm diversification // Am. J. Bot. 2009. V. 96(1). P. 336–348.
Soltis D.E., Bell C.D., Kim S. et al. Origin and early evolution of angiosperms // Ann. N.Y. Acad. Sci. 2008. V. 1133. P. 3–25.
Song X., Tang Y., Wang Y. Genesis of the vertebrate FoxP subfamily member genes occurred during two ancestral whole genome duplication events // Gene. 2016. V. 588(2). P. 156–162.
Song X., Wang Y., Tang Y. Rapid diversification of FoxP2 in teleosts through gene duplication in the teleost-specific whole genome duplication event // PLoS One. 2013. V. 8. P. e83858.
Spring J. Vertebrate evolution by interspecific hybridization – are we polyploid? // FEBS Lett. 1997. V. 400. P. 2–8.
Stoltzfus A. On the possibility of constructive neutral evolution // J. Mol. Evol. 1999. V. 49. P. 169–181.
Sugino R.P., Innan H. Selection for more of the same product as a force to enhance concerted evolution of duplicated genes // Trends Genet. 2006. V. 22. P. 642–644.
Takahashi H., Takahashi K., Liu F.C. FOXP genes, neural development, speech and language disorders // Adv. Exp. Med. Biol. 2009. V. 665. P. 117–129.
Tang H., Wang X., Bowers J.E. et al. Unraveling ancient hexaploidy through multiply-aligned angiosperm gene maps // Genome Res. 2008. V. 18(12). P. 1944–1954.
Tank E.M., Dekker R.G., Beauchamp K. et al. Patterns and consequences of vertebrate Emx gene duplications // Evol. Dev. 2009. V. 11(4). P. 343–353.
Taylor J.S., Van de Peer Y., Braasch I. et al. Comparative genomics provides evidence for an ancient genome duplication event in fish // Philos Trans R. Soc. Lond B Biol. Sci. 2001. V. 356. P. 1661–1679.
Valerius M.T., Li H., Stock J.L. et al. Gsh-1: a novel murine homeobox gene expressed in the central nervous system // Dev. Dyn. 1995. V. 203(3). P. 337–351.
Van de Peer Y., Maere S., Meyer A. The evolutionary significance of ancient genome duplications // Nat. Rev. Genet. 2009. V. 10(10). P. 725–732.
Van de Peer Y., Mizrachi E., Marchal K. The evolutionary significance of polyploidy // Nat. Rev. Genet. 2017. V. 18. P. 411–424.
Vanneste K., Baele G., Maere S. et al. Analysis of 41 plant genomes supports a wave of successful genome duplications in association with the Cretaceous-Paleogene boundary // Genome Res. 2014. V. 24(8). P. 1334–1347.
Veitia R.A. Gene dosage balance: deletions, duplications and dominance // Trends Genet. 2005. V. 21. P. 33–35.
Vuolo F., Mentink R.A., Hajheidari M. et al. Coupled enhancer and coding sequence evolution of a homeobox gene shaped leaf diversity // Genes Dev. 2016. V. 30. P. 2370–2375.
Wagner A. The molecular origins of evolutionary innovations // Trends Genet. 2011. V. 27(10). P. 397–410.
Wilkins A.S., Wrangham R.W., Fitch W.T. The “domestication syndrome” in mammals: a unified explanation based on neural crest cell behavior and genetics // Genetics. 2014. V. 197. P. 795–808.
Wittbrodt J., Meyer A., Schartl M. More genes in fish? // Bioessays. 1998. V. 20. P. 511–515.
Woolfe A., Elgar G. Comparative genomics using Fugu reveals insights into regulatory subfunctionalization // Genome Biol. 2007. V. 8(4). P. R53.
Xanthos J.B., Kofron M., Tao Q. et al. The roles of three signaling pathways in the formation and function of the Spemann Organizer // Development. 2002. V. 129(17). P. 4027–4043.
Yang M., Wang Y., Wang X. et al. Characterization of grass carp (Ctenopharyngodon idellus) Foxp1a/1b/2: evidence for their involvement in the activation of peripheral blood lymphocyte subpopulations // Fish Shellfish Immunol. 2010. V. 28. P. 289–295.
York J.R., McCauley D.W. Functional genetic analysis in a jawless vertebrate, the sea lamprey: insights into the developmental evolution of early vertebrates // J. Exp. Biol. 2020. V. 223(Pt Suppl. 1). P. jeb206433.
Zhang G., Fang X., Guo X. et al. The oyster genome reveals stress adaptation and complexity of shell formation // Nature. 2012. V. 490. P. 49–54.
Zhang H., Ravi V., Tay B.H. et al. Lampreys, the jawless vertebrates, contain only twoParaHox gene clusters // Proc. Natl. Acad. Sci. USA. 2017. V. 114(34). P. 9146–9151.
Zhang X., Zhang Y., Zheng X. et al. A consensus linkage map provides insights on genome character and evolution in common carp (Cyprinus carpio L.) // Mar. Biotechnol. 2013. V. 15. P. 275–312.
Zhang X.G., Hou X.G. Evidence for a single median fin-fold and tail in the Lower Cambrian vertebrate, Haikouichthys ercaicunensis // J. Evol. Biol. 2004. V. 17. P. 1162–1166.
Дополнительные материалы отсутствуют.



