Онтогенез, 2021, T. 52, № 3, стр. 220-232
Механизм участия MSX1 и PAX9 в развитии зубов. Результаты анализа взвешенных сетей ко-экспрессии генов
Ф. Ванг a, В. Джианг c, d, Б. Чен e, f, g, *, Р. Ли b, c, d, **
a Кафедра стоматологии
200237 Шанхай, госпиталь Дахуа, КНР
b Кафедра онкологии полости рта, челюстнолицевой области головы и шеи,
медицинский факультет ДжиаоТонг университета Шанхая
200011 Шанхай, КНР
c Национальный клинический исследовательский центр заболеваний полости рта
200011 Шанхай, КНР
d Шанхайская центральная лаборатория стоматологии и шанхайский
институт стоматологии
200011 Шанхай, КНР
e Кафедра отоларингологии и хирургии лица и головы медицинский
факультет ДжиаоТонг университета Шанхая
200011 Шанхай, КНР
f Институт уха, ДжиаоТонг университет Шанхая
200011 Шанхай, КНР
g Шанхайская центральная лаборатория трансляционной медицины
заболеваний уха и носа
200011 Шанхай, КНР
* E-mail: liaochengzhuikao@163.com
** E-mail: chbent@163.com
Поступила в редакцию 04.11.2020
После доработки 26.01.2021
Принята к публикации 29.01.2021
Аннотация
Изучен механизм участия генов Msx1 и Pax9 в развитии зубов у мыши. Данные микрочипов GSE32321, которые содержали образцы орального эпителия (Epi) и зубных мезенхимальных клеток (Mes) мышей, экспрессирующих Msx1 и Pax9 и нокаутных по данным генам, были использованы для идентификации дифференциально экспрессируемых генов (DEGs). Затем наиболее значимые генные модули были исследованы в группах Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax с помощью анализа взвешенных сетей ко-экспрессии генов (WGCNA). Анализ базы данных Gene Ontology (GO) и скрининг генов-центральных узлов регуляторной сети были выполнены на модулях с наибольшей релевантностью. Всего выявлено 1467, 986, 1212, и 1293 DEGs соответственно в группах Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax. Были идентифицированы четыре ассоциированных генных модуля. Применение метода анализа обогащения по функциональной принадлежности (база данных GO) позволило показать, что эти гены оказывают негативное регуляторное воздействие на пролиферацию клеток, клеточную адгезию, развитие кровеносных сосудов и морфогенез кровеносных сосудов. В качестве основных генов определены IDH3A, SSPN, F13A1 и CBLN1, уровень экспрессии которых варьировал в разные моменты времени. Более того, показано, что IDH3A и CBLN1 вовлечены в окислительно-восстановительные метаболические процессы с участием коэнзимов, а также в межклеточную адгезию. Мы предполагаем, что Msx1 и Pax9 являются ключевыми, но не единственными регуляторами процесса формирования зубов у мышей. В этот процесс также могут быть вовлечены гены IDH3A, SSPN, F13A1 и CBLN1.
ВВЕДЕНИЕ
Развитие зубов, начиная с пролиферации и дифференцировки клеток и до формирования зуба на макроморфологическом уровне, регулируется серией точно скоординированных сигналов (Jheon et al., 2013; Li et al., 2013). Развитие зубов млекопитающих происходит в результате взаимодействия клеток эпителия эктодермы челюсти (Epi) и клеток одонтогенной мезенхимы (Mes), производных нервного гребня (Thesleff, 2003). Важную роль в регуляции морфогенеза и взаимного расположения зубов играют взаимодействия между эпителием зачатка зуба и клетками одонтогенной мезенхимы, формирование эктодермального эпителия челюсти, дифференциальная экспрессия генов в мезенхимальных клетках, а также локальные сигналы, регулирующие морфогенетические движения клеток зачатка зуба (Bei, 2009; Jussila, Thesleff, 2012). Исследования молекулярных механизмов развития дисплазии зубов у мышей подтвердили, что формирование зубов строго контролируется генетически. На сегодняшний день известно не менее 200 генов, которые детерминируют расположение, количество, размер и форму каждого зуба (Thesleff, 1998; Arte, 2001).
Мутантная форма Paired Box Gene 9 (Pax9) выявлена в локусе 14q12-13 генома, длина мРНК этого гена составляет 1630 п.о. Гомозиготные мыши с дефицитом Pax9 умирают вскоре после рождения (Peters et al., 1998). У таких мышей наблюдается полная потеря зубов и дефекты развития, такие как расщелина верхнего неба и деформация хрящевых структур. Эмбриональное развитие зубов при нокауте Pax9 оказывается заблокировано на стадии почки. Это указывает на то, что Pax9 необходим для развития зубов на более поздних стадиях, чем стадия почки (Peters et al., 1998). Отсутствие Msx1 приводило к краниофациальным дефектам развития, включая расщелину неба, нарушение развития резцов, недоразвитие альвеолярной кости нижней челюсти и окончание эмбрионального развития зубов на стадии почки (Satokata, Maas, 1994). На основании этих данных можно сделать вывод о том, что Pax9 и Msx1 играют важную роль в регуляции развитии зубов, однако механизм их действия неизвестен.
Метод анализа взвешенных сетей ко-экспрессии (Weighted gene co-expression network analysis, WGNCA) – это мощный метод системной биологии для анализа молекулярных механизмов регуляции развития и взаимодействий компонентов генетических регуляторных сетей (Langfelder, Horvath, 2008). Этот метод позволяет выделить модули ко-экспрессирующихся генов на основе матрицы попарных корреляций между экспрессией генов, а затем идентифицировать клинически значимые модули, выявить ключевые регуляторные гены в сигнальном пути и оценить внутримодульные связи и роли генов (Oldham et al., 2008). Благодаря использованию метода WGNCA, Канг и соавторы (Kang et al., 2016) успешно идентифицировали гены TMEM229A, LEPREL1, and GAD1 как узловые гены (hub-гены), продукты которых необходимы для онтогенетического перехода от молочных к постоянным зубам. Они сделали вывод о ключевой роли в регуляции этого процесса следующих модулей: кальциевого сигнального пути (calcium signaling pathway), взаимодействий рецепторных белков клетки с внеклеточным матриксом (ECM-receptor interactions) и нейроактивных взаимодействий лиганд-рецептор (neuroactive ligand-receptor interactions). Другой коллектив авторов (Jia et al., 2016), используя WGNCA, показали, что hsa-miR-376a-2, hsa-miR-376a-1, hsa-miR-15a и hsa-miR-424 могут быть ассоциированы с болезнью Альцгеймера. Использование метода WGCNA в комбинации с данными по генетическим маркерам позволило идентифицировать USIF и EADs3 как гены, участвующие в возникновении семейной комплексной гиперлипидемии (Plaisier et al., 2009).
Ранее О’Коннелл и соавторы (O’Connell et al., 2012) применили подход системной биологии для реконструкции развития зуба как сложного органа, состоящего из нескольких тканей. Авторы использовали данные, полученные с помощью микрочипов (GEO accession: GSE32321), анализ профиля экспрессии для генома в целом, данные из литературы, интегративный биоинформатический анализ и генетические модели in vivo. В результате были выявлены ключевые взаимодействия между эпителиальным и мезенхимальным компартментами формирующегося зуба, основанные на обратной связи между сигнальными путями Wnt и Bmp.
В настоящем исследовании мы использовали данные GSE32321 из базы данных Gene Expression Omnibus (GEO) и применили метод WGNCA для всестороннего изучения механизма взаимодействия Pax9 и Msx1 в процессе развития зубов. Цель нашего исследования была следующая: выявить, с использованием различных биоинформатических методов анализа, возможные механизмы взаимодействия Pax9 и Msx1 в развитии зубов мыши.
МАТЕРИАЛЫ И МЕТОДЫ
Получение данных
Данные микрочипов GSE32321 (O’Connell et al., 2012), были загружены из GEO (http://www.ncbi. nlm.nih.gov/geo/). Данные представляли собой 105 связанных образцов эпителия ротовой полости и дентальных мезенхимальных клеток мыши, собранных на E10.0, E11, E11.5, E12.0, E12.5, E13.0, E13.5, и E14.5 дни развития. Мы сконцентрировались на роли Msx1 и Pax9 в развитии зубов мыши на E13.5 день онтогенеза. Были проведены следующие сравнения: эпителий, Msx1 wildtype (n = 3) vs. knockout (n = 3); мезенхима, Msx1 wildtype (n = 3) vs. knockout (n = 3); эпителий Pax9 wildtype (n = 3) vs. knockout (n = 3); мезенхима, Pax9 wildtype (n = 3) vs. knockout (n = 3). Кроме того, образцы, собранные на других стадиях развития, использовались для проведения анализа динамики экспрессии ключевых генов (табл. 1).
Таблица 1.
Информационная таблица GSE32321
| Мезенхима | Эпителий | |
|---|---|---|
| E10 | 2 | 2 |
| E10_Bmp4 | 3 | – |
| E10_FGF8 | 3 | – |
| E10-Gsk3b | 3 | – |
| E10-SSH | 3 | – |
| E11 | 2 | 2 |
| E11.5 | 3 | 3 |
| E12 | 3 | 3 |
| E12.5 | 3 | 3 |
| E13 | 5 | 5 |
| E13_Bmp4 | – | 3 |
| E13_FGF8 | – | 3 |
| E13-Gsk3b | – | 3 |
| E13-SSH | – | 3 |
| E13.5 | 3 | 3 |
| E13.5_Msx1_null | 3 | 3 |
| E13.5_wildtype_Msx1_Epi_1 | 3 | 3 |
| E13.5_Pax9_null_Epi_1 | 3 | 3 |
| E13.5_wildtpye_Pax9_Epi1 | 3 | 3 |
| E14 | – | 2 |
Предварительная обработка данных
Файлы аннотаций зондов использовались для сопоставления зондов с соответствующими генами. Те зонды, для которых было выявлено более одного гена, были отброшены, но за начальное значение экспрессии гена, соответствующего нескольким зондам, было взято среднее значение по всем соответствующим зондам. Чтобы приблизить смещенное распределение данных к нормальному, данные были log2-трансформированы, а затем нормализованы методом среднеквадратичной нормализации (Troyanskaya et al., 2001; Fujita et al., 2006).
Скрининг дифференциально экспрессирующихся генов (DEGs)
Пакет limma (Gentleman et al., 2006) в R 3.1.3 был использован для определения дифференциально экспрессирующихся генов и соответствующих уровней значимости (p-value) в группах сравнения: эпителий, Msx1 wildtype vs. knockout; мезенхима, Msx1 wildtype vs. knockout; эпителий Pax9 wildtype vs. knockout; мезенхима, Pax9 wildtype vs. knockout. Метод Benjamini–Hochberg (BH) (Benjamini, Hochberg, 1995) использовался для оценки уровня ложноположительных результатов (false discovery rate, FDR) (Benjamini, 2010) и поправки на множественное тестирование.
Гены с FDR <0.05 и |lgFC| > 1 считались дифференциально экспрессирующимися.
С помощью R-пакета pheatmap для генов, дифференциально экспрессирующихся в группах Epi-Msx, Epi-Pax, Mes-Msx, и Mes-Pax, был проведен иерархический кластеринг (Szekely, Rizzo, 2005; Press et al., 2007) и построены тепловые карты на основе Евклидовых расстояний (Deza, Deza, 2009).
Построение сети ко-экспрессии и идентификация клинически значимых модулей
Пакет R программы WGCNA (https://labs.genetics. ucla.edu/horvath/CoexpressionNetwork/Rpackages/ WGCNA/) был использован для построения сети ко-экспрессии для дифференциально экспрессирующихся генов в 4 группах. Сначала была использована функция WGCNA pickSoftThreshold для вычисления мягкого порогового значения β с учетом критерия безмасштабности сети. Затем была построена взвешенная матрица смежности по формуле amn = |cmn|β (cmn = корреляция Пирсона для гена m и гена n; amn = значение функции смежности для пары генов m и n, параметр сходства). Затем матрица смежности была заменена на матрицу топологического сходства (TOM). Топологическое перекрытие проведено с учетом степени несходства. Для идентификации модулей генов использовался гибридный метод динамического обрезания дендрограммы. Минимальный размер для полученных дендрограмм составлял 30.
Идентификация клинически значимых модулей
Для того, чтобы определить, какие модули имеют отношение к развитию зубов, был использован T test для определения уровня значимости (P-value) для мРНК-кандидатов в группах Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax. Значимость для каждого гена (GS) определялась как lg10 от P‑value. В качестве значения модуля (MS) использовали среднее значение GS данного модуля. Как правило, модуль с самым высоким значением MS оценивался как связанный с заболеванием, а наиболее релевантный в отношении заболевания модуль в 4 группах был построен при помощи программы Cytoscape 2.8.2 (Smoot et al., 2010).
Анализ функционального обогащения
Гены, входящие в генетическую регуляторную сеть, были сопоставлены с функциональными узлами в GO, а для анализа биологических процессов использовался програмный пакет GOstat (Falcon and Gentleman, 2006). P-value < 0.05 был принят как пороговое значение.
Поиск узловых (hub) генов и горизонтальное сравнение значений экспрессии hub-генов
Степень связности узла – это количество межгенных связей, сходящихся в нем (Estrada, 2006). Узлы с более высокой степенью связности играют значительную роль в построенной сети (Jeong et al., 2001) и рассматриваются в качестве hub-генов. При подсчете степеней узлов были выбраны hub-гены из сетей Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax соответственно. Кроме того, значения экспрессии hub-генов выбирались и сравнивались в разных временных точках. Анализ обогащения GO для 10 ведущих hub-генов также проводился с использованием GOstat (Falcon, Gentleman, 2006) с P‑value < 0.05 в качестве порогового значения.
РЕЗУЛЬТАТЫ
Скрининг дифференциально экспрессирующихся генов
В результате скрининга в общей сложности 1467 дифференциально экспрессирующихся генов (DEGs) были обнаружены в группе Epi-Msx, 986 DEGs – в Epi-Pax, 1212 – в Mes-Msx, и 1293 – в Mes-Pax. Иерархическая кластеризация DEGs в четырех группах показана на рис. 1. Значения экспрессии DEGs значительно различались между контрольными и нокаутными образцами в каждой группе. Поэтому все образцы были надежно дифференцированы на основании экспрессии DEGs.
Построение сети ко-экспрессии и идентификация ее ключевых модулей
Для того, чтобы проверить, содержались ли в образцах “выпадающие” значения, были проанализированы графики кластеризации DEGs в группах Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax (рис. 2). Анализ показал, что образцы всех четырех групп не содержали существенных “выбросов”. В целях соответствия требованию безмасштабности сети, было проанализировано значение “мягкого” порога β. Значение β при достижении R2 0.9 составляло 16 для Epi-Msx, 14 – для Epi-Pax, 18 – для Mes-Msx и 16 – для Mes-Pax (рис. 3).
Рис. 2.
Анализ кластеризации генов по группам: (а) – Epi-Msx, (б) – Epi-Pax, (в) – Mes-Msx , (г) – Mes-Pax. Красным выделены образцы от нокаутных особей, черным – норма, контрольные образцы.

Рис. 3.
Определение мягкого порогового значения для взвешенного анализа генетической регуляторной сети ко-экспрессии для групп: (а) – Epi-Msx, (б) – Epi-Pax, (в) – Mes-Msx , (г) – Mes-Pax.

В соответствии с кластеризацией после динамического обрезания дендрограммы, высоко коррелированные гены были сгруппированы в модули. Всего в Epi-Msx было 6 модулей, в Epi-Pax – 5, в Mes-Msx – 6, в Mes-Pax – 8 (табл. 2). Кроме того, количество генов, соответствующих собственным цветовым модулям, несколько отличалось друг от друга (рис. 4).
Таблица 2.
Количество модулей и количество генов в четырех выделенных группах
| № модуля | Черный | Синий | Коричневый | Зеленый | Серый | Красный | Бирюзовый | Желтый | |
|---|---|---|---|---|---|---|---|---|---|
| EPI-13.5-Msx | 6 | – | 237 | 198 | 79 | 265 | – | 588 | 100 |
| EPI-13.5-Pax | 5 | – | 206 | 157 | – | 205 | – | 295 | 123 |
| MES-13.5-Msx | 6 | – | 173 | 84 | 58 | 221 | – | 615 | 61 |
| MES-13.5-Pax | 8 | 54 | 308 | 152 | 61 | 192 | 58 | 406 | 62 |
Далее был проведен анализ модулей, которые наиболее значимо ассоциировались с клиническими признаками, и, следовательно, имели биологическое значение. Группы Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax содержали многочисленные модули генов, которые были достоверно связаны с заболеваниями. Кроме того, было установлено, что коричневый (рис. 5а), коричневый (рис. 5б), синий (рис. 5в) и коричневый модули (рис. 5г) являются наиболее релевантными модулями генов, имеют наибольшую значимость (с коэффициентом корреляции >0.9) в группах Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax соответственно (табл. 3). Модули, обладающие высокой ассоциативностью с определенными клиническими признаками в различных группах, показаны в ко-экспрессионной сети (рис. 6).
GO-анализ обогащения
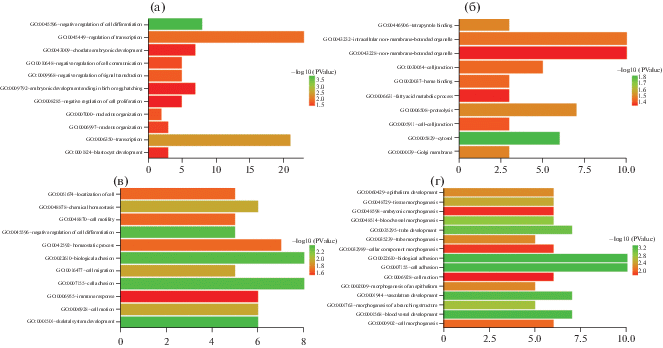
Основные DEGs в ключевых модулях были проанализированы с помощью анализа оверпредставленных терминов GO. В частности, в “коричневом” модуле группы Epi-Msx было выявлено обогащение термина “отрицательная регуляция пролиферации клеток” (GO:0008285). В “коричневом” модуле группы Epi-Pax был обогащен термин “цитозоль” (GO:0005829). В “синем” модуле группы Mex-Msx был в значительной степени обогащен термин “клеточная адгезия” (GO:0007155). В “коричневом” модуле группы Mex-Pax были достоверно обогащены термины “клеточная адгезия” (GO:0007155), “развитие кровеносных сосудов” (GO:0001568) и “морфогенез кровеносных сосудов” (GO:0048514) (рис. 7).
Рис. 5.
Распределение среднего GS и ошибок в модулях групп: (а) – Epi-Msx, (б) – Epi-Pax, (в) – Mes-Msx , (г) – Mes-Pax.

Рис. 6.
Сети ко-экспрессии: (а) – Epi-Msx, (б) – Epi-Pax, (в) – Mes-Msx, (г) – Mes-Pax. Розовым и красным обозначены узлы с повышенным уровнем экспрессии, зеленым и желтым – узлы со сниженной экспрессией. Красные и зеленые узлы являются hub-узлами, которые имеют более высокий ранг в сети и являются предметом данного исследования. Размер круга соответствует уровню узла в сети. Линиями обозначены взаимодействия между узлами.

Уровни экспрессии hub-генов на различных стадиях развития зубов t
На основе сети, показанной на рис. 6, были выявлены hub-гены с наиболее высокой степенью достоверности. Такими hub-генами в сетях Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax групп были, соответственно, каталитическая субъединица 3-альфа (IDH3A) изоцитрат-дегидрогеназы (NAD(+)), саркоспан (SSPN), альфа цепь коагуляционного фактора XIII (F13A1) и предшественник Cerebellin 1 (CBLN1). На рис. 8 показаны уровни экспрессии вышеупомянутых hub-генов на разных стадиях развития зуба. Экспрессия гена IDH3 была самой высокой на начальной стадии Е10, но постепенно снижалась с течением времени и снова увеличивалась со стадии Е11.5. Экспрессия гена F13A1 поднялась до самого высокого уровня на стадии E11, затем постепенно снижалась и достигла самого низкого уровня на стадии E13. На стадии Е13.5 экспрессия гена F13A1 внезапно значительно увеличилась. Экспрессия гена SSPN была самой высокой на начальной стадии E11 по сравнению с другими временными точками. С течением времени экспрессия постепенно снижалась, а затем усиливалась, начиная со стадии Е12.5. После этого уровень экспрессии выравнивался, оставаясь в пределах небольшого диапазона изменений. Показатели экспрессии гена CBLN1 медленно снижались со стадии Е10–Е11.5 и внезапно резко возросли на стадии Е12, после чего тенденция к быстрому росту продолжилась до конца изученного временного интервала. Кроме того, был проведен анализ оверпредставленных терминов GO для 10 ведущих hub-генов (рис. 9). Было обнаружено, что в hub-узлах группы Epi-Msx обогащены такие ключевые функции как “окислительно-восстановительный метаболический процесс коферментов” (GO:0006733). Примечательно, что ген IDH3A также был вовлечен в реализацию этих функций. Для hub-узлов в группе Epi-Pax была в основном характерна “клеточная адгезия через молекулы адгезии, ассоциированные с цитоплазматической мембраной” (GO:0098742). Hub-узлы в группе Mex-Msx были значительно обогащены термином “дифференцировка шванновских клеток” (GO:0014037), а в группе Mex-Pax – термином “клеточная адгезия через молекулы адгезии, ассоциированные с цитоплазматической мембраной” (GO:0098742), как и hub-узел CBLN1. В то же время SSPN и F13A1 не получили какого-либо обогащения ни по одной из функций.
Таблица 3.
Коэффициенты корреляции между каждым цветовым модулем и заболеваниями в четырех исследованных группах
| № модуля | Черный | Синий | Коричневый | Зеленый | Серый | Красный | Бирюзовый | Желтый | |
|---|---|---|---|---|---|---|---|---|---|
| EPI-13.5-Msx | 6 | – | 0.86 | 0.92 | 0.71 | 0.28 | – | 0.78 | 0.8 |
| EPI-13.5-Pax | 5 | – | 0.87 | 0.91 | – | 0.3 | – | 0.9 | 0.82 |
| MES-13.5-Msx | 6 | – | 0.93 | 0.79 | 0.8 | 0.25 | – | 0.9 | 0.81 |
| MES-13.5-Pax | 8 | 0.81 | 0.89 | 0.92 | 0.82 | 0.27 | 0.63 | 0.83 | 0.71 |
ОБСУЖДЕНИЕ
Зубы млекопитающих формируются в процессе органогенеза, основанного на саморегулирующихся взаимодействиях между эпителием и мезенхимой (Saxen, Thesleff, 1998). У мышей наблюдается несколько стадий развития зуба. Во-первых, на месте будущего зуба формируется эпителиальная плакода (локальное утолщение эпителия челюстной эктодермы) (день E11.5). Затем утолщенный зубной эпителий начинает вторгаться в лежащую под ним одонтогенную мезенхиму, которая затем конденсируется вокруг него (день Е12). Этот процесс приводит к формированию почки зуба (день Е13). Недавние исследования показали, что диффундирующие факторы роста могут служить сигнальными молекулами, обеспечивающими взаимодействие эпителия и мезенхимальных клеток в органогенезе зуба, а также в активацию или ингибирование экспрессии генов, связанных с развитием зуба. В процессе развития зуба транскрипционный фактор Msx в основном играет роль медиатора передачи сигнала (Chen et al., 1996). Кроме того, Pax9, который непрерывно экспрессируется в одонтогенной мезенхиме, является важным регулятором морфогенеза именно мезенхимальной, а не эпителиальной ткани зуба. Этот ген может быть использован в качестве маркера одонтогенной мезенхимы. Пониженная экспрессия Pax9 может привести к апоптозу зачатка зуба. Более того, экспрессия Pax9 также позволяет мезенхимальному зачатку на стадии почки зуба приобрести способность индуцировать эктопический эпителий к формированию зуба (Tucker et al., 1998).
При сравнении профилей экспрессии генов Msx1 и Pax9, были показаны значительные различия между образцами дикого типа и нокаут-образцами эпителия и мезенхимы. Таким образом, мы подтвердили, что Msx1 и Pax9 играют важную роль в развитии зуба. Анализ оверпредставленных терминов GO позволил установить, что DEGs участвуют в негативной регуляции клеточной пролиферации, адгезии клеток, развитии кровеносных сосудов и морфогенезе кровеносных сосудов. Hub-генами групп Epi-Msx, Epi-Pax, Mes-Msx и Mes-Pax были, соответственно, IDH3A, SSPN, F13A1 и CBLN1. При сравнении экспрессии этих генов в каждой временной точке можно убедиться, что средние значения экспрессии IDH3A, F13A1, SSPN и CBLN1 в разных временных точках были разными. Кроме того, IDH3A был значительно обогащен термином GO, связанным с метаболическим процессом окисления кофермента, а CBLN1 – термином GO, связанным с клеточной адгезией.
Сравнение профиля экспрессии генов здоровых и патологических тканей десны выявило 61 группу дифференциально экспрессирующихся генов (TOP 2). Гены этих групп негативным образом регулируют клеточную пролиферацию (Demmer et al., 2008). Аналогичным образом, негативная регуляция клеточной пролиферация достоверно коррелировала с процессами развития зубов в группе Epi-Msx. Предыдущее исследование выявило изменения ранней экспрессии генов в клетках пульпы зуба человека (HDPC), обработанных димета- крилатом триэтиленгликоля (TEGDMA) (использовалась концентрация с низкой токсичностью). Кластерный анализ обогащения групп GO выявил, что DEGs были связаны с адгезией клеток, развитием кровеносных сосудов и морфогенезом кровеносных сосудов (Cho et al., 2014). Это объясняется тем, что изменения в тканях пульпы влияют на рост зубов. Аналогичным образом, анализ обогащения терминами GO для группы Mes-Pax показал связь с клеточной адгезией, морфогенезом кровеносных сосудов, развитием кровеносных сосудов. Анализ обогащения для группы Mes-Msx выявил связь с адгезией клеток. По результатам этих исследований, регуляция развития зубов может быть связана с негативной регуляцией клеточной пролиферации, адгезии клеток, развития кровеносных сосудов и морфогенеза кровеносных сосудов.
CBLN1 (cerebellin 1 precursor) является одним из компонентов генетической регуляторной сети гена TP63. Он участвует в регуляции одонтогенеза и ранних стадий развития зуба через модуляцию клеточной адгезии, клеточной сигнализации и эпителиально-мезенхимных взаимодействий (Rostampour, 2018). Предыдущие исследования показали, что CBLN1 необходим для регуляции развития первичного тройничного нерва на ранней стадии формирования зуба. Нерв мог достигать эпителиальных клеток зуба примерно на E10 день, и аксон начинал расти до зуба на стадии прорастания (Lumsden, 1988; Kettunen et al., 2005). CBLN1 был охарактеризован в этом исследовании как hub-ген и участник ко-экспрессионной сети. Было показано, что уровень экспрессии гена CBLN1 был максимален в начале стадии Е10. Примечательно, что CBLN1 был значительно обогащен GO термином “адгезия клеток”. Действительно, взаимодействия клетка-клетка и клетка-внеклеточный матрикс играют ключевую роль в модуляции пластичности/жесткости определенных участков эмалевого органа, что влияет на морфогенез зуба (Lesot, Brook, 2009). Основываясь на наших результатах, можно предположить, что в случае снижения уровня экспрессии Msx1 и Pax9, CBLN1 может играть важную роль на стадии развития зуба Е10 посредством регуляции адгезии клеток.
Кроме того, показано, что коэнзим Q10 (rCoQ10) оказывает ранозаживляющее действие на мягкие ткани после удаления зуба у крыс путем регуляции окислительного стресса и эффективности работы митохондрий (Yoneda et al., 2014). Эти данные позволяют предположить, что в развитии зуба может быть задействован процесс метаболизма этого коэнзима. В данном исследовании функция IDH3A оказалась значительно обогащена именно для процесса метаболизма данного кофермента. Это позволяет предположить, что IDH3A может быть вовлечен в развитие зуба при снижении регуляции Msx1 и Pax9 путем участия в метаболическом процессе кофермента. У мышей-мутантов по IDH3A была исследована роль IDH3 в развитии дегенеративного фенотипа сетчатки, но данных об изменении фенотипа зубов у этих мышей нет (Findlay et al., 2018). В настоящее время практически не имеется данных о роли IDH3A, SSPN, F13A1 в развитии зубов. Однако на основании наших данных может быть сделано предположение, что IDH3A, SSPN, F13A1 действительно могут принимать участие в регуляции развития зубов.
Вместе с тем, профиль экспрессии CBLN1, IDH3A, SSPN и F13A1 не был проверен с использованием таких экспериментальных подходов, как количественный ПЦР анализ, иммуногистохимия или гибридизация in situ. Более того, взаимодействия между Msx1 и IDH3A или SSPN, а также Pax9 и F13A1 или CBLN1 не были доказаны в исследованиях in vivo или in vitro. Наконец, потенциальная роль IDH3A, F13A1, SSPN и CBLN1 в развитии зуба была показана с помощью биоинформатических методов, но не была экспериментально установлена. Для подтверждения наших результатов необходимы дальнейшие исследования.
Список литературы
Arte S. Phenotypic and genotypic features of familial hypodontia: Academic Dissertation. Helsinki: Helsinki University, 2001. 80 p.
Bei M. Molecular genetics of tooth development // Curr. Opin. Genet. Dev. 2009. V. 19. № 5. P. 504–510.
Benjamini Y. Discovering the false discovery rate // J. R. Stat. Soc. Series B Stat. Methodol. 2010. V. 72. № 4. P. 405–416.
Benjamini Y., Hochberg Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing // J. R. Stat. Soc.: Series B Methodol. 1995. V. 57. № 1. P. 289–300.
Chen Y., Bei M., Woo I., Satokata I., Maas R. Msx1 controls inductive signaling in mammalian tooth morphogenesis // Development. 1996. V. 122. № 10. P. 3035–3044.
Cho S., Lee J., Heo J.S., Kim S. Gene expression change in human dental pulp cells exposed to a low level toxic concentration of triethylene glycol dimethacrylate: an RNAseq analysis // Basic & Clinical Pharmacology & Toxicology. 2014. V. 115. № 3. P. 282–290.
Demmer R.T., Behle J.H., Wolf D.L. et al. Transcriptomes in healthy and diseased gingival tissues // J. Periodontology. 2008. V. 79. № 11. P. 2112–2124.
Deza M.M., Deza E. Encyclopedia of Distances. Berlin: Springer, 2009. 583 p.
Estrada E. Virtual identification of essential proteins within the protein interaction network of yeast // Proteomics. 2006. V. 6. № 1. P. 35–40.
Falcon S., Gentleman R. Using GOstats to test gene lists for GO term association // Bioinformatics. 2006. V. 23. № 2. P. 257–258.
Findlay A.S., Carter R.N., Starbuck B. et al. Mouse Idh3a mutations cause retinal degeneration and reduced mitochondrial function // Dis. Model. Mech. 2018. V. 11. № 12. P. 036426.
Fujita A., Sato J.Ż.R., de Oliveira Rodrigues L., Ferreira C.E., Sogayar M.C. Evaluating different methods of microarray data normalization // BMC Bioinformatics. 2006. V. 7. № 1. P. 469.
Gentleman R., Carey V., Huber W., Irizarry R., Dudoit S. Bioinformatics and Computational Biology Solutions Using R and Bioconductor. Springer Science & Business Media, 2006. 474 p.
Jeong H., Mason S.P., Barabási A.-L., Oltvai Z.N. Lethality and centrality in protein networks // Nature. 2001. V. 411. № 6833. P. 41.
Jheon A.H., Seidel K., Biehs B., Klein O.D. From molecules to mastication: the development and evolution of teeth // Wiley Interdiscip. Rev. Dev. Biol. 2013. V. 2. № 2. P. 165–182.
Jia Y., Nie K., Li J., Liang X., Zhang X. Identification of therapeutic targets for Alzheimer’s disease via differentially expressed gene and weighted gene co-expression network analyses // Molecular Medicine Reports. 2016. V. 14. № 5. P. 4844–4848.
Jussila M., Thesleff I. Signaling networks regulating tooth organogenesis and regeneration, and the specification of dental mesenchymal and epithelial cell lineages // Cold Spring Harb Perspect. Biol. 2012. V. 4. № 4. P. a008425.
Kang J., Bai R., Liu K. et al. Identification of significantly different modules between permanent and deciduous teeth by network and pathway analyses // Genet. Mol. Res. 2016. V. 15. № 4. P. gmr15047959.
Kettunen P., Løes S., Furmanek T. et al. Coordination of trigeminal axon navigation and patterning with tooth organ formation: epithelial-mesenchymal interactions, and epithelial Wnt4 and Tgfβ1 regulate semaphorin 3a expression in the dental mesenchyme // Development. 2005. V. 132. № 2. P. 323–334.
Langfelder P., Horvath S. WGCNA: an R package for weighted correlation network analysis // BMC Bioinformatics. 2008. V. 9. № 1. P. 559.
Lesot H., Brook A. Epithelial histogenesis during tooth development // Archives of Oral Biology. 2009. V. 54. P. S25–S33.
Li Z., Yu M., Tian W. An inductive signalling network regulates mammalian tooth morphogenesis with implications for tooth regeneration // Cell Proliferation. 2013. V. 46. № 5. P. 501–508.
Lumsden A. Spatial organization of the epithelium and the role of neural crest cells in the initiation of the mammalian tooth germ // Development. 1988. V. 103(S). P. 155–169.
O’Connell D.J., Ho J.W., Mammoto T. et al. A Wnt-bmp feedback circuit controls intertissue signaling dynamics in tooth organogenesis // Science Signaling. 2012. V. 5. № 206. P. ra4-ra4.
Oldham M.C., Konopka G., Iwamoto K. et al. Functional organization of the transcriptome in human brain // Nature Neuroscience. 2008. V. 11. № 11. P. 1271.
Peters H., Neubüser A., Kratochwil K., Balling R. Pax9-deficient mice lack pharyngeal pouch derivatives and teeth and exhibit craniofacial and limb abnormalities // Genes & Development. 1998. V. 12. № 17. P. 2735–2747.
Plaisier C.L., Horvath S., Huertas-Vazquez A. et al. A systems genetics approach implicates USF1, FADS3, and other causal candidate genes for familial combined hyperlipidemia // PLoS Genetics. 2009. V. 5. № 9. P. e1000642.
Press W., Teukolsky S., Vetterling W., Flannery B. Hierarchical Clustering by Phylogenetic Trees. Numerical Recipes. The Art of Scientific Computing. Cambridge: Cambridge University Press, 2007. 1235 p.
Rostampour N. Linking new genes to an odontogenesis TP63-mediated gene regulatory network that is peripheral to jaw morphogenesis: Academic Dissertation. Saskatoon: University of Saskatchewan, 2018. 71 p.
Satokata I., Maas R. Msx1 deficient mice exhibit cleft palate and abnormalities of craniofacial and tooth development // Nature Genetics. 1994. V. 6. № 4. P. 348.
Saxén L., Thesleff I. Epithelial-mesenchymal interactions in murine organogenesis // Ciba Found. Symp. 1992. V. 165. P. 183–193.
Smoot M.E., Ono K., Ruscheinski J., Wang P.-L., Ideker T. Cytoscape 2.8: new features for data integration and network visualization // Bioinformatics. 2010. V. 27. № 3. P. 431–432.
Szekely G.J., Rizzo M.L. Hierarchical clustering via joint between-within distances: Extending Ward’s minimum variance method // J. Classification. 2005. V. 22. № 2. P. 151–183.
Thesleff I. The genetic basis of normal and abnormal craniofacial development // Acta Odontologica Scandinavica. 1998. V. 56. № 6. P. 321–325.
Thesleff I. Epithelial-mesenchymal signalling regulating tooth morphogenesis // J. Cell. Sci. 2003. V. 116. № 9. P. 1647–1648.
Troyanskaya O., Cantor M., Sherlock G. et al. Missing value estimation methods for DNA microarrays // Bioinformatics. 2001. V. 17. № 6. P. 520–525.
Tucker A.S., Matthews K.L., Sharpe P.T. Transformation of tooth type induced by inhibition of BMP signaling // Science. 1998. V. 282. № 5391. P. 1136–1138.
Wang L., Cao C., Ma Q. et al. RNA-seq analyses of multiple meristems of soybean: novel and alternative transcripts, evolutionary and functional implications // BMC Plant Biology. 2014. V. 14. № 1. P. 169.
Yoneda T., Tomofuji T., Kawabata Y. et al. Application of coenzyme Q10 for accelerating soft tissue wound healing after tooth extraction in rats // Nutrients. 2014. V. 6. № 12. P. 5756–5769.
Дополнительные материалы отсутствуют.