Онтогенез, 2021, T. 52, № 6, стр. 397-418
Парадоксы эпигенетики растений
Т. А. Ежова *
Московский государственный университет им. М.В. Ломоносова, биологический факультет
119234 Москва, Ленинские горы, 1, стр. 12, Россия
* E-mail: ezhova2001@mail.ru
Поступила в редакцию 05.06.2021
После доработки 27.06.2021
Принята к публикации 04.07.2021
Аннотация
Растения обладают уникальной способностью приспосабливать онтогенез к изменяющимся условиям среды и воздействию стрессовых факторов. Эта способность основана на существовании у растений двух особенностей эпигенетической регуляции, которые на первый взгляд выглядят взаимоисключающими. С одной стороны, растения способны к частичному эпигенетическому перепрограммированию генома, что может приводить к адаптации физиологии и метаболизма к изменившимся условиям среды, а также к изменению программ онтогенеза. С другой стороны, растения могут проявлять удивительную стабильность эпигенетических модификаций и способность передавать их вегетативным и половым поколениям. Сочетание этих неразрывно связанных между собой особенностей эпигенетики не только обеспечивает выживаемость в условиях прикрепленного/неподвижного образа жизни, но и лежит в основе удивительно широкого морфологического разнообразия растений, которое может приводить к появлению морф внутри одной популяции и существованию межпопуляционных морфологических различий. В обзоре обсуждаются молекулярно-генетические механизмы, которые обуславливают парадоксальное сочетание свойств стабильности и лабильности эпигенетических модификаций и лежат в основе поливариантности онтогенеза. Рассматриваются также существующие подходы для изучения роли эпигенетической регуляции в проявлении поливариантности онтогенеза, обсуждаются их ограничения и перспективы.
ВВЕДЕНИЕ
В 2022 г. исполняется 150 лет со дня рождения Николая Константиновича Кольцова – предтечи многих основополагающих открытий и научных направлений ХХ века, включая эпигенетику (Morange, 2011; Раменский, 2018). Среди предсказаний великого ученого есть не только рассуждения об эпигенетических явлениях, но и идея о влиянии метилирования на проявление наследственных признаков. В 1915 г. он утверждал, что “во всяком органическом соединении атом водорода может быть скачкообразно заменен группой СН3” (Кольцов, 1915). Позже, идея о влиянии негенетических факторов на реализацию генотипа, о множественности “эпигенетических траекторий”, возможных для одного генотипа, была развита Конрадом Х. Уодингтоном (Waddington, 1968), а открытие роли метилирования ДНК и гистоновых белков в эпигенетической регуляции действия генов полностью подтвердили предсказание Н.К. Кольцова. Сегодня, благодаря значимым успехам в изучении молекулярно-генетических механизмов эпигенетических изменений, эпигенетика стала одним из важнейших направлений генетики, биологии развития и молекулярной биологии. Хотя само понятие эпигенетики трактуется в разных источниках по-разному (см. обзор Тиходеев, 2015), большинство исследователей согласны с тем, что эпигенетические изменения – это модификационные изменения не связанные с изменениями последовательности ДНК, но способные к сохранению в ряду клеточных поколений даже в отсутствие факторов, вызвавших эти изменения (Jablonka, Raz, 2009; Ptashne, 2013; Noble, 2015). Наследуемые изменения экспрессии аллелей генов, вызванные эпигенетическими модификациями (ЭМ), называют эпиаллелями.
Эпигенетические механизмы регуляции генной экспрессии лежат в основе целого ряда биологических процессов, обеспечивающих существование и развитие растений и животных (Эллис и др., 2010). Без поддержания гетерохроматинового состояния центромерных и теломерных районов, невозможно функционирование хромосомного аппарата в ходе митоза и мейоза. Состояние хроматина, которое определяется наличием “эпигенетических меток” (модифицированных вариантов гистоновых белков и метилцитозина в ДНК), влияет на процессы репарации и транскрипции (Разин, Быстрицкий, 2009). ЭМ защищают целостность генома, подавляя транскрипцию генов мобильных элементов, а значит и возможность их перемещения и размножения (Fultz et al., 2015). Эпигенетическая инактивация (замолкание) дуплицированных генов после алло- и автоплоидии, геномных дупликаций позволяет обеспечить геномный гомеостаз и открывает возможности для эволюции функции генов (Song, Chen, 2015). Компенсация дозы генов половых хромосом, хорошо исследованная у животных, имеет место и у двудомных растений с половыми хромосомами и также основана на ЭМ (Muyle et al., 2017, 2018).
Ключевую роль ЭМ играют в процессах развития. Переход на новый этап онтогенеза и развитие каждой новой структуры связаны с локальным эпигенетическим перепрограммированием, т.е. подавлением экспрессии генов, активных на предшествующей стадией, и, одновременно – активацией новой группы генов, определяющих специфику нового органа/стадии (Xiao, Wagner, 2015; Xu et al., 2018; Yan et al., 2020). Установленный профиль генной экспрессии сохраняется клеточными потомками. Именно клеточная память, основанная на эпигенетических механизмах, лежит в основе поддержания на протяжении всего онтогенеза структурно-функциональных особенностей всех тканей и органов многоклеточных организмов (Nashun et al., 2015; Birnbaum, Roudier, 2016).
Эпигенетические процессы, которые являются неотъемлемой частью генетической программы развития и инициируются внутренними сигналами, называют конститутивным эпигенетическим контролем развития (Bräutigam, Cronk, 2018). При нарушении функции ключевых эпигенетических регуляторов клетки растений утрачивают способность поддерживать клеточную идентичность (дифференцированное состояние), приобретают каллусные характеристики и способность к соматическому эмбриогенезу (Aichinger et al., 2009; Chen et al., 2010; Bratzel et al., 2010).
Все перечисленные функции ЭМ являются общими для животных и растений. Вместе с тем, ЭМ у растений выполняют дополнительную функцию адаптации к локальным условиям среды, к воздействиям неблагоприятных факторов за счет изменения программ развития. Взаимодействие между программами развития и программами адаптивного (стрессового) ответа позволяет быстро находить оптимальную жизненную стратегию в условиях ограниченного пула ресурсов (Herms, Mattson, 1992; Huot et al., 2014). Это возможно благодаря тому, что разнообразные неблагоприятные условия (дефицит света, воды, питания, гипо- или гипертермия, воздействие патогенов и пр.) могут инициировать частичное эпигенетическое перепрограммирование генома, которое открывает возможность для изменения программы развития. Эпигенетическую корректировку генетически обусловленной программы развития в ответ на действие внешних сигналов, называют факультативным эпигенетическим контролем развития (Bräutigam, Cronk, 2018).
Возможность изменения программы развития принципиально важна в условиях прикрепленного или пассивного (у водных растений) образа жизни и обусловливает удивительную пластичность онтогенеза, характерную именно для растений (Серебряков, Серебрякова, 1972; Жукова, Глотов, 2001; Калинкина, 2017; Sultan, 2017; Нотов, Жукова, 2019). Более того, у растений фенотипические изменения, вызванные внешними воздействиями, могут поддерживаться вегетативными и половыми потомками (Hauser et al., 2011; Heard, Martienssen, 2014; Lämke, Bäurle, 2017; He, Li, 2018). В этом обзоре основное внимание будет уделено рассмотрению роли ЭМ в проявлении этой удивительной способности к перепрограммированию онтогенеза и анализу механизмов, которые могут инициировать эпигенетическое перепрограммирование онтогенеза и появление морфотипов под действием сингналов окружающей среды.
ОСОБЕННОСТИ БИОЛОГИИ РАСТЕНИЙ, ЛЕЖАЩИЕ В ОСНОВЕ ПЛАСТИЧНОСТИ РАЗВИТИЯ
Пластичность развития – важнейшее условие выживания в условиях прикрепленного образа или пассивного (у водных растений) образа жизни. Она обусловлена способностью растительных клеток изменять свою идентичность в ответ на сигналы от окружающих клеток, физиологические сигналы и сигналы из окружающей среды. Эта способность, установленная с использованием химерных растений (Poethig,1989; Irish, 1991; Szymkowiak, Sussex, 1996) и в экспериментах по регенерации de novo корней (обзоры Xu, 2018; Li, 2021) и побегов (Shin et al., 2020; Lardon, Geelen, 2020) из различных органов, свидетельствует о том, что растительные клетки с легкостью меняют генетическую программу развития.
У растения образование новых органов происходит на всем протяжении онтогенеза, поэтому изменения программы онтогенеза могут происходить на любой стадии жизненного цикла, в том числе и у взрослого растения. Эти изменения облегчаются тем, что они могут затрагивать не весь организм, а отдельные его части, поскольку растения имеют модульное строение (Hallé, 1986; Oborny, 2019). Модули образуются последовательно как результат функционирования апикальных меристем побега и корня. Модуль побега – это междоузлие с листом и пазушной меристемой. Корневой модуль – фрагмент с корнем и отходящим от него боковым корешком. При изменении условий (внешних или внутренних) может меняться судьба отдельных, наиболее молодых модулей. Такая локальная перестройка морфогенеза может обеспечить адаптацию образующихся частей растения к новым условиям, не изменяя всей структуры растения.
Благодаря эпигенетическим механизмам новая программа может сохраняться не только клеточным клоном, но и вегетативными поколениями, поскольку многие растения способны к вегетативному размножению. Получено много экспериментальных данных о наследовании вегетативным потомством профиля метилирования ДНК, вызванного стрессовыми воздействиями на материнские растения. У клевера ползучего Trifolium repens (Leguminósae) изменения метилирования ДНК, вызванные воздействием засухи на материнское растение, сохранялись у 5 исследованных вегетативных поколений (Rendina González et al., 2018). Специфичный профиль метилирования у клонов инвазивного растения очереднопы́льника, прозванного сорняком аллигатора, Alternanthera philoxeroides (Amaranthaceae) из разных эколого-географических мест Китая сохранялся при перенесении в общие условия в течении 2–3 поколений, хотя часть ЭМ (38%) сохранялись на протяжении 10 вегетативных поколений (Shi et al., 2019).
Стабильная передача отдельных эпиаллелей потомству показана и на многих видах с облигатным генеративным размножением – у льнянки Linaria vulgaris, арабидопсис Arabidopsis thaliana, курурузы Zea mays, риса Oryza sativa, томатов Solanum lycopersicum и др. (Cubas et al., 1999; Kakutani, 2002; Manning et al., 2006; Miura et al., 2009; O’Malley, Ecker, 2012; Zhang et al., 2012; Weigel, Colot, 2012; Johannes, Schmitz, 2019). Некоторые эпиаллели растений настолько стабильны, что демонстрируют моногенные расщепления и их сложно отличить от моногенных мутаций (Bondada et al., 2020).
Передаче половому потомству ЭМ способствует отсутствие характерного для животных глобального деметилирования генома в процессе гаметогенеза и раннего эмбриогенеза (Pikaard, Scheid, 2014). Эта особенность и действующие в клетках растений молекулярные механизмы поддержания ЭМ (см. далее) объясняют удивительно высокую долю стабильно поддерживающихся сайтов метилцитозина (91%) и районов метилирования (99.998%) в генетически однородных имбредных линиях арабидопсис (МА-линиях), которые получены от единственного растения – основателя путем самоопыления в течение 30 поколений (Schmitz et al., 2011; Hofmeister et al., 2017).
Важной особенностью растений является отсутствие рано обособляющихся клеток зародышевого пути. У растений генеративные клетки образуются из инициальных клеток зародышевого пути, которые отличаются от окружающих соматических клеток меристем только положением в апексе побега, но не характером детерминации, и в случае гибели могут быть заменены соматическими (Whipple, 2012; Pikaard, Scheid, 2014). Cледовательно, генетическая и эпигенетическая информация соматических клеток потенциально может быть передана потомству.
Таким образом, особенности растений обусловливают возможность их выживания при прикрепленном/пассивным образе жизни. Изменяя генетическую программу развития, растения могут адаптировать онтогенез к условиям среды, проявляя пластичность развития. Новый вариант онтогенеза может сохраняться за счет эпигенетических механизмов и даже передаваться вегетативному и половому потомству. Благодаря этим особенностям вариабельность фенотипа растений может принимать форму поливариантности, т.е. существования определенных морфотипов на основе одного генотипа.
КАК ЭПИГЕНЕТИЧЕСКИЕ МОДИФИКАЦИИ МОГУТ ВЛИЯТЬ НА ФЕНОТИП
Основным механизмом эпигенетических модификаций у растений является метилирование ДНК и модификация гистоновых белков. К эпигенетическим явлениям относят и так называемое белковое наследование, т.е. наследование клеточным клоном измененной конформации белковых молекул, описанное у дрожжей, животных и человека (Harvey et al., 2018). Прионо-подобные белки обнаружены и у растений. Однако в норме они не образуют агрегаты в клетках, поскольку этому препятствуют метаболиты растений (Surguchov et al., 2019).
ЭМ ДНК и гистонов осуществляют 3 группы генов (Pikaard, Scheid, 2014; Allis, Jenuwein, 2016). Гены 1-ой группы (writers) кодируют ферменты, осуществляющие модификации ДНК (метилирование цитозина) и гистонов (метилирование, ацетилирование, убиквитинирование, фосфорилирование, сумоилирование гистонов и пр.). Гены 2-ой группы (erasers) осуществляют противоположную функцию, убирая эти метки. Гены 3-ей группы (адапторные белки или readers) распознают эпигенетические метки и через взаимодействие с другими белками влияют на разнообразные клеточные процессы (рис. 1). Некоторые белки могут совмещать в себе разные функции. Например, хромометилазы СМТ являются одновременно белками 1-ой и 3-ей групп, поскольку распознают гистоновые репрессивные метки и метилируют ДНК в непосредственной близости от этих меток (Zhang et al., 2018). Точно также, гистоновые Н3К9-метилтрансферазы осуществляют свою главную функцию после распознавания метилцитозина 5mC (Li et al., 2018). Однако приведенная выше классификация помогает систематизировать обширную информацию по эпигенетической регуляции.
Рис. 1.
Основные группы эпигенетических регуляторов и их влияние на экспрессию генов. (а) – эпигенетические регуляторы 1-ой группы (1 – writers, стрелки) осуществяют метилирование ДНК (вверху) и модификацию гистонов (внизу); регуляторы 2-ой группы (2 – erasers, тупые стрелки) удаляют эпигенетические метки с ДНК и гистоновых белков; регуляторы 3-ей группы (3 – readers) распознают метки; взаимодействуя с белками РНК-полимеразного комплекса (РНК-пол II), они могут влиять на выбор промотора (показаны альтернативные промоторы Р1, Р2) и скорость транскрипции. Взаимодействуя с белками сплайсеосомы и комплекса полиаденилирования (полиА), белки 3-ей группы могут вмешиваться в процессы сплайсинга и полиаденилирования пре-мРНК; (б) – за счет таких взаимодействий может наблюдаться исключение экзонов и использование альтернативных сайтов сплайсинга (справа), а также образование укороченных мРНК из-за использования альтернативных сайтов полиаденилирования (справа). Это приводит к формированию разных изоформ зрелой мРНК и белка. Широкие стрелки указывают на альтернативные сайты сплайсинга или полиаденилирования.
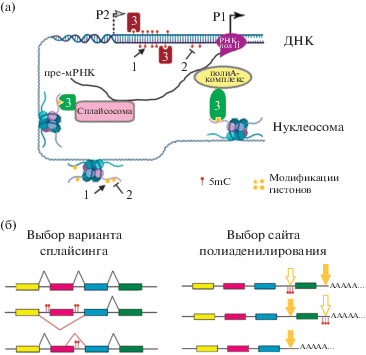
1 группа – writers. К этой группе относятся гены, кодирующие ДНК-метилтрансферазы и гистоновые метилтрансферазы. В отличие от животных, у которых метилируется главным образом цитозин, соседствующий с гуанином (CG-сайты), у растений может метилироваться цитозин в любом окружении (CG, CHG и CHH, где H это A, C или T). Это связано с существованием уникального семейства генов хромометилаз CHROMOMETHYLASE (CMT), а также генов, контролирующих путь РНК-зависимого метилирования ДНК, или RdDM (RNA-directed DNA Methylation pathway). RdDM-путь основан на механизме РНК-интерференции, в котором участвуют варианты консервативных комплексов Dicer и Rinc (включающие белки DLC и AGO, соответственно), а также специфические для растений РНК-полимеразные комплексы IV и V (Zhang et al., 2018; Raju et al., 2019; Gallego-Bartolomé, 2020).
Влияние метилирования ДНК на экспрессию генов зависит от того, в каком контексте метилируется цитозин, от места расположения сайтов метилирования. Метилирование цитозина в регуляторных частях гена чаще всего ассоциировано со снижением уровня транскрипции (Ванюшин, 2006; Law, Jacobsen, 2010). Однако в отдельных случаях, за счет взаимодействия с белками из 3-ей группы, эффект может быть и противоположным (Harris et al., 2018). Метилирование гистонов ассоциировано либо с активацией, либо с репрессией генов в зависимости от позиции и числа метильных групп. H3K4me3, H3K9me3 и H3K36me3 коррелирует с активацией транскрипции, в то время как присутствие метки H3K27me3 маркирует гены со сниженным уровнем транскрипции (Zhang et al., 2007, 2009; Roudier et al., 2011). Метки H3K9me2 и H3K27me1 обычно локализованы в центромерных районах хромосом и типичны для повторов и мобильных элементов, которые содержат и высоко метилированную ДНК (Bernatavichute et al., 2008; Zhang et al., 2009; Roudier et al., 2011).
Метилирование гистонов осуществляют несколько семейств гистоновых метилтрансфераз, которые метилируют разные лизиновые остатки и работают в составе разных белковых комплексов, среди которых наиболее изучены PRC1-, PRC2-комплексы (Liu et al., 2010). В состав этих комплексов входят и гистоновые деацетилазы (присутствуют в PRC1, PRC2), и гистоновые убиквитиназы (PRC1), которые также вызывают компактизацию хроматина и репрессию генов (Derkacheva, Hennig, 2014; Mozgova, Hennig, 2015; Xiao, Wagner, 2015; Förderer et al., 2016).
Гистоновые ацетилазы разрыхляют хроматин и активируют генную экспрессию, а убиквитинирование оказывает либо активирующее, либо репрессирующее влияние на состояние хроматина. На состояние хроматина могут влиять и другие модификации гистоновых белков (Berger, 2007; Fenley et al., 2018), хотя их участие в эпигенетических процессах менее изучено. Гистоновые ацетилазы и убиквитиназы также могут работать в составе различных белковых комплексов, включая комплексы, поддерживающие активное состояние хроматина (Fletcher, 2017; Ornelas-Ayala et al., 2021).
2 группа – erasers. Активное удаление метильных групп с цитозина у растений осуществляется специфическими для растений ферментами с гликозилазной активностью, которые вырезают метилированный цитозин и инициируют его замену на неметилированный цитозин (Li et al., 2018; Parrilla-Doblas et al., 2019; Liu, Lang, 2019). Удаление ацетильных и метильных групп с гистоновых белков осуществляют несколько семейств гистоновых деацетилаз (Ma et al., 2013; Chen et al., 2020) и деметилаз (Prakash et al., 2014), среди которых есть специфические для растений.
3 группа – readers распознают (“прочитывают”) установленные метки или их отсутствие. Это самая разнообразная группа эпигенетических регуляторов (ЭР), которые также чаще всего действуют в составе белковых комплексов (Torres, Fujimori, 2015; Grimanelli, Ingouff, 2020). Нередко readers считывают одновременно разные гистоновые метки, включая фосфорные и убиквитиновые. К эпигенетическим регуляторам этой группы часто относят и хроматиновые белки, которые могут распознавать ЭМ и влияют на нековалентиное взаимодействие ДНК с гистоновыми белками, изменяют плотность упаковки и локализацию нуклеосом, а также катализируют включение гистоновых вариантов (Li et al., 2007; Narlikar et al., 2013).
В составе белков 3-ей группы имеются не только сайты связывания с метилцитозином ДНК или гистоновыми метками, но и сайты связывания с негистоновыми белками. За счет этого readers обеспечивают взаимодействие между эпигенетическими метками (модификациями гистонов и ДНК) и другими клеточными процессами, в которых участвует негистоновый белок (Li, Li, 2012; Patel, 2016; Grimanell, Ingouff, 2020). Например, белки readers могут взаимодействовать с белками, устанавливающими или убирающими метки с ДНК и гистонов, способствуя распространению меток или их снятию (Grimanell, Ingouff, 2020; Scheid et al., 2021). Могут способствовать открытию хроматина, за счет включения гистоновых вариантов, изменяющих состояние хроматина (Kouzarides, 2007; Sijacic et al., 2019). Они могут прямо влиять на транскрипцию, взаимодействуя с транскрипционными факторами (ТФ) или их кофакторами или сами иметь ДНК-связывающий домены, выступая в качестве ТФ-пионеров, аффинность и специфичность действия которых зависят от присутствия в ДНК метилцитозина (Kribelbauer et al., 2019; Grimanell, Ingouff, 2020; Zhu et al., 2016).
Белки 3-ей группы способны влиять на выбор промоторов (Le et al., 2020), а также на пост-транскрипционные события (рис. 1). Взаимодействуя с белками РНК-полимеразного комплекса, белками, контролирующими созревание пре-мРНК (белками, вовлеченными в полиаденилирование, сплайсинг), ридеры определяют выбор сайтов сплайсинга (Ullah et al., 2018; Wei et al., 2018; Wu et al., 2020) и сайтов полиаденилирования (Duan et al., 2017; Zhang et al., 2021). Из этого следует, что readers являются участниками регуляции экспрессии генов на разных этапах реализации генетической информации, сопрягая эпигенетические метки ДНК и гистонов с базовыми механизмами регуляции генной экспрессии. По сути, эпигеном определяет, какие гены транскрибируются в данной ткани, какой из альтернативных промоторов, сайтов сплайсинга, полиаденилирования будет выбираться в данном клеточном клоне, а значит – и какие из изоформ белка будут функционировать в данной ткани, на данной стадии онтогенеза и при данных условиях окружающей среды. Таким образом, ЭМ не только сохраняют профиль экспрессии, который установлен в результате действия ТФ, но и участвуют в его установленовлении на всех этапах образования и созревания мРНК.
Рассмотренные 3 группы генов контролируют ЭМ не только у растений, но и у животных. Однако регуляция экспрессии этих групп генов у растений должна иметь свои особенности. С одной стороны, работа этих генов должна зависеть от условий среды. Т.е. у растений должны существовать механизмы, которые могут инициировать частичное эпигенетическое репрограммирование генома и установление новой программы развития в изменившихся условиях среды. Но с другой стороны – растения должны иметь способность сохранять новую адаптивную программу развития и передавать ее потомкам. Это парадоксальное сочетание стабильности и лабильности ЭМ указывает на существование у растений особых механизмов, контролирующих динамику ЭМ. Рассмотрим имеющиеся данные, которые хотя бы частично могут объяснить уникальные особенности эпигенетической регуляции у растений.
МОЛЕКУЛЯРНЫЕ МЕХАНИЗМЫ, ЛЕЖАЩИЕ В ОСНОВЕ НАСЛЕДОВАНИЯ РАСТЕНИЯМИ ЭПИГЕНЕТИЧЕСКИХ МОДИФИКАЦИЙ
ЭМ различаются по стабильности наследования половыми поколениями и, как правлило, не подчиняются Менделевским закономерностям. Тем не менее, наследуемость, хотя бы в форме “наследуемой нестабильности” (“heritable changeability” Le Goff et al., 2021) – это характерная особенность ЭМ, присущая именно растениям.
Способность ЭМ сохраняться в мейозе отчасти объясняется особенностями биологии растений (см. выше). Тем не менее, есть и молекулярные механизмы, которые поддерживают эту способность. Их действие зависит от типа эпиаллели (“молчащая” гиперметилированная аллель или активно работающая гипометилированная эпиаллель), от копийности генов, от наличия в локусах специальных мотивов, которые привлекают ДНК-метилтрансферазы или другие ЭР, от числа сайтов метилирования ДНК, их типа (CG, CHG, CHH) и локализации в гене, от присутствия модификаций гистонов и их типа (Catoni et al., 2017; Li et al., 2020; Williams, Gehring, 2020).
Эти зависимости сложны и до конца не исследованы. Тем не менее, главными инструментами сохранения ЭМ являются поддерживающие ДНК-метилтрансферазы, которые есть и у животных, а также специфические для растений механизмы положительной обратной связи между хроматиновыми метками и метками метилцитозина (5mС). При поддержании установленного эпигенетического профиля хромометилазы CMT метилируют ДНК, ориентируясь на присутствие хроматиновых меток Н3К9me2, а гистоновые метилтрансферазы KYP/SUVH4 и др. – на присутствие 5mC (Baubec et al., 2010; Du et al., 2015; Stoddard et al., 2019). Этот механизм объясняет стабильное “молчание” гиперметилированных аллелей (например, в гене арабидопсис SUP).
Положительная обратная связь имеется при установлении метилирования de novo с участием малых РНК (RdDM путь). В этом многокомпонентном процессе важную роль играют белки 3‑ей группы (readers). Одни readers распознают хроматиновые метки метилированного Н3К9me2 и ориентируют работу РНК-полимеразы IV, участвующей в образовании малых РНК. Другие – распознают 5mC и привлекают РНК-полимеразу V, которая через посредников (прежде всего – белка AGO с загруженной в него малой РНК) может привлекать к участкам ДНК, с которых считывается пре-мРНК, специфическую ДНК-метилтрансферазу de novo DRM2 (Matzke, Mosher, 2014; Erdmann, Picard, 2020). Благодаря этим механизмам, появившиеся при репрограммировании генома ЭМ, будут поддерживаться до тех пор, пока в процесс не вмешаются другие регуляторы, которые могут активироваться изменением окружающих условий (см. далее). Активирующие гистоновые метки H3K4me3 и H3K18ac препятствуют работе RdDM пути и привлекают ДНК-деметилазу/гликозилазу ROS1, которая снимает метки 5mC и создают условия для стабильного поддержания гипометилированных эпиаллелей (Li et al., 2020).
Образовавшиеся малые РНК, постоянно генерирующиеся в ходе позитивной обратной петли, могут передвигаться по растению по плазмодес-мам и по флоэме (Tamiru et al., 2018; Pagliarani, Gambino, 2019), а возможно и передаваться потомкам, как это показано для нематоды C. elegans (Xu et al., 2018). У растений пока не получено четких доказательств участия малых РНК, вызванных стрессовым воздействием на родителей, в передаче эпигенетической информации потомкам (Morgado et al., 2017; Colicchio et al., 2020). Тем не менее, возникшие мобильные малые РНК могут проникать в герминальные клетки (Duempelmann et al., 2020) и инициировать в них метилирование ДНК и последующие модификации гистонов, которые могут передаваться потомкам и приводить к наследованию фенотипических изменений – онтогенетических или физиолого-биохимических изменений, связанных с устойчивостью к стрессу, испытанному родительскими растениями (Tamiru et al., 2018; Liegard et al., 2019).
Поддержанию в череде поколений эпигенетических меток в определенных генных локусах могут способствовать повторы, которые часто представляют собой в той или иной степени деградировавшие мобильные элементы, встроившиеся в регуляторную область генов. Так, наличие повторов в регуляторной области гена арабидопсис FWA и кукурузы B1 объясняет частое возникновения эпиаллелей этих генов (Henderson, Jacobsen, 2007). О стабильности ЭМ таких генов говорит тот факт, что на основе аналогичных ЭМ создан ряд сортов хозяйственно ценных культур (обзор Lisch, 2013).
В геномах млекопитающих найдены нуклеотидные мотивы, которые способствуют привлечению ДНК-метилтрансфераз к регуляторным участкам определенных локусов (Handa et al., 2005; Wienholz et al., 2010; Wang et al., 2019). Они есть и у растений, хотя менее изучены (Gouil, Baulcombe, 2016). Особенности структуры локусов, делающие их излюбленными мишенями ЭРегуляторов, могут объяснить неоднократно наблюдающиеся случаи возникновения стабильных эпиаллелей некоторых генов. Однако знания об особенностях ДНК или хроматина, которые могут превращать локусы в “горячие” и “холодные” точки ЭМ не достаточны. Например, до сих пор не ясно, почему такой “горячей” точкой ЭМ является ген SUP арабидопсис (Arabidopsis thaliana), который участвует в разграничении в цветке участков с мужскими и женскими репродуктивными органами (Bowman et al., 1992). Гиперметилированные “молчащие” эпиаллели в этом гене возникали при использовании индуцированного мутагенеза (нейтроны, Х-лучи, этилметансульфонат и др. химические мутагены), инсерционного Т-ДНК-мутагенеза (Jacobsen, Meyerowitz, 1997; Rohde et al., 2002), на фоне деметилирования генома у трансгенных линий с подавленной активностью гена ДНК-метилтрансферазы MET1 (Kishimoto et al., 2001), а также спонтанно (Bondada et al., 2020). Причем эпиаллели гена SUP с разной степенью экспрессивности и пенетрантности были обнаружены в 11 (из 1028) экотипах арабидопсис из самых разных мест обитания (Bondada et al., 2020).
Поддержание ЭМ в определенных локусах может обусловливаться наличием в их регуляторных участках цис-элементов для связывания ТФ, которые привлекают с собой ЭР (например, ДНК-метилтрансферазы или деметилазы), поддерживая гиперметилированное или наоборот – гипометилированное состояние локусов. Длинные некодирующие РНК (lncРНК) также могут привлекать ЭР к определенным локусам. Это показано для lncРНК COLDAIR, которая эпигенетически подавляет ген арабидопсис FLC, контролирующий потребность в яровизации (Kim et al., 2017; Wu et al., 2020), а также для lncRNA AG-incRNA4, которая вызывает замолкание в вегетативной части побега гена AG, контролирующего развитие репродуктивных органов в цветке (Wu et al., 2018). Благодаря взаимодействию с ЭР, ТФ и lncРНК могут вызывать ЭМ в генах-мишенях, у которых есть либо цис-элементы для привлечения ТФ, либо участки гомологии с lncРНК. Такие эффекторы ЭМ, экспрессия которых активируется в определенных условиях (или при стрессовых воздействиях), по-видимому, могут играть важную роль как в инициации ЭМ под действием новых условий/стрессов, так и в поддержании возникших эпиаллелей в ряду поколений при сохранении изменившихся условий (например, в 11 поколениях имбредных линий риса выращиваемых в условиях засухи (Zheng et al., 2017).
Ярким свидетельством наследования ЭМ у растений является установленное при изучении имбредных линий арабидопсис (МА-линии, полученных от самоопыления одного растения экотипа Col) стабильное наследование 99.998% районов метилирования на протяжении 30 генеративных поколений в неизменных условиях среды (Hofmeister et al., 2017). Такая стабильность позволила осуществить создание эпигенетической карты на основе анализа F2 поколения от скрещивания двух МА-линий и показать, что примерно половина дифференциально метилированных районов наследуется в соответствии с Менделевскими закономерностями (Hofmeister et al., 2017).
МОЛЕКУЛЯРНЫЕ МЕХАНИЗМЫ, ИНИЦИИРУЮЩИЕ ИЗМЕНЕНИЕ ПРОГРАММЫ РАЗВИТИЯ
Среди факторов, которые влияют на работу ЭР и инициируют частичное репрограммирование генома – многие стрессовые факторы (как абиотические, так и биотические). Они вызывают изменения метаболических процессов и появление в клетках активных форм кислорода (АФК) и азота, которые измененяют окислительно-восстановительный баланс клетки и активируют редокс-зависимые сигнальные системы (Foyer, Noctor, 2013; Locato et al., 2018). Следствием этих процессов являются изменение содержания молекул – доноров метильных групп, необходимых для модификации ДНК и гистонов (прежде всего – S‑aденозилметионина), а также воздействие АФК и метаболитов на белки – регуляторы эпигенетических модификаций (Locato et al., 2018; Saravana Kumar et al., 2020; Lindermayr et al., 2020). Среди последних – ДНК-деметилирующие ферменты (Couturier et al., 2013; Duan et al., 2015), белки, участвующие в РНК-зависимом метилировании ДНК (Charbonnel et al., 2017; Seta et al., 2017). Описано влияние редокс-потенциала и оксида азота, который также является мощным оксидантом, на белки, вовлеченные в метилирование ДНК и модификацию гистоновых белков (обзоры Locato et al., 2018; Saravana Kumar et al., 2020; Lindermayr et al., 2020). Поскольку у растений метилируется цитозин в любом окружении, растения особенно чувствительны к метаболическим изменениям, вызывющим снижение уровня доноров метильных групп в клетках (Lindermayr et al., 2020).
Недавно выявлен дополнительный механизм связи между стрессовыми воздействиями и репрограммированием генома. Показано, что в активации стрессового ответа (тепловой шок, гипотермия, засуха, дифицит фосфатов, атака патогенов), важную роль играет сумоилирование – обратимая посттрансляционная модификация различных белков (Jmii, Cappadocia, 2021). Важнейшей мишенью Сумо-E3 лигазы SIZ1 является ДНК-гликозилаза ROS1, которая стабилизируется в результате этой модификации и осуществляет деметилирование ДНК. Мутации в гене SIZ1 у арабидопсис приводят к повышению уровня метилирования ДНК за счет уменьшения содержания белка ROS1 (Kong et al., 2020). Сумоилирование белков – динамичный и обратимый процесс, который активируется стрессовыми воздействиями. Поэтому в условиях стрессовых воздействий сумоилирование белков через влияние на деметилирование ДНК может играть важную роль в репрограммировании генома растений.
Условия среды/стрессы могут изменять активность ЭР как на посттрансляционном уровне, так и на транскрипционном. Многие ЭР транскрибируются специфически, только при определенных условиях/стрессовых воздействиях или в определенных тканях и органах на определенных стадиях онтогенеза. Специфичность транскрипции показана у разных видов растений для ДНК-метилтрансфераз (Rohini et al., 2014; Guo et al., 2020; Yu et al., 2021) и других ЭР. Более того, среди ЭР, которые относятся к одному классу (например, ДНК-деметилаз/гликозилаз) наблюдаются и некоторые различия между генами-мишенями, что может объясняться наличием в мишенях специфических мотивов, привлекающих к себе ЭР, или тем, что в привлечении к генам-мишеням участвуют ТФ или lncРНК, которые взаимодействуют с определенным набором генов-мишеней (см. выше). Заметим лишь, что среди ТФ, которые взаимодействуют с ЭР, есть и компоненты гормональных сигнальных путей. Привлекая ЭР, изменяющие состояние хроматина к своим генам-мишеням, ТФ способны либо активировать (Wu et al., 2015), либо подавлять экспрессию генов (Hasegawa et al., 2018) в ответ на действие гормонов.
Большинство упомянутых факторов, которые способствуют изменению программы развития при изменении условий среды, действуют в той или иной мере на эпигенетические процессы и в клетках животных. Вместе с тем, у растений появляется информация об особенностях работы некоторых ЭР, а также об обнаружении новых специфических для растений ЭР. Среди последних – 4 гена арабидопсис, возникших в результате доместикации транспозонов семейства Harbinger. Продукты двух таких генов HDP1 и HDP2 работают в составе белкового комплекса IDM1, который обладает активирующей гистон-ацетилтрансферазной активностью и, одновременно, участвует в привлечении ДНК-деметилазы/гликозилазы ROS1 (Duan et al., 2016). Продукты двух других генов – ALP1 и ALP2 – также работают вместе и мешают компллексу PRC2 осуществлять репрессивное метилирование гистонов H3K27me3 (Liang et al., 2015; Velanis et al., 2020).
Гены ICU11 и CP2, найденные у арабидопсис, также взаимодействуют с белками PRC2-комплекса (Mateo-Bonmatí et al., 2018; Bloomer et al., 2020). Однако это взаимодействие приводит к усилению эффективности эпигенетического подавления генов-мишеней. Предполагается, что эти гены, кодирующие 2-оксоглутарат/Fe(II)-зависимые диоксигеназы, каким-то образом помогают удалять с гистонов активирующие метки H3K4me2/3 и H3K36me3, что облегчает дальнейшее установление репрессивных меток (Bloomer et al., 2020). Недавно у арабидопсис найден ген CFK1, кодирующий белок с F-боксом, который взаимодействует с de novo ДНК-метилтрансферазой и запускает процесс ее деградации (Chen et al., 2021).
Кроме этих новых генов, которые вмешиваются в работу уже описанных ЭР, у растений выявлены гены, которые являются представителями известных семейств эпигенетических регуляторов, но осуществляют функцию противоположную той, что выполняют их паралоги. Многие ЭР у растений представлены несколькими генами, что связано с характерной для растений задуплицированностью генома. Функция далеко не всех представителей исследована, и их изучение приносит сюрпризы. Так, например, семейство SUVH гистоновых H3K9-метилтрансфераз (SU(VAR)3-9 HOMOLOG) у арабидопсис представлено 9 паралогами, среди которых SUVH4/KYP, SUVH5 и SUVH6 действительно осуществляют репрессивное метилирование H3K9. Благодаря присутствию SRA-домена они распознают метилированную ДНК и метилируют гистоны в непосредственной близости от этих участков (Li et al., 2018). В то же время, SUVH1, SUVH3, SUVH7 и SUVH8 выполняют противоположную функцию. Они утратили метил-трансферазные домены, но за счет SRA-домена распознают метилированную ДНК в промоторной области ДНК-деметилазы/гликозилазы ROS1. Взаимодействуя с белками – активаторами транскрипции с DnaJ-доменом, белки SUVH1, SUVH3, SUVH7, SUVH8 активируют транскрипцию ROS1, а значит – и последующее деметилирование ДНК (Xiao et al., 2019). Примеров такой субнеофункциолизации генов, по-видимому, будет больше по мере углубления знаний о функции паралогов ЭР у растений.
В заключение раздела отметим, что результатом частичного репрограммирования генома под действиями окружающей среды может быть активация генов защитного ответа или/и изменение программы онтогенеза растения (его растущих модулей). Изменение программы развития того или иного модуля/органа реализуется на ранних этапах его онтогенеза, чаще всего – в клетках апикальных меристем побега и корня, которые содержат стволовые (плюрипотентные) клетки и дают начало всем примордиям органов. Однако, запуск (или перезапуск) программы развития у растений может наблюдаться даже в зрелых органах (Ежова, Куприянова, 2019). Это объясняется тем, что плюрипотентные клетки у растений могут появляться de novo за счет снятия эпигенетической репрессии с генов стволовости, которая в дифференцированных тканях поддерживается многочисленными ЭР (Альберт, Ежова, 2013; Gaillochet, Lohmann, 2015). Снятие эпигенетической репрессии с генов стволовости может обусловить развитие дополнительных лопастей и гребневидных выростов даже на зрелых листьях растений (Fedotov et al., 2017; Куприянова и др., 2021). Онтогенетические реверсии, которые вызываются погодными условиями, также отражают возможность перезапуска генетических прогамм развития за счет возабновления пула стволовых клеток (рис. 2).
Рис. 2.
Онтогенетические реверсии у календулы лекарственной Calendula officinalis (сорт, созданный В.В. Сахаровым в Институте биологии развития РАН имени Н.К. Кольцова) и гравилата речного Geum rivale в условиях влажного холодного лета 2019 г. На одном растении календулы наряду с соцветиями с нормальным онтогенезом (а) можно наблюдать соцветия, формирующие новые апикальные меристемы (б, в), которые также образуют соцветия и семена ((в) – внизу – корзинка 1-ого порядка с созревшими семенами, а вверху – образованные ею 15 дополнительных соцветий 2‑ого порядка, которые находятся на разных этапах онтогенеза). На одном растении гравилата с нормальным онтогенезом цветка (г) встречаются цветки, в центре которых формируются новые побеги с соцветиями (д, е). Фото автора.

ОСНОВНЫЕ ЭКСПЕРИМЕНТАЛЬНЫЕ ПОДХОДЫ ДЛЯ ИЗУЧЕНИЯ РОЛИ ЭПИГЕНЕТИЧЕСКИХ МОДИФИКАЦИЙ В ПОЛИВАРИАНТНОСТИ РАЗВИТИЯ
Пластичность развития растений проявляется в ярко выраженной вариабельности признаков у растений одного вида. Стохастические колебания уровня экспрессии, а также изменения экспрессии генов, вызванные внешними и внутренними факторами, изменяющие развитие, далеко не всегда связаны с ЭМ. Для доказательства причинно-следственной связи между ЭМ и фенотипом необходимо убедиться в отсутствии связи фенотипа с генетическим полиморфизмом и отличить временные изменения экспрессии генов, которые не сохраняются при делении клеток, от длительно сохраняющихся ЭМ. Существуют несколько подходов для изучения роли ЭМ в проявлении поливариантности развития, имеющих разную степень доказательности (табл. 1).
Таблица 1.
Экспериментальные подходы, которые используются для изучения роли эпигенетических модификаций в проявлении поливариантности развития растений
| Объекты | Методы | Возможности | |
|---|---|---|---|
| 1. Рекомбинантные имбредные линии epiRIL, полученные на едином генетическом фоне, среди которых есть фенотипические различия | 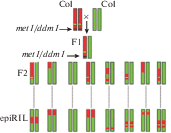 |
Анализ генома, метилома, транскриптома, генетическое картирование (epiQTL) | Выявление ДМУ/генов (эпиаллелей), обусловливающих фенотипические различия |
| 2. Клоны природных и культурных видов из разных эколого-географических зон; клоны, отличающиеся по фенотипу |  |
Анализ уровня и особенностей метилирования генома (MSAP и др. методы) | Демонстрация связи фенотипа с эпигенотипом |
| Анализ уровня генетической изменчивости (AFLP) | |||
| 3. Единичные растения, у которых разные модули или органы имеют фенотипические различия |  |
Те же, что и во 2-ом подходе | Выявление эпигенетического мозаицизма и его связи с фенотипическими различиями |
| 4. Популяции одного вида из разных эколого-географических зон, отличающиеся по фенотипу | 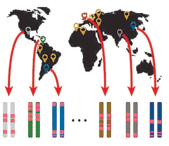 |
Те же, что и во 2-ом подходе (немодельные виды) | Демонстрация внутривидовых генетических и эпигенетических различий |
| Анализ генома, метилома, транскриптома, EWAS (Epigenome-Wide Association Studies, модельные виды) | Выявление ассоциаций фенотипа с ДМУ/генами | ||
Первый подход основан на изучении геномов, метиломов и транскриптомов у генетически идентичных рекомбинантных имбредных линий (epiRIL) арабидопсис (A. thaliana), отличающихся только эпигеномами. Получены epiRIL путем скрещивания растений экотипа Col c растениями того же экотипа, но с мутацией в одном из генов – регуляторов метилирования ДНК (Johannes et al., 2009; Reinders et al., 2009). Эти мутации приводят к снижению уровня метилирования генома – почти полному при мутации met1 и ∼70% при мутации ddm1, что вызывает изменение экспрессии генов и активацию мобильных элементов (Kakutani et al., 1995; Zhang et al., 2006; Lister et al., 2008). Самоопыление F1 и последующий имбридинг потомков F2 путем посева в каждом поколении всего одного семени привел к созданию линий, гомозиготных по дифференциально метилированным участкам генома (ДМУ), которые стабильно сохраняются в течение многих поколений. Растения F2 с мутацией (met1 или ddm1) убирают из популяции. Поэтому полученные в результате имбридинга (до 9 поколений) epiRIL в результате рекомбинаций имеют разные эпигеномы, но практически идентичные геномы. Немногочисленные генетические изменения, возникающие в результате спонтанных мутаций и активации мобильных элементов на фоне общего снижения уровня метилирования ДНК (Miura et al., 2001; Mirouze et al., 2009; Marí-Ordóñez et al., 2013), выявляют секвенированием генома. Благодаря рекомбинации хромосом, которая позволяет использовать принципы генетического картирования, выявляют ДМУ генома, которые обусловливают фенотипические различия между линиями, изменяя экспрессию генов. Для этого линии группируют по фенотипическим показателям (например, линии рано- и поздноцветущие) и сравнивают группы по распределению ДМУ в геномах разных групп epiRIL. Как правило, для одного признака обнаруживаются несколько ДМУ из разных хромосом (как при картировании локусов количественных признаков QTL). Анализ таких epiQTL, определение положения 5mC относительно рядом локализованных генов, вместе с анализом генома и транскриптома может обнаружить эпиаллели, которые изменяют уровень транскрипцию из-за ЭМ и обусловливают фенотипические различия между линиями.
Многочисленные эксперименты с epiRIL показывают, что стабильные ЭМ обусловливают различия по морфологическим признакам (Johannes et al., 2009; Roux et al., 2011; Zhang et al., 2013; Cortijo et al., 2014), продуктивности (Latzel et al., 2013), устойчивости растений к разнообразным стрессовым воздействиям, включая патогены (Zhang et al., 2013; Latzel et al., 2013; Furci et al., 2019; Liégard et al., 2019), по реакции на экзогенные гормоны (Latzel et al., 2012) и по содержанию метаболитов (Kooke et al., 2019).
Эксперименты с epiRIL доказали, что ЭМ могут быть важным источником фенотипических изменений. Однако сегодня этот подход практически исчерпал себя: морфологические изменения между разными имбредными линиями не велики, и исследования большинства признаков уже проведено. Поскольку уровень и спектр ЭМ определяется не столько внешними воздействиями, сколько генетическими различиями (аллельным составом генов – эпигенетических регуляторов), создание epiRIL на основе разных экотипов могло бы расширить фенотипическое разнообразие линий и возможности анализа. Однако у арабидопсис есть и еще одно важное ограничение (Alonso et al., 2015). Растение имеет компактный геном с небольшой долей повторяющихся последовательностей и с самым низким средним уровнем метилирования генома (5% 5mC) по сравнению с другими покрытосеменными растениями (у риса и кукурузы % 5mC составляет 16 и 27%). Следовательно, этот объект далеко не идеален для изучения роли ЭМ в фенотипической изменчивости, хотя именно в исследованиях на арабидопсис открыты все основные гены – ЭР.
Второй подход базируется на исследованиях немодельных видов растений, которые имеют клоновое происхождение (клоны инвазивных видов, клоны культурных растений), что также гарантирует их генетическую идентичность, но не исключает эпигенетические различия. Для подтверждения связи фенотипа с ЭМ анализируют уровень и особенности метилирования генома. Чаще всего используют MSAP-анализ (methylation-sensitive amplified fragment length polymorphism), т.е. анализируют спектр амплифицированных фрагментов ДНК, полученных с использованием пары рестриктаз (часто HpaII и MspI), которые узнают одну последовательность, но обладают разной чувствительностью к присутствию 5mC (Reyna-Lopez et al., 1997). В отдельных случаях используют высокоразрешающую жидкостную хроматографию гидролизованной ДНК (HPLC), иммуноцитохимический анализ хроматина на присутствие 5mC, полногеномное бисульфитное секвенирование, а также другие методы (Kurdyukov, Bullock, 2016).
Результаты большинства исследований, проведенных на клонах инвазивных растений, а также на успешных видах, сочетающих вегетативное размножение с половым, подтвердили, что у таких видов уровень эпигенетической изменчивости существенно превышает генетический и является важным (Gao et al., 2010; Richards et al., 2012; Spens, Douhovnikoff, 2016; Guarino et al., 2019), а иногда и практически единственным источником адаптивной изменчивости (Shi et al., 2019). Эти данные свидетельствуют о том, что адаптивные эпигенетические изменения, которые сохраняются при вегетативном размножении (Rendina González et al., 2018; Shi et al., 2019), обеспечивают дивергенцию и успешную экспансию многих инвазивных видов в разные эколого-географические зоны (Verhoeven, Preite, 2014; Douhovnikoff, Dodd, 2015; Latzel et al., 2016; Münzbergová et al., 2019). Сходные результаты получены в исследованиях растений пирамидального тополя и банана, которые имеют единое клоновое происхождение, но культивируются в разных эколого-географических условиях (Vanden Broeck et al., 2018; Kitavi et al., 2020). В исследованиях паттерна метилирования ДНК восточно-африканских сортов банана, различавшихся по морфологическим признакам, показано сохранение части MSAP-маркеров метилирования ДНК и при половом размножении (Kitavi et al., 2020).
Сегодня много экспериментальных работ и обзоров посвящены анализу роли эпигенетической памяти в устойчивости растений к биотическим и абиотическим стрессорам (Ashapkin et al., 2020; Ueda, Seki, 2020; Oberkofler et al., 2021; Srikant, Drost, 2021). Онтогенетическим признакам в этих исследованиях уделяется мало внимания. Однако есть экспериментальные работы, выполненные на будре (Quan et al., 2021) Glechoma longituba (Lamiaceae) и землянике Fragaria vesca (Rosaceae) (Latzel, Münzbergová, 2018), в которых исследовали число, общую массу, площадь листа и, самое интересное – направление роста вегетативных побегов после воздействия на материнское растение разных условий освещения (света, затемнения, УФ-облучения) и питания (обычный субстрат и обогащенный NH4, NO3, P, K, Mg, Ca) или сочетания разных воздействий. Наблюдая за развитием рамет (вегетативных побегов, которые должны дать начало новому организму), авторы пытались узнать, способны ли растения при формировании рамет “учитывать” прошлый опыт и формировать раметы в том направлении, которое оптимально для развития потомков.
Эта работа и другие иследования “интеллекта растений” подвергаются критике – статистические данные экспериментов и их интерпретация не всегда убедительны (Mallatt et al., 2021). Тем не менее, исследование таких клоновых систем очень интересно. Формируя сложные сети из десятков и сотен взаимодействующих полуавтономных рамет, клоны могут оказываться эпигенетическими мозаиками (González et al., 2016). Такой факультативный эпигенетический мозаицизм (связанный не с направлением дифференцировки тканей, а с действием условий среды) может возникать из-за того, что разные части сети могут оказываться в разных условиях и иметь разный эпигенотип, адаптированный к определенному воздействию. Эксперименты на таких клонах могут оценить роль факультативного эпигенетического мозаицизма в поддержании жизнеспособности всего клона и исследовать возможность обмена эпигенетической информацией разными частями клона. В качестве мобильных эффекторов ЭМ могут рассматриваться малые РНК (miРНК и siРНК), участвующие в РНК-зависимом метилировании ДНК и способные передвигаться между побегом и корнем одного растения по плазмодесмам и по флоэме (Tamiru et al., 2018; Pagliarani, Gambino, 2019).
Третий основан на изучении единичных растений. Анализируют изменения онтогенеза органов при разных воздействиях или анализируют разные части одного растения, которые находятся под воздействием разных микроусловий. Исследования показали, что факультативный эпигенетический мозаицизм выявляется не только у вегетативно размножающихся растений. Различия по уровню и особенностям метилирования ДНК обнаружены и между разными частями отдельных растений дикой лаванды Lavandula latifolia (Lamiaceae) (Alonso et al., 2018; Herrera et al., 2021), падуба остроли́стного Ilex aquifolium (Aquifoliaceae) (Herrera, Bazaga, 2013), тополя волосистоплодного Populus trichocarpa (Salicaceae) (Hofmeister et al., 2020). Хотя различия не велики (между ветвями лаванды различие было по 6–8% MSAP-маркеров по данным Herrera et al., 2021), они свидетельствуют об относительной автономности разных частей многолетних растений. Эти различия могут являться следствием разного воздействия окружающих условий (доступность света, повреждение патогенами, животными и пр.) и зависеть от пространственно-временных характеристик развития того или иного модуля.
Яркое свидетельство связи факультативного эпигенетического мозаицизма и морфогенеза листа получено при изучении многолетних древесных растений падуба остролистого Ilex aquifolium (Herrera, Bazaga, 2013). Для падуба характерна гетерофилия – наличие на одном растении как простых цельнокрайных, так и лопастных листьев с шипами по краю (колючие). Причем ветки растений, которые объедаются оленями и козами, гораздо чаще содержат колючие листья (76% веток), чем ветки неповрежденных деревьев (19%). Кроме того, чем ниже ветки расположены, тем чаще они содержат колючие листья (rs = –0.698), что авторы объясняют большей досягаемостью таких веток для животных, т.е. развитие шипов на листьях – это ответ растения на повреждение растений животными (Herrera, Bazaga, 2013). MSAP-анализ листьев разной морфологии с одних и тех же веток показал, что геном цельнокрайних листьев метилирован сильнее, чем колючих листьев. Эти результаты свидетельствуют о том, что ЭМ (в данном случае – изменения уровня метилирования генома) могут обусловить адаптивные изменения программы онтогенеза листьев. Они также показывают, что факультативный эпигенетический мозаицизм может становиться основным фактором вариации морфологии модулей в пределах одного растения и, возможно, фактором эпигенетического разнообразия потомков одного растения (Herrera, 2017). В отличие от конститутивного эпигенетического мозаицизма, который связан с реализацией генетических программ развития вида, с поддержанием идентичности органов и тканей внутри растения, факультативный эпигенетический мозаицизм отражает возможность адаптивного изменения этих программ в отдельных модулях растения в ответ на воздействие внешних условий.
Гетерофилия особенно распространена среди водных растений. Листья, расположенные над водой и под водой, могут качественно различаться по форме, анатомическим и функциональным особенностям. Погруженные в воду листья как правило более рассеченные, чем надводные. Исследования более 20 видов растений с гетерофилией выявили основные внешние факторы (погруженность, температура) и внутренние факторы (гормоны), определяющие тип формирующегося листа (обзоры Nakayama et al., 2017; Li et al., 2019, 2021; van Veen, Sasidharan, 2021). Одно из первых исследований связи уровня метилирования ДНК с гетерофилией выполнено на водяном орехе Trapa natans (Lythraceae), имеющем сильно рассеченные подводные листья. С использованием иммуно-цитохимического определения 5mC показан более низкий уровень метилирования ДНК в почках, дающих начало рассеченным листьям по сравнению с почками, из которых формируются цельнокрайние листья надводной части (Bitonti et al., 1996). Эти результаты сходны с результатами исследований колючих листьев падуба (Herrera, Bazaga, 2013) и также указывает на существововании факультативного эпигенетического мозаицизма и его роль в проявлении гетерофилии.
Рассмотренные примеры показывают, что 2-ой и 3-ий подходы, в отличие от 1-ого, не позволяет выявить те конкретные дифференциально метилированные участки (ДМУ) генома, которые обусловливают изменения фенотипа. Однако по мере углубления знаний о генетическом контроле онтогенеза будут появляться новые возможности, связанные с тестированием уровня экспрессии и профиля метилирования не всего генома, а ключевых консервативных генов, изменения работы которых потенциально могут обусловливать похожий фенотип. Примером таких исследований является анализ влияния водного дефицита на развитие устьиц на листьях растений арабидопсис (Tricker et al., 2012). Показано, что в этих условиях наблюдается гиперметилирование гена SPCH, который контролирует развитие устьиц. Это приводило к уменьшению их числа на листе. Высокий уровень метилирования ДНК гена SPCH и сниженное число устьиц наследовался потомкам (Tricker et al., 2013).
Одной из наиболее полно изученных и консервативных генетических программ сегодня является программа развития листа (Fritz et al., 2018; Satterlee, Scanlon, 2019).) В этой связи, особенно хорошие перспективы открываются для изучения генетических и эпигенетических основ гетерофилии. Анализ экспрессии генов, играющих центральную роль в консервативных генетических программах развития листа, был проведен на 2 видах гетерофильных растений (Nakayama et al., 2014; Li et al., 2017; Kim et al., 2018). У растений Rorippa aquatica (Brassicaceae) (Nakayama et al., 2014) и Hygrophila difformis (Acanthaceae) (Li et al., 2017) рассеченность погруженных листьев оказалась результатом активации в примордиях листьев высококонсервативных генов, которые поддерживают плюрипотентность клеток листа. Это гены класса KNOX I, экспрессия которых поддерживает деление клеток и определяет возможность усложнения структуры листа (Blein et al., 2008). У обоих видов эти гены не работают в надводных цельнокрайних листьях, но экспрессируются при развитии рассеченных (Nakayama et al., 2014; Li et al., 2017). Хотя в этих работах нет анализа метилированиия ДНК или модификации гистонов в листьях разного типа, можно предполагать, что именно эпигенетическая регуляция KNOX I-генов под влиянием условий среды лежит в основе гетерофилии. Гены KNOX I – известная мишень эпигенетической регуляции (Lodha et al., 2008; Gaillochet, Lohmann, 2015). Поэтому, нет сомнений, что в регуляторных участках KNOX I- генов этих видов можно выявить репрессивные метки в надводных листьев и их отсутствие – в погруженных. В этих исследованиях показано, что главным внешним фактором, вызывающим изменение морфологии листьев, является температура. Понижение температуры с 26 до 20°С достаточно, чтобы растение начинало формировать рассеченные листья вместо цельных. Поэтому R. aquatica и H. difformis являются идеальными моделями для будущих исследований динамики эпигенетических модификаций в KNOX I-генах при изменении внешних условий. Возможно, именно эти модели позволят расшифровать последовательность молекулярных событий, которые запускаются внешними воздействиями и прямо или опосредованно активируют или блокируют работу генов, вовлеченных в эпигенетическую регуляцию генов KNOX I.
Отметим, что помимо гетерофилии у растений есть и другие примеры качественных разлий морфологии органов у одного вида или у одного и того же растения – полигамия, гетерокарпия, диаспория. Изучению генетических и эпигенетических основ полигамии у растений будет посвящен отдельный обзор. Работ по изучению роли ЭМ в проявлении гетерокарпии и диаспории автору обнаружить не удалось.
Четвертый подход основан на анализе внутривидового фенотипического полиморфизма. При работе с немодельными видами определить истинные причины фенотипических различий сложно из-за наличия внутривидового генетического полиморфизма. Поэтому упомянем лишь одну из работ, которая выполнена на мангровых деревьях Laguncularia racemosa (Combretaceae), произрастающих в приливно-отливной полосе морских побережий и устьях рек (Lira-Medeiros et al., 2010). Растения с участков с высоким засолением отличаются от тех, что растут в благоприятных условиях по многим морфологическим показателям, среди которых самые яркие различия наблюдались по высоте (соответственно, до 3 и до 40 м). Анализ генетической (AFLP-анализ – amplified fragment length polymorphism) и эпигенетической (MSAP-анализ) изменчивости показал, что популяции растений с разных участков различаются как по генетическим, так и по эпигенетическим маркерам, хотя уровень изменчивости между популяциями по эпигенетическим маркерам был выше. Кроме того, выявлен более низкий уровень метилирования ДНК у растений с засоленных участков по сравнению с растущими в более благоприятных условиях (Lira-Medeiros et al., 2010). Хотя эти данные указывают на возможную важную роль ЭМ в адаптации растений к разным условиям существования, они не доказывают существование связи между фенотипом и выявленными изменениями уровня метилирования ДНК.
Для подтверждения связи фенотипических различий с ЭМ анализируют фенотипические изменения между линиями на фоне использования ингибиторов метилирования ДНК 5-азацитидина или зебуралина (см. например, Alonso et al., 2017; Münzbergová et al., 2018; Машкина, Табацкая, 2020). При исчезновении или сглаживании фенотипических различий делается заключение об участии эпигенетических механизмов в их появлении в исходных популяциях. Однако действие этих ингибиторов может нарушать развитие растений и затруднять трактовку результатов. Таким образом, исследования, выполненные на природных (не клоновых) популяциях, в которых всегда имеется генетический полиморфизм, не могут исключить его определяющего влияния на любые фенотипические признаки.
Эту проблему помогают решить постоянно расширяющиеся возможности широкомасштабного анализа геномов, метиломов и транскриптомов. Сегодня они успешно реализуются не только на арабидопсис, но и на дикорастущих и культурных растениях с использованием разных подходов (обзор Gahlaut et al., 2020). В исследованиях на образцах дикорастущих и коллекционных растений, на сортах и имбредных линиях культурных растений выявлены ДМУ генома, ассоциированные с определенным фенотипическим признаком, а в некоторых случаях – выявлены гены-кандидаты, которые локализованы рядом с этими ДМУ и изменяют свою экспрессию (Ha et al., 2020; Lu et al., 2020).
ЗАКЛЮЧЕНИЕ
Благодаря использованию разнообразных подходов для изучения роли эпигенетических механизмов в поливариантности развития растений сегодня экспериментально доказаны следующие положения: (1) условия среды произрастания, стрессовые воздействия вызывают изменение эпигенома; часть возникающих ЭМ адаптивна; (2) эпигенетические изменения наследуются при сохранении условий, вызвавших изменения; примерно половина ЭМ стабильно передается потомкам в соответствие с законами Менделя; (3) при изменении условий прежние ЭМ постепенно исчезают, хотя уровень стабильности разных ЭМ различен; (4) внутривидовая морфологическая изменчивость растений обусловлена не только генетическим полиморфизмом, но и эпигенетическими различиями между растениями; (5) многие морфологические и физиолого-биохимические признаки, отличающие разные части одного растения – результат факультативного эпигенетического мозаицизма, который возникает из-за различий микроусловий, воздействующих на разные части растения.
Эти положения указывают на существование у растений уникальных особенностей функционирования эпигенетических механизмов. Действительно, работа ЭР у растений находится под контролем условий окружающей среды. Воздействие окружающей среды может быть не специфическим (за счет снижения уровня доноров метильных групп) и специфическим, основанном на активации экспрессии отдельных генов – ЭР и их воздействии на определенные гены-мишени. Эти и другие рассмотренные в обзоре механизмы могут обусловливать изменение экспрессии определенных групп генов и объяснять адаптивность ЭМ. Сохранение адаптивных ЭМ у потомков обеспечивается поддерживающими метилтрансферазами и положительными обратными взаимодействиями между разными эпигенетическими метками. На работу уже известных ЭР могут влиять и недавно открытые специфические для растений гены. Продукты этих генов могут либо нарушать, либо активировать работу ЭР. Нет сомнения, что у растений будут открыты и новые ЭР, влияющие на динамику ЭМ, поскольку даже у арабидопсис до сих пор не известна функция ~30% генов ядерного генома.
Исследований эпигенетических основ внутривидовой фенотипической изменчивости становится все больше, однако даже при “омиксном” исполнении, требуются дополнительные подтверждения того, что выявленные ассоциации между фенотипом и эпиаллелями генов-кандидатов обусловлены причинно-следственными связями. Сегодня разрабатываются новые подходы для таких подтверждений. Используя специальные системы для эпигенетического редактирования геномов, можно с их помощью привлекать белки – ЭР к генам-кандидатам, и изучать влияние этих модификаций на фенотип (Gallego-Bartolomé et al., 2020; Fal et al., 2021; Selma, Orzáez, 2021). Омиксные подходы и биотехнологии направленного управления эпигенетическими процессами открывают новые перспективы в изучении научных основ пластичности развития растений и практическом использовании наследуемых ЭМ.
Список литературы
Альберт Е.B., Ежова Т.А. Стволовые клетки побега растений – генетическая регуляция // Генетика. 2013. Т. 49. № 2. С. 149–163.
Ванюшин Б.Ф. Метилирование ДНК и эпигенетика // Генетика. 2006. Т. 42. № 9. С. 1186–1199.
Ежова Т.А., Куприянова Е.В. Изучение роли ауксина в регуляции развития эктопических выростов на листьях мутанта taeniata Arabidopsis thaliana // Онтогенез. 2019. Т. 50. С. 312–318.
Жукова Л.А., Глотов Н.В. Морфологическая поливариантность онтогенеза в природных популяциях растений // Онтогенез. 2001. № 6. С. 455–461.
Калинкина В.А. Поливариантность онтогенеза представителей рода Trifolium L. секции Lupinaster (Fabr.) Ser. // Онтогенез. 2017. Т. 48. № 2. С. 172–180.
Кольцов Н.К. Взгляды Лотси на эволюцию организмов // Природа. 1915. № 10. С. 1253. (Цитировано по Раменский, 2018).
Куприянова Е.В., Денисов Е.Р., Байер М.А., Ежова Т.А. Различия в проявлении плюрипотентности клеток in vivo и in vitro у мутанта Arabidopsis thaliana с фенотипом нарушения клеточной памяти // Физиология растений. 2021. Т. 68. № 1. С. 53–62.
Машкина О.С., Табацкая Т.М. Морфогенез рассеченного листа березы в культуре in vitro // Онтогенез. 2020. Т. 51. № 6. С. 460–472.
Нотов А.А., Жукова Л.А. Концепция поливариантности онтогенеза и современная эволюционная морфология // Известия РАН, серия биологическая. 2019. № 1. С. 1–9.
Разин С.В., Быстрицкий А.А. Хроматин: упакованный геном. М.: Бином. Лаборатория знаний, 2009. 176 с.
Раменский Е.В. Эпигенетика: Уоддингтон или Кольцов? // Онтогенез. 2018, Т. 49. № 6. С. 391–396.
Серебряков И.Г., Серебрякова Т.И. Некоторые вопросы эволюции жизненных форм цветковых растений // Ботан. журн. 1972. Т. 57. № 5. С. 417–433.
Тиходеев О.Н. Эпигенетические и эугенетические процессы // Успехи современной биологии. 2015. Т. 135. № 6. С. 542–553.
Эллис С.Д., Дженювейн Т., Рейнберг Д. Эпигенетика. Издательство Техносфера. 2010. 496 с.
Aichinger E., Villar C.B.R., Farrona S. et al. CHD3 proteins and polycomb group proteins antagonistically determine cell identity in Arabidopsis // PLoS Genet. 2009. V. 5. P. e1000605.
Allis C.D., Jenuwein T. The molecular hallmarks of epigenetic control // Nature Rev. Genet. 2016. V. 17. № 8. P. 487–500.
Alonso C., Medrano M., Pérez R. et al. Tissue-specific response to experimental demethylation at seed germination in the non-model herb Erodium cicutarium // Epigenomes. 2017. V. 1. P. 16.
Alonso C., Pérez R., Bazaga P. et al. Within-plant variation in seed size and inflorescence fecundity is associated with epigenetic mosaicism in the shrub Lavandula latifolia (Lamiaceae) // Annals of Botany. 2018. V. 121. № 1. P. 153–160.
Alonso C., Pérez R., Bazaga P., Herrera C.M. Global DNA cytosine methylation as an evolving trait: phylogenetic signal and correlated evolution with genome size in angiosperms // Front. Genet. 2015. V. 6. P. 4.
Ashapkin V.V., Kutueva L.I., Aleksandrushkina N.I., Vanyu-shin B.F. Epigenetic mechanisms of plant adaptation to biotic and abiotic stresses // Int. J. Mol. Sci. 2020. V. 21. № 20. P. 7457.
Baubec T., Dinh H.Q., Pecinka A. et al. Cooperation of multiple chromatin modifications can generate unanticipated stability of epigenetic states in Arabidopsis // Plant Cell. 2010. V. 22. P. 34–47.
Berger S.L. The complex language of chromatin regulation during transcription // Nature. 2007. V. 447. P. 407–412.
Bernatavichute Y.V., Zhang X., Cokus S. et al. Genomewide association of histone H3 lysine nine methylation with CHG DNA methylation in Arabidopsis thaliana // PLoS One. 2008. V. 3. P. e3156.
Birnbaum K.D., Roudier F. Epigenetic memory and cell fate reprogramming in plants // Regeneration. 2016. V. 4. P. 15–20.
Blein T., Pulido A., Vialette-Guiraud A. et al. A conserved molecular framework for compound leaf development // Science. 2008. V. 322. № 5909. P. 1835–1839.
Bloomer R.H., Hutchison C.E., Bäurle I. et al. The Arabidopsis epigenetic regulator ICU11 as an accessory protein of Polycomb Repressive Complex 2 // PNAS. 2020. V. 117. № 28. P. 16660–16666.
Bratzel F., López-Torrejón G., Koch M. et al. Keeping cell identity in Arabidopsis requires PRC1 RING-finger homologs that catalyze H2A monoubiquitination // Curr. Biol. 2010. V. 26. № 20. P. 1853–1859.
Bitonti M.B., Cozza R., Wang G. et al. Nuclear and genomic changes in floating and submerged buds and leaves of heterophyllous waterchestnut (Trapa natans) // Physiologia Plantarum. 1996. V. 97. № 1. P. 21–27.
Bondada R., Somasundaram S., Marimuthu M.P. et al. Natural epialleles of Arabidopsis SUPERMAN display superwoman phenotypes // Commun. Biol. 2020. V. 3. № 1. P. 772.
Bowman J.L., Sakai H., Jack T. et al. SUPERMAN, a regulator of floral homeotic genes in Arabidopsis // Development. 1992. V. 114. P. 599–615.
Bräutigam K., Cronk Q. DNA Methylation and the evolution of developmental complexity in plants // Front. Plant Sci. 2018. V. 9. 1447.
Catoni M., Griffiths J., Becker C. et al. DNA sequence properties that predict susceptibility to epiallelic switching // EMBO J. 2017. V. 36. P. 617–628.
Charbonnel C., Niazi A.K., Elvira-Matelot E. et al. The siRNA suppressor RTL1 is redox-regulated through glutathionylation of a conserved cysteine in the double-stranded-RNA-binding domain // Nucleic Acids Res. 2017. V. 45. P. 11891–11907.
Chen J., Liu J., Jiang J. et al. F-box protein CFK1 interacts with and degrades de novo DNA methyltransferase in Arabidopsis // New Phytol. 2021. V. 229. № 6. P. 3303–3317.
Chen D., Molito A., Liu C. et al. The Arabidopsis PRC1-like ring-finger proteins are necessary for repression of embryonic traits during vegetative growth // Cell Res. 2010. V. 20. P. 1332–1344.
Chen X., Ding A.B., Zhong X. Functions and mechanisms of plant histone deacetylases // Sci. China Life Sci. 2020. V. 63. P. 206–216.
Colicchio J., Kelly J., Hileman L. Mimulus sRNAs Are wound responsive and associated with transgenerationally plastic genes but rarely both // Int. J. Mol. Sci. 2020. V. 21. № 20. P. 7552.
Cortijo S., Wardenaar R., Colomé-Tatché M. et al. Mapping the epigenetic basis of complex traits // Science. 2014. V. 343. P. 1145–1148.
Couturier J., Chibani K., Jacquot J.P., Rouhier N. Cysteine-based redox regulation and signaling in plants // Front. Plant Sci. 2013. V. 4. P. 105.
Cubas P., Vincent C., Coen E. An epigenetic mutation responsible for natural variation in floral symmetry // Nature. 1999. V. 401. P. 157–161.
Derkacheva M., Hennig L. Variations on a theme: Polycomb group proteins in plants // J. Exp. Botany. 2014. V. 65. № 10. P. 2769–2784.
Douhovnikoff V., Dodd R.S. Epigenetics: a potential mechanism for clonal plant success // Plant Ecology. 2015. V. 216. P. 227–233.
Du J., Johnson L.M., Jacobsen S.E., Patel D.J. DNA methylation pathways and their crosstalk with histone methylation // Nature Rev. Mol. Cell Biol. 2015. V. 16. P. 519–532.
Duan C.G., Wang X., Zhang L. et al. A protein complex regulates RNA processing of intronic heterochromatin-containing genes in Arabidopsis // PNAS. 2017. V. 114. № 35. P. E7377–E7384.
Duan C.G., Wang X.G., Tang K. et al. MET18 connects the cytosolic iron-sulfur cluster assembly pathway to active DNA demethylation in Arabidopsis // PLoS Genet. 2015. V. 11. P. e1005559.
Duan C.G., Wang X., Xie S. et al. A pair of trans poson-derived proteins function in a histone acetyltransferase complex for active DNA demethylation // Cell Res. 2016. V. 27. № 2. P. 226–240.
Duempelmann L., Skribbe M., Bühler M. Small RNAs in the transgenerational inheritance of epigenetic information // Trends Genet. 2020. V. 36. P. 203–214.
Erdmann R.M., Picard C.L. RNA-directed DNA methylation // PLoS Genet. 2020. V. 16. № 10. P. e1009034.
Fal K., Tomkova D., Vachon G. et al. Chromatin manipulation and editing: Challenges, new technologies and their use in plants // Int. J. Mol. Sci. 2021. V. 22. № 2. P. 512.
Fedotov A.P., Ezhova T.A., Timonin A.C. Bizarre lamina margins in tae mutant of Arabidopsis thaliana (L.) Heynh. (Brassicaceae) // Wulfenia. 2017. V. 24. P. 163–170.
Fenley A.T., Anandakrishnan R., Kidane Y.H., Onufriev A.V. Modulation of nucleosomal DNA accessibility via chargealtering post-translational modifications in histone core // Epigenet Chromatin. 2018. V. 11. № 1. P. 11.
Fletcher J.C. State of the art: trxG factor regulation of post-embryonic plant development // Front. Plant Sci. 2017. V. 8. P. 1925.
Förderer A., Zhou Y., Turck F. The age of multiplexity: recruitment and interactions of Polycomb complexes in plants // Curr. Opin. Plant Biol. 2016. V. 29. P. 169–178.
Foyer C.H., Noctor G. Stress-triggered redox signalling: what’s in pROSpect? // Plant, Cell and Environment. 2016. V. 39. P. 951–964.
Fritz M.A., Rosa S., Sicard A. Mechanisms underlying the environmentally induced plasticity of leaf morphology // Front. Genet. 2018. V. 9. P. 478.
Fultz D., Choudury S.G., Slotkin R.K. Silencing of active transposable elements in plants // Curr. Opin. Plant Biol. 2015. V. 27. P. 67–76.
Furci L., Jain R., Stassen J. et al. Identification and characterisation of hypomethylated DNA loci controlling quantitative resistance in Arabidopsis // eLife. 2019. V. 8. P. e40655.
Gahlaut V., Zinta G., Jaiswal V., Kumar S. Quantitative epigenetics: A new avenue for crop improvement // Epigenomes. 2020. V. 4. № 4. P. 25.
Gaillochet C., Lohmann J.U. The never-ending story: from pluripotency to plant developmental plasticity // Development. 2015. V. 142. № 13. P. 2237–2249.
Gallego-Bartolomé J., Liu W., Kuo P.H. et al. Co-targeting RNA polymerases IV and V promotes efficient de novo DNA methylation in Arabidopsis // Cell. 2019. V. 176. № 5. P. 1068–1082.
Gallego-Bartolomé J. DNA methylation in plants: mechanisms and tools for targeted manipulation // New Phytologist. 2020. V. 227. P. 38–44.
Gao L.X., Geng Y.P., Li B. et al. Genome-wide DNA methylation alterations of Alternanthera philoxeroides in natural and manipulated habitats: implications for epigenetic regulation of rapid responses to environmental fluctuation and phenotypic variation // Plant Cell and Environment. 2010. V. 33. P. 1820–1827.
González R.A.P., Chrtek J., Dobrev P.I. et al. Stress-induced memory alters growth of clonal off spring of white clover (Trifolium repens) // Am. J. Bot. 2016. V. 103. P. 1567–1574.
Gouil Q., Baulcombe D.C. DNA methylation signatures of the plant chromomethyltransferases // PLoS Genet. 2016. V. 12. № 12. P. e1006526.
Grimanelli D., Ingouff M. DNA methylation readers in plants // J. Mol. Biol. 2020. V. 432. P. 1706–1717.
Guarino F., Heinze B., Castiglione S., Cicatelli A. Epigenetic analysis through MSAP-NGS coupled technology: the case study of white poplar monoclonal populations/stands // Int. J. Mol. Sci. 2020. V. 21. № 19. P. 7393.
Guo X., Xie Q., Li B., Su H. Molecular characterization and transcription analysis of DNA methyltransferase genes in tomato (Solanum lycopersicum) // Genet. Mol. Biol. 2020. V. 43. № 1. P. e20180295.
Ha J., Kwon H., Cho K.H. et al. Identification of epigenetic variation associated with synchronous pod maturity in mungbean (Vigna radiata L.) // Sci. Rep. 2020. V. 10. № 1. P. 17414.
Hallé F. Modular growth in seed plants // Phil. Trans. R. Soc. Lond. B. 1986. V. 313. P. 77–88.
Handa V., Jeltsch A. Profound flanking sequence preference of Dnmt3a and Dnmt3b mammalian DNA methyltransferases shape the human epigenome // J. Mol. Biol. 2005. V. 348. P. 1103–1112.
Harris C.J., Scheibe M., Wongpalee S.P. et al. A DNA methylation reader complex that enhances gene transcription // Science. 2018. V. 362. P. 1182–1186.
Harvey Z.H., Chen Y., Jarosz D.F. Protein-based inheritance: epigenetics beyond the chromosome // Mol. Cell. 2018. V. 69. P. 195–202.
Hasegawa J., Sakamoto T., Fujimoto S. et al. Auxin decreases chromatin accessibility through the TIR1/AFBs auxin signaling pathway in proliferative cells // Sci. Rep. 2018. V. 8. P. 7773.
Hauser M.T., Aufsatz W., Jonak C., Luschnig C. Transgenerational epigenetic inheritance in plants // Biochimica Et Biophysica Acta. 2011. V. 1809. P. 459–468.
He Y., Li Z. Epigenetic environmental memories in plants: establishment, maintenance, and reprogramming // Trends Genet. 2018. V. 34. P. 856–866.
Heard E., Martienssen R.A. Transgenerational epigenetic inheritance: myths and mechanisms // Cell. 2014. V. 157. P. 95–109.
Henderson I.R., Jacobsen S.E. Epigenetic inheritance in plants // Nature. 2007. V. 447. P. 418–424.
Herms D.A., Mattson W.J. The dilemma of plants: to grow or defend // Q. Rev. Biol. 1992. V. 67. P. 283–335.
Herrera C.M. The ecology of subindividual variability in plants: Patterns, processes, and prospects // Web Ecology. 2017. V. 17. № 2. P. 51–64.
Herrera C. M., Bazaga P. Epigenetic correlates of plant phenotypic plasticity: DNA methylation differs between prickly and nonprickly leaves in heterophyllous Ilex aquifolium (Aquifoliaceae) trees // Bot. J. Linn. Soc. 2013. V. 171. P. 441–452.
Herrera C.M., Bazaga P., Pérez R., Alonso C. Lifetime genealogical divergence within plants leads to epigenetic mosaicism in the shrub Lavandula latifolia (Lamiaceae) // New Phytol. 2021.
Hofmeister B.T., Denkena J., Colomé-Tatché M. et al. A genome assembly and the somatic genetic and epigenetic mutation rate in a wild long-lived perennial Populus trichocarpa // Genome Biol. 2020. V. 21. P. 259.
Hofmeister B.T., Lee K., Rohr N.A. et al. Stable inheritance of DNA methylation allows creation of epigenotype maps and the study of epiallele inheritance patterns in the absence of genetic variation // Genome Biol. 2017. V. 18. № 1. P. 155.
Huot B., Yao J., Montgomery B.L., He S.Y. Growth-defense tradeoffs in plants: a balancing act to optimize fitness // Molecular Plant. 2014. V. 7. № 8. P. 1267–1287.
Irish V.F. Cell lineage in plant development // Curr. Opin. Cell Biol. 1991. V. 3. P. 983–987.
Jablonka E., Raz G. Transgenerational epigenetic inheritance: prevalence, mechanisms, and implications for the study of heredity and evolution // Q. Rev. Biol. 2009. V. 84. P. 131–176.
Jacobsen S.E., Meyerowitz E.M. Hypermethylated SUPERMAN epigenetic alleles in Arabidopsis // Science. 1997. V. 277. № 5329. P. 1100–1103.
Jmii S., Cappadocia L. Plant SUMO E3 ligases: Function, structural organization, and connection with DNA // Front. Plant Sci. 2021. V. 12. P. 652170.
Johannes F., Porcher E., Teixeira F.K. et al. Assessing the impact of transgenerational epigenetic variation on complex traits // PLoS Genet. 2009. V. 5. P. e1000530.
Johannes F., Schmitz R.J. Spontaneous epimutations in plants // New Phytol. 2019. V. 221. P. 1253–1259.
Kakutani T. Epi-alleles in plants: Inheritance of epigenetic information over generations // Plant Cell Physiol. 2002. V. 43. Iss. 10. P. 1106–1111.
Kakutani T., Jeddeloh J.A., Richards E.J. Characterization of an Arabidopsis thaliana DNA hypomethylation mutant // Nucleic Acids Res. 1995. V. 23. P. 130–137.
Kim J., Joo Y., Kyung J. et al. A molecular basis behind heterophylly in an amphibious plant // PLoS Genet. 2018. V. 14. № 2. P. e1007208.
Kim D.H., Xi Y., Sung S. Modular function of long noncoding RNA, COLDAIR, in the vernalization response // PLoS Genet. 2017. V. 13. P. 1–18.
Kishimoto N., Sakai H., Jackson J. et al. Site specificity of the Arabidopsis METI DNA methyltransferase demonstrated through hypermethylation of the superman locus // Plant Mol. Biol. 2001. V. 46. № 2. P. 171–183.
Kitavi M., Cashell R., Ferguson M. et al. Heritable epigenetic diversity for conservation and utilization of epigenetic germplasm resources of clonal East African Highland banana (EAHB) accessions // Theor. Appl. Genet. 2020. V. 133. P. 2605–2625.
Kong X., Hong Y., Hsu Yi-F. et al. SIZ1-Mediated SUMOylation of ROS1 enhances its stability and positively regulates active DNA demethylation in Arabidopsis // Mol Plant. 2020. V. 13. № 12. P. 1816–1824.
Kooke R., Morgado L., Becker F. et al. Epigenetic mapping of the Arabidopsis metabolome reveals mediators of the epigenotype-phenotype map // Genome Res. 2019. V. 29. № 1. P. 96–106.
Kribelbauer J.F., Lu X.J., Rohs R. et al. Toward a mechanistic understanding of DNA methylation readout by transcription factors // J. Mol. Biol. 2019. V. 432. Iss. 6. P. 1801–1815.
Kurdyukov S., Bullock M. DNA methylation analysis: Choosing the right method // Biology (Basel). 2016. V. 5. № 1. P. 3.
Kouzarides T. Chromatin modifications and their function // Cell. 2007. V. 128. P. 693–705.
Lämke J., Bäurle I. Epigenetic and chromatin-based mechanisms in environmental stress adaptation and stress memory in plants // Genome Biol. 2017. V. 18. № 1. P. 124.
Lardon R., Geelen D. Natural variation in plant pluripotency and regeneration // Plants (Basel). 2020. V. 9. № 10. P. 1261.
Latzel V., Allan E., Silveira A. B. et al. Epigenetic diversity increases the productivity and stability of plant populations // Nature Commun. 2013. V. 4. P. 2875.
Latzel V., Münzbergová Z. Anticipatory behavior of the clonal plant Fragaria vesca //Front. Plant Sci. 2018. V. 9. P. 1847.
Latzel V., Rendina González A.P., Rosenthal J. Epigenetic memory as a basis for intelligent behavior in clonal plants // Front. Plant Sci. 2016. V. 7. P. 1354.
Latzel V., Zhang Y., Karlsson Moritz K. et al. Epigenetic variation in plant responses to defence hormones // Annals of Botany. 2012. V. 110. № 7. P. 1423–1428.
Law J.A., Jacobsen S.E. Establishing, maintaining and modifying DNA methylation patterns in plants and animals // Nature Rev. Genet. 2010. V. 11. P. 204–220.
Le N.T., Harukawa Y., Miura S. et al. Epigenetic regulation of spurious transcription initiation in Arabidopsis // Nature Commun. 2020. V. 11. P. 3224.
Le Goff A., Allard P., Landecker H. Heritable changeability: Epimutation and the legacy of negative definition in epigenetic concepts // Studies in History and Philosophy of Sci. 2021. V. 86. P. 35–46.
Li G., Hu S., Yang J. et al. Water-Wisteria as an ideal plant to study heterophylly in higher aquatic plants // Plant Cell Rep. 2017. V. 36. P. 1225–1236.
Liégard B., Baillet V., Etcheverry M. et al. Quantitative resistance to clubroot infection mediated by transgenerational epigenetic variation in Arabidopsis // New Phytologist. 2019. V. 222. № 1. P. 468–479.
Li X., Harris C.J., Zhong Z. et al. Mechanistic insights into plant SUVH family H3K9 methyltransferases and their binding to context-biased non-CG DNA methylation // PNAS. 2018. V. 115. P. E8793–E8802.
Li J., Yang D.L., Huang H. et al. Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation // Nature Plants. 2020. V. 6. P. 661–674.
Lindermayr C., Rudolf E.E., Durner J., Groth M. Interactions between metabolism and chromatin in plant models // Molecular Metabolism. 2020. V. 38. P. 100951.
Li B., Carey M., Workman J.L. The role of chromatin during transcription // Cell. 2007. V. 128. P. 707–719.
Li G., Hu S., Hou H., Kimura S. Heterophylly: Phenotypic plasticity of leaf shape in aquatic and amphibious plants // Plants. 2019. V. 8. P. 420.
Li G., Hu S., Zhao X. et al. Mechanisms of the morphological plasticity induced by phytohormones and the environment in plants // Int. J. Mol. Sci. 2021. V. 22. № 2. P. 765.
Li G., Hu S., Yang J. et al. Water-Wisteria as an ideal plant to study heterophylly in higher aquatic plants // Plant Cell Rep. 2017. V. 36. P. 1225–1236.
Li S.W. Molecular bases for the regulation of adventitious root generation in plants // Front. Plant Sci. 2021. V. 12. P. 614072.
Li Y., Kumar S., Qian W. Active DNA demethylation: mechanism and role in plant development // Plant Cell Rep. 2018. V. 37. № 1. P. 77–85.
Li Y., Li H. Many keys to push: diversifying the ‘readership’ of plant homeodomain fingers // Acta Biochim. Biophys. Sin. (Shanghai). 2012. V. 44. № 1. P. 28–39.
Li J., Yang D.-L., Huang H. et al. Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation // Nature Plants. 2020. V. 6. P. 661–674.
Liang S.C., Hartwig B., Perera P. et al. Kicking against the PRCs – A domesticated transposase antagonises silencing mediated by polycomb group proteins and is an accessory component of polycomb repressive complex 2 // PLoS Genet. 2015. V. 11. № 12. P. e1005660.
Liegard B., Baillet V., Etcheverry M. et al. Quantitative resistance to clubroot infection mediated by transgenerational epigenetic variation in Arabidopsis // New Phytol. 2019. V. 222. P. 468–479.
Lisch D. How important are transposons for plant evolution? // Nature Rev. Genet. 2013. V. 14. P. 49–61.
Lister R., O’Malley R.C., Tonti-Filippini J. et al. Highly integrated single-base resolution maps of the epigenome in Arabidopsis // Cell. 2008. V. 133. P. 523–536.
Liu R., Lang Z. The mechanism and function of active DNA demethylation in plants // J Integr. Plant Biol. 2019. V. 62. P. 148–159.
Liu C., Lu F., Cui X., Cao X. Histone methylation in higher plants // Annu. Rev. Plant Biol. 2010. V. 61. P. 395–420.
Locato V., Cimini S., De Gara L. ROS and redox balance as multifaceted players of cross-tolerance: Epigenetic and retrograde control of gene expression // J. Exp. Bot. 2018. V. 69. P. 3373–3391.
Lu W., Xiao L., Quan M. et al. Linkage-linkage disequilibrium dissection of the epigenetic quantitative trait loci (epiQTLs) underlying growth and wood properties in Populus // New Phytol. 2020. V. 225. P. 1218–1233.
Lira-Medeiros C.F., Parisod C., Fernandes R.A. et al. Epigenetic variation in mangrove plants occurring in contrasting natural environment // PLoS One. 2010. V. 5. P. e10326.
Lodha M., Marco C.F., Timmermans M.C. Genetic and epigenetic regulation of stem cell homeostasis in plants // Cold Spring Harb. Symp. Quant. Biol. 2008. V. 73. P. 243–251.
Mallatt J., Blatt M.R., Draguhn A. et al. Debunking a myth: plant consciousness // Protoplasma. 2021. V. 258. № 3. P. 459–476.
Marí-Ordóñez A., Marchais A., Etcheverry M. et al. Reconstructing de novo silencing of an active plant retrotransposon // Nature Genet. 2013. V. 45. № 9. P. 1029–1039.
Mateo-Bonmatí E., David Esteve-Bruna D., Juan-Vicente L. et al. INCURVATA11 and CUPULIFORMIS2 are redundant genes that encode epigenetic machinery components in Arabidopsis // Plant Cell. 2018. V. 30. P. 1596–1616.
Matzke M.A., Mosher R.A. RNA-directed DNA methylation: an epigenetic pathway of increasing complexity// Nature Reviews Genet. 2014. V.15. P. 394–408.
Mirouze M., Reinders J., Bucher E. et al. Selective epigenetic control of retrotransposition in Arabidopsis // Nature. 2009. V. 461. P. 427–430.
Miura A., Yonebayashi S., Watanabe K. et al. Mobilization of transposons by a mutation abolishing full DNA methylation in Arabidopsis // Nature. 2001. V. 411. № 6834. P. 212–214.
Morange M. The attempt of Nikolai Koltzoff (Koltsov) to link genetics, embryology and physical chemistry // J. Bioscience. 2011. V. 36. P. 211–214. Перевод: “Николай Кольцов и молекулярная биология” // Природа. 2015. № 12. С. 78–82.
Morgado L., Preite V., Oplaat C. et al. Small RNAs reflect grandparental environments in apomictic dandelion // Mol. Biol. Evol. 2017. V. 34. P. 2035–2040.
Münzbergová Z., Latzel V., Šurinová M., Hadincová V. DNA methylation as a possible mechanism affecting ability of natural populations to adapt to changing climate // Oikos. 2019. V. 128. P. 124–134.
Ma X., Lv S., Zhang C. et al. Histone deacetylases and their functions in plants // Plant Cell Rep. 2013. V. 32. P. 465–478.
Manning K., Tör M., Poole M. et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening // Nature Genet. 2006. V. 38. № 8. P. 948–952.
Miura K., Agetsuma M., Kitano H. et al. A metastable DWARF1 epigenetic mutant affecting plant stature in rice // PNAS. 2009. V. 106. № 27. P. 11218–11223.
Mozgova I., Hennig L. The polycomb group protein regulatory network // Annu. Rev. Plant Biol. 2015. V. 66. P. 269–296.
Münzbergová Z., Latzel V., Šurinová M., Hadincová V. DNA methylation as a possible mechanism affecting ability of natural populations to adapt to changing climate // Oikos. 2019. V. 128. № 1. P. 124–134.
Muyle A., Shearn R., Marais G.A. The evolution of sex chromosomes and dosage compensation in plants // Genome Biol. Evol. 2017. V. 9. № 3. P. 627–645.
Muyle A., Zemp N., Fruchard C. et al. Genomic imprinting mediates dosage compensation in a young plant XY system // Nature Plants. 2018. V.4. № 9. P. 677–680.
Nakayama H., Nakayama N., Seiki S. et al. Regulation of the KNOX-GA gene module induces heterophyllic alteration in North American lake cress // Plant Cell. 2014. V. 26. P. 4733–4748.
Nakayama H., Sinha N.R., Kimura S. How do plants and phytohormones accomplish heterophylly, leaf phenotypic plasticity, in response to environmental cues // Front. Plant Sci. 2017. V. 8. P. 1717.
Narlikar G.J., Sundaramoorthy R., Owen-Hughes T. Mechanisms and functions of ATP-dependent chromatin-remodeling enzymes // Cell. 2013. V. 154. P. 490–503.
Nashun B., Hill P.W., Hajkova P. Reprogramming of cell fate: epigenetic memory and the erasure of memories past // The EMBO Journal. 2015. V. 34. № 10. P. 1296–1308.
Noble D. Conrad Waddington and the origin of epigenetics // J. Exp. Biol. 2015. V. 218. P. 816–818.
Oberkofler V., Pratx L., Bäurle I. Epigenetic regulation of abiotic stress memory: maintaining the good things while they last // Curr. Opin. Plant Biol. 2021. V. 8. № 61. P. 102007.
Oborny B. The plant body as a network of semi-autonomous agents: a review // Philos. Trans. R. Soc. Lond. B. Biol. Sci. 2019. V. 374. № 1774. P. 20180371.
O’Malley R.C., Ecker J.R. Epiallelic variation in Arabidopsis thaliana // Cold Spring Harb. Symp. Quant. Biol. 2012. V. 77. P. 135–145.
Ornelas-Ayala D., Garay-Arroyo A., García-Ponce B. et al. The epigenetic faces of ULTRAPETALA1 // Front. Plant Sci. 2021. V. 12. P. 637244.
Pagliarani C., Gambino G. Small RNA mobility: Spread of RNA silencing effectors and its effect on developmental processes and stress adaptation in plants // Int. J. Mol. Sci. 2019. V. 20. № 17. P. 4306.
Parrilla-Doblas J.T., Roldán-Arjona T., Ariza R.R., Córdoba-Cañero D. Active DNA demethylation in plants // Int. J. Mol. Sci. 2019. V. 20. № 19. P. 4683.
Patel D.J.A. Structural perspective on readout of epigenetic histone and DNA methylation marks // Cold Spring Harb. Perspectives Biol. 2016. V. 8. № 3. P. a018754.
Pikaard C.S., Scheid O.M. Epigenetic regulation in plants // Cold Spring Harb. Perspectives Biol. 2014. V. 6. № 12. P. a019315.
Poethig S. Genetic mosaics and cell lineage analysis in plants // Trends Genet. 1989. V. 5. P. 273–277.
Prakash S., Singh R., Lodhi N. Histone demethylases and control of gene expression in plants // Cell. Mol. Biol. 2014. V. 60. № 5. P. 97–105.
Ptashne M. Epigenetics: core misconcept // PNAS. 2013. V. 110. P. 7101–7103.
Quan J., Latzel V., Tie D. et al. Ultraviolet B radiation triggers DNA methylation change and affects foraging behavior of the clonal plant Glechoma longituba // Front. Plant Sci. 2021. V. 12. P. 633982.
Raju S.K.K., Ritter E.J., Niederhuth C.E. Establishment, maintenance, and biological roles of non-CG methylation in plants // Essays in Biochemistry. 2019. V. 63. P. 743–755.
Reinders J., Wulff B.B.H., Mirouze M. et al. Compromised stability of DNA methylation and transposon immobilization in mosaic Arabidopsis epigenomes // Genes Dev. 2009. V. 23. P. 939–950.
Rendina González A.P., Preite V., Verhoeven K.J.F., Latzel V. Transgenerational effects and epigenetic memory in the clonal plant Trifolium repens // Front. Plant Sci. 2018. V. 9. P. 1677.
Reyna-Lopez G.E., Simpson J., Ruiz-Herrera J. Differences in DNA methylation patterns are detectable during the dimorphic transition of fungi by amplification of restriction polymorphism // Mol. Genet. Genomics. 1997. V. 253. P. 703–710.
Richards C.L., Schrey A.W., Pigliucci M. Invasion of diverse habitats by few Japanese knotweed genotypes is correlated with high epigenetic differentiation // Ecol. Lett. 2012. V. 15. P. 1016–1025.
Rohde A., Grunau C., De Beck L. et al. Carpel, a new Arabidopsis epi-mutant of the SUPERMAN gene: phenotypic analysis and DNA methylation status // Plant Cell Physiol. 1999. V. 40. № 9. P. 961–972.
Rohini G., Romika K., Sneha T., Shweta G. Genomic survey, gene expression analysis and structural modeling suggest diverse roles of DNA methyltransferases in legumes // PLoS One. 2014. V. 2. e88947.
Roudier F., Ahmed I., Berard C. et al. Integrative epigenomic mapping defines four main chromatin states in Arabidopsis // EMBO Journal. 2011. V. 30. P. 1928–1938.
Roux F., Colomé-Tatché M., Edelist C. et al. Genome-wide epigenetic perturbation jump-starts patterns of heritable variation found in nature // Genetics. 2011. V. 188. P. 1015–1017.
Saravana Kumar R.M., Wang Y., Zhang X. et al. Redox components: Key regulators of epigenetic modifications in plants // Int. J. Mol. Sci. 2020. V. 21. № 4. P. 1419.
Satterlee J.W., Scanlon M.J. Coordination of leaf development across developmental axes // Plants (Basel). 2019. V. 8. № 10. P. 433.
Scheid R., Chen J., Zhong X. Biological role and mechanism of chromatin readers in plant // Curr. Opin. Plant Biol. 2021. V. 10. № 61. P. 102008.
Schmitz R.J., Schultz M.D., Lewsey M.G. et al. Transgenerational epigenetic instability is a source of novel methylation variants // Science. 2011. V. 334. № 6054. P. 369–373.
Selma S., Orzáez D. Perspectives for epigenetic editing in crops // Transgenic Res. 2021.
Seta A., Tabara M., Nishibori Y. et al. Post-translational regulation of the dicing activities of Arabidopsis DICER-LIKE 3 and 4 by inorganic phosphate and the redox state // Plant Cell Physiol. 2017. V. 58. P. 485–495.
Shi W., Chen X., Gao L. et al. Transient stability of epigenetic population differentiation in a clonal invader // Front. Plant Sci. 2019. V. 9. P. 1851.
Shin J., Bae S., Seo P.J. De novo shoot organogenesis during plant regeneration // J. Exp. Bot. 2020. V. 71. № 1. P. 63–72. https://doi.org/10.1093/jxb/erz395
Sijacic P., Holder D. H. Bajic M., Deal R.B. Methyl-CpG-binding domain 9 (MBD9) is required for H2A.Z incorporation into chromatin at a subset of H2A.Z-enriched regions in the Arabidopsis genome // PLoS Genetics. 2019. V. 15. № 8. P. 1008326.
Song Q., Chen Z.J. Epigenetic and developmental regulation in plant polyploids // Curr. Opin. Plant Biol. 2015. V. 24. P. 101–109.
Spens A.E., Douhovnikoff V. Epigenetic variation within Phragmites australis among lineages, genotypes, and ramets // Biological Invasions. 2016. V. 18. P. 2457–2462.
Srikant T., Drost H.G. How stress facilitates phenotypic innovation through epigenetic diversity // Front. Plant Sci. 2021. V. 11. P. 606800. https://doi.org/10.3389/fpls.2020.606800
Stoddard C.I., Feng S., Campbell M.G. et al. A nucleosome bridging mechanism for activation of a maintenance DNA methyltransferase // Mol. Cell. 2019. V. 73. P. 73–83.
Sultan S.E. Developmental plasticity: re-conceiving the genotype // Interface Focus. 2017. V. 7. № 5. P. 20170009.
Surguchov. A., Emamzadeh F.N., Surguchev A.A. Amyloidosis and longevity: A lesson from plants //Biology. 2019. V. 8. № 2. P. 43.
Szymkowiak E.J., Sussex I.M. What chimeras can tell us about plant development // Annu. Rev. Plant Physiol. Plant Mol. Biol. 1996. V. 47. P. 351–376.
Tamiru M., Hardcastle T.J., Lewsey M.G. Regulation of genome-wide DNA methylation by mobile small RNAs // New Phytol. 2018. V. 217. P. 540–546.
Torres I.O., Fujimori D.G. Functional coupling between writers, erasers and readers of histone and DNA methylation // Curr. Opin. Struct. Biol. 2015. V. 35. P. 68–75.
Tricker P.J., Gibbings J.G., Rodríguez et al. Low relative humidity triggers RNA-directed de novo DNA methylation and supression of genes controlling stomatal development // J. Exp. Bot. 2012. V. 63. P. 3799–3813.
Tricker P.J., López C.M., Gibbings G. et al. Transgenerational, dynamic methylation of stomata genes in response to low relative humidity // Int. J. Mol. Sci. 2013. V. 14. P. 6674–6689.
Ueda M., Seki M. Histone modifications form epigenetic regulatory networks to regulate abiotic stress response // Plant Physiol. 2020. V. 182. № 1. P. 15–26.
Ullah F., Hamilton M., Reddy A.S.N., Ben-Hur A. Exploring the relationship between intron retention and chromatin accessibility in plants // BMC Genomics. 2018. V. 19. P. 21.
Vanden Broeck A., Cox K., Brys R. et al. Variability in DNA methylation and generational plasticity in the Lombardy poplar, a single genotype worldwide distributed since the eighteenth century // Front. Plant Sci. 2018. V. 9. P. 1635.
van Veen H., Sasidharan R. Shape shifting by amphibious plants in dynamic hydrological niches // New Phytol. 2021. V. 229. P. 79–84.
Velanis C.N., Perera P., Thomson B. et al. The domesticated transposase ALP2 mediates formation of a novel Polycomb protein complex by direct interaction with MSI1, a core subunit of Polycomb Repressive Complex 2 (PRC2) // PLoS Genet. 2020. V. 16. № 5. P. e1008681.
Verhoeven K.J.F., Preite V. Epigenetic variation in asexually reproducing organisms // Evolution. 2014. V. 68. P. 644–655.
Waddington C.H. The basic ideas of biology // Towards a Theoretical Biology / Ed. Waddington C.H. V. 1. Edinburgh University Press, Edinburgh, 1968. P. 1–32. Перевод Уоддингтон К.X. Основные биологические концепции. В кн.: На пути к теоретической биологии. М.: Мир, 1970. С. 11–46.
Wang M., Zhang K., Ngo V. et al. Identification of DNA motifs that regulate DNA methylation // Nucleic Acids Res. 2019. V. 47. № 13. P. 6753–6768.
Wei G., Liu K., Shen T. et al. Position-specific intron retention is mediated by the histone methyltransferase SDG725 // BMC Biol. 2018. V. 16. P. 44.
Weigel D., Colot V. Epialleles in plant evolution // Genome Biol. 2012. V. 13. № 10. P. 249.
Wienholz B.L., Kareta M.S., Moarefi A.H. et al. DNMT3L modulates significant and distinct flanking sequence preference for DNA methylation by DNMT3A and DNMT3B in vivo // PLoS Genet. 2010. V. 6. P. e1001106.
Whipple C. Defining the plant germ line-nature or nurture? // Science. 2012. V. 337. Iss. 6092. P. 301–302.
Williams B.P., Gehring M. Principles of epigenetic homeostasis shared between flowering plants and mammals // Trends Genet. 2020. V. 36. № 10. P. 751–763.
Wu H.W., Deng S., Xu H. et al. A noncoding RNA transcribed from the AGAMOUS (AG) second intron binds to CURLY LEAF and represses AG expression in leaves // New Phytol. 2018. V. 219. P. 1480–1491.
Wu M.F., Yamaguchi N., Xiao J. et al. Auxin-regulated chromatin switch directs acquisition of flower primordium founder fate // Elife. 2015. V. 4. P. e09269.
Wu Z., Fang X., Zhu D., Dean C. Autonomous pathway: FLOWERING LOCUS C repression through an antisense-mediated chromatin-silencing mechanism // Plant Physiol. 2020. V. 182. P. 27–37.
Xiao J., Wagner D. Polycomb repression in the regulation of growth and development in Arabidopsis // Curr. Opin. Plant Biol. 2015. V.23. P. 15–24.
Xiao X., Zhang J., Li T. et al. A group of SUVH methyl-DNA binding proteins regulate expression of the DNA demethylase ROS1 in Arabidopsis // Journal of Integrative Plant Biology. 2019. V. 61. № 2. P. 110–119.
Xu F., Feng X., Chen X. et al. Cytoplasmic Argonaute protein promotes the inheritance of RNAi // Cell Rep. 2018. V. 23. P. 2482–2494.
Xu L. De novo root regeneration from leaf explants: wounding, auxin, and cell fate transition // Curr. Opin. Plant Biol. 2018. V. 41. P. 39–45.
Xu Y., Zhang L., Wu G. Epigenetic regulation of juvenile-to-adult transition in plants // Front. Plant Sci. 2018. V. 9. P. 1048.
Yan B., Lv Y., Zhao C., Wang X. Knowing when to silence: roles of polycomb-group proteins in SAM maintenance, root development, and developmental phase transition // Int. J. Mol. Sci. 2020. V. 21. № 16. P. 5871.
Yu Z., Zhang G., Teixeira da Silva J.A. et al. DNA methyltransferase and demethylase gene families in Dendrobium officinale reveal their potential functions in polysaccharide accumulation // BMC Plant Biology. 2021. V. 21. P. 21.
Zhang H., Lang Z., Zhu J.K. Dynamics and function of DNA methylation in plants // Nat. Rev. Mol. Cell. Biol. 2018. V. 19. P. 489–506.
Zhang H.M., Lang Z.B., Zhu J.K. Dynamics and function of DNA methylation in plants // Nature Rev. Mol. Cell Biol. 2018. V. 19. P. 489–506.
Zhang L., Cheng Z., Qin R. et al. Identification and characterization of an epi-allele of FIE1 reveals a regulatory linkage between two epigenetic marks in rice // Plant Cell. 2012. V. 24. № 11. P. 4407–4421.
Zhang X., Bernatavichute Y.V., Cokus S. et al. Genomewide analysis of mono-, di- and trimethylation of histone H3 lysine 4 in Arabidopsis thaliana // Genome Biol. 2009. V. 10. № 6. P. R62.
Zhang X., Clarenz O., Cokus S. et al. Whole-genome analysis of histone H3 lysine 27 trimethylation in Arabidopsis // PLoS Biol. 2007. V. 5. P. e129.
Zhang X., Yazaki J., Sundaresan A. et al. Genome-wide high-resolution mapping and functional analysis of DNA methylation in Arabidopsis // Cell. 2006. V. 126. № 6. P. 1189–1201.
Zhang Y.Y., Fischer M., Colot V., Bossdorf O. Epigenetic variation creates potential for evolution of plant phenotypic plasticity // New Phytol. 2013. V. 197. P. 314–322.
Zhang Y.Z., Lin J., Ren Z. et al. Genome-wide distribution and functions of the AAE complex in epigenetic regulation in Arabidopsis // J. Integr. Plant Biol. 2021. V. 63. № 4. P. 707–722.
Zheng X., Chen L., Xia H. et al. Transgenerational epimutations induced by multi-generation drought imposition mediate rice plant’s adaptation to drought condition // Sci. Rep. 2017. V. 7. P. 39843.
Zhu H., Wang G., Qian J. Transcription factors as readers and effectors of DNA methylation // Nature Rev. Genet. 2016. V. 17. P. 551–565.
Дополнительные материалы отсутствуют.
Инструменты
Онтогенез


