Поверхность. Рентгеновские, синхротронные и нейтронные исследования, 2022, № 11, стр. 3-8
Моделирование взаимодействия человеческой липоксигеназы LOX 15 со свободной арахидоновой кислотой
П. Д. Короткова a, *, А. А. Юрченко b, В. И. Тимофеев c, d, **, А. Б. Шумм a, e, Ю. А. Владимиров a, b, c
a Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
b Российский национальный исследовательский медицинский университет
им. Н.И. Пирогова Министерства здравоохранения Российской Федерации
117997 Москва, Россия
c Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
119333 Москва, Россия
d Национальный исследовательский центр “Курчатовский институт”
123182 Москва, Россия
e Физический институт им. П.Н. Лебедева Российской академии наук
119991 Москва, Россия
* E-mail: korotkovapol@gmail.com
** E-mail: tostars@mail.ru
Поступила в редакцию 14.01.2021
После доработки 22.02.2022
Принята к публикации 28.02.2022
- EDN: RTKNAT
- DOI: 10.31857/S1028096022100089
Аннотация
Человеческая липоксигеназа ассоциирована с процессом ферроптоза – одного из путей запрограммированной смерти клетки. Это делает изучение механизма функционирования данного фермента крайне значимым, так как процесс ферроптоза связан с рядом заболеваний, прежде всего, злокачественных новообразований. Вместе с тем экспериментальное исследование этого фермента является затруднительным, так как липоксигеназа человека является мембранноассоциированным белком. В настоящей работе для изучения липоксигеназы были применены методы, основанные на молекулярном моделировании. Методом молекулярного докинга получена модель комплекса человеческой липоксигеназы с арахидоновой кислотой. С помощью моделирования на основе молекулярной механики изучена динамика полученного комплекса. Получены значения среднеквадратичных отклонений атомов С-α, а также значения среднеквадратичных флуктуаций атомов С-α, что позволило сделать вывод об устойчивости модельного комплекса. Описаны подвижные участки молекулы. Методом MM/GBSA, основанным на комбинации молекулярной механики и решения обобщенного уравнения Борна, рассчитано изменение свободной энергии при взаимодействии человеческой липоксигеназы с арахидоновой кислотой. Показано, что значение изменения свободной энергии соответствует неспецифическому связыванию лиганда, что объясняет относительно слабую каталитическую активность человеческой липоксигеназы по отношению к свободной арахидоновой кислоте.
ВВЕДЕНИЕ
Программируемая смерть клеток – один из важнейших регуляторных механизмов в организме млекопитающих. В 2008 г. были идентифицированы соединения RSL3 и RSL5, которые повышали смертность клеток в присутствии активных форм кислорода. Было показано, что эти соединения активировали особый механизм смерти мутировавших раковых клеток, который оказался связанным с клеточными хранилищами железа и отличался от апоптотического [1]. В 2012 г. введено понятие “ферроптоз” для обозначения железо-зависимой формы неапоптотической смерти клеток [2].
В дальнейшем проблеме ферроптоза было посвящено значительное количество обзорных статей [3–6]. Гибель клеток при ферроптозе связывают с нарушением их функционирования в результате пероксидации липидов в мембранах эндоплазматического ретикулума, но механизм этой гибели, в отличие от механизмов апоптоза, до сих пор окончательно не выяснен. Немногим яснее на данный момент представляется механизм самих реакций липидной пероксидации, вовлеченных в ферроптоз. В обзоре [7] указаны два потенциальных химических пути: ферментативное окисление арахидоновой кислоты (AA), входящей в состав фосфатидилэтаноламина, с образованием гидропероксида AAOOH с перокисдной группой в пятнадцатом положении при помощи липоксигеназ и неферментативное цепное окисление полиненасыщенных жирных кислот, катализируемое ионами железа, не входящего в активный центр гемопротеинов. В пользу первого предположения говорят данные хромато-масс-спектрометрии, в пользу второго – ферроптоз комплексонами железа [3] и активация ферроптоза при нарушении гомеостаза железа, в частности, при его избытке в организме [2, 7, 8]. Таким образом, построение модели взаимодействия человеческой 15-липоксигеназы (LOX 15) с арахидоновой кислотой и исследование ее свойств представляет несомненную важность для исследования механизма ферроптоза.
В настоящей работе методом молекулярного докинга получена модель LOX 15 с арахидоновой кислотой, исследована молекулярная динамика полученного комплекса. Методом молекулярной механики MM/GBSA, сочетающим молекулярную механику и решение обобщенного уравнения Борна, показано, что изменение свободной энергии соответствует неспецифическому связыванию лиганда.
МАТЕРИАЛЫ И МЕТОДЫ
Молекулярный докинг
Начальная модель системы была получена путем молекулярного докинга с использованием программы HADDOCK [9, 10]. Топология арахидоновой кислоты сгенерирована с использованием сервиса PRODRG [11]. В качестве модели LOX 15 использована модель кристалла [12].
Алгоритм докинга состоит из трех этапов. На первом этапе выполняли докинг твердых тел, при котором взаимодействующие молекулы вращали и перемещали случайным образом, чтобы минимизировать межмолекулярную энергию. После этого следовала вторая стадия, на которой выполняли оптимизацию пространства торсионных углов для уточнения ориентации лиганда и взаимодействующих с ним аминокислотных остатков белка. Последним этапом было уточнение структуры в явном растворителе.
На первом этапе было получено 1000 конфигураций структуры, из которых только 200 наиболее энергетически выгодных были проанализированы на третьем этапе. На третьем этапе 10 лучших структур уточняли в модельной сфере, заполненной водой. В качестве модели воды была выбрана TIP3P, которая представляет собой жесткую молекулу, состоящую из трех атомов. Каждый атом этой модели имеет свой заряд и свои параметры Леннарда-Джонса [13].
Молекулярная динамика
Для моделирования молекулярной динамики в качестве стартовой модели была выбрана наилучшая согласно оценочной функции конфигурация комплекса LOX 15 и арахидоновой кислоты. Для параметризации лиганда использовали программу Antechamber [14]. Моделирование проводили с помощью программы GROMACS 2020.3 [15]. Одним из способов описания взаимодействия между атомами является силовое поле. В настоящей работе было выбрано одно из стандартных полей – ff14SB: в нем существенно улучшена точность моделирования боковых цепей аминокислотных остатков по сравнению с альтернативным вариантом ff99SB [16]. Кроме того, выбранная модель воды является наиболее подходящей для использования с силовым полем ff14SB.
Молекула белка была помещена в ячейку в форме параллелепипеда таким образом, чтобы расстояние от молекулы белка до краев ячейки составляло 1.5 нм. Свободное пространство ячейки было заполнено молекулами воды. Кроме того, в систему было добавлено 0.15 моль/л KCl, а также несколько ионов калия для уравновешивания заряда системы.
На первом этапе для каждой из систем была проведена минимизация энергии с шагом 1 фс до максимальной силы 1000.0 кДж/моль · нм. Далее системы были уравновешены в условиях ограниченной подвижности тяжелых атомов при температуре 298 К и давлении 1 атм путем моделирования в “каноническом” (NVT) и изотермоизобарическом (NTP) ансамблях (100 пс в каждом) соответственно. Температура и давление в системах контролировали с использованием модифицированного термостата Берендсена [17] и баростата Паринелло–Рахмана [18]. Продуктивную симуляцию молекулярной динамики длительностью 20 нс для каждой из систем проводили в изотермоизобарическом ансамбле с шагом в 2 фс. Алгоритм LINCS [19] использовали для ограничения связей с участием атомов водорода. Дальнодействующие электростатические взаимодействия рассчитывали с использованием схемы суммирования по Эвальду [20]. Ван-дер-Ваальсовские и кулоновские взаимодействия были усечены до 1.4 нм, что является наиболее оптимальным для используемого силового поля [16]. Расчет молекулярной динамики проводили три раза.
Расчет изменения свободной энергии при образовании комплекса LOX 15 с арахидоновой кислотой
Для расчета энергии взаимодействия липоксигеназы с арахидоновой кислотой использовали (на основе вычисления свободной энергии образования комплекса) метод MM/GBSA [21]. Изначально для биомакромолекул использовали метод MM/PBSA, предполагающий решение уравнения Пуассона–Больцмана [21], но в дальнейшем нашел широкое применение метод MM/GBSA с использованием обобщенного уравнения Борна, что позволило на порядок увеличить скорость расчетов, что особенно важно в случае биомакромолекул [22, 23]. Суть метода заключается в том, чтобы сравнить свободную энергию сольватированной молекулы в двух разных состояниях (связанном и свободном). Известны различные модификации этого метода [24, 25]. В настоящей работе применили модификацию метода MM/GBSA, описанную в [26, 27].
Примененный модуль gmx_MMPBSA [28] версии 1.4.0, который используют для адаптации скрипта MMPBSA.py [29] и пакета AmberTools20 [30] к формату GROMACS, предоставляет возможность использовать метод MM/GBSA с обобщенной моделью Борна [26] при обработке результатов моделирования молекулярной динамики, проведенного с помощью программы GROMACS 2020.3 [15]. Вычисление свободной энергии для каждой траектории проводили на 1000 фреймах.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
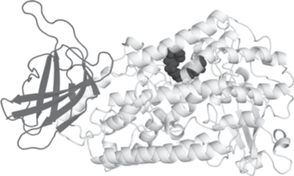
В результате молекулярного докинга было получено 10 моделей комплекса липоксигеназы с арахидоновой кислотой. Их кластеризация с использованием алгоритма FCC [31] показала, что все модели принадлежат одному кластеру, что говорит об их пространственной схожести. Критерием качества модели служит оценочная функции, которая представляет собой взвешенную сумму функций, учитывающих энергии различной природы, включая энергии Ван-дер-Ваальса, электростатического взаимодействия, десольватацию и энергию нарушения ограничений, задаваемых при докинге [9, 10]. По значению этой функции была выбрана наилучшая модель (рис. 1) для дальнейшего молекулярно-динамического исследования. Видно, что молекула липоксигеназы состоит из двух доменов: малый β-домен (аминокислотные остатки 1–120) и большой α-спиральный домен (аминокислотные остатки 121–676). Активный центр фермента расположен в “кармане” α-спирального домена [32].
Рис. 1.
Модель молекулы липоксигеназы LOX 15: темно-серым выделен малый β-домен (аминокислотные остатки 1–120); светло-серым – большой α-спиральный домен (аминокислотные остатки 121–676). Черным – молекула арахидоновой кислоты.
Анализ траектории проводили при длительности симуляции 20 нс. При выполнении периодических граничных условий во время расчетов некоторые части белковых молекул оказывались в соседних ячейках, что приводило к усложнению вычислений, это потребовало повторной центрировки молекул и возвращения в ячейку моделирования. Для улучшения точности полученных данных расчеты проводили три раза, после чего результат был усреднен. Затем был проведен первоначальный анализ траекторий, включая расчет среднеквадратичного отклонения, радиуса инерции и среднеквадратичных колебаний. Соответствующие графики показаны на рис. 2. Из рис. 2а видно, что среднеквадратичное отклонение быстро стабилизируется на уровне 0.2 нм. При таком размере системы быстрый выход значения на плато и низкий уровень флуктуации значения этой величины свидетельствует о высокой стабильности системы в процессе моделирования. На рис. 2б показано, что радиус инерции для моделируемого комплекса находится в диапазоне от 2.79 до 2.84 нм, другими словами, практически не меняется в процессе симуляции. Это указывает на то, что компактность системы существенно не изменяется во время моделирования. Было отмечено, что наибольшие значения среднеквадратичных колебаний атомов C-α моделируемых комплексов соответствуют малому домену фермента, что можно объяснить наличием относительно длинных, порядка 20–30 аминокислотных остатков, подвижных петель в малом домене. На рис. 2в также видно наличие подвижных участков и в большом домене. Следует отметить, что липоксигеназа является мембранно-ассоциированным белком. Кроме того, как следует из базы данных OPM [33], фрагменты молекулы, предположительно, взаимодействующие с мембраной, встречаются как в малом, так и в большом домене [34]. Учитывая, что вычисление молекулярной динамики проводили в водном растворе, такое поведение мембранно-ассоциированных участков поверхности фермента объяснимо. Как бы то ни было, участок активного центра фермента является достаточно стабильным для проведения расчетов методом MM/GBSA. Для улучшения статистики расчет свободной энергии проводили три раза. Полученные значения изменения свободной энергии приведены в табл. 1.
Рис. 2.
Графики временны́х зависимостей среднеквадратичного отклонения D (а) и радиуса инерции Rg (б); среднеквадратичной величины колебаний F атомов C-α в зависимости от номера аминокислотного остатка (в).
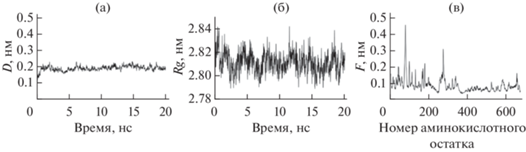
Таблица 1.
Изменения свободной энергии при образовании комплекса арахидоновой кислоты
| Номер эксперимента | Расчетное значение изменения свободной энергии, ккал/моль |
|---|---|
| 1 | –5 ± 2 |
| 2 | –5 ± 2 |
| 3 | –5.4 ± 1.9 |
| Среднее значение | –5 ± 2 |
Известно, что связывание субстратов с ферментами обычно сопровождается большим изменением свободной энергии [35–37]. Напротив, значения энергии связывания около 5–6 ккал/моль соответствуют непродуктивному связыванию субстрата. В качестве примера можно привести белковые кристаллы. Известно, что они образуются в специфических условиях и крайне нестабильны, что соответствует тому факту, что изменение свободной энергии при формировании связей в кристалле мало [38]. С другой стороны, известно, что человеческая липоксигеназа в природных условиях работает в комплексе с белком-партнером и липидным бислоем, а также характеризуется наличием малого β-домена [34], что отличает ее от растительных и бактериальных липоксигеназ [39, 40]. Вероятно, эти особенности объясняют слабую каталитическую активность свободной человеческой липоксигеназы.
ЗАКЛЮЧЕНИЕ
В работе установлено, что изменение свободной энергии при связывании человеческой липоксигеназой арахидоновой кислоты говорит о непродуктивном связывании. Полученные данные объясняют тот факт, что свободная арахидоновая кислота является очень слабым субстратом для каталитического окисления в присутствии человеческой липоксигеназы и позволяют прояснить связанный с этим аспект механизма реакций липидной пероксидации, вовлеченных в ферроптоз.
Список литературы
Yang W.S., Stockwell B.R. // Chem. Biol. 2008. V. 15. № 3. P. 234. https://www.doi.org/10.1016/j.chembiol.2008.02.010
Dixon S.J., Lemberg K.M., Lamprecht M.R., Skouta R., Zaitsev E.M., Gleason C.E., Patel D.N., Bauer A.J., Cantley A.M., Yang W.S., Morrison B., Stockwell B.R. // Cell. 2012. V. 149. № 5. P. 1060. https://www.doi.org/10.1016/j.cell.2012.03.042
Dixon S.J., Stockwell B.R. // Nat. Chem. Biol. 2014. V. 10. № 1. P. 9. https://www.doi.org/10.1038/nchembio.1416
Wu D., Chen L. // Acta Biochim. Biophys. Sin. 2015. V. 47. № 10. P. 857. https://www.doi.org/10.1093/abbs/gmv086
Xie Y., Hou W., Song X., Yu Y., Huang J., Sun X., Kang R., Tang D. // Cell Death Differ. 2016. V. 23. № 3. P. 369. https://www.doi.org/10.1038/cdd.2015.158
Seibt T.M., Proneth B., Conrad M. // Free Radical Biol. Med. 2019. V. 133. P. 144. https://www.doi.org/10.1016/j.freeradbiomed.2018.09.014
Stoyanovsky D.A., Tyurina Y.Y., Shrivastava I., Bahar I., Tyurin V.A., Protchenko O., Jadhav S., Bolevich S.B., Kozlov A.V., Vladimirov Y.A., Shvedova A.A., Philpott C.C., Bayir H., Kagan V.E. // Free Radical Biol. Med. 2019. V. 133. P. 153. https://www.doi.org/10.1016/j.freeradbiomed.2018.09.008
Latunde-Dada G.O. // Biochim. Biophys. Acta. Gen. Subj. 2017. V. 1861. № 8. P. 1893. https://www.doi.org/10.1016/j.bbagen.2017.05.019
Dominguez C., Boelens R., Bonvin A.M.J.J. // J. Am. Chem. Soc. 2003. V. 125. № 7. P. 1731. https://www.doi.org/10.1021/ja026939x
De Vries S.J., van Dijk A.D.J., Krzeminski M., van Dijk M., Thureau A., Hsu V., Wassenaar T., Bonvin A.M.J.J. // Proteins: Struct. Funct. Bioinf. 2007. V. 69. № 4. P. 726. https://www.doi.org/10.1002/prot.21723
Schüttelkopf A.W., van Aalten D.M.F. // Acta Crystallogr. D: Biol. Crystallogr. 2004. V. 60. № 8. P. 1355. https://www.doi.org/10.1107/S0907444904011679
Kobe M.J., Neau D.B., Mitchell C.E., Bartlett S.G., Newcomer M.E. // J. Biol. Chem. 2014. V. 289. № 12. P. 8562. https://www.doi.org/10.1074/jbc.M113.543777
Jorgensen W.L., Chandrasekhar J., Madura J.D., Impey R.W., Klein M.L. // J. Chem. Phys. 1983. V. 79. № 2. P. 926. https://www.doi.org/10.1063/1.445869
Salomon-Ferrer R., Case D.A., Walker R.C. // Wiley Interdiscip. Rev.: Comput. Mol. Sci. 2013. V. 3. № 2. P. 198. https://www.doi.org/10.1002/wcms.1121
Abraham M.J., Murtola T., Schulz R., Páll S., Smith J.C., Hess B., Lindahl E. // SoftwareX. 2015. V. 1–2. P. 19. https://www.doi.org/10.1016/j.softx.2015.06.001
Maier J.A., Martinez C., Kasavajhala K., Wickstrom L., Hauser K.E., Simmerling C. // J. Chem. Theory Comput. 2015. V. 11. № 8. P. 3696. https://www.doi.org/10.1021/acs.jctc.5b00255
Berendsen H.J.C., Postma J.V., van Gunsteren W.F., DiNola A.R.H.J., Haak J.R. // J. Chem. Phys. 1984. V. 81. № 8. P. 3684. https://www.doi.org/10.1063/1.448118
Parrinello M., Rahman A. // J. Chem. Phys. 1982. V. 76. № 5. P. 2662. https://www.doi.org/10.1063/1.443248
Hess B., Bekker H., Berendsen H.J.C., Fraaije J.G.E.M. // J. Comput. Chem. 1997. V. 18. № 12. P. 1463. https://www.doi.org/10.1002/(SICI)1096-987X(199709) 18:12<1463::AID-JCC4>3.0.CO;2-H
Darden T., York D., Pedersen L. // J. Chem. Phys. 1993. V. 98. № 12. P. 10 089. https://www.doi.org/10.1063/1.464397
Kollman P.A., Massova I., Reyes C., Kuhn B., Huo S., Chong L., Lee M., Lee T., Duan Y., Wang W., Donini O., Cieplak P., Srinivasan J., Case D.A., Cheatham T.E. // Acc. Chem. Res. 2000. V. 33. № 12. P. 889. https://www.doi.org/10.1021/ar000033j
Dominy B.N., Brooks C.L. // J. Phys. Chem. B. 1999. V. 103. № 18. P. 3765. https://www.doi.org/10.1021/jp984440c
Srinivasan J., Trevathan M.W., Beroza P., Case D.A. // Theor. Chem. Acc. 1999. V. 101. № 6. P. 426. https://www.doi.org/10.1007/s002140050460
Foloppe N., Hubbard R. // Curr. Med. Chem. 2006. V. 13. № 29. P. 3583. https://www.doi.org/10.2174/092986706779026165
Wang J., Hou T., Xu X. // Curr. Comput.-Aided Drug Des. 2006. V. 2. № 3. P. 287. https://www.doi.org/10.2174/157340906778226454
Onufriev A., Bashford D., Case D.A. // Proteins: Struct. Funct. Bioinf. 2004. V. 55. № 2. P. 383. https://www.doi.org/10.1002/prot.20033
Onufriev A., Bashford D., Case D.A. // J. Phys. Chem. B. 2000. V. 104. № 15. P. 3712. https://www.doi.org/10.1021/jp994072s
Valdés-Tresanco M.S., Valdés-Tresanco M.E., Valiente P.A., Moreno E. // J. Chem. Theory Comput. 2021. V. 17. № 10. P. 6281. https://doi.org/10.5281/zenodo.4569307
Miller III B.R., McGee Jr. T.D., Swails J.M., Homeyer N., Gohlke H., Roitberg A.E. // J. Chem. Theory Comput. 2012. V. 8. № 9. P. 3314. https://www.doi.org/10.1021/ct300418h
Case D.A., Belfon K., Ben-Shalom I.Y., Brozell S.R., Cerutti D.S., Cheatham III T.E., Cruzeiro V.W.D., Darden T.A., Duke R.E., Giambasu G., Gilson M.K., Gohlke H., Goetz A.W., Harris R., Izadi S., Izmailov S.A., Kasavajhala K., Kovalenko A., Krasny R., Kurtzman T., Lee T.S., LeGrand S., Li P., Lin C., Liu J., Luchko T., Luo R., Man V., Merz K.M., Miao Y., Mikhailovskii O., Monard G., Nguyen H., Onufriev A., Pan F., Pantano S., Qi R., Roe D.R., Roitberg A., Sagui C., Schott-Verdugo S., Shen J., Simmerling C.L., Skrynnikov N.R., Smith J., Swails J., Walker R.C., Wang J., Wilson L., Wolf R.M., Wu X., Xiong Y., Xue Y., York D.M., Kollman P.A. Amber 2020. San Francisco: University of California, 2020. 918 p.
Rodrigues J.P.G.M., Trellet M., Schmitz C., Kastritis P., Karaca E., Melquiond A.S.J., Bonvin A.M.J.J. // Proteins: Struct. Funct. Bioinf. 2012. V. 80. № 7. P. 1810. https://www.doi.org/10.1002/prot.24078
Kobe M.J., Neau D.B., Mitchell C.E., Bartlett S.G., Newcomer M.E. // J. Biol. Chem. 2014. V. 289. № 12. P. 8562. https://www.doi.org/10.1074/jbc.M113.543777
Orientation of Proteins in Membranes. University of Michigan, 2005. https://opm.phar.umich.edu/proteins/2345
Lomize M.A., Pogozheva I.D., Joo H., Mosberg H.I., Lomize A.L. // Nucleic Acids Res. 2012. V. 40. № D1. P. D370. https://www.doi.org/10.1093/nar/gkr703
Fatriansyah J.F., Rizqillah R.K., Yandi M.Y., Fadilah, Sahlan M. // J. King Saud Univ. Sci. 2022. V. 34. № 1. P. 101707. https://www.doi.org/10.1016/j.jksus.2021.101707
Azam F., Eid E.E.M., Almutairi A. // J. Mol. Struct. 2021. V. 1246. P. 131124. https://www.doi.org/10.1016/j.molstruc.2021.131124
Rants'o T.A., van der Westhuizen C.J., van Zyl R.L. // J. Mol. Graphics Modell. 2022. V. 110. P. 108054. https://www.doi.org/10.1016/j.jmgm.2021.108054
Kordonskaya Y.V., Timofeev V.I., Dyakova Y.A., Marchenkova M.A., Pisarevsky Y.V., Kovalchuk, M.V. // Crystals. 2021. V. 11. № 9. P. 1121. https://www.doi.org/10.3390/cryst11091121
Peng Y. L., Shirano Y., Ohta H., Hibino T., Tanaka K., Shibata D. // J. Biol. Chem. 1994. V. 269. № 5. P. 3755. https://www.doi.org/10.1016/S0021-9258(17)41924-7
Jiang Y., Wang S., He F., Fan Q., Ma Y., Yan W., Tang Y., Yang R., Zhao W. // Int. J. Food Sci. Technol. 2018. V. 53. № 12. P. 2738. https://www.doi.org/10.1111/ijfs.13885
Дополнительные материалы отсутствуют.
Инструменты
Поверхность. Рентгеновские, синхротронные и нейтронные исследования


