Прикладная биохимия и микробиология, 2019, T. 55, № 3, стр. 231-236
Получение и очистка рекомбинантной дипептидилпептидазы 4 Tenebrio molitor
В. Ф. Терещенкова 1, Е. В. Клячко 2, С. В. Беневоленский 2, М. А. Белозерский 3, Я. Е. Дунаевский 3, И. Ю. Филиппова 1, Е. Н. Элпидина 3, *
1 Московский государственный университет им. М.В. Ломоносова, химический факультет
119991 Москва, Россия
2 Институт биохимии имени А.Н. Баха, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” Российской академии наук
119071 Москва, Россия
3 Московский государственный университет им. М.В. Ломоносова, Научно-исследовательский
институт физико-химической биологии имени А.Н. Белозерского
119991 Москва, Россия
* E-mail: elp@belozersky.msu.ru
Поступила в редакцию 26.10.2018
После доработки 30.11.2018
Принята к публикации 18.12.2018
Аннотация
Дипептидилпептидаза 4 – уникальная пролин-специфичная пептидаза, способная гидролизовать связи, образованные остатком пролина, отщепляя с N-конца пептидов и белков дипептид, содержащий остаток этой иминокислоты в положении P1. В экспрессионной системе Pichia pastoris впервые получен препарат рекомбинантной дипептидилпептидазы 4 из насекомого Tenebrio molitor и предложен способ его очистки. Аутентичность полученного рекомбинантного фермента подтверждена масс-спектрометрически. Использование полученного препарата фермента T. molitor перспективно для проведения гидролиза труднорасщепляемых богатых пролином пептидов и белков, в частности проламинов – запасных белков семян злаковых растений, не гидролизуемых полностью пищеварительными ферментами человека и вызывающих у предрасположенных людей аутоиммунное заболевание желудочно-кишечного тракта (ЖКТ) – целиакию.
Пролин является единственной иминокислотой среди двадцати природных протеиногенных аминокислот. В связи с этим богатые пролином белки обладают рядом структурных особенностей. Находясь внутри полипептидной цепи, пролин формирует такую ее конформацию, которая препятствует деградации богатых пролином белков пептидазами широкого спектра действия. В природе гидролиз пролинсодержащих белков осуществляется преимущественно под действием особой группы протеолитических ферментов – пролин-специфичных пептидаз (ПСП). Важными представителями богатых пролином белков являются проламины – запасные белки семян злаковых растений, составляющие основу рациона большинства населения. В структуре проламинов содержится до 30% аминокислотных остатков пролина и до 50% остатков глутамина [1, 2]. Некоторые пептиды проламинов, главным образом, глиадинов из семян пшеницы, устойчивые к протеолизу пищеварительными пептидазами человека [3, 4], вызывают у предрасположенных людей аутоиммунное заболевание тонкого кишечника – целиакию.
Дипептидилпептидаза 4 (ДПП4, КФ: 3.4.14.5) является наиболее изученной ПСП, которая может быть использована для гидролиза труднорасщепляемых богатых пролином пептидов и белков, в частности проламинов семян, частичный гидролиз которых приводит к появлению токсических пептидов, вызывающих у предрасположенных людей заболевание ЖКТ. Фермент относится к сериновым пептидазам и гидролизует связи, образованные остатком пролина, отщепляя с N-конца пептидов и белков дипептид, содержащий остаток этой иминокислоты в положении P1 [5, 6], и может рассматриваться в качестве потенциального кандидата для создания препарата, способного скорректировать недостатки в работе пищеварительной системы человека.
Цель работы – получение рекомбинантной формы пищеварительной ДПП4 насекомого Tenebrio molitor [7], вредителя зерновых культур, для которого проламины являются главными пищевыми белками.
МЕТОДИКА
Материалы. В работе использовали следующие реактивы: акриламид, метилен-бис-акриламид, N,N,N',N'-тетраметилэтилендиамин (“Reanal”, Венгрия), персульфат аммония, додецилсульфатнатрия (ДДС-Na), 2-амино-2-гидроксиметил-пропан-1,3-диол (“ICN Biomedicals”, США), дитиотреитол (ДTT, “Fluka”, Швейцария), Кумасси G-250 (“Serva”, Германия), 2-[4-(2-гидроксиэтил)пиперазин-1-ил]этансульфоновая кислота (HEPES, “Carl Roth”, Германия); имидазол (“Panreac”, США); Ni2+-NTA сефароза (“GE Healthcare”, Великобритания); трипсин для масс-спектрометрического анализа (“Promega”, США); субстраты: Ala-Pro-AMC (AMC ‑ остаток 7-амино-4-метилкумарина) (“GeneCust”, Люксембург), Ala-Pro-pNA (pNA ‑ остаток п-нитроанилина) был синтезирован по стандартным методикам [8] в лаборатории химии белка кафедры ХПС химического факультета МГУ им. М.В. Ломоносова; зеоцин (“Invitrogen”, США); дрожжевой экстракт без аминокислот (“BD Difco”, США).
Клонирование гена ДПП4 T. molitor. Для выделения суммарной РНК были извлечены кишечники из 10 личинок T. molitor. Кишечники были очищены от содержимого, заморожены в жидком азоте и гомогенизированы. Суммарная РНК была выделена с использованием набора PureLink RNA Mini Kit “Thermo Fisher Scientific Inc.” (США) согласно протоколу производителя.
Рекомбинантная плазмида, содержащая вставку гена, кодирующего целевой белок, была получена в “Еврогене” (Россия). Для клонирования заданной последовательности были синтезированы специфические праймеры:
DPP-dir1 5'-ATACCCCACGGAAGTCAGG,
DPP-dir2 5'-ACCTGGACCCTACGTCATCG,
DPP-rev1 5'-TTCCGTGGGGTATTGGTCCTC,
DPP-rev2 5'-AGGGTCCAGGTCCTTGACAGAC.
С помощью праймеров:
DPP-out/d 5'-CATTCAGCTATGATTCTTCATAAGG,
DPP-out/r 5'-AAGGACACTGCTGCGACAG
была проведена амплификация полноразмерной кДНК ДПП4. Для удобства дальнейшей работы были введены сайты узнавания рестриктазами SalI/NheI, сайт KEX2, на С-конец фермента была добавлена последовательность, соответствующая 6-гистидиновому тегу (His6-tag) для упрощения процедуры очистки.
Полученная последовательность была отсеквенирована и протестирована на наличие случайных замен, которые могли быть устранены в случае обнаружения, и вставлена в плазмиду pPICZalfaA.
Получение препарата рекомбинантной ДПП4 T. molitor в экспрессионной системе Pichia pastoris. Для конструирования продуцента рекомбинантной ДПП4 T. molitor, полученная плазмида была линеаризована и введена в клетки прототрофа P. pastoris GS115-II-3 электропорацией. Отбор трансформантов осуществляли на чашках с агаризованной средой YPD (г/л): дрожжевой экстракт – 10, пептон соевый – 20, глюкоза – 20, бактоагар – 20, содержавшей зеоцин для селекции. Клоны с интегрированным в хромосому геном ДПП4 отбирали с помощью ПЦР. Отобранные клоны ферментировали в качалочных колбах с отбойниками на качалке Innova 43 250 об./мин в течение 65 ч при 28°С. Ферментационная среда содержала 1% дрожжевого экстракта, 2% ферментационного пептона, 1.34% YNB, 100 мМ К-фосфатный буфер, pH 6.0, 1% метанола, 1% сорбитола и 4 × 10–5% биотина. Для индукции экспрессии гена рекомбинантной ДПП4 T. molitor в среду добавляли метанол до 1% по объему каждые 24 ч. По окончании ферментации культуральную жидкость, содержащую рекомбинантную ДПП4 T. molitor, отделяли от клеток центрифугированием. Аликвоты культуральной жидкости хранили при –20°C.
Определение активности рекомбинантной ДПП4 T. molitor по хромогенному субстрату Ala-Pro-pNA. Ферментативную активность ДПП4 определяли по начальным скоростям гидролиза хромогенного пептидного субстрата, содержащего остаток п‑нитроанилина для детекции. В ячейку микропланшета вносили от 3 до 50 мкл препарата фермента и добавляли 0.1 М ацетат-фосфат-боратный универсальный буфер (УБ) [9], pH 7.9, до конечного объема 195 мкл. Затем вносили 5 мкл 10 мМ раствора субстрата Ala-Pro-pNA в диметилформамиде (ДМФА), конечная концентрация субстрата составляла 0.25 мМ, концентрация ДМФА в реакционной смеси – 2.5 об. %. Количество образовавшегося п-нитроанилина определяли в 96-луночных планшетах на микропланшетном фотометре ELx808 (“BioTek Instruments, Inc.”, США), измеряя оптическое поглощение раствора при 405 нм в нулевой момент времени и через различные промежутки. Смесь инкубировали при 37°C во встроенном термостате прибора ELx808 или в термостатируемом шкафу (“Binder”, Германия).
Активность пептидазы рассчитывали по формуле:
Очистка рекомбинантной ДПП4 T. molitor. Рекомбинантную ДПП4 T. molitor очищали методом металл-хелатной аффинной хроматографии на колонке с Ni2+-NTA сефарозой, поскольку в ее последовательность был введен His6-tag.
Для предварительной очистки 40 мл культуральной среды концентрировали в 8 раз (до 5 мл) и промывали 20 мМ Tрис-НCl буфером, рН 7.9, с использованием ячейки для ультрафильтрации с мембранными фильтрами YM-30 (“Amicon”, Нидерланды). Затем наносили на колонку (1 × 5 см), содержащую 3 мл Ni2+-NTA сефарозы. Элюцию проводили ступенчато с использованием градиента имидазола 0, 40, 150, 1000 мМ в 20 мМ Трис-НCl буфере, рН 7.9. Активность проб определяли, как описано выше. Последующее обессоливание и концентрирование препарата проводили в ячейке для ультрафильтрации Amicon с фильтрами YM-30.
Проведение электрофореза и пост-электрофоретического тестирования активности пептидазы в геле. Для проведения электрофореза использовали вертикальные пластинки размером 80 × 70 × 1 мм для заливки полиакриламидного геля и электрофоретическую ячейку с источником тока Miniprotean III (“BioRad”, США). Электрофорез проводили в нативных условиях в 4%-ном концентрирующем и 8%-ном разделяющем ПААГ, содержащем 35 мМ HEPES и 43 мМ имидазола при рН 7.2, как описано в [10]. Аликвоты фермента с активностью 1 нмоль/мин по субстрату Ala-Pro-pNA вносили в ячейку геля. Электрофорез проводили в направлении от катода к аноду при постоянном токе 20 мА до момента выхода красителя (40–60 мин) при 4°С.
Постэлектрофоретическое тестирование активности проводили непосредственно в геле с использованием специфического флуорогенного субстрата ДПП4 ‑ Ala-Pro-AMC. После электрофореза гель инкубировали в 0.125 мМ Ala-Pro-AMC в 0.1 М УБ при рН 7.9 в течение 10 мин при 37°С. Флуоресцирующие полосы продукта реакции визуализировали под УФ-лампой при 366 нм.
Электрофорез также осуществляли в денатурирующих условиях по методу Леммли в 4%-ном концентрирующем и 8%-ном разделяющем ПААГ. Перед нанесением на гель пробы инкубировали в буфере для приготовления образца согласно методу. Электрофорез проводили в направлении от катода к аноду при напряжении 200 В до момента выхода красителя (примерно 60 мин). После завершения электрофореза для визуализации белковых полос блок геля окрашивали Кумасси G-250.
Масс-спектрометрический анализ. Для проведения масс-спектрометрического анализа белков препараты очищенных ферментов подвергали электрофорезу в нативных условиях, как описано выше. Окрашенную Кумасси G-250 белковую полосу, соответствующую активности по флуорогенному субстрату Ala-Pro-AMC, вырезали из геля, и содержащийся в ней белок гидролизовали трипсином [11].
Масс-спектрометрический анализ триптических гидролизатов был проведен на тандемном MALDI-времяпролетном масс-спектрометре Ultraflextreme MALDI-TOF-TOF BRUKER (“Bruker Daltonik”, Германия), оснащенном УФ лазером (Nd). Масс-спектры получены в режиме положительных ионов с использованием рефлектрона. Точность измеренных масс после докалибровки по пикам автолиза трипсина составляла 30 ppm (0.01%). Спектры фрагментации получены в тандемном режиме Lift, точность измерения масс фрагментов 1 Да. Полученные масс-спектры анализировали с использованием сервера Mascot [12] и внутренней базы последовательностей из транскриптома кишечника личинок T. molitor [13].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Наиболее простыми системами для получения рекомбинантных белков являются бактериальные клетки. Однако такие системы подходят, в основном, для получения бактериальных белков, поскольку не могут обеспечить необходимые пост-трансляционные модификации, характерные для белков более сложных организмов. Анализ литературных данных показал, что в бактериальных системах E. coli были экспрессированы только бактериальные ДПП4 [14, 15]. Для экспрессии ферментов млекопитающих использовали более сложные системы на основе дрожжевых клеток Pichia pastoris [16], клеточных линий насекомых Sf9 [17] и Hi5 [18], а также клеток из яичников млекопитающего китайского хомячка (CHO, Chinese Hamster Ovary) [19, 20].
Для получения рекомбинантной ДПП4 T. molitor была выбрана система экспрессии P. pastoris, обеспечивающая получение активного фермента с высоким выходом и оптимальными пост-трансляционными модификациями. Преимуществом этой системы являлось отсутствие секреции в культуральную жидкость собственных дрожжевых цистеиновых и пролин-специфичных пептидаз.
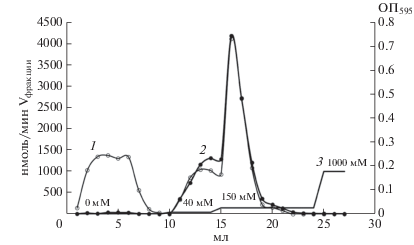
Для получения рекомбинантного белка ДПП4 T. molitor клетки прототрофа P. pastoris GS115-II-3 были трансформированы полученным вектором pPICZalfaA-TmDPP4 (TmDPP4 – dipeptidylpeptidase 4 Tenebrio molitor) методом электропорации. Схема конструкции представлена на рис. 1. Рекомбинантный белок был получен в пяти независимых параллелях. На основании анализа ферментативной активности по специфическому субстрату ДПП4 Ala-Pro-pNA в полученных культуральных средах, были отобраны штаммы, содержащие целевой фермент с максимальной ферментативной и удельной активностями. Выход белка составил 44 мг из 1 л культуральной среды. Благодаря введенному в последовательность His6-tag белок был очищен в одну стадию с использованием металл-хелатной аффинной хроматографии на колонке с Ni2+-NTA сефарозой (рис. 2). Фракция активного фермента элюировалась 150 мМ имидазолом. Сразу после очистки фракции, содержащие активный фермент, были обессолены и сконцентрированы. Выход по белку составил 44%, по активности – 84%.
Рис. 2.
Очистка рекомбинантной ДПП4 T. molitor методом металл-хелатной аффинной хроматографии на Ni2+-NTA сефарозе: 1 – содержание белка; 2 – активность фермента по специфическому субстрату Ala-Pro-pNA; 3 – градиент имидазола (мМ).
Аутентичность полученного рекомбинантного фермента подтверждали масс-спектрометрически. Для этого полученный препарат фермента подвергали электрофоретическому анализу в ПААГ. Пост-электрофоретическое тестирование активности с флуорогенным субстратом Ala-Pro-AMC и окрашивание Кумасси G-250 показало наличие одной белковой полосы в геле. С использованием масс-спектрометрических подходов было подтверждено, что полученная последовательность рекомбинантной ДПП4 T. molitor соответствовала последовательности нативной ДПП4 T. molitor (NCBI: MH265052, score 321, [12]) (рис. 3).
Рис. 3.
Идентификация рекомбинантной ДПП4 T. molitor. 1 – пост-электрофоретическое тестирование активности с субстратом Ala-Pro-AMC после проведения ЭФ в ПААГ в нативных условиях; 2 – окрашивание Кумасси G-250 после электрофореза в ПААГ в денатурирующих условиях. На приведенной последовательности ДПП4 T. molitor подчеркнуты пептиды, найденные методом пептидного фингерпринта с помощью MALDI-TOF масс-спектрометрии, аминокислотные остатки активного центра отмечены*.

Таким образом, в данной работе был разработан метод получения рекомбинантной ПСП– ДПП4 T. molitor. Ранее было показано, что нативный фермент ДПП4 T. molitor, полученный при фракционировании экстракта средней кишки, способен эффективно гидролизовать глиадины и, по-видимому, может участвовать в пищеварении насекомого [7].
Полученный рекомбинантный фермент может быть использован для разработки терапевтических средств и биологически-активных добавок, улучшающих усвоение глютен-содержащей пищи, а также для предобработки сырья при изготовлении безглютеновых напитков и продуктов питания, включая питание для детей. В зависимости от требуемого результата предлагаемый рекомбинантный фермент можно применять как в качестве индивидуального препарата, так и в комбинации с другими ферментами.
Известны работы по гидролизу глиадинов отдельными пептидазами, которые способны расщеплять пептидные связи, образованные пролином. К ним относятся пролилолигопептидазы из Myxococcus xanthus, Sphingomonas capsulata и Flavobacterium meningosepticum [21], пролилэндопептидазы из Aspergillus niger [22, 23], аминопептидаза и Х-пролилдипептидилпептидаза из A. oryzae [24, 25] и ПСП молочнокислых бактерий Lactobacillus plantarum и Pediococcus pentosaceus [26], а также других штаммов молочнокислых бактерий [27]. Описан гидролиз глиадинов цистеиновыми пептидазами. Так, в настоящее время разработаны способы получения в виде профермента (pro-ЕР-В2) такого типа цистеиновой пептидазы из ячменя Hordeum vulgare [28, 29] и усеченной формы цистеиновой пептидазы тритикаина-альфа пшеницы Triticum aestivum [30], которые рекомбинантно экспрессировали в бактериальной системе E. coli. Преимуществом рекомбинантной ДПП4 T. molitor является то, что полученный фермент входит в состав природной энзиматической системы ‑ пищеварительного комплекса насекомых-вредителей запасов зерновых культур, целенаправленно приспособленной для гидролиза проламинов, что может обеспечить большую эффективность в расщеплении глиадинов и их токсических пептидов по сравнению с приведенными аналогами.
Авторы выражают благодарность к. х. н. М.В. Серебряковой за проведение масс-спектрометрического анализа препарата рекомбинантной ДПП4.
Работа выполнена при финансовой поддержке Российского Фонда фундаментальных исследований совместно с Фондом поддержки научно-проектной деятельности студентов, аспирантов и молодых ученых “Национальное интеллектуальное развитие” (проект № 17-34-80158 мол_эв_а), Российским Фондом фундаментальных исследований (проекты № 17-54-61008 Египет_а и 18-04-01221_а), и Фондом содействия инновациям (УМНИК № 8874ГУ/2015 (0018984)).
Список литературы
Shewry P.R., Tatham A.S. // Biochem. J. 1990. V. 267. № 1. P. 1–12.
Shewry P.R., Halford N.G. // J. Exp. Bot. 2002. V. 53. № 370. P. 947–958.
Shan L., Molberg O., Parrot I., Hausch F., Filiz F., Gray G.M., Sollid L.M., Khosla C. // Science. 2002. V. 297. № 5590. P. 2275–2279.
Shan L., Qiao S.W., Arentz-Hansen H., Molberg O., Gray G.M., Sollid L.M., Khosla C.J. // Proteome Res. 2005a. V. 4. № 5. P. 1732–1741.
Cunningham D.F., O’Connor B. // Biochim Biophys Acta. 1997. V. 1343. № 2. P. 160–186.
Misumi Y., Ikehara Y. Dipeptidyl-peptidase IV (chapter 745). In: Rawlings N.D., Salvesen G.S. (eds). Handbook of Proteolytic Enzymes 3rd ed., V. 3.Amsterdam: Elsevier, 2013. P. 3374–3379.
Tereshchenkova V.F., Goptar I.A., Kulemzina I.A., Zhuzhikov D.P., Serebryakova M.V., Belozersky M.A., Dunaevsky Y.E., Oppert B., Filippova I.Y., Elpidina E.N. // Insect Biochem. Mol. Biol. 2016. V. 76. P. 38–48.
Houben-Weyl Methods of Organic Chemistry: Synthesis of Peptides and Peptidomimetics, 4th edition. V. E22a. In: Goodman M., Toniolo C., Moroder L., Felix A. (eds). Stuttgart, NY: Thieme, 2004. 785 p.
Frugoni J.A.C. // Gazz. Chem. Ital. 1957. V. 87. P. 403–407.
McLellan T. // Anal. Biochem. 1982. V. 126. № 1. P. 94–99.
Prabhakar S., Chen M.S., Elpidina E.N., Vinokurov K.S., Smith C.M., Marshall J., Oppert B. // Insect Mol. Biol. 2007. V. 16. № 4. P. 455–468.
Perkins D.N., Pappin D.J., Creasy D.M. Cottrell J.S. // Electrophoresis. 1999. V. 20. № 18. P. 3551–3567. http://www.matrixscience.com.
Martynov A.G., Elpidina E.N., Perkin L., Oppert B. // BMC Genomics. 2015. V. 16. № 1. P. 75.
Kabashima T., Yoshida T., Ito K., Yoshimoto T. // Arch. Biochem. Biophys. 1995. V. 320. № 1. P. 123–128.
Jobin M.C., Martinez G., Motard J., Gottschalk M., Grenier D. // J. Bacteriol. 2005. V. 187. № 2. P. 795–799.
Baer J.W., Gerhartz B., Hoffmann T., Rosche F., Demuth HU. Characterization of human DP IV produced by a Pichia pastoris expression system. In: Back N., Cohen I.R., Kritchevsky D., Lajtha A., Paoletti R. (eds). Dipeptidyl Aminopeptidases in Health and Disease. Advances in Experimental Medicine and Biology. V. 524. Springer, Boston, MA, 2004. P. 103–108.
Dobers J., Zimmermann-Kordmann M., Leddermann M., Schewe T., Reutter W., Fan H. // Protein Expr. Purif. 2002. V. 25. № 3. P. 527–532.
Lee B., Shi L., Kassel D.B., Asakawa T., Takeuchi K., Christopher R.J. // Eur. J. Pharmacol. 2008. V. 589. № 1–3. P. 306–314.
Hong W.J., Piazza G.A., Hixson D.C., Doyle D. // Biochemistry. 1989. V. 28. № 21. P. 8474–8479.
Yu D.M., Slaitini L., Gysbers V., Riekhoff A.G., Kahne T., Knott H.M., De Meester I., Abbott C.A., McCaughan G.W., Gorrell M.D. // Scand. J. Immunol. 2011. V. 73. № 2. P. 102–111.
Shan L., Marti T., Sollid L.M., Gray G.M., Khosla C. // Biochem. J. 2004. V. 383. № Pt 2. P. 311–318.
Stepniak D., Spaenij-Dekking L., Mitea C., Moester M., de Ru A., Baak-Pablo R., van Veelen P., Edens L., Koning F. // Am. J. Physiol. Gastrointest. Liver Physiol. 2006. V. 291. № 4. P. G621–629.
Konig J., Holster S., Bruins M.J., Brummer R.J. // Sci. Rep. 2017. V. 7. № 1. P. 13100.
Byun T., Kofod L., Blinkovsky A. // J. Agric. Food Chem. 2001. V. 49. № 4. P. 2061–2063.
Eugster P.J., Salamin K., Grouzmann E., Monod M. // Microbiology. 2015. V. 161. № 12. P. 2277–2288.
Gerez C.L., Dallagnol A., Rollan G., Font de Valdez G. // Food Microbiol. 2012. V. 32. № 2. P. 427–430.
De Angelis M., Cassone A., Rizzello C., Gagliardi F., Minervini F., Calasso M., Di Cagno R., Francavilla R., Gobbetti M. // Appl. Enviroment. Microbiol. 2010. V. 76. № 2. P. 508–518.
Gass J., Vora H., Bethune M.T., Gray G.M., Khosla C. // J. Pharmacol. Exp. Ther. 2006. V. 318. № 3. P. 1178–1186.
Vora H., McIntire J., Kumar P., Deshpande M., Khosla C. // Biotechnol. Bioeng. 2007. V. 98. № 1. P. 177–185.
Savvateeva L.V., Gorokhovets N.V., Makarov V.A., Serebryakova M.V., Solovyev A.G., Morozov S.Y., Reddy V.P., Zernii E.Y., Zamyatnin A.A. Jr, Aliev G. // Int. J. Biochem. Cell. Biol. 2015. V. 62. P. 115–124.
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология