Российский физиологический журнал им. И.М. Сеченова, 2019, T. 105, № 9, стр. 1113-1121
Регуляция метаболизма скелетных мышц эпигенетическими факторами
И. В. Астратенкова 1, *, И. И. Ахметов 2, Н. Д. Гольберг 2, В. А. Рогозкин 2
1 Санкт-Петербургский государственный университет
Санкт-Петербург, Россия
2 Санкт-Петербургский Научно-исследовательский институт
физической культуры
Санкт-Петербург, Россия
* E-mail: astratenkova@mail.ru
Поступила в редакцию 02.07.2018
После доработки 10.06.2019
Принята к публикации 20.06.2019
Аннотация
В регуляции метаболизма скелетных мышц участвует сложный набор молекулярных механизмов, включая генетические и эпигенетические факторы. Каскады реакций, связанные с посттрансляционной модификацией белков, являются универсальным механизмом контроля внутриклеточного обмена веществ, обеспечивающего дифференцировку, трансформацию, гипертрофию и атрофию мышечных клеток. Эпигенетическая регуляция экспрессии генов в скелетных мышцах человека включает три процесса: метилирование ДНК, модификацию гистонов и взаимодействие мРНК с микроРНК. В статье рассматриваются возможные механизмы эпигенетической регуляции внутриклеточного метаболизма скелетных мышц при выполнении однократных и систематических физических нагрузок. Основное внимание обращено на эпигенетическую регуляцию экспрессии структурных и метаболических генов в скелетных мышцах при различных функциональных состояниях человека.
Изучение молекулярных механизмов экспрессии генов в скелетных мышцах на протяжении многих лет остается одной из актуальных проблем молекулярной генетики и физиологии человека. До недавнего времени в генетике доминировало представление о том, что ДНК является единственным носителем наследственной информации. Расшифровка геномов человека и других организмов вскрыла не только принципы организации ДНК, но и показала сложность процессов регуляции экспрессии структурных и метаболических генов. Оказалось, что внешние воздействия такие как питание, стресс, физические нагрузки и другие стимулы изменяют экспрессию генов и их действие контролируется сложным набором регуляторных механизмов.
Возникла необходимость в более четком определении понятий, и кроме термина геном сейчас широкое распространение получил термин эпигеном, который включает совокупность элементов, регулирующих экспрессию генов. К эпигенетическим модификациям относят метилирование ДНК и гистонов, модификации гистонов и регуляторных белков в реакциях ацетилирования, фосфорилирования, убиквитинирования, сумоилирования, ацетилглюкозоаминирования и протеолитическое расщепление. Каскады реакций, связанные с эпигенетической посттрансляционной модификацией белков, являются универсальным механизмом контроля внутриклеточного метаболизма, обеспечивающего дифференцировку, трансформацию, гипертрофию и атрофию мышечных клеток.
Цель статьи – показать участие эпигенетических факторов в регуляции метаболизма скелетных мышц при различных функциональных состояниях человека.
Многочисленные генетические исследования, проводимые в разных странах, показали, что регуляция метаболизма в организме человека представлена не только экспрессией генов на уровне ДНК, но включает более сложный механизм с участием эпигенетических факторов. Кроме того, стало понятно, что помимо структурных особенностей ДНК (полиморфизмы, мутации), на предрасположенность человека к выполнению физических нагрузок влияют эпигенетические модификации, которые по своей природе являются обратимыми процессами и могут оказывать положительное либо отрицательное действие на физическую работоспособность.
Рассмотрим кратко последние достижения в эпигенетике человека, связанные с проявлением физической активности. Мышечные клетки обладают врожденными и индивидуальными эпигенетическими признаками, называемыми эпигеномом, который составляет два общих эпигенетических события: метилирование ДНК и модификацию гистонов. Эти процессы контролируют конформацию ДНК в двух разных состояниях: открытом с транскипционно активным эухроматином и закрытом с транскрипционно неактивным гетерохроматином. Таким образом, регулируется участие ДНК в процессах транскрипции, репарации и репликации.
Метилирование представляет собой временную химическую модификацию нуклеотидной последовательности без нарушения кодирующей способности ДНК. В этом случае обратимое метилирование рассматривается как эпимутация в отличие от мутации, приводящей к нуклеотидным заменам, делециям или вставкам нуклеотидов. Процесс метилирования и деметилирования ДНК в скелетных мышцах катализируют ДНК-метилтрансферазы (DNMT) и деметилазы, называемые десять-одиннадцать транслоказными энзимами (TET). В скелетных мышцах человека процесс метилирования ДНК включает перенос метильной группы (–CH3) в положение С5 нуклеотида цитозина, который катализирует семейство DNMTs, состоящее из трех ферментов, различающихся по интенсивности экспрессии под влиянием физической нагрузки. Метилирование происходит в CpG-островках, в результате чего образуются метилСpG-последовательности на обеих цепях ДНК [1]. Семейство TETs включает 3 фермента TET1, TET2 и TET3, которые катализируют каскад окислительных реакций с участием 5-метилцитозина (5mC). На первом этапе 5-метилцитозин превращается в 5-гидроксиметилцитозин (5-hmC), который в дальнейшем окисляется в 5-формилцитозин (5fC) и 5-кабоксицитозин (5caC) [2, 3]. Теперь 5fC и 5caC становятся субстратами для фермента тимин-ДНК-гликозилазы (TDG), способного удалять их из молекулы ДНК. Восстановление цитозина в структуре ДНК происходит с участием мультибелкового комплекса BER (base excision repair) [4].
Из семи возможных модификаций гистонов в статье рассматриваются только процессы метилирования и ацетилирования гистонов в скелетных мышцах, поскольку именно они наиболее интенсивно изменяются при выполнении физических нагрузок. Метилирование гистонов имеет более разнообразный характер и приводит к разным изменениям интенсивности экспрессии генов. Метилирование лизина 4 в гистоне H3 (H3K4me3) вызывает увеличение экспрессии генов. При метилировании лизина в положении 9 и 27 гистона H3 (H3K9me2/3 и H3K27me3) и лизина 20 в гистоне H4 (H4K20me3) происходит угнетение экспрессии генов. Предполагается, что монометилирование гистонов приводит к активации процессов транскрипции, тогда как ди- и триметилирование, напротив, сопровождается угнетением транскрипции. Управление этими процессами происходит в реакциях взаимодействия между модификацией гистонов и метилированием ДНК [5]. Эпигенетические модификации, связанные с метилированием ДНК и гистонов, сопровождаются изменением экспрессии генов при наличии сигналов из внешней среды, в особенности это проявляется на моделях различных физических нагрузок.
Важным шагом в понимании роли метилирования ДНК в экспрессии генов в скелетных мышцах стало исследование, выполненное на разных типах мышечных волокон человека [6]. В медленных и быстрых типах мышечных волокон выявлены разные места метилирования ДНК и показана экспрессия ряда структурных и метаболических генов. Так, в медленных мышечных волокнах обнаружено гиперметилирование генов, участвующих в анаэробном метаболизме, таких как ген Са-АТФазы эндоплазматического ретикулума быстрой изоформы (SERCA1), фосфофруктокиназы (PEKM), альдолазы А (ALDOA) и разобщающего белка 3 (UCP3). С другой стороны, в быстрых мышечных волокнах выявлено гиперметилирование генов, которые участвуют в аэробном метаболизме, такие как ген Са-АТФазы эндоплазматического ретикулума медленной изоформы (SERCA2) и лактатдегидрогеназы B (LDHH). Предложенный авторами метод определения мест метилирования в специализированных мышечных волокнах открывает новые возможности для выявления молекулярных различий в тяжелых цепях миозина I и IIa скелетных мышц.
Выполнение физической нагрузки вызывает изменение метилирования промоторов метаболических и структурных генов в скелетных мышцах. Так, в мышцах человека после однократной физической нагрузки происходит снижение метилирования промоторов генов PGC-1α, TFAM, MEF2A, CS и PDK4 [7]. В процессе метилирования промоторов генов в скелетных мышцах принимают участие многочисленные метаболические пути, регулирующие освобождение Ca2+, усиление окислительного фосфорилирования и повышение продукции ATФ; оказывают влияние изменение отношения AMФ/ATФ, активация 5-AMФ протеинкиназы (AMPK), увеличение активных форм кислорода, изменение содержания S-аденозилметионина, основного поставщика метильных групп, а также возможное усиление реакций деметилирования с участием ферментов TET/BER [8]. Совокупный эффект этих метаболических реакций создает условия для успешного осуществления реакции метилирования с участием семейства ферментов метилтрансфераз.
В результате выполнения тренировочной программы в течение 6 месяцев произошло изменение профиля метилирования ДНК в скелетных мышцах испытуемых. Большинство исследованных генов после такой тренировки находились в различной степени гипометилирования [9]. Таким образом, достаточно убедительно показано, что однократные и систематические физические нагрузки сопровождаются изменениями степени метилирования ДНК в скелетных мышцах, и это является наиболее значимым эпигенетическим фактором [10–12].
В посттрансляционной модификации белков два типа ферментов контролируют обратимость ацетилирования остатков аминокислоты лизина в молекуле гистонов: гистоацетилтрансфераза (HAT) и гистондеацетилаза (HDAC). Эти ферменты катализируют перенос ацетильной группы на аминокислоту лизин в N-концевом участке молекулы гистонов H2A, H2B, H3 и H4. В настоящее время идентифицировано 18 ферментов HDACs, которые разделены на 4 класса. В регуляции метаболизма скелетных мышц участвуют ферменты, включенные в класс IIa. Эти ферменты имеют существенное отличие от других классов гистондеацетилаз, прежде всего потому, что являются тканеспецифичными и участвуют в регуляции метаболизма скелетных и сердечной мышц. Ферменты класса IIa участвуют в реакциях со специфическими транскрипционными факторами и их зависимое от сигналов ядерно-цитоплазматическое перемещение оказывает эффект на регуляцию генов в скелетных мышцах [13].
В состоянии покоя ферменты HDACs класса IIa находятся в ядрах в дефосфорилированном виде и взаимодействуют с транскрипционными факторами и регуляторными белками в качестве регуляторов активности генов. При наличии информационных сигналов, поступающих в ядро, происходит фосфорилирование HDACs, приводящее к распаду белковых комплексов с транскрипционными факторами и за этим следует выход ферментов в цитоплазму.
Регуляция метаболизма в скелетных мышцах при выполнении физических нагрузок различной интенсивности и длительности происходит с участием ферментов HDACs класса IIa. Роль этих ферментов изучалась на разных моделях как при однократном, так и систематическом физическом воздействии. Фосфорилирование HDAC и транспорт фермента из ядра способствует усилению транскрипционного ответа при выполнении физической нагрузки. Следовательно, группу ферментов HDACs можно рассматривать как ключевые регуляторы метаболизма в скелетных мышцах, контролирующие процессы метаболической адаптации к внешним воздействиям, в том числе к физическим нагрузкам [13].
В последние годы появились факты, которые показывают, что связь между эпигенетикой и метаболизмом является двухсторонней с участием метаболитов из общих метаболических путей, используемых для модификации белков и ДНК. Ключевым фактором для регуляции метаболизма скелетных мышц является транскрипционный контроль экспрессии метаболических ферментов. Это положение можно рассматривать как основное для эпигенетического регулирования [8, 14]. В качестве подтверждающего примера можно привести содержание ацетил-СоА в цитоплазме и ядре мышечных клеток, поскольку именно он служит субстратом в реакции ацетилирования белков. При наличии в мышечной клетке ферментов ацетилтрансфераз и деацетилаз фактически процесс регулируется высоким уровнем ацетил-СоА, при низком содержании этого субстрата процесс становится неэффективным [1]. Таким образом, доступность ацетил-СоА для ацетилирования гистонов во многом определяет интенсивность экспрессии генов и уровень метаболизма в скелетных мышцах как в покое, так и при функциональной активности. Увеличение активности фермента АТФ-цитратлиазы (ACL) в мышцах повышает метаболизм в митохондриях, и при этом возрастает продукция АТФ. Таким образом, повышение доступности ацетил-СоА приводит к увеличению метаболизма скелетных мышц.
В недавно опубликованной статье австралийских ученых обсуждается проблема наследования потомством метаболических фенотипов скелетных мышц [8]. Подробно рассматриваются изменения в метаболизме скелетных мышц, обусловленные посттрансляционными модификациями гистонов в реакциях ацетилирования/деацетилирования и метилированием ДНК, в моделях на клеточных культурах, экспериментах на животных и физических нагрузках у людей. Метилирование ДНК и модификация гистонов в реакциях ацетилирования и метилирования, а также участие микроРНК выполняют важную функцию в эпигенетической регуляции экспрессии генов и метаболизма скелетных мышц при различных функциональных состояниях человека [8, 14].
Классическая триада РНК-компонентов синтеза белков (информационные, транспортные и рибосомальные) постепенно расширялась, и выявлялись новые некодирующие белок РНК с различными функциями. Решающий прорыв произошел в начале 2000 годов, когда был открыт класс малых РНК, микроРНК, содержащих ~22 нуклеотида. Эта группа некодирующих белок РНК принимает участие в регуляции многих клеточных процессов у разных представителей животного и растительного мира. Синтез микроРНК включает несколько этапов [15]. На первом этапе фермент РНК полимераза II синтезирует длинную двухцепочечную первичную примикроРНК из независимых геномных транскрипционных единиц или из интронов белок-кодирующих генов. На втором этапе фермент РНКаза III – Drоsha вырезает более короткую премикроРНК из примикроРНК, которая состоит из 60 нуклеотидов. Из одной примикроРНК может быть образована одна или несколько премикроРНК. Фермент Drosha является компонентом большого комплекса-микропроцессора. Этот ядерный комплекс существует в двух формах: структура 600 кДа, функция которой пока не установлена, и гетеродимер, включающий Drosha и РНК-связывающий белок DGCR8. Субклеточное распределение микроРНК-синтезирующих ферментов и их субстратов показывает, что примикроРНК образуются в ядрах, тогда как премикроРНК используются в цитоплазме. Транспорт премикроРНК из ядра в цитоплазму через ядерные поры происходит с участием ядерного транспортного рецептора Экспортина 5. Кроме транспортной функции белок Экспортин 5 защищает премикроРНК от деградации экзонуклеазами [16]. В цитоплазме РНКаза III эндонуклеаза Dicer вместе с РНК-связывающим партнером TRBR (транспортным РНК-связывающим белком) вырезают из премикроРНК двуцепочечный фрагмент длиной ~22 нуклеотида. Эта молекула зрелой микроРНК, при участии белков шаперонов HSP70 и HSP90 присоединяется к белку Аргонавту (AGO2). Для связывания с AGO2 на 5' конце молекулы микроРНК имеется домен, состоящий из 6–8 нуклеотидов. Затем происходит разделение нитей РНК, одна из них – проводник переносится на РНК-индуцируемый комплекс выключения гена (RISC), а другая нить РНК подвергается деградации. В дальнейшем зрелая микроРНК в комплексе с RISC через 3'-UTR и очень короткий участок молекулы (seed) взаимодействует с мРНК-мишенью [15]. Активность ферментов Dicer и Drosha может изменяться при взаимодействии с различными факторами. Так, белок холодового шока (RBM3, РНК-связывающий мотив белок 3) способствует усилению ассоциации премикроРНК с Dicer, что приводит к ускорению образования зрелой микроРНК. Кроме того, показано, что белок RBM3 способен участвовать в регуляции размеров мышц [17]. Хотя основное количество микроРНК образуется по стандартному пути, имеется также и альтернативный путь, в котором синтез микроРНК происходит без участия ферментов Drosha и Dicer [18]. Так, синтез miR-451 происходит с участием премикроРНК и AGO2 [19]. Следует отметить, что образование микроРНК может осуществляться также с участием нуклеаз других сигнальных путей, в том числе основного механизма деградации РНК и факторов сплайсинга премикроРНК [15]. МикроРНК осуществляют посттранскрипционный сайленсинг генов – РНК-интерференцию. Они способны регулировать интенсивность процессов транскрипции, процессинга РНК и трансляции посредством комплементарного взаимодействия с ДНК или мРНК в разных органах и тканях человека. По данным биоинформационной оценки мишеней микроРНК до 60% всех генов человека находятся под контролем микроРНК. Каждая микроРНК имеет сайты связывания со многими мРНК, а одна мРНК представляет собой возможную мишень для нескольких микроРНК. Таким образом, микроРНК и мРНК образуют сложную сеть регуляторных взаимодействий, которая участвует в эпигенетической модификации экспрессии генов [20].
В скелетных мышцах тканеспецифическими микроРНК являются miR-1, miR-133a, miR-133b, miR-206, которые получили название миомиРНК. Эти четыре микроРНК принадлежат к семейству miR-1, которое может быть на основе различий специфических последовательностей в структуре молекулы разделено на две группы: miR-1/206 и miR-133a/b. Следует отметить, что микроРНК регулируют основные функции скелетных мышц, такие как миогенез, энергетический метаболизм, синтез белков, гипертрофию и атрофию. Семейство миомиРНК постепенно расширяется и в настоящее время включает 7 микроРНК: 1, 133, 206, 208а, 208b, 486, 499. Важной характеристикой этого класса РНК является возможность одной микроРНК регулировать экспрессию многих других микроРНК и белков-мишеней, которые участвуют в метаболизме скелетных мышц [21, 22]. Это свойство указывает на наличие у некоторых микроРНК избыточных функций, и в этом случае потеря регуляторной функции одной микроРНК не всегда приводит к изменению экспрессии белка-мишени. В процессе метаболизма разные микроРНК взаимодействовуют между собой и совместно создают важную молекулярную систему, которая регулирует и поддерживает клеточный баланс [23]. МикроРНК регулируют метаболизм и биогенез митохондрий не только в скелетных мышцах, но и в других органах и тканях человека [24, 25]. В митохондриях существуют два вида миомиРНК, контролирующих митохондриальный геном: во-первых, ядерные микроРНК, которые транспортируются в митохондрии, и во-вторых, митохондриальные микроРНК, ингибирующие функции тРНК [26]. Открытие локализованных в митохондриях микроРНК, получивших название митомиРНК, поставило вопрос о механизмах их транслокации из цитоплазмы и возможности образования непосредственно с участием митохондриальной ДНК. Эти процессы требуют обеспечения энергией и относятся к АТФ-зависимым. Установлено, что продукция АТФ в процессе окислительного фосфорилирования в митохондриях регулируется с участием miR-156, miR-16, miR-195 и miR-424 [27]. Кроме того, miR-181c, miR-210 и miR-338 регулируют отдельные звенья цепи транспорта электронов в митохондриях [28]. Исследования, выполненные на животных и контингентах людей разного возраста, использовавших различные виды физических нагрузок, показали участие эпигенетической модификации в экспрессии структурных и метаболических генов в скелетных мышцах. Применение однократных и систематических физических нагрузок разной энергетической направленности позволило не только выявить новые миомиРНК, но и определить интенсивность экспрессии генов, участвующих в метаболизме скелетных мышц [27, 29, 30].
В процессе систематического выполнения аэробных физических нагрузок возрастает уровень экспрессии микроРНК в скелетных мышцах, что сопровождается увеличением биогенеза митохондрий, повышением содержания ферментов, обеспечивающих аэробные метаболические реакции и расширением сети капилляров [31–33]. Каждая микроРНК имеет много мишеней, и это затрудняет определение влияния отдельных молекул в разных условиях метаболизма тканей и органов человека. Реакция микроРНК в организме человека на разные внешние воздействия существенно различается, на что указывают результаты, полученные при разных видах физических нагрузок и применения разных пищевых веществ [34–36]. Можно предположить, что по мере расширения диапазона исследований микроРНК будут использоваться в качестве эпигенетических маркеров разных фенотипов и в частности, для выявления повреждений скелетных мышц и для оценки реакции организма на адекватность физической нагрузки функциональному состоянию человека [20].
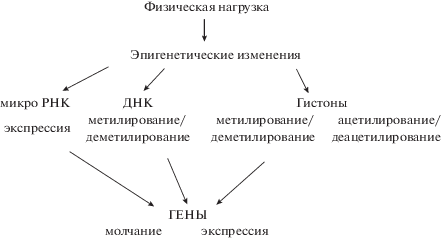
Участники эпигенетической регуляции метаболизма в скелетных мышцах представлены на рис. 1. Первым компонентом сложной системы эпигенетической регуляции метаболизма скелетных мышц является молекула ДНК, на которой происходят реакции метилирования и деметилирования с участием семейств специфических ферментов. Второй компонент включает гистоны и регуляторные белки, модификация которых осуществляется в реакциях ацетилирования, метилирования и фосфорилирования, что приводит к посттрансляционной модификации белков и вызывает изменение экспрессии регуляторных и метаболических генов. В качестве третьего компонента выступает многочисленное семейство микроРНК, которые участвуют в регуляции экспрессии генов и при взаимодействии с мРНК осуществляют контроль процесса трансляции и деградации зрелых мРНК.
В заключение необходимо отметить, что за последние два года заметно увеличился поток статей по теме эпигенетическая регуляция. Это связано не только с повышенным интересом к данному научному направлению, но и с организацией международного консорциума эпигеном человека (www.cell.com/consortium/IHEC). Состав консорциума и основные направления работы подробно изложены на сайте (http://ihec-epigenomes.org). Среди научных направлений планируется интеграция эпигеномики с другими омикс-технологиями, такими как протеомика, транскриптомика, секретомика, фосфопротеомика и ацетиломика, метаболомика и липидомика. Участие этих технологий в физиологии мышечной деятельности подробно изложено в обзоре [10].
Дальнейший прогресс в физиологии мышечной деятельности человека возможен при объединении и анализе данных геномики, эпигеномики и транскриптомики с привлечением инструментов биоинформатики, что позволит расширить представление о молекулярных механизмах, регулирующих метаболизм в скелетных мышцах человека при различных функциональных состояниях. Широкий диапазон метаболизма скелетных мышц при выполнении различных по интенсивности и длительности физических нагрузок создает возможности для улучшения функциональной деятельности и дальнейшего более глубокого изучениям молекулярных механизмов эпигенетической регуляции.
Список литературы
Su X., Wellen K.E., Rabinowitz J.D. Metabolic control of methylation and acetylation. Current Opinion in Chem. Biol. 30: 52–60. 2016.
Dean W. Pathways of DNA demetylation. Adv. Exp. Med. Biol. 945: 247–274. 2016.
Yin X., Xu Y. Structure and function of TET enzymes. Adv. Exp. Med. Biol. 945: 275–302. 2016.
Ravichandran M., Jurkowska R.Z., Jurkowski T.P. Target specificity of mammalian DNA methylation and demethylation machinery. Org. Biomol. Chem. 16(9): 1419–1435. 2018. https://doi.org/10.1039/c7ob02574b
Sharples A.P., Stewart C.E., Seaborne R.A. Does skeletal muscle have an epi-memory? The role of epigenetics in nutritional programming, metabolic disease, aging and exercise. Aging Cell. 15: 603–616. 2016.
Begue G., Rane U., Jemiolo B., Trappe S. DNA mutilation assessment from human slow-and fast-twitch skeletal muscular fibers. J. Apply. Physiol. 122(4): 952–967. 2017.
Barres R., Yan J., Edan B., Treebak J.T., Rasmunssen M., Fritz T., Caidahl K., Krook A., O’Gorman D.J., Zierath J.R. Acute exercise remodels promoter methylation in human skeletal muscule. Cell Metab. 15(3): 405–411. 2012.
Howletd K.F., McGee S.L. Epigenetic regulation of skeletal muscle metabolism. Clin. Sci. 130: 1051–1063. 2016.
Lindholm M.E., Marabita F., Gomez-Cabrero D., Rundqvist H., Ekstrom T.J., Tenger J., Sudberg C.I. An integrative analysis reveals coordinates reprogramming of the epigenome and the transcriptome in human skeletal muscle offer training. Epigenetics. 9(12): 1557–1569. 2014.
Hoffman N.J. Omics and exercise: Global approaches for mapping exercise biological networks. Cold Spring Harbor Perspect Med. 7. a 029884. 2017.
Ling C., Ronn T. Epigenetic adaptation to regular exercise in human. Drug Discov. Today. 19(7): 1015–1018. 2014.
Rasmussen M., Zierath J., Barres R. Dynamic epigenetic responses to muscle contraction. Drug Discov. Today. 19(7): 1010–1014. 2014.
Астратенкова И.В., Рогозкин В.А. Роль ацетилирования/деацетилирования гистонов и транскрипционных факторов в регуляции метаболизма в скелетных мышцах. Рос. физиол. журн. им. И.М. Сеченова. 103(6): 593–605. 2017. [Astratenkova I.V., Rogozkin V.A. The role of acetylation/deacetylation of histones and transcription factors in the regulation of skeletal muscle metabolism. Russ. J. Physiol. 103(6): 593–605. 2017. (In Russ.)].
McGee S.L., Walder K.R. Exercise and the skeletal muscle epigenome. Cold Spring Harb. Perspect. Med. 7(9). a029876. 2017.
Ameres S., Zamore P. Diversifying microRNA sequence and function. Nat. Rev. Biol. Mol. Cell. 14(8): 475–488. 2013.
Finnegan E.F., Pasquinelli A.E. MicroRNA biogenesis: Regulating the regulators. Crit. Rev. Biochem. Mol. Biol. 48(1): 51–68. 2013.
Pilotte J., Dupont-Versteegden E.E., Vanderklish P.W. Widespread regulation of miRNA biogenesis at the Dicer step by the cold-inducible RNA-binding protein, RBM3. PLoS ONE. 6(12): e8446. 2011.
Langenberger D., Cakir M.V., Hoffman S., Slaider P.F. Dicer-processed small RNAs rules and exceptions. J. Exp. Zool. B Mol. Dev. Evol. 320(1): 35–46. 2013.
Cheloufi S., Dos Santos C.O., Chong M.M., Hannon G.J. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis. Nature. 465(7296): 584–589. 2010.
Астратенкова И.В., Рогозкин В.А. Участие микроРНК в регуляции метаболизма скелетных мышц. Рос. физиол. журн. им. И.М. Сеченова. 101(7): 745–757. 2015. [Astratenkova I.V., Rogozkin V.A. The role of microRNAs in the regulation of muscle metabolism. Russ. J. Physiol. 101(7): 745–757. 2015. (In Russ.)].
Pasquielli A.E. MicroRNAs and their targets: recognition, regulation and an emerging reciprocal relationship. Nat. Rev. Genet. 13(4): 271–282. 2012.
Roberts T.C., Godfrey C., McClorey G., Vader P., Briggs D., Gardinre C. Extracellular microRNAs are dynamic non-vesicular biomarkers of muscle turnover. Nucleic Acids Res. 41(20): 9500–9513. 2013.
Aoi W. Frontier impact of microRNAs in skeletal muscle research: A future perspective. Front Physiol. 5: 495. 2014.
Bandiera S., Mategot R., Girard M., Demongeot J., Herion-Caude A. MitomiRs delineating the intracellular localization of microRNAs at mitochondria. Free Radic. Biol. Med. 64: 12–19. 2013.
Tomasetti M., Neuzil J., Dong L. MicroRNAs as regulators of mitochondrial function: Role in cancer suppression. Biochem. Biophys. Acta. 1840(4): 1441–1453. 2014.
Demongeot J., Hazgui H., Bandiera S., Cohen O., Herion-Caude A. MitomiRs, chloromiRs and modeling of the microRNA inhibition. Acta Biother. 61(3): 367–383. 2013.
Duare F., Palmeira C., Rolo A. The role of microRNAs in mitochondria: Small players acting wide. Genes. 5 (4): 865–886. 2014.
Das S., Ferlito M., Kent O.A., Fox-Talbot K., Wang R., Liu D., Raghavachari N., Yang Y., Wheelan S.J., Murphy E. Nuclear miRNA regulates the mitochondrial genome in the heart. Circ. Res. 110 (12): 1596–1603. 2012.
Kirby T.J., Chaillou T., McCarthy J.J. The role of microRNAs in skeletal muscle health and disease. Front. Biosci. 20: 37–77. 2015.
Rowlands D.S., Page R.A., Sukala W.R., Giri M., Ghimbovschi S.D., Hayat I. Multi-Omic integrated networks connect DNA methylation and microRNA with skeletal muscle plasticity to chronic exercise in type 2 diabetic obesity. Physiol. Genomics. 46(20): 747–765. 2014.
Nielsen S., Scheele C., Yfanti C., Akerstrom T., Nielsen A.R., Pedersen B.K. Muscle specific microRNAs are regulated by endurance exercise in human skeletal muscle. J. Physiol. (London) 588(20): 4029–4037. 2010.
Russell A.P., Lamon S., Boon H., Wada S., Guller I., Brown E.L., Chibalin A.V. Regulation of miRNAs in human skeletal muscle following acute endurance exercise and short-term endurance training. J. Physiol. 591(18): 4637–4653. 2013.
van Rooij E., Quiat D., Johnson B.A., Suthherland L.B., Qi X., Richardson J.A. A family of microRNAs encoded by myosin genes governs myosin expression and muscle performance. Dev. Cell. 17(5): 662–673. 2009.
Cui S., Wang C., Yin X., Tian D., Lu Q., Zhang C., Chen X., Ma J. Similar responses of circulating microRNAs to acute high-intensity interval exercise and vigorous-intensity continuous exercise. Front. Physiol. 7. 102. 2016.
Sapp R.M., Shill D.D., Rofh S.M., Hagberg J.M. Circulating microRNAs in acute and chronic exercise: More than mere biomarkers. J. Appl. Physiol. 122: 702–717. 2017.
Wardle S., Baiey M.E., Kilikevicius A., Malkova D., Wilson R.H., Venckunas T., Morgan C.N. Plasma microRNA levels differ between endurance and strength athletes. PLoS One. 10(4): e0122107. 2015.
Дополнительные материалы отсутствуют.
Инструменты
Российский физиологический журнал им. И.М. Сеченова