Российский физиологический журнал им. И.М. Сеченова, 2023, T. 109, № 12, стр. 1856-1869
Экспрессия генов апоптоза и аутофагии в клетках HeLa и HEK 293 в условиях недостатка питательных веществ
А. Д. Трубникова 1, Е. С. Прокопенко 1, Т. В. Соколова 1, О. В. Надей 1, Н. И. Агалакова 1, *
1 Институт эволюционной физиологии и биохимии им. И.М. Сеченова РАН
Санкт-Петербург, Россия
* E-mail: nagalak@mail.ru
Поступила в редакцию 10.10.2023
После доработки 25.10.2023
Принята к публикации 26.10.2023
- EDN: CHNZTJ
- DOI: 10.31857/S0869813923120117
Аннотация
Целью исследования было сравнение степени развития аутофагии в клетках карциномы шейки матки человека HeLa-V и HeLa-R и в неопухолевых клетках эмбриональных почек человека HEK 293 в условиях голодания двух типов – 24- и 48-часового культивирования в среде DMЕM без сыворотки и 4-часовой инкубации в минимальной среде Эрла. В работе оценивали жизнеспособность клеток методом МТТ и экспрессию генов апоптоза (BCL2, BAX, CASP3) и аутофагии (ULK1, BECN1, ATG5, ATG14, MAP1LC3B) методом ПЦР в реальном времени. Культивирование в условиях сывороточного голодания и в среде Эрла привело к значительному снижению жизнеспособности клеток HEK 293, но не оказало влияния на клетки HeLa-V и HeLa-R. В опухолевых клетках обеих линий увеличивалась экспрессия гена антиапоптотического белка BCL2, в то время как в клетках HEK 293 снижалось соотношение генов BCL2/BAX и активировался ген CASP3. В клетках HeLa-V и HeLa-R в условиях недостатка питательных веществ наблюдались различные комбинации стимуляции генов начальных этапов аутофагии ULK1, BECN1, ATG5 и ATG14, но ни один из вариантов обработки не влиял на экспрессию гена MAP1LC3B. В клетках HEK 293 сывороточное голодание привело к увеличению уровня экспрессии генов BECN1, ATG5, ATG14 и MAP1LC3B. Таким образом, стимуляция аутофагии в клетках HeLa, особенно HeLa-R, препятствует развитию процессов апоптоза, в то время как в клетках HEK 293 процессы апоптоза и аутофагии происходят параллельно. Культивирование в среде DMEM без сыворотки в течение 48 ч является наиболее эффективным способом индукции аутофагии в клетках опухолевых линий и соответственно наиболее подходящей моделью для изучения роли аутофагии в развитии их резистентности к апоптотическому пути гибели.
ВВЕДЕНИЕ
В течение многих лет для лечения онкологических заболеваний используются химиотерапевтические препараты, снижающие образование метастазов и увеличивающие срок жизни пациентов. Однако высокая токсичность таких препаратов и появление фенотипов опухолей, устойчивых к применяемой терапии, стали серьезной клинической проблемой [1]. Поэтому одной из важнейших задач в настоящее время является определение путей взаимодействия между гибелью опухолевых клеток и механизмами их выживания, определяющими эффективность лечения. Кроме того, для снижения доз цитотоксических препаратов необходима разработка новых типов комбинированной терапии из соединений, действующих на различные внутриклеточные мишени раковых клеток.
Одним из механизмов, позволяющим раковым клеткам избегать гибели (апоптоза), может быть аутофагия – процесс, при котором поврежденные или ненужные белки, или даже целые органеллы, доставляют в лизосомы для деградации и элиминации [2, 3]. Адаптируя внутриклеточные процессы к недостатку энергетических ресурсов или стрессу (повреждения ДНК, увеличение продукции свободных радикалов), аутофагия активируется как альтернативный источник метаболитов для пролиферации раковых клеток, поэтому они способны выживать в центральных областях опухолей, которые обычно слабо васкуляризованы и плохо снабжаются питательными веществами и кислородом. Утилизируя поврежденные органеллы и накопившиеся токсичные белки, аутофагия способствует росту опухоли. Часто химиотерапевтические препараты индуцируют накопление аутофагосом в клетках раковых линий, что является причиной развития резистентности к такому воздействию [4–6]. Поэтому изучение механизмов аутофагии и ее ингибирование фармакологическими препаратами или генетическими манипуляциями представляет интерес как метод повышения чувствительности раковых клеток к цитотоксическим воздействиям противоопухолевых препаратов и открывает перспективы для разработки новых средств уничтожения раковых клеток.
Выбор экспериментальных условий культивирования клеток, наиболее близких к условиям in vivo и адекватных для изучения эффективности фармакологических препаратов, является ключевым этапом любого исследования. В случае аутофагии это должны быть такие условия, при которых ее стимуляция происходит раньше, чем активация апоптоза [7]. Кроме того, время обработки клеток должно быть таким, чтобы аутофагия сама по себе не стала причиной их гибели, они должны иметь возможность восполнить недостаток питательных веществ и вернуться к нормальной жизнедеятельности, поэтому необходимо индуцировать только начальные этапы этого процесса, не допуская критического уровня накопления аутофагосом. Одним из традиционных способов индуцировать аутофагию является голодание, в частности, культивирование в средах без сывороток, глюкозы, аминокислот и витаминов [8]. В этом случае аутофагия необходима для адаптации к временному недостатку питательных веществ. В предыдущих работах для изучения процессов аутофагии и динамики образования аутофагосом и аутолизосом в клетках HeLa и HEK 293 использовали инкубацию в средах Хэнкса или Эрла, в том числе в комбинации с ионизирующим излучением [9–11]. Целью данного исследования было сравнить интенсивность процессов аутофагии и апоптоза в клетках карциномы шейки матки HeLa (HeLa-R и HeLa-V) и клетках эмбриональных почек человека HEK 293 в условиях недостатка питательных веществ двух типов (культивирование в среде DMEM без сыворотки и в минимальной среде Эрла) для выявления экспериментальных условий, наиболее подходящих для последующего поиска способов сенсибилизации к противоопухолевым препаратам. В работе сравнивали жизнеспособность клеток и оценивали изменения экспрессии генов, кодирующих маркеры апоптоза (BAX, BCL2, CASP3) и аутофагии (ULK1, ATG5, ATG14, BECN1 и MAP1LC3B).
МЕТОДЫ ИССЛЕДОВАНИЯ
Клеточные линии HeLa-V и HEK 293 были получены из коллекции клеточных культур фирмы “Биолот” (Россия). Клетки HeLa-R были любезно предоставлены А.П. Трашковым (Центр доклинических и клинических исследований, НИЦ “Курчатовский институт” – ПИЯФ). Клетки выращивали в среде DMEM (4.5 г/л глюкозы) (Биолот, Россия), содержащей 10% эмбриональной телячьей сыворотки, 100 ЕД/мл пенициллина и 100 мкг/мл стрептомицина (Биолот, Россия) при температуре 37°C и 5% CO2 до достижения плотности 60–70% монослоя. Пассирование проводили с помощью раствора Трипсина-Версена (1 : 1).
Клетки рассеивали на 24-, 12- или 6-луночные культуральные планшеты (Jet Biofil, Китай) с плотностью ~30 × 103, 70 × 103 или 150 × 103 клеток на лунку соответственно. Через сутки опытные лунки дважды промывали буфером DPBS (Биолот, Россия) и заменяли среду на DMEM без сыворотки или на среду Эрла (Биолот, Россия), представляющей собой раствор неорганических солей. В бессывороточной среде DMEM клетки выращивали в течение 24 и 48 ч, в среде Эрла – в течение 4 ч. После этого оценивали их жизнеспособность с помощью МТТ-теста или лизировали для выделения РНК. Каждый эксперимент проводили не менее 4 раз.
Жизнеспособность клеток определяли методом МТТ, который основан на способности живых клеток превращать желтый тетразолиевый краситель MTT (3-(4,5-диметилтетразол-2-ил)-2,5-дифенил-тетразолиум бромид) в фиолетовый кристаллический продукт формазан. Раствор МТТ (5 мг/мл PBS) вносили в каждую лунку планшета в конечной концентрации 0.056% за 2 ч до окончания эксперимента. Для растворения кристаллов формазана в лунки добавляли лизирующий раствор (20% SDS, 50% N,N-диметилформамида на соляной кислоте) и оставляли в инкубаторе на 24 ч. Интенсивность фиолетового окрашивания определяли на планшетном ридере CLARIOstar Plus (BMG Labtech, Германия) при длине волны 570 нм. Процент жизнеспособных клеток в каждой опытной лунке рассчитывали, принимая экстинкцию контрольных клеток за 100%.
Для выделения суммарной РНК монослой клеток лизировали с помощью однофазного водного реагента ExtractRNA (Евроген, Россия) из расчета 1 мл на 10 см2 поверхности лунки. Полученную суспензию центрифугировали в течение 10 мин при 4°С и 12 000 g. Для экстракции РНК к супернатанту добавляли хлороформ (0.2 мл на 1 мл раствора ExtractRNA), инкубировали в течение 5–6 мин при комнатной температуре и центрифугировали 15 мин при 4°С и 12 000 g. К водной фазе, содержащей РНК, добавляли изопропанол (0.5 мл на 1 мл раствора ExtractRNA), инкубировали 10 мин при комнатной температуре, центрифугировали 10 мин при 4°С и 12 000 g и удаляли супернатант. Осадок заливали 75%-ным этиловым спиртом и хранили при –20°С. Общее содержание РНК в пробах (поглощение при 260 нм) и чистоту (отношение коэффициентов поглощения 260/280 нм) оценивали с помощью спектрофотометра NanoPhotometer–N50 (IMPLEN, Германия). Коэффициент поглощения при 260/280 нм превышал 1.8 во всех образцах, что указывало на их высокую чистоту.
Для синтеза кДНК РНК (1 мкг) смешивали с 1 мкл смеси случайных декануклеотидных праймеров (Random(dN)10-primer) и инкубировали 2 мин при 75°С, реакцию останавливали на льду. К смеси добавляли 4 мкл Storage Buffer 5х (Евроген, Россия), 2 мкл смеси дезоксинуклеотидов (dNTP), 2 мкл дитиотреитола (DTT), 2 мкл деионизированной воды (dH2O) и 1 мкл MMLV ревертазы из набора MMLV RT (Евроген, Россия). Реакционную смесь инкубировали в течение 5 мин при 25°C, 60 мин при 42°C и 5 мин при 70°C. Полученную кДНК разбавляли в 10 раз dH2O и хранили при –20°C.
Все реакции ПЦР проводили в режиме реального времени на термоциклере C1000 Touch, совмещенном с блоком обнаружения CFX96 (Bio-Rad Laboratories, Inc., Геркулес, Калифорния, США). Реакционная смесь объемом 25 мкл содержала 17 мкл dH2O, 5 мкл qPCRmix-HS SYBR Master Mix (Евроген, Россия), по 1 мкл прямого и обратного праймеров (10 нМ) и 1 мкл раствора исследуемого образца кДНК. Программа амплификации включала начальную денатурацию при 95°C в течение 5 мин и 45 циклов амплификации, каждый из которых состоял из этапа денатурации при 95°C в течение 5 с, этапа отжига при 57–63°C в течение 10–50 с и этапа элонгации при 72°C в течение 30 с. Интенсивность флуоресценции регистрировали в конце каждого этапа элонгации. Результаты ПЦР анализировали с помощью программного обеспечения CFX Manager. Специфичность амплификации определяли по количеству пиков кривых плавления. Для этого ампликоны подвергали термической денатурации, включающей инкубацию при 65°C в течение 5 с и медленное нагревание до 95°C, после чего измеряли флуоресценцию. Все реакции проводили в трех повторах. Все гены также были проанализированы без ДНК-матрицы.
Последовательности использованных праймеров (синтезированных компанией “Евроген”, Россия) представлены в табл. 1. Температура и продолжительность отжига праймеров варьировались в зависимости от оптимальных условий для каждой пары праймеров. Для анализа были отобраны пары праймеров, имеющие эффективность амплификации, близкую к идеальному значению 2, и коэффициент корреляции 0.98 или выше. Эффективность амплификации тестируемых праймеров определяли путем трехкратных последовательных разведений кДНК (с коэффициентом разведения 10), для каждого разведения выполняли три повтора. Температура плавления была оптимизирована с помощью программы Primer Blast tool (https://www.ncbi.nlm.nih.gov/tools/primer-blast/). Содержание GC составляло 40–60%, размер продукта был ограничен 70–250 парами оснований. Значения Ct для каждого гена были менее 45. Значения Ct, полученные для каждого набора праймеров, использовали для построения стандартных кривых в зависимости от концентраций кДНК. Эффективность амплификации рассчитывали по формуле E = 10(1/–α), где α – угол наклона стандартной кривой.
Таблица 1.
Нуклеотидные последовательности, температура и время отжига праймеров, использованных для ПЦР-анализа
| Ген | Номер последовательности гена (NCBI) | Прямой праймер (5'→3') Обратный праймер (3′→5′) |
Температура (°С) и время отжига (с) праймеров |
|---|---|---|---|
| BAX | NM_001291429.2 | Forward: TCCCCCCGAGAGGTCTTTT Reverse: CGGCCCCAGTTGAAGTTG |
57, 10 |
| BCL2 | NM_000633.3 | Forward: AGTACCTGAACCGGCACCT Reverse: GGCCGTACAGTTCCACAAA |
57, 50 |
| CASP3 | NM_032991.2 | Forward: AACTGGACTGTGGCATTGAG Reverse: ACAAAGCGACTGGATGAACC |
56, 30 |
| ULK1 | NM_003565.4 | Forward: GGCAAGTTCGAGTTCTCCCG Reverse: CGACCTCCAAATCGTGCTTCT |
59, 50 |
| ATG5 | NM_004849.4 | Forward: GTATCATCCCACAGCCAAC Reverse: GCAAAGTAAGACCAGCCC |
53, 50 |
| ATG14 | NM_014924.5 | Forward: GCGCCAAATGCGTTCAGAG Reverse: AGTCGGCTTAACCTTTCCTTCT |
57, 30 |
| BECN1 | NM_001313998.2 | Forward: CCATGCAGGTGAGCTTCGT Reverse: GAATCTGCGAGAGACACCATC |
57, 50 |
| MAP1LC3B | NM_022818.5 | Forward: TTCAGGTTCACAAAACCCGC Reverse: TCTCACACAGCCCGTTTACC |
57, 30 |
| ERN1 | NM_001433.5 | Forward: CAGCAGACTTTGTCATCGGC Reverse: CTCTCGGGTTTTGGTGTCGT |
59, 30 |
| EIF2AK3 | NM_004836.7 | Forward: TGTCGCCAATGGGATAGTGACGAA Reverse: AATCCGGCTCTCGTTTCCATGTCT |
59, 20 |
| CNOT4* | NM_001393371.1 | Forward: GTCCAAAACCTGACTGCATGTATC Reverse: GGTGTTTACCCGCCTGCAT |
57, 10 |
Относительную экспрессию целевых генов оценивали методом 2–ΔΔCT [12]. Выбор референсных генов не проводили, для нормализации результатов ПЦР использовали ген СNOT4, который, согласно литературным данным, стабильно экспрессируется как в нормальных, так и в раковых клетках [13]. Результаты выражали в относительных единицах, где уровень экспрессии в контрольном образце принимался за единицу.
Результаты работы анализировали в программе GraphPad Prism 6 (San Diego, Калифорния, США) с применением теста Манна–Уитни с предварительной проверкой на выбросы и нормальность распределения. Данные представлены в виде графиков boxplot, показаны медиана, верхний и нижний квартили, максимальное и минимальное значения, а также индивидуальные значения. Различия считались достоверными при р < 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Результаты теста МТТ показали, что выживаемость клеток обеих опухолевых линий HeLa-V и HeLa-R в условиях длительного голодания (24- и 48-часового культивирования в среде без сыворотки) не отличалась достоверно от таковой в полной ростовой среде (рис. 1). Жизнеспособность клеток неопухолевой линии HEK 293 снижалась до 62 и 53% после 24- и 48-часовой инкубации в бессывороточной среде соответственно.
Рис. 1.
Жизнеспособность клеток HeLa-V, HeLa-R и HEK 293 после культивирования в среде DMEM без сыворотки (SF) в течение 24 и 48 ч и в среде Эрла в течение 4 ч. Представлены медианы и межквартильные диапазоны (n = 9–14) для каждой линии клеток. Уровень жизнеспособности клеток, растущих в полной среде (контроль), принят за 100% и показан в виде прерывистой линии. ** р < 0.01, *** р < 0.001 по сравнению с контролем.
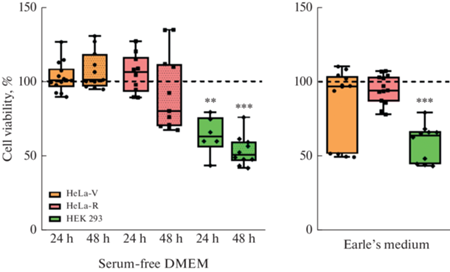
Культивирование клеток HeLa-R в среде Эрла в течение 4 ч также не оказало влияния на их выживаемость. Жизнеспособность клеток HeLa-V в этой среде в среднем не отличалась достоверно от контроля, однако в некоторых экспериментах она снизилась примерно наполовину. Жизнеспособность клеток HEK 293 в среде Эрла упала до 58%.
Чтобы сравнить чувствительность опухолевых и неопухолевых клеток к разным вариантам недостатка питательных веществ, в работе оценивали экспрессию генов, кодирующих два важнейших белка митохондриального проапоптотического сигнального каскада – Bcl-2 и Bax, и эффекторную каспазу апоптоза – каспазу 3. В клетках линий HeLa-V и HeLa-R уровень экспрессии гена BCL2, кодирующего антиапоптотический белок Bcl-2, оставался стабильным как после 24-часового, так и 48-часового культивирования в среде без сыворотки (рис. 2a). В противоположность этому, длительное голодание (48 ч) привело к статистически достоверному снижению содержания BCL2 мРНК в клетках HEK 293. Инкубация в среде Эрла в течение 4 ч не оказала влияния на активность гена BCL2 в клетках HEK 293, однако привела к значительному увеличению уровня экспрессии этого гена в клетках HeLa-V и HeLa-R.
Рис. 2.
Изменение относительной экспрессии генов BCL2 (a) и BAX (b) в клетках трех линий после 24- и 48-часовой инкубации в бессывороточной среде DMEM или 4-часовой инкубации в среде Эрла. Данные нормализованы относительно референсного гена (CNOT4). Показаны медианы и межквартильные диапазоны (n = 5–7). Con – контроль, FS – среда DMEM без сыворотки, EM – среда Эрла. * р < 0.05, ** p < 0.01 по сравнению с контролем для клеток каждой линии.
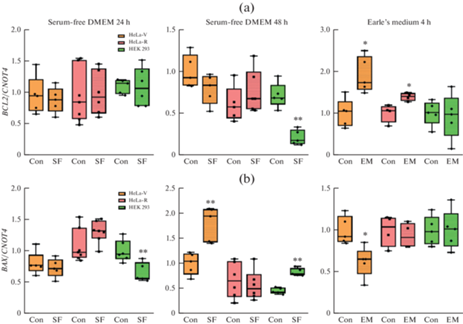
Экспрессия гена BAX в клетках HeLa-V не изменилась после 24-часового, но увеличилась после 48-часового культивирования в среде без сыворотки (рис. 2b). В то же время в клетках HeLa-R уровень ВAX мРНК не отличался от контроля ни после 24, ни после 48 ч голодания. В клетках HEK 293 экспрессия гена BAX снижалась после 24 ч, но увеличивалась после 48 ч культивирования в бессывороточной среде, что хорошо согласуется с изменениями уровня гена BCL2 и снижает соотношение BCL2/BAX в сторону большей активности проапоптотического гена. Инкубация в среде Эрла не оказала влияния на активность BAX в клетках HeLa-R и HEK 293, но привела к снижению уровня экспрессии этого гена в клетках HeLa-V, что увеличивает соотношение BCL2/BAX и указывает на активацию антиапоптотических процессов.
Уровень экспрессии гена CASP3, кодирующего эффекторную каспазу-3, в клетках обеих опухолевых линий (HeLa-V и HeLa-R), культивируемых в среде без сыворотки, не отличалcя от такового в клетках, растущих в контрольных условиях в полной среде (рис. 3). В противоположность этому, содержание мРНК каспазы-3 в клетках HEK 293 значительно увеличивалось как после 24-часового, так и после 48-часового сывороточного голодания, что указывает на активацию апоптотических процессов. Вопреки ожиданиям, в среде Эрла уровень экспрессии гена CASP3 оставался стабильным в клетках всех клеточных линий.
Рис. 3.
Относительная экспрессия гена CASP3 в клетках трех линий в условиях голодания разного типа. Con – контроль, FS – среда DMEM без сыворотки, EM – среда Эрла. * р < 0.05, ** p < 0.01 по сравнению с контролем для клеток каждой линии (n = 5–7).

Для оценки взаимосвязи между культивированием в условиях недостатка питательных веществ и возможной стимуляцией процессов аутофагии в клетках различного происхождения была охарактеризована экспрессия генов, кодирующих некоторые белки ключевых этапов аутофагии.
В клетках HeLa-V уровень мРНК белка Ulk-1, инициирующего процесс аутофагии, принимая сигналы от различных протеинкиназ, вовлеченных в регуляцию клеточного метаболизма, не изменялся ни после 24- или 48-часового культивирования в среде DMEM без сыворотки, ни после 4-часовой обработки в среде Эрла (рис. 4a). В противоположность этому, экспрессия этого гена в клетках HeLa-R значительно увеличивалась в ответ на инкубацию во всех трех средах. В клетках контрольной линии HEK 293 уровень экспрессии гена ULK1 значительно снижался после 24-часового голодания в бессывороточной среде, но не изменялся после 48-часового культивирования в условиях сывороточного голодания и в среде Эрла.
Рис. 4.
Экспрессия генов аутофагии ULK1 (a) и BECN1 (b) после инкубации в среде DMEM без сыворотки в течение 24 и 48 ч или в течение 4 ч в среде Эрла. Данные нормализованы относительно референсного гена (CNOT4). Con – контроль, FS – среда DMEM без сыворотки, EM – среда Эрла. * р < 0.05, ** p < 0.01 по сравнению с контролем для клеток каждой линии (n = 4–7).
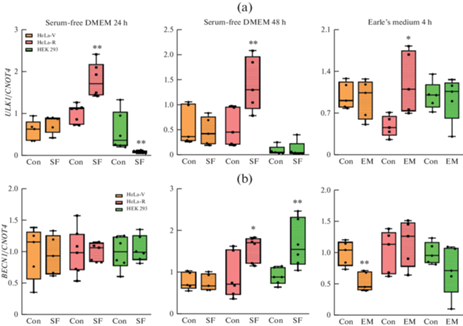
В отличие от ULK1 после 24-часового сывороточного голодания активность гена BECN1, продукт которого Beclin-1 играет ключевую роль в процессе аутофагии, запуская формирование и созревание фагофоры, ни в одной из клеточных линий достоверно не изменялась. 48-часовое голодание не приводило к изменению экспрессии гена BECN1 в клетках HeLa-V, но сопровождалось значительным увеличением уровня мРНК в клетках HeLa-R и HEK 293 (рис. 4b). В среде Эрла уровень экспрессии гена BECN1 не изменялся в клетках HeLa-R и HEK 293, однако снижался в клетках HeLa-V, что, возможно, может быть связано со снижением жизнеспособности этих клеток в некоторых экспериментах (рис. 1).
Экспрессия гена ATG5, кодирующего белок Atg5 (autophagy related 5), повышалась в клетках обеих опухолевых линий HeLa-V и HeLa-R, растущих в бессывороточной среде в течение 24 и 48 ч, и в клетках HeLa-V в среде Эрла (рис. 5a). В клетках HEK 293 экспрессия гена ATG5 увеличивалась только после 2-дневного культивирования в бессывороточной среде.
Рис. 5.
Изменения экспрессии генов аутофагии ATG5 (a) и ATG14 (b) в условиях голодания в культуральных средах разного состава. Данные нормализованы относительно референсного гена (CNOT4). Con – контроль, FS – среда DMEM без сыворотки, EM – среда Эрла. * р < 0.05, ** p < 0.01 по сравнению с контролем для клеток каждой линии (n = 4–6).
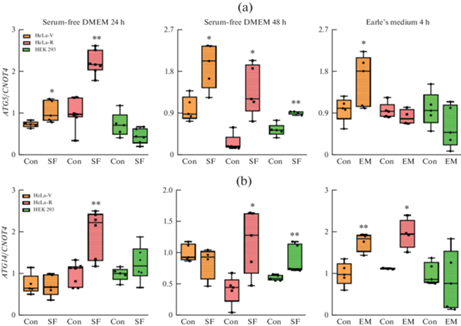
Уровень мРНК гена ATG14, кодирующего белок Atg14 (autophagy related 14), достоверно не изменялся в клетках HeLa-V, культивируемых в условиях сывороточного голодания в течение одних и двух суток, но увеличивался в среде Эрла (рис. 5b). В клетках HeLa-R увеличение экспрессии этого гена наблюдалось после как 24- и 48-часового голодания в бессывороточной среде, так и в среде Эрла. Повышение уровня ATG14 мРНК в клетках HEK 293 наблюдалось только в ответ на двухдневную инкубацию в среде DMEM без сыворотки.
Экспрессия гена MAP1LC3B в клетках HeLa-V и HeLa-R оставалась стабильной независимо от продолжительности и типа недостатка питательных веществ, но достоверно увеличивалась в клетках HEK 293 в ответ на 24- и 48-часовую инкубацию в среде DMEM без сыворотки (рис. 6).
Рис. 6.
Экспрессия гена аутофагии MAP1LC3B в клетках трех линий после инкубации в различных средах. Данные нормализованы относительно референсного гена (CNOT4). Con – контроль, FS – среда DMEM без сыворотки, EM – среда Эрла. ** p < 0.01, *** p < 0.001 по сравнению с контролем для клеток каждой линии (n = 4–6).

ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В работе была проанализирована эффективность индукции аутофагии в клетках карциномы шейки матки человека двух сублиний (HeLa-V и HeLa-R) и неопухолевой клеточной линии эмбриональных почек человека HEK 293, культивируемых в условиях недостатка питательных веществ двух типов (в среде DMEM без сыворотки и в среде Эрла) с целью дальнейшего поиска способов сенсибилизации раковых клеток к противоопухолевым препаратам.
Сравнительный анализ жизнеспособности показал, что клетки опухолевого происхождения HeLa-V и HeLa-R практически не чувствительны к длительному культивированию в среде без сыворотки, а HeLa-R способны выживать в течение 4 ч даже в минимальной среде Эрла (рис. 1), что в целом подтверждает их низкую потребность в питательных веществах. В противоположность этому, выживаемость клеток неопухолевой линии HEK 293 значительно снижалась при всех вариантах инкубации.
Эти результаты подтвердились после оценки экспрессии генов (BCL2, BAX, CASP3), кодирующих три ключевых посредника клеточной гибели (Bcl-2, Bax, каспазу-3) (рис. 2, 3). Белки суперсемейства Bcl-2 (B-cell lymphoma/leukemia-2) располагаются на внешней мембране митохондрий и являются важнейшими регуляторами митохондриального пути апоптоза, функционирующими как активаторы каспаз [14, 15]. Антиапоптотический белок Bcl-2 напрямую связывает проапоптотические белки (в том числе Bax), таким образом обеспечивая целостность митохондриальной мембраны и предотвращая высвобождение нескольких молекул, например цитохрома С, из межмембранного пространства митохондрий в цитозоль. Если уровня Bcl-2 в клетке недостаточно для ингибирования активности Bax, последний олигомеризуется и образует поры на внешней мембране митохондрий, что увеличивает ее проницаемость и приводит к выходу цитохрома С, являясь сигналом к апоптозу. Каспазы, семейство цистеиновых протеаз, играют центральную роль в цепи молекулярных процессов, регулирующих запуск и прогрессирование гибели клеток [16, 17]. В этих каскадах инициаторные каспазы (например, 8 и 9) стимулируются множеством летальных стимулов, генерируемых внутри клеток или на клеточной мембране, в то время как эффекторные каспазы (3 и 7) ответственны за протеолитическое расщепление специфических субстратов и разрушение клеточных компонентов, приводя к морфологическим изменениям, типичным для апоптоза.
Результаты данной работы показали, что длительное культивирование в условиях недостатка питательных веществ не инициирует апоптоз в клетках HeLa-R, они способны увеличивать активность антиапоптотического гена BCL2 даже в минимальной среде Эрла (рис. 2). В клетках HEK 293 и HeLa-V повышение экспрессии гена BАХ на фоне падения уровня BCL2 мРНК в условиях сывороточного голодания, т.е. снижение соотношения BCL2/BAX, косвенно указывает на стимуляцию митохондриального пути апоптоза (рис. 2). В противоположность этому, в среде Эрла соотношение BCL2/BAX в клетках HeLa-V увеличивалось, что указывает на сдвиг между активностью кодируемых ими проапоптотического белка Bax и антиапоптотического белка Bcl-2 в сторону антиапоптотических процессов, увеличивая жизнеспособность клеток этой линии. Однако увеличение экспрессии гена CASP3, кодирующего ключевой эффекторный белок клеточной гибели каспазу-3, наблюдалось только в клетках неопухолевого происхождения HEK 293 (рис. 3). В целом это подтверждает меньшую чувствительность HeLa-V и высокую резистентность клеток HeLa-R к индукции апоптоза, и может объяснить хорошо известную нечувствительность опухолевых клеток к химиотерапевтическим препаратам.
Анализ экспрессии нескольких генов, кодирующих белки ключевых этапов аутофагии (ULK1, BECN1, ATG5, ATG14, MAPLC3B), показал, что метаболическое голодание приводит к активации этого механизма гибели в клетках всех трех линий. Аутофагия не включает активацию каспаз, для нее характерно изменение уровней молекулярных маркеров Ulk1, Beclin-1, белков семейства Atg, процессинг белка LC3 и накопление аутофагосом [18, 19]. Белок Ulk1 активируется в условиях недостатка питательных веществ и является критически необходимым для индукции аутофагии и ранних этапов биогенеза аутофагосом [20]. Ulk1 напрямую фосфорилирует белок Beclin-1 и активирует некоторые другие киназы. Beclin-1 играет ключевую роль в процессе аутофагии, запуская формирование и созревание фагофоры. В комплексе с Bcl-2 Beclin-1 ингибирует аутофагию, а нарушение этого взаимодействия вследствие конкурентного образования связи Bcl-2/Bax ведет к ее активации. Свободный Beclin-2 является ключевым эффектором инициаторного комплекса аутофагии наряду с PI3K киназой и белком р150 [21, 22]. Таким образом, Beclin-1 в комплексе с Bcl-2 функционирует как молекулярный “переключатель” между аутофагией и апоптозом. Белок Atg14 контролирует фосфорилирование Beclin-1 и инициирует его транслокацию из сети Гольджи к аутофагосомам в процессе аутофагии [23, 24]. Белок Atg5 (autophagy related 5) участвует в расширении мембраны фагофоры в аутофагических везикулах. В комплексе с другими членами семейства Atg, например Atg-12, он участвует в процессах инициации и элонгации фагофоры, а на поздних стадиях необходим для образования липидной мембраны аутофагосомы и конъюгации LC3 с фосфатидилэтаноламином мембран [25, 26]. Активный Atg5 предотвращает активацию каспаз и апоптоз, увеличивая интенсивность аутофагии, однако в митохондриях он может действовать и как проапоптотическая молекула. Белок LC3 (Microtubule-associated protein 1A/1B-light chain 3) является одним из центральных структурных белков мембран аутофагосом, участвующим в их биогенезе и отборе субстрата аутофагии [27, 28]. На стадии инициации цитоплазматическая форма LC3А (18 kDa) подвергается протеолизу с присоединением фосфатидилэтаноламина, что приводит к появлению активной формы LC3В (16 kDa), необходимой для удлинения мембраны фагофора, терминального слияния аутофагосомы с мембраной-мишенью и взаимодействия аутофагосомы с компонентами цитоскелета.
В нашей работе время и вариант недостатка питательных веществ (24- или 48-часовое культивирование в среде DMEM без сыворотки или 4 ч в среде Эрла), необходимых для стимуляции аутофагии, а также тип клеток и природа экспрессируемых молекул различались. В неопухолевых клетках HEK 293 однодневное культивирование в бессывороточной среде подавляло активность гена ULK1 (рис. 4), что может быть связано с активацией апоптотических процессов (рис. 3). Однако после 48-часовой инкубации в этих клетках также увеличивалась активность генов BECN1 (рис. 4), ATG5 и ATG14 (рис. 5), что указывает на параллельную или вторичную индукцию процессов аутофагии. Более того, в клетках HEK 293 оба варианта сывороточного голодания привели к увеличению экспрессии ключевого гена аутофагии, маркера зрелых аутофагосом MAP1LC3B (рис. 6). В опухолевых клетках обеих линий наблюдались различные комбинации стимуляции генов начальных этапов аутофагии ULK1 и BECN1 (рис. 4), ATG5 и ATG14 (рис. 5), однако ни один из вариантов метаболического голодания не привел к увеличению экспрессии гена MAP1LC3B, что могло бы указывать на полноценное завершение процесса аутофагии (рис. 6).
Таким образом, стимуляция аутофагии в клетках HeLa, особенно HeLa-R, препятствует развитию процессов апоптоза, в то время как в клетках HEK 293 процессы апоптоза и аутофагии происходят параллельно и приводят к их гибели. Этот феномен по крайней мере частично может объяснить высокую резистентность раковых клеток некоторых типов к химиотерапевтическим препаратам. Сравнительный анализ активации генов аутофагии в разных условиях недостатка питательных веществ показал, что наиболее подходящим вариантом для изучения особенностей этого процесса в опухолевых клетках является 48-часовое культивирование в среде DMEM без сыворотки. Именно такой тип голодания индуцировал начальные этапы аутофагии, но не приводил к ее завершению, что указывает на адаптацию клеток к недостатку питательных веществ, и может быть использован для поиска способов сенсибилизации раковых клеток к цитотоксическим препаратам.
Список литературы
Chern YJ, Tai IT (2020) Adaptive response of resistant cancer cells to chemotherapy. Cancer -Biol Med 17(4): 842–863. https://doi.org/10.20892/j.issn.2095-3941.2020.0005
Noguchi M, Hirata N, Tanaka T, Suizu F, Nakajima H, Chiorini JA (2020) Autophagy as a modulator of cell death machinery. Cell Death Dis 11: 517. https://doi.org/10.1038/s41419-020-2724-5
Thorburn A (2018) Autophagy and disease. J Biol Chem 293(15): 5425–5430. https://doi.org/10.1074/jbc.R117.810739
Goldsmith J, Levine B, Debnath J (2014) Autophagy and cancer metabolism. Methods Enzymol 542: 25–57. https://doi.org/10.1016/B978-0-12-416618-9.00002-9
Zhang T, Yu J, Cheng S, Zhang Y, Zhou CH, Qin J, Luo H (2023) Research Progress on the Anticancer Molecular Mechanism of Targets Regulating Cell Autophagy. Pharmacology 108(3): 224–237. https://doi.org/10.1159/000529279
Rahman MA, Saikat AS, Rahman MS, Islam M, Parvez MA, Kim B (2023) Recent Update and Drug Target in Molecular and Pharmacological Insights into Autophagy Modulation in Cancer Treatment and Future Progress. Cells 12(3): 458. https://doi.org/10.3390/cells12030458
Su M, Mei Y, Sinha S (2013) Role of the Crosstalk between Autophagy and Apoptosis in Cancer. J Oncol 2013: 102735. https://doi.org/10.1155/2013/102735
King KE, Losier TT, Russell RC (2021) Regulation of Autophagy Enzymes by Nutrient Signaling. Trends Biochem Sci 46(8): 687–700. https://doi.org/10.1016/j.tibs.2021.01.006
Kong EY, Cheng SH, Yu KN (2018) Induction of autophagy and interleukin 6 secretion in bystander cells: metabolic cooperation for radiation-induced rescue effect? J Radiat Res 59(2): 129–140. https://doi.org/10.1093/jrr/rrx101
Bampton ET, Goemans CG, Niranjan D, Mizushima N, Tolkovsky AM (2005) The dynamics of autophagy visualized in live cells: from autophagosome formation to fusion with endo/lysosomes. Autophagy 1(1): 23–36. https://doi.org/10.4161/auto.1.1.1495
Chen Y, Azad MB, Gibson SB (2009) Superoxide is the major reactive oxygen species regulating autophagy. Cell Death Differ 16(7): 1040–1052. https://doi.org/10.1038/cdd.2009.49
Racz GA, Nagy N, Tovari J, Apati A, Vertessy BG (2021) Identification of new reference genes with stable expression patterns for gene expression studies using human cancer and normal cell lines. Sci Rep 11: 19459. https://doi.org/10.1038/s41598-021-98869-x
Rao X, Huang X, Zhou Z, Lin X (2013) An improvement of the 2^(-delta delta CT) method for quantitative real-time polymerase chain reaction data analysis. Biostat Bioinforma Biomath 3(3): 71–85.
Eichhorn JM, Alford SE, Sakurikar N, Chambers TC (2014) Molecular analysis of functional redundancy among anti-apoptotic Bcl-2 proteins and its role in cancer cell survival. Exp Cell Res. 322: 415–424. https://doi.org/10.1016/j.yexcr.2014.02.010
Singh R, Letai A, Sarosiek K (2019) Regulation of apoptosis in health and disease: the balancing act of BCL-2 family proteins. Nat Rev Mol Cell Biol. 20: 175–193. https://doi.org/10.1038/s41580-018-0089-8
Dixit VM (2023) The road to death: Caspases, cleavage, and pores. Sci Adv 9(17): eadi2011. https://doi.org/10.1126/sciadv.adi2011
Sahoo G, Samal D, Khandayataray P, Murthy MK (2023) A Review on Caspases: Key Regulators of Biological Activities and Apoptosis. Mol Neurobiol 60(10): 5805–5837. https://doi.org/10.1007/s12035-023-03433-5
Lamark T, Johansen T (2021) Mechanisms of Selective Autophagy. Annu Rev Cell Dev Biol 37: 143–169. https://doi.org/10.1146/annurev-cellbio-120219-035530
Majeed ST, Majeed R, Andrabi KI (2022) Expanding the view of the molecular mechanisms of autophagy pathway. J Cell Physiol 237(8): 3257–3277. https://doi.org/10.1002/jcp.30819
Rong Z, Zheng K, Chen J, Jin X (2022) Function and regulation of ULK1: From physiology to pathology. Gene 840: 146772. https://doi.org/10.1016/j.gene.2022.146772
Vega-Rubín-de-Celis S (2019) The Role of Beclin 1-Dependent Autophagy in Cancer. Biology 9(1): 4. https://doi.org/10.3390/biology9010004
Prerna K, Dubey VK (2022) Beclin1-mediated interplay between autophagy and apoptosis: New understanding. Int J Biol Macromol 204: 258–273. https://doi.org/10.1016/j.ijbiomac.2022.02.005
Menon MB, Dhamija S (2018) Beclin 1 Phosphorylation – at the Center of Autophagy Regulation. Front Cell Dev Biol 6: 137. https://doi.org/10.3389/fcell.2018.00137
Ohashi Y (2021) Activation Mechanisms of the VPS34 Complexes. Cells 10(11): 3124. https://doi.org/10.3390/cells10113124
Zhou P, Zhang Z, Liu M, Li P, Zhu Y (2023) Effects of autophagy‑related gene 5 on tumor development and treatment (Review). Oncol Rep 50(2): 155. https://doi.org/10.3892/or.2023.8592
Changotra H, Kaur S, Yadav SS, Gupta GL, Parkash J, Duseja A (2022) ATG5: A central autophagy regulator implicated in various human diseases. Cell Biochem Funct 40(7): 650–667. https://doi.org/10.1002/cbf.3740
Chen KD, Lin CC, Tsai MC, Huang KT, Chiu KW (2018) Tumor microenvironment mediated by suppression of autophagic flux drives liver malignancy. Biomed J 41(3): 163–168. https://doi.org/10.1016/j.bj.2018.03.002
Tanida I, Ueno T, Kominami E (2004) LC3 conjugation system in mammalian autophagy. Int J Biochem Cell Biol 36(12): 2503–2518. https://doi.org/10.1016/j.biocel.2004.05.009
Дополнительные материалы отсутствуют.
Инструменты
Российский физиологический журнал им. И.М. Сеченова


