Успехи современной биологии, 2020, T. 140, № 4, стр. 333-346
Разнообразие и структура митохондриальных генофондов славян в этногенетическом аспекте
Б. А. Малярчук 1, *, М. В. Деренко 1
1 Институт биологических проблем Севера ДВО РАН
Магадан, Россия
* E-mail: malyarchuk@ibpn.ru
Поступила в редакцию 14.03.2020
После доработки 14.03.2020
Принята к публикации 14.03.2020
Аннотация
Приводится обзор данных об изменчивости нуклеотидных последовательностей целых митохондриальных геномов в популяциях русских, поляков и сербов, представляющих соответственно группы восточных, западных и южных славян, в сравнении с аналогичными данными для других европейских народов. Показано, что результаты анализа изменчивости целых митогеномов позволяют существенно детализировать представления о структуре и разнообразии митохондриальных генофондов, полученные ранее с помощью других подходов, например анализа распределения гаплотипов главной некодирующей области мтДНК или частот митохондриальных гаплогрупп. Продемонстрировано, что байесовский анализ больших наборов данных об изменчивости целых митогеномов у славян позволяет выявлять в Европе межрегиональные различия динамики эффективной численности популяций во времени. Применение филогеографического анализа современных и палеогеномных данных об изменчивости целых митогеномов дает возможность выделять уникальные филогенетические кластеры мтДНК, специфичные для отдельных этнических групп и их общностей. На примере трех этнических групп славян продемонстрирована общность их генетического происхождения, истоки которой уходят в эпохи бронзового и железного веков.
ВВЕДЕНИЕ
Согласно археологическим и палеоантропологическим данным, расселение славян с территории прародины, располагавшейся предположительно в Центральной Европе, наиболее интенсивно происходило в эпоху раннего средневековья (в VI–VII вв. н. э.) и сопровождалось генетическим взаимодействием с народами, населявшими территории миграционных путей славян (Алексеева, 1973; Седов, 1995, 2003). На западе они соприкасались с германскими племенами, на юге – с фракийцами, кельтами и иллирийцами, на севере – с балтами, на востоке – с иранцами и финно-уграми (Алексеева, Алексеев, 1989). Формирование же восточных славян (русских, украинцев и белорусов) происходило в лесной и лесостепной зоне Восточной Европы и сопровождалось ассимиляцией древнейших народов этого региона. Поэтому вполне логичным является ожидание в генофонде современных восточных славян генетических компонентов, принадлежащих и собственно славянам, и финно-угорским, и скифо-сарматским, и летто-литовским народам.
Согласно молекулярно-генетическим данным, распространение славян в эпоху раннего средневековья сопровождалось активной ассимиляцией автохтонных популяций Европы, и поэтому субстратные генетические компоненты преобладают над компонентами, привнесенными собственно славянскими миграциями (Malyarchuk, Derenko, 2001; Malyarchuk et al., 2004; Balanovsky et al., 2008; Woźniak et al., 2010; Morozova et al., 2012; Sarac et al., 2014; Kushniarevich et al., 2015). Благодаря данным об изменчивости микросателлитных локусов Y-хромосомы были выявлены два основных генетических субстрата славян: первый представлен у западных и восточных славян, а также в некоторых южнославянских группах (у словенцев и западных хорватов), а второй характерен для других южнославянских этнических групп (Rebała et al., 2007). Аналогично, по результатам анализа полиморфизма Y-хромосомы и аутосомных локусов, были выявлены: центрально-восточноевропейский генетический субстрат в восточно- и западнославянских популяциях Восточной и Центральной Европы и южно-восточноевропейский субстрат – у южных славян Балканского полуострова (Kushniarevich et al., 2015).
Результаты исследований изменчивости мтДНК оказались менее информативными, что обусловлено, по-видимому, невысокой информативностью использованных в большинстве исследований маркеров мтДНК. В качестве последних главным образом выступали гипервариабельный сегмент 1 (ГВС1) главной некодирующей области и отдельные варианты полиморфизма кодирующей области, определяющие гаплогруппы мтДНК. Тем не менее, именно благодаря исследованиям изменчивости мтДНК получены некоторые важные выводы относительно генетической истории славян. Так, при исследовании полиморфизма современной и древней мтДНК обнаружена (Juras et al., 2014) генетическая преемственность популяций Польши, по меньшей мере, с эпохи римского железного века до настоящего времени. Эти результаты свидетельствуют в пользу гипотезы о формировании первых славянских групп на территории между рр. Одер и Висла (Лер-Сплавинский, 1960; Седов, 1979).
Ранее по данным об изменчивости нуклеотидных последовательностей ГВС1 мтДНК было показано, что межэтнические взаимодействия (смешение и ассимиляция) играли большую роль в формировании различных групп славян (Малярчук, 2001). Так, западные славяне, несмотря на их центральное положение среди славян в целом, проявляют выраженное сходство с германскими народами, южные славяне генетически родственны неславянским народам Балкан, а русские – генетически сходны с финно-угорскими популяциями Восточной Европы. На основании данных об изменчивости мтДНК, Y-хромосомы и аутосомных локусов было обнаружено также, что русские популяции центральной и южной частей Восточной Европы отличаются генетически от русских северной части Восточной Европы (Malyarchuk et al., 2004; Balanovsky et al., 2008; Morozova et al., 2012; Khrunin et al., 2013; Kushniarevich et al., 2015). Кроме этого, оказалось, что русские популяции Северо-Запада европейской части России по распределению митохондриальных линий близки популяциям Северо-Восточной Польши и Прибалтики (Grzybowski et al., 2007). Южнославянские популяции также в большой степени гетерогенны в генетическом отношении (Malyarchuk et al., 2003; Tömöry et al., 2007; Karachanak et al., 2012; Sarac et al., 2014; Davidović et al., 2015; Kushniarevich et al., 2015). Это обусловлено сложной демографической историей народов Балканского полуострова, поскольку в этот регион проникали многие популяции из различных частей Евразии. Так, на протяжении двух последних тысячелетий это были, например, гунны, авары, мадьяры, булгары, готы и, конечно, славяне. Влияние всех этих народов сказалось на генетическом облике современного населения Балканского полуострова.
Таким образом, несмотря на генетическое сходство между различными славянскими популяциями (они всегда кластеризуются вблизи друг от друга по результатам анализа главных компонент или многомерного шкалирования генетических дистанций), между ними существуют и различия, что объясняется проявлением, как отмечалось выше, двух основных субстратов, ассимилированных славянами: центрально-восточноевропейского и южно-восточноевропейского (Kushniarevich et al., 2015). Между тем, в генетическом отношении структура собственно славянского компонента остается загадкой. Очевидно, что для его идентификации нужны более глубокие исследования генофондов (в масштабе изменчивости целых ядерных и митохондриальных геномов) различных групп славян, а также соседних неславянских народов. О реальности существования генетических маркеров славян, по которым можно проследить славянские миграции, свидетельствуют результаты исследований изменчивости целых молекул мтДНК (Малярчук и др., 2017, 2019; Malyarchuk et al., 2008; Mielnik-Sikorska et al., 2013; Davidović et al., 2015; Davidovic et al., 2017). Так, в митохондриальном генофонде русских присутствуют подгруппы мтДНК, характерные для славян в целом (частота этого компонента – 12.2%); эволюционный возраст таких подгрупп мтДНК составил примерно 2.7–3.5 тыс. лет (Малярчук и др., 2019). Полученные в этой работе результаты свидетельствуют о том, что предки славян населяли территории Восточной и Центральной Европы в конце бронзового века и, по-видимому, генетически могли быть связаны с носителями культуры боевых топоров. По всей видимости, популяционно-специфичные маркеры могут быть обнаружены и на Y-хромосоме в случае проведения более детальных популяционных исследований полиморфизма на уровне целых Y-хромосом. Что касается аутосомных локусов, то результаты выполненных к настоящему времени исследований свидетельствуют о том, что сигналы славянской экспансии в раннем средневековье проявляются довольно отчетливо (Ralph, Coop, 2013; Hellenthal et al., 2014; Busby et al., 2015). Однако результаты других работ (Kushniarevich et al., 2015) позволили зарегистрировать лишь очень слабый сигнал в виде общих сегментов ДНК (размером 2–3 сМ), присутствующих как у восточных и западных, так и у южных славян. Таким образом, поиск аутосомных маркеров славян и славянских миграций еще предстоит осуществить в будущих исследованиях.
Несмотря на большое количество митохондриальных геномов, опубликованных в настоящее время (в базе данных MITOMAP (www.mitomap.org) на 1.01.2020 присутствуют 50 175 митогеномов), популяционных наборов данных достаточно большого размера (от 100 митогеномов и выше) довольно мало. Да и сугубо популяционных исследований полногеномной изменчивости мтДНК у европейцев пока выполнено не очень много: сардинцы (Fraumene et al., 2006), поволжские татары (Malyarchuk et al., 2010), русские (Malyarchuk et al., 2017), поляки (Malyarchuk et al., 2017; Skonieczna et al., 2018; Piotrowska-Nowak et al., 2019a,b), сербы (Davidović et al., 2020), финны, итальянцы, датчане (Raule et al., 2014), эстонцы (Stoljarova et al., 2016), венгры (Malyarchuk et al., 2018). Лишь в отдельных случаях в европейских популяциях, например у датчан и сардинцев, определены нуклеотидные последовательности более 1 000 митогеномов (Li et al., 2014; Olivieri et al., 2017).
Целью настоящей работы является, таким образом, обзор данных об изменчивости нуклеотидных последовательностей целых митохондриальных геномов в популяциях восточных, западных и южных славян (на примере русских, поляков и сербов соответственно) в сравнении с другими европейскими народами. Приводятся основные результаты использования данных об изменчивости современных и древних митохондриальных геномов в исследовании истории формирования славян.
РАЗНООБРАЗИЕ МИТОХОНДРИАЛЬНЫХ ГЕНОФОНДОВ СЛАВЯН
Как видно из данных (табл. 1), славянские популяции незначительно отличаются по параметрам генетического разнообразия как друг от друга, так и от других европейских популяций.
Таблица 1.
Генетическое разнообразие и результаты тестирования на нейтральность изменчивости мтДНК в популяциях человека
| Популяция | N | H | S | HD (%) | ND (%) | K | Tajima’s D (p) |
|---|---|---|---|---|---|---|---|
| Русские1 | 376 | 361 | 1097 | 100 ± 0.0 | 0.18 ± 0.01 | 29.02 | –2.57 (<0.001) |
| Поляки1, 2 | 300 | 287 | 1053 | 100 ± 0.0 | 0.19 ± 0.00 | 30.91 | –2.56 (<0.001) |
| Сербы3 | 226 | 207 | 790 | 99.9 ± 0.0 | 0.17 ± 0.08 | 27.82 | –2.51 (<0.001) |
| Венгры4 | 80 | 78 | 475 | 99.9 ± 0.2 | 0.18 ± 0.09 | 30.33 | –2.35 (<0.001) |
| Эстонцы5 | 114 | 108 | 493 | 99.9 ± 0.1 | 0.17 ± 0.08 | 28.39 | –2.32 (<0.001) |
| Татары поволжские6 | 73 | 68 | 509 | 99.8 ± 0.3 | 0.21 ± 0.10 | 35.18 | –2.32 (<0.001) |
| Сардинцы7 | 63 | 50 | 234 | 99.2 ± 0.4 | 0.15 ± 0.01 | 24.31 | –1.78 (<0.05) |
| Тосканцы8 | 118 | 117 | 730 | 99.9 ± 0.1 | 0.19 ± 0.09 | 30.83 | –2.59 (<0.001) |
| Испанцы8 | 101 | 97 | 541 | 99.9 ± 0.1 | 0.18 ± 0.09 | 29.96 | –2.42 (<0.001) |
| Европа9 | 816 | 755 | 1749 | 100 ± 0.0 | 0.18 ± 0.00 | 29.32 | –2.62 (<0.001) |
| Западная Азия9 | 439 | 391 | 1411 | 99.9 ± 0.0 | 0.18 ± 0.00 | 29.92 | –2.65 (<0.001) |
| Сибирь10 | 697 | 395 | 915 | 99.7 ± 0.0 | 0.19 ± 0.00 | 31.48 | –2.26 (<0.001) |
Примечание: N – размер выборки; H – количество выявленных гаплотипов; S – число полиморфных сайтов; HD – гаплотипическое разнообразие и стандартное отклонение (без учета делеционно-инсерционного полиморфизма); ND – нуклеотидное разнообразие и стандартное отклонение (без учета делеционно-инсерционного полиморфизма); K – среднее число попарных нуклеотидных различий; Tajima’s D – результат теста на нейтральность Таджимы и статистическая значимость (p). По данным следующих работ: 1 – Malyarchuk et al., 2017; 2 – Skonieczna et al., 2018; Piotrowska-Nowak et al., 2019b; 3 – Davidović et al., 2020; 4 – Malyarchuk et al., 2018; 5 – Stoljarova et al., 2016; 6 – Malyarchuk et al., 2010; 7 – Fraumene et al., 2006; 8 – проект 1000 Genomes Project (http://www.internationalgenome.org); 9 – Derenko et al., 2018; 10 – Derenko et al., 2019.
Во всех выборках обнаружен высокий уровень генетического разнообразия, определенного как по частотам гаплотипов мтДНК (H, HD), так и по степени попарных нуклеотидных различий между митогеномами (ND, K). Для сравнения в табл. 1 приводятся аналогичные сведения для региональных выборок населения Евразии – европейской, западноазиатской и сибирской. Как видно, сходные значения генетического разнообразия наблюдаются как в этнических, так и в региональных выборках. Следует отметить, что, согласно результатам полномитогеномных исследований, вероятность обнаружения двух одинаковых митогеномов среди неродственных по материнской линии индивидов в популяциях очень низка и составляет, например, для русских 0.28%, N = 376 (Malyarchuk et al., 2017), а для американцев европейского происхождения – 0.39%, N = 263 (Just et al., 2015). Величина этого показателя зависит от размера исследованных популяций: так, для выборки европейцев N = 1281 вероятность составляет уже 0.098%.
Тестирование с помощью теста Таджимы характера изменчивости мтДНК для всех исследованных популяций, кроме небольшой сардинской выборки, показало наличие значимо отрицательных значений D-статистики (p < 0.01), что обусловлено избытком низкочастотных вариантов полиморфизма в популяциях (табл. 1). В рамках существующих моделей (Tajima, 1989, 1993) это свидетельствует о недавней популяционной экспансии, случившейся после эпизода редукции численности (бутылочного горлышка), и/или о воздействии на популяционные генофонды отрицательного (очищающего) отбора. Кроме того, установлено, что величина D-статистики зависит также и от характера мутационного процесса: чем ниже гетерогенность мутационных скоростей в нуклеотидных сайтах, тем ниже величина D (Aris-Brosou, Excoffier, 1996). Таким образом, целый ряд факторов, включая факторы популяционной динамики, может объяснить значимо отрицательные значения D-статистики, наблюдаемые в европейских популяциях, по данным об изменчивости целых митогеномов.
Анализ распределения попарных нуклеотидных различий между последовательностями мтДНК также используется для исследования демографической истории популяций. Согласно результатам моделирования, в состоянии популяционного равновесия наблюдается мультимодальный характер распределения попарных нуклеотидных различий, однако для популяций, прошедших через этапы быстрого роста численности или через экспансию с высокой скоростью миграции между соседними группами особей, характерно унимодальное распределение этого параметра (Rogers, Harpending, 1992; Ray et al., 2003). Проведенный нами анализ показал, что распределение попарных нуклеотидных различий между целыми митогеномами в различных группах населения Европы – у русских, поляков, сербов или европейцев в целом (N = 1281) – имеет строго бимодальный характер (рис. 1). Первый пик на графике функции распределения находится в районе 12–13 нуклеотидных различий, а второй пик – в районе 32–33 нуклеотидных различий.
Рис. 1.
Графики распределений попарных нуклеотидных различий для митохондриальных геномов населения Европы, Западной Азии и Восточной Азии. Для построения использован пакет программ DnaSP 5.10 (Librado, Rozas, 2009). На оси X указаны попарные нуклеотидные различия, на оси Y – их частота.

В более раннем исследовании изменчивости мтДНК у населения США (Just et al., 2015) также было выявлено бимодальное распределение попарных нуклеотидных различий для митогеномов европейского и америндского происхождения и мультимодальное распределение – для митогеномов африканского происхождения. Авторы предположили, что такого рода распределения возникают вследствие главной особенности организации митохондриальных генофондов популяций, а именно существования митохондриальных генофондов в виде гаплогрупп мтДНК, то есть групп филогенетически родственных гаплотипов. Поэтому при попарном сравнении гаплотипов мтДНК должны выявляться, как минимум, две группы попарных нуклеотидных различий: одна из них соответствует различиям между недавно дивергировавшими гаплотипами в пределах гаплогрупп мтДНК, а другая – различиям между гаплогруппами (Just et al., 2015). Мультимодальное распределение попарных нуклеотидных различий мтДНК, наблюдаемое у африканцев, отражает, по мнению авторов (Just et al., 2015), многокомпонентность структуры их митохондриальных генофондов и больший эволюционный возраст африканских генетических линий. Между тем результаты проведенного нами анализа нуклеотидных последовательностей митогеномов населения Евразии показывают строго унимодальный характер распределения попарных нуклеотидных различий у населения Западной Азии (персов, кашкайцев, ливанцев, армян, азербайджанцев, грузин, N = 1085) и Восточной Азии (китайцев, японцев, уйгуров, сибирских этнических групп, N = 2045) (рис. 1). Таким образом, если следовать этой логике (Just et al., 2015), то можно констатировать, что в азиатских популяциях, в отличие от европейских, распределение попарных нуклеотидных различий в пределах гаплогрупп мтДНК соответствует таковому между гаплогруппами. В европейских же популяциях, по всей видимости, баланс в распределении нуклеотидного разнообразия внутри гаплогрупп мтДНК и между ними оказался нарушенным под действием климатических или иных факторов, способствовавших дрейфу генов во время демографических кризисов в истории европейцев.
ДЕМОГРАФИЧЕСКИЙ АНАЛИЗ ИЗМЕНЧИВОСТИ мтДНК В ПОПУЛЯЦИЯХ СЛАВЯН
В последние десятилетия большую популярность приобрел байесовский анализ динамики эффективной численности популяций (Ne), позволяющий реконструировать изменения популяционных размеров в прошлом, основываясь на данных об изменчивости ДНК в современных популяциях (Drummond et al., 2002, 2005). Использование этого подхода в анализе изменчивости мтДНК русских показало (рис. 2), что относительное снижение эффективной численности населения, предкового по отношению к русским, началось примерно 50 тыс. лет назад (95%-ный доверительный интервал (95% ДИ): 46–61 тыс. лет назад) и продолжалось вплоть до примерно 24.5 тыс. лет назад (95% ДИ: 21.7–26.1 тыс. лет назад) (Malyarchuk et al., 2017). Этому периоду соответствует фаза максимального похолодания в Европе во время поздневалдайского оледенения. Последующие изменения численности населения Восточной Европы связаны с ростом Ne, первый максимум которого наблюдается во время потепления примерно 13–11 тыс. лет назад (рис. 2). Последний резкий рост численности (десятикратный) произошел примерно 4.3 тыс. лет назад (95% ДИ: 2.9–5.8 тыс. лет назад) в эпоху бронзового века. Данные археологии и палеогеномики свидетельствуют, что самая значительная экспансия этого времени связана с распространением носителей курганной культуры из Понто-Каспийской степной зоны в соседние регионы Европы и Азии (Gimbutas, 1956; Allentoft et al., 2015; Haak et al., 2015). Таким образом, результаты байесовского анализа динамики Ne свидетельствуют о том, что экспансия населения Восточной Европы, ставшего основой для русских популяций, началась в бронзовом веке и, возможно, была обусловлена интенсификацией миграционных процессов в Европе того времени.
Рис. 2.
Байесовский график динамики эффективной численности популяций во времени по данным изменчивости целых митохондриальных геномов русского населения, N = 376 (Malyarchuk et al., 2017). Сплошная черная линия – медиана, сплошные серые линии – 95% ДИ. График показывает изменение параметра Ne × μ (ось Y) во времени (ось X, в тыс. лет; 0 – настоящее время). В анализе использовано значение скорости накопления мутаций в митохондриальном геноме человека (μ), равное 1.665 × 10–8 замен на нуклеотидную позицию в год (Soares et al., 2009). Использован пакет программ BEAST 1.10.2 (Drummond et al., 2005).

Байесовский анализ изменчивости мтДНК у поляков, N = 300, показал, что локальный минимум эффективной численности предкового по отношению к полякам населения наблюдался примерно 28 тыс. лет назад (95% ДИ: 26.3–28.9 тыс. лет назад), а затем выявляется только рост численности, который еще более усилился примерно 7.6–8.5 тыс. лет назад (рис. 3). Этому мог способствовать климатический оптимум голоцена, а также произошедшие в то время изменения хозяйственного уклада европейского населения, связанные с переходом от охоты и собирательства к сельскому хозяйству. Предполагается, что этому переходу могли поспособствовать произошедшие примерно 8 тыс. лет назад миграции в Европу переселенцев из Анатолии (анатолийских фермеров), где находился один из центров возникновения сельского хозяйства (Hofmanová et al., 2016; Lipson et al., 2017).
Рис. 3.
Байесовский график динамики эффективной численности популяций во времени по данным изменчивости целых митохондриальных геномов поляков, N = 300 (Malyarchuk et al., 2017; Skonieczna et al., 2018; Piotrowska-Nowak et al., 2019b). Обозначения как на рис. 2.
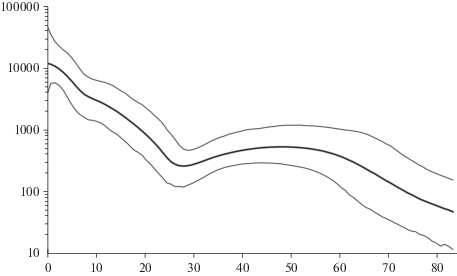
Между тем, байесовский анализ изменчивости мтДНК у сербов, представляющих балканское население, показывает, что резкий рост численности начался примерно 23 тыс. лет тому назад (95% ДИ: 22.3–24.6 тыс. лет назад) и достиг максимума 7.1 тыс. лет назад (95% ДИ: 6.4–7.9 тыс. лет назад) (рис. 4). Однако после этого момента времени наблюдается снижение численности вплоть до раннего средневековья – 1.6 тыс. лет назад (95% ДИ: 0.8–2.4 тыс. лет назад), после чего вновь регистрируется рост численности. Такая динамика Ne может объясняться тем, что в некоторых регионах Европы, действительно, наблюдалась депопуляция энеолитического населения примерно 5–6 тыс. лет назад (например в мегапоселениях балкано-дунайского и соседних восточно-европейских регионов), еще до периода миграций носителей курганной культуры из восточноевропейских степей (Shennan et al., 2013; Müller et al., 2016). Одной из возможных причин депопуляции населения является излишний рост численности и скученность населения, что привело к распространению инфекций, в том числе чумы (Rascovan et al., 2019). Популяционный рост численности предкового по отношению к сербам населения, зарегистрированный примерно 1.6 тыс. лет назад, совпадает с миграционным периодом IV–VII вв., когда на Балканах осели и смешались с автохтонным иллирийским и фракийским населением многие пришлые племена, включая готов, славян и аваров (Седов, 1995). Самые ранние миграции славян на Балканский полуостров датируются первой половиной VI в. – согласно археологическим данным, они были направлены из Подунавья, Прикарпатья и Северного Причерноморья (Седов, 2003).
Рис. 4.
Байесовский график динамики эффективной численности популяций во времени по данным изменчивости целых митохондриальных геномов сербов, N = 226 (Davidović et al., 2020). Обозначения как на рис. 2.

Таким образом, проведенное с помощью анализа изменчивости целых митогеномов исследование динамики эффективной численности популяций славян из различных регионов Европы приводит к различающимся результатам. Общей чертой является рост численности в послеледниковое время, что соответствует результатам проведенных ранее исследований динамики Ne в популяциях Европы и Западной Азии (Zheng et al., 2012; Derenko et al., 2013; Li et al., 2014; Lippold et al., 2014; Karmin et al., 2015; Batini et al., 2017). Однако анализ больших выборок митогеномов из популяций восточных, западных и южных славян позволил выявить региональные особенности динамики Ne: на востоке Европы выявляется резкий рост численности популяций в бронзовом веке, в центре Европы – увеличение численности в энеолите, а на юге Европы – увеличение численности в раннем средневековье, последовавшее после снижения популяционного размера, которое началось еще в энеолите.
Необходимо отметить, что ранее региональные особенности динамики Ne в популяциях Европы не были выявлены, что, скорее всего, связано с небольшими размерами исследованных выборок митогеномов. Так, в исследовании (Batini et al., 2017) размеры выборок из 15 европейских популяций не превышали 20 человек. Авторами этой работы был сделан вывод о том, что в митохондриальных генофондах европейцев нашла отражение только послеледниковая экспансия населения Европы, а сигналы, связанные с миграциями неолитических фермеров из Анатолии и носителей курганной культуры эпохи бронзового века с востока на запад Европы, были утрачены из-за воздействия на мтДНК дрейфа генов. Между тем, результаты недавнего исследования динамики Ne в финской популяции (размеры проанализированных выборок варьировали от 281 до 562 человек для разных видов анализа) показали рост численности населения в последние 4–5 тыс. лет, что, по мнению авторов, связано как с распространением сельского хозяйства, так и с миграциями носителей культуры боевых топоров в эпоху бронзового века (Översti et al., 2017). Результаты исследования динамики Ne для 2000 митогеномов датчан также продемонстрировали периоды спада и роста популяционной численности в последние 3 тыс. лет (Li et al., 2014). Аналогично, анализ изменчивости митогеномов исторических образцов – большой выборки викингов VIII–XI вв., N = 420, также позволил выявить сложную динамику Ne, показавшую рост численности населения, начиная от 15 тыс. лет тому назад, затем резкое ее увеличение – 6–7 тыс. лет назад и спад – примерно 4 тыс. лет назад (Margaryan et al., 2019). Таким образом, по всей видимости, только расширение размеров выборок в исследованиях разнообразия мтДНК позволит в перспективе получить наиболее корректные выводы относительно динамики эффективной численности европейских популяций.
ЭТНОСПЕЦИФИЧНЫЕ КОМПОНЕНТЫ МИТОХОНДРИАЛЬНОГО ГЕНОФОНДА СЛАВЯН
Еще в самых ранних популяционных исследованиях полиморфизма мтДНК человека было установлено, что в связи с наследованием митохондриальных геномов по материнской линии и без рекомбинаций полиморфные варианты мтДНК характеризуются этнорасовой специфичностью распределения (Denaro et al., 1981; Johnson et al., 1983). По мере достижения полномитогеномного уровня исследований изменчивости мтДНК стали выявляться генетические компоненты, специфичные для этнотерриториальных групп или даже отдельных этнических групп. Например, при исследовании полиморфизма мтДНК евреев-ашкенази были обнаружены подгруппы мтДНК, специфичные только для этого народа, и выявлены географические источники происхождения большинства гаплотипов мтДНК ашкенази, что позволило существенно прояснить историю происхождения этого народа (Costa et al., 2013). Очень информативным оказался анализ большого массива данных полномитогеномной изменчивости у населения Сардинии (около 3.5 тыс. митогеномов) (Olivieri et al., 2017). Это исследование показало, что 78.4% митогеномов островитян формируют подгруппы мтДНК, возникшие в разное время в Сардинии (начиная с мезолита) на основе различных предковых линий мтДНК. Высокая информативность анализа в случае сардинцев связана с большим количеством митогеномов и относительной изоляцией населения Сардинии. Между тем, подобного рода анализ 843 митохондриальных геномов финнов, которые тоже характеризуются довольно обособленной популяционной историей, позволил установить, что почти 30% митогеномов финнов относятся к финно-специфичным подгруппам мтДНК (Översti et al., 2017). Примерно треть митогеномов пришлась на этноспецифичную фракцию и у армян, у которых широко распространены эндогамные браки (Derenko et al., 2019).
В отношении русского населения Восточной Европы также проводились подобного рода исследования, и был выявлен ряд подгрупп мтДНК, характерных как только для русских, так и для славян в целом (Малярчук и др., 2017, 2019). Так, анализ показал, что 9.7% митохондриальных геномов, обнаруженных у русских, формируют подгруппы мтДНК, специфичные только для русских. Между тем, понятие такого рода популяционной специфичности является довольно условным, поскольку восточная часть Европы в отношении полиморфизма целых молекул мтДНК изучена намного хуже, чем западная. Поэтому нельзя исключить, что по мере увеличения объема данных о полномитогеномной изменчивости в популяциях Восточной Европы часть вариантов мтДНК, характерных на нынешнем этапе исследований для русских, перейдет в разряд специфичных для финно-угорских, тюркских или балтских популяций Восточной Европы. У русского населения выявлены также подгруппы мтДНК, характерные для славян в целом (частота 12.2%), поскольку гаплотипы, принадлежащие этим подгруппам, были обнаружены в различных славянских популяциях (Малярчук и др., 2019). В Дополнительных материалах (доступно только в электронной версии на сайте https://elibrary.ru) приводятся филогенетические деревья ряда подгрупп мтДНК, специфичных для славян. Таким образом, довольно большая фракция гаплотипов мтДНК у русских является частью общеславянского генетического пула. Это, в свою очередь, свидетельствует о реальности существования в прошлом славянской общности, в которую входили и предки русских.
Результаты анализа изменчивости целых митогеномов у поляков из различных районов Польши, N = 814 (Malyarchuk et al., 2017; Skonieczna et al., 2018; Piotrowska-Nowak et al., 2019a,b), показывают, что в польском генофонде 10.1% митохондриальных геномов относятся к подгруппам мтДНК, распространенным только среди поляков. У сербов, N = 226 (Davidović et al., 2020), частота этноспецифичного компонента составила 15%. Результаты молекулярного датирования показали, что возраст подгрупп мтДНК, специфичных для русских, составляет, в среднем, 2.7 ± 0.2 тыс. лет, для поляков – 2.9 ± 0.2 тыс. лет и для сербов – 2.0 ± ± 0.4 тыс. лет.
Проведенные ранее исследования изменчивости мтДНК у русских показали, что возраст обнаруженных в русском генофонде славянских подгрупп мтДНК оценивается примерно в 3.5 ± 0.3 тыс. лет, возраст славяно-германских подгрупп мтДНК составляет 4.2 ± 0.4 тыс. лет, а возраст славяно-финских подгрупп мтДНК – примерно 2.8 ± 0.2 тыс. лет (Малярчук и др., 2019). Таким образом, молекулярные датировки возраста различных популяционно-специфичных подгрупп мтДНК, выявленных в генофондах восточных, западных и южных славян, соответствуют эпохе бронзового и железного веков.
ПАЛЕОГЕНОМНЫЕ ДАННЫЕ О ПРОИСХОЖДЕНИИ ПРЕДКОВ СЛАВЯН
Палеогеномные исследования последних лет показали, что на формирование генетического облика современных европейцев большое влияние оказали миграции населения Понто-Каспийских степей эпохи бронзового века с востока на запад Европы (Allentoft et al., 2015; Haak et al., 2015). Результаты исследований изменчивости митохондриальных геномов у современного русского населения Восточной Европы также позволили выявить рост эффективной численности популяций в эпоху бронзового века, что, по всей видимости, могло быть связано с миграционными процессами именно бронзового века (Malyarchuk et al., 2017). Для исследования вопроса об истоках митохондриального генофонда русских целесообразно проведение анализа данных об изменчивости целых митохондриальных геномов у современного русского населения в сравнении с распределением гаплогрупп мтДНК у древнего населения Европы и Кавказа эпохи неолита и бронзового века. Такого рода анализ показал, что миграции древнего населения Понто-Каспийских степей, произошедшие ранее 4–3 тыс. лет до н. э. в западном направлении, могли привести к формированию в Центральной Европе смешанных популяций, характеризующихся восточноевропейскими (понто-каспийскими) компонентами аутосомного генофонда, но центрально-европейскими гаплотипами мтДНК (гаплогруппы H, J, T, K, W и др.); последние были унаследованы от анатолийских фермеров еще в энеолите (табл. 2).
Таблица 2.
Спектры гаплогрупп мтДНК у населения Восточной Европы в нескольких временных интервалах
| Гаплогруппы мтДНК (U2e, U4, U5) европейских охотников-собирателей | Гаплогруппы мтДНК (H, J, T, W и др.) анатолийских фермеров |
|---|---|
| 6–4 тыс. лет до н.э. (население современных территорий России, Украины и Латвии) |
|
| U2e, U4a, U4b, U4d, U5a1, U5a2, U5b2 | H1*, H2a1, T2 |
| 4–3 тыс. лет до н.э. (население современных территорий России, Украины и Латвии) |
|
| U2e, U5a1, U5a2 | H*, H1b, H2b, H5a, H6a1, H13a1a1, H15b, HV, R0a1, J2b1, T1a1, T2b, T2c, U8b1, W3a1, W6c |
| 3–2 тыс. лет до н.э. (население современных территорий России, Украины, Эстонии и Литвы) |
|
| U2e, U4*, U4a, U4c, U5a1, U5a2, U5b2 | H2a1, H5a1, H6a2, H13a1, J1c, J1c1, J1c2, J1c3, J2a, J2b1, T2a1, T2b, T2c, W3a1, I3a, N1a1a, K1b, U2d, X4 |
Дальнейшая экспансия этих популяций в обратном направлении – на восток Европы и далее в Азию – объясняет появление примерно 4–3 тыс. лет до н. э. у восточных европейцев гаплотипов, относящихся к анатолийскому набору гаплогрупп мтДНК (Малярчук, 2019). Собственно, с этого момента начинается формирование современной структуры митохондриальных генофондов русского населения.
Общим происхождением генетических компонентов в эпоху бронзового века можно объяснить и сходство митохондриальных генофондов населения западной и восточной частей Европы. Гомогенность европейского населения по распределению митохондриальных линий была обнаружена еще в ранних исследованиях полиморфизма нуклеотидных последовательностей ГВС1 мтДНК (Comas et al., 1996). Аналогичные исследования русских популяций также показали, что по распределению гаплотипов мтДНК русские мало отличаются от других европейских популяций (Malyarchuk, Derenko, 2001; Malyarchuk et al., 2004). Это, по всей видимости, объясняется тем, что сложившаяся в конце бронзового века генетическая структура европейского населения в последующем уже мало изменялась, и поэтому все более поздние миграционные процессы в Европе, включая миграции славян в раннем средневековье, приводили лишь к небольшому перераспределению одних и тех же гаплотипов мтДНК в различных популяциях.
Некоторое своеобразие сохраняют только те европейские популяции, которые сформировались в средние века под воздействием генетически отличающегося населения Сибири и Центральной Азии (гунны, авары, булгары, татары и др.), которое несло с собой на запад Европы (вплоть до Паннонии) восточноазиатские гаплотипы мтДНК. Например, польско-литовские татары – потомки выходцев из Золотой Орды, осевших в XIV в. на землях Великого княжества Литовского, до сих пор отличаются от соседних польских, литовских и белорусских популяций наличием высокой частоты восточноазиатских линий мтДНК (более 30%) (Pankratov et al., 2016). Между тем, для современного населения Венгрии характерны довольно низкие частоты восточноазиатских вариантов мтДНК (3–5%) (Egyed et al., 2007; Tömöry et al., 2007; Malyarchuk et al., 2018). Однако палеогенетические исследования показали, что население Венгрии эпохи раннего средневековья характеризовалось существенной долей восточноазиатского компонента митохондриального генофонда (15–23%) (Csősz et al., 2016; Neparáczki et al., 2017), который явно преобладал у аварской элиты (до 70%) (Csáky et al., 2020). Между тем, в последующее тысячелетие у венгров произошло снижение частоты восточноазиатских линий мтДНК вследствие доминирования внутриевропейских брачных связей (Tömöry et al., 2007).
Палеогеномных исследований, направленных на изучение вопроса о происхождении славян, к сожалению, пока мало. В одной из первых работ, посвященной проблеме генетической преемственности населения Польши на протяжении последних двух тысячелетий, было выявлено сходное распределение гаплогрупп мтДНК в популяциях римского железного века (2200–1500 лет назад) и средневековья (1000–600 лет назад), а также у современных поляков (Juras et al., 2014). Авторы этого исследования заключили, что западные славяне (поляки, чехи и словаки) сформировались на основе центрально- и восточноевропейского населения эпохи железного века. В другом исследовании населения Центральной Европы эпохи средневековья было установлено, что по распределению гаплогрупп мтДНК наиболее близкими оказываются выборки средневекового населения нынешней Польши и Словакии (IX–XII вв.); также они кластеризуются вместе с выборкой населения X в. из Венгрии и венгерскими лонгобардами (Csákyová et al., 2016). Последние представляют собой древнегерманское племя, обитавшее в VI–VIII вв. на территориях между рр. Эльба и Дунай. Между тем, было установлено, что по распределению гаплогрупп мтДНК средневековые словаки довольно далеки от современных словаков (Csákyová et al., 2016). В то же время, следует отметить, что оба исследования (Juras et al., 2014; Csákyová et al., 2016) основывались на результатах анализа полиморфизма ГВС1 и скрининга маркеров гаплогрупп мтДНК, и поэтому полученные результаты не могут рассматриваться в качестве надежного источника информации о генетическом сходстве между древними и современными популяциями. Тем не менее, это пока единственные данные о генетической структуре западных славян эпохи средневековья, представляющих, по всей видимости, так называемых венедов – северную ветвь славян раннего средневековья (VI–VIII вв.), проживавших к востоку от р. Висла – от Балтийского побережья до Карпат и низовьев р. Дунай.
Недавно получены первые сведения о генетической структуре антов, представляющих южную ветвь ранних славян (Järve et al., 2019). Согласно археологическим данным, предполагается, что анты формировались на основе черняховской культуры III–V вв. в процессе межэтнического взаимодействия славян (восточных венедов), германцев (остготов) и потомков скифского населения Северного Причерноморья (Седов, 1995). Результаты исследования (Järve et al., 2019) показали, что три изученных представителя черняховской культуры с территории современной Украины характеризуются высокой долей (18–45%) центрально-европейского генетического компонента. В пространстве главных компонент аутосомных вариантов полиморфизма черняховцы располагаются вместе с различными представителями современных народов Европы, однако авторы (Järve et al., 2019) делают вывод о генетическом сходстве между черняховцами и древнегерманскими готами. Между тем, согласно данным (Järve et al., 2019), один из черняховских образцов располагается вблизи от современных боснийцев и французов, другой – рядом со словаками и оркнейцами, а третий – рядом с поляками, русскими и белорусами. Таким образом, каждый из черняховских образцов проявляет некоторое сходство с представителями современных славян, а не германоязычных народов, в связи с чем предложенный вывод (Järve et al., 2019) о преимущественном генетическом родстве черняховцев и готов, по всей видимости, нельзя считать достаточно обоснованным.
Митохондриальные геномы трех исследованных (Järve et al., 2019) представителей черняховской культуры относятся к гаплогруппам H1c, H1n6 и T2g1. Известно, что довольно редкая гаплогруппа T2g1 обнаружена, в основном, на юге – у армян, палестинцев и таджиков (Pereira et al., 2017). Гаплогруппа H1c, наоборот, главным образом распространена в Европе, в том числе у русских, поляков, сербов, чехов и словаков (Malyarchuk et al., 2017). Аналоги черняховскому H1n6-варианту мтДНК обнаружены у современного населения Дании и Испании (Behar et al., 2012; Raule et al., 2014) (рис. 5). Поэтому, по всей видимости, нельзя однозначно решить вопрос о происхождении этого черняховского гаплотипа мтДНК и, тем более, ассоциировать его только с готами. Таким образом, на нынешнем этапе исследований очевидно, что представители черняховской культуры были очень разнообразны в генетическом отношении, а для выяснения происхождения черняховцев необходимо существенно увеличить размеры выборок.
Рис. 5.
Филогенетическое дерево гаплогруппы H1n6, построенное с помощью программы mtPhyl 4.015. На ветвях указаны позиции мтДНК, в которых произошли транзиции; для трансверсии показан результат нуклеотидной замены. Эволюционный возраст подгруппы мтДНК (в тыс. лет) приводится в соответствии со скоростью мутаций в целом митогеноме (Soares et al., 2009). Для митохондриальных геномов приводятся номера в GenBank и страна происхождения. Митогеном черняховского индивидуума MJ-19 реконструирован из геномных данных (Järve et al., 2019).

ЗАКЛЮЧЕНИЕ
Обзор данных об изменчивости нуклеотидных последовательностей целых митохондриальных геномов в популяциях славян (русских, поляков и сербов) свидетельствует о том, насколько более детальными стали представления о структуре и разнообразии митохондриальных генофондов, которые ранее базировались на результатах анализа лишь небольшого участка главной некодирующей области мтДНК или распределения частот митохондриальных гаплогрупп. Намного улучшились также и результаты демографического анализа, основанного на данных об изменчивости мтДНК – особенно для байесовского анализа динамики эффективной численности популяций во времени, позволившего в случае анализа больших наборов генетических данных выявлять межрегиональные различия динамики Ne у европейцев. Применение филогеографического анализа современных и палеогеномных данных об изменчивости мтДНК дало возможность максимально точно (благодаря использованию полноразмерных митохондриальных геномов) выявлять общие компоненты генофондов различных популяций, а также уникальные филогенетические кластеры мтДНК, специфичные для отдельных этнических групп и их общностей. На примере трех этнических групп славян продемонстрирована общность их генетического происхождения, истоки которой уходят в эпохи бронзового и железного веков. Вместе с тем, существующий дефицит данных об изменчивости целых молекул мтДНК у населения Европы, особенно ее восточной части, препятствует на данном этапе формированию более целостной картины межэтнических генетических отношений и реконструкции генетической истории славян в эпоху раннего средневековья. Таким образом, дальнейшие исследования полиморфизма целых митохондриальных геномов у современного и древнего населения Восточной Европы представляются очень важными и перспективными в историческом отношении.
Список литературы
Алексеева Т.И. Этногенез восточных славян по данным антропологии. М.: МГУ, 1973. 332 с.
Алексеева Т.И., Алексеев В.П. Антропология о происхождении славян // Природа. 1989. № 1. С. 60–62.
Лер-Сплавинский Т. К современному состоянию проблемы происхождения славян // Вопр. языкозн. 1960. № 4. С. 20–30.
Малярчук Б.А. Дифференциация славян и их генетическое положение среди народов Евразии по данным об изменчивости митохондриальной ДНК // Генетика. 2001. Т. 37. С. 1705–1712.
Малярчук Б.А. Истоки митохондриального генофонда русских по результатам анализа палеогеномных данных для населения Европы и Кавказа эпохи неолита и бронзового века // Вавилов. журн. генет. селекц. 2019. Т. 23. С. 588–593.
Малярчук Б.А., Деренко М.В., Литвинов А.Н. Структура макрогаплогруппы U у русских // Генетика. 2017. Т. 53. С. 488–494.
Малярчук Б.А., Литвинов А.Н., Деренко М.В. Структура и формирование митохондриального генофонда русского населения Восточной Европы // Генетика. 2019. Т. 55. С. 574–582.
Седов В.В. Происхождение и ранняя история славян. М.: Наука, 1979. 158 с.
Седов В.В. Славяне в раннем средневековье. М.: Наука, 1995. 416 с.
Седов В.В. Этногенез ранних славян // Вестн. РАН. 2003. Т. 73. С. 594–605.
Allentoft M.E., Sikora M., Sjogren K.G. et al. Population genomics of Bronze Age Eurasia // Nature. 2015. V. 522. P. 167–172.
Aris-Brosou S., Excoffier L. The impact of population expansion and mutation rate heterogeneity on DNA sequence polymorphism // Mol. Biol. Evol. 1996. V. 13. P. 494–504.
Balanovsky O., Rootsi S., Pshenichnov A. et al. Two sources of the Russian patrilineal heritage in their Eurasian context // Am. J. Hum. Genet. 2008. V. 82. P. 236–250.
Batini C., Hallast P., Vågene Å.J. et al. Population resequencing of European mitochondrial genomes highlights sex-bias in Bronze Age demographic expansions // Sci. Rep. 2017. V. 7. P. e12086.
Behar D.M., van Oven M., Rosset S. et al. A “Copernican” reassessment of the human mitochondrial DNA tree from its root // Am. J. Hum. Genet. 2012. V. 90. P. 675–684.
Busby G.B.J., Hellenthal G., Montinaro F. et al. The role of recent admixture in forming the contemporary West Eurasian genomic landscape // Curr. Biol. 2015. V. 25. P. 2878.
Comas D., Calafell F., Mateu E. et al. Geographic variation in human mitochondrial DNA control region sequence: the population history of Turkey and its relationship to the European populations // Mol. Biol. Evol. 1996. V. 13. P. 1067–1077.
Costa M.D., Pereira J.B., Pala M. et al. A substantial prehistoric European ancestry amongst Ashkenazi maternal lineages // Nat. Commun. 2013. V. 4. P. e2543.
Csáky V., Gerber D., Koncz I. et al. Genetic insights into the social organisation of the Avar period elite in the 7th century AD Carpathian Basin // Sci. Rep. 2020. V. 10. P. e948.
Csákyová V., Szécsényi-Nagy A., Csősz A. et al. Maternal genetic composition of a medieval population from a Hungarian-Slavic contact zone in Central Europe // PLoS One. 2016. V. 11. P. e0151206.
Csősz A., Szécsényi-Nagy A., Csákyová V. et al. Maternal genetic ancestry and legacy of 10th century AD Hungarians // Sci. Rep. 2016. V. 6. P. e33446.
Davidović S., Malyarchuk B., Aleksić J. et al. Mitochondrial DNA perspective of Serbian genetic diversity // Am. J. Phys. Anthropol. 2015. V. 156. P. 449–465.
Davidovic S., Malyarchuk B., Aleksić J. et al. Mitochondrial super-haplogroup U diversity in Serbians // Ann. Hum. Biol. 2017. V. 44. P. 408–418.
Davidović S., Malyarchuk B., Grzybowski T. et al. Complete mitogenome data for the Serbian population: the contribution to high quality forensic databases // Int. J. Leg. Med. 2020. https://doi.org/10.1007/s00414-020-02324-x
Denaro M., Blanc H., Johnson M.J. et al. Ethnic variation in Hpa I endonuclease cleavage patterns of human mitochondrial DNA // PNAS USA. 1981. V. 78. P. 5768–5772.
Derenko M., Denisova G., Malyarchuk B. et al. Mitogenomic diversity and differentiation of the Buryats // J. Hum. Genet. 2018. V. 63. P. 71–81.
Derenko M., Denisova G., Malyarchuk B. et al. Insights into matrilineal genetic structure, differentiation and ancestry of Armenians based on complete mitogenome data // Mol. Genet. Genomics. 2019. V. 294. P. 1547–1559.
Derenko M., Malyarchuk B., Bahmanimehr A. et al. Complete mitochondrial DNA diversity in Iranians // PLoS One. 2013. V. 8. P. e80673.
Drummond A.J., Nicholls G.K., Rodrigo A.G., Solomon W. Estimating mutation parameters, population history and genealogy simultaneously from temporally spaced sequence data // Genetics. 2002. V. 161. P. 1307–1320.
Drummond A.J., Rambaut A., Shapiro B., Pybus O.G. Bayesian coalescent inference of past population dynamics from molecular sequences // Mol. Biol. Evol. 2005. V. 22. P. 1185–1192.
Egyed B., Brandstätter A., Irwin J.A. et al. Mitochondrial control region sequence variations in the Hungarian population: analysis of population samples from Hungary and from Transylvania (Romania) // Forensic Sci. Int. Genet. 2007. V. 1. P. 158–162.
Fraumene C., Belle E.M., Castri L. et al. High resolution analysis and phylogenetic network construction using complete mtDNA sequences in Sardinian genetic isolates // Mol. Biol. Evol. 2006. V. 23. P. 2101–2111.
Grzybowski T., Malyarchuk B.A., Derenko M.V. et al. Complex interactions of the Eastern and Western Slavic populations with other European groups as revealed by mitochondrial DNA analysis // Forensic Sci. Int. Genet. 2007. V. 1. P. 141–147.
Haak W., Lazarids I., Patterson N. et al. Massive migration from the steppe was a source for Indo-European languages in Europe // Nature. 2015. V. 522. P. 207–211.
Hellenthal G., Busby G.B.J., Band G. et al. A genetic atlas of human admixture history // Science. 2014. V. 343. P. 747–751.
Hofmanová Z., Kreutzer S., Hellenthal G. et al. Early farmers from across Europe directly descended from Neolithic Aegeans // PNAS USA. 2016. V. 113. P. 6886–6891.
Gimbutas M. The prehistory of Eastern Europe. Part I: Mesolithic, Neolithic and Copper Age cultures in Russia and the Baltic area. Cambridge: The Peabody Museum, 1956. 241 p.
Järve M., Saag L., Scheib C.L. et al. Shifts in the genetic landscape of the Western Eurasian steppe associated with the beginning and end of the Scythian dominance // Curr. Biol. 2019. V. 29. P. 2430–2441.
Johnson M.J., Wallace D.C., Ferris S.D. et al. Radiation of human mitochondria DNA types analyzed by restriction endonuclease cleavage patterns // J. Mol. Evol. 1983. V. 19. P. 255–271.
Juras A., Dabert M., Kushniarevich A. et al. Ancient DNA reveals matrilineal continuity in present-day Poland over the last two millennia // PLoS One. 2014. V. 9. P. e110839.
Just R.S., Scheible M.K., Fast S.A. et al. Full mtGenome re-ference data: development and characterization of 588 forensic-quality haplotypes representing three U.S. populations // Forensic Sci. Int. Genet. 2015. V. 14. P. 141–155.
Karachanak S., Carossa V., Nesheva D. et al. Bulgarians vs the other European populations: a mitochondrial DNA perspective // Int. J. Leg. Med. 2012. V. 126. P. 497–503.
Karmin M., Saag L., Vicente M. et al. A recent bottleneck of Y chromosome diversity coincides with a global change in culture // Genome Res. 2015. V. 25. P. 459–466.
Khrunin A.V., Khokhrin D.V., Filippova I.N. et al. A genome-wide analysis of populations from European Russia reveals a new pole of genetic diversity in northern Europe // PLoS One. 2013. V. 8. P. e58552.
Kushniarevich A., Utevska O., Chuhryaeva M. et al. Genetic heritage of the Balto-Slavic speaking populations: a synthesis of autosomal, mitochondrial and Y-chromosomal data // PLoS One. 2015. V. 10. P. e0135820.
Li S., Besenbacher S., Li Y. et al. Variation and association to diabetes in 2000 full mtDNA sequences mined from an exome study in a Danish population // Eur. J. Hum. Genet. 2014. V. 22. P. 1040–1045.
Librado P., Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. P. 1451–1452.
Lippold S., Xu H., Ko A. et al. Human paternal and maternal demographic histories: insights from high-resolution Y chromosome and mtDNA sequences // Investig. Genet. 2014. V. 5. P. e13.
Lipson M., Szécsényi-Nagy A., Mallick S. et al. Parallel palaeogenomic transects reveal complex genetic history of early European farmers // Nature. 2017. V. 551. P. 368–372.
Malyarchuk B.A., Derenko M.V. Mitochondrial DNA variability in Russians and Ukrainians: implication to the origin of the Eastern Slavs // Ann. Hum. Genet. 2001. V. 65. P. 63–78.
Malyarchuk B., Derenko M., Denisova G., Kravtsova O. Mitogenomic diversity in Tatars from the Volga-Ural region of Russia // Mol. Biol. Evol. 2010. V. 27. P. 2220–2226.
Malyarchuk B., Derenko M., Denisova G. et al. Whole mitochondrial genome diversity in two Hungarian populations // Mol. Gen. Genomics. 2018. V. 293. P. 1255–1263.
Malyarchuk B., Derenko M., Grzybowski T. et al. Differentiation of mitochondrial DNA and Y chromosomes in Russian populations // Hum. Biol. 2004. V. 76. P. 877–900.
Malyarchuk B.A., Grzybowski T., Derenko M.V. et al. Mitochondrial DNA variability in Bosnians and Slovenians // Ann. Hum. Genet. 2003. V. 67. P. 412–425.
Malyarchuk B.A., Grzybowski T., Derenko M. et al. Mitochondrial DNA phylogeny in Eastern and Western Slavs // Mol. Biol. Evol. 2008. V. 25. P. 1651–1658.
Malyarchuk B., Litvinov A., Derenko M. et al. Mitogenomic diversity in Russians and Poles // Forensic Sci. Int. Genet. 2017. V. 30. P. 51–56.
Margaryan A., Lawson D., Sikora M. et al. Population genomics of the Viking world // bioRxiv. 2019. https://doi.org/10.1101/703405
Mielnik-Sikorska M., Daca P., Malyarchuk B. et al. The history of Slavs inferred from complete mitochondrial genome sequences // PLoS One. 2013. V. 8. P. e54360.
Morozova I., Evsyukov A., Kon’kov A. et al. Russian ethnic history inferred from mitochondrial DNA diversity // Am. J. Phys. Anthropol. 2012. V. 147. P. 341–351.
Müller J., Rassmann K., Videiko M. Trypillia mega-sites and European prehistory: 4100–3400 BCE. London: Routledge, 2016. 330 p.
Neparáczki E., Juhász Z., Pamjav H. et al. Genetic structure of the early Hungarian conquerors inferred from mtDNA haplotypes and Y-chromosome haplogroups in a small cemetery // Mol. Genet. Genomics. 2017. V. 292. P. 201–214.
Olivieri A., Sidore C., Achilli A. et al. Mitogenome diversity in Sardinians: a genetic window onto an Island’s past // Mol. Biol. Evol. 2017. V. 34. P. 1230–1239.
Översti S., Onkamo P., Stoljarova M. et al. Identification and analysis of mtDNA genomes attributed to Finns reveal long-stagnant demographic trends obscured in the total diversity // Sci. Rep. 2017. V. 7. P. e6193.
Pankratov V., Litvinov S., Kassian A. et al. East Eurasian ancestry in the middle of Europe: genetic footprints of steppe nomads in the genomes of Belarusian Lipka Tatars // Sci. Rep. 2016. V. 6. P. e30197.
Piotrowska-Nowak A., Elson J.L., Sobczyk-Kopciol A. et al. New mtDNA association model, MutPred variant load, suggests individuals with multiple mildly deleterious mtDNA variants are more likely to suffer from atherosclerosis // Front. Genet. 2019a. V. 9. P. 702.
Piotrowska-Nowak A., Kosior-Jarecka E., Schab A. et al. Investigation of whole mitochondrial genome variation in normal tension glaucoma // Exp. Eye Res. 2019b. V. 178. P. 186–197.
Pereira J.B., Costa M.D., Vieira D. et al. Reconciling evidence from ancient and contemporary genomes: a major source for the European Neolithic within Mediterranean Europe // Proc. Biol. Sci. 2017. V. 284. P. e20161976.
Ralph P., Coop G. The geography of recent genetic ancestry across Europe // PLoS Biol. 2013. V. 11. P. e1001555.
Rascovan N., Sjogren K.G., Kristiansen K. et al. Emergence and spread of basal lineages of Yersinia pestis during the Neolithic decline // Cell. 2019. V. 176. P. 295–305.
Raule N., Sevini F., Li S. et al. The co-occurrence of mtDNA mutations on different oxidative phosphorylation subunits, not detected by haplogroup analysis, affects human longevity and is population specific // Ag. Cell. 2014. V. 13. P. 401–407.
Ray N., Currat M., Excoffier L. Intra-deme molecular diversity in spatially expanding populations // Mol. Biol. Evol. 2003. V. 20. P. 76–86.
Rebała K., Mikulich A.I., Tsybovsky I.S. et al. Y-STR variation among Slavs: evidence for the Slavic homeland in the middle Dnieper basin // J. Hum. Genet. 2007. V. 52. P. 406–414.
Rogers A.R., Harpending H. Population growth makes waves in the distribution of pairwise genetic differences // Mol. Biol. Evol. 1992. V. 9. P. 552–569.
Sarac J., Saric T., Augustin D.H. et al. Maternal genetic heritage of Southeastern Europe reveals a new Croatian isolate and a novel, local sub-branching in the X2 haplogroup // Ann. Hum. Genet. 2014. V. 78. P. 178–194.
Shennan S., Downey S.S., Timpson A. et al. Regional population collapse followed initial agriculture booms in mid-Holocene Europe // Nat. Commun. 2013. V. 4. P. e2486.
Skonieczna K., Malyarchuk B., Jawień A. et al. Mitogenomic differences between the normal and tumor cells of colorectal cancer patients // Hum. Mut. 2018. V. 39. P. 691–701.
Soares P., Ermini L., Thomson N. et al. Correcting for purifying selection: an improved human mitochondrial molecular clock // Am. J. Hum. Genet. 2009. V. 84. P. 740–759.
Stoljarova M., King J.L., Takahashi M. et al. Whole mitochondrial genome genetic diversity in an Estonian population sample // Int. J. Leg. Med. 2016. V. 130. P. 67–71.
Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism // Genetics. 1989. V. 123. P. 585–595.
Tajima F. Statistical analysis of DNA polymorphism // Jpn. J. Genet. 1993. V. 68. P. 567–595.
Tömöry G., Csányi B., Bogácsi-Szabó E. et al. Comparison of maternal lineage and biogeographic analyses of ancient and modern Hungarian populations // Am. J. Phys. Anthropol. 2007. V. 134. P. 354–368.
Woźniak M., Malyarchuk B., Derenko M. et al. Similarities and distinctions in Y chromosome gene pool of Western Slavs // Am. J. Phys. Anthropol. 2010. V. 142. P. 540–548.
Zheng H.-X., Yan S., Qin Z.-D., Jin L. mtDNA analysis of global populations support that major population expansions began before Neolithic Time // Sci. Rep. 2012. V. 2. P. e745.
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии


