Успехи современной биологии, 2021, T. 141, № 3, стр. 211-226
Белок ядрышка SURF6 – известные свойства и возможные функции
А. А. Моралева 1, *, М. А. Ползиков 1, М. Ю. Кордюкова 1, О. В. Зацепина 1
1 Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Москва, Россия
* E-mail: a_moraleva@mail.ru
Поступила в редакцию 22.12.2020
После доработки 23.12.2020
Принята к публикации 23.12.2020
Аннотация
Ядрышко – основной субъядерный компартмент, где происходит биогенез рибосом. Также ядрышко является центром стрессовой реакции, вовлеченным в регуляцию множества процессов, в частности, прогрессирования клеточного цикла, старения и апоптоза. Новые данные связывают ядрышко также с контролем стабильности генома и развитием злокачественных новообразований человека. В пролиферирующих клетках млекопитающих основную массу ядрышка составляют белки, большинство из которых представлено факторами, участвующими в транскрипции рибосомных генов, в процессинге новообразованных транскриптов пре-рРНК и в сборке рибосомных частиц. Однако около трети белков, обнаруженных в составе выделенных ядрышек, изучены плохо, а их роль в клеточном метаболизме до сих пор остается невыясненной. К таким белкам относится, в частности, эволюционно консервативный белок ядрышка SURF6, описываемый в данной работе.
ВВЕДЕНИЕ
Ядрышко представляет собой основной домен клеточного ядра, не отделенный мембраной от нуклеоплазмы и сформированный около участков хромосом, кодирующих повторы рибосомных генов (рДНК). Канонической функцией ядрышка является биосинтез рибосом. В настоящее время к функциям ядрышка относят участие в регуляции клеточного цикла, апоптоза, в развитии вирусных инфекций и процессов клеточного старения (Núñez Villacís et al., 2018). Новые данные связывают ядрышко также с контролем стабильности генома и развитием злокачественных новообразований человека (Lindström et al., 2018). Таким образом, ядрышко – многофункциональный и значимый клеточный компартмент, неканонические функции которого в настоящее время активно изучаются. Согласно данным масс-спектрометрического анализа, в ядрышке клеток человека содержится около 6000 белков, большинство из которых представлено факторами, участвующими в транскрипции рибосомных генов, в процессинге новообразованных транскриптов пре-рРНК и в сборке рибосомных частиц. Около трети белков, выявляемых в составе ядрышек, изучены плохо, а их роль в клеточном метаболизме до сих пор остается невыясненной (Tafforeau et al., 2013). К таким белкам относится, в частности, белок ядрышка SURF6. SURF6 впервые был описан в 1996 г. как продукт экспрессии гена Surf-6, являющегося одним из членов Surfeit-локуса в геноме мыши (Magoulas, Fried, 1996). Имеющиеся на настоящий момент данные позволяют предположить, что SURF6 участвует как в биогенезе рибосом, так и в регуляции клеточного цикла. Однако существующая на сегодня информация не позволяет сделать более определенных выводов о роли SURF6 в метаболизме клеток высших эукариот, и этот белок до сих пор относится к наименее изученным белкам ядрышка у млекопитающих.
Surfeit-ЛОКУС
Surfeit-локус – консервативный локус генов – широко распространен у позвоночных, он выявлен в геномах человека, мыши, курицы (Duhig et al., 1998), шпорцевой лягушки (Wolff et al., 2002), рыбы Фугу (Armes et al., 1997; Wolff et al., 2002) (рис. 1). Это дает основания предполагать, что организация локуса Surfeit играет существенную роль в генной экспрессии позвоночных. У рыбы Фугу Fugu rubripes гены локуса Surfeit расположены в трех различных участках генома, но их структура гомологична структуре генов Surfeit-локуса у млекопитающих (Armes et al., 1997). Беспозвоночные виды, например, фруктовая мушка D. melanogaster и нематода C. elegans, не имеют Surfeit-локуса, характерного для позвоночных, но обладают генами, гомологичными генам Surfeit-локуса, которые располагаются на разных хромосомах, а продукты экспрессии этих генов также имеют гомологов у позвоночных видов (Armes et al., 1997).
Рис. 1.
Surfeit-локусы мыши M. musculus и человека H. sapiens. Геномная ДНК обозначена толстой линией, CpG-обогащенные участки представлены в виде черных прямоугольников. Направление транскрипции генов Surfeit-локуса и псевдогенов ψ указано стрелками, также указано расстояние между генами, где п.о. – пара нуклеотидных оснований, т.п.о. – тысяча пар нуклеотидных оснований (Magoulas, Fried, 2000).

Surfeit-локус млекопитающих представляет собой плотный кластер шести не связанных друг с другом генов “домашнего хозяйства” (Surf-1, Surf-2, … Surf-6) (Huxley, Fried, 1990). Как было показано, нуклеотидные последовательности шести генов Surfeit-локуса и аминокислотные последовательности продуктов их экспрессии не имеют гомологии между собой и с другими генами и белками. Surfeit-локус обладает уникальными свойствами: перекрывающиеся генные последовательности, двунаправленные промоторы транскрипции и очень плотное расположение генов (Armes et al., 1997).
Направление транскрипции для пяти из шести генов Surfeit-локуса (кроме гена Surf-6) противоположно направлению транскрипции соседнего гена, причем, 5'-концы каждого из шести генов ассоциированы с участками геномной ДНК, обогащенными цитозином и гуанином (CpG-обогащенные участки) и не содержат консенсусной ТАТА-последовательности (рис. 1), что характерно для генов “домашнего хозяйства” (Armes et al., 1997). В частности, метилирование таких участков приводит к подавлению активности промотора, расположенного в этой области (Antequera et al., 1989).
По сравнению с расположением других генов, гены Surfeit-локуса имеют достаточно малые межгенные расстояния: их разделяют не более 73 пар оснований, тогда как большинство близкорасположенных генов млекопитающих разделяют десятки и сотни тысяч пар оснований. Необычно плотное расположение генов, характерное для генов локуса Surfeit, является консервативным для млекопитающих и птиц. Этот факт свидетельствует в пользу того, что подобная организация может иметь биологическое значение и играть важную функциональную или регуляторную роль. Такое плотное расположение генов в локусе может также свидетельствовать о цис-взаимодействии генов или взаимной регуляции экспрессии генов данного локуса у позвоночных (Armes et al., 1997).
Необходимо заметить, что промоторная область генов Surf-1 и Surf-2 человека содержит четыре сайта связывания транскрипционных факторов. Два из них идентифицированы и представляют собой белки Sp1 и YY1 (Cole, Gaston, 1997). Известно, что транскрипционные факторы Sp1 и YY1 взаимодействуют между собой и с транс-крипционным фактором с-Myc (Seto et al., 1993). Транскрипционный фактор с-Myc – ядерный белок, регулирующий экспрессию генов, которые участвуют в процессах клеточной пролиферации, дифференцировки и апоптоза (Marcu et al., 1992; Stine et al., 2015). Показано, что активация промотора Surf-1 достигается путем связывания транс-крипционных факторов YY1 и с-Myc в ответ на добавление ростовых факторов. Кроме того, обнаружено, что экспрессия гена Surf-6 у D. melanogaster также активируется транскрипционным фактором с-Myc (Orian et al., 2003). С использованием фибробластов крысы, постоянно экспрессирующих c-Myc, были идентифицированы 38 генов, кодирующих белки с ядрышковой локализацией, экспрессия которых находилась в зависимости от уровня эндогенного фактора c-Myc (Schlosser et al., 2003). К числу таких генов относится ген Surf-6 человека, а также гены других ядрышковых белков: нуклеолина, В23/нуклеофозмина, фибрилларина и Nopp140. Все эти белки играют ключевые роли в биогенезе рибосом. Можно предположить, что, несмотря на отсутствие Surfeit-локуса у беспозвоночных и, в частности, у D. melanogaster, Surf-гены могут обладать схожими механизмами Myc-зависимой активации.
К настоящему моменту продукты экспрессии каждого из шести Surf-генов идентифицированы. Белки, кодирующиеся Surf-генами, обладают различными функциями и имеют межвидовые гомологи, принадлежащие к SURF1, SURF2, … SURF6 белковым семействам, представленным в базе данных белковых семейств PFam (http://www.sanger.ac.uk/Software/Pfam/).
Так, белки, принадлежащие семейству SURF1, являются трансмембранными митохондриальными белками, предположительно участвующими в биогенезе цитохрома с. Белок, кодируемый Surf-1, – компонент промежуточного звена митохондриальной регуляции трансляции комплекса MITRAC (mitochondrial translation regulation assembly intermediate of cytochrome c oxidase), который участвует в регуляции сборки цитохром с-оксидазы. Дефекты Surf-1 являются причиной синдрома Ли – тяжелого неврологического расстройства, которое обычно связано с системным дефицитом цитохром с-оксидазы (комплекс IV), и болезни Шарко–Мари–Тута – моторно-сенсорной полиневропатии (Smith et al., 2005).
SURF2 – консервативный белок, локализующийся в ядре и ядрышке, функция которого неизвестна.
Ген Surf-3 кодирует рибосомный белок L7a.
Surf-4 кодирует консервативный интегральный мембранный белок, который содержит несколько предполагаемых трансмембранных областей и связан с эндоплазматическим ретикулумом. Специфическая функция этого белка не определена, но его дрожжевой гомолог непосредственно необходим для упаковки гликозилированного про-альфа-фактора в везикулы COPII. Этот ген использует несколько сайтов полиаденилирования, что приводит к изменению длины транскрипта (Yin et al., 2018).
Surf-5 кодирует белковый компонент MED22 медиаторного комплекса, который функционирует в регуляции транскрипции за счет мостиковых взаимодействий между ген-специфическими регуляторными факторами, РНК-полимеразой II и общими факторами транскрипции. Наблюдались альтернативно сплайсированные варианты транскриптов, кодирующие разные изоформы (Sato et al., 2003).
И, наконец, ген Surf-6 кодирует белок SURF6, являющийся белком ядрышкового матрикса (Magoulas et al., 1998). Surf-6 экспрессируется во всех типах тканей (Trott et al., 2001), но его функция до сих пор точно неизвестна.
Таким образом, Surfeit-локус уникален как по строению и взаимному расположению генов, так и по разнообразию функций белков, кодируемых Surf-генами.
Необходимо заметить, что в локусе Surfeit мыши был обнаружен псевдоген, расположенный на расстоянии в 68 п.о. от 3'-конца гена Surf-5. Псевдогенная последовательность также содержится в локусе человека и соответствует рибосомному белку rpL21 (Trott et al., 2001). Наличие псевдогенов, рассеянных по геному, типично для рибосомных белков, в частности rpL21 обладает 145 псевдогенами (Коваленко, Патрушев, 2018).
Также стоит отметить, что в геноме человека содержится один псеводген Surf-6, выявлены 53 псевдогена для Surf-3 (рибосомный белок L7a), псевдогенов для других членов Surfeit-локуса у человека не выявлено (http://pseudofam.pseudogene.org/).
Анализ последовательности кДНК Surf-6 человека показал, что открытая рамка считывания начинается с первого ATG, который окружен последовательностью, подобной последовательности, выведенной Козак, и служащей для эффективной инициации трансляции у млекопитающих. Выше сайта начала транскрипции гена Surf-6 находится несколько сайтов связывания транскрипционных факторов, однако, как и в случае с другими генами локуса Surfeit, отсутствует консенсусная TATA-последовательность (Magoulas, Fried, 2000).
СВОЙСТВА БЕЛКА SURF6
К признакам семейства белков SURF6 относят: сравнительно небольшие размеры (у мыши и человека 355 и 361 а.о. соответственно); обогащенность остатками лизина и аргинина (28%), которые придают белкам большой положительный заряд (изоэлектрическая точка pI около 10.5); отсутствие каких-либо консенсусных функциональных последовательностей; преимущественно ядрышковая локализация. У человека экспрессия гена Surf-6 описана в тканях всех органов, использованных для анализа: поджелудочной железе, почке, мышцах, печени, легком, плаценте, мозге и сердце (Magoulas, Fried, 2000). Было показано, что SURF6 человека содержит 10 потенциальных сайтов фосфорилирования (результаты анализа аминокислотной последовательности SURF6 с использованием базы данных Prosite (http://prosite.expasy.org)).
С помощью Вестерн-блотов было определено, что белок SURF6 имеет электрофоретическую подвижность в районе 46 кДа у человека и 43 кДа у мыши. SURF6 является высокогидрофильным белком (Magoulas, Fried, 2000). Аминокислотная последовательность белка SURF6 обладает пятью потенциальными сайтами фосфорилирования казеин киназой II (Magoulas, Fried, 2000). По-видимому, эти сайты являются активными, поскольку методом дрожжевой двухгибридной системы было показано связывание SURF6 c казеин киназой II у D. melanogaster. Считается, что фосфорилирование казеин киназой II может влиять на активность функций ядрышка в течение клеточного цикла и контролировать активность специфических ядрышковых белков (Trott et al., 2001).
Нуклеотидная последовательность гена Surf-6 уникальна, поскольку при анализе баз данных нуклеотидных последовательностей не было обнаружено генов, гомологичных гену Surf-6. Но, как было показано, белки различных таксономических групп (от человека до дрожжей) обладают высококонсервативным доменом, который позволяет объединить их в одно семейство – семейство SURF6-белков (Polzikov et al., 2005) (рис. 2).
Рис. 2.
Выровненные аминокислотные последовательности С-концевого консервативного участка молекулы SURF6 (SURF6-домен) у представителей различных таксономических групп. Средняя длина SURF6-домена – 191 аминокислотный остаток (а.о.); процент идентичности а.о. в пределах домена у разных видов – 36%. Указаны продолжительность SURF6-домена и количество а.о. в молекуле SURF6 для каждого вида. Выравнивание последовательностей осуществлено методом ClustalW. Затемнение выполнено для консервативных аминокислотных остатков (черный цвет – 100% идентичность а.о. во всех видах; серый цвет – меньше 100%, но более 70% идентичности а.о.; светло-серый цвет – менее 70%, но более 50% идентичности а.о.). Вторичная структура обозначена на рисунке следующим образом: прямоугольник – α-спиральные участки, стрелка – β-складчатый слой, прямая линия – неупорядоченные области (Polzikov et al., 2005).
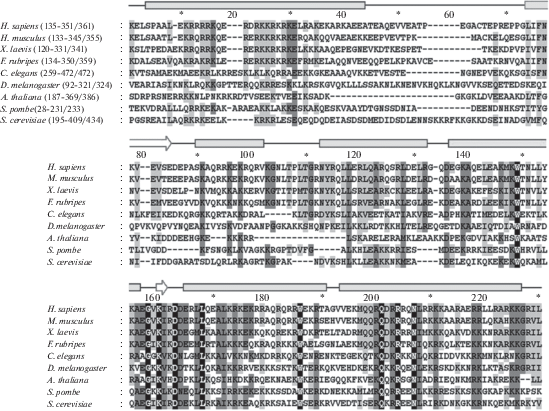
SURF6-домен находится в С-концевой области и имеет среднюю длину 191 а.о. с 36%-ной межвидовой идентичностью аминокислотных остатков. N-концевая область белков семейства SURF6 имеет разную длину и не проявляет значительной гомологии между белками семейства у разных видов. Внутри домена находится высококонсервативное ядро, состоящее примерно из 60 аминокислот, 9 из которых сохраняются у всех видов и располагаются между остатками триптофана и аспарагина. Консервативный домен SURF6 имеет, в основном, α-спиральную структуру. Высказано предположение, что аминокислотные остатки, составляющие ядро домена, могут играть важную роль как в процессе фолдинга белка, так и в процессах молекулярного взаимодействия SURF6 c белковыми партнерами и нуклеиновыми кислотами in vivo (Polzikov et al., 2005).
С помощью аффинной хроматографии и нитроцеллюлозных фильтров с сорбированными на них ДНК или РНК была показана способность SURF6 мыши связываться с нуклеиновыми кислотами in vitro, причем связь с РНК была сильнее, чем с ДНК (Magoulas et al., 1998). Также было показано, что воздействие РНКазы А на клетки полностью препятствует иммуноцитохимическому окрашиванию ядрышек антителами к SURF6. Поскольку основным типом РНК ядрышек является рРНК на разных стадиях созревания, это наблюдение является свидетельством в пользу возможного взаимодействия SURF6 с рРНК in situ. При этом обработка клеток ДНКазой I не влияла на локализацию SURF6 (Гурченков и др., 2005), что может говорить об отсутствии связи SURF6 с рДНК in situ. Об этом также косвенно говорит поведение SURF6 в митозе: все специфические белки ядрышка, которые ассоциированы с рДНК, включая РНК-полимеразу I и ее факторы транс-крипции, – белки UBF, TBP, TAF во время митоза сохраняют связь с районами ядрышковых организаторов. В отличие от этих белков, SURF6 располагался на поверхности хромосом, в цитоплазме и в составе предъядрышек, подобно белкам, участвующим в созревании рРНК, – фибрилларину и В23 (Гурченков и др., 2005).
ВНУТРИКЛЕТОЧНАЯ ЛОКАЛИЗАЦИЯ БЕЛКА SURF6
Методом иммуноцитохимической электронной микроскопии обнаружено, что в клетках мыши белок SURF6 локализуется преимущественно в гранулярном компоненте (ГК) (Magoulas et al., 1998) (рис. 3), где происходит процессинг рРНК, и входит в состав ядрышкового матрикса. SURF6 присутствует в ядрышках в течение клеточного цикла, и его локализация остается практически одинаковой во всех фазах цикла. При этом области локализации SURF6 значительно, но не полностью перекрываются с областями локализации В23 и фибрилларина, причем степень колокализации SURF6 и B23 оказывается выше степени колокализации SURF6 и фибриларина (Magoulas et al., 1998).
Рис. 3.
Иммуноцитохимическое выявление белка SURF6 в клетках мыши линии NIH/3T3 с помощью специфических антител (Polzikov et al., 2012).

Распределение SURF6 в течение митоза подобно распределению белков, участвующих в процессинге рРНК. При вступлении клеток в митоз и начале распада ядрышка в профазе белок мигрирует из ядрышек в ядро, где располагается в межхромосомных районах. В прометафазе и метафазе SURF6 локализуется на поверхности хромосом и в цитоплазме, частично оставаясь ассоциированным с остаточными ядрышками. В анафазе SURF6 располагается в основном в цитоплазме и слабо декорирует хромосомы. В ранней телофазе белок находится на перихромосомной области хромосом. В телофазе и поздней телофазе белок отчетливо обнаруживается в предъядрышковых тельцах и практически отсутствует в цитоплазме. Было выявлено, что белок SURF6 не ассоциирован с митотическими ядрышковыми организаторами (рДНК). Также белок SURF6 был найден в составе цитоплазматических включений, в которых локализуются В23 и фибрилларин, хотя динамика его перемещений отличается от поведения этих белков. Так, при переходе в начале митоза SURF6 покидает остаточное ядрышко позже В23, но раньше фибрилларина, а в конце митоза SURF6 исчезает из цитоплазмы позже фибрилларина, но раньше В23. В целом, поведение SURF6 в митозе говорит о том, что он проявляет свойства, присущие белкам, вовлеченным в процессинг рРНК, а не транс-крипцию (Гурченков и др., 2005).
Было показано, что миграция белков семейства SURF6 в ядрышки обеспечивается эволюционно консервативными сигналами ядерной локализации и адресной доставки белков в ядрышко. В первичной структуре данных белков присутствуют множественные сигналы ядерной локализации – NLS (nuclear localization signal). NLS могут быть как одинарными, так и двойными. Наибольшее количество NLS содержится в консервативных SURF6-доменах белков (Polzikov et al., 2005).
Для экспериментального подтверждения присутствия NLS в последовательности SURF6 мыши была создана серия генетических конструкций, кодирующих зеленый флуоресцентный белок EGFP с различными участками последовательности белка SURF6. Локализацию химерных белков изучали в клетках мыши линии P19. При этом подавляющее большинство укороченных форм SURF6 обладали способностью мигрировать в ядра и ядрышки клеток. Однако отчетливой ядрышковой локализацией обладали только полипептиды, содержащие N-концевую часть полноразмерного белка SURF6.
Консервативный SURF6-домен насыщен остатками аргинина и лизина, которые также входят в состав двойных перекрывающихся NLS, составляющих от 20 до 45% последовательности SURF6-домена у разных видов. Как показано более чем для 79% проанализированных белков ядра и ядрышка с известными функциями, NLS входят в состав или непосредственно примыкают белковым доменам, отвечающим за ДНК- или РНК-связывающие свойства белковых молекул (Polzikov et al., 2005).
ВЗАИМОДЕЙСТВИЕ SURF6 С ДРУГИМИ БЕЛКАМИ
С помощью двух альтернативных методов: коиммунопреципитации с использованием специфичных антител к SURF6 и аффинной хроматографии было выявлено несколько белковых партнеров SURF6 человека в опухолевых клетках HeLa. Оба подхода показали, что SURF6 человека ассоциирован с многофункциональными белками В23/нуклеофозмином и нуклеолином, основным кофактором РНК-полимеразы I, UBF, а также с фактором процессинга рРНК – Ebp2 (Кордюкова и др., 2014а,б).
С помощью масс-спектрометрии и аффинной хроматографии было идентифицировано 14 белков, связывающихся с консервативным доменом SURF6 (Surf6_dom). Эти белки образуют разные функциональные группы. Основным местом локализации и функционирования этих белков являются цитоплазма (для рибосомных белков RPS9, RPL17, RPL26, трансляционных факторов EIF3D, eEF1γ, виментина) или ядро (для остальных белков). Пять белковых партнеров Surf6_dom участвуют в биогенезе рибосом: структурный белок малой (40S) субъединицы рибосомы RPS9; структурные белки большой (60S) субъединицы рибосомы RPL17, RPL26; белки Ku70 и Ku80, вовлеченные в транскрипцию рДНК. Surf6_dom образует также комплекс с белками, участвующими в процессинге мРНК (SF3B3, hnRNP С, hnRNPH, hnRNPU), что соответствует современным представлениям об участии ядрышек высших эукариот в созревании мРНК (Кордюкова и др., 2014а). Таким образом, SURF6 взаимодействует с факторами биогенеза рибосом, участвующими в регуляции транскрипции рДНК, процессинге рРНК и сборке рибосомных субъединиц. Также он взаимодействует с белками, принимающими участие в регуляции сплайсинга и клеточного цикла. В целом эти данные говорят в пользу многофункциональности белка SURF6.
Было предсказано (Ferrolino et al., 2018), что SURF6 человека содержит множественные богатые аргинином короткие поливалентные линейные мотивы (названные R-мотивами) в своей первичной структуре, благодаря которым SURF6 напрямую взаимодействует и совместно локализуется с NPM1. Исследования показали, что мультивалентные R-мотивы в неупорядоченном N-концевом фрагменте SURF6 (S6N; остатки 1–182) взаимодействуют с двумя кислотными участками внутри IDR-домена NPM1. При концентрации выше насыщения эти взаимодействия за счет электростатической комплиментарности мотивов вызывают гетеротипическое разделение жидкой и жидкой фаз (ЖЖРФ) (Ferrolino et al., 2018; Mitrea et al., 2018). Интересно, что в каплях, подобных жидкостям, NPM1–S6N активны два конкурирующих механизма: гетеротипный ЖЖРФ NPM1 с S6N (формирующий молекулярные сети NPM1–S6N) и гомотипный ЖЖРФ NPM1 (формирующий молекулярные сети NPM1–NPM1). Ранее предполагалось (Mitrea et al., 2018), что способность NPM1 претерпевать множественные типы ЖЖРФ с различными классами ядрышковых компонентов (например, с рРНК, с рибосомными и нерибосомными белками, содержащими R-мотив) играет роль буфера в поддержании жидкоподобного структурного каркаса ГК ядрышка. Эта буферная способность может компенсировать вариации в сети партнеров NPM1, присутствующих в ядре, поскольку прерибосомные частицы векторно собираются от фибриллярных центров в центре ядрышка к ГК на периферии. Было показано, что композиционные и физические свойства капель NPM1–S6N модулируются за счет конкуренции между гетеротипными механизмами NPM1–S6N и гомотипическими механизмами создания каркасов NPM1–NPM1, и что взаимодействие между этими механизмами обеспечивает динамическую адаптацию к изменениям концентраций партнеров SURF6 в модулировании доступной валентности NPM1 и NPM1-зависимого молекулярного каркаса в жидкоподобных каплях. Таким образом, NPM1 и SURF6 совместно вносят вклад в формирование и функциональную регуляцию каркаса ГК (Ferrolino et al., 2018).
Также было высказано предположение (Ferrolino et al., 2018), что роль SURF6 в регулировании состава и биофизических свойств ядрышкового матрикса распространяется не только на ГК, но также на фибриллярные центры и плотный фибриллярный компонент. Благодаря множеству конкурентных взаимодействий с множественными ядрышковыми белками, а также ДНК и РНК, SURF6 может динамически модулировать особенности ядрышкового каркаса во время биогенеза рибосом, возможно, способствуя образованию градиента ядрышкового каркаса, который направляет путь сборки рибосомных частиц. Выяснилось, что включение SURF6 в гомотипные капли NPM1 изменяет подвижность NPM1, вязкость капли, ее состав и гидрофобность. Таким образом, нарушение непрерывности концентрации SURF6 в ядрышке и связанное с этим влияние на вязкость и гидрофобность каркаса могут вносить вклад в этот гипотетический градиент, способствующий сборке рибосом (Ferrolino et al., 2018). Интересно, что резкие различия в локальной вязкости, гидрофобности и поверхностном натяжении, как было показано, опосредуют компартментализацию плотного фибриллярного компонента внутри ГК (Correll et al., 2019). Однако потребуются дальнейшие исследования, чтобы проверить гипотезы относительно того, как различные типы конкурирующих каркасов, включающие многочисленные белки и нуклеиновые кислоты, влияют на молекулярные перестройки внутри ядрышка, которые сопровождают биогенез рибосом.
ДРОЖЖЕВОЙ ГОМОЛОГ SURF6 – Rrp14
Наличие консервативного SURF6-домена позволило считать гомологом SURF6 мыши белок ядрышка Rrp14 у пекарских дрожжей S. сerevisiae – белок, кодируемый геном ykl082c. Идентичность аминокислотных остатков между консервативными доменами белков SURF6 мыши и Rrp14 составляет 23%. Подобно консервативным доменам всех белков SURF6-семейства, домен белка Rrp14 обладает высококонсервативным “ядром” из 60 а.о., девять из которых сохраняются неизменными у всех видов (Polzikov et al., 2005). Известно, что экспрессия гена ykl082c осуществляется специфическим дрожжевым транскрипционным фактором Rap1, вовлеченным в транс-крипцию генов белков, участвующих в биогенезе рибосом (Planta, 1997).
Сборка рибосомных субъединиц у дрожжей также начинается в ядрышке, где РНК-полимераза I транскрибирует предшественник 35S пре-рРНК, из которого после обработки и удаления внешних и внутренних транскрибируемых спейсеров (ETS и ITS) в ходе созревания генерируется 18S, 5.8S и 25S рРНК (рис. 4а). После эндонуклеолитического расщепления предшественника рРНК по сайту A2 у дрожжей субъединицы пре-40S и пре-60S следуют разными путями биогенеза (Klinge, Woolford, 2019). Модульная сборка частицы 90S обеспечивает основу для доменного складывания рРНК-предшественника 18S. В отличие от субъединицы 40S, архитектура субъединицы 60S с ее шестью сильно переплетенными доменами 25S рРНК является более сложной (рис. 4б) (Woolford, Baserga, 2013). В ранней пре-рРНК домен VI связывается с доменами I и II и с участком 5.8S предшественника рРНК (рис. 4в). С этого начинается формирование туннеля выхода полипептида. Его созревание прогрессирует по мере складывания доменов рРНК в следующем порядке: VI, V, III и IV. Полная сборка туннеля выхода полипептида достигается только тогда, когда домен V полностью свернут (рис. 4в). После этого требуется несколько нуклеоплазматических стадий для расщепления ITS2 прежде, чем частицы будут экспортированы в цитоплазму, где они окончательно созревают (Kater et al., 2017). Особенностью ядрышковой частицы пре-60S является ее открытая архитектура, в которой открытые для растворителя домены I, II и VI инкапсулированы рядом факторов сборки рибосом, формирующих кольцеобразную структуру на стороне, открытой для растворителя (рис. 5а). При этом различные факторы, действуя совместно, стерически предотвращают преждевременные контакты РНК–белок и РНК–РНК (Sanghai et al., 2018).
Рис. 4.
Структура дрожжевой пре-рРНК (Oeffinger et al., 2007). 5'ETS и 3'ETS – внешние транскрибируемые спейсеры; ITS – внутренние транскрибируемые спейсеры рибосомного гена; 18S, 5.8S, 25S рРНК – участки, кодирующие зрелые рРНК. Участки ферментативного расщепления обозначены буквами A, B, C, D, E (а). Вторичная структура 25S и 5.8S рРНК S. cerevisiae (б). Справа 25S рРНК содержит шесть доменов (I–VI) вторичной структуры. Пары оснований 5.8S рРНК (черные) с доменом I (адаптировано из https://crw-site.chemistry.gatech.edu/). Эти вторичные структуры филогенетически консервативны во всех царствах, хотя эукариотические рРНК содержат сегменты, не обнаруженные в прокариотических или архейных рРНК (Woolford, Baserga, 2013). Последовательность сборки доменов пре-рРНК пре-60S (в). Сборка рибосомных белков и факторов биогенеза на формирующемся предшественнике 35S рРНК начинается котранскрипционно. Формирование туннеля выхода полипептида (показано здесь как черный кружок) начинается с того, что домен VI связывается с доменами I и II и с участком 5.8S предшественника рРНК. Его созревание прогрессирует по мере складывания доменов рРНК в следующем порядке: VI, V, III и IV и завершается только тогда, когда домен V полностью свернут, как это наблюдается в состоянии F (Kater et al., 2017).

Рис. 5.
Структура ранней ядрышковой частицы пре-60S S. cerevisiae, полученной с помощью криоэлектронной микроскопии (а). Подбор плотности крио-ЭМ белка Rrp14, входящего в состав предшественников больших субъединиц рибосом пре-60S S. cerevisiae (б). Изображение создано в PyMO (Sanghai et al., 2018).

При помощи метода равновесного центрифугирования в градиенте сахарозы доказано, что белок Rrp14 является компонентом ранних 90S пре-рибосомных комплексов и связан с предшественниками 60S рибосомных частиц (Oeffinger et al., 2007a). Истощение пула Rrp14 с помощью метода генного нокаута приводит к почти полной блокировке процессинга 20S пре-рРНК, предшественника 18S рРНК, а также 27S пре-рРНК, предшественника 25S рРНК, и сопровождается гибелью клеток.
Также показано, что нокаут белка SURF6 в дрожжах приводит к накоплению аберрантных A2–C2 фрагментов (рис. 4а). Таким образом, Rrp14 может прямо или опосредованно препятствовать преждевременному расщеплению ITS2 по сайту С2, с которого начинается деградация ITS2 у дрожжей, не затрагивая другие стадии созревания пре-рРНК (Oeffinger et al., 2007a).
Недавно с помощью криоэлектронной микроскопии было показано, что Rrp14 совместно с гетеродимером Ssf1–Rrp15 образуeт кольцевую структуру, инкапсулирующую домены I, II и VI 25S рРНК, которые выравнивают две стороны формируемого туннеля выхода полипептида. В то время как длинный комплекс Ssf1–Rrp15 расположен на границе раздела доменов I и VI, С-концевая консервативная спираль Rrp14 соединяет домены II и VI. При этом Ssf1 занимает ту же позицию, что и Rpl31. Rpl31 в более поздних частицах пре-60S связывается на границе раздела доменов III и VI около туннеля выхода полипептида, который создается доменами I, III и VI на стороне, подверженной воздействию растворителя (Sanghai et al., 2018; Klinge, Woolford, 2019). На этом этапе c пре-рРНК уже связаны рибосомные белки Rpl7, Rpl13, Rpl17, Rpl24, Rpl26. Последующая стадия созревания состояний, как полагают, включает соединение доменов III и VI и образование туннеля выхода полипептида на стороне, открытой для растворителя. Это может сопровождаться вставкой N-конца белка Nog1 в формирующийся туннель и заменой Ssf1–Rrp15 на Rpl31 (Sanghai et al., 2018). С использованием техники экспрессии и очистки усеченной пре-рРНК пре-60S была определена иерархическая последовательность присоединения факторов раннего биогенеза большой субъединицы рибосом. Было показано, что Rrp14 связан с IV доменом 25S рРНК (Chaker-Margot, Klinge, 2019).
Таким образом, белок Rrp14 совместно с белками Ssf1 и Rrp15 (у человека PPAN и Rrp15 соответственно) действуют как шапероны доменов I и VI, образующих две стороны формирующегося туннеля выхода полипептида до связывания Rpl31 и удаления ITS2. Одновременно с Rrp14 c пре-рРНК связаны белки Ssf1/PPAN, Rrp15, Rrp1/Nop52, Ebp2/EBNA1BP2, Brx1/Brix1, NSA1/WDR74, YTM1/WDR12, Rpf1, Erb1/BOP1, Nop7/Pes1, Nop15/MKI67IP, Nop16, Mak16, Nog1/GTPBP4 (Sanghai et al., 2018; Klinge, Woolford, 2019).
Раннее с помощью аффинной хроматографии и дрожжевой двугибридной системы показано, что у дрожжей S. cerevisiae с белком Rrp14 взаимодействуют следующие белки (табл. 1). Как видно, большинство белковых партнеров Rrp14 участвуют в процессинге рибосом. И часть из них (Rrp1, Ebp2, Ssf1, NSA1, Nop15, Mak11) была идентифицирована в том же рибонуклеиновом комплексе, что и Rrp14 (Sanghai et al., 2018). Причем белки Ssf1, LOC1, DBP9, Mak11, как и Rrp14, связаны с IV доменом пре-60S (Chaker-Margot, Klinge, 2019).
Таблица 1.
Список белков пекарских дрожжей S. cerevisiae, взаимодействующих с белком Rrp14, членом эволюционно консервативного белкового семейства SURF6
| Гомолог у дрожжей |
Гомолог у человека |
Функция белка у дрожжей |
|---|---|---|
| Ssf1 | PPAN | Белок, принадлежащий Brix-семейству и являющийся компонентом 66S пре-рибосомальной частицы, необходим для созревания большой субъединицы (Fatica et al., 2002; Dez, Tollervey, 2004; Krogan et al., 2006; Klinge, Woolford, 2019) |
| Nop12 | RBM34 | Белок ядрышка, вовлеченный в процессинг пре-25S рРНК и биогенез большой 60S рибосомной субъединицы, содержит РНК-распознающий мотив (RRM) и связан с белком Ebp2 (Wu et al., 2001; Yamada et al., 2007; Klinge, Woolford, 2019) |
| LOC1 | Нет | Ядерный белок, вовлеченный в асимметричную локализацию ASH1 мРНК, связывает двухцепочную РНК in vitro, является компонентом 66S пре-рибо-сомальной частицы (Urbinati et al., 2006; Yamada et al., 2007) |
| NSA1 | WDR74 | Компонент 66S пре-рибосомальной частицы, вовлеченный в биогенез 60S рибосомной субъединицы (Kressler et al., 2008; Klinge, Woolford, 2019) |
| Rrp36 | Rrp36 | Компонент 90S пре-рибосом вовлечен в процесс раннего расщепления 35S пре-рРНК и биогенез 40S субъединицы. Гомолог белка Rrp36 у человека участвует в расщеплении по сайту A0, A1 (Gérus et al., 2010; Klinge, Woolford, 2019) |
| FAF1 | Нет | Белок, необходимый для пре-рРНК процессинга и сборки 40S рибосомной субъединицы, участвует в расщеплении по A1-, A0-, A2-сайтам (Karkusiewicz et al., 2004; Yamada et al., 2007) |
| Nop15 | MKI67IP | Компонент 66S пре-рибосомных частиц, вовлеченный в биогенез 60S рибосомной субъединицы, локализован и в ядрышке, и в цитоплазме (Oeffinger et al., 2007b; Kressler et al., 2008; Breitkreutz et al., 2010; Klinge, Woolford, 2019) |
| Ebp2 | EBNA1BP2 | Белок, существенно необходимый для созревания 25S рРНК и сборки 60S рибосомной частицы, участвует в процессинге 27SA3 в 27SB пре-рРНК (у человека 36S в 32S пре-рРНК соответственно), локализуется в ядрышке, является компонентом 66S пре-рибосомных частиц, играет ключевую роль в митотическом разделении вируса Эпштейна–Барр (Huber et al., 2000; Yamada et al., 2007; Klinge, Woolford, 2019) |
| Mak11 | PAK1IP1 | Белок, вовлеченный в ранний биогенез 60S рибосомной субъединицы (Chaker-Margot, Klinge, 2019), является существенным для клеточного роста и репликации M1 дцРНК вируса (Saveanu et al., 2007; Lebreton et al., 2008; Klinge, Woolford, 2019) |
| DBP9 | DDX56 | АТФ-зависимая РНК-хеликаза, принадлежащая к DEAD-box-семейству, вовлеченная в биогенез 60S рибосомной субъединицы (Daugeron et al., 2001; Kikuma et al., 2004; Yamada et al., 2007) |
| UTP11 | UTP11L | Субъединица U3 комплекса SSU – процессомы, вовлеченной в биогенез 18S и сборку малой рибосомной субъединицы (Gallagher et al., 2004; Yamada et al., 2007; Turner et al., 2009) |
| Rrp1 | Rrp1/Nop52 | Эволюционно консервативный белок, необходимый для биогенеза 60S рибосомной субъединицы и процессинга пре-рРНК в рРНК, ассоциирован с несколькими различными 66S пре-рибосомными частицами (Klinge, Woolford, 2019). Гомолог у человека – Nop52, который вовлечен в биогенез 25S рРНК (Savino et al., 1999; Horsey et al., 2004; Yoshikawa et al., 2015). Интересно, что белок Rrp1 взаимодействует с белком SIC1, участвующим в регуляции перехода клеток из G1- в S-фазу клеточного цикла (Cross et al., 2007; Costanzo et al., 2010) |
| GIC1 GIC2 |
Нет | Rho-подобные ГТФ-связывающие белки GIC1 и GIC2 – эффекторы белка CDC42 (Uetz et al., 2000; Drees et al., 2001; Höfken, Schiebel, 2004) |
| ZDS2 | Нет | Белок, участвующий в организации теломер и клеточной пролиферации, взаимодействующий с молчащими белками теломер, вовлеченный в транскрипционный сайленсинг, участвует в регуляции вхождения клеток в митоз посредством активации CDC14p-фосфатазы в ранней анафазе. В течение интерфазы ZDS2 локализован в ядре, преимущественно в ядрышке. В течение интерфазы ZDS2 локализован в ядре, а CDC14p – преимущественно в ядрышке. Любопытно, что белок ZDS2, в свою очередь, взаимодействует с белком Sir2, принадлежащим к семейству SIR-белков, играющих активную роль в регуляции транскрипции рибосомных генов (Yakura et al., 2006). Кроме того, белки GIC1 и GIC2, а также ZDS2 взаимодействуют с фосфотазой CDC14, принимающей участие в регуляции вхождения клеток в митоз и в инициации репликации (Yakura et al., 2006; Queralt, Uhlmann, 2008) |
| BUD7 | Нет | Белок, играющий роль в установлении клеточной полярности (Zahner et al., 1996; Drees et al., 2001) |
| KIN4 | Нет | Серин/триониновая белковая киназа, ингибирующая митотическую входную сеть, когда веретено деления активировано (Pereira, Schiebel, 2005; Krogan et al., 2006; Breitkreutz et al., 2010) |
| CKA1 | CSNK2A1 | Альфа 1 субъединица казеин киназы II – серин/триониновая белковая киназа, играющая роль в процессах клеточного роста и пролиферации, является голо-ферментом и содержит CKA2, CKB1 и CKB2, транскрипционные факторы и РНК-полимеразы (Ho et al., 2002; Breitkreutz et al., 2010) |
| YPL141C | Нет | Белок, предположительно являющийся белковой киназой, имеет подобие с KIN4p (Gallagher et al., 2004; Krogan et al., 2006; Breitkreutz et al., 2010) |
| PRP43 | DHX15 | РНК-хеликаза, принадлежащая DEAH-box-семейству, функционирует и как полимераза I, и как полимераза II, вовлечена в вырезание лариат-интрона из сплайсосомы (Lebaron et al., 2005) |
| RPS16A и RPS16B |
Нет | Белки с неясной функцией (Yamada et al., 2007) |
Важно отметить, что часть белковых партнеров Rrp14 участвует в установлении клеточной полярности и регуляции пролиферации. Было показано, что штаммы дрожжей, у которых было пониженное содержание Rrp14, имели дефекты в расположении и элонгации веретена деления в течение митоза, чего не наблюдалось ранее при понижении содержания других факторов биогенеза рибосом (Yamada et al., 2007). Логично предположить поэтому, что белок Rrp14 может регулировать и координировать важные биологические процессы – биогенез рибосом, клеточный цикл и клеточную полярность.
Как описано ранее, гомологи белков Rrp1, Ebp2 человека также являются белковыми партнерами SURF6 (Кордюкова и др., 2014б), что говорит в пользу участия SURF6 в биогенезе большой субъединицы рибосом у млекопитающих.
УЧАСТИЕ SURF6 В БИОГЕНЕЗЕ РИБОСОМ МЛЕКОПИТАЮЩИХ
В отличие от низших эукариот, биогенез рибосом млекопитающих изучен недостаточно. В основном это связано с невозможностью провести очистку пре-рибосомных комплексов (так как ядрышки не солюбилизируются эффективно, без нарушений) с использованием протоколов экстракции, существующих в настоящее время (Nieto et al., 2020).
С помощью равновесного центрифугирования в градиенте сахарозы было показано, что SURF6 человека кофракционируется с предшественниками большой субъединицы (Couté et al., 2006). Тем не менее скрининг ядрышковых белков на основе РНК-интерференции с оценкой изменения количественного содержания промежуточных продуктов процессинга пре-рРНК показал, что нокдаун SURF6, как и его партнера NPM1, не приводит к существенным изменениям в биогенезе рибосом (Tafforeau et al., 2013). Что, возможно, связано с недостаточной эффективностью нокдауна.
Было показано, что в фибробластах мыши оверэкспрессия SURF6 заметно увеличивает количество всех промежуточных продуктов рРНК, за исключением 36S рРНК, являющегося самым длинным общим предшественником для 5.8S и 28S рРНК. Наиболее заметные изменения касаются накопления 45S пре-рРНК (общий предшественник для 18S, 5.8S и 28S рРНК, образованной ниже 47S пре-рРНК), 34S и 20S рРНК (предшественников 18S рРНК) и 32S рРНК (Moraleva et al., 2017).
Также было показано, что оверэкспрессия SURF6 в клетках мыши приводит к многократному (более чем в семь раз) увеличению содержания фрагментов второго внутреннего транскрибируемого спейсера ITS2 и почти двукратному увеличению содержания долгоживущих участков 5'-внешнего транскрибируемого спейсера 5'ETS. Содержание же фрагментов, соответствующих 18S рРНК, ITS1, 5.8S рРНК и 28S рРНК, изменяется менее заметно, по сравнению с ITS2 и 5'ETS.
Также методом FISH продемонстрировано, что SURF6 колокализуется с ITS2 и 5'ETS. Имеющиеся данные не дают сделать заключение об участии SURF6 в сайт-специфическом расщеплении ITS2, но позволяют предположить, что SURF6 мыши может принимать участие в стабилизации ITS2 и 5'ETS (Ползиков и др., 2010), что соответствует данным, полученным для дрожжевого гомолога SURF6 Rrp14. Вероятно, стабилизация ITS2 является эволюционно консервативной функцией семейства белков SURF6. Вопрос о механизмах стабилизации ITS2 с участием SURF6/Rrp14 остается открытым.
Предполается, что SURF6 непосредственно связывается с ITS2, препятствуя его расщеплению рибонуклеазами. Однако среди белковых партнеров SURF6 на сегодня не описаны рибонуклеазы, и среди белковых партнеров Rrp14 не выявлено белков, обладающих рибонуклеазной активностью.
АКТИВНОСТЬ SURF6 ПРИ КЛЕТОЧНОЙ ДИФФЕРЕНЦИРОВКЕ И ПРОЛИФЕРАЦИИ
Экспериментальным путем доказано, что истощение пула белка Rrp14 у дрожжей (Oeffinger et al., 2007a; Yamada et al., 2007), в соматических клетках (Polzikov et al., 2007) и эмбрионах мыши (Romanova et al., 2006) приводит к гибели клеток. Эти данные позволяют рассматривать белки семейства SURF6 как эволюционно консервативные и жизненно необходимые белки “домашнего хозяйства” эукариот. Однако изучение дрожжевого SURF6 – белка Rrp14 – показало, что этот белок многофункционален и необходим не только для сборки субъединиц рибосомы и процессинга рРНК, но также для поляризации и деления клеток (Oeffinger et al., 2007a; Yamada et al., 2007).
С помощью дифференциального дисплея мРНК и нозерн-блотов было обнаружено, что экспрессия гена Surf-6 на уровне транскриптов регулируется в процессе дифференцировки эмбриональных стволовых клеток. Было исследовано влияние на экспрессию Surf-6 эритропоэтина – цитокина, играющего важную роль в процессе деления и дифференцировки предшественников эритроидных клеток. Добавление эритропоэтина в среду культивирования эмбриональных стволовых клеток, экспрессирующих рецептор эритропоэтина, и к гематопоэтическим стволовым клеткам-предшественникам сильно повышало уровень экспрессии гена Surf-6 (Xia et al., 2000).
При анализе экспрессии гена Surf-6 в процессе эмбриогенеза у шпорцевой лягушки (Wolff et al., 2002) установлено, что наибольший уровень экспрессии наблюдался на 30–38 стадиях эмбриогенеза, соответствующих латеральной фазе, а падение экспрессии – на 8–9 стадиях, соответствующих этапу бластулы, и 40–45 стадиях, соответствующих этапу начала генерации нейронов в таламусе до завершения формирования таламуса. Высокий уровень экспрессии гена Surf-6 на большинстве стадий эмбриогенеза, а также уменьшение экспрессии на терминальных стадиях эмбриогенеза может свидетельствовать в пользу участия белка SURF6 в процессах клеточной дифференцировки у эмбрионов шпорцевой лягушки. Сходные результаты были получены при изучении экспрессии Surf-6 у взрослых особей D. melanogaster по сравнению с эмбриональной стадией и личинкой: пик экспрессии наблюдается на 0–12 стадиях эмбриона и на сроке 18–24 ч у взрослых самок, процессы клеточной дифференцировки сопровождались снижением уровня содержания SURF6 (Arbeitman et al., 2002).
Таким образом, ген Surf-6 и его возможные гомологи – важные участники клеточного метаболизма, ассоциированные предположительно с процессами клеточной дифференцировки и пролиферации.
Обнаружено, что индукция нокдауна SURF6 в фибробластах мыши приводит к гибели клеток путем апоптоза. Также методом проточной цитофлуориметрии показано, что нокдаун увеличивает на ~8% содержание клеток в G1-периоде и снижает на ~7% долю клеток в S-периоде, по сравнению с контролем. Доля клеток, находящихся в G2-периоде клеточного цикла и митозе, в обеих популяциях клеток оставалась практически на одном уровне. Эти наблюдения свидетельствуют о том, что нокдаун SURF6 препятствует переходу клеток из G1- в S-период клеточного цикла (Polzikov et al., 2007).
Как было указано ранее, гомологами белка SURF6 млекопитающих у дрожжей S. cerevisiae и нематоды C. elegans являются гены ykl082c и ZK546.14, кодирующие белки Rrp14 и CE02914 соответственно. Оказалось, что удаление с помощью гомологичной рекомбинации двух копий гена ykl082c приводит к летальному исходу у дрожжей (Giaever et al., 2002). Подавление экспрессии гена ZK546.14 у нематоды сопровождается замедлением роста и аномалиями в развитии взрослых особей. Истощение белка CE02914 приводит к остановке развития зародышей на стадии личинки и их последующей гибели (Piano et al., 2002). В совокупности эти наблюдения говорят о том, что белок ядрышка SURF6 относится к жизненно необходимым белкам эукариот.
БИОЛОГИЧЕСКАЯ РОЛЬ SURF6 В ЭМБРИОНАЛЬНОМ РАЗВИТИИ МЛЕКОПИТАЮЩИХ
В отличие от соматических клеток, раннее развитие зародышей млекопитающих характеризуется периодом транскрипционного покоя и присутствием ядрышковых предшественников, которые собираются внутри ядра в зрелые ядрышки, начиная со стадии двухклеточного зародыша. В доимплантационном периоде развития зародышей мыши формирование ядрышка заканчивается не раньше стадии морулы. мРНК Surf-6 была идентифицирована методом ПЦР на стадии одноклеточного зародыша, но ее количество значительно уменьшалось при переходе от одноклеточной стадии к двухклеточной. Кроме того, было обнаружено, что количество мРНК Surf-6 нарастает при переходе от двухклеточной стадии к стадии морулы. Также было показано, что распределение SURF6 в зародышах мыши подобно, хотя и не идентично, распределению других белков ядрышка – B23 и фибрилларина. Истощение SURF6 методом РНК-интерференции приводит к снижению содержания 18S мРНК, к остановке развития зародышей на стадии морулы и к их последующей гибели. В совокупности эти наблюдения говорят о том, что SURF6 участвует в раннем эмбриогенезе не только плодовой мушки и нематоды, но и млекопитающих (Romanova et al., 2006).
СВЯЗЬ SURF6 С ПРОЛИФЕРАТИВНЫМ СТАТУСОМ КЛЕТОК
Было показано, что в течение клеточного цикла наибольший уровень SURF6 наблюдается в G2-фазе клеточного цикла, а уровень SURF6 в клетках, находящихся в состоянии пролиферативного покоя (G0-фазе), заметно снижен, по сравнению с асинхронной культурой (Гурченков и др., 2005). При анализе различных типов клеток особенно высокий уровень экспрессии SURF6 описан в активно пролиферирующих клетках – в эмбриональных, в предшественниках, в гемопоэтических стволовых линиях (Ringwald et al., 2012) и в некоторых опухолевых клетках (http://www.proteinatlas.org/ENSG00000148296-SURF6/cancer). Интересно, что SURF6 отсутствует в лимфоцитах селезенки мыши, но его содержание прогрессивно нарастает после активации к пролиферации in vitro с помощью митогена. При этом экспрессия SURF6 начинается раньше белков PCNA и Ki-67, которые являются основными маркерами пролиферации, используемыми в онкодиагностике (Моралева и др., 2009). Исследование содержания SURF6 на иммуноцитохимическом уровне и иммуноблотах в лимфоцитах человека: нормальных, или покоящихся; активированных к пролиферации митогеном in vitro; в нативных лимфоцитах, полученных от больных лимфопролиферативными заболеваниями, – показало, что при определенных условиях белок ядрышка SURF6 не выявляется в нормальных лимфоцитах, но обнаруживается в активированных лимфоцитах, а также в лимфоцитах больных, при этом наибольший уровень отмечен у больных с диагнозом лимфома мантийной зоны. Таким образом, SURF6 может служить новым активационным маркером лимфоцитов (Малышева и др., 2010а,б; Моралева и др., 2020).
Неупорядоченная первичная структура и высокий позитивный заряд молекулы SURF6 способствуют его взаимодействию с NPM1, что, по мнению некоторых авторов, является ключевым условием формирования и функционирования ядрышка и его отдельных субдоменов (Smetana, 2002; Ferrolino et al., 2018). Это может быть причиной изменения содержания SURF6 при активации, бласттрансформации и дедифференцировке клеток.
На основании крупномасштабного скрининга образования колоний с помощью трансфекции кДНК SURF6 был идентифицирован как белок, предположительно связанный с онкогенезом в культивируемых фибробластах мыши и опухолевых клетках человека (Wan et al., 2004). Обнаружено, что оверэкспрессия SURF6 в фибробластах мыши не имеет заметной цитотоксичности, но значительно ускоряет пролиферацию за счет сокращения времени G1/S-перехода (Moraleva et al., 2017). Эти данные подтверждают ранее высказанную гипотезу о том, что SURF6 млекопитающих является предполагаемым онкобелком (Wan et al., 2004).
ЗАКЛЮЧЕНИЕ
Суммируя литературные данные, можно заключить, что белок ядрышек млекопитающих SURF6 относится к белкам, необходимым для поддержания клеток в жизнеспособном состоянии, подобно дрожжевому гомологу Rrp14, участвует в биогенезе рибосом, способствуя стабилизации внутригенного транскрибируемого спейсера ITS2 и, возможно, 5'ETS. Также анализ имеющихся литературных данных позволяет предположить участие белка ядрышка SURF6 не только в биогенезе рибосом, но и в регуляции клеточной пролиферации и дифференцировке. Однако прямые данные о функциональной значимости белка SURF6 в клетках млекопитающих до сих пор отсутствуют.
Список литературы
Гурченков В.В., Ползиков М.А., Магоулас К. и др. Свойства и функции нового белка ядрышка SURF-6 в клетках мыши 3T3 // Биоорг. химия. 2005. Т. 31. № 6. С. 521–528.
Кордюкова М.Ю., Ползиков М.А., Шишова К.В., Зацепина О.В. Анализ белковых партнеров белка ядрышка человека SURF6 в клетках HeLa методом аффинной адсорбции // Биоорг. химия. 2014а. Т. 40. № 4. С. 421.
Кордюкова М.Ю., Ползиков М.А., Шишова К.В., Зацепина О.В. Функциональное значение белка ядрышка SURF6 человека – ключевого белка одноименного семейства эукариот // Докл. РАН. 2014б. Т. 455. № 4. 471–473.
Коваленко Т.Ф., Патрушев Л.И. Псевдогены как функционально значимые элементы генома // Биохимия. 2018. Т. 83. № 11. P. 1643–1662.
Малышева М.В., Булычева Т.И., Дейнеко Н.Л. и др. Содержание ключевых белков ядрышка в лимфоидных клетках здоровых лиц и больных с лимфопролиферативными заболеваниями // Клин. лаб. диагн. 2010а. Т. 12. С. 23–27.
Малышева М.В., Моралева А.А., Дейнеко Н.Л. и др. Сравнительный анализ экспрессии ключевых белков ядрышка в лимфоцитах периферической крови здоровых доноров, активированных к пролиферации in vitro // Иммунология. 2010б. Т. 31. № 1. С. 13–17.
Моралева А.А., Малышева М.В., Магоулас Х. и др. Ранняя экспрессия белка ядрышка SURF-6 в лимфоцитах селезенки мыши, активированных к пролиферации in vitro // Бюл. экспер. биол. мед. 2009. Т. 147. № 5. С. 507–511.
Моралева А.А., Малышева М.В., Хайдуков С.В. и др. Повышенное содержание белка ядрышка SURF6 в активированных лимфоцитах доноров и в лимфоцитах больных лимфопролиферативными заболеваниями // Докл. РАН. Науки о жизни. 2020. Т. 494. № 1. С. 532–536.
Ползиков М.А., Вейко Н.Н., Жарская О.О. и др. Сверхпродукция белка ядрышка SURF-6 в фибробластах мыши NIH/3T3 приводит к стабилизации внутренних транскрибируемых спейсеров пре-рРНК // Биоорг. хим. 2010. Т. 36. № 5. С. 661–671.
Antequera F., Macleod D., Bird A.P. Specific protection of methylated CpGs in mammalian nuclei // Cell. 1989. V. 58. № 3. 509–517.
Arbeitman M.N., Furlong E.E.M., Imam F. et al. Gene expression during the life cycle of Drosophila melanogaster // Science. 2002. V. 297. № 5590. P. 2270–2275.
Armes N., Gilley J., Fried M. The comparative genomic structure and sequence of the surfeit gene homologs in the puffer fish Fugu rubripes and their association with CpG-rich islands // Genome Res. 1997. V. 7. № 12. P. 1138–1152.
Breitkreutz A., Choi H., Sharom J.R. et al. A global protein kinase and phosphatase interaction network in yeast // Science. 2010. V. 328. № 5981. P. 1043–1046.
Chaker-Margot M., Klinge S. Assembly and early maturation of large subunit precursors // RNA. 2019. V. 25. № 4. P. 465–471.
Cole E.G., Gaston K. A functional YY1 binding site is necessary and sufficient to activate Surf-1 promoter activity in response to serum growth factors // Nucl. Acids Res. 1997. V. 25. № 18. P. 3705–3711.
Correll C.C., Bartek J., Dundr M. The nucleolus: a multiphase condensate balancing ribosome synthesis and translational capacity in health, aging and ribosomopathies // Cells. 2019. V. 8. № 8. P. 869.
Costanzo M., Baryshnikova A., Bellay J. et al. The genetic landscape of a cell // Science. 2010. V. 327. № 5964. P. 425–431.
Couté Y., Burgess J.A., Diaz J.-J. et al. Deciphering the human nucleolar proteome // Mass Spectrom. Rev. 2006. V. 25. № 2. P. 215–234.
Cross F.R., Schroeder L., Bean J.M. Phosphorylation of the Sic1 inhibitor of B-type cyclins in Saccharomyces cerevisiae is not essential but contributes to cell cycle robustness // Genetics. 2007. V. 176. № 3. P. 1541–1555.
Daugeron M.C., Kressler D., Linder P. Dbp9p, a putative ATP-dependent RNA helicase involved in 60S-ribosomal-subunit biogenesis, functionally interacts with Dbp6p // RNA. 2001. V. 7. № 9. P. 1317–1334.
Dez C., Tollervey D. Ribosome synthesis meets the cell cycle // Curr. Opin. Microbiol. 2004. V. 7. № 6. P. 631–637.
Drees B.L., Sundin B., Brazeau E. et al. A protein interaction map for cell polarity development // J. Cell Biol. 2001. V. 154. № 3. P. 549–571.
Duhig T., Ruhrberg C., Mor O., Fried M. The human Surfeit locus // Genomics. 1998. V. 52. № 1. P. 72–78.
Fatica A., Cronshaw A.D., Dlakić M., Tollervey D. Ssf1p prevents premature processing of an early pre-60S ribosomal particle // Mol. Cell. 2002. V. 9. № 2. P. 341–351.
Ferrolino M.C., Mitrea D.M., Michael J.R., Kriwacki R.W. Compositional adaptability in NPM1–SURF6 scaffolding networks enabled by dynamic switching of phase separation mechanisms // Nat. Commun. 2018. V. 9. № 1. P. 5064.
Gallagher J.E.G., Dunbar D.A., Granneman S. et al. RNA polymerase I transcription and pre-rRNA processing are linked by specific SSU processome components // Genes Dev. 2004. V. 18. № 20. P. 2506–2517.
Gérus M., Bonnart C., Caizergues-Ferrer M. et al. Evolutionarily conserved function of RRP36 in early cleavages of the pre-rRNA and production of the 40S ribosomal subunit // Mol. Cell. Biol. 2010. V. 30. № 5. P. 1130–1144.
Giaever G., Chu A.M., Ni L. et al. Functional profiling of the Saccharomyces cerevisiae genome // Nature. 2002. V. 418. № 6896. P. 387–391.
Ho Y., Gruhler A., Heilbut A. et al. Systematic identification of protein complexes in Saccharomyces cerevisiae by mass spectrometry // Nature. 2002. V. 415. № 6868. P. 180–183.
Höfken T., Schiebel E. Novel regulation of mitotic exit by the Cdc42 effectors Gic1 and Gic2 // J. Cell Biol. 2004. V. 164. № 2. P. 219–231.
Horsey E.W., Jakovljevic J., Miles T.D. et al. Role of the yeast Rrp1 protein in the dynamics of pre-ribosome maturation // RNA. 2004. V. 10. № 5. P. 813–827.
Huber M.D., Dworet J.H., Shire K. et al. The budding yeast homolog of the human EBNA1-binding protein 2 (Ebp2p) is an essential nucleolar protein required for pre-rRNA processing // J. Biol. Chem. 2000. V. 275. № 37. P. 28764–28773.
Huxley C., Fried M. The mouse surfeit locus contains a cluster of six genes associated with four CpG-rich islands in 32 kilobases of genomic DNA // Mol. Cell. Biol. 1990. V. 10. № 2. P. 605–614.
Karkusiewicz I., Rempola B., Gromadka R. et al. Functional and physical interactions of Faf1p, a Saccharomyces cerevisiae nucleolar protein // Biochem. Biophys. Res. Comm. 2004. V. 319. № 2. P. 349–357.
Kater L., Thoms M., Barrio-Garcia C. et al. Visualizing the assembly pathway of nucleolar pre-60S ribosomes // Cell. 2017. V. 171. № 7. P. 1599–1610.
Kikuma T., Ohtsu M., Utsugi T. et al. Dbp9p, a member of the DEAD box protein family, exhibits DNA helicase activity // J. Biol. Chem. 2004. V. 279. № 20. P. 20692–20698.
Klinge S., Woolford J.L. Ribosome assembly coming into focus // Nat. Rev. Mol. Cell. Biol. 2019. V. 20. № 2. P. 116–131.
Kressler D., Roser D., Pertschy B., Hurt E. The AAA ATPase Rix7 powers progression of ribosome biogenesis by stripping Nsa1 from pre-60S particles // J. Cell Biol. 2008. V. 181. № 6. P. 935–944.
Krogan N.J., Cagney G., Yu H. et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae // Nature. 2006. V. 440. № 7084. P. 637–643.
Lebaron S., Froment C., Fromont-Racine M. et al. The splicing ATPase prp43p is a component of multiple preribosomal particles // Mol. Cell. Biol. 2005. V. 25. № 21. P. 9269–9282.
Lebreton A., Rousselle J. C., Lenormand P. et al. 60S ribosomal subunit assembly dynamics defined by semi-quantitative mass spectrometry of purified complexes // Nucl. Acids Res. 2008. V. 36. № 15. P. 4988–4999.
Lindström M.S., Jurada D., Bursac S. et al. Nucleolus as an emerging hub in maintenance of genome stability and cancer pathogenesis // Oncogene. 2018. V. 37. № 18. P. 2351–2366.
Magoulas C., Fried M. The Surf-6 gene of the mouse surfeit locus encodes a novel nucleolar protein // DNA Cell Biol. 1996. V. 15. № 4. P. 305–316.
Magoulas C., Fried M. Isolation and genomic analysis of the human surf-6 gene: a member of the Surfeit locus // Gene. 2000. V. 243. № 1–2. P. 115–123.
Magoulas C., Zatsepina O.V., Jordan P.W. et al. The SURF-6 protein is a component of the nucleolar matrix and has a high binding capacity for nucleic acids in vitro // Eur. J. Cell Biol. 1998. V. 75. № 2. P. 174–183.
Marcu K.B., Bossone S.A., Patel A.J. Myc function and regulation // Ann. Rev. Biochem. 1992. V. 61. P. 809–860.
Mitrea D.M., Cika J.A., Stanley C.B. et al. Self-interaction of NPM1 modulates multiple mechanisms of liquid–liquid phase separation // Nat. Commun. 2018. V. 9. № 1. P. 842.
Moraleva A., Magoulas C., Polzikov M. et al. Involvement of the specific nucleolar protein SURF6 in regulation of proliferation and ribosome biogenesis in mouse NIH/3T3 fibroblasts // Cell Cycle. 2017. V. 16. № 20. P. 1979–1991.
Nieto B., Gaspar S.G., Moriggi G. et al. Identification of distinct maturation steps involved in human 40S ribosomal subunit biosynthesis // Nat. Commun. 2020. V. 11. № 1. P. 156.
Núñez Villacís L., Wong M.S., Ferguson L.L. et al. New roles for the nucleolus in health and disease // BioEssays. 2018. V. 40. № 5. P. 8–10.
Oeffinger M., Fatica A., Rout M.P., Tollervey D. Yeast Rrp14p is required for ribosomal subunit synthesis and for correct positioning of the mitotic spindle during mitosis // Nucl. Acids Res. 2007a. V. 35. № 4. P. 1354–1366.
Oeffinger M., Wei K.E., Rogers R. et al. Comprehensive analysis of diverse ribonucleoprotein complexes // Nat. Meth. 2007b. V. 4. № 11. P. 951–956.
Orian A., van Steensel B., Delrow J. et al. Genomic binding by the drosophila Myc, Max, Mad/Mnt transcription factor network // Genes Dev. 2003. V. 17. № 9. P. 1101–1114.
Pereira G., Schiebel E. Kin4 kinase delays mitotic exit in response to spindle alignment defects // Mol. Cell. 2005. V. 19. № 2. P. 209–221.
Piano F., Schetter A.J., Morton D.G. et al. Gene clustering based on RNAi phenotypes of ovary-enriched genes in C. elegans // Curr. Biol. 2002. V. 12. № 22. P. 1959–1964.
Planta R.J. Regulation of ribosome synthesis in yeast // Yeast. 1997. V. 13. № 16. P. 1505–1518.
Polzikov M., Zatsepina O., Magoulas C. Identification of an evolutionary conserved SURF-6 domain in a family of nucleolar proteins extending from human to yeast // Biochem. Biophys. Res. Commun. 2005. V. 327. № 1. P. 143–149.
Polzikov M., Magoulas C., Zatsepina O. The nucleolar protein SURF-6 is essential for viability in mouse NIH/3T3 cells // Mol. Biol. Rep. 2007. V. 34. № 3. P. 155–160.
Polzikov M.A., Kordyukova M.Y., Zavalishina L.E. et al. Development of novel mouse hybridomas producing monoclonal antibodies specific to human and mouse nucleolar protein SURF-6 // Hybridoma. 2012. V. 31. № 1. P. 48–53.
Queralt E., Uhlmann F. Separase cooperates with Zds1 and Zds2 to activate Cdc14 phosphatase in early anaphase // J. Cell Biol. 2008. V. 182. № 5. P. 873–883.
Ringwald M., Wu C., Su A.I. BioGPS and GXD: mouse gene expression data – the benefits and challenges of data integration // Mamm. Genome. 2012. V. 23. № 9–10. P. 550–558.
Romanova L.G., Anger M., Zatsepina O.V., Schultz R.M. Implication of nucleolar protein SURF6 in ribosome biogenesis and preimplantation mouse development // Biol. Reprod. 2006. V. 75. № 5. P. 690–696.
Sanghai Z.A., Miller L., Molloy K.R. et al. Modular assembly of the nucleolar pre-60S ribosomal subunit // Nature. 2018. V. 556. № 7699. P. 126–129.
Sato S., Tomomori-Sato C., Banks C.A.S. et al. Identification of mammalian mediator subunits with similarities to yeast mediator subunits Srb5, Srb6, Med11, and Rox3 // J. Biol. Chem. 2003.V. 278. № 17. P. 15123–15127.
Saveanu C., Rousselle J.-C., Lenormand P. et al. The p21-activated protein kinase inhibitor Skb15 and its budding yeast homologue are 60S ribosome assembly factors // Mol. Cell. Biol. 2007. V. 27. № 8. P. 2897–2909.
Savino T.M., Bastos R., Jansen E., Hernandez-Verdun D. The nucleolar antigen Nop52, the human homologue of the yeast ribosomal RNA processing RRP1, is recruited at late stages of nucleologenesis // J. Cell Sci. 1999. V. 112. Pt. 12. P. 1889–1900.
Schlosser I., Hölzel M., Mürnseer M. et al. A role for c-Myc in the regulation of ribosomal RNA processing // Nucl. Acids Res. 2003. V. 31. № 21. P. 6148–6156.
Seto E., Lewis B., Shenk T. Interaction between transcription factors Sp1 and YY1 // Nature. 1993. V. 365. № 6445. P. 462–464.
Smetana K. Structural features of nucleoli in blood, leukemic, lymphoma and myeloma cells // Eur. J. Histochem. 2002. V. 46. № 2. P. 125–132.
Smith D., Gray J., Mitchell L. et al. Assembly of cytochrome-c oxidase in the absence of assembly protein Surf1p leads to loss of the active site heme // J. Biol. Chem. 2005. V. 280. № 18. P. 17652–17656.
Stine Z.E., Walton Z.E., Altman B.J. et al. MYC, metabolism, and cancer // Canc. Discov. 2015. V. 5. № 10. P. 1024–1039.
Tafforeau L., Zorbas C., Langhendries J.-L. et al. The complexity of human ribosome biogenesis revealed by systematic nucleolar screening of pre-rRNA processing factors // Mol. Cell. 2013. V. 51. № 4. P. 539–551.
Trott R.L., Kalive M., Karandikar U. et al. Identification and characterization of proteins that interact with Drosophila melanogaster protein kinase CK2 // Mol. Cell. Biochem. 2001. V. 227. № 1–2. P. 91–98.
Turner A.J., Knox A.A., Prieto J.-L. et al. A novel small-subunit processome assembly intermediate that contains the U3 snoRNP, nucleolin, RRP5, and DBP4 // Mol. Cell. Biol. 2009. V. 29. № 11. P. 3007–3017.
Uetz P., Giot L., Cagney G. et al. A comprehensive analysis of protein–protein interactions in Saccharomyces cerevisiae // Nature. 2000. V. 403. № 6770. P. 623–627.
Urbinati C.R., Gonsalvez G.B., Aris J.P., Long R.M. Loc1p is required for efficient assembly and nuclear export of the 60S ribosomal subunit // Mol. Genet. Genom. 2006. V. 276. № 4. P. 369–377.
Wan D., Gong Y., Qin W. et al. Large-scale cDNA transfection screening for genes related to cancer development and progression // PNAS USA. 2004. V. 101. № 44. P. 15724–15729.
Wolff C.-M., Nguyen V.K., Remy P. Cloning and expression of the surfeit locus member Surf-6 during embryogenesis in Xenopus laevis // DNA Seq. 2002. V. 13. № 3. P. 149–152.
Woolford J.L., Baserga S.J. Ribosome biogenesis in the yeast Saccharomyces cerevisiae // Genetics. 2013. V. 195. № 3. P. 643–681.
Wu K., Wu P., Aris J.P. Nucleolar protein Nop12p participates in synthesis of 25S rRNA in Saccharomyces cerevisiae // Nucl. Acids Res. 2001. V. 29. № 14. P. 2938–2949.
Xia Z.B., Dai M.S., Magoulas C. et al. Differentially expressed genes during in vitro differentiation of murine embryonic stem cells transduced with a human erythropoietin receptor cDNA // J. Hematother. Stem Cell Res. 2000. V. 9. № 5. P. 651–658.
Yakura M., Ozoe F., Ishida H. et al. Zds1, a novel gene encoding an ortholog of Zds1 and Zds2, controls sexual differentiation, cell wall integrity and cell morphology in fission yeast // Genetics. 2006. V. 172. № 2. P. 811–825.
Yamada H., Horigome C., Okada T. et al. Yeast Rrp14p is a nucleolar protein involved in both ribosome biogenesis and cell polarity // RNA. 2007. V. 13. № 11. P. 1977–1987.
Yin Y., Garcia M.R., Novak A.J. et al. Surf4 (Erv29p) binds amino-terminal tripeptide motifs of soluble cargo proteins with different affinities, enabling prioritization of their exit from the endoplasmic reticulum // PLoS Biol. 2018. V. 16. № 8. P. e2005140.
Yoshikawa H., Ishikawa H., Izumikawa K. et al. Human nucleolar protein Nop52 (RRP1/NNP-1) is involved in site 2 cleavage in internal transcribed spacer 1 of pre-rRNAs at early stages of ribosome biogenesis // Nucl. Acids Res. 2015. V. 43. № 11. P. 5524–5536.
Zahner J.E., Harkins H.A., Pringle J.R. Genetic analysis of the bipolar pattern of bud site selection in the yeast Saccharomyces cerevisiae // Mol. Cell. Biol. 1996. V. 16. № 4. P. 1857–1870.
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии


