Успехи физиологических наук, 2020, T. 51, № 1, стр. 58-71
Генетические аспекты фМРТ исследований головного мозга
П. Д. Лисачев a, М. Е. Мельников b, c, М. Б. Штарк b, c, *
a Институт вычислительных технологий СО РАН
630090 Новосибирск, Россия
b Федеральное государственное бюджетное научное учреждение “Федеральный исследовательский центр фундаментальной и трансляционной медицины.
Институт молекулярной биологии и биофизики” Министерства науки
и высшего образования Российской Федерации
630117 Новосибирск, Россия
c Федеральное государственное автономное образовательное учреждение высшего образования
“Новосибирский национальный исследовательский государственный университет”
630090 Новосибирск, Россия
* E-mail: mark@niimbb.ru
Поступила в редакцию 01.07.2019
После доработки 20.08.2019
Принята к публикации 05.10.2019
Аннотация
Функциональная магнитнорезонансная томография (фМРТ) предоставляет уникальную возможность неинвазивной динамической оценки активности мозговых структур с высоким пространственным разрешением. Интеграция нейровизуализационных данных и генетической информации позволяет глубже понять принципы работы мозга. В обзоре представлены недавние работы, посвященные изучению генетических основ организации мозга и механизмов нейропсихических заболеваний с использованием фМРТ. В частности, рассмотрены ассоциации транскриптома со структурным коннектомом у животных и человека. Показано, что архитектура глобальных сетей в существенной степени является наследуемой. Охарактеризовано влияние генов, детерминирующих нейрофизиологические процессы на клеточном и субклеточном уровнях, на параметры глобальной активности мозга, включая структуру сетей покоя.
ВВЕДЕНИЕ
Появление в арсенале исследователей метода функциональной магнитнорезонансной томографии в значительной мере способствовало прогрессу в изучении функциональной организации мозга человека за последние 25 лет. С принципами метода, его преимуществами и ограничениями можно ознакомиться во многих обзорах (см., например, [4]). Вкратце, фМРТ обеспечивает неинвазивную динамическую оценку активности мозговых структур на основе анализа локальной гемодинамики, зависимой от уровня оксигенации крови, – сигнала BOLD (blood oxygenation level dependent). ФМРТ обладает относительно высоким пространственным разрешением (по сравнению с ЭЭГ) и позволяет оценивать локализацию церебрального ответа с точностью до нескольких миллиметров.
Несмотря на большую популярность активационных исследований, очерчивающих топографию ответа головного мозга на определенные стимулы, известны и другие приложения метода фМРТ. В первую очередь, речь идет об изучении функциональных связей между различными регионами мозга как в состоянии покоя, так и при выполнении испытуемым тех или иных задач (например, [88]). Специальные статистические техники, такие, как причинность по Грэнджеру (GCA), позволяют грубо оценить направление этих связей, то есть последовательность вовлечения различных мозговых звеньев в ту или иную деятельность. Для широкого ознакомления с этой темой мы рекомендуем читателю классический обзор [41]. Важно заметить, что разрешение, предлагаемое фМРТ, является в этом случае даже несколько избыточным, поскольку головной мозг человека может быть достаточно надежно сведен к конечному числу относительно функционально независимых субъединиц [23].
Анализ записей фМРТ в состоянии покоя позволил выявить регионы мозга, активность которых изменялась достаточно согласованно, и объединить их на этом основании в крупные системы, обозначаемые как функциональные сети (FCN, functional connectivity network). В основном, математический аппарат выделения сетей сводится к анализу независимых или главных компонент. В большинстве исследований идентифицируется от 10 до 20 таких компонент, для каждой из которых описано функциональное значение (см., например, [18, 40, 63]). В работе Buckholtz, Meyer-Lindenberg [19] охарактеризован конкретный вклад четырех крупнейших сетей мозга в регуляцию конкретных классов когнитивных процессов и в возникновение отдельных симптомов психических расстройств вкупе с генетическими факторами, влияющими на активность данных сетей (рис. 1).
Рис. 1.
Влияние генов DRD2, MAOA и ZNF804A на функциональные сети мозга. Сверху приведены названия сетей, в скобках указаны гены, полиморфизм которых ассоциируется с изменениями активности данной сети. Ниже – схема, изображающая основные узлы сети и связи между ними. Узлы: 1 – передняя поясная извилина, 2 – латеральная префронтальная кора, 3 – внутритеменная борозда, 4 – медиальная префронтальная кора, 5 – предклинье, 6 – височно-теменное сочленение, 7 – орбитофронтальная кора, 8 – миндалевидный комплекс, 9 – полосатое тело. Ниже – базовые когнитивные функции, реализуемые при участии сети. Внизу – психопатологические симптомы, ассоциированные с нарушением функционирования сети. Адаптировано из [19].
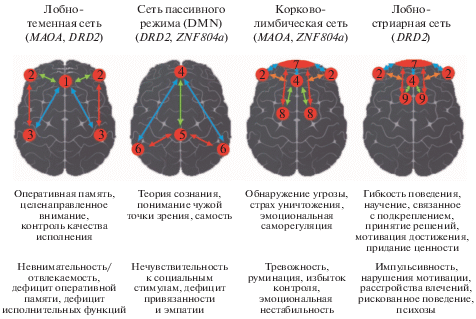
Отдельно взятая сеть может быть представлена как топографически, в виде трехмерной карты, наложенной на анатомический снимок мозга, так и схематически как система участков мозга, объединенных связями определенной силы, знака и направленности. Для последнего варианта крайне органичным решением является применение теории графов. При этом узлами графов становятся регионы, входящие в состав сети, а ребрами – связи между ними. Описание основных сетевых характеристик, таких как степень узла (node degree), сила связи (connection strength), хабы, влияния (authorities), центральность (centrality), взаимодействие (betweenness) представлено в обзорах [20, 82]. Также ознакомиться с использованием теории графов в нейровизуализации можно в работе Wang et al. [101], а применительно к ЭЭГ – Vecchio et al. [97]. Высокая информативность этих метрик подтверждается исследованиями, демонстрирующими перспективность использования сетевых характеристик в качестве эндофенотипов – количественных биологических маркеров генетического риска нейропсихических расстройств [11, 64, 71, 92].
Актуальные представления о структурно-функциональном устройстве головного мозга, его системной (сетевой) организации предполагают, что индивидуальные вариации в выраженности конкретных связей структур внутри сети и сетей между собой должны быть в существенной степени генетически детерминируемыми и наследуемыми. Формирование и поддержание индивидуальной картины особенностей гемодинамического ответа в ответ на стимулы и спонтанной гемодинамики в покое обеспечивается генетическими переменными, которые могут быть исследованы вкупе с собственно фМРТ-данными. На стыке нейровизуализационных и генетических исследований можно выделить небольшую область “визуализационной генетики” (imaging genetics), уже включающую в себя ощутимый корпус публикаций. Эти работы, в основном, реализуют преимущества интеграции структурных, функциональных и генетических данных для понимания принципов работы мозга и механизмов нейропсихических заболеваний [11, 44, 68, 87]. Количество исследований в этой области быстро растет [2, 21, 24, 33, 62, 68, 87]. В данной публикации, не претендуя на полноту изложения, мы представляем обзор работ, посвященных изучению генетических основ организации мозга визуализационными методами на примере фМРТ.
ВЗАИМООТНОШЕНИЯ ГЕННОЙ ЭКСПРЕСИИ И СТРУКТУРНЫХ СВЯЗЕЙ ВНУТРИ ГОЛОВНОГО МОЗГА
Хотя в общем случае согласованная активность двух областей мозга не означает наличия между ними прямых синаптических коммуникаций, в основе функциональной синхронии различных церебральных регионов, безусловно, лежит тот или иной тип структурных связей. Поэтому анализ генетических аспектов исследований функциональных сетей целесообразно предварить рассмотрением работ, в которых была сделана попытка выявить ассоциации транскриптома и анатомических связей нейронов и целых областей мозга. Базовый дизайн подобных экспериментов представлен на рисунке 2. Первые работы такого рода [10, 50, 95] были выполнены на нематоде Caenorhabditis elegans с использованием данных об экспрессии нескольких сотен генов в отдельных клетках и карты межнейронных связей [103]. Выбор объекта исследования был мотивирован небольшим (302) числом нейронов в нервной системе животного и принципиальной возможностью оценить все возможные связи между ними. Результаты этих работ позволили сделать заключение, что профили генной экспрессии нейронов несут значительную информацию об их связях [50], и в данном контексте продемонстрировали преимущество анализа наборов взаимодействующих генов по сравнению с изучением отдельных генов [10, 95].
Рис. 2.
Дизайн исследования связей между генной экспрессией и структурными или функциональными сетями мозга. Анализ начинается с изучения генной экспрессии (в случае человеческого мозга речь идет о посмертных образцах), структурных (например, с помощью диффузионной МРТ) и функциональных связей. Данные могут быть представлены в виде трехмерной анатомической карты, схематически как система участков мозга, объединенных связями определенной силы, знака и направленности (для коннектома) и, в конечном счете, в виде соответствующих матриц, предназначенных для дальнейшего математического анализа, например, исследования корреляции транскриптома и коннектома. В качестве “узлов” могут выступать как пространственные воксели, так и анатомические отделы мозга.

Опираясь на эти результаты, две группы авторов [39, 104] независимо друг от друга сопоставили коннектомы корковых и подкорковых областей крысы [16] и данные по экспрессии генов в мозге мыши [56] и пришли к выводу, что анатомически определяемые области мозга с близкими паттернами экспрессии генов имеют сходные профили связей, т.е. закономерность, выявленная в работах на нематоде на клеточном уровне, может быть обнаружена и на более высоком структурном уровне. Аналогичный результат был получен для мозга мыши с использованием промежуточного пространственного разрешения – 200 мкм [32]. При этом включение в модель всего 25 наиболее информативных генов позволяло получить предсказание коннектома участка мозга мыши с 91% точностью, оцененной как площадь под кривой ROC, характеризующей отношения чувствительности и избирательности [49].
Кроме того, отделы мозга со сходными транскриптомами с более высокой вероятностью оказываются связанными друг с другом [38, 42]. Исследования, проведенные как на грызунах, так и на человеке, показали, что области мозга, функционально связанные между собой в рамках одной функциональной сети, также демонстрируют значительное сходство транскриптомов [6, 46, 54, 65, 79, 100].
Отчасти это можно объяснить тем, что пространственно близкие нейроны и области мозга, с одной стороны, имеют относительно высокую вероятность формирования связей [9, 17, 48, 85], а с другой – часто имеют сходные профили генетической экспрессии, что может быть связано, например, с их общим эмбриональным происхождением [12, 72, 105]. Так, Pantazatos, Li [70] убедительно продемонстрировали, что в основе результатов Richiardi et al. [79] лежит именно пространственная автокорреляция генетической экспрессии, и, следовательно, данную работу нельзя рассматривать в качестве доказательства специфической корреляции генетической экспрессии в пределах функциональных сетей. Pantazatos, Li [70] также указывают на несовершенство модели линейной регрессии, использованной French, Pavlidis [38] для коррекции эффекта пространственной близости. Наилучшим решением этой проблемы авторы считают исключение близко расположенных участков (менее 3 см для человеческого мозга) из корреляционного анализа.
Другое возможное объяснение ассоциаций между профилями транскриптома и структурно-функциональных связей может заключаться в следующем. Как известно, даже кратковременная активность нейронов порождает в них самих и окружающих глиальных клетках волны возмущения генетической экспрессии, длящиеся много часов и даже дней [1, 5, 59]. Поэтому ожидаемо, что регулярная синхронная активность функционально связанных областей приводит к тому, что их транскриптомы становятся более похожими между собой и отличными от транскриптомов областей, работающих в другом режиме. В частности, к генам, чувствительным к режиму работы нейронов, относятся регуляторы окислительного метаболизма, что объясняет их высокий уровень экспрессии в хабах – областях мозга, формирующих большое количество связей с другими структурами [42, 96]. Хабы характеризуются также высоким уровнем экспрессии генов, важных для когнитивных функций [83] и процессов межнейронной коммуникации, таких как глутамат- и ГАМК-эргическая передача [8].
Транскриптом участка мозга отражает также его специфический клеточный состав (соотношение нейронов, глии и “транзитных” аксонов), который также влияет на вероятность формирования связей этого участка с другими [102]. Наконец, еще одним фактором, повышающим биохимическое сходство связанных нейронов, может быть зависимое от нейронной активности прионоподобное распространение макромолекул [37].
ГЕНЕТИЧЕСКИЕ ОСНОВЫ ФОРМИРОВАНИЯ ФУНКЦИОНАЛЬНЫХ СЕТЕЙ
Влияние генов на структурно-функциональную организацию мозга представляет существенный интерес как для решения фундаментальных вопросов нейронауки, так и в связи с поиском маркеров генетического риска психических и неврологических заболеваний, поскольку многие из них характеризуются отклонениями от нормы в сетевой архитектуре мозга [25, 64, 67, 71, 92, 94, 101].
Важным признаком существенного влияния генотипа на характеристики функциональных сетей является их наследуемость. Наследуемость сетевых параметров мозга у человека может быть оценена близнецовым методом [22, 35, 86, 89, 90, 107]. Так, исследование 574 одно- и разнояйцевых близнецов и одиночных сиблингов близнецовых пар показало, что классические метрики “тесного мира” (small world) функциональных сетей покоя, полученные из данных ЭЭГ, имеют высокую наследуемость [90]. В другом исследовании характеристик анатомических сетей, выявленных на основе данных МРТ коры мозга 308 близнецов, 64 одиночных сиблингов близнецов и 228 одиночных детей также обнаружены генетические детерминанты характеристик “тесного мира” лобно-теменных и затылочных сетей [86]. Glahn et al. [45] использовали другой подход: с помощью фМРТ авторы исследовали сеть пассивного режима работы мозга (DMN, default mode network) у 333 испытуемых из больших семей (от 5 до 32 человек) и также продемонстрировали высокую наследуемость параметров этой сети. Miranda-Dominguez et al. [66] исследовали наследуемость сетей покоя, используя алгоритм, основанный на машинном обучении, и показали, в частности, что индивидуальный “коннектотип” может быть идентифицирован через несколько лет после первой записи.
Некоторые из генов, экспрессия которых демонстрирует ковариацию в удаленных участках функциональных сетей, широко распределенных в пределах коры, по-видимому, отвечают за формирование и поддержку функционирования длинных аксонов, поскольку их экспрессия в мозге приматов выше, чем у грызунов [54].
Diez, Sepulcre [26] исследовали динамические перестройки сетей покоя для выявления рекуррентной (аттрактор-подобной) нейронной активности. С целью повышения временного разрешения BOLD-сигнала вместо оценки связей по всей длине фМРТ-записи применялся метод скользящего окна. Оказалось, что для областей с рекуррентной активностью, к которым относится, в частности, DMN, характерна повышенная экспрессия генов, связанных с синаптической пластичностью (долговременной потенциацией и депрессией).
Richiardi et al. [79] продемонстрировали влияние генетического полиморфизма на структуру связей между узлами внутри сетей покоя у человека. Их классификация на основе предложенной авторами интегральной оценки эффекта полиморфизма 136 генов продемонстрировала значительные различия сетевого фенотипа в группах испытуемых с разными значениями эффекта полиморфизма. Pantazatos, Li [70] критикуют методику идентификации группы генов и указывают на то, что авторы не провели статистическое исследование, доказывающее, что полиморфизм любой другой группы не будет демонстрировать сходных результатов. Однако авторы комментария констатируют, что рассматриваемая группа генов оказалась на 75% состоящей из “дифференциально стабильных” генов [46], которые характеризуются локально высоким и неравномерно распределенным в коре мозга уровнем экспрессии и с высокой вероятностью являются функционально важными [70]. Выявленные Richiardi et al. [79] гены связаны с активностью ионных каналов, нейромедиаторами и синаптической функцией, что предполагает ассоциации между функциональными связями внутри мозга и сложными синаптическими механизмами. Интересно, что идентифицированная группа генов характеризуется значимыми связями с вероятностью возникновения девяти заболеваний, включая болезнь Альцгеймера и шизофрению.
Программный комплекс NeuroSynth [108] позволяет на основе сопоставления карт экспрессии гена с данными из обширной базы фМРТ-исследований формулировать гипотезы о его вовлечении в функции мозга. Fox et al. [36] провели анализ связи экспрессии генов в участках мозга человека, для которых на основе данных фМРТ было продемонстрировано вовлечение в 48 категорий когнитивных процессов. Многие из полученных корреляций воспроизвели уже известные ассоциации генов с когнитивными функциями, что свидетельствует о правомерности применения такого подхода, как минимум, для независимого подтверждения уже имеющихся выводов о функциях генов. Кроме того, были выявлены ранее неизвестные связи, что демонстрирует возможность использования подобного рода анализа для генерации перспективных гипотез и их дальнейшего более детального исследования. Quintana et al. [77] с помощью NeuroSynth изучили корреляцию пространственного распределения экспрессии генов, отвечающих за синтез, секрецию и рецепцию окситоцина, с данными фМРТ и продемонстрировали связь оцситоциновой сигнальной системы с антиципацией, удовлетворением потребностей и избеганием угрожающих стимулов.
ФУНКЦИОНАЛЬНЫЕ СЕТИ МОЗГА И ФАКТОРЫ РИСКА ПСИХИЧЕСКИХ ЗАБОЛЕВАНИЙ
Важным источником информации о роли конкретных генов в формировании сетевой архитектуры мозга являются нейровизуализационные работы на материале пациентов с генетическими особенностями в рамках так называемых “сетевых болезней” и генетически модифицированных модельных животных. Например, Liégeois et al. [57, 58] исследовали с помощью фМРТ пациентов с нарушениями речи, связанными с мутацией гена транскрипционного фактора FOXP2, и выявили изменения их мозговой активности, по сравнению со здоровыми родственниками, при произнесении слов. В другой работе показано, что у пациентов, страдающих рецидивирующим рассеянным склерозом, гомозиготный аллель rs6795970A гена SCN10A ассоциируется с нарушением функциональных связей мозжечка с другими отделами мозга и более тяжелым течением болезни [81]. Ген SCN10A кодирует один из типов натриевых каналов (Nav1.8), эктопически экспрессирующийся в клетках Пуркинье мозжечка при рассеянном склерозе. Еще в одном исследовании рассмотрено влияние на сетевую организацию мозга гена, кодирующего GPR88 (G protein-coupled receptor 88), ассоциированного с психическими расстройствами. У мышей-нокаутов по гену Gpr88 наблюдались значительные изменения коннектома и структуры функциональных связей, сопровождавшиеся нарушениями поведения – гиперактивностью и дефицитом обучения [7].
Интерпретация данных о влиянии генетических маркеров психических расстройств на активность мозга у пациентов затруднена, поскольку сложно отделить непосредственные эффекты генетического нарушения от вторичных патологических изменений, связанных, например, с дегенерацией церебральной ткани. Однако в некоторых случаях исследования функциональных сетей позволяют выявить влияние генетических факторов риска когнитивных нарушений на работу мозга у здоровых носителей неблагоприятных аллелей, что, с одной стороны, позволяет приблизиться к пониманию механизма патогенеза, а с другой – может оказаться полезным для ранней диагностики и разработки средств предупреждения заболевания. Так, в ряде работ показана связь нескольких генетических маркеров риска развития болезни Альцгеймера (варианты генов APOE, PICALM, CLU и BIN1) с изменениями сетей покоя у здоровых испытуемых (см. обзоры [21, 74]). К наиболее типичным изменениям можно отнести снижение силы функциональных связей в средней и затылочной областях DMN, включая заднюю поясную извилину и предклинье, и повышенную связность передней части DMN, включая среднюю височную и префронтальную кору [21].
Val66Met полиморфизм мозгового нейротрофического фактора, ассоциированный с развитием различных когнитивных отклонений, в частности, ускоренным развитием деменции у пожилых людей, также отражается на характеристиках функциональных связей в мозге здоровых людей, что было показано с помощью фМРТ [99, 109] и магнитоэнцефалографии [80]. Kepa et al. [51] выявили локусы однонуклеотидного полиморфизма (SNP, single nucleotide polymorphism), связанные со сниженной экспрессией транскрипционного фактора MYT1L, играющего важную роль в нейрогенезе. У носителей более редкого T-аллеля rs17338519 наблюдалось снижение объема правого гиппокампа и увеличение его активности в задаче извлечения эпизодической памяти, по сравнению с GG-гомозиготами. В таблице 1 приведены дополнительные примеры работ, демонстрирующих ассоциацию некоторых риск-аллелей с мозговой активностью у здоровых людей.
Таблица 1.
Ассоциации генетических факторов риска болезней мозга с характеристиками мозговой активности у здоровых испытуемых
| Генетический полиморфизм | Кодируемый белок | Экспериментальная парадигма | BOLD ответ, параметры которого ассоциированы с полиморфизмом (связь = функциональная связь) | Ссылки |
|---|---|---|---|---|
| ANK3 rs10994336 | Анкирин 3 | Распознавание эмоций по лицу | Активация сети распознавания эмоционального выражения лица, связь между зрительной и вентральной префронтальной корой | [27] |
| APOE ε2, ε3, ε4 |
Аполипопротеин E | Состояние покоя | Связи дорзолатеральной префронтальной коры | [106] |
| BDNF Val66Met |
Мозговой нейротрофический фактор | Состояние покоя | Связь между дорзолатеральной префронтальной и передней островковой корой | [99] |
| Связь гиппокампа с мозжечком | [109] | |||
| CACNA1C rs1006737 | Субъединица α1С кальциевого канала L-типа | Распознавание эмоций по лицу | Активация сети распознавания эмоционального выражения лица, связь между зрительной и вентральной префронтальной корой | [27] |
| Ассоциативная эпизодическая память | Активация гиппокампа, передней поясной коры и дорзолатеральной префронтальной коры; связь между левым и правым гиппокампом | [29, 30] | ||
| Формирование и извлечение следов эпизодической памяти | Активация правого гиппокампа | [55] | ||
| MAD1L1 rs11764590 | Белок контрольной точки сборки митотического веретена MAD1L1 | Потребностно-мотивационная сфера (контроль реакций, связанных с немедленным получением вознаграждения) | Активация вентральной области покрышки, стриатума, лобной и теменной коры; связь между префронтальными областями и вентральным стриатумом | [93] |
| MYT1L rs17338519 |
Транскрипционный фактор MYT1L | Извлечение следов эпизодической памяти | Активация правого гиппокампа | [51] |
| NCAN rs1064395 | Хондроитинсульфат протеогликан нейрокан | Словарный запас (порождение слов требуемой категории) | Инактивация большого левого латерального височного кластера: от средней височной извилины до височного полюса | [78] |
| SLC6A4 L/S |
Транспортер серотонина | Сложная зрительно-моторная реакция (Go/No-Go) | Активация клина, медиальной и нижней лобных извилин | [3] |
| S100A10 rs72993629 | Кальций-связывающий белок S100A10 | Состояние покоя | Амплитуда низкочастотных флуктуаций в левой парацентральной дольке и правом бледном шаре | [75] |
| S100B rs11542311 |
Кальций-связывающий белок S100B | Распознавание сцен, связанных с пространственной навигацией | Активация правой ретроспленальной коры | [53] |
| S100B rs3788266 |
Активация правой ретроспленальной коры и левой парагиппокампальной навигационной зоны | |||
| WWC1/KIBRA rs17070145 |
WW- и C2-домен содержащий белок 1 | Состояние покоя | Связи дорзолатеральной префронтальной коры | [106] |
| ZNF804A rs1344706 | Цинк-пальцевый белок 804A | Задача на оперативную память (N-back) | Связи в пределах дорзолатеральной префронтальной коры, связи дорзолатеральной префронтальной коры с гиппокампом | [31] |
| Заспознавание эмоций по лицу | Связи миндалины |
Эффекты генетического полиморфизма не всегда воспроизводятся в разных популяциях. Одной из причин могут быть ложно-положительные или ложно-отрицательные статистические оценки результатов исследования [68]. Для повышения надежности выводов иногда прибегают к репликации исследования на независимой группе испытуемых. Другая причина – влияние генетического фона или среды на эффект конкретного гена [76]. Так, была выявлена связь полиморфизма rs72993629 в регуляторной области кластера генов S100A10/S100A11 с рискованным половым поведением у афроамериканцев, причем знак связи определялся наличием или отсутствием зависимости испытуемого от каннабиса, тогда как у американцев-европеоидов такой ассоциации обнаружено не было [75]. В той же группе, состоявшей из 3350 человек, у женщин обеих рас, в отличие от мужчин, аналогичным эффектом обладал полиморфизм гена CLTC rs12944716. При этом фМРТ-исследование в независимой группе из 24-х афроамериканцев (с неизвестными параметрами наркотической зависимости и сексуального поведения) показало влияние rs72993629 на активность структур мозга, связанных с половой функцией и поведением, – левой парацентральной дольки и правого бледного шара.
Другой пример зависимости эффекта гена от фона – взаимодействие APOE и KIBRA (WWC1). У KIBRA (rs17070145) TT-гомозигот наблюдается зависимое от полиморфизма APOE уменьшение (ε2 > ε3 > ε4) плотности функциональных связей (functional connectivity density) дорзолатеральной префронтальной коры, тогда как у носителей KIBRA C-аллеля – увеличение (ε2 < ε3 < ε4) [106].
АНАЛИЗ ВАРИАБЕЛЬНОСТИ КОГНИТИВНЫХ ФУНКЦИЙ
Генотип оказывает существенное влияние на когнитивные способности [61, 69, 98]. Современные методы нейровизуализации достаточно чувствительны, чтобы выявить не только потенциально патологические изменения в мозге, но и различия в нейросетевой архитектуре, связанные с успешностью решения когнитивных задач здоровыми людьми [28, 52, 60, 91]. Один из подходов к поиску причин индивидуальных различий заключается в изучении эффектов генетического полиморфизма. Разумеется, в отсутствие детального генотипирования испытуемых и с учетом неполноты имеющейся информации об эффектах генетического полиморфизма, невозможно провести четкую грань между этими работами и упомянутыми выше исследованиями риск-аллелей у здоровых волонтеров.
Berry et al. [14] обнаружили, что люди с генетическим полиморфизмом, ограничивающим активность транспортера холина (Ile89Val вариант гена SLC5A7, rs1013940), более уязвимы для отвлекающих воздействий. Оказалось, что этот эффект связан с отсутствием активации правой префронтальной коры в условиях, требующих повышенного внимания [13]. Кроме того, указанный полиморфизм отражался в активности орбитофронтальной коры и парагиппокампальной извилины при решении задач, связанных с поддержанием концентрации внимания.
Sadaghiani et al. [84] исследовали полиморфизм rs1044396 гена CHRNA4, кодирующего субъединицу никотинового рецептора, на нейронную активность в цингуло-оперкулярной сети, вовлеченной в базовые процессы когнитивного контроля, включая поддержание бдительности. Все области сети показали более высокую активность у гетерозигот по сравнению с обоими типами гомозигот во время когнитивной нагрузки. Кроме того, гетерозиготы более точно выполняли поведенческие задачи, которые в первую очередь зависели от постоянной концентрации.
Berto et al. [15], используя интракраниальную ЭЭГ, идентифицировали 163 гена, дифференциально экспрессирующихся в областях мозга, активность которых коррелировала с успешностью запоминания слов у пациентов, страдающих ненаследственной эпилепсией. Представленный авторами список частично перекрывается с генами, экспрессия которых коррелирует с деятельностью сетей покоя [79, 100].
Pergola et al. [73] идентифицировали в гене глутаматного рецептора GluN2B пять локусов SNP, комбинация которых позволяла надежно предсказывать уровень экспрессии гена в префронтальной коре в посмертных образцах, и показали связь предсказанной таким образом экспрессии GluN2B с активностью префронтальной коры и функционированием рабочей памяти у здоровых испытуемых. В другой работе было показано, что на основе анализа 13-ти SNPs в наборе генов, коэкспрессирующихся с геном дофаминового рецептора D1 (DRD1), можно предсказать эффективность рабочей памяти и соответствующую активность префронтальной коры у здоровых индивидов [34]. Авторы идентифицировали локусы полиморфизма DRD1, ассоциированные с уровнем его экспрессии. Интересно, что ассоциированный с экспрессией полиморфизм одного DRD1 недостаточно надежно коррелировал с исследованными параметрами, несмотря на известное значительное влияние этого рецептора на рабочую память и активность префронтальной коры. Это еще раз демонстрирует более высокую информативность изучения ассоциаций фенотипа с большим набором генетических маркеров, о чем уже упоминалось выше и что было показано многими авторами [10, 43, 47, 73, 95].
ЗАКЛЮЧЕНИЕ
1. Визуализационная генетика – относительно новая и быстро развивающаяся область, в рамках которой исследуются генетические факторы, формирующие фенотип мозга. Влияние генома на структурно-функциональную организацию мозга представляет существенный интерес для решения широкого круга вопросов нейронауки, включая поиск генетических факторов риска нейропсихических заболеваний, поскольку многие психические заболевания связаны с нарушением сетевой архитектуры мозга.
2. У лабораторных животных и у людей продемонстрировано сходство транскриптомов областей мозга, структурно связанных между собой. Этот феномен может быть следствием пространственных автокорреляций, или “синхронизации” генетической экспрессии церебральных структур, работающих сопряженно, или гистологического сходства (р.п.) регионов, принадлежащих к одной сети, но может отражать и специфичные закономерности.
3. Функциональные связи внутри сетей покоя человеческого мозга и между ними достаточно индивидуальны и стабильны. Получены убедительные данные в пользу наследуемости характеристик связей внутри сети пассивного режима работы мозга (DMN) и других глобальных церебральных сетей.
4. Могут быть выделены конкретные гены, наиболее сильно связанные с характеристиками внутри- и межсетевых функциональных связей. Эти гены преимущественно кодируют белки, вовлеченные в синаптическую функцию, включая синтез нейромедиаторов, и работу ионных каналов. Полиморфизм этих генов часто ассоциируется с риском психических и неврологических расстройств.
5. Генетические вариации, ассоциированные с повышенным риском развития болезни Альцгеймера и других деменций, рассеянного склероза, шизофрении, расстройств поведения и влечений, речевых нарушений, отражаются также в изменении церебральных ответов испытуемых в фМРТ-пробах, модификации функциональных связей, включая нарушение архитектуры глобальных сетей, в особенности, сети пассивного режима работы мозга (DMN).
6. Генотипические детерминанты функционирования нейромедиаторных систем (холин-, глутамат-, серотонин- и дофаминэргической) проявляются в аномалиях функционирования мозга при выполнении когнитивных задач и могут быть предикторами уровня памяти и внимания у здоровых людей.
Список литературы
Анохин К.В. Молекулярная генетика памяти: когнитивная регуляция экспрессии генов в мозге при обучении // В сб.: Успехи функциональной нейрохимии. Санкт-Петербург: Издательство Санкт-Петербургского государственного университета, 2003. С. 33–45.
Малых С.Б. Генетически информативные исследования нейрофизиологических характеристик // В кн.: Геномика поведения: детское развитие и образование / Под ред. Малых С.Б., Ковас Ю.В., Гайсиной Д.А. – Томск: Издательский Дом Томского государственного университета, 2016. С. 332–381.
Петровский Е.Д., Савостьянов А.Н., Савелов А.А. и др. Влияние полиморфизма аллелей серотонинового транспортера на индивидуальные особенности мозговой гемодинамики у людей в условиях экспериментальной парадигмы “стоп–сигнал” // Вавиловский журнал генетики и селекции. 2014. Т. 18. № 4/3. С. 1281–1288.
Штарк М.Б., Коростышевская А.М., Резакова М.В., Савелов А.А. Функциональная магнитно-резонансная томография и нейронауки // Усп. физиол. наук. 2012. Т. 43. № 1. С. 3–29.
Alberini C.M., Kandel E.R. The regulation of transcription in memory consolidation // Cold Spring Harb. Perspect. Biol. 2015. V. 7. № 1. Article № a021741. https://doi.org/10.1101/cshperspect.a021741
Anderson K.M., Krienen F.M., Choi E.Y. et al. Gene expression links functional networks across cortex and striatum // Nat. Commun. 2018. V. 9. № 1. Article № 1428. https://doi.org/10.1038/s41467-018-03811-x
Arefin T.M., Mechling A.E., Meirsman A.C. et al. Remodeling of sensorimotor brain connectivity in Gpr88-deficient mice // Brain Connect. 2017. V. 7. № 8. P. 526–540. https://doi.org/10.1089/brain.2017.0486
Arnatkevi$\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\smile}$}}{c} $iūtė A., Fulcher B.D., Pocock R., Fornito A. Hub connectivity, neuronal diversity, and gene expression in the Caenorhabditis elegans connectome // PLoS Comput. Biol. 2018. V. 14. № 2. Article № e1005989. https://doi.org/10.1371/journal.pcbi.1005989
Averbeck B.B., Seo M. The statistical neuroanatomy of frontal networks in the macaque // PLoS Comput. Biol. 2008. V. 4. № 4. Article № e1000050. https://doi.org/10.1371/journal.pcbi.1000050
Baruch L., Itzkovitz S., Golan-Mashiach M. et al. Using expression profiles of Caenorhabditis elegans neurons to identify genes that mediate synaptic connectivity // PLoS Comput. Biol. 2008. V. 4. № 7. Article № e1000120. https://doi.org/10.1371/journal.pcbi.1000120
Bassett D.S., Sporns O. Network neuroscience // Nat. Neurosci. 2017. V. 20. № 3. P. 353–364. https://doi.org/10.1038/nn.4502
Bernard A., Lubbers L.S., Tanis K.Q. et al. Transcriptional architecture of the primate neocortex // Neuron. 2012. V. 73. № 6. P. 1083–1099. https://doi.org/10.1016/j.neuron.2012.03.002
Berry A.S., Blakely R.D., Sarter M., Lustig C. Cholinergic capacity mediates prefrontal engagement during challenges to attention: evidence from imaging genetics // Neuroimage. 2015. V. 108. P. 386–395. https://doi.org/10.1016/j.neuroimage.2014.12.036
Berry A.S., Demeter E., Sabhapathy S. et al. Disposed to distraction: genetic variation in the cholinergic system influences distractibility but not time-on-task effects // J. Cogn. Neurosci. 2014. V. 26. № 9. P. 1981–1991. https://doi.org/10.1162/jocn_a_00607
Berto S., Wang G.Z., Germi J. et al. Human genomic signatures of brain oscillations during memory encoding // Cereb. Cortex. 2018. V. 28. № 5. P. 1733–1748. https://doi.org/10.1093/cercor/bhx083
Bota M., Dong H.W., Swanson L.W. Brain architecture management system // Neuroinformatics. 2005. V. 3. № 1. P. 15–48. https://doi.org/10.1385/NI:3:1:015
Braitenberg V., Schüz A. Cortex: Statistics and geometry of neuronal connectivity. Berlin: Springer, 1998. 249 p. https://doi.org/10.1007/978-3-662-03733-1
Bressler S.L. Large-scale cortical networks and cognition // Brain Res. Brain Res. Rev. 1995. V. 20. № 3. P. 288–304. https://doi.org/10.1016/0165-0173(94)00016-I
Buckholtz J.W., Meyer-Lindenberg A. Psychopathology and the human connectome: toward a transdiagnostic model of risk for mental illness // Neuron. 2012. V. 74. № 6. P. 990–1004. https://doi.org/10.1016/j.neuron.2012.06.002
Bullmore E., Sporns O. Complex brain networks: graph theoretical analysis of structural and functional systems // Nat. Rev. Neurosci. 2009. V. 10. № 3. P. 186–198. https://doi.org/10.1038/nrn2575
Chiesa P.A., Cavedo E., Lista S. et al. Revolution of resting-state functional neuroimaging genetics in Alzheimer’s disease // Trends Neurosci. 2017. V. 40. № 8. P. 469–480. https://doi.org/10.1016/j.tins.2017.06.002
Colclough G.L., Smith S.M., Nichols T.E. et al. The heritability of multi-modal connectivity in human brain activity // Elife. 2017. V. 6. Article № e20178. https://doi.org/10.7554/eLife.20178
Constable R.T., Scheinost D., Finn E.S. et al. Potential use and challenges of functional connectivity mapping in intractable epilepsy // Front. Neurol. 2013. V. 4. Article № 39. https://doi.org/10.3389/fneur.2013.00039
Contreras J.A., Goñi J., Risacher S.L. et al. The structural and functional connectome and prediction of risk for cognitive impairment in older adults // Curr. Behav. Neurosci. Rep. 2015. V. 2. № 4. P. 234–245. https://doi.org/10.1007/s40473-015-0056-z
Crossley N.A., Mechelli A., Scott J. et al. The hubs of the human connectome are generally implicated in the anatomy of brain disorders // Brain. 2014. V. 137. № 8. P. 2382–2395. https://doi.org/10.1093/brain/awu132
Diez I., Sepulcre J. Neurogenetic profiles delineate large-scale connectivity dynamics of the human brain // Nat. Commun. 2018. V. 9. № 1. Article № 3876. https://doi.org/10.1038/s41467-018-06346-3
Dima D., Jogia J., Collier D. et al. Independent modulation of engagement and connectivity of the facial network during affect processing by CACNA1C and ANK3 risk genes for bipolar disorder // JAMA Psychiatry. 2013. V. 70. № 12. P. 1303–1311. https://doi.org/10.1001/jamapsychiatry.2013.2099
Dresler M., Shirer W.R., Konrad B.N. et al. Mnemonic training reshapes brain networks to support superior memory // Neuron. 2017. V. 93. № 5. P. 1227–1235. https://doi.org/10.1016/j.neuron.2017.02.003
Erk S., Meyer-Lindenberg A., Linden D.E.J. et al. Replication of brain function effects of a genome-wide supported psychiatric risk variant in the CACNA1C gene and new multi-locus effects // Neuroimage. 2014. V. 94. P. 147–154. https://doi.org/10.1016/j.neuroimage.2014.03.007
Erk S., Meyer-Lindenberg A., Schnell K. et al. Brain function in carriers of a genome-wide supported bipolar disorder variant // Arch. Gen. Psychiatry. 2010. V. 67. № 8. P. 803–811. https://doi.org/10.1001/archgenpsychiatry.2010.94
Esslinger C., Walter H., Kirsch P. et al. Neural mechanisms of a genome-wide supported psychosis variant // Science. 2009. V. 324. № 5927. Article № 605. https://doi.org/10.1126/science.1167768
Fakhry A., Ji S. High-resolution prediction of mouse brain connectivity using gene expression patterns // Methods. 2015. V. 73. P. 71–78. https://doi.org/10.1016/j.ymeth.2014.07.011
Fan C.C., Smeland O.B., Schork A.J. et al. Beyond heritability: improving discoverability in imaging genetics // Hum. Mol. Genet. 2018. V. 27. № R1. P. R22–R28. https://doi.org/10.1093/hmg/ddy082
Fazio L., Pergola G., Papalino M. et al. Transcriptomic context of DRD1 is associated with prefrontal activity and behavior during working memory // Proc. Natl. Acad. Sci. USA. 2018. V. 115. № 21. P. 5582–5587. https://doi.org/10.1073/pnas.1717135115
Fornito A., Zalesky A., Bassett D.S. et al. Genetic influences on cost-efficient organization of human cortical functional networks // J. Neurosci. 2011. V. 31. № 9. P. 3261–3270. https://doi.org/10.1523/JNEUROSCI.4858-10.2011
Fox A.S., Chang L.J., Gorgolewski K.J., Yarkoni T. Bridging psychology and genetics using large-scale spatial analysis of neuroimaging and neurogenetic data // bioRxiv. 2014. (https://www.biorxiv.org/content/early/2014/12/09/012310) https://doi.org/10.1101/012310
Franzmeier N., Rubinski A., Neitzel J. et al. Functional connectivity associated with tau levels in ageing, Alzheimer’s, and small vessel disease // Brain. 2019. V. 142. № 4. P. 1093–1107. https://doi.org/10.1093/brain/awz026
French L., Pavlidis P. Relationships between gene expression and brain wiring in the adult rodent brain // PLoS Comput. Biol. 2011. V. 7. № 1. Article № e1001049. https://doi.org/10.1371/journal.pcbi.1001049
French L., Tan P.P., Pavlidis P. Large-scale analysis of gene expression and connectivity in the rodent brain: insights through data integration // Front. Neuroinf. 2011. V. 5. Article № 12. https://doi.org/10.3389/fninf.2011.00012
Fries P. Rhythms for cognition: Communication through coherence // Neuron. 2015. V. 88. № 1. P. 220–235. https://doi.org/10.1016/j.neuron.2015.09.034
Friston K.J. Functional and effective connectivity: a review // Brain Connect. 2011. V. 1. № 1. P. 13–36. https://doi.org/10.1089/brain.2011.0008
Fulcher B.D., Fornito A. A transcriptional signature of hub connectivity in the mouse connectome // Proc. Natl. Acad. Sci. USA. 2016. V. 113. № 5. P. 1435–1440. https://doi.org/10.1073/pnas.1513302113
Gamazon E.R., Wheeler H.E., Shah K.P. et al. A gene-based association method for mapping traits using reference transcriptome data // Nat. Genet. 2015. V. 47. № 9. P. 1091–1098. https://doi.org/10.1038/ng.3367
Ganglberger F., Kaczanowska J., Penninger J.M. et al. Predicting functional neuroanatomical maps from fusing brain networks with genetic information // NeuroImage. 2018. V. 170. P. 113–120. https://doi.org/10.1016/j.neuroimage.2017.08.070
Glahn D.C., Winkler A.M., Kochunov P. et al. Genetic control over the resting brain // Proc. Natl. Acad. Sci. USA. 2010. V. 107. № 3. P. 1223–1228. https://doi.org/10.1073/pnas.0909969107
Hawrylycz M., Miller J.A., Menon V. et al. Canonical genetic signatures of the adult human brain // Nat. Neurosci. 2015. V. 18. № 12. P. 1832–1844. https://doi.org/10.1038/nn.4171
Heck A., Fastenrath M., Ackermann S. et al. Converging genetic and functional brain imaging evidence links neuronal excitability to working memory, psychiatric disease, and brain activity // Neuron. 2014. V. 81. № 5. P. 1203–1213. https://doi.org/10.1016/j.neuron.2014.01.010
Hellwig B. A quantitative analysis of the local connectivity between pyramidal neurons in layers 2/3 of the rat visual cortex // Biol. Cybern. 2000. V. 82. № 2. P. 111–121. https://doi.org/10.1007/PL00007964
Ji S., Fakhry A., Deng H. Integrative analysis of the connectivity and gene expression atlases in the mouse brain // Neuroimage. 2014. V. 84. P. 245–253. https://doi.org/10.1016/j.neuroimage.2013.08.049
Kaufman A., Dror G., Meilijson I., Ruppin E. Gene expression of Caenorhabditis elegans neurons carries information on their synaptic connectivity // PLoS Comput. Biol. 2006. V. 2. № 12. Article № e167. https://doi.org/10.1371/journal.pcbi.0020167
Kepa A., Martinez Medina L., Erk S. et al. Associations of the intellectual disability gene MYT1L with Helix-Loop-Helix gene expression, hippocampus volume and hippocampus activation during memory retrieval // Neuropsychopharmacology. 2017. V. 42. № 13. P. 2516–2526. https://doi.org/10.1038/npp.2017.91
Kong R., Li J., Orban C. et al. Spatial topography of individual-specific cortical networks predicts human cognition, personality, and emotion // Cereb. Cortex. 2019. V. 29. № 6. P. 2533–2551. https://doi.org/10.1093/cercor/bhy123
Kong X.Z., Song Y., Zhen Z., Liu J. Genetic variation in S100B modulates neural processing of visual scenes in Han Chinese // Cereb. Cortex. 2017. V. 27. № 2. P. 1326–1336. https://doi.org/10.1093/cercor/bhv322
Krienen F.M., Yeo B.T.T., Ge T. et al. Transcriptional profiles of supragranular-enriched genes associate with corticocortical network architecture in the human brain // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 4. P. E469–E478. https://doi.org/10.1073/pnas.1510903113
Krug A., Witt S.H., Backes H. et al. A genome-wide supported variant in CACNA1C influences hippocampal activation during episodic memory encoding and retrieval // Eur. Arch. Psychiatry Clin. Neurosci. 2014. V. 264. № 2. P. 103–110. https://doi.org/10.1007/s00406-013-0428-x
Lein E.S., Hawrylycz M.J., Ao N. et al. Genome-wide atlas of gene expression in the adult mouse brain // Nature. 2007. V. 445. № 7124. P. 168–176. https://doi.org/10.1038/nature05453
Liégeois F., Baldeweg T., Connelly A. et al. Language fMRI abnormalities associated with FOXP2 gene mutation // Nat. Neurosci. 2003. V. 6. № 11. P. 1230–1237. https://doi.org/10.1038/nn1138
Liégeois F., Morgan A.T., Connelly A., Vargha-Khadem F. Endophenotypes of FOXP2: dysfunction within the human articulatory network // Eur. J. Paediatr. Neurol. 2011. V. 15. № 4. P. 283–288. https://doi.org/10.1016/j.ejpn.2011.04.006
Lisachev P.D., Shtark M.B. Long-term potentiation-associated gene expression: involvement of the tumour protein p53 // In: The Hippocampus – Plasticity and Functions / Ed. A. Stuchlik – London: IntechOpen, 2018. P. 49–64. https://doi.org/10.5772/intechopen.73219
Mattar M.G., Wymbs N.F., Bock A.S. et al. Predicting future learning from baseline network architecture // Neuroimage. 2018. V. 172. P. 107–117. https://doi.org/10.1016/j.neuroimage.2018.01.037
McClearn G.E., Johansson B., Berg S. et al. Substantial genetic influence on cognitive abilities in twins 80 or more years old // Science. 1997. V. 276. № 5318. P. 1560–1563. https://doi.org/10.1126/science.276.5318.1560
McFarland D.J. How neuroscience can inform the study of individual differences in cognitive abilities // Rev. Neurosci. 2017. V. 28. № 4. P. 343–362. https://doi.org/10.1515/revneuro-2016-0073
McIntosh A.R. Towards a network theory of cognition // Neural Netw. 2000. V. 13. № 8–9. P. 861–870. https://doi.org/10.1016/S0893-6080(00)00059-9
Mears D., Pollard H.B. Network science and the human brain: Using graph theory to understand the brain and one of its hubs, the amygdala, in health and disease // J. Neurosci. Res. 2016. V. 94. № 6. P. 590–605. https://doi.org/10.1002/jnr.23705
Mills B.D., Grayson D.S., Shunmugavel A. et al. Correlated gene expression and anatomical communication support synchronized brain activity in the mouse functional connectome // J. Neurosci. 2018. V. 38. № 25. P. 5774–5787. https://doi.org/10.1523/JNEUROSCI.2910-17.2018
Miranda-Dominguez O., Feczko E., Grayson D.S. et al. Heritability of the human connectome: A connectotyping study // Netw. Neurosci. 2018. V. 2. № 2. P. 175–199. https://doi.org/10.1162/netn_a_00029
Moseley R.L., Ypma R.J., Holt R.J. et al. Whole-brain functional hypoconnectivity as an endophenotype of autism in adolescents // Neuroimage Clin. 2015. V. 9. P. 140–152. https://doi.org/10.1016/j.nicl.2015.07.015
Mufford M.S., Stein D.J., Dalvie S. et al. Neuroimaging genomics in psychiatry – a translational approach // Genome Med. 2017. V. 9. № 1. Article № 102. https://doi.org/10.1186/s13073-017-0496-z
Panizzon M.S., Neale M.C., Docherty A.R. et al. Genetic and environmental architecture of changes in episodic memory from middle to late middle age // Psychol. Aging. 2015. V. 30. № 2. P. 286–300. https://doi.org/10.1037/pag0000023
Pantazatos S.P., Li X. Commentary: BRAIN NETWORKS. Correlated gene expression supports synchronous activity in brain networks. Science 348, 1241-4. // Front. Neurosci. 2017. V. 11. Article № 412. https://doi.org/10.3389/fnins.2017.00412
Park M.T.M., Raznahan A., Shaw P. et al. Neuroanatomical phenotypes in mental illness: identifying convergent and divergent cortical phenotypes across autism, ADHD and schizophrenia // J. Psychiatry Neurosci. 2018. V. 43. № 3. P. 201–212. https://doi.org/10.1503/jpn.170094
Peng Q., Schork A., Bartsch H. et al. Conservation of distinct genetically-mediated human cortical pattern // PLoS Genet. 2016. V. 12. № 7. Article № e1006143. https://doi.org/10.1371/journal.pgen.1006143
Pergola G., Di Carlo P., Andriola I. et al. Combined effect of genetic variants in the GluN2B coding gene (GRIN2B) on prefrontal function during working memory performance // Psychol. Med. 2016. V. 46. № 6. P. 1135–1150. https://doi.org/10.1017/S0033291715002639
Pietzuch M., King A.E., Ward D.D., Vickers J.C. The influence of genetic factors and cognitive reserve on structural and functional resting-state brain networks in aging and Alzheimer’s disease // Front. Aging Neurosci. 2019. V. 11. Article № 30. https://doi.org/10.3389/fnagi.2019.00030
Polimanti R., Meda S.A., Pearlson G.D. et al. S100A10 identified in a genome-wide gene × cannabis dependence interaction analysis of risky sexual behaviours // J. Psychiatry Neurosci. 2017. V. 42. № 4. P. 252–261. https://doi.org/10.1503/jpn.160189
Polimanti R., Yang C., Zhao H., Gelernter J. Dissecting ancestry genomic background in substance dependence genome-wide association studies // Pharmacogenomics. 2015. V. 16. № 13. P. 1487–1498. https://doi.org/10.2217/pgs.15.91
Quintana D.S., Rokicki J., van der Meer D. et al. Oxytocin pathway gene networks in the human brain // Nat. Commun. 2019. V. 10. № 1. Article № 668. https://doi.org/10.1038/s41467-019-08503-8
Raum H., Dietsche B., Nagels A. et al. A genome-wide supported psychiatric risk variant in NCAN influences brain function and cognitive performance in healthy subjects // Hum. Brain Mapp. 2015. V. 36. № 1. P. 378–390. https://doi.org/10.1002/hbm.22635
Richiardi J., Altmann A., Milazzo A.C. et al. Correlated gene expression supports synchronous activity in brain networks // Science. 2015. V. 348. № 6240. P. 1241–1244. https://doi.org/10.1126/science.1255905
Rodríguez-Rojo I.C., Cuesta P., López M.E. et al. BDNF Val66Met polymorphism and gamma band disruption in resting state brain functional connectivity: A magnetoencephalography study in cognitively intact older females // Front. Neurosci. 2018. V. 12. Article № 684. https://doi.org/10.3389/fnins.2018.00684
Roostaei T., Sadaghiani S., Park M.T. et al. Channelopathy-related SCN10A gene variants predict cerebellar dysfunction in multiple sclerosis // Neurology. 2016. V. 86. № 5. P. 410–417. https://doi.org/10.1212/WNL.0000000000002326
Rubinov M., Sporns O. Complex network measures of brain connectivity: uses and interpretations // Neuroimage. 2010. V. 52. № 3. P. 1059–1069. https://doi.org/10.1016/j.neuroimage.2009.10.003
Rubinov M., Ypma R.J., Watson C., Bullmore E.T. Wiring cost and topological participation of the mouse brain connectome // Proc. Natl. Acad. Sci. USA. 2015. V. 112. № 32. P. 10032–10037. https://doi.org/10.1073/pnas.1420315112
Sadaghiani S., Ng B., Altmann A. et al. Overdominant effect of a CHRNA4 polymorphism on cingulo-opercular network activity and cognitive control // J. Neurosci. 2017. V. 37. № 40. P. 9657–9666. https://doi.org/10.1523/JNEUROSCI.0991-17.2017
Scannell J.W., Blakemore C., Young M.P. Analysis of connectivity in the cat cerebral cortex // J. Neurosci. 1995. V. 15. № 2. P. 1463–1483. https://doi.org/10.1523/JNEUROSCI.15-02-01463.1995
Schmitt J.E., Lenroot R.K., Wallace G.L. et al. Identification of genetically mediated cortical networks: a multivariate study of pediatric twins and siblings // Cereb. Cortex. 2008. V. 18. № 8. P. 1737–1747. https://doi.org/10.1093/cercor/bhm211
Scult M.A., Hariri A.R. A brief introduction to the neurogenetics of cognition-emotion interactions // Curr. Opin. Behav. Sci. 2018. V. 19. P. 50–54. https://doi.org/10.1016/j.cobeha.2017.09.014
Shtark M.B., Kozlova L.I., Bezmaternykh D.D. et al. Neuroimaging study of alpha and beta EEG biofeedback effects on neural networks // Appl. Psychophysiol. Biofeedback. 2018. V. 43. № 2. P. 169-178. https://doi.org/10.1007/s10484-018-9396-2
Sinclair B., Hansell N.K., Blokland G.A. et al. Heritability of the network architecture of intrinsic brain functional connectivity // Neuroimage. 2015. V. 121. P. 243–252. https://doi.org/10.1016/j.neuroimage.2015.07.048
Smit D.J., Stam C.J., Posthuma D. et al. Heritability of “small-world” networks in the brain: a graph theoretical analysis of resting-state EEG functional connectivity // Hum. Brain Mapp. 2008. V. 29. № 12. P. 1368–1378. https://doi.org/10.1002/hbm.20468
Stevens A.A., Tappon S.C., Garg A., Fair D.A. Functional brain network modularity captures inter- and intra-individual variation in working memory capacity // PLoS One. 2012. V. 7. № 1. Article № e30468. https://doi.org/10.1371/journal.pone.0030468
Suo X.S., Lei D.L., Li L.L. et al. Psychoradiological patterns of small-world properties and a systematic review of connectome studies of patients with 6 major psychiatric disorders // J. Psychiatry Neurosci. 2018. V. 43. № 6. Article № 427. https://doi.org/10.1503/jpn.170214
Trost S., Diekhof E.K., Mohr H. et al. Investigating the impact of a genome-wide supported bipolar risk variant of MAD1L1 on the human reward system // Neuropsychopharmacology. 2016. V. 41. № 11. P. 2679–2687. https://doi.org/10.1038/npp.2016.70
van den Heuvel M.P., Hulshoff Pol H.E. Exploring the brain network: a review on resting-state fMRI functional connectivity // Eur. Neuropsychopharmacol. 2010. V. 20. № 8. P. 519–534. https://doi.org/10.1016/j.euroneuro.2010.03.008
Varadan V., Miller D.M. 3rd, Anastassiou D. Computational inference of the molecular logic for synaptic connectivity in C. elegans // Bioinformatics. 2006. V. 22. № 14. P. e497–e506. https://doi.org/10.1093/bioinformatics/btl224
Vértes P.E., Rittman T., Whitaker K.J. et al. Gene transcription profiles associated with inter-modular hubs and connection distance in human functional magnetic resonance imaging networks // Philos. Trans. R. Soc. Lond. B Biol. Sci. 2016. V. 371. № 1705. Article № 20150362. https://doi.org/10.1098/rstb.2015.0362
Vecchio F., Miraglia F., Rossini P.M. Connectome: Graph theory application in functional brain network architecture // Clinical Neurophysiology Practice. 2017. V. 2. P. 206–213. doi.org/https://doi.org/10.1016/j.cnp.2017.09.003
Volk H.E., McDermott K.B., Roediger H.L.3rd, Todd R.D. Genetic influences on free and cued recall in long-term memory tasks // Twin Res. Hum. Genet. 2006. V. 9. № 5. P. 623–631. https://doi.org/10.1375/183242706778553462
Wang C., Zhang Y., Liu B. et al. Dosage effects of BDNF Val66Met polymorphism on cortical surface area and functional connectivity // J. Neurosci. 2014. V. 34. P. 2645–2651. https://doi.org/10.1523/JNEUROSCI.3501-13.2014
Wang G.Z., Belgard T.G., Mao D. et al. Correspondence between resting-state activity and brain gene expression // Neuron. 2015. V. 88. № 4. P. 659–666. https://doi.org/10.1016/j.neuron.2015.10.022
Wang J., Zuo X., He Y. Graph-based network analysis of resting-state functional MRI // Front. Syst. Neurosci. 2010. V. 4. Article № 16. https://doi.org/10.3389/fnsys.2010.00016
Wen J., Goyal M.S., Astafiev S.V. et al. Genetically defined cellular correlates of the baseline brain MRI signal // Proc. Natl. Acad. Sci. USA. 2018. V. 115. № 41. P. E9727–E9736. https://doi.org/10.1073/pnas.1808121115
White J.G., Southgate E., Thomson J.N., Brenner S. The structure of the nervous system of the nematode Caenorhabditis elegans // Philos. Trans. R. Soc. Lond. B Biol. Sci. 1986. V. 314. № 1165. P. 1–340. https://doi.org/10.1098/rstb.1986.0056
Wolf L., Goldberg C., Manor N. et al. Gene expression in the rodent brain is associated with its regional connectivity // PLoS Comput Biol. 2011. V. 7. № 5. Article № e1002040. https://doi.org/10.1371/journal.pcbi.1002040
Zapala M.A., Hovatta I., Ellison J.A. et al. Adult mouse brain gene expression patterns bear an embryologic imprint // Proc. Natl. Acad. Sci. USA. 2005. V. 102. № 29. P. 10357–10362. https://doi.org/10.1073/pnas.0503357102
Zhang N., Liu H., Qin W. et al. APOE and KIBRA interactions on brain functional connectivity in healthy young adults // Cereb. Cortex. 2017. V. 27. № 10. P. 4797–4805. https://doi.org/10.1093/cercor/bhw276
Yang Z., Zuo X.-N., McMahon K.L. et al. Genetic and environmental contributions to functional connectivity architecture of the human brain // Cereb. Cortex. 2016. V. 26. № 5. P. 2341–2352. https://doi.org/10.1093/cercor/bhw027
Yarkoni T., Poldrack R.A., Nichols T.E. et al. Large-scale automated synthesis of human functional neuroimaging data // Nat. Methods. 2011. V. 8. № 8. P. 665–670. https://doi.org/10.1038/nmeth.1635
Yin Y., Hou Z., Wang X. et al. The BDNF Val66Met polymorphism, resting-state hippocampal functional connectivity and cognitive deficits in acute late-onset depression // J. Affect. Disord. 2015. V. 183. P. 22–30. https://doi.org/10.1016/j.jad.2015.04.050
Дополнительные материалы отсутствуют.
Инструменты
Успехи физиологических наук


