Успехи физиологических наук, 2020, T. 51, № 4, стр. 64-81
Механизмы возникновения и эволюции белок-кодирующих генов
Р. Н. Мустафин *
Федеральное государственное бюджетное образовательное учреждение высшего образования “Башкирский государственный медицинский университет”
450008 г. Уфа, Россия
* E-mail: ruji79@mail.ru
Поступила в редакцию 12.04.2018
После доработки 10.10.2019
Принята к публикации 10.07.2020
Аннотация
В обзорной статье рассмотрены различные механизмы возникновения белок-кодирующих генов в эволюции. У прокариот важными источниками новых белков служат внегеномные элементы – плазмиды, интегроны, CRISPR и бактериофаги, гены вспомогательного набора которых в ходе эволюции переходят в базовые. В интеграции новых последовательностей в геном при этом ключевую роль играют транспозоны, ретроэлементы и некодирующие РНК, оказывающие регуляторный эффект в адапатации. Возникновение эукариот связано с глобальным распространением мобильных элементов, которые способствовали увеличению размеров геномов за счет формирования сложных регуляторных структур и генов некодирующих РНК, формируя интроны и межгенные области. Кроме того, транспозоны у эукариот оказались важными источниками белок-кодирующих генов за счет образования ретрокопий, экзонизации инсерций, одомашнивания генов транспозонов. Эффективность распространения мобильных элементов связана с образованием доменных структур их транскриптов (в том числе представленных некодирующими РНК) и продуктов трансляции, способных взаимодействовать с регуляторными последовательностями транспозонного происхождения в геноме и другими молекулами. Обнаруженная способность некодирующих РНК транслироваться в функциональные пептиды позволяет предположить данный механизм в качестве возможного источника формирования новых белок-кодирующих генов. Предполагается, что эволюция эукариот обусловлена совершенствованием универсальных систем регуляции работы геномов, развившихся в качестве систем защиты хозяев от транспозонов и вирусов. К данным системам относятся РНК-интерференция, сплайсинг, контроль структуры генома посредством гистонов, теломер и метилирования ДНК. Одной из причин возникновения комплекса систем могло быть физическое разобщение генов, участвующих в единых биохимических процессах (оперон у прокариот) в связи с многочисленными инсерциями с привлечением регуляторных элементов транспозонов, которые не только компенсировали данное разобщение, но и предоставили возможность более координированной динамической регуляции работы генома.
ВВЕДЕНИЕ
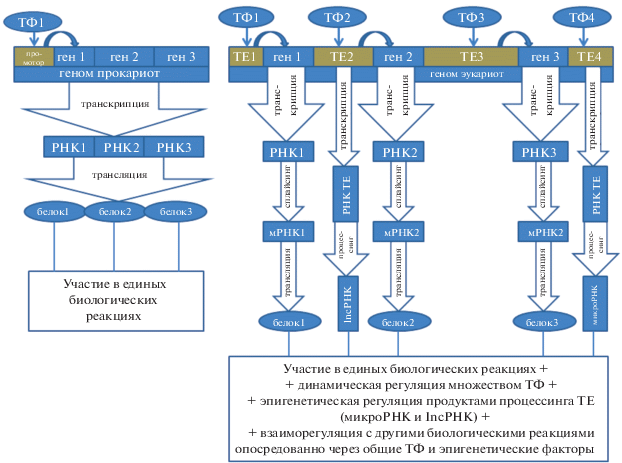
Структурное разнообразие геномов эукариот формируется благодаря мобильным элементам (TE – transposable elements), которые оказались важными источниками образования их белок-кодирующих генов [29], длинных некодирующих РНК (нкРНК) [47, 48, 67] и микроРНК [18, 37, 60, 65, 83]. Классификация ТЕ подробно описана в обзоре [9]. Последовательности ТЕ обнаруживаются в экзонах [52, 53] и интронах [56, 94] белок-кодирующих генов, а также в регуляторных структурах [17, 35, 57, 79]. Описаны механизмы, посредством которых формируются белок-кодирующие гены в эволюции из последовательностей ТЕ, включая одомашнивание целых генов ТЕ [17, 29, 30, 58, 62], экзонизацию инсерций [91, 94], неофункционализацию дуплицированных при помощи ТЕ копий генов [53], преобразование генов длинных нкРНК [16, 77, 101] и микроРНК в кодирующие функциональные пептиды [26, 27, 55].
Механизмы формирования генов у прокариот и эукариот в эволюции кардинально отличаются, что накладывает свой отпечаток на особенностях организации их геномов, которые обусловлены главным образом распространением у эукариот ТЕ. Благодаря транспозонам сформировались практически все глобальные отличительные черты организации геномов эукариот: мозаичная интрон-экзонная структура генов [81], альтернативные сплайсинговые варианты генов [87], ретрогены [41], нуклеосомная организация хроматина с управлением транскрипцией при помощи модификаций гистонов [72, 73], эпигенетическая регуляция при помощи консервативных для эукариот микроРНК [53], дискретная линейная структура хромосом с теломерами на концах [3, 23, 36, 51]. Формирование данных систем обусловлено эволюцией защитных механизмов хозяев, направленных против распространения чужеродных последовательностей вирусов и ТЕ. Однако совершенствование комплексных координированных систем взаиморегуляции белков с ТЕ у эукариот позволили им выйти на эволюционно новый уровень преобразований геномов. К примеру, отдельные компоненты РНК-интерференции (РНКи) обнаружены у прокариот, но не развились в единый взаимосвязанный комплекс ферментов из Dicer, Argonaute, RdRP или их гомологов [88].
Предположительной причиной универсализации систем контроля ТЕ может быть необходимость массового возникновения новых генов и регуляторных структур в жестких условиях существования предка всех эукариот. Действительно, согласно многочисленным исследованиям, эукариоты произошли от архей, для которых характерно проживание в агрессивной среде [13, 15, 40]. В этих условиях возможность выживания приобретают те особи, у которых формируется максимальное количество возможных белков, пептидов и РНК-рибозимов, обеспечивающих новые адаптивные свойства. Источником данных преобразований могли быть ТЕ, в особенности ретроэлементы, увеличивающие размеры геномов и обеспечивающие организмы не только новыми вариантами белков, в том числе транскрипционными факторами (ТФ), но и регуляторными последовательностями [17, 35, 57, 79], связывающимися с данными факторами. Интересно, что в эволюции передача генов от бактерий археям происходила в 5 раз чаще, чем от архей бактериям. В исследовании Nelson-Sathi с соавторами при изучении филогенеза 267 568 белок-кодирующих генов из 134 секвенированных геномов архей, выявлено 2264 группо-специфических случаев приобретения генов от бактерий [78]. Это говорит о более успешной работе транспозонов у архей, способных участвовать в передаче генов вспомогательного набора в базовые.
ВОЗНИКНОВЕНИЕ ГЕНОВ ПРОКАРИОТ
Эволюция геномов прокариот происходит благодаря вертикальному и горизонтальному переносам генов [78]. Широко известной моделью объяснения происхождения новых генов в эволюции прокариот является их дупликация. Согласно недавним филогенетическим исследованиям, формирование генов de novo может быть и альтернативным. Это подтверждается выводами о том, что длинные некодирующие РНК (нкРНК) могут служить пулом новизны, а рибосомы могут транслировать новые ORF. Прокариотические геномы отличаются плотной упаковкой генов, и межгенные пространства весьма ограничены. Поэтому еще в 1977 г. Grasse предложил механизм эволюции новых генов под названием “перекрывание”, подтвержденный в последующем работами Ohno. Данный механизм заключается в том, что некодирующие нуклеотидные последовательности перекрывают существующие гены по альтернативной рамке считывания, преобразуясь в кодирующие последовательности путем создания нового промотора рядом с подходящим сайтом связывания рибосом и стартовым кодоном. Тривиальные перекрывания только нескольких пар оснований обнаружено у 30% генов бактерий. При нетривиальных перекрываниях (характерных для вирусов, но редких для прокариот) гены белок-кодирующих областей внедряются полностью или частично в аннотированные “материнские гены” [34].
В формировании генов у прокариот ключевую роль играют ферменты ретроэлементов (РЭ), обеспечивающие возможность в процессе эволюции генам базового набора переходить в категорию вспомогательных, а генам вспомогательного набора – в базовые [13]. Данное универсальное свойство обеспечивает выживание бактерий и архей за счет обмена необходимыми для адаптации генами. Наиболее активными в межклеточном переносе и распространении чужеродных генов являются плазмиды, особенно конъюгативные. Обычно плазмиды несут широкое разнообразие генов, помогающих прокариотам выживать в определенных условиях, например в присутствии антибиотиков или солей тяжелых металлов, или обеспечивать их проникновение и существование внутри инфицируемых организмов. Появление таких генов в плазмидах в большинстве случаев связывают с ТЕ и интегронами, способными включать кассетные гены. Обнаружены также CR-элементы (common regions) бактерий, вызывающие появление и распространение целых блоков генов и способные к независимой транспозиции с помощью механизма репликации по типу “катящегося кольца” (rolling circle – RC) [5].
Механизм, обеспечивающий возможность в процессе эволюции генам базового набора переходить в категорию вспомогательных, а генам вспомогательного набора – в базовые, сформированный благодаря РЭ, является наиболее усовершенствованной универсальной системой, обеспечивающий выживание отдельных особей и эволюцию прокариот. Гены, поступающие в бактерии и археи путем горизонтального переноса, и передающие адаптационные признаки, формируются за счет мутационных процессов. РЭ характерны для прокариот, но их особенности функционирования значительно отличаются от эукариотических. Впервые РЭ, известные как ретроны, открыты в 1989 году у Myxococcus xanthus [54] и E. coli [61]. Другой тип бактериальных ретроэлементов, DGR (diversity-generating retroelements), впервые был описан в 2002 году у представителей рода Bordetella [63]. DGR состоят из обратной транскриптазы (RT – reverse transcriptase), дополнительного белка (кодируемого геном atd), матрицы РНК и кодирующего целевой белок гена mtd, который содержит С-концевую вариабельную область (VR – variable region). DGR не проявляют мобильность, но производят различные последовательности в области VR, что потенциально обеспечивает устойчивость к фагам. Бактерии имеют и другие механизмы иммунитета против фагов, включая CRISPR (clustered regularly interspaced short palindromic repeats) и Abi (abortive bacteriophage infection), также содержащие в своем составе RT. Было показано, что два не охарактеризованных класса бактериальных RT ассоциированы с CRISPR/cas элементами. Система Abi блокирует фаги на разных стадиях инфекционного цикла и обычно обеспечивается одним плазмидным геном. Системы AbiA, AbiK, Abi-P2 включают RT, а фермент AbiK обладает подлинной RT-активностью [92]. Существование РЭ у прокариот и их отличительные свойства от эукариотических говорят об их возможном участии в формировании новых генов за счет того, что, помимо защиты от фагов, их RT могут участвовать в переходе генов вспомогательного набора в базовые. В эволюции это могло быть причиной возникновения предка эукариот, характеризующегося постоянным противоборством ТЕ с геномами хозяев, в результате которого возникли системы контроля ТЕ с возможностью их экзаптации для нужд хозяев.
ВОЗНИКНОВЕНИЕ УНИВЕРСАЛЬНЫХ СИСТЕМ УПРАВЛЕНИЯ ГЕНОМОМ
Геномы бактерий и архей представлены нуклеоидом и способными к автономной репликации плазмидами. Они организованы главным образом кольцевыми двухцепочечными молекулами ДНК. Встречаются редкие исключения. Например, у рода Borrelia геномы имеют линейную структуру с замкнутыми шпилечными концами, у Streptomyces – с ковалентно соединенными к 5'-концам ДНК белками инициации репликации. У некоторых бактерий могут быть два нуклеоида, например, у Agrobacterium tumefaciens 1 линейный и 1 кольцевой нуклеоиды, у Vibrio cholera и архея Haloarcula marismortui 2 кольцевых нуклеоида [13]. Некоторые бактерии обладают множеством плазмид размером с хромосому (например, Vibrio). Определенные прокариоты проявляют полиплоидию в связи с содержанием множества копий генома, а некоторые могут даже содержать эндосимбионтов с их собственными геномами. Однако эукариоты отличаются тем, что проявляют все отличительные признаки одновременно [32].
Гены прокариот организованы в структурно-функциональные единицы – опероны, в связи с чем любое внедрение в них чужеродных последовательностей может грозить гибели. Это обуславливает поддержание жестких контрмер против инсерций в геномы [88]. Инсерции происходят, но они жестко урегулированы при помощи сайстспецифической и гомологичной рекомбинации и генетических структур, таких как CRISPR/cas, плазмиды, инсерционные последовательности (IS-элементы), транспозоны, интегроны с их генными кассетами, сложно организованные модульные структуры (интегрирующие конъюгативные (ICE) и мобилизуемые элементы (IME), геномные острова). Эти механизмы участвуют в переносе генов как в пределах одной молекулы ДНК, так и между разными молекулами (хромосомно-плазмидная, хромосомно-фаговая), находящимися в одной клетке, способными в дальнейшем обеспечивать межклеточное распространение генов посредством конъюгации, трансформации или трансдукции [5]. Некоторые инсерции ТЕ в регуляторных последовательностях геномов бактерий могут сохраняться в виде “направленных мутаций”, если они выгодны хозяину. Например, у мутантных (делеция гена crp) E. coli, которые не могут расти на глицерине, инсерция IS5 в специфическую область перед промотором glpFK способствует эффективному использованию глицерина со скоростями, которые увеличиваются при добавлении глицерина или при потере репрессора глицерина [100]. Это говорит о том, что отдельные принципы использования последовательностей ТЕ геномами хозяев для управления экспрессией генов, формировались уже у прокариот. Однако в полной мере данные принципы в виде универсальных систем реализованы у эукариот, что связано со значительным усложнением организации и функционирования их геномов.
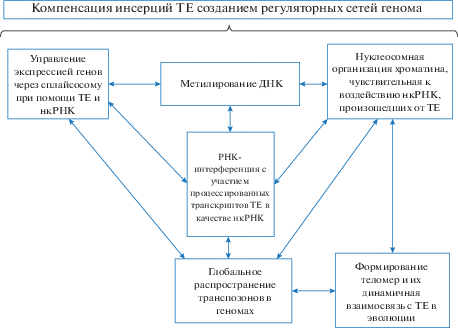
Гены у эукариот не формируют оперонов, несмотря на функциональное взаимодействие, они разобщены в пространственном отношении в ДНК, что свидетельствует о важном значении регуляторных структур, обеспечивающих взаимосвязь продуктов экспрессии, которые участвуют в единых биологических реакциях. Глобальное распространение ТЕ в геномах эукариот объясняет данный феномен, так как многие компонетны транспозонов экзаптируются хозяином в качестве собственных регуляторных элементов [17, 35, 57, 79]. Пространственное разобщение генов обусловлено распространением ТЕ, при котором сохраняют жизнеспособность те особи, у которых внедряющиеся последовательности обеспечивали возможность координации в работе продуктов экспрессии за счет регуляторных функций последовательностей ТЕ (рис. 1). Данное универсальное свойство, сформированное на одном из этапов эволюции, совершенствовалось в дальнейшем с регуляцией эпигенетических факторов транспозонами (подробнее в обзоре [10]), обеспечивая образование новых механизмов регуляции экспрессии генов при условии отбора особей с оптимальными фенотипическими проявлениями полученных регуляторных комплексов.
Рис. 1.
Схема компенсации разобщения генов, составляющих оперон, созданием множества регуляторных связей транспозонами, инсерции которых вызывают данные разобщения.
Специфические особенности количества и структуры хромосом каждого вида эукариот могут свидетельствовать об отборе оптимального состава и количества ТЕ относительно генов и регуляторных структур (подробнее в обзоре [8]). Таким образом, первой универсальной системой, сыгравшей ключевую роль в возникновении эукариот, стал механизм компенсации инсерций ТЕ в различные сайты генома созданием новых регуляторных сетей, обеспечивающих адаптирующее управление экспрессии генов в онтогенезе. При этом сами ТЕ стали важными источниками и регуляторных последовательностей и ТФ, с ними связывающихся [35], играя важную роль в формировании геномов эукариот за счет способности ТЕ ремоделировать регуляторные сети [70]. Предполагается, что ТЕ являются основными факторами генной регуляции, являясь источниками сайтов связывания с ТФ (TFBS – transcription factor binding site), которые служат в качестве энхансеров и промоторов [28]. При исследовании TFBS у 29 видов млекопитающих было выявлено, что геномы кооптируют фрагменты ТЕ для использования в качестве регуляторов в самых разнообразных процессах [66]. При этом ТЕ, содержащие TFBS, могут переписывать новые гены в существующие транскрипционные сети. Например, ТЕ значительно амплифицировали E2F TFBS при видообразовании капусты, в результате чего у некоторых видов капусты более 85% всех E2F TFBS находятся в пределах ТЕ [39]. В геноме человека энхансеры, произошедшие от MIR (mammalian wide interspersed repeat), являются богатыми источниками TFBS, которые играют важную регуляторную роль, что объясняет их сохранение в эволюции [46].
Регуляторные последовательности, произошедшие от ТЕ, принимают участие как в поддержании плюрипотентности, так и в управлении дифференцировкой клеток, что согласуется с активацией ТЕ при данных процессах. Например, в геноме человека около половины активных элементов произошли от ТЕ, а 44% открытых областей хроматина содержат ТЕ в своем составе. При этом тысячи TFBS, произошедших от ТЕ, характеризуются тканеспецифическим характером активации [45]. В геноме человека выявлено 794 972 TFBS, которые возникли от LTR (long terminal repeat – длинные концевые повторы) содержащих ТЕ – эндогенных ретровирусов ERV (endogenous retrovirus). Анализ кластеризации показал, что HERV/LTR можно сгруппировать в соответствии с шаблонами связывания с ТФ, к примеру, плюрипотентными ТФ (SOX2, POU5F1, NANOG), ТФ эмбриональной эндодермы/мезодермы (например, GATA4/6, SOX17, FOXA1/2) и гематопоэтические ТФ (например, SPI1(PU1), GATA1/2, TAL1) [43]. Использование РЭ в качестве альтернативных промоторов является распространенным феноменом, проявляющимся преимущественно в эмбриональных стволовых клетках (СК). В эмбриональном развитии до 20% транскриптома инициируется из РЭ – их альтернативные промоторы проявляют склонность к тканеспецифической активности. Многие из этих РЭ были одомашнены путем экзонизации [70]. Например, у мышей активация неавтономных ТЕ MER130 вызывает формирование неокортекса у эмбрионов на 14 день развития. MER130 функционируют в качестве энхансеров, расположенных вблизи генов неокортекса и сохранены в эволюции в связи с данной регуляторной ролью [80]. Распространение определенных ТЕ у разных таксонов в эволюции способствовало установлению линиеспецифических паттернов генной экспрессии. Данные гены ТЕ характеризуются группировкой вокруг генов, вовлеченных в развитие и регуляцию транскрипции [35].
Второй важной системой, сыгравшей роль в возникновении и эволюции эукариот, является участие теломер в регуляторных генных сетях. Кардинальным отличием всех эукариот является организация их ДНК в линейную структуру за счет образования на концах хромосом теломер, происхождение которых обязано РЭ. Так как теломераза в эволюции произошла от РЭ [36, 51], в данном отношении большое значение могли иметь особенности состава и распределения ТЕ в геномах, влияющие на распределение теломер. Можно предположить, что специфическое распределения теломер в геномах отражают отбор наиболее оптимальных регуляторных стратегий в их работе. Интересно, что концевые участки хромосом впервые названы теломерами у дрозофил, у которых они имеют истинное транспозонное происхождение (ретроэлементы HeT-A, TART и TAHRE) вследствие потери теломеразы в эволюции [23], что доказывает роль ТЕ в происхождении теломер. Кроме того, одним из доказательств роли ТЕ в происхождении теломер являются исследования злокачественных опухолей, в которых обнаружен альтернативный механизм их удлинения за счет экспрессии L1, осуществляющего синтез теломерной ДНК “проскальзыванием” [3]. На важную роль теломер в управлении работой генома указывает их взаимосвязь с гистонами. Например, во всех опухолях, несущих мутации ATRX, влияющих на взаимодействие хвостов H3/H3.3, выявлено массивное удлинение длины теломер без индукции теломеразы – альтернативное удлинение теломер [40]. Интересно, что у растений сигналы теломерных последовательностей обнаруживаются также в интерстициальных сигналах хромосом, что объясняет уникальный феномен редукции числа хромосом за счет слияния теломер [2].
Третьей системой, участвующей в управлении работой генома, стала РНК-интерференция, которая первоначально возникла как универсальный консервативный механизм защиты от инсерций ТЕ и является обязательным условием для существования видов [88]. Несмотря на универсальность и консервативность, что говорит об ее эффективности, система РНКи приобрела важное значение для отбора особей с оптимальным составом ТЕ в их геномах. Эффективность данной системы для большинства известных многоклеточных эукариот является отправной точкой для понимания происхождения их генов, а также понимания происхождения самих многоклеточных, ключевую роль в котором сыграли ТЕ (подробнее в обзоре [11]). Объяснить это можно тем, что РНКи необходима для подавления экспрессии ТЕ путем процессинга продуктов экспрессии и направления их в составе RISC против гомологичных последовательностей. В эволюции одомашнивание ТЕ приводит к образованию новых генов хозяев [17, 20, 24, 30, 76, 99], в том числе ТФ [35, 62]. Экзонизация ТЕ в интронах приводит к появлению новых доменов белков [17, 56, 91] или регуляторных последовательностей [31, 91]. Ретрогены, образованные при помощи ТЕ, содержат в своем составе последовательности транспозонов [38, 53, 90]. В связи с этим многие гены содержат ТЕ и их участки в своем составе или происходят непосредственно от ТЕ. Поэтому параллельно с ингибированием самих ТЕ происходит подавление экспрессии белок-кодирующих генов (БКГ), содержащих гомологичные им последовательности.
Четвертая универсальная система – управление экспрессией генов через сплайсинговую машину. Большинство генов всех эукариот содержат интроны [75]. В значительно меньших количествах интроны содержатся у прокариот, которые являются мобильными генетическими элементами и обладают способностью к самосплайсингу [81, 89]. Интроны генов эукариот также происходят от ТЕ – они непосредственно являются результатом многочисленных “бомбардировок” генов транспозонами [53, 98]. При условии, когда все особи подвергались подобным “бомбардировкам” в эволюции выжили лишь те немногочисленные особи, геномы которых выработали способность вместе с защитой от инсерций использовать их для управления экспрессией генов. Это объясняет тот факт, что у эукариот успешно сохранились лишь сплайсосомные интроны, так как могли сохраниться лишь те инсертированные ТЕ, которыми можно было управлять и использовать для оптимизации адаптационных возможностей через сплайсинговую машину (подробнее в обзоре [7]).
Пятая система – это нуклеосомная организация хроматина. Сохранение специфичного для всех эукариот универсального консервативного октамера гистонов непосредственно связано с необходимостью оптимизации управления экспрессией генов [40, 71]. Данная система имеет ключевое значение в управлении работой генных сетей и позволяет скоординировано управлять генной экспрессией за счет изменения модификацией гистонов. Данная система имеет тесную взаимосвязь с ТЕ, которые являются источниками нкРНК [18, 37, 60, 65, 83], воздействующими на модификации гистонов [86]. Кроме того, в эволюции ТЕ участвуют в возникновении генов, продукты которых модифицируют гистоны и метилируют ДНК, что говорит о потенциале ТЕ в непосредственном влиянии на эпигенетическую регуляцию генома [25, 29].
Шестая система (рис. 2) – метилирование ДНК, которое непосредственно зависит от других систем и динамично меняется с каждым клеточным делением при формировании органов и тканей у многоклеточных организмов (подробнее в обзоре [11]). Ключом к управлению метилированием являются последовательные активации ТЕ, запрограммированные видоспецифически в соответствии с составом и расположением ТЕ относительно белок-кодирующих генов, теломер и генов некодирующих РНК [11]. На метилирование воздействуют нкРНК [74, 86, 102], в происхождении которых важную роль играют ТЕ [18, 37, 60, 65, 83]. Показано непосредственное влияние нкРНК, произошедших от ТЕ, в модификации функции специфических локусов [97, 104]. В связи с этим активация определенных ТЕ ведет к метилированию как самих ТЕ, так и БКГ, содержащих гомологичные последовательности. На метилирование влияют модификации гистонов под действием нкРНК [86] и под влиянием непосредственного расположения TE в геноме [4].
ВОЗНИКНОВЕНИЕ СИСТЕМЫ МОДИФИКАЦИИ ГИСТОНОВ
У бактерий и архей ДНК упаковывается белком HU, у эукариот и некоторых архей – гистонами. Однако структурная организация и функциональные возможности нуклеосом эукариот (управление экспрессией генов, сеграгацией хромосом и репарацией ДНК) значительно отличается от таковой у архей. Нуклеосомы архей значительно проще и представляют собой предковую частицу, состоящую из белков с гистоновыми доменами, которые не содержат хвостов и формируют тетрамерную частицу (у эукариот октамерная) лишь однократно обернутую цепочкой ДНК. Считается, что эукариотические нуклеосомы эволюционировали от археального предка путем дублирования количества субъединиц для обеспечения двойного обертывания ДНК и получения хвостов гистонов. Хотя ДНК обертывается правосторонней суперспиралью вокруг архейных нуклеосом, а у эукариот – левосторонней, структурная суперпозиция димерных субъединиц показывает, что эукариотические гистоны могли эволюционировать от более простых археальных предков [40].
Возникновение характерного для всех эукариот гистоновых комплексов, формирующих универсальную нуклеосому, является важнейшим фактором в эволюции. У многоклеточных эукариот гистоновые мРНК не полиаденилированы, а заканчиваются консервативными вторичными структурами типа “стебель-петля”. Белок SLBP (stem-loop binding protein) связывается со структурами “стебель-петля” и используется для всех этапов метаболизма гистоновых мРНК. Гистоновые мРНК быстро деградируют при ингибировании репликация ДНК по 3'–5' пути, при котором требуется обширное уридилирование промежуточных продуктов распада мРНК [73]. Для генов гистонов характерны отсутствие интронов и кластерная организация в виде тандемных повторов [71]. Указанные особенности гистонов позволяют предположить роль ТЕ в их возникновении и формировании в эволюции с сохранением в отборе наиболее оптимальных вариантов, ставших консервативными для всех эукариот. Хотя, несмотря на консервативность гистонов, у многоклеточных эукариот два семейства, гистон H2A и гистон Н3, значительно диверсифицировали по ключевым остаткам. При филогенетическом анализе по линии зеленых растений показана ранняя диверсификация семейства Н2А у одноклеточных зеленых водорослей и значительные экспансия Н2А вариантов у цветковых растений [50]. Новые варианты гистонов были выявлены у множества видов, и многие сходные с гистонами последовательности были аннотированы в качестве псевдогенов в геномных базах данных, однако их тканеспецифическая экспрессия была подтверждена путем секвенирования 3'-нетранслируемых областей (3'UTR) транскриптов. Предполагается, что разнообразие вариантов Н3, приобретенных после разделения видов, сыграло роль в регулировании тканеспецифической экспрессии генов отдельных видов [72]. Канонические белки гистонов кодируются репликационно-зависимыми генами (организованными в тандемные кластеры) и должны быстро достигать высоких уровней экспрессии в S фазу. Напротив, синтез вариантов репликационно-независимых, не организованных в кластеры и содержащих интроны, кодируемых полиаденилированными мРНК, происходит во все фазы клеточного цикла [71]. Это говорит о продолжающемся эволюционном отборе функциональных взаимосвязей ретрокопий генов гистонов, сформированных при помощи РЭ.
Можно предположить, что обширная колонизация транспозонами геномов при возникновении эукариот способствовала различным комбинациям с образованием ретрогенов, среди которых оказались гистоновые. Оптимальная комбинация гистонов, образованных при помощи РЭ, дала возможность возникновению нового универсального многофункционального комплекса, в дальнейшем принимающего важнейшее участие во всех преобразованиях геномов. У эукариот октамерный комплекс нуклеосом (в отличие от архей) имеет важное значение в регуляции экспрессии генов. Гистон Н3.3 обычно распределяется по транскрипционно активным генам и несет модификации, связанные с активацией, такие как Н3K4me3, тогда как Н3.1 и Н3.2 распределены по другим частям генома и связаны с подавлением экспрессии. Также известно, что Н3.3 включается в промоторные области до активации транскрипции в клеточной дифференцировке комплексами гистоновых шаперонов, включая HIRA и Chd1. Интересно, что Н3.3 также вовлечен в сайленсинг генома путем включения в перицентромерный гетерохроматин и теломерные области, во взаимосвязи с DAXX/ATRX. В данных случаях селективное включение гистоновых вариантов может быть молекулярной платформой последующих модификаций и ремоделирования хроматина для получения потенциала дифференцировки [72].
ФОРМИРОВАНИЕ СИСТЕМЫ УПРАВЛЕНИЯ СПЛАЙСИНГОМ
Известно минимум четыре класса интронов: 1) интроны ядерных белок-кодирующих генов, которые удаляются сплайсосомой (сплайсосомные интроны); 2) интроны тРНК, которые удаляются белками; 3) самосплайсируемые интроны группы I и 4) группы II, удаляемые РНК катализом [1]. Сплайсосомные интроны являются вездесущими элементами ядерных геномов – их эволюционный рост связан с происхождением эукариот. Хотя интроны не кодируют белок, они являются неотъемлемой частью регуляции экспрессии генов. Некоторые интроны сами кодируют функциональные РНК путем дальнейшего процессинга после сплайсинга интронных нуклеотидных последовательностей. Были обнаружены ассоциации тысяч нкРНК – snoРНК, микроРНК, siРНК, piРНК и длинных нкРНК с интронами, имеющие ярко выраженный синергетический эффект для тонкой настройки паттернов генной экспрессии во всем геноме [82].
Интроны обеспечивают альтернативный сплайсинг (АС) молекул пре-мРНК, что способствует увеличению как белкового разнообразия, так и специализации внутри протеома. Кроме того, некоторые интроны играют важную роль в широком спектре регуляторных функций экспрессии генов, таких как нонсенс-опосредованный распад и экспорт мРНК. Исследования геномов широкого круга организмов показали, что интрон-экзонная структура гомологичных генов разных организмов широко варьирует, а длина и плотность интронов может значительно различаться даже между родственными видами. Например, у одноклеточных грибов Encephalitozoon cuniculi всего 15 интронов на весь геном, что в среднем составляет 0.0075 интронов на ген, тогда как у человека в геноме 139 418 интронов – в среднем 8.4 интрона на ген [75]. Очевидна положительная корреляция между количеством интронов и нкРНК со сложностью организмов, тогда как количество БКГ относительно постоянно от червей до человека [82].
Мобильные интроны и их хоминг-эндонуклеазы были открыты еще в 70-х гг. XX в. Мобильность самосплайсируемых интронов группы I и интеинов обеспечивается кодируемыми ими сайт-специфическми эндонуклеазами, способными выявлять безинтронный аллель и вносить в него разрыв, репарация которого производится по интрон-содержащему аллелю, приводя к генной конверсии [89]. Интроны группы II встречаются во всех доменах жизни и поэтому считаются исключительно успешными мобильными элементами. Они обнаруживаются у бактерий, хлоропластов и митохондрий растений и грибов, но отсутствуют в ядерных геномах. Предполагается, что двойственность интронов группы II – способность выполнять как реакции самосплайсинга, так и ретромобильности, сыграла ключевую роль в эволюции эукариот. Эти интроны, вероятно, обеспечили основу для появления эукариотических РЭ, сплайсосомных интронов и других ключевых компонентов сплайсосомы [81, 93]. Выделяют три большие подгруппы интронов группы II с последующим подразделением по распределению между 8 филогенетическими линиями. Каждая подгруппа характеризуется специфическими вторичными структурами РНК и особенностями сплайсинга и мобильности. В частности, хотя бактериальные интроны обычно высоко активны и функциональны, интроны эукариотических органелл зачастую не имеют важных вторичных доменов и/или кодируют дегенеративные IEP (intron-encoded protein). Важно отметить, что сплайсинг этих вырожденных интронов зависит от действия транс-действующих РНК других интронов или кодируемых хозяином белков [42].
Наиболее объяснимой причиной отсутствия интронов группы II в ядерной ДНК и их преобразование в сплайсосомные может быть преимущество последних в возможности использования сплайсосомы и АС для глобальных регуляторных программ управления экспрессией генов и онтогенезом. Этим объясняется общая зависимость эукариот от сплайсосомных интронов [93]. Сплайсосома состоит из пяти мяРНП (малых ядреных рибонуклеопротеинов U1, U2, U3, U4, U5) вместе с многочисленными менее стабильно связанными белками. Ядро сплайсосомы консервативно для всех эукариот. Сплайсосома взаимодействует со специфическими сайтами интронов и фланкированных экзонов для обеспечения точного и эффективного сплайсинга. Нуклеотиды на концах интронов и смежные нуклеотиды экзонов вовлечены в данные взаимодействия, предоставляя сигналы сплайсинга. Последовательности [A/C]AG|GU[A/G]AGU (сайт сплайсинга показан вертикальной полосой, а первые два нуклеотида интрона подчеркнуты) на донорском сигнале сплайсинга комплементарны 5' концу U1 мяРНК, и данное взаимодействие, по-видимому, является основным требованием для сплайсинга. Последовательности [C,U]AG|G (последние два нуклеотида интрона подчеркнуты) предшествуют полипиримидиновому тракту, типичному для акцепторного сплайсингового сигнала и распознаются U5 мяРНК [84].
Предполагается, что появление эукариот на ранних этапах эволюции связано с инвазией интронов, так как последний общий предок эукариот (LECA – last eukaryotic common ancestor) характеризовался чрезвычайной плотностью интронов в геноме, сходной с современными богатыми интронами геномами. Из этой богатой интронами LECA различные линии эукариот проявляли четкие эволюционные пути, ведущие к глубоко расходящимся современным структурам геномов [42]. Согласно многочисленным сравнительным исследованиям ортологичных генов, интроны могут теряться и вновь обретаться в течение эволюции. Известно два точных механизма потери интронов: опосредованная RT потеря интрона и геномные делеции [84]. Обретение новых интронов происходит различными путями: транспозицией, инсерциями ТЕ, дублированием тандемных повторов, переносом интронов, инсерцией интронов группы II, получением интрона при репарации разрывов двойной спирали (DSBR). Наиболее распространенным способом появления новых интронов, как предполагается, является транспозиция интрона [98]. Таким образом, основные пути распространения интронов связаны с ТЕ. Роль ТЕ в регуляции экспрессии генов при инсерции в интроны или при образовании новых интронов путем ретротранспозиции связана с тем, что в ТЕ содержатся последовательности донорных и акцепторных сайтов сплайсинга [17]. Выявлено также множество высококонсервативных ТЕ в качестве энхансеров транскрипции и альтернативных сплайсинговых экзонов, способствующих гомеостазу мРНК в клетке [35]. При этом сами ТЕ могут служить источниками сплайсинговых энхансеров и сайленсеров – малых РНК длиной 10 нуклеотидов, управляющих сплайсингом путем связывания с SR-белками или мяРНП [57]. Сплайсосомные интроны играют важную роль в АС (подробнее в обзоре [7]), обеспечивая грандиозное белковое разнообразие при сохранении значительно меньшего количества БКГ. Так, около 95% мультиэкзонных генов млекопитающих проявляют АС [42].
ФОРМИРОВАНИЕ ГЕНОВ ПУТЕМ ЭКЗОНИЗАЦИИ ТРАНСПОЗОНОВ
Перемещения ТЕ часто служат в качестве движущей силы для превращения интронов в экзоны или для получения новых интронов, а также в качестве альтернативных сплайсинговых транскриптов. Это способствует белковому разнообразию [94]. Новые сплайсинговые варианты уже существующих генов имеют важное значение в усложнении высших животных и растений. Этим объясняется, например, менее выраженные вариации в количестве хромосом у высших растений (2n от 10 до 50) по сравнению с папоротниками и мхами (2n более 500) [2], так как у последних большее значение в эволюции играет WGD (whole-genome duplication – полногеномная дупликация). Несмотря на относительно малое число хромосом, геномы высших растений характеризуются значительными размерами, что говорит о важной роли ТЕ в их преобразованиях и в регуляции сплайсинга. Действительно, только LTR-содержащие РЭ у высших растений занимают до 91% генома у Asparagus officinalis, более 76% – у Hordeum vulgare, до 58% – у Allium cepa, 55% – у Zea mays, что превышает долю всех ТЕ в геномах большинства животных [53]. Экзонизацией называют процесс приобретения новых экзонов генами из некодирующих, прежде всего интронных, последовательностей. Точковые мутации или инсерции (а также мутации инсертированных ТЕ) могут образовывать сайты АС, вызывая включение новых последовательностей нуклеотидов в качестве экзонов. Если образующийся в результате вариант АС является предпочтительным, отбор может работать для оптимизации новых сайтов сплайсинга (обычно являющихся минорными фракциями) и увеличения их пропорции или даже полной замены новыми вариантами [87]. Таким образом, ТЕ способствуют белковому разнообразию и могут быть экзонизированы. В данном отношении чрезвычайно активными в эволюции являются неавтономные SINE (short interspersed nuclear elements – короткие диспергированные элементы ядра), которые при инсерции в антисмысловой ориентации в транскрибируемую область становятся источниками множества латентных сигналов сплайсинга. Выявлено, например, что около 5% всех внутренних альтернативных экзонов в геноме человека образовались путем активности латентных сигналов в процессе, названном экзонизацией Alu [91]. В интронах Arabidopsis thaliana выявлено более 3% ТЕ, сохранение которых указывает на их адаптационное значение [56]. Помимо SINE, L1-элементы также содержат доноры и акцепторы сайтов сплайсинга и сигналы полиаденилирования, используемые в транскрипции и способствующие экспрессии генов и образованию гибридных L1-транскриптов [17]. Одним из объяснений экзонизации может быть способность продуктов трансляции ТЕ образовывать ДНК-связывающие домены и другие пространственные структуры, позволяющие белку обретать новые свойства [35].
3'-UTR большинства БКГ и генов длинных нкРНК (lncРНК – long noncoding РНК) расщепляются и полиаденилируется. Недавние открытия показали, что значительная часть генов содержат более одного сайта полиаденилирования – альтернативные варианты являются широко распространенным явлением. Альтернативное полиаденилирование способствует усложнению транскриптома путем образования изоформ, которые различаются либо их кодирующей последовательностью, либо их 3'-UTR, что потенциально регулирует эффективность, стабильность, локализацию и значимость трансляции целевых РНК. 3'-UTR играют важную роль в регуляции экспрессии генов. Так, более 50% генов человека приводят к появлению множества изоформ с альтернативным 3'-UTR, тем самым способствуя тканеспецифической и стадиеспецифической регуляции транскриптов, которые в противном случае кодировали бы белок с одной и той же функцией [31]. У транскриптов 3'-UTR служат важными концентраторами посстранскрипционной регуляции экспрессии гена. Обнаружено, что, помимо экзонизации ТЕ, инсертированных в интроны, экзонизация межгенных ТЕ в ходе эволюции эукариот способствовала образованию новых терминальных экзонов и сайтов полиаденилирования [91]. Например, у Leishmania major почти 1000 копий семейства РЭ LmSIDER2 локализованы в 3'-UTR различных мРНК, что говорит о глобальной функции в посттранскрипционной регуляции [35].
ВОЗНИКНОВЕНИЕ ГЕНОВ ПУТЕМ ДУПЛИКАЦИИ
РЭ являются важной эволюционной силой для создания новых и потенциально функциональных безинтронных генов (ретрогенов) с видоспецифическими фенотипическими проявлениями их функционирования. Ретрогены могут быть реактивированы при помощи предполагаемых промоторов и других транскрипционных регуляторных элементов, подходящим образом расположенными вверх по течению от сайта их инсерции. Многие ретрогены функциональны. Например, в геноме человека не менее 1000 транскрибируемых ретрокопий, более десятой части из которых биологически активны. Многие ретрогены экспрессируются в яичках и их продукты активно участвуют в сперматогенезе и других уникальных функциях мужской зародышевой линии [41]. Эволюционный смысл сохранения ретрогенов в геномах эукариот может быть связан с наличием фланкирующих их последовательностей, происходящих от ТЕ, что позволяет включать данные копии генов в новые регуляторные сети, управляемые с участием ТЕ, что служит динамическим источником новых адаптационных свойств. Например, у дрозофилы выявлено 15 полиморфных химерных ретрокопий, фланкированных LTR. У млекопитающих ретрокопии, полученные с помощью RT автономных ретроэлементов LINE-1, содержат специфичные для LINE-1 последовательности в их фланкирующих областях: сайт расщепления эндонкулеазы TTTT/AA и поли(А) хвост [90]. У растений ретрогены фланкированы главным образом LTR, но могут встречаться также последовательности non-LTR РЭ [104].
Дупликация генов ответственна за увеличение разновидностей белков в эволюции. В среднем около 10% БКГ содержат не менее 1 ретрокопии в геноме, хотя в реальности их гораздо больше, так как фактическое количество не определяется из-за 5'-усечения при ретротранспозиции [38]. Образование ретрокопий и их сохранение в эволюции логично в связи с возможностью их регуляции, так как они содержат последовательности РЭ – новые взаимосвязи в генных сетях могут способствовать лучшей адаптации при отборе. При этом ретрогены могут кодировать белки, сохранившие функцию родительского гена (субфункционализация), развить новую (неофункционализация) или даже функционально заместить родительский ген [53]. Дупликация генов с помощью РЭ характерна для всех эукариот. Помимо этого, у растений большое значение приобрело WGD в результате слияния нередуцированных гамет в процессе мейотической реституции, приводящее к полиплоидизации при межвидовой и межродовой гибридизации [6]. В связи с этим растения отличаются более выраженной изменчивостью числа хромосом от 2n = 4 у Halopappus gracilis до 2n = = 1440 у Ophioglossum reticulatum [2]. У животных полиплоидизация вызывает нарушение хромосомного механизма определения пола, поэтому данный процесс не имел важного значения в их эволюции – для большинства видов характерно партеногенетическое размножение [68]. Филогенетический анализ показал, что эволюция и диверсификация семенных растений произошла как следствие двух древних событий WGD – одно предшествовало расхождению голосеменных и покрытосеменных, а другое – диверсификации покрытосеменных [44]. Несмотря на это, содержание ТЕ в геномах растений значительно превышает таковое у животных и грибов, достигая 90% всех последовательностей ДНК. Например, у Asparagus officinalis только LTR-элементы занимают до 91% генома [53]. Данный феномен можно объяснить важной ролью ТЕ в перестройке регуляции новых генных сетей, возникающих в результате удвоения геномного материала.
Таким образом, активность ТЕ имеет ключевое значение в возникновении новых генов путем дупликаций, создавая измененный генетический материал, содержащий регуляторные последовательности, произошедшие от РЭ. Одним из объяснений важнейшей роли РЭ в формировании новых генов может быть усиленный мутагенез РНК по сравнению с ДНК (выше на несколько порядков, составляя 10–3–10–4), в результате чего обратно транскрибированная последовательность имеет большую вероятность изменения, проходя в дальнейшем эволюционный отбор на пригодность [68]. Помимо этого, ТЕ способны изменять ретрогены путем повторных инсерций в них с последующей экзонизацией. Например, выявлены ретрокопии, комбинирующие последовательности двух и более родительских генов, что говорит о наличии механизмов перетасовки экзонов при помощи ретротранспозиций [90]. Экзонизация последовательностей ТЕ является частым событием при транскрипции ретрогенов в раннем эмбриональном развитии и может способствовать посттранскрипционной регуляции экспрессии ретрогенов [41].
ОДОМАШНИВАНИЕ ТРАНСПОЗОНОВ
ТЕ были и остаются богатыми источниками материала для сборки и изменений регуляторной системы эукариотических генов. Многие промоторы и сигналы полиаденилирования происходят от видоспецифических ТЕ [35]. Обширное распространение ТЕ в геномах, как предполагается, было основной причиной происхождения и эволюции эукариот. Причем, помимо регуляторной функции, гены ТЕ стали использоваться геномами хозяев для собственных нужд. Во многих случаях благодаря данным генам таксоны обретали универсальные консервативные свойства. Пример наиболее древнего одомашнивания ТЕ с формированием универсального консервативного механизма, присущего всем эукариотам – возникновение теломеразы от обратной РЭ [17, 23], что привело к универсальному свойству геномов всех эукариот – линейной структуре ДНК с теломерами на концах. Теломеры отвечают за фиксацию хромосом к ядерной оболочке, сегрегацию и стабилизацию поврежденных хромосом, управление экспрессией генов [14]. Они состоят из повторяющихся участков ДНК и G-цепи – одноцепочечного 3'-выступающего участка, который образует t-петлю путем переплетения с двухцепочечным участком [12]. Другой пример – одомашнивание гена env гликопротеина оболочки эндогенных ретровирусов, способствовавшее образованию трофобласта на поверхности матки при беременности у млекопитающих [30].
Любая из активностей или доменов, кодируемых белками ТЕ, способна к одомашниванию. Наиболее склонными к этому оказались транспозазы, что было замечено при первоначальном анализе генома человека при идентификации 47 генов целиком или главным образом произошедших от кодирующих транспозонами последовательностей. Причем 43 из них оказались произошедшими от транспозаз, несмотря на то, что ДНК-транспозоны составляют лишь 7% всех ТЕ человека. Даже при использовании строгих критериев для оценки функциональности генов, полученных от ТЕ, у животных, грибов и растений, выявляются множество различных функциональных для хозяина белков, произошедших от ТЕ [35]. При исследовании наборов генов позвоночных животных было обнаружено, что около 1000 БКГ произошли от РЭ, а фрагменты ТЕ способствуют образованию не менее 4% существующих кодирующих областей [99]. При полногеномном анализе ДНК человека было идентифицировано 85 генов, кодирующих 103 белковые изоформы, напоминающие ретровирусные белки Gag. Данные гены были одомашнены из РЭ по меньшей мере в пяти независимых событиях во время эволюции позвоночных и впоследствии были дуплицированы у млекопитающих. Гомологи Gag у млекопитающих выполняют важные функции, такие как регуляция апоптоза и связывание с убиквитинлигазой [20].
Наследуемая способность транспозаз образовывать петли и узлы ДНК в области их присоединения предрасполагает к их вербовке в качестве белков, которые упаковывают и организуют геном в функционально независимые домены хроматина подобно белкам “инсуляторам”. Действительно, BEAF-32 – белок инсулятор дрозофилы, который связывает пограничный хроматиновый элемент scs и хроматин с ядерным матриксом, полностью кооптирован от транспозазы hAT. У делящихся дрожжей центромерсвязывающие белки, вовлеченные в хромосомную сегрегацию, Abp1 и два его паралога, известные как CENP-B гомологи, произошли от pogo-подобных транспозаз линии грибов. Согласно последним данным, CENP-B делящихся грибов взаимодействует с ТЕ и способствует их связыванию с периферией ядра. Более того, Abp1 непосредственно взаимодействует с гистоновыми деацетилазами и направляет их к связанной с ними ДНК, тем самым вызывая локальное зарождение гетерохроматина и подавляя транскрипцию соседних генов в нескольких хромосомных локусах [35]. Суперсемейство ДНК-транспозонов PIF/IS5, известное также как Harbinger, впервые открытое у кукурузы, выявлено в геномах многих цветковых растений, некоторых грибов и разнообразных животных (нематоды, комары, морские ежи, рыбы). Распространенность и консервативность данного ТЕ говорит о возможностях его использования хозяином для создания регуляторных последовательностей и БКГ. Так, PIF-подобная транспозаза была завербована у общего предка позвоночных для создания нового гена HARBI1, который активно экспрессируется у птиц и млекопитающих [24]. Растения, благодаря одомашниванию древней Mutator-подобной транспозазы, обрели транскриционные факторы FHY3 и FAR1, позволяющие регулировать реакцию на свет [62]. Было идентифицировано экспрессируемое семейство транспозаз-подобного гена hobo/Ac/Tam [hAT] у злаковых трав. Данный ген, названный gary, найден в количестве 1–2-х копий у ячменя, 2-х расходящихся копий у риса и 2-х сходных копий у гексаплоидной пшеницы. У всех трех видов злаковых присутствует полная открытая транспозаз-подобная рамка считывания размером в 2.5 килобаз. Экспрессия gary обнаружена также у овса, ржи, кукурузы, сорго и сахарного тростника. Генетическое картирование показало, что gary находился в одном локусе генома около 60 млн лет и потерял способность к перемещению, однако экспрессирует транспозаз-связанный белок [76]. При исследовании эволюционной истории химерного гена приматов SETMAR, выявлено, что он образован в результате слияния гена гистоновой метилтрансферазы SET с геном транспозазы ТЕ. Ген транспозазы был завербован как часть SETMAR около 40 миллионов лет назад после инсерции ТЕ Hsmar1 ниже ранее существовавшего гена SET, за которым следует de novo экзонизация ранее некодирующей последовательности и создание нового интрона [25]. Лизиновая метилтрансфераза SETMAR участвует в реакции на повреждение и репарацию ДНК и связана с диметилированием лизина 36 в гистоне Н3 (Н3K36) в местах повреждения ДНК. Кроме того, SETMAR метилирует Lys-130 в мяРНП70, который является ключевым в регуляции выбора 5'-сайта сплайсинга, а метилирование мяРНП70 при помощи SETMAR участвует в управлении конститутивным и альтернативным сплайсингом [21].
Одомашнивание ТЕ часто сопряжено с образованием белков, способствующих глобальным преобразованиям в регуляции генов и в особенностях онтогенетического развития, являясь источником мощной эволюционной изменчивости. Например, белки HDP1 и HDP2 транспозонного происхождения (совместно одомашнены из транспозазы и ДНК-связывающего белка, кодируемых транспозоном Harbinger) функционируют в качестве ацетилтрансферазного комплекса гистонов для активного деметилирования ДНК как факторы антисайленсинга у арабидопсиса [29]. Центромерсвязывающие белки, вовлеченные в хромосомную сегрегацию, CENP-B, произошли от pogo-подобной транспозазы у грибов [35, 99]. Инсуляторы (регуляторные элементы, способствующие организации хроматина эукариот) также могут быть результатом одомашнивания ТЕ. Например, MIR-инсуляторы рекрутируют транскрипционные комплексы и ферменты модификации хроматина у человека для модуляции генных регуляторных сетей [95]. Учитывая важное значение ТЕ в качестве источников микроРНК [18, 37, 60, 65, 83] и длинных нкРНК [47, 49], управляющих экспрессией генов на транскрипционном и посттранскрипционном уровнях, полученные данные говорят о важнейшей роли ТЕ одновременно с образованием новых генов, в формировании регуляторной сети геномов у эукариот.
ТРАНСЛЯЦИЯ ФУНКЦИОНАЛЬНЫХ ПЕПТИДОВ ИЗ НЕКОДИРУЮЩИХ РНК
Возрастает количество доказательств того, что ранее аннотированные lncРНК обладают способностью кодировать пептиды [69], хотя они не содержат длинные или консервативные ORF. Еще в 2006 г. Levine et al. описали примеры образования новых БКГ у дрозофилы за счет отбора возможных вариантов экспрессии некодирующих РНК [59]. В 2008 г. Cai et al. обнаружили новый БКГ у Saccharomyces cerevisiae, который, вероятно, был сформирован из некодирующей последовательности [19]. В 2012 г. Carvunis et al. на дрожжах продемонстрировали трансляцию коротких ORF, расположенных в некодирующих областях [22]. В 2012 г. Xie et al. обнаружили 24 специфических для гоминидов белок-кодирующих генов, образованных de novo из lncРНК [96]. Оказалось, что образование новых БКГ из некодирующих последовательностей относительно распространенное явление у всех эукариот. В 2014 г. Ruiz et al. при исследовании Mus musculus, Homo sapiens, Danio rerio, D. melanogaster, A. thaliana, S. cerevisiae, обнаружили, что большая часть lncРНК, экспрессируемая в клетках шести разных видов, связана с рибосомами. При этом lncРНК проявляли сходный потенциал кодирования с эволюционно молодыми белок-кодирующими последовательностями, свидетельствуя о том, что они играют важную роль в de novo эволюции белков [85].
РНК, сама по себе являющаяся продуктом транскрипции, служит основным регулятором транскрипционного процесса. В частности, lncРНК функционируют путем связывания с гистон-модифицирующими комплексами, с ДНК-связывающими белками (включая ТФ), и даже с РНК-полимеразой-II. Некоторые lncРНК могут функционировать на РНК-уровне, например, как рибозимы или рибосвитчи. Однако чаще всего lncРНК выполняют свои функции в составе РНП. Предполагается несколько путей регуляции транскрипции при помощи lncРНК: вербовка регуляторных белковых комплексов in cis, ингибирование связывания транскрипционного регуляторного фактора, косвенно через акт транскрипции и косвенно через организацию генома и архитектуры ядра [64]. Предполагается, что lncРНК обладают модульной организацией, подобно белкам, состоящим из дискретных доменов, комбинация которых определяет их функцию. При этом фрагменты ТЕ внутри lncРНК способствуют их функционированию в качестве РНК-, ДНК- и белок-связывающих доменов. Это отражает более общий феномен экзаптации во время эволюции lncРНК, когда инсертированные последовательности ТЕ переориентируются в качестве сайтов распознавания как для белков, так и для нуклеиновых кислот [47].
У млекопитающих пептид миорегулин (MLN) образуется при трансляции lncРНК и выполняет контроль эффективности мышц, тонко регулируя поглощение кальция через физическое взаимодействие с кальциевой АТФазой SERCA [16]. Была обнаружена также специфичная для мышц lncРНК, кодирующая пептид из 34 аминокислот, названный DWORF (dwarf open reading frame). DWORF локализуется в мембране SERCA (саркоплазматический ретикулум), усиливая ее активность путем вытеснения ингибиторов SERCA, фосфоламбана, сарколипина и миорегулина [77]. Пептиды, кодируемые генами lncРНК, оказывают ингибирующее или стимулирующее воздействие на целевые гены у млекопитающих [69]. Функционально охарактеризованные кодируемые генами lncРНК пептиды у растений включают ENOD40 (ранний нодулин 40, который необходим для симбиотического взаимодействия растений и бактерий), IPS1 (индуцированный фосфатным голоданием-1, участвующий в поглощении фосфатов), LDMAR (контролирует чувствительную к фотопериодам мужскую стерильность), COOLAIR и COLDAIR (управляют временем цветения арабидопсиса, влияя на транскрипцию FLC) [101]. Некоторые lncРНК могут быть прекурсорами малых РНК, таких как piРНК, snoРНК и микроРНК. Например, lncРНК BORDERLINE является прекурсором малой РНК, вовлеченной в демаркацию эпигенетически различного хромосомного домена S. pombe [49].
Пример саморегуляции lncРНК продуктами собственной трансляции с учетом активности самих lncРНК либо в качестве рибозимов либо в комплексе с белками показывает механизм, по которому могли образовываться первые признаки живого. Принцип, по которому миллионы лет отбирались молекулы функциональных РНК в качестве источников белка на заре зарождения жизни действует до сих пор. У эукариот геном характеризуется динамичностью, обусловленной транспозициями TE, что дает основу для формирования множества новых нкРНК, продукты транскрипции которых, обретая пространственную конфигурацию, проходят отбор по “поиску” возможных мишеней для взаимодействия одновременно с отбором их продуктов трансляции. Наиболее оптимальные комбинации сохраняются в борьбе за существование при условии приобретения адаптивных признаков. Отличием эукариот является большая степень мобилизации TE у всех представителей, наиболее объяснимой причиной которой могло быть воздействие экстремальных природных факторов на заре зарождения предка всех эукариот – в экстремальных условиях наибольший потенциал выживания получали те особи, которые характеризовались возможностью быстрого приобретения новых признаков за счет мобилизации ТЕ с образованием новых конфигураций РНК и белков с адаптивными функциями. Предполагается, что эукариоты произошли от архей, для которых характерно обитание в экстремальных условиях. Археи имеют сходные с эукариотами, а не с бактериями аппарат репликации и репарации ДНК, а также компоненты транскрипционного комплекса [13].
У растений выявлено, что микроРНК тоже могут кодировать пептиды, обозначаемые miPEP, которые усиливают транскрипцию связанных с ними микроРНК [26]. При-микроРНК содержат короткую открытую рамку считывания последовательностей, кодирующих регуляторные пептиды. То есть при-микроРНК могут как транслироваться в пептиды, так и образовывать путем процессинга зрелые микроРНК. Pri-miR171b у Medicago truncatula и pri-miR165a у Arabidopsis thaliana продуцируют пептиды, обозначенные как miPEP171b и miPEP165a, которые усиливают аккумулирование соответствующих им зрелых микроРНК. Помимо этого, у A. thaliana и M. truncatula выявлено еще 5 микроРНК, кодирующих активные miPEP. При исследовании Arabidopsis выявлено, что по меньшей мере каждая из 50 при-микроРНК содержит одну короткую открытую рамку считывания (smORF), используемую для трансляции в пептид [55]. Пептиды miPEP являются функциональными и могут способствовать накоплению ассоциированных с ними при-микроРНК и, в конечном счете, к подавлению целевых генов. Природная экспрессия miPEP может быть обнаружена с использованием специфических антител, а их паттерны экспрессии сходны с таковыми для соответствующих микроРНК [55]. Существование эндогенных miPEP было экспериментально продемонстрировано с использованием иммуноблота, репортерного анализа GUS и исследований сверхэкспрессии для miPEP171b и miPEP165a. Трансляция pri-miPEP171b и pri-miPEP165a была подтверждена профилированием рибосом. Идентификация miPEP с использованием только вычислительного прогнозирования является сложной задачей. Как было показано, для обнаружения кодируемых smORF, комбинация подходов включает высокопроизводительное секвенирование РНК (RNA-seq), профилирование рибосом, протеомные и биоинформационные исследования [69]. Поскольку miPEP специфически способствуют транскрипции соответствующих им при-микроРНК, которые приводят к подавлению целевых генов, они представляют собой эффективное средство для изучения соответствующих им семейств микроРНК и повышения урожайности в агрономических культурах. Например, экзогенное применение синтетического miPEP172c, который стимулирует экспрессию miR172c, в конечном итоге способствует формированию узелков (симбиоз-специфические органы для взаимодействия с ризобиями) у сои. В связи с этим miPEP могут быть использованы в качестве альтернативных инструментов для оптимизации агрономических характеристик сельскохозяйственных культур [27].
ТЕ являются важными источниками микроРНК [18, 37, 60, 65, 83], а также вносят сигналы, необходимые для биогенеза многих lncРНК, в то же время являясь основой структуры самих генов lncРНК. Транспозоны составляют не менее 30% общих последовательностей и более 2/3 зрелых транскриптов lncРНК [48], играя ключевую роль в формировании их функциональных доменов. Более того, сами ТЕ могут использоваться в качестве генов lncРНК, продукты транскрипции которых регулируют дифференцировку клеток. Например, у человека HERVH экспрессируется в lncРНК, которая поддерживает идентичность эмбриональных стволовых клеток [67]. Так как в эволюции lncРНК могут быть источниками формирования белков de novo, логично предположить, что ТЕ играют ключевую роль в качестве важного источника преобразования геномов путем образования новых регуляторных структур и генов, а также динамической настройки их взаимосвязей (рис. 3).
ЗАКЛЮЧЕНИЕ
Таким образом, в возникновении и эволюции генов ключевую роль играют гармоничные взаимоотношения между существующими белок-кодирующими последовательностями и некодирующими областями ДНК, происходящими от ТЕ. Данные взаиморегуляторные сети имеют глобальный характер и способствуют возникновению новых белок-кодирующих генов и регуляторных сетей в естественном отборе. У прокариот наиболее удачным универсальным глобальным механизмом, способствующим их выживанию и приобретению новых признаков, стала система, обеспечивающая взаимопереход генов базового набора со вспомогательными за счет РЭ, ДНК-транспозонов, плазмид, интегронов, CRISPR и бактериофагов. Возникновению эукариот от архей, характеризующихся обитанием в агрессивной среде, способствовало ужесточение условий существования, при которых отбирались особи с максимальным количеством вариантов регуляторных взаимосвязей и возможностью образования новых белков и РНК-рибозимов. Этому способствовало глобальное распространение в геномах ТЕ. Результатом множественных инсерций стало разобщение генов (составляющих оперон у прокариот), участвующих в единых биохимических реакциях. Однако появлялись все новые регуляторные сети и универсальные защитные системы хозяев, настраиваемые при участии ТЕ и систем их контроля. Данные системы включают РНК-интерференцию, образование теломер и нуклеосом, управление сплайсингом, метилированием и модификациями гистонов. В результате кооперации данных систем сформировалась динамичность геномов, позволившая эволюционировать в огромное разнообразие форм жизни за счет формирования из ТЕ как регуляторных последовательностей, так и белков путем экзонизации и дупликации генов, одомашнивания ТЕ, преобразования генов нкРНК (источниками которых являются транспозоны) в белок-кодирующие.
Список литературы
Альбертс Б. Основы молекулярной биологии клетки; пер. с англ. М.: БИНОМ. Лаборатория знаний, 2015. 768 с.
Бадаева Е.Д., Салина Е.А. Структура генома и хромосомный анализ растений // Вавиловский журн. генетики и селекции. 2013. Т. 17. С. 1017–1042.
Бондарев И.Э., Хавинсон В.Х. Подавление альтернативного механизма удлинения теломер в раковых клетках с помощью ингибиторов обратной транскриптазы // Успехи геонтол. 2016. Т. 29. № 2. № 218–221.
Ванюшин Б.Ф. Эпигенетика сегодня и завтра // Вавиловский журн. генетики и селекции. 2013. Т. 17. № 4/2. С. 805-832.
Ильина Т.С. Мобильные ISCR-элементы: структура, функции и роль в создании, наращивании и распространении блоков бактериальных генов множественной резистентности к антибиотикам // Молекулярная генетика, микробиология и вирусология. 2012. V. 4. С. 3–13.
Логинова Д.Б. Молекулярно-цитогенетический анализ мейотических механизмов восстановления фертильности у пшенично-ржаных гибридов (ABDR, 4x = 28). Дисс. ... к.б.н. Новосибирск, 2016. 153 с.
Мустафин Р.Н., Хуснутдинова Э.К. Роль интронов в эволюции // Успехи физиологических наук. 2015. № 1. С. 90–102.
Мустафин Р.Н. Роль вирусов в эволюции геномов // Успехи физиологических наук. 2016. № 2. С. 77–97.
Мустафин Р.Н., Хуснутдинова Э.К. Роль мобильных элементов генома в онтогенезе // Успехи физиологических наук. 2016. № 3. С. 109–135.
Мустафин Р.Н., Хуснутдинова Э.К. Взаимосвязь эпигенетических факторов в механизмах старения и малигнизации // Успехи физиологических наук. 2017. № 3. С. 92–120.
Мустафин Р.Н., Хуснутдинова Э.К. Общие универсальные генетические закономерности, лежащие в основе возникновения и эволюции многоклеточности // Успехи физиологических наук. 2018. Т. 49. № 2. С. 87–116.
Павлов К.И., Мухин В.Н., Клименко В.М., Анисимов В.Н. Система теломера-теломераза и психические процессы при старении, в норме и патологии // Успехи геронтол. 2017. № 1. С. 17–26.
Равин Н.В., Шестаков С.В. Геном прокариот. Вавиловский журн. генетики и селекции. 2013 // Т. 17. № 4/2. С. 972-984.
Рунов А.Л., Вонский М.С., Михельсон В.М. Уровень метилирования ДНК и длина теломер как основа для построения модели биологических часов старения // Цитология. 2015. Т. 57. № 3. С. 192–196.
Шестаков С.В. Роль архей в происхождении эукариот // Экологическая генетика. 2017. Т. 15. № 4. С. 52–59.
Anderson D.M., Anderson K.M., Cang C.L. et al. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance // Cell. 2015. V. 160. № 4. P. 595–606.
Belancio V.P., Roy-Engel A.M., Deininger P.L. All y’all need to know ‘bout retroelements in cancer // Semin. Cancer Biol. 2010. V. 20. P. 200–210.
Borchert G.M., Holton N.W., Williams J.D. et al. Comprehensive analysis of microRNA genomic loci identifies pervasive repetitive-element origins // Mobile Genetic Elements. 2011. V. 1. P. 8–17.
Cai J., Zhao R., Jiang H. Wang W. De novo origination of a new protein-coding gene in Saccharomyces cerevisiae // Genetics. 2008. V. 179. P. 487–496.
Campillos M., Doerks T., Shah P.K., Bork P. Computational characterization of multiple Gag-like human protein // Trends Genet. 2006. V. 22. № 11. P. 585-9.
Carlson S.M., Moore K.E., Sankaran S.M. et al. Proteomic Strategy Identifies Lysine Methylation of Splicing Factor snRNP70 by the SETMAR Enzyme // J. Biol. Chem. 2015. V. 290. № 19. P. 12040-7.
Carvunis A.R., Rolland T., Wapinski I. et al. Proto-genes and de novo gene birth // Nature. 2012. V. 487. P. 370–374.
Casacuberta E. Drosophila: Retrotransposons Making up Telomeres // Viruses. 2017. V. 9. № 7. pii:E192. )https://doi.org/10.3390/v9070192
Casola C., Lawing A.M., Betran E., Feschotte C. PIF-like transposons are common in drosophila and have been repeatedly domesticated to generate new host genes // Mol. Biol. Evol. 2007. V. 24. № 8. P. 1872-88.
Cordaux R., Udit S., Batzer M.A., Feschotte C. Birth of a chimeric primate gene by capture of the transposase gene from a mobile element // Proc. Natl. Acad. Sci. USA. 2006. V. 103. № 21. P. 8101–8106.
Couzigou J.M., Lauressergues D., Becard G., Comier J.P. miRNA-encoded peptides (miPEPs): A new tool to analyze the role of miRNAs in plant biology // RNA Biol. 2015. V. 12. P. 1178-80.
Couzigou J.M., Andre O., Cuillotin B. et al. Use of microRNA-encoded peptide miPEP172c to stimulate nodulation in soybean // New Phytol. 2016. V. 211. P. 379–381.
de Souza F.S., Franchini L.F., Rubinstein M. Exaptation of transposable elements into novel cis-regulatory elements: is the evidence always strong // Mol. Biol. Evol. 2013. V. 30. P. 1239–1251.
Duan C.G., Wang X., Pan L. et al. A pair of transposon-derived proteins function in a histone acetyltransferase complex for active DNA demethylation // Cell. Res. 2017. V. 27. № 2. P. 226–240.
Dupressoir A., Lavialle C., Heidmann T. From ancestral infectious retroviruses to bona fide cellular genes: role of the captured syncytins in placentation // Placenta. 2012. V. 33. № 9. P. 663-71.
Elkon R., Ugalde A.P., Agami R. Alternative cleavage and polyadenylation: extent, regulation and function // Nat. Rev. Genet. 2013. V. 14. № 7. P. 496–506.
Elliott T.A., Gregory T.R. Do larger genomes contain more diverse transposable elements // BMC Evol. Biol. 2015. V. 15. P. 69–81. (a).
Elliott T.A., Gregory T.R. What’s in a genome? The C-value enigma and the evolution of eukaryotic genome content // Philos. Trans. R. Soc. Lond. B. Biol. Sci. 2015. V. 370. № 1678. P. 20140331. doi . (b).https://doi.org/10.1098/rstb.2014.0331
Fellner L., Simon S., Scherling C. et al. Evidence for the recent origin of a bacterial protein-coding, overlapping orphan gene by evolutionary overprinting // BMC Evol. Biol. 2015. V. 15. P. 283. doi 10. 1186/s12862-015-0558-z
Feschotte C. Transposable elements and the evolution of regulatory networks // Nat. Rev. Genet. 2008. V. 9. № 5. P. 397–405.
Garavis M., Gonzalez C., Villasante A. On the origin of the eukaryotic chromosome: the role of noncanonical DNA structures in telomere evolution // Genome Biol. Evol. 2013. V. 5. P. 1142–1150.
Gim J., Ha H., Ahn K. et al. Genome-Wide Identification and Classification of microRNAs derived from repetitive elements // Genomic Inform. 2014. V. 12. P. 261–267.
Grandi F.C., Rosser J.M., Newkirk S.J. et al. Retrotransposition creates sloping shores: a graded influence of hypomethylated CpG islands on flanking CpG sites // Genome Res. 2015. V. 25. № 8. P. 1135-46.
Henaff E., Vives C., Desvoyes B. et al. Extensive amplification of the E2F transcription factor binding sites by transposons during evolution of Brassica species // Plant J. 2014. V. 77. P. 852–862.
Henikoff S., Smith M.M. Histone variants and epigenetics // Cold. Spring Harb. Perspect. Biol. 2015. V. 7. № 1. :a019364. https://doi.org/10.1101/cshperspect.a019364
Huang C.J., Lin W.Y., Chang C.M., Choo K.B. Transcription of the rat testis-specific Rtdpoz-T1 and –T2 retrogenes during embryo development: co-transcription and frequent exonisation of transposable element sequences // BMC Mol. Biol. 2009. V. 10. P. 74–88.
Irimia M., Roy S.W. Origin of spliceosomal introns and alternative splicing // Cold. Spring Harb. Perspect. Biol. 2014. V. 6. № 6. pii: a016071.
Ito J., Suqimoto R., Nakaoka H. et al. Systematic identification and characterization of regulatory elements derived from human endogenous retroviruses // PLoS Genet. 2017. V. 13. e1006883.
Jamsheer M., Mannully C.T., Gopan N., Laxmi A. Comprehensive Evolutionary and Expression Analysis of FCS-Like Zinc finger Gene Family Yields Insights into Their Origin, Expansion and Divergence // PLoS One. 2015. V. 10. № 8. e0134328.
Jacques P.E., Jeyakani J., Bourgue G. The majority of primate-specific regulatory sequences are derived from transposable elements // PLoS Genet. 2013. V. 9. e1003504.
Jjingo D., Conley A.B., Wang J. et al. Mammalian-wide interspersed repeat (MIR)-derived enhancers and the regulation of human gene expression // Mob. DNA. 2014. V. 5. P. 5–14.
Johnson R., Guigo R. The RIDL hypothesis: transposable elements as functional domains of long noncoding RNAs // RNA. 2014. V. 20. № 7. P. 959–976.
Kapusta A., Kronenberg Z., Lynch V.J. et al. Transposable elements are major contributors to the origin, diversification, and regulation of vertebrate long noncoding RNAs // PLoS Genet. 2013. V. 9. № 4. e1003470.
Kapusta A., Feschotte C. Volatile evolution of long noncoding RNA repertoires: mechanisms and biological implications // Trends Genet. 2014. V. 30. № 10. P. 439-52.
Kawashima T., Lorkovic Z.J., Nishihama R. et al. Diversification of histone H2A variants during plant evolution // Trends Plant. Sci. 2015. V. 20. № 7. P. 419-25.
Kopera H.C., Moldovan J.B., Morrish T.A. et al. Similarities between long interspersed element-1 (LINE-1) reverse transcriptase and telomerase // Proc. Natl. Acad. Sci. USA. 2011. V. 108. P. 20345–20350.
Kralovicova J., Patel A., Searle M., Vorechovsky I. The role of short RNA loops in recognition of a single-hairpin exon derived from a mammalian-wide interspersed repeat // RNA Biology. 2015. V. 12. № 1. P. 54–69.
Kubiak M.R., Makalowska I. Protein-Coding Genes’ Retrocopies and Their Functions // Viruses. 2017. V. 9. pii: E80.
Lampson B.C., Inouye M., Inouye S. Reverse transcriptase with concomitant ribonuclease H activity in the cell-free synthesis of branched RNA-linked msDNA of Myxococcus xanthus // Cell. 1989. V. 56. № 4. P. 701-7.
Lauressergues D., Couzigou J.M., Clemente H.S. et al. Primary transcripts of microRNAs encode regulatory peptides // Nature. 2015. V. 520. № 7545. P. 90-3.
Le T.N., Miyazaki Y., Takuno S. et al. Epigenetic regulation of intragenic transposable elements impacts gene transcription in Arabidopsis thaliana // Nucleic Acids Res. 2015. V. 43. № 8. P. 3911-21.
Lei H., Vorechovsky I. Identification of splicing silencers and enhancers in sense Alus: a role for pseudoacceptors in splice site repression // Mol. Cell. Biol. 2005. V. 25. № 16. P. 6912-20.
Lescale C., Deriano L. The RAG recombinase: Beyond breaking // Mich. Ageing. Dev. 2016. № 16. P. 30263-9. https://doi.org/10.1016/j.mad.2016.11.003
Levine M.T., Jones C.D., Kern A.D. et al. Novel genes derived from noncoding DNA in Drosophila melanogaster are frequently X-linked and exhibit testis-biased expression // Proc. Natl. Acad. Sci. USA. 2006. V. 103. P. 9935–9939.
Li Y., Li C., Xia J., Jin Y. Domestication of transposable elements into microRNA genes in plants // PLoS One. 2011. V. 6. e19212.
Lim D., Maas W.K. Reverse transcriptase-dependent synthesis of a covalently linked, branched DNA-RNA compound in E. coli B // Cell. 1989. V. 56. № 5. P. 891–904.
Lin R., Ding L., Casola C. et al. Transposase-derived transcription factors regulate light signaling in Arabidopsis // Sciende. 2007. V. 318. P. 1302-5.
Liu M., Deora R., Doulatov S.R. et al. Reverse transcriptase-mediated tropism switching in Bordetella bacteriophage // Science. 2002. V. 295. № 5562. P. 2091-4.
Long Y., Wang X., Youmans D.T., Cech T.R. How do lncRNAs regulate transcription? // Sci. Adv. 2017. V. 3. № 9. eaao2110.
Lorenzetti A.P., Antonio G.Y., Paschoal A.R., Domingues D.S. Plant TE-MIR DB: a database for transposable element-related microRNAs in plant genomes // Funct. Integr. Genomics. 2016. V. 16. P. 235–242.
Lowe C.B., Haussler D. 29 mammalian genomes reveal novel exaptations of mobile elements for likely regulatory functions in the human genome // PLoS One. 2012. V. 7. e43128.
Lu X., Sachs F., Ramsay L. et al. The retrovirus HERVH is a long noncoding RNA required for human embryonic stem cell identity // Nat. Struct. Mol. Biol. 2014. V. 21. P. 423–425.
Lukash L.L. Mutagenesis induced by integration processes and evolution of nuclear genome // Biopolymers and cell. 2007. V. 23. № 3. P. 172–187.
Lv S., Pan L., Wang G. Commentary: Primary Transcripts of microRNAs Encode Regulatory Peptides // Front Plant Sci. 2016. V. 7. P. 1436.
Mak K.S., Burdach J., Norton L.J. et al. Repression of chimeric transcripts emanating from endogenous retrotransposons by a sequence-specific transcription factor // Genome Biol. 2014. V. 15. R 58.
Marzluff W.F., Wagner E.J., Duronio R.J. Metabolism and regulation of canonical histone mRNAs: life without a poly(A) tail // Nat. Rev. Genet. 2008. V. 9. № 11. P. 843–854.
Maehara K., Harada A., Sato Y. et al. Tissue-specific expression of histone H3 variants diversified after species separation // Epigenetics Chromatin. 2015. V. 8. P. 35. https://doi.org/10.1186/s13072-015-0027-3
Marzluff W.F., Koreski K.P. Birth and Death of Histone mRNAs // Trends Genet. 2017. V. 33. № 10. P. 745–759.
Morita S., Horii T., Kimura M. et al. MiR-29 Represses the Activities of DNA Methyltransferases and DNA Demethylases // Int. J. Mol. Sci. 2013. V. 14. P. 14 647–14 658.
Mourier T., Jeffares D.C. Eukaryotic intron loss // Science. 2003. V. 300. № 5624. P. 1393.
Muehlbauer G.J., Bhau B.S., Syed N.H. et al. A hAT superfamily transposase recruited by the cereal grass genome. Mol. Genet. Genomics // 2006. V. 274. № 6. P. 553-63.
Nelson B.R., Makarewich C.A., Anderson D.M. et al. A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle // Science. 2016. V. 351. № 6270. P. 271-5.
Nelson-Sathi S., Sousa F.L., Roettger M. et al. Origins of major archaeal clades correspond to gene acquisitions from bacteria // Nature. 2015. V. 517. № 7532. P. 77–80.
Nishihara H., Smit A., Okada N. Functional noncoding sequences derived from SINEs in the mammalian genome // Genome Res. 2006. V. 16. № 7. P. 864-74.
Notwell J.H., Chung T., Heavner W., Bejerano G. A family of transposable elements co-opted into developmental enhancers in the mouse neocortex // Nat. Commun. 2015. V. 6. P. 6644.
Novikova O., Belfort M. Mobile Group II Introns as Ancestral Eukaryotic Elements // Trends Genet. 2017. pii: S0168-9525(17)30126-9. https://doi.org/10.1016/j.tig2017.07.009
Rearick D., Prakash A., McSweeny A. et al. Critical association of ncRNA with introns // Nucleic Acids Res. 2011. V. 39. № 6. P. 2357–2366.
Roberts J.T., Cooper E.A., Favreau C.J. Formation from transposable element insertions and nonboding RNA mutations // Mobile Genetic Elements. 2013. V. 1. № 6. e27755.
Rogozin I.B., Carmel L., Csuros M., Koonin E. Origin and evolution of spliceosomal introns // Biology Direct. 2012. V. 7. P. 11.
Ruiz-Orera J., Messeguer X., Subirana J.A., Alba M.M. Long non-coding RNAs as a source of new peptides // Elife. 2014. V. 3. e03523. https://doi.org/10.7554/eLife.03523
Samantarrai D., Dash S., Chhetri B., Mallick B. Genomic and epigenomic cross-talks in the regulatory landscape of miRNAs in breast cancer // Mol. Cancer Res. 2013. V. 11. P. 315–328.
Schmitz J., Brosius J. Exonization of transposed elements: A challenge and opportunity for evolution // Biochimie. 2011. V. 93. № 11. P. 1928-34.
Shabalina S.A., Koonin E.V. Origins and evolution of eukaryotic RNA interference // Trends. Ecol. Evol. 2008. V. 23. P. 578-87.
Stoddard B.L. Homing endonucleases from mobile group I introns: discovery to genome engineering // Mob. DNA. 2014. V. 5. P. 7. https://doi.org/10.1186/1759-8753-5-7
Tan S., Cardoso-Moreira M., Shi W. et al. LTR-mediated retroposition as a mechanism of RNA-based duplication in metazoans // Genome Res. 2016. V. 26. № 12. P. 1663–1675.
Tajnik M., Vigilante A., Braun S. et al. Inergenic Alu exonisation facilitates the evolution of tissue-specific transcript ends // Nucleic Acids Res. 2015. V. 43. № 21. P. 10492-505.
Toro N., Nisa-Martinez R. Comprehensive phylogenetic analysis of bacterial reverse transcriptases // PLoS One. 2014. V. 9. № 11. e114083. https://doi.org/10.1371/journal.pone.0114083
Vesteg M., Sandorova Z., Krajcovic J. Selective forces for the origin of spliceosomes // J. Mol. Evol. 2012. V. 74. P. 226–231.
Wang D., Su Y., Wang X. et al. Transposon-Derived and Satellite-Derived Repetitive Sequences Play Distinct Functional Roles in Mammalian Intron Size Expansion // Evol. Bioinform. Online. 2012. V. 8. P. 301-19.
Wang J., Vicente-Garcia C., Seruqqia D. et al. MIR retrotransposons sequences provide insulators to the human genome // Proc. Natl. Acad. Sci. USA. 2015. V. 112. № 32. P. 4428-37.
Xie C., Zhang Y.E., Chen J.Y. et al. Hominoid-specific de novo protein-coding genes originating from long non-coding RNAs // PLoS Genet. 2012. V. 8. e1002942.
Xu C., Tian J. SiRNA-mediated DNA methylation and H3K9 dimethylation in plants // Protein Cell. 2013. V. 4. P. 656–663.
Yenerall P., Zhou L. Identifying the mechanisms of intron gain: progress and trends // Biol. Direct. 2012. V. 7. P. 29. https://doi.org/10.1186/1745-6150-7-29
Zdobnov E.M., Campillos M., Harrington E.D. et al. Protein coding potential of retroviruses and other transposable elements in vertebrate genomes // Nucleic Acids Res. 2005. V. 33. P. 946-54.
Zhang Z., Saier M.H.Jr. Transposon-mediated adaptive and directed mutations and their potential evolutionary benefits // J. Mol. Microbiol. Biotechnol. 2012. V. 21. № 1–2. P. 59–70.
Zhang J., Mujahid H., Hou Y. et al. Plant Long ncRNAs: A New Frontier for Gene Regulatory Control // American J. Plant Sciences. 2013. V. 4. № 5. ID:32139.
Zhang G., Esteve P., Chin H.G. et al. Small RNA-mediated DNA (cytosine-5) methyltransferase 1 inhibition leads to aberrant DNA methylation // Nucleic Acids Res. 2015. V. 43. P. 6112–6124.
Zhang H., Tao Z., Hong H. et al. Transposon-derived small RNA is responsible for modified function of WRKY45 locus // Nat. Plants. 2016. V. 2. P. 16 016–1623.
Zhu Z., Tan S., Zhang Y., Zhang Y.E. LINE-1-like retrotransposons contribute to RNA-based gene duplication in dicots // Sci. Rep. 2016. V. 6. P. 24755.
Дополнительные материалы отсутствуют.
Инструменты
Успехи физиологических наук