Биоорганическая химия, 2022, T. 48, № 2, стр. 217-222
Тканеспецифическая экспрессия изоформ нейрексина-1α в органах крысы
О. В. Серова 1, *, Е. А. Ганцова 1, И. Е. Деев 1, 2, А. Г. Петренко 1
1 ФГБУН “Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова” РАН
117997 Москва, ул. Миклухо-Маклая, 16/10, Россия
2 Научно-технологический университет “Сириус”
354340 Сочи, Олимпийский просп., 1, Россия
* E-mail: oxana.serova@gmail.com
Поступила в редакцию 06.06.2021
После доработки 21.07.2021
Принята к публикации 24.07.2021
- EDN: HOABOF
- DOI: 10.31857/S0132342322020191
Аннотация
Нейрексины представляют собой семейство белков синаптической адгезии, которые играют ключевую роль в формировании и поддержании синапсов. Нейрексины подвергаются обширному альтернативному сплайсингу в шести сайтах (SS1–SS6), что приводит к экспрессии множества различных изоформ. Альтернативный сплайсинг регулирует функциональную активность нейрексинов в различных типах тканей и клеток и, предположительно, играет ключевую роль в определении специфичности взаимодействия различных нейронов. В этом исследовании мы провели анализ тканевой экспрессии изоформ мРНК нейрексина-1α, содержащих вставку в недавно обнаруженном сайте сплайсинга SS6, с использованием TaqMan ПЦР в реальном времени в различных органах крыс линии Wistar. Изоформа, содержащая вставку в сайте SS6, была обнаружена только в нейрональных тканях, что указывает на ее потенциальную функциональную важность. Положение вставки SS6 в шарнирной области между доменами LNS5 и LNS6 увеличивает вариабельность возможных конформаций молекулы, что может представлять собой дополнительный механизм регулирования функциональной активности нейрексина-1α в головном мозге.
ВВЕДЕНИЕ
Нейрексины (Nrxn1–3) представляют собой семейство белков синаптической адгезии, которые играют ключевую роль в формировании и стабилизации синапсов [1]. Нейрексины и их лиганды образуют сложные сети взаимодействия, опосредуя многие регуляторные функции. Нарушения в работе нейрексинов и взаимодействующих с ними белков вызывают расстройство аутистического спектра (РАС), шизофрению и умственную отсталость [2]. Нейрексины представляют собой мембранные белки 1-го типа и первоначально были обнаружены как рецепторы α-латротоксина [3]. Нейрексины кодируются тремя гомологичными генами Nrxn1–3. Каждый ген, в свою очередь, транскрибируется с двух независимых промоторов (α и β), что приводит к образованию длинной (α) и короткой (β) форм нейрексинов [4, 5]. Внеклеточная область α-нейрексинов состоит из трех модулей, содержащих два домена LNS (Laminin, Neurexin, Sex-hormone binding protein domain, который также называют доменом Laminin G), между которыми находится EGF-подобный домен. Внеклеточный фрагмент β-нейрексинов содержит только один домен LNS [4].
Нейрексины подвергаются обширному альтернативному сплайсингу, в результате чего экспрессируется множество различных изоформ. Альтернативный сплайсинг нейрексинов по-разному регулируется в областях мозга и зависит от стадии развития и синаптической активности [6, 7]. Считается, что альтернативный сплайсинг регулирует функциональную активность нейрексинов и, предположительно, играет ключевую роль в определении специфичности взаимодействия различных нейронов. Первоначально были известны пять сайтов сплайсинга (SS1–SS5) в α‑нейрексинах, два из которых (SS4 и SS5) также были обнаружены в β-нейрексинах. Позже был идентифицирован шестой сайт сплайсинга (SS6) нейрексина-1α (Nrxn1α), который расположен между пятым LNS и третьим EGF-подобным доменами. Вставка в этой области соответствует пептиду из 9 а.о. VALMKADLQ, который консервативен у животных [8, 9].
Первоначально было обнаружено, что гены Nrxn экспрессируются в головном мозге, но в более поздних исследованиях выявлена экспрессия Nrxn не только в нервных тканях. В меньших количествах экспрессия мРНК Nrxn1 была обнаружена в почках, печени, сердце, желудке и легких [10]. Нейрексины также были обнаружены в сосудистой системе [11] и в β-клетках поджелудочной железы [12], где они играют функциональную роль. Nrxn1α – компонент механизма регуляции экзоцитоза, он необходим для стыковки инсулиновых гранул с мембраной в β-клетках поджелудочной железы [13]. мРНК Nrxn3 была обнаружена в тканях легких, поджелудочной железы, сердца, плаценты, печени и почек. Более того, специфические варианты сплайсинга Nrxn3 были идентифицированы в сердце [14]. Неясно, что именно запускает экспрессию изоформ нейрексина в разных тканях.
Цель данной работы – анализ экспрессии нейрексина-1α с альтернативным сплайсингом по сайту SS6 с помощью TaqMan ПЦР в реальном времени в различных органах крысы для выявления потенциальной роли альтернативного сплайсинга в регуляции функциональной активности нейрексинов в различных тканях.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Поскольку нейрексины обнаруживаются не только в нервных тканях, мы исследовали экспрессию Nrxn1α со вставкой в недавно обнаруженном сайте сплайсинга SS6 в различных органах крыс линии Wistar с использованием анализа TaqMan ПЦР в реальном времени. Из четырех самок крыс были выделены органы: почки, печень, сердце, поджелудочная железа и головной мозг. Из каждого извлеченного органа крыс была выделена тотальная РНК. Головной мозг делили на три части: кору мозга, мозжечок и оставшуюся часть. Тотальную РНК из каждой части мозга выделяли по отдельности. С помощью реакции обратной транскрипции были получены образцы кДНК из различных органов крысы, которые далее анализировали методом TaqMan ПЦР в реальном времени.
Для обнаружения изоформ Nrxn1α (SS6+) и (SS6–) мы использовали одинаковые прямой праймер и зонд, но разные обратные праймеры (табл. 1). Один из них гибридизировался на границе экзонов 17 и 18, что позволило амплифицировать только фрагмент кДНК, содержащий вставку в SS6-сайте. Другой праймер отжигался на границе экзонов 16 и 18, что позволило получить фрагмент кДНК без вставки SS6. В качестве референсного гена использовали ген домашнего хозяйства Gapdh.
Таблица 1.
Последовательности олигонуклеотидных праймеров и зондов, использованных в TaqMan ПЦР в реальном времени
| Ген | Праймер | Последовательность (5'–3') |
|---|---|---|
| Nrxn1 | Nx_probe | FAM-TGATGCTCTTTTCTGCAATGGGCAAATTGAG-BHQ1 |
| Nx_fw | CTTTCAAGGTTGCCTGGCATCTGT | |
| Nx_(SS6+)_rev | GTTGTGCTGGGCCCTTGCAAG | |
| Nx_(SS6–)_rev | GTTGTGCTGGGCCCTTCGCAT | |
| Gapdh | Gapdh_probe | FAM-CCTGGAGAAACCTGCCAAGTATGATG-BHQ1 |
| Gapdh_fw | CATGGCCTTCCGTGTTCCTA | |
| Gapdh_rev | CGCCTGCTTCACCACCTTCT |
Изоформа рецептора, содержащая вставку SS6, была обнаружена только в головном мозге, тогда как вариант сплайсинга без вставки – во всех пяти анализируемых органах (рис. 1). В мозге уровень экспрессии изоформы нейрексина без вставки SS6 значительно превышает уровень экспрессии изоформы, содержащей вставку (рис. 1). Соотношение экспрессии этих двух изоформ в разных частях головного мозга также было разным. Наибольшее количество Nrxn1α (SS6–) было обнаружено в мозжечке, тогда как максимальная экспрессия Nrxn1α (SS6+) наблюдалась в коре головного мозга. Если общее обнаруженное в каждой части мозга количество Nrxn1α представить как 100%, то значения экспрессии изоформ (SS6–) и (SS6+) были бы следующими: 87 и 13% в коре головного мозга, 96 и 4% в мозжечке, 94 и 6% в остальной части мозга.
Рис. 1.
Анализ экспрессии изоформ Nrxn1α (SS6+) и (SS6–) в органах крыс линии Wistar (n = 4) с использованием TaqMan ПЦР в реальном времени. Отрезками отмечены значения среднеквадратической ошибки (SEM). ПЦР с каждым образцом кДНК проводили в трех повторностях. * p < 0.001. Цифрами обозначены органы: 1 – кора мозга, 2 – мозжечок, 3 – остальная часть мозга, 4 – сердце, 5 – почка, 6 – печень, 7 – поджелудочная железа.
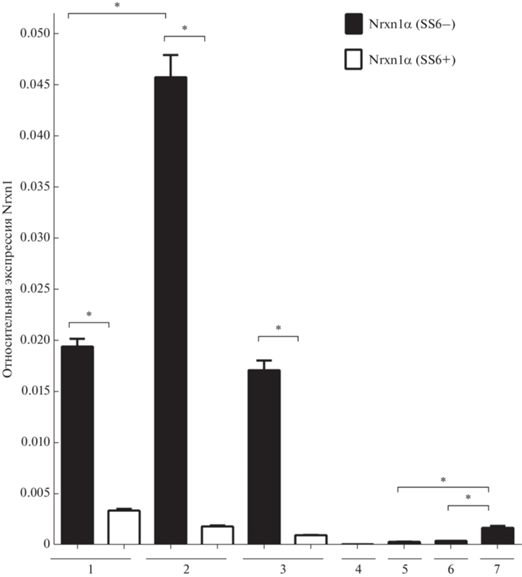
В отличие от изоформы Nrxn1α (SS6+), которая была обнаружена только в ткани головного мозга, изоформа (SS6–) экспрессировалась и в других тканях, таких как сердце, почки, печень и поджелудочная железа, но в гораздо меньших количествах. Среди последних названных органов наибольшее количество изоформы (SS6–) наблюдалось в поджелудочной железе.
Обнаруженная нами экспрессия гена Nrxn1 в ненейрональных тканях подтверждается современными литературными данными секвенирования РНК из различных органов. Согласно данным секвенирования РНК одиночных клеток, ген Nrxn1 экспрессируется в островках поджелудочной железы человека [15] , экспрессия Nrxn1 была выявлена в альфа-, бета-, дельта-, ацинарных клетках и клетках протоков поджелудочной железы (табл. 2). Также есть данные об экспрессии Nrxn1 в редких гамма-клетках поджелудочной железы [16]. Секвенирование РНК почки мыши выявило экспрессию Nrxn1 в клетках собирательных трубочек и в клетках толстой восходящей части петли Генле. Среднее значение экспрессии гена Nrxn1 в собирательных трубочках составило 1.8 транскриптов на миллион (TPM) [17].
Таблица 2.
Экспрессия гена Nrxn1 в различных типах клеток островков поджелудочной железы человека [15]
| Типы клеток островков поджелудочной железы | Значения экспрессии гена Nrxn1, TPM |
|---|---|
| Ацинарные клетки | 1.758 |
| Альфа-клетки | 2.718 |
| Бета-клетки | 2.343 |
| Дельта-клетки | 2.841 |
| Клетки протоков | 2.642 |
Альтернативный сплайсинг по-разному регулируется среди генов нейрексинов, несмотря на их гомологию. Каждая изоформа Nrxn демонстрирует уникальный профиль экспрессии в зависимости от области, типа клеток и сенсорной системы [18]. Механизм регуляции экспрессии изоформ нейрексина изучен недостаточно хорошо. Недавние исследования показывают, что альтернативный сплайсинг сайтов SS3 и SS4 регулируется нейрональной активностью [1]. Мы обнаружили изоформу Nrxn1α со вставкой в сайте SS6 только в головном мозге, тогда как вариант сплайсинга без вставки в SS6 был выявлен во всех протестированных органах. Наибольшее количество Nrxn1α (SS6–) было обнаружено в мозжечке, тогда как максимальная экспрессия Nrxn1α (SS6+) наблюдалась в коре головного мозга. Можно предположить, что альтернативный сплайсинг в сайте сплайсинга SS6 представляет собой механизм, контролирующий функциональную активность Nrxn1α в различных тканях.
Недавние исследования с использованием комбинации электронной томографии отдельных частиц (IPET), рентгеновской кристаллографии и малоуглового рентгеновского рассеяния (SAXS) показали, что эктодомен Nrxn1α принимает несколько дискретных конформаций [19]. Разнообразие наблюдаемых конформаций обеспечивается за счет двух основных шарнирных областей внутри Nrxn1α. Один из шарниров расположен между доменами LNS1 и LNS2, а другой – между доменами LNS5 и LNS6 [19]. Было показано, что вставка в сайте SS6, расположенная непосредственно в молекулярном шарнире между доменами LNS5 и LNS6, увеличивает вариабельность конформаций молекулы [19]. Можно предположить, что включение вставки SS6 может влиять на связывание белков-партнеров в синаптической щели, изменяя фактические сайты связывания или доступность к этим сайтам связывания. Сообщается, что вставка SS6 чувствительна к протеолизу. Таким образом, вероятная функция вставки SS6 может заключаться в том, чтобы сделать молекулу нейрексина-1α чувствительной к протеолизу, позволяя отщепляться области L1–L5 [19] с образованием секретируемой формы рецептора. Подобная растворимая секретируемая форма была обнаружена для рецептора Nrxn3 [20, 21]. Функциональное значение растворимой формы Nrxn3 подтверждается сниженной экспрессией и соотношением трансмембранных и растворимых изоформ Nrxn3 postmortem в мозге при болезни Альцгеймера [22].
Тканеспецифическая экспрессия различных изоформ Nrxn1α указывает на потенциальную роль альтернативного сплайсинга SS6 в регуляции функциональной активности Nrxn1α.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Эксперименты с животными. В экспериментах использовали самок крыс линии Wistar (n = 4) в возрасте 3–4 месяца (Питомник лабораторных животных “Столбовая”, Россия). Животные были здоровыми и содержались в стандартных условиях. Перед хирургическим удалением органов крыс анестезировали золетилом (20 мг/кг массы тела) и ксилазином (5 мг/кг массы тела) в 0.9%-ном растворе NaCl.
Органы крысы: почки, печень, поджелудочную железу, сердце и головной мозг – вырезали, каждый орган помещали в отдельную пробирку IKA DT-20-М (IKA, Германия) с TRIzol Reagent (Invitrogen, США) и сразу же целиком гомогенизировали с использованием гомогенизатора IKA Ultra-Turrax Tube Drive. Головной мозг делили на три части: кору головного мозга, мозжечок и оставшуюся часть мозга. Каждую часть мозга гомогенизировали по отдельности.
Получение кДНК. Тотальную РНК из органов крысы выделяли с помощью реагента TRIzol (Invitrogen, США) согласно протоколу производителя. Для удаления примесей геномной ДНК образцы тотальной РНК обрабатывали ДНКазой I (Thermo Scientific, США) согласно протоколу производителя. кДНК получали реакцией обратной транскрипции с использованием набора для обратной транскрипции RevertAid RT Reverse Transcription Kit (Thermo Scientific, США) и случайных гексамерных праймеров (random hexamer primer), входящих в состав набора, согласно протоколу производителя.
TaqMan ПЦР в реальном времени. Праймеры и специфический зонд для каждого конкретного гена были выбраны с использованием программ Gene Runner (http://www.generunner.net/), Oligo Explorer (https://www.genelink.com/tools/gl-oe.asp) и Oligo Analyzer (https://eu.idtdna.com/pages/tools/oligoanalyzer), специфичность праймеров и зондов подтверждали с помощью BLAST (https://blast.ncbi.nlm.nih.gov/). Ген Gapdh использовали в качестве референсного гена. Праймеры были синтезированы ЗАО “Евроген” (Россия), зонды – ООО “Люмипроб РУС” (Россия). Для обнаружения изоформ Nrxn1α (SS6+) и (SS6–) использовали одни и те же прямой праймер и зонд, но разные обратные праймеры. Зонд на 5'-конце содержал метку флуоресценции FAM (Fluorescein amidites), на 3'-конце – гаситель BHQ1 (Black Hole Quencher 1).
Для каждой пары праймеров рассчитывали эффективность реакции (Е) по формуле: E = 10 – 1/k, где k берется из уравнения прямой линии: CT = = klogP0 + b, в котором P0 – концентрация кДНК, СT – количество циклов, в которых флуоресценция достигает порогового уровня, а значения k и b получены из линейной аппроксимации экспериментальных данных. Эффективность реакции для генов-мишеней и референсного гена Gapdh составляла 2, что соответствует максимальной эффективности реакции.
TaqMan ПЦР в реальном времени с каждым образцом кДНК проводили в трех повторностях на приборе DTprime (ДНК-Технология, Россия) в 20 мкл реакционной смеси, использовали 1 ед. Taq ДНК-полимеразы с “горячим стартом” (ЗАО “Евроген”, Россия), концентрация праймеров составляла 0.4 мкМ, зонда – 0.2 мкМ. Параметры реакции: предварительная денатурация 95°С – 5 мин; 45 циклов амплификации: 95°С – 15 с, 60°С – 10 с, 72°С – 10 с.
Относительный уровень экспрессии (NE) вычисляли по формуле [23]:
Статистическую обработку данных ПЦР в реальном времени проводили с помощью программного обеспечения Prism 6 (GraphPad Software, США), используя критерий Стьюдента (t-тест). Статистически значимыми считали отличия при p < 0.05.
ЗАКЛЮЧЕНИЕ
Проведен подробный анализ экспрессии изоформ нейрексина-1α (Nrxn1α) с альтернативным сплайсингом по сайту SS6 с помощью TaqMan ПЦР в реальном времени в различных органах крыс линии Wistar. Показана тканеспецифичная экспрессия изоформ Nrxn1α. Изоформа, содержащая вставку в сайте SS6, была обнаружена только в нейрональных тканях, что указывает на ее потенциальную функциональную важность. Положение вставки SS6 в шарнирной области между доменами LNS5 и LNS6 увеличивает вариабельность возможных конформаций молекулы, что может представлять собой дополнительный механизм регулирования функциональной активности Nrxn1α в головном мозге.
Список литературы
Sudhof T.C. // Cell. 2017. V. 171. P. 745–769. https://doi.org/10.1016/j.cell.2017.10.024
Rudenko G. // Curr. Opin. Struct. Biol. 2019. V. 54. P. 112–121. https://doi.org/10.1016/j.sbi.2019.01.009
Petrenko A.G., Kovalenko V.A., Shamotienko O.G., Surkova I.N., Tarasyuk T.A., Ushkaryov Yu.A., Grishin E.V. // Embo J. 1990. V. 9. P. 2023–2027.
Ushkaryov Y.A., Petrenko A.G., Geppert M., Sudhof T.C. // Science. 1992. V. 257. P. 50–56. https://doi.org/10.1126/science.1621094
Geppert M., Ushkaryov Y.A., Hata Y., Davletov B., Petrenko A.G., Sudhof T.C. // Cold Spring Harb. Symp. Quant. Biol. 1992. V. 57. P. 483–490. https://doi.org/10.1101/sqb.1992.057.01.053
Ullrich B., Ushkaryov Y.A., Sudhof T.C. // Neuron. 1995. V. 14. P. 497–507. https://doi.org/10.1016/0896-6273(95)90306-2
Schreiner D., Nguyen T.M., Russo G., Heber S., Patrignani A., Ahrne E., Scheiffele P. // Neuron. 2014. V. 84. P. 386–398. https://doi.org/10.1016/j.neuron.2014.09.011
Serova O.V., Radionov N.V., Shayahmetova D.M., Deyev I.E., Petrenko A.G. // Dokl. Biochem. Biophys. 2015. V. 463. P. 239–242. https://doi.org/10.1134/S1607672915040110
Treutlein B., Gokce O., Quake S.R., Sudhof T.C. // Proc. Natl. Acad. Sci. USA. 2014. V. 111. P. E1291–E1299. https://doi.org/10.1073/pnas.1403244111
Saito A., Miyauchi N., Hashimoto T., Karasawa T., Han G.D., Kayaba M., Sumi T., Tomita M., Ikezumi Y., Suzuki K., Koitabashi Y., Shimizu F.,Kawachi H. // Am. J. Physiol. Regul. Integr. Comp. Physiol. 2011. V. 300. P. R340–R348. https://doi.org/10.1152/ajpregu.00640.2009
Bottos A., Destro E., Rissone A., Graziano S., Cordara G., Assenzio B., Cera M.R., Mascia L., Bussolino F., Arese M. // Proc. Natl. Acad. Sci. USA. 2009. V. 106. P. 20782–20787. https://doi.org/10.1073/pnas.0809510106
Suckow A.T., Comoletti D., Waldrop M.A., Mosedale M., Egodage S., Taylor P., Chessler S.D. // Endocrinology. 2008. V. 149. P. 6006–6017. https://doi.org/10.1210/en.2008-0274
Mosedale M., Egodage S., Calma R.C., Chi N.W., Chessler S.D. // J. Biol. Chem. 2012. V. 287. P. 6350–6361. https://doi.org/10.1074/jbc.M111.299081
Occhi G., Rampazzo A., Beffagna G., Antonio Danieli G. // Biochem. Biophys. Res. Commun. 2002. V. 298. P. 151–155. https://doi.org/10.1016/s0006-291x(02)02403-8
Li J., Klughammer J., Farlik M., Penz T., Spittler A., Barbieux C., Berishvili E., Bock C., Kubicek S. // EMBO Rep. 2016. V. 17. P. 178–187. https://doi.org/10.15252/embr.201540946
Segerstolpe A., Palasantza A., Eliasson P., Andersson E.M., Andreasson A.C., Sun X., Picelli S., Sabirsh A., Clausen M., Bjursell M.K., Smith D.M., Kasper M., Ämmälä C., Sandberg R. // Cell Metab. 2016. V. 24. P. 593–607. https://doi.org/10.1016/j.cmet.2016.08.020
Chen L., Lee J.W., Chou C.L., Nair A.V., Battistone M.A., Paunescu T.G., Merkulova M., Breton S., Verlander J.W., Wall S.M., Brown D., Burg M.B., Knepper M.A. // Proc. Natl. Acad. Sci. USA. 2017. V. 114. P. E9989–E9998. https://doi.org/10.1073/pnas.1710964114
Uchigashima M., Cheung A., Suh J., Watanabe M., Futai K. // J. Comp. Neurol. 2019. V. 527. P. 1940–1965. https://doi.org/10.1002/cne.24664
Liu J., Misra A., Reddy M., White M.A., Ren G., Rudenko G. // J. Mol. Biol. 2018. V. 430. P. 4325–4343. https://doi.org/10.1016/j.jmb.2018.08.026
Ushkaryov Y.A., Sudhof T.C. // Proc. Natl. Acad. Sci. USA. 1993. V. 90. P. 6410–6414. https://doi.org/10.1073/pnas.90.14.6410
Hishimoto A., Liu Q.R., Drgon T., Pletnikova O., Walther D., Zhu X.G., Troncoso J.C., Uhl G.R. // Hum. Mol. Genet. 2007. V. 16. P. 2880–2891. https://doi.org/10.1093/hmg/ddm247
Hishimoto A., Pletnikova O., Lang D.L., Troncoso J.C., Egan J.M., Liu Q.R. // Alzheimer’s Res Ther. 2019. V. 11. P. 28. https://doi.org/10.1186/s13195-019-0475-2
Simon P. // Bioinformatics. 2003. V. 19. P. 1439–1440. https://doi.org/10.1093/bioinformatics/btg157
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


