Журнал аналитической химии, 2021, T. 76, № 12, стр. 1100-1106
Определение максимальной длины днк, расшифровываемой с точностью 99%, в полимере на основе линейного N,N-полидиметилакриламида методом капиллярного гель-электрофореза с лазер-индуцируемой флуоресценцией
Д. В. Бочарова a, *, Я. И. Алексеев a, b, А. А. Волков c, Г. С. Лавров a, А. Г. Плугов b, И. А. Волков b, А. А. Чемигов b, Б. В. Бардин a, В. Е. Курочкин a
a Институт аналитического приборостроения Российской академии наук
198095 Санкт-Петербург, ул. Ивана Черных, 31/33, Россия
b ООО “Научно-производственная фирма Синтол”
127434 Москва, ул. Тимирязевская, 42, корп. Б, офис 316, Россия
c Всероссийский научно-исследовательский институт рыбного хозяйства и океанографии (ВНИРО)
107140 Москва, ул. В. Красносельская, 17, Россия
* E-mail: souldarydary@gmail.com
Поступила в редакцию 16.03.2021
После доработки 01.04.2021
Принята к публикации 02.04.2021
Аннотация
Расшифровка последовательности (секвенирование) ДНК – одна из важнейших задач современных молекулярно-генетических исследований. Точность расшифровки является основополагающим параметром секвенирования ДНК. В настоящей работе установлены максимальные длины фрагментов ДНК, полученные по методу Сенгера, расшифровываемые с точностью 99% в полимере на основе линейного N,N-полидиметилакриламида ПДМА-6 для капилляров длиной 50 см до оптического окна детектора на отечественном генетическом анализаторе Нанофор 05 и на импортном генетическом анализаторе GA 3500xL. Проведено сравнение результатов расшифровки последовательности ДНК при использовании различных алгоритмов анализа. Подобраны параметры алгоритмов анализа, которые позволят пользователям генетических анализаторов получать лучшие результаты расшифровки ДНК при использовании полимера ПДМА-6.
Расшифровка последовательности фрагментов ДНК по методу Сенгера [1] с использованием капиллярного гель-электрофореза является классической технологией с помощью которой были прочитаны первые геномы различных организмов, начиная с простейших фагов [2] и заканчивая геномом человека [3, 4]. Расшифровка генома любого организма позволяет понять его структурную организацию, выявить гены или группы генов, ответственные за те или иные функции и признаки исследуемого организма. В ходе популяционных исследований, представляющих собой сравнение множества геномов родственных организмов, удается выявлять более тонкие различия на уровне замены единичных нуклеотидов, которые также определяют разнообразие функций и признаков организмов. Так, например, в геноме человека размером шесть миллиардов пар нуклеотидов найдено более 25 тысяч генов, содержащих более трех миллионов однонуклеотидных замен, обусловливающих фенотипическое разнообразие вида Homo sapiens [5]. Таким образом, замена всего одного нуклеотида может изменять функцию изучаемого организма, поэтому точность расшифровки ДНК – один из важнейших параметров, требующих тщательной оценки [6]. Исторически массовое секвенирование геномов методом Сенгера началось с внедрением автоматических секвенаторов ДНК. Полученные с помощью этих приборов “сырые” данные требовали разработки и применения специального программного обеспечения, позволяющего расшифровать последовательности ДНК с высокой точностью, а также ввести критерии оценки точности. Так, с 1998 года использовалось программное обеспечение “Phred base calling” [7, 8]. Показатель качества Phred определяется как свойство, логарифмически связанное с вероятностями ошибок при присвоении пику на электрофореграмме соответствующего ему наименования нуклеотида A, T, G или С по характеристичному для каждого нуклеотида сигналу флуоресценции (цвет пика). Для расчета показателя качества используют следующую формулу [8]:
где P – вероятность ошибки при определении нуклеотида. В табл. 1 представлены варианты значений показателя качества Q, вероятности некорректного определения нуклеотида и достоверности секвенирования ДНК.Таблица 1.
Показатели качества Phred [8]
| Phred Quality Score, Q | Вероятность некорректного определения нуклеотида | Достовер- ность, % |
|---|---|---|
| 10 | 1 из 10 | 90 |
| 20 | 1 из 100 | 99 |
| 30 | 1 из 1000 | 99.9 |
| 40 | 1 из 10000 | 99.99 |
| 50 | 1 из 100000 | 99.999 |
Однако в 2010 году было статистически доказано превосходство алгоритма, реализованного в программном обеспечении KB™ Basecaller, разработанном производителем автоматических секвенаторов ДНК серии ABIPrizm компанией “Applied Biosystem” (сейчас “ThermoFisher Scientific”), в сравнении с “Phred base calling” [9]. С тех пор вышло несколько новых версий программы KB™ Basecaller, однако техническая часть алгоритма расшифровки последовательности ДНК до сих пор не опубликована.
В начале 2000-х годов в России были созданы первые отечественные приборы для капиллярного электрофореза серии “Нанофор”, предназначенные для автоматического количественного определения состава жидких проб практически любых неорганических и органических веществ, в том числе биологически активных [10]. Приборы Нанофор 01 со спектрофотометрическим детектором и Нанофор 02 с высокочувствительным флуоресцентным детектором выпускались малыми сериями в 2000–2005 гг., а в 2005 г. был разработан первый однокапиллярный автоматический секвенатор ДНК Нанофор 03-С. Прибор снабжен четырехканальным детектором лазер-индуцированной флуоресценции и устройством автоматического заполнения капилляра раствором разделяющего полимера. В 2011 году был создан первый отечественный 8-капиллярный секвенатор ДНК Нанофор 05 [11], предназначенный для решения задач определения длин фрагментов ДНК в исследуемом образце по стандарту с известными длинами (фрагментный анализ ДНК или генетическое типирование ДНК) [12] и расшифровки первичной структуры ДНК по методу Сенгера (секвенирование ДНК) [13]. Одновременно с разработкой прибора Нанофор 05 велась разработка собственного алгоритма расшифровки последовательности ДНК, реализованного в программе “ДНК Анализ”. Детальное описание данного алгоритма будет представлено в отдельной публикации.
Для эффективного решения широкого спектра пользовательских задач в области капиллярного гель-электрофоретического анализа нуклеиновых кислот необходимо использовать оптимизированные параметры программ электрофореза: напряжение, температуру, время и т.д. [14]. В данном исследовании в качестве полимера, разделяющего фрагменты ДНК, использовали коммерческий полимер на основе линейного N,N-полидиметилакриламида ПДМА-6, предназначенный для секвенирования ДНК.
Цель настоящего исследования – определение длины фрагмента ДНК, последовательность нуклеотидов в котором расшифровывается с точностью 99%, при разделении продуктов реакции секвенирования в полимере ПДМА-6 на приборах капиллярного гель-электрофореза с детекцией лазер-индуцированной флуоресценции с использованием различных алгоритмов анализа.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Реагенты. Полимер ПДМА-6 (ООО “НПФ Синтол”, Россия), формамид Hi-Di (ThermoFisher Scientific, США), буферный раствор для электрофореза ТАПС (ООО “НПФ Синтол”, Россия), спектральный калибратор BigDye Terminator v3.1 Matrix Standard (ThermoFisher Scientific, США), сиквенсный стандарт BigDye Terminator v3.1 Sequencing Standard (ThermoFisher Scientific, США).
Приборы и материалы. Капиллярный гель-электрофорез с детекцией лазер-индуцируемой флуоресценции проводили на генетических анализаторах Нанофор 05 (Институт аналитического приборостроения РАН, Россия) и 3500хL Genetic Analyzer (ThermoFisher Scientific, США). Для исследований на приборе Нанофор 05 использовали капилляры внутренним диаметром 50 мкм, внешним диаметром 192 мкм, длиной 60 см (длина до окна детектора – 50 см) (Molex, США). Капилляры были собраны в линейки из 8 капилляров каждая (Институт аналитического приборостроения РАН, Россия). Для исследования на 3500хL Genetic Analyzer использовали стандартную (50 см до оптического окна) 8-капиллярную линейку 3500 Genetic Analyzer 8-Capillary Array (ThermoFisher Scientific, США). Для денатурации продуктов реакции секвенирования использовали амплификатор GeneAmp 2720 Thermal Cycler (ThermoFisher Scientific, США). Для растворения и центрифугирования образцов использовали низкоскоростную микроцентрифугу-вортекс Циклотемп-901 (ООО “Циклотемп”, Россия). Для центрифугирования образцов в стрипах применяли низкоскоростную микроцентрифугу Циклотемп-903 (ООО “Циклотемп”, Россия).
Подготовка проб и анализ данных. Спектральный калибратор BigDye Terminator v3.1 Matrix Standard готовили следующим образом: 8 мкл раствора калибратора смешивали с 72 мкл деионизованного формамида и раскапывали по 10 мкл в каждую пробирку стрипа, состоящего из 8 пробирок объемом 0.2 мл. Закрывали крышки микропробирок и центрифугировали их в течение 2 мин. Затем стрипы помещали в амплификатор и денатурировали ДНК в следующих условиях: 95°C – 5 мин, 4°С – 5 мин. Затем центрифугировали пробирки в течение 2 мин со скоростью вращения 4500 об/мин.
Сиквенсный стандарт BigDye Terminator v3.1 Sequencing Standard готовили следующим образом: лиофилизированный сиквенсный стандарт растворяли в 300 мкл деионизованного формамида. Перемешивали на вортексе в течение 1 мин. Затем раскапывали по 10 мкл в каждую пробирку стрипа. Последующие действия осуществляли аналогично описанному выше протоколу подготовки образцов спектрального калибратора.
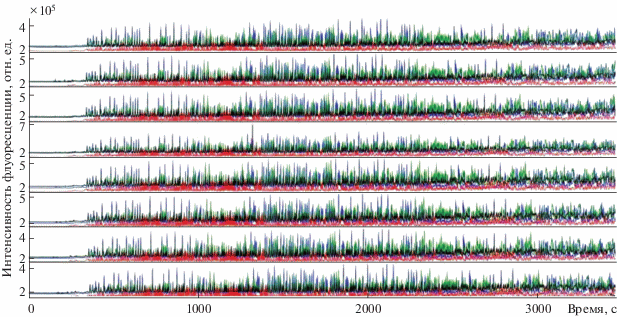
Гель-электрофоретическое разделение сиквенсного стандарта BigDye Terminator v3.1 проводили с помощью программы управления прибором Нанофор 05 – SeqPI версия 174 (Институт аналитического приборостроения РАН, Россия) и программы 3500 Data Collection Software v.1.0 (ThermoFisher Scientific, США) для прибора 3500xL Genetic Analyzer по стандартному протоколу: напряжение нанесения образца – 1.8 кВ, время нанесения 24 с, напряжение электрофореза 12.2 кВ. Пример разделения сиквенсного стандарта BigDye Terminator v3.1 в 8 капиллярах линейки на приборе Нанофор 05 приведен на рис. 1.
Рис. 1.
Результат электрофоретического разделения сиквенсного стандарта BigDye Terminator v3.1 на генетическом анализаторе Нанофор 05 по 8 капиллярам.
Экспериментальные данные, полученные на приборах Нанофор 05 и 3500xL Genetic Analyzer, анализировали с помощью программы “ДНК Анализ”, версия 5.0.4.3 (Институт аналитического приборостроения РАН, Россия) с визуализацией в программе “ПАР2СЕК”, версия 1.7.0 (Институт аналитического приборостроения РАН, Россия), а также с помощью программы Sequencing Analysis Software v6.0 Built Id:FC3 (ThermoFisher Scientific, США).
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
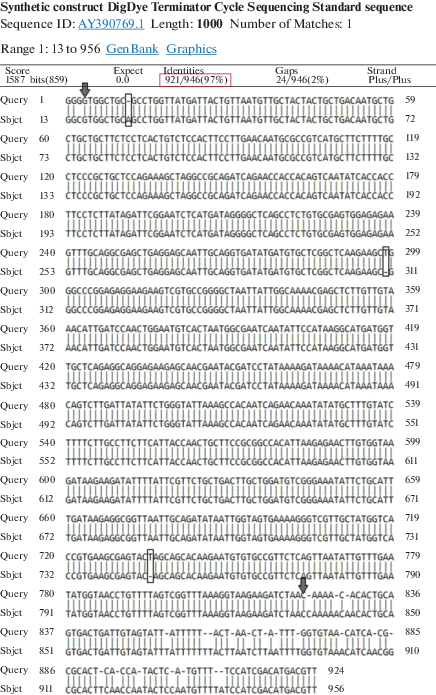
Точность секвенирования ДНК в каждом из 8 капилляров оценивали путем выравнивания расшифрованных последовательностей на референс – известную последовательность сиквенсного стандарта Synthetic construct BigDye Terminator Cycle Sequencing Standard sequence длиной 1000 нуклеотидов с помощью программы BLAST (Basic Local Alignment Search Tool) на сайте NCBI [14]. Экспериментально полученную последовательность (Query) выравнивали на референсную (Sbjct). Максимальную длину, соответствующую точности 99% (Phred Q20), рассчитывали как расстояние между первым из двух неверно определенных нуклеотидов с 5' конца и вторым из двух неверно определенных нуклеотидов с 3' конца. В обоих случаях в расчет принимали только те пары неверно определенных нуклеотидов, которые находились между собой на расстоянии, не превышающем 100 нуклеотидов. Пример определения максимальной длины фрагмента ДНК, последовательность нуклеотидов в котором расшифровывается с точностью 99%, приведен на рис. 2.
Рис. 2.
Результат выравнивания экспериментально полученной (Query) и референсной (Sbjct) последовательностей сиквенсного стандарта на сайте NCBI. Стрелками показаны первый (позиция 4) и последний (позиция 825) нуклеотиды, определяющие максимальную длину расшифровываемого с точностью 99% фрагмента ДНК. Прямоугольниками выделены ближайшие к ним внутри окна длиной в 100 нуклеотидов ошибки определения (в данном примере в позициях расшифрованной последовательности пропущен нуклеотид А (позиция 12 Query) и ошибочно определен нуклеотид Т (позиция 739 Query)).
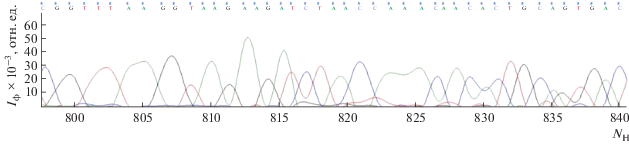
Для получения результата, приведенного на рис. 2, использовали данные, полученные на генетическом анализаторе Нанофор 05 и автоматически конвертированные в формат .ab1. Для данного образца максимальная длина расшифровки последовательности сиквенсного стандарта с точностью 99% (Phred Q20) составила 800 нуклеотидов при использовании алгоритма анализа KB версия 1.4.1.8 (ThermoFisher Scientific, США), файл мобильности KB_3130_POP6_BDTv3.mob, значение “Spacing” выставляли равным 24.0. Данные, полученные для этого же образца (в формате .srd) анализировали также в программе “ДНК Анализ”, версия 5.0.4.3 (Институт аналитического приборостроения РАН, Россия) и визуализированы в программе ПАР2СЕК (Институт аналитического приборостроения РАН, Россия). Величина максимальной, расшифровываемой с точностью 99%, длины сиквенсного стандарта составила 821 нуклеотид (рис. 3).
Рис. 3.
Электрофореграмма фрагмента расшифрованной последовательности сиквенсного стандарта BigDye Terminator v3.1 от 798 до 840 нуклеотида в капилляре А, выравненного на референсную последовательность синтетической конструкции Synthetic construct BigDye Terminator Cycle Sequencing Standard sequence длиной 1000 нуклеотидов, программа ПАР2СЕК.
В табл. 2 приведены результаты выравнивания на референс данных, полученных с помощью двух разных алгоритмов расшифровки последовательности ДНК. Последовательность 53 образцов расшифрована с помощью алгоритма, реализованного в программе “ДНК Анализ”, версия 5.0.4.3. Последовательность 61 образца расшифрована с помощью алгоритма KB версия 1.4.1.8 (файл мобильности KB_3130_POP6_BDTv3.mob, значение “Spacing” равно 24.0), реализованного в программе “Sequencing Analysis”, v. 6.0. Данные получены на приборе Нанофор 05 с использованием геля ПДМА-6 при напряжении электрофореза 12.2 кВ.
Таблица 2.
Результаты выравнивания экспериментально расшифрованных последовательностей сиквенсного стандарта на референсную последовательность, полученные на приборе Нанофор 05 и проанализированные разными алгоритмами
| Параметры расшифровки последовательности ДНК | ДНК Анализ, версия 5.0.4.3 | KB, версия 1.4.1.8 |
|---|---|---|
| Совпадение с референсной последовательностью, % | 97.2 ± 0.1* | 96.9 ± 0.2** |
| Максимальная длина расшифровки с точностью Q20*, нуклеотид | 798.4 ± 6.8* | 776.8 ± 3.2** |
Аналогичным образом были получены данные на приборе 3500xL Genetic Analyzer. Анализ с использованием алгоритма KB версия 1.4.1.8 (файл мобильности KB_3130_POP7_BDTv3.mob, значение “Spacing” равно 12.0) позволил установить следующие параметры расшифровки последовательности ДНК: совпадение с референсной последовательностью составило 97.9 ± 0.2%, максимальная длина расшифровки с точностью Q20 составила 813.4 ± 6.6 нуклеотидов (доверительный интервал при α = 0.05, P = 93).
Как видно из табл. 2, длина расшифрованной с точностью Q20 последовательности сиквенсного стандарта на приборе Нанофор 05, полученная с использованием алгоритма “ДНК Анализ”, версия 5.0.4.3 больше в среднем на 22 нуклеотида по сравнению с полученной с помощью алгоритма KB 1.4.1.8. Совпадение с референсной последовательностью также больше на 0.3% при обработке данных с использованием программы “ДНК Анализ”.
При обработке данных, полученных на приборе 3500xL GA, с помощью программы “Sequencing Analysis”, v. 6.0 отмечается увеличение длины расшифровываемой последовательности ДНК на 15 нуклеотидов по сравению с данными, полученными на приборе Нанофор 05 и обработанными алгоритмом “ДНК Анализ”, версия 5.0.4.3. Разница в совпадении с референсной последовательностью также оказалась незначительно (на 0.7%) лучше в случае данных, полученных на приборе 3500xL GA при обработке алгоритмом KB версия 1.4.1.8.
Данные, полученные в настоящей работе, авторы планируют использовать для дальнейшей оптимизации алгоритма расшифровки пиков в программе “ДНК Анализ” для полимера ПДМА-6. Кроме того, будет изучена возможность использования для секвенирования ДНК полимера ПД-МА-4, разделяющего фрагменты ДНК за более короткое время, который ранее применяли только для фрагментного анализа ДНК.
* * *
Установлены максимальные длины прочтения последовательности ДНК, полученные по методу Сенгера и расшифровываемые с точностью 99% в полимере на основе линейного N,N-полидиметилакриламида ПДМА-6 для капилляров длиной 50 см до оптического окна детектора на отечественном генетическом анализаторе Нанофор 05 и импортном генетическом анализаторе 3500xL Genetic Analyzer. Проведено сравнение максимальных значений длин ДНК, расшифровываемых с точностью Q20 для стандартных протоколов электрофоретического разделения продуктов реакции секвенирования ДНК. Полученные данные позволят пользователям генетических анализаторов получать лучшие результаты расшифровки последовательности ДНК с использованием полимера ПДМА-6.
Работа выполнена по Государственному заданию Министерства науки и высшего образования Российской Федерации № 075-00780-19-00 по теме № 0074-2019-0017.
Список литературы
Sanger F., Niclein S., Coulson A.R. DNA sequencing with chain-terminating inhibitors // Proc. Natl. Acad. Sci. USA. 1977. V. 74. P. 5463. https://doi.org/10.1073/pnas.74.12.5463
Sanger F., Air G.M., Barrell B.G., Brown N.L., Coulson A.R., Fiddes J.C., Hutchison C.A., Slocombe P.M., Smith M. Nucleotide sequence of bacteriophage phi X174 DNA // Nature. 1977. V. 265. № 5596. P. 687. https://doi.org/10.1038/265687a0
International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome // Nature. 2001. V. 409. P. 860. https://doi.org/10.1038/35057062
Venter J.C. et al. The sequence of the human genome // Science. 2001. V. 291. P. 1304. https://doi.org/10.1126/science.1058040
International HapMap Consortium. A second generation human haplotype map of over 3.1 million SNPs // Nature. 2007. V. 449. № 7164. P. 851. https://doi.org/10.1038/nature06258
Волков А.А., Волков И.А., Плугов А.Г., Кулябина Е.В., Мелкова О.Н., Лавров Г.С., Бочарова Д.В., Алексеев Я.И. Генетический анализатор Нанофор 05 в качестве средства измерений при секвенировании ДНК // Измерительная техника. 2021. № 1. С. 60. https://doi.org/10.32446/0368-1025it.2021-1-60-65
Ewing B., Hillier L., Wendl M.C., Green P. Base-calling of automated sequencer traces using Phred. I. Accuracy assessment // Genome Res. 1998. V. 8. P. 175.
Ewing B., Green P. Base-calling of automated sequencer traces using Phred. II. Error probabilities // Genome Research. 1998. V. 3. № 3. P. 186.
Hyman R.W., Jiang H., Fukushima M., Davis R.W. A direct comparison of the KB™ Basecaller and phred for identifying the bases from DNA sequencing using chain termination chemistry // BMC Research Notes. 2010. V. 3. № 257. https://doi.org/10.1186/1756-0500-3-257
Беленький Б.Г., Курочкин В.Е. Высокоэффективный капиллярный электрофорез и микрофлюидные чип-анализаторы. I. Приборы высокоэффективного капиллярного электрофореза // Научное приборостроение. 2002. Т. 12. № 4. С. 42.
Алексеев Я.И., Белов Ю.В., Малюченко О.П., Монахова Ю.А., Натыров А.Н., Орехов В.А., Коновалов С.В., Курочкин В.Е., Петров А.И. Генетический анализатор для фрагментного анализа ДНК // Научное приборостроение. 2012. Т. 22. № 4. С. 86.
Sokolenko A.P., Savonevich E.L., Ivantsov A.O., Raskin G.A., Kuligina E.S., Gorodnova T.V., Preobrazhenskaya E.V., Kleshchov M.A., Tiurin V.I., Mukhina M.S., Kotiv K.B., Shulga A.V., Kuznetsov S.G., Berlev I.V., Imyanitov E.N. Rapid selection of BRCA1-proficient tumor cells during neo adjuvant therapy for ovarian cancer in BRCA1 mutation carriers // Cancer Lett. 2017. V. 397. P. 127. https://doi.org/10.1016/j.canlet.2017.03.036
Slatko B.E., Kieleczawa J., Ju J., Gardner A.F., Hendrickson C.L., Ausubel F.M. “First Generation” automated DNA sequencing technology // Curr. Protocols Mol. Biol. 2011. V. 96. № 1. P. 7.2.1. https://doi.org/10.1002/0471142727.mb0702s96
Бочарова Д.В., Алексеев Я.И., Курочкин В.Е. Изучение влияния напряжения электрического поля на эффективность разделения одноцепочечных фрагментов ДНК при капиллярном гель-электрофорезе в полимерах на основе линейного поли-N,N-диметилакриламида // Журн. аналит. химии. 2020. Т. 75. № 12. С. 1148. https://doi.org/10.31857/S0044450220100035
https://www.ncbi.nlm.nih.gov/ (23.02.2021).
Дополнительные материалы отсутствуют.
Инструменты
Журнал аналитической химии


