Биоорганическая химия, 2019, T. 45, № 2, стр. 155-165
Прямой молекулярный фишинг в лизате клеток HepG2 потенциальных партнеров белок-белковых взаимодействий для белков, кодируемых генами 18-й хромосомы человека
П. В. Ершов 1, Ю. В. Мезенцев 1, Е. О. Яблоков 1, Л. А. Калужский 1, А. В. Флоринская 1, О. В. Гнеденко 1, В. Г. Згода 1, И. В. Вахрушев 1, О. С. Раева 1, К. Н. Ярыгин 1, А. А. Гилеп 2, С. А. Усанов 1, А. Е. Медведев 1, А. С. Иванов 1
1 Научно-исследовательский институт биомедицинской химии имени В.Н. Ореховича (ИБМХ)
119121 г. Москва, Погодинская ул., д. 10, стр. 8, Россия
2 Институт биоорганической химии Национальной академии наук Беларуси (ИБОХ)
220141 г. Минск, ул. Академика В.Ф. Купревича, д.5, корп.2, Беларусь
Поступила в редакцию 30.03.2018
После доработки 23.05.2018
Принята к публикации 02.04.2018
Аннотация
Целью настоящей работы было выявление спектра возможных белок-белковых взаимодействий (ББВ) для 6 целевых белков (CYB5A, RAB27B, SMAD4, CXXC1, RNMT, ТТR), кодируемых генами 18-й хромосомы человека, которые имеют определенную медицинскую значимость. Для этого был использован комплексный подход, основанный на совместном применения трех технологий: прямого молекулярного фишинга – аффинной хроматографии, белковой масс-спектрометрии и SPR-анализа (поверхностный плазмонный резонанс, от англ. surface plasmon resonance). В результате из лизата клеточной линии карциномы человека HepG2 было выделено от 1 до 11 потенциальных белков-партнеров для каждого целевого белка. На биосенсоре поверхностного плазмонного резонанса Biacore 3000 были валидированы 10 потенциальных ББВ, для которых были доступны препараты рекомбинантных белков. Для пяти белковых пар (СYB5A/CPR, СYB5A/CYP2C9, СYB5A/CYCS, CPR/СYP2C9 и CXXC1/ CYCS) были получены позитивные результаты, подтверждающие прямое взаимодействие выявленных белков-партнеров с целевыми белками. Для данных ББВ были определены величины равновесных констант диссоциации белковых комплексов (значения Kd порядка 10–7–10–5 М). Для оценки специфичности выявленных ББВ были протестированы 16 дополнительных различных ББВ с участием целевых белков. Эффективность прямого молекулярного фишинга была оценена на примере цитохрома b5 (CYB5A) как наиболее исследованного из всей выборки целевых белков с точки зрения структурно-функциональных связей с его белками-партнерами. Полученные новые данные расширяют наши знания в области интерактомики белков, кодируемых генами 18-й хромосомы человека, и сокращают количество непроверенных биоинформационных предсказаний возможных ББВ, которые содержатся в доступных Интернет-ресурсах.
ВВЕДЕНИЕ
Исследование белок-белковых взаимодействий (ББВ) актуальное научное направление, являющееся одним из немногих путей выяснения функций белков, большинство из которых находится в составе стабильных или динамичных белковых комплексов. Наиболее достоверные данные о ББВ исследователи получают с использованием подходов и методов, которые по приемам очень схожи с принципом рыбной ловли, что и нашло свое отражение в используемой терминологии: белок-наживка (bait protein), белок-добыча (prey protein), молекулярная рыбалка или фишинг (molecular fishing). Белком-наживкой называют известный целевой белок, который используют для выделения из клеточного или тканевого лизата его потенциальных белков-партнеров по ББВ (белки-добыча).
Ранее нами был разработан вариант прямого молекулярного фишинга, который является разновидностью твердофазной аффинной хроматографии, с использованием иммобилизованного на носителе белка-наживки в качестве аффинного лиганда. Выделение его потенциальных белков-партнеров из лизата биологического материала базируется на их прямом специфическом взаимодействии [1–5].
Одним из доступных объектов для исследования белкового интерактома человека является клеточная линия карциномы человека HepG2 (клетки HepG2), которая часто рассматривается в качестве модели гепатоцитов человека. В настоящее время с использованием современных протеомных технологий, основанных на методах белковой масс-спектрометрии, в гепатоцитах человека и клетках HepG2 выявлено более 9000 белков [6, 7], а в силу гетерогенности протеома HepG2, возникающей в результате реализации аберраций на белковом уровне, были установлены 2399 канонических и иных протеоформ [8]. Изучение ББВ такого большого числа белков сопряжено с определенными проблемами в постановке эксперимента и интерпретации получаемых массивов данных. Поэтому исследователи часто идут по пути изучения интерактома отдельных целевых белков, как, например, в работе [9].
В рамках Российского сегмента международного проекта “Протеом человека” были выполнены исследования по протеомному профилированию и инвентаризации белков, кодированных генами 18-й хромосомы человека, в ткани печени, клеточной культуре HepG2 и плазме крови [10–14], а также выборочное исследование интерактомов шести целевых белков 18-й хромосомы человека [1, 4], играющих определенную роль в развитии социально значимых заболеваний: микросомального цитохрома b5 (CYB5A) [15]; белка Ras семейства Rab27B (Rab27B) [16, 17]; белка 4 семейства SMAD (SMAD4) [18, 19], фактора транскрипции CXXC1, содержащго домен типа “цинковые пальцы” (CXXC1) [20]; мРНК-кэп-гуанин-N7-метилтрансферазы (RNMT) [21] и транстиретина (TTR) [22]. Для этих целей была использована комбинация прямого молекулярного фишинга потенциальных белков-партнеров из лизата ткани печени человека, технологии масс-спектрометрической идентификации белков и экспериментальной валидации возможных ББВ с помощью технологии поверхностного плазмонного резонанса (SPR) [1, 4]. В результате для каждого целевого белка-наживки было идентифицировано довольно большое число потенциальных белков-партнеров (до нескольких десятков).
Сравнительный анализ списков обнаруженных белков-партнеров для целевых белков-наживок показал крайне низкий уровень совпадения, что свидетельствует о высокой специфичности прямого молекулярного фишинга. Большое же число обнаруженных белков может быть обусловлено рядом причин: 1) наличие действительно большого репертуара взаимодействий иммобилизованного белка-лиганда с индивидуальными белками-партнерами; 2) выделение из лизата HepG2 сложных природных мультимолекулярных комплексов, содержащих белковые партнеры разных уровней (“партнеры партнеров”), в которых прямой партнер белка-наживки выступает в качестве своеобразной аффинной метки, позволяющей выделить весь комплекс; 3) выделение из лизата неспецифических комплексов и/или мицелл, которые образуются в ходе лизиса биологического материала и которые также содержат в своем составе прямой партнер белка-наживки. Выборочная SPR-валидация взаимодействия некоторых идентифицированных белков показала, что среди обнаруженных белков имеются реальные белки-партнеры, которые специфически взаимодействуют с белком-наживкой [4].
Очевидно, что количество ББВ с участием целевых белков не может быть постоянным и зависит от множества факторов, определяющих белковый состав конкретного биологического материала [23]. Образцы ткани печени человека представляют собой довольно гетерогенный по клеточному составу биоматериал (гепатоциты, клетки формирующие стенки желчных протоков и кровеносных сосудов, эндотелиоциты, макрофаги, клетки Купфера), содержащий также примесь компонентов желчи и крови. Поэтому было вполне целесообразно выполнить аналогичное исследование на более простом объекте – клеточной линии карциномы печени человека HepG2. Данный объект позволяет в определенной степени стандартизовать протеомные исследования, так как определяются усредненные показатели для миллионов однотипных клеток, выращенных в одних условиях, в отличие от одиночного образца ткани печени одного индивидуума. Настоящая работа является логическим продолжением цикла наших исследований по прямому молекулярному фишингу белков-партнеров, взаимодействующих с целевыми белками, кодированными генами 18-й хромосомы человека [1, 4, 24].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Аналитический вариант прямого молекулярного фишинга позволил оценить присутствие в лизате клеток HepG2 белкового материала, который может связываться с иммобилизованными целевыми белками на чипе оптического биосенсора. Все целевые белки были ковалентно иммобилизованы на оптическом чипе до одного и того же уровня (приблизительно 4000 ± 200 RU), что эквивалентно 4.0 ± 0.2 нг белка/мм2. Как видно из табл. 1, позитивные сигналы связывания белкового материала из образцов клеточного лизата HepG2 регистрировались со всеми целевыми белками в диапазоне от 33 до 186 RU.
Таблица 1.
SPR-aнализ связывания белкового материала лизата клеток HepG2*
| Целевой белок | Сигнал биосенсора, RU | Число выявленных потенциальных белков-партнеров |
|---|---|---|
| RAB27B | 34 ± 4 | 3 |
| CYB5A | 141 ± 7 | 10 |
| SMAD4 | 33 ± 4 | 2 |
| RNMT | 95 ± 6 | 1 |
| CXXC1 | 186 ± 9 | 11 |
| TTR | 135 ± 7 | 7 |
Препаративный вариант молекулярного фишинга с использованием выделения белков на аффинной колонке с иммобилизованными целевыми белками и последующего масс-спектрометрического анализа белков в полученных элюатах позволил идентифицировать потенциальные белки-партнеры для всех исследуемых целевых белков (табл. 2). Стоит отметить, что число выделенных и идентифицированных белков (от 1 до 11 белков на один целевой белок) было прямо пропорционально уровню связывания белкового материала из лизата в аналитическом фишинге, за исключением целевого белка RNMT (табл. 1 и 2).
Таблица 2.
Потенциальные белки-партнеры целевых белков, кодируемых генами 18-й хромосомы человека, выделенные из лизата клеток HepG2 человека
| № | Ген белка-партнера* | Название белка-партнера | M, Дa | Mascot | ||
|---|---|---|---|---|---|---|
| входящий номер в Uniprot | score** | пеп-тиды*** | ||||
| CYB5A | ||||||
| 1 | CAT | Catalase | 59 947 | P04040 | 86 | 5 |
| 2 | CYCS | Cytochrome c | 11 855 | P99999 | 63 | 4 |
| 3 | LSS | Lanosterol synthase | 84 453 | P48449 | 51 | 2 |
| 4 | HSPH1 | Heat shock protein 105 kDa | 97 716 | Q92598 | 71 | 4 |
| 5 | CYB5R3 | NADH-cytochrome b5 reductase 3 | 34 441 | P00387 | 50 | 4 |
| 6 | СPR | NADPH-cytochrome P450 reductase | 77 097 | P16435 | 63 | 3 |
| 7 | CYP2C9 | Сytochrome P450 2C9 | 55 628 | P11712 | 101 | 5 |
| 8 | RAB5C | Ras-related protein Rab-5C | 23 696 | P51148 | 58 | 3 |
| 9 | ARHGAP18 | Rho GTPase-activating protein 18 | 75 215 | Q8N392 | 50 | 4 |
| 10 | UGP2 | UTP-glucose-1-phosphate uridylyltransferase | 57 076 | Q16851 | 59 | 3 |
| TTR | ||||||
| 1 | CP | Ceruloplasmin | 122 983 | P00450 | 178 | 8 |
| 2 | CRP | C-reactive protein | 25 194 | P02741 | 68 | 3 |
| 3 | ITIH3 | Inter-alpha-trypsin inhibitor heavy chain H3 | 100 072 | Q06033 | 62 | 4 |
| 4 | LUM | Lumican | 38 747 | P51884 | 54 | 2 |
| 5 | RNMT | mRNA cap guanine-N7 methyltransferase | 55 494 | O43148 | 210 | 8 |
| 6 | F2 | Prothrombin | 71 475 | P00734 | 55 | 3 |
| 7 | VTN | Vitronectin | 55 069 | P04004 | 184 | 8 |
| СXXC1 | ||||||
| 1 | DDX1 | ATP-dependent RNA helicase DDX1 | 83 349 | Q92499 | 71 | 4 |
| 2 | DDX3Y | ATP-dependent RNA helicase DDX3Y | 73 564 | O15523 | 371 | 13 |
| 3 | DDX46 | Probable ATP-dependent RNA helicase DDX46 | 117 803 | Q7L014 | 78 | 4 |
| 4 | DHX9 | ATP-dependent RNA helicase A | 142 181 | Q08211 | 64 | 4 |
| 5 | FABP1 | Fatty acid-binding protein, liver | 14 256 | P07148 | 59 | 4 |
| 6 | GTF2I | General transcription factor II-I | 112 859 | P78347 | 124 | 6 |
| 7 | NQO1 | NAD(P)H dehydrogenase [quinone] 1 | 30 905 | P15559 | 95 | 5 |
| 8 | RAB27B | Ras-related protein Rab-27B | 24 820 | O00194 | 169 | 7 |
| 9 | SLIRP | SRA stem-loop-interacting RNA-binding protein, mitochondrial | 12 398 | Q9GZT3 | 78 | 3 |
| 10 | SRSF1 | Serine/arginine-rich splicing factor 1 | 27 842 | Q07955 | 51 | 2 |
| 11 | ZCCHC3 | Zinc finger CCHC domain-containing protein 3 | 44 389 | Q9NUD5 | 82 | 4 |
| RAB27B | ||||||
| 1 | HBA1 | Hemoglobin subunit alpha | 15 305 | P69905 | 228 | 12 |
| 2 | HBB | Hemoglobin subunit beta | 16 102 | P68871 | 79 | 4 |
| 3 | NQO1 | NAD(P)H dehydrogenase [quinone] 1 | 30 905 | P15559 | 79 | 5 |
| SMAD4 | ||||||
| 1 | BHMT | Betaine-homocysteine S-methyltransferase 1 | 45 426 | Q9H2M3 | 115 | 6 |
| 2 | CYP2C9 | Cytochrome P450 2C9 | 56 333 | P11712 | 136 | 8 |
| RNMT | ||||||
| 1 | EEF1A1P5 | Putative elongation factor 1-alpha-like 3 | 50 495 | Q5VTE0 | 100 | 5 |
Ранее нами было выполнено исследование потенциальных ББВ с участием тех же целевых белков в лизате ткани печени человека [4, 24], где было идентифицировано примерно на порядок большее число потенциальных белков-партнеров, чем в лизате клеток HepG2. Это различие в числе выявленных потенциальных белков-партнеров может быть обусловлено рядом причин: 1) более селективным фишингом на аффинном сорбенте на базе СNBr-сефароза 4В, 2) более полном отмывании связавшихся неспецифически или с низкой аффинностью белков в микроколонке по сравнению с парамагнитными частицами, которые были использованы ранее, 3) объективными различиями в белковом составе лизатов ткани печени и клеточной культуры HepG2. В конечном итоге данные факторы могли и комбинировано повлиять на кратное снижение уровня неспецифического связывания белков из клеточного лизата.
В целом, результаты прямого молекулярного фишинга с использованием сорбента СNBr-сефароза 4В можно охарактеризовать как специфичные, что следует из отсутствия совпадений в списках идентифицированных потенциальных белков-партнеров для разных целевых белков. Среди белков, выделенных из лизата клеток HepG2, обращает на себя внимание некоторые маркерные белки, которые экспрессируются только в опухолевидных клетках и отсутствуют в нормальных гепатоцитах – например, белки NQO1 [25], RAB5C [26] и UGP2 [27, 28]. В контексте прямого молекулярного фишинга, данная группа белков может рассматриваться в качестве “контаминирующих” белков-партнеров целевых белков, либо как отражение реальных сдвигов профиля белок-белковых взаимодействий в культуре клеток.
Результаты выборочной SPR-валидации ББВ между потенциальными белками-партнерами (CYCS, CYP2C9, CPR) и панелью целевых белков CYB5A, SMAD4, RNMT, CXXC1 представлены в табл. 3. Выбор валидируемых пар белков был ограничен доступностью для подобных анализов препаратов высокоочищенных рекомбинантных белков.
Таблица 3.
SPR-анализ взаимодействий целевых белков с белками, выделенными из лизата клеток HepG2*
| Потенциальные белки-партнеры из лизата | Целевые белки | Другое | |||
|---|---|---|---|---|---|
| CYB5A | SMAD4 | CXXC1 | RNMT | СPR | |
| CYCS | 7.2 ± 1.3 × 10–5 | Н/о | 2.5 ± 0.7 × 10–5 | Нет | Нет |
| CYP2C9 | 1.1 ± 0.3 × 10–6 | Нет | Нет | Н/о | 1.0 ± 0.2 × 10–7 |
| CPR | 3.4 ± 0.2 × 10–6 | Нет | Н/о | Н/о | Нет |
| Контрольные белки** | Целевые белки 18-й хромосомы человека | ||||
| GST | Нет | Нет | Нет | Нет | Н/о |
| B2M | Нет | Нет | Нет | Нет | Н/о |
| aFP | Нет | Нет | Нет | Нет | Н/о |
| BSA | Нет | Нет | Нет | Нет | Н/о |
В качестве контроля на специфичность взаимодействий с целевыми белками были использованы препараты белков GST, B2M, aFP и БСА, которые в SPR-анализе не показали значимого связывания. Поскольку белки CPR, CYP2C9 и CYCS были выделены из лизата клеток HepG2 как потенциальные белки-партнеры для CYB5A, был выполнен SPR-анализ бинарных взаимодействий: СYB5A/CPR, СYB5A/CYP2C9, СYB5A/CYCS, а также CPR/СYP2C9 (табл. 3). Серии типичных сенсограмм взаимодействий для белковой пары CPR/СYP2C9 и CYB5A/CPR представлены на рис. 1 и 2 соответственно.
Рис. 1.
SPR-анализ белок-белкового взаимодействия на примере СPR/CYP2C9. Сенсограммы взаимодействия c иммобилизованной на оптическом чипе СPR c CYP2C9 в концентрациях, мкМ: 2.5 (1), 1 (2), 0.25 (3), 0.1 (4). Уровень иммобилизации СPR составил в среднем 9000 RU. Раствор CYP2C9 инжектировали в течение 10 мин при скорости потока жидкости 5 мкл/мин. Регенерацию поверхности оптического чипа выполняли инжекцией раствора, содержавшего 2 M NaCl и 0.4% CHAPS в течение 30 с при скорости потока жидкости 15 мкл/мин. Выходной сигнал биосенсора (RU) представлял собой разницу сигналов между измерительным и контрольным (без иммобилизованного белка) каналами биосенсора.

Рис. 2.
Сенсограммы взаимодействия CYB5A, иммобилизованного на оптическом чипе, c СPR в концентрациях, мкМ: 1 (1); 5 (2); 7 (3); 10 (4). Уровень иммобилизации CYB5A составил в среднем 5100 RU. Инжекция СPR была в течение 5 мин при скорости потока жидкости 10 мкл/мин. Регенерацию поверхности оптического чипа выполняли при инжекции раствора содержавшего 2 M NaCl и 0.4% CHAPS в течение 30 с при скорости потока жидкости 10 мкл/мин. Выходной сигнал биосенсора (RU) представлял собой разницу сигналов между измерительным и контрольным (без иммобилизованного белка) каналами биосенсора.

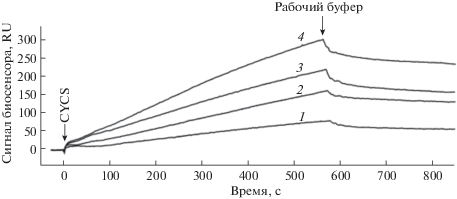
Взаимодействие CYB5A/CYP2C9 было количественно охарактеризовано нами на оптическом биосенсоре Biacore 3000 ранее [4]. Равновесная константа диссоциации (Kd) комплекса СYB5A/CYCS согласно нашим пилотным экспериментам составила порядка 70 мкМ, что оказалось сопоставимо с 30 мкМ по данным Сан и др. [29]. Из-за отсутствия белкового препарата CYB5R3 (выделенного из клеточного лизата на СYB5A, см. табл. 2), нам не удалось на оптическом биосенсоре подтвердить взаимодействие СYB5A/CYB5R3, которое было описано ранее другими авторами [30–32]. SPR-анализом было также подтверждено взаимодействие CXXC1/CYCS (рис. 3). Таким образом, выделенные из лизата клеток HepG2 белки CPR, CYP2C9, CYCS, CYB5R3 можно считать прямыми партнерами целевого белка СYB5A. С другой стороны, SPR-валидация взаимодействия SMAD4/CYP2C9 показала негативный результат (табл. 3), что, в свою очередь, позволяет исключить CYP2C9 из списка прямых партнеров SMAD4.
Рис. 3.
Сенсограммы взаимодействия белка CXXC1, иммобилизованного на оптическом чипе, с СYCS в концентрациях, мкМ: 4 (1); 20 (2); 40 (3); 80 (4). Уровень иммобилизации CXXC1 составил в среднем 3800 RU. Инжекция аналита была в течение 10 мин при скорости потока жидкости 10 мкл/мин. Регенерацию поверхности оптического чипа выполняли при инжекции 5 мM NaOH в течение 30 с при скорости потока жидкости 20 мкл/мин. Выходной сигнал биосенсора (RU) представлял собой разницу сигналов между измерительным и контрольным (без иммобилизованного белка) каналами биосенсора.
При прямом молекулярном фишинге, основанном на аффинном выделении потенциальных белков-партнеров целевого белка, иммобилизованного на сорбенте, возможно прохождение как минимум двух процессов: 1) взаимодействие прямого белка-партнера с целевым белком (например, образование бинарных комплексов CYB5A/CYP2C9 и CYB5A/CPR); 2) взаимодействие с целевым белком его прямого белка-партнера, ассоциированного со своими белками-партнерами (например, образование тройного комплекса CYB5A/[CYP2C9/CPR]). В связи с этим важно отметить, что существует определенная проблема интерпретации результатов прямого молекулярного фишинга и их дальнейшей валидации из-за присутствия в перечне выделенных белков как прямых, так и непрямых партнеров (“партнеры партнеров”), входящих в состав сложных белковых комплексов более высокого порядка. Однако, как было успешно продемонстрировано в экспериментах с SPR-анализом, она решается с привлечением дополнительного экспериментального инструментария для подтверждения наличия/отсутствия прямого парного взаимодействия выделенных белков с целевым белком, а также между собой.
Перечень белков-партнеров для CYB5A, выделенных при использовании аналогичного по методическому выполнению прямого молекулярного фишинга из лизата ткани печени человека (табл. 4), предлагается для сравнения с перечнем белков-партнеров из лизата клеток HepG2 (табл. 2). Интересно отметить, что при сравнении этих списков имеет место полное несовпадение, которое может быть обусловлено многими факторами, определяющими белковый состав конкретного биологического материала. Так, образец ткани печени человека представляет собой гетерогенный биоматериал как по клеточному составу, так и по наличию примесей. Гепатоциты являются основными клетками по массе, но в ткани печени также содержится заметное количество клеток, формирующих стенки желчных протоков и кровеносных сосудов, эндотелиоцитов, макрофагов, клеток Купфера и других. В качестве примесей в лизате несомненно присутствуют клетки крови, а также белковые компоненты желчи и плазмы. Клеточная линия HepG2 была выделена из опухоли печени человека в 1979 году и охарактеризована как первичная гепатоцеллюлярная карцинома [33] и в течение 30 лет использовалась как модель гепатоцита печени человека. Однако в 2009 году была выявлена ошибка в определении типа опухоли [34], и тип опухоли, из которой происходит линия HepG2, был определен как гепатобластома и, соответственно, клетки HepG2 были охарактеризованы как эпителиальные по своему происхождению. В последующих исследованиях были выявлены многочисленные и разнообразные различия между гепатоцитами человека и клетками HepG2 [6, 35–37]. Следовательно, обнаруженные нами различия в результатах прямого молекулярного фишинга из лизатов ткани печени и клеток HepG2 вполне объяснимы и их нужно рассматривать не как противоречивые результаты, а как взаимодополняющие, востребованные для формирования более целостной картины интерактома конкретного целевого белка. Тем не менее, остается открытым вопрос о масштабах экстраполяции интерактомных данных с модельного объекта на тканевой уровень и наоборот.
Таблица 4.
Потенциальные белки-партнеры для микросомального цитохрома CYB5A, выделенные из лизата ткани печени человека*
| № | Ген | Название белка | M, Дa | Mascot | ||
|---|---|---|---|---|---|---|
| входящий номер в Uniprot | scoreа | пептидыб | ||||
| CYB5A | ||||||
| 1 | ACTG1 | Actin, cytoplasmic 2 | 41 793 | P63261 | 187 | 10 |
| 2 | ANXA2 | Annexin A2 | 38 604 | P07355 | 163 | 6 |
| 3 | RDX | Radixin | 68 564 | P35241 | 57 | 2 |
| 4 | MSN | Moesin | 67 820 | P26038 | 120 | 6 |
| 5 | CYP4A11 | Cytochrome P450 4A11 | 59 348 | Q02928 | 135 | 6 |
| 6 | S100A9 | Protein S100-A9 | 13 242 | P06702 | 141 | 7 |
| 7 | ACAA1 | 3-Ketoacyl-CoA thiolase, peroxisomal | 44 292 | P09110 | 146 | 7 |
| 8 | FABP1 | Fatty acid-binding protein | 14 208 | P07148 | 212 | 9 |
| 9 | HSPA5 | 78 kDa glucose-regulated protein | 72 333 | P11021 | 132 | 5 |
| 10 | EPHX1 | Epoxide hydrolase 1, microsomal | 52 949 | P07099 | 102 | 5 |
| 11 | ALDH1A1 | Retinal dehydrogenase 1 | 54 862 | P00352 | 61 | 3 |
* См. примечание к табл. 2.
В качестве примера мы проанализировали с участием выявленных потенциальных белков-партнеров возможные ББВ цитохрома b5 (CYB5A), интерактом которого наиболее изучен по сравнению с другими используемыми в работе целевыми белками. Функциональная роль взаимодействий СYB5A с его прямыми белками партнерами – CPR, СYB5R, СYCS и различными изоформами цитохрома Р450 может заключаться в переносе электронов в окислительных реакциях многих эндогенных и экзогенных соединений [38–44]. Возможная функциональная взаимосвязь CYB5A c ланостеролсинтазой (LSS) обусловлена участием этих белков в пути синтеза ланостерола. SPR-анализ показал, что CYB5A связывается с ланостеролдеметилазой (CYP51) [45], а последняя, в свою очередь, может взаимодействовать с LSS (по гомологии с комплексообразованием ERG7/ERG11 у дрожжей [46]).
В ряде работ [47–49] было обнаружено, что шапероны (HSPA5, HSPH1), микросомальная эпоксидгидролаза (EPHX1) и ретинальдегидрогеназа 1 (ALDH1A1) методом копреципитации выделялись совместно с микросомальными цитохромами Р450, которые являются прямыми белками-партнерами CYB5A. Можно предположить, что эти белки могут быть для CYB5A непрямыми партнерами второго порядка.
Таким образом, приведенные литературные данные и наши результаты SPR-валидации подтверждают функциональные связи CYB5A с более чем половиной выделенных из лизатов потенциальных/реальных белков-партнеров, что однозначно свидетельствует о релевантности и достаточно высокой специфичности прямого молекулярного фишинга для выявления ББВ отдельных целевых белков. Можно заключить, что полученные в настоящей работе новые данные расширяют наши знания в области интерактомики белков, кодируемых генами 18-й хромосомы человека, и сокращают количество непроверенных биоинформационных предсказаний возможных ББВ, которые содержатся в доступных Интернет-ресурсах типа STRINGdb (http://string-db.org), FunCoup (http://funcoup.sbc.su.se) и других.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
В работе были использованы пять коммерчески доступных препаратов белков человека: транстиретин (TTR) и бычий сывороточный альбумин (BSA) от фирмы “Sigma” (США); глутатион-S-трансфераза (GST), бета-2-микроглобулин (B2M), альфа-фетопротеин (aFP) от фирмы “USBio” (США). Следующие белковые препараты (чистота >95% по гель-электрофорезу в денатурирующих условиях) были получены в ИБОХ НАН Беларуси [50–52]: микросомальный цитохром b5 (CYB5A), белок Rab27B из Ras-семейства (Rab27B), белок 4 семейства SMAD (SMAD4), фактор транскрипции CXXC1, содержащий домен типа “цинковые пальцы” (CXXC1), мРНК-кэп-гуанин-N7-метилтрансфераза (RNMT), цитохром С (CYCS), цитохром Р450 2С9 (CYP2C9) и цитохром-Р450-редуктаза (СPR).
Выращивание клеток HepG2 и подготовка образцов. Культивирование клеток HepG2 проводили согласно описанному [53]. Замороженные образцы клеток линии HepG2 были взяты из коллекции клеточных культур ИБМХ. После размораживания клетки культивировали в ростовой среде (DMEM/F12 (1 : 1) с добавлением 10% фетальной сыворотки крупного рогатого скота, 100 ед./мл пенициллина, 100 ед./мл стрептомицина и 2 мМ L-глутамина (все реактивы – Gibco, США)) в стандартных условиях (37°С, 5% СО2, 80% влажности), меняя среду два раза в неделю. По достижении монослоя (80–90% конфлюэнтности) культуры подвергали пассированию в соотношении 1 : 3. Для перевода клеток в суспензию флаконы промывали 0.25% раствором трипсина в EDTA (ПанЭко, РФ), затем вносили в них 3 мл вышеуказанного раствора и инкубировали при 37°С в течение 5–10 мин. При подготовке клеточного материала для последующих исследований клетки снимали с пластика, переносили клеточную суспензию в конические пробирки объемом 15 мл, а затем удаляли трипсин и компоненты ростовой среды в трех циклах отмывки путем центрифугирования с последующим ресуспендированием осадка в 10 мл фосфатно-солевого буфера. По окончании подсчитывали концентрацию клеток и переносили аликвоты (по 1 мл) в конические пробирки объемом 1.5 мл. После осаждения в настольной центрифуге и удаления супернатанта ампулы с клеточным осадком замораживали в парах жидкого азота. Всего таким образом был подготовлен экспериментальный материал в количестве 1 млрд. клеток.
Получение лизата клеток HepG2. Лизат клеток HepG2 был приготовлен путем гомогенизации клеточной массы (примерно 100 млн клеток) в ручном гомогенизаторе Sample griding kit (GE Healthcare, США), содержащем специальный абразивный материал, с 1 мл лизирующего буфера CellLytic Mammalian Tissue Lysis/Extraction Reagent и 10 мкл коктейля ингибиторов протеаз (Sigma, США, кат. номера C3228 и R8340, соответственно). После центрифугирования при 13 400 g (4°С) в течении 25 мин, к отобранному супернатанту добавляли 25% глицерина (v/v) и хранили при температуре –80°С. Содержание общего белка в образцах лизата клеток HepG2, определенное спектрофотометрически по методу Брэдфорд, составляло порядка 5 мг/мл.
Прямой молекулярный фишинг. Аналитический вариант прямого молекулярного фишинга выполняли на четырехканальном оптическом биосенсоре Biacore 3000 (GE Healthcare, США), работа которого основана на эффекте поверхностного плазмонного резонанса (SPR). Ковалентную иммобилизацию целевых белков на поверхности оптического чипа СМ5 выполняли в соответствии с протоколом производителя [54] путем формирования амидных связей между карбоксильными группами карбоксиметилированного декстрана на поверхности чипа и свободными аминогруппами белка. Измерения выполнялись в стандартном рабочем буфере HBS-EP+ (150 мМ NaCl, 3 мМ EDTA, 0.05% Tween 20, 10 мМ HEPES, pH 7.4). Образец лизата клеток HepG2 разбавляли в 20 раз рабочим буфером и осуществляли инжекцию последовательно через контрольный (без белка) и измерительный (с иммобилизованным целевым белком) каналы биосенсора в течение 10 мин при скорости потока 5 мкл/мин. Выходной сигнал биосенсора (в резонансных единицах, RU (1 RU эквивалентна изменению поверхностной концентрации белка в расчете на 1 мм2 примерно на 1 пг)) представлял собой разницу сигналов между измерительным и контрольным каналами. Уровень связывания белкового материала из клеточного лизата с иммобилизованным целевым белком оценивался по возрастанию сигнала биосенсора между начальной и конечной точками инжекции.
Препаративный вариант прямого молекулярного фишинга осуществляли в оригинальных микроколонках объемом 200 мкл, заполненных аффинным сорбентом, в качестве которого была использована CNBr-сефароза 4В (GE Healthcare, США) с иммобилизованным целевым белком. Иммобилизацию всех целевых белков выполняли в соответствии с протоколом фирмы-производителя сорбента (GE Healthcare, США). К 200 мкл раствора белка в буферном растворе 500 мM NaCl, 100 мМ NaHCO3, рН 8.3) добавляли 100 мкл суспензии CNBr-сефарозы 4В (100 мг сухого сорбента) и инкубировали в течение 2 ч при комнатной температуре. Инактивацию сорбента выполняли путем его инкубации в буфере, содержащем 150 мМ NaCl, 100 мМ Tris-HCl (рН 7.4) в течение ночи при 4°С. В среднем, количество иммобилизованного белка на сорбенте, оцененное по остаточной концентрации не связавшегося белка в растворе, составляло около 3 мг белка на 1 г сорбента. Заполненные сорбентом микроколонки помещали в хроматограф AKTA Purifier 10 (GE Healthcare, США) и далее уравновешивали 30 мин рабочим буфером HBS-EP+ при скорости потока 50 мкл/мин и температуре 15°С. Далее через микроколонку пропускали 2 мл лизата, разбавленного в 2 раза рабочим буфером HBS-EP+ (0.5 мг/мл белка), при скорости потока 50 мкл/мин в течение 80 мин. Связавшиеся на сорбенте белки элюировали 4% раствором HCOOH (pH 2.5) при скорости потока 50 мкл/мин в течение 100 мин. Содержание общего белка в элюатах, определенное спектрофотометрически по методу Брэдфорд, составляло порядка 30 мкг/мл. Для контроля возможного неспецифического связывания белков лизата с сорбентом использовали микроколонки, заполненные инактивированной CNBr-сефарозой 4В без иммобилизованного белка. Эксперименты по аффинному выделению потенциальных белков-партнеров для каждого целевого белка повторяли три раза.
Масс-спектрометрическая идентификация белков. Для осуществления масс-спектрометрического анализа выделенных белков выполняли специальную подготовку проб, полученных в результате молекулярного фишинга. Образцы элюатов, содержащие белковый материал (30 мкг общего белка), подвергали стандартной процедуре трипсинолиза с предварительным алкилированием и восстановлением сульфгидрильных групп белков. Все процедуры выполняли в центрифужных концентраторах Vivaspin 500 Centrifugal Concentrator, 10 кДа MWCO (GE Healthcare, США) по методу FASP (Filter Aided Sample Preparation) [1, 55]. Для трипсинолиза белков использовали лиофилизованный препарат трипсина из поджелудочной железы свиньи (активность 15 600 ед./мг, Promega (США) кат. номер V5111). разделение пептидов осуществляли в хроматографической системе Ultimate 3000 RSLCnano (“Thermo Scientific”). Пептиды разделяли на колонке Acclaim Pepmap® C18 (75 мкм × 150 мм, 2 мкм размер частиц) (“Thermo Scientific”) в градиенте подвижной фазы А (0.02% уксусная кислота, 0.1% муравьиная кислота, рН 2.66) и подвижной фазы Б (80% ацетонитрил, 0.02% уксусная кислота, 0.1% муравьиная кислота) при скорости потока 0.3 мкл/мин. Пептиды перед аналитическим разделением наносили на обогащающую колонку Accalaim µ-Precolumn (0.5 мм × 3 мм, 5 мкм размер частиц) (Thermo Scientific) при скорости потока 10 мкл/мин в течение 5 мин в изократическом режиме подвижной фазы В (2% ацетонитрил, 0.02% уксусная кислота, 0.1% муравьиная кислота, рН 2.63). Разделение проводили в линейном градиенте подвижной фазы Б (от 2 до 37% в течение 55 мин), линейно повышали концентрацию буфера B до 90% за 5 мин, далее промывали колонку при 90% подвижной фазы Б в течение 10 мин, снижали содержание буфера В до 2% за 5 мин и уравновешивали в начальных условиях градиента при 2% подвижной фазы Б в течение 15 мин.
Масс-спектрометрический анализ выполняли на гибридном орбитальном масс-спектрометре Orbitrap Exactive (“Thermo Scientific”, США) в режиме положительной ионизации в источнике NSI (“Thermo Scientific”). Температура осушающего газа (азот) составляла 280оС, напряжение на эмиттере 2.1 кВ. Панорамное сканирование проводили в диапазоне m/z от 400 до 1200, тандемное сканирование фрагментных ионов от нижней границы m/z 110 до верхней границы, определяемой зарядным состоянием прекурсорного иона, но не более m/z 2100. Для тандемного сканирования учитывали только ионы от z = 2+ до z = 6+ по зарядному состоянию. Максимальное число разрешенных для синхронной изоляции ионов в режиме MS2 было установлено, как не более 20. Максимальное время накопления для прекурсорных ионов составило не более 50 мс, для фрагментных ионов не более 110 мс.
Идентификацию белков выполняли с помощью программы Mascot (www.matrixscience.com) с использованием базы данных SwissProt (www.uniprot.org) для таксона Homo sapiens (декабрь 2017 г). Были использованы следующие параметры поиска: протеолитический фермент-трипсин, допустимые отклонения по массе моноизотопных пептидов ±20 ppm (пропромиле, от англ. рarts per million), допустимые отклонения MS/MS ±0.06 Да, число допустимых пропусков сайтов расщепления трипсином – 2, вариабельные модификации – окисленный метионин, фиксированные модификации – карбамидометил. В результирующий список достоверно обнаруженных белков включали только те белки, которые идентифицировались при 3 технических повторах c достоверностью p < 0.01 и Mascot Score >50.
Поверхностный плазмонный резонанс (SPR). Исследование ББВ выполняли на четырехканальном оптическом биосенсоре Biacore 3000 (GE Healthcare, США) используя в качестве рабочего буфера HBS-EP+. Образцы тестируемых белков инжектировали по поверхности оптического чипа с ковалентно иммобилизованными целевыми белками в трех измерительных каналах биосенсора в течение 5 или 10 мин при скорости потока жидкости от 5 или 10 мкл/мин, при этом первый канал был в качестве контрольного. Регенерацию поверхности оптического чипа выполняли раствором, содержащем 2 M NaCl, 0.4% CHAPS (3-((3-холамидопропил)диметиламмоний)1-пропансульфонат), в течение 30 с при скорости потока жидкости 15 мкл/мин. Регистрируемый сигнал биосенсора (в резонансных единицах RU) представлял собой разницу сигналов между измерительным и контрольным (без иммобилизованного белка) каналами. Расчет равновесных констант диссоциации белковых комплексов выполняли путем анализа серии сенсограмм с помощью лицензионного программного обеспечения Biacore BiaEvaluation software v. 4.1 с использованием математической модели поверхностного связывания 1 : 1 по Лэнгмюру.
БЛАГОДАРНОСТИ
Работа выполнена в рамках Программы фундаментальных научных исследований государственных академий наук на 2013–2020 годы. Оптико-биосенсорный анализ белок-белковых взаимодействий и масс-спектрометрическая идентификация белков были выполнены с использованием оборудования ЦКП “Протеом человека” (ИБМХ), поддержанного Минобрнауки России (уникальный идентификатор проекта RFMEFI62117X0017).
Список литературы
Иванов А.С., Ершов П.В., Мольнар А.А., Мезенцев Ю.В., Калужский Л.А., Яблоков Е.О., Флоринская А.В., Гнеденко О.В., Медведев А.Е., Козин С.А., Митькевич В.А., Макаров А.А., Гилеп А.А., Лущик А.Я., Гайдукевич И.В., Усанов С.А. // Биоорган. химия. 2016. Т. 42(1). С. 1–10. [Ivanov A.S., Ershov P.V., Molnar A.A., Yu Mezentsev V., Kaluzhskiy L.A., Yablokov E.O., Florinskaya A.V., Gnedenko O.V., Medvedev A.E., Kozin S.A., Mitkevich V.A., Makarov A.A., Gilep A.A., Ya Luschik A., Gaidukevich I.V., Usanov S.A. // Russ. J.Bioorgan. Chem. 2016. Т. 42(1). P. 14–21.]
Иванов А.С., Згода В.Г, Арчаков А.И. // Биоорган. химия. 2011. Т. 37. С. 8–21. [Ivanov A.S., Zgoda V.G., Archakov A.I. // Russ. J. Bioorgan. Chem. 2011. V. 37(1). P. 4–16.]
Ivanov A.S., Medvedev A.E., Ershov P.V., Mezentsev Y.V., Yablokov E.O., Kaluzhsky L.A., Gnedenko O.V., Buneeva O.A., Molnar A.A., Gilep A.A., Yantsevich A.V., Usanov S.A., Zgoda V.G., Archakov A.I. // FEBS J. 2013. V. 280 (Suppl. 1). P. 633.
Ivanov A.S., Medvedev A., Ershov P., Molnar A., Mezentsev Y., Yablokov E., Kaluzhsky L., Gnedenko O., Buneeva O., Haidukevich I., Sergeev G., Lushchyk A., Yantsevich A., Medvedeva M., Kozin S., Popov I., Novikova S., Zgoda V., Gilep A., Usanov S., Lisitsa A., Archakov A. // Proteomics. 2014. V. 14. P. 2261–2274.
Medvedev A.E., Kopylov A.T., Buneeva O.A., Zgoda V.G., Archakov A.I. // Proteomics. 2012. V. 12(4–5). P. 621–637.
Wiśniewski J.R., Vildhede A., Norén A., Artursson P. // J. Proteomics. 2016. V. 136. P. 234–247.
Rowe C., Gerrard D.T., Jenkins R., Berry A., Durkin K., Sundstrom L., Goldring C.E., Park B.K., Kitteringham N.R., Hanley K.P., Hanley N.A. // Hepatology. 2013. V. 58(2). P. 799–809.
Поверенная Е.В., Киселева О.И., Пономаренко Е.А., Нарыжный С.Н., Згода В.Г., Лисица А.В. // Биомедицинская химия. 2017. Т. 63(5). С. 373–378.
Hou C., Li Y., Liu H., Dang M., Qin G., Zhang N., Chen R. // Proteome Sci. 2018. V. 16. P. 5.
Zgoda V.G., Kopylov A.T., Tikhonova O.V., Moisa A.A., Pyndyk N.V., Farafonova T.E., Novikova S.E., Lisitsa A.V., Ponomarenko E.A., Poverennaya E.V., Radko S.P., Khmeleva S.A., Kurbatov L.K., Filimonov A.D., Bogolyubova N.A., Ilgisonis E.V., Chernobrovkin A.L., Ivanov A.S., Medvedev A.E., Mezentsev Y.V., Moshkovskii S.A., Naryzhny S.N., Ilina E.N., Kostrjukova E.S., Alexeev D.G., Tyakht A.V., Govorun V.M., Archakov A.I. // J. Proteome Res. 2013. V. 12(1). P. 123–134.
Ponomarenko E.A., Kopylov A.T., Lisitsa A.V., Radko S.P., Kiseleva Y.Y., Kurbatov L.K., Ptitsyn K.G., Tikhonova O.V., Moisa A.A., Novikova S.E., Poverennaya E.V., Ilgisonis E.V., Filimonov A.D., Bogolubova N.A., Averchuk V.V., Karalkin P.A., Vakhrushev I.V., Yarygin K.N., Moshkovskii S.A., Zgoda V.G., Sokolov A.S., Mazur A.M., Prokhortchouck E.B., Skryabin K.G., Ilina E.N., Kostrjukova E.S., Alexeev D.G., Tyakht A.V., Gorbachev A.Y., Govorun V.M. Archakov A.I. // J. Proteome Res. 2014. V. 13(1). P. 183–190.
Poverennaya E.V., Kopylov A.T., Ponomarenko E.A., Ilgisonis E.V., Zgoda V.G., Tikhonova O.V., Novikova S.E., Farafonova T.E., Kiseleva Y.Y., Radko S.P., Vakhrushev I.V., Yarygin K.N., Moshkovskii S.A., Kiseleva O.I., Lisitsa A.V., Sokolov A.S., Mazur A.M., Prokhortchouk E.B., Skryabin K.G., Kostrjukova E.S., Tyakht A.V., Gorbachev A.Y., Ilina E.N., Govorun V.M., Archakov A.I. // J. Proteome Res. 2016. V. 15(11). P. 4030–4038.
Пономаренко Е.А., Згода В.Г., Копылов А.Т., Поверенная Е.В., Ильгисонис Е.В., Лисица А.В., Арчаков А.И. // Биомедицинская химия. 2015. Т. 61(2). С. 169–175.
Kopylov A.T., Ilgisonis E.V., Moysa A.A., Tikhonova O.V., Zavialova M.G., Novikova S.E., Lisitsa A.V., Ponomarenko E.A., Moshkovskii S.A., Markin A.A., Grigoriev A.I., Zgoda V.G., Archakov A.I. // J. Proteome Res. 2016. V. 15(11). P. 4039–4046.
Giordano S.J., Kaftory A., Steggles A.W. // Hum. Genet. 1994. V. 93(5). P. 568–570.
Bao J., Ni Y., Qin H., Xu L., Ge Z., Zhan F., Zhu H., Zhao J., Zhou X., Tang X., Tang L. // Gastroenterol. Res. Pract. 2014. V. 2014. P. 913106.
Hendrix A., Braems G., Bracke M., Seabra M., Gahl W., De Wever O., Westbroek W. // Oncotarget. 2010. V. 1(4). P. 304–308.
Zhu Y., Feng F., Yu J., Song B., Hu M., Gao X., Wang Y., Zhang Q. // DNA and Cell Biology. 2013. V. 32(9). P. 531–540.
Houlston R., Bevan S., Williams A., Young J., Dunlop M., Rozen P., Eng C., Markie D., Woodford–Richens K., Rodriguez–Bigas M.A., Leggett B., Neale K., Phillips R., Sheridan E., Hodgson S., Iwama T., Eccles D., Bodmer W., Tomlinson I. // Hum. Mol. Genet. 1998. V. 7(12). P. 1907–1912.
Blackledge N.P., Thomson J.P., Skene P.J. // Cold Spring Harb. Perspect. Biol. 2013. V. 5(11). P. a018648.
Stefanska B., Cheishvili D., Suderman M., Arakelian A., Huang J., Hallett M., Han Z.G., Al–Mahtab M., Akbar S.M., Khan W.A., Raqib R., Tanvir I., Khan H.A., Rabbani S.A., Szyf M. // Clin. Cancer Res. 2014. V. 20(12). P. 3118–3132.
Hawkins P.N., Ando Y., Dispenzeri A., Gonzalez–Duarte A., Adams D., Suhr O.B. // Ann. Med. 2015. V. 47(8). P. 625–638.
Pu S., Vlasblom J., Turinsky A., Marcon E., Phanse S., Trimble S.S., Olsen J., Greenblatt J., Emili A., Wodak S.J. // J. Proteomics. 2015. V. 118. P. 63–80.
Ershov P., Mezentsev Y., Gnedenko O., Mukha D., Yantsevich A., Britikov V., Kaluzhskiy L., Yablokov E., Molnar A., Ivanov A., Lisitsa A., Gilep A., Usanov S., Archakov A. // Proteomics. 2012. V. 12(22). P. 3295–3298.
Dinkova-Kostova A.T., Talalay P. // Arch. Biochem. Biophys. 2010. V. 501(1). P. 116–123.
Onodera Y., Nam J.M., Hashimoto A., Norman J.C., Shirato H., Hashimoto S., Sabe H. // J. Cell Biol. 2012. V. 197(7). P. 983–996.
Tan G.S., Lim K.H., Tan H.T., Khoo M.L., Tan S.H., Toh H.C., Ching Ming Chung M. // J. Proteome Res. 2014. V. 13(11). P. 4833–4846.
Wang Q., Yang Z.L., Zou Q., Yuan Y., Li J., Liang L., Zeng G., Chen S. // CancerInvest. 2016. V. 34(6). P. 255–264.
Sun M.H., Liu S.Q., Du K.J., Nie C.M., Lin Y.W. // Spectrochim. Acta A Mol. Biomol. Spectrosc. 2014. V. 118. P. 130–137.
Shirabe K., Nagai T., Yubisui T., Takeshita M. // Biochim. Biophys. Acta. 1998. V. 1384(1). P. 16–22.
Dailey H.A., Strittmatter P. // J. Biol. Chem. 1979. V. 254(12). P. 5388–5396.
Strittmatter P., Hackett C.S., Korza G., Ozols J. // J. Biol. Chem. 1990. V. 265(35). P. 21709–21713.
Aden D.P., Fogel A., Plotkin S., Damjanov I., Knowles B.B. // Nature. 1979. V. 282. P. 615–617.
López-Terrada D., Cheung S.W., Finegold M.J., Knowles B.B. // Human Pathology. 2009. V. 40(10). P. 1512–1515.
Gerets H.H.J., Tilmant K., Gerin B., Chanteux H., Depelchin B.O., Dhalluin S., Atienzar F.A. // Cell Biol. Toxicol. 2012. V. 28. P. 69.
Costantini S., Di Bernardo G., Cammarota M., Castello G., Colonna G. // Gene. 2013. V. 518(2). P. 335–345.
Fasolino I., Guarino V., Marrese M., Cirillo V., Vallifuoco M., Tamma M.L., Vassallo V., Bracco A., Calise F., Ambrosio L. // Biomed. Mater. 2017. V. 13(1). P. 015017.
Guengerich F.P. // Arch. Biochem. Biophys. 2005. V. 440(2). P. 204–211.
Guengerich F.P., Johnson W.W. // Biochemistry. 1997. V. 36(48). P. 14741–14750.
Shimada T., Mernaugh R.L., Guengerich F.P. // Arch. Biochem. Biophys. 2005. V. 435(1). P. 207–216.
Gnedenko O.V., Yablokov E.O., Usanov S.A., Mukha D.V., Sergeev G.V., Bulko T.V., Kuzikov A.V., Moskaleva N.E., Shumyantseva V.V., Ivanov A.S., Archakov A.I. // Chemical Physical Letters. 2014. V. 593. P. 40–44.
Jakobs H.H., Mikula M., Havemeyer A., Strzalkowska A., Borowa-Chmielak M., Dzwonek A., Gajewska M., Hennig E.E., Ostrowski J., Clement B. // PLoS One. 2014. V. 9(8). P. e105371.
Qian W., Sun Y.L., Wang Y.H., Zhuang J.H., Xie Y., Huang Z.X. // Biochemistry. 1998. V. 37(40). P. 14137–14150.
Nq S., Smith M.B., Smith H.T., Millett F. // Biochemistry. 1977. V. 16(23). P. 4975–4978.
Yablokov E., Florinskaya A., Medvedev A., Sergeev G., Strushkevich N., Luschik A., Shkel T., Haidukevich I., Gilep A., Usanov S., Ivanov A. // Archives of Biochemistry and Biophysics. 2017. V. 619(1). P. 10–15.
Mo C., Bard M. // Biochim. Biophys. Acta. 2005. V. 1737(2–3). P. 152–160.
Li B., Yau P., Kemper B. // Proteomics. 2011. V. 11(16). P. 3359–3368.
Imaoka S., Wan J., Chow T., Hiroi T., Eyanagi R., Shigematsu H., Funae Y. // Arch. Biochem. Biophys. 1998. V. 353. P. 331–336.
Taura K.I., Yamada H., Hagino Y., Ishii Y., Mori M.A., Oguri K. // Biochem. Biophys. Res. Communs. 2000. V. 273. P. 1048–1052.
Gilep A.A., Guryev O.L., Usanov S.A., Estabrook R.W. // J. Inorg. Biochem. 2001. V. 87. P. 237–244.
Usanov S.A., Graham S.E., Lepesheva G.I., Azeva T.N., Strushkevich N.V., Gilep A.A., Estabrook R.W., Peterson J.A. // Biochemistry. 2002. V. 41. P. 8310–8320.
Сергеев Г.В., Гилеп А.А., Усанов С.А. // Биохимия. 2014. Т. 79. С. 520–531. [Sergeev G.V., Gilep A.A., Usanov S.A. // Biochemistry (Moscow). 2014. V. 79. P. 406–416.]
Naryzhny S.N., Maynskova M.A., Zgoda V.G., Ronzhina N.L., Kleyst O.A., Vakhrushev I.V., Archakov A.I. // J. Proteome Res. 2016. V. 15(2). P. 525–530.
Biacore Sensor Surface Handbook BR–1005–71. Edition AB. 2007.
Wiśniewski J.R., Zougman A., Nagaraj N., Mann M. // Nat. Methods. 2009. V. 6(5). P. 359–362.
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


