Биоорганическая химия, 2020, T. 46, № 5, стр. 550-552
Мультиплексная ПЦР для выявления бактериальных возбудителей инфекционной пневмонии
С. А. Лапа 1, *, Е. С. Клочихина 1, Р. А. Мифтахов 1, А. М. Золотов 1, А. С. Заседателев 1, А. В. Чудинов 1
1 ФГБУН Институт молекулярной биологии им. В.А. Энгельгардта РАН
119991 Москва, ул. Вавилова 32, Россия
* E-mail: lapa@biochip.ru
Поступила в редакцию 04.04.2020
После доработки 16.04.2020
Принята к публикации 20.04.2020
Аннотация
Разработана и оптимизирована система мультиплексной ПЦР для быстрого выявления пяти основных возбудителей бактериальной пневмонии. Система может быть расширена для анализа возбудителей пневмонии вирусной (ДНК- и РНК-содержащие вирусы), а также грибковой природы.
ВВЕДЕНИЕ
Инфекционная пневмония – острый воспалительный процесс легочной ткани, вызываемый широким рядом возбудителей бактериальной, вирусной и грибковой природы. Вспышки инфекционных пневмоний, вызванных коронавирусами SARS-CoV (2002–2003 г.), MERS-CoV (2012–2013 г.), SARS-CoV-2 (2019–?), вирусами гриппа А и В, а также бактериальных пневмоний, становятся реальной социальной угрозой. Бактериальные пневмонии характеризуются летальностью около 15%, при этом летальность в случаях тяжелого течения внебольничной пневмонии (ТВП) может достигать 21–58% [1], что сравнимо с таковыми показателями SARS (т.н. атипичная пневмония, ТОРС), MERS [2] и превышает этот показатель для CoViD-19 и гриппа.
Специализированные лечебные учреждения, в которые поступают пациенты с клиническим диагнозом “пневмония”, сталкиваются с проблемой установления этиологии заболевания и быстрой идентификации возбудителя, поскольку часто вирусные и бактериальные пневмонии характеризуются сходной клинической картиной. Ситуация резко обострилась с возникновением пандемии CoViD-19, для которого необходима своевременная дифференциальная диагностика вторичной бактериальной инфекции, часто возникающей как одно из осложнений при течении болезни.
От своевременной постановки точного диагноза зависит правильный подбор препаратов и стратегия лечения пациента [3]. Сложившаяся ситуация сигнализирует об острой необходимости создания экспресс-методов дифференциальной диагностики.
Разработана мультиплексная ПЦР для видового определения пяти основных возбудителей бактериальной пневмонии человека. Система ориентирована на применение в клинической диагностике для определения этиологии заболевания благодаря возможности дифференцировать бактериальную от вирусной и грибковой пневмонии.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Одними из наиболее распространенных возбудителей бактериальной пневмонии являются представители нескольких родов: Staphylococcus aureus, Pseudomonas aeruginosa, Haemophilus influenza, Legionella pneumophila, Klebsiella pneumonia и некоторые другие [4]. Для перечисленных пяти бактериальных видов были выбраны генетические мишени и сконструированы праймеры для осуществления мультиплексной ПЦР. При конструировании праймеров руководствовались требованием видовой специфичности и внутривидовой консервативности выбранных участков генетических мишеней, необходимым для надежной дифференциальной диагностики, например, ген ebpS для идентификации S. aureus [5] и ген sidA для L. pneumophila [6]. Учитывали необходимость получения различных длин ПЦР-продуктов для удобства идентификации возбудителя при электрофоретическом разделении.
Для каждого праймера проводили процедуру BLAST-анализа, определяли его физико-химические характеристики, включая тестирование на наличие как внутри- так и межмолекулярных вторичных структур.
Проводили оптимизацию температурно-временного профиля ПЦР с применением градиентной ПЦР, а также состава компонентов буфера и концентрации каждого из праймеров в смеси. Экспериментально определяли специфичность праймеров к целевым и нецелевым мишеням (используя тотальную геномную ДНК каждого из тестируемых бактериальных штаммов), как в режиме применения индивидуальных ДНК-матриц, так и в режиме смесей ДНК нескольких возбудителей в одной пробирке. Установили, что праймеры обладают высокой специфичностью в смеси, содержащей ДНК различных микроорганизмов и способны выявлять исключительно свои специфичные мишени, не давая ложноположительный результат при наличии в смеси неспецифичной ДНК.
Определяли чувствительность сконструированной тест-системы раститровкой ДНК каждого из анализируемых возбудителей, которая составила от 102 до 103 копий геномной ДНК на реакционную пробирку, в том числе при одновременном введении в смесь нескольких матриц.
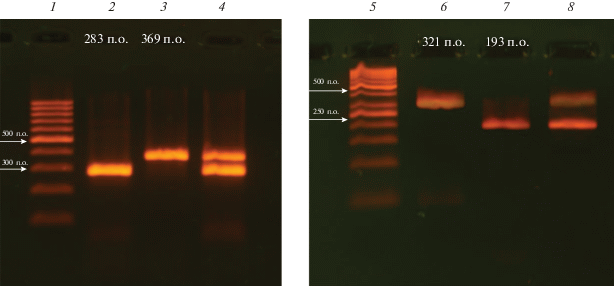
Результат дифференциального обнаружения ДНК возбудителя в образце методом мультиплексной ПЦР показан на рис. 1.
Рис. 1.
Определение ДНК возбудителя электрофоретическим разделением ПЦР-продуктов. 1 – маркер длин ДНК GeneRuler 100bp (Thermo Scientific, США), 2 – S. aureus, 3 – L. pneumophila, 4 – S. aureus + L. pneumophila, 5 – маркер длин ДНК GeneRuler 50bp (Thermo Scientific, США), 6 – P. aeruginosa, 7 – H. influenza, 8 – P. aeruginosa + H. influenza. 4% агарозный гель, окрашивание бромистым этидием.
Разработанная система может быть расширена для выявления возбудителей пневмонии вирусной и грибковой природы. В настоящее время планируется проведение испытаний на клинических образцах, применение меченых производных dNTP для последующего анализа на биочипах, а также разработка математического алгоритма расчетов сигналов амплификации. Система подходит для диагностических лабораторий, специализирующихся на клинических анализах с использованием ПЦР.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы
В работе использована деконтаминированная полногеномная ДНК бактериальных штаммов из коллекции ГНЦ прикладной микробиологии и биотехнологии (п. Оболенск). Выделение ДНК из клеточных культур осуществляли CTAB-методом [7].
Праймеры
Множественное выравнивание последовательностей геномных мишеней проводили с помощью алгоритма ClustalW (www.clustal.org). Конструирование праймеров осуществляли с помощью сетевого ресурса www.idtdna.com, анализ специфичности проводили с помощью алгоритма BLAST (NIH, США). Последовательности, видоспецифичность, генетические мишени и длина ПЦР-продуктов для всех использованных пар праймеров приведены ниже: S. aureus, ген ebpS, прямой ebpS-f (5'-ACTCGACTGAGGATAAAGCGTCT-3'), обратный ebpS-r (5'-CCTCCAAATATCGCTAATGCACC-3'), длина продукта – 283 п.о.; L. pneumophila, ген sidA, прямой sidA-f (5'-TTCCACTGGTGGGTGGGGTTTTG-3'), обратный sidA-r (5'-TCATGTTGGAGTTCTATGGCACG-3'), длина ПЦР-продукта – 369 п.о.; H. influenza, ген fucK, прямой fucK-f (5'-TGCTCACTCAACGCTTAACTGGT-3'), обратный fucK-r (5'-TTCTGGGCTAATGGTGTACGTAA-3'), длина ПЦР‑продукта – 193 п.о.; P. aeruginosa, ген oprL, прямой oprL-f (5'-GCGTGCGATCACCACCTTCTACT-3'), обратный oprL-r (5'-TTCTTCAGCTCGACGCGACGGTT-3'), длина ПЦР-продукта – 321 п.о.; K. pneumonia, ген rmpA, прямой rmpA-f (5'-ATCAATAGCAATTAAGCACAAAA-3'), обратный rmpA-r (5'-TCATAATCACACCCTTTAGGATA-3'), длина ПЦР-продукта – 177 п.о.
Мультиплексная ПЦР
Реакционная смесь (30 мкл) содержала 1.5 единицы Taq-полимеразы (Thermo Scientific, США) в буфере той же фирмы, dNTP в концентрации 200 мкМ каждого, пять пар специфичных праймеров и полногеномную бактериальную матрицу (либо смесь бактериальных ДНК). Реакцию проводили на ДНК-амплификаторе MiniCycler (MJResearch, США) при следующих условиях: 95°C в течение 5 мин (начальная денатурация); 36 циклов по 20 с при 95°C, 30 с при 66°C и 40 с при 72°C; завершающая инкубация в течение 5 мин при 72°C. Градиентную ПЦР и определение чувствительности системы с помощью ПЦР в режиме реального времени проводили на амплификаторе IQ5 (Bio-Rad, США). Продукты ПЦР разделяли в 4% агарозном геле, окрашивали бромистым этидием. Тип анализируемой ДНК определяли по длинам продуктов амплификации (рис. 1).
Список литературы
Sligl W.I., Marrie T.J. // Crit. Care. Clin. 2013. V. 29. P. 563–601. https://doi.org/10.1016/j.ccc.2013.03.009
Song Z., Xu Y., Bao L., Zhang L., Yu P., Qu Y., Zhu H., Zhao W., Han Y., Qin C. // Viruses. 2019. V. 11. E59. https://doi.org/10.3390/v11010059
Harris M., Clark J., Coote N., Fletcher P., Harnden A., McKean M., Thomson A. // Thorax. 2011. 66:ii1eii23. https://doi.org/10.1136/thoraxjnl-2011-200598
Han Y.C., Woo J.H. // Respirology. 1996. V. 1. P. 115–122. https://doi.org/10.1111/j.1440-1843.1996.tb00019.x
Liu Y., Cao Y., Wang T., Dong Q., Li J., Niu C. // Front. Microbiol. 2019. V. 10. A222. https://doi.org/10.3389/fmicb.2019.00222
Tabatabaei M., Hemati Z., Moezzi M.O., Azimzadeh N. // Mol. Biol. Res. Commun. 2016. V. 5. P. 215–223.
Doyle J.J., Doyle J.L. // Phytochemical Bullettin. 1987. V. 19. P. 11–15.
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


