Биоорганическая химия, 2021, T. 47, № 1, стр. 135-143
Физико-химические свойства фотоконвертируемого флуоресцентного белка из Montastraea cavernosa
А. Ю. Фролова 1, 2, А. А. Пахомов 1, 2, *, В. И. Мартынов 1
1 ФГБУН “Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова” РАН
117997 Москва, ул. Миклухо-Маклая, 16/10, Россия
2 ФГБУН Институт элементоорганических соединений им. А.Н. Несмеянова РАН
119334 Москва, ул. Вавилова, 28, Россия
* E-mail: alpah@mail.ru
Поступила в редакцию 08.06.2020
После доработки 19.06.2020
Принята к публикации 20.06.2020
Аннотация
Среди широкого разнообразия известных на сегодняшний день флуоресцентных зондов особое место занимают фотоконвертируемые флуоресцентные белки (ФКФБ), способные к необратимому изменению положения максимума эмиссии флуоресценции под действием света. Были изучены основные физико-химические свойства ФКФБ, выделенного из Montastraea cavernosa (mcavGR). Показано, что mcavGR обладает высокой яркостью и контрастностью фотопревращения, сравнимой с лучшими гомологичными ФКФБ, такими как Kaede, dendFP, Dendra2, EosFP и др., и в то же время обладает значительной фотостабильностью.
ВВЕДЕНИЕ
Флуоресцентные метки – одни из основных инструментов для визуализации процессов в живых системах [1, 2]. Генетически кодируемые зонды на основе флуоресцентных белков (ФБ) обладают рядом уникальных свойств и перекрывают практически весь видимый диапазон от синего до красного цвета [3–7]. Несмотря на определенный “провал” в желтой области (максимумы эмиссии ~545 нм) и невысокую яркость белков с эмиссией в красном диапазоне (>620 нм), ФБ используются для решения самого широкого спектра задач in vitro и in vivo [8–12]. Отдельный класс представляют собой зонды на основе фотоконвертируемых флуоресцентных белков (ФКФБ), способных к изменению цвета за счет батохромного смещения максимумов возбуждения и эмиссии флуоресценции в ответ на облучение УФ-светом [13, 14]. В основном эти зонды представлены белками семейства Kaede и способны к фотопревращению из зеленого флуоресцентного состояния в красное [15, 16]. Такая фотоконверсия происходит за счет расширения π-системы хромофора белка в результате реакций фотоэлиминирования [17, 18] (рис. 1). Эти свойства в сочетании с возможностью генетического кодирования позволяют применять ФКФБ для прицельного мечения и наблюдения за перемещением объектов внутри клетки [19–21], а также в рамках целого организма [22, 23]. ФКФБ были использованы также при разработке новых методов суперразрешающей флуоресцентной микроскопии, которые позволяют преодолеть дифракционный барьер и получать снимки с разрешением до нескольких десятков нанометров [24–27]. В то же время ФКФБ имеют ряд характеристик, ограничивающих их применение. В частности, фотоконверсия, как правило, происходит в результате облучения УФ-светом, токсичного для клеток или модельного организма. Тетрамерная природа большинства флуоресцентных белков дикого типа вносит дополнительные ограничения для их использования из-за склонности к агрегации. При помощи направленного мутагенеза были получены мономерные варианты ряда ФКФБ [19, 21, 28–30], однако после мономеризации часто ухудшается эффективность их созревания и фотофизические характеристики, такие как квантовый выход флуоресценции, фотостабильность, степень фотоконверсии. На сегодняшний день поиск и получение ФКФБ с улучшенными свойствами по-прежнему остаются актуальными.
Ранее сообщалось, что флуоресцентный белoк, выделенный из кораллового полипа Montastraea cavernosa (mcavGR), способен к смещению максимумов возбуждения/эмиссии флуоресценции из зеленой области в красную [31], однако полная информация о его физико-химических свойствах с тех пор не появлялась. В настоящей работе мы попытались восполнить этот пробел и подвергли всестороннему изучению физико-химические свойства белка mcavGR в сравнении с другими ФКФБ.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Согласно проведенному нами филогенетическому анализу, mcavGR принадлежит к той же группе ФБ, что и ФКФБ Kaede [15], EosFP [32], dendFP [33], а также ФБ Dronpa [34], AzamiGreen [35], kikG [36] и cFP484 [37]. Ближайшими гомологами, помимо других ФБ из Montastraea cavernosa [38, 39], являются Kaede и EosFP (степень гомологии 77 и 79% соответственно), а также неприродный белок-предшественник LEA (least evolved ancestor, степень гомологии 88%) [40, 41]. В работе Labas et al. [31] mcavGR, как и другой гомологичный белок rfloRFP, был описан впервые как красный флуоресцентный белок (mcavRFP) из-за наблюдаемой окраски в конечном состоянии после фотопревращения, хотя в отсутствие света белок синтезируется в зеленой форме. Мы не стали переименовывать белок в mcavFP, т.к. под mcavFP иногда подразумевается другой гомолог, также выделенный из Montastraea cavernosa [42, 43].
Как и у других Kaede-подобных белков, хромофор mcavGR образован из аминокислотной последовательности -His-Tyr-Gly- [31]. Способность к фотоконверсии под действием УФ-света, вероятно, обусловлена реакцией фотоэлиминирования, сопровождающейся расширением π‑системы исходного хромофора за счет имидазолэтиленильной группы (рис. 1). В настоящей работе фотоконверсионные свойства mcavGR сравнивали с широко используемым ФКФБ Dendra2, выступающими мономерным, ярким и, в отличие от других гомологов, способным также к ограниченной фотоконверсии под действием цианового (488 нм) света, а также с природным предшественником Dendra2 – dendFP. Для сравнения с другими ФКФБ использовали литературные данные (табл. 1).
Таблица 1.
Физико-химические свойства mcavGR в сравнении с другими фотоконвертируемыми флуоресцентными белками
| Флуоресцентный белок | Олигомерное состояние | До фотоконверсии | После фотоконверсии | Ссылки | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| λ возб./эм., нм | ε*, М–1 см–1 | QY | яркость/1000 | рКа | λ возб./эм., нм | ε*, М–1 см–1 | QY | яркость/1000 | рКа | |||
| mcavGR | Тетрамер | 506/517 | 106 000 | 0.79 | 83.74 | 6.7 | 572/581 | 87 200 | 0.70 | 61.04 | 6.8 | Данная работа |
| Kaede | Тетрамер | 508/518 | 98 800 | 0.80 | 79.04 | 5.6 | 572/580 | 60 400 | 0.33 | 19.93 | 5.7 | [15, 16] |
| dendFP | Димер** | 492/508 | 82 600 | 0.76 | 62.78 | 6.5 | 557/575 | 73 000 | 0.64 | 46.72 | 6.2 | [44, 45] |
| Dendra2 | Мономер | 490/507 | 45 000 | 0.50 | 22.50 | 6.6 | 553/573 | 35 000 | 0.55 | 19.25 | 6.9 | [19] |
| KikGR | Тетрамер | 507/517 | 28 200 | 0.70 | 19.74 | 7.8 | 583/593 | 32 600 | 0.65 | 21.19 | 5.5 | [36] |
| KikGRX | Тетрамер | 507/517 | 20 300 | 0.70 | 14.21 | 8.0 | 583/593 | 28 600 | 0.65 | 18.20 | 5.7 | [46] |
| mKikGR | Мономер | 505/515 | 49 000 | 0.69 | 33.81 | 6.6 | 580/591 | 28 000 | 0.63 | 17.64 | 5.2 | [28] |
| mClavGR2 | Мономер | 488/504 | 19 000 | 0.77 | 14.63 | 8.0 | 566/583 | 32 000 | 0.53 | 16.96 | 7.3 | [21] |
| mMaple | Мономер | 489/505 | 15 000 | 0.74 | 11.10 | 8.2 | 566/583 | 30 000 | 0.56 | 16.80 | 7.3 | [30] |
| EosFP | Тетрамер | 506/516 | 72 000 | 0.70 | 50.40 | 5.8 | 571/581 | 41 000 | 0.55 | 22.55 | 6.8 | [32] |
| mEosFP | Мономер | 505/516 | 67 200 | 0.64 | 43.01 | 5.3 | 569/581 | 37 000 | 0.62 | 22.94 | 6.5 | [29, 32] |
| mEos4b | Мономер | 505/516 | 78 200 | 0.84 | 65.69 | 5.5 | 570/580 | 55 500 | 0.71 | 39.40 | 5.8 | [43, 47] |
| LEA | Тетрамер | 504/513 | 86 800 | 0.81 | 70.31 | 6.3 | 571/580 | ND | ND | ND | ND | [40] |
Прежде всего мы провели анализ олигомерного состояния mcavGR с помощью гель-фильтрации. С использованием EGFP, dendFP и DsRed в качестве белков сравнения было показано, что mcavGR – тетрамер, как и большинство других природных представителей белков семейства Kaede.
В исходном состоянии mcavGR – типичный зеленый флуоресцентный белок с максимумами возбуждения/эмиссии флуоресценции при 506/517 нм соответственно, которые под действием света смещаются на ~65 нм в красную область спектра (рис. 2а, 2б). При понижении рН наблюдается уменьшение поглощения при 506 нм с одновременным ростом при 385 нм зеленой формы белка до фотопревращения (рис. 3а). Ранее было показано, что такое поведение связано с кислотно-основным равновесием фенольной группы хромофора [44, 48]. рКа этого перехода оказалось равным 6.66 ± 0.03, что является сопоставимым со значениями рКа для других гомологов. Отметим, что фотоконверсия белка запускается светом, возбуждающим протонированную форму хромофора, поэтому низкие значения рКа кислотно-основного равновесия хромофора затрудняют конверсию при физиологических рН ~ 7.5 за счет смещения равновесия в сторону анионного хромофора. С другой стороны, при высоких значениях рКа яркость белка падает при нейтральных значениях рН за счет снижения концентрации анионного хромофора. В этом ключе mcavGR имеет “средние” значения рКа относительно других Kaede-подобных белков (табл. 1). Фотоконвертированный белок при закислении среды, аналогично другим Kaede-подобным белкам, переходит из красной формы в зеленую со сдвигом максимумов поглощения от 572 к 460–490 нм и эмиссии флуоресценции от 581 к 510–540 нм с рКа = 6.78 ± 0.02 (рис. 2в, 3б).
Рис. 2.
Фотофизические свойства mcavGR. (а) – Спектры поглощения в ходе фотоконверсии при облучении УФ-светом (365 нм); (б) – спектры возбуждения и эмиссии флуоресценции до (зеленый цвет) и после (красный цвет) фотоконверсии при рН 8; (в) – спектры возбуждения и эмиссии флуоресценции фотоконвертированного белка при рН 5. Положение максимумов в нанометрах показано числами над соответствующими пиками.

Рис. 3.
Зависимость спектров поглощения mcavGR от рН до (а) и после (б) фотоконверсии. Кривые титрования, рассчитанные по максимуму поглощения анионной формы (506 нм (а) и 572 нм (б)), приведены на вставках.

Одним из ключевых параметров ФБ, характеризующих их применимость в флуоресцентной микроскопии, выступает яркость, определяемая как произведение квантового выхода и молярного коэффициента экстинкции. Согласно полученным данным, mcavGR имеет коэффициенты экстинкции в максимумах поглощения анионных форм хромофора 106 000 и 87 200 М–1 см–1 и квантовые выходы 0.79 и 0.70 для исходной и фотоконвертированной форм соответственно. С учетом данных, описанных в литературе (табл. 1), mcavGR – один из самых ярких ФКФБ, имеющих значительные перспективы для дальнейшего применения.
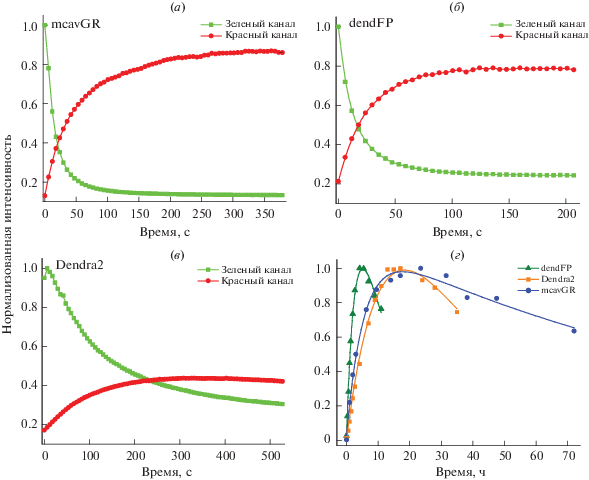
Кинетические параметры фотопревращения ФБ зависят в значительной степени от используемого оборудования и его настройки в конкретном эксперименте, поэтому оценку фотоконверсионных свойств mcavGR проводили в сравнении с другими реперными белками в тех же условиях. Как было отмечено выше, в качестве реперных белков использовали ФКФБ Dendra2 и его природный предшественник dendFP. Анализ кинетики фотопревращения белков под действием синего света (405 нм) в условиях микроскопии показал, что mcavGR конвертируется несколько медленнее dendFP, но быстрее его мономерного варианта Dendra2 (табл. 2, рис. 4а–4в). В то же время из рис. 4а–4в видно, что mcavGR демонстрирует более высокий контраст в ходе фотопревращения. Анализ фотостабильности белков при длительном облучении УФ-светом (365 нм) в одинаковых условиях показал, что mcavGR дольше других сохраняет флуоресцентные свойства (рис. 4г, табл. 2).
Таблица 2.
Фотоконверсионные свойства mcavFP в сравнении с белками dendFP и Dendra2
| Флуоресцентный белок | k (G → R, 405 нм, микро)*, мин–1 | k (G → R, 365 нм)**, ч–1 | k (обесцвеч., 365 нм)**, ч–1 |
|---|---|---|---|
| mcavGR | 1.11 ± 0.03 | 0.18 ± 0.01 | 0.008 ± 0.001 |
| dendFP | 2.22 ± 0.04 | 0.40 ± 0.04 | 0.076 ± 0.013 |
| Dendra2 | 0.51 ± 0.04 | 0.12 ± 0.02 | 0.018 ± 0.007 |
Рис. 4.
Кинетика фотопревращений mcavGR в сравнении с белками dendFP и Dendra2. Представлены кривые зелено-красной фотоконверсии mcavGR (а), dendFP (б) и Dendra2 (в) под действием синего (405 нм) света в условиях широкопольной флуоресцентной микроскопии; (г) – динамика изменения поглощения при 570 нм в ходе зелено-красной конверсии и фотодеградации белков под действием УФ-света (365 нм).
Таким образом, нами были исследованы основные физико-химические свойства белка mcavGR. Показано, что в растворе он – тетрамер. Изучена кинетика превращения белка mcavGR из зеленого состояния в красное под действием света, а также его фотостабильность. Проанализированы основные фотофизические характеристики, такие как положение максимумов поглощения и флуоресценции, коэффициенты экстинкции, квантовые выходы флуоресценции, яркость. Показано, что в сравнении с другими белками Kaede-семейства mcavGR обладает высокой яркостью в исходном и фотоконвертированном состояниях, а также является фотостабильным. Эти свойства подтверждают, что mcavGR имеет значительный потенциал для дальнейшего применения в качестве фотоконвертируемого генетически кодируемого флуоресцентного зонда, особенно в случае получения его мономерного варианта.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Филогенетический анализ проводили с использованием последовательностей белков, взятых из базы данных FPbase [43]. Выравнивание последовательностей осуществляли при помощи утилиты MAFFT v.7.467 [49]. Для построения филогенетического дерева использовали програму Seaview4 [50] совместно с программой PhyML 3.0 [51].
Наработка и выделение белков. Клетки Escherichia сoli штамма JM-109 трансформировали плазмидой pQE-30, несущей ген целевого флуоресцентного белка (mcavGR, dendFP, Dendra2, EGFP, DsRed), и выращивали в среде LB, содержавшей ампициллин в концентрации 100 мкг/мл. Клеточную культуру выращивали при постоянном перемешивании (250 об./мин) и температуре 37°С в течение ночи, затем температуру снижали до 22°С и продолжали перемешивание клеточной суспензии в течение суток для максимального созревания белка. Осаждение клеточной биомассы осуществляли центрифугированием (3000 g) в течение 15 мин при 4°С, осадок затем ресуспендировали в промывочном буфере (50 мМ Tris-HCl, 300 мМ NaCl, 20 мМ имидазол, pH 8.0) и охлаждали во льду. Далее клетки лизировали при помощи френч-пресса (Ashcroft, США) при давлении 2000 psi. Получившийся клеточный лизат центрифугировали при 75 000 g в течение 20 мин при 4°С. Целевые белки на своем N-конце содержали гексагистидиновую последовательность, поэтому их выделение осуществляли методом металл-хелатной хроматографии на Ni-NTA-агарозе (Qiagen, США). После нанесения клеточного лизата на хроматографическую колонку, предварительно уравновешенную промывочным буфером, проводили промывку 4-кратным объемом того же буфера. Элюирование белков осуществляли буфером, содержавшим 50 мМ Tris-HCl, 300 мМ NaCl, 250 мМ имидазол, pH 8.0.
Гель-фильтрационный анализ проводили с использованием хроматографа ACTA FPLC (GE Healthcare, Швеция) со скоростью потока 0.5 мл/мин. Колонку Superdex 200 (300 × 10 мм; GE Healthcare, Швеция) уравновешивали буфером, содержавшим 20 мМ Tris-HCl, 100 мМ NaCl (pH 8.0), и наносили образцы белков с концентрацией ~1.5 мг/мл. Детекцию белковых фракций осуществляли при 280 нм. В качестве белков сравнения для определения олигомерного состояния mcavGR использовали мономерный EGFP [52] и тетрамерный DsRed [53], в качестве димера – dendFP [45].
Фотофизическая характеризация. Спектры поглощения исследуемых белков регистрировали на спектрофотометре Cary 50 Bio (Varian, США) при комнатной температуре. Флуоресцентные свойства белков изучали в тех же условиях при помощи спектрофлуориметра Cary Eclipse (Varian, США). Определение квантовых выходов и молярных коэффициентов экстинкции белка mcavGR до и после фотоконверсии проводили в соответствии с методикой, использованной в работе Pletneva et al. [45]. В качестве стандартов для определения квантового выхода до и после фотоконверсии использовали EGFP и DsRed соответственно. Для определения коэффициента экстинкции mcavGR до фотопревращения концентрацию полностью созревшего белка рассчитывали методом Ward [54] по поглощению белка после денатурации в щелочной среде и с учетом коэффициента экстинкции GFP-хромофора 44 000 М–1 см–1. Коэффициент экстинкции красной формы белка определяли с учетом остаточного количества неконвертированного белка согласно Pletneva et al. [45]. Яркость белка определяли как произведение значений квантового выхода флуоресценции и молярного коэффициента экстинкции.
Фотометрическое титрование. Для получения кривых титрования белок с помощью гель-фильтрации на колонке NAP-10 (GE Healthcare, США) переводили в буферный раствор, содержавший 12.5 мМ CH3COONa, 12.5 мМ СAPS, 12.5 мМ NaH2PO4, 12.5 мМ Tris-HCl, 100 мМ NaCl, pH 7.5. Титрование осуществляли добавлением аликвот растворов 0.5 М HCl либо 0.5 М NaOH к 100 мкл белка. После каждого добавления раствора кислоты или щелочи образцы белка ресуспендировали, измеряли pH при помощи pH-метра Orion Star A211 (Thermo Fisher Scientific, США) с использованием микро-рН-электрода Orion 8220BNWP (Thermo Fisher Scientific, США) и снимали спектр поглощения белка. Для определения константы кислотно-основного равновесия хромофора mcavGR cтроили зависимость поглощения анионной формы хромофора от изменения рН с учетом разбавления анализируемых образцов и проводили аппроксимацию согласно уравнению Henderson–Hasselbalch [55].
Изучение скорости реакции зелено-красной фотоконверсии выполняли на белках, иммобилизованных на гранулах Ni-NTA-агарозы, в промывочном буфере при комнатной температуре. Исследование проводили на инвертированном флуоресцентном микроскопе DMI6000 (Leica, Германия). Образец гранул с отдельным белком облучали фиолетовым светом (405 ± 20 нм). Использовали объектив с 63-кратным увеличением и набор светофильтров для регистрации зеленого и красного флуоресцентных сигналов (GFP и CY3 соответственно). Константы скорости зелено-красной фотоконверсии определяли при помощи программного обеспечения биохимического симулирования кинетики COPASI [56] с использованием схемы необратимых реакций первого порядка G → R → C, где G – исходная зеленая форма, R – красная форма, C – продукт фотообесцвечивания красной формы. Параметры реакций были определены по анализу кинетики для красной формы белка при помощи методов дифференциальной эволюции (Differential Evolution).
Исследование динамики фотоконверсии и фотообесцвечивания проводили с использованием УФ-трансиллюминатора CL-215 (Ultra-Violet Products, США) при температуре 4°С для минимизации фотообесцвечивания. Эксперименты по изучению фотодеградации осуществляли при комнатной температуре. Образцы белков с одинаковыми концентрациями (~0.2 мг/мл) облучали светом с длиной волны 365 нм в течение заданного времени, после чего снимали спектры поглощения белков. Кинетические параметры реакций определяли с использованием программы COPASI, как описано выше.
Список литературы
Chudakov D.M., Matz M.V., Lukyanov S., Lukyanov K.A. // Physiol. Rev. Am. Physiological Soc. 2010. V. 90. P. 1103–1163. https://doi.org/10.1152/physrev.00038.2009
Martynov V.I., Pakhomov A.A., Popova N.V., Deyev I.E., Petrenko A.G. // Acta Naturae. 2016. V. 8. P. 33–46.
Tomosugi W., Matsuda T., Tani T., Nemoto T., Kotera I., Saito K., Horikawa K., Nagai T. // Nat. Methods. 2009. V. 6. P. 351–353. https://doi.org/10.1038/nmeth.1317
Shinoda H., Ma Y., Nakashima R., Sakurai K., Matsuda T., Nagai T. // Cell Chem. Biol. 2018. V. P. 330e7–338e7. https://doi.org/10.1016/j.chembiol.2017.12.005
Pakhomov A.A., Martynov V.I. // Biochem. Biophys. Res. Commun. 2011. V. 407. P. 230–235. https://doi.org/10.1016/j.bbrc.2011.03.004
Pakhomov A.A., Frolova A.Y., Tabakmakher V.M., Chugunov A.O., Efremov R.G., Martynov V.I. // J. Photochem. Photobiol. B. 2020. V. 206. P. 111 853. https://doi.org/10.1016/j.jphotobiol.2020.111853
Fabritius A., Ng D., Kist A.M., Erdogan M., Portugues R., Griesbeck O. // Cell Chem. Biol. 2018. V. 25. P. 1554e8–1561e8. https://doi.org/10.1016/j.chembiol.2018.08.008
Kostyuk A.I., Demidovich A.D., Kotova D.A., Belousov V.V., Bilan D.S. // Int. J. Mol. Sci. 2019. V. 20. P. 4200. https://doi.org/10.3390/ijms20174200
Martynov V.I., Pakhomov A.A., Deyev I.E., Petrenko A.G. // Biochim. Biophys. Acta Gen. Subj. 2018. V. 1862. P. 2924–2939. https://doi.org/10.1016/j.bbagen.2018.09.013
Bukhari H., Müller T. // Trends Cell Biol. 2019. V. 29. P. 912–928. https://doi.org/10.1016/j.tcb.2019.08.004
Можаев А.А., Серова О.В., Орса А.Н., Бойко А.А., Горященко А.С., Деев И.Е., Петренко А.Г. // Биоорг. химия. 2019. Т. 45. С. 213–217. [Mozhaev A.A., Serova O.V., Orsa A.N., Boyko A.A., Goryashchenko A.S., Deyev I.E., Petrenko A.G. // Russ. J. Bioorg. Chem. 2019. V. 45. P. 179–182.] https://doi.org/10.1134/S1068162019020080
Злобовская О.А., Ширманова М.В., Ковалева Т.Ф., Саркисян К.С., Загайнова Е.В., Лукьянов К.А. // Биоорг. химия. 2019. Т. 45. С. 17–26. [Zlobovskaya O.A., Shirmanova M.V., Kovaleva T.F., Sarkisyan K.S., Zagaynova E.V., Lukyanov K.A. // Russ. J. Bioorg. Chem. 2018. V. 44. P. 645–652.] https://doi.org/10.1134/S1068162018060109
Adam V. // Histochem. Cell Biol. 2014. V. 142. P. 19–41. https://doi.org/10.1007/s00418-014-1190-5
Nemet I., Ropelewski P., Imanishi Y. // Photochem. Photobiol. Sci. 2015. V. 14. P. 1787–1806. https://doi.org/10.1039/C5PP00174A
Ando R., Hama H., Yamamoto-Hino M., Mizuno H., Miyawaki A. // Proc. Natl. Acad. Sci. USA. 2002. V. 99. P. 12 651–12 656. https://doi.org/10.1073/pnas.202320599
Fron E., Sliwa M., Adam V., Michiels J., Rocha S., Dedecker P., Hofkens J., Mizuno H. // Photochem. Photobiol. Sci. 2014. V. 13. P. 867–874. https://doi.org/10.1039/c3pp50335f
Mizuno H., Mal T.K., Tong K.I., Ando R., Furuta T., Ikura M., Miyawaki A. // Mol. Cell. 2003. V. 12. P. 1051–1058. https://doi.org/10.1016/S1097-2765(03)00393-9
Li X., Chung L.W., Mizuno H., Miyawaki A., Morokuma K. // J. Phys. Chem. B. 2010. V. 114. P. 16 666–16 675. https://doi.org/10.1021/jp1101779
Chudakov D.M., Lukyanov S., Lukyanov K.A. // Nat. Protoc. 2007. V. 2. P. 2024–2032. https://doi.org/10.1038/nprot.2007.291
Baker S.M., Buckheit R.W., Falk M.M. // BMC Cell Biol. 2010. V. 11. P. 15. https://doi.org/10.1186/1471-2121-11-15
Hoi H., Shaner N.C., Davidson M.W., Cairo C.W., Wang J., Campbell R.E. // J. Mol. Biol. 2010. V. 401. P. 776–791. https://doi.org/10.1016/j.jmb.2010.06.056
Moeyaert B., Holt G., Madangopal R., Perez-Alvarez A., Fearey B.C., Trojanowski N.F., Ledderose J., Zolnik T.A., Das A., Patel D., Brown T.A., Sachdev R.N.S., Eickholt B.J., Larkum M.E., Turrigiano G.G., Dana H., Gee C.E., Oertner T.G., Hope B.T., Schreiter E.R. // Nat. Commun. 2018. V. 9. P. 4440. https://doi.org/10.1038/s41467-018-06935-2
Osugi T., Sasakura Y., Satake H. // Sci. Rep. 2020. V. 10. P. 1892. https://doi.org/10.1038/s41598-020-58884-w
Betzig E., Patterson G.H., Sougrat R., Lindwasser O.W., Olenych S., Bonifacino J.S., Davidson M.W., Lippincott-Schwartz J., Hess H.F. // Science. 2006. V. 313. P. 1642–1645. https://doi.org/10.1126/science.1127344
McKinney S.A., Murphy C.S., Hazelwood K.L., Davidson M.W., Looger L.L. // Nat. Methods. 2009. V. 6. P. 131–133. https://doi.org/10.1038/nmeth.1296
Nienhaus K., Ulrich Nienhaus G. // Chem. Soc. Rev. 2014. V. 43. P. 1088–1106. https://doi.org/10.1039/C3CS60171D
Robert A., Hookway C., Gelfand V.I. // BioEssays. 2016. V. 38. P. 232–243. https://doi.org/10.1002/bies.201500142
Habuchi S., Tsutsui H., Kochaniak A.B., Miyawaki A., van Oijen A.M. // PLoS One. 2008. V. 3. P. 1–9. https://doi.org/10.1371/journal.pone.0003944
Adam V., Moeyaert B., David C.C., Mizuno H., Lelimousin M., Dedecker P., Ando R., Miyawaki A., Michiels J., Engelborghs Y., Hofkens J. // Chem. Biol. 2011. V. 18. P. 1241–1251.
McEvoy A.L., Hoi H., Bates M., Platonova E., Cranfill P.J., Baird M.A., Davidson M.W., Ewers H., Liphardt J., Campbell R.E. // PLoS One. 2012. V. 7. P. e51 314. https://doi.org/10.1371/journal.pone.0051314
Labas Y.A., Gurskaya N.G., Yanushevich Y.G., Fradkov A.F., Lukyanov K.A., Lukyanov S.A., Matz M.V. // Proc. Natl. Acad. Sci. USA. 2002. V. 99. P. 4256–4261. https://doi.org/10.1073/pnas.062552299
Wiedenmann J., Ivanchenko S., Oswald F., Schmitt F., Rocker C., Salih A., Spindler K.-D., Nienhaus G.U. // Proc. Natl. Acad. Sci. USA. 2004. V. 101. P. 15 905–15 9910. https://doi.org/10.1073/pnas.0403668101
Пахомов А.А., Мартынова Н.Ю., Гурская Н.Г., Балашова Т.А., Мартынов В.И. // Биохимия. 2004. Т. 69. С. 1108–1117. [Pakhomov A.A. // Biochemistry (Moscow). 2004. V. 69. P. 901–908.] https://doi.org/10.1023/B:BIRY.0000040223.09641.29
Ando R., Mizuno H., Miyawaki A. // Science. 2004. V. 306. P. 1370–1373. https://doi.org/10.1126/science.1102506
Karasawa S., Araki T., Yamamoto-Hino M., Miyawaki A. // J. Biol. Chem. 2003. V. 278. P. 34 167–34 171. https://doi.org/10.1074/jbc.M304063200
Tsutsui H., Karasawa S., Shimizu H., Nukina N., Miyawaki A. // EMBO Rep. 2005. V. 6. P. 233–238. https://doi.org/10.1038/sj.embor.7400361
Ai H.W. // Biochem. J. 2006. V. 400. P. 531–540.
Kelmanson I.V., Matz M.V. // Mol. Biol. Evol. 2003. V. 20. P. 1125–1133. https://doi.org/10.1093/molbev/msg130
Field S.F., Bulina M.Y., Kelmanson I.V, Bielawski J.P., Matz M.V. // J. Mol. Evol. 2006. V. 62. P. 332–339. https://doi.org/10.1007/s00239-005-0129-9
Kim H., Grunkemeyer T.J., Modi C., Chen L., Fromme R., Matz M.V., Wachter R.M. // Biochemistry. 2013. V. 52. P. 8048–8059. https://doi.org/10.1021/bi401000e
Kim H., Zou T., Modi C., Dörner K., Grunkemeyer T.J., Chen L., Fromme R., Matz M.V., Ozkan S.B., Wachter R.M. // Structure. 2015. V. 23. P. 34–43. https://doi.org/10.1016/j.str.2014.11.011
Mazel C.H., Lesser M.P., Gorbunov M.Y., Barry T.M., Farrell J.H., Wyman K.D., Falkowski P.G. // Limnol. Oceanogr. 2003. V. 48. P. 402–411. https://doi.org/10.4319/lo.2003.48.1_part_2.0402
Lambert T.J. // Nat. Methods. 2019. V. 16. P. 277–278. https://doi.org/10.1038/s41592-019-0352-8
Пахомов А.А., Черткова Р.В., Мартынов В.И. // Биоорг. химия. 2015. Т. 41. С. 669–674. [Pakhomov A.A., Chertkova R. V., Martynov V.I. // Russ. J. Bioorg. Chem. 2015. V. 41. P. 602–606.] https://doi.org/10.1134/S1068162015060114
Pletneva N.V., Pletnev S., Pakhomov A.A., Chertkova R.V., Martynov V.I., Muslinkina L., Dauter Z., Pletnev V.Z. // Acta Crystallogr. Sect. D Struct. Biol. 2016. V. 72. P. 922–932. https://doi.org/10.1107/S205979831601038X
Tsutsui H., Shimizu H., Mizuno H., Nukina N., Furuta T., Miyawaki A. // Chem. Biol. 2009. V. 16. P. 1140–1147. https://doi.org/10.1016/j.chembiol.2009.10.010
Paez-Segala M.G., Sun M.G., Shtengel G., Viswanathan S., Baird M.A., Macklin J.J., Patel R., Allen J.R., Howe E.S., Piszczek G., Hess H.F., Davidson M.W., Wang Y., Looger L.L. // Nat. Methods. 2015. V. 12. P. 215–218. https://doi.org/10.1038/nmeth.3225
Pakhomov A.A., Martynov V.I., Orsa A.N., Bondarenko A.A., Chertkova R.V., Lukyanov K.A., Petrenko A.G., Deyev I.E. // Biochem. Biophys. Res. Commun. 2017. V. 493. P. 1518–1521. https://doi.org/10.1016/j.bbrc.2017.09.170
Katoh K., Toh H. // Bioinformatics. 2007. V. 23. P. 372–374. https://doi.org/10.1093/bioinformatics/btl592
Gouy M., Guindon S., Gascuel O. // Mol. Biol. Evol. 2010. V. 27. P. 221–224. https://doi.org/10.1093/molbev/msp259
Guindon S., Dufayard J.-F., Lefort V., Anisimova M., Hordijk W., Gascuel O. // Syst. Biol. 2010. V. 59. P. 307–321. https://doi.org/10.1093/sysbio/syq010
Orm M., Cubitt A.B., Kallio K., Gross L.A., Tsien R.Y., Remington S.J. // Science. 1996. V. 273. P. 1392–1395. https://doi.org/10.1126/science.273.5280.139
Baird G.S., Zacharias D.A., Tsien R.Y. // Proc. Natl. Acad. Sci. USA. 2000. V. 97. P. 11 984–11 989. https://doi.org/10.1073/pnas.97.22.11984
Ward W.W. // Green Fluorescent Protein: Properties, Applications, and Protocols. 2nd ed. / Eds. Chalfie M., Kain S.R. John Wiley & Sons, Inc., 2005. P. 39–65. https://doi.org/10.1002/0471739499.ch3
Sniegowski J.A., Phail M.E., Wachter R.M. // Biochem. Biophys. Res. Commun. 2005. V. 332. P. 657–663. https://doi.org/10.1016/j.bbrc.2005.04.166
Hoops S., Sahle S., Gauges R., Lee C., Pahle J., Simus N., Singhal M., Xu L., Mendes P., Kummer U. // Bioinformatics. 2006. V. 22. P. 3067–3074. https://doi.org/10.1093/bioinformatics/btl485
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия



