Биоорганическая химия, 2021, T. 47, № 2, стр. 276-286
Фотоактивируемая система CRISPR/Cas9
Е. А. Ахметова 1, 3, 2, В. М. Голышев 1, 3, И. П. Вохтанцев 1, 3, М. И. Мещанинова 1, А. Г. Веньяминова 1, Д. С. Новопашина 1, 3, 2, *
1 ФГБУН “Институт химической биологии и фундаментальной медицины” СО РАН
630090 Новосибирск, пр. акад. Лаврентьева, 8, Россия
2 Научно-технологический университет “Сириус”
354383 Сочи, Олимпийский просп., 1, Россия
3 Новосибирский государственный университет
630090 Новосибирск, ул. Пирогова, 1, Россия
* E-mail: danov@niboch.nsc.ru
Поступила в редакцию 18.09.2020
После доработки 28.09.2020
Принята к публикации 30.09.2020
Аннотация
Разработан подход к созданию фотоактивируемой системы CRISPR/Cas9, содержащей белок Cas9, синтетическую 102-звенную sgРНК или пару направляющих crРНК/tracrРНК и блокирующие фоторасщепляемые олигодезоксирибонуклеотиды. Фоторасщепляемые олигодезоксирибонуклеотиды содержали два или три фоторасщепляемых линкера в своем составе. Термическая стабильность комплексов таких модифицированных олигонуклеотидов с направляющими РНК уменьшалась с увеличением количества линкеров. Исследована кинетика фоторасщепления фотомодифицированных блокирующих олигодезоксирибонуклеотидов. Продемонстрирована in vitro возможность фотоактивации геномного редактирования в результате УФ-облучения при использовании фоторасщепляемых блокирующих олигодезоксирибонуклеотидов в системе CRISPR/Cas9.
ВВЕДЕНИЕ
Облучение светом определенной длины волны позволяет эффективно управлять активностью различных молекулярно-биологических систем in vitro и in vivo [1–3]. Введение светочувствительных молекул в состав олигонуклеотидов или белков дает возможность изменять их активность в клетках с высоким пространственно-временным разрешением. В литературе описаны конструкции, которые можно активировать, например, за счет удаления фотоблокирующей группы под действием света, изменения их вторичной структуры в результате смены конформации введенного фоточувствительного линкера или внесения разрывов в их структуру путем гидролиза фоторасщепляемого линкера. На сегодняшний день известны разнообразные фоторегулируемые конструкции на основе малых интерферирующих РНК (siРНК) [4, 5], микроРНК [6, 7], антисмысловых олигонуклеотидов [8, 9], каталитических нуклеиновых кислот [10, 11], аптамеров [12], а также направляющих РНК для системы CRISPR/Cas9 [13–18].
CRISPR-системы, в частности системы CRISPR/ Cas9 [19, 20], являются перспективным инструментом редактирования генома. Эффекторные комплексы направляющих РНК и Cas-белков позволяют вносить разрывы в заданные положения ДНК или РНК. В качестве направляющих РНК в системе CRISPR/Cas9 используют природный вариант из двух молекул РНК – crРНК, содержащей участок связывания с ДНК-мишенью и участок связывания со второй молекулой РНК, а именно tracrРНК, взаимодействующей с белком Cas9. Другим вариантом является использование химерной протяженной sgРНК, объединяющей в себе последовательности crРНК и tracrРНК [19].
На данный момент предложены два варианта создания фотоактивируемых систем CRISPR/Cas9. Первый вариант подразумевает создание направляющих РНК, содержащих фотогруппы в гетероциклических основаниях, которые блокируют активность CRISPR-системы до облучения светом [13–17]. Во втором варианте используют блокирующие олигонуклеотиды, содержащие один или несколько фоторасщепляемых линкеров на основе 1-(2-нитрофенил)-пропандиола (PL), комплементарные направляющей sgРНК [18]. Такие блокирующие олигонуклеотиды связываются с sgРНК, образуя комплементарный дуплекс, не позволяющий sgРНК сформировать комплекс с ДНК-мишенью и инициировать работу системы CRISPR/Cas9 вплоть до облучения светом.
Целью данного исследования являлась разработка фотоактивируемой системы CRISPR/Cas9, содержащей белок Cas9, синтетическую 102-звенную sgРНК или пару направляющих crРНК/ tracrРНК и блокирующие фоторасщепляемые олигодезоксирибонуклеотиды, и сравнительное изучение влияния длины блокирующего олигонуклеотида и количества фоторасщепляемых линкеров в его составе на эффективность активации обоих вариантов систем.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Для создания фотоактивируемой системы CRISPR/Cas9 нами были синтезированы фоторасщепляемые олигодезоксирибонуклеотиды, комплементарные фрагменту направляющей РНК в области связывания с ДНК-мишенью по аналогии с работой Jain et al. [18].
Для введения фоторасщепляемого линкера был синтезирован фосфитамид на основе 1-(2-нитрофенил)-1,2-этандиола (PL) [4, 8]. С его использованием в ходе автоматического твердофазного фосфитамидного синтеза была получена серия фоторасщепляемых олигодезоксирибонуклеотидов, комплементарных фрагменту направляющей РНК, участвующей в образовании комплекса с ДНК-мишенью (табл. 1). Кроме того, были получены немодифицированные олигодезоксирибонуклеотиды такой же последовательности, не содержащие фоторасщепляемых линкеров.
Таблица 1.
Последовательности и молекулярные массы блокирующих олигодезоксирибонуклеотидов
| Шифр | Последовательность, 5'–3' | Молекулярная масса | |
|---|---|---|---|
| рассчитано | найдено* | ||
| B20_PL3 | TTTTTT-PL-ACAAA-PL-TTGAG-PL-TTATCC | 7421.71 | – |
| B30_PL2 | GCTCTAAAAC-PL-TTTTTTACAA-PL-ATTGAGTTAT | 9657.20 | – |
| B30_PL3 | GCTCTAAA-PL-ACTTTTT-PL-TACAAAT-PL-TGAGTTAT | 9901.32 | – |
| B30_PL2_F | Flu-GCTCTAAAAC-PL-TTTTTTACAA-PL-ATTGAGTTAT | 10 225.69 | – |
| B30_PL3_F | Flu-GCTCTAAA-PL-ACTTTTT-PL-TACAAAT-PL-TGAGTTAT | 10 469.81 | – |
| B20 | TTTTTTACAAATTGAGTTATCC | 6689.36 | 6690.34 |
| B30 | GCTCTAAAACTTTTTTACAAATTGAGTTAT | 9168.94 | 9169.02 |
Все олигодезоксирибонуклеотиды и их производные выделяли методом препаративного гель-электрофореза. Следует отметить, что электрофоретическая подвижность олигодезоксирибонуклеотидов, содержащих фоторасщепляемый линкер, была меньше в сравнении с подвижностью немодифицированных олигодезоксирибонуклеотидов с такой же нуклеотидной последовательностью. Строение олигонуклеотидов подтверждали методом MALDI-TOF-масс-спектрометрии (табл. 1). Олигодезоксирибонуклеотиды, содержащие фоторасщепляемый линкер, скорее всего, гидролизуются по положениям введения фоторасщепляемых линкеров в процессе ионизации лазером, в результате чего не удалось получить спектры целых молекул. В масс-спектрах обнаруживали лишь пики, соответствующие массам фрагментов олигодезоксирибонуклеотидов, образующихся при гидролизе.
Кинетику расщепления олигодезоксирибонуклеотидов, содержащих фоторасщепляемые линкеры, исследовали путем анализа облученных в течение определенного времени светом с длиной волны 365 нм водных растворов их 5'-флуоресцеин-модифицированных аналогов методом денатурирующего гель-электрофореза с флуоресцентной визуализацией геля (рис. 1а).
Рис. 1.
(а) – Пример электрофоретического анализа расщепления флуоресцентно-меченых фотомодифицированных олигодезоксирибонуклеотидов B30_PL3_F и B30_PL2_F; (б) – кинетические кривые расщепления B30_PL3_F и B30_PL2_F.

Сравнительное исследование расщепления флуоресцентно-меченых олигодезоксирибонуклеотидов (B30_PL3_F и B30_PL2_F), содержащих фоторасщепляемые линкеры, показало, что полное расщепление обоих олигодезоксирибонуклеотидов происходит уже через 5 мин облучения (рис. 1б). При этом полное расщепление олигодезоксирибонуклеотида с тремя фотолабильными вставками происходит быстрее.
В качестве направляющих РНК для системы CRISPR/Cas9 автоматическим твердофазным фосфитамидным методом были синтезированы 102‑звенная sgРНК и пара crРНК/tracrРНК (табл. 2).
Таблица 2.
Последовательности химически синтезированных направляющих РНК
| РНК (длина) | Последовательность олигорибонуклеотида*, 5'–3' |
|---|---|
| sgРНК (102 нт) | GGAUAACUCAAUUUGUAAAAAAGUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGG-CUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUU |
| crРНК (42 нт) | AUAACUCAAUUUGUAAAAAAGUUUUAGAGCUAUGCUGUUUUG |
| tracrРНК (74 нт) | AACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCAC-CGAGUCGGUGCUUUUUUU |
С использованием плазмиды pBS2SKM Psp2 TTG на основе вектора pBluescript II SK(–) со вставкой протоспейсерной последовательности и PAM (5'-TGG-3') в качестве ДНК-мишени проводили исследование способности фоторасщепляемых олигодезоксирибонуклеотидов, комплементарных направляющей РНК, блокировать работу системы CRISPR/Cas9 до облучения и активировать ее действие после облучения. Блокирующие олигонуклеотиды были сконструированы таким образом, чтобы образовывать комплементарный дуплекс с направляющей РНК и препятствовать формированию комплекса с двуцепочечной ДНК-мишенью (плазмидой) (рис. 2).
Рис. 2.
Система CRISPR/Cas9, включающая двуцепочечную ДНК-мишень, направляющие РНК и блокирующие фоторасщепляемые ДНК до облучения (а, в) и после облучения (б, г). (а, б) – Система, содержащая пару направляющих crРНК/tracrРНК; (в, г) – система, содержащая химерную направляющую sgРНК.

В результате облучения блокирующий олигодезоксирибонуклеотид гидролизуется по сайтам введения фоторащепляемых линкеров, и РНК высвобождается из дуплекса, после чего запускается действие системы CRISPR/Cas9.
Предварительно была проверена термическая стабильность дуплексов блокирующих олигодезоксирибонуклеотидов, содержащих фоторасщепляемые линкеры, с направляющей РНК на примере crРНК (рис. 3).
Рис. 3.
Температуры плавления структурированной РНК-мишени (crРНК) и ее дуплексов с блокирующими олигонуклеотидами (B20 и B30) и их фотомодифицированными аналогами (B20_PL3, B30_PL2 и B30_PL3). Условия см. в “Эксперим. части”.

Сравнительное исследование показало, что введение фоторасщепляемых линкеров уменьшает термическую стабильность дуплексов блокирующих олигонуклеотидов с crРНК, при этом увеличение числа линкеров также отрицательно сказывается на стабильности. Как и ожидалось, более протяженные олигодезоксирибонуклеотиды формировали более стабильные дуплексы.
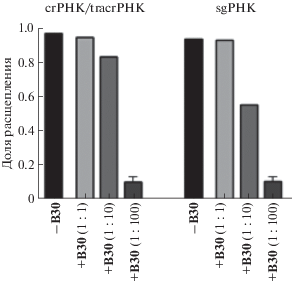
В результате исследования расщепления плазмидной ДНК белком Cas9 в присутствии направляющих РНК и блокирующих ДНК оказалось, что необходим достаточно большой избыток блокирующих ДНК для ингибирования системы CRISPR/ Cas9. Так, 10-кратный избыток блокирующего олигонуклеотида B30 снижал эффективность расщепления мишени лишь незначительно (рис. 4). Для сравнительного исследования нами был выбран вариант с использованием 100-кратного избытка блокирующих олигодезоксирибонуклеотидов.
Рис. 4.
Анализ расщепления плазмидной ДНК-мишени комплексами crРНК/tracrРНК/Cas9 и sgРНК/Cas9 в присутствии 10-кратного (1 : 10) и 100‑кратного (1 : 100) избытка 30-звенного блокирующего немодифицированного олигодезоксирибонуклеотида B30. Условия см. в “Эксперим. части”.
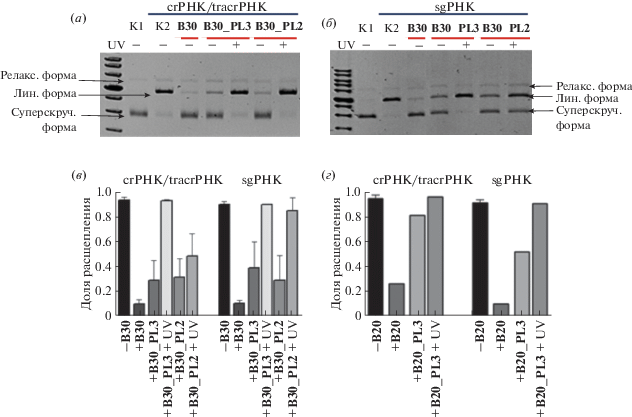
При расщеплении суперскрученной плазмидной ДНК белком Cas9 в присутствии направляющих РНК регистрировали образование в основном линейной формы плазмиды и незначительное количество релаксированной формы плазмиды, содержащей один ник (разрыв в одной из цепей) (рис. 5а, 5б). Результаты сравнительного анализа расщепления ДНК-мишени показали, что в большей степени снижается активность CRISPR-системы при использовании 30-звенных блокирующих олигонуклеотидов по сравнению с 20-звенными олигонуклеотидами, что согласуется со стабильностью их гетеродуплексов с crРНК. Введение фотолабильных модификаций в структуру блокирующих олигонуклеотидов снижает уровень ингибирования действия CRISPR-системы. При этом наблюдали более высокий уровень ингибирования для комплексов Cas9 с sgРНК. Во всех случаях облучение светом длиной волны 365 нм в течение 30 мин позволяло восстановить активность CRISPR-системы (рис. 5в, 5г).
Рис. 5.
Электрофореграммы продуктов расщепления ДНК-мишени (релаксированной формы плазмиды, содержащей один ник (одноцепочечный разрыв), и линейной формы плазмиды, содержащей двуцепочечный разрыв) комплексами crРНК/tracrРНК (а) и sgРНК (б) с Cas9 в присутствии 30-звенных блокирующих олигодезоксирибонуклеотидов. K1 – исходная ДНК-мишень, K2 – продукты расщепления ДНК-мишени белком Cas9 в присутствии соответствующих направляющих РНК. Анализ расщепления плазмидной суперскрученной ДНК-мишени белком Cas9 с парой направляющих crРНК/tracrРНК или с направляющей sgРНК в присутствии 100-кратного избытка фоточувствительных блокирующих 30-звенных (в) и 20-звенных (г) олигодезоксирибонуклеотидов до и после облучения светом с длиной волны 365 нм.
Кинетический анализ расщепления плазмиды также проводили, используя 100-кратный избыток блокирующих олигодезоксирибонуклеотидов по отношению к направляющим РНК (рис. 6).
Рис. 6.
Кинетические кривые расщепления плазмидной ДНК-мишени комплексом crРНК/tracrРНК/Cas9 в присутствии блокирующих олигодезоксирибонуклеотидов B30_PL3 (а) и B30_PL2 (б) и комплексом sgРНК/Cas9 в присутствии блокирующих олигодезоксирибонуклеотидов B30_PL3 (в) и B30_PL2 (г).

При использовании блокирующих фоторасщепляемых олигодезоксирибонуклеотидов B30_PL3 и B30_PL2 наблюдали значительное снижение уровня расщепления мишени. Облучение олигонуклеотида с тремя фотолинкерами в течение 30 мин приводило к практически полному восстановлению активности CRISPR-системы, в то время как облучение олигонуклеотидов с двумя линкерами восстанавливало уровень расщепления лишь частично.
Полученные результаты позволяют сделать заключение, что предложенная система CRISPR/ Cas9 может быть использована для фотоконтролируемого редактирования генов in vitro. Для использования этой системы in vivo необходимы дополнительные исследования с учетом особенностей проведения геномного редактирования в живых клетках.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалы. В работе были использованы следующие реактивы: перхлорат натрия, N-метилимидазол (Acros Organics, США); 4,4'-диметокситритилхлорид (Alfa Aesar, США), полимеры с присоединенным первым нуклеозидным звеном, 5'-O-(4,4'-диметокситритил), 2'-O-третбутилдиметилсилил-N-ацетилзащищенные рибонуклеозид-3'-фосфитамиды, 5'-O-(4,4'-диметокситритил), 5'-O-(4,4'-диметокситритил)-N-ацетилзащищенные дезоксирибонуклеозид-3'-фосфитамиды (ChemGenes, США); краситель “Stains-all”, персульфат аммония, дихлоруксусная кислота, акриламид, N,N '-метиленбисакриламид, 2,6-лутидин, бромистый этидий, Трис(гидроксиметил)аминометан (Fluka, Швейцария); фосфитамид флуоресцеина (GlenResearch, США); мочевина, 40%-ный водный раствор метиламина, молекулярные сита “Trap-PacTM Molecular Sieve Bag” 3 Å (Millipore, США); агароза (MP, США); ацетон, пиридин, тетрагидрофуран, формамид, пропионовый ангидрид (PanReac, Испания); ксиленцианол FF, бромфеноловый синий, N,N,N ',N '-этилендиаминтетрауксусная кислота (Serva, Германия); 1-(2-нитрофенил)-1,2-этандиол, триэтиламин, 5-этилтио-1H-тетразол, триэтиламинтригидрофторид, этокситриметилсилан, 2-цианоэтил-N,N,N ',N '-тетраизопропилфосфитамид (Sigma-Aldrich, США); хлористый метилен, бикарбонат натрия, йод кристаллический, гексан (Реахим, Россия); ацетонитрил (ХимМед, Россия), а также другие реактивы и растворители отечественного и зарубежного производства.
Рекомбинантная эндонуклеаза Cas9 и плазмида pBS2SKM Psp2 TTG на основе вектора pBluescript II SK(–) со вставкой протоспейсерной последовательности и PAM (5'-TGG-3') получены согласно стандартному протоколу [21].
Оборудование. Все водные растворы были приготовлены с использованием деионизованной воды, полученной с помощью прибора Millipore Simplicity System (Millipore, США). Концентрирование растворов олигонуклеотидов производили на вакуумном концентраторе Concentrator Plus (Eppendorf, США). Осажденные из растворов олигонуклеотиды центрифугировали на центрифугах MiniSpin Plus (Eppendorf, Германия). Перемешивание растворов осуществляли с помощью Thermomixer Comfort (Eppendorf, Германия). Оптическую плотность растворов олигонуклеотидов измеряли на спектрофотометре NanoDrop 1000 (Thermo Scientific, США). Тонкослойную хроматографию проводили на пластинках DC-Alufolien Kieselgel 60 F254 (Merck, Германия).
Синтез 2-О-4,4'-диметокситритил-1-(2-нитрофенил)-1,2-этандиола (I). Синтез проводили по аналогии с работами Ji et al. [4] и Tang et al. [8] с небольшими изменениями. К раствору 200 мг 1‑(2-нитрофенил)-1,2-этандиола (1.1 ммоль) в 1 мл абс. пиридина добавляли раствор 406.6 мг 4,4'-диметокситритилхлорида (1.2 ммоль) в 3 мл абс. пиридина и проводили реакцию в течение часа. За глубиной протекания реакции следили с помощью тонкослойной хроматографии (система А этилацетат : гексан, 2 : 8). После прохождения реакции добавляли 0.5 мл метанола для разложения избытка 4,4'-диметокситритилхлорида. Реакционную смесь после упаривания растворяли в хлористом метилене и проводили экстракцию насыщенным раствором NaHCO3. Объединенные органические слои сушили безводным Na2SO4 и упаривали досуха. Выделение продукта проводили методом колоночной хроматографии на силикагеле в градиенте концентрации этилацетата в гексане (0–35%). Rf 0.11. Выход реакции составил 85%.
1H-ЯМР (500 MГц, CDCl3) δ, м.д.: 7.85 (d, 1H), 7.82 (d, 1H), 7.62 (t, 1H), 7.42–7.35 (m, 3H), 7.30–7.16 (m, 11H), 6.78 (m, 4H), 5.50 (dd, 1H), 3.78 (s, 7H), 3.62 (dd, 1H), 3.16 (dd, 1H).
Синтез 1-(2-циано-N,N-диизопропилфосфит-амид)-2-О-4,4'-диметокситритил-1-(2-нитрофенил)-1,2-этандиола (II). Синтез проводили по аналогии с работой Ji et al. [4] с небольшими изменениями. К раствору 480 мг (1 ммоль) соединения (I) в 3 мл абс. ацетонитрила добавляли раствор, содержавший 195 мг 5-этилтиотетразола в 1.2 мл абсолютного ацетонитрила и 300 мкл N,N-диизопропилэтиламина, а затем 625 мкл (2 ммоль) 2-цианоэтил-N,N,N ',N '-тетраизопропилфосфитамида. За прохождением реакции следили с помощью ТСХ (система Б хлороформ : гексан : этилацетат, 4 : 4 : 2). После прохождения реакции к реакционной смеси добавляли 6 мл этилацетата, раствор над осадком отбирали, осадок промывали несколько раз этилацетатом. Реакционную смесь экстрагировали насыщенным водным раствором NaHCO3. Объединенные органические слои сушили безводным Na2SO4, фильтровали и упаривали досуха. Продукт выделяли из реакционной смеси методом колоночной хроматографии на силикагеле в градиенте концентрации этилацетата в гексане (0–30%). Rf 0.80. Выход реакции составил 61%.
1H-ЯМР (300 MГц, CDCl3) δ, м.д.: 7.89 (d, 1H), 7.80–7.76 (d, 1H), 7.59–7.51 (t, 2H), 7.41–7.33 (m, 3H), 7.29–7.10 (m, 13H), 6.80–6.72 (m, 5H), 5.81–5.72 (m, 1H), 3.90–3.80 (m, 10H), 3.56–3.38 (m, 3H), 3.27–3.20 (m, 1H), 2.63–2.38 (m, 2H), 2.04–1.02 (m, 1H), 1.28–1.11 (m, 12H), 0.94–0.80 (d, 7H). 31P-ЯМР (150 MГц, CDCl3) δ 150.1.
Фосфитамидный синтез олигонуклеотидов. Олигонуклеотиды были получены на автоматическом ДНК/РНК-синтезаторе ASM-800 (Биоссет, Россия) твердофазным фосфитамидным методом в реакторах объемом 50 мкл согласно оптимизированному для данного прибора синтетическому протоколу. Введение фосфитамида (II) в олигодезоксирибонуклеотиды проводили с использованием его 0.1 М раствора в абс. ацетонитриле в течение 30 мин. Флуоресцеин вводили на 5'-конец олигонуклеотидов в ходе фосфитамидного синтеза по протоколу, рекомендованному производителем. Олигонуклеотиды деблокировали и отделяли от полимерного носителя 40%-ным раствором метиламина в течение 15 мин (20 мин для 102-звенного олигорибонуклеотида) при 65°C и постоянном перемешивании или, в случае 5'-флуоресцеин-содержащих олигонуклеотидов, 30%-ным водным раствором аммиака при комнатной температуре в течение 16 ч. В случае олигорибонуклеотидов удаление 2'-O-TBDMSi-защитных групп с олигорибонуклеотидов проводили с использованием свежеприготовленного раствора N-метилпирролидинон : Et3N : Et3N ⋅ 3HF (1.5 : 0.75 : 1, v/v/v) при 65°C при перемешивании в течение 1.5 ч, добавляли этокситриметилсилан, через 10 мин проводили осаждение олигорибонуклеотидов диэтиловым эфиром.
Анализ гомогенности олигонуклеотидов. Гомогенность полученных олигонуклеотидов подтверждали методом гель-электрофореза в 12%-ном (акриламид : N,N '-метиленбисакриламид 30 : 0.5) или 15%-ном (акриламид : N,N '-метиленбисакрил-амид 29 : 1) ПААГ в денатурирующих условиях (8 М мочевина, 50 мМ Трис-H3BO3, pH 8.3, 0.1 M Na2ЭДТА). Для нанесения на гель использовали 5 мкл раствора 8 М мочевины с содержанием 0.025% ксиленцианолового FF. Для визуализации олигонуклеотидов использовали раствор красителя “Stains-all”, приготовленный из 50 мг красителя “Stains-all” и 100 мл смеси вода–формамид (1 : 1). После окрашивания гели сушили на приборе GelDryer model 583 (Bio-Rad, США).
Выделение олигонуклеотидов и их конъюгатов методом препаративного гель-электрофореза. Деблокированные олигонуклеотиды и конъюгаты выделяли с помощью препаративного гель-электрофореза в денатурирующем 12%-ном или 15%-ном ПААГ в вышеуказанных условиях. Визуализировали олигонуклеотиды в геле при наложении геля на пластину DC-Alufolien Kieselgel 60 F254 (Merck, Германия) в свете УФ-лампы (λ = 254 нм). В случае фоточувствительных олигодезоксирибонуклеотидов во избежание активации реакции расщепления под воздействием УФ-излучения закрывали стеклом основную часть геля, содержавшую продукт, оставляя только небольшую его часть, достаточную для визуализации. Олигонуклеотиды элюировали из геля 0.3 М раствором перхлората натрия или, в случае протяженных РНК, 0.3 М раствором ацетата натрия (pH 5.2), после чего проводили обессоливание с использованием Tet-Pak C18 Cartrige (Millipore, США) или осаждение спиртом соответственно.
Осаждали олигонуклеотиды из водных растворов в виде натриевых солей десятикратным объемом 2%-ного раствора перхлората натрия в ацетоне. Протяженные олигорибонуклеотиды осаждали из 0.3 M раствора ацетата натрия (рН 5.2) четырехкратным объемом этилового спирта при –20°C как минимум в течение 2 ч. Супернатант после центрифугирования отбирали, осадок промывали охлажденным 80%-ным этиловым спиртом и сушили досуха на воздухе.
Исследование кинетики расщепления фотомодифицированных олигодезоксирибонуклеотидов при облучении. Олигодезоксирибонуклеотиды, содержащие фоторасщепляемые линкеры и остаток флуоресцеина на 5'-конце (B30_PL2_F и B30_PL3_F), в виде растворов с концентрацией 10 мкМ в воде или в буфере (20 мМ HEPES, pH 7.5, 100 мМ KCl, 1 мМ дитиотреитол, 0.5 мМ Na2ЭДТА, 2 мМ MgCl2, 25% глицерина) облучали светом с длиной волны 365 нм в течение 1, 2, 5, 10, 15 и 30 мин. Продукты расщепления анализировали в денатурирующем 15%-ном ПААГ с последующей визуализацией УФ-облучением на длине волны 312 нм с использованием прибора для гель-документации Quantum Vilber Lourmat (Vilber Lourmat, Франция). Для получения количественных характеристик изображения переводили в цифровую форму в программном пакете Quantity Оne (Bio-Rad, США). Долю расщепления модифицированного олигодезоксирибонуклеотида рассчитывали в программном продукте Microsoft Excel. Параметры рассчитывали в программном пакете GraphPad Prism 5.0.4.533 (GraphPad, США) с использованием уравнения:
где fa – доля продукта реакции, Pst – доля продукта при переходе реакции в стационарную фазу (предельная степень расщепления), k1 – константа реакции псевдопервого порядка, t – время реакции.Термическая денатурация дуплексов направляющих РНК с блокирующими олигодезоксирибонуклеотидами. Дифференциальные кривые термической денатурации дуплексов были получены на установке с терморегулируемой оптической кюветой на базе спектрофотометра Cary 300 BioMelt (Varian Inc., Австралия). Концентрации олигонуклеотидных компонентов 1 мкМ в буфере 0.1 М NaCl, 10 мМ какодилат натрия (рН 7.4), 1 мМ Na2ЭДTA. Изменение температуры проводили со скоростью 0.5°С/мин. Детекцию поглощения осуществляли на двух длинах волн (260 и 270 нм). Обработку кривых термической денатурации и ренатурации проводили с использованием программного обеспечения Microsoft Office 2010 (Microsoft Corporation, США). Температуры плавления были определены как максимумы первых производных кривых плавления по температуре.
Расщепление плазмидной ДНК белком Cas9 в присутствии направляющих РНК. Реакцию проводили в 10 мкл раствора, содержавшего 20 мМ HEPES (pH 7.5), 100 мМ KCl, 1 мМ дитиотреитол, 0.5 мМ Na2ЭДТА, 2 мМ MgCl2, 25% глицерина. Контрольный раствор содержал все компоненты, кроме направляющей РНК и белка Cas9. Первоначально проводили сборку эффекторного комплекса. Для этого в буфер добавляли 1.35 мкл (1 мкМ, 1.35 пмоль) направляющей РНК, а в случае пары направляющих crРНК/tracrРНК – по 1.35 мкл (1 мкМ, 1.35 пмоль) каждой РНК и 0.602 мкл раствора белка Cas9 (2.24 мкМ, 1.35 пмоль) в 50-кратном избытке по отношению к плазмиде pBS2SKM. Растворы перемешивали и инкубировали 15 мин при 37°C. Пары crРНК/tracrРНК до добавления Cas9 предварительно инкубировали 5 мин при 90°C и охлаждали до 25°C. Затем в каждую пробирку добавляли 1 мкл раствора (50 нг/мкл, 27 фмоль) плазмиды pBS2SKM, содержащей протоспейсер и последовательность PAM (5'-TGG-3'). Перемешивали реакционные смеси и инкубировали в течение часа при 37°C в условиях отсутствия прямого солнечного света. Для остановки реакции к реакционной смеси добавляли 2.5 мкл раствора, содержавшего 250 мМ Na2ЭДТА, 1.2% SDS, 0.01% бромфенолового синего и 30% глицерина.
При анализе активности CRISPR-систем в присутствии блокирующих олигодезоксирибонуклеотидов сначала инкубировали 5 мин при 90°C гетеродуплекс 1.35 мкл направляющей crРНК или sgРНК (1 мкМ, 1.35 пмоль) с эквимолярным количеством блокирующего олигонуклеотида или с 10- или 100-кратным его избытком, добавляя 1.35 мкл 1, 10 или 100 мкМ раствора блокирующего олигодезоксирибонуклеотида соответственно, и остужали до 25°C. Затем добавляли Cas9 и инкубировали эффекторный рибонуклеопротеиновый комплекс 15 мин при 37°C.
Дезактивацию блокирующих фоторасщепляемых олигодезоксирибонуклеотидов проводили путем облучения их растворов в течение 30 мин УФ-светом (λ = 365 нм).
Исследование кинетики расщепления плазмидной ДНК белком Cas9 в присутствии направляющих РНК. Реакцию проводили в 85 мкл раствора, содержавшего 20 мМ HEPES (pH 7.5), 100 мМ KCl, 1 мМ дитиотреитол, 0.5 мМ Na2ЭДТА, 2 мМ MgCl2, 25% глицерина. Для сборки эффекторного комплекса в буфер добавляли 3.5 мкл (1 мкМ, 3.5 пмоль) направляющей sgРНК, а в случае пары направляющих crРНК/tracrРНК – по 3.5 мкл (1 мкМ, 3.5 пмоль) каждой РНК, и 1.56 мкл раствора белка Cas9 (5.8 мкМ, 3.5 пмоль) в 15-кратном избытке по отношению к плазмиде pBS2SKM. Растворы перемешивали и инкубировали 15 мин при 37°C. Пары crРНК/tracrРНК до добавления Cas9 предварительно инкубировали 5 мин при 90°C и остужали до 25°C. При анализе активности CRISPR-систем в присутствии блокирующих олигодезоксирибонуклеотидов сначала инкубировали 5 мин при 90°C гетеродуплекс 3.5 мкл направляющей crРНК или sgРНК (1 мкМ, 3.5 пмоль) с 3.5 мкл (100 мкМ, 0.35 нмоль, 100-кратный избыток) раствора блокирующего олигодезоксирибонуклеотида и остужали до 25°C. После этого добавляли 1.56 мкл раствора Cas9 (5.8 мкМ, 3.50 пмоль) и инкубировали комплекс 15 мин при 37°C. Аналогичным образом готовили растворы, содержащие блокирующие фоторасщепляемые олигодезоксирибонуклеотиды, дезактивированные УФ-облучением в течение 30 мин (λ = 365 нм).
После сборки эффекторного комплекса в каждую пробирку добавляли 8.5 мкл раствора (50 нг/мкл, 0.233 пмоль) плазмиды pBS2SKM. Реакционные смеси перемешивали и инкубировали в течение 1.5 ч при 37°C в условиях отсутствия прямого солнечного света. Для остановки реакции отбирали аликвоты 10 мкл через 2, 5, 10, 20, 30, 45, 60 и 90 мин и добавляли их в пробирки с 2.5 мкл раствора, содержавшего 250 мМ Na2ЭДТА, 1.2% SDS, 0.01% бромфенолового синего и 30% глицерина.
Анализ эффективности расщепления плазмиды. Эффективность реакции расщепления суперскрученной формы плазмиды до релаксированнной и линейной форм плазмиды белком Cas9 в присутствии направляющих РНК анализировали методом гель-электрофореза в 1%-ном агарозном геле в TAE-буфере (4 мМ Трис, 3 мМ CH3COOH, 0.07 мМ Na2ЭДТА) с добавлением 2.5 мкл этидий бромида (концентрация – 10 мг/мл). На гель наносили 10 мкл реакционной смеси в Quenching Buffer (250 мМ Na2ЭДТА, 1.2% SDS, 0.01% бромфенолового синего и 30% глицерина). Для контроля подвижности в геле продуктов расщепления использовали ДНК-маркер 1 kb, длина фрагментов от 250 до 10 000 п.н. (СибЭнзим, Россия). Проводили визуализацию с использованием системы гель-документации Quantum (Vilber Lourmat, Франция). Для получения количественных характеристик изображения переводили в цифровую форму в программном пакете Quantity Оne (Bio-Rad, США).
Долю расщепления плазмиды рассчитывали по следующим формулам:
Список литературы
Hartmann D., Smith J.M., Mazzotti G., Chowdhry R., Booth M.J. // Biochem. Soc. Trans. 2020. V. 48. P. 1645–1659. https://doi.org/10.1042/BST20200014
Wu Y., Yang Z., Lu Y. // Curr. Opin. Chem. Biol. 2020. V. 57. P. 95–104. https://doi.org/10.1016/j.cbpa.2020.05.003
Ruble B.K., Yeldell S.B., Dmochowski I.J. // J. Inorg. Biochem. 2015. V. 150. P. 182–188. https://doi.org/10.1016/j.jinorgbio.2015.03.010
Ji Y., Yang J., Wu L., Yu L., Tang X. // Angew. Chem. Int. Ed. 2016. V. 55. P. 2152–2156. https://doi.org/10.1002/anie.201510921
Shah S., Jain P.K., Kala A., Karunakaran D., Friedman S.H. // Nucleic Acids Res. 2009. V. 37. P. 4508–4517. https://doi.org/10.1093/nar/gkp415
Griepenburg J.C., Ruble B.K., Dmochowski I.J. // Bioorg. Med. Chem. 2013. V. 21. P. 6198–6204. https://doi.org/10.1016/j.bmc.2013.04.082
Connelly C.M., Uprety R., Hemphill J., Deiters A. // Mol. Biosyst. 2012. V. 8. P. 2987–2993. https://doi.org/10.1039/c2mb25175b
Tang X., Su M., Yu L., Lv C., Wang J., Li Z. // Nucleic Acids Res. 2010. V. 38. P. 3848–3855. https://doi.org/10.1093/nar/gkq079
Wu L. Wang Y., Wu J., Lv C., Wang J., Tang X. // Nucleic Acids Res. 2013. V. 41. P. 677–686. https://doi.org/10.1093/nar/gks996
Korman A., Sun H., Hua B., Yang H., Capilato J.N., Paul R., Panja S., Ha T., Greenberg M.M., Woodson S.A. // Proc. Natl. Acad. Sci. USA. 2020. V. 117. P. 12 080–12 086. https://doi.org/10.1073/pnas.2003425117
Young D.D., Lively M.O., Deiters A. // J. Am. Chem. Soc. 2010. V. 132. P. 6183–6193. https://doi.org/10.1021/ja100710j
Feng M. Ruan Z., Shang J., Xiao L., Tong A., Xiang Y. // Bioconjug. Chem. 2017. V. 28. P. 549–555. https://doi.org/10.1021/acs.bioconjchem.6b00646
Zhou W., Brown W., Bardhan A., Delaney M., Ilk A.S., Rauen R.R., Kahn S.I., Tsang M., Deiters A. // Angew. Chem. Int. Ed. 2020. V. 59. P. 8998–9003. https://doi.org/10.1002/anie.201914575
Taemaitree L., Brown T. // ACS Cent. Sci. 2020. V. 6. P. 616–618. https://doi.org/10.1021/acscentsci.0c00350
Moroz-Omori E.V., Satyapertiwi D., Ramel M.-C., Høgset H., Sunyovszki I.K., Liu Z., Wojciechowski J.P., Zhang Y., Grigsby C.L., Brito L., Bugeon L., Dallman M.J., Stevens M.M. // ACS Cent. Sci. 2020. V. 6. P. 695–703. https://doi.org/10.1021/acscentsci.9b01093
Liu Y., Zou R.S., He S., Nihongaki Y., Li X., Razavi S., Wu B., Ha T. // Science. 2020. V. 368. P. 1265–1269. https://doi.org/10.1126/science.aay8204
Zhang Y., Ling X., Su X., Zhang S., Wang J., Zhang P., Feng W., Zhu Y.Y., Liu T., Tang X. // Angew. Chem. Int. Ed. 2020. V. 59. https://doi.org/10.1002/anie.202009890
Jain P.K., Ramanan V., Schepers A.G., Dalvie N.S., Panda A., Fleming H.E., Bhatia S.N. // Angew. Chem. Int. Ed. 2016. V. 55. P. 12 440–12 444. https://doi.org/10.1002/anie.201606123
Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J.A., Charpentier E. // Science. 2012. V. 337. P. 816–821. https://doi.org/10.1126/science.1225829.2012
Khadempar S., Familghadakchi S., Motlagh R.A., Farahani N., Dashtiahangar M., Rezaei H., Gheibi Hayat S.M. // J. Cell Physiol. 2019. V. 234. P. 5751–5761. https://doi.org/10.1002/jcp.27476
Anders C., Jinek M. // Methods Enzymol. 2014. V. 546. P.1–20. https://doi.org/10.1016/B978-0-12-801185-0.00001-5
Shubsda M.F., Goodisman J., Dabrowiak J.C. // J. Biochem. Biophys. Methods. 1997. V. 34. P. 73–79. https://doi.org/10.1016/S0165-022X(96)01204-3
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


