Биоорганическая химия, 2021, T. 47, № 2, стр. 259-269
Направляющие РНКазу Р конъюгаты олигонуклеотидов с олиго(N-метилпирролом) как перспективные антибактериальные препараты
Н. А. Данилин 1, 2, А. Л. Матвеев 2, Н. В. Тикунова 2, А. Г. Веньяминова 2, Д. С. Новопашина 1, 2, *
1 Новосибирский государственный университет
630090 Новосибирск, ул. Пирогова, 1, Россия
2 ФГБУН “Институт химической биологии и фундаментальной медицины” СО РАН
630090 Новосибирск, просп. акад. Лаврентьева, 8, Россия
* E-mail: danov@niboch.nsc.ru
Поступила в редакцию 27.08.2020
После доработки 01.09.2020
Принята к публикации 06.09.2020
Аннотация
Впервые получены конъюгаты направляющих РНКазу Р олиго(2'-О-метилрибо)- и олигодезоксирибонуклеотидов (EGS-олигонуклеотидов) с олиго(N-метилпирролом). В модельной системе с использованием флуоресцентно-меченых химически синтезированных РНК-мишеней, соответствующих фрагментам мРНК генов ftsZ и gyrA Acinetobacter baumannii, продемонстрирована способность РНКазы Р расщеплять РНК в присутствии направляющих олигонуклеотидов и их конъюгатов с олиго(N-метилпирролом). Введение одного остатка олиго(N-метилпиррола) на 5'-конец направляющего EGS-олигодезоксирибонуклеотида значительно повышает эффективность гидролиза РНК-мишени РНКазой Р, а его введение на 5'-конец направляющего EGS-олиго(2'-О-метилрибонуклеотида) незначительно уменьшает эффективность гидролиза РНК-мишени РНКазой Р. Показано, что присоединение олиго(N-метилпиррола) на 5'-конец олигонуклеотидов увеличивает эффективность их проникновения в клетки бактерий.
ВВЕДЕНИЕ
Создание принципиально новых антибактериальных препаратов является актуальной задачей современной биоорганической химии, молекулярной биологии и фундаментальной медицины. В настоящее время до конца не решенными остаются проблемы возникновения лекарственной устойчивости бактерий к антимикробным препаратам и побочных эффектов, возникающих при применении антибиотиков [1, 2]. Перспективным подходом к разработке антибактериальных препаратов является создание антисмысловых олигонуклеотидов, мишенью которых являются жизненно важные бактериальные РНК (см., например, [3]). Использование таких олигонуклеотидов позволяет блокировать экспрессию соответствующих бактериальных генов и приводить к подавлению роста бактерий.
Ранее было показано, что ключевой фермент созревания тРНК – РНКаза Р, природным субстратом которой является пре-тРНК, – способен гидролизовать РНК-мишень в составе комплекса с комплементарным ей антисмысловым олигонуклеотидом, содержащим ССА-последовательность на 3'-конце [4]. Эта способность РНКазы Р легла в основу EGS-технологии (external guide sequences technology), а соответствующие антисмысловые олигонуклеотиды, направляющие действие РНКазы Р, получили название EGS-олигонуклеотиды [5–7]. Было продемонстрировано, что использование EGS-олигонуклеотидов, направляющих бактериальную РНКазу Р на расщепление мРНК определенных бактериальных генов, позволяет подавить рост бактерий (см., например, [8–10]). В качестве основы для создания EGS-олигонуклеотидов часто используют замкнутые нуклеиновые кислоты (LNA – locked nucleic acid, BNA – bridged nucleic acid) [11, 12], морфолиновые олигонуклеотиды [9, 10, 13–16], олиго(2'-О-метилрибонуклеотиды) и фосфорилгуанидиновые олигонуклеотиды [17]. Одним из подходов к улучшению проникновения олигонуклеотидов в бактерии является их конъюгирование с молекулами-доставщиками (см., например, [18]). В качестве доставляющей группировки мы предлагаем использовать полиамид олиго(N-метилпиррол). Олиго(N-метилпирролы) являются аналогами пептидных антибиотиков нетропсина и дистамицина. Они положительно заряжены, сиквенс-специфически связываются с малой бороздкой ДНК и способны в отсутствие трансфектанта проникать в эукариотические клетки [19–22].
В данной работе предложено использование направляющих РНКазу Р конъюгатов олиго(2'-О-метилрибонуклеотидов) и олигодезоксирибонуклеотидов с олиго(N-метилпирролом) в качестве потенциальных антибактериальных препаратов.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В качестве бактериальной мишени нами выбрана грамотрицательная бактерия Acinetobacter baumannii, вызывающая тяжелые внутрибольничные инфекции [23, 24], отличающаяся высокой устойчивостью к антибиотикам и включенная Всемирной организацией здравоохранения (ВОЗ) в список наиболее опасных бактерий [25–27]. Мы предлагаем в качестве альтернативы классическим антибиотикам использовать EGS-олигонуклеотиды, способные ингибировать экспрессию жизненно важных генов этой бактерии. В качестве мишени для EGS-олигонуклеотидов были выбраны фрагменты мРНК генов домашнего хозяйства ftsZ и gyrA A. baumannii в области сайта инициации трансляции. Ген ftsZ кодирует нуклеотидсвязывающий белок ftsZ, играющий ключевую роль при делении прокариот [28]. ДНК-гираза, одна из субъединиц которой кодируется геном gyrА, является необходимым элементом процесса репликации бактерий [29]. Ингибирование экспрессии этих важных для жизнедеятельности бактерии генов должно вызывать нарушение жизненного цикла и подавление роста бактерий.
В качестве основы для EGS-олигонуклеотидов нами были выбраны олигодезоксирибонуклеотиды и аналоги олигорибонуклеотидов, содержащих в 2'-положении рибозы 2'-О-метильную группу. Олиго(2'-О-метилрибонуклеотиды) полностью устойчивы к действию ДНК- и РНК-специфических нуклеаз, обладают повышенной устойчивостью к действию нуклеаз двойного спектра и способны образовывать стабильные комплексы с РНК-мишенью [30, 31]. Ранее было показано, что для эффективного взаимодействия РНКазы Р с комплексом РНК–EGS-олигонуклеотид необходимо наличие четырех немодифицированных рибонуклеотидов с 3'-конца EGS-олигонуклеотида [32–34].
Синтез конъюгатов олигонуклеотидов с олиго(N-метилпирролом) проводили, используя в качестве исходных 5'-фосфаты олиго(2'-О-метилрибонуклеотидов), содержащих четыре немодифицированных рибонуклеотида с 3'-конца, и 5'-фосфатов олигодезоксирибонуклеотидов. Присоединение лиганда проводили путем активации 5'-фосфата олигонуклеотида окислительно-восстановительной парой трифенилфосфин/дипиридилдисульфид (PPh3/(PyS)2) в присутствии диметиламинопиридина (DMAP) с последующим взаимодействием с аминопроизводным олиго(N-метилпиррола) по аналогии с работой Новопашиной с соавт. [35] (схема 1).
Активацию 5'-фосфата проводили в диметилсульфоксиде, для чего олигонуклеотид предварительно переводили в цетавлоновую соль. Затем к активированому фосфату олигонуклеотида добавляли олиго(N-метилпиррол) и инкубировали при перемешивании при температуре 37°C (схема 1). Через 16 ч реакционную смесь, содержащую конъюгаты с одним и двумя остатками олиго(N-метилпиррола), осаждали. Конъюгаты выделяли путем электрофореза в денатурирующем препаративном ПААГ с последующей элюцией и обессоливанием и подтверждали строение методом MALDI-TOF-масс-спектрометрии (табл. 1).
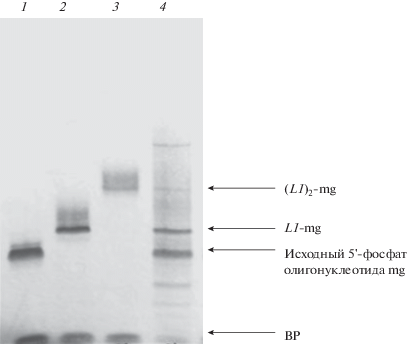
Аналогичным образом, используя в качестве исходных 5'-фосфаты 3'-флуоресцеинсодержащих олигонуклеотидов, получали конъюгаты 3'-флуоресцеин-меченых EGS-олигонуклеотидов с одним остатком олиго(N-метилпиррола) (табл. 1). Строение конъюгатов подтверждали методом MALDI-TOF-масс-спектрометрии. В результате реакции получали смесь моно- и бис-конъюгатов, с последующим выделением их в индивидуальном виде, выходы конъюгатов после выделения в расчете на первое нуклеотидное звено составили 1.1–7.1%. Подвижность конъюгатов при гель-электрофорезе была значительно ниже подвижности исходных олигонуклеотидов и зависела от количества присоединенных лигандов (рис. 1).
Таблица 1.
Синтезированные конъюгаты EGS-олигонуклеотидов с олиго(N-метилпирролом)
| Шифр | Последовательность (5'-3') | Выход*, % | Молекулярная масса | |
|---|---|---|---|---|
| рассчитано | получено** | |||
| (L1)2-mf | 5'-(L1)2-pAmUmUmCmAmAmAmUmGmAmGmGmCmCmACCA | 1.1 | 7973.09 | 7974.05 |
| L1-mf | 5'-L1-pAmUmUmCmAmAmAmUmGmAmGmGmCmCmACCA | 2.4 | 6985.97 | 6983.83 |
| L1-mf-Flu | 5'-L1-pAmUmUmCmAmAmAmUmGmAmGmGmCmCmACCA-Flu | 7.1 | 7475.44 | 7476.10 |
| (L1)2-mg | 5'-(L1)2-pUmUmUmCmCmGmAmUmAmCmGmCmUmCmACCA | 2.7 | 7862.96 | 7862.01 |
| L1-mg | 5'-L1-pUmUmUmCmCmGmAmUmAmCmGmCmUmCmACCA | 4.5 | 6875.85 | 6874.11 |
| L1-mg-Flu | 5'-L1-pUmUmUmCmCmGmAmUmAmCmGmCmUmCmACCA-Flu | 2.3 | 7033.16 | 7034.70 |
| (L1)2-df | 5'-(L1)2-pATTCAAATGAGGCCACCA | 6.8 | 7530.81 | 7526.00 |
| L1-df | 5'-L1-pATTCAAATGAGGCCACCA | 6.3 | 6543.69 | 6547.15 |
| L1-df-Flu | 5'-L1-pATTCAAATGAGGCCACCA-Flu | 1.5 | 7033.16 | 7034.70 |
| (L1)2-dg | 5'-(L1)2-pTTTCCGATACGCTCACCA | 5.3 | 7448.73 | 7452.32 |
| L1-dg | 5'-L1-pTTTCCGATACGCTCACCA | 2.1 | 6461.61 | 6461.99 |
| L1-dg-Flu | 5'-L1-pTTTCCGATACGCTCACCA-Flu | 2.7 | 6951.08 | 6952.12 |
* Выход в расчете на первое нуклеозидное звено. ** По результатам масс-спектрометрического анализа. Обозначения: L1 – олиго(N-метилпиррол); m – олиго(2'-О-метилрибонуклеотид); d – олигодезоксирибонуклеотид; f – EGS-олигонуклеотид, направляющий РНКазу Р на мРНК гена ftsZ; g ‒ EGS-олигонуклеотид, направляющий РНКазу Р на мРНК гена gyrA; Flu – остаток флуоресцеина; p – фосфат; Nm – 2'-О-метил-рибонуклеотид. Структуру конъюгатов см. на схеме 1.
Рис. 1.
Электрофореграмма, полученная при анализе исходного олигонуклеотида 5'-р-UmUmUmCmCmGmAmUmAmCm- GmCmUmCmACCA (1), выделенных конъюгатов L1-mg, содержащего один остаток олиго(N-метилпиррола) (2), и (L1)2-mg, содержащего два остатка олиго(N-метилпиррола) (3), и реакционной смеси при их получении (4). Структуру конъюгатов см. в табл. 1 и на схеме 1. Условия: денатурирующий 15%-ный ПААГ, окрашивание “Stains-all”. BP – бромфеноловый синий.
При исследовании способности РНКазы Р гидролизовать РНК-мишени в присутствии EGS-олигонуклеотидов и их конъюгатов с олиго(N-метилпирролом) в качестве РНК-мишеней нами были использованы два модельных 5'-флуоресцеин-меченых олигорибонуклеотида, представляющие собой фрагменты мРНК генов ftsZ и gyrA A. baumannii (табл. 2) в районе сайта инициации трансляции.
Схема 1. Схема синтеза конъюгатов олигонуклеотидов с олиго(N-метилпирролом). PPh3 – трифенилфосфин, (PyS)2 – дипиридилдисульфид, DMAP – диметиламинопиридин.
Таблица 2.
Последовательности 5'-флуоресцентно-меченых РНК-мишеней
| Шифр | Последовательность (5'-3') | Выход*, % |
|---|---|---|
| r24-ftsZ | 5'-Flu-AGGUCAUGGCCUCAUUUGAAUUUA | 45.4 |
| r24-gyrA | 5'-Flu-UAUGCAUGAGCGUAUCGGAAAUCC | 42.0 |
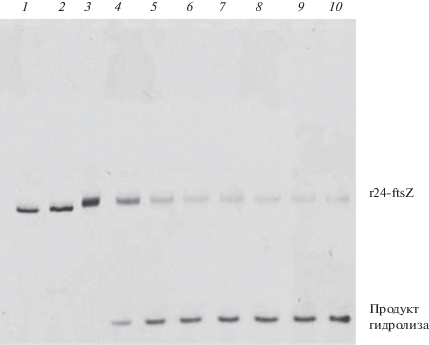
Расщепление РНК-мишеней проводили в присутствии 10-кратного избытка EGS-олигонуклеотидов или их конъюгатов при 37°С. На рис. 2 представлен пример электрофоретического разделения реакционных смесей, полученных в результате расщепления РНК-мишени r24-ftsZ РНКазой Р в присутствии конъюгата L1-mf.
Рис. 2.
Электрофореграмма, полученная при разделении продуктов гидролиза 5'-флуоресцеин-меченой РНК-мишени (r24-ftsZ) РНКазой Р в присутствии конъюгата EGS-олиго(2'-О-метилрибонуклеотида) L1-mf: 1 – контроль без EGS-олигонуклеотида через 0 мин, 2 – контроль без EGS-олигонуклеотида через 60 мин, 3 – контроль без РНКазы Р, 4–10 – реакционная смесь через 2, 5, 10, 15, 30, 60 и 120 мин после начала реакции с ферментом. Условия: 200 нМ РНК М1, 2 мкМ белок С5, 2 мкМ РНК-мишень, 20 мкМ EGS-олигонуклеотид, 10 мМ Трис-HCl, pH 7.5, 10 мМ MgCl2, 100 мМ NH4Cl, 37°C.
При проведении расщепления РНК-мишеней РНКазой Р в присутствии конъюгатов EGS-олигонуклеотидов, содержащих два остатка олиго(N-метилпиррола), продуктов гидролиза не регистрировали, на электрофорезе наблюдали комплексы, обладающие меньшей подвижностью в сравнении с исходными РНК-мишенями (данные не приведены).
В результате сравнительного исследования способности РНКазы Р расщеплять модельные РНК-мишени в составе их дуплексов с конъюгатами EGS-олиго(2'-О-метилрибонуклеотидов) и EGS-олигодезоксирибонуклеотидов c олиго(N-метилпирролом) было обнаружено, что введение одного лиганда на 5'-конец направляющего EGS-олигодезоксирибонуклеотида значительно повышает эффективность гидролиза РНК-мишени РНКазой Р, а введение одного лиганда на 5'-конец направляющего EGS-олиго(2'-О-метилрибонуклеотида) незначительно уменьшает эффективность гидролиза РНК-мишени РНКазой Р (рис. 3). Такая разница, вероятно, связана с различиями в структурах дуплексов РНК-мишеней с олигодезоксирибонуклеотидами и олиго(2'-О-метилрибонуклеотидами) и со способностью олиго(N-метилпиррола) взаимодействовать с малой бороздкой формирующейся двойной спирали. Для обоих типов EGS-олигонуклеотидов эффективность гидролиза РНК мишени r24-ftsZ была выше, чем эффективность гидролиза РНК-мишени r24-gyrA.
Рис. 3.
Кривые расщепления 5'-флуоресцеин-меченых РНК-мишеней r24-ftsZ (а) и r24-gyrA (б) РНКазой Р в присутствии направляющих олигодезоксирибонуклеотидов (df и dg) и (2'-О-метилрибонуклеотидов) (mf и mg), а также их 5'‑конъюгатов c олиго(N-метилпирролом) (L1-df, L1-dg, L1-mf и L1-mg). Условия: 200 нМ РНК М1, 2 мкМ белок С5, 2 мкМ флуоресцентно-меченая РНК-мишень, 20 мкМ EGS-олигонуклеотид, 10 мМ Трис-HCl, pH 7.5, 10 мМ MgCl2, 100 мМ NH4Cl, 37°C. Данные получены путем усреднения результатов как минимум трех экспериментов. За 100% принимали полное превращение РНК-мишени в продукты гидролиза. Стрелкой указан сайт расщепления РНК-мишени РНКазой Р.

Несколько неожиданным для нас оказался результат, полученный на РНК-мишени r24-gyrA. Расщепление этой мишени в присутствии конъюгата EGS-олигодезоксирибонуклеотида с олиго(N-метилпирролом) РНКазой Р происходило более эффективно, чем для аналогичного конъюгата EGS-олиго(2'-О-метилрибонуклеотида) (рис. 4). Возможно, данный эффект связан со значительной термической стабилизацией комплекса EGS-олигодезоксирибонуклеотид–РНК-мишень за счет дополнительного взаимодействия введенного остатка олиго(N-метилпиррола) с дуплексом.
Рис. 4.
Предельная степень гидролиза модельных РНК-мишеней r24-ftsZ и r24-gyrA в присутствии соответствующих EGS-олигодезоксирибонуклеотидов (df и dg), EGS-олиго(2'-О-метилрибонуклеотидов) (mf и mg), а также их 5'-конъюгатов c олиго(N-метилпирролом) (L1-df, L1-dg, L1-mf и L1-mg). Предельную степень гидролиза рассчитывали по уравнению (1) (см. “Эксперим. часть”), принимая за 100% полное превращение РНК-мишени в продукты гидролиза.

Полученные результаты демонстрируют возможность эффективного гидролиза РНК-мишеней РНКазой Р в присутствии EGS-олигонуклеотидов и их конъюгатов с олиго(N-метилпирролом) in vitro.
Для исследования проникновения конъюгатов EGS-олигонуклеотидов с олиго(N-метилпирролом) в бактерии была использована специально полученная серия их 3'-флуоресцеин-меченых аналогов (табл. 1) и контрольные 3'-флуоресцеин-меченые EGS-олиго(2'-О-метилрибонуклеотиды) (mf-Flu и mg-Flu) и EGS-олигодезоксирибонуклеотиды (df-Flu и dg-Flu). Сравнительное исследование проникновения конъюгатов EGS-олигонуклеотидов с олиго(N-метилпирролом) в клетки бактерий Escherichia coli и Acinetobacter baumannii методом проточной цитофлуорометрии показало, что введение олиго(N-метилпиррола) в большинстве случаев улучшает эффективность проникновения конъюгатов (рис. 5).
Рис. 5.
Эффективность проникновения (%) 5'-конъюгатов направляющих олигонуклеотидов с олиго(N-метилпирролом), содержащих остаток флуоресцеина на 3'-конце, в A. baumannii и E. coli по данным проточной цитофлуорометрии.

Полученные результаты подтверждают перспективность использования созданных в данной работе направляющих РНКазу Р конъюгатов EGS-олиго(2'-О-метилрибонуклеотидов) и EGS-олигодезоксирибонуклеотидов с олиго(N-метилпирролом) в качестве потенциальных антибактериальных препаратов.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
В работе были использованы следующие реактивы: перхлорат натрия, N-метилимидазол (Acros Organics, США); 5-этилтио-1H-тетразол (Biosset, Россия); CPG-полимеры с присоединенным первым нуклеозидным звеном, фосфитамиды N-ацетилзащищенных 5'-O-(4,4'-диметокситритил)-2'-O-метилрибонуклеотидов, N-ацетилзащищенных 5'-O-(4,4'-диметокситритил)-2'-O-третбутилдиметилсилилрибонуклеозидов, N-ацетилзащищенных 5'-O-(4,4'-диметокситритил)-2'-дезоксирибонуклеотидов (ChemGenes, США); фосфитамид 2-[2-(4,4'-диметокситритилокси)этилсульфонил]этанола, фосфитамид 1-диметокситритилокси-2-(N-тиомочевина-(ди-O-пивалоил-флуоресцеин)-4-аминобутил)-пропанола, модифицированный CPG-полимерный носитель с флуоресцеином (Glen Research, США); краситель “Stains-all”, персульфат аммония, дихлоруксусная кислота, 2,6-лутидин, трифенилфосфин, 4-(N,N-диметил)аминопиридин, 2,2'-дипиридилдисульфид, цетилтриметиламмоний бромид, N,N,N ',N '-тетраметилэтилендиамин (Fluka, Швейцария); мочевина, 40%-ный водный раствор метиламина (Merck, Германия); молекулярные сита Trap-PacTM Molecular Sieve Bag 3 Å (Millipore, США); пиридин, ацетон, тетрагидрофуран, ацетонитрил (Panreac, Испания); ксиленцианол FF, бромфеноловый синий (Serva, Германия); Na2EDTA (Amresco, США); акриламид, N,N '-метиленбисакриламид, пропионовый ангидрид (Acros Organics, Бельгия); трис(гидроксиметил)аминометан, триэтиламин, триэтиламинтригидрофторид, этокситриметилсилан, хлорид магния (Sigma-Aldrich, США); хлористый метилен, йод кристаллический (Криохром, Россия), а также другие реактивы и растворители отечественного и зарубежного производства.
Олиго(N-метилпиррол) был получен с.н.с. В.А. Рябининым и в.н.с. А.Н. Синяковым, лаборатория биоорганической химии ферментов Института химической биологии и фундаментальной медицины СО РАН [35].
Для ферментативных реакций использовали РНКазу Р E. coli (К.Ф. 3.1.26.5), компоненты которой получены в лаборатории биоорганической химии ферментов Института химической биологии и фундаментальной медицины СО РАН: каталитически активная РНК М1 синтезирована по методике, описанной в работе Guerrier-Takada et al. [36], и любезно предоставлена д.х.н. Н.А. Моор, а белковый кофактор С5 получен по протоколу, приведенному в работе Guerrier-Takada et al. [37], и любезно предоставлен д.б.н. С.Н. Ходыревой.
В работе использовали следующие приборы: спектрофотометр Nano Drop 1000 (Thermo Fisher Scientific, США), термостатируемый шейкер Thermomixer F1.5 (Eppendorf, Германия), центрифуги MiniSpin plus и Сentrifuge 5415R (Eppendorf, Германия), Speed-Vac Concentrator SVC-100H (Savant, США), термостатируемый шейкер (Bioer, США), камеру для вертикального электрофореза (Хеликон, Россия), прибор для сушки гелей Gel Dryer B35 (Bio-Rad, США).
Олигодезоксирибонуклеотиды, олигорибонуклеотиды и их модифицированные аналоги были синтезированы с использованием соответствующих мономерных синтонов и полимерных носителей твердофазным фосфитамидным методом на автоматическом ДНК/РНК-синтезаторе АСМ-800 (Биосет, Россия) по оптимизированным для данного прибора протоколам в лаборатории химии РНК Института химической биологии и фундаментальной медицины СО РАН. Деблокирование олигонуклеотидов проводили в стандартных условиях. Выделение олигорибонуклеотидов и их модифицированных аналогов проводили методом препаративного гель-электрофореза.
Все водные растворы были приготовлены с использованием деионизованной воды (с удельным сопротивлением 18 × 106 Oм/см), полученной с помощью прибора Millipore Simplicity System (Millipore, США).
Олигонуклеотиды, их конъюгаты и производные осаждали из водных растворов в виде натриевых солей десятикратным объемом 2%-ного раствора NaClO4 в ацетоне. Супернатант после центрифугирования отбирали, осадок промывали ацетоном и высушивали на воздухе.
Расчет коэффициентов молярного поглощения. Значения молярных коэффициентов поглощения для 2'-О-метилрибонуклеотидов при 260 нм принимали равными соответствующим молярным коэффициентам поглощения для рибонуклеотидов. Молярные коэффициенты поглощения флуоресцентно-меченых производных олигонуклеотидов считали равными сумме значений молярных коэффициентов поглощения олигонуклеотидов и молярного коэффициента поглощения флуоресцеина, присоединенного к олигомеру (20 900 M–1 cм–1 при 260 нм). Молярный коэффициент олиго(N-метилпиррола) – 36 610 M–1 cм–1 при 260 нм [35]. Молярные коэффициенты были рассчитаны с учетом гипохромного эффекта в программном пакете OligoAnalyzer 3.1.
Аналитический гель-электрофорез в денатурирующих условиях. Анализ гомогенности олигонуклеотидов, их производных и конъюгатов проводили методом гель-электрофореза в 15%-ном ПААГ в денатурирующих условиях (акриламид : : N,N '-метиленбисакриламид (30 : 0.5), 8 М мочевина, 50 мМ Трис-Н3ВО3, рН 8.3, 0.1 мМ Na2ЭДTA) при напряжении 50 В/см и толщине геля 0.4 мм. К олигонуклеотидным образцам (~0.05 о.е.) для нанесения на гель добавляли 4–5 мкл раствора 8 М мочевины, содержавшего 0.05% ксиленцианолового голубого и 0.05% бромфенолового синего. Для визуализации олигонуклеотидов и их производных использовали раствор, приготовленный из 50 мг красителя “Stains-all” и 100 мл смеси формамид : вода (1 : 1). После прокрашивания гели высушивали в течение 40–70 мин на приборе GelDryer 583 (Bio-Rad, США).
Выделение олигонуклеотидов и их конъюгатов препаративным гель-электрофорезом. Деблокированные олигонуклеотиды, их конъюгаты и производные выделяли с помощью препаративного электрофореза в 15%-ном ПААГ в указанных выше условиях, при толщине геля 0.4 мм. После электрофореза гель визуализировали в УФ-свете с использованием пластин DC-Alufolien Kieselgel 60 F254, а в случае флуоресцентно-меченых олигонуклеотидов ориентировались на соответствующую окрашенную полосу. Участки геля, содержащие продукт, вырезали и проводили элюцию нуклеотидного материала из ПААГ 0.3 М водным раствором NaClO4 в течение 16 ч при 25°С. Обессоливание олигонуклеотидов проводили на фазе C18 (Waters, США).
Строение полученных олигонуклеотидов подтверждали методом MALDI-TOF-масс-спектрометрии с использованием масс-спектрометра REFLEX III (Bruker Daltonics, Германия; Центр коллективного пользования Центра масс-спектрометрического анализа Института химической биологии и фундаментальной медицины СО РАН).
Синтез конъюгатов олигонуклеотидов, содержащих остатки олиго(N-метилпиррола) на 5'-конце. 5'-Фосфат олигонуклеотида в количестве 20 ОЕ260 осаждали в виде цетилтриметиламмониевой соли, добавляя 8%-ный водный раствор бромида цетилтриметиламмония порциями 10, 5, 3 и далее по 1 мкл до прекращения выделения осадка. Осадок олигонуклеотида сушили в течение часа в вакууме в эксикаторе с P2O5. К осадку добавляли 5 мг (41 мкмоль) DMAP в 50 мкл DMSOабс, 5.3 мг (PyS)2 (25 мкмоль) в 25 мкл DMSOабс и 6.8 мг Ph3P (25 мкмоль) в 25 мкл DMSOабс и перемешивали в течение 15 мин при температуре 37°С. Затем добавляли раствор 1 мг (1 мкмоль) олиго(N-метилпиррола) в 105 мкл смеси TEA : DMSOабс (1 : 20). Реакцию проводили при перемешивании при температуре 37°С в течение 16 ч. Реакционную смесь осаждали в виде натриевой соли.
Приготовление раствора холофермента РНКазы Р. Раствор РНК М1 инкубировали в течение 5 мин при 65°С, затем медленно охлаждали до комнатной температуры в течение 30 мин. После этого готовили свежий раствор белка С5 в буфере, содержавшем 10 мМ Трис-HCl (pH 7.5), 10 мМ MgCl2, 100 мМ NH4Cl, смешивали растворы РНК М1 и белка С5 и инкубировали их в течение 5 мин при 37°С. Использовали 10-кратный избыток белка С5 по отношению к РНК М1, концентрацию холофермента принимали равной концентрации РНК М1. Полученный раствор холофермента РНКазы Р сразу же использовали для проведения требуемых реакций.
Расщепление флуоресцентно-меченых модельных РНК-мишеней РНКазой Р в присутствии направляющих олигонуклеотидов. Реакцию проводили в 20 мкл раствора, содержавшего 10 мМ Трис-HCl (pH 7.5), 10 мМ MgCl2, 100 мМ NH4Cl, 0.2 мкМ РНК M1, 2 мкМ белок C5. Контрольный раствор содержал все компоненты, кроме РНК М1 и белка С5. Готовили 10 мкл раствора РНК-субстрата, содержавшего 20 мкМ флуоресцентно-меченую РНК-мишень и 200 мкМ направляющий олигонуклеотид. Предварительно для разрушения устойчивых вторичных структур, образовавшихся при хранении РНК, и образования комплекса РНК-мишени с направляющим олигонуклеотидом раствор РНК-субстрата инкубировали 2 мин при 90°С, затем охлаждали 5 мин при 25°С. Для начала реакции добавляли к приготовленной реакционной смеси холофермента РНКазы Р раствор субстрата. Через 0, 2, 5, 10, 15, 30, 60, 120 и 180 мин отбирали из реакционной смеси пробы объемом 2 мкл и смешивали их с 4 мкл денатурирующего буфера для нанесения (по 0.05% ксиленцианолового голубого и бромфенолового синего в 8 М мочевине). Продукты реакции анализировали электрофорезом в 15%-ном ПААГ в денатурирующих условиях. Гель сканировали с использованием системы визуализации гелей Quantum St5 (Vilber Lourmat, Франция) и полученное изображение обрабатывали в программном пакете Quantity One (Bio-Rad, США).
Расчет кинетических параметров расщепления РНК-субстрата РНКазой Р. Данные, полученные по результатам расщепления РНК-субстрата РНКазой Р, обрабатывали в программе Microsoft Excel. Долю продукта реакции рассчитывали как отношение интенсивности полосы, соответствующей продукту реакции, к суммарной интенсивности полос, соответствующих РНК-субстрату и продукту реакции. Расчет проводили с использованием программного обеспечения Graph Pad Prizm 7.00.159 по уравнению для реакции псевдопервого порядка:
где ${{f}_{a}}$ – доля продукта реакции, $Pl$ – доля продукта при переходе реакции в стационарную фазу (предельная степень расщепления), ${{k}_{{obs}}}$ – константа реакции псевдопервого порядка, t – время реакции.Исследование проникновения 5'-конъюгатов олигонуклеотидов с олиго(N-метилпирролом) в бактериальные клетки. Исследование проникновения 3'-флуоресцентно-меченых конъюгатов EGS-олигонуклеотидов с олиго(N-метилпирролом) в клетки проводили на культурах E. coli и A. baumannii. Ночную культуру бактериальных клеток разводили в 100 раз ростовой средой LB (лизогенный бульон, среда Лурия–Бертани) и инкубировали клетки (3–5 × 106 кл./мл) в течение 2 ч при 37°С до достижения OD600 0.35. Культуру клеток готовили в экспоненциальной фазе роста (5–6 × 106 кл./мл), клетки осаждали центрифугированием при 4000 g в течение 4 мин и ресуспендировали в среде LB, содержавшей конъюгат. Конечные концентрации конъюгатов в среде составляли 0.2 или 1 мкМ. Клетки инкубировали в течение 1 ч при 37°С при качании в темноте. Клетки осаждали центрифугированием при 4000 g в течение 4 мин и добавляли 100 мкл 0.9%-ного раствора NaCl. Клеточный осадок ресуспендировали в 100 мкл 4%-ного раствора формальдегида в фосфатном буфере и инкубировали в течение 30 мин при комнатной температуре при качании. Затем клетки трижды промывали стерильным фосфатным буфером и инкубировали с 4',6-диамидино-2-фенилиндолом (DAPI) в течение дополнительных 15 мин. Проточную цитометрию проводили с использованием прибора NovoCyte (ACEA Biosciences, США).
Список литературы
Llor C., Bjerrum L. // Ther. Adv. Drug Saf. 2014. V. 5. P. 229–241. https://doi.org/10.1177/2042098614554919
Guidry C.A., Mansfield S.A., Cook C.H., Sawyer R.G. // Surg. Clin. North Am. 2014. V. 94. P. 1195–1218. https://doi.org/10.1016/j.suc.2014.08.010
Bai H., Xue X., Hou Z., Zhou Y., Meng J., Luo X. // Curr. Drug Discov. Technol. 2010. V. 7. P. 76–85. https://doi.org/10.2174/157016310793180594
Walker S.C., Engelke D.R. // Crit. Rev. Biochem. Mol. Biol. 2006. V. 41. P. 77–102. https://doi.org/10.1080/10409230600602634
Förster A.C., Altman S. // Science. 1990. V. 249. P. 783–786. https://doi.org/10.1126/science.1697102
Davies-Sala C., Soler-Bistué A., Bonomo R.A., Zorreguieta A., Tolmasky M.E. // Ann. N.Y. Acad. Sci. 2015. V. 1354. P. 98–110. https://doi.org/10.1111/nyas.12755
Derksen M., Mertens V., Pruijn G.J. // Biomolecules. 2015. V. 5. P. 3029–3050. https://doi.org/10.3390/biom5043029
Lundblad E.W., Altman S. // Nat. Biotechnol. 2010. V. 27. P. 212–221. https://doi.org/10.1016/j.nbt.2010.03.003
Wesolowski D., Tae H.S., Gandotra N., Llopis P., Shen N., Altman S. // Proc. Natl. Acad. Sci. USA. 2011. V. 108. P. 16 582‒16 587. https://doi.org/10.1073/pnas.1112561108
Wesolowski D., Alonso D., Altman S. // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 8686–8689. https://doi.org/10.1073/pnas.1306911110
Soler Bistué A.J.C., Martín F.A., Vozza N., Ha H., Joaquín J.C., Zorreguieta A., Tolmasky M.E. // Proc. Natl. Acad. Sci. USA. 2009. V. 106. P. 13 230–13 235. https://doi.org/10.1073/pnas.0906529106
Jackson A., Jani S., Sala C.D., Soler-Bistué A.J., Zorreguieta A., Tolmasky M.E. // Biol. Methods Protoc. 2016. V. 1. P. bpw001. https://doi.org/10.1093/biomethods/bpw001
Shen N., Ko J., Xiao G., Wesolowski D., Shan G., Geller B., Izadjoo M., Altman S. // Proc. Natl. Acad. Sci. USA. 2009. V. 106. P. 8163–8168. https://doi.org/10.1073/pnas.0903491106
Augagneur Y., Wesolowski D., Tae H.S., Altman S., Ben Mamoun C. // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. 6235–6240. https://doi.org/10.1073/pnas.1203516109
Sala C.D., Soler-Bistué A.J.C., Korprapun L., Zorreguieta A., Tolmasky M.E. // PLoS One. 2012. V. 7. P. e47 690. https://doi.org/10.1371/journal.pone.0047690
Sawyer A.J., Wesolowski D., Gandotra N., Stojadinovic A., Izadjoo M., Altman S., Kyriakides T.R. // Int. J. Pharm. 2013. V. 453. P. 651–655. https://doi.org/10.1016/j.ijpharm.2013.05.041
Danilin N.A., Koroleva L.S., Novopashina D.S., Venyaminova A.G. // Russ. J. Bioorg. Chem. 2019. V. 45. P. 825–832. https://doi.org/10.1134/S106816201906013X
Jani S., Jackson A., Davies-Sala C., Chiem K., Soler-Bistué A., Zorreguieta A., Tolmasky M.E. // Methods Mol. Biol. 2018. V. 1737. P. 89–98. https://doi.org/10.1007/978-1-4939-7634-8_6
Ahmad A., Ranjan S., Zhang W., Zou J., Pyykkö I., Kinnunen P.K.J. // Biochim. Biophys. Acta. 2015. V. 1848. P. 544–553. https://doi.org/10.1016/j.bbamem.2014.11.008
Kawamoto Y., Bando T., Sugiyama H. // Bioorg. Med. Chem. 2018. V. 26. P. 1393–1411. https://doi.org/10.1016/j.bmc.2018.01.026
Lin J., Nagase H. // Biomolecules. 2020. V. 10. P. 544. https://doi.org/10.3390/biom10040544
Dervan P.B., Doss R.M., Marques M.A. // Curr. Med. Chem. Anti-Cancer Agents. 2005. V. 5. P. 373–387. https://doi.org/10.2174/1568011054222346
Wong D., Nielsen T.B., Bonomo R.A., Pantapalangkoor P., Luna B., Spellberg B. // Clin. Microbiol. Rev. 2017. V. 30. P. 409–447. https://doi.org/10.1128/CMR.00058-16
Harding C.M., Hennon S.W., Feldman M.F. // Nat. Rev. Microbiol. 2018. V. 16. P. 91–102. https://doi.org/10.1038/nrmicro.2017.148
Doi Y., Bonomo R.A., Hooper D.C., Kaye K.S., Johnson J.R., Clancy C.J., Thaden J.T., Stryjewski M.E., van Duin D. // Clin. Infect. Dis. 2017. V. 64 (Suppl. 1). P. S30–S35. https://doi.org/10.1093/cid/ciw829
Tacconelli E., Carrara E., Savoldi A., Harbarth S., Mendelson M., Monnet D.L., Pulcini C., Kahlmeter G., Kluytmans J., Carmeli Y., Ouellette M., Outterson K., Patel J., Cavaleri M., Mcox E., Rhouchens C.R., Grayson L., Hansen P., Singh N., Theuretzbacher U., Magrini N., and the WHO Pathogens Priority List Working Group Aboderin A.O., Al-Abri S.S., Jalil N.A., Benzonana N., Bhattacharya S., Brink A.J., Burkert F.R., Cars O., Cornaglia G., Dyar O.J., Wfriedrich A.W., Gales A.C., Gandra S., Giske C.G., Goff D.A., Goossens H., Gottlieb T., Blanco M.G., Hryniewicz W., Kattula D., Jinks T., Skanj S.S., Kerr L., Kieny M.-P., Kim Y.S., Kozlov R.S., Labarca J., Laxminarayan R., Leder K., Leibovici L., Levy-Hara G., Littman J., Malhotra-Kumar S., Manchanda V., Moja L., Ndoye B., Pan A., Paterson D.L., Paul M., Qiu H., Ramon-Pardo P., Rodríguez-Baño J., Sanguinetti M., Sengupta S., Sharland M., Si-Mehand M., Lsilver L.L., Song W., Steinbakk M., Thomsen J., Thwaites G.E., van der Meer J.W.M., Kinh N.V., Vega S., Villegas M.V., Wechsler-Fördös A., Wertheim H.F.L., Wesangula E., Woodford N., Oyilmaz F.O., Zorzet A. // Lancet. Infect. Dis. 2018. V. 18. P. 318–327. https://doi.org/10.1016/S1473-3099(17)30753-3
Tacconelli E., Magrini N. // Global Priority List of Antibiotic-Resistant Bacteria to Guide Research, Discovery, and Development of New Antibiotics. Geneva: World Health Organization, 2018.
Вишняков И.Е., Борхсениус С.Н. // Цитология. 2007. Т. 49. С. 421–429. [Vishnyakov I.E., Borchsenius S.N. // Cell Tissue Biol. 2007. V. 1. P. 206–214.] https://doi.org/10.1134/S1990519X07030029
Сидоренко С.В., Тишков В.И. // Усп. биол. химии. 2004. Т. 44. С. 263–306.
Cummins L.L., Owens S.R., Risen L.M., Lesnik E.A., Freier S.M., McGee D., Guinosso C.J., Cook P.D. // Nucleic Acids Res. 1995. V. 23. P. 2019–2024. https://doi.org/10.1093/nar/23.11.2019
Boutorine A.S., Venyaminova A.G., Repkova M.N., Sergueyeva Z.A., Pyshnyi D.V. // Biochimie. 1994. V. 76. P. 23–32. https://doi.org/10.1016/0300-9084(94)90059-0
Новопашина Д.С., Назаров А.С., Воробьева М.А., Купрюшкин М.С., Давыдова А.С., Ломзов А.А., Пышный Д.В., Альтман С., Веньяминова А.Г. // Мол. биология. 2018. Т. 52. С. 1045–1054. [Novopashina D.S., Nazarov A.S., Vorobjeva M. A., Kuprushkin M.S., Davydova A.S., Lomzov A.A., Pyshnyi D.V., Altman S., Venyaminova A.G. // Mol. Biol. 2018. V. 52. Р. 905–912.] https://doi.org/10.1134/S0026893318060134
Jiang X., Sunkara N., Lu S., Liu F. // Methods Mol. Biol. 2014. V. 1103. P. 45–56. https://doi.org/10.1007/978-1-62703-730-3_4
Perreault J.P., Altman S. // J. Mol. Biol. 1992. V. 226. P. 399–409. https://doi.org/10.1016/0022-2836(92)90955-j
Новопашина Д.С., Синяков А.Н., Рябинин В.А., Перpуо Л., Жиованнанжели К., Веньяминова А.Г., Буторин А.С. // Биоорг. химия. 2013. Т. 39. С. 159–174. [Novopashina D.S., Sinyakov A.N., Ryabinin V.A., Perrouault L., Giovannangeli C., Venyaminova A.G., Boutorine A.S. // Russ. J. Bioorg. Chem. 2013. V. 39. P. 138–152.] https://doi.org/10.1134/S1068162013010081
Guerrier-Takada C., Lumelsky N., Altman S. // Science. 1989. V. 246. P. 1578–1584. https://doi.org/10.1126/science.2480641
Guerrier-Takada C., Gardiner K., Marsh T., Pace N., Altman S. // Cell. 1983. V. 35. P. 849–857. https://doi.org/10.1016/0092-8674(83)90117-4
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия



