Биоорганическая химия, 2021, T. 47, № 2, стр. 235-249
ДНК с 2-пиридилдитиогруппой при С2'-атоме – перспективные инструменты для фиксации белка MutS с сохранением его функциональной активности
С. А. Перри 1, Е. А. Кубарева 1, М. В. Монахова 1, *, Р. М. Трикин 2, Е. М. Косарецкий 1, Е. А. Романова 1, В. Г. Метелев 1, П. Фридхофф 3, Т. С. Орецкая 1
1 Научно-исследовательский институт физико-химической биологии имени А.Н. Белозерского, химический факультет и факультет биоинженерии и биоинформатики Московского государственного университета имени М.В. Ломоносова
119991 Москва, Ленинские горы, 1, Россия
2 ФГАОУ ВО Первый МГМУ им. И.М. Сеченова Минздрава России (Сеченовский Университет)
119991 Москва, ул. Трубецкая, 8, стр. 2, Россия
3 Институт биохимии, FB 08, Университет Ю. Либиха
D-35392 Гиссен, Heinrich-Buff-Ring, 17, Германия
* E-mail: monakhovamv@gmail.com
Поступила в редакцию 01.10.2020
После доработки 18.10.2020
Принята к публикации 20.10.2020
Аннотация
Нековалентные ДНК-белковые взаимодействия лежат в основе жизнедеятельности клеток. Ковалентные связи между ДНК и белком, возникающие при воздействии различных эндогенных и экзогенных факторов, а также химиотерапевтических агентов, как правило, нарушают нормальное функционирование ДНК-связывающих белков. Олигодезоксирибонуклеотиды с реакционноспособной группировкой используются как инструменты для подтверждения элементов структуры и механизмов действия таких белков. Для фиксации белка MutS, являющегося сенсором системы MMR (система репарации “мисматчей”, mismatch repair system) из Escherichia coli, через остаток цистеина предложено использовать ДНК с 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]группой в составе нуклеозида в заданном положении олигомерной цепи и с двумя флуорофорами. Дуплекс представляет собой набор комплементарных олигонуклеотидов с разрывами в “верхней” и “нижней” цепях. Это позволяет варьировать место введения реакционноспособной группировки, синтезируя только центральный фрагмент ДНК минимальной длины. Конъюгат MutS c ДНК был получен реакцией тиол-дисульфидного обмена с количественным выходом и очищен от непрореагировавшей ДНК с использованием эксклюзионной хроматографии. Методом флуоресцентного резонансного переноса энергии показано, что белок MutS в составе конъюгата сохраняет способность изменять свою конформацию и конформацию ДНК, что свидетельствует о сохранении его активности.
ВВЕДЕНИЕ
Метод ковалентного связывания (“кросслинкинг”) белков с ДНК позволяет зафиксировать комплекс, образующийся в результате взаимодействия биомолекул, а также изучить особенности его формирования, локализацию белков на ДНК и расположение всех компонентов комплекса относительно друг друга и ДНК. Данный подход предусматривает следующие этапы исследования: 1) конструирование модельной системы, состоящей из белка и нуклеиновой кислоты, содержащих реакционноспособные группировки, и позволяющей с высоким выходом получить конъюгат белка с ДНК-лигандом; 2) выделение конъюгата в гомогенном состоянии; 3) проверка функциональной активности белка в составе конъюгата.
Для ковалентного связывания компонентов белково-нуклеинового комплекса предложено множество реакционноспособных группировок, различающихся по природе образующейся химической связи, скорости и эффективности ее формирования [1–8].
Мы сосредоточили свои усилия на разработке методов “кросслинкинга” белков с ДНК, основанных на реакции тиол-дисульфидного обмена [9–16]. В данной работе продолжено изучение свойств ДНК, содержащих 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]группу в составе нуклеозида в заданном положении цепи. Эта группировка позволяет зондировать электростатические контакты белка с углеводофосфатным остовом ДНК. Она успешно использовалась при изучении свойств фактора транскрипции NF-κВ [14] и никующей эндонуклеазы BspD6I [15]. В данной работе мы поставили задачу выделить ковалентно связанный комплекс белка с ДНК, содержащей 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]- группу, и выяснить, сохраняет ли он свою функциональную активность в составе конъюгата.
В качестве модели для изучения свойств разрабатываемых нами ДНК-реагентов хорошо зарекомендовал себя белок MutS из системы репарации неканонических пар нуклеотидов или “мисматчей” (MMR) Escherichia coli. MutS представляет собой сенсор, сканирующий ДНК в поисках “мисматча”. В присутствии ADP MutS узнает и связывает “мисматч”-содержащую ДНК, образуя с ней стабильный “начальный” комплекс. Связывание MutS приводит к изгибу линейной ДНК на 60° в случае пары G/T. Известно, что большинство контактов белка MutS с ДНК являются электростатическими, и аминокислотные остатки в районе “мисматча” сближены именно с углеводофосфатным остовом [17–21].
В присутствии ATP происходит формирование “окончательного” комплекса, в котором MutS переходит в конформацию “скользящий зажим”, что сопровождается уходом белка от некомплементарной пары и распрямлением ДНК [22–24]. Описанные конформационные переходы ДНК в комплексе с MutS можно наблюдать по изменению сигнала флуоресцентного резонансного переноса энергии [9, 25, 26] (рис. 1).
Ранее нами уже были получены и выделены конъюгаты MutS с ДНК и показана возможность проверки функциональной активности зафиксированного белка [9, 16]. В первом случае конъюгаты представляли собой мутантные формы MutS, “сшитые” с 3′-концевой [3-(3-гидроксипропилдитио)пропил]фосфатной группой, что являлось определенным ограничением для последующих исследований взаимодействия MutS с другими белковыми партнерами. Во втором случае модификация в ДНК вводилась по С5-атому тимидина, содержащему гексаметиленовый линкер. В зависимости от модифицирующего агента в работе были синтезированы ДНК, содержащие пиридилдитиогруппу на различном расстоянии от ДНК (18–50 Å). Однако такие ДНК-реагенты не позволяют реализовать “сшивку нулевой длины”, т.е. фиксировать контакты белка на более близком (~10 Å) расстоянии от ДНК.
Использование пиридилдитиогруппы, включенной в 2′-положение нуклеотида, дает возможность синтезировать ДНК-реагент без разрыва в месте модификации и получить конъюгат MutS–ДНК на коротком линкере (~7 Å). 17-Звенный олигодезоксирибонуклеотид, несущий 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]группу, являлся фрагментом дуплекса (I), составленного из четырех комплементарных олигонуклеотидов, два из которых содержат флуорофоры (рис. 2). Такой ДНК-дуплекс может работать как “конструктор”, аналогично предложенному нами ранее в работе Perevozchikova et al. [25]. Дизайн дуплекса позволяет менять только один олигонуклеотид с реакционноспособной группой, если нужно варьировать положение модификации относительно “мисматча” и флуоресцентных меток.
Рис. 2.
Модифицированные ДНК-дуплексы, использованные в данной работе. U – 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]уридин.

В представленной работе подобраны оптимальные условия ковалентного связывания мутантных форм MutS из E. coli – MutS(A469C/Δ801–853) и MutS(N497С/Δ801–853) – с ДНК. Оптимизирован метод эксклюзионной хроматографии, позволивший выделить конъюгат MutS–ДНК. Основываясь на изменении интенсивности сигнала FRET, мы показали, что зафиксированный на ДНК белок MutS является функционально активным: находясь в составе конъюгата, он способен изменять конформацию ДНК в присутствии ATP.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Конструирование модельных реакционноспособных фрагментов ДНК. В качестве базового ДНК-лиганда был использован 45-звенный дуплекс (II) (рис. 2), представляющий собой фрагмент последовательности плазмиды pUC-MMR, который хорошо себя зарекомендовал в экспериментах по исследованию инициации репарации “мисматчей” ДНК с помощью белков MutS, MutL и MutH in vitro [27]. Этот дуплекс в центральной позиции содержит G/T-пару, которая является одним из повреждений, наиболее эффективно узнаваемых и репарируемых системой MMR [28].
Предложенный ДНК-дуплекс имеет важную особенность: он содержит в своем составе FRET-пару, состоящую из флуорофоров Alexa-488 (донор, максимумы λпогл/λисп = 495/519 нм) и Alexa-594 (акцептор, максимумы λпогл/λисп = 590/617 нм). Эти флуорофоры присоединены к С5-атому остатка тимидина через гексаметиленовые линкеры и находятся в разных цепях ДНК-дуплекса на расстоянии 12 п.н. от “мисматча”. В работе Cristóvão et al. [17] было показано, что такие модифицированные гетероциклические основания в составе ДНК не являются субстратами системы репарации ММR и могут быть использованы в качестве флуоресцентных меток. Ранее нами было продемонстрировано, что FRET-пара в составе дуплекса (II) позволяет следить за изменением его пространственной структуры [25].
Для получения 45-звенного ДНК-дуплекса (II) мы использовали гибридизацию на олигонуклеотиде-матрице (“верхняя” цепь), содержащем остаток G в “мисматче” и флуорофор Alexa-594, трех олигонуклеотидов (“нижняя” цепь): 13-мера с Alexa-488, 17-мера с остатком Т в “мисматче” и 15‑мера. Предложенная конструкция дуплекса позволяет варьировать структуру как 15-, так и 17‑мера, вводя в заданную позицию модифицированное звено с пиридилдисульфидной группой, не меняя олигонуклеотид-матрицу и сохраняя оптимальное расположение FRET-пары (например, дуплекс (I), рис. 2). Наличие разрывов в цепи ДНК не влияет на характер связывания MutS с ДНК-лигандом, содержащим некомплементарную пару [17, 25].
Следующим этапом работы был выбор места введения пиридилдисульфидной группировки в 17-звенный фрагмент “нижней” цепи 45-звенного дуплекса. Ранее для “кросслинкинга” белка с дисульфид-содержащей ДНК нами были успешно использованы мутантные формы MutS, содержащие остаток Cys в положениях А469 или N497, локализованных в составе домена “зажим”. Эти аминокислотные остатки эффективно взаимодействовали с ДНК, несущими дисульфидную группировку, присоединенную к 3′-концу седьмого нуклеотида в 3′-направлении от остатка Т “мисматча” (положение –7) [16].
В базе данных Protein Data Bank (PDB) найдены 10 структур комплексов MutS с ДНК [20, 29–32] (табл. 1). Анализ трех кристаллических структур MutS в комплексе с дуплексом, содержащим “мисматч” (PDB: 1W7A, 10H7, 1E3M), а также данных литературы [20, 30] показал, что А469 или N497 сближены с нуклеотидами не только в положении –7, но и в пятом положении в 5′- или 3′-направлении от Т “мисматча” (положения +5 и –5), независимо от молекулы кофактора, а также длины дуплекса (табл. 2, рис. 3). Мы рассчитали расстояния (Å) от первого атома углерода (СВ) боковой цепи А469 и N497 до атома С2′ нуклеотидов в 5′- или 3′-направлении от Т “мисматча” (табл. 2). При замене А469 и N497 на Cys и введении в нуклеотид модифицированной группировки расстояние между реагирующими группами может сократиться до 3–4 Å. Такое расстояние должно обеспечить возможность протекания химической реакции между MutS и 2-пиридилдисульфидной группой в 2′-положении углеводного фрагмента ДНК.
Таблица 1.
Кристаллические структуры MutS с ДНК*
| ДНК-лиганд (5′–3′) (3′–5′) |
Кофакторы | Разрешение, Å | Код PDB | Ссылка |
|---|---|---|---|---|
| AGCTGCCAGGCACCAGTGTCAGCGTCCTAT TCGACGGTTCGTGGTCACAGTCGCAGGATA |
ATP, Mg2+ | 2.27 | 1W7A | [29] |
| AGCTGCCAAGCACCAGTGTCAGCGTCCTAT TCGACGGTACGTGGTCACAGTCGCAGGATA |
ADP, Mg2+ | 2.40 | 1OH6 | [20] |
| AGCTGCCAGGCACCAGTG TCGACGGTTCGTGGTCA |
ADP, Mg2+ | 2.10 | 1WB9 | [30] |
| AGCTGCCAGGCACCAGTG TCGACGGTTCGTGGTCA |
ADP, Mg2+ | 2.50 | 1WBB | [30] |
| AGCTGCCAGGCACCAGTGTCAGCGTCCTAT TCGACGGTTCGTGGTCACAGTCGCAGGATA |
ADP, Mg2+ | 2.20 | 1E3M | [31] |
| AGCTGCCACGCACCAGTGTCAGCGTCCTAT TCGACGGTACGTGGTCACAGTCGCAGGATA |
ADP, Mg2+ | 2.90 | 1OH5 | [20] |
| AGCTGCCAGGCACCAGTGTCAGCGTCCTAT TCGACGGTGCGTGGTCACAGTCGCAGGATA |
ADP, Mg2+ | 2.50 | 1OH7 | [20] |
| AGCTGCCA-GGCACCAGTGTCAGCGTCCTAT TCGACGGTTCCGTGGTCACAGTCGCAGGATA |
ADP, Mg2+ | 2.90 | 1OH8 | [20] |
| AGCTGCCAGGCACCAGTG TCGACGGTTCGTGGTCA |
ADP, Mg2+ | 2.40 | 1WBD | [20] |
| AGCTGCCAGGCACCAGTGTCA TCGACGGTTCGTGGTCACAGT |
ADP, Mg2+ | 3.10 | 3ZLJ | [32] |
Таблица 2.
Рассчитанные расстояния от атома CB боковой цепи А469 и N497 до атома С2ꞌ нуклеотида в структурах комплексов MutS–ДНК*
| Положение нуклеотида** | Расстояние, Å | |||
|---|---|---|---|---|
| А469 | N497 | |||
| субъединица А | субъединица Б | субъединица А | субъединица Б | |
| –7 | 19.29 19.18 19.30 |
8.51 9.92 9.71 |
9.05 8.29 8.14 |
22.22 22.55 22.61 |
| –5 | 9.54 9.62 9.61 |
15.44 15.25 15.23 |
13.19 13.39 13.40 |
17.96 17.89 17.98 |
| +5 | 22.43 22.54 22.37 |
28.05 27.90 27.83 |
30.27 30.55 30.51 |
8.56 8.54 8.48 |
Рис. 3.
Контакты углеводного фрагмента и фосфатного остатка нуклеотидов в положениях –7, –5 и +5 ДНК-дуплекса с субъединицами А и Б гомодимера MutS в белково-нуклеиновом комплексе (PDB: 1WB9).

Для проверки этого предположения была сконструирована серия 17-звенных ДНК-дуплексов (рис. 2). ДНК-дуплекс (III) входит в состав предложенной нами 45-звенной системы. Последовательность в дуплексах (IV) и (V) соответствует последовательности фрагмента дуплекса, использовавшегося при кристаллизации комплекса MutS с ДНК [31]. В этих дуплексах единственный остаток U введен в 5-ю позицию с одной или другой стороны от Т “мисматча”. Поскольку контакты MutS с ДНК (кроме области “мисматча”) носят неспецифический характер, мы посчитали возможным заменить в “нижней” цепи остаток А (5-й с 3′-конца от “мисматча”) на U. В качестве контроля синтезирован модифицированный дуплекс (VI), который является аналогом дуплекса (III), но не содержит “мисматч”. Белок MutS, отвечающий за узнавание “мисматча”, должен взаимодействовать с таким дуплексом менее эффективно по сравнению с остальными. Для получения дуплексов (III–VI) 2′-дисульфидсодержащие 17-звенные олигонуклеотиды, синтезированные по методике Metelev et al. [15], гибридизовали с комплементарными матрицами.
Характеристика мутантных форм белка MutS с единичным остатком цистеина. Для создания белков MutS, содержащих единственный остаток Cys (SC, Single Cys), мы использовали вариант белка MutS – CFMutS(Δ801–853), который не имеет остатков Cys (Сys Free), а также не содержит 53 С‑концевые аминокислоты, практически не важные для процесса репарации “мисматчей” [33], но участвующие в тетрамеризации MutS [31]. В реакциях ковалентного связывания MutS с модифицированными ДНК, требующих высокой концентрации биомолекул, олигомеризация белка нежелательна. Ранее было доказано, что CFMutS(Δ801–853) имеет то же сродство к ДНК с G/T-парой, что и белок дикого типа (WTMutS) [34]. Далее для белка CFMutS и мутантных форм, полученных на его основе, обозначение (Δ801–853) опущено. В качестве контроля в данной работе использовали полноразмерный WTMutS, содержащий шесть остатков Cys на мономер белка.
Генетические конструкции для экспрессии мутантных форм SCMutS(A469C) и SCMutS(N497C) были получены ранее сайт-направленным мутагенезом [16]. Белки выделяли, как описано в работах Heinze et al. [16] и Feng et al. [35]. Характеристика их свойств в сравнении с WTMutS и CFMutS приведена ниже. Далее по тексту аббревиатура SC опускается. Согласно имеющимся данным [16, 25, 36], при связывании белка MutS с ДНК даже канонического строения происходит ускорение обмена ADP (вплоть до 10‑кратного) в АТРазных доменах гомодимера WTMutS. Сравнительную характеристику мутантных форм по этому признаку выполняли с использованием флуоресцентного аналога ADP – 2′/3′-O-(N-метилантранилоил)аденозин-5′-дифосфата (МАНТ-ADP) – в отсутствие и в присутствии 30-звенного дуплекса (VII) 5′-CAAGCCTATGCCCTCAGCACCCAGGGTGCC-3′/3′-GTTCGGATACGGGAGTCGTGGGTCCCACGG-5′, не содержащего “мисматч” (концентрация 500 нМ).
В отсутствие ДНК в реакционной смеси значения констант скорости высвобождения МАНТ-ADP оказались сходными (в пределах погрешности) для всех вариантов MutS (0.007–0.012 с–1) (табл. 3). В присутствии ДНК для WTMutS, CFMutS и MutS(A469C) получены близкие значения koff (0.043–0.049 с–1). Таким образом, обмен МАНТ-ADP на ADP в АТРазных доменах этих вариантов MutS в присутствии ДНК в условиях наших экспериментов происходил в 4–6 раз быстрее по сравнению с реакционной смесью в отсутствие ДНК. Для MutS(N497C) наблюдалось не столь эффективное ускорение обмена ADP в АТРазных доменах – в 2.3 раза (табл. 3), что может быть связано либо с меньшим сродством рассматриваемой мутантной формы MutS к ДНК, либо с некоторым нарушением взаимной регуляции АТРазных и ДНК-связывающего доменов в этом белке.
Таблица 3.
Эффективность обмена МАНТ-ADP на ADP в АТРазных доменах различных форм MutS
| Белок | Скорость высвобождения МАНТ-ADP из комплекса с белком MutS, koff, с–1* | Ускорение обмена ADP в MutS в присутствии ДНК, koff (+ДНК)/koff (–ДНК) |
|
|---|---|---|---|
| в отсутствие дуплекса (VII), koff (–ДНК) |
в присутствии дуплекса (VII), koff (+ДНК) |
||
| WTMutS | 0.012 ± 0.001 | 0.043 ± 0.002 | 3.6 |
| CFMutS | 0.008 ± 0.002 | 0.047 ± 0.001 | 5.6 |
| MutS(A469C) | 0.011 ± 0.005 | 0.049 ± 0.007 | 4.3 |
| MutS(N497C) | 0.007 ± 0.003 | 0.015 ± 0.003 | 2.3 |
Для характеристики функциональных свойств полученных мутантных форм белков была исследована способность MutS(A469C), MutS(N497C) и CFMutS активировать гидролиз суперскрученной (сс) плазмиды pUC-MMR, содержащей неканоническую G/T-пару и последовательность 5′-GATC-3′/3′-CTm6AG-5′, эндонуклеазой MutH в присутствии белка MutL (рис. 4). Об активности MutH судили по образованию ДНК с разрывом только в одной из цепей (оц – одноцепочечный разрыв). Реакционные смеси анализировали в агарозном геле, содержавшем бромид этидия. Оценивали увеличение в ходе реакции интенсивности зоны, соответствующей плазмиде с одноцепочечным разрывом.
Рис. 4.
Анализ продуктов гидролиза белком MutH плазмиды pUC-MMR, содержащей неканоническую пару G/T и монометилированную последовательность 5′-GATC-3′/3′-CTm6AG-5′, в присутствии MutL и мутантных форм MutS методом электрофореза в 1%-ном агарозном геле с бромидом этидия. Верхние зоны соответствуют ДНК с разрывом только в одной из цепей (оц), а нижние – суперскрученной плазмиде (сс). М – маркер длины ДНК GeneRuler™ 1 kb.
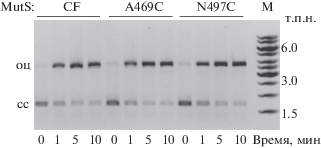
В наших экспериментах уже за 5 мин наблюдался практически полный переход ДНК-субстрата с G/T-“мисматчем” в ДНК с одноцепочечным разрывом в присутствии CFMutS (рис. 4). Эффективность гидролиза ДНК эндонуклеазой MutH в присутствии MutS(A469C) и MutS(N497C) в пределах ошибки близка к таковой для CFMutS. Интересно, что мутантная форма MutS(N497C), характеризующаяся пониженной способностью к обмену ADP в присутствии ДНК, активировала последующие этапы репарации.
Таким образом, мутантные формы MutS, содержащие единственный остаток Cys на мономер в положениях 469 и 497, сохраняют основные биохимические свойства белка MutS и в дальнейшем могут использоваться для структурно-функциональных исследований системы MMR.
Взаимодействие мутантных форм MutS с 17-звенными ДНК-дуплексами с 2'-пиридилдисульфидной группировкой. Схема реакции между гомодимером мутантной формы MutS, содержащим единственный остаток Cys на мономер, и ДНК с одним остатком 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]уридина представлена на рис. 5.
Рис. 5.
Схема реакции тиол-дисульфидного обмена между остатком цистеина MutS и 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]уридином в составе ДНК-дуплекса.

45-Звенный дуплекс (I), предложенный нами для последующих экспериментов, содержит в своем составе 17-звенный дуплекс (III) с реакционноспособной группировкой. Поэтому взаимодействие именно дуплекса (III) с мутантными формами MutS было охарактеризовано нами в первую очередь. В экспериментах использовали белок MutS(А469С), т.к. он проявлял наибольшее сходство с белком дикого типа. Более того, остаток 469 субъединицы А гомодимера MutS находится также на расстоянии <10 Å от положения ‒5 G/T-содержащего дуплекса, использованного в РСА (рис. 3, табл. 2).
Ковалентное связывание мутантной формы MutS(А469С) с 5′-32Р-меченным ДНК-дуплексом (III) проводили в буфере, содержавшем 1 мМ ADP, в течение 30 мин при 37°С. Для достижения максимального выхода конъюгата использовали 5-кратный избыток ДНК по отношению к белку. Как видно из изображений геля, окрашенного раствором кумасси G250, и его радиоавтографа (рис. 6), кроме зоны, соответствующей белку (молекулярная масса ~90 кДа), наблюдалась дополнительная зона с большей молекулярной массой (MutS × ДНК). В этой зоне обнаруживалась радиоактивность, что подтверждало наличие в ней ДНК и позволяло считать эту зону продуктом “кросслинкинга”. Из двух остатков Cys гомодимеров мутантных форм MutS только один способен образовывать ковалентную связь с реакционноспособной ДНК. Таким образом, 100%-ному выходу конъюгатов мутантных форм MutS с модифицированными ДНК в расчете на белок соответствует 50%-ная модификация этих белков. Выход конъюгата MutS(А469С) с ДНК-дуплексом (III) составлял >80%, т.к. >40% белка находилось в составе зоны, соответствующей конъюгату (рис. 6a, дорожка 3). Кроме зоны, соответствующей конъюгату MutS(А469С) с ДНК-дуплексом, наблюдалась зона с очень низкой подвижностью в 8%-ном SDS-ПААГ (>200 кДа), соответствующая димеру MutS (MutS × MutS). Образованию белок-белковой сшивки через дисульфидный мостик в данном случае способствует сближение остатков Cys в гомодимере мутантной формы MutS(А469С). При добавлении 10 мМ DTT в реакционную смесь зоны, соответствующие конъюгату и димеру белка, исчезают (рис. 6, дорожки 4), что подтверждает наличие в них дисульфидной связи.
Рис. 6.
Анализ продуктов ковалентного связывания мутантной формы MutS(А469С) и дуплекса (III) методом электрофореза в 8%-ном SDS-ПААГ. (а) – Фотография геля, окрашенного раствором кумасси G250; (б) – радиоавтограф геля. Дорожка 1 – препарат MutS(А469С) с 10 мМ DTT (а); дорожка 2 – препарат дуплекса (III); дорожки 3 и 4 – реакционные смеси в буфере А, не содержащем и содержащем 10 мМ DTT соответственно. М – маркер молекулярной массы PageRuler™ (10–200 кДа).

Для оценки специфичности предложенного ДНК-реагента было изучено взаимодействие дуплексов (IV)–(VI), содержащих единичный остаток 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]уридина, с белками CFMutS, MutS(A469C) и MutS(N497C). В качестве примера на рис. 7 приведены результаты анализа реакционных смесей для дуплекса (IV) и различных форм белка MutS. CFMutS, не содержащий остатков Cys, как и ожидалось, не образовывал ковалентный комплекс ни с одним ДНК-дуплексом (рис. 7a). Эффективность образования ковалентно связанного комплекса при оценке по белку для всех дуплексов и мутантных форм MutS(A469C) и MutS(N497C) составляла 60–100%. Тем не менее наблюдались различия в эффективности образования конъюгатов для MutS(N497C) и MutS(A469C) с дуплексами (III)–(VI). Например, по данным РСА, реакционноспособная группировка ДНК-дуплекса (V) должна быть сближена с остатком 497 из субъединицы Б MutS (табл. 2, рис. 3). Образование конъюгата этого ДНК-дуплекса с мутантной формой MutS(N497C) происходило с максимальной эффективностью (90–100% по белку) и менее эффективно – с мутантной формой MutS(A469C) (60–70% по белку) (рис. 7). В условиях наших экспериментов образование ковалентного комплекса с ДНК-дуплексом (VI) канонического строения также происходило с высоким выходом (до 60% в расчете на белок). Ранее такой факт наблюдался в случае фактора транскрипции NF-κВ и никующей эндонуклеазы BspD6I, которые имеют высокое сродство к неспецифической ДНК [14, 15]. Этим же свойством обладает белок MutS [31, 33, 34]. Возможно, остатки Cys мутантных форм MutS сближаются с реакционноспособной группировкой ДНК в процессе поиска “мисматча” и успевают образовать с ней ковалентную связь. В случае MutS при взаимодействии с ДНК-дуплексом (VI) на геле наблюдаются несколько зон, соответствующих конъюгатам. Это связано с тем, что 17-звенный дуплекс канонического строения более устойчив по сравнению с “мисматч”-содержащим и не полностью денатурирует в условиях гель-электрофореза. Наблюдаемые продукты, по-видимому, соответствуют конъюгатам MutS с одной цепью и ДНК-дуплексом. Вместе с тем кинетический анализ реакции “кросслинкинга” MutS(A469C) с ДНК-реагентами, содержащими 3′-концевую пиридилдисульфидную группу, показал в 4 раза большую скорость образования конъюгата с “мисматч”-содержащим дуплексом по сравнению с каноническим [16]. В то же время гомодимер WTMutS, имеющий в своем составе 12 остатков Cys, не формировал конъюгаты с дуплексами (III)–(VI), содержащими 2′-пиридилдисульфидное звено.
Рис. 7.
Взаимодействие WTMutS и мутантных форм CFMutS, MutS(A469C), MutS(N497C) с 32P-меченными ДНК-дуплексами (IV)–(VI), содержащими единичный остаток 2′-дезокси-2′-[3-(2-пиридилдитио)пропионамидо]уридина. Радиоавтограф (а) и фотография (б) 8%-ного SDS-ПААГ, окрашенного раствором кумасси G250. Дорожки 1–3 – реакционные смеси, содержащие CFMutS, MutS(A469C), MutS(N497C) и дуплекс (IV) соответственно; дорожка 4 – препарат WTMutS; дорожки 5 и 6 – реакционные смеси, содержащие WTMutS и дуплекс (V) или (VI) соответственно; дорожка 7 – препарат MutS(A469C); дорожки 8 и 9 –реакционные смеси, содержащие MutS(A469C) и дуплекс (V) или (VI) соответственно. М – маркер молекулярной массы PageRuler™ (10–200 кДа).

В совокупности представленные в данной работе и описанные нами ранее результаты [9, 14–16] подтверждают, что для образования ковалентной связи необходимо непосредственное сближение Cys с описываемой реакционноспособной группировкой – либо в процессе сканирования ДНК, либо при образовании специфического ДНК-белкового комплекса.
“Кросслинкинг” 45-звенного дуплекса (I) с MutS(A469C) и выделение ДНК-белкового конъюгата. Выбранный и оптимизированный нами метод ковалентного присоединения мутантных форм MutS к 17-звенным дуплексам оказался очень эффективным. Мы использовали его для получения конъюгата 45-звенного дуплекса (I), содержащего FRET-пару, с MutS(A469C), а также для выделения полученного комплекса и проверки функциональной активности его белкового компонента.
В составе ДНК-дуплекса (I) присутствуют довольно короткие ДНК-фрагменты. Наиболее короткие из них – 15-звенный и 13-звенный, содержащий Alexa-488 – имеют расчетную температуру плавления ~44.5°С (рассчитана в программе OligoAnalyzer, см. “Эксперим. часть”). По результатам гель-электрофореза в неденатурирующих условиях наблюдалась частичная диссоциация этих коротких дуплексов. Было подобрано молярное соотношение олигонуклеотидов, содержащих флуорофоры, при котором наблюдалась максимальная эффективность FRET, и при этом избыток 13-звенного или 15-звенного олигонуклеотида был минимален (45-мер : 13-мер = = 45-мер : 15-мер = 1.0 : 1.1). Поскольку в данной работе измерялись относительные изменения флуоресценции, незначительный избыток однотяжевых ДНК, присутствовавший во всех экспериментах в равном количестве, не влиял на общую тенденцию. Образование дуплекса (I) контролировали методом гель-электрофореза в неденатурирующих условиях, как описано в работе Ede et al. [37]. Для повышения стабильности ДНК-дуплексов их формировали и хранили в присутствии 5 мМ MgCl2. Кроме того, эксперименты выполняли при температуре 20°С.
“Кросслинкинг” проводили в буфере А (см. “Эксперим. часть”), содержавшем 1 мМ ADP, 5 мкМ ДНК-дуплекс (I) и 1 мкМ MutS(A469C). Выход продукта был близок к количественному (рис. 8). Для выделения конъюгата MutS(A469C) c дуплексом (I) использовали эксклюзионную хроматографию с условиями разделения, описанными ранее [16]. Отметим, что выделение конъюгатов MutS с протяженным дуплексом, имеющим составную “нижнюю” цепь и флуорофоры в разных цепях, методом эксклюзионной хроматографии выполнено впервые. Наличие флуорофоров в составе этого дуплекса позволяет эффективно контролировать разделение компонентов и оценивать стабильность ДНК в процессе очистки. За ходом разделения следили не только при 260 и 280 нм, но и при 480 нм (поглощение Alexa-488) и 590 нм (поглощение Alexa-594). На рис. 8а приведен пример хроматограммы разделения компонентов реакции между MutS(A469C) и дуплексом (I).
Рис. 8.
Выделение конъюгата MutS(A469C) c дуплексом (I) методом эксклюзионной ВЭЖХ на колонке с сорбентом Superdex200 TM. (а) – Хроматограммы смеси, содержащей ДНК-белковый конъюгат. Детектировали оптическую плотность раствора при 260, 280, 490 и 590 нм; (б) – электрофоретический анализ фракций, полученных в результате эксклюзионной хроматографии конъюгата в градиентном 4–20%-ном SDS-ПААГ. Дорожки 1–4 соответствуют временам удерживания 22.0–23.5 мин. М – маркер молекулярной массы PageRuler™ (10–200 кДа). Гель окрашивали раствором солей серебра.
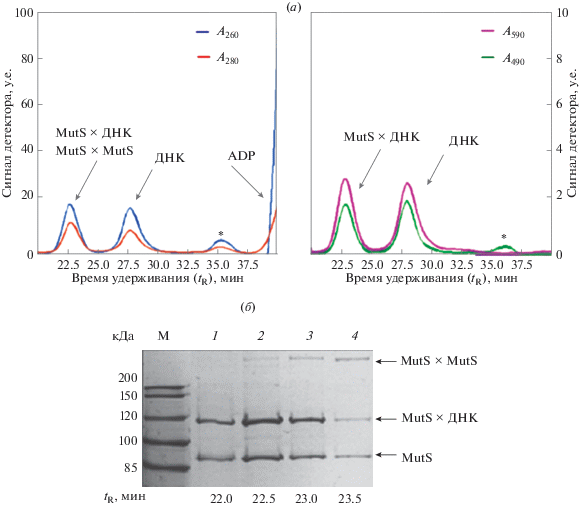
Для определения состава фракций использовали соотношение A260/A280. Время удерживания (tR) конъюгата MutS(A469C) с дуплексом (I) с массой ~200 кДа составляло ~22.5 мин (A260/A280 = 1.4). Время удерживания ДНК-дуплекса (I) (с массой ~30 кДа) составляло ~28 мин (A260/A280 = 1.9).
На хроматограмме наблюдался небольшой пик (на рис. 8а он обозначен звездочкой) с tR ≈ 36 мин и увеличенным поглощением в области 488 нм. По его поглощению можно предположить, что он соответствует 13-звенному ДНК-фрагменту, содержащему флуорофор Alexa-488, т.к. значение A260/A280 для него составляло 1.8. Как упоминалось, этот олигонуклеотид добавляли в незначительном избытке при составлении ДНК-дуплекса, что обеспечивало наиболее эффективное формирование ДНК-системы (I).
При электрофоретическом анализе образцов собранных фракций в градиентном 4–20%-ном SDS-ПААГ (рис. 8б) наблюдалось образование двух зон, соответствующих конъюгату мономера MutS(A469C) с дуплексом (I) и свободному белку в мономерной форме, а также образование зоны стабильного димера MutS(A469C), обладающего низкой подвижностью в условиях электрофореза. Во фракции пика с tR = 22 мин практически не наблюдалось примеси димера белка (рис. 8б, дорожка 1), поэтому именно эта фракция была использована в дальнейшем для функциональных исследований конъюгата. Таким образом, в ходе эксклюзионной хроматографии конъюгат MutS c ДНК был очищен от непрореагировавшей ДНК (двуцепочечной ДНК и олигонуклеотидов), однако полностью отделить его от непрореагировавшего белка не удалось.
Изменение флуоресцентных свойств дуплекса (I), ковалентно связанного с MutS(A469C), при добавлении АТР. В следующей серии экспериментов мы использовали выделенный конъюгат дуплекса (I) с MutS(A469C) и контрольный образец – “несшитый” комплекс MutS(A469C) с дуплексом (II), который аналогичен дуплексу (I), но не содержит реакционноспособную группировку (рис. 2). Эти образцы применяли для изучения конформационных перестроек ДНК при переходе “начального” комплекса, узнающего “мисматч”, в “окончательный”, в котором MutS находится в конформации “скользящий зажим” (рис. 1). Как упоминалось выше, в ходе этого процесса, протекающего в присутствии ATP, происходит разгибание ДНК, что сопровождается значительным уменьшением эффективности переноса энергии между флуорофорами в дуплексах (I) и (II).
Для регистрации образования “начального” комплекса, узнающего некомплементарную пару, записывали спектры испускания при возбуждении Alexa-488 и определяли значения анизотропии обоих флуорофоров. В условиях контрольного эксперимента вся ДНК была связана с белком MutS(A469C): соотношение концентраций дуплекса (II) и MutS(A469C) составляло 1 : 10 в расчете на мономер белка.
Для количественной интерпретации изменений, наблюдавшихся в спектрах флуоресценции, использовали отношение Δ(Fmax/Red), где Fmax – максимум интенсивности флуоресценции акцептора при возбуждении донора флуоресценции, а Red – максимум флуоресценции донора в условиях его возбуждения; Δ указывает на изменение величины Fmax/Red в ходе связывания MutS с ДНК. При расчетах вычитали значения Fmax/Red после связывания с MutS из значений Fmax/Red для раствора ДНК-дуплекса. Значение Δ(Fmax/Red) нивелирует эффект разведения реакционной смеси, что неизбежно в ходе эксперимента, и позволяет говорить об изменении эффективности переноса энергии между донором и акцептором флуоресценции во FRET-паре. Поскольку Δ(Fmax/Red) является отношением двух значений интенсивности флуоресценции, эта величина не имеет размерности.
Из спектров флуоресценции (рис. 9, образцы 1) следует, что для конъюгата MutS(A469C) с дуплексом (I) и не связанного ковалентно комплекса МutS(A469C) с дуплексом (II) наблюдался эффективный перенос энергии между флуорофорами. Значение Δ(Fmax/Red) составляло ~0.13 для дуплексов (I) и (II), что свидетельствует о наличии в растворах “начального” комплекса, узнающего “мисматч” (где ДНК изогнута на 60°) [31]. После добавления 1 мМ АТР к этим комплексам наблюдалось значительное уменьшение эффективности переноса энергии, что видно по изменению спектра флуоресценции (рис. 9, образцы 2). Значения Δ(Fmax/Red) изменились до 0.012. Такое изменение эффективности переноса энергии, по-видимому, свидетельствует об исчезновении изгиба ДНК, характерного для “начального” комплекса. “Скользящий зажим” MutS, формирующийся после добавления АТP, сразу диссоциирует, и ДНК-белковый комплекс разрушается (за исключением ДНК-фрагментов с заблокированными концами [26]). Именно поэтому в случае комплекса MutS(A469C) с дуплексом (II) значения анизотропии уменьшались с 0.3 до 0.15: Δr(Alexa-594) = 0.15 (рис. 9). Однако в случае “сшитого” комплекса наблюдалось практически полное отсутствие изменения анизотропии Alexa-594: Δr(Alexa-594) = 0.01. Добавление в реакционную смесь восстановителя (10 мМ DTT) приводило к разрушению конъюгата (данные не приведены). Значения анизотропии флуорофоров для такого препарата, равные r(Alexa-488) = 0.06 и r(Alexa-594) = 0.15, свидетельствуют о наличии в растворе свободного ДНК-дуплекса. Полученные результаты показывают, что ковалентная cвязь между ДНК и Cys469 в домене “зажим” белка MutS не препятствует изменению конформации белка и его переходу в “скользящий зажим” при связывании ATP. В дальнейшем важно понять, возможны ли в конъюгате MutS–ДНК другие конформационные перестройки белка, например, изменение положения “мисматч”-связывающего [38, 39] и “коннекторного” доменов [40], а также взаимодействие MutS с белком MutL [40–42].
Рис. 9.
Анализ конформационных перестроек ДНК при образовании “окончательного” узнающего комплекса методами, основанными на измерениях интенсивности и анизотропии флуоресценции. На верхних панелях представлены спектры испускания (в диапазоне 500–800 нм) при возбуждении Alexa-488; нижние панели – диаграммы, демонстрирующие изменение анизотропии флуорофора Alexa-594 в ходе эксперимента; 1 – конъюгат MutS(A469C) с дуплексом (I) и не связанный ковалентно комплекс МutS(A469C) с дуплексом (II); 2 – те же образцы после добавления ATP (1 мМ).

Полученные результаты согласуются с предшествующими данными. В работе Monakhova et al. [9] ДНК содержала пиридилдисульфидную группу в 5-м положении Т, а конъюгат MutS–ДНК выделяли методом анионообменной хроматографии (препарат не содержал примеси белка). Наблюдаемые нами эффекты не противоречат общепринятой модели: в присутствии АТP происходят конформационные изменения в MutS и “выпрямление” ДНК в белково-нуклеиновом комплексе [24].
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Реактивы. N-сукцинимидил-3-(2-пиридилдитио)пропионат, наборы белков-маркеров молекулярной массы PageRuler™ (10–200 кДа), набор ДНК-маркеров GeneRuler™ 1 kb (250–10 000 п.н.), раствор PageBlue™ на основе кумасси G250, АDP, АТP (Thermo Fisher Scientific, США); трео-2,3-дигидрокси-1,4-димеркаптобутан (1,4-дитиотреит, DTT); γ-32P-АТP с удельной радиоактивностью 1000 Кюри/моль и концентрацией 2 мкМ (ИМБ РАН, Россия); МАНТ-ADP – 2′/3′-O-(N-метилантранилоил)аденозин-5′-дифосфат (BIOLOG Life Science Institute, Германия).
Ферменты и белки. Т4 полинуклеотидкиназа (Thermo Fisher Scientific, США). MutS и мутантные варианты получены согласно методикам, опубликованным ранее [16, 35].
Плазмидная ДНК (pUC-MMR), содержащая G/T-пару и участок узнавания MutH, описана и получена согласно методике, опубликованной ранее [27].
Олигодезоксирибонуклеотиды 5′-GTCATCCTCGTAlexa594CTCAGGCACCCTGGGTGCTGAGGGCATAGGCTTG-3′ и 5′-CAAGCCTATAlexa488GCCC-3′ синтезированы компанией Eurogentec (Бельгия). Флуорофоры Alexa-488 и Alexa-594 присоединены по С5-атому тимидина через 2-(6-аминогексилкарбамоил)виниловый линкер. Олигодезоксирибонуклеотиды с включениями 2′-дезокси-2′-аминоуридина получали по стандартному протоколу амидофосфитного олигонуклеотидного синтеза на автоматическом синтезаторе ABI 380B (Applied Biosystems, США) с использованием 5′‑O-диметокситритил-2′-трифторацетамидо-2′-дезокси-3′-(N,N-диизопропиламидо)цианэтилфосфита уридина (ChemGenes, США). Немодифицированные олигодезоксирибонуклеотиды синтезированы ЗАО “Евроген” (Россия).
Синтез олигодезоксирибонуклеотидов с единичным 2′-дисульфид-содержащим уридиновым звеном. Исходными соединениями для получения 2-пиридилдисульфид-содержащих олигонуклеотидов служили синтетические олигонуклеотиды с единичным 2′-амино-2′-дезоксиуридиновым звеном. Модификацию проводили согласно методике, опубликованной ранее [15]. Процент превращения исходного соединения в целевой 2-пиридилдисульфид-содержащий олигонуклеотид оценивали методом ВЭЖХ в ион-парном варианте или методом электрофореза в 20%-ном ПААГ, содержавшем 7 М мочевину.
Конструирование флуоресцентно- и 32P-меченных ДНК-дуплексов. 32Р-метку в олигонуклеотиды вводили с помощью полинуклеотидкиназы Т4 (10 ед. акт.) и [γ-32P]ATP (200 нМ) в 10 мкл буфера с 50 мМ Трис-HCl (pH 7.6), 10 мМ MgCl2, 5 мМ DTT, 100 мкМ спермидина в течение 30 мин при 37°С. Непрореагировавший [γ-32P]ATP удаляли из смеси, используя электрофорез в 20%-ном ПААГ, содержавшем 7 М мочевину, с последующим извлечением олигонуклеотида из геля. Растворы ДНК-дуплексов готовили смешиванием эквимолярных количеств комплементарных олигонуклеотидов. При приготовлении составных ДНК-дуплексов использовали 10%-ный избыток 13- и 15-звенных олигонуклеотидов по отношению к 45-звенной ДНК-матрице. Смесь нагревали до 80°С и медленно охлаждали до комнатной температуры. В раствор флуоресцентно-меченных ДНК-дуплексов добавляли MgCl2 до концентрации 5 мМ для повышения стабильности ДНК-дуплексов. Готовые ДНК-дуплексы хранили при 4°С.
Кинетика обмена ADP в АТРазных доменах белка MutS и его мутантных форм. Обмен ADP на ATP в АТРазных доменах белка MutS изучали с использованием МАНТ-ADP. Кинетику изменения интенсивности флуоресценции регистрировали на спектрофлуориметре Varian Cary Eclipse (США). Измерения проводили при комнатной температуре в кварцевой кювете (Hellma, Германия). Реакционная смесь представляла собой буфер А (25 мМ HEPES (pH 7.5), 125 мМ KCl, 5 мМ MgCl2) с 0.05% (v/v) Tween-20 и 500 нМ МАНТ-ADP, а также содержала (или не содержала) ДНК-дуплекс (VII) (концентрация 500 нМ). Добавляли MutS (или мутантную форму белка) в концентрации 500 нМ (в расчете на мономер). Зависимость интенсивности флуоресценции от времени измеряли при λвозб. = 355 нм и λисп. = 448 нм и напряжении детектора 600 В. После записи начального участка спектра (белок, связанный с МАНТ-ADP) в кювету добавляли ADP до концентрации 1 мМ, быстро перемешивали и продолжали запись спектра до выхода на плато. Суммарный объем смеси составлял 75 мкл. Измерения повторяли не менее трех раз.
Кривые обрабатывали с помощью программы GraphPad Prism® 5 (http://www.graphpad.com). Экспоненциальные участки аппроксимировали согласно уравнению, соответствующему кинетической схеме процесса:
Во всех случаях коэффициент корреляции составлял >0.98.
Активация начальных этапов репарации “мисматчей” в ДНК. Процесс репарации, включая этап гидролиза “дочерней” цепи ДНК, наблюдали in vitro с использованием рекомбинантных белков MutS (дикого типа или описанных в данной работе мутантных форм), MutL и MutH. Для этого раствор pUC-MMR (25 нМ) инкубировали с MutS (400 нМ в расчете на мономер белка), а затем добавляли равный объем смеси белков MutL (400 нМ в расчете на мономер белка) и MutH (100 нМ), предварительно выдержанных с 1 мМ ATP при 0°С. Реакцию проводили в 20 мкл в буфере А при 37°С. В интервале 1–10 мин отбирали аликвоты по 5 мкл, к которым добавляли 5 мкл раствора, содержавшего 250 мМ EDTA, 50% глицерина и 0.1% красителя бромфенолового синего. Реакционные смеси анализировали методом электрофореза в 1%-ном агарозном геле, содержавшем бромид этидия. Фотографии гелей, полученные при облучении УФ-светом, обрабатывали с помощью программы GelAnalyser 2010A (http://www.gelanalyzer.com). Измеряли интенсивность зон, соответствующих ДНК с одноцепочечным разрывом.
Ковалентное связывание белка MutS с реакционноспособными ДНК-лигандами. “Кросслинкинг” мутантных форм MutS с различными модифицированными ДНК-дуплексами (I), (III)–(VI) проводили в буфере А, содержавшем 1 мМ АDP. Концентрация мутантной формы MutS в расчете на мономер составляла 1 мкМ, концентрация ДНК-дуплекса – 5 мкМ. Продукты реакции разделяли в 6%-ном или 8%-ном денатурирующем SDS-ПААГ с 4%-ным концентрирующим слоем. При проведении реакций c 32P-меченными фрагментами ДНК гель после электрофореза высушивали и закладывали в кассету BAS Сassette2 2340 с экраном, чувствительным к β-излучению. Электрофореграммы получали с помощью прибора FLA-3000 (FujiFilm, Япония). Затем гель помещали в воду и окрашивали раствором кумасси G250 для визуализации белка. Выход конъюгата определяли денситометрически с использованием фотографий гелей, окрашенных раствором кумасси G250. Рассчитывали интенсивность зоны геля, соответствующей конъюгату, по отношению к суммарной интенсивности зон, содержащих белок в дорожке. Этот метод не позволяет получить точные данные о количестве вещества в той или иной зоне геля (ошибка ~10%), однако дает возможность проследить общую тенденцию.
Выделение ДНК-белковых конъюгатов методом эксклюзионной хроматографии. Конъюгаты мутантных форм MutS(A469C) и MutS(N497C) с 2-пиридилдисульфид-содержащим ДНК-дуплексом (I) выделяли с использованием системы для ВЭЖХ Elite LaChrom VWR-Hitachi L-2455 (Япония). Перед нанесением на колонку препарат конъюгата центрифугировали 15 мин при 12 000 об./мин. Использовали хроматографическую колонку размерами 10 × 300 мм с сорбентом Superdex200 TM (GE Healthcare Life Sciences, США). Колонку уравновешивали буфером Б (10 мМ HEPES (pH 7.9), 1 мМ EDTA, 200 мМ KCl, 10% (v/v) глицерина), который затем служил элюентом. Объем наносимого на колонку препарата составлял 100 мкл, расход элюента – 0.5 мл/мин. За ходом разделения следили по поглощению при 260, 280, 490 и 590 нм. Рассчитывали значения A260/A280 для элюата. При элюции белка с колонки соотношение A260/A280 составляло 0.7, при элюции конъюгата – 1.4, при элюции ДНК – 1.9. Концентрацию белка во фракциях определяли на приборе NanoDrop ND-1000 (PeqLab, США). Гомогенность конъюгата анализировали в 6%-ном SDS-ПААГ. Гель окрашивали раствором кумасси G250 или раствором солей серебра с использованием набора The PageSilver™ (Thermo Fisher Scientific, США). Раствор очищенного конъюгата хранили при 0°С. Все эксперименты с конъюгатом проводили в течение 4 ч после выделения, т.к. далее активность белка значительно уменьшалась.
Измерение анизотропии и сигнала FRET в конъюгате MutS–ДНК. Раствор конюгата MutS(A469C)–ДНК (50 нМ) в 100 мкл буфера А с 0.05% (v/v) Tween-20 помещали в кварцевую кювету и измеряли сигнал флуоресценции и анизотропию для двух флуорофоров – Alexa-488 (донор) и Alexa-594 (акцептор) – на спектрофлуориметре FluoroMax‑4 (Horiba, Япония) при комнатной температуре. Затем к раствору добавляли ATP до конечной концентрации 1 мМ, инкубировали 5 мин при комнатной температуре и проводили измерения повторно. В контрольном эксперименте измеряли интенсивность флуоресценции не зафиксированного ковалентно комплекса MutS–ДНК. Концентрации белка и ДНК в этом случае составляли 20 и 200 нМ соответственно. Флуорофоры возбуждали при длинах волн 470 и 575 нм (Alexa-488 и Alexa-594 соответственно). При записи спектров флуоресценции излучение детектировали в диапазоне длин волн 490–800 или 595–800 нм для донора и акцептора соответственно. Максимальное значение интенсивности флуоресценции измеряли при 535 нм для Alexa-488 и 620 нм для Alexa-594. Для измерения FRET препарат облучали светом с длиной волны 470 нм, детектировали испускание флуоресценции в диапазоне 500–800 нм (при 525 нм наблюдался максимум испускания Alexa-488, при 625 нм – максимум испускания Alexa-594, Fmax). Также образец возбуждали при 575 нм и детектировали испускание флуоресценции Alexa-594 в диапазоне 600–800 нм (для вычисления значения Red, интенсивности испускания Alexa-594 при 625 нм). В ходе экспериментов вычисляли Δ(Fmax/Red).
Расстояние от аминокислоты до ДНК (от первого углеродного атома боковой цепи аминокислотного остатка (CB) до атома С2′ ближайшего нуклеотида в ДНК) в комплексах MutS–ДНК (PDB: 1W7A, 10H7 и 1E3М) определяли в программе Jmol [43] с помощью команды “measure”.
ЗАКЛЮЧЕНИЕ
Таким образом, на примере двух мутантных форм MutS нами продемонстрировано, что ДНК-фрагменты с 2-пиридилдитиогруппой при С2′‑атоме могут быть использованы для фиксации белка MutS через остаток цистеина, сближенный с ДНК. Высокого выхода ДНК-белкового конъюгата с последующим его выделением в количествах, необходимых для дальнейшего анализа активности MutS, удалось достигнуть благодаря следующим факторам: 1) химической природе реакционноспособной группировки, 2) правильно подобранному месту модификации в ДНК, 3) дополнительной детекции белково-нуклеинового конъюгата в ходе эксклюзионной хроматографии за счет наличия флуорофоров в ДНК. Установлено, что MutS в составе конъюгата сохраняет способность изменять свою конформацию и конформацию ДНК. Таким образом, предложенный нами дизайн конъюгатов с функционально активным белковым компонентом может быть использован для изучения последовательности и характера перестроек в белково-нуклеиновых комплексах.
Список литературы
Dolinnaya N., Zubin E., Kubareva E., Zatsepin T., Oretskaya T. // Curr. Org. Chem. 2009. V. 13. P. 1029–1049. https://doi.org/10.2174/138527209788680745
Ilina E.S., Khodyreva S.N., Lavrik O.I. // Biochimie. 2018. V. 150. P. 88–99. https://doi.org/10.1016/j.biochi.2018.04.027
Ide H., Nakano T., Salem A.M.H., Shoulkamy M.I. // DNA Repair. 2018. V. 71. P. 190–197. https://doi.org/10.1016/j.dnarep.2018.08.024
Stasińska A.R., Putaj P., Chmielewski M.K. // Bioorg. Chem. 2020. V. 95. P. 103 518.https://doi.org/10.1016/j.bioorg.2019.103518
Wong S.S., Jameson D.M. // Chemistry of Protein and Nucleic Acid Cross-Linking and Conjugation, Second Edition. CRC Press, Boca Raton, 2011. 622 pp.
Khodyreva S., Lavrik O. // DNA Repair (Amst). 2020. V. 90. P. 102 847.https://doi.org/10.1016/j.dnarep.2020.102847
Hyjek-Składanowska M., Stasińska A.R., Napiórkowska-Gromadzka A., Bartłomiejczak A., Seth P.P., Chmielewski M.K., Nowotny M. // Bioorganic Med. Chem. 2020.https://doi.org/10.1016/j.bmc.2020.115741
Friedhoff P., Manelyte L., Giron-Monzon L., Winkler I., Groothuizen F.S., Sixma T.K. // Methods in Enzymology. 2017. V. 592. P. 77–101. https://doi.org/10.1016/bs.mie.2017.03.025
Monakhova M., Ryazanova A., Hentschel A., Viryasov M., Oretskaya T., Friedhoff P., Kubareva E. // J. Chromatogr. A. 2015. V. 1389. P. 19–27. https://doi.org/10.1016/j.chroma.2015.02.045
Monakhova M., Ryazanova A., Kunetsky V., Li P., Shilkin E., Kisil O., Rao D.N., Oretskaya T., Friedhoff P., Kubareva E. // Biochimie. 2020. V. 171–172. P. 43–54. https://doi.org/10.1016/j.biochi.2020.02.004
Metelev V.G., Kubareva E.A., Vorob’eva O. V., Romanenkov A.S., Oretskaya T.S. // FEBS Lett. 2003. V. 538. P. 48–52. https://doi.org/10.1016/S0014-5793(03)00122-4
Воробьева О.В., Романенков А.С., Метелев В.Г., Карягина А.С., Лаврова Н.В., Орецкая Т.С., Кубарева Е.А. // Молекуляр. биология. 2003. Т. 37. С. 906–915. [Vorob’eva O.V., Romanenkov A.S., Metelev V.G., Kariagina A.S., Lavrova N.V., Oretskaia T.S., Kubareva E.A. // Mol. Biol. (Mosk.). 2003. V. 37. P. 906–915.]
Романенков А.С., Кисиль О.В., Зацепин Т.С., Ямскова О.В., Карягина А.С., Метелев В.Г., Орецкая Т.С., Кубарева Е.А. // Биохимия. 2006. Т. 1. С. 1648–1658. [Romanenkov A.S., Kisil O.V., Zatsepin T.S., Yamskova O.V., Karyagina A.S., Metelev V.G., Oret-skaya T.S., Kubareva E.A. // Biochemistry (Mosc.). 2006. V. 71. P. 1341–1349.] https://doi.org/10.1134/s0006297906120091
Абросимова Л.А., Самсонова А.А., Перевязова Т.А., Юнусова А.К., Артюх Р.И., Романова Е.А., Железная Л.А., Орецкая Т.С., Кубарева Е.А. // Молекуляр. биология. 2020. Т. 54. С. 599–610. [Abrosimova L.A., Samsonova A.R., Perevyazova T.A., Yunusova A.K., Artyukh R.I., Romanova E.A., Zheleznaya L.A., Oret-skaya T.S., Kubareva E.A. // Mol. Biol. (Mosk.). 2020.V. 54. P. 599–610.] https://doi.org/10.1134/S0026893320040020
Metelev V., Romanenkov A., Kubareva E., Zubin E., Polouchine N., Zatsepin T., Molochkov N., Oretskaya T. // IUBMB Life. 2006. V. 58. P. 654–658. https://doi.org/10.1080/15216540600981750
Heinze R.J., Sekerina S., Winkler I., Biertümpfel C., Oretskaya T.S., Kubareva E., Friedhoff P. // Mol. Biosyst. 2012. V. 8. P. 1861–1864. https://doi.org/10.1039/c2mb25086a
Cristóvão M., Sisamakis E., Hingorani M.M., Marx A.D., Jung C.P., Rothwell P.J., Seidel C.A.M., Friedhoff P. // Nucleic Acids Res. 2012. V. 40. P. 5448–5464. https://doi.org/10.1093/nar/gks138
Warren J.J., Pohlhaus T.J., Changela A., Iyer R.R., Modrich P.L., Beese L.S. // Mol. Cell. 2007. V. 26. P. 579–592. https://doi.org/10.1016/j.molcel.2007.04.018
Biswas I., Hsieh P. // J. Biol. Chem. 1997. V. 272. P. 13 355–13 364. https://doi.org/10.1074/jbc.272.20.13355
Natrajan G. // Nucleic Acids Res. 2003. V. 31. P. 4814–4821. https://doi.org/10.1093/nar/gkg677
Перевозчикова С.А., Романова Е.А., Орецкая Т.С., Фридхофф П., Кубарева Е.А. // Acta Naturae. 2013. Т. 5. С. 18–35. [Perevoztchikova S.A., Romanova E.A., Oretskaya T.S., Friedhoff P., Kubareva E.A. // Acta Naturae. 2013. V. 5. P. 17–34.] https://doi.org/10.32607/20758251-2013-5-3-17-34
Putnam C.D. // Proc. Natl. Acad. Sci. 2020. V. 117. P. 20 351–20 353. https://doi.org/10.1073/pnas.2013560117
Bhairosing-Kok D., Groothuizen F.S., Fish A., Dharadhar S., Winterwerp H.H.K., Sixma T.K. // Nucleic Acids Res. 2019. V. 47. P. 8888–8898. https://doi.org/10.1093/nar/gkz649
Liu J., Hanne J., Britton B.M., Bennett J., Kim D., Lee J.-B., Fishel R. // Nature. 2016. V. 539. P. 583–587. https://doi.org/10.1038/nature20562
Perevozchikova S.A., Trikin R.M., Heinze R.J., Romanova E.A., Oretskaya T.S., Friedhoff P., Kubareva E.A. // PLoS One. 2014. V. 9. P. e104 963. https://doi.org/10.1371/journal.pone.0104963
Hura G.L., Tsai C.-L., Claridge S.A., Mendillo M.L., Smith J.M., Williams G.J., Mastroianni A.J., Alivisatos A.P., Putnam C.D., Kolodner R.D., Tainer J.A. // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 17 308–17 313. https://doi.org/10.1073/pnas.1308595110
Heinze R.J., Giron-Monzon L., Solovyova A., Elliot S.L., Geisler S., Cupples C.G., Connolly B.A., Friedhoff P. // Nucleic Acids Res. 2009. V. 37. P. 4453–4463. https://doi.org/10.1093/nar/gkp380
Kramer B., Kramer W., Fritz H.-J. // Cell. 1984. V. 38. P. 879–887. https://doi.org/10.1016/0092-8674(84)90283-6
Lamers M.H., Georgijevic D., Lebbink J.H., Winter-werp H.H.K., Agianian B., de Wind N., Sixma T.K. // J. Biol. Chem. 2004. V. 279. P. 43879–43885. https://doi.org/10.1074/jbc.M406380200
Lebbink J.H.G., Georgijevic D., Natrajan G., Fish A., Winterwerp H.H.K., Sixma T.K., de Wind N. // EMBO J. 2006. V. 25. P. 409–419. https://doi.org/10.1038/sj.emboj.7600936
Lamers M.H., Perrakis A., Enzlin J.H., Winter-werp H.H.K., de Wind N., Sixma T.K. // Nature. 2000. V. 407. P. 711–717. https://doi.org/10.1038/35037523
Groothuizen F.S., Fish A., Petoukhov M.V., Reumer A., Manelyte L., Winterwerp H.H.K., Marinus M.G., Lebbink J.H.G., Svergun D.I., Friedhoff P., Sixma T.K. // Nucleic Acids Res. 2013. V. 41. P. 8166–8181. https://doi.org/10.1093/nar/gkt582
Bjornson K.P., Modrich P. // J. Biol. Chem. 2003. V. 278. P. 18557–18562. https://doi.org/10.1074/jbc.M301101200
Manelyte L., Urbanke C., Giron-Monzon L., Friedhoff P. // Nucleic Acids Res. 2006. V. 34. P. 5270–5279. https://doi.org/10.1093/nar/gkl489
Feng G., Winkler M.E. // Biotechniques. 1995. V. 19. P. 956–965.
Lebbink J.H.G., Fish A., Reumer A., Natrajan G., Winterwerp H.H.K., Sixma T.K. // J. Biol. Chem. 2010. V. 285. P. 13131–13141. https://doi.org/10.1074/jbc.M109.066001
Ede N.J., Tregear G.W., Haralambidis J. // Bioconjug. Chem. 1994. V. 5. P. 373–378. https://doi.org/10.1021/bc00028a016
Qiu R., Sakato M., Sacho E.J., Wilkins H., Zhang X., Modrich P., Hingorani M.M., Erie D.A., Weninger K.R. // Proc. Natl. Acad. Sci. USA. 2015. V. 112. P. 10 914–10 919. https://doi.org/10.1073/pnas.1505655112
LeBlanc S.J., Gauer J.W., Hao P., Case B.C., Hingorani M.M., Weninger K.R., Erie D.A. // Nucleic Acids Res. 2018. V. 46. P. 10 782–10 795. https://doi.org/10.1093/nar/gky865
Groothuizen F.S., Winkler I., Cristóvão M., Fish A., Winterwerp H.H., Reumer A., Marx A.D., Hermans N., Nicholls R.A., Murshudov G.N., Lebbink J.H., Friedhoff P., Sixma T.K. // Elife. 2015. V. 4. https://doi.org/10.7554/eLife.06744
Hao P., LeBlanc S.J., Case B.C., Elston T.C., Hingorani M.M., Erie D.A., Weninger K.R. // Proc. Natl. Acad. Sci. USA. 2020. V. 117. P. 17 775–17 784. https://doi.org/10.1073/pnas.1918517117
Liu J., Lee R., Britton B.M., London J.A., Yang K., Hanne J., Lee J.-B., Fishel R. // Nat. Commun. 2019. V. 10. P. 5294. https://doi.org/10.1038/s41467-019-13191-5
Herráez A. // Biochem. Mol. Biol. Educ. 2006. V. 34. P. 255–261. https://doi.org/10.1002/bmb.2006.494034042644
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия



