Биоорганическая химия, 2022, T. 48, № 6, стр. 677-693
Петлевая изотермическая амплификация: от теории к практике
Ф. В. Ширшиков 1, *, Ю. А. Беспятых 1
1 ФГБУ “Федеральный научно-клинический центр физико-химической медицины” ФМБА России
119435 Москва, ул. Малая Пироговская, 1А, Россия
* E-mail: shrshkv@ya.ru
Поступила в редакцию 18.05.2022
После доработки 01.06.2022
Принята к публикации 17.06.2022
- EDN: AJNHCM
- DOI: 10.31857/S0132342322060227
Аннотация
Вопросы повышения точности видовой идентификации патогенов и сокращения продолжительности анализа и по сей день остаются актуальными для современной молекулярной диагностики. В лабораторной и клинической практике для обнаружения патогенов наиболее востребованы методы амплификации нуклеиновых кислот, “золотым стандартом” среди которых считается полимеразная цепная реакция (ПЦР). Тем не менее в ряде случаев альтернативой классической ПЦР-диагностике выступает группа методов изотермической амплификации. За более чем 30-летнюю историю развития способов синтеза ДНК при постоянной температуре появление метода петлевой изотермической амплификации (LAMP) сделало возможным развитие новых направлений в области мобильной диагностики бактериальных и вирусных инфекций. В настоящем обзоре рассмотрены ключевые характеристики данного метода и особенности практической работы с ним. Впервые обсуждается пространственное строение ампликонов реакции LAMP с одноцепочечными петлевыми структурами, благодаря которым становится возможным отжиг праймеров при изотермических условиях. Проанализированы последние достижения по модификации метода LAMP, позволяющие рассматривать его в качестве уникальной платформы для разработки способов молекулярной диагностики нового поколения.
СОДЕРЖАНИЕ
ВВЕДЕНИЕ........................................................677
ПРИНЦИП МЕТОДА LAMP...........................678
ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ......680
ФЕРМЕНТЫ ДЛЯ МЕТОДА LAMP................682
ИНЖЕНЕРИЯ НОВЫХ ФЕРМЕНТОВ..........683
ОПТИМИЗАЦИЯ АМПЛИФИКАЦИИ.........684
СПОСОБЫ ДЕТЕКЦИИ.................................686
КОЛИЧЕСТВЕННЫЙ АНАЛИЗ.....................687
ЗАКЛЮЧЕНИЕ.................................................688
СПИСОК ЛИТЕРАТУРЫ................................689
ВВЕДЕНИЕ
Методы амплификации нуклеиновых кислот нашли широкое применение в различных областях молекулярной биологии, стали незаменимым фундаментом для генной инженерии и ценным источником информации о причинах инфекционных и наследственных заболеваний. Метод полимеразной цепной реакции (ПЦР) по-прежнему крайне востребован при решении широкого спектра научно-исследовательских задач. Несмотря на важную роль этого метода в области здравоохранения, при создании способов мобильной диагностики привлекательной альтернативой ПЦР становится группа разнообразных методов, основанных на изотермической амплификации [1, 2].
При изотермической амплификации, в отличие от ПЦР, нет необходимости в температурных циклах для денатурации нуклеиновой кислоты, отжига и элонгации олигонуклеотидных праймеров, поскольку все эти этапы протекают последовательно и без разделения каждого процесса во времени. Возможность осуществления амплификации при изотермических условиях в значительной степени зависит от структуры и количества праймеров, использования в реакционной смеси особых ферментов и других вспомогательных белков, а также от тщательного подбора остальных компонентов реакции [3]. Следует отметить, что принцип некоторых вариантов изотермической амплификации, не без существенных модификаций, был заимствован из различных механизмов репликации вирусных геномов. Так, исторически первым методом изотермической амплификации стал метод 3SR (от англ. self-sustained sequence replication), появившийся благодаря исследованиям по моделированию ретровирусной репликации в условиях in vitro [4]. Амплификация по принципу “катящегося кольца” (от англ. rolling circle amplification, RCA) [5] основана на механизме репликации вироидов и некоторых вирусов, обладающих кольцевым геномом [6–8]. Петлевая изотермическая амплификация (от англ. loop-mediated isothermal amplification, LAMP) имеет много структурных гомологий с механизмом репликации генома поксвирусов [9, 10]. В этой связи совершенно очевидна важность фундаментальных исследований прокариотических и вирусных ферментов репликации, поскольку обнаружение новых термостабильных, точных и процессивных обратных транскриптаз и ДНК-полимераз способно вывести технологию LAMP на совершенно иной уровень практического применения, как это произошло когда-то в области ПЦР с открытием ДНК-полимеразы I из Thermus aquaticus, или Taq-полимеразы [11].
Метод LAMP оказался наиболее популярным способом изотермической амплификации по целому ряду причин: в силу малого количества компонентов реакционной смеси, их доступности для рутинной практики, а также благодаря высокой специфичности реакции, которая обеспечивается 4–6 праймерами [12, 13]. Простота реакции амплификации позволила осуществить проведение LAMP в максимально ограниченных условиях на борту Международной космической станции [14]. Несмотря на то что у технологии LAMP пока остаются недостатки, связанные с применяемыми на сегодняшний день ферментами, метод обладает некоторыми преимуществами перед ПЦР, особенно ценными для клинической практики. Технология LAMP нашла множество применений в “полевой” диагностике, когда необходимо проводить амплификацию вне лабораторных условий [15–19].
В настоящем обзоре рассмотрены возможности метода LAMP и некоторые его особенности, которые необходимо учитывать при разработке новых тест-систем. Многие упомянутые в обзоре методики апробированы авторами при разработке тест-систем на основе LAMP, а соответствующие теоретические и практические аспекты метода проиллюстрированы на примере конкретного набора праймеров (табл. 1) [20]. Впервые демонстрируются результаты молекулярного моделирования пространственного строения гантелеобразных стартовых структур, образующихся в ходе реакции LAMP. Детально описаны процедуры подбора генов-мишеней и дизайна праймеров, отмечена важность такого этапа, как предсказание нежелательных вторичных структур в нуклеотидных последовательностях мишени и ампликона. Рассмотрены основные этапы оптимизации состава реакционной смеси и условий проведения амплификации, приведены основные характеристики ферментов, используемых в методе LAMP, описаны способы детекции положительного сигнала амплификации и увеличения специфичности реакции.
Таблица 1.
Нуклеотидные последовательности праймеров набора DAT [20], разработанные с помощью программ MorphoCatcher и PrimerExplorer
| Праймер | Нуклеотидная последовательность, 5′→3′ |
|---|---|
| FIP | TCTTTTTCCCAACGTCCGGCGTCGTATCGCAGAAGAGG |
| BIP | CACCGAGGACGCTGATTACCTTCTCATCTTCCGCTTCAC |
| F3 | CGTCGTAAGGTCGAGGAA |
| B3 | CCTTCCACCTGACGATCA |
| LF | ATTTTCTTCTGCCATCTGG |
| LB | ACGTAACCACCTCTCATCAC |
ПРИНЦИП МЕТОДА LAMP
Метод LAMP был разработан и запатентован группой японских исследователей из компании Eiken Chemical в 1998 г. [21]. Одной из главных целей создания нового метода амплификации было повышение специфичности детекции нуклеотидных полиморфизмов по сравнению с возможностями ПЦР. В настоящее время патент на технологию LAMP утратил свою охранную силу, чем отчасти и объясняется повышенный интерес к данному способу амплификации.
Механизм реакции LAMP со множеством промежуточных вторичных структур ДНК обеспечивается необычной структурой праймеров и напоминает японское искусство оригами [9]. Для амплификации методом LAMP необходимо найти в гене-мишени шесть сайтов отжига для двух пар праймеров, которые при средней длине одного сайта отжига в 20 нт покрывают ~120 п.н. гена-мишени. Это минимум в 3 раза увеличивает потенциальную аналитическую специфичность метода LAMP, если сравнить его с ПЦР на основе прямого и обратного праймеров.
Метод LAMP основан на особой структуре внутренних праймеров (от англ. forward inner primer, FIP и backward inner primer, BIP), участвующих в формировании двух терминальных инвертированных повторов в нуклеотидной последовательности ампликонов. Вторая пара внешних праймеров (F3 и B3) участвует только на первых стадиях амплификации, поэтому их концентрация существенно ниже, чем у внутренних праймеров [10]. Благодаря последовательному отжигу внутренних и внешних праймеров, ампликон на начальных стадиях реакции LAMP формирует два типа гантелеобразных стартовых структур (рис. 1), имеющих по две одноцепочечные петли и одному сайту отжига для комплементарного внутреннего праймера. С целью ускорения амплификации разработчики метода предложили использовать третью пару петлевых праймеров (от англ. loop forward, LF и loop backward, LB), сайты отжига которых тоже расположены в петлевых участках стартовых структур между сайтами F1/B1 и F2/B2 [22]. Следует, однако, учитывать, что вероятность обнаружения сайтов отжига петлевых праймеров напрямую зависит от длины соответствующих петель стартовых структур. Для некоторых наборов “ко́ровых” праймеров (так называют совокупность внутренних и внешних праймеров [23]) можно сконструировать только один петлевой праймер, а для других ни одного. Именно по этой причине петлевыми праймерами удается дополнить не каждую тест-систему.
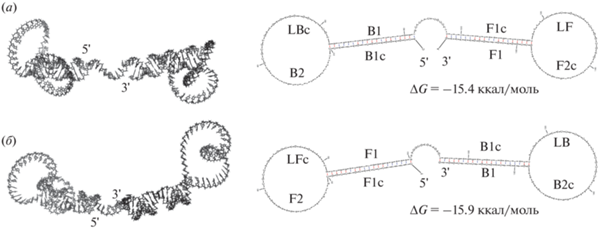
Рис. 1.
Молекулярные модели пространственного строения стартовых структур, образующихся в реакции LAMP на основе набора праймеров DAT (табл. 1). Моделирование структуры ДНК проводили с помощью программ mFold и RNAComposer при температуре 65°C, концентрации ионов натрия – 50 мМ, ионов магния – 8 мМ. Приведены третичная (слева) и вторичная (справа) структуры ампликонов, имеющих комплементарные нуклеотидные последовательности. На вторичной структуре ампликонов показаны все сайты отжига для внутренних и петлевых праймеров, а также их комплементарные участки. Длина нуклеотидной последовательности составляет 176 нт, GC-состав: 55%. (а) – Стартовая структура F, получившая название благодаря наличию сайта отжига (F2c) для внутреннего праймера FIP; (б) – стартовая структура B с сайтом отжига (B2c) для внутреннего праймера BIP.
Процесс амплификации на основе метода LAMP происходит в изотермических условиях (60–65°C) и обычно занимает не более 15–30 мин, а при высокой концентрации целевой нуклеиновой кислоты выход реакции на плато возможен за 5–7 мин. Вместо термической денатурации молекулы ДНК в реакции LAMP используется цепь-вытесняющая активность большого фрагмента Bst-ДНК-полимеразы из Geobacillus stearothermophilus (Bst LF), при этом непрерывный во времени отжиг внутренних и петлевых праймеров обусловлен одноцепочечным состоянием соответствующих сайтов отжига и их постоянной стерической доступностью для праймеров. При температуре 65°C, оптимальной для фермента Bst LF, двойная спираль ДНК становится локально денатурированной благодаря эффекту “дыхания” цепей [24, 25], облегчающему процесс отжига праймеров на начальных стадиях. Цепь-вытесняющая активность Bst LF позволяет высвобождать ранее синтезированную цепочку ДНК, которая гибридизуется сама на себя с помощью комплементарных участков и образует новую точку инициации синтеза ДНК. Образующиеся стартовые структуры после отжига соответствующего внутреннего праймера могут многократно служить матрицей для синтеза комплементарной версии самих себя без необходимости в исходной нуклеиновой кислоте, добавляемой к реакционной смеси. Следовательно, метод LAMP представляет собой цепную биохимическую реакцию, количество ампликонов в которой увеличивается экспоненциально.
Быстрое накопление ДНК-продуктов в методе LAMP обеспечивается в основном нуклеотидными последовательностями внутренних и петлевых праймеров. При визуализации в агарозном геле совокупность ампликонов разной длины формирует характерную картину в виде регулярной “лесенки”, плавно переходящей в “шмер” высокомолекулярных продуктов амплификации (рис. 2). В ходе реакции LAMP на 25 мкл реакционной смеси образуется до 10 мкг ДНК, что примерно на два порядка превосходит суммарный выход ПЦР [26]. Поэтому при работе с содержимым пробирок после реакции LAMP следует помнить о возможной контаминации лаборатории и реагентов.
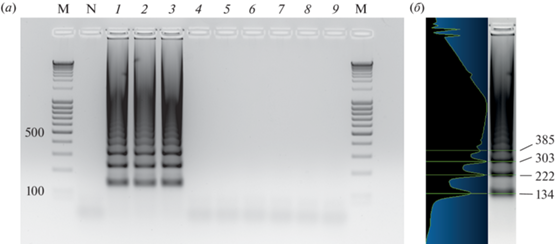
Рис. 2.
Визуализация продуктов реакции LAMP на основе праймеров DAT с помощью агарозного гель-электрофореза [20]. (а) – Характерная “лесенка” продуктов реакции амплификации с периодичностью молекулярных масс (п.н.). M – маркер молекулярных масс ДНК (MassRuler DNA Ladder Mix, ThermoFisher Scientific, Литва); N – отрицательный контроль без добавления ДНК-матрицы; 1–3 – реакция на матрице геномной ДНК разных штаммов бактерий Dickeya solani; 4–9 – реакции с добавлением в качестве матрицы геномной ДНК представителей Pectobacterium spp., встречающихся на картофеле вместе с D. solani; (б) – профиль локализации полос ДНК и рассчитанные с помощью программы Vision-Capt (Vilber Lourmat, Франция) молекулярные массы продуктов амплификации (п.н.).
Интересная особенность реакции LAMP заключается в необязательности включения в состав ампликона нуклеотидной последовательности, фланкированной праймерами. Если в ПЦР прямой и обратный праймеры всегда фланкируют участок выбранного для амплификации гена, то в случае LAMP между сайтами F1 и B1 может не оставаться ни одного нуклеотида. При такой конфигурации внутренних праймеров фрагменты исходного гена-мишени сохраняются только в сайтах отжига петлевых праймеров (участки между F1 и F2, а также между B1 и B2). В этой связи следует подчеркнуть, что метод LAMP малопригоден для генной инженерии и в значительной степени адаптирован для диагностики.
Поскольку в составе ампликонов разной длины многократно присутствует одна и та же нуклеотидная последовательность [27, 28], то в качестве характеристики специфичности реакции можно использовать анализ кривых плавления, отслеживая однажды измеренную и воспроизводимую для конкретного набора праймеров температуру плавления (Тпл) получаемого продукта амплификации. Наличие в ампликоне хотя бы одного сайта рестрикции вне последовательностей праймеров позволяет получить в агарозном геле всего одну полосу с характерной молекулярной массой [9] и тем самым дает возможность оценить специфичность реакции. Любые отклонения в количестве пиков плавления продуктов реакции LAMP, в получаемом значении Тпл, в регулярности “лесенки” ампликонов в агарозном геле и количестве полос после обработки амплификата рестриктазами свидетельствуют о наличии в реакционной смеси фоновой неспецифической амплификации, причины которой будут рассмотрены далее.
ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ
В методах амплификации определяющим фактором высокой специфичности и чувствительности детекции является дизайн олигонуклеотидных праймеров. Метод LAMP, основанный на синхронной работе 4–6 праймеров, требует особого внимания к их последовательностям. Если рассматривать LAMP как мультиплексную реакцию из нескольких пар праймеров, то становится очевидным влияние на эффективность реакции различных термодинамических характеристик праймеров, таких как стабильность гибридизации растущих 3′-концов и предрасположенность праймеров к димеризации. В условиях постоянной ферментативной активности Bst LF димеризация 3′-концов праймеров способствует их элонгации и, следовательно, появлению в праймерах новой нуклеотидной последовательности. Элонгированные праймеры могут непредсказуемо влиять на эффективность и специфичность реакции. Поэтому при обнаружении признаков неспецифической фоновой амплификации в некоторых случаях помогает сдвиг сайтов отжига проблемных праймеров на несколько нуклеотидов для сокращения или устранения комплементарности в предполагаемых димерах [29]. Экспериментально показана применимость такого подхода для сокращения фоновой амплификации, однако при оценке потенциальных димеров следует помнить о существовании комплементарности не только между парами A·T и G·C, которые обычно предсказываются in silico, но и о термодинамическом вкладе в гибридизацию праймеров неканонических пар нуклеотидов [30].
Стандартная конфигурация праймеров для реакции LAMP, состоящая из коровых и петлевых праймеров, со временем была пересмотрена, и появились новые типы праймеров:
1) “стволовые” праймеры (от англ. stem forward, StF и stem backward, StB), сайты отжига которых локализованы в промежутке между сайтами F1 и B1 [31]. Данный тип праймеров, как и в случае с петлевыми праймерами, сильно зависит от длины нуклеотидной последовательности, фланкируемой сайтами F1 и B1;
2) “роевые” (от англ. swarm) праймеры F1S/B1S, которые отжигаются на сайты F1c/B1c [32]. Отметим, что идентичные сайты отжига были использованы позднее другими авторами для праймеров под названиями FC/RC [33]. В данной работе было показано, что сокращение расстояния между сайтами отжига F2/B2 и F1/B1 позволяет значительно снизить неспецифичную фоновую амплификацию и повысить аналитическую чувствительность. Преимущество “роевых” праймеров заключается в том, что их сайты отжига не требуется искать отдельно, т.к. они совпадают с сайтами отжига внутренних праймеров.
Как мы видим, модификация архитектуры праймеров и физического расстояния между их сайтами отжига являются дополнительными факторами в оптимизации специфичности или чувствительности метода. При этом каждая дополнительная пара праймеров увеличивает мультиплексность реакции и требует предварительной проверки in silico на совместимость с другими олигонуклеотидами для исключения возможных димеров.
Эволюция метода LAMP в отношении состава праймеров, по всей видимости, происходит не только по пути создания новых типов праймеров. В недавней работе авторам удалось провести амплификацию только на основе внутренних праймеров, не добавляя внешние праймеры, при этом в остальном состав реакционной смеси остался неизменным [34].
Выбранная мишень и ампликон, находясь на некоторых стадиях реакции в одноцепочечном состоянии, не должны содержать нежелательных вторичных структур в сайтах отжига праймеров, снижающих эффективность амплификации. Основной вклад в образование вторичных структур вносит неравномерное распределение GC-состава и, следовательно, локальное повышение Тпл в таких участках ДНК. Предсказание вторичной и третичной структур одноцепочечных нуклеиновых кислот (как РНК, так и ДНК) можно проводить с приближением к оптимальным для Bst LF значениям температуры и концентрациям ионов магния с помощью веб-сервисов mFold (http://www.unafold.org) [35] и RNAComposer (http://rnacomposer.ibch.poznan.pl) [36]. Таким образом, следует тщательно проверять каждый потенциальный набор праймеров и его физико-химические свойства с помощью методов биоинформатики.
Трудности в дизайне и совместимости разных наборов праймеров являются, пожалуй, самым главным недостатком метода LAMP, усложняющий процесс разработки мультиплексных тест-систем [37–40]. Учитывая эти особенности, следует отметить, что на сегодняшний день оптимальным вариантом для LAMP-диагностики является тест-система, состоящая из отрицательного контроля без матрицы, положительного контроля на основе плазмидной ДНК или армированной РНК в составе фаговой частицы и, наконец, оптимизированной реакционной смеси для исследуемого образца.
Многие критерии скрининга мишеней в геноме и подбора праймеров для LAMP удалось формализовать на языках программирования, что привело к созданию новых высокоэффективных инструментов для решения различных задач молекулярной диагностики. Для упрощения процедуры дизайна LAMP-праймеров создано несколько удобных веб-сервисов, позволяющих проводить предварительный анализ потенциальных генов-мишеней, а затем использовать полученную информацию при конструировании праймеров с необходимыми параметрами. Среди таких сервисов получили особую популярность PrimerExplorer (http://primerexplorer.jp/e) [10] японской компании Eiken Chemical, а также LAMP Primer Design Tool (https://lamp.neb.com) [13] американской компании New England Biolabs (NEB). Эти программы позволяют получить кандидатные наборы праймеров по интересующей нуклеотидной последовательности, имеют опции сортировки праймеров по GC-составу и другим физико-химическим параметрам (например, по термодинамической стабильности 3′-концов или риску димеризации). Несмотря на сходство этих веб-сервисов, PrimerExplorer обладает некоторыми преимуществами. Например, предоставляет ряд уникальных возможностей для дизайна праймеров с различной степенью специфичности к гомологичным нуклеотидным последовательностям. Сервис позволяет вручную выбрать те нуклеотиды, которые при расположении на концах праймеров повысят их специфичность в отношении конкретного таксона. Если же такие нуклеотидные позиции окажутся в середине последовательности праймера, это приведет к повышению толерантности тест-системы, т.е. отжиг праймеров будет происходить с большей эффективностью, несмотря на различия в нуклеотидных последовательностях близкородственных генов-мишеней.
В научной литературе часто встречаются методики дизайна праймеров, когда после выбора гена-мишени анализируются гомологичные гены во всех штаммах целевого патогена, а далее проводится множественное выравнивание с целью определения консервативных областей без полиморфизмов [41, 42]. При таком подходе, однако, некоторые участки множественного выравнивания, идентифицируемые как консервативные, на самом деле могут и не содержать видоспецифичных нуклеотидов. Таким образом, в процессе дизайна праймеров необходимо включать в множественное выравнивание последовательности близкородственных ортологичных генов, чтобы дополнить консервативные участки праймеров только видоспецифичными нуклеотидами, которых нет в генах-ортологах.
Для устранения необходимости картировать множественное выравнивание вручную нами была разработана программа [43] и онлайн-сервис MorphoCatcher (http://morphocatcher.ru) [44]. Данный сервис позволяет извлечь из множественного выравнивания информацию о локализации таксон-специфичных нуклеотидов и выбрать наиболее полиморфный в сравнении с ортологами участок гена-мишени. Производительность сервиса ограничена только алгоритмами множественного выравнивания, поэтому он полезен на этапе скрининга как отдельных генов-ортологов, так и при анализе множественного выравнивания полных вирусных геномов.
В последнее время намечается тенденция совмещения инструментов для дизайна и скрининга праймеров с международными базами геномных данных вирусов человека в рамках глобальной инициативы GISAID (https://www.gisaid.org) [45], что привело к появлению высокопроизводительных аналитических сервисов, таких как COVID-19 CG (https://covidcg.org) [46] и Primer Monitor Tool (https://primer-monitor.neb.com) [47]. Со временем алгоритмы дизайна праймеров для LAMP будут становиться более удобными для пользователей, учитывать множество термодинамических параметров олигонуклеотидов, их вторичную и третичную структуру, а также осуществлять поиск подходящих для диагностики мишеней на основе актуальных данных со всего мира.
ФЕРМЕНТЫ ДЛЯ МЕТОДА LAMP
Исторически первым и наиболее часто используемым ферментом в технологии LAMP стал большой фрагмент ДНК-полимеразы I из термофильной бактерии Geobacillus stearothermophilus (ранее вид относили к роду Bacillus [48, 49], что теперь отражено в названии фермента). Фермент Bst LF обладает 5′→3′ полимеразной, цепь-вытесняющей и ревертазной активностью, но не имеет 5′→3′ экзонуклеазной активности [50, 51]. Со временем были разработаны гомологи Bst LF с точечными изменениями аминокислотной последовательности, повысившими процессивность, термостабильность и ревертазную активность фермента при температуре >65°C [13, 52].
Использование технологии олигонуклеотидных аптамеров [53] позволяет обратимо ингибировать проявление полимеразной активности Bst-полимеразы при комнатной температуре, что повышает специфичность реакций на основе фермента Bst 2.0 WarmStart (NEB, США) [13, 23]. Таким образом, в настоящее время существуют гомологи Bst-полимеразы, совместимые с форматом амплификации по принципу “горячего” старта, широко используемым в современных приложениях ПЦР. Стоит отметить, что если Taq-полимераза обратимо инактивируется с помощью TP7-антитела [54], диссоциирующего при первой стадии денатурации ДНК, то применяемые для ингибирования Bst-полимеразы специфические олигонуклеотидные аптамеры диссоциируют от фермента при температуре >45°C [13].
Дальнейшие исследования показали, что ДНК-полимеразы из других прокариот и вирусов также применимы в методе LAMP. Примерами могут служить термостабильная ДНК-полимераза OmniAmp с ревертазной активностью из бактериофага PyroPhage 3173 горячих источников Йеллоустонского национального парка [55, 56], а также большой фрагмент ДНК-полимеразы I из почвенной бактерии Ureibacillus thermosphaericus [57].
При использовании в качестве матрицы молекул РНК, как и в случае с методом ПЦР, необходимо включать в протокол амплификации стадию обратной транскрипции и добавлять в реакционную смесь один из следующих ферментов:
1) ревертазу из миелобластоз-ассоциированного вируса-сателлита (от англ. myeloblastosis-associated virus, MAV), происхождение которой часто ошибочно приписывают вирусу миелобластоза птиц (от англ. avian myeloblastosis virus, AMV) [58];
2) ревертазу вируса лейкемии мышей Молони (от англ. Moloney murine leukemia virus, MMLV) [59];
3) ревертазу с "горячим" стартом WarmStart RTx (NEB, США), которая может работать при 65°C [13].Альтернативным вариантом для тест-систем на РНК-мишень может быть использование ДНК-полимеразы Bst 3.0 (NEB, США), обладающей выраженной ревертазной активностью [13].
При использовании ревертаз в методе LAMP важно помнить об их различиях в процессивности, стабильности и температурном диапазоне активности, который не всегда пересекается с температурным оптимумом Bst-полимеразы [59]. Таким образом, при совмещении ревертаз AMV/MAV и MMLV с методом LAMP возникает необходимость в двух температурных режимах, отводимых на обратную транскрипцию и собственно амплификацию, что делает саму концепцию изотермического теста несколько противоречивой, поскольку в данном случае его проведение потребует программируемого термостата или термоциклера. В этой связи оправдано использование ревертаз, способных осуществлять РНК-зависимый синтез ДНК в температурном оптимуме используемой ДНК-полимеразы [13, 60].
В некоторых задачах диагностики, когда допустима идентификация патогена с низким таксономическим разрешением [47], оправдан подход, направленный на снижение специфичности тест-системы к нуклеотидным полиморфизмам в гене-мишени. Повысить толерантность праймеров к незначительным отличиям в нуклеотидной последовательности удалось сначала для ПЦР [61], a через некоторое время этот способ был адаптирован для LAMP [62]. Методика предполагает использование в реакционной смеси двух полимераз: классической Bst-полимеразы и ДНК-полимеразы с 3′→5′ экзонуклеазной активностью. Второй фермент выполняет при этом функцию удаления из последовательности праймеров всех некомплементарных матрице нуклеотидов. Такая модификация метода LAMP уже нашла применение при создании тест-систем для диагностики вируса лихорадки денге [62] и коронавируса SARS-CoV-2 [63].
Среди других ферментов, успешно адаптированных для метода LAMP, следует отметить хеликазу UvrD [64, 65], способную снизить вероятность фоновой амплификации, а также урацил-ДНК-гликозилазу [66], применяемую в технологии ПЦР для предотвращения контаминации продуктами амплификации.
ИНЖЕНЕРИЯ НОВЫХ ФЕРМЕНТОВ
Эра поиска новых ферментов дикого типа для решения актуальных задач биотехнологии постепенно уходит в прошлое. На первое место выходят современные методы инженерной энзимологии и способы предсказания третичной структуры белков, позволяющие с помощью единичных модификаций аминокислотной последовательности добиться изменения их активности [67–69]. Применение подобных подходов помогло создать термостабильные гомологи Bst LF, переносящие нагрев до 90°C в течение 2 мин, что превосходит по этому критерию существующие коммерческие ферменты (табл. 2) [70–72]. Стабильность третичной структуры гомологов Bst LF может быть полезна как для экспресс-методов прямой амплификации с термическим лизисом мембран патогенов [73], так и при создании сухих реакционных смесей по протоколам лиофилизации или вакуумной сушки [74, 75]. Кроме того, рациональный дизайн Taq-полимеразы открыл для этого фермента новую сферу применения в изотермической амплификации (табл. 2). Таким образом, методы направленной эволюции [81, 82] становятся необходимыми инструментами при адаптации известных и новых ферментов под широкий спектр узкоспециализированных задач молекулярной диагностики.
Таблица 2.
Модификации аминокислотной последовательности известных ДНК-полимераз, повышающие их применимость в методе LAMP
| Фермент | Модификация | Наблюдаемый эффект | Ссылки |
|---|---|---|---|
| PB | D422A | Повышение термостабильности при 65°C | [57] |
| Bst LF | T493N A552G |
Повышение термостабильности и возможность повышения температуры реакции до 73°C | [71] |
| Taq | G46D | Снижение 5′→3′ экзонуклеазной активности | [76–78] |
| K738F A743F |
Появление цепь-вытесняющей активности и возможность применения в реакции LAMP | ||
| D732N | Увеличение процессивности и скорости амплификации, появление цепь-вытесняющей и ревертазной активности | [79] | |
| D119A D119N |
Снижение 5′→3′ экзонуклеазной активности | ||
| E507K | Ускорение полимеразной активности и повышение толерантности фермента к цельной крови | [80] |
Интересно, что поиск ДНК-полимераз с цепь-вытесняющей активностью оказывается возможным не только в геномах обитателей геотермальных источников, но и среди арктических микроорганизмов. В качестве примера может служить ДНК-полимераза из морской психрофильной бактерии Psychrobacillus sp. (PB-полимераза) с оптимумом активности при 25–37°C, которой необходима всего одна модификация (табл. 2) для совместимости с методом LAMP [57]. Тем не менее в своей нативной форме низкотемпературная PB-полимераза может стать основой для тест-систем, использующих в качестве источника постоянной температуры тепло человеческого тела. Прототипы таких тест-систем на основе метода RPA (от англ. recombinase polymerase amplification) были разработаны для диагностики фитопатогенных вирусов [83]. Поиск других ДНК-полимераз с низкотемпературным оптимумом активности может привести к созданию методов амплификации, не требующих нагревательных приборов, что может быть востребовано при организации мобильных пунктов диагностики.
Таким образом, идеальная ДНК-полимераза для амплификации LAMP, по всей видимости, должна объединять в себе сразу несколько свойств: сильно выраженную цепь-вытесняющую активность, высокую скорость синтеза ДНК и процессивность фермента, наличие активности обратной транскриптазы, отсутствие экзонуклеазной активности, а также устойчивость фермента к различным ингибиторам амплификации.
ОПТИМИЗАЦИЯ АМПЛИФИКАЦИИ
При создании и оптимизации тест-систем на основе LAMP важным этапом является скрининг праймеров, не подверженных фоновой амплификации и ложноположительному срабатыванию при отсутствии в реакционной смеси ДНК-матрицы. При этом влияние конкретного фактора или компонента реакционной смеси необходимо контролировать не только по скорости появления сигнала амплификации, но и по его специфичности. В наиболее быстром сигнале амплификации может остаться незамеченным вклад фоновой амплификации, который при использовании интеркалирующих красителей может быть выявлен только при анализе кривых плавления финального амплификата.
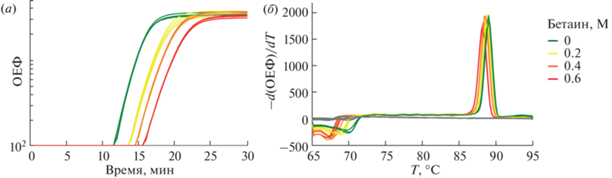
Оптимизация амплификации LAMP минимально включает в себя определение температурного режима проведения реакции (рис. 3а) и необходимой концентрации ионов магния (рис. 3б), а также подбор концентраций всех пар праймеров, которые планируется включить в состав реакционной смеси (рис. 4). Использование в качестве дополнительных компонентов реакционной смеси различных энхансеров амплификации, обычно применяемых для оптимизации ПЦР, допустимо и при работе с методом LAMP. Для каждого нового компонента реакционной смеси необходимо демонстрировать его положительное влияние на эффективность амплификации. Например, добавляя в состав буфера гуанидин гидрохлорид, можно значительно ускорить реакцию LAMP и увеличить чувствительность праймеров для детекции РНК или ДНК [84]. Примером негативного влияния на скорость реакции LAMP может служить бетаин, который, снижая температуру плавления пар G·C [85], ингибирует неспецифическую и специфическую амплификацию, что подтверждается некоторыми авторами и нашими наблюдениями (рис. 5) [23, 86].
Рис. 3.
Влияние температуры (а) и концентрации ионов магния (б) на скорость (слева) и специфичность (справа) реакции LAMP на основе набора праймеров DAT. ОЕФ – относительные единицы флуоресценции. Каждый фактор оптимизации тестировали в трех технических повторах (n = 3). Кривые амплификации даны в логарифмическом масштабе. Для измерения сигналов флуоресценции использовали термоциклер CFX96 Touch (Bio-Rad, США) и интеркалирующий краситель EvaGreen (Biotium, США). Кривые плавления финального амплификата только в случае повышенной температуры (66°C) и концентрации ионов магния (10 мМ) позволяют заметить изменение специфичности амплификации по появлению минорных пиков или смещению основных пиков плавления. Сигналы отрицательных контролей без добавления ДНК-матрицы показаны серым цветом.
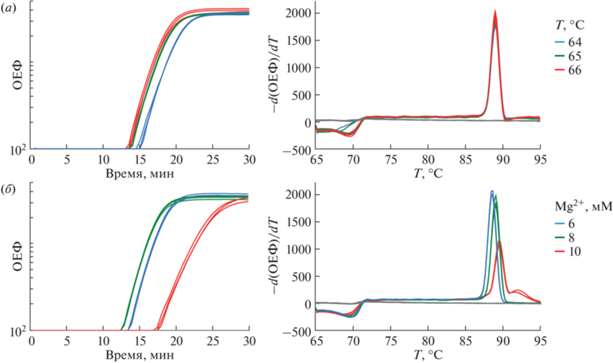
Рис. 4.
Влияние петлевых праймеров на скорость и специфичность реакции LAMP. CP – коровые праймеры (FIP, BIP, F3 и B3); LF – прямой петлевой праймер; LB – обратный петлевой праймер; LP – прямой и обратный петлевые праймеры. (а) – Кривые амплификации в логарифмическом масштабе. Каждый петлевой праймер в концентрации 0.8 мМ способен ускорять амплификацию, поэтому при добавлении двух праймеров наблюдается выраженный синергетический эффект на скорость реакции; (б) – кривые плавления финального амплификата с идентичной Тпл, свидетельствующей о сохранении уровня специфичности при увеличении скорости реакции.
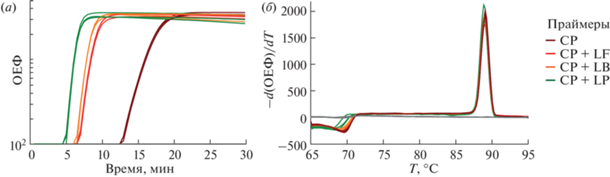
Рис. 5.
Влияние бетаина на скорость, а также на специфичность и температуру плавления амплификата в реакции LAMP. (а) – Кривые амплификации в логарифмическом масштабе. При увеличении концентрации бетаина наблюдается ингибирование реакции; (б) – кривые плавления финального амплификата. Наблюдается постепенное снижение Тпл амплификата при увеличении концентрации бетаина в реакционной смеси.
При разработке многокомпонентных реакционных смесей, требующих нахождения сбалансированного сочетания всех реагентов, существует способ значительно сократить количество необходимых экспериментов по поиску оптимальных значений факторов, влияющих на эффективность амплификации. Для этого следует выполнять дизайн экспериментов с помощью метода построения ортогональных матриц, принцип которого был исходно предложен японским инженером Гэнъити Тагути для оптимизации производственных процессов и контроля качества продукции [87, 88]. В применении к методам амплификации ортогональная матрица Тагути будет содержать информацию о минимальном количестве комбинаций различных факторов, позволяющем малым числом экспериментальных проверок наиболее широко оценить ландшафт оптимальных значений каждого интересующего фактора, измеряя отношение сигнал/шум.
Дизайн экспериментов на основе классической факториальной матрицы при наличии пяти факторов оптимизации (например, температура и четыре компонента реакционной смеси) в трех различных переменных значениях предписывает экспериментально проверить 243 (35) комбинации. Метод Тагути позволяет сократить количество необходимых экспериментов до 27. Создаваемая на основе дисперсионного анализа экспериментальных данных математическая модель позволяет определить оптимальные значения факторов, а также вычислить процентный вклад каждого фактора в эффективность амплификации [89]. Несмотря на очевидные преимущества этого подхода в области разработки тест-систем, метод Тагути используется незаслуженно редко.
СПОСОБЫ ДЕТЕКЦИИ
Метод LAMP оказался совместим с используемыми ранее в ПЦР способами детекции, такими как агарозный гель-электрофорез и детекция амплификации в режиме реального времени с интеркалирующими красителями [9]. С целью выявления подходящих интеркалирующих красителей (например, некоторые красители семейства SYTO, EvaGreen и др.) была проведена серия сравнительных исследований их ингибирующего действия на реакцию LAMP [90, 91]. Позднее для детекции LAMP была предложена методика, основанная на мечении внутренних и петлевых праймеров для последующей визуализации амплификата с помощью бумажных тест-полосок [92, 93]. Благодаря особенностям прохождения реакции, для LAMP были разработаны принципиально новые способы визуальной детекции, которые на сегодняшний день недоступны в технологии ПЦР. Рассмотрим эти способы детекции более подробно.
Рис. 6.
Аналитическая чувствительность набора праймеров DAT в диапазоне концентраций 4 × 100–4 × 105 копий геномной ДНК D. solani в ходе реакции LAMP. (а) – Кривые амплификации в логарифмическом масштабе; (б) – кривые плавления финального амплификата; (в) – стандартная кривая с визуализацией линейного динамического диапазона и предела детекции. Эффективность амплификации составила 99.4%, R2 = 0.948; (г) – детекция LAMP с помощью гидроксинафтола синего. ГЭ – геном-эквивалент; РС – реакционная смесь; N – отрицательный контроль без добавления ДНК-матрицы; 1–6 – реакции с добавлением геномной ДНК в порядке падения концентрации. На дне пробирок с положительными реакциями (1–5) виден осадок пирофосфата магния. Предел детекции (40 ГЭ/РС) на основе гидроксинафтола синего совпадает с таковым, полученным в формате детекции в режиме реального времени.
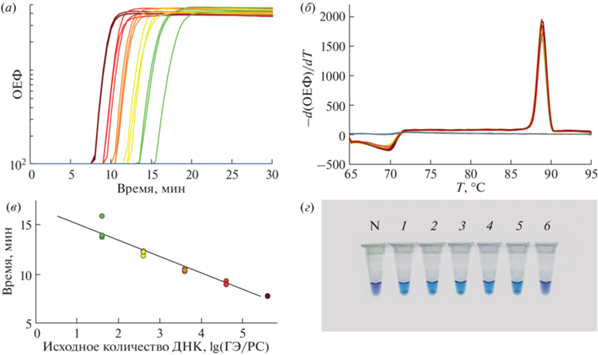
Первым качественным признаком положительной реакции, обнаруженным и характерным для реакции LAMP, стало образование на дне пробирок белого нерастворимого осадка пирофосфата магния. Динамику повышения мутности реакционной смеси с протекающей амплификацией было предложено оценивать на основе турбидиметрии с помощью специально созданных для этого портативных приборов-термостатов [26]. Таким образом, дополнительным признаком прохождения амплификации оказалось снижение концентрации свободных ионов магния в реакционной смеси.
Визуализация сигнала реакции LAMP по магнию основана на применении веществ-индикаторов двухвалентных ионов металлов, которые при изменении концентрации свободных ионов способны менять цвет реакционной смеси в видимом или ультрафиолетовом свете [94, 95]. Исторически первым таким металлоиндикатором стал кальцеин [10], механизм действия которого требует добавления в реакционную смесь ионов марганца. Немного позднее стали применять краситель гидроксинафтоловый синий (рис. 6г), широко используемый для мобильной диагностики и не требующий дополнительных модификаций состава реакционной смеси [96]. В последнее время, особенно при детекции LAMP в микрофлюидных чипах, набирает популярность индикатор эриохромовый черный Т [74, 97]. Другой металлоиндикатор, известный под названием кислотный хром синий K, был также успешно апробирован для детекции LAMP [98].
Альтернативный способ детекции LAMP основан на использовании различных pH-чувствительных красителей, меняющих цвет при подкислении реакционной смеси в процессе синтеза ДНК. К этой группе индикаторов можно отнести феноловый красный, крезоловый красный, нейтральный красный, а также метакрезол фиолетовый [99]. Недавно было показано применение в детекции LAMP ксиленолового оранжевого [100] и некоторых производных флуоресцеина [101].
При использовании описанных выше красителей-индикаторов мы имеем дело с непрямой детекцией амплификации, поскольку в механизме их работы важную роль играют побочные продукты реакции LAMP: пирофосфат магния и ионы водорода. Поэтому присутствие в образце различных интерферирующих веществ (например, двухвалентных ионов металлов) или неподходящий уровень pH образца может привести к изменению окраски реакционной смеси LAMP до начала амплификации. Так, даже атмосферный углекислый газ может оказать негативное влияние на срок хранения реагентов на основе фенолового красного, подкисляя слабозабуференную реакционную смесь [102].
К индикаторам, которые меняют свою окраску в ответ на повышение концентрации амплифицируемой ДНК, относят берберин [103], кристаллический фиолетовый [104], малахитовый зеленый [105], а также метиловый зеленый [106].
Подводя итог, отметим, что использование любых видов визуальной детекции в клинической диагностике требует максимально объективной оценки изменения цвета, основанной на спектрофотометрическом анализе [84, 102].
Актуальной задачей по-прежнему является совершенствование способов прямой детекции LAMP посредством особой структуры праймеров, меченных флуорофором и гасителем флуоресценции [12]. Отсутствие у Bst LF 5′→3′ экзонуклеазной активности затрудняет использование в методе LAMP линейных разрушаемых TaqMan-проб, а также проб с инвертированным концевым повтором, или “молекулярных маяков” [107, 108], для детекции накопления ДНК в режиме реального времени. Технология TaqMan работает в LAMP с некоторой долей фоновой амплификации в отрицательных контролях без матрицы [109], а “молекулярные маяки” характеризуются крайне низкой специфичностью [110]. Поэтому наиболее оптимальным решением проблемы прямой детекции представляется мечение флуорофором внутренних праймеров [37].
КОЛИЧЕСТВЕННЫЙ АНАЛИЗ
Преимущество количественного анализа нуклеиновых кислот на основе ПЦР состоит в том, что удвоение количества ампликонов в оптимизированных условиях реакции, при которых эффективность амплификации составляет ~100%, строго связано со стадиями термоциклирования и происходит в каждом новом цикле [111]. В реакции LAMP кинетические параметры отжига праймеров и элонгации ДНК могут сильно варьировать, поскольку они определяются термодинамическими свойствами праймеров и амплифицируемого участка гена-мишени. Таким образом, высокая эффективность реакции LAMP способствует снижению разрешающей способности тест-системы на образцах со слабыми отличиями в концентрации целевой нуклеиновой кислоты [112]. Парадоксально, но метод LAMP может быть одновременно охарактеризован как весьма чувствительный метод с низким значением предела детекции, а с другой стороны, он имеет слабую разрешающую способность при попытке оценить количество копий мишени в образцах с менее чем 10-кратной разницей в концентрации исходной нуклеиновой кислоты. Во многом это связано с тем, что на прохождение всего линейного динамического диапазона реакции LAMP требуется лишь несколько минут (рис. 6в) [112]. И если значения пороговых циклов ПЦР можно с достаточно высокой точностью пересчитать в количество копий гена-мишени, то в случае с пороговыми значениями времени LAMP мы можем гарантированно различать только 10-кратную разницу концентраций искомой нуклеиновой кислоты. В этой связи вопрос применимости метода LAMP для количественной диагностики в режиме реального времени представляется довольно спорным, несмотря на предложенные математические модели кинетики амплификации [27, 113]. Означает ли это, что метод LAMP абсолютно неприменим для количественной диагностики?
Для понимания ответа на этот вопрос следует рассмотреть технологию LAMP в свете метода цифровой ПЦР, как наиболее совершенного способа количественного анализа нуклеиновых кислот [114, 115]. Суть цифровой ПЦР состоит в том, что стандартная реакционная смесь разделяется на тысячи индивидуальных микрореакций, в каждой из которых может содержаться единичное количество генов-мишеней. Обнаружение мишени в таком случае происходит на пределе аналитической чувствительности, поскольку множество индивидуальных микрореакций устраняет возможные различия концентраций гена-мишени в каждой из них. Следовательно, это позволяет использовать для детекции нуклеиновой кислоты любой чувствительный метод амплификации, среди которых следует рассматривать и метод LAMP [116].
Таким образом, метод LAMP может составить конкуренцию ПЦР именно в формате цифровой амплификации. Во-первых, для реакции LAMP достаточно простейшего резистивного нагревательного элемента, поэтому из стоимости устройства для цифровой LAMP можно смело вычитать затраты на разработку точного и высокоскоростного термоциклера. Во-вторых, высокий выход реакции LAMP в сочетании с яркими интеркалирующими красителями способен обеспечить более высокую интенсивность флуоресценции индивидуальных микрореакций с положительным сигналом, что позволит более точно отличать их от микрореакций с отрицательным сигналом. Пожалуй, не останется значимых преимуществ у цифровой LAMP только на этапах разработки и производства чипов, необходимых для генерации капель реакционной смеси в масле [117] или внутри отдельных микрореакторов [74].
ЗАКЛЮЧЕНИЕ
Гибкость методов изотермической амплификации с возможностью тонкой настройки каждого компонента реакции является одной из движущих характеристик, способствовавших появлению огромного количества модификаций метода LAMP. На сегодняшний день многие из них претендуют на роль “золотого стандарта” для мобильной диагностики [118, 119]. Неприхотливость изотермической амплификации к источнику температуры и совместимость с сухим форматом реакционной смеси открывает перед методом LAMP широкие перспективы. Существует множество различных портативных платформ, созданных в том числе благодаря усилиям российских научных групп, для проведения реакции LAMP:
1) мобильные термостаты для пробирок 0.2 мл с детекцией сигнала амплификации в режиме реального времени [13, 65, 120, 121];
2) микрофлюидные лаборатории “на чипе”, имеющие отсеки для выделения нуклеиновых кислот, проведения LAMP и детекции амплификации с помощью бумажной тест-полоски [122];
3) одноразовые чипы со множеством микрореакционных камер и специальные картриджи для реакционной смеси LAMP, позволяющие анализировать результаты амплификации с помощью смартфона [74, 97, 123].
В настоящее время обнаружение однонуклеотидных полиморфизмов так и не стало главным преимуществом LAMP, как планировалось разработчиками метода [21]. Тем не менее весьма оригинальный способ аллель-специфичной детекции был предложен на основе LAMP и двух олигонуклеотидных зондов, каждый из которых представляет собой половину стартовой структуры [124]. Отжиг таких зондов на однонуклеотидном полиморфизме приводит к их лигированию и формированию полноценной стартовой структуры, которая запускает реакцию LAMP только при наличии в гене-мишени искомой нуклеотидной замены. Возможно, развитие именно этого способа амплификации поможет в адаптации метода LAMP к исследованиям в области генетических заболеваний.
Особенности строения стартовых структур и архитектура праймеров LAMP послужили важной отправной точкой в создании других способов изотермической амплификации [33, 125]. Некоторые результаты представляются весьма перспективными для внедрения в метод LAMP с целью повышения его чувствительности к нуклеотидным заменам. В частности, особый интерес представляет ДНК-связывающий белок MutS из T. aquaticus, ставший основой для детекции нуклеотидных полиморфизмов в одном из ассиметричных методов изотермической амплификации [126]. Совершенствование алгоритмов дизайна праймеров и рациональное включение в их последовательность модифицированных нуклеотидов, таких как LNA и PNA [127, 128], может повысить стабильность праймеров при хранении и привести к появлению более специфичных методик аллель-специфичной LAMP.
Пандемия коронавирусной инфекции COVID-19 послужила важным стимулом для развития метода LAMP, вдохновив исследователей на совершенствование экспресс-методов пробоподготовки [129, 130], разработку методов диагностики без выделения и очистки нуклеиновых кислот [131, 132], оптимизацию альтернативных ДНК-полимераз и ревертаз [60], создание новых способов детекции на основе различных нуклеаз CRISPR-Cas [133–136]. Именно в последние годы началось широкомасштабное тестирование технологии LAMP в клинической практике [19, 102, 137].
Все эти исследования могут способствовать постепенному внедрению метода LAMP в систему здравоохранения как недорогого, специфичного и быстрого способа проведения скринингового обследования больших групп населения [138]. Впереди перед ним множество испытаний на пути в сферу молекулярной диагностики, возникающих по причине повсеместного внедрения ПЦР в диагностических лабораториях и доминирующего положения ПЦР в нормативно-правовых документах, регулирующих эту область в медицине и сельском хозяйстве. Можно рассчитывать, что именно экономический фактор сыграет важную роль в интенсивности мер по внедрению технологии LAMP во многих странах мира.
Таким образом, метод LAMP стал во многом революционным решением в области мобильной молекулярной диагностики, создав целую плеяду новых способов детекции амплификации нуклеиновых кислот. В перспективе технология LAMP имеет все шансы стать полноценной альтернативой ПЦР в области молекулярной диагностики патогенов, а новые модификации проведения LAMP обязательно помогут найти этому интересному способу амплификации свою уникальную сферу применения.
Список литературы
Esbin M.N., Whitney O.N., Chong S., Maurer A., Darzacq X., Tjian R. // RNA. 2020. V. 26. P. 771–783. https://doi.org/10.1261/rna.076232.120
Chaouch M. // Rev. Med. Virol. 2021. V. 31. P. e2215. https://doi.org/10.1002/rmv.2215
Islam M.M., Koirla D. // Anal. Chim. Acta. 2021. P. 339338. https://doi.org/10.1016/j.aca.2021.339338
Guatelli J.C., Whitfield K.M., Kwoh D.Y., Barringer K.J., Richman D.D., Gingeras T.R. // Proc. Natl. Acad. Sci. USA. 1990. V. 87. P. 1874–1878. https://doi.org/10.1073/pnas.87.5.1874
Mohsen M.G., Kool E.T. // Acc. Chem. Res. 2016. V. 49. P. 2540–2550. https://doi.org/10.1021/acs.accounts.6b00417
Kusumoto-Matsuo R., Kanda T., Kukimoto I. // Genes Cells. 2011. V. 16. P. 23–33. https://doi.org/10.1111/j.1365-2443.2010.01458.x
Flores R., Serra P., Minoia S., Di Serio F., Navarro B. // Front. Microbiol. 2012. V. 3. P. 217. https://doi.org/10.3389/fmicb.2012.00217
Wawrzyniak P., Płucienniczak G., Bartosik D. // Front. Microbiol. 2017. V. 8. P. 2353. https://doi.org/10.3389/fmicb.2017.02353
Notomi T., Okayama H., Masubuchi H., Yonekawa T., Watanabe K., Amino N., Hase T. // Nucleic Acids Res. 2000. V. 28. P. e63. https://doi.org/10.1093/nar/28.12.e63
Tomita N., Mori Y., Kanda H., Notomi T. // Nat. Protoc. 2008. V. 3. P. 877–882. https://doi.org/10.1038/nprot.2008.57
Green M.R., Sambrook J. // Cold Spring Harb. Protoc. 2019. V. 2019. P. 436–456. https://doi.org/10.1101/pdb.top095109
Becherer L., Borst N., Bakheit M., Frischmann S., Zengerle R., von Stetten F. // Anal. Methods. 2020. V. 12. P. 717–746. https://doi.org/10.1039/C9AY02246E
Moore K.J.M., Cahill J., Aidelberg G., Aronoff R., Bektaş A., Bezdan D., Butler D.J., Chittur S.V., Codyre M., Federici F., Tanner N.A., Tighe S.W., True R., Ware S.B., Wyllie A.L., Afshin E.E., Bendesky A., Chang C.B., Dela Rosa R., 2nd, Elhaik E., Erickson D., Goldsborough A.S., Grills G., Hadasch K., Hayden A., Her S.Y., Karl J.A., Kim C.H., Kriegel A.J., Kunstman T., Landau Z., Land K., Langhorst B.W., Lindner A.B., Mayer B.E., McLaughlin L.A., McLaughlin M.T., Molloy J., Mozsary C., Nadler J.L., D’Silva M., Ng D., O’Connor D.H., Ongerth J.E., Osuolale O., Pinharanda A., Plenker D., Ranjan R., Rosbash M., Rotem A., Segarra J., Schürer S., Sherrill-Mix S., Solo-Gabriele H., To S., Vogt M.C., Yu A.D., Mason C.E., The gLAMP Consortium // J. Biomol. Tech. 2021. V. 32. P. 228–275. https://doi.org/10.7171/jbt.21-3203-017
Rubinfien J., Atabay K.D., Nichols N.M., Tanner N.A., Pezza J.A., Gray M.M., Wagner B.M., Poppin J.N., Aken J.T., Gleason E.J., Foley K.D., Copeland D.S., Kraves S., Alvarez Saavedra E. // FASEB Bioadv. 2020. V. 2. P. 160–165. https://doi.org/10.1096/fba.2019-00088
Tomlinson J. // Methods Mol. Biol. 2013. V. 938. P. 291–300. https://doi.org/10.1007/978-1-62703-089-2_25
Suea-Ngam A., Bezinge L., Mateescu B., Howes P.D., DeMello A.J., Richards D.A. // ACS Sens. 2020. V. 5. P. 2701–2723. https://doi.org/10.1021/acssensors.0c01488
Yadav S., Sharma N.N., Akhtar J. // Analyst. 2021. V. 146. P. 3422–3439. https://doi.org/10.1039/d1an00214g
Liu W., Yue F., Lee L.P. // Acc. Chem. Res. 2021. V. 54. P. 4107–4119. https://doi.org/10.1021/acs.accounts.1c00385
Mozsary C., McCloskey D., Babler K.M., Boza J., Butler D., Currall B., Williams S., Wiley A., Afshin E.E., Grills G.S., Sharkey M.E., Premsrirut P., Solo-Gabriele H., Cardentey Y., Erickson D., Mason C.E. // J. Biomol. Tech. 2021. V. 32. P. 221–227. https://doi.org/10.7171/jbt.21-3203-019
Мирошников К.А., Ширшиков Ф.В. // Патент RU 2642313 C1, 2018.
Notomi T., Hase T. // Patent US 6410278 B1, 2002.
Nagamine K., Hase T., Notomi T. // Mol. Cell. Probes. 2002. V. 16. P. 223–229. https://doi.org/10.1006/mcpr.2002.0415
Tanner N.A., Evans T.C. // Curr. Protoc. Mol. Biol. 2014. V. 105. P. 15.14.1–15.14.14. https://doi.org/10.1002/0471142727.mb1514s105
von Hippel P.H., Johnson N.P., Marcus A.H. // Biopolymers. 2013. V. 99. P. 923–954. https://doi.org/10.1002/bip.22347
Beyerle E.R., Dinpajooh M., Ji H., von Hippel P.H., Marcus A.H., Guenza M.G. // Nucleic Acids Res. 2021. V. 49. P. 1872–1885. https://doi.org/10.1093/nar/gkab015
Mori Y., Nagamine K., Tomita N., Notomi T. // Biochem. Biophys. Res. Commun. 2001. V. 289. P. 150–154. https://doi.org/10.1006/bbrc.2001.5921
Schneider L., Blakely H., Tripathi A. // Electrophoresis. 2019. V. 40. P. 2706–2717. https://doi.org/10.1002/elps.201900167
Kaur N., Thota N., Toley B.J. // Comput. Struct. Biotechnol. J. 2020. V. 18. P. 2336–2346. https://doi.org/10.1016/j.csbj.2020.08.020
Meagher R.J., Priye A., Light Y.K., Huang C., Wang E. // Analyst. 2018. V. 143. P. 1924–1933. https://doi.org/10.1039/c7an01897e
SantaLucia J., Hicks D. // Annu. Rev. Biophys. Biomol. Struct. 2004. V. 33. P. 415–440. https://doi.org/10.1146/annurev.biophys.32.110601.141800
Gandelman O., Jackson R., Kiddle G., Tisi L. // Int. J. Mol. Sci. 2011. V. 12. P. 9108–9124. https://doi.org/10.3390/ijms12129108
Martineau R.L., Murray S.A., Ci S., Gao W., Chao S.-H., Meldrum D.R. // Anal. Chem. 2017. V. 89. P. 625–632. https://doi.org/10.1021/acs.analchem.6b02578
Ding X., Xu Z., Yin K., Sfeir M., Liu C. // Anal. Chem. 2019. V. 91. P. 12852–12858. https://doi.org/10.1021/acs.analchem.9b02582
Mao R., Wang T., Zhao Y., Wu X., Zhang S., Cai T. // Talanta. 2022. V. 240. P. 123217. https://doi.org/10.1016/j.talanta.2022.123217
Zuker M. // Nucleic Acids Res. 2003. V. 31. P. 3406–3415. https://doi.org/10.1093/nar/gkg595
Popenda M., Szachniuk M., Antczak M., Purzycka K.J., Lukasiak P., Bartol N., Blazewicz J., Adamiak R.W. // Nucleic Acids Res. 2012. V. 40. P. e112. https://doi.org/10.1093/nar/gks339
Tanner N.A., Zhang Y., Evans T.C. // Biotechniques. 2012. V. 53. P. 81–89. https://doi.org/10.2144/0000113902
Ball C.S., Light Y.K., Koh C.-Y., Wheeler S.S., Coffey L.L., Meagher R.J. // Anal. Chem. 2016. V. 88. P. 3562–3568. https://doi.org/10.1021/acs.analchem.5b04054
Mayboroda O., Katakis I., O’Sullivan C.K. // Anal. Biochem. 2018. V. 545. P. 20–30. https://doi.org/10.1016/j.ab.2018.01.005
Zhang Y., Tanner N.A. // PLoS One. 2022. V. 17. P. e0254324. https://doi.org/10.1371/journal.pone.0254324
Yan W. // Methods Mol. Biol. 2015. V. 1275. P. 91–101. https://doi.org/10.1007/978-1-4939-2365-6_6
Skuza L. // Methods Mol. Biol. 2022. V. 2392. P. 83–91. https://doi.org/10.1007/978-1-0716-1799-1_6
Ширшиков Ф.В., Пеков Ю.А., Карпец П.Ю., Мирошников К.А. // Свидетельство о государственной регистрации программы для ЭВМ RU 2018616252, 2018.
Shirshikov F.V., Pekov Y.A., Miroshnikov K.A. // PeerJ. 2019. V. 7. P. e6801. https://doi.org/10.7717/peerj.6801
Elbe S., Buckland-Merrett G. // Glob. Chall. 2017. V. 1. P. 33–46. https://doi.org/10.1002/gch2.1018
Chen A.T., Altschuler K., Zhan S.H., Chan Y.A., Deverman B.E. // eLife. 2021. V. 10. P. e63409. https://doi.org/10.7554/elife.63409
Tamanaha E., Zhang Y., Tanner N.A. // PLoS One. 2022. V. 17. P. e0259610. https://doi.org/10.1371/journal.pone.0259610
Nazina T.N., Tourova T.P., Poltaraus A.B., Novikova E.V., Grigoryan A.A., Ivanova A.E., Lysenko A.M., Petrunyaka V.V., Osipov G.A., Belyaev S.S., Ivanov M.V. // Int. J. Syst. Evol. Microbiol. 2001. V. 51. P. 433–446. https://doi.org/10.1099/00207713-51-2-433
Coorevits A., Dinsdale A.E., Halket G., Lebbe L., De Vos P., Van Landschoot A., Logan N.A. // Int. J. Syst. Evol. Microbiol. 2012. V. 62. P. 1470–1485. https://doi.org/10.1099/ijs.0.030346-0
Oscorbin I.P., Boyarskikh U.A., Filipenko M.L. // Mol. Biotechnol. 2015. V. 57. P. 947–959. https://doi.org/10.1007/s12033-015-9886-x
Jackson L.N., Chim N., Shi C., Chaput J.C. // Nucleic Acids Res. 2019. V. 47. P. 6973–6983. https://doi.org/10.1093/nar/gkz513
Oscorbin I.P., Belousova E.A., Boyarskikh U.A., Zakabunin A.I., Khrapov E.A., Filipenko M.L. // Nucleic Acids Res. 2017. V. 45. P. 9595–9610. https://doi.org/10.1093/nar/gkx645
Xiao X., Li H., Zhao L., Zhang Y., Liu Z. // Biomed. Pharmacother. 2021. V. 143. P. 112232. https://doi.org/10.1016/j.biopha.2021.112232
Murali R., Sharkey D.J., Daiss J.L., Murthy H.M. // Proc. Natl. Acad. Sci. USA. 1998. V. 95. P. 12562–12567. https://doi.org/10.1073/pnas.95.21.12562
Moser M.J., DiFrancesco R.A., Gowda K., Klingele A.J., Sugar D.R., Stocki S., Mead D.A., Schoenfeld T.W. // PLoS One. 2012. V. 7. P. e38371. https://doi.org/10.1371/journal.pone.0038371
Chander Y., Koelbl J., Puckett J., Moser M.J., Klingele A.J., Liles M.R., Carrias A., Mead D.A., Schoenfeld T.W. // Front. Microbiol. 2014. V. 5. P. 395. https://doi.org/10.3389/fmicb.2014.00395
Piotrowski Y., Gurung M.K., Larsen A.N. // BMC Mol. cell Biol. 2019. V. 20. P. 31. https://doi.org/10.1186/s12860-019-0216-1
Perbal B. // Retrovirology. 2008. V. 5. P. 49. https://doi.org/10.1186/1742-4690-5-49
Rittié L., Perbal B. // J. Cell Commun. Signal. 2008. V. 2. P. 25–45. https://doi.org/10.1007/s12079-008-0026-2
Alekseenko A., Barrett D., Pareja-Sanchez Y., Howard R.J., Strandback E., Ampah-Korsah H., Rovšnik U., Zuniga-Veliz S., Klenov A., Malloo J., Ye S., Liu X., Reinius B., Elsässer S.J., Nyman T., Sandh G., Yin X., Pelechano V. // Sci. Rep. 2021. V. 11. P. 1820. https://doi.org/10.1038/s41598-020-80352-8
Li Y., Wan Z., Hu Y., Zhou Y., Chen Q., Zhang C. // Biotechniques. 2019. V. 66. P. 225–230. https://doi.org/10.2144/btn-2018-0184
Zhou Y., Wan Z., Yang S., Li Y., Li M., Wang B., Hu Y., Xia X., Jin X., Yu N., Zhang C. // Front. Microbiol. 2019. V. 10. P. 1056. https://doi.org/10.3389/fmicb.2019.01056
Lu R., Wu X., Wan Z., Li Y., Zuo L., Qin J., Jin X., Zhang C. // Virol. Sin. 2020. V. 35. P. 344–347. https://doi.org/10.1007/s12250-020-00218-1
Bokelmann L., Nickel O., Maricic T., Pääbo S., Meyer M., Borte S., Riesenberg S. // Nat. Commun. 2021. V. 12. P. 1467. https://doi.org/10.1038/s41467-021-21627-0
Natoli M.E., Kundrod K.A., Chang M.M., Smith C.A., Paul S., Coole J.B., Butlin N.G., Tanner N.A., Baker E., Schmeler K.M., Richards-Kortum R. // J. Biomol. Tech. 2021. V. 32. P. 180–185. https://doi.org/10.7171/jbt.21-3203-013
Hsieh K., Mage P.L., Csordas A.T., Eisenstein M., Soh H.T. // Chem. Commun. (Camb). 2014. V. 50. P. 3747–3749. https://doi.org/10.1039/c4cc00540f
Jumper J., Evans R., Pritzel A., Green T., Figurnov M., Ronneberger O., Tunyasuvunakool K., Bates R., Žídek A., Potapenko A., Bridgland A., Meyer C., Kohl S.A.A., Ballard A.J., Cowie A., Romera-Paredes B., Nikolov S., Jain R., Adler J., Back T., Petersen S., Reiman D., Clancy E., Zielinski M., Steinegger M., Pacholska M., Berghammer T., Bodenstein S., Silver D., Vinyals O., Senior A.W., Kavukcuoglu K., Kohli P., Hassabis D. // Nature. 2021. V. 596. P. 583–589. https://doi.org/10.1038/s41586-021-03819-2
Coulther T.A., Stern H.R., Beuning P.J. // Trends Biotechnol. 2019. V. 37. P. 1091–1103. https://doi.org/10.1016/j.tibtech.2019.03.011
Nikoomanzar A., Chim N., Yik E.J., Chaput J.C. // Q. Rev. Biophys. 2020. V. 53. P. e8. https://doi.org/10.1017/s0033583520000050
Milligan J.N., Shroff R., Garry D.J., Ellington A.D. // Biochemistry. 2018. V. 57. P. 4607–4619. https://doi.org/10.1021/acs.biochem.8b00200
Paik I., Ngo P.H.T., Shroff R., Diaz D.J., Maranhao A.C., Walker D.J.F., Bhadra S., Ellington A.D. // Biochemistry. 2021. https://doi.org/10.1021/acs.biochem.1c00451
Paik I., Bhadra S., Ellington A.D. // ACS Synth. Biol. 2022. V. 11. P. 1488–1496. https://doi.org/10.1021/acssynbio.1c00559
Hu L.X., Yang Z.H., Zhang D., Zhao D.M., Zhu J.H. // Lett. Appl. Microbiol. 2016. V. 63. P. 289–296. https://doi.org/10.1111/lam.12618
Rodriguez-Manzano J., Karymov M.A., Begolo S., Selck D.A., Zhukov D. V., Jue E., Ismagilov R.F. // ACS Nano. 2016. V. 10. P. 3102–3113. https://doi.org/10.1021/acsnano.5b07338
García-Bernalt Diego J., Fernández-Soto P., Crego-Vicente B., Alonso-Castrillejo S., Febrer-Sendra B., Gómez-Sánchez A., Vicente B., López-Abán J., Muro A. // Sci. Rep. 2019. V. 9. P. 14744. https://doi.org/10.1038/s41598-019-51342-2
Ignatov K., Kramarov V. // Patent US 9896671 B2, 2018.
Ignatov K.B., Barsova E.V., Fradkov A.F., Blagodatskikh K.A., Kramarova T.V., Kramarov V.M. // Biotechniques. 2014. V. 57. P. 81–87. https://doi.org/10.2144/000114198
Varlamov D.A., Blagodatskikh K.A., Smirnova E.V., Kramarov V.M., Ignatov K.B. // Front. Bioeng. Biotechnol. 2020. V. 8. P. 604793. https://doi.org/10.3389/fbioe.2020.604793
Barnes W.M., Zhang Z., Kermekchiev M.B. // Front. Bioeng. Biotechnol. 2020. V. 8. P. 553474. https://doi.org/10.3389/fbioe.2020.553474
Arezi B., McKinney N., Hansen C., Cayouette M., Fox J., Chen K., Lapira J., Hamilton S., Hogrefe H. // Front. Microbiol. 2014. V. 5. P. 408. https://doi.org/10.3389/fmicb.2014.00408
Ouaray Z., Singh I., Georgiadis M.M., Richards N.G.J. // Protein Sci. 2020. V. 29. P. 455–468. https://doi.org/10.1002/pro.3762
Ouaray Z., Benner S.A., Georgiadis M.M., Richards N.G.J. // J. Biol. Chem. 2020. V. 295. P. 17046–17059. https://doi.org/10.1074/jbc.rev120.013745
Verosloff M., Chappell J., Perry K.L., Thompson J.R., Lucks J.B. // ACS Synth. Biol. 2019. V. 8. P. 902–905. https://doi.org/10.1021/acssynbio.8b00526
Zhang Y., Ren G., Buss J., Barry A.J., Patton G.C., Tanner N.A. // Biotechniques. 2020. V. 69. P. 178–185. https://doi.org/10.2144/btn-2020-0078
Vasudevamurthy M.K., Lever M., George P.M., Morison K.R. // Biopolymers. 2009. V. 91. P. 85–94. https://doi.org/10.1002/bip.21085
Ma C., Wang Y., Zhang P., Shi C. // Anal. Biochem. 2017. V. 530. P. 1–4. https://doi.org/10.1016/j.ab.2017.04.017
Cobb B.D., Clarkson J.M. // Nucleic Acids Res. 1994. V. 22. P. 3801–3805. https://doi.org/10.1093/nar/22.18.3801
Thanakiatkrai P., Welch L. // Int. J. Legal Med. 2012. V. 126. P. 161–165. https://doi.org/10.1007/s00414-011-0558-5
Morero M., Ramirez M.R., Oyhenart J. // Vet. Parasitol. 2021. V. 295. P. 109462. https://doi.org/10.1016/j.vetpar.2021.109462
Oscorbin I.P., Belousova E.A., Zakabunin A.I., Boyarskikh U.A., Filipenko M.L. // Biotechniques. 2016. V. 61. P. 20–25. https://doi.org/10.2144/000114432
Quyen T.L., Ngo T.A., Bang D.D., Madsen M., Wolff A. // Front. Microbiol. 2019. V. 10. P. 2234. https://doi.org/10.3389/fmicb.2019.02234
Phillips E.A., Moehling T.J., Bhadra S., Ellington A.D., Linnes J.C. // Anal. Chem. 2018. V. 90. P. 6580–6586. https://doi.org/10.1021/acs.analchem.8b00269
Li J., Macdonald J., von Stetten F. // Analyst. 2019. V. 144. P. 31–67. https://doi.org/10.1039/c8an01621f
Scott A.T., Layne T.R., O’Connell K.C., Tanner N.A., Landers J.P. // Anal. Chem. 2020. V. 92. P. 13343–13353. https://doi.org/10.1021/acs.analchem.0c02666
Varona M., Anderson J.L. // Anal. Chem. 2019. V. 91. P. 6991–6995. https://doi.org/10.1021/acs.analchem.9b01762
Goto M., Honda E., Ogura A., Nomoto A., Hanaki K.-I. // Biotechniques. 2009. V. 46. P. 167–172. https://doi.org/10.2144/000113072
Nguyen H.Q., Nguyen V.D., Van Nguyen H., Seo T.S. // Sci. Rep. 2020. V. 10. P. 15123. https://doi.org/10.1038/s41598-020-72095-3
Wang X., Fu Z., Chen X., Peng C., Xu X., Wei W., Li F., Xu J. // Anal. Bioanal. Chem. 2017. V. 409. P. 881–889. https://doi.org/10.1007/s00216-016-0084-x
Tanner N.A., Zhang Y., Evans T.C. // Biotechniques. 2015. V. 58. P. 59–68. https://doi.org/10.2144/000114253
Jaroenram W., Cecere P., Pompa P.P. // J. Microbiol. Methods. 2019. V. 156. P. 9–14. https://doi.org/10.1016/j.mimet.2018.11.020
Brown T.A., Schaefer K.S., Tsang A., Yi H.A., Grimm J.B., Lemire A.L., Jradi F.M., Kim C., McGowan K., Ritola K., Armstrong D.T., Mostafa H.H., Korff W., Vale R.D., Lavis L.D. // J. Biomol. Tech. 2021. V. 32. P. 121–133. https://doi.org/10.7171/jbt.21-3203-007
Dao Thi V.L., Herbst K., Boerner K., Meurer M., Kremer L.P., Kirrmaier D., Freistaedter A., Papagiannidis D., Galmozzi C., Stanifer M.L., Boulant S., Klein S., Chlanda P., Khalid D., Barreto Miranda I., Schnitzler P., Kräusslich H.G., Knop M., Anders S. // Sci. Transl. Med. 2020. V. 12. P. eabc7075. https://doi.org/10.1126/scitranslmed.abc7075
Fischbach J., Xander N.C., Frohme M., Glökler J.F. // Biotechniques. 2015. V. 58. P. 189–194. https://doi.org/10.2144/000114275
Miyamoto S., Sano S., Takahashi K., Jikihara T. // Anal. Biochem. 2015. V. 473. P. 28–33. https://doi.org/10.1016/j.ab.2014.12.016
Lucchi N.W., Ljolje D., Silva-Flannery L., Udhayakumar V. // PLoS One. 2016. V. 11. P. e0151437. https://doi.org/10.1371/journal.pone.0151437
Thapa J., Maharjan B., Malla M., Fukushima Y., Poudel A., Pandey B.D., Hyashida K., Gordon S. V., Nakajima C., Suzuki Y. // Tuberculosis (Edinb). 2019. V. 117. P. 1–6. https://doi.org/10.1016/j.tube.2019.05.004
Liu W., Huang S., Liu N., Dong D., Yang Z., Tang Y., Ma W., He X., Ao D., Xu Y., Zou D., Huang L. // Sci. Rep. 2017. V. 7. P. 40125. https://doi.org/10.1038/srep40125
Sherrill-Mix S., Hwang Y., Roche A.M., Glascock A., Weiss S.R., Li Y., Haddad L., Deraska P., Monahan C., Kromer A., Graham-Wooten J., Taylor L.J., Abella B.S., Ganguly A., Collman R.G., Van Duyne G.D., Bushman F.D. // Genome Biol. 2021. V. 22. P. 169. https://doi.org/10.1186/s13059-021-02387-y
Liang R., Liang L., Ren X., Jia Y., Han K., Zhao J., Song C., Cui S. // Arch. Virol. 2021. V. 166. P. 1599–1605. https://doi.org/10.1007/s00705-021-04963-w
Hardinge P., Murray J.A.H. // BMC Biotechnol. 2019. V. 19. P. 55. https://doi.org/10.1186/s12896-019-0549-z
Bustin S.A., Benes V., Garson J.A., Hellemans J., Huggett J., Kubista M., Mueller R., Nolan T., Pfaffl M.W., Shipley G.L., Vandesompele J., Wittwer C.T. // Clin. Chem. 2009. V. 55. P. 611–622. https://doi.org/10.1373/clinchem.2008.112797
Schoepp N.G., Schlappi T.S., Curtis M.S., Butkovich S.S., Miller S., Humphries R.M., Ismagilov R.F. // Sci. Transl. Med. 2017. V. 9. P. eaal3693. https://doi.org/10.1126/scitranslmed.aal3693
Subramanian S., Gomez R.D. // PLoS One. 2014. V. 9. P. e100596. https://doi.org/10.1371/journal.pone.0100596
Huggett J.F., Foy C.A., Benes V., Emslie K., Garson J.A., Haynes R., Hellemans J., Kubista M., Mueller R.D., Nolan T., Pfaffl M.W., Shipley G.L., Vandesompele J., Wittwer C.T., Bustin S.A. // Clin. Chem. 2013. V. 59. P. 892–902. https://doi.org/10.1373/clinchem.2013.206375
Salipante S.J., Jerome K.R. // Clin. Chem. 2020. V. 66. P. 117–123. https://doi.org/10.1373/clinchem.2019.304048
Khorosheva E.M., Karymov M.A., Selck D.A., Ismagilov R.F. // Nucleic Acids Res. 2016. V. 44. P. e10. https://doi.org/10.1093/nar/gkv877
Schuler F., Siber C., Hin S., Wadle S., Paust N., Zengerle R., von Stetten F. // Anal. Methods. 2016. V. 8. P. 2750–2755. https://doi.org/10.1039/C6AY00600K
Wong Y.-P., Othman S., Lau Y.-L., Radu S., Chee H.-Y. // J. Appl. Microbiol. 2018. V. 124. P. 626–643. https://doi.org/10.1111/jam.13647
Land K.J., Boeras D.I., Chen X.-S., Ramsay A.R., Peeling R.W. // Nat. Microbiol. 2019. V. 4. P. 46–54. https://doi.org/10.1038/s41564-018-0295-3
Coole J., Kortum A., Tang Y., Vohra I., Maker Y., Kundrod K., Natoli M., Richards-Kortum R. // J. Vis. Exp. 2021. V. 168. P. 1–14. https://doi.org/10.3791/62148
Мезин А.В., Наумов С.А., Прусаков К.А., Назаренко К.А., Алдаров К.Г., Бажутов М.Н., Наумов А.Ю., Ильина Е.Н., Говорун В.М., Басманов Д.В. // Патент RU 210215 U1, 2022.
Phillips E.A., Moehling T.J., Ejendal K.F.K., Hoilett O.S., Byers K.M., Basing L.A., Jankowski L.A., Bennett J.B., Lin L.-K., Stanciu L.A., Linnes J.C. // Lab Chip. 2019. V. 19. P. 3375–3386. https://doi.org/10.1039/c9lc00506d
Priye A., Bird S.W., Light Y.K., Ball C.S., Negrete O.A., Meagher R.J. // Sci. Rep. 2017. V. 7. P. 44778. https://doi.org/10.1038/srep44778
Fu Y., Duan X., Huang J., Huang L., Zhang L., Cheng W., Ding S., Min X. // Sci. Rep. 2019. V. 9. P. 5955. https://doi.org/10.1038/s41598-019-42542-x
Ding X., Wang G., Mu Y. // Anal. Chim. Acta. 2019. V. 1081. P. 193–199. https://doi.org/10.1016/j.aca.2019.07.055
Mitani Y., Lezhava A., Kawai Y., Kikuchi T., Oguchi-Katayama A., Kogo Y., Itoh M., Miyagi T., Takakura H., Hoshi K., Kato C., Arakawa T., Shibata K., Fukui K., Masui R., Kuramitsu S., Kiyotani K., Chalk A., Tsunekawa K., Murakami M., Kamataki T., Oka T., Shimada H., Cizdziel P.E., Hayashizaki Y. // Nat. Methods. 2007. V. 4. P. 257–262. https://doi.org/10.1038/nmeth1007
Itonaga M., Matsuzaki I., Warigaya K., Tamura T., Shimizu Y., Fujimoto M., Kojima F., Ichinose M., Murata S.-I. // PLoS One. 2016. V. 11. P. e0151654. https://doi.org/10.1371/journal.pone.0151654
Cao G., Kong J., Xing Z., Tang Y., Zhang X., Xu X., Kang Z., Fang X., Guan M. // Anal. Chim. Acta. 2018. V. 1024. P. 123–135. https://doi.org/10.1016/j.aca.2018.04.022
Paul R., Ostermann E., Wei Q. // Biosens. Bioelectron. 2020. V. 169. P. 112592. https://doi.org/10.1016/j.bios.2020.112592
Mason M.G., Botella J.R. // Nat. Protoc. 2020. V. 15. P. 3663–3677. https://doi.org/10.1038/s41596-020-0392-7
Nagura-Ikeda M., Imai K., Tabata S., Miyoshi K., Murahara N., Mizuno T., Horiuchi M., Kato K., Imoto Y., Iwata M., Mimura S., Ito T., Tamura K., Kato Y. // J. Clin. Microbiol. 2020. V. 58. P. e01438-20. https://doi.org/10.1128/jcm.01438-20
Fakheran O., Dehghannejad M., Khademi A. // Infect. Dis. Poverty. 2020. V. 9. P. 100. https://doi.org/10.1186/s40249-020-00728-w
Kellner M.J., Koob J.G., Gootenberg J.S., Abudayyeh O.O., Zhang F. // Nat. Protoc. 2019. V. 14. P. 2986–3012. https://doi.org/10.1038/s41596-019-0210-2
Gootenberg J.S., Abudayyeh O.O., Lee J.W., Essletzbichler P., Dy A.J., Joung J., Verdine V., Donghia N., Daringer N.M., Freije C.A., Myhrvold C., Bhattacharyya R.P., Livny J., Regev A., Koonin E.V., Hung D.T., Sabeti P.C., Collins J.J., Zhang F. // Science. 2017. V. 356. P. 438–442. https://doi.org/10.1126/science.aam9321
Li S.-Y., Cheng Q.-X., Wang J.-M., Li X.-Y., Zhang Z.-L., Gao S., Cao R.-B., Zhao G.-P., Wang J. // Cell Discov. 2018. V. 4. P. 20. https://doi.org/10.1038/s41421-018-0028-z
Chen J.S., Ma E., Harrington L.B., Da Costa M., Tian X., Palefsky J.M., Doudna J.A. // Science. 2018. V. 360. P. 436–439. https://doi.org/10.1126/science.aar6245
Butler D., Mozsary C., Meydan C., Foox J., Rosiene J., Shaiber A., Danko D., Afshinnekoo E., MacKay M., Sedlazeck F.J., Ivanov N.A., Sierra M., Pohle D., Zietz M., Gisladottir U., Ramlall V., Sholle E.T., Schenck E.J., Westover C.D., Hassan C., Ryon K., Young B., Bhattacharya C., Ng D.L., Granados A.C., Santos Y.A., Servellita V., Federman S., Ruggiero P., Fungtammasan A., Chin C.S., Pearson N.M., Langhorst B.W., Tanner N.A., Kim Y., Reeves J.W., Hether T.D., Warren S.E., Bailey M., Gawrys J., Meleshko D., Xu D., Couto-Rodriguez M., Nagy-Szakal D., Barrows J., Wells H., O’Hara N.B., Rosenfeld J.A., Chen Y., Steel P.A.D., Shemesh A.J., Xiang J., Thierry-Mieg J., Thierry-Mieg D., Iftner A., Bezdan D., Sanchez E., Campion T.R., Jr., Sipley J., Cong L., Craney A., Velu P., Melnick A.M., Shapira S., Hajirasouliha I., Borczuk A., Iftner T., Salvatore M., Loda M., Westblade L.F., Cushing M., Wu S., Levy S., Chiu C., Schwartz R.E., Tatonetti N., Rennert H., Imielinski M., Mason C.E. // Nat. Commun. 2021. V. 12. P. 1660. https://doi.org/10.1038/s41467-021-21361-7
Vandenberg O., Martiny D., Rochas O., van Belkum A., Kozlakidis Z. // Nat. Rev. Microbiol. 2021. V. 19. P. 171–183. https://doi.org/10.1038/s41579-020-00461-z
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


