Биологические мембраны: Журнал мембранной и клеточной биологии, 2022, T. 39, № 3, стр. 186-194
Латеральное взаимодействие цилиндрических трансмембранных пептидов в одномерном приближении
О. В. Кондрашов a, С. А. Акимов a, *
a Институт физической химии и электрохимии им. А.Н. Фрумкина РАН
119071 Москва, Россия
* E-mail: akimov_sergey@mail.ru
Поступила в редакцию 12.01.2022
После доработки 03.03.2022
Принята к публикации 04.03.2022
- EDN: CUSUWX
- DOI: 10.31857/S0233475522030069
Аннотация
Различные мембранные включения могут индуцировать деформации бислойных липидных мембран. Характерная длина распространения деформаций по мембране составляет несколько нанометров. Перекрывание деформаций, индуцированных различными мембранными включениями, приводит к их эффективному латеральному взаимодействию. Энергия взаимодействия может быть рассчитана в рамках адекватной теории упругости. Однако практически такой расчет может быть проведен в аналитическом виде только для эффективно одномерных систем, например, обладающих трансляционной или вращательной симметрией. В общем случае систем с низкой симметрией задача аналитически не решается. Мы теоретически рассмотрели взаимодействие двух цилиндрических трансмембранных пептидов, опосредованное деформациями мембраны. Энергии взаимодействия были получены путем численной минимизации функционала упругой энергии. Кроме того, мы рассчитали энергии взаимодействия в одномерном приближении, предполагая, что система обладает трансляционной симметрией. Было показано, что одномерное приближение достаточно хорошо воспроизводит результаты точного численного расчета в липидных бислоях различной толщины и жесткости.
ВВЕДЕНИЕ
Бислойные липидные мембраны являются основным структурным элементом клеточных мембран. За счет чрезвычайно низкой проницаемости, мембраны выполняют в клетках барьерную функцию, изолируя содержимое клеточных органелл от цитоплазмы и цитоплазмы от окружающей среды. Барьерные свойства мембран позволяют поддерживать неравномерное пространственное распределение различных веществ и электрических потенциалов, необходимое для жизнедеятельности клеток, являющихся открытыми неравновесными системами.
Обмен клетки с окружающей средой небольшими молекулами и ионами обеспечивается встроенными в мембрану специализированными каналами и обменниками (пассивный транспорт) или насосами (активный транспорт). Проникновение в клетку или выход из нее крупных молекул или надмолекулярных объектов происходит посредством эндоцитоза и экзоцитоза, соответственно. Примечательно, что при экзоцитозе и эндоцитозе барьерная функция мембран не нарушается: как правило, в этих процессах цитоплазма и окружающая среда ни в какой момент времени не бывают непрерывно связаны, т.е. слияние (экзоцитоз) и деление (эндоцитоз) мембран в норме происходят без утечки [1–3]. Отсутствие утечки подразумевает тонкую перестройку липидного матрикса в этих процессах, сопровождающуюся сильными деформациями мембран [1–5]. Энергия, необходимая для реализации деформаций, обеспечивается работой специализированных белков слияния и деления. Характерная величина энергии деформаций, как правило, настолько велика, что именно она определяет высоты активационных барьеров и, соответственно, скорость протекания процессов [1, 3, 6].
Энергия деформации может быть количественно оценена в рамках адекватной теории упругости. По-видимому, первая теория упругости липидных мембран была разработана Хельфрихом в 1973 году [7]. В ней учитывалась только деформация, аналогичная поперечному изгибу в смектических жидких кристаллах, а также вклад гауссовой кривизны, являющийся топологическим инвариантом. Деформации считались малыми, и энергия вычислялась в квадратичном приближении (закон Гука). По мере развития экспериментальной техники становились доступны для наблюдения дополнительные деформационные моды. Оказалось, что для описания промежуточных структур процесса слияния мембран необходимо учитывать деформацию наклона, при которой оси липидных молекул отклоняются от нормали к поверхности липидного монослоя. Деформация наклона была введена в теорию упругости мембран в работе Хамма и Козлова [8]. Более строгий учет вкладов деформаций во втором порядке был сделан в работе Терци и Дезерно [9], в которой было введено перекрестное слагаемое по деформациям наклона и поперечному изгибу. Однако полученный функционал энергии не мог описывать стабильную упругую систему, поскольку он не являлся положительно определенной квадратичной формой малых деформаций [10], и упругая энергия, в принципе, могла понижаться при бесконечном увеличении амплитуды деформаций. Для получения функционала упругой энергии, стабильного в указанном смысле, необходимо дополнительно учитывать вклад, квадратичный по градиенту деформации поперечного изгиба [10]. При использовании функционалов упругой энергии различной степени полноты и точности вычисляется энергия деформаций липидных монослоев. При этом деформирующие мембрану белки и пептиды сами, как правило, считаются твердыми и недеформируемыми. В рамках теории упругости белки и пептиды учитываются через условия на их границе с липидным монослоем, которые налагаются на деформации мембраны, например на толщину бислоя, ориентацию липидных молекул, краевой угол поверхности липидного монослоя на границе с белком и т.д. [5, 11, 12].
Периферические белки и амфипатические пептиды частично встраиваются в липидный монослой, раздвигая в стороны полярные головки липидов [13, 14]. Трансмембранные белки и пептиды с приблизительно цилиндрической формой трансмембранной части деформируют липидный бислой, если длина трансмембранного участка не совпадает с толщиной гидрофобной части мембраны [15–17]. Когда твердые мембранные включения расположены далеко друг от друга, вызываемые ими деформации мембраны независимы, и их энергия аддитивна. При сближении включений деформации перекрываются, что приводит к эффективному латеральному взаимодействию; характерная длина затухания деформаций в мембране составляет несколько нанометров [12, 18, 19].
Для расчета энергии деформаций обычно используются квадратичные функционалы упругой энергии, применимые лишь в случае малых деформаций [7–10]. Для нахождения пространственного распределения деформаций функционал упругой энергии минимизируется, что приводит к системе линейных дифференциальных уравнений, которая решается в заданных граничных условиях. Для систем, обладающих трансляционной или вращательной симметрией, т.е. являющихся практически одномерными, минимизация функционала упругой энергии приводит к системе обыкновенных линейных дифференциальных уравнений, решение которой может быть получено в аналитическом виде [11, 12]. Однако в случае низкой симметрии рассматриваемой системы получаются уравнения в частных производных, которые, как правило, не удается решить аналитически. В частности, даже простейшая задача о нахождении энергии взаимодействия двух цилиндрических трансмембранных пептидов аналитически не решается [11]. В таких случаях функционал упругой энергии может быть минимизирован численно для каждого заданного набора граничных условий.
Ранее было показано, что латеральное взаимодействие двух частично встроенных в мембрану амфипатических пептидов может быть с хорошей точностью описано в рамках одномерного приближения [12]. В этом приближении предполагается, что оси пептидов параллельны друг другу, длина пептидов бесконечна и система обладает трансляционной симметрией вдоль направления осей пептидов. В такой эффективно одномерной системе энергия деформаций мембраны, отнесенная к единице длины вдоль направления трансляционной симметрии, может быть рассчитана аналитически. Для получения абсолютной величины энергии деформаций мембраны, индуцируемых пептидами конечной длины, необходимо погонную энергию, рассчитанную в одномерном приближении, умножить на эффективную “деформационную” длину пептида. Если оси пептидов практически параллельны друг другу, то “деформационная” длина превышает фактическую длину пептида приблизительно на 1 нм [12]. Если же оси пептидов существенно не параллельны и пересекаются под значительным углом, условно от π/4 до 3π/4, то эффективная “деформационная” длина приблизительно равна размеру (диаметру) пептида в направлении, перпендикулярном его продольной оси. Данное одномерное приближение оказывается близко к подходу Дерягина для расчета энергии взаимодействия, опосредованного быстро (экспоненциально) монотонно убывающими силами в конфигурациях с относительно низкой симметрией [20].
В настоящей работе мы рассмотрели латеральное взаимодействие двух цилиндрических трансмембранных пептидов, опосредованное упругими деформациями мембраны. В данной системе распределение деформаций и их энергия не могут быть получены в аналитическом виде, поэтому мы находили их путем численной минимизации функционала упругой энергии. Энергия взаимодействия двух трансмембранных пептидов была рассчитана аналитически в одномерном приближении в предположении, что система обладает трансляционной симметрией в направлении, перпендикулярном отрезку, соединяющему центры двух пептидов. Мы показали, что взаимодействие цилиндрических трансмембранных пептидов достаточно точно описывается в одномерном приближении.
ПОСТАНОВКА И РЕШЕНИЕ ЗАДАЧИ
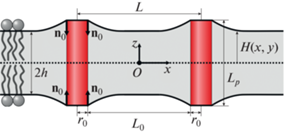
Энергию деформаций мембраны будем рассчитывать в рамках теории упругости Хамма–Козлова [8], обобщенной для учета дополнительных деформационных мод [11, 12]. Состояние деформированного участка липидного монослоя определяется векторным полем единичных векторов n, называемых директорами, характеризующими среднюю ориентацию липидных молекул. Поле директоров задается на некоторой поверхности, называемой нейтральной, проходящей внутри монослоя параллельно его границе с водой в области сочленения полярных головок с гидрофобными цепями липидов. Форма нейтральной поверхности характеризуется векторным полем ее единичных нормалей N. Введем декартову систему координат Oxyz таким образом, чтобы плоская межмонослойная поверхность в исходном недеформированном состоянии совпадала с плоскостью Oxy, ось Oz была перпендикулярна плоскости недеформированной мембраны, начало координат O было расположено в середине отрезка, соединяющего центры трансмембранных пептидов, а ось Ox была направлена вдоль этого отрезка (рис. 1). Монослой, находящийся в области z > 0 будем называть верхним, а в области z < 0 – нижним. Форму нейтральной поверхности верхнего монослоя будем задавать z-координатами ее точек, функцией H(x, y) (рис. 1). Будем учитывать следующие деформации: 1) поперечный изгиб, характеризующийся дивергенцией директора вдоль нейтральной поверхности, div(n); 2) наклон, характеризующийся вектором наклона t = n – N; 3) латеральное растяжение-сжатие, характеризующееся относительным изменением площади нейтральной поверхности, α = (a – a0)/a0, где a, a0 – текущая и исходная площади нейтральной поверхности, приходящиеся на одну липидную молекулу, соответственно; 4) гауссову кривизну $K = \frac{{\partial {{n}_{x}}}}{{\partial x}}\frac{{\partial {{n}_{y}}}}{{\partial y}} - \frac{{\partial {{n}_{x}}}}{{\partial y}}\frac{{\partial {{n}_{y}}}}{{\partial x}}$ где nx, ny – проекции директора на соответствующие оси; 5) кручение, характеризующееся ротором директора, rot(n); 6) латеральное натяжение, определяющее работу, которую необходимо совершить для увеличения площади липидного монослоя, пропорциональную [grad(H(x, y))]2. Деформации считаются малыми, и энергия записывается во втором порядке по ним следующим образом [11, 12]:
(1)
$\begin{gathered} W = \int {dS\left\{ {\frac{B}{2}{{{\left[ {\operatorname{div} \left( {\mathbf{n}} \right) + {{J}_{0}}} \right]}}^{2}} - \frac{B}{2}J_{0}^{2}} \right.} + \\ + \,\,\frac{{{{K}_{t}}}}{2}{{{\mathbf{t}}}^{2}} + \frac{\sigma }{2}{{\left[ {{\mathbf{grad}}\left( H \right)} \right]}^{2}} + \\ \left. { + \,\,\frac{{{{K}_{a}}}}{2}{{\alpha }^{2}} + {{K}_{G}}K + \frac{{{{K}_{{{\text{rot}}}}}}}{2}{{{\left[ {{\mathbf{rot}}\left( {\mathbf{n}} \right)} \right]}}^{2}}} \right\}, \\ \end{gathered} $(3)
$\begin{gathered} W = \int {dS\left\{ {\frac{B}{2}{{{\left[ {\operatorname{div} \left( {\mathbf{n}} \right) + {{J}_{0}}} \right]}}^{2}} - \frac{B}{2}J_{0}^{2}} \right.} + \\ + \,\,\frac{{{{K}_{t}}}}{2}{{\left[ {{\mathbf{n}} - {\mathbf{grad}}\left( H \right)} \right]}^{2}} + \frac{\sigma }{2}{{\left[ {{\mathbf{grad}}\left( H \right)} \right]}^{2}} + \\ + \,\,\frac{{{{K}_{a}}}}{{2{{h}^{2}}}}{{\left[ {h - \frac{{{{h}^{2}}}}{2}\operatorname{div} \left( {\mathbf{n}} \right) - H} \right]}^{2}} + \\ \left. { + \,\,{{K}_{G}}K + \frac{{{{K}_{{{\text{rot}}}}}}}{2}{{{\left[ {{\mathbf{rot}}\left( {\mathbf{n}} \right)} \right]}}^{2}}} \right\}. \\ \end{gathered} $(4)
${\mathbf{n}}\left( \infty \right) = {\mathbf{N}}\left( \infty \right) = {{\left( {0,0, - 1} \right)}^{T}},\,\,\,\,H\left( \infty \right) = h.$(5)
${{n}_{n}}(r = {{r}_{0}}) = 0,\,\,\,\,{{n}_{t}}(r = {{r}_{0}}) = 0,\,\,\,\,H(r = {{r}_{0}}) = {{{{L}_{p}}} \mathord{\left/ {\vphantom {{{{L}_{p}}} 2}} \right. \kern-0em} 2},$Рис. 1.
Мембрана с двумя трансмембранными пептидами. Пептиды радиуса r0 и длины Lp расположены на расстоянии L (между центрами) друг от друга; расстояние между границами пептидов L0 = L – 2r0. Толщина гидрофобной части бислоя 2h. Введена декартова система координат, начало O которой расположено в плоскости межмонослойной поверхности посередине отрезка, соединяющего центры пептидов. Форма нейтральной поверхности верхнего монослоя задается функцией H(x, y). На границах пептидов директор n0 вертикален.
Вариация функционала (3) по директору и форме нейтральной поверхности монослоя приводит к системе линейных уравнений в частных производных, которая не может быть решена аналитически при граничных условиях (4), (5). Поэтому минимизация функционала проводилась численно с использованием метода конечных элементов с адаптивной расчетной сеткой, аналогично работам [12, 22, 23]. Плоскость Oxy разбивалась на элементарные треугольники. В каждом треугольнике поля деформаций, определяемые функциями n(x, y) = (nx(x, y), ny(x, y), –1)T и H(x, y), аппроксимировались линейными функциями координат (x, y), т.е. поля деформаций заменялись их линейными интерполянтами, заданными на узлах расчетной сетки (вершинах элементарных треугольников). Линейные интерполянты деформаций подставлялись в функционал упругой энергии (3), и поверхностная плотность энергии аналитически интегрировалась по площади каждого элементарного треугольника. Далее бралась сумма энергий по всем треугольным ячейкам расчетной сетки, т.е. по всей нейтральной поверхности липидного монослоя, и получалась полная упругая энергия монослоя Wtotal. В это выражение для Wtotal квадратично входят неизвестные значения деформационных полей в узлах расчетной сетки. Чтобы получить численное значение полной упругой энергии, функция Wtotal аналитически минимизировалась по значениям полей деформации в узлах сетки, за исключением тех, которые задаются граничными условиями. Граничные условия (4) при x, y → ∞ фактически задавались на расстоянии не менее 25 нм от трансмембранных пептидов. Это расстояние существенно превышает характерные длины затухания деформаций мембраны, которые обычно составляют несколько нанометров [12, 22].
Для повышения точности приближенного расчета энергии использовались неоднородные сетки: поверхностная плотность узлов расчетной сетки в окрестности трансмембранных пептидов была повышенной. Границы трансмембранных пептидов в верхнем и нижнем монослоях представлялись в виде кусочно-линейных функций. Окрестность границы пептида разделялась на пять областей; каждая область определялась неравенством ri – 1 ≤ d ≤ ri, где d – расстояние до границы пептида; ri – 1, ri – константы, определяющие внутреннюю и внешнюю границы областей соответственно для i = 1, 2, 3, 4, 5. Численные значения ri были следующими: r0 = 0; r1 = 1 нм; r2 = = 1.5 нм; r3 = 4 нм; r4 = 11 нм; r5 = ∞. Максимальная площадь элементарного треугольника расчетной сетки ограничивалась величиной 0.5 γ (в нм2). Введенные пять областей разбивались на элементарные треугольники, площадь которых не превышала γ θi, где θ1 = 0.01, θ2 = 0.02, θ3 = 0.04, θ4 = = 0.05, θ5 = 0.5. Этот алгоритм позволяет получить численное значение Wtotal для каждого конкретного значения γ, устанавливаемого вручную. Для повышения точности расчетов величина энергии Wtotal рассчитывалась при пяти последовательно уменьшающихся значениях γ = 1.5; 1.25; 1.05; 0.85; 0.62. Затем полученная таким образом зависимость Wtotal от γ аппроксимировалась полиномом второй степени и экстраполировалась на нулевой размер ячейки расчетной сетки, γ → 0. Однако оказалось, что значение Wtotal, рассчитанное для самой грубой сетки, соответствующей γ = 1.5, отличается от величины Wtotal, получаемой в результате экстраполяции при γ → 0, не более чем на 1.5%. В связи с этим большая часть расчетов проводились на самой грубой сетке γ = 1.5. В результате были получены зависимости энергии деформаций мембраны от расстояния между двумя цилиндрическими трансмембранными пептидами.
В одномерном приближении в предположении о трансляционной симметрии системы вдоль оси Oy все деформации зависят только от координаты x, и функционал упругой энергии (3) может быть переписан в следующем виде:
(6)
$\begin{gathered} W = \int {dS\left\{ {\frac{B}{2}{{{\left[ {\frac{{dn}}{{dx}} + {{J}_{0}}} \right]}}^{2}} - \frac{B}{2}J_{0}^{2}} \right.} + \\ + \,\,\frac{{{{K}_{t}}}}{2}{{\left[ {n - \frac{{dH}}{{dx}}} \right]}^{2}} + \frac{\sigma }{2}{{\left[ {\frac{{dH}}{{dx}}} \right]}^{2}} + \\ \left. { + \,\,\frac{{{{K}_{a}}}}{{2{{h}^{2}}}}{{{\left[ {h - \frac{{{{h}^{2}}}}{2}\frac{{dn}}{{dx}} - H} \right]}}^{2}}} \right\}, \\ \end{gathered} $(7)
$\begin{gathered} \left( {\frac{B}{{{{K}_{t}}}} + \frac{{{{K}_{a}}{{h}^{2}}}}{{4{{K}_{t}}}}} \right)n{\kern 1pt} ''\, - n + \left( {1 + \frac{{{{K}_{a}}}}{{2{{K}_{t}}}}} \right)H{\kern 1pt} ' = 0, \\ h\left( {1 + \frac{\sigma }{{{{K}_{t}}}}} \right)H{\kern 1pt} ''\, - \frac{{{{K}_{a}}}}{{h{{K}_{t}}}}H - h\left( {1 + \frac{{{{K}_{a}}}}{{2{{K}_{t}}}}} \right)n{\kern 1pt} ''\, + \frac{{{{K}_{a}}}}{{{{K}_{t}}}} = 0, \\ \end{gathered} $(8)
$\begin{gathered} {{h}^{2}}\left( {4{{l}^{2}} + A{{h}^{2}}} \right){{n}^{{\left( 4 \right)}}} + \\ + \,\,4\left( {A{{h}^{2}} - A{{l}^{2}} - {{h}^{2}}s} \right)n{\kern 1pt} ''\, + An = 0, \\ \end{gathered} $(9)
$n(x) = {{c}_{1}}{{e}^{{qx}}} + {{c}_{2}}{{e}^{{ - qx}}} + {{c}_{3}}{{e}^{{px}}} + {{c}_{4}}{{e}^{{ - px}}},$(10)
$\begin{gathered} q = \sqrt {\frac{{2\left( {A{{l}^{2}} + {{h}^{2}}s - A{{h}^{2}} - \sqrt {{{A}^{2}}\left( {{{l}^{4}} - 2{{l}^{2}}{{h}^{2}} - {{h}^{4}}s} \right) - 2A{{h}^{2}}\left( {{{h}^{2}}s + {{l}^{2}}s + 2{{l}^{2}}} \right) + {{h}^{4}}s} } \right)}}{{h\left( {1 + s} \right)(4{{l}^{2}} + A{{h}^{2}})}}} , \\ p = \sqrt {\frac{{2\left( {A{{l}^{2}} + {{h}^{2}}s - A{{h}^{2}} + \sqrt {{{A}^{2}}\left( {{{l}^{4}} - 2{{l}^{2}}{{h}^{2}} - {{h}^{4}}s} \right) - 2A{{h}^{2}}\left( {{{h}^{2}}s + {{l}^{2}}s + 2{{l}^{2}}} \right) + {{h}^{4}}s} } \right)}}{{h\left( {1 + s} \right)(4{{l}^{2}} + A{{h}^{2}})}}} . \\ \end{gathered} $(11)
$\begin{gathered} H = \frac{{4{{l}^{2}}{{q}^{2}} + A{{h}^{2}}{{q}^{2}} - 4}}{{2q\left( {2 + A} \right)}}\left( {{{c}_{1}}{{e}^{{qx}}} - {{c}_{2}}{{e}^{{ - qx}}}} \right) + \\ + \,\,\frac{{4{{l}^{2}}{{p}^{2}} + A{{h}^{2}}{{p}^{2}} - 4}}{{2p\left( {2 + A} \right)}}\left( {{{c}_{3}}{{e}^{{px}}} - {{c}_{4}}{{e}^{{ - px}}}} \right). \\ \end{gathered} $Четыре комплексных коэффициента c1, c2, c3, c4 эквивалентны восьми действительным. Однако при наложении условия вещественности функций n(x) и H(x) при любом действительном x остается только четыре независимых действительных коэффициента, которые следует определить из граничных условий. В одномерном приближении граничные условия ставятся следующим образом:
(12)
$n({{{{L}_{0}}} \mathord{\left/ {\vphantom {{{{L}_{0}}} 2}} \right. \kern-0em} 2}) = 0,\,\,\,\,H({{{{L}_{0}}} \mathord{\left/ {\vphantom {{{{L}_{0}}} 2}} \right. \kern-0em} 2}) = {{{{L}_{p}}} \mathord{\left/ {\vphantom {{{{L}_{p}}} {2,}}} \right. \kern-0em} {2,}}\,\,\,\,n(0) = 0,$РЕЗУЛЬТАТЫ
Расчеты энергии деформаций мембраны, индуцированных двумя трансмембранными цилиндрическими пептидами, были проведены при следующих параметрах мембран и пептидов. Длина трансмембранной части пептидов считалась равной Lp = 3.6 нм; радиус трансмембранного пептида – r0 = 0.65 нм, что приблизительно соответствует радиусу α-спирали. Были рассмотрены бислои четырех различных толщин: 2h = 3.0, 3.2, 4.0, 4.2 нм; при 2h = Lp = 3.6 нм пептиды не деформируют мембрану. При каждой толщине бислоя были рассмотрены случаи “мягкой” (обозначаемой индексом “s”) и “жесткой” (обозначаемой индексом “r”) мембран, характеризующихся двумя различными наборами модулей упругости: модули поперечного изгиба Bs = 10 kBT [23], Br = = 5Bs = 50 kBT; модули латерального растяжения-сжатия Ks = 133 мН/м [23], Kr = 5Ks = 665 мН/м. Модули гауссовой кривизны и кручения выражались через модуль поперечного изгиба: KG = –B/2 [24], Krot = B/2 [12, 22]. Модуль наклона определяется величиной поверхностного натяжения границы раздела вода – гидрофобные цепи липидов и должен слабо зависеть от конкретной химической структуры и состояния липида; он считался равным Kt = 40 мН/м [8]. Латеральное натяжение считалось равным σ = 0.1 мН/м; получаемые результаты слабо зависят от его конкретной величины. Спонтанная кривизна дает постоянный вклад в упругую энергию, не зависящий от расстояния между пептидами [22]. Поскольку мы рассматриваем энергии взаимодействия, отсчитываемые от энергии состояния бесконечно удаленных друг от друга пептидов (см. (13)), постоянные вклады в упругую энергию не учитывались. Функционал упругой энергии (3) минимизировался методом конечных элементов при следующих дискретных расстояниях между центрами пептидов: L = {1.4, 1.7, 2.0, 2.4, 2.8, 3.2, 3.6, 4.0, 4.5, 5.0, 5.5, 6.0, 6.5, 7.0, 7.5, 8.0, 8.5, 9.0, 9.5, 10.0, 11.0, 12.0, 13.0, 14.0, 15.0, 16.0, 18.0, 20.0, 24.0, 28.0} нм. При аппроксимации расчетные энергии при первых трех расстояниях брались с уменьшенным весом.
Результаты расчетов энергии взаимодействия двух цилиндрических трансмембранных пептидов методом конечных элементов и в рамках одномерного приближения приведены на рис. 2. Значения энергии, полученные численно методом конечных элементов, аппроксимировались кривыми, рассчитанными в одномерном приближении согласно соотношению (14). В случае, когда Lp = 2h = 3.6 нм, пептиды не деформируют мембрану и энергия взаимодействия пептидов постоянна и равна нулю. При Lp ≠ 2h возникают упругие деформации мембраны, которые опосредуют латеральное взаимодействие пептидов. При всех значениях параметров пептиды отталкиваются (энергия взаимодействия положительна) на больших расстояниях (L > 4–6 нм) и притягиваются (энергия взаимодействия отрицательна) на малых расстояниях (L < 4 нм) (рис. 2). Индуцируемые пептидами деформации затухают на расстояниях порядка нескольких нанометров, и при L > > 10 нм энергия взаимодействия становится пренебрежимо малой (рис. 2). Таким образом, для сближения пептидов необходимо преодолеть некоторый энергетический барьер, высота которого практически не зависит от жесткости мембраны (ср. соответствующие кривые на рис. 2а и 2б), но зависит от разности (Lp – 2h). Положение вершины барьера смещается вправо приблизительно на 1 нм при увеличении толщины бислоя с 2h = = 3.0 нм до 2h = 4.2 нм (рис. 2).
Рис. 2.
Энергии взаимодействия двух цилиндрических трансмембранных пептидов, опосредованного деформациями мембраны: а – в мягкой мембране, б – в жесткой мембране. Черные круги соответствуют точкам, полученным численно методом конечных элементов; сплошные кривые получены в одномерном приближении и преобразованы в соответствии с соотношением (14) для аппроксимации результатов численных расчетов. Длина пептида Lp = 3.6 нм; толщина гидрофобной части бислоя 2h = 3.0 нм (зеленые кривые); 3.2 нм (синие кривые); 4.0 нм (серые кривые); 4.2 нм (красные кривые). Потенциалы были сдвинуты на постоянную величину (2 kBT – зеленые кривые, 3 kBT – синие кривые, 4 kBT – серые кривые, 6 kBT – красные кривые) для лучшей иллюстрации. Горизонтальные штриховые линии соответствуют потенциалу взаимодействия при L → ∞. Черные вертикальные штриховые линии соответствуют расстоянию L = 2r0, при котором пептиды касаются друг друга.
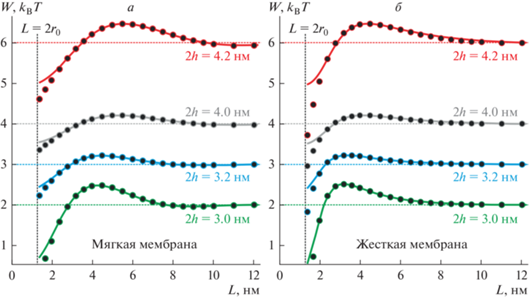
Из графиков (рис. 2) видно, что одномерное приближение достаточно хорошо описывает взаимодействие цилиндрических трансмембранных пептидов при расстояниях между центрами пептидов L ≥ 2 нм независимо от жесткости и толщины мембраны. При расстоянии между центрами L = 2 нм расстояние между границами пептидов составляет L0 = L – 2r0 = 0.7 нм, что приблизительно соответствует латеральному размеру одной липидной молекулы. Применение теории упругости сплошной среды для описания состояния участка мембраны между пептидами при меньших расстояниях между ними, вообще говоря, не вполне корректно, особенно в одномерном приближении. Параметры Deff , α и β аппроксимации энергий взаимодействия, полученных численно методом конечных элементов, кривыми, рассчитанными в одномерном приближении согласно соотношению (14), приведены в табл. 1.
Таблица 1.
Параметры аппроксимации энергий взаимодействия, полученных численно методом конечных элементов, кривыми, рассчитанными в одномерном приближении (Deff, α, β). Характерные длины затухания (ld) и осцилляций (lo) деформаций мембраны
| 2h | Мягкая мембрана | Жесткая мембрана | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Deff | α | β | ld | lo | Deff | α | β | ld | lo | |
| 3.0 | 0.69 | 0.88 | 0.45 | 2.25 | 1.39 | 0.34 | 1.09 | 0.15 | 1.19 | 11.7 |
| 3.2 | 0.70 | 0.88 | 0.50 | 2.50 | 1.44 | 0.33 | 1.11 | 0.16 | 1.32 | 3.78 |
| 4.0 | 0.72 | 0.87 | 0.72 | 3.64 | 1.67 | 0.32 | 1.13 | 0.35 | 1.94 | 2.42 |
| 4.2 | 0.72 | 0.85 | 0.80 | 3.96 | 1.72 | 0.32 | 1.13 | 0.41 | 2.11 | 2.38 |
Из табл. 1 видно, что параметры аппроксимации Deff и α практически не зависят от толщины бислоя. В настоящей работе мы фиксировали длину трансмембранного пептида (Lp = 3.6 нм) и варьировали толщину бислоя. Независимость Deff и α от h позволяет применять разработанный подход в другой постановке: при фиксированной толщине бислоя можно аналогичным образом рассматривать взаимодействие цилиндрических трансмембранных пептидов различной длины. При увеличении жесткости мембраны в 5 раз параметр α меняется слабо (с ∼0.9 до ∼1.1), однако Deff уменьшается приблизительно в 2 раза, c ∼0.70 до ∼0.33. В подходе Дерягина [20] для приближенного расчета энергии взаимодействия кривых поверхностей на основе одномерного приближения эффективная длина leff, по которой взаимодействуют кривые поверхности, в наших обозначениях определяется выражением:
где ld – характерная длина затухания сил, опосредующих взаимодействие. В табл. 1 приведены значения длин затухания (ld) и осцилляций (lo) деформаций мембраны, полученные из (10) как ld = 1/|Re(q)|, lo = 1/|Im(q)|. Из таблицы видно, что Deff не связано напрямую с ld, например, потому, что ld сильно зависит от толщины липидного монослоя, в то время как Deff от нее практически не зависит. Таким образом, несмотря на то что одномерное приближение позволяет достаточно точно находить энергии взаимодействия цилиндрических трансмембранных пептидов (рис. 2), эффективный “деформационный” диаметр пептидов Deff не может быть определен в рамках подхода Дерягина согласно соотношению (15). Это, по-видимому, связано с тем, что leff в виде (15) получается в предположении r0 ⪢ ld, что в нашем случае заведомо не выполняется. Кроме того, подход Дерягина был обоснован для монотонно затухающих с расстоянием сил, опосредующих взаимодействие [20], в то время как деформации мембраны затухают и осциллируют, т.е. затухают немонотонно, поскольку |Im(q)| = 1/lo ≠ 0 (табл. 1).Отметим, однако, что при увеличении жесткости мембраны в 5 раз эффективный “деформационный” диаметр пептидов уменьшился приблизительно в (0.72/0.32) = 2.25 ≈ $\sqrt 5 $ раз, причем практически независимо от толщины бислоя. Таким образом, на основании результатов расчетов, приведенных в табл. 1, можно предварительно предположить, что эффективный диаметр пептидов, определяющий их латеральное взаимодействие в одномерном приближении, должен быть обратно пропорционален корню квадратному из жесткости мембраны. При этом наиболее вероятно, что деформацией, определяющей величину Deff, является поперечный изгиб. Модули поперечного изгиба и латерального растяжения–сжатия имеют разную размерность. Для сравнения “жесткостей” этих деформаций необходимо привести их модули к одной размерности. Можно модуль поперечного изгиба разделить на квадрат характерного параметра липидного монослоя, имеющего размерность длины, т.е., например, на его толщину h. Тогда B/h2 ≈ (10 kBT)/(2 нм)2 = = 0.625 kBT/нм2, в то время как Ka = 133 мН/м ≈ ≈ 32 kBT/нм2, т.е. Ka ⪢ B/h2. Таким образом, деформация латерального растяжения–сжатия значительно более “жесткая”, и ее энергия, соответственно, существенно меньше, чем энергия деформации поперечного изгиба.
Таким образом, в настоящей работе мы обосновали применение одномерного приближения для расчета энергии взаимодействия цилиндрических трансмембранных пептидов в липидных бислоях различной толщины и жесткости. Величины энергии, рассчитанные в одномерном приближении, значительно отклоняются от точных значений, когда расстояние между границами пептидов становится меньше латерального размера липидной молекулы. Однако в таких условиях применение теории упругости сплошной среды в целом не вполне корректно. При больших расстояниях между пептидами одномерное приближение достаточно хорошо воспроизводит результаты точного (численного) расчета. Эффективный “деформационный” диаметр трансмембранного пептида практически не зависит от толщины бислоя и, по-видимому, обратно пропорционален корню квадратному из модуля поперечного изгиба.
Работа выполнена при поддержке Российского научного фонда (грант № 22-24-00834).
Список литературы
Kozlovsky Y., Kozlov M.M. 2003. Membrane fission: Model for intermediate structures. Biophys. J. 85, 85–96.
Martens S., Kozlov M.M., McMahon H.T. 2007. How synaptotagmin promotes membrane fusion. Science. 316, 1205–1208.
Chernomordik L., Kozlov M.M., Zimmerberg J. 1995. Lipids in biological membrane fusion. J. Membr. Biol. 146, 1–14.
Kozlovsky Y., Kozlov M.M. 2002. Stalk model of membrane fusion: Solution of energy crisis. Biophys. J. 82, 882–895.
Pannuzzo M., McDargh Z.A., Deserno M. 2018. The role of scaffold reshaping and disassembly in dynamin driven membrane fission. Elife. 7, e39441.
Kozlovsky Y., Chernomordik L.V., Kozlov M.M. 2002. Lipid intermediates in membrane fusion: Formation, structure, and decay of hemifusion diaphragm. Biophys. J. 83, 2634–2651.
Helfrich W. 1973. Elastic properties of lipid bilayers: Theory and possible experiments. Z. Naturforsch. C. 28, 693–703.
Hamm M., Kozlov M.M. 2000. Elastic energy of tilt and bending of fluid membranes. Eur. Phys. J. E. 3, 323–335.
Terzi M.M., Deserno M. 2017. Novel tilt-curvature coupling in lipid membranes. J. Chem. Phys. 147, 084702.
Pinigin K.V., Kuzmin P.I., Akimov S.A., Galimzyanov T.R. 2020. Additional contributions to elastic energy of lipid membranes: Tilt-curvature coupling and curvature gradient. Phys. Rev. E. 102, 042406.
Kondrashov O.V., Galimzyanov T.R., Pavlov K.V., Kotova E.A., Antonenko Y.N., Akimov S.A. 2018. Membrane elastic deformations modulate gramicidin A transbilayer dimerization and lateral clustering. Biophys. J. 115, 478–493.
Kondrashov O.V., Galimzyanov T.R., Jiménez-Munguía I., Batishchev O.V., Akimov S.A. 2019. Membrane-mediated interaction of amphipathic peptides can be described by a one-dimensional approach. Phys. Rev. E. 99, 022401.
Perrin Jr. B.S., Fu R., Cotten M.L., Pastor R.W. 2016. Simulations of membrane-disrupting peptides II: AMP piscidin 1 favors surface defects over pores. Biophys. J. 111, 1258–1266.
Chen C.H., Wiedman G., Khan A., Ulmschneider M.B. 2014. Absorption and folding of melittin onto lipid bilayer membranes via unbiased atomic detail microsecond molecular dynamics simulation. Biochim. Biophys. Acta. 1838, 2243–2249.
Bories F., Constantin D., Galatola P., Fournier J.B. 2018. Coupling between inclusions and membranes at the nanoscale. Phys. Rev. Lett. 120, 128104.
Lin X., Gorfe A.A., Levental I. 2018. Protein partitioning into ordered membrane domains: Insights from simulations. Biophys. J. 114, 1936–1944.
Park S., Yeom M.S., Andersen O.S., Pastor R.W., Im W. 2019. Quantitative characterization of protein–lipid interactions by free energy simulation between binary bilayers. J. Chem. Theor. Comput. 15, 6491–6503.
Молотковский Р.Ю., Акимов С.А. 2009. Расчет линейного натяжения в различных моделях кромки поры в липидном бислое. Биол. мембраны. 26, 149–158.
Карпунин Д.В., Акимов С.А., Фролов В.А. 2005. Формирование пор в плоских липидных мембранах, содержащих лизолипиды и холестерин. Биол. мембраны. 22, 429–432.
Israelachvili J. 2011. Intermolecular and Surface Forces. New York: Academic Press, ISBN 9 780 123 919 274.
Nagle J.F., Wilkinson D.A. 1978. Lecithin bilayers. Density measurement and molecular interactions. Biophys. J. 23, 159–175.
Kondrashov O.V., Akimov S.A. 2022. Regulation of antimicrobial peptide activity via tuning deformation fields by membrane-deforming inclusions. Int. J. Mol. Sci. 23, 326.
Rawicz W., Olbrich K.C., McIntosh T., Needham D., Evans E. 2000. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophys. J. 79, 328–339.
Hu M., de Jong D.H., Marrink S.J., Deserno M. 2013. Gaussian curvature elasticity determined from global shape transformations and local stress distributions: A comparative study using the MARTINI model. Faraday Discuss. 161, 365–382.
Дополнительные материалы отсутствуют.
Инструменты
Биологические мембраны: Журнал мембранной и клеточной биологии


