Биологические мембраны: Журнал мембранной и клеточной биологии, 2022, T. 39, № 3, стр. 205-214
Ингибитор PI3K и mTOR воксталисиб нарушает сопряжение мускаринового рецептора М3 с мобилизацией Ca2+
О. О. Лямин a, П. Д. Котова a, *, Е. А. Дымова a, П. Ю. Фадеев a, О. А. Рогачевская a, Е. А. Воронова a, С. С. Колесников a
a Институт биофизики клетки РАН, ФИЦ ПНЦБИ РАН
142290 Пущино, Московская обл., Россия
* E-mail: p.d.kotova@gmail.com
Поступила в редакцию 12.12.2021
После доработки 24.01.2022
Принята к публикации 25.01.2022
- EDN: JUEYZV
- DOI: 10.31857/S0233475522030070
Аннотация
Ингибитор PI3K и mTOR киназ воксталисиб считается перспективным лекарственным средством для терапии различных опухолей. Между тем, использование этого соединения в терапевтических целях ассоциируется с рядом побочных эффектов, причины которых не вполне ясны. В данной работе было показано, что в присутствии воксталисиба нарушается внутриклеточная Ca2+-сигнализация, инициируемая ацетилхолином в клетках HEK 293. Данные ингибиторного анализа свидетельствовали о том, что за чувствительность этих клеток к ацетилхолину ответственны преимущественно М3-мускариновые рецепторы и что воксталисиб подавляет Ca2+-ответы на ацетилхолин по механизму, не связанному с ингибированием PI3K и/или mTOR. Результаты физиологических экспериментов свидетельствовали о возможности непосредственного влияния воксталисиба на взаимодействие М3-рецепторов с агонистами. С помощью методов вычислительной биофизики, включая докинг и молекулярную динамику, моделировали взаимодействие М3-рецептора с воксталисибом и другими соединениями, которые были использованы в физиологических экспериментах. Вычислительные эксперименты показали, что воксталисиб способен связываться с ортостерическим сайтом М3-рецептора и тем самым препятствовать его активации агонистом. Представляется, что побочные эффекты, возникающие у пациентов при применении воксталисиба, могут быть частично обусловлены ингибированием холинергической сигнальной системы клеток, экспрессирующих М3-рецептор.
ВВЕДЕНИЕ
Фосфорилирование и дефосфорилирование белков, катализируемые разнообразными киназами и фосфатазами, вовлечены в регуляцию большинства клеточных функций и внутриклеточных процессов, включая сигнальные. Сигнальные каскады, протекающие с участием фосфатидилинозитол-3-киназы (PI3K), Akt-киназы и мишени рапамицина млекопитающих (mTOR), регулируют множество ключевых процессов, в том числе рост, пролиферацию и метаболизм клеток. Кроме того, показано, что PI3K/Akt/mTOR-путь патологически активирован в клетках опухолей различного происхождения, а ингибирование этого пути ведет к регрессии опухолей. В настоящее время в качестве лекарственных средств для противоопухолевой терапии используют ингибиторы сигнальных молекул, вовлеченных в PI3K/Akt/mTOR-сигнализацию [1].
К таким соединениям относится воксталисиб (другие названия этого вещества SAR245409, XL765), ингибирующий одновременно PI3K и mTOR. В данный момент воксталисиб находится на стадии клинических испытаний терапии опухолей различной природы и применяется как самостоятельно, так и в сочетании с другими противоопухолевыми препаратами и радиотерапией. Установлено, что воксталисиб, используемый даже в качестве монотерапии, вызывает многочисленные побочные эффекты, такие как тошнота, диарея, гипергликемия, снижение аппетита, повышенная утомляемость, сыпь, сухость кожи, астения, рвота и нарушение баланса ферментов печени [2]. Побочные эффекты воксталисиба могут быть связаны с тем, что помимо установленного ингибирования PI3K и mTOR, это вещество может также воздействовать и на другие клеточные мишени. Так, например, в предыдущих исследованиях нами было показано, что такие ингибиторы PI3K, как LY294002, LY303511 и PI828, подавляют Ca2+-сигналы, вызываемые агонистами аминергических рецепторов по механизму, не зависящему от ингибирования PI3K, и, по всей вероятности, воздействуя непосредственно на рецепторы [3, 4]. В данной работе в физиологических экспериментах мы исследовали ингибирующее воздействие воксталисиба на Ca2+-сигналы, инициируемые ацетилхолином, а также попытались объяснить природу этого явления, используя методы вычислительной биофизики.
МАТЕРИАЛЫ И МЕТОДЫ
Микрофотометрия. В работе использовали клетки линии HEK 293, которые культивировали в среде DMEM (Gibco), содержащей 4.5 г/л глюкозы, 10% эмбриональной бычьей сыворотки (HyClone), 100 мг/мл гентамицина (Sigma), 2 мМ глутамина (Sigma) во влажной атмосфере c 5% CO2 в воздухе при 37°C. Для эксперимента клетки снимали с культурального пластика 0.25% раствором трипсина (Sigma), а затем прикрепляли ко дну фотометрической камеры с помощью адгезивного материала Cell Tak (Corning). При всех дальнейших манипуляциях клетки находились во внеклеточном растворе, содержащем (мМ): 130 NaCl, 5 KCl, 2 CaCl2, 1 MgCl2, 10 HEPES, pH 7.4, 10 глюкозы (все соли и буфер произведены Sigma). Для загрузки флуоресцентным Ca2+-зондом Fluo-4 клетки инкубировали в присутствии проникающего через мембрану Fluo-4 AM (4 мкМ) и детергента Pluronic (0.02%) (оба Molecular Probes) при комнатной температуре в течение 30 мин. Затем клетки отмывали внеклеточным раствором и выдерживали в нем при комнатной температуре в течение 1 ч.
Фотометрические эксперименты проводили с использованием инвертированного флуоресцентного микроскопа Axiovert 135 (Zeiss), оборудованного объективом Plan NeoFluar 20×/0.75 и цифровой EMCCD камерой LucaR (Andor Technology). Флуоресценцию клеток возбуждали на длине волны 480 ± 10 нм, эмиссию регистрировали в области 520 ± 20 нм, что соответствует спектральным характеристикам Fluo-4. Изменение уровня Са2+ в цитоплазме оценивали по относительному изменению интенсивности флуоресценции Fluo-4 ΔF/F0, где ΔF = F – F0, F и F0 – интенсивность эмиссии Са2+-индикатора в текущий момент времени и в начале регистрации, соответственно. Количественный фотометрический анализ изображений осуществляли с использованием программы NIS Elements (Nikon). Полученные экспериментальные данные обрабатывали с помощью программы Sigma Plot 12.5 (Systat Software Inc).
Все химические соединения, использованные в описанных ниже экспериментах, применяли путем полной замены раствора в фотометрической камере с помощью системы перфузии. В работе использовали: ацетилхолин (Acetylcholine, Tocris), воксталисиб (Voxtalisib, Selleckchem), вортманнин (Wortmannin, Tocris), рапамицин (Rapamycin, InvivoGen) и 4-DAMP (Tocris).
Компьютерное моделирование. При компьютерном моделировании механизмов взаимодействия исследуемых соединений с мускариновым М3-рецептором применяли два метода вычислительной биофизики: докинг – для определения энергетически наиболее выгодных положений исследуемых соединений на рецепторе; молекулярную динамику – для анализа вероятной эволюции во времени предсказанных докингом комплексов.
Для этого использовали 3D-структуру мускаринового M3-рецептора, полученную из базы данных кристаллографических структур RCSB (https://www.rcsb.org) [5], PDB ID: 4U15 [6]. Из исходной структуры предварительно были удалены все небелковые молекулы, в частности антагонист тиотропиум и белковый фрагмент между 5-й и 6-й трансмембранными спиралями, способствующий кристаллизации. Образовавшийся промежуток заполнили аминокислотным линкером GSGSGSGS; выбор последовательности из глицина и серина мотивирован распространенностью этих аминокислот в природных линкерах, электронейтральностью, короткой боковой цепью и практикой использования в молекулярно-динамическом моделировании лиганд-рецепторного взаимодействия [7]. Количество GS-повторов выбрано минимальным, обеспечивающим свободную динамику 5-й и 6-й спиралей рецептора. Положение линкера на концах 5-й и 6-й спиралей смоделировано с помощью SWISS-MODEL [8]. Исходная структура была получена для М3-рецептора крысы (Rattus norvegicus), поэтому в SWISS-MODEL было дополнительно проведено гомологическое моделирование по аминокислотной последовательности рецептора человека, более чем на 90% совпадающей с аминокислотной последовательностью рецептора модельного организма. 3D-структуры соединений ацетилхолин, воксталисиб, вортманнин, рапамицин и 4-DAMP были получены из баз данных ZINC [9] и PubChem [10]. Подготовку всех структур к докингу проводили в AutoDockTools-1.5.6. Докинг осуществляли с помощью Autodock Vina 1.1.2 [11], позволяющего уточнять область на рецепторе, в границах которой надлежит искать наиболее выгодные для образования комплекса конформации лиганда. В качестве такой области было выбрано пространство между трансмембранными спиралями рецептора, ограниченное центром рецептора с одной стороны и внеклеточными петлями с другой. Данный выбор мотивирован наличием в базах данных нескольких экспериментально полученных структур, в которых антагонисты мускаринового рецептора образуют комплекс с рецептором именно в этой области. Ввиду отсутствия кристаллографических структур мускаринового М3-рецептора в комплексе с ацетилхолином, положение ацетилхолина, принимаемое за ортостерический сайт рецептора, также моделировали докингом. Константы диссоциации соединений рассчитывали по формуле Kd = exp(1000ΔG/RT), где ΔG – значение скоринг-функции Autodock Vina 1.1.2, используемое как аппроксимация свободной энергии связи, R – газовая постоянная, равная 1.98 кал/(моль К), и T = = 298.15; К – значение абсолютной температуры, используемое Autodock Vina.
Для устранения возникающих при докинге стерических конфликтов, уточнения конформации лиганда на рецепторе и проверки устойчивости предсказанных комплексов было проведено моделирование методом молекулярной динамики. Молекулярно-динамические расчеты выполняли на видеокартах на базе графических процессоров Nvidia 2080 и Nvidia 3080 при помощи инструмента pmemd.cuda, входящего в программное обеспечение Amber 20 [12]. Предварительно для аминокислотных остатков рецептора были определены формы протонирования. Аспартат в положении 113 был протонирован всегда, подобно аспартату в положении 83 в родопсине, протонированному в течение всего фотоцикла [13]. Расчет состояний остальных аминокислотных остатков был осуществлен с использованием PROPKA [14] при pH 7.0. Для сохранения электронейтральности N- и C-концы содержали ацетильную и N-метиламидную группы соответственно.
Ориентирование структуры рецептора с целью ее корректного размещения в мембране было проведено с привлечением инструмента OPM [15]. Погружение лиганд-рецепторного комплекса в модель мембраны и водного раствора NaCl в концентрации 0.15 моль/л выполнено с помощью инструмента Packmol Memgen 1.1.0 [16, 17]. В качестве образующих мембрану фосфолипидов использовали молекулы фосфатидилхолина (POPC). Полученные структуры имели размер примерно 100 × 100 × 100 Å и содержали порядка 100 000 атомов.
Параметризацию модели осуществляли с помощью входящих в Amber Tools 20 инструментов tleap, antechamber и parmchk2. При параметризации использовали следующие силовые поля: ff19SB – для аминокислотных остатков, lipid17 – для молекул мембраны, gaff2 – для лигандов. В качестве модели воды использовали четырехточечную модель OPC. Оптимизацию построенных структур проводили методом итеративной минимизации потенциальной энергии с помощью входящего в Amber Tools 20 инструмента pmemd. Количество итераций составляло 5000, из них 2500 проводили методом наискорейшего спуска. Затем система была разогрета до 310 К (37°С) и приведена в равновесное состояние в два этапа: вначале в условиях канонического ансамбля (NVT) на протяжении 1 нс с временны́м шагом 0.002 пс, а затем для выравнивания плотности дополнительно в условиях изотерм-изобарического ансамбля (NPT) при температуре 310 К и давлении 1 атм на протяжении 1 нс с временны́м шагом 0.002 пс. Полученные структуры использовали в качестве исходных для молекулярно-динамического моделирования в ансамбле NPT при 310 К и 1 атм. на протяжении не менее 100 нс с временны́м шагом 0.002 пс. Динамика ковалентных связей с участием атомов водорода ограничивалась в рамках алгоритма SHAKE. Радиус отсечения нековалентных взаимодействий был взят равным 9 Å. Использовали ланжевеновский термостат и баростаты Берендсена (получение равновесного состояния) и Монте-Карло (собственно моделирование). Дальнодействующие электростатические взаимодействия рассчитывали методом Эвальда (Particle Mesh Ewald method).
Моделирование методом ускоренной молекулярной динамики (accelerated molecular dynamics, или aMD) выполняли при тех же условиях в соответствии с алгоритмом, описанным в руководстве к Amber. Данный алгоритм ускоряет кинетику конформационных блужданий за счет сглаживания пиков и впадин функции потенциальной энергии системы, что позволяет перебирать значительно большее количество состояний системы, чем в стандартной молекулярной динамике при том же времени вычислений. Параметры пороговых значений энергии и факторы ускорения ethreshp, ethreshd, alphap, alphad рассчитывали по формулам, предложенным в руководстве к Amber.
Изображения формировали с помощью программного обеспечения PyMOL [18].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
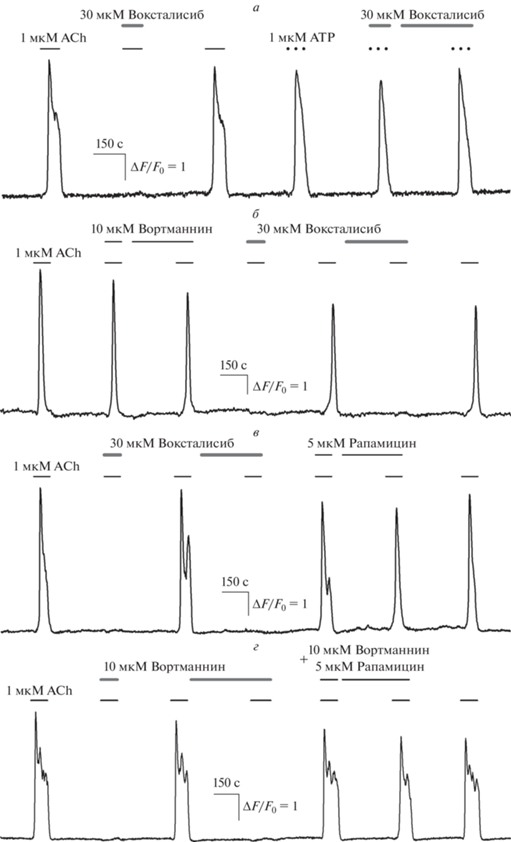
Физиологические эксперименты показали, что 50–80% клеток HEK 293 отвечают на стимуляцию ацетилхолином в концентрации 1 мкМ мобилизацией внутриклеточного Ca2+, при этом ответы практически всех холинергических клеток полностью подавляются в присутствии 30 мкМ воксталисиба (рис. 1а). Специфичность действия воксталисиба по отношению к холинергической сигнальной системе подтверждается отсутствием ингибирующего действия этого соединения на Ca2+-сигнализацию, инициируемую пуринергическим агонистом ATP (рис. 1а). Поскольку воксталисиб является ингибитором PI3K и mTOR, подавление Ca2+-ответов на ацетилхолин формально можно интерпретировать как свидетельство участия PI3K/Akt/mTOR пути в их генерации. Для проверки участия PI3K и mTOR в генерации Ca2+-ответов на ацетилхолин мы использовали ингибиторы этих молекул другой химической природы. Оказалось, что классический ингибитор PI3K вортманнин не влиял на способность клеток отвечать на ацетилхолин даже в случае предварительной инкубации с ним (рис. 1б). Стоит отметить, что вортманнин ингибирует PI3K необратимо [19], и подавление Ca2+-ответов в присутствии воксталисиба, апплицированного после вортманнина (рис. 1б), может быть объяснено только воздействием воксталисиба на мишень, отличную от PI3K. Таким образом, эксперименты с вортманнином показали, что PI3K не играет заметной роли в генерации клеточных ответов на ацетилхолин, но оставляли возможность того, что ингибирующее действие воксталисиба может быть связано с воздействием на другую его мишень – mTOR. Такая возможность была проверена с помощью классического ингибитора mTOR рапамицин, который, как оказалось, также не блокировал ответы на ацетилхолин при различных протоколах его использования (рис. 1в). Поскольку раздельное применение ингибиторов PI3K и mTOR не приводило к подавлению ответов, аналогичному эффекту воксталисиба, они были применены совместно. Оказалось, что и в этом случае клетки генерировали полноценные ответы на ацетилхолин (рис. 1г). Таким образом, результаты экспериментов с классическими ингибиторами PI3K и mTOR однозначно свидетельствовали о том, что PI3K/Akt/mTOR путь не играет принципиальной роли в генерации Ca2+-сигналов, вызываемых ацетилхолином в клетках HEK 293. Следовательно, воксталисиб блокирует Ca2+-ответы на ацетилхолин не за счет ингибирования PI3K и/или mTOR, а воздействует на другие клеточные мишени.
Рис. 1.
Влияние ингибиторов PI3K и mTOR на Ca2+-ответы клеток HEK 293 на ацетилхолин. На рисунках представлены репрезентативные регистрации Ca2+-ответов одиночных клеток; моменты и продолжительность аппликаций веществ обозначены горизонтальными линиями выше экспериментальной кривой. Изменение внутриклеточного Ca2+ оценивали по относительному изменению флуоресценции Fluo-4 согласно формуле: ΔF/F0, где ΔF = F – F0, F и F0 – текущая интенсивность эмиссии зонда и его эмиссия в начале регистрации соответственно. а – Ответы клетки на ацетилхолин (АСh) (1 мкМ) и ATP (1 мкМ) в контроле и в присутствии ингибитора PI3K и mTOR воксталисиба (30 мкМ) (n = 72); б – ответы клетки на ацетилхолин (1 мкМ) в контроле и в присутствии ингибитора PI3K вортманнина (10 мкМ) и воксталисиба (30 мкМ) (n = 46); в – ответы клетки на ацетилхолин (1 мкМ) в контроле и в присутствии ингибитора mTOR рапамицина (5 мкМ) и воксталисиба (30 мкМ) (n = 32); г – ответы клетки на ацетилхолин в контроле в присутствии воксталисиба (30 мкМ) и смеси вортманнина (10 мкМ) и рапамицина (5 мкМ) (n = 19).
При анализе других возможностей обращал на себя внимание тот факт, что воксталисиб блокировал ответы на ацетилхолин при одновременной аппликации с ним (рис. 1а), тогда как обычно ингибирование внутриклеточных мишеней в интактных клетках происходит с некоторой задержкой. Последняя определяется временем, требуемым для проникновения ингибитора через плазматическую мембрану и его накопления в цитозоле до необходимого уровня. Тот факт, что воксталисиб блокировал клеточные ответы без предварительной инкубации, указывал на возможность внеклеточного действия этого соединения, т.е. в качестве вероятной мишени могли выступать сами мускариновые рецепторы.
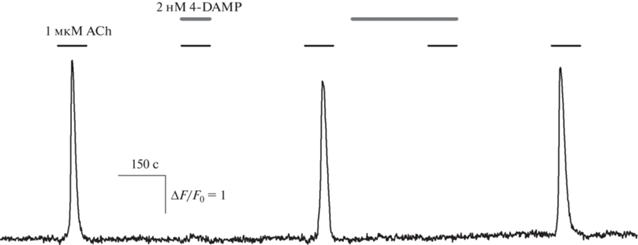
Известно, что клетки линии HEK 293 эндогенно экспрессируют мускариновые рецепторы, из которых M3-изоформа является доминантной [20]. Наши эксперименты также показали, что за чувствительность HEK 293 к ацетилхолину ответственны преимущественно M3-рецепторы. Так, антагонист M3-рецептора 4-DAMP оказался весьма эффективным и блокировал Ca2+-ответы всех холинергических клеток уже при концентрации 5–10 нМ при одновременной аппликации с ацетилхолином (1 мкМ) (рис. 2). Таким образом, физиологические эксперименты косвенно подтверждают идею о том, что М3-рецепторы могли быть мишенью действия воксталисиба. Для независимого подкрепления такой возможности было проведено компьютерное моделирование взаимодействия М3-рецептора с веществами, которые были использованы в экспериментах.
Рис. 2.
Подавление Ca2+-ответов клеток HEK 293 на ацетилхолин антагонистом M3-рецепторов 4-DAMP. Репрезентативная регистрация Ca2+-ответов клеток на ацетилхолин (1 мкМ) в контроле и в присутствии 4-DAMP (2 нМ) (n = 50).
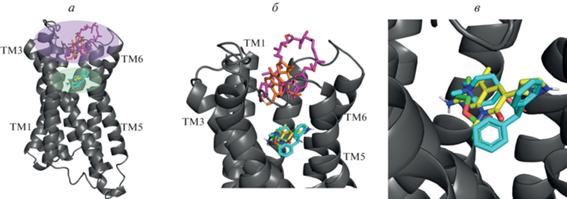
Моделирование методом докинга показало, что молекулы рапамицина и вортманнина могут занимать зоны в области внеклеточных петель рецептора, так называемом вестибюле, а молекулы воксталисиба и 4-DAMP могут локализоваться глубоко в трансмембранной полости рецептора, располагаясь в непосредственной близости от ортостерического сайта (рис. 3). Количественной характеристикой локализации соединений на рецепторе могут служить расстояния между геометрическими центрами соединений и геометрическим центром ацетилхолина, рассчитанные усреднением предсказанных докингом координат тяжелых атомов. Оценки этих расстояний для воксталисиба, 4-DAMP, рапамицина и вортманнина дают значения 2.49, 3.04, 14.44 и 11.66 Å соответственно.
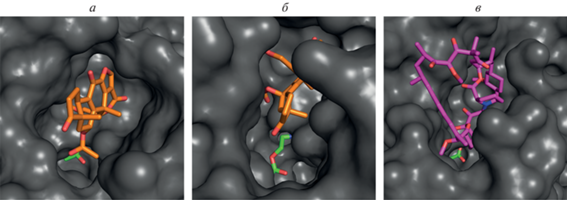
Рис. 3.
Комплексы M3-рецептора с исследовавшимися соединениями. Представлены предсказанные докингом конформации, энергетически наиболее выгодные для образования комплексов. Воксталисиб изображен желтым цветом, 4-DAMP – бирюзовым, рапамицин – фиолетовым, вортманнин – оранжевым, ацетилхолин – зеленым. ТМ1, ТМ3, ТМ5 и ТМ6 – трансмембранные 1, 3, 5 и 6-я спирали рецептора соответственно. а – Общий вид рецептора, фиолетовым выделена область внеклеточных петель с расположенными в ней молекулами рапамицина и вортманнина, зеленым – область ортостерического сайта, содержащая молекулы воксталисиба и 4-DAMP; б – увеличенный вид ортостерического сайта и внеклеточных петель, связанных с молекулами указанных соединений; в – ортостерический сайт рецептора, связанный с 4-DAMP и воксталисибом.
Для оценки аффинности соединений воксталисиб и 4-DAMP к рецептору в положении ортостерического сайта мы использовали значения скоринг-функции, полученные Autodock Vina при докинге, которые для воксталисиба и 4-DAMP оказались равны –8.9 и –10.1 соответственно. С точностью до ранее установленной погрешности 2.85 ккал/моль [11] эти величины могут быть использованы как оценки свободной энергии связи для воксталисиба и 4-DAMP и тем самым позволяют оценить константы диссоциации Kd (см. Материалы и методы). Следует отметить, что хотя оцениваемая таким образом величина Kd может существенно отклоняться от истинного значения константы диссоциации, относительные расчетные величины могут давать вполне реалистичные оценки. Так, например, для мускариновых рецепторов ранее было показано, что отношение констант диссоциации ингибитора PI3-киназы LY294002 и его аналога LY303511, полученных из расчетов, согласуется с результатами эксперимента [3]. Отношение расчетных констант диссоциации для воксталисиба и 4-DAMP составляет 7.63. Такое значение в целом отражает результаты экспериментов, в соответствии с которыми 4-DAMP подавлял Ca2+-ответы на ацетилхолин в дозах, значительно меньших, чем те, которые требовались в случае воксталисиба.
Построенные молекулярно-динамические траектории длительностью 100 нс продемонстрировали, что предсказанные докингом комплексы в целом устойчивы и что локализация лигандов в структуре рецептора существенно не меняется за указанное время. Так, молекулы воксталисиба и 4-DAMP оставались в области ортостерического сайта рецептора, а рапамицин и вортманнин – в вестибюле (рис. 4). На примере тиотропиума молекулярно-динамическое моделирование ранее выявило, что антагонисты мускариновых рецепторов не сразу следуют в ортостерический сайт, а способны задерживаться в вестибюле [21]. Чтобы проверить возможность перемещения рапамицина и вортманнина из вестибюля в ортостерический сайт рецептора на временны́х интервалах длиннее 100 нс, конформационные блуждания соответствующих комплексов анализировали методом ускоренной молекулярной динамики. Нами было получено несколько ускоренных траекторий для комплексов рецептора с рапамицином и вортманнином на длительностях в несколько сотен наносекунд. Данное моделирование показало, что вортманнин остается в вестибюле, не перемещаясь в область ортостерического сайта, но и не диссоциируя в раствор. При этом рецептор мог принимать конформации, при которых в вестибюле, занятом молекулой вортманнина, сохраняется свободное пространство. По-видимому, размеры этого пространства достаточны для проникновения ацетилхолина в область ортостерического сайта рецептора (рис. 5а, 5б), как это предполагают физиологические эксперименты (рис. 1б). Для окончательного вывода требуется построение траекторий в микросекундном диапазоне.
Рис. 4.
Эволюция комплексов мускаринового M3-рецептора с исследуемыми соединениями. Представлены последовательности кадров молекулярно-динамических траекторий комплексов, полученных через 25, 50, 75 и 100 нс после начала симуляции. Начальная конфигурация определялась докингом. Видно, что молекулы воксталисиба и 4-DAMP сохраняют положение в области ортостерического сайта рецептора, вортманнина и рапамицина – в области внеклеточных петель. Соединения изображены с атомами водорода.
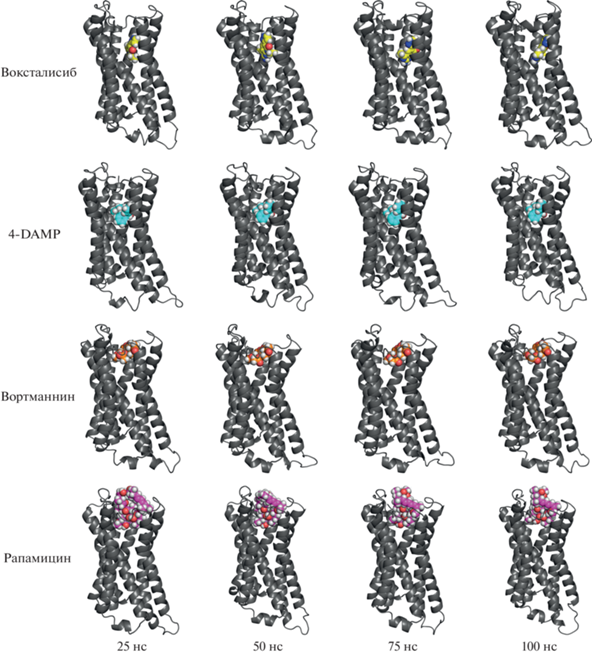
Рис. 5.
Локализация молекул вортманнина и рапамицина в вестибюле М3-рецептора (вид из внеклеточного пространства). а – Локализация молекулы вортманнина (оранжевый), предсказанная докингом. При этой конфигурации вортманнин, вероятно, препятствует прохождению ацетилхолина (зеленый) в область ортостерического сайта; б – положение молекулы вортманнина, полученное при симуляции методом ускоренной молекулярной динамики; в – положение молекулы рапамицина (фиолетовый) в области ортостерического сайта, предсказанное докингом. Видно, что рапамицин стерически блокирует вестибюль и препятствует прохождению ацетилхолина (зеленый) в область ортостерического сайта.
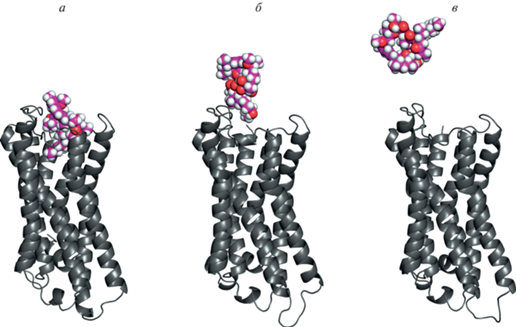
В исходном положении, предсказанном докингом, рапамицин занимает все пространство вестибюля и не оставляет возможности для прохода ацетилхолина (рис. 5в). Между тем, как показало моделирование с применением ускоренной молекулярной динамики, при достаточно продолжительной эволюции молекулярная система может принимать такие конформации, при которых рапамицин диссоциирует в раствор, освобождая вестибюль агонисту для его свободного перемещения в область ортостерического сайта (рис. 6).
Рис. 6.
Диссоциация рапамицина из области вестибюля М3-рецептора в раствор. Последовательность кадров молекулярно-динамической траектории комплекса мускаринового М3-рецептора с рапамицином; моделирование методом ускоренной молекулярной динамики. а – Исходное положение, в котором рапамицин расположен в вестибюле рецептора; б – промежуточная конфигурация, при которой рапамицин еще взаимодействует со 2-й внеклеточной петлей рецептора, но вестибюль уже практически свободен; в – полная диссоциация рапамицина в раствор.
В целом, компьютерное моделирование показало, что воксталисиб и 4-DAMP способны конкурировать с агонистом за ортостерический сайт рецептора и тем самым препятствовать его активации. В присутствии рапамицина и вортманнина ортостерический сайт, напротив, доступен для ацетилхолина, что позволяет активировать рецептор. При этом доступность ортостерического сайта для агониста в присутствии рапамицина и вортманнина обеспечивается по различным сценариям. Представляется, что в вестибюле связанный вортманнин оставляет, по крайней мере при некоторых достижимых рецептором конформациях, достаточно пространства для прохода ацетилхолина в ортостерический сайт рецептора. Хотя вестибюль блокируется при связывании рапамицина, этот ингибитор не способен надолго задерживаться в этой области и диссоциирует в раствор, оставляя рецептор для свободного взаимодействия с ацетилхолином. Предложенные по результатам моделирования механизмы вполне объясняют, почему воксталисиб и 4-DAMP способны ингибировать Ca2+-ответы клеток на ацетилхолин, в то время как рапамицин и вортманнин были неэффективны (рис. 1, 2).
Таким образом, полученные в данной работе результаты свидетельствуют о том, что воксталисиб способен взаимодействовать с мускариновыми рецепторами М3-типа и блокировать внутриклеточную Ca2+-сигнализацию, индуцированную ацетилхолином. Эти данные указывают на то, что побочные эффекты, возникающие у пациентов при приеме воксталисиба, могут быть связаны не только с его воздействием на сигнальный каскад PI3K/Akt/mTOR, но и с угнетающим влиянием воксталисиба на холинергическую систему, играющую ключевую роль во многих физиологических процессах [22, 23].
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Источники финансирования. Работа выполнена при поддержке гранта РНФ № 20-74-00056.
Соответствие принципам этики. Настоящая статья не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Список литературы
Porta C., Paglino C., Mosca A. 2014. Targeting PI3K/Akt/mTOR signaling in cancer. Front Oncol. 4, 64.
Mishra R., Patel H., Alanazi S., Kilroy M.K., Garrett J.T. 2021. PI3K inhibitors in cancer: Clinical implications and adverse effects. Int. J. Mol. Sci. 22, 3464.
Kotova P.D., Kochkina E.N., Lyamin O.O., Rogachevskaja O.A., Kovalenko N.P., Ivashin D.S., Bystrova M.F., Enukashvily N.I., Kolesnikov S.S. 2020. Calcium signaling mediated by aminergic GPCRs is impaired by the PI3K inhibitor LY294002 and its analog LY303511 in a PI3K-independent manner. Eur. J. Pharmacol. 880, 173182.
Дымова Е.А., Рогачевская О.А., Воронова Е.А., Котова П.Д. 2021. PI828 подавляет Ca2+-сигнализацию, инициируемую аминергическими агонистами, по механизму, независимому от ингибирования PI3-киназы. Биол. мембраны. 38 (5), 265–273.
Berman H.M., Westbrook J., Feng Z., Gilliland G., Bhat T.N., Weissig H., Shindyalov I.N., Bourne P.E. 2000. The Protein Data Bank. Nucleic Acids Res. 28, 235–242.
Thorsen T.S., Matt R., Weis W.I., Kobilka B.K. 2014. Modified T4 lysozyme fusion proteins facilitate G protein-coupled receptor crystallogenesis. Structure. 22 (11), 1657–1664.
Reddy Chichili V.P., Kumar V., Sivaraman J. 2013. Linkers in the structural biology of protein-protein interactions. Protein Sci. 22 (2), 153–167.
Waterhouse A., Bertoni M., Bienert S., Studer G., Tauriello G., Gumienny R., Heer F.T., de Beer T.A.P., Rempfer C., Bordoli L., Lepore R., Schwede T. 2018. SWISS-MODEL: Homology modelling of protein structures and complexes. Nucleic Acids Res. 46, W296–W303.
Sterling T., Irwin J.J. 2015. ZINC 15 – Ligand discovery for everyone. J. Chem. Inf. Model. 55 (11), 2324–2337.
Kim S., Chen J., Cheng T., Gindulyte A., He J., He S., Li Q., Shoemaker B.A., Thiessen P.A., Yu B., Zaslavsky L., Zhang J., Bolton E.E. 2021. PubChem in 2021: New data content and improved web interfaces. Nucleic Acids Res. 49 (D1), D1388–D1395.
Trott O., Olson A.J. 2010. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading. J. Comput. Chem. 31, 455–461.
Case D.A., Aktulga H.M., Belfon K., Ben-Shalom I.Y., Brozell S.R., Cerutti D.S., Cheatham T.E. III, Cisne-ros G.A., Cruzeiro V.W.D., Darden T.A., Duke R.E., Giambasu G., Gilson M.K., Gohlke H., Goetz A.W., Harris R., Izadi S., Izmailov S.A., Jin C., Kasavajhala K., Kaymak M.C., King E., Kovalenko A., Kurtzman T., Lee T.S., LeGrand S., Li P., Lin C., Liu J., Luchko T., Luo R., Machado M., Man V., Manathunga M., Merz K.M., Miao Y., Mikhailovskii O., Monard G., Nguyen H., O’Hearn K.A., Onufriev A., Pan F., Pantano S., Qi R., Rahnamoun A., Roe D.R., Roitberg A., Sagui C., Schott-Verdugo S., Shen J., Simmerling C.L., Skrynnikov N.R., Smith J., Swails J., Walker R.C., Wang J., Wei H., Wolf R.M., Wu X., Xue Y., York D.M., Zhao S., Kollman P.A. 2021. Amber 2021. University of California, San Francisco.
Fahmy K., Jäger F., Beck M., Zvyaga T.A., Sakmar T.P., Siebert F. 1993. Protonation states of membrane-embedded carboxylic acid groups in rhodopsin and metarhodopsin II: A Fourier-transform infrared spectroscopy study of site-directed mutants. Proc. Natl. Acad. Sci. USA. 90, 10206–10210.
Jurrus E., Engel D., Star K., Monson K., Brandi J., Felberg L.E., Brookes D.H., Wilson L., Chen J., Liles K., Chun M., Li P., Gohara D.W., Dolinsky T., Konecny R., Koes D.R., Nielsen J.E., Head-Gordon T., Geng W., Krasny R., Wei G.W., Holst M.J., McCammon J.A., Baker N.A. 2018. Improvements to the APBS biomolecular solvation software suite. Protein Sci. 27 (1), 112–128.
Lomize M.A., Pogozheva I.D, Joo H., Mosberg H.I., Lomize A.L. 2012. OPM database and PPM web server: Resources for positioning of proteins in membranes. Nucleic Acids Res. 40, D370–376.
Martinez L., Andrade R., Birgin E.G., Martinez J.M. 2009. Packmol: A package for building initial configurations for molecular dynamics simulations. J. Comput. Chem. 30 (13), 2157–2164.
Nugent T., Jones D.T. 2013. Membrane protein orientation and refinement using a knowledge-based statistical potential. BMC Bioinformatics. 14, 276.
Schrodinger, LLC. 2015. The PyMOL molecular graphics system. Version 2.2.
Powis G., Bonjouklian R., Berggren M.M., Gallegos A., Abraham R., Ashendel C., Zalkow L., Matter W.F., Dodge J., Grindey G. 1994. Wortmannin, a potent and selective inhibitor of phosphatidylinositol-3-kinase. Cancer Res. 54 (9), 2419–2423.
Atwood B.K., Lopez J., JWager-Miller J., Mackie K., Straiker A. 2011. Expression of G protein-coupled receptors and related proteins in HEK293, AtT20, BV2, and N18 cell lines as revealed by microarray analysis. BMC Genomics. 12, 14.
Kruse A.C., Hu J., Pan A.C., Arlow D.H., Rosenbaum D.M., Rosemond E., Green H.F., Liu T., Chae P.S., Dror R.O., Shaw D.E., Weis W.I., Wess J., Kobilka B.K. 2012. Structure and dynamics of the M3 muscarinic acetylcholine receptor. Nature. 482 (7386), 552–556.
Abrams P., Andersson K.E., Buccafusco J.J., Chapple C., de Groat W.C., Fryer A.D., Kay G., Laties A., Nathanson N.M., Pasricha P.J., Wein A.J. 2006. Muscarinic receptors: Their distribution and function in body systems, and the implications for treating overactive bladder. Br. J. Pharm. 148, 565–578.
Saternos H.C., Almarghalani D.A., Gibson H.M., Meqdad M.A., Antypas R.B., Lingireddy A., AbouAlaiwi W.A. 2018. Distribution and function of the muscarinic receptor subtypes in the cardiovascular system. Physiol. Genomics. 50, 1–9.
Дополнительные материалы отсутствуют.
Инструменты
Биологические мембраны: Журнал мембранной и клеточной биологии


