Биологические мембраны: Журнал мембранной и клеточной биологии, 2023, T. 40, № 1, стр. 43-54
Клеточные линии с единственной функциональной изоформой IP3-рецептора
Е. Е. Копылова a, Е. А. Воронова a, Н. В. Кабанова a, О. А. Рогачевская a, М. Ф. Быстрова a, С. С. Колесников a, *
a Институт биофизики клетки РАН ФИЦ ПНЦБИ РАН
142290 Пущино, Московская обл., Россия
* E-mail: staskolesnikov@yahoo.com
Поступила в редакцию 29.08.2022
После доработки 29.09.2022
Принята к публикации 03.10.2022
- EDN: NSLVHI
- DOI: 10.31857/S0233475523010036
Аннотация
Многие агонисты регулируют клеточные функции, стимулируя поверхностные рецепторы сопряженные фосфоинозитидным каскадом с мобилизацией внутриклеточного Са2+. В невозбудимых клетках генерация внутриклеточных Са2+ сигналов протекает преимущественно за счет выброса Са2+ из Са2+-депо, локализованного в эндоплазматическом ретикулуме (ЭР). В этой системе IP3-рецепторы, являющиеся внутриклеточным IP3-активируемыми Са2+-каналами, обеспечивают регулируемый выброс депонированного Са2+ в ответ на стимуляцию клеток. Ряд факторов затрудняет анализ специфической роли IP3-рецепторов в физиологии клетки и их регуляторных механизмов. Во-первых, три гена кодируют IP3-рецепторы, и в клетках обычно экспрессированы два из них или даже все гены. При этом разные изоформы IP3-рецепторов находятся под контролем различных механизмов. Кроме того, изоформоспецифичные антагонисты IP3-рецепторов не идентифицированы на данный момент. Клеточные линии, экспрессирующие IP3-рецепторы только одного типа, представляют собой эффективную клеточную модель для исследования регуляторных механизмов, фармакологии и физиологической роли изоформ IP3-рецепторов. В данной работе мы использовали CRISPR/Cas9 технологию для инактивации генов IP3-рецепторов в клетках HEK-293, экспрессирующих все три гена этих белков. Были получены моноклоны генетически модифицированых клеток HEK-293 и идентифицированы те из них, которые содержали биаллельные инактивирующие мутации в двух из трех генов IP3-рецепторов. В результате были получены моноклональные клеточные линии с единственной функциональной изоформой IP3-рецептора, которые можно использовать для исследования роли данного подтипа IP3-рецептора в агонист-индуцированной Са2+-сигнализации и анализа его регуляторных механизмов.
ВВЕДЕНИЕ
Внутриклеточный Са2+ вовлечен в регуляцию разнообразных клеточных функций, таких как экспрессия генов, подвижность, дифференцировка и пролиферация, апоптоз, синаптическая передача, мышечная сократимость [1]. В невозбудимых клетках генерация внутриклеточных Са2+-сигналов протекает за счет выброса Са2+ из Са2+-депо, локализованных преимущественно в эндоплазматическом ретикулуме (ЭР) [2, 3]. Гомеостаз Са2+ в ЭР контролируется достаточно сложной системой транспортных и регуляторных белков, которая обеспечивает поддержание стационарного уровня депонированного Са2+ в покое, контролируемый выброс Са2+ в ответ на стимуляцию клеток и эффективное пополнение Са2+-депо [4–6]. В этой системе IP3-рецептор, представляющий собой Са2+-канал ЭР, обеспечивает регулируемый выброс депонированного Са2+ при стимуляции невозбудимых клеток [7, 8]. Ключевым в этом процессе является стимул-рецептор-G-белок-зависимая активация фосфолипазы С, которая катализирует продукцию вторичного медиатора IP3 с последующей стимуляцией IP3-рецепторов. У позвоночных субъединицы IP3-рецепторов кодируют три гена: IP3R1, IP3R2 и IP3R3. В разных клетках обычно функциональны 2–3 изоформы IP3-рецепторов, активность каждой из которых находится под контролем различных механизмов. Это определяет индивидуальные особенности IP3-зависимых Са2+-сигналов и специфическую роль IP3-рецепторов данного типа в физиологии различных клеток [1, 7, 8].
В клеточных исследованиях все в большей мере используется инструментарий редактирования генома для адресной модификации генов, кодирующих компоненты рецепторных систем, систем трансдукции сигналов и регуляторных путей [9]. В настоящее время используют несколько систем сайт-специфичного узнавания и разрезания ДНК: цинковопальцевые нуклеазы (ZFN, Zinc-Finger Nuclease), TALE-ассоциированные нуклеазы (Transcription Activator-like Effector Nucleases, TALEN) и CRISPR/Cas9 (Clustered Regularly Interspaced Shortpalindromic Repeats/CRISPR-associated protein) [10]. Все эти системы осуществляют направленный двухцепочечный разрыв в целевом участке ДНК. В настоящий момент наибольшее распространение получила технология CRISPR/Cas9 в силу универсальности и относительной простоты реализации: в ней ключевым является конструирование короткого (17–20 нуклеотидов) РНК-гида (sg-РНК), который будет направлять нуклеазу в место внесения разрыва в ДНК [11]. Нуклеаза Cas9 в комплексе с sg-РНК распознает последовательность PAM (Protospacer Adjacent Motif) и вносит разрыв в целевой локус, комплементарный sg-РНК, на расстоянии 3–4 нуклеотидов от PAM. В целом, при наличии последовательности PAM комплекс CRISPR/Cas9 можно направить на любую выбранную исследователем последовательность-мишень. После внесения двухцепочечных разрывов в ДНК клетка активирует эндогенную систему репарации разными путями – за счет гомологичной и негомологичной рекомбинации, что приводит к возникновению целевых мутаций, хотя нецелевые повреждения ДНК (off-target) также возможны. Нецелевые мутации возникают при посадке системы CRISPR/Cas9 на области генома, частично комплементарные sg-РНК, при наличии мотива PAM.
Клеточные линии, экспрессирующие IP3-рецепторы только определенного типа, представляют собой эффективную модель для исследования регуляторных механизмов, фармакологии и физиологической роли изоформ этих лиганд-управляемых внутриклеточных Са2+-каналов. В данной работе мы использовали систему CRISPR/Cas9 для редактирования генома клеток HEK-293, которые экспрессируют все три гена IP3-рецепторов [12]. В гены IP3-рецепторов вносились мутации, которые приводили к смещению открытой рамки считывания, что приводило к трансляции дефектного рецепторного белка. Были выявлены моноклоны генетически модифицированных клеток HEK-293, которые содержали биаллельные инактивирующие мутации в двух из трех генов IP3-рецепторов. В результате были получены стабильные клеточные линии с единственной функциональной изоформой IP3-рецептора, т.е. либо IP3R1, либо IP3R2, либо IP3R3.
МАТЕРИАЛЫ И МЕТОДЫ
1.1. Конструкция pGuide-it-tdTomato Vector/IP3R3 для направленного редактирования гена IP3R3 (эндонуклеазная система)
Поиск оптимального расположения протоспейсера и мотива PAM проводили на смысловой и антисмысловой цепях ДНК гена IP3R3 на последовательности мРНК IP3R3 (GenBank NM_002224.4). При дизайне sg-РНК учитывались данные по влиянию протяженности и нуклеотидного состава sg-РНК на эффективность и специфичность редактирования [13]. Для редактирования был выбран участок с контекстом 5'-ATGTCCAGCTTTCTTCACAT, расположенный в первом экзоне гена справа от инициирующего кодона. Для внесения редактирующего комплекса в клетки использован вектор pGuide-it-tdTomato Vector, содержащий ген cas9 и позволяющий экспрессировать требуемую sg-РНК под контролем промотора U6. Для синтеза спейсерной последовательности были получены два взаимно-комплементарных олигонуклеотида 5'-ccggATGTCCAGCTTTCTTCACAT-3' и 5'‑aaacATGTGAAGAAAGCTGGACAT-3'. Все олигонуклеотиды содержали на 5'‑концах адаптерные последовательности для клонирования в вектор (подчеркнуты). Каждый олигонуклеотид (50 пмоль/мкл) фосфорилировали по 5'- концам при помощи Т4-полинуклеотидкиназы Anza™ T4 PNK kit (Invitrogen) в соответствии с рекомендацией производителя. Реакцию отжига фосфорилированных олигонуклеотидов проводили в буфере Annealing Buffer из набора Guide-it CRISPR/Cas9 System (Takara) путем нагревания до 94°С и постепенного охлаждения в течение 40 мин. Олигонуклеотидный дуплекс с концентрацией 100 фмоль/мкл клонировали в линеаризованную плазмиду pGuide-it-tdTomato Vector по липким концам адаптерных последовательностей с использованием набора для быстрого лигирования Guide-it CRISPR/Cas9 System (Takara). Компетентные клетки Stellar Competent Cells (Takara) трансформировали лигазной смесью, полученные плазмиды (pGuide-it-tdTomato/IP3R3) тестировали на наличие вставки при помощи набора для ПЦР (ScreenMix-HS (UDG), Евроген) с праймерами 5'-ccggATGTCCAGCTTTCTTCACAT-3' и 5'‑AAAAGCACCGACTCGGTGCC‑3' (коммерческий праймер к плазмиде). Наличие вставки спейсера и правильность полученной конструкции подтверждали секвенированием (Евроген).
1.2. Конструкция pGuide-it-tdTomato Vector/IP3R2 для направленного редактирования гена IP3R2 (эндонуклеазная система)
В последовательности мРНК гена IP3R2 (база данных GenBank NM_002223.4) для редактирования был выбран потенциальный протоспейсер, содержащий с 3'-конца мотив PAM – СGG. Для конструирования вставки, кодирующей sg-РНК, синтезированы взаимно-комплементарные олигонуклеотиды 5'-ccggGGACATCGTGTCCCTGTACG-3' и 5'‑ aaacCGTACAGGGACACGATGTCC-3', содержащие на 5'-концах адаптерные последовательности для клонирования в плазмидный вектор (подчеркнуты). Фосфорилирование олигонуклеотидов, формирование дуплекса и клонирование в вектор pGuide-it-tdTomato Vector проводили аналогично тому, как описано для плазмиды pGuide-it-tdTomato Vector/IP3R3. Полученной лигазной смесью трансформировали компетентные клетки Stellar Competent Cells (Takara), полученные плазмиды тестировали на наличие вставки при помощи набора для ПЦР с колоний (ScreenMix‑HS (UDG), Евроген) с праймерами 5'‑ccggGGACATCGTGTCCCTGTACG-3' и 5'‑AAAAGCACCGACTCGGTGCC‑3'. Для подтверждения правильности полученной конструкции (pGuide-it-tdTomato/IP3R2) плазмиды секвенировали (Евроген).
1.3. Конструкция AIO-GFP/IP3R1 для направленного редактирования IP3R1 (никазная система)
Редактирование гена IP3R1 осуществляли при помощи вектора AIO-GFP, обеспечивающего экспрессию Cas9-D10A никазы, слитой с EGFP и двух sg-РНК [14]. Вектор AIO-GFP был предоставлен Steve Jackson (Addgene plasmid # 74119; http://n2t.net/addgene:74119). Поиск мишени для редактирования на смысловой и антисмысловой нитях ДНК проводили по последовательностям NM_001099952.4; NM_001168272.2; NM_001378452.1; NM_002222.7 (GenBank), соответствующим всем четырем вариантам транскриптов гена. Для редактирования были выбраны участки в составе третьего экзона, идентичные для всех вариантов транскриптов: 1) на расстоянии 68 пар нуклеотидов (п.н.) справа от инициирующего кодона (пара взаимно-комплементарных олигонуклеотидов: 5'-accgAAATGGATTTATTAGCACCT-3' и 5'‑aaacAGGTGCTAATAAATCCATTT-3', “смысловой дуплекс”) и 2) на расстоянии 25 п.н. справа от инициирующего кодона (пара взаимно‑комплементарных олигонуклеотидов 5'‑accgAACAAATGTCTCCAATATGT‑3' и 5'‑aaacACATATTGGAGACATTTGTT-3' “антисмысловой дуплекс”). Все синтезированные олигонуклеотиды содержат адаптерные последовательности для последующего клонирования в вектор (подчеркнуты). Олигонуклеотиды были фосфорилированы на 5'-концах, как описано выше, и проведена реакция отжига для формирования дуплексов перед клонированием в линеаризированную плазмиду AIO-GFP. В вектор последовательно встраивали сначала антисмысловой дуплекс фосфорилированных олигонуклеотидов по сайту BbsI (Thermo Scientific), затем смысловой дуплекс по сайту BsaI (Eco31I) (Thermo Scientific). При встраивании “антисмыслового” дуплекса результат лигирования контролировали при помощи ПЦР с колоний (ScreenMix-HS (UDG), Евроген) с праймерами 5'‑accgAACAAATGTCTCCAATATGT-3' и 5'‑CTTGATGTACTGCCAAGTGGGC-3' после трансформации вектором клеток Stellar Competent Cells (Takara). При лигировании “смыслового” дуплекса по сайту BsaI плазмидную ДНК вектора со вставкой “антисмыслового” дуплекса обрабатывали рестриктазой, дефосфорлировали при помощи щелочной фосфатазы Fast-AP (Thermo Scientific) и проводили вторую реакцию лигирования. Колонии после трансформации проверяли при помощи ПЦР с колоний с праймерами 5'‑accgAAATGGATTTATTAGCACCT-3' и 5'‑CTTGATGTACTGCCAAGTGGGC-3'. Наличие вставок, соответствующих последовательностям sg-РНК в векторе AIO-GFP/ IP3R1, подтверждали секвенированием (Евроген).
2. Получение моноклонов НЕК-293
Трансфекцию клеток НЕК-293 проводили с использованием набора реагентов Lipofectamin 3000 (Invitrogen) в соответствии с протоколом производителя. Через 72 ч после трансфекции клетки анализировались по интенсивности флуоресценции с помощью сортера клеток FACSAria SORP (BD Biosciences). Для получения моноклонов отбирали клетки, обладающие наибольшей интенсивностью флуоресценции, переносили их по одной в лунку 96-луночного планшета и по достижении 50–70% конфлюэнтности клеточные клоны переносили в лунки с большей ростовой поверхностью (последовательно в 24-, 12- и 6-луночные планшеты). После достижения монослоя в лунке 6-луночного планшета клетки использовались для генетического анализа. Клеточные моноклоны с выявленными биаллельными мутациями целевого гена культивировали в среде DMEM (Gibco) с высоким содержанием глюкозы с добавлением 10–15% эмбриональной бычьей сыворотки (HyClone), 100 мг/мл гентамицина (Sigma), 4 мМ глутамина (Sigma) во влажной атмосфере c 5% содержанием CO2 в воздухе при 37°C.
3. Идентификация клеточных моноклонов с биаллельными мутациями целевых генов
Для обнаружения моноклонов с биаллельными мутациями сначала выявляли клоны с мутациями в модели in vitro с коммерческим ферментом Cas9 и синтезированными sg-РНК, а затем секвенировали по Сэнгеру область редактирования выбранных клонов. Из суспензии клеток каждого из полученных моноклонов HEK293 выделяли геномную ДНК набором реактивов Quick-gDNA MiniPrep, (ZymoResearch) и амплифицировали фрагмент гена (размером от 700 до 900 п.н.) с и-спользованием высокоточной ДНК-полимеразы Phusion Hot Start II (F549; Thermo Scientific) в соответствии с инструкциями производителя. Для этого использовали следующие праймеры: 5'‑GGGATTTGCATGTGTGTGGTG-3' и 5'‑CTACTGAAGCTGGGAAGAACAGG-3' для IP3R3 (ожидаемый размер ампликона 962 п.н.); 5'‑CTGGTCCAAAAATTCCCCTGAG-3' и 5'-AAGCCAGCGAAGACCTCTTC-3' для IP3R2 (ожидаемый размер ампликона 848 п.н.); и 5'‑CATTCCCTGAAGCAAATTGAATATG-3' и 5'-AGTGGAATAAAGGCACTCTC-3' для IP3R1 (ожидаемый размер ампликона 769 п.н.). Амплифицированный участок геномной ДНК, содержащий предполагаемую мутацию, подвергался анализу в модели in vitro комплексом sg-РНК/Cas9.
sg-РНК для трех генов рецептора получали методом транскрипции in vitro, используя набор реактивов Guide-it sgRNA In Vitro Transcription Kit (Takara), содержащего РНК-полимеразу фага Т7. Для синтеза ДНК-матрицы, содержащей последовательность промотора Т7, спейсерные последовательности редактируемых генов и scaffold-матрицу, включающую транс-активирующую РНК, были сконструированы смысловые (прямые) олигонуклеотиды: CCTCTAATACGACTCACTATAGGATGTCCAGCTTTCTTCACATGTTTAAGAGCTATGC для IP3R3, CCTCTAATACGACTCACTATAGGACATCGTGTCCCTGTACGGTTTAAGAGCTATGC для IP3R2, CCTCTAATACGACT- CACTATAGGAAATGGATTTATTAGCACCTGTTTAAGAGCTATGC для IP3R1 (последовательности, соответствующие sg-РНК в составе олигонуклеотидов подчеркнуты). Антисмысловой (обратный) праймер входил в состав реакционной смеси использованного коммерческого набора. Полученные с помощью ПЦР ДНК-матрицы использовали для синтеза (транскрипции) sg-РНК in vitro, очищали продукты с использованием набора реагентов (Guide-it IVT RNA Clean-Up Kit, Takara).
Реакцию гидролиза фрагментов амплифицированной геномной ДНК проводили комплексом Cas9/sgRNA in vitro согласно протоколу производителя (Guide-it™ Genotype Confirmation Kit, Takara). По характеру гидролиза отбирали моноклоны, в которых редактируемый участок не содержит мишеней для Cas9 в комплексе с sg-РНК и, следовательно, несет мутации, затрагивающие область, комплементарную sg-РНК. Для проверки точной локализации мутаций для каждого моноклона была приготовлена клонотека ПЦР-фрагментов редактируемого участка, содержащих продукты амплификации, соответствующие обеим аллелям. Очищенные ПЦР- фрагменты моноклонов с выявленными in vitro биаллельными мутациями клонировали в вектор pJET1.2/blunt по тупым концам с использованием набора CloneGet PCR Cloning Kit (Thermo Schientific), лигазной смесью трансформировали компетентные клетки E. coli XL1-blue (Евроген), колонии тестировали на присутствие вставки ПЦР с праймерами pjet F/R (ScreenMix-HS (UDG), Евроген). Продукты ПЦР очищали при помощи набора QIAqui-ck PCR Purification Kit (Qiagen). Для выявления indel-мутаций продукты ПЦР не менее чем 10 индивидуальных клонов для каждой клонотеки были секвенированы по Сенгеру в компании Евроген с праймерами pjet F/R (5'-CGACTCACTATAGGGAGAGCGGC-3', 5'‑AAGAACATCGATTTTCCATGGCAG-3').
4. Мониторинг внутриклеточного Ca2+
Для фотометрического эксперимента клетки снимали с культурального пластика 0.25% раствором трипсина (Sigma-Aldrich), а затем прикрепляли ко дну фотометрической камеры с помощью адгезивного материала Cell Tak (Corning). При дальнейших манипуляциях клетки находились во внеклеточном растворе, содержащем (мМ): 130 NaCl, 5 KCl, 2 CaCl2, 1 MgCl2, 10 HEPES, pH 7.4, 10 глюкозы. Для загрузки флуоресцентным Ca2+-зондом Fluo-4 клетки инкубировали в присутствии проникающего предшественника Fluo-4 AM (4 мкМ) и детергента Pluronic (0.02%) (оба Molecular Probes) при комнатной температуре в течение 30 мин. Затем клетки отмывали внеклеточным раствором и выдерживали в нем при комнатной температуре в течение 1 ч.
Фотометрические эксперименты проводили с использованием инвертированного флуоресцентного микроскопа Axiovert 135 (Zeiss), оборудованного объективом Plan NeoFluar 20×/0.75 и цифровой EMCCD камерой LucaR (Andor Technology). В соответствии со спектральными характеристиками Fluo-4 флуоресценцию клеток возбуждали на длине волны 480 ± 10 нм, эмиссию регистрировали в области 520 ± 20 нм. Изменение уровня Са2+ в цитоплазме оценивали по относительному изменению интенсивности флуоресценции Fluo-4 ΔF/F0, где ΔF = F – F0, F и F0 – интенсивность эмиссии Са2+-индикатора в текущий момент времени и в начале регистрации, соответственно. Количественный фотометрический анализ изображений осуществляли с использованием программы NIS Elements (Nikon). Полученные экспериментальные данные обрабатывали с помощью программы Sigma Plot 12.5 (Systat Software Inc).
Вещества, использовавшиеся в физиологических экспериментах, приобретались в Sigma-Aldrich (соли, буферы, глюкоза) и Tocris Bioscience (ацетилхолин, ACh).
РЕЗУЛЬТАТЫ
Ряд факторов затрудняет анализ регуляторных механизмов индивидуальных изоформ IP3-рецепторов и их специфической роли в физиологии клетки. Во-первых, в клетках обычно экспрессированы два или даже все три гена, кодирующие IP3-рецепторы. Кроме того, разные изоформы IP3-рецепторов находятся под контролем различных механизмов. Ингибиторный анализ не позволяет выявить индивидуальный вклад IP3R1, IP3R2 или IP3R3 изоформ в генерацию клеточного ответа, поскольку на данный момент изоформо-специфичные антагонисты IP3-рецепторов не идентифицированы. Поэтому целью данной работы было получение клеточных линий, экспрессирующих лишь один ген IP3-рецептора, с целью дальнейшего использования этих клеточных моделей для анализа роли различных изоформ IP3-рецептора в сигнальных процессах. Клетки HEK-293 традиционно используются как модельная система для физиологических тестов и гетерологической экспрессии рекомбинантных белков, включая ионные каналы и рецепторные, сигнальные и регуляторные белки. Учитывая это обстоятельство, а также то, что эти клетки экспрессируют все три гена IP3 рецепторов, именно эта клеточная система была выбрана для геномного редактирования и получения клеточных линий с единственной функциональной изоформой IP3-рецептора.
Технология CRISPR/Cas9 – удобный и надежный инструмент для направленного мутагенеза ДНК в различных объектах [9, 11]. Суть технологии заключается в способности рибопротеинового комплекса нуклеазы Cas9 с sg-РНК узнавать целевую последовательность в геноме и вносить двунитевые разрывы, которые затем репарируются с внесением делеций и инсерций. Направленный характер мутаций определяется последовательностью sg-РНК, комплементарной редактируемому локусу и наличием мотива PAM, обеспечивающего пространственную ориентацию фермента, оптимальную для гидролиза ДНК-мишени. Широкое применение метода связано с возможностью доставки в клетку системы, продуцирующей и Cas9, и sg-РНК с заданной последовательностью в составе одного вектора. Однако в каждом случае, в зависимости от локализации на хромосоме, структурного состояния ДНК, особенностей нуклеотидных последовательностей в области редактирования, результаты направленного мутагенеза остаются плохо предсказуемыми. Поэтому помимо важности для клеточных исследований, получение двойных биаллельных мутаций IP3-рецепторов в клетках млекопитающих представлял интерес для отработки стратегии выбора мишеней и оптимального пути верификации результатов.
Получение линии клеток HEK293/ΔIP3R2/ΔIP3R1
Получение клеточных линий с двойным нокаутом IP3-рецепторов подробно рассматривается на примере линии HEK293/ΔIP3R2/ΔIP3R1, в клетках которой были инактивированы гены IP3R2 и IP3R1. Аналогично были получены линии HEK293/ ΔIP3R3/ΔIP3R2 и HEK293/ΔIP3R1/ΔIP3R3, в клетках которых функциональными оставались лишь IP3R1 и IP3R2, соответственно. В целом получение стабильной клеточной линии в каждом случае было проведено по схеме, представленной на рис. 1.
Рис. 1.
Основные этапы получения и верификации биаллельных мутаций, приводящих к инактивации целевого гена. а – Создание генетических конструкций; б – получение и идентификация клеточных моноклонов; в – анализ характера и локализации мутаций.
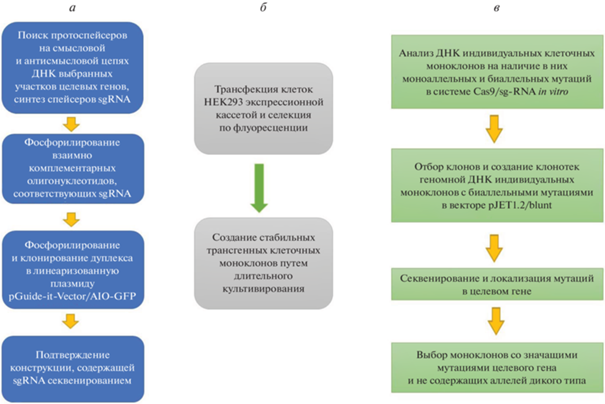
Инактивация двух из трех генов IP3-рецепторов проводилась последовательно. В рассматриваемом случае на первом этапе был получен клеточный моноклон HEK293/ΔIP3R2 с биаллельными мутациями, приводящими к смещению открытой рамки считывания IP3R2 человека, затем в клетках этой линии с использованием системы CRISPR/Cas9-D10A был инактивирован ген IP3R1. Для редактирования IP3R2 был использован вектор pGuide-it-td Tomato (Takara), обеспечивающий синтез sg-РНК для IP3R2 и нуклеазы Cas9. Красный флуоресцентный белок tdTomato, который экспрессировался независимо, являлся индикатором успешной трансфекции и обеспечивал эффективный первичный отбор клонов для последующего анализа мутаций. Участок для редактирования был выбран вблизи ATG-кодона IP3R2 в последовательности первого экзона.
После подтверждения интеграции олигонуклеотидного дуплекса, кодирующего sg-РНК, полученной плазмидой трансфицировали клетки HEK-293. Моноклоны анализировали на наличие мутаций в два этапа: 1) тестированием in vitro доступности редактируемого участка для гидролиза Cas9 в комплексе с соответствующей sg-РНК и 2) секвенированием редактируемой области в клонотеках, полученных для ПЦР-продуктов гена-мишени.
Первый этап, RGEN-RFLP, (RNA-guided engineered nucleases) существенно сужает круг моноклонов, для которых целесообразно приготовление клонотек и последующая процедура секвенирования [15]. Этот метод гораздо более точно отражает число событий редактирования, чем другие методы оценки эффективности работы систем CRISPR/Cas9 при геномном редактировании [16]. Суть метода заключается в том, что аналогично процессу, произошедшему в клетках, в условиях in vitro проводится внесение разрыва комплексом Cas9/sg-RNA на амплифицированном фрагменте гена (рис. 2). Праймеры для амплификации участка гена подбираются таким образом, чтобы место предполагаемой мутации было ассиметрично, и продукты гидролиза эффективно разделялись в агарозном геле. Во фрагментах ДНК дикого типа визуализируется два продукта неравной длины, поскольку комплекс Cas9/sg-RNA узнает как PAM, так и последовательность протоспейсера и вносит двунитевой разрыв. Три продукта на электрофореграмме свидетельствуют о наличии моноаллельной мутации в целевом участке: одна аллель была отредактирована и не подвергалась гидролизу (один непорезанный продукт), участок на второй неизмененной аллели был доступен для гидролиза (два продукта). Отсутствие гидролиза указывает на наличие в ДНК биаллельной мутации, приводящей к изменению последовательности, узнаваемой комплексом Cas9/sg-RNA (рис. 2).
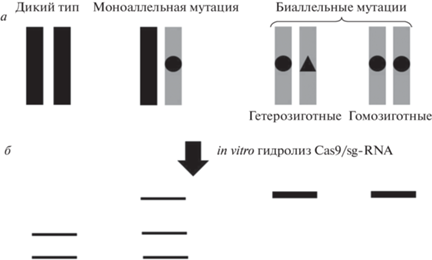
Рис. 2.
Варианты гидролиза амплифицированных фрагментов ДНК моноклонов после после обработки in vitro комплексом Cas9/sg-RNA. а – Типы аллельных мутаций; б – ожидаемые продукты расщепления, соответствующие типу мутаций. Представлены три различные картины рестрикции, получаемые на электрофореграмме. Если мутации присутствуют в аллели, то стандартный Cas9-sgRNA комплекс не сможет расщепить ПЦР-фрагмент, содержащий редактируемую область, тогда как аллели дикого типа будут опознаны и порезаны.
Для линии HEK293/ΔIP3R2 моноаллельные мутации выявляются на дорожках под номерами 1, 2, 4, 6 и 7; дикий тип – 3; биаллельные мутации – 5, 8 и 9 (рис. 3а). Клон, соответствующий паттерну разрезания, приведенному на дорожке 5, был выбран для получения клонотеки ПЦР-продуктов и последующего секвенирования.
Рис. 3.
Примеры гидролиза ПЦР-фрагментов, содержащих редактируемый участок ДНК, in vitro комплексом Cas9/sg-RNA. ДНК-маркер (М) – 1 kb Plus Ladder (Ферментас). а – Гидролиз фрагментов геномной ДНК для IP3R2, амплифицированных из индивидуальных моноклонов (дорожки 1–9). б – гидролиз фрагментов геномной ДНК для IP3R1, амплифицированных из индивидуальных моноклонов (дорожки 1–10).
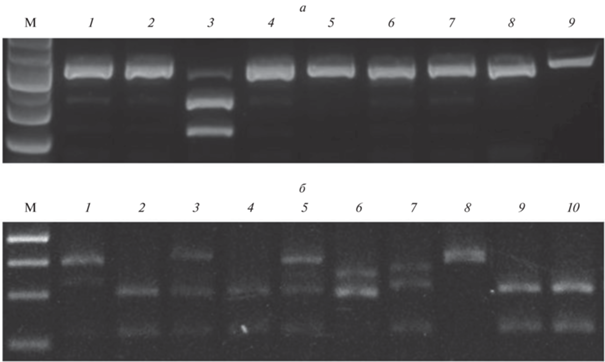
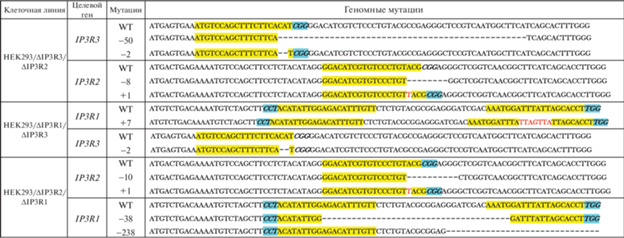
В моноклоне с генотипом HEK293/ΔIP3R2 было проведено редактирование гена IP3R1. Для минимизации off-target эффектов была использована система Cas9‑D10А с никазной активностью в составе вектора AIO-GFP [14]. Действие фермента Cas9-D10A, приводящее к двунитевому разрыву ДНК, обеспечивается кооперацией двух направляющих sg-РНК, что повышает точность взаимодействия редактирующего комплекса с мишенью [17]. Протоспейсеры для CRISPR/Cas9-10А, содержащие на 3'-конце смысловой цепи мотив PAM – ТGG и на 3'-конце антисмыловой цепи мотив PAM – CCТ, были выбраны в третьем экзоне гена IP3R1 справа от ATG-кодона (табл. 1). Вектор на основе AIO-GFP, обеспечивающий экспрессию “смыслового” и “антисмыслового” протоспейсеров использовали для трансфекции линии клеток HEK293/ΔIP3R2. Тестирование in vitro (рис. 3б) указывает на наличие биаллельной мутации в одном клоне (дорожка 8). Моноаллельным мутациям соответствуют дорожки 1, 3, 5, 6, 7; дикому типу – 2, 4, 9, 10. После секвенирования клонотеки с фрагментом гена IP3R1 для выбранного клона установлено, что одна аллель имеет делецию 38 пар нуклеотидов, приводящую к смещению открытой рамки считывания и появлению стоп-кодона после 14-й аминокислоты, во второй аллели обнаружена делеция 238 пар нуклеотидов, приводящая к смещению открытой рамки считывания и появлению стоп-кодона после 70-й аминокислоты (табл. 1). При получении двойных мутаций HEK-293/ΔIP3R2/ΔIP3R1 инвариантность мутаций, внесенных на первом этапе при редактирования гена IP3R2, вновь подтверждали секвенированием клонотек для ПЦР-продуктов IP3R2. Этот шаг служил для проверки стабильности клеточной линии с одним мутированным геном при последующих редактировании и культивировании. Результаты секвенирования подтверждали устойчивость первоначальной indel-мутации и демонстрировали, что в течение нескольких пассажей не происходит реверсии внесенных изменений.
Таблица 1.
Нуклеотидная последовательность участков целевого гена после редактирования клеточных линий HEK293/ΔIP3R3/ΔIP3R2, HEK293/ΔIP3R1/ΔIP3R3, HEK293/ΔIP3R2/ΔIP3R1
Таким образом, с помощью технологии CRISPR/ Cas9 получена стабильная линия клеток HEK293/ ΔIP3R2/ΔIP3R1 с биаллельными мутациями генов IP3R2 и IP3R1, приводящими к их инактивации, при сохранении функционального гена IP3R3.
Получение линии клеток HEK293/ΔIP3R3/ΔIP3R2
В данном случае первоначально инактивировался ген IP3R3 с использованием вектора pGuide-it-tdTomato Vector/IP3R3. Участок геномной ДНК, выбранный для редактирования, представляет собой GC-богатую последовательность, что затрудняло амплификацию. Для оптимизации амплификации участка редактированного гена применяли полимеразу Qiagen Taq Hot Start DNA polymerase, буфер с добавлением Q-solution и подбирали оптимальный температурный режим. После двухэтапного тестирования был отобран моноклон с биаллельными мутациями, приводящими к смещению открытой рамки считывания гена IP3R3 (табл. 1). На одной аллели обнаружена делеция 50 п.н., приводящая к образованию укороченного белка – 16 аминокислот. В другой аллели имеется делеция 2 п.н., приводящая к смещению открытой рамки считывания после 9-й аминокислоты и появлению стоп-кодона после 32-й аминокислоты.
В полученной линии HEK293/ΔIP3R3 с использованием вектора pGuide-it-tdTomato Vector/ IP3R2 затем инактивировался ген IP3R2. Инактивирующие мутации включали делецию 8 п.н., приводящую к смещению открытой рамки считывания после 17-й аминокислоты, и появление стоп-кодона после 31-й аминокислоты, а также инсерцию 1 п.н., приводящую к смещению открытой рамки считывания после 17-й аминокислоты и появлению стоп-кодона после 34-й аминокислоты (табл. 1). В полученной линии (HEK293/ ΔIP3R2/ΔIP3R3) интакным остается ген IP3R1.
Появление одной и той же мутации (инсерция 1 п.н. в гене IP3R2) при независимых манипуляциях (получение линий HEK293/ΔIP3R2/ΔIP3R1 и HEK293/ΔIP3R3/ΔIP3R2) с редактируемым участком с использованием одного и того же инструмента не является неожиданным эффектом. Характер мутации зависит от особенностей используемой ферментативной системы, и для Cas9 описана высокая частота как делеций > 3 п.н. (58%), так и инсерционных замен 1 п.н. (13%), в то время как протяженные инсерции встречаются гораздо реже (около 1%) [18, 19].
Получение линии клеток HEK293/ΔIP3R1/ΔIP3R3
При получении данной клеточной линии сначала нокаутировали ген IP3R1 с помощью плазмиды AIO-GFP/IP3R1, а затем редактировали ген IP3R3 с использованием вектора pGuide-it-tdTomato/IP3R3. После первого этапа редактирования был отобран моноклон, имеющий только один вариант мутации (гомозигота) по гену IP3R1, который воспроизводимо идентифицируется в нескольких секвенированных клонотеках: инсерция 7 п.н., приводящая к смещению открытой рамки считывания после 28-й аминокислоты и появлению стоп-кодона (табл. 1).
Эту клеточную линию (HEK293/ΔIP3R1) использовали на втором этапе последовательного редактирования с использованием плазмиды pGuide-it-tdTomato/IP3R3. В полученном моноклоне выявлена гомозиготная мутация – делеция 2 п.н., приводящая к смещению открытой рамки считывания после 9-й аминокислоты и появлению стоп-кодона после 32-й аминокислоты (табл. 1). Таким образом, полученная клеточная линия HEK293/ΔIP3R1/ΔIP3R3 имеет по обоим генам биаллельные идентичные мутации, инактивирующие трансляцию обоих белков.
Полученные клеточные линии были протестированы на устойчивость направленных мутаций через 1 год хранения при –80°С. Для каждого гена-мишени были приготовлены и секвенированы клонотеки ПЦР-фрагментов редактированных областей. Во всех случаях было подтверждено наличие полученных ранее инактивирующих мутаций и отсутствие дополнительных нуклеотидных замен.
ОБСУЖДЕНИЕ
Первичные медиаторы, вовлеченные в паракринные и автокринные регуляции, модулируют клеточные функции, взаимодействуя с поверхностными рецепторами и стимулируя сопряженные с ними внутриклеточные сигнальные каскады. Трансдукция многих агонистов, рецепторы которых сопряжены с фосфоинозитидным каскадом, сопровождается мобилизацией внутриклеточного Ca2+ [3, 7, 20]. Ключевым событием в трансдукции Са2+-мобилизующих агонистов является стимуляция фосфолипазы С (PLC), которая гидролизует липидный субстрат фосфатидилинозитол-4,5-бифосфат и продуцирует два вторичных медиатора, IP3 и диацилглицерин. Первичная роль IP3 состоит в активации IP3-рецепторов и высвобождении Ca2+ из Ca2+-депо [3, 7]. Помимо связывания коагонистов IP3 и Ca2+, активность IP3-рецепторов модулируется рядом механизмов, включая фосфорилирование несколькими киназами и взаимодействие со специализированными белками [21]. Это обстоятельство, а также то, что в клетках обычно функционируют несколько изоформ IP3-рецепторов [8], затрудняют анализ их регуляции и физиологической роли в нативных клетках. Существенно более однозначную интерпретацию результатов могут обеспечить клеточные модели с единственной функциональной изоформой IP3-рецепторов.
Первая такая модель была разработана на основе клеток DT40, полученных из лимфомы цыпленка, в которых были инактивированы собственные гены IP3-рецепторов (DT40-TKO клетки) [22]. Гетерологическая экспрессия индивидуальных изоформ IP3-рецепторов млекопитающих в клетках DT40-TKO позволила выявить многие особенности функционирования этих IP3-активируемых Са2+-каналов. В частности, было показано, что обмен Са2+ между ЭР и митохондриями через IP3-рецепторы необходим для поддержания биоэнергетики клетки [23]. Ряд фактов свидетельствовал о том, что, хотя все изоформы IP3-рецепторов могут участвовать в формировании контакта между ЭР и митохондриями, IP3R2 обеспечивает наиболее эффективный перенос Ca2+ в митохондрии [24]. Используя пермеабилизованные клетки DT40-TKO, экспрессирующие индивидуальные изоформы IP3-рецепторов, были получены кривые доза–ответ, характеризующие выброс Са2+, который индуцировался IP3 и его синтетическими аналогами [25]. Фактически это было первое исследование связи структуры лиганда с активностью IP3-рецепторов. Анализ Ca2+-сигналов в клетках DT40-TKO с рекомбинантными IP3-рецепторами млекопитающих позволил прийти к заключению, что IP3-индуцированный выброс Ca2+ достаточно эффективен только, если каждая субъединица тетрамерного IP3-рецептора связана с IP3 [26].
Клетки различных линий в культуре представляют собой традиционный объект для исследований рецепторных систем и сигнальных процессов. По сравнению с нативными клетками, выделенными из диссоциированных биологических тканей, культивируемые клетки более стабильны, и их можно эффективно модифицировать за счет гетерологичной экспрессии целевых белков или редактирования целевых генов. Клетки линии HEK-293 часто используются для клеточных исследований, поскольку они экспрессируют широкий набор сигнальных и регуляторных белков, остаточно стабильны при различных условиях культивирования, быстро достигают требуемой плотности (время удвоения культуры 36 ч). Кроме того, клетки HEK-293 с высокой эффективностью могут быть трансфицированы разными методами (химическая и электрическая трансфекция) с использованием плазмидных и вирусных векторов и обеспечивают точную трансляцию рекомбинантных белков и достаточную продуктивность их синтеза. Совокупность этих характеристик делает клетки линии HEK-293 хорошей тест-системой для экспериментов, сочетающих функциональные физиологические тесты и гетерологичную экспрессию белков, вовлеченных в сигнальные процессы [27].
В данной работе мы использовали клетки HEK-293, CRISPR/Cas9 технологию и методы клеточной селекции для генерации моноклональных клеточных линий с единственной функциональной изоформой IP3-рецептора. Были получены стабильные клеточные моноклоны HEK293/ΔIP3R2/ΔIP3R1, HEK293/ΔIP3R1/ΔIP3R3, HEK293/ΔIP3R3/ΔIP3R2 с биаллельными мутациями, приводящими к сдвигу открытой рамки считывания, с использованием одного sg-РНК для генов IP3R3 и IP3R2, двух sg-РНК для никазной системы редактирования гена IP3R1. Описан простой и удобный способ выявления полученных мутаций в модели in vitro при гидролизе участков геномной ДНК моноклонов комплексом Cas9-sgRNA, с подтверждением целевых мутаций секвенированием.
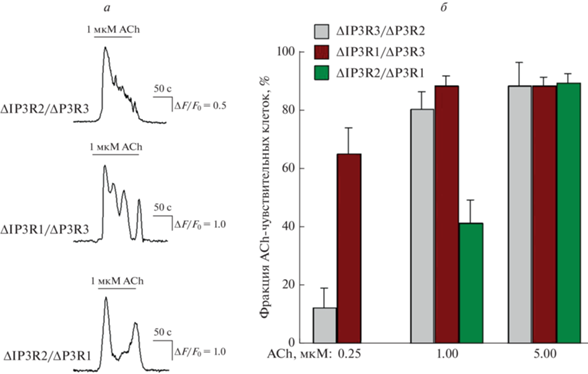
Физиологические тесты показали, что клетки в этих линиях способны отвечать на агонисты мобилизацией Са2+. В случае ACh этот агонист мобилизовал Са2+ примерно в 90% популяции клеток каждой из полученных линий (рис. 4). Это свидетельствует о том, что фосфоинозитидный каскад с единственной функциональной изоформой IP3-рецептора способен обеспечить сопряжение клеточных рецепторов с мобилизацией Са2+. В дальнейшем полученные нами клеточные линии могут быть использованы для исследования роли индивидуальных изоформ IP3-рецептора во внутриклеточной Са2+-сигнализации.
Рис. 4.
Ca2+-ответы клеток на ACh. а – Репрезентативнный мониторинг внутриклеточного Са2+ в клетках HEK293/ΔIP3R2/ΔIP3R1 (n = 62), HEK293/ΔIP3R1/ΔIP3R3 (n = 51) и HEK293/ΔIP3R3/ΔIP3R2 HEK-293WT (n = 58), загруженных Fluo-4. Моменты и продолжительность аппликаций ACh (1 мкМ) обозначены горизонтальными линиями выше экспериментальных кривых. Изменение внутриклеточного Са2+ оценивали по относительному изменению флуоресценции Fluo-4 ΔF/F0, где ΔF = F – F0, F – текущая интенсивность флуоресценции, F0 – средняя интенсивность флуоресценции в начальный момент регистрации. б – Фракция клеток для каждой из линий, генерирующих Са2+-ответы на ACh при концентрациях 0.25, 1 и 5 мкМ.
Авторы благодарят Д.М. Поташникову за помощь в проведении работ по сортировке клеток (поддерживаются Программой развития МГУ). Авторы признательны И.С. Масулис и Ю.А. Ковалицкой за плодотворную дискуссию и сделанные замечания.
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Источники финансирования. Работа выполнена при поддержке гранта РНФ № 22-14-00031.
Соответствие принципам этики. Настоящая статья не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Список литературы
Foskett J.K., White C., Cheung K.-H., Mak D.O. 2007. Inositol trisphosphate receptor Ca2+ release channels. Physiol Rev. 87, 593–658.
Berridge M.J., Bootman M.D., Roderick H.L. 2003. Calcium signalling: dynamics, homeostasis and remodelling. Nat. Rev. Mol. Cell. Biol. 4, 517–529.
Clapham D.E. Calcium signaling. 2007. Cell 131, 1047– 1058.
Zampese E., Pizzo P. 2012. Intracellular organelles in the saga of Ca2+ homeostasis: different molecules for different purposes? Cell. Mol. Life Sci. 69, 1077–1104.
Takeshima H., Venturi E., Sitsapesan R. New and notable ion-channels in the sarcoplasmic/endoplasmic reticulum: Do they support the process of intracellular Ca2+ release? 2015. J. Physiol. 593, 3241–3251.
Carreras-Sureda A, Pihán P, Hetz C. 2018. Calcium signaling at the endoplasmic reticulum: Fine-tuning stress responses. Cell Calcium 70, 24–31.
Berridge M.J. 2016. The inositol trisphosphate/calcium signaling pathway in health and disease. Physiol. Rev. 96, 1261–1296.
Hamada K., Mikoshiba K. 2020. IP3 receptor plasticity underlying diverse functions. Annu. Rev. Physiol. 82, 151–176.
Milligan G., Inoue A. 2018. Genome editing provides new insights into receptor-controlled signalling pathways. Trends Pharm. Sci. 39, 481–493. https://doi.org/10.1016/j.tips.2018.02.005
Wijshake T., Baker D.J., van de Sluis B. 2014. Endonucleases: new tools to edit the mouse genome. Biochim. Bioph. Acta. 1842, 1942–1950.
Jiang F., Doudna J.A. 2017. CRISPR–Cas9 structures and mechanisms. Annu. Rev. Biophys. 46, 505–529.
Lock J.T., Alzayady K.J., Yule D.I., Parker I. 2018. All three IP3 receptor isoforms generate Ca2+ puffs that display similar characteristics. Sci. Signal. 11, eaau0344.
Sledzinski P., Dabrowska M., Nowaczyk M., Olejniczak M. 2021. Paving the way towards precise and safe CRISPR genome editing. Biotechnol Adv. 49, 107737.
Chiang T.W., le Sage C., Larrieu D., Demir M., Jackson S.P. 2016. CRISPR-Cas9(D10A) nickase-based genotypic and phenotypic screening to enhance genome editing. Sci. Rep. 6, 24356.
Kim J.M., Kim D., Kim S., Kim J.S. 2014. Genotyping with CRISPR-Cas-derived RNA-guided endonucleases. Nat. Commun. 5, 3157.
Ломов Н.А., Вьюшков В.С., Петренко А.П., Сыркина М.С., Рубцов М.А. 2019. Методы оценки эффективности работы систем CRISPR/Cas 9 при геномном редактировании. Мол. биол. 53, 982–997.
Смирнов А.В., Юнусова А.М., Лукьянчикова В.А., Баттулин Н.Р. 2016. Система CRISPR/Cas9 – универсальный инструмент геномной инженерии. Вавиловский журн. генетики и селекции. 20, 493–510.
Allen F., Crepaldi L., Alsinet C., Strong A.J., Kleshchevnikov V., De Angeli P., Páleníková P., Khodak A., Kiselev V., Kosicki M., Bassett A.R., Harding H., Galanty Y., Muñoz-Martínez F., Metzakopian E., Jackson S.P., Parts L. 2018. Predicting the mutations generated by repair of Cas9-induced double-strand breaks. Nat. Biotechnol. 27, 10.
Bennett E.P., Petersen B.L., Johansen I.E., Niu Y., Yang Z., Chamberlain Ch.A., Met Ö., Wandall H.H., Frödin M. 2020. INDEL detection, the ‘Achilles heel’ of precise genome editing: A survey of methods for accurate profiling of gene editing induced indels. Nucleic Acids Res. 48, 11958–11981.
Lemmon M.A., Schlessinger J. 2010. Cell signaling by receptor tyrosine kinases. Cell. 141, 1117–1134.
Parys J.B., Vervliet T. 2020. New insights in the IP3 receptor and its regulation. Adv. Exp. Med. Biol. 1131, 243–270.
Sugawara H., Kurosaki M., Takata M., Kurosaki T. 1997. Genetic evidence for involvement of type 1, type 2 and type 3 inositol 1,4,5-trisphosphate receptors in signal transduction through the B-cell antigen receptor. EMBO J. 16, 3078–3088.
Cardenas C., Miller R.A., Smith I., Bui T., Molgó J., Müller M., Vais H., Cheung K.H., Yang J., Parker I., Thompson C.B., Birnbaum M.J., Hallows K.R., Foskett J.K. 2010. Essential regulation of cell bioenergetics by constitutive InsP3 receptor Ca2+ transfer to mitochondria. Cell. 142, 270–283.
Bartok A., Weaver D., Golenár T., Nichtova Z., Katona M., Bánsághi S., Alzayady K.J., Thomas V.K., Ando H., Mikoshiba K., Joseph S.K., Yule D.I., Csordás G., Hajnóczky G. 2019. IP3 receptor isoforms differently regulate ER-mitochondrial contacts and local calcium transfer. Nat. Commun. 10, 3726.
Saleem H., Tovey S.C., Rahman T., Riley A.M., Potter B.V., Taylor C.W. 2013. Stimulation of inositol 1,4,5-trisphosphate (IP3) receptor subtypes by analogues of IP3. PLoS One. 8, 54877.
Alzayady K.J., Wang L., Chandrasekhar R., Wagner II L.E., Petegem F.V., Yule D.I. 2016. Defining the stoichiometry of inositol 1,4,5-trisphosphate binding required to initiate Ca2+ release. Sci. Signal. 9, ra35.
Hu J., Han J., Li H., Zhang X., Liu L.L., Chen F., Zen-g B. 2018. Human embryonic kidney 293 cells: A vehicle for biopharmaceutical manufacturing, structural biology, and electrophysiology. Cells Tissues Organs. 205, 1–8.
Дополнительные материалы отсутствуют.
Инструменты
Биологические мембраны: Журнал мембранной и клеточной биологии


