Биология моря, 2019, T. 45, № 1, стр. 44-51
Сборка и анализ референсной базы нуклеотидных последовательностей митохондриального гена COI для разграничения видов песчанок (Uranoscopiformes: Ammodytidae) Северного полушария
1 Институт биологии моря им. А.В. Жирмунского, Национальный научный центр морской биологии
(ННЦМБ) ДВО РАН
690041 Владивосток, Россия
2 Дальневосточный государственный технический рыбохозяйственный университет
690087 Владивосток, Россия
* E-mail: sturcoal@mail.ru
Поступила в редакцию 28.03.2018
После доработки 19.06.2018
Принята к публикации 18.10.2018
Аннотация
Песчанки Северного полушария (Uranoscopiformes: Ammodytidae) – рыбы шельфовой зоны, характеризующиеся криптическим разнообразием на видовом уровне и демонстрирующие парафилетические отношения на уровне родов, в последнее время привлекают внимание таксономистов в связи с широким распространением молекулярно-генетических методов в систематике. В настоящей работе сформирована и проанализирована референсная библиотека нуклеотидных последовательностей митохондриального гена COI восьми видов песчанок рода Ammodytes и двух видов рода Hyperoplus с применением дистанционных (ABGD – сопоставление генетических расстояний) и топологических (PTP) критериев. Показана надежность данных методов идентификации при высоких значениях межвидовой дивергенции (пять видов Ammodytes и один вид Hyperoplus), тогда как для видов, проявляющих незначительную взаимную дивергенцию (A. tobianus, H. lanceolatus), топологические критерии превосходят дистанционные. Вид A. personatus впервые отмечен в восточной части Охотского моря.
Песчанки Северного полушария (Uranoscopiformes: Ammodytidae) хорошо изучены с позиций классической таксономии (Kitaguchi, 1979; Ida et al., 1994; Robards et al., 1999; Mecklenburg et al., 2002; Hatooka, 2002; Orr et al., 2015; Thiel, Knebelsberger, 2016). В настоящее время они представлены 10 видами, которые объединены в два рода: Ammodytes Linnaeus, 1758 и Hyperoplus Günther, 1862 (Fricke et al., 2018). Песчанки – стайные рыбы-планктофаги, населяющие шельфовую зону Тихого, Атлантического и Северного Ледовитого океанов (Мельников, Худя, 1998; Robards et al., 1999; Парин и др., 2014). Виды этих родов играют существенную роль в питании большинства ихтиофагов Северного полушария (Sherman et al., 1981; Springer, Speckman, 1997; Мельников, Худя, 1998; Чикелев, Датский, 2000; Черешнев и др., 2001), а также являются важным объектом промысла во многих странах.
Молекулярно-генетические (Kim et al., 2006, 2010; Han et al., 2012; Li et al., 2013; Orr et al., 2015) и биохимические (Okamoto, 1989) методы давно используются для изучения разнообразия песчанок. Первой на возможное криптическое разнообразие и недостаточную информативность морфологических признаков указала Мекленбург (Mecklenburg et al., 2011) при формировании библиотеки ДНК-штрихкодов рыб Арктики. Позже с применением аналогичных методов удалось восстановить ареалы современных песчанок рода Ammodytes, исключив их амфипацифическое распространение и номенклатурные противоречия в отношении вида A. personatus Girard, 1856 (Туранов, Картавцев, 2014; Turanov et al., 2016). Данные о количестве видов песчанок и сведения об их ареалах были подтверждены в ходе проведения комплексного анализа с описанием нового вида рода (Orr et al., 2015). В результате изучения видового разнообразия песчанок в Северном и Балтийском морях было показано, что морфологически обособленный род Hyperoplus является парафилетическим (Thiel, Knebelsberger, 2016), обнаруживая минимальные различия по отношению к виду A. tobianus Linnaeus, 1758 на уровне изменчивости нуклеотидных последовательностей гена COI. Продолжающееся накопление данных посредством глобального ДНК-штрихкодирования (Hebert et al., 2003) обусловливает необходимость формирования унифицированной референсной библиотеки для возможности идентификации видов песчанок Северного полушария на основе современных методов молекулярной систематики. В настоящей работе обсуждаются преимущества и недостатки использования митохондриального маркера COI для разграничения видов песчанок.
МАТЕРИАЛ И МЕТОДИКА
При формировании референсной библиотеки для идентификации песчанок использовали набор данных, включавших как оригинальные последовательности митохондриального гена COI (44 последовательности от трех видов из семи локальностей; Баренцево, Охотское и Берингово моря, 2008–2015 годы вылова), так и привлеченные из NCBI (Benson et al., 2018) и BOLD (Ratnasingham, Hebert, 2007, 2013) (рис. 1, табл. 1). Внутривидовая выборка составляла от трех (Ammodytes americanus DeKay, 1842) до 53 (A. hexapterus Pallas, 1814) последовательностей (среднее количество – 6). Тотальную ДНК выделяли из фиксированной в 95% этиловом спирте ткани скелетных мышц песчанок, используя набор К-Сорб (Синтол, Москва). Амплификация последовательностей гена COI на основе праймеров FishF1 и FishR1 (Ward et al., 2005) выполнена в реакционной смеси объемом 25 мкл, включавшей 5 мкл 5x Taq Red буфера (Евроген), 0.5 мкл 50x смеси dNTP (Евроген, Москва), 0.1 мкл Taq ДНК полимеразы (Евроген), 1 мкл раствора тотальной ДНК образца, по 0.12 мкМ прямого и обратного праймеров, а также деионизированную воду до конечного объема. Термический алгоритм полимеразной цепной реакции (ПЦР) включал предварительный нагрев при температуре 94°C в течение 2 мин и 35 циклов по следующей схеме: денатурация при 94°C – 50 с, отжиг при 52°C – 40 с, элонгация при 72°C – 1 мин и заключительная элонгация при 72°C в течение 10 мин. Качество амплифицированных фрагментов проверяли посредством электрофореза в 1% агарозном геле с последующей экспозицией в растворе бромистого этидия и с визуализацией в проходящем ультрафиолетовом свете. Циклосеквенирование ампликонов выполнено с использованием набора BrightDye™ Terminator Cycle Sequencing Kit v3.1 (NimaGen) в прямом и обратном направлениях с праймерами, задействованными при амплификации. Капиллярный электрофорез фрагментов проведен на базе секвенатора ABI Prism 3500 DNA sequencer в ННЦМБ ДВО РАН. Сборку консенсусных последовательностей проводили в программе ChromasPro 1.34 (ChromasPro; http://www.technelysium.com.au/chromas.html/). Данные о проанализированном материале хранятся в открытом доступе (табл. S1 )11. Конечная матрица состояла из 151 последовательности от восьми видов рода Ammodytes и двух видов рода Hyperoplus. В качестве внешней группы использованы Uranoscopus fuscomaculatus Kner, 1868 и Kathetostoma averruncus Jordan & Bollman, 1890. Последовательности выравнены в программе MEGA 7 (Kumar et al., 2016) с использованием алгоритма Muscle (Edgar, 2004). Генетические расстояния с коррекцией по модели нуклеотидных замещений рассчитаны в MEGA 7. Для обеспечения надежности топологического критерия при определении видовых границ песчанок использовано несколько методов филогенетических построений: метод ближайшего соседства (NJ), максимальной парсимонии (MP) и байесовский метод (BI). Поиск оптимальных топологий NJ и MP выполнен с использованием соответствующего набора функций из пакетов APE (Paradis et al., 2004) и phangorn (Schliep, 2011) в среде R (R Core Team, 2017). Устойчивость топологии оценивали посредством 1000 реплик непараметрического бутстреп-теста с конденсированием узлов, существующих в менее 50% полученных топологий. Байесовское филогенетическое древо построено в программе MrBayes 3.2 (Ronquist et al., 2012) с конденсированием узлов, маргинальные значения апостериорной вероятности которых составляли менее 50%. Поиск оптимальной модели нуклеотидных замещений (TrN + G) с использованием информационного критерия Акаике выполнен при помощи функции modeltest пакета phangorn (Schliep, 2011). Количество операционных таксономических единиц было оценено также с использованием альтернативных дистанционных – ABGD (Puillandre et al., 2012) и топологических – PTP (Zhang et al., 2013) методов. Анализ ABGD выполнен с выставлением порога априорных значений межвидовой дивергенции от 0.001 до 0.010 при коррекции генетических расстояний по модели K2P (Kimura, 1980) и без коррекции. Генерализованный анализ разграничения видов на основе стартовой топологии, предварительно полученной BI-методом, был выполнен в веб-приложении bPTP server (Zhang et al., 2013). Произведен запуск в 500 000 поколений цепей Маркова с частотой выборки 1 на 100 поколений. Первые 500, приходившиеся на отжиг, были отброшены. По результатам предварительного запуска, следуя рекомендациям разработчиков, из анализа были исключены ветви внешней группы с повторным проведением расчетов. Результаты анализа (рис. 3) визуализированы с помощью PhyloMap (Zhang et al., 2011).
Рис. 1.
Карта Северного полушария с указанием мест поимки песчанок, использованных в анализе. Цифрами обозначены номера выборок материала автора (табл. 1); более подробная информация о материале доступна в табл. S1 .
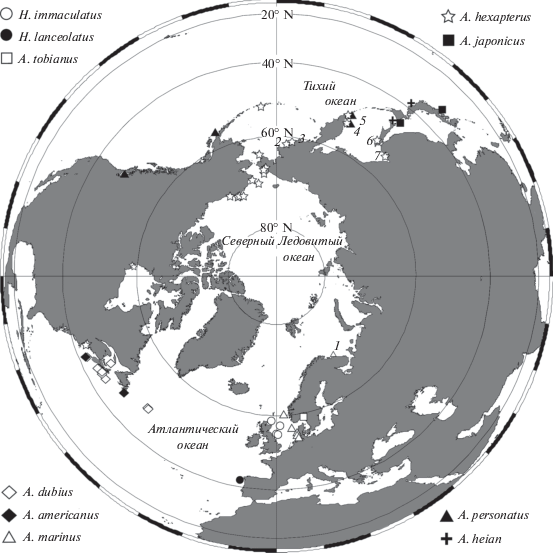
Рис. 2.
Филогенетическое BI-дерево, укорененное по средней точке. Иллюстрирует филогенетические отношения между видами песчанок Северного полушария на основе изменчивости последовательностей гена COI. В узлах приведены оценки устойчивости топологии, выраженные в процентах.
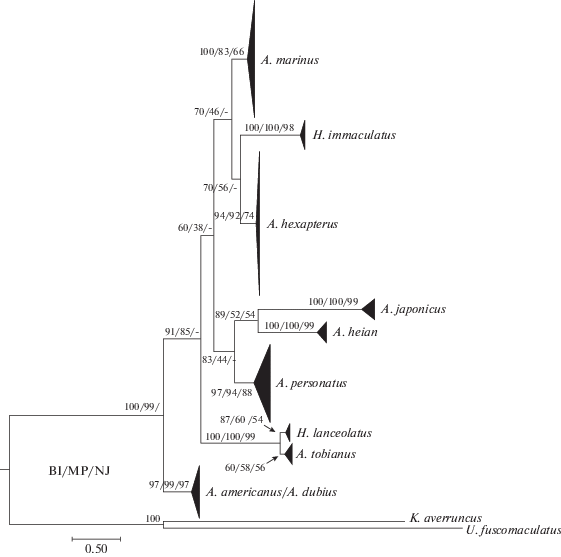
Рис. 3.
Визуализация (PhyloMap) результатов генерализованного анализа разграничения видов (bPTP) на основе наилучшей байесовской топологии (рис. 2) взаимоотношений песчанок Северного полушария.
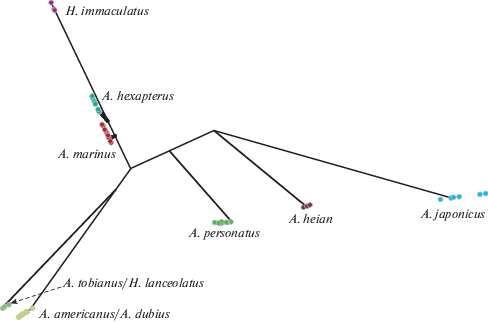
Таблица 1.
Характеристика выборок песчанок из соответствующих локальностей (подробная информация представлена в табл. S1 )
| Вид | Номер выборки* | Место сбора | Год | Объем выборки, экз. |
|---|---|---|---|---|
| Ammodytes americanus | – | Провинция Ньюфаундленд и Лабрадор | 2002, 2006 | 3 |
| A. dubius | – | Новая Шотландия | 2005–2007 | 12 |
| A. heian | – | О-в Хоккайдо, Вакканай | 2015 | 6 |
| – | О-в Хонсю, б. Оппа | 2008 | 1 | |
| A. hexapterus | 7 | Охотское море, Шантарские острова, б. Якшина | 2010 | 8 |
| 6 | Охотское море, о-в Сахалин, зал. Пильтун | 2015 | 2 | |
| 4 | Охотское море, юго-западное побережье п-ва Камчатка | 2015 | 1 | |
| 2 | Берингово море, восточная часть | 2015 | 11 | |
| 3 | Берингово море, восточная часть | 2008 | 2 | |
| 5 | Охотское море, юго-западное побережье п-ва Камчатка | 2011 | 1 | |
| – | Берингово море, северная часть | 2007 | 1 | |
| – | Чукотское море | 2007, 2009, 2012 | 7 | |
| – | Море Бофорта | 2011, 2012 | 6 | |
| – | Атлантический океан, зал. Мэн, р. Сако | 2014, 2016 | 2 | |
| – | Гудзонов залив, Черчилл | 2004 | 3 | |
| – | Алеутские острова, о-в Атка | 2012 | 9 | |
| A. japonicus | – | О-в Хоккайдо, Вакканай | 2015 | 4 |
| – | О-в Хонсю, б. Осака | 2006 | 1 | |
| A. marinus | – | Северное море | 2010, 2011 | 24 |
| 1 | Баренцево море | 2013 | 12 | |
| A. personatus | 4 | Охотское море, юго-западное побережье п-ва Камчатка | 2015 | 5 |
| 5 | Охотское море, юго-западное побережье п-ва Камчатка | 2011 | 4 | |
| – | Тихий океан, северо-восточная часть, море Селиш | 2006, 2013 | 3 | |
| – | Зал. Аляска, о-в Агаюк | 2007 | 2 | |
| A. tobianus | – | Северное море, прол. Скагеррак | 2009 | 6 |
| Hyperoplus immaculatus | – | Северное море | 2011 | 8 |
| H. lanceolatus | – | Атлантический океан, побережье Португалии | 2005 | 5 |
РЕЗУЛЬТАТЫ
Длина конечной матрицы последовательностей составила 701 пару нуклеотидов (пн), из которых 233 были изменчивыми (141 парсимонийно информативных и 72 синглтонных). Внутривидовые генетические расстояния изменялись от 0 (A. hexapterus) до 0.007 (A. japonicus Duncker & Mohr, 1939) при среднем значении 0.002, в то время как межвидовые расстояния варьировали от 0.038 [без учета дивергенции между A. tobianus и H. lanceolatus (Le Sauvage, 1824)] до 0.204 при среднем значении 0.09 (табл. 2). Топология полученного дерева (рис. 2) позволяет выявить 9 кластеров видового уровня (здесь и далее без учета внешней группы) с высокой поддержкой во всех алгоритмах построения. Кластер с последовательностями, принадлежащими видам A. americanus и A. dubius Reinhardt, 1837, не обнаруживает межвидовой изменчивости (значение дивергенции 0.002). Виды A. tobianus и H. lanceolatus формируют наиболее тесный общий кластер. В то же время H. immaculatus (Corbin, 1950) последовательно объединяется с A. hexapterus и A. marinus Raitt, 1934, что указывает на парафилетичность рода Hyperoplus. В общий кластер объединяются A. personatus, а также A. heian Orr, Wildes & Kai, 2015 и A. japonicus. Объединенный полифилетический кластер A. americanus и A. dubius занимает в данной схеме базальное положение по отношению ко всем остальным песчанкам. Узлы надвидового уровня менее надежны и поддержаны не во всех алгоритмах построения.
Таблица 2.
Внутривидовые (значения по диагонали) и межвидовые (все остальные) генетические расстояния между последовательностями COI песчанок с корректировкой значений по модели нуклеотидных замещений TrN + G
| Вид | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1. Ammodytes dubius/A. americanus | 0.002 | – | – | – | – | – | – | – | – | – | – |
| 2. A. marinus | 0.051 | 0.002 | – | – | – | – | – | – | – | – | – |
| 3. A. hexapterus | 0.057 | 0.018 | 0.000 | – | – | – | – | – | – | – | – |
| 4. A. personatus | 0.069 | 0.055 | 0.038 | 0.005 | – | – | – | – | – | – | – |
| 5. Hyperoplus immaculatus | 0.077 | 0.046 | 0.037 | 0.065 | 0.001 | – | – | – | – | – | – |
| 6. H. lanceolatus | 0.100 | 0.091 | 0.095 | 0.126 | 0.124 | 0.001 | – | – | – | – | – |
| 7. Ammodytes tobianus | 0.101 | 0.092 | 0.096 | 0.128 | 0.139 | 0.006 | 0.002 | – | – | – | – |
| 8. A. heian | 0.106 | 0.093 | 0.085 | 0.092 | 0.106 | 0.137 | 0.138 | 0.005 | – | – | – |
| 9. A. japonicus | 0.141 | 0.149 | 0.139 | 0.140 | 0.144 | 0.202 | 0.204 | 0.138 | 0.007 | – | – |
| 10. Uranoscopus fuscomaculatus | 0.860 | 0.975 | 1.098 | 1.018 | 0.919 | 1.260 | 1.262 | 0.958 | 1.254 | в.о. | – |
| 11. Kathetostoma averruncus | 0.913 | 0.962 | 0.979 | 0.951 | 1.110 | 1.318 | 1.296 | 1.211 | 1.746 | 0.908 | в.о. |
Анализ ABGD выявил 8 филогрупп (H. lanceolatus и A. tobianus попадают в одну группу) в широком диапазоне априорных значений внутривидовой дивергенции (от 0.001 до 0.01) при использовании коррекции по модели K2P, однако при использовании некорректированных генетических расстояний в зависимости от априорных значений внутривидовой дивергенции было найдено от восьми (0.001–0.052) до девяти (0.001–0.019) филогрупп. При этом в отдельную, группу 9, попадала последовательность из кластера A. personatus. Определение видовых границ c позиций ABGD ожидаемо совпадало c данными, основанными на анализе генетических расстояний, рассчитанных с использованием оптимальной модели нуклеотидных замещений (табл. 2), однако отличалось от топологического (рис. 1) ввиду объединения в группу видового ранга A. tobianus и H. lanceolatus.
Результаты генерализованного анализа разграничения видов (рис. 3) указывают на выявление восьми групп видового уровня. В данном случае, как и по результатам других подходов, к формированию единых видовых групп склонны последовательности A. tobianus и H. lanceolatus, а также A. americanus и A. dubius.
ОБСУЖДЕНИЕ
Анализ референсной базы нуклеотидных последовательностей песчанок Северного полушария показал высокую надежность любого из представленных методов разграничения видов песчанок при высоких значениях межвидовой дивергенции (пять видов Ammodytes и один вид Hyperoplus), тогда как для видов, проявляющих незначительную межвидовую дивергенцию (A. tobianus и H. lanceolatus), топологические критерии превосходят дистанционные. Это следует учитывать при идентификации видов песчанок, что предполагает обязательное использование полной референсной базы (табл. S1 ) с реконструкцией филогенетических отношений (рис. 2). Наши данные показывают, что фрагмент митохондриального гена COI, который успешно используется для идентификации песчанок, не подходит для выведения высокоустойчивой филогении этой группы в ее полном составе, что согласуется с ранее полученными результатами (Туранов, Картавцев, 2014). Необходимо также отметить, что особенности получаемой таким образом топологии (часто встречающаяся политомия в узлах надвидового уровня) не позволяют использовать методы разграничения видов, основанные на коалесценции (Fujisawa, Barraclough, 2013). Кроме того, последние данные о возможной интрогрессивной гибридизации песчанок в зоне вторичного контакта, формирующейся в районе слияния Куросио и Оясио (Kim et al., 2017), указывают на необходимость привлечения и других молекулярно-генетических маркеров для разграничения видов песчанок. Результаты анализа позволили получить новые сведения о распространении вида A. personatus в восточной части Охотского моря (юго-западное побережье п-ва Камчатка).
Автор выражает глубокую признательность П.П. Стрелкову (СПбГУ) за предоставление материала из Баренцева моря, а также А.Д. Кухлевскому (ННЦМБ ДВО РАН) за помощь при проведении секвенирования последовательностей COI.
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (проект № 16-34-00298, секвенирование и формирование референсной базы), а также гранта ДВО РАН № 18-4-040 (филогенетический анализ).
Список литературы
Мельников И.В., Худя В.Н. Дальневосточная песчанка (Ammodytes hexapterus Pallas) в Охотском и западной части Берингова морей // Изв. ТИНРО. 1998. Т. 124. С. 344–359.
Парин Н.В., Евсеенко С.А., Васильева Е.Д. Рыбы морей России: аннотированный каталог. М.: Товарищество науч. изд. КМК. 2014. 733 с.
Туранов С.В., Картавцев Ю.Ф. Таксономический состав и распространение песчанок рода Ammodytes (Perciformes: Ammodytidae) в северной части Тихого океана // Биол. моря. 2014. Т. 40. № 6. С. 455–462.
Черешнев И.А., Волобуев В.В., Хованский И.Е., Шестаков А.В. Прибрежные рыбы северной части Охотского моря. Владивосток: Дальнаука. 2001. 197 с.
Чикелев В.Г., Датский А.В. Дальневосточная многопозвонковая песчанка Ammodytes hexapterus (Ammodytidae) в Анадырском заливе и прилегающих водах // Вопр. ихтиологии. 2000. Т. 40. № 6. С. 772–779.
Benson D.A., Cavanaugh M., Clark K. et al. GenBank // Nucleic Acids Res. 2018. V. 46. № D1. P. D41–D47.
Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput // Nucleic Acids Res. 2004. V. 32. № 5. P. 1792–1797.
Fricke R., Eschmeyer W.N., van der Laan R. Catalog of fishes: genera, species, references. (http://researcharchive. calacademy.org/research/ichthyology/catalog/fishcatmain.asp). Electronic version accessed 25 March 2018.
Fujisawa T., Barraclough T.G. Delimiting species using single-locus data and the Generalized Mixed Yule Coalescent approach: a revised method and evaluation on simulated data sets // Syst. Biol. 2013. V. 62. № 5. P. 707–724.
Han Z., Yanagimoto T., Zhang Y., Gao T. Phylogeography study of Ammodytes personatus in northwestern Pacific: Pleistocene isolation, temperature and current conducted secondary contact // PLoS ONE. 2012. V. 7. № 5. e37425. doi 10.1371/journal.pone.0037425
Hatooka K. Ammodytidae // Fishes of Japan with pictorial keys to the species. Tokyo: Tokai Univ. Press. 2002. V. 2. P. 1974.
Hebert P.D.N., Cywinska A., Ball S.L., deWaard J.R. Biological identifications through DNA barcodes // Proc. Roy. Soc. B. 2003. V. 270. № 1512. P. 313–321. doi 10.1098/rspb.2002.2218
Ida H., Sirimontaporn P., Monkolprasit S. Comparative morphology of the fishes of the family Ammodytidae, with a description of two new genera and two new species // Zool. Stud. 1994. V. 33. № 4. P. 251–277.
Kim J.-K., Bae S.E., Lee S.J., Yoon M.G. New insight into hybridization and unidirectional introgression between Ammodytes japonicus and Ammodytes heian (Trachiniformes, Ammodytidae) // PLoS ONE. 2017. V. 12. № 6. e0178001.
Kim J.-K., Park J.Y., Kim Y.S. Genetic diversity, relationships and demographic history of three geographic populations of Ammodytes personatus (Ammodytidae) from Korea inferred from mitochondrial DNA control region and 16S rRNA sequence data // Korean J. Genet. 2006. V. 28. P. 343–351.
Kim J.-K., Watson W., Hyde J. et al. Molecular identification of Ammodytes (Pisces, Ammodytidae) larvae, with ontogenetic evidence on separating populations // Genes Genomics. 2010. V. 32. P. 437–445.
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. 1980. V. 16. № 2. P. 111–120.
Kitaguchi T.A. A taxonomic study of sand lances (genus Ammodytes) in the waters of northern coast of Hokkaido, Japan // Sci. Rep. Hokkaido Fish. Exp. Stn. 1979. V. 21. P. 17–30.
Kumar S., Stecher G., Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874.
Li N., Zhang Z., Zhao L., Gao T. Complete mitochondrial DNA sequence of the Pacific sand lance Ammodytes hexapterus (Perciformes: Ammodytidae): Mitogenomic perspective on the distinction of Ammodytes hexapterus and Ammodytes personatus // Mitochondrial DNA. 2013. V. 24. № 5. P. 463–465. doi 10.3109/19401736.2013.766180
Mecklenburg C.W., Mecklenburg T.A., Thorsteinsson L.K. Fishes of Alaska. Bethesda, Maryland: Am. Fish. Soc. 2002. 1037 p.
Mecklenburg C.W., Møller P.R., Steinke D. Biodiversity of arctic marine fishes: taxonomy and zoogeography // Mar. Biodiversity. 2011. V. 41. P. 109–140. doi 10.1007/s12526-010-0070-z
Okamoto H. A genetic comparison of sympatric populations of sand lance (genus Ammodytes) from the region east of Cape Soya, Japan // Can. J. Fish. Aquat. Sci. 1989. V. 46. P. 1945–1951.
Orr J.W., Wildes S., Kai Y. et al. Systematics of North Pacific sand lances of the genus Ammodytes based on molecular and morphological evidence, with the description of a new species from Japan // Fish. Bull. 2015. V. 113. № 2. P. 129–157.
Paradis E., Claude J., Strimmer K. APE: analyses of phylogenetics and evolution in R language // Bioinformatics. 2004. V. 20. № 2. P. 289–290.
Puillandre N., Lambert A., Brouillet S., Achaz G. ABGD, Automatic Barcode Gap Discovery for primary species delimitation // Mol. Ecol. 2012. V. 21. № 8. P. 1864–1877.
R Core Team. R: A language and environment for statistical computing. Vienna, Austria: R Foundation for Statistical Computing. 2017. https://www.R-project.org/. Дата просмотра 25.08.2017.
Ratnasingham S., Hebert P.D.N. BOLD: The Barcode of Life Data System (http://www.barcodinglife.org) // Mol. Ecol. Notes. 2007. V. 7. № 3. P. 355–364.
Ratnasingham S., Hebert P.D.N. A DNA-Based Registry for All Animal Species: The Barcode Index Number (BIN) System // PLoS ONE. 2013. V. 8. № 7. e66213.
Robards M.D., Willson M.F., Armstrong R.H., Piatt J.F. Sand lance: a review of biology and predator relations and annotated bibliography // Portland, Oregon: U.S. Department of Agriculture, Forest Service, Pacific Northwest Research Station. 1999. Res. Pap. PNW-RP-521. 327 p.
Ronquist F., Teslenko M., van Der Mark P. et al. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space // Syst. Biol. 2012. V. 61. № 3. P. 539–542.
Schliep K.P. Phangorn: phylogenetic analysis in R // Bioinformatics. 2011. V. 27. № 4. P. 592–593.
Sherman K., Jones C., Sullivan L. et al. Congruent shifts in sand eel abundance in western and eastern North Atlantic ecosystems // Nature. 1981. V. 291. P. 486–489. doi 10.1038/291486a0
Springer A.M., Speckman S.G. A forage fish is what? Summary of the symposium // Forage fishes in marine ecosystems: Proc. Int. Symp. Role Forage Fishes Mar. Ecosyst. 1997. P. 773–807.
Thiel R., Knebelsberger T. How reliably can northeast Atlantic sand lances of the genera Ammodytes and Hyperoplus be distinguished? A comparative application of morphological and molecular methods // ZooKeys. 2016. № 617. P. 139–164.
Turanov S.V., Kartavtsev Yu.Ph., Lipinsky V.V. et al. DNA-barcoding of perch-like fishes (Actinopterygii: Perciformes) from far-eastern seas of Russia with taxonomic remarks for some groups // Mitochondrial DNA, Part A. 2016. V. 27. № 2. P. 1188–1209.
Ward R.D., Zemlak T.S., Innes B.H. et al. DNA barcoding Australia’s fish species // Phil. Trans. Roy. Soc., B. 2005. V. 360. № 1462. P. 1847–1857.
Zhang J., Kapli P., Pavlidis P., Stamatakis A. A general species delimitation method with applications to phylogenetic placements // Bioinformatics. 2013. V. 29. № 22. P. 2869–2876.
Zhang J., Mamlouk A.M., Martinetz T. et al. PhyloMap: an algorithm for visualizing relationships of large sequence data sets and its application to the influenza A virus genome // BMC Bioinformatics. 2011. V. 12. P. 248.
Дополнительные материалы отсутствуют.


