Биология моря, 2019, T. 45, № 5, стр. 323-333
Гены wnt и frizzled у иглокожих
А. С. Гирич 1, *, А. В. Бойко 1
1 Национальный научный центр морской биологии им. А.В. Жирмунского ДВО РАН
690041 Владивосток, Россия
* E-mail: astromoon@mail.ru
Поступила в редакцию 19.06.2018
После доработки 27.12.2018
Принята к публикации 28.03.2019
Аннотация
С помощью методов молекулярной биологии и биоинформатики изучены гены wnt и frizzled у 17 видов иглокожих. Показано, что у иглокожих имеется от 11 до 13 генов wnt и 4 гена frizzled. В целом для типа Echinodermata характерна утрата отдельных генов семейства Wnt. В частности, у представителей всех классов, кроме Asteroidea, отсутствует ген wnt11, а у исследованных представителей Echinoidea утрачен и ген wnt2. Гены wnt являются консервативными, однако аминокислотные последовательности соответствующих белков лишь на 49–73% идентичны таковым гомологичных белков у разных классов иглокожих. В то же время семейство Frizzled характеризуется большей степенью сходства – 63–76%. В данной работе также проведен анализ филогении генных семейств Wnt и Frizzled.
Wnt сигналинг является одним из наиболее важных механизмов, с помощью которого осуществляется дистанционное межклеточное взаимодействие (Huelsken, Behrens, 2002; Mo, Cui, 2012). Он необходим как при формировании организма в эмбриогенезе (Logan, Nusse, 2004; Reya, Clevers, 2005; Козин и др., 2019), так и для поддержания жизнедеятельности взрослого животного (Almuedo-Castillo et al., 2012). Данный сигнальный путь также принимает участие в регуляции регенерации, а нарушение этого механизма может приводить к формированию опухолей или к смерти (Reya, Clevers, 2005; Poustka et al., 2007). Вероятно, Wnt сигналинг появляется у наиболее примитивных многоклеточных, поскольку у Ctenophora и Placozoa уже имеется 2–3 лиганда Wnt и 1–2 рецептора Frizzled (Adamska et al., 2007; Srivastava et al., 2008; Lapebie et al., 2009; Pang et al., 2010). Причем на этом этапе эволюции количество лигандов больше, чем рецепторов. Смысл такого “неравноправия” до сих пор не ясен, так как в результате взаимодействий лиганда и рецептора активируется один из трех сигнальных путей Wnt (Komiya, Habas, 2008). Возможно, разные лиганды связываются с рецептором с разной эффективностью, что приводит к конкуренции между ними, вследствие чего осуществляется регуляция процессов (Kestler, Kühl, 2008; Grumolato et al., 2010).
Одним из интереснейших процессов, в котором принимает участие Wnt сигналинг, является регенерация. Исследования последних лет показывают, что белки семейств Wnt и Frizzled не только контролируют заживление ран, но и координируют формирование утраченной конечности (Kawakami et al., 2006; Yokoyama et al., 2011; Takeo et al., 2013). Среди беспозвоночных хорошие восстановительные способности выявлены у иглокожих. Эти животные могут регенерировать после значительных повреждений, причем восстановление утраченных органов происходит без участия стволовых клеток за счет дедифференцировки сохранившихся дифференцированных клеток (Mladenov et al., 1989; García-Arrarás et al., 1998; Dolmatov, Ginanova, 2001; Долматов, Машанов, 2007; Mashanov et al., 2008; García-Arrarás, Dolmatov, 2010). Недавно было показано, что при регенерации у иглокожих активируется сигнальный путь Wnt (Sun et al., 2011; Mashanov et al., 2012; Girich et al., 2017; Li et al., 2017).
Иглокожие являются удобными модельными объектами для изучения генных механизмов развития (Poustka et al., 2007; Robert et al., 2014) и регенерации (Sun et al., 2011; Mashanov et al., 2012), однако полноценные данные о генах wnt и frizzled ограничены исследованиями на морских ежах Strongylocentrotus purpuratus и Paracentrotus lividus (Croce et al., 2006; Robert et al., 2014), а также на морских звездах Patiria miniata и P. pectinifera (McCauley et al., 2013; Kawai et al., 2016). Для голотурий имеются сведения лишь об отдельных генах wnt (Mashanov et al., 2012; Sun et al., 2013; Li et al., 2017), а данные для офиур и морских лилий отсутствуют. Кроме морских ежей, для других классов иглокожих общее число генов wnt и frizzled неизвестно. Поэтому интересно выяснить, является ли отсутствие гена wnt2 у морского ежа S. purpuratus уникальным или оно характерно для всего класса Echinoidea. В этой связи определены цели настоящего исследования: поиск на собственном материале и в базах данных последовательностей транскриптов wnt и frizzled, а также анализ филогении этих генных семейств у иглокожих.
МАТЕРИАЛ И МЕТОДИКА
Для анализа использованы особи голотурии Eupentacta fraudatrix, собранные в зал. Петра Великого Японского моря, а также морской лилии Himerometra robustipinna и голотурии Cladolabes schmeltzii из зал. Нячанг Южно-Китайского моря. Из животных была выделена РНК с помощью TRI reagent (MRC). Синтез кДНК, секвенирование транскриптома (Illumina Hiseq) и последующая сборка контигов осуществлены компанией Евроген (ЗАО Евроген). Параметры секвенированных транскриптомов для E. fraudatrix: N 50 = 830, программа сборки Trinity 2.1.11, объем контигов 827 Mb; для C. schmeltzii: N 50 = 771, программа сборки Trinity 2.4.0, объем контигов 100 Mb; для H. robustipinna: N 50 = 800, программа сборки Trinity 2.4.0, объем контигов 148 Mb. Полные последовательности транскриптов были получены методом RACE.
Дополнительно проведен поиск транскриптов генов wnt и frizzled иглокожих с помощью базы данных SPbase (http://www.echinobase.org/Echinobase) и NCBI (https://www.ncbi.nlm.nih.gov) по алгоритмам tblastx и blastn. Базы данных транскриптомов были загружены и переведены в аминокислотные последовательности (по 6 аминокислотных последовательностей на одну нуклеотидную) с помощью mEMBOSS (так как аминокислотные последовательности имеют большую гомологию). Подобный алгоритм действий применяли и к собственным транскриптомам. Далее, используя алгоритм blastp в программе Ugene, проводили поиск последовательностей искомых генов. Были обнаружены последовательности искомых генов у голотурий Apostichopus japonicus, Parastichopus parvimensis, Sclerodactyla briareus, E. fraudatrix и C. schmeltzii, у морских ежей Eucidaris tribuloides, Lytechinus variegatus и Sphaerichinus granulatus, у морских звезд P. miniata, P. pectinifera, Henricia sp. и Leptasterias sp., у офиуры Ophiothrix spiculata и у морской лилии H. robustipinna. Транскрипты искомых генов не были найдены лишь у морской лилии Anneissia japonica.
Окончательную сборку контигов и выравнивание последовательностей производили в программе Mega 5.1 с дальнейшей корректировкой вручную по консервативным остаткам цистеина у белков Wnt и по консервативным доменам у белков Frizzled. Последовательности, использованные для выравнивания, доступны по ссылке (https://docs.google.com/document/d/e/2PACX-1vRndofrU8A1U7WG7fgKCY1ySoNLKJvrMJoXvroZZZJXZbhP7XyP-UWAvtRzmLbUzzooe7BbOs2keDNQ/pub). Использовали также программу GeneRunner для определения рамок считывания. Полученные последовательности загружены в базу данных NCBI.
Сравнение предполагаемых аминокислотных последовательностей белков проводили через онлайн сервис Align Sequences Protein BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM= blastp&PAGE_TYPE= BlastSearch&BLAST_SPEC = blast2seq&LINK_LOC=blasttab).
Для построения филогенетических деревьев нуклеотидные и аминокислотные последовательности генов wnt, frizzled и соответствующих им белков анализировали с помощью Partitionfinder2.1.1 (Lanfear et al., 2016); аминокислотные последовательности анализировали также в программе Gblocks_0.91b. После выявления консервативных блоков были созданы выравнивания с удаленными участками малой схожести, которые также использовали для построения филогенетических деревьев.
Деревья, отражающие эволюцию генов wnt и frizzled, были построены с помощью трех алгоритмов: MrBayes c 1 500 000 генерациями (Guindon et al., 2010), Maximum Likelihood и онлайн сервиса CIPRES (http://www.phylo.org), а также с помощью Neighbor-Joining и программы MegaX. Для этого были использованы полные кодирующие последовательности транскриптов, аминокислотные последовательности и выравнивание с удаленными участками малой схожести от всех пяти классов иглокожих: голотурии (A. japonicus), морские ежи (S. purpuratus), морские звезды (P. miniata), морские лилии (H. robustipinna) и офиуры (O. spiculata). Идентичным методом было построено филогенетическое дерево генов wnt с добавлением последовательностей книдарий (Nematostella vectensis и Acropora digitifera), полухордовых (Saccoglossus kowalevskii), рыб (Danio rerio), амфибий (Xenopus laevis) и млекопитающих (Mus musculus).
Полученные деревья загружены в базу данных https://www.treebase.org и доступны по ссылке http://purl.org/phylo/treebase/phylows/study/TB2: S23695?x-access-code=86605374091d2e5f83bbe12 abab19577&format=html
РЕЗУЛЬТАТЫ
Почти во всех транскриптомах иглокожих были обнаружены транскрипты генов wnt и frizzled. Наиболее полный набор генов wnt выявлен у морской звезды P. miniata (табл. 1). У данного вида обнаружен также ген wnt11, который отсутствовал у всех других изученных нами представителей иглокожих. У морской звезды Leptasterias sp. найдены транскрипты четырех генов wnt: wnt1, wnt5, wnt7 и wntA, а у морской звезды Henricia sp. – пяти: wnt4, wnt5, wnt8, wnt16 и wntA. В геноме офиуры O. spiculata обнаружены гены wntA, wnt1–10 и wnt16. У морских ежей E. tribuloides, L. variegatus и S. granulatus найдены разные наборы wnt (табл. 1). У этих видов так же, как у S. purpuratus и P. lividus, отсутствовали гены wnt2 и wnt11.
Таблица 1.
Гены wnt у представителей разных классов иглокожих
| Класс | Вид | wntA | wnt1 | wnt2 | wnt3 | wnt4 | wnt5 | wnt6 | wnt7 | wnt8 | wnt9 | wnt10 | wnt11 | wnt16 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Морские звезды | P. miniata | + | + | + | + | + | + | + | + | + | + | + | + | + |
| P. pectinifera | – | + | + | + | + | + | – | + | + | – | + | + | + | |
| Henricia sp. | + | – | – | – | + | + | – | – | + | – | – | – | + | |
| Leptasterias sp. | + | + | – | – | – | + | – | + | – | – | – | – | – | |
| Морские ежи | E. tribuloides | – | + | – | – | + | + | + | + | + | – | + | – | + |
| L. variegatus | – | – | – | + | – | + | + | – | + | + | – | – | + | |
| S. granulatus | – | – | – | + | – | + | – | – | – | – | – | – | – | |
| S. purpuratus* | + | + | – | + | + | + | + | + | + | + | + | – | + | |
| P. lividus* | + | + | – | + | + | + | + | + | + | + | + | – | + | |
| Голотурии | A. japonicus* | + | + | + | + | + | + | + | + | + | + | + | – | + |
| P. parvimensis | + | + | – | + | + | + | + | – | – | – | + | – | – | |
| S. briareus | + | + | – | + | – | – | – | + | – | + | – | – | – | |
| E. fraudatrix | + | - | + | + | + | + | + | + | + | + | – | – | + | |
| C. schmeltzii | + | + | + | + | + | + | + | + | – | – | + | – | + | |
| Морские лилии | H. robustipinna | + | + | + | + | + | + | + | + | + | + | + | – | + |
| A. japonica | – | – | – | – | – | – | – | – | – | – | – | – | – | |
| Офиуры | O. spiculata* | + | + | + | + | + | + | + | + | + | + | + | – | + |
Анализ транскриптомов голотурий E. fraudatrix и C. schmeltzii показал, что у данных видов экспрессируется немного различающийся набор генов wnt. У E. fraudatrix мы не обнаружили транскрипты генов wnt1 и wnt11, а у C. schmeltzii – wnt8, wnt9 и wnt11 (табл. 1). Среди голотурий у A. japonicus обнаружено наибольшее число генов семейства Wnt, однако и у этого вида отсутствовал ген wnt11. У остальных голотурий выявлены разные наборы генов семейства Wnt, при этом транскрипты wnt11 также не обнаружены. Набор wnt в транскриптоме морской лилии H. robustipinna был сходен с таковым у A. japonicus. У H. robustipinna обнаружены транскрипты wnt1–10, wnt16 и wntA. У морской лилии A. japonica транскрипты генов wnt не найдены.
Построенные филогенетические деревья генов wnt показали, что wnt10 и wnt9 располагаются у основания дерева (рис. 1, 2). В большинстве деревьев близко к основанию также расположены группы wnt1–wnt6 и wntA–wnt8. Кроме этого формируется группа генов wnt2–wnt4–wnt11 (рис. 2), расположенная на удалении от основания деревьев. Достоверность таких пар достаточно высокая и составляет 97–100%. Исходя из построенного дерева, невозможно судить об эволюции иглокожих, так как наиболее древними классами оказываются то морские лилии, то голотурии или морские звезды. Подобный результат получен, вероятно, из-за большой изменчивости генов wnt. Группа wnt2–wnt4–wnt11 наиболее изменчива у иглокожих. У представителей всех классов, кроме морских звезд, отсутствовал ген wnt11, а у морских ежей – и wnt2. Для подтверждения правильности построения деревьев были использованы три разных метода и три разные программы (рис. 2). Хотя порядок ветвления у большинства деревьев различался, все же большая часть филогенетических деревьев показала формирование сходных групп.
Рис. 1.
Филогенетическое дерево дивергенции генов wnt, построенное на основе полных кодирующих последовательностей транскриптов генов wnt, всех пяти классов иглокожих, а также S. kowalevskii, M. musculus, X. laevis, D. rerio, N. vectensis и A. digitifera. Дерево сгенерировано с помощью метода MrBayes.
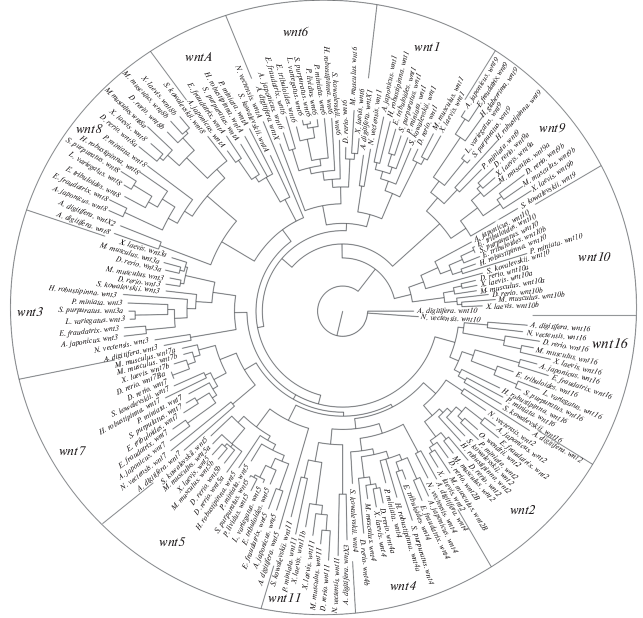
Рис. 2.
Схемы филогенетических деревьев дивергенции генов wnt, построенные на основе полных кодирующих последовательностей транскриптов генов wnt, иглокожих, губок, кишечнополостных, полухордовых, рыб, амфибий и млекопитающих. Деревья сгенерированы с помощью метода MrBayes, Maximum Likelihood и Neighbor-Joining; использованы нуклеотидные последовательности транскриптов (Nucleotid), аминокислотные последовательности белков (Amino acid) и консервативные области аминокислотных последовательностей (Conservative part). Обведены группы генов, формирующиеся в большинстве построенных деревьев.

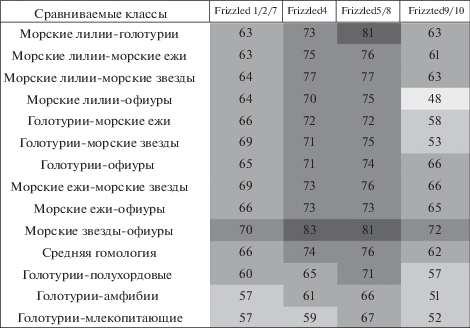
Интересные результаты были получены при сравнении аминокислотных последовательностей белков Wnt у разных классов иглокожих (рис. 3). Гомология основной части белков Wnt у разных классов иглокожих составляла 60–65%. Wnt9 характеризовался наименьшей гомологией – 49%, а белки Wnt2 и Wnt4 наибольшей – 71–73%. Гомология между отдельными белками Wnt у одного животного составляла 30–46%. Следовательно, Wnt9 показал наибольшую изменчивость у разных классов иглокожих, приблизительно такую же, как изменчивость разных белков Wnt в пределах одного вида.
Рис. 3.
Гомология предполагаемых аминокислотных последовательностей белков Wnt разных классов иглокожих, полухордовых, амфибий и млекопитающих (в %).

Кроме лигандов Wnt у всех 17 изученных видов иглокожих обнаружены транскрипты генов frizzled1/2/7 и frizzled5/8. Транскрипты frizzled4 не были найдены у S. briareus и L. variegatus, транскрипты frizzled9/10 – у L. variegatus, S. granulatus и Leptasterias sp. (табл. 2). Анализ филогении генов frizzled показал, что ближе к основанию филогенетического дерева располагался frizzled1/2/7 (рис. 4). Далее располагались паралоги frizzled5/8, frizzled4 и frizzled9/10.
Таблица 2.
Гены frizzled у представителей разных классов иглокожих
| Класс | Вид | frizzled1/2/7 | frizzled4 | frizzled5/8 | frizzled9/10 |
|---|---|---|---|---|---|
| Морские звезды | P. miniata | + | + | + | + |
| P. pectinifera | + | + | + | + | |
| Henricia sp. | + | + | + | + | |
| Leptasterias sp. | + | + | + | – | |
| Морские ежи | E. tribuloides | + | + | + | + |
| L. variegatus | + | + | – | – | |
| S. granulatus | + | + | + | – | |
| S. purpuratus* | + | + | + | + | |
| P. lividus* | + | + | + | + | |
| Голотурии | A. japonicus* | + | + | + | + |
| P. parvimensis | + | + | + | + | |
| S. briareus | + | + | – | + | |
| E. fraudatrix | + | + | + | + | |
| C. schmeltzii | + | + | + | + | |
| Морские лилии | H. robustipinna | + | + | + | + |
| A. japonica | + | + | + | + | |
| Офиуры | O. spiculata* | + | + | + | + |
Рис. 4.
Схемы филогенетических деревьев дивергенции генов frizzled, построенные на основе полных кодирующих последовательностей транскриптов генов frizzled, иглокожих, губок, кишечнополостных, полухордовых, рыб, амфибий и млекопитающих. Деревья сгенерированы с помощью метода MrBayes, Maximum Likelihood и Neighbor-Joining; использованы нуклеотидные последовательности транскриптов (Nucleotid), аминокислотные последовательности белков (Amino acid) и консервативные области аминокислотных последовательностей (Conservative part). Обведены группы генов, формирующиеся в большинстве построенных деревьев.

Выполненное сравнение аминокислотных последовательностей белков Frizzled показало, что максимальную гомологию среди иглокожих имели Frizzled5/8 и Frizzled4 (74–76%), а минимальную – Frizzled9/10 и Frizzled1/2/7 (63–66%) (рис. 5). Изменчивость последовательностей белков рецепторов Frizzled была меньше, чем таковая у лигандов Wnt.
ОБСУЖДЕНИЕ
В результате проведенного исследования установлено, что у всех иглокожих имеется от 11 до 13 генов лигандов Wnt и 4 гена рецептора Frizzled. Максимальное количество генов wnt обнаружено у морских звезд, у которых выявлен уникальный среди иглокожих ген wnt11 (Kawai et al., 2016). То, что wnt11 не обнаружен у представителей других классов иглокожих, в том числе и у более эволюционно древних морских лилий, можно объяснить несколькими причинами. Наиболее простая причина – отсутствие аннотированных геномов. Анализ на основе транскриптомов может дать отрицательный результат из-за низкого уровня экспрессии или ошибок секвенирования. Другой причиной может быть неоднократная и независимая утрата этого гена в разных группах иглокожих. Возможно, в эволюции иглокожих происходило уменьшение функциональной значимости гена wnt11, что сопровождалось/вызывалось снижением его активности. Сходную функцию мог выполнять другой ген данного семейства. Это могло стать причиной полной утраты гена wnt11, поскольку без его экспрессии животное могло полноценно развиваться и давать потомство. С этой гипотезой согласуются сведения о том, что ген wnt11 не экспрессируется в эмбриогенезе морской звезды Patiria pectinifera (Kawai et al., 2016).
Существует небольшая путаница в названиях генов wnt у иглокожих, вызванная неточным определением того или иного гена. Вероятно, это связано с низкой гомологией данных генов. В частности, у морского ежа S. purpuratus ген wntA сначала был определен как wnt4, a ген wnt2 у офиуры Ophiocoma wendtii загружен в базу данных как wnt8 (Vaughn et al., 2012). Поэтому для правильного определения последовательности необходимо опираться не только на данные Blast, но и на филогенетические деревья.
Наш анализ показал, что морские звезды имеют полный набор из 13 генов wnt, морские лилии, офиуры и голотурии имеют 12 генов wnt, а морские ежи – 11, поскольку у них отсутствует не только wnt11, но и wnt2 (Croce et al., 2006). Возможно, по аналогии с геном wnt11, ген wnt2 не имел какой-либо жизненно важной функции и его утрата не стала фатальной для предков морских ежей. У других иглокожих данный ген, вероятно, имеет большое значение в регуляции морфогенезов, поскольку экспрессируется при регенерации нервной системы у Holothuria glaberrima (Mashanov et al., 2014) и в эмбриогенезе морских звезд (McCauley et al., 2013; Kawai et al., 2016).
Таким образом, у иглокожих наблюдается тенденция к утрате генов семейства Wnt. Если wnt11 отсутствует у Crinoidea, Ophiuroidea, Echinoidea и Holothuroidea, можно предположить, что утрата этого гена в эволюции Echinodermata происходила как минимум дважды. Вероятно, вначале он был потерян общим предком всех современных морских лилий, но сохранился у представителей Eleuterozoa. Если предположить, что верна гипотеза “Cryptosyringida” (Smith, 1984), то вторая утрата wnt11 произошла у общего предка офиур, морских ежей и голотурий. Если же верна гипотеза “Asterozoa–Echinozoa” (Reich et al., 2015), то следует предположить, что утрата wnt11 происходила независимо у офиур и Echinozoa. Для выяснения данного вопроса необходимы дополнительные исследования. В настоящее время четкого понимания эволюции генного семейства Wnt нет. В работах исследователи используют либо полные последовательности генов wnt, но тогда они сильно ограничены выбором животных (Croce et al., 2006; Pang et al., 2010), либо консервативные области транскриптов, транслируя их в аминокислотные последовательности (Prud’homme et al., 2002; Jannsen et al., 2010). При этом исследователи получают разные результаты, когда наиболее древними признаются разные гены семейства Wnt: wnt11 и wnt4 (Croce et al., 2006), wnt3 (Cho et al., 2010), wnt16, wnt2 и wnt7 (Pang et al., 2010), wntA и wnt6 (Riddiford, Olson, 2011). Правильно было бы изучать разнообразие генов wnt у губок как у самых простых многоклеточных, однако современные данные показывают, что гены wnt губок уникальны и не имеют ортологов у вторичноротых (Borisenko et al., 2016).
Для нашего анализа были выбраны полные последовательности кодирующих участков транскриптов генов wnt, так как только они несут смысловую нагрузку и преимущественно подвержены действию естественного отбора. Отдельные классы иглокожих разошлись примерно 550–450 млн лет назад (Wada, Satoh, 1994) и имеют достаточно много различий. В этой связи 5 монофилетичных групп служат хорошей моделью для оценки эволюции генного семейства Wnt. Для сравнения нами построено филогенетическое дерево генов wnt с учетом последовательностей транскриптов wnt млекопитающих, земноводных, рыб, полухордовых и кишечнополостных. Для семейства Wnt построение филогении, основанной только на консервативных областях, не дало значимых результатов (рис. 2). Вероятно, это связано с тем, что аминокислотные последовательности Wnt невелики (300–350 аминокислот), а консервативные области имеют длину около 123 аминокислот, что недостаточно для корректного анализа. Наилучший результат получен при использовании полных нуклеотидных последовательностей транскриптов. Построенные на их основе филогенетические деревья генов wnt имеют много общих свойств.
Мы предполагаем, что наиболее древним геном семейства является wnt10. Гомология аминокислотных последовательностей Wnt10 иглокожих варьирует в достаточно широких пределах в зависимости от вида. Это может говорить о том, что у разных животных функции данного белка могут сильно различаться. Однако у иглокожих этот ген исследован слабо. В эмбриогенезе морских звезд экспрессия wnt10 незначительна и наблюдается лишь на поздних стадиях развития (Kawai et al., 2016). У человека описано участие Wnt10 в таких процессах, как фиброз тканей, рост опухоли и регенерация (Kuma et al., 2014).
К предковым генам следует также отнести ген wnt9. Различие аминокислотной последовательности белка Wnt9 у разных классов иглокожих составляет 44–54% (рис. 3). Такой низкий процент гомологии больше характерен для паралогов у одного животного, чем для ортологичных генов. В то же время гомология последовательностей белков Wnt морского ежа с ортологами позвоночных составляет 50–70%, что иногда превышает гомологию с ортологичными белками других иглокожих (Ferkowicz, Raff, 2001). Это говорит о том, что функции Wnt9 могут кардинально различаться в разных классах иглокожих. Например, при развитии морского ежа P. lividus ген wnt9 экспрессируется в клетках будущей энтодермы еще на стадии бластулы (Robert et al., 2014), в то время как при развитии морских звезд P. pectinifera и P. miniata экспрессия wnt9 не была обнаружена (McCauley et al., 2013; Kawai et al., 2016). Экспрессию этого гена исследовали также при регенерации голотурии H. glaberrima (Mashanov, García-Arrarás, 2011). Остальные гены wnt сформировали отдельные группы, однако старшинство данных групп достоверно определить не удалось (Kuma et al., 2014).
Отдельную группу на филогенетических деревьях формировали гены wnt1 и wnt6 (рис. 2). Они находятся на одной хромосоме друг за другом (Holstein, 2012) и с большой вероятностью появились в результате дупликации (Cho et al., 2010). Кроме этого данные гены имеют сходные паттерны экспрессии при регенерации внутренних органов голотурии (Girich et al., 2017) и развитии морского ежа (Robert et al., 2014), а также при развитии насекомых (Hogvall et al., 2014). Сходство происхождения и экспрессии говорит о сходных функциях, которые должны выполнять эти гены. Так, оба гена задействованы при формировании органов нервной и мышечной систем у млекопитающих (Quinlan et al., 2009; Hitchins et al., 2013). У иглокожих исследование гена wnt6 проводилось на эмбрионах морских ежей и звезд (Croce et al., 2006; McCauley et al., 2013; Robert et al., 2014; Kawai et al., 2016), а также при регенерации A. japonicus (Sun et al., 2013).
Группа родственных генов wnt2, wnt4 и wnt11 имеет наибольшую гомологию по аминокислотному составу среди иглокожих, а значит, и наиболее сходные функции. В то же время эта группа наиболее изменчива и склонна к утрате генов. По-видимому, если функции данных генов взаимозамещаемы, то при утрате одного гена его функцию выполняет ближайший “родственник” (Long et al., 2013). Какой-либо зависимости положения генов wnt на дереве от гомологии последовательностей соответствующих им белков не выявлено.
Для генов frizzled анализ филогении по консервативным областям белков показал более стабильные результаты, вероятно, из-за относительно большой длины используемых доменов (около 325 аминокислот). Построение филогении семейства Frizzled с помощью метода Neighbor-Joining показало большой разброс значений и не дало корректных результатов. Остальные методы дали сходный результат: имеется предковый frizzled1/2/7, от которого дивергировал frizzled5/8, а в дальнейшем образовались frizzled4 и frizzled9/10. Почти во всех исследованных транскриптомах были обнаружены все 4 гена рецептора frizzled, что говорит о важности их экспрессии. У иглокожих гомология белковых последовательностей Frizzled больше, чем таковая у Wnt, и составляет 63–76%. В эмбриональном развитии морских звезд и морских ежей паттерны экспрессии генов frizzled схожи, что не отмечено у большинства генов wnt. Так, frizzled1/2/7 экспрессируется по средней линии зародыша, захватывая эктодерму и крышу архентерона; frizzled4 экспрессируется в небольшом количестве клеток вокруг бластопора, транскрипты frizzled5/8, имеющего максимальную гомологию последовательности среди иглокожих, обнаружены на анимальном полюсе, а также на кончике архентерона; frizzled9/10 экспрессируется в клетках формирующейся энтодермы и эктодермы вегетативного полюса на стадии гаструлы (McCauley et al., 2013; Robert et al., 2014; Kawai et al., 2016). Таким образом, экспрессия генов frizzled имеет более выраженные паттерны, чем у генов wnt, что, по-видимому, необходимо для формирования тканевых линий зародыша.
Эволюция сигнальных путей происходит за счет генных дупликаций либо в результате изменения белковых взаимодействий (Tsuda, Kawata, 2010). При этом иглокожие за 400 млн лет раздельной эволюции не обрели новых членов семейств Wnt и Frizzled, как, например, губки или позвоночные (Holsten, 2012; Borisenko et al., 2016), и проявляют тенденцию к утрате генов wnt, что не вписывается в общую тенденцию эволюции данных генов у животных. При этом все гены frizzled, имеющиеся у предка иглокожих, присутствуют у представителей каждого класса, что еще раз доказывает необходимость работы именно рецепторов Frizzled, так как количество лигандов Wnt избыточно и утрата одного из членов семейства не оказывается летальной.
Таким образом, гены wnt и frizzled у иглокожих участвуют в процессах жизнедеятельности, контролируют эмбриогенез и регенерацию. Наиболее важными являются гены рецепторов Frizzled, так как у разных классов иглокожих они экспрессируются в эмбриогенезе и локализация экспрессии четко детерминирована. Предполагаемые аминокислотные последовательности белков Frizzled очень консервативны; их гомология составляет 63–76% в отличие от большинства белков Wnt (49–73%). Гены wnt зачастую не имеют четко определенной локализации экспрессии. Количество этих генов больше; наблюдается утрата одного или двух членов семейства Wnt, что, вероятно, связано со схожими функциями генов общего происхождения. Интересно, что гомология аминокислотных последовательностей белков Wnt и Frizzled примерно одинакова при сравнении как ортологов иглокожих, так и ортологов иглокожих с позвоночными и полухордовыми (рис. 3, 5). Очевидно, это указывает на то, что расхождение классов иглокожих произошло на ранних этапах формирования Echinodermata, а эволюционное расстояние между классами Echinoidea и Asteroidea может быть сопоставимо с расстоянием между морскими ежами и позвоночными. Вся изменчивость последовательностей Wnt и Frizzled происходит преимущественно за счет аминокислотных замен в вариабельных участках, что не затрагивает основных функций Wnt как лиганда, однако может иметь значение для регуляции его активности.
Список литературы
Долматов И.Ю., Машанов В.С. Регенерация у голотурий. Владивосток: Дальнаука. 2007. 212 с.
Козин В.В., Борисенко И.Е., Костюченко Р.П. Участие канонического сигнального пути Wnt в определении полярности тела и клеточной идентичности у Metazoa: Новые данные о развитии губок и аннелид // Изв. РАН. Сер. биол. 2019. № 1. С. 1–12.
Adamska M., Degnan S.M., Green K.M. et al. Wnt and TGF-beta expression in the sponge Amphimedon queenslandica and the origin of metazoan embryonic patterning // PloS One. 2007. V. 2. № 10. https://doi.org/10.1371/journal.pone.0001031
Almuedo-Castillo M., Sureda-Gómez M., Adell T. Wnt signaling in planarians: new answers to old questions // Int. J. Dev. Biol. 2012. V. 56. P. 53–65. https://doi.org/10.1387/ijdb.113451ma
Borisenko I., Adamaski M., Ereskovsky A., Adamaska M. Suprisingly rich repertoire of Wnt genes in the demosponge Halisarca dujardini // BMC Evol. Biol. 2016. V. 123. № 16. P. 1–7. https://doi.org/10.1186/s12862-016-0700-6
Cho S., Vallès Y., Giani V.C. Jr et al. Evolutionary dynamics of the wnt gene family: a lophotrochozoan perspective // Mol. Biol. Evol. 2010. V. 27. P. 1645–1658.
Croce J.C., Wu S.Y., Byrum C. et al. A genome-wide survey of the evolutionarily conserved Wnt pathways in the sea urchin Strongylocentrotus purpuratus // Dev. Biol. 2006. V. 300. P. 121–131.
Dolmatov I.Y., Ginanova T.T. Regeneration in holothurians // Microsc. Res. Tech. 2001. V. 55. P. 452–463.
Ferkowicz M.J., Raff R.A. Wnt gene expression in sea urchin development: heterochronies associated with the evolution of developmental mode // Evol. Dev. 2001. V. 3. № 1. P. 24–33.
García-Arrarás J.E., Estrada-Rodgers L., Santiago R. et al. Cellular mechanisms of intestine regeneration in the sea cucumber, Holothuria glaberrima Selenka (Holothuroidea: Echinodermata) // J. Exp. Zool. 1998. V. 281. № 4. P. 288–304.
García-Arrarás J.E., Dolmatov I.Yu. Echinoderms: potential model systems for studies on muscle regeneration // Cur. Pharm. Des. 2010. V. 16. P. 942–955.
Girich A.S., Isaeva M.P., Dolmatov I.Yu. Wnt and frizzled expression during regeneration of internal organs in the holothurian Eupentacta fraudatrix // Wound Repair Regen. 2017. V. 25. P. 828–835.
Grumolato L., Liu G., Mong P. et al. Canonical and noncanonical Wnts use a common mechanism to activate completely unrelated coreceptors // Genes Dev. 2010. V. 24. P. 2517–2530.
Guindon S., Dufayard J.F., Lefort V. et al. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0 // Syst. Biol. 2010. V. 59. № 3. P. 307–321.
Hitchins L., Fletcher F., Allen S., Dhoot G.K. Role of Sulf1A in Wnt1- and Wnt6-induced growth regulation and myoblast hyperelongation // FEBS Open Bio. 2013. V. 3. P. 30–34.
Hogvall M., Schönauer A., Budd G. et al. Analysis of the Wnt gene repertoire in an onychophoran provides new insights into the evolution of segmentation // EvoDevo. 2014. V. 5. № 14. https://doi.org/10.1186/2041-9139-5-14
Holstein T.W. The evolution of Wnt pathway // Cold Spring Harb Perspect Biol. 2012. V. 4. № 7. https://doi.org/10.1101/cshperspect.a007922
Huelsken J., Behrens J. The Wnt signaling pathway // J. Cell Sci. 2002. V. 115. P. 3977–3978.
Janssen R., Gouar M.L., Pechmann M. et al. Conservation, loss, and redeployment of Wnt ligands in protostomes: implication for understanding the evolution of segment formation // Evol. Biol. 2010. V. 10. https://doi.org/10.1186/1471-2148-10-374
Kawai N., Kuraishi R., Kaneko H. Wnt, Frizzled, and sFRP gene expression patterns during gastrulation in the starfish Patiria (Asterina) pectinifera // Gene Expr. Patterns. 2016. V. 21. P. 19–27.
Kawakami Y., Esteban C.R., Raya M. et al. Wnt/beta-catenin signaling regulates vertebrate limb regeneration // Genes Dev. 2006. V. 20. P. 3232–3237.
Kestler H.A., Kühl M. From individual Wnt pathways towards a Wnt signalling network // Philos. Trans. R. Soc. Lond. B. Biol. Sci. 2008. V. 363. P. 1333–1347.
Komiya Y., Habas R. Wnt signal transduction pathways // Organogenesis. 2008. V. 4. № 2. P. 68–75.
Kuma A., Yamada S., Wang K. et al. Role of WNT10A-expressing kidney fibroblasts in acute interstitial nephritis // PloS One. 2014. V. 9(7). https://doi.org/10.1371/journal.pone.0103240
Lanfear R., Frandsen P.B., Wright A.M. et al. PartitionFinder 2: new methods for selecting partitioned models of evolution for molecular and morphological phylogenetic analyses // Mol. Biol. Evol. 2016. V. 34. № 3. P. 772–773. dx.doi.org/https://doi.org/10.1093/molbev/msw260
Lapebie P., Gazave E., Ereskovsky A. et al. WNT/beta-catenin signalling and epithelial patterning in the homoscleromorph sponge Oscarella // PloS One. 2009. V. 4. № 6. https://doi.org/10.1371/journal.pone.0005823
Li X., Sun L., Yang H. et al. Identification and expression characterization of WntA during intestinal regeneration in the sea cucumber Apostichopus japonicus // Comp. Biochem. Physiol., Part B. 2017. V. 210. P. 55–63.
Long M., VanKuren N.W., Chen S., Vibranovski M.D. New gene evolution: little did we know // Annu. Rev. Genet. 2013. V. 47. P. 307–333.
Logan C.Y., Nusse R. The Wnt signaling pathway in development and disease // Annu. Rev. Cell. Dev. Biol. 2004. V. 20. P. 781–810.
Mashanov V.S., Zueva O.R., Heinzeller T. Regeneration of the radial nerve cord in a holothurian: a promising new model system for studying post-traumatic recovery in the adult nervous system // Tissue Cell. 2008. V. 40. P. 351–372.
Mashanov V.S., García-Arrarás J.E. Gut regeneration in holothurians: a snapshot of recent developments // Biol. Bull. 2011. V. 221. P. 93–109.
Mashanov V.S., Zueva O.R., García-Arrarás J.E. Expression of wnt9, tctp, and bmp1/tii in sea cucumber visceral regeneration // Gene Expr. Patterns. 2012. V. 12. P. 24–35.
Mashanov V.S., Zueva O.R., García-Arrarás J.E. Transcriptomic changes during regeneration of the central nervous system in an echinoderm // BMC. Genomics. 2014. V. 15. P. 357. https://doi.org/10.1186/1471-2164-15-357
McCauley B.S., Akyar E., Filliger L., Hinman V.F. Expression of wnt and frizzled genes during early sea star development // Gene Expr. Patterns. 2013. V. 13. P. 437–444.
Mladenov P.V., Bisgrove B., Asotra S., Burke R.D. Mechanisms of arm tip regeneration in the sea star Leptasterias hexactis // Rouxs Arch. Dev. Biol. 1989. V. 198. № 1. P. 19–28.
Mo S., Cui Z. Regulation of canonical wnt signaling during development and diseases // Embryogenesis. Dr. Ken-Ichi Sato (Ed.). 2012. ISBN: 978-953-51-0466-7, InTech. 652 p.
Pang K., Ryan J.F., Mulliken J.C. et al. Genomic insights into Wnt signaling in an early diverging metazoan, the ctenophore Mnemiopsis leidyi // EvoDevo. 2010. V. 1. P. 1–15. https://doi.org/10.1186/2041-9139-1-10
Poustka A.J., Kuhn A., Groth D. et al. A global view of gene expression in lithium and zinc treated sea urchin embryos: new components of gene regulatory networks // Genome Biol. 2007. V. 8. № 5. https://doi.org/10.1186/gb-2007-8-5-r85
Quinlan R., Graf M., Mason I. et al. Complex and dynamic patterns of Wnt pathway gene expression in the developing chick forebrain // Neural Devel. 2009. 4:35. https://doi.org/10.1186/1749-8104-4-35
Prud’homme B., Lartillot N., Balavoine G. et al. Phylogenetic analysis of the Wnt gene family: Insights from Lophotrochozoan members // Curr. Biol. 2002. V. 12. P. 1395–1400.
Reich A., Dunn C., Akasaka K., Wessel G. Phylogenomic analyses of Echinodermata support the sister group of Asterozoa and Echinozoa // PloS One. 2015. V. 10. https://doi.org/10.1371/journal.pone.0119627
Reya T., Clevers H. Wnt signaling in stem cells and cancer // Nature. 2005. V. 434. P. 843–850.
Riddiford N., Olson P. Wnt genes loss in flatworms // Dev. Genes Evol. 2011. V. 221. P. 187–197.
Robert N., Lhomond G., Schubert M., Croce J.C. A comprehensive survey of wnt and frizzled expression in the sea urchin Paracentrotus lividus // Genesis. 2014. V. 52. P. 235–250.
Smith A.B. Classification of the Echinodermata // Paleontology. 1984. V. 27. P. 431–459.
Srivastava M., Begovic E., Chapman J. et al. The Trichoplax genome and the nature of placozoans // Nature. 2008 . V. 454. P. 955–960.
Sun L., Chen M., Yang H. et al. Large scale gene expression profiling during intestine and body wall regeneration in the sea cucumber Apostichopus japonicus // Comp. Biochem. Physiol. Part D. 2011. V. 6. P. 195–205.
Sun L.N., Yang H.S., Chen M.Y., Hu D.X. Cloning and expression analysis of Wnt6 and Hox6 during intestinal regeneration in the sea cucumber Apostichopus japonicus // Genet. Mol. Res. 2013. V. 12. № 4. P. 5321–5334.
Takeo M., Chou W.C., Sun Q. et al. Wnt activation in nail epithelium couples nail growth to digit regeneration // Nature. 2013. V. 499. P. 228–232.
Tsuda M.E., Kawata M. Evolution of gene regulatory Networks by fluctuating selection and intrinsic constraints // PloS Comput. Biol. 2010. V. 6. № 8. https://doi.org/10.1371/journal.pcbi.1000873
Vaughn R., Garnhart N., Garey J.R. et al. Sequencing and analysis of the gastrula transcriptome of the brittle star Ophiocoma wendtii // EvoDevo. 2012. V. 3. https://doi.org/10.1186/2041-9139-3-19
Wada H., Satoh M. Phylogenetic relationships among extant classes of echinoderms, as inferred from sequences of 18S rDNA, coincide with relationships deduced from the fossil record // J. Mol. Evol. 1994. V. 38. P. 41–49.
Yokoyama H., Maruoka T., Ochi H. et al. Different requirement for Wnt/β-catenin signaling in limb regeneration of larval and adult Xenopus // PloS One. 2011. V. 6. P. 1–13. https://doi.org/10.1371/journal.pone.0021721
Дополнительные материалы отсутствуют.