Биология моря, 2023, T. 49, № 1, стр. 45-55
Генетическое разнообразие мтДНК дальневосточного трепанга Apostichopus japonicus (Selenka, 1867) (Echinodermata: Holothuroidea) в заливе Петра Великого Японского моря
В. Д. Ягодина 1, *, В. А. Брыков 1
1 Национальный научный центр морской биологии им. А.В. Жирмунского (ННЦМБ) ДВО РАН
690041 Владивосток, Россия
* E-mail: iagodinavd@gmail.com
Поступила в редакцию 27.08.2022
После доработки 30.09.2022
Принята к публикации 06.10.2022
- EDN: LSPRDP
- DOI: 10.31857/S0134347523010102
Аннотация
Изучено генетическое разнообразие дальневосточного трепанга Apostichopus japonicus (Selenka, 1867), обитающего в зал. Петра Великого Японского моря. Проанализированы 5 выборок с использованием фрагмента гена COI митохондриальной ДНК. Всего выявлено 16 гаплотипов, при этом гаплотипическое разнообразие было высоким (0.86767 ± 0.01800), а разнообразие нуклеотидов – низким (0.00759 ± 0.00025). Результаты AMOVA и попарного Fst не выявили существенных генетических различий между выборками из зал. Петра Великого. На основе полученных данных и структуры гаплотипической сети, высказано предположение, что дальневосточный трепанг обитает в неравновесных условиях. Это связано с неравномерным расселением молоди, зависящим от гидрологического режима, типа грунта и развития марикультуры в акватории, а также со значительным браконьерским выловом.
Голотурии, или морские огурцы (Holothuroidea), представляют собой морфологически разнообразную, экологически и экономически важную группу иглокожих (Echinodermata). Они распространены по Мировому океану от мелководных прибрежных районов до значительных глубин (Tyler et al., 1992). Голотурии обитают на дне, где питаются водорослями, мелкими животными или детритом. При этом они снижают органическую нагрузку на экосистемы, выделяя неорганический азот и фосфор, играя важную роль в рециркуляции питательных веществ. Некоторые виды могут повышать щелочность воды и концентрацию растворенного неорганического углерода за счет процессов пищеварения и выделения аммиака (Simões et al., 2019).
Морские огурцы, обитающие в Японском море, не являются исключением. В их число входит и дальневосточный трепанг Apostichopus japonicus (Selenka, 1867), тело которого может достигать 40 см в длину и 9 см в диаметре (Левин, 2000). Он распространен в прибрежных водах Восточной Азии, где является одним из важнейших объектов аквакультуры благодаря пищевой ценности и широкому использованию в медицине (Oh et al., 2017). Дальневосточный трепанг – важный промысловый вид в Северо-Восточной Азии, имеющий самую долгую историю эксплуатации в этом регионе (Purcell et al., 2012). Однако из-за расширения искусственного воспроизводства и резкого сокращения природных ресурсов в последние 30 лет запасы диких популяций A. japonicus сокращаются (Chen, Yang, 2008). Уменьшение численности видов, как правило, приводит к снижению генетического разнообразия, что, в свою очередь, может быть причиной ухудшения выживаемости и увеличения смертности. В настоящее время исследования направлены на определение запасов A. japonicus и восстановление его численности, в том числе при помощи искусственного выращивания в условиях марикультуры (Yan et al., 2013).
В связи с этим представляется важным проведение исследований популяционной генетики A. japonicus на значительной части его ареала с использованием молекулярных маркеров. Для оценки генетического разнообразия и популяционной структуры этого вида часто использовали микросателлитные маркеры (Kanno et al., 2006; Kim et al., 2008; Chang et al., 2009; Soliman et al., 2012) и однонуклеотидные полиморфизмы (Du et al., 2012; Dong et al., 2016). Применялся также анализ изменчивости последовательности субъединицы I цитохром с-оксидазы (COI) митохондриальной ДНК (Valente et al., 2014; Soliman et al., 2016; Hamamoto et al., 2021), что позволяло оценивать не только популяционную структуру, но и демографическую историю вида.
Большая часть исследований на Дальнем Востоке России посвящена изучению биологии дальневосточного трепанга, его образа жизни, а также процессов, определяющих снижение численности (Селин, 2001; Лысенко и др., 2018). Популяционную структуру и уровень генетического разнообразия A. japonicus на Дальнем Востоке России ранее не изучали. В настоящей работе, используя частичные последовательности гена COI мтДНК, мы исследовали генетическое разнообразие и структуру популяции дальневосточного трепанга в зал. Петра Великого Японского моря.
МАТЕРИАЛ И МЕТОДИКА
Сбор проб
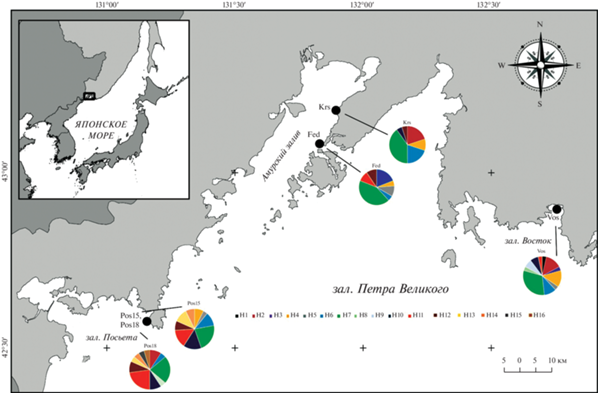
В работе использовано 5 выборок дальневосточного трепанга Apostichopus japonicus, собранных водолазным способом в зал. Петра Великого Японского моря в 2015 и 2018 гг.: зал. Восток (Vos, n = 29, июль 2015 г.); мыс Красный, Амурский залив (Krs, n = 20, июнь 2015 г.); зал. Посьета (Pos15, n = 25, июль 2015 г.); б. Федорова, Амурский залив (Fed, n = 21, июнь 2015 г.) и зал. Посьета (Pos18, n = 22, июль 2018 г.). Две выборки из зал. Посьета (3 и 5) были взяты с интервалом в три года из одной локальности (рис. 1).
Выделение ДНК, ПЦР и секвенирование
Выделение геномной ДНК из фиксированной в 96% этаноле ткани проводили методом щелочного лизиса (Truett, 2006). Фрагмент гена COI амплифицировали с использованием праймеров COIce-F (5'-ACTGCCCACGCCCTAGTAATGATATTTTT TATGGTNATGCC-3') и COIce-R (5'- TCGTGTGTCTACGTCCATTCCTAC TGTRAACATRTG-3') (Hoareau, Boissin, 2010). ПЦР для каждого образца ДНК проводилась в реакционной смеси объемом 25 мкл с использованием Go Taq Green Master Mix (Promega Corp, Madison, WI, USA) и 10 мМ dNTPs (каждого); 10 мМ прямого и обратного праймера (каждого); 40–60 нг тотальной ДНК. Амплификацию осуществляли по следующему протоколу: начальная денатурация при температуре 94°С (2 мин) с последующими 35 циклами денатурации при 94°С в течение 30 с; отжиг при 49°С (45 с) и элонгация при 72°С в течение 1 мин, с конечной элонгацией при 72°С в течение 10 мин.
Продукты ПЦР визуализировали с помощью горизонтального электрофореза в 1.5% агарозном геле в трис-боратном буфере (TBE), содержавшем бромид этидия (10 мг/мл).
ПЦР-продукты очищали с помощью экзонуклеазы I (ExoI) и термочувствительной щелочной фосфатазы (FastAP; ThermoFisher Scientific, Waltham, Massachusetts, USA). Реакцию секвенирования проводили с использованием набора BigDye® Terminator v3.1 Cycle Sequencing Kits (Applied Biosystems, Карлсбад, Калифорния, США) и анализировали в секвенаторе ABI3130xl (Институт Мирового океана, Дальневосточный федеральный университет).
Анализ данных
Сборку последовательности проводили с использованием программного обеспечения ChromasPro 2.1.10 (http://technelysium.com.au; бесплатный пробный период составлял 14 дней). Все полученные последовательности были выровнены в программе MEGA 11 (Tamura et al., 2021) с применением алгоритма MUSCLE (Edgar, 2004). Итоговая длина последовательности составила 554 п.н. AIC (информационный критерий Акаике) показал, что среди методов максимального правдоподобия наиболее подходящей моделью была двухпараметрическая модель Кимуры (K2) (см.: Kimura, 1980). Надежность ветвей дерева оценивали с помощью бутстрэп-анализа (Felsenstein, 1985) – 1000 повторов.
DnaSP 6.12.03 (Rozas et al., 2017) использовали для расчета описательной статистики – количество гаплотипов, частоту гаплотипов, количество полиморфных сайтов (S), разнообразие гаплотипов (h), разнообразие нуклеотидов (π) и среднее количество нуклеотидных различий (k) при параметрах по умолчанию. На основе результатов оценки гаплотипов была создана медианная сеть гаплотипов (MJ; Bandelt et al., 1999) с визуализацией в программе Network 10.2.0.0 (http://fluxus-engineering.com) с 1000 бутстрэп-репликами (Felsenstein, 1985). Нами был проведен анализ молекулярной дисперсии (AMOVA; Excoffier et al., 1992), а также выполнены тесты нейтральности на основе FS Фу (Fu, 1997) и D Таджимы (Tajima, 1989) с использованием программы Arlequin 3.5.1.2 (Excoffier, Lischer, 2010). Значимость проверена путем случайной перестановки с использованием 10 000 повторов. Показатель Fst основан на попарных различиях гаплотипов, вероятность оценена с использованием 10 000 случайных перестановок (Arlequin). Р-значение (P < 0.05) было скорректировано по минимальному объему выборки (коррекция Бонферрони). Распределение частот попарных нуклеотидных различий (распределение несоответствия) для оценки демографического расширения популяции было рассчитано в соответствии с рекомендациями (Schneider, Excoffier, 1999) для каждой выборки. Кроме этого были рассчитаны индекс неоднородности Харпендинга (Harpending, 1994) и сумма квадратичных отклонений (Schneider, Excoffier, 1999).
РЕЗУЛЬТАТЫ
Изменчивость фрагмента мтДНК Apostichopus japonicus в заливе Петра Великого
Всего для фрагмента гена COI мтДНК во всех выборках было обнаружено 16 гаплотипов (рис. 1); все последовательности были депонированы в GenBank (№ OP077239–OP077254). В целом гаплотипическое разнообразие было высоким COI (H = 0.86767), в то время как нуклеотидное разнообразие было низким (π = = 0.00759 ± 0.00025) (табл. 1). Общее число полиморфных сайтов составило 21, наименьшее – для Fed (12), наибольшее – для Pos18 (18). Среднее число нуклеотидных различий варьировало от 3.83810 ± 2.00950 (Fed) до 4.57333 ± 2.32447 (Pos15). Наибольшее число гаплотипов наблюдалось в выборках из заливов Восток (2015 г.) и Посьета, собранных в 2018 г. (по 11), наименьшее – в выборке с мыса Красный (6). Самым распространенным гаплотипом был H7, найденный во всех выборках. Уникальные гаплотипы обнаружены в выборках Vos, Pos15 и Pos18.
Таблица 1.
Молекулярное разнообразие выборок Apostichopus japonicus, отобранных в зал. Петра Великого, на основе последовательности COI мтДНК
| Выборка | Размер выборки | Число гаплотипов | S | Гаплотипы (частота встречаемости) | k ± SD | h ± SD | π ± SD |
|---|---|---|---|---|---|---|---|
| Vos | 29 | 11 | 17 | H1 (1), H2 (4), H3 (1), H4 (4), H5 (1), H6 (3), H7 (9), H8 (1), H9 (2), H10 (2), H11 (1) | 4.16749 ± 2.13323 | 0.86946 ± 0.04300 | 0.00752 ± 0.00054 |
| Krs | 20 | 6 | 13 | H2 (4), H4 (2), H6 (4), H7 (8), H10 (1), H12 (1) | 3.91053 ± 2.04637 | 0.78421 ± 0.06400 | 0.00706 ± 0.00061 |
| Pos15 | 25 | 9 | 17 | H4 (2), H5 (1), H6 (3), H7 (2), H10 (2), H11 (7), H12 (4), H13 (3), H14 (1) | 4.57333 ± 2.32447 | 0.88000 ± 0.03900 | 0.00826 ± 0.00057 |
| Fed | 21 | 7 | 12 | H2 (4), H4 (1), H5 (2), H6 (1), H7 (9), H11 (2), H12 (2) | 3.83810 ± 2.00950 | 0.78571 ± 0.07400 | 0.00693 ± 0.00054 |
| Pos18 | 22 | 11 | 18 | H2 (2), H6 (1), H7 (5), H8 (1), H10 (2), H11 (5), H12 (2), H13 (1), H14 (1), H15 (1), H16 (1) | 4.42857 ± 2.27070 | 0.90043 ± 0.04100 | 0.00799 ± 0.00098 |
| Общее | 117 | 16 | 21 | H1 (1), H2 (14), H3 (1), H4 (9), H5 (4), H6 (12), H7 (33), H8 (2), H9 (2), H10 (7), H11 (15), H12 (9), H13 (4), H14 (2), H15 (1), H16 (1) | 4.20336 ± 0.31863 | 0.86767 ± 0.01800 | 0.00759 ± 0.00025 |
Попарные оценки Fst, основанные на последовательностях COI мтДНК (табл. 2), в большинстве продемонстрировали отрицательные значения и были статистически незначимы (P > 0.05). Р-дистанции (табл. 2) имели низкие и близкие величины (от 0.00692 между Krs и Fed до 0.00836 между Pos15 и Pos18).
Таблица 2.
Значения Fst и p-дистанций для исследованных выборок Apostichopus japonicus
| Vos | Krs | Pos15 | Fed | Pos18 | |
|---|---|---|---|---|---|
| Vos | 0.00716 | 0.00802 | 0.00716 | 0.00788 | |
| Krs | –0.03007 | 0.00788 | 0.00692 | 0.00779 | |
| Pos15 | 0.00638 | 0.01670 | 0.00784 | 0.00836 | |
| Fed | –0.02081 | –0.02146 | 0.02090 | 0.00742 | |
| Pos18 | 0.00430 | 0.02276 | 0.01637 | –0.01642 |
Результаты оценки изменчивости мтДНК в выборках методом AMOVA показали (табл. 3), что значительное разнообразие гаплотипов наблюдалось внутри выборок (99.93%), а не между ними (0.07%), т.е. различий между группами особей практически не наблюдалось. Индекс фиксации F = 0.00070 был статистически незначим, оценки между и внутри групп были также незначимыми (P > 0.05).
Таблица 3.
Результат анализа молекулярной дисперсии (AMOVA) пяти выборок Apostichopus japonicus на основе фрагмента гена COI мтДНК
| Дисперсия | Степень свободы | Сумма квадратов | Компоненты вариансы | Процент изменчивости |
|---|---|---|---|---|
| Между выборками | 4 | 8.539 | 0.00147 | 0.07 |
| Внутри выборок | 112 | 235.256 | 2.10050 | 99.93 |
| Общее | 116 | 243.795 | 2.10197 |
Тесты на нейтральность (табл. 4), основанные на последовательностях COI мтДНК, показали, что значения D и FS согласованы между собой. Средние для всех выборок показатели D и FS были отрицательными. Однако ни одно из представленных значений не было статистически значимым (P > 0.05).
Таблица 4.
Результаты тестов на нейтральность Таджимы (D) и Фу (FS) для выборок Apostichopus japonicus, включая соответствующие P-значения
| Выборка | D | P | FS | P |
|---|---|---|---|---|
| Vos | –0.12838 | 0.46430 | –1.05419 | 0.34590 |
| Krs | 0.24374 | 0.62930 | 1.76809 | 0.81930 |
| Pos15 | 0.05617 | 0.54990 | 0.31968 | 0.60640 |
| Fed | 0.53424 | 0.73670 | 0.88963 | 0.69770 |
| Pos18 | –0.37936 | 0.37180 | –1.78021 | 0.21530 |
| Общее | 0.06528 | 0.55040 | 0.02860 | 0.53692 |
С помощью метода максимального правдоподобия была получена дендрограмма гаплотипов на основе фрагмента гена COI мтДНК (рис. 2). На основе топологии ветвей можно выделить две основные клады: кластер 1 (гаплотипы H1, H2, H3, H5, H6, H10, H12) и кластер 2 (гаплотипы H4, H7, H8, H9, H11, H13, H14, H15, H16).
Рис. 2.
Дендрограмма для гаплотипов Apostichopus japonicus из зал. Петра Великого, реконструированная с помощью метода максимального правдоподобия по данным секвенирования 554 п.н. фрагмента гена COI мтДНК. Цифры в узлах – значение бутстрэп-поддержки (1000 повторов) более 40%.

В сети гаплотипов, основанной на MJ-алгоритме (рис. 3), найдено не более трех нуклеотидных замен между гаплотипами, при этом 3 гаплотипа, являющиеся промежуточным звеном между другими, не были выявлены. Часто встречавшиеся гаплотипы находились преимущественно на периферии сетки. Исходя из данных дендрограммы, также формировались 2 кластера (кластер 1 – гаплотипы H1, H2, H3, H5, H6, H10, H12 и кластер 2 – гаплотипы H4, H7, H8, H9, H11, H13, H14, H15, H16), соответствовавшие ML-дереву. Как видно из рис. 3, гаплотипы или кластеры гаплотипов не ассоциированы с той или иной локальностью.
Рис. 3.
Медианная сеть гаплотипов на основе последовательностей COI Apostichopus japonicus. На ветвях указаны мутационные различия между гаплотипами; размер окружностей пропорционален абсолютным частотам гаплотипов. Все мутации имеют равный вес, интервал поиска медианных векторов ε равен нулю.

Межгодовые вариации в выборках дальневосточного трепанга
Были проанализированы две выборки из зал. Посьета, отобранные в разные годы (в 2015 и 2018 гг.). Два гаплотипа H13 и H14 оказались общими и присутствовали только в этой локальности (табл. 1). В выборке Pos18 выявлено 11 гаплотипов, на 2 больше, чем в 2015 г. При этом самого распространенного для других групп гаплотипа (H7) за 3 года стало больше. Всего A. japonicus, собранного в этом районе за 3 года, сохранилось 7 общих гаплотипов. Несмотря на то, что гаплотипический состав различался (табл. 1), статистически значимых различий (показатель Fst, табл. 2) не выявлено.
Исторические демографические вариации использовали при изучении частотных распределений попарных различий между последовательностями (рис. 4). В настоящем исследовании график распределения несоответствия для всех популяций показал мультимодальную и неровную форму. Мы также рассчитали индекс неоднородности (r) и сумму квадратов отклонений (SSD) в соответствии с моделью демографического расширения для каждой выборки (табл. 5) и обнаружили, что все они были незначимы (P < 0.05).
Рис. 4.
Распределение частот числа попарных нуклеотидных различий (несоответствий) между гаплотипами COI мтДНК для каждой выборки. По оси абсцисс – количество парных различий, по оси ординат – частота парных сравнений. Наблюдаемые частоты представлены синей линией. Частота, ожидаемая в рамках гипотезы модели расширения популяции, изображена оранжевой линией.

Таблица 5.
SSD (сумма квадратов отклонения) и индекс неоднородности (r) для выборок Apostichopus japonicus, включая соответствующие P-значения
| Выборка | SSD | P | r | P |
|---|---|---|---|---|
| Vos | 0.06095 | 0.00990 | 0.14884 | 0.00960 |
| Krs | 0.12822 | 0.00470 | 0.31155 | 0.00510 |
| Pos15 | 0.05227 | 0.00670 | 0.13182 | 0.00620 |
| Fed | 0.10554 | 0.00360 | 0.26025 | 0.01220 |
| Pos18 | 0.06314 | 0.04420 | 0.15714 | 0.03100 |
ОБСУЖДЕНИЕ
Общее число гаплотипов у дальневосточного трепанга Apostichopus japonicus, собранного в зал. Петра Великого, составило 16. При этом наименьшее число гаплотипов наблюдалось в выборках Krs и Fed (6 и 7 соответственно). Эти величины свидетельствуют об относительно низком генетическом разнообразии данного вида в исследованной акватории по сравнению с A. japonicus из префектуры Иватэ (Япония); был обнаружен 81 гаплотип (Adachi et al., 2018). Среди потенциально значимых причин снижения генетического разнообразия в данных локальностях мы рассматриваем изменение прибрежной зоны (Nehemia, Kochzius, 2017) и развитие прибрежных районов (Soliman et al., 2016), а также незаконный промысел. Наше исследование частично подтверждает это наблюдение, так как выборки Krs и Fed были собраны в местах с хорошо развитыми береговыми линиями.
Исследование фрагмента гена COI мтДНК A. japonicus показало высокое гаплотипическое разнообразие (от 0.78421 ± 0.06400 в Krs до 0.90043 ± 0.04100 в Pos18) и низкое нуклеотидное разнообразие (от 0.00693 ± 0.00054 для Fed до 0.00826 ± 0.00057 для Pos15). Сходные данные получены при изучении мтДНК у этого вида дальневосточного трепанга с побережья Японии (Adachi et al., 2018), а также у таких представителей голотурий, как Holothuria arguinensis (Rodrigues et al., 2015), H. edulis (Soliman et al., 2016) и H. atra (Hamamoto et al., 2021). Число полиморфных сайтов (S) в нашем исследовании было ниже, чем в выборке с побережья Японии (Adachi et al., 2018). В целом низкие значения разнообразия нуклеотидов и высокие значения гаплотипического разнообразия указывают на большое количество близкородственных гаплотипов. Возможно, что они претерпевают интенсивные стохастические процессы в короткие временные интервалы, или же в этом участвуют и селективные процессы.
Результаты AMOVA и попарного сравнения Fst не выявили существенных генетических различий между выборками из разных локальностей зал. Петра Великого. Значения Fst и индекса фиксации были статистически незначимы (P > 0.05). Для других видов голотурий получен сходный результат, например, не обнаружено гетерогенности между популяциями Holothuria nobilis (см.: Uthicke, Benzie, 2003). Анализ молекулярной дисперсии не выявил существенных различий между субпопуляциями Cucumaria frondosa в западной части Атлантики (So et al., 2011), это позволило предположить, что она составляет единую панмиктическую популяцию. Однако в некоторых исследованиях генетические дистанции были значительно выше: для H. atra с центральных и южных островов Рюкю AMOVA выявил высокий уровень дисперсии среди групп (Hamamoto et al., 2021). При попарном сравнении популяций обнаружена высокая степень генетической структурированности H. edulis, обитающей у о-ва Окинава (Soliman et al., 2016). Возможные причины панмиксии или, наоборот, структурированности внутри видов могут быть связаны с длительностью личиночного периода у разных видов. Виды с короткой планктонной стадией характеризуются большей подразделенностью и наличием структуры по сравнению с видами, у которых период свободноплавающей личинки длится дольше (So et al., 2011). Пелагический период у A. japonicus длится около двух недель (Левин, 2000; Qiu et al., 2015), что может быть причиной низкого уровня дифференциации между выборками. Различия в истории расселения, генетическом составе новых особей и отборе в период после оседания – это факторы, которые могут вызывать генетическую неоднородность (Watts et al., 1990; Hedgecock, Pudovkin, 2011; Karl et al., 2012). Как правило, морские беспозвоночные даже с низкой подвижностью на взрослой стадии могут характеризоваться высоким потоком генов, если способность к расселению высока на планктонной стадии (Palumbi, Wilson, 1990).
Полученная сеть гаплотипов в нашем случае характеризуется некоторыми особенностями. Типично, когда в сетке у большинства наземных и морских видов центральный гаплотип является наиболее часто встречающимся, от него “звездообразно” отходят менее часто встречающиеся гаплотипы (Avise, 2000). Этот аспект часто используется для филогеографических реконструкций происхождения и расселения видов и таксонов, а центральные гаплотипы рассматриваются как предковые (Avise, 2000). В нашем случае часто встречающиеся гаплотипы, как правило, находятся на периферии сетки, а центральными являются редко встречающиеся или гипотетические, т.е. не обнаруженные нами. Поскольку предковые гаплотипы отсутствуют, так же как и длинные ветви (максимальное число мутаций между гаплотипами не более трех), это может свидетельствовать о частом обновлении производителей в популяциях дальневосточного трепанга в исследованном регионе. Данный факт подтверждается также мультимодальностью распределения различий между гаплотипами (рис. 4). Анализ распределения несоответствий для всех выборок продемонстрировал мультимодальное распределение, что свидетельствует о демографическом равновесии или стабильной популяции (Ray et al., 2003). В то же время мультимодальное (включая бимодальное) распределение несоответствия указывает на периодические уменьшение размера популяции (Marjoram, Donnelly, 1994; Rogers, 1995) и даже на возможную изоляцию популяций в прошлом (Harpending, 1994).
Состояние популяций дальневосточного трепанга в разных частях зал. Петра Великого неодинаковое. Это связано со значительным выловом вида в недавнем прошлом и с браконьерством, что привело к снижению эффективного размера популяции. Во всех районах расселение молоди дальневосточного трепанга неравномерное, оно зависит от гидрологического режима, типа грунта, наличия полей анфельции или зарослей зостеры (Левин, 2000). Напомним, что на личинок A. japonicus влияют такие факторы среды, как температура, соленость воды и концентрация взвешенного осадка. Эти факторы могут как благоприятствовать (в определенной степени) их росту и расселению, так и противодействовать.
ЗАКЛЮЧЕНИЕ
С использованием фрагмента гена COI мтДНК было оценено генетическое разнообразие в выборках трепанга Apostichopus japonicus в зал. Петра Великого Японского моря. Мы не обнаружили дифференциации между выборками, что может быть связано с наличием свободноплавающей (до двух недель) личинки в жизненном цикле.
Интересными представляются филогенетические отношения между гаплотипами, которые не были обнаружены ранее в популяциях других видов. Это явление, очевидно, может быть обусловлено несколькими причинами. Вероятны стохастические факторы, влияющие на некоторую селективность по отношению к гаплотипам. Можно предположить, что в определенном регионе оседают личинки от немногих производителей и вследствие снижения численности данная вероятность увеличивается во времени. На это могут накладываться факторы отбора. Один из таких факторов – эпизоотии дальневосточного трепанга, наблюдавшиеся в 2005–2010 гг. (Терехова, Белькова, 2016). Эпизоотии могут приводить к быстрым сменам пула взрослых производителей и вариациям гаплотипического набора. Немаловажным селективным фактором может быть температура воды в период размножения. Косвенные доказательства наличия отбора, основанные на клинальной изменчивости нейтральных аллельных вариантов в некоторых микросателлитных локусах, приведены нами ранее (Yagodina et al., 2022). Еще одним фактором остается нелегальный вылов дальневосточного трепанга в зал. Петра Великого. Показано, что численность A. japonicus в этом регионе, начиная с конца 1990-х годов, снизилась более чем в 20 раз (Лысенко и др., 2018). Очевидно, что селективное изъятие из популяций крупных половозрелых особей может приводить к быстрой смене пула производителей и вариациям гаплотипического состава у потомков.
Список литературы
Левин В.С. Дальневосточный трепанг: биология, промысел, воспроизводство. СПб.: Голанд. 2000. 200 с.
Лысенко В.Н., Жариков В.В., Лебедев А.М. Современное состояние поселений дальневосточного трепанга Apostichopus japonicus (Selenka, 1867) в Дальневосточном морском заповеднике // Биол. моря. 2018. Т. 44. № 2. С. 134–140.
Селин Н.И. Вертикальное распределение дальневосточного трепанга Apostichopus japonicus в заливе Восток Японского моря // Биол. моря. 2001. Т. 27. № 4. С. 297–299.
Терехова В.Е., Белькова Н.Л. Идентификация оппортунистических патогенов трепанга (Apostichopus japonicus), культивируемого в Приморском крае // Вода: химия и экология. 2016. № 1. С. 36–42.
Adachi K., Okumura S., Moriyama S. Genetic structure of Japanese sea cucumbers (Apostichopus japonicus) along the Sanriku coast supports the effect of earthquakes and related tsunamis // Genetica. 2018. V. 146. № 6. P. 497–503. https://doi.org/10.1007/s10709-018-0041-z
Avise J.C. Phylogeography: The History and Formation of Species. Harvard: Harvard Univ. Press. 2000. 464 p.
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Chang Y., Feng Z., Yu J., Ding J. Genetic variability analysis in five populations of the sea cucumber Stichopus (Apostichopus) japonicus from China, Russia, South Korea and Japan as revealed by microsatellite markers // Mar. Ecol. 2009. V. 30. P. 455–461. https://doi.org/10.1111/j.1439-0485.2009.00292.x
Chen L., Yang J. Microsatellite genetic variation in wild and hatchery populations of the sea cucumber (Apostichopus japonicus Selenka) from northern China // Aqua. Res. 2008. V. 39. P. 1541–1549. https://doi.org/10.1111/j.1365-2109.2008.02027.x
Dong Y., Li Q., Zhong X., Kong L. Development of gene-derived SNP markers and their application for the assessment of genetic diversity in wild and cultured populations in sea cucumber, Apostichopus japonicus // J. World Aqua. Soc. 2016. V. 47. № 6. P. 873–888. https://doi.org/10.1007/s12686-013-9858-z
Du H., Bao Z., Yan J. et al. Development of 101 gene-based single nucleotide polymorphism markers in sea cucumber, Apostichopus japonicus // Int. J. Mol. Sci. 2012. V. 13. P. 7080–7097. https://doi.org/10.3390/ijms13067080
Edgar R.C. MUSCLE: a multiple sequence alignment method with reduced time and space complexity // BMC Bioinform. 2004. V. 5. P. 113. https://doi.org/10.1186/1471-2105-5-113
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Excoffier L., Smouse P., Quattro J. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data // Genetics. 1992. V. 131. P. 479–491. https://doi.org/10.1093/genetics/131.2.479
Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap // Evolution. 1985. V. 39. P. 783–791. https://doi.org/10.1111/j.1558-5646.1985.tb004
Fu Y.X. Statistical test of neutrality of mutation against population growth, hitchhiking and background selection // Genetics. 1997. V. 147. № 2. P. 915–925. https://doi.org/10.1093/genetics/147.2.915
Hamamoto K., Soliman T., Poliseno A., Iria Fernandez-Silva I., Reimer J.D. Higher genetic diversity of the common sea cucumber Holothuria (Halodeima) atra in marine protected areas of the Central and Southern Ryukyu Islands // Front. Conserv. Sci. 2021. V. 2. P. 736633. https://doi.org/10.3389/fcosc.2021.736633
Harpending R.C. Signature of ancient population growth in a low-resolution mitochondrial DNA mismatch distribution // Hum. Biol. 1994. V. 66. P. 591–600.
Hedgecock D., Pudovkin A.I. Sweepstakes reproductive success in highly fecund marine fish and shellfish: a review and commentary // Bull. Mar. Sci. 2011. V. 87. № 4. P. 971–1002. https://doi.org/10.5343/bms.2010.1051
Hoareau T.B., Boissin E. Design of phylum-specific hybrid primers for DNA barcoding: addressing the need for efficient COI amplification in the Echinodermata // Mol. Ecol. Res. 2010. V. 10. P. 960–967. https://doi.org/10.1111/j.1755-0998.2010.02848.x
Kanno M., Li Q., Kijima A. Microsatellite analysis of Japanese sea cucumber, Stichopus (Apostichopus) japonicus, supports reproductive isolation in color variants // Mar. Biotech. 2006. V. 8. P. 672–685. https://doi.org/10.1007/s10126-006-6014-8
Karl S.A., Toonen R.J., Grant W.S., Bowen B.W. Common misconceptions in molecular ecology: echoes of the modern synthesis // Mol. Ecol. 2012. V. 21. P. 4171–4189. https://doi.org/10.1111/j.1365-294X.2012.05576.x
Kim M., Choi T., An H.S. Population genetic structure of sea cucumber, Stichopus japonicus in Korea using microsa-tellite markers // Aqua. Res. 2008. V. 39. P. 1038–1045. https://doi.org/10.1111/j.1365-2109.2008.01962.x
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. 1980. V. 16. № 2. P. 111–120. https://doi.org/10.1007/BF01731581
Marjoram P., Donnelly P. Pairwise comparisons of mitochondrial DNA sequences in subdivided populations and implications for early human evolution // Gene-tics. 1994. V. 136. P. 673–683. https://doi.org/10.1093/genetics/136.2.673
Nehemia A., Kochzius M. Reduced genetic diversity and alteration of gene flow in a fiddler crab due to mangrove degradation // PLoS One. 2017. V. 12. P.8. https://doi.org/10.1371/journal.pone.0182987
Oh G.-W., Ko S.-C., Lee D.H. et al. Biological activities and biomedical potential of sea cucumber (Stichopus japo-nicus): a review // Fish. Aqua. Sci. 2017. V. 20. https://doi.org/10.1186/s41240-017-0071-y
Palumbi S.R., Wilson A.C. Mitochondrial DNA diversity in the sea urchins Strongylocentrotus purpuratus and S. droebachiensis // Evolution. 1990. V. 44. P. 403–415. https://doi.org/10.1111/j.1558-5646.1990.tb05208.x
Purcell S.W., Samyn Y., Conand C. Commercially important sea cucumbers of the world // FAO Species Catalogue for Fishery Purposes № 6 / Eds. N. De Angelis, A. Lovatelli. 2012. P. 223.
Qiu T., Zhang T., Hamel J.-F., Mercier A. Development, settlement, and post-settlement growth // The Sea Cucumber Apostichopus japonicus History, Biology and Aquaculture / Eds. H. Yang, J.-F. Hamel, A. Mercier. Academic Press. 2015. P. 111–131. https://doi.org/10.1016/B978-0-12-799953-1.00008-8
Ray N., Currat M., Excoffier L. Intra-deme molecular diversity in spatially expanding populations // Mol. Biol. Evol. 2003. V. 20. P. 76–86. https://doi.org/10.1093/molbev/msg009
Rodrigues F., Valente S., González-Wanguemert M. Genetic diversity across geographical scales in marine coastal ecosystems: Holothuria arguinensis a model species // J. Exp. Mar. Biol. Ecol. 2015. V. 463. P. 158–167. https://doi.org/10.1016/j.jembe.2014.12.006
Rogers A.R. Genetic evidence for a Pleistocene population expansion // Evolution. 1995. V. 49. P. 608–615. https://doi.org/10.1111/j.1558-5646.1995.tb02297.x
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. DnaSP 6: DNA sequence polymorphism analysis of large datasets // Mol. Biol. Evol. 2017. V. 34. № 12. P. 3299–3302.https://doi.org/10.1093/molbe v/msx248
Schneider S., Excoffier L. Estimation of past demographic parameters from the distribution of pairwise differences when the mutation rates vary among sites: application to human mitochondrial DNA // Genetics. 1999. V. 152. P. 1079–1089. https://doi.org/10.1093/genetics/152.3.1079
Selenka E. Beiträge zur Anatomie und Systematik der Holothurien // Zoology. 1867. V. 17. P. 291–374.
Simões T.D., Azevedo E., Silva F.H. et al. Ecological traits of sea cucumbers with commercial relevance from the north-eastern Atlantic coast // Front. Mar. Sci. Conference Abstract: IMMR'18. 2019. https://doi.org/10.3389/conf.FMARS.2018.06.00147
So J., Uthicke S., Hamel J.-F., Mercier A. Genetic population structure in a commercial marine invertebrate with long-lived lecithotrophic larvae: Cucumaria frondosa (Echinodermata: Holothuroidea) // Mar. Biol. 2011. V. 158. P. 859–870. https://doi.org/10.1007/s00227-010-1613-3
Soliman T., Kanno M., Kijima A., Yamazaki Y. Population genetic structure and gene flow in the Japanese sea cucumber Apostichopus japonicus across Toyama Bay, Japan // Fish. Sci. 2012. V. 78. P. 775–783. https://doi.org/10.1007/s12562-012-0509-1
Soliman T., Fernandez-Silva I., Reimer J.D. Genetic population structure and low genetic diversity in the over-exploited sea cucumber Holothuria edulis Lesson, 1830 (Echinodermata: Holothuroidea) in Okinawa Island // Conserv. Genetics. 2016. V. 17. P. 811–821. https://doi.org/10.1007/s10592-016-0823-8
Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism // Genetics. 1989. V. 123. P. 585–595. https://doi.org/10.1093/genetics/123.3.585
Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis version 11 // Mol. Biol. Evol. 2021. V. 38. P. 3022–3027. https://doi.org/10.1093/molbev/msab120
Truett G.E. Preparation of Genomic DNA from Animal Tissues // DNA Sequencing II: Optimizing Preparation and Cleanup / Ed. J. Kieleczawa. Sudbury: Jones and Bartlett Publishers. 2006. P. 33–46.
Tyler P.A., Young C.M.D., Billett S.M., Giles L.A. Pairing behaviour, reproduction and diet in the deep-sea holothurian genus Paroriza (Holothurioidea: Synallactidae) // J. Mar. Biol. Assoc. U. K. 1992. V. 72.2. P. 447–462. https://doi.org/10.1017/S0025315400037814
Uthicke S., Benzie J.A.H. Gene flow and population history in high dispersal marine invertebrates: Mitochondrial DNA analysis of Holothuria nobilis (Echinodermata: Holothuroidea) populations from the Indo-pacific // Mol. Ecol. 2003. V. 12. P. 2635–2648. https://doi.org/10.1046/j.1365-294X.2003.01954.x
Valente S., Serrão E.A., González-Wangüemert M. West versus East Mediterranean Sea: origin and genetic diffe-rentiation of the sea cucumber Holothuria polii // Mar. Ecol. 2014. V. 36. № 3. P. 485–495. https://doi.org/10.1111/maec.12156
Watts R.J., Johnson M.S., Black R. Effects of recruitment on genetic patchiness in the urchin Echinometra mathaei in Western Australia // Mar. Biol. 1990. V. 105. P. 145–151.
Yagodina V.D., Bondar E.I., Brykov V.A. Genetic variability and population structure of the Japanese sea cucumber, Apostichopus japonicus Selenka, 1867 revealed by microsatellites in Peter the Great Gulf, Sea of Japan // Mar. Biodivers. 2022. V. 52. P. 40. https://doi.org/10.1007/s12526-022-01278-0
Yan J., Jing J., Mu X. et al. A genetic linkage map of the sea cucumber (Apostichopus japonicus) based on microsa-tellites and SNPs // Aquaculture. 2013. V. 404–405. P. 1–7. https://doi.org/10.1016/j.aquaculture.2013.04.011
Дополнительные материалы отсутствуют.